水产养殖环境复杂多变,生活在其中的生物因长期受到细菌、病原体、寄生虫及重金属和微塑料等环境胁迫,严重影响生存生长和繁育等功能,可能导致其大量死亡,直接影响水产养殖业的产量、经济收益及生态保护[1]。尽管水产动物的先天免疫系统(innate immune system)能够抵御外界环境胁迫及侵染[2],但面对长期环境胁迫时,仅从先天免疫系统角度难以全面解释水产动物的表型变化及对环境适应的表型差异。大量研究表明,表观遗传修饰研究主要集中在水产动物性腺发育、遗传育种及免疫调控机制等方面。据报道,环境胁迫诱导生物发生DNA甲基化,并通过调控抗性基因表达激活生物体免疫防御,增强生物对环境胁迫的适应能力[3]。本文将围绕环境胁迫如何通过DNA甲基化调控抗性基因表达,以及改变生物的生理生态效应以适应环境胁迫进行综述,以期通过揭示DNA甲基化在抗逆反应中的修饰作用机制,为水产动物品种选育和生态保护提供有益参考。
1 表观遗传修饰概述
表观遗传修饰能够在不改变DNA序列的情况下调控基因表达,并改变生物表型[4]。表观遗传修饰包括DNA甲基化、组蛋白修饰和非编码RNA调控[5]。在水产动物中,环境胁迫诱导DNA甲基化发生并调控基因表达,使生物的生理活动及表型发生变化以适应环境[6]。从表观遗传角度解析DNA甲基化调控的抗逆基因表达机制,可为分子标记辅助育种和抗逆品种选育提供靶点,助力水产养殖业可持续发展。
1.1 DNA甲基化
环境胁迫下,DNA甲基化通过甲基转移酶(DNA methyltransferase,DNMTs)催化S-腺苷甲硫氨酸(SAM)向胞嘧啶(cytosine,C)第5位碳原子(C5位)添加甲基(—CH3)形成5-甲基胞嘧啶(5mC),同时SAM向S-腺苷同型半胱氨酸(SAH)转化,呈现动态调控基因表达模式(图1),起到抑制基因启动子区和转录起始位点的作用[7]。DNA甲基化通常发生在胞嘧啶“C”-磷酸“p”-鸟嘌呤“G”(CpG)二核苷酸上,也有甲基化胞嘧啶发生在非CpG位点,包括CHG和CHH位点(其中H = A、C或T)[8]。脊椎动物DNA甲基化水平通常为70%~80%,呈现出“全局”模式,即不同基因组元件均发生DNA甲基化,CpG含量较高的片段(CpG岛)(CpG islands)除外[9],如斑马鱼(Danio rerio)[10]、小鼠(Mus musculus)[11]等脊椎动物。无脊椎动物的DNA甲基化模式分为“昆虫型”,几乎没有甲基化;“棘皮动物型”,其基因组同时包含甲基化和非甲基化部分[12]。其中昆虫的DNA甲基化水平相对较低[13],海鞘(Didemnum lahillei)[14]属于“棘皮动物型”甲基化模式。栉孔扇贝(Chlamys farreri)有14.9%~16.5%的CpG位点发生DNA甲基化[15]。长牡蛎(Crassostrea gigas)基因组甲基化水平约2%,参与调控牡蛎的生长发育和环境适应过程[16]。不同基因组元件DNA甲基化发挥不同调控基因表达功能,其中,基因启动子CpG岛甲基化程度较低时,不影响转录因子与DNA的结合,从而促进基因表达,但启动子CpG岛甲基化程度较高时,阻遏转录因子与DNA的结合,或5 mC结合蛋白(MBP)招募蛋白复合物使转录沉默,抑制基因表达[17](图2)。
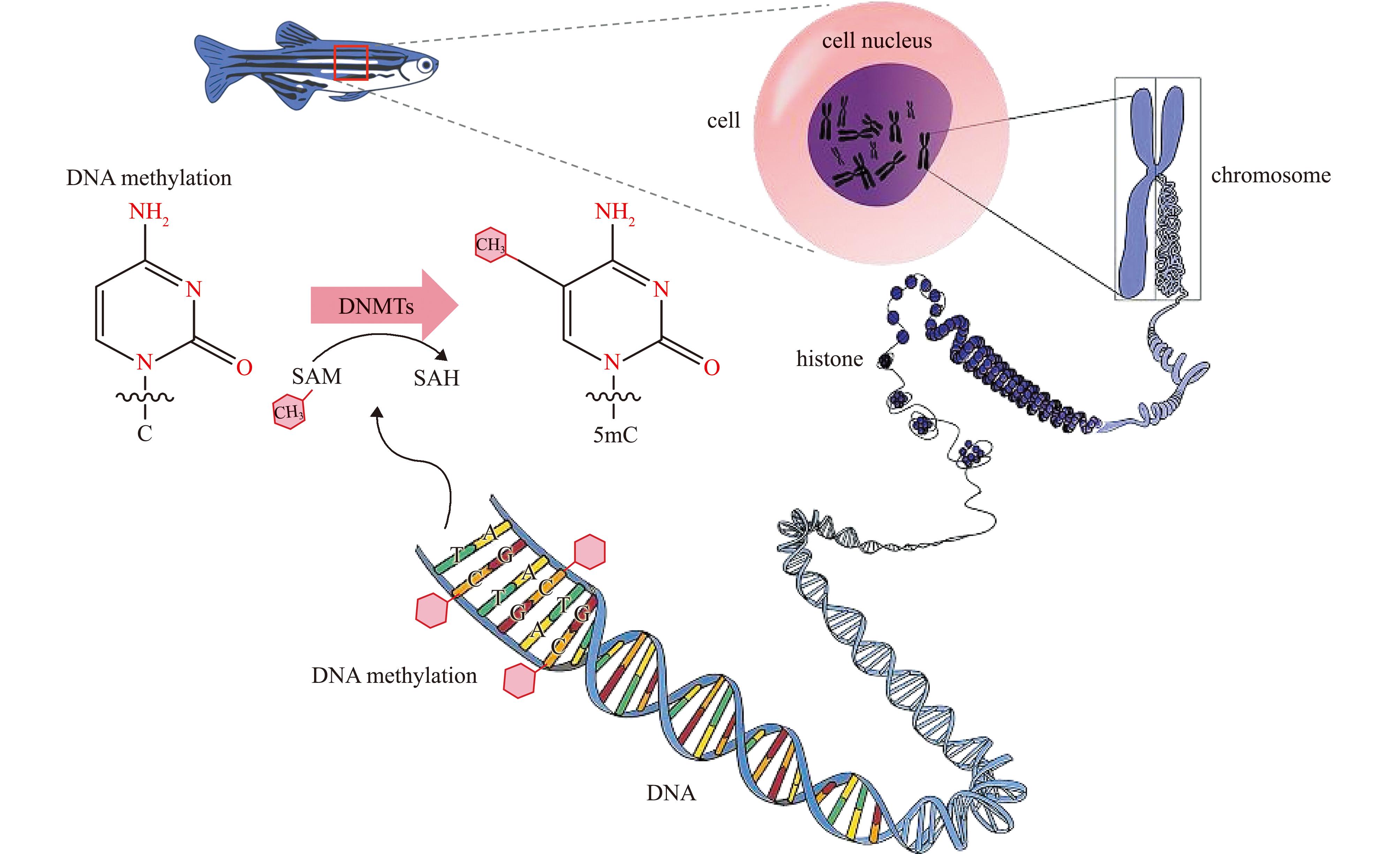
图1 DNA甲基化修饰
Fig.1 DNA methylation modification
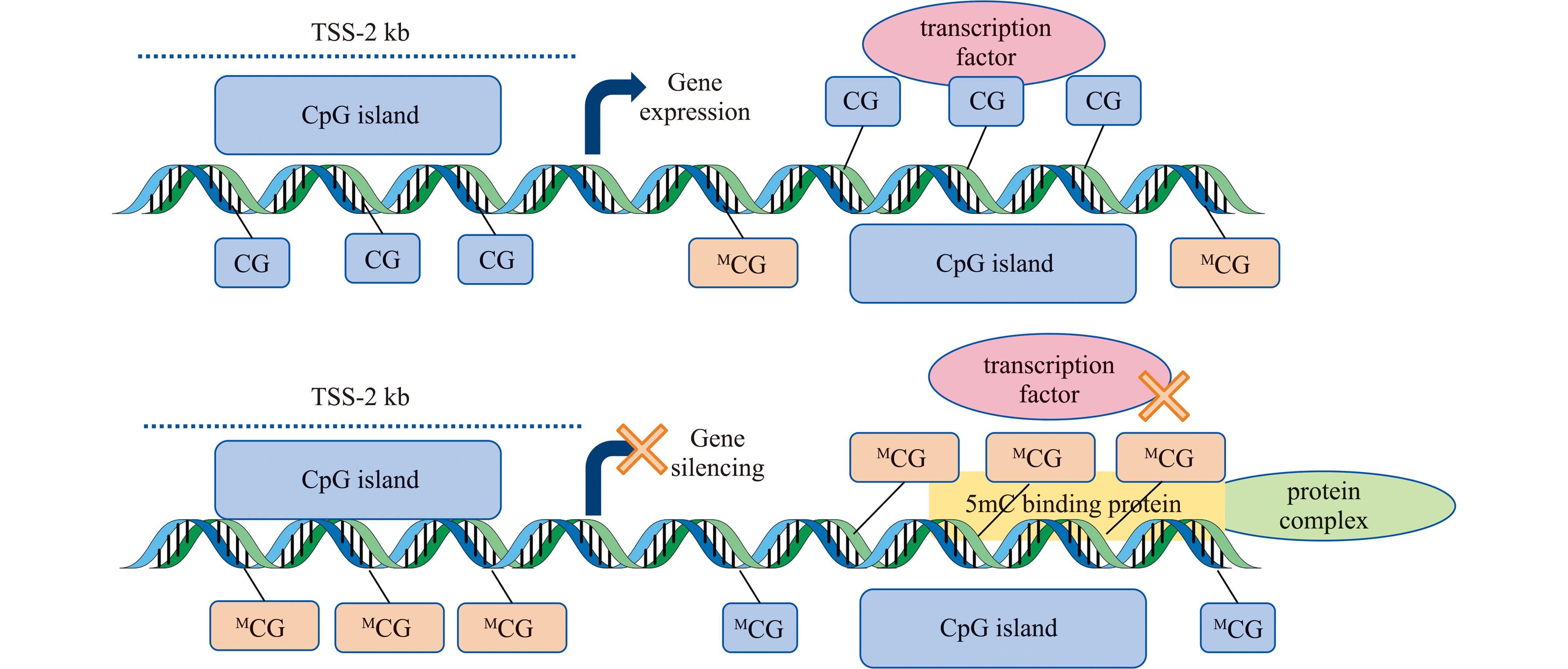
图2 启动子区域DNA甲基化调控基因表达
Fig.2 DNA methylation in promoter region regulates gene expression
1.2 DNA甲基化调控酶
DNA甲基转移酶(DNMT)负责建立和维持DNA甲基化模式[18]。DNA甲基化分为两种方式,分别为从头甲基化和维持(半保留)甲基化[19]。不同类型的甲基化由不同种类的甲基转移酶行使功能,DNA甲基转移酶包括Dnmt1、Dnmt2、Dnmt3a/3b和DnmtL,但只有Dnmt1和Dnmt3a/3b有甲基转移活性。两种甲基化方式分别由 Dnmt3和 Dnmt1催化完成。Dnmt1在UHRF1介导下维持半甲基化CpG位点的甲基化,Dnmt3在早期发育建立甲基化模式,或在应对急性环境胁迫时发生重头甲基化,Dnmt3a/3b的从头甲基化有利于维持DNA甲基化模式,可能是经过Dnmt1缺失的CpG位点来维持[20](图3)。DNA去甲基化的过程包括在半保留复制后使新DNA链不发生甲基化,使用地西他滨抑制剂或者通过不依赖于复制的过程主动去甲基化[21]。DNA去甲基化酶是被一组名为10~11易位家族酶(TET)的双加氧酶,包括TET1、TET2和TET3,双加氧酶结构域能将DNA中5-甲基胞嘧啶(5mC)的甲基氧化为5-羟甲基胞嘧啶(5hmC)、进一步氧化为5-甲酰胞嘧啶(5fC)和5-羧基胞嘧啶(5caC),随后进行复制依赖性稀释和胸腺嘧啶DNA糖基化酶(TDG)依赖性碱基切除来完成去甲基化[22](图4)。DNMT1和DNMT3建立的DNA甲基化修饰会被甲基CpG结合域蛋白(MBD)识别[23]。MBD家族的大多数成员专门靶向甲基化胞嘧啶,并参与DNA甲基化和去甲基化过程。据报道,DNMTs与TETs之间相互作用共同调控水产动物的表观遗传修饰,是生物适应环境变化及调控基因表达的关键作用机制[24]。DNMTs与TETs间的动态平衡与环境胁迫密切相关,当水产动物受到环境胁迫时,DNMT3b过表达导致免疫基因高甲基化水平抑制基因表达,TETs则通过去甲基化激活基因表达,增强生物响应环境胁迫的抗性反应[25]。因此,DNA甲基化调控酶DNMTs和TETs在水产动物抗逆过程中具有维持生物体甲基化稳态及调控基因表达的功能和协同作用。
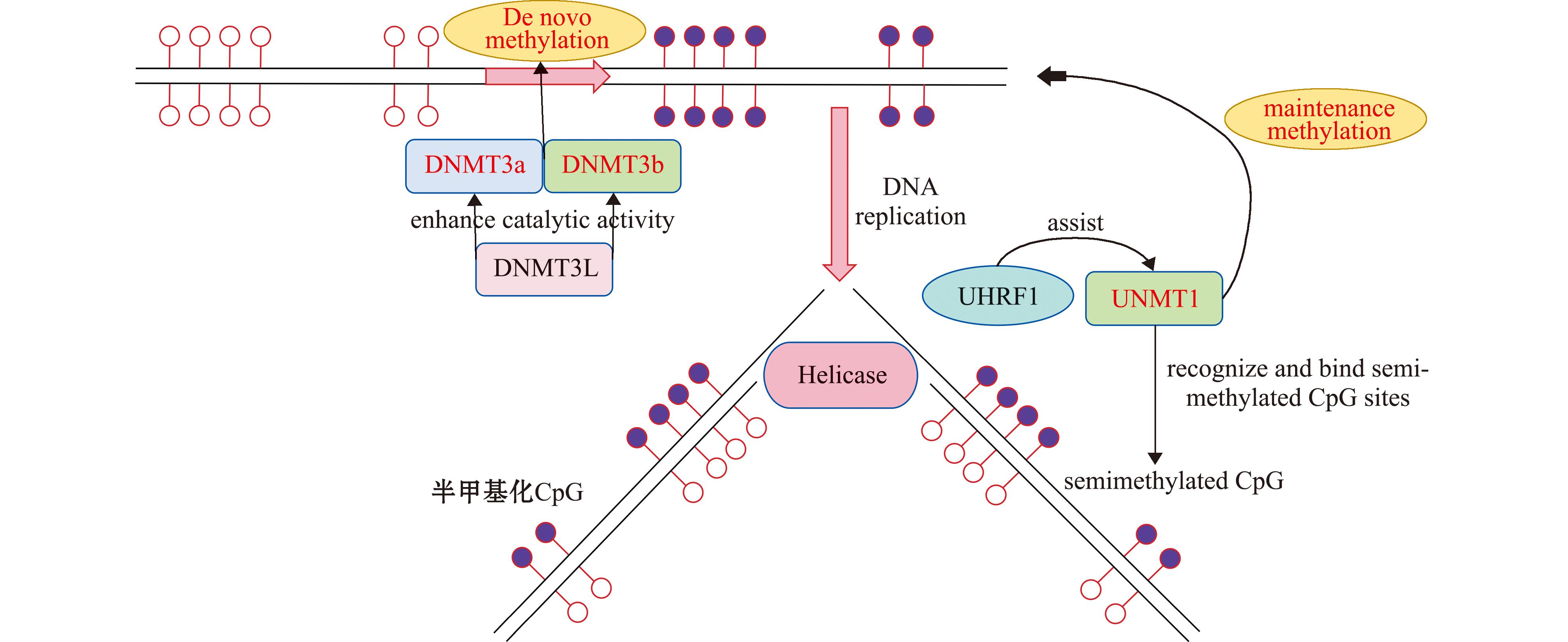
图3 DNA甲基转移酶作用机制
Fig.3 Mechanism of DNA methyltransferase
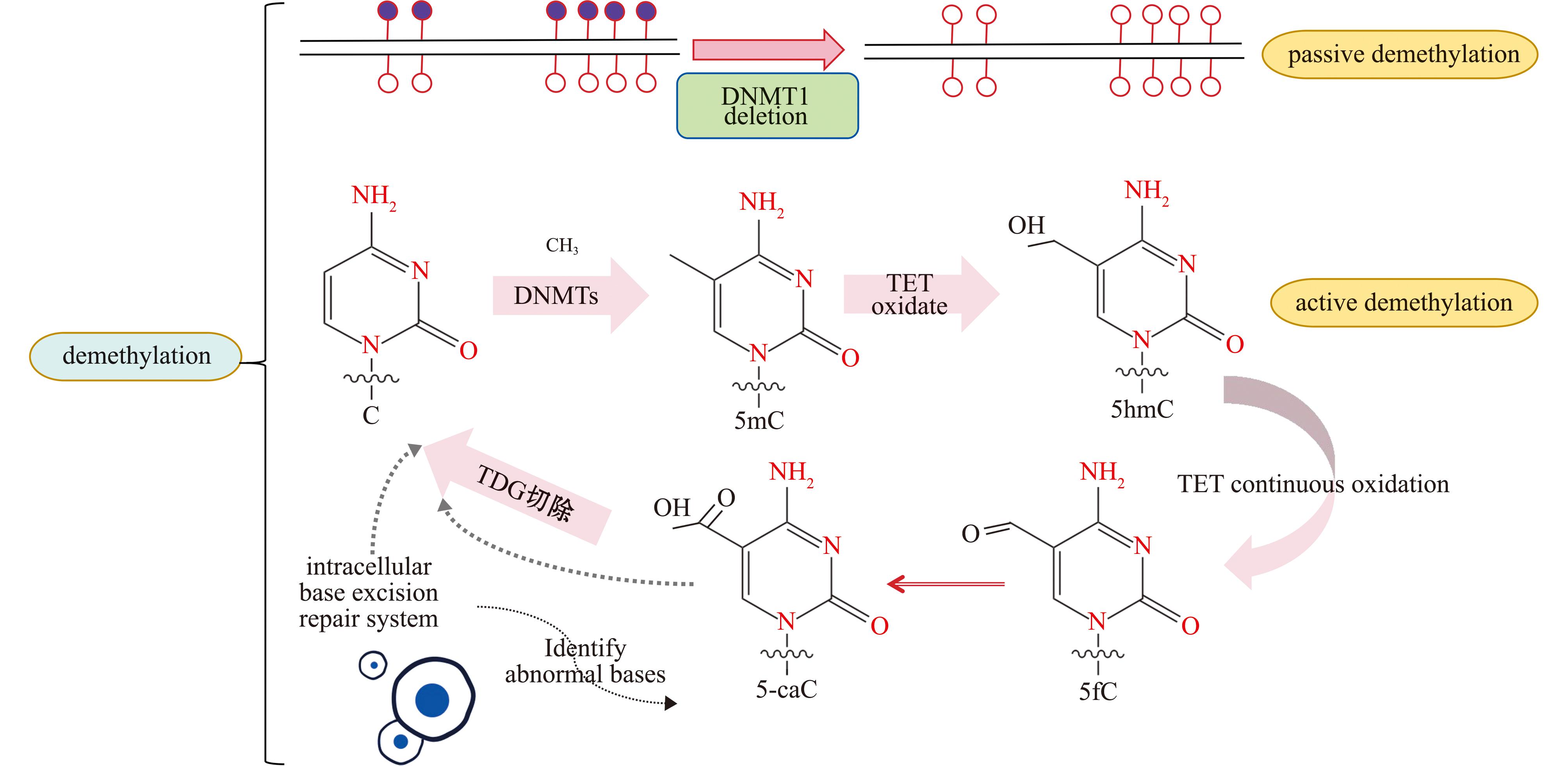
图4 去甲基化酶作用机制
Fig.4 Mechanism of demethylase action
2 环境胁迫诱导DNA甲基化调控水产动物抗性或免疫反应
自然水体中常见的环境胁迫因子包括非生物因子(温度、盐度、低氧、污染物)和生物因子(病毒、细菌和寄生虫),这些因子对水产动物的生长发育、生理代谢、抗氧化能力、耐受性和适应性及抗病免疫功能均有较大影响[26]。环境胁迫诱导水产动物发生DNA甲基化的同时,甲基化通过调控基因转录在细胞分化、胚胎发育、环境适应和免疫防御中发挥重要作用[7]。一般来说,DNA甲基化调控基因转录机制有两种,首先是DNA甲基化会阻碍基因与其他调控因子的结合,从而影响基因的转录表达;其次是DNA甲基化后能募集其他蛋白或组蛋白修饰相关酶,导致染色质失去活性[27]。在水产动物中,探究环境胁迫通过DNA甲基化调控基因表达,改变生物的生理功能及表型对生物适应环境具有重要意义。
2.1 非生物因子
2.1.1 温度 极端温度变化对水产动物的生理功能、新陈代谢和抗性免疫具有较大影响,热应激是极端温度变化中致使水产动物产量降低的环境胁迫因子之一,DNA甲基化的发生可以帮助水产动物快速适应环境胁迫。已有研究表明,DNA甲基化通过调控热应激和抗氧化功能相关基因表达,改变水产动物应对环境变化的适应性和表型可塑性。如大西洋鲑(Salmo salar)幼鱼进行缺氧和升温处理后[28],热休克蛋白(HSP70)基因启动子区域发生甲基化,调控其转录活性升高以维持细胞稳态。无脊椎动物中,刺参(Apostichopus japonicus)的HSP70基因受热应激诱导的DNA甲基化负调控表达,整体甲基化水平显著增加[29]。Yang等[30]同样发现,DNA甲基化可能通过影响免疫和热休克蛋白关键基因转录调控参与刺参响应高温胁迫。中间棘海胆(Strongylocentrotus intermedius)的DNA甲基化水平在热应激下显著升高,其氧化磷酸化、凋亡和代谢相关基因(MOAP-1,GGT1、HSP70和RDH8)受DNA甲基化调控表达下调,从而抑制海胆响应热应激免疫反应[31]。长牡蛎DNMT1、DNMT3A迅速响应热应激刺激,但通过DNA甲基化抑制剂(5-Aza)处理后其甲基化可塑性降低,导致热应激相关基因表达降低,使长牡蛎耐热性产生负面影响[32]。此外,Roberto等[33]通过长牡蛎对热应激的耐受性区分了耐热性和热敏性两组家系,发现耐热性家系具有高甲基化区域,差异甲基化基因(DMGs)数量远超于热敏性,这些与DNA甲基化过程相关的基因通过离子相互作用参与受甲基化调控的热应激反应,热应激诱导的DNA甲基化在无脊椎动物中出现了跨代表观遗传修饰。卤虫(Artemia franciscana)后代即使未经环境胁迫也可以产生抗性表型,并调控HSP90、HSP70基因表达,推测DNA甲基化在无脊椎动物跨代表观遗传修饰中具有不可逆的作用[34]。总的来说,热应激上调DNMTs表达以促使DNA甲基化发生,而DNA甲基化能够调控HSP70等热应激相关基因表达,进而维持生物体细胞稳态,防止细胞损伤,改变水产动物表型可塑性,帮助其适应热应激环境,并在跨代表观遗传中起着重要作用。低温变化引起的冷应激同样对生物的生理代谢和免疫功能有严重影响,通过DNA甲基化修饰调控冷应激期间的基因表达有助于生物响应环境胁迫的适应性及耐受性。低温(18 ℃和10 ℃)处理后凡纳滨对虾(Litopenaeus vannamei)的甲基化主要发生在基因体区域(外显子和内含子),差异甲基化区域相关基因(DMRGs)显著富集在氧化磷酸化和mTOR 信号通路,推测甲基化调控相关抗性基因表达以响应低温胁迫[35]。斑马鱼胚胎在短期冷应激后全基因组DNA甲基化水平升高,特别是在基因组的非编码区域,其中参与抗氧化、凋亡、发育、染色质修饰及免疫过程的相关基因受甲基化显著调控,这些生物过程通过调控DNA甲基化应对冷应激[36]。
总而言之,极端温度变化是水产动物面临的重要环境胁迫因子,热应激和冷应激通过诱导DNA甲基化调控机制,影响生物细胞稳态和氧化应激等生理功能,调控热休克蛋白相关基因表达,帮助生物在应对环境胁迫的耐受性、适应性及跨代遗传中做出表观遗传响应,可为提高水产动物特别是经济物种的抗性能力及抗性品种选育提供参考。
2.1.2 盐度 水体环境中盐度胁迫通过破坏渗透压稳态、诱导氧化应激、影响免疫指标和代谢等生理过程,限制水产动物的生存、生长和抗性反应。已有研究表明,高盐或低盐胁迫在影响水产动物生理表型的同时,还通过DNA甲基化调控水产动物关键基因表达参与免疫过程。如盐度15胁迫下,半滑舌鳎(Cynoglossus semilaevis)肝脏组织严重受损,与细胞生长分化及抑制凋亡功能相关的基因IGF1外显子甲基化负调控其表达水平[37]。大黄鱼(Larimichthys crocea)在低盐(5)胁迫下的甲基化水平显著高于高盐度组(25),DNA甲基化参与其鳃组织和肾脏组织的渗透压调节、能量代谢[38]等生物过程。通过富集分析发现,大黄鱼DMGs在细胞凋亡、NF-kB和PI3K-Akt等免疫通路中显著富集[39]。此外,生物体DNA甲基化水平与盐度胁迫下甲基转移酶的表达、不同甲基化基因组元件及组织特异性密切相关。大黄鱼Dnmt1和Dnmt3a2在肝脏、肠道和大脑组织中显著表达[39]。尼罗罗非鱼(Oreochromis niloticus)的 DNA 甲基化水平在高盐胁迫后略有升高,LTB4R与ACOT4基因同属脂酰辅酶 A 硫酯酶家族,且均参与脂质代谢过程,LTB4R基因因启动子区域高甲基化而表达下调,ACOT4基因则因内含子区域高甲基化导致表达水平下调,推测不同基因组元件甲基化调控基因机制并非绝对,内含子区域的高甲基化可能通过抑制增强子功能,导致基因表达下调[40]。盐度变化显著影响欧洲鲈(Dicentrarchus labrax)鳃组织DNA甲基化水平,甲基转移酶DNMT3a的表达受到抑制,基因表达水平与启动子、第一内含子和第一外显子中的 DNA 甲基化水平呈负调控关系[41]。三疣梭子蟹(Portunus trituberculatus)鳃组织中的DNA甲基化水平受低盐胁迫影响而降低,在不同组织中,肌肉和鳃中的甲基化水平降低,但肝胰腺中的甲基化水平增加,3种组织的甲基化水平分别为47.31%、17.69%和22.94%,说明DNA 甲基化参与三疣梭子蟹响应低盐胁迫反应[42]。
因此,高盐和低盐胁迫动态调节DNA甲基化水平变化、甲基转移酶和抗性免疫基因表达,启动子、内含子及第一内含子和第一外显子等不同基因组元件对基因表达具有不同调控作用,这些调控机制使水产动物能响应环境胁迫并最终适应环境。探究DNA甲基化与盐度胁迫后基因表达模式间及甲基化水平变化与组织特异性的调控关系,有助于阐明表观遗传调控在生物适应环境胁迫中的调控作用,辅助选育耐盐品种分子标记,提升养殖动物响应盐度胁迫的抗逆能力。
2.1.3 溶解氧 氧气是自然环境中最基本的元素,当溶解氧(DO)浓度低于2.8 mg/L时,水体出现低氧或缺氧状态,而缺氧会使水产动物产生大量活性氧(ROS),并通过表观遗传调控机制在水产动物抗氧化应激及免疫反应中发挥作用。低氧胁迫下日本牙鲆(Paralichthys olivaceus)激活HIF-1ɑ、Nrf2 和 STAT3等转录因子来应对急性缺氧胁迫,免疫基因STAT3和VEGFA启动子区域DNA甲基化负调控基因表达。进一步研究发现,日本牙鲆DNA甲基化通过调控HIF-1/VEGFA或HIF-1/LDH-A调控轴来参与牙鲆适应缺氧环境的代谢和免疫相关生物学过程[43]。Liu等[44]还发现,急性缺氧胁迫会激活牙鲆骨骼肌细胞免疫反应,早期生长反应基因2(Egr2)通过结合FasL启动子位点上调其表达,甲基化通过负调控FasL基因参与Egr2-FasL-Fas信号通路调节免疫反应,诱导细胞凋亡,维持牙鲆免疫稳态。急性缺氧诱导DNA甲基化正调控虹鳟(Oncorhynchus mykiss)胚胎中参与线粒体介导细胞凋亡和自噬功能相关的基因以促进细胞凋亡活性,参与虹鳟应激反应[45]。刺参受到高温、低氧和高温低氧联合处理后,3组CRY1a和CRY1b启动子中CpG岛区域甲基化水平上调,调控CRY1a和CRY1b基因表达降低,促进下游炎症因子的表达,激活水产动物的免疫防御系统[46]。缺氧还损伤水产动物的发育繁殖功能,通过表观遗传机制将这种损伤跨代遗传,影响子代水产动物的繁殖能力,并对物种种群的长期稳定和生态系统的稳态造成巨大影响。此外,DNA甲基转移酶和去甲基化酶在水产动物响应缺氧胁迫时发挥重要调控作用。如缺氧胁迫下,大黄鱼DNA去甲基化酶基因(tet1、 tet2和tet3)及甲基转移酶基因(dnmt1和dnmt3bb)的表达显著上调,氧化应激相关基因HIF1A、HIF3A、CAMK2B和MAP2K2受到启动子甲基化水平负调控基因表达,揭示了表观遗传调控机制在大黄鱼响应缺氧胁迫中的重要作用[47]。
总的来说,低氧胁迫会对生物组织造成损伤,破坏其正常免疫代谢功能,甚至导致死亡。水产动物通过表观遗传修饰调控抗氧化基因表达,影响生理代谢和免疫抗性维持体内稳态。研究低氧胁迫对生物免疫的影响及表观遗传修饰参与的抗性反应,对于水产养殖业的可持续发展具有重要意义。
2.1.4 污染物 海洋环境中污染物的潜在来源包括工业排放、石油泄漏和重金属污染等。有机污染物通过食物链进行生物累积,对海洋生物甚至人类健康造成极大危害。长期暴露于有机污染物会导致水产动物出现性逆转、代谢紊乱、生殖能力下降、性成熟时间延长、后代存活率低、神经发育受损和免疫抑制等问题,影响DNA甲基化水平,改变表观遗传调控[48]。三丁基锡(TBT)和三苯基锡(TPT)及其混合物诱导褐菖鲉(Sebastiscus marmoratus)肝脏DNA甲基化水平降低,推测低甲基化水平是甲基(—CH3)转移反应受到干扰所致[49]。17α-乙炔基雌二醇(EE2)处理使斑马鱼编码卵黄蛋白原I(Vitellogenin I,VtgI)基因DNA甲基化水平降低,肝脏中甲基化水平变化相较于脑中更为明显,雌性斑马鱼VtgI表达显著高于雄性,推测污染物诱导DNA甲基化参与生物性别分化且具有组织特异性[50]。多环芳烃(PAHs)是一类沉积在海底的有机污染物,苯并[a]芘(B[a]P)作为一种典型的PAHs,对海洋生物具有致癌、致畸、致突变性等影响。雌性栉孔扇贝在苯并[a]芘低浓度组(0.4 μg/L)甲基化水平上升,中高浓度组(2、10 μg/L)DNA甲基化水平下降,与性激素合成代谢相关基因hsd17β受启动子甲基化调控表达下调,推测DNA甲基化调控机制在贝类性别分化及繁殖中具有重要作用[51]。苯并[a]芘胁迫后泥蚶(Tegillarca granosa)抗氧化酶活性显著升高,神经毒性生物标志物相关酶活性被干扰抑制,DNA 甲基化水平显著降低,推测B[a]P代谢物会损害DNMTs的活性,导致5-甲基胞嘧啶含量下降,甲基化水平受到抑制可增加抗氧化基因的表达水平,从而提高泥蚶抗氧化活性以应对B[a]P毒性[52]。
重金属是常见的环境污染物之一,其通过水体进入海洋生态系统中,可对水生生物造成极大危害。周新文等[53]研究发现,重金属离子混合物能提高鱼类肝脏的DNA甲基化总水平,推测该混合物刺激机体产生大量活性氧,导致DNA损伤,进而影响DNMTs与DNA胞嘧啶碱基的结合作用及甲基化水平。研究表明,不同浓度铜离子对长牡蛎胚胎的毒性作用可能会通过改变DNA甲基化水平来影响胚胎发育相关基因(Hoxa1)的表达[54]。另有研究发现,不同浓度镉离子长期胁迫(45 d和90 d)会抑制尼罗罗非鱼幼鱼的生长和发育,并激活抗氧化系统发生氧化应激反应,胁迫后幼鱼DNA甲基化水平显著降低,DNMT3a和DNMT3b表达下调,去甲基化酶TET1和TET2表达上调,DNA甲基化变化及甲基化调控酶间的相互作用与抗氧化防御系统共同揭示了生物体应对污染物胁迫的毒性作用机制[55]。此外,亚砷酸盐胁迫导致金鱼肝脏中DNA甲基化水平降低,且通过谷胱甘肽和抗氧化酶的适应性变化来应对污染物引起的氧化应激反应[56]。
因此,通过研究污染物对水产动物的生理功能及抗性免疫的影响,以及表观遗传修饰在机体中的调控作用,对于评估环境污染物对海洋生物生存和适应的影响及表观遗传调控机制的阐明具有重要意义。
2.2 生物因子
2.2.1 病毒 DNA甲基化作为关键的表观遗传修饰之一,通过不同基因组元件甲基化水平调控免疫基因表达,在生物抗病毒免疫应答中发挥着重要作用。如大西洋鲑头肾组织受鲑立克次体败血症(salmonid rickettsial septicemia,SRS)侵染后,与吞噬相关的模式受体基因启动子和第一个外显子区域中的甲基化强烈抑制基因表达[57]。大量关于草鱼(Ctenopharyngodon idellus)的研究表明,草鱼抗病毒基因黑色素瘤分化相关基因5(MDA5)和维甲酸诱导基因I(RIG-I)表达受到草鱼呼肠孤病毒(grass carp reovirus,GCRV)诱导的DNA甲基化负调控,其中易感组RIGI-I甲基化水平显著高于抗性组,表达水平受甲基化负调控而降低,草鱼MDA5的第一个外显子包含了更多的候选CpG位点,这些位点的高甲基化可负调控易感组MDA5的表达,同时调控下游基因MAVS的表达[58]。GCRV感染后,草鱼全基因组DNA甲基化水平和DNMTs、TETs及甘氨酸N-甲基转移酶(GNMTs)的表达水平均升高,启动子区域低甲基化负调控补体基因C3和KNG1表达[59]。草鱼肾细胞中易感细胞的全基因组DNA甲基化、mRNA和microRNA表达水平高于对照细胞和抗性细胞,内含子区域甲基化负调控差异表达免疫相关基因,同时LGP2、NLRC5、SOCS3B和TBK1结合蛋白分别被 miR-34-y、miR-722-y和 new-m0076-5p负调控,以上结果较好地从表观遗传学的角度揭示了水产动物抗病毒调节机制,为今后的抗性育种研究提供了潜在的生物标志物[60]。
在硬骨鱼中,与病毒防御相关基因MDA5和TRIM的DNA甲基化已被证明负调控MDA5的表达[61]。传染性脾肾坏死病毒(Infectious spleen and kidney necrosis,ISKNV)侵染后,鳜(Siniperca chuatsi)的DNA甲基化水平为23.44%,这种高甲基化基因组增强了ISKNV在鳜体内的增殖能力,5-氮杂胞苷处理后的低甲基化使ISKNV激活了细胞强烈的免疫反应,并降低体内的病毒致病性[62]。虹鳟单核细胞(RTS11)在感染传染性胰脏坏死病毒(infectious pancreatic necrosis virus,IPNV)后,甲基化调控酶(DNMT1)和组蛋白乙酰转移酶(HAT、HDA)显著高表达,干扰素基因IFN1、IFNγ启动子发生表观遗传变化,推测IPNV感染后水产动物发生了表观遗传重编程,并通过启动子甲基化和去甲基化,以及与启动子相关的组蛋白甲基化共同在病毒诱导的免疫应答和抗病毒过程中起作用[63]。对虾可通过改变表观遗传标记,激活抗病毒免疫相关基因,进而调控免疫通路的基因表达,使其能迅速应对病毒的侵染并进行有效防御[64]。
由此可知,水产动物在受到病毒侵染后,可通过调控DNMTs的表达,诱导抗病毒等免疫相关基因发生DNA甲基化,并靶向修饰其特定甲基化区域及位点,或与组蛋白修饰协同作用以调控基因表达等方式抵御病毒侵染。
2.2.2 细菌 水体环境中细菌的存在严重影响着水产动物的生理、生长、发育及免疫抗性。其中,部分有益细菌能够与水产动物共生,有助于机体抵抗病原体入侵和维持肠道菌群平衡。然而,部分致病菌的侵染会引发各种细菌性疾病,可导致水产动物组织损伤,并引起大规模的细菌性疾病感染及经济损失[65]。此外,病原体感染可引发DNA甲基化变化,从而调控生物体基因表达升高或下降,这些变化在激活下游免疫转录因子及生物体免疫调节机制中发挥重要作用[66]。据报道,弧菌胁迫导致半滑舌鳎促炎细胞因子白细胞介素-17C(IL-17C)的表达受DNA甲基化调控而降低,蛋白酶激活受体3(PAR3)表达呈上调趋势,诱导IL8表达[67],哈维氏弧菌(Vibrio harveyi)胁迫后半滑舌鳎抗性家系全基因组DNA甲基化水平高于易感家系,肿瘤坏死因子(TNF-ɑ)和脂多糖结合蛋白样基因的(LBP)DNA甲基化水平负调控基因表达,帮助抗性家系在应对弧菌胁迫时发挥免疫应答作用[68]。DNA甲基化通过调控免疫相关基因过敏毒素趋化受体1样(C5ar1-Like)和嗜酸性粒细胞过氧化物酶样(EPX-Like)在大菱鲆响应杀鲑气单胞菌(Aeromonas salmonicida)免疫机制中发挥作用[69]。鳗弧菌(Vibrio anguillarum)胁迫下大菱鲆(Scophthalmus maximus)与细胞凋亡和免疫功能相关的Rho GTP酶基因(Rho)表达受启动子低甲基化负调控[70],牙鲆肿瘤坏死因子α(TNFα)基因表达与DNA甲基化呈负相关关系,并激活Nuclear factor kappa-B(NF-κB)转录因子发挥免疫功能[71]。灿烂弧菌(Vibrio splendidus)胁迫导致刺参体壁组织DNA甲基化水平升高,且负调控LRR、HSP20、CARD等抗氧化、免疫和凋亡基因表达,此外使用5-氮杂胞苷处理后,牡蛎甲基化水平降低,DNMT3和白细胞介素-17(IL-17)表达均显著下降[72]。副溶血弧菌(Vibrio parahaemolyticus)胁迫下三疣梭子蟹TETs与Dnmt3B表达呈现负相关关系,推测甲基转移酶和去甲基化酶基因在弧菌胁迫中调控DNA甲基化的动态平衡,从而影响生物体的免疫应答和胁迫适应机制[73]。类似地,尼罗罗非鱼[74]、红鳍东方鲀(Takifugu rubripes)[75]及长牡蛎[76]在分别应对无乳链球菌(Streptococcus agalactiae)、哈维氏弧菌和溶藻弧菌(Vibrio alginolyticus)感染时,DNA 甲基化亦可通过调控免疫基因的表达来激活其免疫防御系统。
因此,细菌胁迫在引起水产动物先天免疫系统防御和炎症反应的同时,还通过影响甲基转移酶和去甲基化酶动态平衡、改变DNA甲基化模式及调控抗性免疫基因表达,帮助水产动物增强应对细菌侵染的适应性和生存能力,对抗性品系选育和保护具有重要意义。
2.2.3 寄生虫 寄生虫是影响生物种群遗传多样性、经济物种健康、产量和经济效益的环境胁迫生物因子之一。大部分表观遗传修饰在细胞分化和发育过程中起决定作用,但寄生虫侵染可能会诱导机体成熟的免疫细胞发生DNA 甲基化变化,从而改变转录因子对基因的调控作用。鱼类体内代谢、发育和细胞凋亡相关基因在抵御寄生虫侵染过程中高度甲基化,基因表达被抑制,推测寄生虫介导DNA甲基化调控机制抑制水产动物免疫反应[77]。刺激隐核虫(Cryptocaryon irritans)侵染下,红鳍东方鲀关键免疫基因肿瘤蛋白p53(TP53)和有丝分裂激活蛋白激酶2α(MAP2K2A)受到基因启动子甲基化负调控基因表达,有助于揭示红鳍东方鲀应对寄生虫侵染的免疫机制,筛选的候选基因可作为基于甲基化的最佳生物标志[78]。在大西洋鲑抵御海虱侵染[79]、美国牡蛎(Crassostrea virginica)感染帕金虫(Perkinsus marinus)[80]的研究中同样阐述了DNA甲基化启动子区域负调控基因表达帮助生物体响应环境胁迫的机制。目前,DNA甲基化在寄生虫与脊椎动物宿主间相互作用的研究已有报道,但在无脊椎动物,特别是双壳类软体动物中的调节机制还有待研究。
因此,寄生虫的排泄物或分泌产物影响生物体的发育和免疫功能。表观遗传修饰机制有助于生物体对环境变化作出反应,其中DNA甲基化变化在宿主与寄生虫侵染的相互作用中发挥重要功能。通过研究寄生虫分泌物干扰宿主细胞的甲基转移酶活性,DNA甲基化调控免疫基因持续低表达,有助于了解水产动物与寄生虫相互作用中表观遗传调控机制的重要作用。
综上所述,环境胁迫包括生物和非生物因子,其通过激活并大量产生ROS来破坏水产动物细胞稳态,导致DNA损伤,促进DNA甲基化调控酶活性升高,相应基因表达上调。DNA甲基转移酶(DNMTs)将甲基(—CH3)转移至胞嘧啶发生DNA甲基化,此时机体先天免疫防御系统启动,激活抗氧化、代谢、抗性和免疫相关基因表达。DNA甲基化则进一步修饰基因的不同基因元件来调控基因表达。推测启动子高DNA甲基化水平导致基因沉默,低甲基化水平促进基因表达,而基因体甲基化调控作用更为复杂,通常与基因表达呈正相关关系,有助于防止非正常转录物的产生。最终帮助水产动物产生表型可塑性,在生存、解毒、耐寒耐热、渗透压调节和炎症反应中发挥作用,维持机体细胞和免疫稳态(图5)。

图5 环境胁迫诱导DNA甲基化调控基因表达示意图
Fig.5 Schematic diagram of DNA methylation regulation gene expression induced by environmental stress
3 结论
本研究中围绕近年来环境生物因子(病毒、细菌、寄生虫)和非生物因子(温度、盐度、低氧、污染物)如何通过DNA甲基化来动态调控水产动物基因表达,以及参与氧化应激、抗性和免疫等功能以维持机体稳态等研究进行了综述。首先,环境胁迫首先通过激活水产动物氧化应激反应,引发DNA损伤、影响甲基转移酶表达,进而促使DNA甲基化发生和影响甲基化模式;其次,环境胁迫通过启动子、内含子及外显子区域的高或低甲基化程度来调控热休克蛋白、氧化应激、细胞凋亡和代谢免疫相关基因的表达,帮助水产动物产生表型可塑性以适应环境变化并增强抗病免疫功能;再者,环境胁迫诱导的DNA甲基化在跨代表观遗传修饰中发挥重要作用,帮助水产动物的子一代具有先天响应环境胁迫的抗性免疫反应;最后,类似寄生虫一类的胁迫因子会对水产动物施加选择压力,DNA甲基化模式的改变可以影响其种群遗传多样性。综上所述,环境胁迫通过表观遗传机制影响水产动物的基因表达,调控其生理和免疫反应,增强其适应性和抗逆性,这些表观遗传调控机制在水产动物抗性品种选育及生态环境评估中具有重要应用价值。
4 展望
水环境中胁迫因子的刺激会显著影响水产动物的生长发育、生理生化和抗性免疫功能,引起DNA甲基化发生。DNA甲基化是重要的表观遗传修饰之一,在氧化应激和抗性免疫等多个生物学过程及应对环境胁迫中发挥关键作用。尽管本研究中对水产动物响应环境胁迫的表观遗传修饰与基因表达的调控关系等进行了总结阐述,但关于揭示甲基化具体调控基因表达的机制还需深入研究。目前,研究人员通过全基因组亚硫酸盐甲基化测序(WGBS)、简化表示亚硫酸盐测序(RRBS)和甲基化DNA免疫沉淀测序(MethylCap-Seq)等多种甲基化测序技术在全基因组层面和单细胞水平揭示了生物体响应环境胁迫的表观遗传动态变化及修饰机制。近年来,已有部分研究将甲基化组学与其他高通量测序组学关联分析,包括但不限于基因组学、转录组学、蛋白质组学和代谢组学等。
未来,DNA甲基化将继续从多组学整合角度进行深入研究,全面揭示环境胁迫与表观遗传修饰间的调控网络机制,构建环境胁迫因子通过表观遗传响应调控代谢、免疫及抗性基因和通路调控轴。为进一步将相关研究应用到实际生产中,未来可以通过上述联合分析,识别并筛选出响应环境胁迫的关键甲基化位点作为甲基化分子标记,广泛应用在耐高温、耐低氧、耐盐碱、抗污染物等抗逆性状,抗病毒、抗弧菌、抗寄生虫等免疫抗病性状,以及生长性状等方面个体的筛选和品系选育,构建可实际应用的抗逆性状筛选技术,包括建立甲基化分子标记与生物表型间的调控模型和开发调控网络预测网站等,为抗逆性状筛选转化到实际种质改良产业中提供应用潜力。
[1] MORLEY N J.Interactive effects of infectious diseases and pollution in aquatic molluscs[J].Aquatic Toxicology,2010,96(1):27-36.
[2] AKIRA S,UEMATSU S,TAKEUCHI O.Pathogen recognition and innate immunity[J].Cell,2006,124(4):783-801.
[3] LIU Z J,ZHOU T,GAO D Y.Genetic and epigenetic regulation of growth,reproduction,disease resistance and stress responses in aquaculture[J].Frontiers in Genetics,2022,13:994471.
[4] GAVERY M R,ROBERTS S B.Epigenetic considerations in aquaculture[J].PeerJ,2017,5:e4147.
[5] ROY S,KUMAR V,BEHERA B K,et al.Epigenetics:perspectives and potential in aquaculture[M]//Advances in Fisheries Biotechnology.Singapore:Springer Nature Singapore,2021:133-150.
[6] 杨震飞,刘波,戈贤平,等.水产养殖环境胁迫对鱼类表观遗传的影响研究进展[J].大连海洋大学学报,2018,33(2):270-282.
YANG Z F,LIU B,GE X P,et al.Research progress of aquaculture environmental stress on epigenetic regulation in fish:a review[J].Journal of Dalian Ocean University,2018,33(2):270-282.(in Chinese)
[7] VALENTE A,VIEIRA L,SILVA M J,et al.The effect of nanomaterials on DNA methylation:a review[J].Nanomaterials,2023,13(12):1880.
[8] IBARRA C A,FENG X Q,SCHOFT V K,et al.Active DNA demethylation in plant companion cells reinforces transposon methylation in gametes[J].Science,2012,337(6100):1360-1364.
[9] HOFFMAN A R,HU J F.Directing DNA methylation to inhibit gene expression[J].Cellular and Molecular Neurobiology,2006,26(4/5/6):425-438.
[10] GOLL M G,HALPERN M E.DNA methylation in zebrafish[M]//Modifications of Nuclear DNA and its Regulatory Proteins.Amsterdam:Elsevier,2011:193-218.
[11] FENG S H,COKUS S J,ZHANG X Y,et al.Conservation and divergence of methylation patterning in plants and animals[J].Proceedings of the National Academy of Sciences of the United States of America,2010,107(19):8689-8694.
[12] GAVERY M R,ROBERTS S B.DNA methylation patterns provide insight into epigenetic regulation in the Pacific oyster (Crassostrea gigas)[J].BMC Genomics,2010,11:483.
[13] RICHARDS S,GIBBS R A,WEINSTOCK G M,et al.The genome of the model beetle and pest Tribolium castaneum[J].Nature,2008,452:949-955.
[14] SIMMEN M W,BIRD A.Sequence analysis of transposable elements in the sea squirt,Ciona intestinalis[J].Molecular Biology and Evolution,2000,17(11):1685-1694.
[15] SUN Y,HOU R,FU X T,et al.Genome-wide analysis of DNA methylation in five tissues of Zhikong scallop,Chlamys farreri[J].PLoS One,2014,9(1):e86232.
[16] RIVIERE G,WU G C,FELLOUS A,et al.DNA methylation is crucial for the early development in the Oyster C.gigas[J].Marine Biotechnology,2013,15(6):739-753.
[17] SMITH J,SEN S,WEEKS R J,et al.Promoter DNA hypermethylation and paradoxical gene activation[J].Trends in Cancer,2020,6(5):392-406.
[18] CHEN Z Y,ZHANG Y.Role of mammalian DNA methyltransferases in development[J].Annual Review of Biochemistry,2020,89:135-158.
[19] GOLL M G,BESTOR T H.Eukaryotic cytosine methyltransferases[J].Annual Review of Biochemistry,2005,74:481-514.
[20] RIGGS A D,XIONG Z G.Methylation and epigenetic fidelity[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(1):4-5.
[21] 姜洁明,刘鹰,刘奇,等.硬骨鱼类性别分化过程的表观遗传机制研究进展[J].水产科学,2023,42(4):726-734.
JIANG J M,LIU Y,LIU Q,et al.Advances on epigenetic mechanism of sex differentiation in teleost fish[J].Journal of Fisheries Sciences,2023,42(4):726-734.(in Chinese)
[22] WU H,ZHANG Y.Reversing DNA methylation:mechanisms,genomics,and biological functions[J].Cell,2014,156(1/2):45-68.
[23] DHASARATHY A,WADE P A.The MBD protein family-reading an epigenetic mark?[J].Mutation Research,2008,647(1/2):39-43.
[24] WANG X X,CONG R H,LI A,et al.Transgenerational effects of intertidal environment on physiological phenotypes and DNA methylation in Pacific oysters[J].Science of the Total Environment,2023,871:162112.
[25] ABDELNOUR S A,NAIEL M A E,BEN SAID M,et al.Environmental epigenetics:Exploring phenotypic plasticity and transgenerational adaptation in fish[J].Environmental Research,2024,252:118799.
[26] CHEN F,JIANG F F,MA J,et al.Intersecting planetary health:Exploring the impacts of environmental stressors on wildlife and human health[J].Ecotoxicology and Environmental Safety,2024,283:116848.
[27] CHOY M K,MOVASSAGH M,GOH H G,et al.Genome-wide conserved consensus transcription factor binding motifs are hyper-methylated[J].BMC Genomics,2010,11:519.
[28] BEEMELMANNS A,RIBAS L,ANASTASIADI D,et al.DNA methylation dynamics in Atlantic salmon (Salmo salar) challenged with high temperature and moderate hypoxia[J].Frontiers in Marine Science,2021,7:604878.
[29] CHANG M Y,GE J L,LIAO M J,et al.Genome-wide DNA methylation and transcription analysis reveal the potential epigenetic mechanism of heat stress response in the sea cucumber Apostichopus japonicus[J].Frontiers in Marine Science,2023,10:1136926.
[30] YANG Y,SUN Y N,ZHOU X Q,et al.Genome-wide analysis of DNA methylation reveals significant impact of two typical high temperature stress on biological functions in Apostichopus japonicus[J].Aquaculture Reports,2023,29:101522.
[31] LIU A Z,ZENG F S,WANG L,et al.High temperature influences DNA methylation and transcriptional profiles in sea urchins (Strongylocentrotus intermedius)[J].BMC Genomics,2023,24(1):491.
[32] WANG X X,CONG R H,LI A,et al.Experimental DNA demethylation reduces expression plasticity and thermal tolerance in Pacific oysters[J].Marine Biotechnology,2023,25(3):341-346.
[33] ROBERTO A E,ANA M I,STEVEN R B,et al.Differentially methylated gene regions between resistant and susceptible heat-phenotypes of the Pacific oyster Crassostrea gigas[J].Aquaculture,2021,543:736923.
[34] ROY S,KUMAR V,BOSSIER P,et al.Phloroglucinol treatment induces transgenerational epigenetic inherited resistance against Vibrio infections and thermal stress in a brine shrimp (Artemia franciscana) model[J].Frontiers in Immunology,2019,10:2745.
[35] LI Z B,WANG W J,YU X L,et al.Integrated analysis of DNA methylome and transcriptome revealing epigenetic regulation of CRIR1-promoted cold tolerance[J].BMC Plant Biology,2024,24(1):631.
[36] STRICHMAN-ALMASHANU L Z,LEE R S,ONYANGO P O,et al.A genome-wide screen for normally methylated human CpG islands that can identify novel imprinted genes[J].Genome Research,2002,12(4):543-554.
[37] LI S P,HE F,WEN H S,et al.Low salinity affects cellularity,DNA methylation,and mRNA expression of igf1 in the liver of half smooth tongue sole (Cynoglossus semilaevis)[J].Fish Physiology and Biochemistry,2017,43(6):1587-1602.
[38] YANG J,LIU M H,ZHOU T T,et al.Genome-wide methylome and transcriptome dynamics provide insights into epigenetic regulation of kidney functioning of large yellow croaker
[39] ZHANG Y,ZHU F,TENG J,et al.Effects of salinity stress on methylation of the liver genome and complement gene in large yellow croaker (Larimichthys crocea)[J].Fish &Shellfish Immunology,2022,129:207-220.
[40] HUANG D D,LIANG X Y,QIAO T F,et al.Genome-wide DNA methylation responses to salinity stress in skin of GIFT Tilapia as revealed by whole-genome bisulfite sequencing[J].Aquaculture,2025,596:741745.
[41] BLONDEAU-BIDET E,BANOUSSE G,L’HONORÉ T,et al.The role of salinity on genome-wide DNA methylation dynamics in European sea bass gills[J].Molecular Ecology,2023,32(18):5089-5109.
[42] LU S K,LI R H,GAO T L,et al.Analysis of DNA methylation level of Portunus trituberculatus subjected to low salinity with methylation-sensitive amplification polymorphism[J].Journal of Ocean University of China,2019,18(5):1158-1162.
[43] LIU B H,LI G L,LI X H,et al.The responsive mechanisms of DNA methylation and transcriptional regulation to acute hypoxia stress in HIF-1/VEGFA signal pathway of Japanese flounder (Paralichthys olivaceus)[J].Aquaculture,2024,578:740021.
[44] LIU B H,LI G L,YANG J,et al.The mechanism of immune related signal pathway Egr2-FasL-Fas in transcription regulation and methylated modification of Paralichthys olivaceus under acute hypoxia stress[J].Fish &Shellfish Immunology,2022,123:152-163.
[45] VERON V,MARANDEL L,LIU J W,et al.DNA methylation of the promoter region of bnip3 and bnip3l genes induced by metabolic programming[J].BMC Genomics,2018,19(1):677.
[46] WU J,ZHANG W W,LI C H.Heat and hypoxia exposure mediates circadian rhythms response via methylation modification in Apostichopus japonicas[J].Frontiers in Marine Science,2021,8:721465.
[47] GAO Z Y,ZHENG C Y,ZHANG Y B,et al.Genome-wide analysis of DNA methylation and gene expression in large yellow croaker (Larimichthys crocea) under hypoxic stress[J].Aquaculture,2025,595:741624.
[48] LISTER K N,LAMARE M D,BURRITT D J.Pollutant resilience in embryos of the Antarctic sea urchin Sterechinus neumayeri reflects maternal antioxidant status[J].Aquatic Toxicology,2015,161:61-72.
[49] WANG Y Q,WANG C G,ZHANG J L,et al.DNA hypomethylation induced by tributyltin,triphenyltin,and a mixture of these in Sebastiscus marmoratus liver[J].Aquatic Toxicology,2009,95(2):93-98.
[50] STRÖMQVIST M,TOOKE N,BRUNSTRÖM B.DNA methylation levels in the 5’ flanking region of the vitellogenin I gene in liver and brain of adult zebrafish (Danio rerio):Sex and tissue differences and effects of 17α-ethinylestradiol exposure[J].Aquatic Toxicology,2010,98(3):275-281.
[51] TANG J,SONG A M,PAN L Q,et al.Study of DNA methylation of hsd17β,er and reproductive endocrine disrupting effects in female Chlamys farreri under benzo [a] Pyrene stress[J].Environmental Pollution,2023,328:121667.
[52] GUO B Y,FENG D,XU Z T,et al.Acute benzo [a] Pyrene exposure induced oxidative stress,neurotoxicity and epigenetic change in blood clam Tegillarca granosa[J].Scientific Reports,2021,11:18744.
[53] 周新文,朱国念,MWALILINO J,等.Cu、Zn、Pb、Cd及其混合重金属离子对鲫鱼(Carassius auratus)DNA甲基化水平的影响[J].中国环境科学,2001,21(6):549-552.
ZHOU X W,ZHU G N,MWALILINO J,et al.Influence of Cu,Zn,Pb,Cd and their heavy metalion mixture on the DNA methylation level of the fish (Carassius auratus)[J].China Environmental Science,2001,21(6):549-552.(in Chinese)
[54] SUSSARELLU R,LEBRETON M,ROUXEL J,et al.Copper induces expression and methylation changes of early development genes in Crassostrea gigas embryos[J].Aquatic Toxicology,2018,196:70-78.
[55] HU F X,YIN L,DONG F L,et al.Effects of long-term cadmium exposure on growth,antioxidant defense and DNA methylation in juvenile Nile Tilapia (Oreochromis niloticus)[J].Aquatic Toxicology,2021,241:106014.
[56] BAGNYUKOVA T V,LUZHNA L I,POGRIBNY I P,et al.Oxidative stress and antioxidant defenses in goldfish liver in response to short-term exposure to arsenite[J].Environmental and Molecular Mutagenesis,2007,48(8):658-665.
[57] MUKIIBI R,PE ALOZA C,GUTIERREZ A,et al.The impact of Piscirickettsia salmonis infection on genome-wide DNA methylation profile in Atlantic Salmon[J].Genomics,2022,114(6):110503.
ALOZA C,GUTIERREZ A,et al.The impact of Piscirickettsia salmonis infection on genome-wide DNA methylation profile in Atlantic Salmon[J].Genomics,2022,114(6):110503.
[58] SHANG X Y,SU J G,WAN Q Y,et al.CpA/CpG methylation of CiMDA5 possesses tight association with the resistance against GCRV and negatively regulates mRNA expression in grass carp,Ctenopharyngodon idella[J].Developmental and Comparative Immunology,2015,48(1):86-94.
[59] XIONG L,HE L B,LUO L F,et al.Global and complement gene-specific DNA methylation in grass carp after grass carp reovirus (GCRV) infection[J].International Journal of Molecular Sciences,2018,19(4):1110.
[60] SHANG X Y,YANG C R,WAN Q Y,et al.The destiny of the resistance/susceptibility against GCRV is controlled by epigenetic mechanisms in CIK cells[J].Scientific Reports,2017,7(1):4551.
[61] CHANG M X.The negative regulation of retinoic acid-inducible gene I (RIG-I)-like receptors (RLRs) signaling pathway in fish[J].Developmental and Comparative Immunology,2021,119:104038.
[62] LIANG M C,PAN W Q,YOU Y L,et al.Hypermethylated genome of a fish vertebrate iridovirus ISKNV plays important roles in viral infection[J].Communications Biology,2024,7(1):237.
[63] MANR QUEZ R A,SANDOVAL M,LONCOMAN C,et al.Epigenetic reprogramming around IFN1 and IFNy2 promoters in rainbow trout cells inoculated with infectious pancreatic necrosis virus (IPNV)[J].Fish &Shellfish Immunology,2023,140:108947.
QUEZ R A,SANDOVAL M,LONCOMAN C,et al.Epigenetic reprogramming around IFN1 and IFNy2 promoters in rainbow trout cells inoculated with infectious pancreatic necrosis virus (IPNV)[J].Fish &Shellfish Immunology,2023,140:108947.
[64] WIKUMPRIYA G C,PRABHATHA M W S,LEE J Y,et al.Epigenetic modulations for prevention of infectious diseases in shrimp aquaculture[J].Genes,2023,14(9):1682.
[65] LIU T T,KANG L,XU J L,et al.PVBase:a MALDI-TOF MS database for fast identification and characterization of potentially pathogenic Vibrio species from multiple regions of China[J].Frontiers in Microbiology,2022,13:872825.
[66] QIN W H,SCICLUNA B P,VAN DER POLL T.The role of host cell DNA methylation in the immune response to bacterial infection[J].Frontiers in Immunology,2021,12:696280.
[67] MA X R,CHEN Q C,CHEN Z F,et al.Genome-wide DNA methylation mediates the resistance to vibriosis in Cynoglossus semilaevis[J].Fish &Shellfish Immunology,2023,142:109144.
[68] XIU Y J,SHAO C W,ZHU Y,et al.Differences in DNA methylation between disease-resistant and disease-susceptible Chinese tongue sole (Cynoglossus semilaevis) families[J].Frontiers in Genetics,2019,10:847.
[69] LI Y R,SU L,LIU X F,et al.Immunity of turbot induced by inactivated vaccine of Aeromonas salmonicida from the perspective of DNA methylation[J].Frontiers in Immunology,2023,14:1124322.
[70] ZHANG K,LIU Y X,LIU X M,et al.A functional polymorphism in the promoter of RhoB is associated with susceptibility to Vibrio anguillarum in turbot (Scophthalmus maximus)[J].Fish &Shellfish Immunology,2019,93:269-277.
[71] YANG J,LIU B H,LI X H,et al.Immune correlates of NF-κB and TNFα promoter DNA methylation in Japanese flounder (Paralichthys olivaceus) muscle and immune parameters change response to Vibrio anguillarum infection[J].Fish &Shellfish Immunology,2021,119:578-586.
[72] ZHAO Q,WANG W L,LI J X,et al.The DNA cytosine-5-methyltransferase 3 (DNMT3) involved in regulation of CgIL-17 expression in the immune response of oyster Crassostrea gigas[J].Developmental &Comparative Immunology,2021,123:104092.
[73] ZHOU X F,SUN D F,GUO J Y,et al.Insights into the DNA methylation of Portunus trituberculatus in response to Vibrio parahaemolyticus infection[J].Fish &Shellfish Immunology,2024,154:109983.
[74] HU Q M,AO Q W,TAN Y,et al.Genome-wide DNA methylation and RNA analysis reveal potential mechanism of resistance to Streptococcus agalactiae in GIFT strain of Nile Tilapia (Oreochromis niloticus)[J].Journal of Immunology,2020,204(12):3182-3190.
[75] 王晨诗,黄馨笛,崔晓玉,等.哈维氏弧菌感染对红鳍东方鲀IL-6基因DNA甲基化的影响[J].大连海洋大学学报,2022,37(2):221-226.
WANG C S,HUANG X D,CUI X Y,et al.Effects of pathogen Vibrio harveyi infection on DNA methylation of IL-6 gene in tiger puffer Takifugu rubripes[J].Journal of Dalian Ocean University,2022,37(2):221-226.(in Chinese)
[76] LI J N,HE Y M,YANG B,et al.Whole-genome DNA methylation profiling revealed epigenetic regulation of NF-κB signaling pathway involved in response to Vibrio alginolyticus infection in the Pacific oyster,Crassostrea gigas[J].Fish &Shellfish Immunology,2024,151:109705.
[77] SAGONAS K,MEYER B S,KAUFMANN J,et al.Experimental parasite infection causes genome-wide changes in DNA methylation[J].Molecular Biology and Evolution,2020,37(8):2287-2299.
[78] XIA Y Q,YANG Y,LIU Y Y,et al.DNA methylation analysis reveals potential mechanism in Takifugu rubripes against Cryptocaryon irritans infection[J].Marine Biotechnology,2024,26(2):288-305.
[79] VALENZUELA-MU OZ V,WANAMAKER
OZ V,WANAMAKER ![]() G,et al.Environmental influence on the Atlantic salmon transcriptome and methylome during sea lice infestations[J].Fish &Shellfish Immunology,2024,151:109692.
G,et al.Environmental influence on the Atlantic salmon transcriptome and methylome during sea lice infestations[J].Fish &Shellfish Immunology,2024,151:109692.
[80] FARIAS N D,DE OLIVEIRA N F P,DA SILVA P M.Perkinsus infection is associated with alterations in the level of global DNA methylation of gills and gastrointestinal tract of the oyster Crassostrea gasar[J].Journal of Invertebrate Pathology,2017,149:76-81.