淡水养殖池塘蓝藻水华,严重危害养殖动物,威胁水产品质量安全。微囊藻(Microcystis)是淡水养殖池塘中最为常见的水华蓝藻。目前,微生物原位控藻被认为是最为绿色、最具发展前景的生态控藻方式[1]。
浮游病毒是水环境中种类和数量最多的生物类群,包括噬藻体、噬菌体、真核藻类病毒等[2]。噬藻体是一类以蓝藻为主要宿主的浮游病毒,能特异性裂解蓝藻细胞。Safferman等[3]首次分离出噬藻体LPP-1,能同时感染鞘丝藻(Lynbya)、席藻(Phormidium)和织线藻(Plectonema)[4]。噬藻体Mae-Yong 924-1能够高效裂解铜绿微囊藻(M.aeruginosa)和华美微囊藻(M.elabena)。
自然水域环境中,微囊藻细胞被胞外多糖包裹以群体形态存在,群体形成对于其抵抗浮游动物牧食和调控垂直迁移等具有重要的生态学意义[5]。微囊藻群体细胞间黏附着大量细菌,这些附生菌在微囊藻群体形成及群体形态维持中发挥着重要作用[6]。附生菌不仅能够作为微囊藻群体化的基质,还可作为微囊藻细胞的能量来源。在微囊藻和附生菌之间存在磷转移现象,附生菌可成为微囊藻的临时磷储备库[7]。筛选以微囊藻关键附生菌为特异性靶标的噬菌体,可通过侵染附生菌来促进微囊藻群体衰解,对于开发新型绿色微生物原位控藻技术具有重要意义。
病毒基因多样性高,基于保守序列的16S rDNA和18S rDNA测序不适用于病毒多样性分析。病毒宏基因组学可无偏好性地检测样本中所有病毒核酸,尽可能完整呈现样本中病毒种类多样性。目前已发现DNA病毒8 800种,RNA病毒7 596种。RNA病毒在水产养殖动物中较为常见[8],但目前养殖水环境中RNA病毒多样性研究还处于空白状态。本研究利用镜检法对团头鲂养殖池塘浮游植物进行定性定量分析,在确认该池塘发生微囊藻水华基础上,采用16S rDNA测序技术分析微囊藻群体附生菌群落组成,并采用宏基因组学测序技术分析浮游病毒群落结构,研究结果可为筛选噬藻体及针对微囊藻群体附生菌的噬菌体提供数据参考。
1 材料与方法
1.1 材料
2023年8月于天津市水产研究所暴发蓝藻水华的团头鲂(Megalobrama amblycephala)养殖池塘采集水样。该池塘面积约为0.67 hm2,平均水深约1.5 m。2022年6月投放1万尾鱼苗,每尾约50 g,期间,每天上下午各投料1次。2023年12月出池,每尾鱼出池质量为650~750 g。
1.2 方法
根据采样池塘形状及采样时风向,分别于上风向、中部及下风向定点采样。
每个采样点用1 L采水器取1 L水样加入15 mL鲁哥试剂,静置24 h后去除上清液,用于后续浮游植物定性和定量分析。参考《中国淡水藻类——系统、分类及生态》和《淡水微型生物图谱》进行浮游植物种类鉴定[9-10],依据《淡水浮游生物研究方法》进行浮游植物定量分析[11]。
使用25号浮游生物网采集浮游植物,3个采样点的样品混合,于镜下随机挑选网采浮游植物中微囊藻群体,无菌水多次冲洗,镜下观察无浮游细菌后,参照Dai等[6]方法进行附生菌分离。将获得的菌悬液先后经3.00、0.22 μm混合纤维素膜过滤,将0.22 μm滤膜放入-80 ℃冰箱保存。使用SDS方法提取0.22 μm膜上DNA[12]。采用27F和1 492R引物扩增细菌16S rDNA V3~V4区,文库经过Qubit和Q-PCR定量,文库合格后,送至北京诺禾致源生物科技有限公司,使用NovaSeq 6000进行上机测序。
使用5 L采水器采集水面以下约0.5 m深水样,每个采样点取10 L水样,水样经38 μm筛绢过滤两次后用1.6 μm混合纤维素膜(Millipore,US)过滤移除颗粒悬浮物等,再用0.22 μm聚醚砜滤膜(Millipore,US)滤除细菌,加入1 mg/L FeCl3黑暗状态下常温静置过夜。经0.8 μm聚碳酸酯(Millipore,US)滤膜抽滤,取滤膜放入50 mL无菌无酶的离心管中。使用RNApre Pure Cell/Bacteria Kit试剂盒提取RNA,质控后去除其中rRNA,在NEB Fragmentation Buffer中将得到的RNA随机打断成250~300 bp短片段,以片段化RNA为模板,以随机寡核苷酸为引物合成cDNA第一条链,随后进行PCR扩增建库;文库构建完成后,经Qubit 2.0 Fluorometer、Agilent 2100生物分析仪和qRT-PCR检测并合格的文库送至北京诺禾致源生物科技有限公司,使用Novaseq-PE150平台进行测序。
1.3 数据处理
参考Lampitt等[13]方法计算浮游植物优势度,以Y≥0.02的浮游植物定为优势种。
16S rDNA测序下机数据使用FLASH对每个样本的reads进行拼接;采用fastp软件(Version 0.23.1)对拼接得到的原始数据经过严格的过滤处理得到高质量数据,然后与物种注释数据库(https://www.arb-silva.de/)比对去除嵌合体序列,得到最终的有效数据(Effective Tags);采用QIIME2 (Version QIIME2-202202)软件中的DADA2模块进行降噪,从而获得最终的ASVs序列[14];采用QIIME2软件Silva 138.1数据库进行物种注释。采用QIIME2软件计算Goods_Coverage指数评估测序深度,所有样本的Coverage值均>0.99,表明测序数据充分覆盖群落多样性。为评估微囊藻附生菌群落的α多样性,基于质量过滤和嵌合体去除后的ASV表,在样本的均一化测序深度下,使用QIIME2计算以下指数:Chao1指数估计物种丰富度;Shannon指数和Simpson指数评估物种多样性;Pielou′s均匀度指数反映物种分布均匀性。
对病毒宏基因组下机原始数据进行预处理,得到高质量的Clean data数据;采用Bowtie2(2.2.5)软件将Clean data与宿主基因组比对去除宿主序列;Trinity(v2.4.0)[15]组装Clean data;采用Diamond(v0.8.22)软件将拼接得到的序列与数据库GenBank、NR、Refseq和CDD进行比对。
2 结果与分析
2.1 浮游植物群落结构特征
从表1可见,取样池塘浮游植物分属于6门27属,其中绿藻门种类最多,共13种,其次为硅藻门和蓝藻门,分别为6种和5种,隐藻门2种,甲藻门和裸藻门各1种。浮游植物平均丰度为8.11×108 cells/L,上风向、中部、下风向采样点浮游植物丰度分别为3.93×108、7.16×108、1.32×109 cells/L。蓝藻门相对丰度最高,为95.50%,其次依次为绿藻门(3.69%)、硅藻门(0.44%)、隐藻门(0.33%)、甲藻门(0.05%)和裸藻门(0.01%)。
表1 团头鲂养殖池塘浮游植物优势度
Tab.1 Predominance index of phytoplankton in the Megalobrama amblycephalus pond
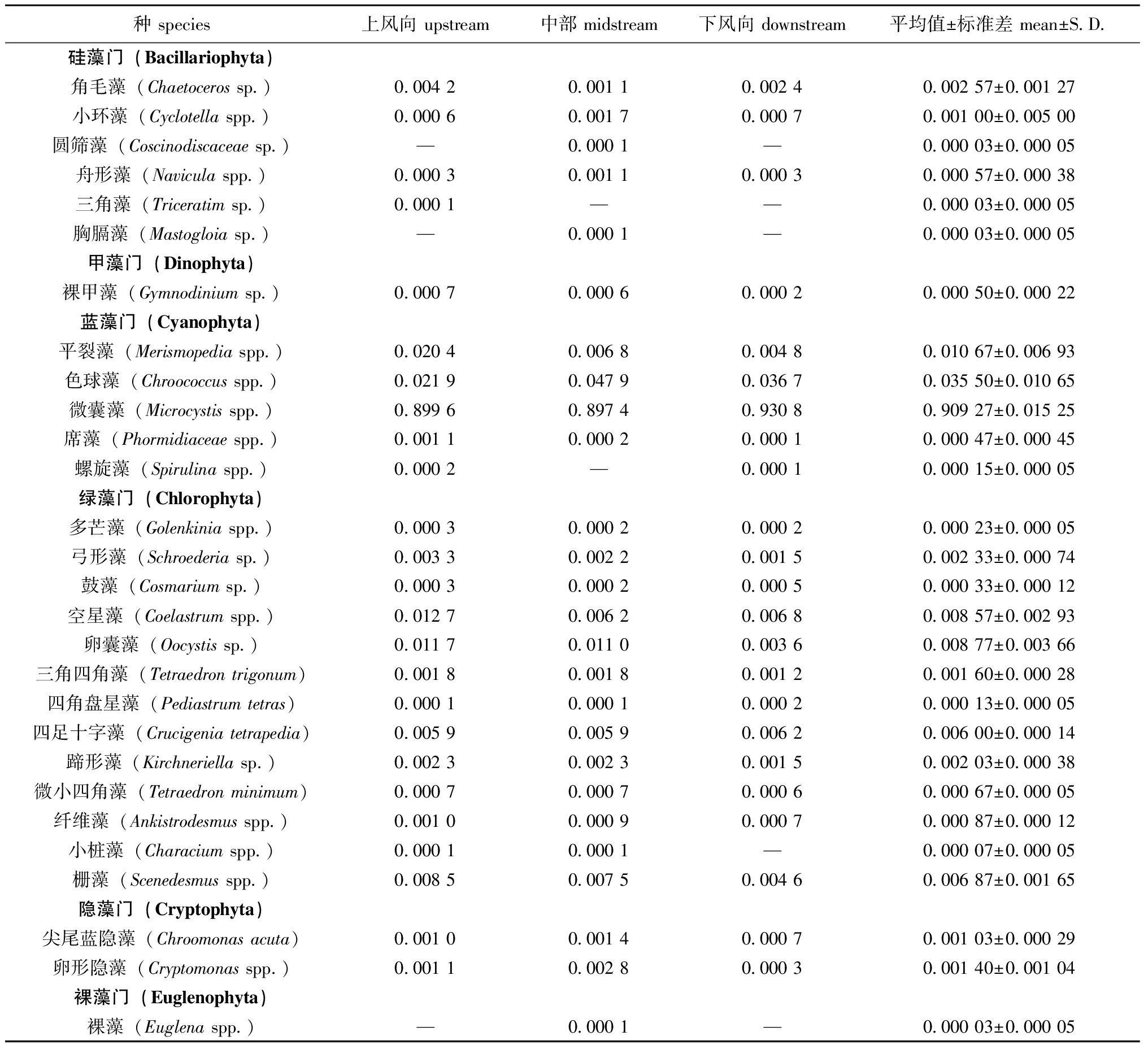
种 species上风向 upstream中部 midstream下风向 downstream平均值±标准差 mean±S.D.硅藻门(Bacillariophyta)角毛藻(Chaetoceros sp.)0.004 20.001 10.002 40.002 57±0.001 27小环藻(Cyclotella spp.)0.000 60.001 70.000 70.001 00±0.005 00圆筛藻(Coscinodiscaceae sp.)—0.000 1—0.000 03±0.000 05舟形藻(Navicula spp.)0.000 30.001 10.000 30.000 57±0.000 38三角藻(Triceratim sp.)0.000 1——0.000 03±0.000 05胸膈藻(Mastogloia sp.)—0.000 1—0.000 03±0.000 05甲藻门(Dinophyta)裸甲藻(Gymnodinium sp.)0.000 70.000 60.000 20.000 50±0.000 22蓝藻门(Cyanophyta)平裂藻(Merismopedia spp.)0.020 40.006 80.004 80.010 67±0.006 93色球藻(Chroococcus spp.)0.021 90.047 90.036 70.035 50±0.010 65微囊藻(Microcystis spp.)0.899 60.897 40.930 80.909 27±0.015 25席藻(Phormidiaceae spp.)0.001 10.000 20.000 10.000 47±0.000 45螺旋藻(Spirulina spp.)0.000 2—0.000 10.000 15±0.000 05绿藻门(Chlorophyta)多芒藻(Golenkinia spp.)0.000 30.000 20.000 20.000 23±0.000 05弓形藻(Schroederia sp.)0.003 30.002 20.001 50.002 33±0.000 74鼓藻(Cosmarium sp.)0.000 30.000 20.000 50.000 33±0.000 12空星藻(Coelastrum spp.)0.012 70.006 20.006 80.008 57±0.002 93卵囊藻(Oocystis sp.)0.011 70.011 00.003 60.008 77±0.003 66三角四角藻(Tetraedron trigonum)0.001 80.001 80.001 20.001 60±0.000 28四角盘星藻(Pediastrum tetras)0.000 10.000 10.000 20.000 13±0.000 05四足十字藻(Crucigenia tetrapedia)0.005 90.005 90.006 20.006 00±0.000 14蹄形藻(Kirchneriella sp.)0.002 30.002 30.001 50.002 03±0.000 38微小四角藻(Tetraedron minimum)0.000 70.000 70.000 60.000 67±0.000 05纤维藻(Ankistrodesmus spp.)0.001 00.000 90.000 70.000 87±0.000 12小桩藻(Characium spp.)0.000 10.000 1—0.000 07±0.000 05栅藻(Scenedesmus spp.)0.008 50.007 50.004 60.006 87±0.001 65隐藻门(Cryptophyta)尖尾蓝隐藻(Chroomonas acuta)0.001 00.001 40.000 70.001 03±0.000 29卵形隐藻(Cryptomonas spp.)0.001 10.002 80.000 30.001 40±0.001 04裸藻门(Euglenophyta)裸藻(Euglena spp.)—0.000 1—0.000 03±0.000 05
注:—表示未检出。
Note:—means not detected.
依据Y≥0.02定为优势种的原则,共鉴定出优势种2种,分别是微囊藻(0.909 3)和色球藻(Chroococcus)(0.035 5)。
2.2 微囊藻群体附生菌群落结构多样性
养殖池塘微囊藻群体附生菌由15门23纲46目87科109属组成。相对丰度排名前10的菌属组成如图1所示,梭杆菌门(Fusobacteriota)梭杆菌纲(Fusobacteriia)鲸杆菌属(Cetobacterium)相对丰度最高(29.96%),其次依次为变形菌门(Proteobacteria)γ-变形菌纲(Gammaproteobacteria)气单胞菌属(Aeromonas)(13.88%)和希瓦氏菌属(Shewanellaceae)(8.09%)。
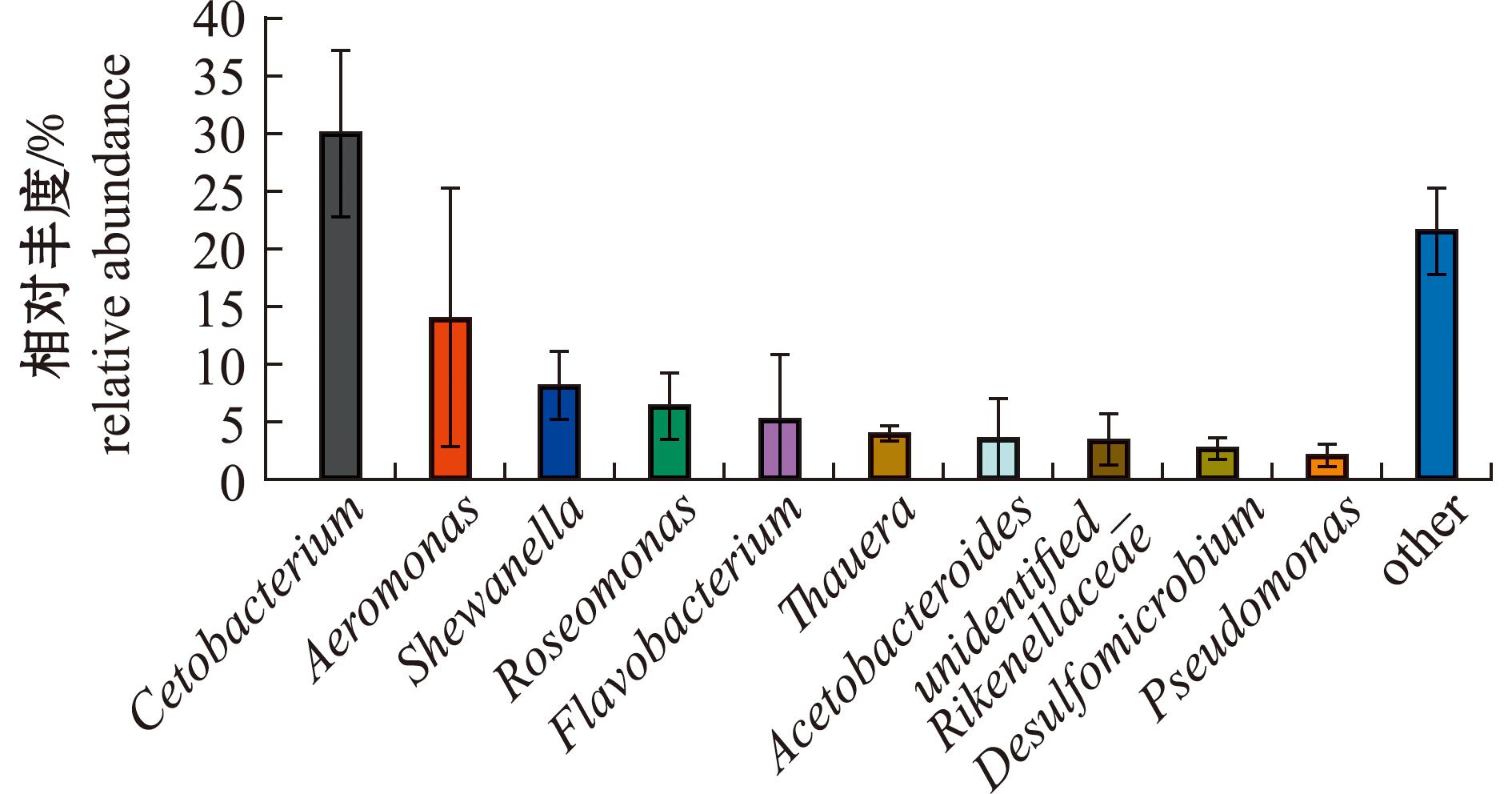
图1 属分类层级上微囊藻群体附生菌的相对丰度(前10)
Fig.1 Relative abundance of epiphytic bacteria in Microcystis colonies at the level of genus (top 10)
养殖池塘微囊藻群体附生菌α多样性指数见表2。各样本的细菌coverage均为1,说明此次测序已经覆盖了样本中所有的细菌种类,证明本次测序数据量已经达到了较为充足的水平,可以表征大多数细菌,能反映真实的细菌群落结构。附生菌Chao1指数平均为256.41,Pielou_e指数为0.63,Shannon指数为5.07,Simpson指数为0.93。
表2 微囊藻附生菌α多样性指数
Tab.2 Alpha diversity indices of epiphytic bacteria in Microcystis colonies
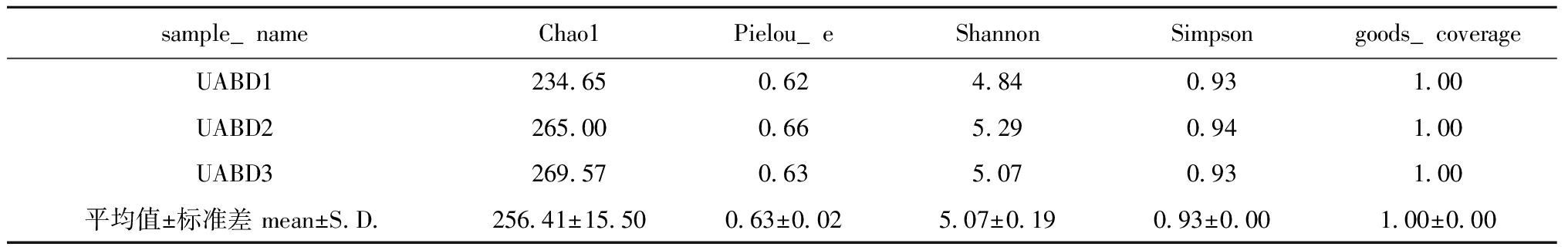
sample_nameChao1Pielou_eShannonSimpsongoods_coverageUABD1234.650.624.840.931.00UABD2265.000.665.290.941.00UABD3269.570.635.070.931.00平均值±标准差 mean±S.D.256.41±15.500.63±0.025.07±0.190.93±0.001.00±0.00
注:UABD为样品编号;1、2、3为平行样品。Note:UABD is the sample ID;1,2,3 represent parallel samples.
2.3 浮游病毒群落结构特征
养殖池塘浮游病毒分属于8门9纲25科179种。从表3可见,相对丰度排名前10的浮游病毒种类中8种均属于有尾噬菌体纲(Caudoviricetes),有尾噬菌体纲在浮游病毒中占绝对优势。其中Klebsiella phage ST13OXA48phi121相对丰度最高(30.25%),随后为Escherichia virus DE3(11.24%)和Caudovirales phage(7.81%),Punavirus P1相对丰度最低(1.79%)。
表3 种分类层级上浮游病毒的相对丰度(前10)
Tab.3 Relative abundance of planktic viruses at the level of species (top 10) %
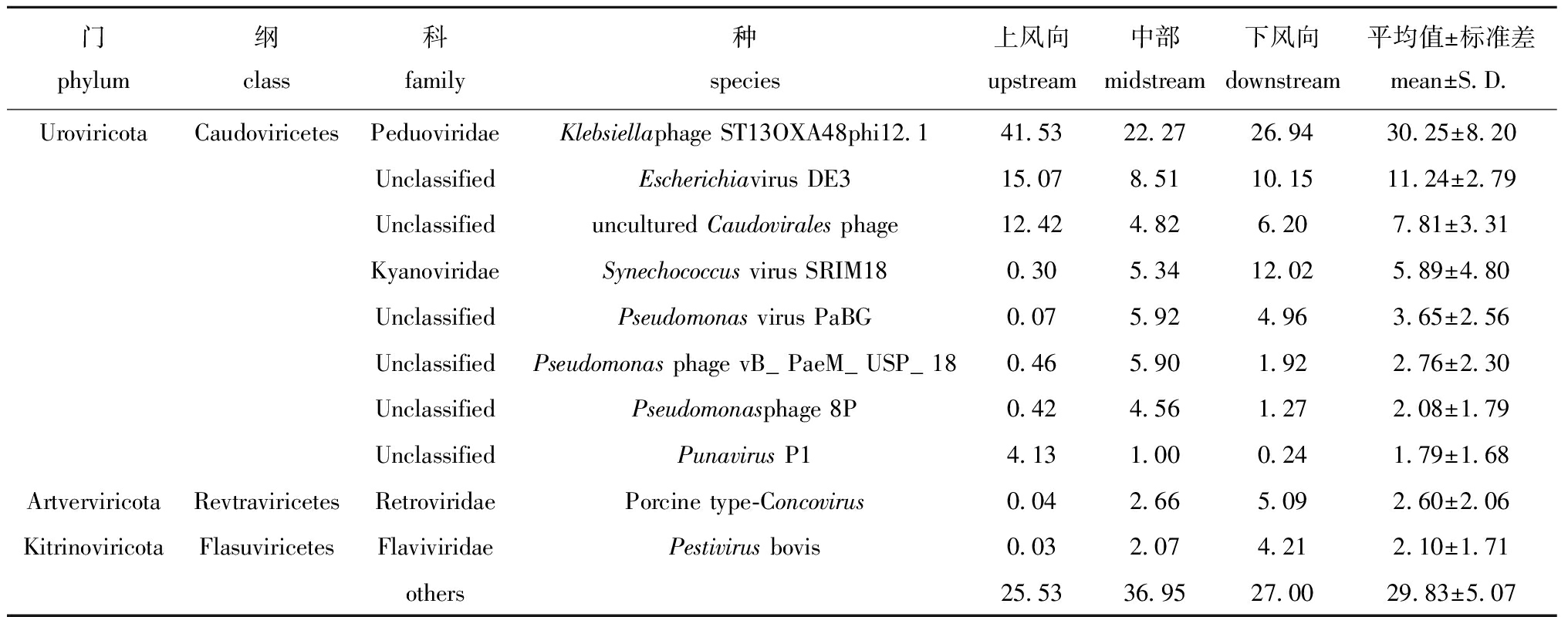
门phylum纲class科family种species上风向upstream中部midstream下风向downstream平均值±标准差mean±S.D.UroviricotaCaudoviricetesPeduoviridaeKlebsiellaphage ST13OXA48phi12.141.5322.2726.9430.25±8.20UnclassifiedEscherichiavirus DE315.078.5110.1511.24±2.79Unclassifieduncultured Caudovirales phage12.424.826.207.81±3.31KyanoviridaeSynechococcus virus SRIM180.305.3412.025.89±4.80UnclassifiedPseudomonas virus PaBG0.075.924.963.65±2.56UnclassifiedPseudomonas phage vB_PaeM_USP_180.465.901.922.76±2.30UnclassifiedPseudomonasphage 8P0.424.561.272.08±1.79UnclassifiedPunavirus P14.131.000.241.79±1.68ArtverviricotaRevtraviricetesRetroviridaePorcine type-Concovirus0.042.665.092.60±2.06KitrinoviricotaFlasuviricetesFlaviviridaePestivirus bovis0.032.074.212.10±1.71others25.5336.9527.0029.83±5.07
对能够侵染微囊藻附生菌的浮游病毒进行统计,结果见表4。针对微囊藻附生菌的噬菌体共有22种,分别侵染附生菌中假单胞菌属(Pseudomonas)、气单胞菌属(Aeromonas)、肠杆菌属(Enterobacterales)及黄杆菌属(Flavobacterium)。
表4 浮游病毒及其侵染的附生菌种类和相对丰度
Tab.4 Species and relative abundances of planktonic viruses and their infected epiphytic bacteria %
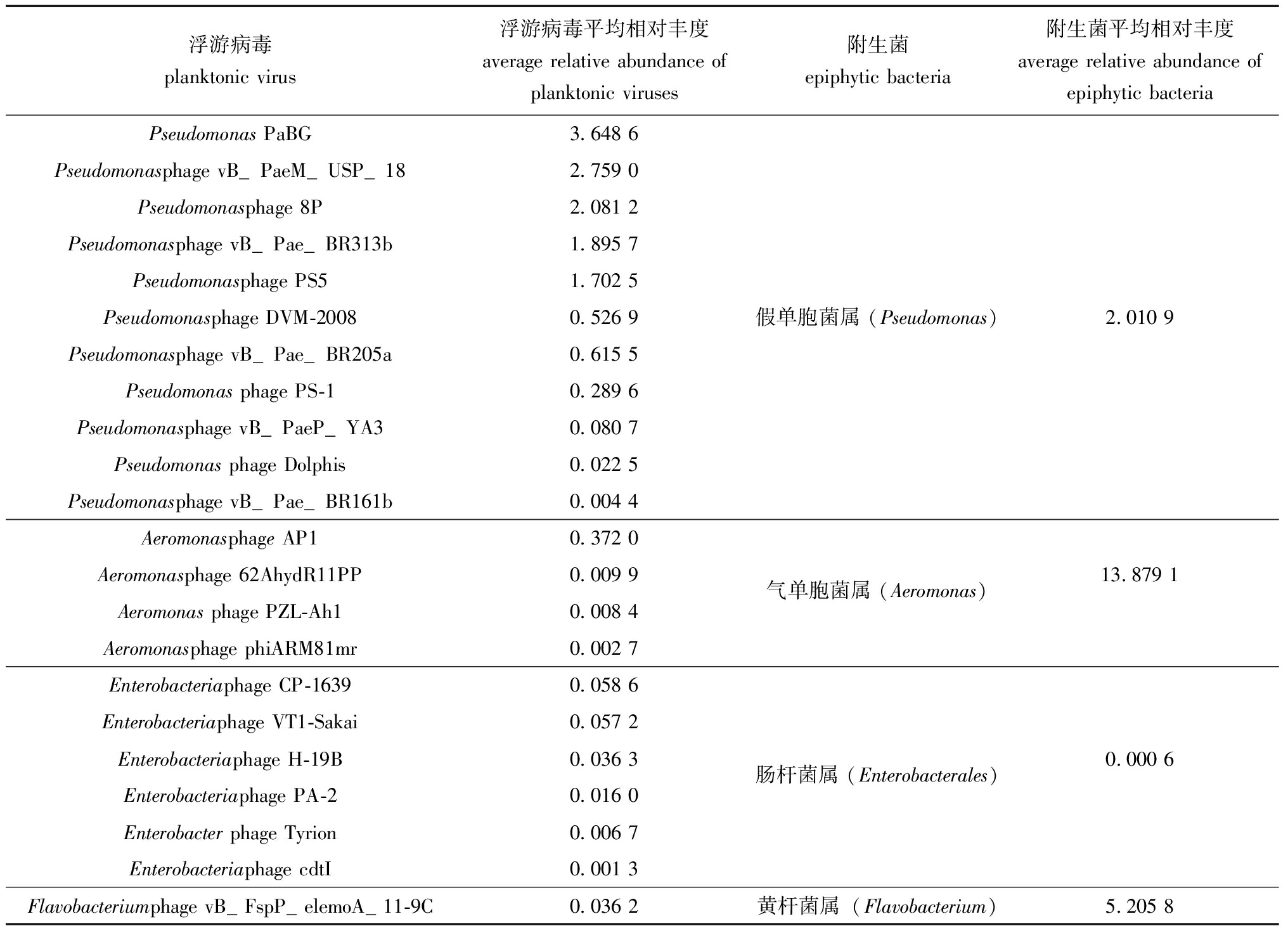
浮游病毒planktonic virus浮游病毒平均相对丰度average relative abundance of planktonic viruses附生菌epiphytic bacteria附生菌平均相对丰度average relative abundance of epiphytic bacteriaPseudomonas PaBG3.648 6Pseudomonasphage vB_PaeM_USP_182.759 0Pseudomonasphage 8P2.081 2Pseudomonasphage vB_Pae_BR313b1.895 7Pseudomonasphage PS51.702 5Pseudomonasphage DVM-20080.526 9假单胞菌属 (Pseudomonas)2.010 9Pseudomonasphage vB_Pae_BR205a0.615 5Pseudomonas phage PS-10.289 6Pseudomonasphage vB_PaeP_YA30.080 7Pseudomonas phage Dolphis0.022 5Pseudomonasphage vB_Pae_BR161b0.004 4Aeromonasphage AP10.372 0Aeromonasphage 62AhydR11PP0.009 9气单胞菌属 (Aeromonas)13.879 1Aeromonas phage PZL-Ah10.008 4Aeromonasphage phiARM81mr0.002 7Enterobacteriaphage CP-16390.058 6Enterobacteriaphage VT1-Sakai0.057 2Enterobacteriaphage H-19B0.036 3肠杆菌属 (Enterobacterales)0.000 6Enterobacteriaphage PA-20.016 0Enterobacter phage Tyrion0.006 7Enterobacteriaphage cdtI0.001 3Flavobacteriumphage vB_FspP_elemoA_11-9C0.036 2黄杆菌属(Flavobacterium)5.205 8
浮游病毒中侵染假单胞菌属噬菌体种类为11种,相对丰度达到13.599 7%;侵染气单胞菌属、肠杆菌属和黄杆菌属噬菌体种类较少,相对丰度较低,分别为0.393 0%、0.176 1%、0.036 2%。
3 讨论
3.1 浮游植物群落结构特征分析
微囊藻水华暴发会破坏养殖水域生态平衡,并给养殖产业造成损失,还会向水体释放微囊藻毒素(microcystins,MCs),造成食品安全隐患。
本研究采样时间为8月份,蓝藻在夏季高温、强光照环境下,极易在富营养化水体中暴发[16]。试验期间共鉴定出浮游植物6门28种,密度平均值为8.11×108 cells/L,其中优势藻类为蓝藻门,相对丰度为95.50%,其中属于蓝藻门的微囊藻属优势度最高,为0.909 3。从种的数量分析,绿藻门>硅藻门>蓝藻门>隐藻门>甲藻门>裸藻门,王旭娜等[17]研究发现,养殖池塘中浮游植物群落绿藻、硅藻、隐藻多为常见种,李景等[18]发现养殖池塘中绿藻为常见种,与本研究结果相似。
3.2 微囊藻附生菌群落结构多样性
研究发现,微囊藻细胞被胞外多糖包裹以群体形态存在[5],其胶被上黏附着大量细菌,附生菌与藻细胞共同构成了独特的“藻际环境”。附生菌还能为微囊藻提供磷等营养物质[7],但在营养元素缺乏时,附生菌又会与微囊藻抢夺营养,甚至产生溶藻杀藻现象[19]。
本研究中鉴定出附生菌15门23纲109属。有研究表明,α变形菌纲、γ-变形菌纲为微囊藻附生菌的主要附生类群[20]。在本研究中,γ-变形菌纲(Gammaproteobacteria)、梭杆菌纲、拟杆菌纲(Bacteroidia)、α变形菌纲(Alphaproteobacteria)为微囊藻附生菌优势类别。在属分类层级上丰度前三的细菌为鲸杆菌属、气单胞菌属和希瓦氏菌属。对太湖微囊藻附生菌进行研究发现,微囊藻附生细菌群落核心优势菌属为红杆菌属(Rhodobacter)、拉尔氏菌属(Ralstonia)、玫瑰单胞菌属(Roseomonas)、紫杆菌属(Porphyrobacter)、产碱菌科(Alcaligenaceae)和嗜纤维菌(Cytophagaceae)[21];孟艳艳等[22]的研究中发现,微囊藻附生菌水族箱产气单胞菌(Aeromonas aquariorum strain KBN1201762),并对微囊藻的生长有促进作用。赵婕等[23]研究发现,赤杆菌属(Erythrobacter)对微囊藻的形成具有促进作用。刘玉梅等[24]发现,1株溶藻气单胞菌(Aeromonas sp.)对微囊藻等多种常见水华藻类均有较好的除藻效果。本研究中相对丰度最高的浮游细菌为鲸杆菌属(Cetobacterium),相对丰度为29.96%。鲸杆菌属可产出维生素B12[25],维生素B12是一类重要的辅助因子,其对藻类的生理功能具有重要意义[26]。太湖及伊利湖西部微囊藻附生菌的Shannon指数分别为0.80~2.17、1.2~2.6,这说明其群落结构多样性低于本研究附生菌群落结构多样性[27-28]。
3.3 养殖池塘浮游病毒群落组成分析
本研究发现,伪病毒科(Peduoviridae)为团头鲂养殖池塘发生微囊藻水华期间的优势类群。前期研究发现,非养殖水体中优势病毒类群一般为长尾病毒科、短尾病毒科、肌尾病毒科[29-30]。养殖水体与非养殖水体存在的类似宿主较少,因此,养殖水体浮游病毒和其他非养殖水体的优势类群存在较大差别[31]。团头鲂养殖池塘除伪病毒科外还存在疱疹病毒科(Herpesviridae),感染细菌的阿克曼噬菌体科(Ackermannviridae),以鱼类为宿主的虹彩病毒科(Iridoviridae)、呼肠孤病毒科(Reoviridae),以植物为宿主的雀麦花叶病毒科(Bromoviridae)、自转录噬菌体科(Autographiviridae)、逆转录病毒科(Retroviridae)、斯基托噬菌体科(Schitoviridae)、拟菌病毒科(Mimiviridae)。养殖池塘与其他非养殖水体水环境条件不同、水体流动性不同,且养殖池塘中的宿主生物多样性较低,可能影响了浮游病毒的种群分布[32]。
本研究中对RNA病毒进行宏基因组测序,53.4%序列比对为DNA病毒,14.4%序列比对为RNA病毒,还有32.2%序列未比对到已知病毒。Djikeng等[33]采用宏基因组测序技术对尼德伍德湖RNA病毒进行分析时亦发现75.01%序列比对为RNA病毒,12.1%为DNA病毒。推测出现此现象的原因为浮游病毒在夏季增殖活跃[34],取样过程中DNA病毒宿主裂解,处于转录过程中DNA病毒的mRNA流出,提取RNA时这部分mRNA被同时提取,因此测序结果中出现DNA病毒。
取样池塘中发现多种已被鉴定的噬菌体。Escherichia virus DE3能够侵染大肠杆菌,Klebsiella phage ST13OXA48phi121为侵染克雷伯氏菌(Klebsiella pneumoniae)的噬菌体。更重要的是浮游病毒中存在多种针对微囊藻附生菌的噬菌体。气单胞菌属、黄杆菌属和假单胞菌分别为微囊藻附生菌中第2、第5和第10优势菌属,在浮游病毒中发现16种能够侵染以上3种菌属的噬菌体,另有6种噬菌体可以侵染附生菌非优势菌属-肠杆菌属。22种附生菌噬菌体共占浮游病毒的14.23%。这些噬菌体能否通过侵染附生菌进而影响微囊藻水华的发生与衰亡降解还有待进一步研究。
噬藻体在蓝藻种群结构调节方面发挥着重要作用,具有抑杀蓝藻的巨大潜力。已知大多数噬藻体具有宿主特异性,如从日本Mikata湖分离得到的MA-LMM01,只能特异性感染一株有毒的铜绿微囊藻(M.aeruginosa),且潜伏期较长[35]。噬藻体PA-SR01仅裂解假鱼腥藻(Pseudanabaena sp.)KCZY-C8,不能侵染其他两种假鱼腥藻藻株[36]。在已报道的近250株噬藻体中,仅21株来自淡水水体,其中微囊藻噬藻体8株,分别为Ma MV-DC、Ma-LMM01、Mic1、v B_Mel S-Me-ZS1、Mae-Yong924-1、Phi Ma05[37]、Myoviridae等[38]和Yong-L2-223[39]。在本研究中未比对到已知的微囊藻噬藻体序列。Angly等[40]对北冰洋、哥伦比亚等海湾进行研究时发现,大部分病毒序列在现有数据库中匹配不到,也说明目前水环境中的病毒研究还存在诸多空白。水华发生时养殖池塘中微囊藻密度高,可为噬藻体提供丰富的宿主,该池塘中可能存在未被鉴定的微囊藻噬藻体。
本研究中检测出聚球藻噬藻体Synechococcus srim18和Synechococcus srim50。聚球藻个体微小(0.6~2 μm),无法通过显微镜检法进行检测,因此未在取样池塘中检出。过去学者多关注于海水聚球藻噬藻体研究。胡嘉琪等[39]以海洋聚球藻(S.sp.PCC 7002)为指示宿主,从淡水中分离出一株新型噬藻体Yong-L2-223,该噬藻体除感染指示藻外,还可以感染绿色微囊藻(M.viridis FACHB-1342)和水华束丝藻(Aphanizomenon flosaquae FACHB-1209),该淡水噬藻体宿主范围广,分离方法独特,为养殖水体中微囊藻噬藻体分离提供了新的思路。
4 结论
1)蓝藻门藻细胞丰度达到7.79×108 cells/L,其中微囊藻细胞丰度高达6.72×108 cells/L,优势度为0.909 3,说明该池塘发生微囊藻水华。
2)采样池塘鉴定出微囊藻附生菌15门87科109属,其中鲸杆菌属相对丰度最高(29.96%);微囊藻附生菌群落结构多样性较高。
3)团头鲂养殖池塘发生微囊藻水华时共鉴定出浮游病毒78属179种,第一优势种为Klebsiella phage ST13OXA48phi121,相对丰度为30.25%。其中22种浮游病毒为能够侵染微囊藻附生菌的噬菌体,但未发现已被鉴定的微囊藻噬藻体。
[1] ANTOSIAK ![]() S,MALEC P,et al.Cyanophage infections reduce photosynthetic activity and expression of CO2 fixation genes in the freshwater bloom-forming Cyanobacterium aphanizomenon Flos-aquae[J].Harmful Algae,2022,116:102215.
S,MALEC P,et al.Cyanophage infections reduce photosynthetic activity and expression of CO2 fixation genes in the freshwater bloom-forming Cyanobacterium aphanizomenon Flos-aquae[J].Harmful Algae,2022,116:102215.
[2] 张奇亚,桂建芳.水生病毒学[M].北京:高等教育出版社,2008. ZHANG Q Y,GUI J F.Aquatic virology[M].Beijing:Higher Education Press,2008.(in Chinese)
[3] SAFFERMAN R S,MORRIS M E.Algal virus:isolation[J].Science,1963,140(3567):679-680.
[4] QIAN M H,LI D F,LIN W,et al.A novel freshwater cyanophage,mae-Yong924-1,reveals a new family[J].Viruses,2022,14(2):283.
[5] BURKERT U,HYENSTRAND P,DRAKARE S,et al.Effects of the mixotrophic flagellate Ochromonas sp. on colony formation in Microcystis aeruginosa[J].Aquatic Ecology,2001,35(1):11-17.
[6] DAI W,RUAN W L,BI X D,et al.The role of attached bacteria in the formation of Microcystis colony in Chentaizi River[J].Water Science and Technology,2022,86(5):968-978.
[7] JIANG L J,YANG L Y,XIAO L,et al.Quantitative studies on phosphorus transference occuring between Microcystis aeruginosa and its attached bacterium (Pseudomonas sp.)[J].Hydrobiologia,2007,581(1):161-165.
[8] 桂朗,张奇亚.中国水产动物病毒学研究概述[J].水产学报,2019,43(1):168-187. GUI L,ZHANG Q Y.A brief review of aquatic animal virology researches in China[J].Journal of Fisheries of China,2019,43(1):168-187.(in Chinese)
[9] 胡鸿钧,魏印心.中国淡水藻类:系统、分类及生态[M].北京:科学出版社,2006. HU H J,WEI Y X.The freshwater algae of China:Systcmatics,Taonomy,Ecology[M].Beijing:Science Press,2006.(in Chinese)
[10] 周凤霞,陈剑虹.淡水微型生物图谱[M].北京:化学工业出版社,2005. ZHOU F X,CHEN J H.Atlas of Freshwater microbiota[M].Beijing:Chemical Industry Press,2005.(in Chinese)
[11] 章宗涉,黄祥飞.淡水浮游生物研究方法[M].北京:科学出版社,1991. ZHANG Z X,HUANG X F.Methods of freshwater plankton research[M].Beijing:Science Press,1991.(in Chinese)
[12] DELLAPORTA S L,WOOD J,HICKS J B.A plant DNA minipreparation:version Ⅱ[J].Plant Molecular Biology Reporter,1983,1(4):19-21.
[13] LAMPITT R S,WISHNER K F,TURLEY C M,et al.Marine snow studies in the northeast Atlantic Ocean:distribution,composition and role as a food source for migrating plankton[J].Marine Biology,1993,116(4):689-702.
[14] WANG Y Y,GUO H,GAO X G,et al.The intratumor microbiota signatures associate with subtype,tumor stage,and survival status of esophageal carcinoma[J].Frontiers in Oncology,2021,11:754788.
[15] GRABHERR M G,HAAS B J,YASSOUR M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome[J].Nature Biotechnology,2011,29(7):644-652.
[16] HUISMAN J,CODD G A,PAERL H W,et al.Cyanobacterial blooms[J].Nature Reviews Microbiology,2018,16(8):471-483.
[17] 王旭娜,江敏,钟锐,等.凡纳滨对虾养殖池塘中浮游植物群落结构与水质因子的关系[J].水产学报,2018,42(11):1787-1803. WANG X N,JIANG M,ZHONG R,et al.The relationship between phytoplankton community structure andwater quality factors in the pond of Litopenaeus vannamei[J].Journal of Fisheries of China,2018,42(11):1787-1803.(in Chinese)
[18] 李景,陈昌福.温棚高产养虾池中浮游植物群落与水化学因子特征[J].中国水产,2015(7):72-76. LI J,CHEN C F.Characteristics of phytoplankton community and hydrochemical factors in high-yield shrimp ponds in greenhouses[J].China Fisheries,2015(7):72-76.(in Chinese)
[19] MANAGE P M,KAWABATA Z,NAKANO S.Dynamics of cyanophage-like particles and algicidal bacteria causing Microcystis aeruginosa mortality[J].Limnology,2001,2(2):73-78.
[20] ADLER J.Chemotaxis in bacteria[J].Science,1966,153(3737):708-716.
[21] 方道艳.蓝藻群体附生细菌群落组成的稳定性及其动态变化[D].南京:南京大学,2021. FANG D Y.Stability and dynamic changes of epiphytic bacteria community composition in cyanobacteria population[D].Nanjing:Nanjing University,2021.(in Chinese)
[22] 孟艳艳,王芳,梁霞,等.微囊藻水华附生菌的代谢特征及对微囊藻生长的影响[J].湖泊科学,2015,27(6):1115-1123. MENG Y Y,WANG F,LIANG X,et al.Metabolic characteristics and effects on Microcystis growth of attached bacteria of Microcystis blooms[J].Journal of Lake Sciences,2015,27(6):1115-1123.(in Chinese)
[23] 赵婕,李建宏,管章玲,等.一株产碱性磷酸酶附生菌对微囊藻生长的影响[J].湖泊科学,2011,23(1):49-55. ZHAO J,LI J H,GUAN Z L,et al.Effect of an attached bacterium of alkaline phosphatase producting on the growth of Microcystis aeruginosa[J].Journal of Lake Sciences,2011,23(1):49-55.(in Chinese)
[24] 刘玉梅,王梦蕙,李力.一株溶藻气单胞菌(Aeromonas sp.)的溶藻特性研究[C].//第五届山东城镇水大会——第四届“黄河杯”城镇饮用水安全保障技术论坛暨城市供水水质监测技术交流会论文集,2010:553-559. LIU Y M,WANG M H,LI L.Study on the algicidal characteristics of an algicidal Aeromonas sp.[C].//The 5th Shandong Urban Water Conference 2010-The 4th “Yellow River Cup” Technical Forum on Urban Drinking Water Safety Assurance and Technical Exchange Meeting on Urban Water Supply Quality Monitoring,2010:553-559.(in Chinese)
[25] SUZUKI A,SHIRAKATA C,ANZAI H,et al.Vitamin B12 biosynthesis of Cetobacterium ceti isolated from the intestinal content of captive common bottlenose dolphins (Tursiops truncatus)[J].Microbiology,2022,168(9).DOI:10.1099/mic.0.001244.
[26] 周璇,李军英,伍刚,等.维生素B12在细菌与真核藻类相互作用中的功能研究进展[J].微生物学通报,2015,42(5):913-919. ZHOU X,LI J Y,WU G,et al.The roles of vitamin B12 in the interaction between bacteria and eukaryotic algae[J].Microbiology China,2015,42(5):913-919.(in Chinese)
[27] WU Q,ZHANG Y P,LI Y M,et al.Comparison of community composition between Microcystis colony-attached and free-living bacteria,and among bacteria attached with Microcystis colonies of various sizes in culture[J].Aquatic Ecology,2019,53(3):465-481.
[28] SMITH D J,TAN J Y,POWERS M A,et al.Individual Microcystis colonies harbour distinct bacterial communities that differ by Microcystis oligotype and with time[J].Environmental Microbiology,2021,23(6):3020-3036.
[29] YANG Q W,GAO C,JIANG Y,et al.Metagenomic characterization of the viral community of the south Scotia ridge[J].Viruses,2019,11(2):95.
[30] DU X P,CAI Z H,ZUO P,et al.Temporal variability of virioplankton during a Gymnodinium catenatum algal bloom[J].Microorganisms,2020,8(1):107.
[31] CORINALDESI C,CREVATIN E,DEL NEGRO P,et al.Large-scale spatial distribution of virioplankton in the Adriatic Sea:testing the trophic state control hypothesis[J].Applied and Environmental Microbiology,2003,69(5):2664-2673.
[32] CLAIRE HORNER-DEVINE M,CARNEY K M,BOHANNAN B J M.An ecological perspective on bacterial biodiversity[J].Proceedings Biological Sciences,2004,271(1535):113-122.
[33] DJIKENG A,KUZMICKAS R,ANDERSON N G,et al.Metagenomic analysis of RNA viruses in a fresh water lake[J].PLoS One,2009,4(9):e7264.
[34] 姜北,周遵春,董颖,等.仿刺参养殖池塘浮游病毒丰度与环境因子的相关性分析[J].生态学杂志,2010,29(4):702-710. JIANG B,ZHOU Z C,DONG Y,et al.Correlation analysis on virioplankton abundance and its environmental factors in Apostichopus japonicus culture pond[J].Chinese Journal of Ecology,2010,29(4):702-710.(in Chinese)
[35] YOSHIDA T,NAGASAKI K,TAKASHIMA Y,et al.Ma-LMM01 infecting toxic Microcystis aeruginosa illuminates diverse cyanophage genome strategies[J].Journal of Bacteriology,2008,190(5):1762-1772.
[36] ZHANG D,YOU F,HE Y L,et al.Isolation and characterization of the first freshwater cyanophage infecting Pseudanabaena[J].Journal of Virology,2020,94(17):e00682-20.
[37] 王飞,潘灵婷,钱敏桦,等.新型淡水微囊藻噬藻体vB_MweS-yong2的分离与基因组分析[J].微生物学报,2022,62(10):3784-3800. WANG F,PAN L T,QIAN M H,et al.Isolation and genome analysis of a new freshwater Microcystis cyanophage vB_MweS-yong2[J].Acta Microbiologica Sinica,2022,62(10):3784-3800.(in Chinese)
[38] WANG J P,BAI P,LI Q,et al.Interaction between cyanophage MaMV-DC and eight Microcystis strains,revealed by genetic defense systems[J].Harmful Algae,2019,85:101699.
[39] 胡嘉琪,潘灵婷,周钦,等.新型聚球藻噬藻体Yong-L2-223的分离及基因组分析[J].微生物学报,2024,64(1):189-207. HU J Q,PAN L T,ZHOU Q,et al.Isolation and genome analysis of a novel Synechococcus cyanophage Yong-L2-223[J].Acta Microbiologica Sinica,2024,64(1):189-207.(in Chinese)
[40] ANGLY F E,FELTS B,BREITBART M,et al.The marine viromes of four oceanic regions[J].Public Library of Science Biology,2006,4(11):e368.