长吻鮠(Leiocassis longirostris)隶属鲶形目(Silurifomes)鲿科(Bagrid)鮠属(Leiocassis),又名“鮰鱼”,俗称“江团”、“肥沱”,主要分布于中国长江流域、珠江流域、闽江流域、淮河流域、辽河流域及其相通的江河湖泊,因其吻部相对于其他鮠属鱼类较长,故称为“长吻鮠”[1]。长吻鮠肉质细嫩,营养丰富,为淡水鱼类的珍品,素有“不食江团,不知鱼味”的美称,备受消费者的青睐。历史上,长江流域长吻鮠的自然资源最为丰富,近年来,由于受到酷捕滥渔、水域环境污染及水利工程建设等因素的影响,其自然种质资源日趋衰竭,养殖群体中的良种亦相对匮乏,亟需进行良种选育研究。
在水产动物中开展良种选育,若采用传统的选育方法,相对费时耗力,特别是长吻鮠等繁殖周期比较长的水产动物,亟需辅以分子标记等辅助育种技术,加速其良种的选育进程。随着分子标记技术的迅猛发展,随机扩增多态性DNA(random amplification polymorphic DNA,RAPD)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)、间简单重复序列(inter simple sequence repeat,ISSR)、简单重复序列(simple sequence repeat,SSR)及单核苷酸多态性(single nucleotide polymorphism,SNP)等分子标记技术陆续在水产动物遗传育种研究中日益突出。其中,微卫星,又称为“简单重复序列”,是基因组中以1~6个核苷酸为基本重复单位组成的串联重复序列,凭借其良好的重复性、较高的多态性和遗传稳定性及共显性遗传等优点[2-3],目前被广泛应用于水产动物群体遗传多样性分析[4]、亲子鉴定[5]、生长性状的关联分析[6-8]、遗传图谱构建[9]及分子标记辅助育种[10]等相关研究。
目前,长吻鮠的研究主要集中于繁殖生物学、养殖模式[11]、低氧胁迫[12-13]及营养成分[14-15]等方面,而对于群体遗传分析方面的研究相对较少。莫艳秀等[16]利用RAPD分子标记技术对洞庭湖野生长吻鮠群体的遗传多样性进行了评估,研究结果显示,与其他淡水鱼类相比,该群体呈现出较低的遗传多样性。相关研究中,肖明松等[17]利用线粒体DNA(mtDNA)控制区序列作为分子标记,对长吻鮠3个养殖群体和4个长江水域野生群体的遗传多样性和遗传结构进行了分析,结果显示,长吻鮠养殖群体的平均单倍型多样性和核苷酸多样性均小于野生群体,但尚未观察到显著的遗传分化现象。王红莹等[1]利用微卫星标记对长江流域4个长吻鮠群体(重庆、石首、武汉和九江)的遗传多样性和遗传结构进行了分析,发现长江水系中的长吻鮠群体遗传多样性相对贫乏。Hou等[18]利用15个微卫星分子标记对来自长江流域和珠江流域的8个长吻鮠野生群体和1个来自四川的长吻鮠养殖群体进行了遗传检测,结果显示,9个长吻鮠群体的遗传多样性仍保持在相对较高水平,且遗传分化程度较小。Zhang等[19]利用微卫星分子标记技术对4个养殖长吻鮠群体(四川眉山群体、四川宜宾群体、湖北武汉群体和湖北石首群体)的遗传多样性和遗传结构开展了研究,同样发现4个群体均具有相对较高的遗传多样性。然而,关于长吻鮠在长江水系上、中、下游养殖群体的种质资源背景研究报道相对较少,限制了对其养殖种质资源的有效开发与利用,阻碍了其养殖产业的健康、可持续发展,特别是在长江流域十年禁捕的政策下,野生群体获得难度相对较大,而对长吻鮠不同养殖群体开展遗传多样性和遗传结构研究,对于解析长吻鮠不同养殖群体的遗传特征、鉴定遗传特性优异的养殖群体及制定科学有效的遗传改良和育种策略具有重要的参考意义。
本研究中利用课题组自主开发的20对具有多态性的EST-SSR分子标记,对长江上游(四川眉山)、长江中游(湖北仙桃)、长江下游(江苏常州、上海宝山)及珠江水系(广东顺德)的长吻鮠养殖群体开展了遗传多样性和遗传结构研究,旨在全面评估不同地理区域长吻鮠养殖群体的种质资源背景,以期为长吻鮠优良新品系的选育提供科学参考,推动长吻鮠养殖产业的健康、可持续发展。
1 材料与方法
1.1 材料
试验用5个长吻鮠养殖群体共计150个样品,分别采集于上海市宝山区(以下简称“BS”,n=30)、江苏省常州市(以下简称“CZ”,n=30)、四川省眉山市(以下简称“MS”,n=30)、湖北省仙桃市(以下简称“XT”,n=30)和广东省佛山市顺德区(以下简称“SD”,n=30)的养殖场。每个样品均剪取其部分尾鳍组织,并立即置于95%乙醇中保存,随后转移至4 ℃下保存备用。
1.2 方法
1.2.1 基因组DNA的提取 每个长吻鮠尾鳍样品剪取30 mg左右,利用灭菌ddH2O清洗后,分别置于1.5 mL离心管中剪碎,然后参照天根生化科技(北京)有限公司的海洋动物组织基因组DNA提取试剂盒(DP324-03)说明书提取每个长吻鮠尾鳍样品的基因组DNA。利用15 g/L琼脂糖凝胶电泳和NanoVue Plus紫外可见分光光度计评估每个长吻鮠尾鳍DNA的质量和浓度,将电泳结果显示无拖尾降解现象、条带清晰,且OD260 nm/OD280 nm比值为1.8~2.0的长吻鮠DNA样品稀释至50 ng/μL,置于-20 ℃下保存备用。
1.2.2 PCR扩增 课题组自主开发了20对多态性较高的长吻鮠EST-SSR引物(表1),并委托上海吉真生物工程有限公司合成,其中,每对EST-SSR标记的正向引物5′端分别使用不同荧光染料(FAM、HEX和TAMRA)进行标记,以便于后续的荧光检测和分析。同时,开展梯度PCR扩增和条件优化研究,以确保引物的扩增效果和特异性。
表1 20个长吻鮠微卫星位点信息
Tab.1 Information of twenty microsatellite loci in Leiocassis longirostris

引物名称primer name引物序列(5′→3′) primer sequences(5′→3′) 重复基序repeat motif退火温度/℃anneal temperate规格/bpproduct sizeLL01F:TCTCTCATTTCTGCTCATGGGTR:GCCTCTGTCCTGTGGAGAGT(TGTC)860105LL02F:TCTCCAAGTGGCAATGCACTR:ACTGTCCAAATCTGCACCACA(GAAA)1365174LL03F:CCTTTCTGCACTTCCCCCATR:GGAGTGAACTGCTGAGTGCT(TTTA)860146LL04F:GCCTAACTATGTTGGAAGGGCR:AGAAAAGAACACATGTAGCACTTT(TAAA)1658133LL05F:GCTTCTTGAGGATTGTGTACCCR:GTCCTGTAAGCGTCACTCTGT(TATT)760164LL06F:TTGGGGGTTAAACTGCTGGTR:GAGAAAAGCCTTTGCCCAGT(GAAGA)859165LL07F:TAGAGGTGAGTGAGGCAGCTR:TTCACCTTTCTCAGCAGCAGT(GATG)753100LL08F:AACCGTCTTGCAAAACAGGAR:AGCTGACATGGATTATCTGCCT(T)1065200LL09F:TAACACAGCGCATACCCCTGR:TCTGATACAATGACTTGTTCTTCCA(TTTG)860127LL10F:TGAGGAACAAGAGAGAGGGAGAR:ACAATGTCCACTAGAGGCGC(AAGA)1563192LL11F:TCCTCCACTGTGTTTAATCCCTR:AGAATTGACGAAGAAGTTGTCACA(TTTC)1664178LL12F:TCCCAGCTTCATCTCCACACR:TCACAAAGGCAAATTTGACGAA(AAAG)1064143LL13F:TCCACACACATAGCTGCACTR:GCTCCTGCTCACCAGAATACA(TATC)1365148LL14F:CGCTCCACTTTTCCAATCAGCR:GCGTTTGGAGGTGGCTAAAA(AAAT)852144LL15F:TGCGTTTCAAGTCACTCAAGGR:TCCCACATTATCTTAGGCTGCA(AATA)1252183LL16F:TCTTCATGGGGGTATTAGCATGTR:TGAGCAACTTTTTAACATTAGATGACA(AAAG)1653193LL17F:TGGTCAGAGACTCAGAGACACTR:TGTTCATGTTTTTCTTGCTTTCTTCT(AAT)1352146LL18F:TGAGGCATGGAGGTTGTGTTR:TCCAAAATTCTCTCTTAATACTCGTCT(TTA)752114LL19F:GCGCTGTTTCTTTATGGCTGTR:TCACCAAGCCGTTCATCTTCT(TAA)1264198LL20F:TCCAAGAATTCACAGAGAAACCACR:ACATGAGTCAAAAGACCCACA(ATT)1364125
PCR反应体系为15 μL,其中包括基因组DNA 1 μL,10×Buffer和MgCl2(25 mmol)各1.5 μL,dNTP(10 mmol)和Taq酶(5 U/μL)各0.3 μL,正向和反向引物(10 μmol)各0.15 μL,不足部分利用去离子水补足至15 μL。
PCR反应条件设定如下:首先,在94 ℃下进行3 min的预变性处理,随后进入循环扩增阶段,每个循环包括在94 ℃下变性15 s,在各引物的最佳退火温度(表1)下退火15 s,72 ℃延伸30 s,共进行35次循环,待循环结束后,再在72 ℃下延伸10 min,最后将反应体系冷却至4 ℃下保存备用。利用琼脂糖凝胶电泳的方法评估PCR产物浓度,对PCR产物浓度进行评估后,将其稀释10倍,以降低背景干扰,然后再与LIZ500内标(大小分别为35,50,75,100,139,150,160,200,250,300,340,350,400,450,490,500 base)均匀混合,最后置于ABI 3730XL测序仪进行毛细管电泳检测(委托上海吉真生物科技有限公司完成)。
1.3 数据处理
使用GeneMarker 2.2软件读取ABI 3730XL基因测序仪测序结果,获取每个样本等位基因的编号、峰值图和基因型等信息后,进行微卫星片段大小的统计,并将结果录入Excel中保存备用。利用GenAlEx 6.0软件[20]计算长吻鮠不同群体的等位基因数(number of allele,Na)、有效等位基因数(effective number of allele,Ne)、观测杂合度(observed heterozygosity,Ho)和期望杂合度(expected heterozygosity,He)、群体间Nei’s遗传距离和遗传相似度、群体间遗传分化指数(genetic differentiation index,Fst)、基因流(gene flow,Nm)及分子方差分析(analysis of molecular variance,AMOVA)等遗传多样性参数,并进行主坐标分析(principal coordinates analysis,PCoA)和哈迪-温伯格平衡(hardy-Weinberg equilibrium,HWE)检验。参照Bostein等[21]的计算公式计算不同微卫星位点的多态信息含量(polymorphism information content,PIC)。利用MEGA软件构建长吻鮠不同群体的UPGMA(unweighted pair-group method with arithmetic means,UPGMA)聚类树。利用Structure (version 2.3.4)软件[22]进行遗传结构作图,采用Structure Harverter 软件[23]确定最适K值,CLUMPP软件[24]对K值矩阵进行合并,采用Distruct软件[25]绘制贝叶斯基因型聚类图。
2 结果与分析
2.1 遗传多样性
在本研究中,长吻鮠5个养殖群体的遗传多样性参数见表2。结果显示,20对EST-SSR引物总共检测到219个多态性位点,平均每对引物检测到10.95个等位基因。CZ群体的平均Ne最多(4.13),BS群体最低(3.03)。长吻鮠5个养殖群体的平均Ho从0.708 3(BS)到0.813 3(SD),而平均He则是从0.624 3(BS)到0.727 6(CZ)。平均PIC则是CZ群体最高(0.686 5),其次分别为MS群体(0.684 7)、XT群体(0.673 3),BS群体最低(0.574 0)。5个长吻鮠养殖群体的平均Ho值均大于平均He值,表明5个长吻鮠养殖群体存在不同程度的杂合子过剩现象。总体而言,长吻鮠CZ群体的各项遗传参数大部分都高于其他养殖群体,有望应用于后续长吻鮠的良种培育研究。
表2 长吻鮠不同群体的遗传多样性参数
Tab.2 Genetic diversity of Leiocassis longirostris from five different cultured populations
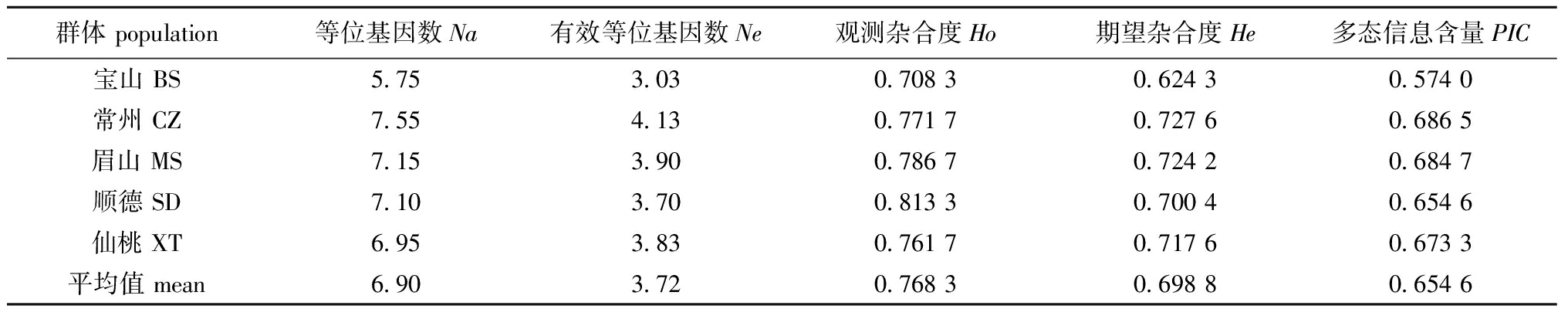
群体 population等位基因数Na有效等位基因数Ne观测杂合度Ho期望杂合度He多态信息含量PIC宝山 BS5.753.030.708 30.624 30.574 0常州 CZ7.554.130.771 70.727 60.686 5眉山 MS7.153.900.786 70.724 20.684 7顺德 SD7.103.700.813 30.700 40.654 6仙桃 XT6.953.830.761 70.717 60.673 3平均值 mean6.903.720.768 30.698 80.654 6
2.2 哈迪-温伯格(Hardy-Weinberg)平衡检测
20个微卫星位点的哈迪-温伯格(Hardy-Weinberg,HWE)平衡检测结果(表3)显示,微卫星位点LL14在5个长吻鮠群体中均显著偏离哈迪-温伯格平衡(P<0.05),BS群体显著偏离哈迪-温伯格平衡位点最多,有12个微卫星位点显著偏离(P<0.05),CZ群体和SD均有9个微卫星位点显著偏离哈迪-温伯格平衡(P<0.05),MS群体有8个微卫星位点显著偏离哈迪-温伯格平衡(P<0.05),XT群体显著偏离哈迪温-伯格平衡位点最少,仅有7个位点显著偏离(P<0.05);在100个群体组合中(5群体×20位点),符合哈迪-温伯格平衡的群体位点有55个;其余45个群体位点显著偏离(P<0.05)。
表3 长吻鮠20个微卫星位点的遗传多样性参数
Tab.3 Genetic diversity in 20 microsatellite loci of Leiocassis longirostris
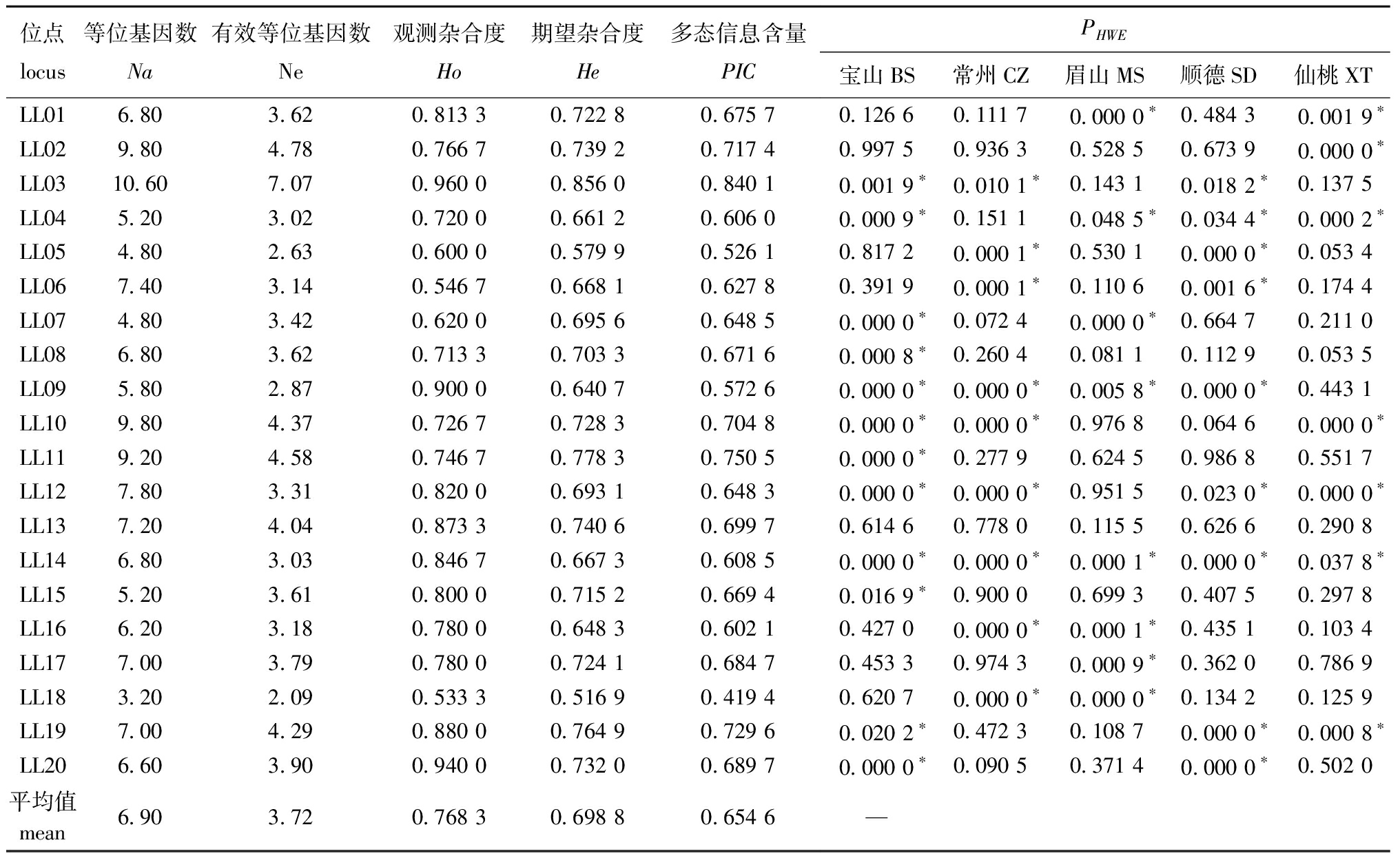
位点locus等位基因数Na有效等位基因数Ne观测杂合度Ho期望杂合度He多态信息含量PICPHWE宝山BS常州CZ眉山MS顺德SD仙桃XTLL016.803.620.813 30.722 80.675 70.126 60.111 7 0.000 0∗0.484 3 0.001 9∗LL029.804.780.766 70.739 20.717 40.997 50.936 30.528 50.673 90.000 0∗LL0310.607.070.960 00.856 00.840 10.001 9∗0.010 1∗0.143 10.018 2∗0.137 5LL045.203.020.720 00.661 20.606 00.000 9∗0.151 10.048 5∗0.034 4∗0.000 2∗LL054.802.630.600 00.579 90.526 10.817 20.000 1∗0.530 10.000 0∗0.053 4LL067.403.140.546 70.668 10.627 80.391 90.000 1∗0.110 60.001 6∗0.174 4LL074.803.420.620 00.695 60.648 50.000 0∗0.072 40.000 0∗0.664 70.211 0LL086.803.620.713 30.703 30.671 60.000 8∗0.260 40.081 10.112 90.053 5LL095.802.870.900 00.640 70.572 60.000 0∗0.000 0∗0.005 8∗0.000 0∗0.443 1LL109.804.370.726 70.728 30.704 80.000 0∗0.000 0∗0.976 80.064 60.000 0∗LL119.204.580.746 70.778 30.750 50.000 0∗0.277 90.624 50.986 80.551 7LL127.803.310.820 00.693 10.648 30.000 0∗0.000 0∗0.951 50.023 0∗0.000 0∗LL137.204.040.873 30.740 60.699 70.614 60.778 00.115 50.626 60.290 8LL146.803.030.846 70.667 30.608 50.000 0∗0.000 0∗0.000 1∗0.000 0∗0.037 8∗LL155.203.610.800 00.715 20.669 40.016 9∗0.900 00.699 30.407 50.297 8LL166.203.180.780 00.648 30.602 10.427 00.000 0∗0.000 1∗0.435 10.103 4LL177.003.790.780 00.724 10.684 70.453 30.974 30.000 9∗0.362 00.786 9LL183.202.090.533 30.516 90.419 40.620 70.000 0∗0.000 0∗0.134 20.125 9LL197.004.290.880 00.764 90.729 60.020 2∗0.472 30.108 70.000 0∗0.000 8∗LL206.603.900.940 00.732 00.689 70.000 0∗0.090 50.371 40.000 0∗0.502 0平均值 mean6.903.720.768 30.698 80.654 6—
注:*表示有显著性差异(P<0.05)。Note:*means significant effect(P<0.05).
2.3 遗传距离、遗传相似度和UPGMA聚类分析
长吻鮠不同群体间的遗传距离和遗传相似系数见表4。遗传距离的结果显示,长吻鮠不同地理群体间的遗传距离为0.033 4~0.226 6,平均每两个群体间的遗传距离为0.129 1;其中BS群体与其他地理群体间的遗传距离较大(0.159 4~0.226 6),平均每两个群体间的遗传距离为0.206 0;其他两两地理群体间的遗传距离为0.033 4~0.097 1,平均遗传距离为0.077 8。遗传相似度的结果显示,长吻鮠CZ群体和XT群体具有最高的遗传相似度(0.967 1),而BS群体和XT群体具有最低的遗传相似度(0.797 2)。
表4 5个长吻鮠群体间的遗传相似系数(对角线上方)和遗传距离(对角线下方)
Tab.4 Genetic similarity coefficient (below diagonal) and genetic distance (above diagonal) of Leiocassis longirostris from five stocks cultured
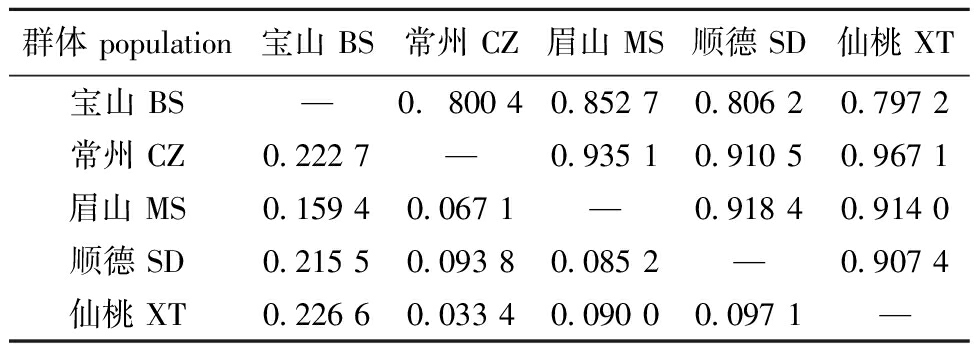
群体 population宝山 BS常州 CZ眉山 MS顺德 SD仙桃 XT宝山 BS—0.800 40.852 70.806 20.797 2常州 CZ0.222 7—0.935 10.910 50.967 1眉山 MS0.159 40.067 1—0.918 40.914 0顺德 SD0.215 50.093 80.085 2—0.907 4仙桃 XT0.226 60.033 40.090 00.097 1—
基于Nei’s遗传距离的UPGMA聚类分析结果(图1)显示,CZ群体先与和XT群体聚为一支,然后与MS群体聚为一支,再与SD群体聚为一支,最后与BS群体聚为一支,表明BS群体与其他地理群体遗传关系相对较远。
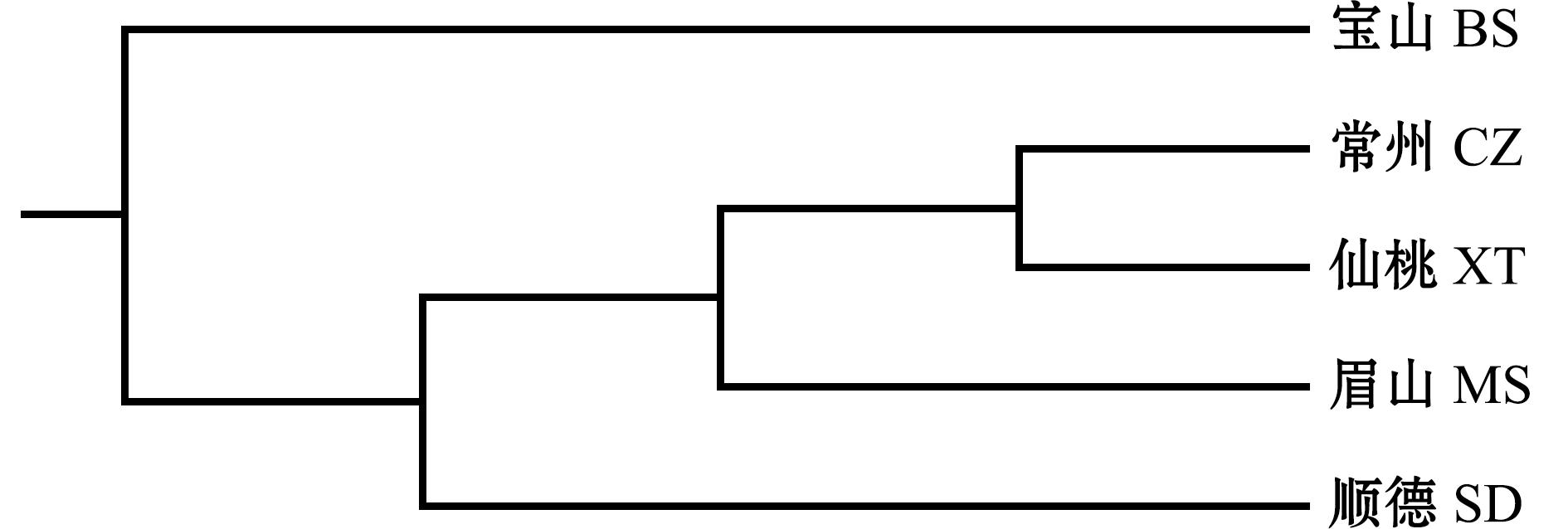
图1 5个不同长吻鮠养殖群体间的UPGMA聚类分析
Fig.1 UPGMA analysis among five different cultured populations in Leiocassis longirostris
2.4 遗传分化和分子方差分析
长吻鮠不同群体间的遗传分化指数(Fst)为0.013 8~0.055 8(表5),长吻鮠BS群体和CZ群体、SD群体和XT群体具有中等程度的遗传分化(0.053 6~0.055 8),而其他两两群体间的遗传分化微弱(0.013 8~0.043 8),表明不同的长吻鮠养殖群体具有不同程度的遗传分化。此外,长吻鮠不同养殖群体间的基因流(Nm)为4.230 3(BS &XT)~17.865 9(CZ &XT)(表5),表明这5个群体之间存在不同程度的基因交流,各群体间的基因流均大于4,在一定程度上降低了遗传分化的发生几率。
表5 长吻鮠两两群体间的遗传分化指数(对角线下方)和基因流(对角线上方)
Tab.5 Index of genetic differentiation (below diagonal) and gene flow (above diagonal) between any two populations in Leiocassis longirostris
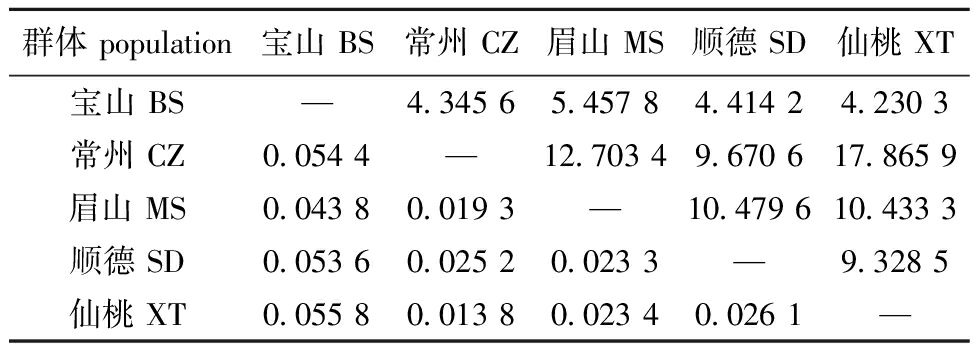
群体 population宝山 BS常州 CZ眉山 MS顺德 SD仙桃 XT宝山 BS—4.345 65.457 84.414 24.230 3常州 CZ0.054 4—12.703 49.670 617.865 9眉山 MS0.043 80.019 3—10.479 610.433 3顺德 SD0.053 60.025 20.023 3—9.328 5仙桃 XT0.055 80.013 80.023 40.026 1—
为进一步探究长吻鮠不同养殖群体遗传变异的来源,对长吻鮠5个养殖群体开展了分子方差分析(AMOVA),结果显示,在总的遗传变异中,仅有4.72%的遗传变异来源群体间,其余95.28%的遗传变异来源于群体内个体间的遗传差异,表明5个长吻鮠养殖群体的遗传变异主要来源于群体内个体间的遗传差异(表6)。
表6 长吻鮠不同群体和个体间的分子方差分析
Tab.6 AMOVA analysis among the different populations and iindividuals in Leiocassis longirostris
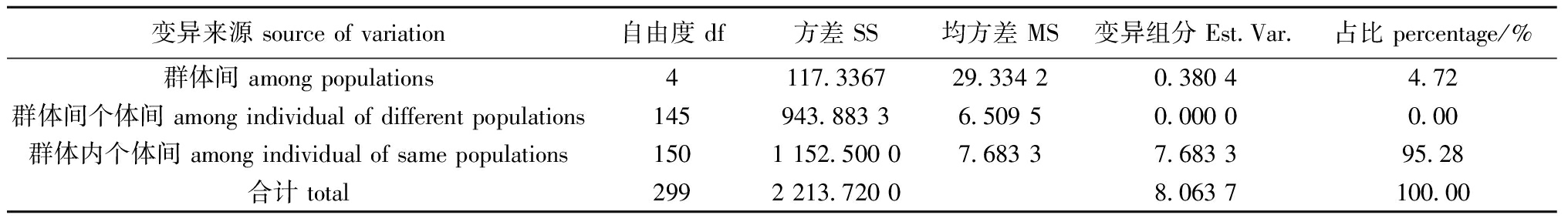
变异来源 source of variation自由度 df方差 SS均方差 MS变异组分 Est.Var.占比 percentage/%群体间 among populations4117.336729.334 20.380 44.72群体间个体间 among individual of different populations145943.883 36.509 50.000 00.00群体内个体间 among individual of same populations1501 152.500 07.683 37.683 395.28合计 total2992 213.720 08.063 7100.00
2.5 长吻鮠不同群体的遗传结构
在动物群体遗传学中,主坐标分析(PCoA)能够可视化的显示不同群体的相似性和差异性。基于长吻鮠不同个体间的Nei’s遗传距离的主坐标分析(PCoA)结果显示(图2),BS群体内的个体与其他群体的个体具有明显的差异,其中,CZ和XT群体内的个体间遗传差异最小,BS和XT群体内个体间的遗传差异最大。

图2 长吻鮠5个群体基于Nei’s遗传距离的主坐标分析
Fig.2 Principal coordinates analysis based on Nei’s genetic distance among five Leiocassis longirostris populations
对长吻鮠不同群体的遗传结构进行分析,通过假设K值为1~10,绘制δK变化曲线图,当δK最大时,得到最佳K值为2(图3),表明150个长吻鮠个体最佳能够划分为2个类群,聚类图中不同颜色代表不同长吻鮠类群个体(图4),结果显示,5个长吻鮠群体的不同个体遗传结构存在不同程度的混杂,不同群体的遗传组成较多样化,且BS群体的遗传结构与其他4个群体具有显著性差异。
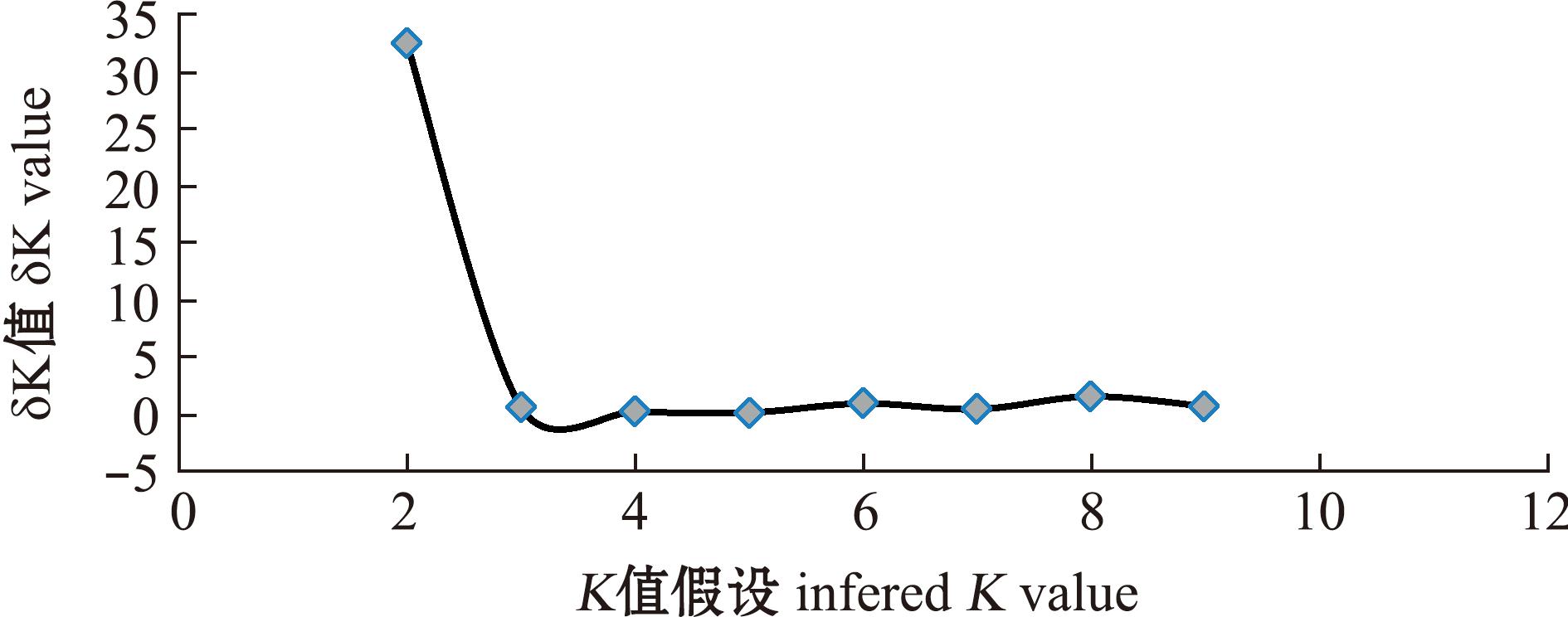
图3 基于不同K值假设的δK计算结果
Fig.3 δK calculated based on different K values
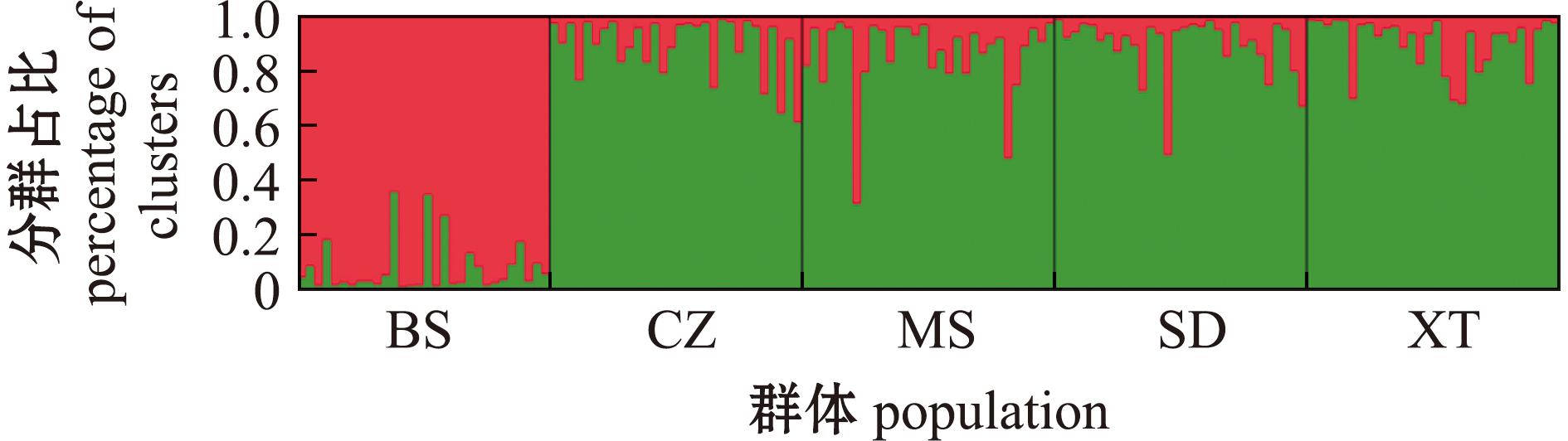
图4 长吻鮠5个养殖群体中个体的遗传结构图(K=2)
Fig.4 Genetic structure of individuals from five cultured populations of Leiocassis longirostris(K=2)
3 讨论
3.1 群体遗传多样性
遗传多样性是水产动物长期生存、适应环境和保持进化的重要基础,亦是种质资源评估的重要理论依据[26],其中,基于微卫星分子标记分析遗传多样性和遗传结构是最常用且有效的分子标记方法,备受水产研究人员关注。近年来,虽然InDel、SNP等新一代分子标记的出现,导致微卫星标记的关注度有所降低,但是相对于SNP标记的二态性特征,微卫星标记凭借其较高的多态性,较好的重复性和共显性遗传特性,仍然被认为是水产动物群体遗传多样性分析的重要工具[27]。有研究表明,与SNP标记相比较,中等密度的微卫星标记研究种群关系的效果相对更好[27],因此微卫星标记仍然具有较为广阔的应用前景。
丰富的遗传多样性是水产动物适应性进化和人工选育的重要基础和先决条件,而等位基因数、观测杂合度、期望杂合度和多态信息含量则是评估群体遗传多样性水平的关键指标[28]。基于75个微卫星分子标记的研究显示,13种淡水鱼类(共计7 755尾个体)的平均Na为7.50,平均He为0.46[29],而在本研究中,5个长吻鮠养殖群体的平均Na为5.75~7.55,略低于上述13种淡水鱼类的平均值;而平均He为0.624 3~0.727 6,高于13种淡水鱼类的平均值,表明尽管5个长吻鮠养殖群体的等位基因数相对较少,但其期望杂合度高,遗传多样性仍然较为丰富。国内部分学者亦对长吻鮠不同群体的遗传多样性开展了深入性研究。例如,王红莹等[1]利用微卫星分子标记技术分析了长江流域长吻鮠(重庆、石首、武汉和九江群体)的遗传多样性,发现4个群体的平均Ho和He分别为0.650 5和0.576 7;Zhang等[19]则对4个养殖长吻鮠群体(四川眉山群体、四川宜宾群体、湖北武汉群体和湖北石首群体)进行了类似分析,发现4个群体的平均Ho和He分别为0.807 2和0.810 4;Jin等[30]利用线粒体DNA和微卫星分子标记技术分析了长江上游长吻鮠不同鱼龄野生群体的遗传多样性,结果显示,观测杂合度和多态信息含量分别为0.734 2和0.676 4;Hou等[18]利用15个微卫星分子标记对来自长江流域和珠江流域的8个长吻鮠野生群体和1个来自四川的长吻鮠养殖群体进行了遗传检测,结果显示,9个长吻鮠群体的平均Ho和He分别为0.73和0.72,且大部分群体的平均Ho和He无显著性差异。将本研究结果与上述研究进行比较显示,本研究中所获得的5个长吻鮠群体的平均Ho和He分别为0.768 3和0.698 8,这一结果略高于王红莹等[1]和Jin等[30]的研究结果,但是略低于Hou等[18]的研究结果,这可能与试验所选微卫星引物的数量、类型、试验样本的数量及来源有关。此外,本研究中所有长吻鮠群体的Ho均高于平均He,且所有位点的平均近交系数均为负值,表明这些群体存在不同程度的杂合子过剩现象,这主要与不同养殖群体的遗传背景差异、繁育亲本选择的选育策略、繁殖方式、基因交流的受限程度及养殖环境的特异性等多种因素有关[3,31];此外,本研究中所选部分养殖长吻鮠样本来源于相关水域的水产养殖市场,由于市场进货渠道的多样性,可能会存在所选取样本来源于当地不同养殖场的情况,这种来源的多样性可能导致所选样本的遗传杂合度相对较高,进而呈现出杂合子过剩的现象。多态信息含量是评估遗传多样性水平高低的重要参考指标,而基于Botstein对于多态信息含量(PIC)的评估标准,当PIC>0.5,0.25<PIC≤0.5,PIC≤0.25时分别代表高度多态、中度多态和低度多态性[21]。本研究中,85%的微卫星位点为高度多态(PIC值为0.552 1~0.789 0),15%的微卫星位点为中度多态(PIC值为0.422 6~0.498 6),而5个长吻鮠群体的平均多态信息含量均大于0.5,表明这些群体具有相对较大的基因丰度和遗传改良潜力。
哈迪-温伯格平衡定律又称为遗传学平衡定律,是群体有性繁殖上下代之间基因频率和基因型频率是否能够保持平衡的检验尺度[32],亦是水产动物开展保种和新品种(系)选育的重要理论依据。在本研究中,BS群体有60%的位点显著偏离哈迪-温伯格平衡(P<0.05),是5个群体中偏离比例最高的群体;相比之下,XT群体仅有35%的位点显著偏离哈迪温伯格平衡(P<0.05),偏离比例最低。值得注意的是,微卫星位点LL14在5个长吻鮠群体中均显著偏离哈迪-温伯格平衡(P<0.05)。总体而言,在100个群体位点组合中,45%的微卫星位点显著偏离哈迪-温伯格平衡(P<0.05),这种现象在水产动物中较为普遍,特别是在水产动物的选育群体中更为突出。类似地,在鲤(Cyprinus carpio)[33]、菲律宾蛤仔(Ruditapes philippinarum)[34]及拟穴青蟹(Scylla paramamosain)[35]等水产动物的研究中,均发现高比例的微卫星标记显著偏离哈迪-温伯格平衡,偏离比例分别为91.7%、52.5%和88.24%,这些偏离现象的原因可能归因于水产动物较高的遗传变异度,以及养殖/选育过程中人工定向选择强度的差异和亲本的配种策略的不同程度影响。
3.2 群体遗传分化和结构
探究水产经济动物不同地理群体间的遗传分化水平和遗传结构,对于指导水产经济动物繁育亲本的遗传配对,以及种质资源的保护、开发和利用具有重要的理论和实践意义。而遗传分化指数(Fst)则是评估群体间遗传分化程度的重要参考指标,当0<Fst<0.05时,代表微弱的遗传分化;当0.05≤Fst<0.15时,代表中等程度的遗传分化;当0.15≤Fst<0.25时,代表高水平的遗传分化[36]。在本研究中,BS群体除与MS群体遗传分化微弱外(FDst=0.043 8),与其他3个群体均具有中等程度的遗传分化(0.053 6~0.055 8),且与XT群体遗传分化程度最高(0.055 8);而其他两两群体间的遗传分化均相对微弱(0.013 8~0.025 2),且CZ群体和XT群体遗传分化程度最小(0.013 8)。基因流(Nm)是影响群体遗传分化水平的重要影响因子,当Nm>4时表明群体间的遗传分化程度较小,能够有效降低遗传分化发生的几率。本研究中,长吻鮠BS群体与其他4群体间基因流相对较小(4.230 3~5.457 8),其他4个群体间的基因流相对较大(9.328 5~17.865 9),表明其他4个群体间基因交流频繁,导致遗传分化水平较低,这与有效繁育亲本的数量差异、人工定向选择强度及可能存在的遗传瓶颈效应等多重因素有关[37];与此同时,相对较大的遗传距离亦存在于BS群体和CZ群体、XT群体,且已产生中等程度的遗传分化,可能是由于BS群体取自原种保种群体的繁育子代,仅定期从四川长吻鮠良种场补充部分繁育亲本,导致与其他群体产生基因交流机会较少,因此遗传结构特征相对独特,且与四川MS群体具有相对较低的遗传分化水平和相对较高的基因流。
Nei’s遗传距离和遗传相似系数不仅能够有效反映不同群体间的基因差异程度,而且能够反映不同群体间的亲缘关系。本研究中长吻鮠不同地理群体间的遗传距离为0.033 4~0.226 6,平均每两个群体间的遗传距离为0.129 1;其中,BS群体与其他地理群体间的遗传距离较大(0.159 4~0.226 6),平均每两个群体间的遗传距离为0.206 0;其他两两地理群体间的遗传距离为0.033 4~0.097 1,平均遗传距离为0.077 8。遗传相似度结果显示,长吻鮠CZ群体和XT群体具有最高的遗传相似度(0.967 1),而BS群体和XT群体具有最低的遗传相似度(0.797 2)。基于Nei’s遗传距离构建的UPGMA聚类分析图和主坐标分析图(PCoA)亦显示,BS群体单独聚为一支,其他4个群体聚为一支。而Structure分析结果显示一个长吻鮠群体最佳效果划分为2个类群,5个长吻鮠群体的不同个体遗传结构存在不同程度的混杂,不同群体的遗传组成亦较多样化,其中BS群体单独为一个类群,其他4个群体为一个类群。这些研究结果表明,不同的长吻鮠养殖群体的遗传结构存在不同程度的混杂和多样化。
4 结论
1)本研究利用20对多态性EST-SSR分子标记对5个长吻鮠养殖群体开展了遗传多样性和遗传结构分析,发现5个长吻鮠养殖群体的遗传多样性相对较高,除长吻鮠BS群体外,其他两两群体间的遗传分化微弱,BS群体具有相对独特的遗传结构。
2)长吻鮠后续遗传育种工作中,在考虑长吻鮠个体大小、营养成分及抗逆性等经济性状的同时,选用遗传距离较大的繁育亲本进行组配,有望培育出抗逆性强、生长性能优的长吻鮠新品种(系)。
[1] 王红莹,黄文清.应用微卫星标记分析长江流域长吻鮠4个群体的遗传多样性[J].河南农业科学,2011,40(2):146-148,160. WANG H Y,HUANG W Q.Preliminary analysis on the genetic diversity in four populations of Leiocassis longirostris by using microsatellite markers[J].Journal of Henan Agricultural Sciences,2011,40(2):146-148,160.(in Chinese)
[2] 于爱清,施永海,徐嘉波,等.长江刀鲚选育群体转录组EST-SSR的分布特征分析[J].渔业科学进展,2019,40(5):101-109. YU A Q,SHI Y H,XU J B,et al.Characteristic analysis of microsatellites in selected Coilia ectenes using a transcriptome dataset[J].Progress in Fishery Sciences,2019,40(5):101-109.(in Chinese)
[3] 张敏莹,方弟安,周彦锋,等.基于微卫星标记的钱塘江中下游三角鲂亲本群体和自然捕捞群体遗传多样性及遗传结构[J].大连海洋大学学报,2022,37(5):775-783. ZHANG M Y,FANG D A,ZHOU Y F,et al.Genetic diversity and genetic structure of four brooder populations and one captive population of black bream Megalobrama terminalis in the middle and lower reaches of Qiantang River by microsatellite marker[J].Journal of Dalian Ocean University,2022,37(5):775-783.(in Chinese)
[4] 徐昊旸,杨雪莹,倪未,等.乌龟选育世代遗传多样性及遗传结构的微卫星分析[J].大连海洋大学学报,2023,38(5):812-818. XU H Y,YANG X Y,NI W,et al.Genetic diversity and genetic structure of selected generations of Chinese pond turtle (Mauremys reevesii) based on microsatellite DNA[J].Journal of Dalian Ocean University,2023,38(5):812-818.(in Chinese)
[5] 鲁弘毅,田海峰,胡乔木,等.基于微卫星多重PCR的黄鳝亲子鉴定技术[J].南方水产科学,2023,19(6):97-106. LU H Y,TIAN H F,HU Q M,et al.Parentage assignment of Monopterus albus using multiplex PCR of microsatallites[J].South China Fisheries Science,2023,19(6):97-106.(in Chinese)
[6] 顾颖,鲁翠云,张芹,等.微卫星QTL标记分析豫选黄河鲤群体遗传结构及生长性状相关性[J].淡水渔业,2016,46(4):9-18. GU Y,LU C Y,ZHANG Q,et al.Analysis of genetic diversity and growth traits in Cyprinus carpio var.haematopterus using microsatellite QTL markers[J].Freshwater Fisheries,2016,46(4):9-18.(in Chinese)
[7] 刘志刚,高风英,佟延南,等.尼罗罗非鱼生长性状相关微卫星标记的筛选与验证[J].大连海洋大学学报,2023,38(6):925-934. LIU Z G,GAO F Y,TONG Y N,et al.Screening and verification of microsatellite markers associated with growth traits in Nile Tilapia (Oreochromis niloticus)[J].Journal of Dalian Ocean University,2023,38(6):925-934.(in Chinese)
[8] 王丹,邹桂伟,罗相忠.鲢EST-SSR标记的开发及其与生长性状关联性分析[J].水生生物学报,2020,44(3):494-500. WANG D,ZOU G W,LUO X Z.Development ofest-ssr markers and analysis of growth trait in silver carp[J].Acta Hydrobiologica Sinica,2020,44(3):494-500.(in Chinese)
[9] 郭香,李琪,孔令锋,等.基于微卫星标记整合长牡蛎遗传图谱[J].水产学报,2013,37(6):823-829. GUO X,LI Q,KONG L F,et al.Microsatellite-based consensus map of the Pacific oyster(Crassostrea gigas)[J].Journal of Fisheries of China,2013,37(6):823-829.(in Chinese)
[10] 鲁翠云,匡友谊,郑先虎,等.水产动物分子标记辅助育种研究进展[J].水产学报,2019,43(1):36-53. LU C Y,KUANG Y Y,ZHENG X H,et al.Advances of molecular marker-assisted breeding for aquatic species[J].Journal of Fisheries of China,2019,43(1):36-53.(in Chinese)
[11] 覃普,姚俊杰,杨云贤,等.陆基推水集装箱循环水养殖长吻鮠、黄颡鱼试验初报[J].科学养鱼,2020(11):42-43. QIN P,YAO J J,YANG Y X,et al.Preliminary report on aquaculture of Leiocassis longirostris and Pseudobagrus fulvidraco with circulating water in land-based water pushing container[J].Scientific Fish Farming,2020(11):42-43.(in Chinese)
[12] 李谣,杨智茹,程景颢,等.低氧胁迫和恢复对长吻鮠脑组织低氧应答基因、生理生化指标和食欲的影响[J].水产学报,2023,47(1):152-164. LI Y,YANG Z R,CHENG J H,et al.Effects of hypoxia stress and reoxygenation on appetite,hypoxic response genes and physiological and biochemical indexes in the brain tissues of Leiocassis longirostris[J].Journal of Fisheries of China,2023,47(1):152-164.(in Chinese)
[13] 李谣,杨智茹,程景颢,等.低氧胁迫和复氧对长吻鮠鳃组织低氧应答基因和生理生化指标的影响[J].中国农学通报,2023,39(2):107-116. LI Y,YANG Z R,CHENG J H,et al.Effects of hypoxic stress and reoxygenation on hypoxic response genes and physiological and biochemical indexes in the gill tissue of Leiocassis longirostris[J].Chinese Agricultural Science Bulletin,2023,39(2):107-116.(in Chinese)
[14] 赵仲孟,周剑,李强,等.不同年龄段养殖长吻鮠肌肉的营养成分比较及评价[J].中国农学通报,2023,39(23):102-108. ZHAO Z M,ZHOU J,LI Q,et al.Analysis and evaluation of nutritive composition in muscles of cultured Leiocassis longirostris at different ages[J].Chinese Agricultural Science Bulletin,2023,39(23):102-108.(in Chinese)
[15] 张升利,孙向军,张欣,等.长吻鮠含肉率及肌肉营养成分分析[J].大连海洋大学学报,2013,28(1):83-88. ZHANG S L,SUN X J,ZHANG X,et al.The dressing rate and nutrient components in muscle of Leiocassis longirostris[J].Journal of Dalian Ocean University,2013,28(1):83-88.(in Chinese)
[16] 莫艳秀,王晓清,莫永亮.长吻鮠遗传多样性的RAPD分析[J].江西水产科技,2010(2):13-16. MO Y X,WANG X Q,MO Y L.RAPD analysis of genetic diversity of Leiocassis longirostris [J].Jiangxi Fishery Science and Technology,2010(2):13-16.(in Chinese)
[17] 肖明松,崔峰,康健,等.长吻鮠养殖群体与野生群体遗传多样性分析[J].水生生物学报,2013,37(1):90-99. XIAO M S,CUI F,KANG J,et al.Analysis on sequence polymorphism of the mitochondrial DNA control region and population genetic diversity of the cultivated and natural Chinese longsnout catfish(Leiocassis longirostris)[J].Acta Hydrobiologica Sinica,2013,37(1):90-99.(in Chinese)
[18] HOU Y L,YE H,SONG X H,et al.Genetic diversity and population structure of Chinese longsnout catfish (Leiocassis longirostris) using microsatellite DNA markers[J].Fishes,2024,9(1):35.
[19] ZHANG L,MOU C Y,ZHOU J,et al.Genetic diversity of Chinese longsnout catfish (Leiocassis longirostris) in four farmed populations based on 20 new microsatellite DNA markers[J].Diversity,2022,14(8):654.
[20] PEAKALL R,SMOUSE P E.Genalex 6:genetic analysis in excel.Population genetic software for teaching and research[J].Molecular Ecology Notes,2006,6(1):288-295.
[21] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[22] EVANNO G,REGNAUT S,GOUDET J.Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J].Molecular Ecology,2005,14(8):2611-2620.
[23] EARL D A,VONHOLDT B M.STRUCTURE HARVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method[J].Conservation Genetics Resources,2012,4(2):359-361.
[24] JAKOBSSON M,ROSENBERG N A.CLUMPP:a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure[J].Bioinformatics,2007,23(14):1801-1806.
[25] ROSENBERG N A.Distruct:a program for the graphical display of population structure[J].Molecular Ecology Notes,2004,4(1):137-138.
[26] 罗宇婷,方弟安,周彦锋,等.基于微卫星标记对长江下游鲢遗传多样性现状的分析[J].南方水产科学,2021,17(6):48-57. LUO Y T,FANG D A,ZHOU Y F,et al.Genetic diversity of silver carp(Hypophthalmichthys molitrix) in lower reaches of Yangtze River based on microsatellite markers[J].South China Fisheries Science,2021,17(6):48-57.(in Chinese)
[27] 杨铭,于洋,张晓军,等.基于转录组数据的凡纳滨对虾微卫星标记开发[J].海洋科学,2017,41(2):96-102. YANG M,YU Y,ZHANG X J,et al.Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp(Litopenaus vannamei)[J].Marine Sciences,2017,41(2):96-102.(in Chinese)
[28] 于爱清,施永海,邓平平.长江刀鲚选育和野生群体遗传多样性的微卫星分析[J].水产科技情报,2019,46(3):121-125. YU A Q,SHI Y H,DENG P P.Genetic diversity analysisof cultured and wild populations of Coilia ectenes by microsatellite markers[J].Fisheries Science &Technology Information,2019,46(3):121-125.(in Chinese)
[29] DEWOODY J A,AVISE J C.Microsatellite variation in marine,freshwater and anadromous fishes compared with other animals[J].Journal of Fish Biology,2000,56(3):461-473.
[30] JIN Y J,HE K,XIANG P,et al.Temporal genetic variation of the Chinese longsnout catfish (Leiocassis longirostris) in the Upper Yangtze River with resource decline[J].Environmental Biology of Fishes,2022,105(9):1139-1151.
[31] BALLOUX F.Heterozygote excess in small populations and the heterozygote-excess effective population size[J].Evolution,2004,58(9):1891-1900.
[32] 管宇,张香云,徐群芳,等.哈迪-温伯格平衡检验及其基因频率的估计[J].浙江林学院学报,2009,26(1):122-126. GUAN Y,ZHANG X Y,XU Q F,et al.Hardy-Weinberg equilibrium testing and estimating of allele frequency[J].Journal of Zhejiang Forestry College,2009,26(1):122-126.(in Chinese)
[33] 董在杰,刘念,傅建军,等.6个野生与选育鲤群体的微卫星遗传分析[J].南方水产科学,2018,14(4):46-55. DONG Z J,LIU N,FU J J,et al.Genetic analysis for six wild and selection populations of common carp (Cyprinus carpio) using microsatellites[J].South China Fisheries Science,2018,14(4):46-55.(in Chinese)
[34] 聂鸿涛,李佳,霍忠明,等.菲律宾蛤仔人工选育群体与野生群体的遗传多样性分析[J].中国水产科学,2016,23(3):538-546. NIE H T,LI J,HUO Z M,et al.Analysis of genetic variability in selected lines and a wild population of Ruditapes philippinarum using microsatellite markers[J].Journal of Fishery Sciences of China,2016,23(3):538-546.(in Chinese)
[35] 舒妙安,周宇芳,朱晓宇,等.中国沿海拟穴青蟹群体遗传多样性的微卫星分析[J].水产学报,2011,35(7):977-984. SHU M A,ZHOU Y F,ZHU X Y,et al.Microsatellite analysis on genetic diversity of seven wild populations of Scylla paramamosain in China[J].Journal of Fisheries of China,2011,35(7):977-984.(in Chinese)
[36] WRIGHT S.The interpretation of population structure by f-statistics with special regard to systems of mating[J].Evolution,1965,19(3):395-420.
[37] SLATKIN M.Gene flow and the geographic structure of natural populations[J].Science,1987,236(4803):787-792.