长牡蛎(Crassostrea gigas)是全球范围内养殖规模最大、产量最高的贝类之一。在其生命周期的早期阶段,即胚胎和幼虫期,个体极为敏感且脆弱。这一时期贝壳的正常形成对于其能否顺利度过变态发育过程至关重要[1]。在长牡蛎的个体发育中,经历了基质壳和钙质壳等形成过程[2],其中,几丁质是贝类早期幼虫合成基质壳的主要部分[3],且对后续钙的沉积具有重要作用,其由几丁质合成酶(chitin synthase,CHS)催化合成。
几丁质是软体动物贝壳的有机组分之一,与蛋白质、多糖等物质共同构成贝壳的有机框架,在此基础上,生物矿化过程得以有序进行[4]。几丁质是由直链N-乙酰-β-D-氨基葡糖胺(GlcNAc)单体通过β-1,4糖苷键连接而成的古老且丰富的结构氨基多糖[5]。几丁质在自然界中广泛存在,根据结构不同,其分为α、β和γ 3种聚合物。目前,在双壳贝类中鉴定到的主要为β型几丁质,其由同向平行排列的GlcNAc聚合长链形成单斜晶系结构[6]。现已从牡蛎、厚壳贻贝(Mytilus coruscus)、乌贼(Sepia officinalis)、珠母贝(Pinctada maxima)、芋螺(Lividoconus muriculatus)和江珧(Atrina pectinata)等软体动物中成功提取并鉴定到β型几丁质[6-10]。长牡蛎幼虫最初的壳场(shell filed)起源于壳腺(shell gland),其是幼虫囊胚后期胚胎腹侧出现的一个微弱凹陷;随着胚胎发育到担轮幼虫早期,壳场在背侧凹陷深处开始形成。在此期间,贝壳骨膜在该区域加工合成,其主要成分是几丁质,这一过程标志着长牡蛎幼虫贝壳形成的开端;胚胎发育至担轮幼虫期,最初的骨膜层从背侧持续扩展到腹侧,中间的狭窄区域将发育为铰合部[11],此时形成了两个贝壳的雏形;贝壳一般在D型幼虫时期完成均匀的钙化过程,标志着其贝壳的主要成分由几丁质过渡到了钙质。因此,几丁质的合成在长牡蛎原肠胚到D型幼虫时期的变态发育过程中发挥着重要作用,其中涉及壳腺的形成、壳腺向壳场的转变、初生骨膜的合成和钙质壳的沉积过程[12]。
几丁质合成是由多种酶协同作用的复杂过程,其中,几丁质合成酶(CHS)扮演着核心角色。CHS是一种膜结合的糖苷转化酶,通过催化尿苷二磷酸酯-N-乙酰氨基葡萄糖(UDP-GlcNAc)生物结合形成几丁质[13],这是几丁质合成过程中最后且最关键的环节[14-15]。CHS最初是在真菌中被鉴定出来,根据氨基酸序列结构的差异,CHS被划分为两个主要分支:分支Ⅰ(包含CHS-1~3)和分支Ⅱ(包含CHS-4~8)[16]。真菌CHS家族成员蛋白均具有多个跨膜结构域(transmembrane domain,TMD)和3种不同的几丁质合成结构域(Chitin_synth_1 domain、Chitin_synth_1N domain和Chitin_synth_2 domain)。其中,分支Ⅰ蛋白的特点是具有疏水的C端和亲水的N端区域,并包含几丁质合成结构域。分支Ⅱ蛋白还包含一个细胞色素b5样的血红素/类固醇结合结构域(Cytochrome b5-like heme/steroid binding domain,Cyt-b5 domain)。此外,分支Ⅱ中CHS-5和CHS-6蛋白均具有N端的肌球蛋白结构域(myosin motor like domain,MMD)[17]。在昆虫中,几丁质合成酶主要包括两种类型:几丁质合成酶1(CHS-1,又称CHSA)和几丁质合成酶2(CHS-2,又称CHSB)[13]。其中,CHS-1主要负责昆虫表皮和气管中几丁质的合成,CHS-2只在中肠围食膜上负责几丁质的形成[14-15]。几丁质是双壳贝类珍珠层形成的关键成分,其主要为钙质壳的形成提供有机框架。CHS在贝类几丁质壳的形成过程中发挥着重要作用。目前已在包括长牡蛎[18]、厚壳贻贝[19]、日本珍珠贝(Pinctada fucata)[9]、栉江珧[20]等在内的多种双壳贝类中鉴定到CHS的存在,且被证明参与了幼虫发育时期的壳形成过程。贝类CHS主要包括两个保守的关键结构域:Chitin_synth_1N和Chitin_synth_2。在长牡蛎中,CHS在幼虫从浮游阶段向附着变态阶段转变的过程中表达显著升高[18]。利用原位杂交检测到厚壳贻贝中的CHS主要在幼虫发育阶段的铰合部表达,后续可能参与铰合部的分化[19]。另外,利用红外光谱等技术鉴定出日本珍珠贝棱柱层中含有几丁质,并证明CHS合成的几丁质有助于壳结构中有机框架的形成[9]。
几丁质是贝类担轮幼虫阶段合成基质壳的主要成分,而CHS是几丁质代谢的关键酶,其在贝类担轮幼虫时期的基质贝壳形成到D型幼虫的钙质贝壳沉积过程中发挥重要作用[21-22]。目前,已在多种软体动物中鉴定到几丁质和CHS的存在,包括长牡蛎、厚壳贻贝等,然而相关研究仍处于初步阶段,关于贝类CHS基因的系统发育关系、亚型分类及其在贝类生长发育等生理过程中的具体作用机制仍有待进一步揭示。长牡蛎是中国重要的海水养殖贝类,壳形成过程对于长牡蛎早期幼虫发育及苗种培育至关重要,因此,研究长牡蛎幼虫阶段壳形成过程对人工养殖海域牡蛎苗种繁育具有重要意义。本研究中对长牡蛎CgCHS基因家族进行了分子和进化等特征分析,检测了其在长牡蛎幼虫发育时期的表达模式,以期为进一步探究长牡蛎CgCHS在幼虫壳形成中的作用机制提供有益参考。
1 材料与方法
1.1 材料
试验用长牡蛎亲贝采自辽宁省大连市长海县獐子岛集团股份有限公司大连海珍品原良种厂生物技术研发部养殖海区,饲养于新鲜海水中,每天换1/2新鲜海水,每日投喂金藻(Chrysophyta),在夏季育苗季节利用新鲜恒温海水繁育长牡蛎幼虫。
1.2 方法
1.2.1 基于HMM模型进行CHS基因家族鉴定 基于CHS的功能结构域,利用隐马尔可夫模型(hidden Markov model,HMM)预测含功能结构域的蛋白序列并去除冗余。具体步骤如下:从NCBI网站上下载长牡蛎蛋白质数据(GCF_902806645.1);去除数据冗余,使用cd-hit软件去除由于基因组可能的拼接错误和可变剪切产生的冗余,参数为“-c 0.8-aS 0.8-d 0”,将相似度大于80%的序列合并,保留最长蛋白序列;建立HMM模型,使用Pfam数据库的Chitin_synth_2结构域信息,下载原始HMM模型文件,用软件HMMER中的Hmmpress命令建立索引;使用HMMER软件中的hmmsearch命令,在蛋白组中搜索含有功能结构域的蛋白序列;编写Perl脚本整理输出结果,从蛋白组文件中提取出所有蛋白序列,共筛选出7个CgCHS基因家族成员序列。
为鉴定不同物种中的CHS基因家族,检索了NCBI(http://www.ncbi.nlm.nih.gov)和Ensembl(http://www.ensembl.org)数据库,收集到4种硬骨鱼[斑马鱼(Danio rerio)、花鲈(Lateolabrax japonicus)、青鳉(Oryzias latipes)、欧洲鳎(Solea solea)],4种节肢动物[果蝇(Drosophila virilis)、赤拟谷盗(Tribolium castaneum)、西方蜜蜂(Apis mellifera)、中华绒螯蟹(Eriocheir sinensis)],5种软体动物[长牡蛎、虾夷扇贝(M.yessoensis)、厚壳贻贝、中华蛸(Octopus sinensis)、澳洲黑边鲍(H.rubra)]、1种假体腔动物[秀丽隐杆线虫(Caenorhabditis elegans)]和4种真菌类[酵母菌(Saccharomyces cerevisiae)、绣球菌(Sparassis crispa)、玉米黑粉菌(Mycosarcoma maydis)、禾谷镰孢霉(Fusarium graminearum)],共18个物种CHS基因的核酸序列。
1.2.2 CgCHS基因染色体定位 依据长牡蛎基因组注释信息判定CgCHS基因在染色体上的分布情况,借助TBtools程序将CgCHS基因锚定在相应染色体上,绘制CgCHS基因的染色体分布。
1.2.3 CgCHS家族蛋白结构域、理化性质、系统进化分析、多序列比对及Motif分析 根据查询得到的CgCHS家族成员的蛋白序列,利用Expasy ProtParam网站(https://web.expasy.org/protparam/)预测CgCHS蛋白质的理化性质和不稳定系数。蛋白质的不稳定系数是指蛋白质在体外测试中稳定性的参考值,其值<40表明稳定性高,≥40表明蛋白质可能不稳定。利用SMART在线工具(http://smart.embl-heidelberg.de)预测CgCHS蛋白结构域。利用TMHMM 2.0(https://services.healthtech.dtu.dk/services/TMHMM-2.0/)对CgCHS家族蛋白跨膜区域进行在线分析。
根据从Ensembl和NCBI数据库获得的34个选定物种(8种硬骨鱼类、1种软骨鱼类、12种节肢动物、6种软体动物、1种假体腔动物、1种两栖纲和5种真菌类)的CHS编码蛋白质序列,利用MEGA X软件对CgCHS和其他不同物种中的CHS蛋白质序列进行多序列比对及系统进化树分析。采用最大似然法(maximum likelihood)和软件默认参数,构建CHS家族蛋白质分子系统进化树。
根据CgCHS基因GFF注释文件信息,在MEME(http://meme-suite.org/tools/meme)网站分析CgCHS蛋白序列的保守基序(Motif),并对其进行可视化分析。
1.2.4 长牡蛎幼虫样品的收集 取20只性腺饱满的长牡蛎,辨别性别后,挤出精子和卵子,分别经过48 μm和75 μm筛绢过滤至5 L量杯中。向卵子中倒入适量精子,用显微镜检查受精情况。将受精卵放在装有50 L 25 ℃过滤海水的培养箱中孵育,发育至担轮幼虫时期前每隔20 min搅动一次。取各发育时期样品:受精卵(0 h)、4细胞期(1 h 32 min)、8细胞期(2 h 25 min)、桑葚胚期(3 h 30 min)、囊胚期(4 h 35 min)、原肠胚期(8 h 30 min)、担轮幼虫期(12 h 35 min)、D型幼虫期(17 h 35 min)。分别用48 μm筛网过滤收集至50 mL管中,以800 r/min离心5 min,吸出多余海水,幼虫样品加500 μL Trizol至1.5 mL管中,-80 ℃下保存。每个发育时期设置3组重复。
1.2.5 RNA提取和cDNA合成 使用TransZol Up Plus RNA Kit(北京全式金生物技术有限公司)提取长牡蛎早期各发育阶段幼虫的总RNA。通过NanoDrop 2000和10 g/L琼脂糖凝胶电泳法对所提取的RNA样品进行定量和定性检测。将质量合格的RNA用于后续cDNA合成。
参照TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix反转录试剂盒(北京全式金生物技术有限公司)配置混合体系,采用PCR仪进行反转录,用于扩增CgCHS序列。反应体系(20 μL):gDNA Removal 1 μL,Anchored Oligo(dT)18 Primer(0.5 μg/μL)1 μL,2×Ts Reaction Mix 10 μL,TransScript qRT/RI Enzyme Mix 1 μL,Total RNA/mRNA 1 μL,RNase-Free dH2O 6 μL。cDNA合成在42 ℃下恒温15 min,85 ℃下加热5 s,42 ℃下恒温30 min,85 ℃加热5 s条件下进行反应,将cDNA模板稀释20 倍并保存在-80 ℃超低温冰箱用于后续试验。
1.2.6 实时荧光定量-聚合酶链式反应(qRT-PCR)分析 根据CgCHS基因的cDNA全长序列,采用Primer Premier 5.0软件设计qRT-PCR引物(表1)。利用Quant Studio 6实时荧光定量PCR仪进行qRT-PCR反应,检测CgCHS在长牡蛎幼虫不同发育阶段(受精卵、4细胞期、8细胞期、桑椹胚期、囊胚期、原肠胚期、担轮幼虫期和D型幼虫期)的表达情况,以EF(NP_001292242.2)为内参基因。qRT-PCR反应体系(10 μL):2×SYBR Green Master Mix 5 μL,ROX Reference Dye(50 ×)0.2 μL,正、反向引物F、R各0.2 μL,20倍稀释的cDNA模板2 μL,DEPC水2.4 μL。反应程序:95 ℃下预变性10 min,95 ℃下变性10 s,60 ℃下退火45 s,共进行40个循环。采用2-ΔΔCt法计算基因的相对表达量。
表1 试验所用引物
Tab.1 Primers used in the experiment
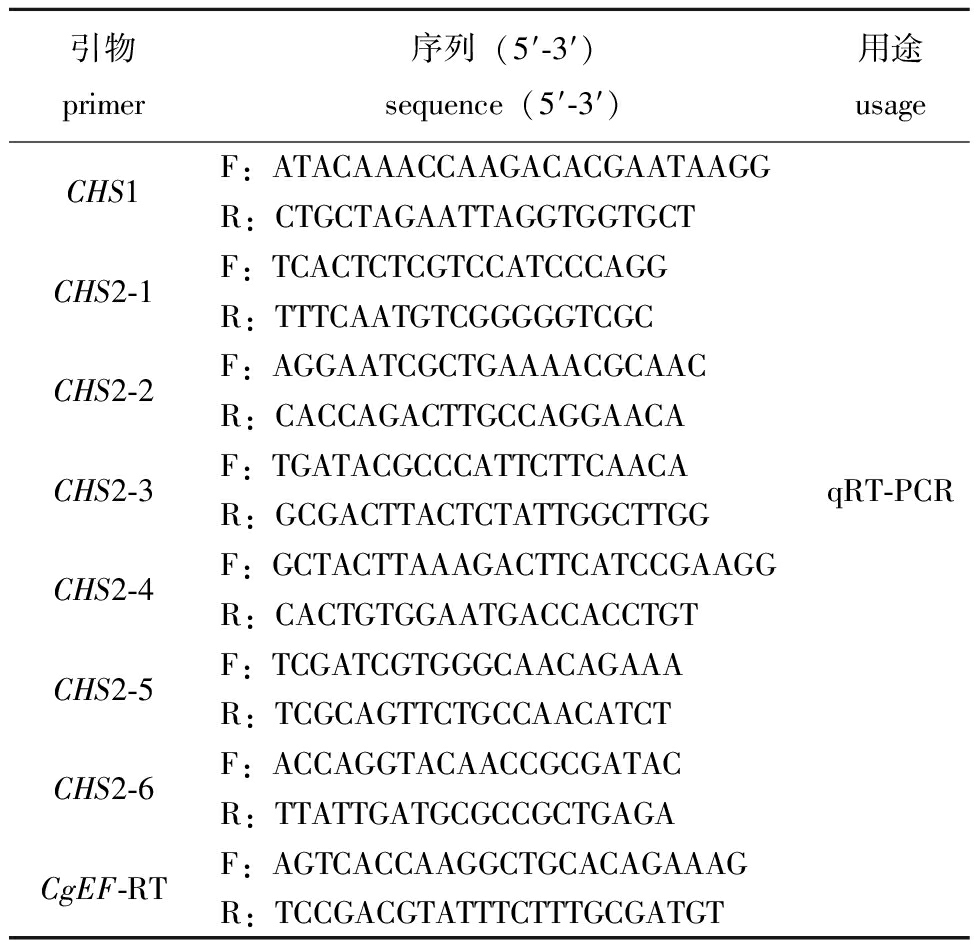
引物primer序列(5′-3′)sequence(5′-3′)用途usageCHS1F:ATACAAACCAAGACACGAATAAGGR:CTGCTAGAATTAGGTGGTGCTCHS2-1F:TCACTCTCGTCCATCCCAGGR:TTTCAATGTCGGGGGTCGCCHS2-2F:AGGAATCGCTGAAAACGCAACR:CACCAGACTTGCCAGGAACACHS2-3F:TGATACGCCCATTCTTCAACAR:GCGACTTACTCTATTGGCTTGGqRT-PCRCHS2-4F:GCTACTTAAAGACTTCATCCGAAGGR:CACTGTGGAATGACCACCTGTCHS2-5F:TCGATCGTGGGCAACAGAAAR:TCGCAGTTCTGCCAACATCTCHS2-6F:ACCAGGTACAACCGCGATACR:TTATTGATGCGCCGCTGAGACgEF-RTF:AGTCACCAAGGCTGCACAGAAAGR:TCCGACGTATTTCTTTGCGATGT
2 结果与分析
2.1 长牡蛎及其他物种中CHS基因家族的数量特征
从NCBI数据库获取了不同物种中CHS基因家族的序列信息,比较了真菌类、脊索动物、节肢动物、软体动物与假体腔动物中CHS基因家族的拷贝数。从表2可见,真菌类中CHS家族成员有5~8个亚家族(CHS-1、CHS-2、CHS-3、CHS-4、CHS-5、CHS-6、CHS-7、CHS-8),但仅在玉米黑粉菌中发现了全部CHS家族成员。多数节肢动物包含1~2个CHS基因家族成员,其中果蝇和赤拟谷盗均含有CHS-1和CHS-2,西方蜜蜂中含有CHS-2和CHS-6,而中华绒螯蟹中只含有CHS-2。在硬骨鱼、线虫和软体动物中只存在CHS-1和CHS-2,其中长牡蛎、厚壳贻贝、中华蛸存在CHS-1和CHS-2,虾夷扇贝只存在CHS-1(表2)。因此,各物种CHS基因家族成员数量上有所差别,仅真菌中CHS基因家族成员的拷贝数为5~8个,在其他物种中CHS-1和CHS-2拷贝数居多,说明CHS-1和CHS-2在进化关系中较为保守。
表2 不同物种CHS基因家族的拷贝数
Tab.2 Copy number of CHS genes family among different species
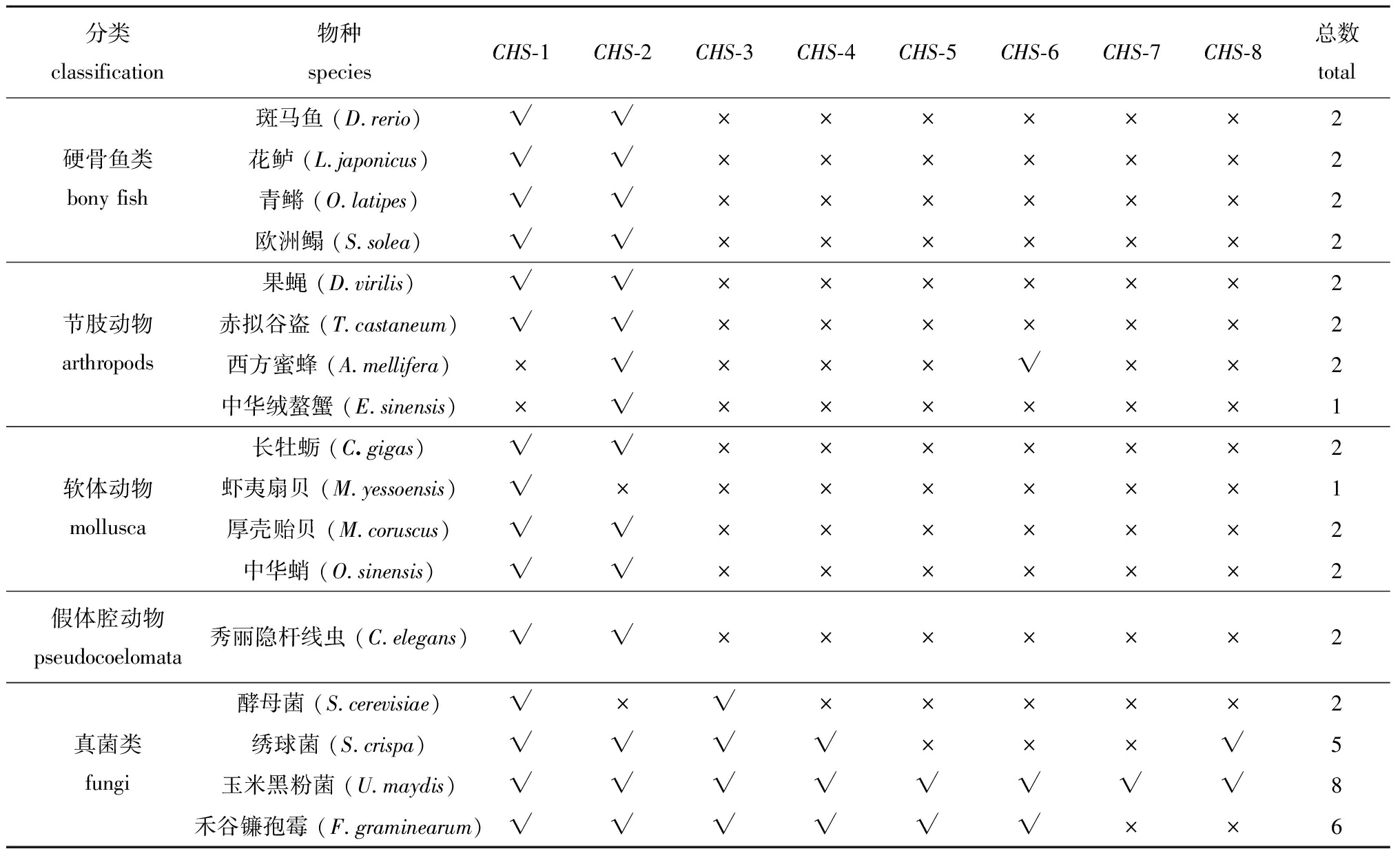
分类classification物种speciesCHS-1CHS-2CHS-3CHS-4CHS-5CHS-6CHS-7CHS-8总数total斑马鱼 (D.rerio)√√××××××2硬骨鱼类bony fish花鲈 (L.japonicus)√√××××××2青鳉 (O.latipes)√√××××××2欧洲鳎 (S.solea)√√××××××2果蝇 (D.virilis)√√××××××2节肢动物arthropods赤拟谷盗 (T.castaneum)√√××××××2西方蜜蜂 (A.mellifera)×√×××√××2中华绒螯蟹 (E.sinensis)×√××××××1长牡蛎 (C.gigas)√√××××××2软体动物mollusca虾夷扇贝 (M.yessoensis)√×××××××1厚壳贻贝 (M.coruscus)√√××××××2中华蛸 (O.sinensis)√√××××××2假体腔动物pseudocoelomata秀丽隐杆线虫 (C.elegans)√√××××××2酵母菌 (S.cerevisiae)√×√×××××2真菌类fungi绣球菌 (S.crispa)√√√√×××√5玉米黑粉菌 (U.maydis)√√√√√√√√8禾谷镰孢霉 (F.graminearum)√√√√√√××6
注:√表示此物种中存在该基因,×表示此物种缺失该基因。本研究所分析的物种为加粗字体。
Note:√ indicates the presence of a gene in the species;×indicates the gene was lost.The species analyzed in this study is in bold.
2.2 长牡蛎CHS基因家族数量和染色体定位
从长牡蛎基因组中筛选并鉴定出7个CHS基因,属于CHS-1和CHS-2两个亚家族,将其命名为CgCHS1(LOC105344864)、CgCHS2-1(LOC105341371)、CgCHS2-2(LOC105334325)、CgCHS2-3(LOC105333743)、长牡蛎具有10条染色体,CgCHS基因主要分布在2、3、4、7号染色体上。其中,CgCHS2-2和CgCHS2-6基因位于2号染色体上,CgCHS2-3和CgCHS2-4基因位于3号染色体上,CgCHS2-1和CgCHS2-5基因位于4号染色体上,CgCHS1基因位于7号染色体上(图1)。
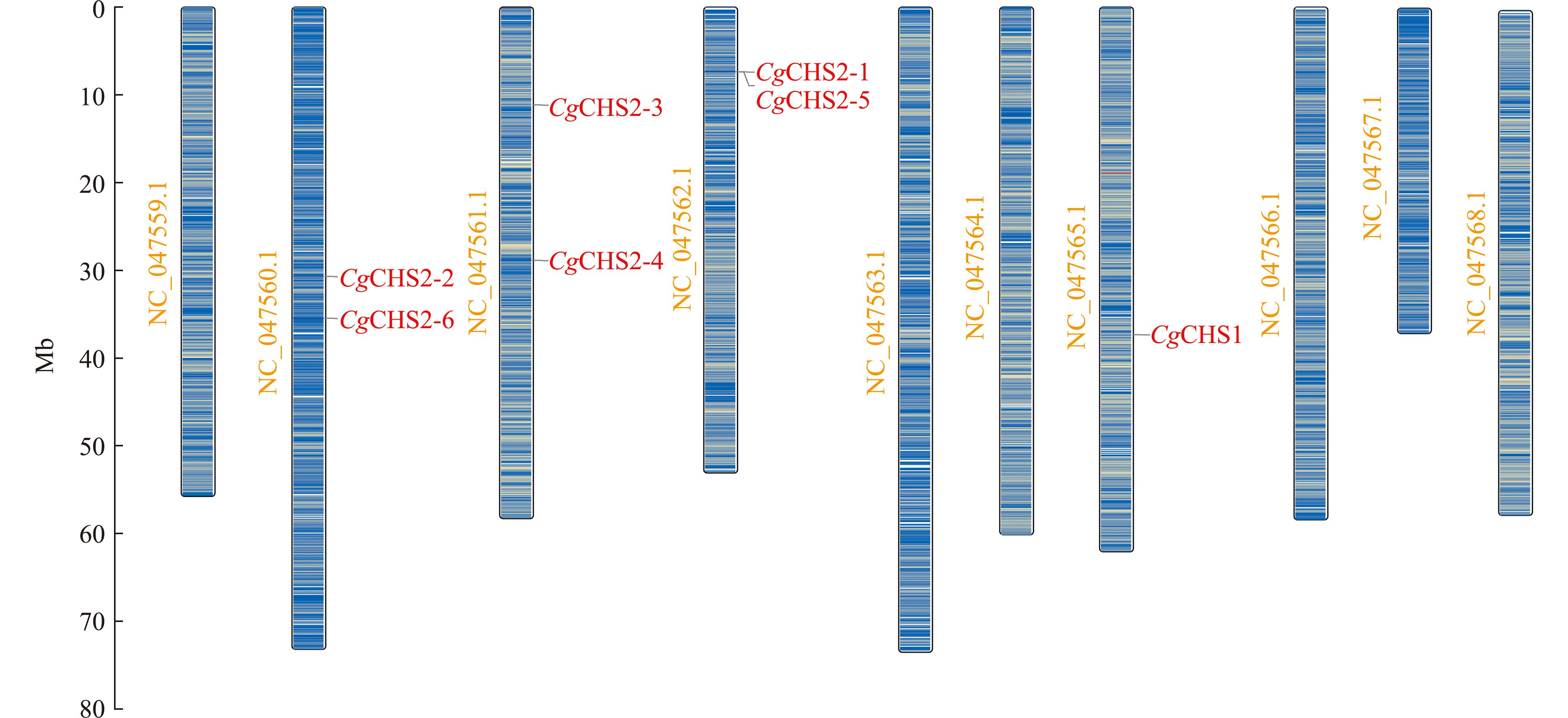
图1 CgCHS家族基因成员的染色体定位
Fig.1 Chromosomal localization of gene members of CgCHS family
CgCHS2-4(LOC105324613)、CgCHS2-5(LOC105341373)、CgCHS2-6(LOC105336663),基因长度分别是5 605、6 124、4 354、4 587、5 250、5 318、4 546(表3)。
表3 长牡蛎CHS家族基因信息
Tab.3 Information of CHS family genes among Crassostrea gigas
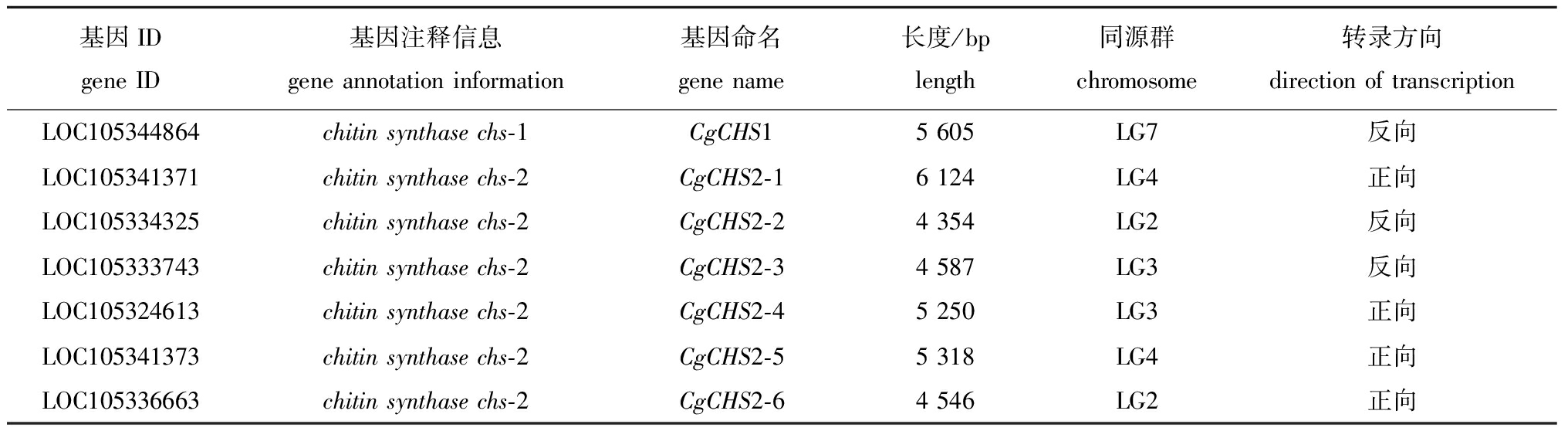
基因IDgene ID基因注释信息gene annotation information基因命名gene name长度/bplength同源群chromosome转录方向direction of transcriptionLOC105344864chitin synthase chs-1CgCHS15 605LG7反向LOC105341371chitin synthase chs-2CgCHS2-16 124LG4正向LOC105334325chitin synthase chs-2CgCHS2-24 354LG2反向LOC105333743chitin synthase chs-2CgCHS2-34 587LG3反向LOC105324613chitin synthase chs-2CgCHS2-45 250LG3正向LOC105341373chitin synthase chs-2CgCHS2-55 318LG4正向LOC105336663chitin synthase chs-2CgCHS2-64 546LG2正向
2.3 CgCHS家族蛋白的序列和结构特征
长牡蛎7个CgCHS基因编码蛋白(CgCHS1和CgCHS2-1~6)序列分别含有1 594、1 443、1 333、1 298、1 698、1 407和1 393个氨基酸,相对分子质量分别为182 700、165 600、151 300、150 600、193 900、161 500和158 600。不稳定系数分析表明,其中6个CgCHS蛋白(CgCHS2-1~6)的不稳定系数≥40,说明这些蛋白质并不稳定;只有CgCHS1蛋白的不稳定系数<40,说明该蛋白质相对稳定。CgCHS蛋白的亲水指数分析发现,CgCHS1、CgCHS2-1、CgCHS2-3、CgCHS2-4、CgCHS2-5和CgCHS2-6均为亲水性蛋白,只有CgCHS2-2为疏水性蛋白(表4)。
表4 长牡蛎CHS家族蛋白质信息
Tab.4 Information of CHS family protein among Crassostrea gigas
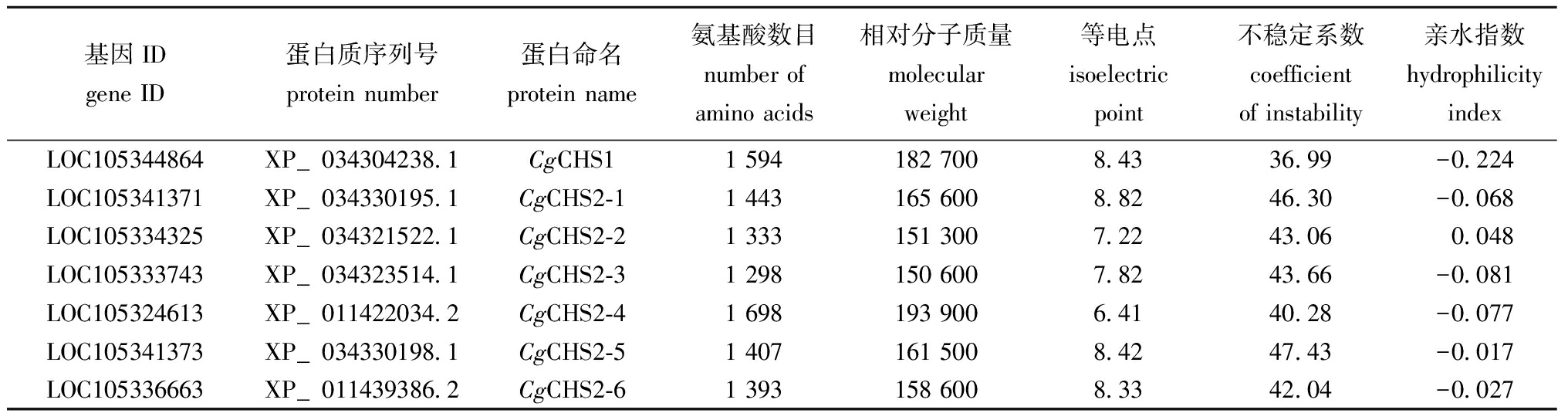
基因IDgene ID蛋白质序列号protein number蛋白命名protein name氨基酸数目number of amino acids相对分子质量molecular weight等电点isoelectric point不稳定系数coefficient of instability亲水指数hydrophilicity indexLOC105344864XP_034304238.1CgCHS11 594182 7008.4336.99-0.224LOC105341371XP_034330195.1CgCHS2-11 443165 6008.8246.30-0.068LOC105334325XP_034321522.1CgCHS2-21 333151 3007.2243.060.048LOC105333743XP_034323514.1CgCHS2-31 298150 6007.8243.66-0.081LOC105324613XP_011422034.2CgCHS2-41 698193 9006.4140.28-0.077LOC105341373XP_034330198.1CgCHS2-51 407161 5008.4247.43-0.017LOC105336663XP_011439386.2CgCHS2-61 393158 6008.3342.04-0.027
采用SMART在线工具预测CgCHS蛋白质的结构域。结果显示,CgCHS重要结构域保守,均含有Chitin_synth_2结构域及多个跨膜结构域(图2和图3)。通过对不同物种的CHS蛋白结构域分析,发现在真菌CHS蛋白有Chitin_synth_1N和Chitin_synth_2关键结构域及跨膜结构域,而在脊索动物、节肢动物、软体动物与假体腔动物中CHS只有Chitin_synth_2关键结构域和跨膜结构域。此外,节肢动物中西方蜜蜂的CHS蛋白还有RPT1(regulatory particle triple-aprotein1)结构域(图4)。
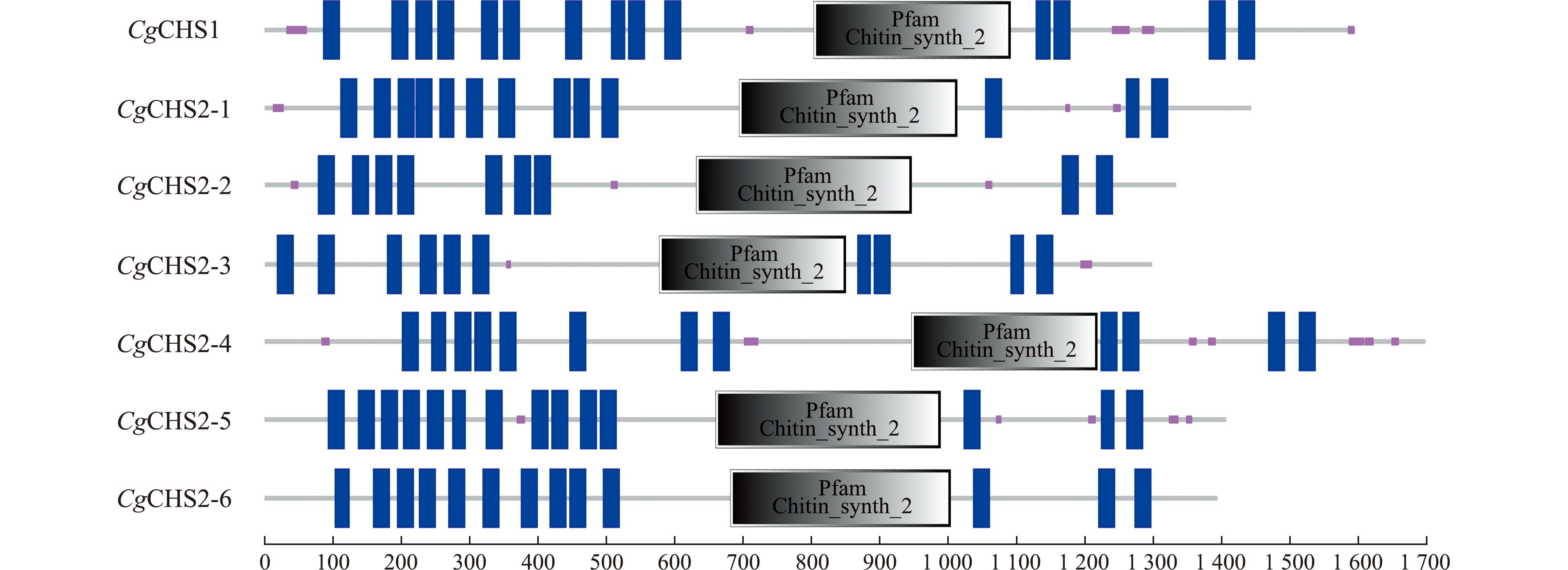
图2 通过SMART预测CgCHS蛋白质的结构域
Fig.2 Domains of CgCHS proteins were predicted by SMART
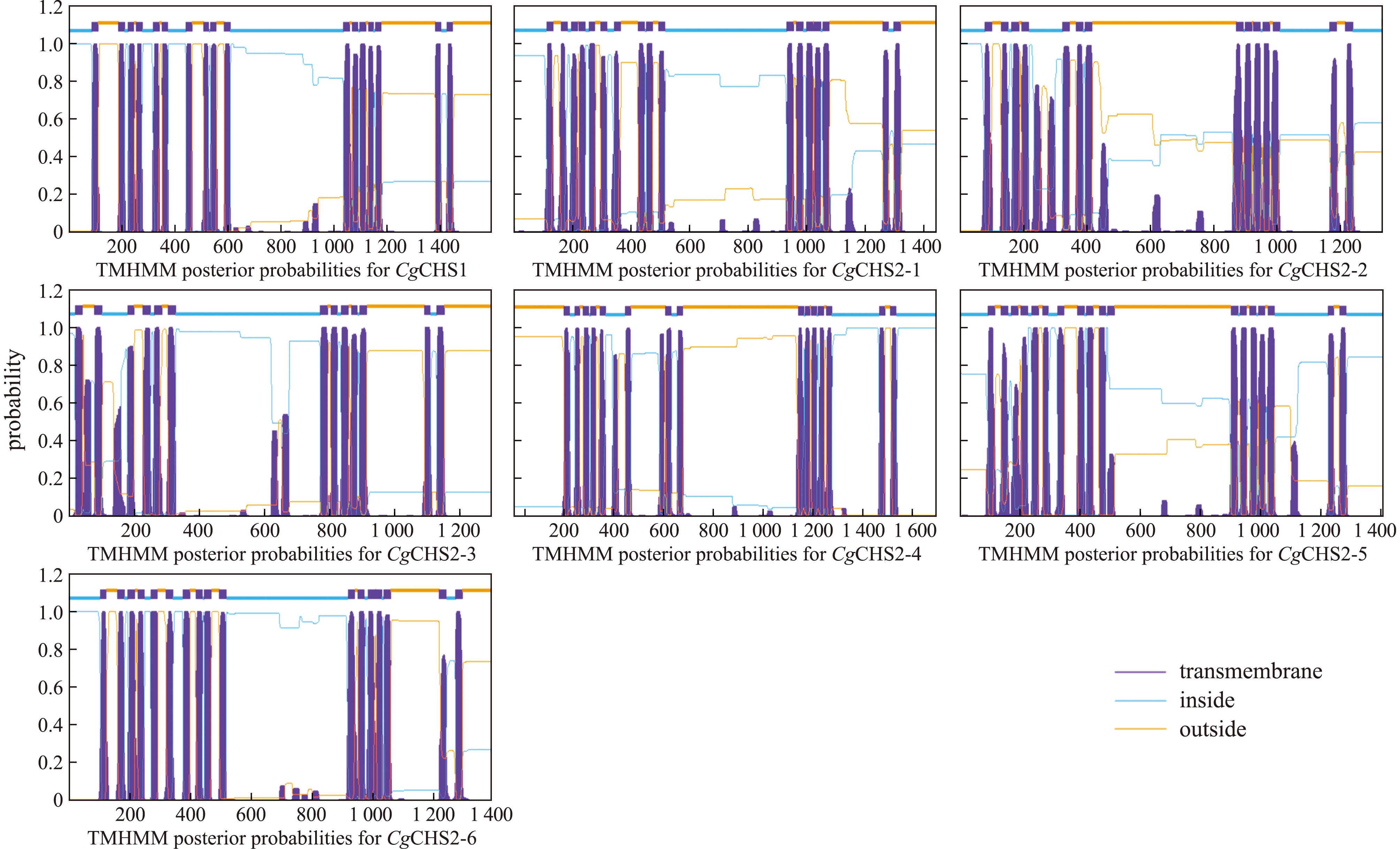
图3 通过TMHMM 2.0分析CgCHS家族蛋白跨膜区域
Fig.3 Transmembrane region of CgCHS family proteins was analyzed by TMHMM 2.0
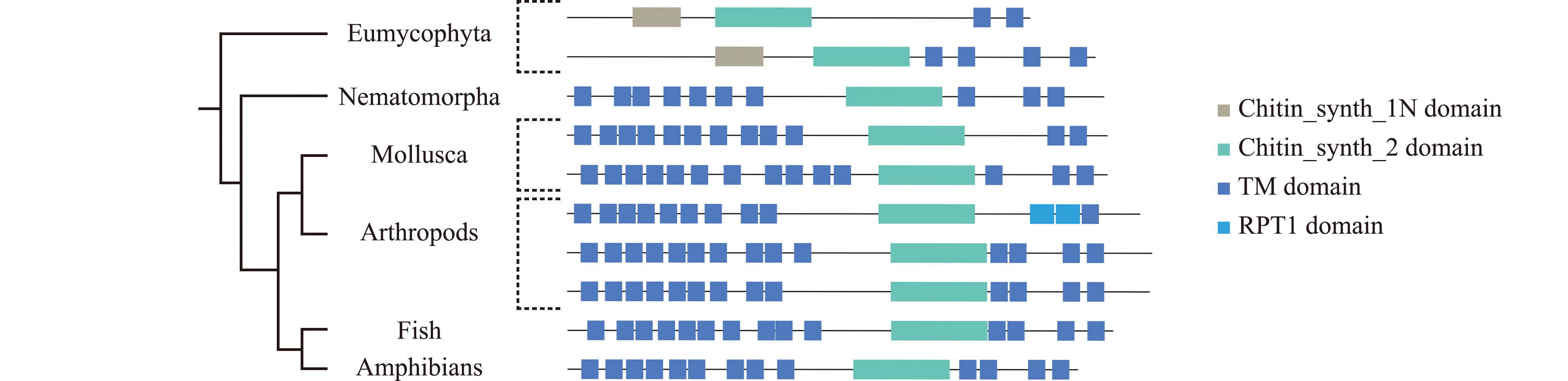
图4 不同物种CHS蛋白结构域的分析
Fig.4 Analysis of CHS protein domains in different species.
为了进一步解析长牡蛎CgCHS家族蛋白的结构特征,使用TBtools软件分析了7个CgCHS蛋白质氨基酸序列的Motif,发现不同CgCHS家族蛋白之间保守Motif基本相同,均存在Motif1~10,关键结构域区域Motif排列顺序相同,均以Motif5、Motif3、Motif6、Motif1、Motif10、Motif2、Motif7、Motif9、Motif4、Motif8的顺序排列,表明CgCHS家族蛋白的结构域在进化上具有极高的保守性和相似性(图5)。
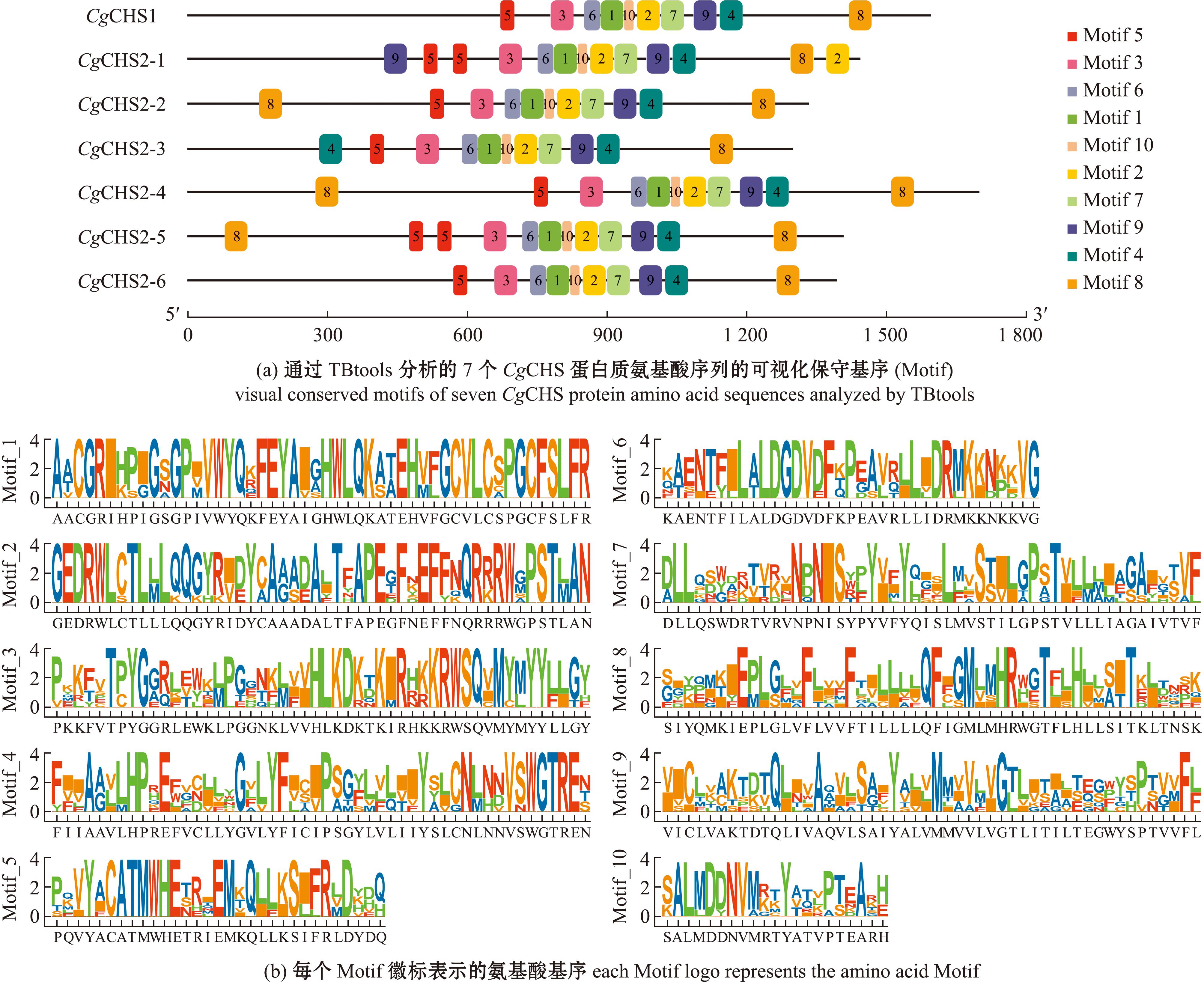
(a)中不同颜色方块代表不同的Motif徽标;(b)中Motif徽标由每个位置的一堆字母组成,字母的相对大小表示它们在序列中的频率。
(a),different colored squares represent different Motif logos;(b),motif logo consists of a bunch of letters for each location,the relative size of the letters indicates their frequency in the sequence.
图5 CgCHS家族基因保守Motif分析
Fig.5 Conserved Motif analysis of CgCHS family genes
2.4 CgCHS家族蛋白的系统进化关系
使用MEGA X构建了CHS家族蛋白的系统进化树,包括真菌[酿酒酵母(Saccharomyces cerevisiae S288c)、玉米黑粉菌(Mycosarcoma maydis)、拟轮枝镰孢(Fusarium verticillioides 7600)、绣球菌(Sparassis crispa)、粟酒裂殖酵母(Schizosaccharomyces pombe)]、硬骨鱼类[白斑狗鱼(Esox lucius)、白沙鳉(Cyprinodon tularosa)、伯氏肩孔南极鱼(Trematomus bernacchii)、斑马鱼、虹鳟(Oncorhynchus mykiss)、条纹狼鲈(Morone saxatilis)、泰国斗鱼(Betta splendens)、欧洲鳎]、软骨鱼类[噬人鲨(Carcharodon carcharias)]、软体动物[长牡蛎、神盾螺(G.aegis)、双斑蛸(O.bimaculatus)、澳洲黑边鲍、美洲牡蛎(C.virginica)、虾夷扇贝]、节肢动物[果蝇、按蚊(Anopheles merus)、家蚕(Bombyx mori)、棕榈蓟马(Thrips palmi)、褐飞虱(Nilaparvata lugens)、红火蚁(Solenopsis invicta)、颚蚁(Odontomachus Brunneus)、西方蜜蜂、克氏原螯虾(Procambarus clarkii)、美洲龙虾(Homarus americanus)、中华绒螯蟹、日本对虾(Penaeus japonicus)]、两栖纲[非洲爪蟾(Xenopus tropicalis)]及假体腔动物(秀丽隐杆线虫)。结果表明,不同门类物种CHS分别聚为大的分枝。长牡蛎CgCHS主要与其他软体动物CHS聚为一支,包括神盾螺、双斑蛸、澳洲黑边鲍、美洲牡蛎和虾夷扇贝。其中,长牡蛎CgCHS1与美洲牡蛎CvCHS-1聚为一支,其次和双斑蛸ObCHS-1聚为一支。CgCHS2-2、CgCHS2-3、CgCHS2-4、CgCHS2-6与神盾螺GaCHS聚为一支。有趣的是,CgCHS2-1、CgCHS2-5与脊索动物CHS聚为一支(图6)。
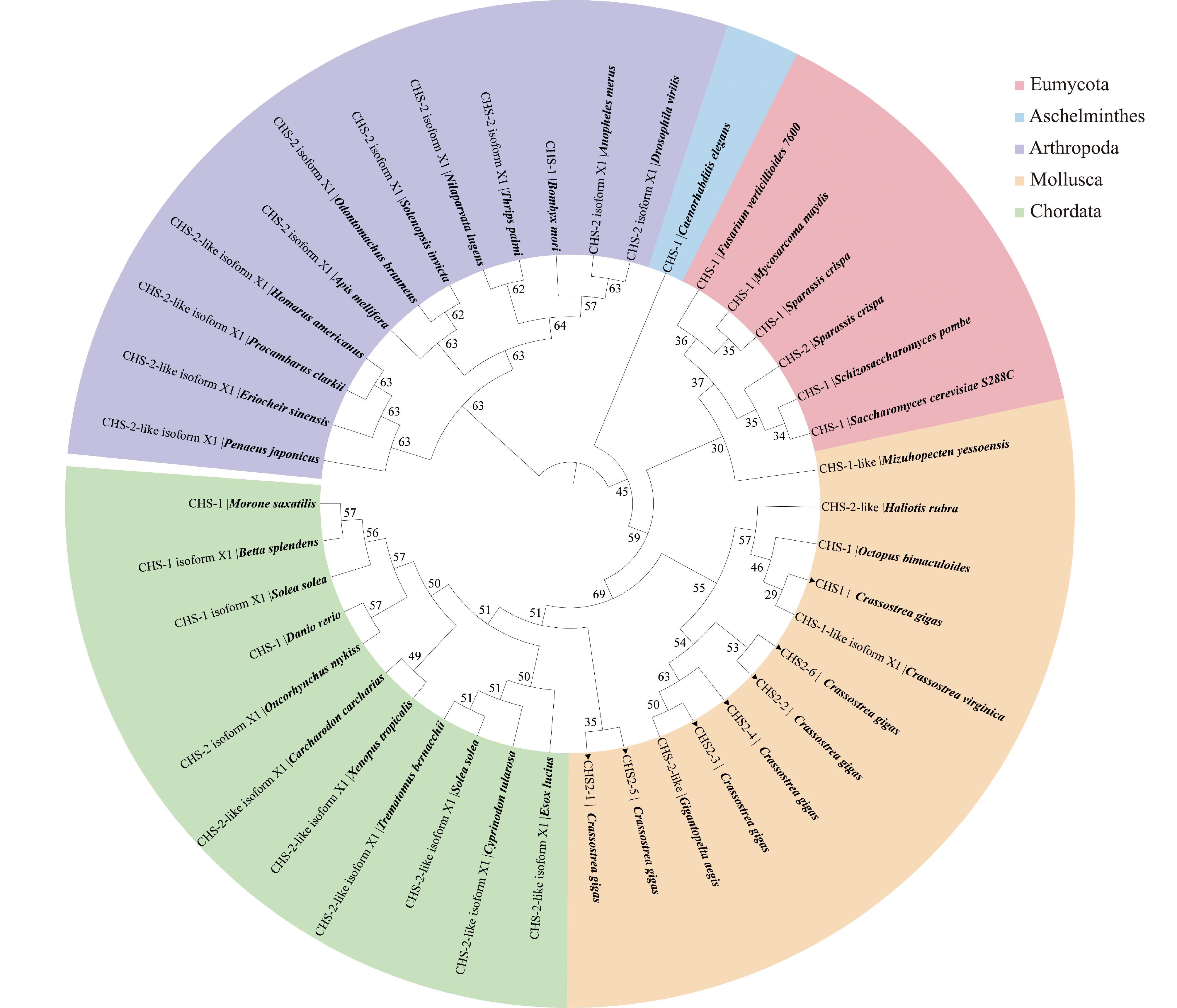
图6 不同物种CHS氨基酸序列的系统进化树
Fig.6 Phylogenetic tree of amino acid sequences of CHS in different species
2.5 CgCHS家族基因在长牡蛎早期发育过程中的表达规律
利用qRT-PCR检测了7个CgCHS基因在长牡蛎幼虫不同发育阶段(受精卵、4细胞期、8细胞期、桑椹胚期、囊胚期、原肠胚期、担轮幼虫期和D型幼虫期)的表达情况。其中,担轮幼虫和D型幼虫时期是长牡蛎壳形成的关键时期。从图7可见,除了CgCHS2-6外(图7(g)),其余6个CgCHS基因在长牡蛎整个早期发育阶段均有表达。其中,CgCHS1的表达量在囊胚期开始升高,在原肠胚时期最高,约为受精卵时期的5.15倍,在担轮幼虫时期降低(图7(a));CgCHS2-1在8细胞期开始表达,其表达量在囊胚期最高,约为受精卵时期的89倍,在原肠胚和担轮幼虫时期均呈现出降低的趋势,在D型幼虫时期再次升高(图7(b));CgCHS2-2在表达量担轮幼虫前期开始升高,在担轮幼虫后期降低,在D型幼虫时期最高,约为受精卵时期的559倍(图7(c));CgCHS2-3在担轮幼虫前期开始表达,在担轮幼虫后期表达量逐渐升高,在D型幼虫时期最高,约为受精卵时期的1 460倍(图7(d));CgCHS2-4表达量在囊胚期开始上升,在担轮幼虫后期达到最高,约为受精卵时期的153倍,在D型幼虫时期降低(图7(e));CgCHS2-5在担轮幼虫后期表达量达到最高,约为受精卵时期的3.29倍(图7(f));CgCHS2-6在长牡蛎幼虫整个发育时期中表达量均处于较低的趋势(图7(g))。
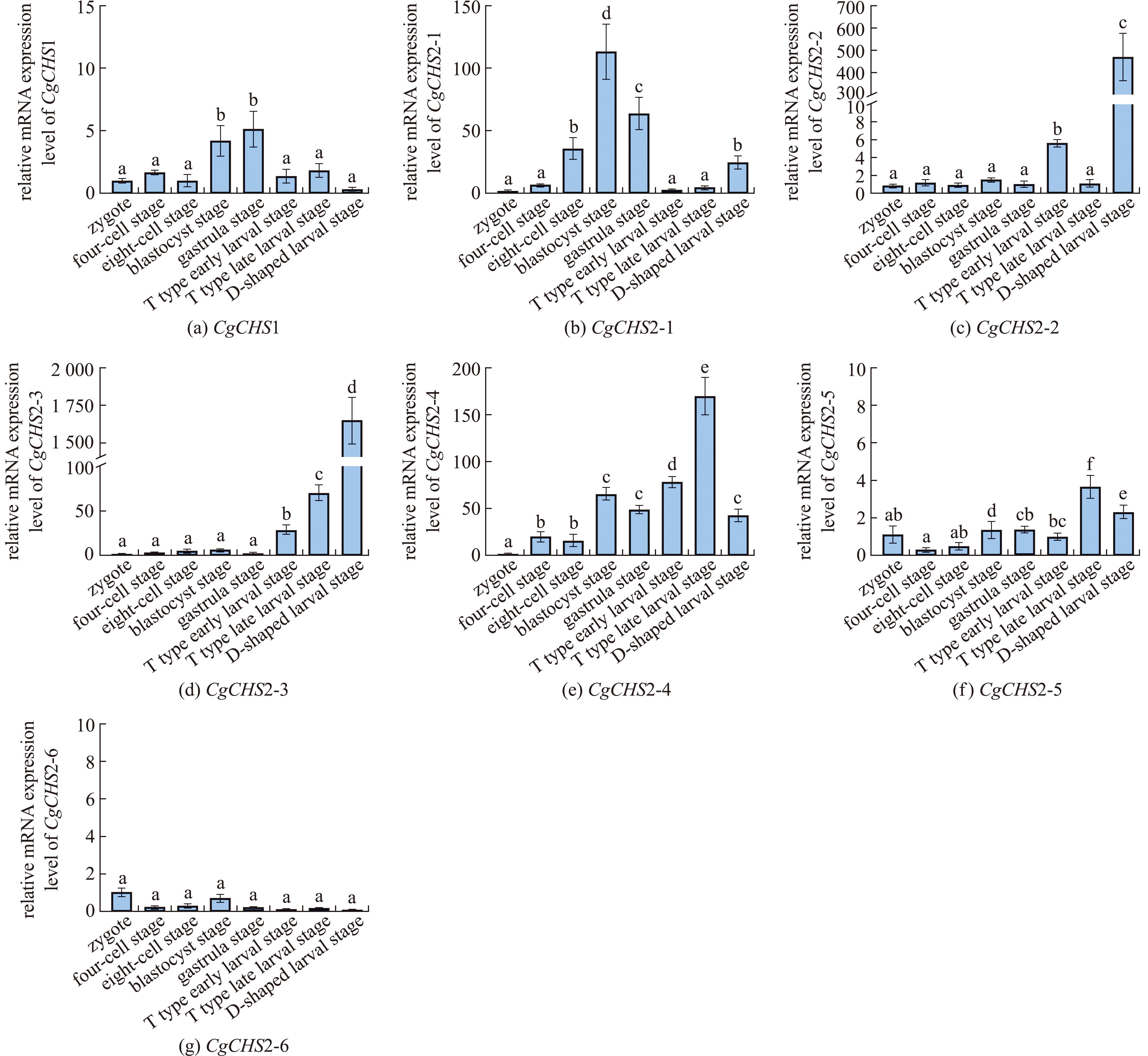
图7 CgCHS家族在长牡蛎早期发育过程中的表达
Fig.7 Expression of the CgCHS family during early development of Crassostrea gigas
综上所述,在长牡蛎壳形成的关键时期担轮幼虫和D型幼虫时期中,CgCHS2-2、CgCHS2-3、CgCHS2-4、CgCHS2-5表达量均出现上调;CgCHS1、CgCHS2-1主要在囊胚期表达量升高;CgCHS2-6表达量在整个发育时期均处于较低水平。
3 讨论
CHS是几丁质代谢过程中的关键酶,在贝类幼虫壳基质的生物合成中发挥重要作用[22]。CHS主要通过催化UDP-GlcNAc生物结合形成几丁质,随后几丁质与大分子物质结合,构建生物框架,参与生物矿化沉积形成钙质贝壳。已有研究表明,CHS在珍珠贝(Pinctada fucata martensii)[23]和紫贻贝(Mytilus galloprovincialis)[19]的壳形成过程中起关键作用。本研究中系统筛查了长牡蛎的CHS基因家族,解析了其分子和进化特征,并检测了其在长牡蛎幼虫壳形成中的表达模式,旨在为研究CgCHS在长牡蛎幼虫生长发育中的动态调控机制提供科学依据。
3.1 CgCHS家族的结构和进化特征
CHS基因家族几乎存在于所有真核生物中,但不同物种中其成员数量和主要生理学功能存在显著差异。有研究首先在真菌中鉴定出8种CHS[16],而在节肢动物和软体动物中仅鉴定出2种CHS[24]。本研究中通过基因组数据库鉴定了多个物种中的CHS家族成员,结果显示,各物种CHS家族成员数量上差别较大,如真菌中CHS基因家族的拷贝数为5~8个,而在其他物种(包括长牡蛎)则主要存在CHS-1和CHS-2两个拷贝。几丁质是真菌细胞壁及隔膜的重要结构成分,因此CHS在真菌中被广泛研究。CHS是催化几丁质合成的关键酶,通过催化UDP-GlcNAc生物合成几丁质从而指导真菌细胞壁的合成[16]。由于几丁质在植物和哺乳动物体内无法合成,几丁质的生物合成被认为是研发杀菌剂、杀虫剂和抗真菌药物的重要靶点[25-26]。大部分物种具有CHS-1和CHS-2两种几丁质合成酶,这可能是由于它们在基因转录调控、基因家族演化和生物学功能上的差异和分化[27]。在真菌中CHS分支Ⅰ和分支Ⅱ的区别在于结构域的差异。具体而言,CHS分支Ⅱ含有一个Cyt-b5结构域,说明它除了参与几丁质的合成,还可能参与脂肪酸的合成和代谢、胆固醇的生物合成、氨基酸的代谢等生物化学过程[17]。因此,在物种进化过程中,真菌中CHS分支Ⅱ发挥的作用可能不断减弱,并伴随基因缺失现象的发生,从而导致大多数物种中仅保留了CHS分支Ⅰ的成员CHS-1和CHS-2。在长牡蛎幼虫发育过程中,从担轮幼虫时期的几丁质壳向D-型幼虫的钙质壳的沉积过程是长牡蛎形成钙质壳的关键过程[28],这个期间能否形成均匀的钙质壳是长牡蛎幼虫能否存活的关键[29-30]。在长牡蛎基因组数据库中,根据关键结构域共鉴定得到2个CHS基因家族成员CHS-1和CHS-2,其中筛选出了7个CHS基因,分别位于2、3、4和7号染色体上。CHS家族蛋白序列和结构特征相对保守。系统进化分析显示,长牡蛎的CgCHS家族与无脊椎动物CHS间存在一定的相似性和同源性,预示在长牡蛎的进化过程中可能存在多样性的适应性进化。
CHS通常包括Chitin_synth_1、Chitin_synth_1N和Chitin_synthase_2保守结构域及多个跨膜结构域[17]。Chitin_synthase_2保守结构域主要发挥生物合成几丁质的功能,其主要参与将GlcNAc从UDP-GlcNAc转移到生长中的几丁质链上的过程[25]。多个跨膜结构域说明CHS为膜结合蛋白,能够调节细胞膜信号传导。在家蚕、西方蜜蜂、果蝇和黄粉虫(Tenebrio molitor)等昆虫的CHS氨基酸序列中均鉴定到1个Chitin_synth_2保守结构域[31-32]。相似地,长牡蛎CgCHS均有保守的Chitin_synth_2结构域,且关键结构域区域Motif以相同的顺序排列。真菌中有Chitin_synth_1、Chitin_synth_1N和Chitin_synth_2关键结构域及跨膜结构域。节肢动物中西方蜜蜂AmCHS蛋白还有RPT1结构域,在长牡蛎等软体动物及脊索动物、假体腔动物中未发现Chitin_synth_1N和RPT1结构域,说明Chitin_synth_2关键结构域在进化关系上较保守。Chitin_synth_1是几丁质合成酶的核心催化结构域,而Chitin_synth_1N和Chitin_synth_2则是与之相关的辅助结构域,它们可能在酶的定位、稳定性、调控或特定的生物学功能中发挥作用[33]。这3个结构域的序列、结构和功能的差异共同决定了几丁质合成酶的多样性和它们在不同生物体中的适应性[25]。
理化性质分析显示,CgCHS2-1~6蛋白质并不稳定,只有CgCHS1蛋白质相对稳定,CgCHS2-2为疏水性蛋白,其余均为亲水性蛋白。由于CHS在不同物种中广泛存在及其多样性,其亲水性特性可能与其在特定物种中的生物学功能紧密相关,如西方蜜蜂等昆虫中CHS为亲水性蛋白[32]。在软体动物和环节动物中,几丁质的作用与它们在湿润环境中的生存和结构完整性有关[34]。因此,多数CHS是亲水性蛋白,其功能依赖于水环境,且CHS合成的几丁质在湿润条件下才能维持其结构和功能的稳定性。CHS被归类为糖基转移酶家族2(GTF2)[35],包含来自细菌、真菌、植物和动物的序列,它能够催化糖基从活化的糖供体转移到特定的受体,从而形成糖苷键。虽然大多数糖基转移酶催化的化学反应相似,但它们在供体、受体和产物特异性方面表现出显著的多样性,从而形成大量不同的糖缀合物、低聚糖和多糖[36]。CHS的N-末端和C-末端残基即使在同一物种的同种型中也可能不同,但这两个基序含有大量的酸性或碱性残基,这是各种软体动物CHS的主要特征[20]。此外,细胞内的关键结构域高度保守,具有催化几丁质合成的功能,在软体动物的精确控制矿物沉积过程中起着关键作用[20]。
3.2 CgCHS家族在长牡蛎早期发育过程中的表达模式分析
双壳贝类早期壳形成过程在不同发育时期受到不同CHS的调控。双壳贝类壳形成起源于囊胚期形成的壳腺,随后壳腺在早期担轮幼虫时期通过向外翻转形成壳场,随着胚胎的发育壳场在担轮幼虫时期形成骨膜,且出现了两个贝壳的雏形,在D型幼虫时期完成壳的钙质化[11]。通过对CgCHS基因家族成员进行长牡蛎早期发育过程中的表达分析发现,在长牡蛎壳形成的关键时期,如担轮幼虫和D型幼虫时期中,CgCHS2-2、CgCHS2-3、CgCHS2-4、CgCHS2-5表达量均出现上调,推测其参与幼虫壳形成及几丁质壳向钙质壳转化的过程;CgCHS1、CgCHS2-1主要在囊胚期表达量上调,推测其参与了囊胚期壳腺的形成;CgCHS2-6表达量在整个发育时期都处于较低的趋势,推测其可能不参与长牡蛎幼虫壳形成。双壳类在幼虫发育过程中形成两种类型的壳,即胚壳Ⅰ(几丁质壳)和胚壳Ⅱ(钙质壳)[37]。胚壳Ⅰ是由担轮幼虫时期幼虫背部外胚层细胞(壳腺)分泌的有机成分形成的[38],随后,幼虫壳胚壳Ⅱ由D型幼虫外套膜生成[37]。在紫贻贝幼虫发育过程中几丁质最开始是由囊胚期形成的壳腺构成,随后翻转为担轮幼虫时期形成的壳场[12]。相似的,CgCHS最先开始表达于囊胚期,其是壳形成的开始时期,在壳形成的关键时期担轮幼虫时期表达量显著上升,其是几丁质壳形成及向钙质壳转化的开始时期。根据不同CgCHS基因的不同表达模式,推测其基因表达差异的主要原因可能是长牡蛎中存在CgCHS的数量较多,在同一个体中,这些基因可能呈现交替表达的现象。因此长牡蛎CgCHS基因在早期壳发育过程中展现了多样化的表达模式。
4 结论
1)长牡蛎中存在两个CgCHS家族(CgCHS1和CgCHS2),共包含7个CgCHS基因家族成员(CgCHS1和CgCHS2-1~6),它们不均匀的分布在4条染色体上。
2)长牡蛎CgCHS表达蛋白结构域保守,均包含Chitin_synth_2关键结构域,含有10个相似的Motif(Motif1~10)基序,有着相似的理化性质,进化关系均有较高的保守性。
3)7个CgCHS在长牡蛎早期壳形成过程中的不同阶段呈现出不同的表达模式。CgCHS1和CgCHS2-1在囊胚期表达水平显著升高,可能参与了囊胚期壳腺的形成;CgCHS2-2~5在担轮幼虫和D型幼虫时期表达水平显著升高,可能参与几丁质壳向钙质壳转化的过程。通过对长牡蛎CgCHS基因家族的分子和表达特性分析,将为后期研究CgCHS基因如何动态调控长牡蛎幼虫生长发育及几丁质壳转化为钙质壳的相关机制提供借鉴。
[1] 杨梅.长牡蛎(Crassostrea gigas)fox基因家族和Wnt信号通路相关基因的进化及表达分析[D].北京:中国科学院大学,2014. YANG M.Evolution and expression analysis of fox gene family and Wnt signaling pathway related genes in Crassostrea gigas[D].Beijing:University of Chinese Academy of Sciences,2014.(in Chinese)
[2] NAKAHARA H,BEVELANDER G.Ingestion of particulate matter by the outer surface cells of the mollusc mantle[J].Journal of Morphology,1967,122(2):139-145.
[3] BIELEFELD U,BECKER W.Embryonic development of the shell in Biomphalaria glabrata (Say)[J].The International Journal of Developmental Biology,1991,35(2):121-131.
[4] ADDADI L,JOESTER D,NUDELMAN F,et al.Mollusk shell formation:a source of new concepts for understanding biomineralization processes[J].Chemistry - A European Journal,2006,12(4):980-987.
[5] LIU X J,ZHANG J Z,ZHU K Y.Chitin in arthropods:biosynthesis,modification,and metabolism[M]//Targeting Chitin-containing Organisms.Singapore:Springer Singapore,2019:169-207.
[6] KAYA M,MUJTABA M,EHRLICH H,et al.On chemistry of γ-chitin[J].Carbohydr Polym,2017,176:177-186.
[7] LAVALL R L,ASSIS O B G,CAMPANA-FILHO S P.β-chitin from the pens of Loligo sp.:extraction and characterization[J].Bioresource Technology,2007,98(13):2465-2472.
[8] MOHAN K,RAVICHANDRAN S,MURALISANKAR T,et al.Extraction and characterization of chitin from sea snail Conus inscriptus (Reeve,1843)[J].International Journal of Biological Macromolecules,2019,126:555-560.
[9] SUZUKI M,SAKUDA S,NAGASAWA H.Identification of chitin in the prismatic layer of the shell and a chitin synthase gene from the Japanese pearl oyster,Pinctada fucata[J].Bioscience,Biotechnology,and Biochemistry,2007,71(7):1735-1744.
[10] SUDATTA B P,SUGUMAR V,VARMA R,et al.Extraction,characterization and antimicrobial activity of chitosan from pen shell,Pinna bicolor[J].International Journal of Biological Macromolecules,2020,163:423-430.
[11] NIELSEN C.Trochophora larvae:Cell-lineages,ciliary bands,and body regions.1.Annelida and Mollusca[J].Journal of Experimental Zoology Part B:Molecular and Developmental Evolution,2004,302B(1):35-68.
[12] ZHANG G F,FANG X D,GUO X M,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[13] 唐斌,张露,熊旭萍,等.海藻糖代谢及其调控昆虫几丁质合成研究进展[J].中国农业科学,2018,51(4):697-707. TANG B,ZHANG L,XIONG X P,et al.Advances in trehalose metabolism and its regulation of insect chitin synthesis[J].Scientia Agricultura Sinica,2018,51(4):697-707.(in Chinese)
[14] TANG B,CHEN J,YAO Q,et al.Characterization of a trehalose-6-phosphate synthase gene from Spodoptera exigua and its function identification through RNA interference[J].Journal of Insect Physiology,2010,56(7):813-821.
[15] CHEN J,TANG B,CHEN H X,et al.Different functions of the insect soluble and membrane-bound trehalase genes in chitin biosynthesis revealed by RNA interference[J].PLoS One,2010,5(4):e10133.
[16] LARSON T M,KENDRA D F,BUSMAN M,et al.Fusarium verticillioides chitin synthases CHS5 and CHS7 are required for normal growth and pathogenicity[J].Current Genetics,2011,57(3):177-189.
[17] CUI Z F,WANG Y H,LEI N,et al.Botrytis cinerea chitin synthase BcChsVI is required for normal growth and pathogenicity[J].Current Genetics,2013,59(3):119-128.
[18] ZHANG Y K,LIU Z Q,SONG X R,et al.The inhibition of ocean acidification on the formation of oyster calcified shell by regulating the expression of Cg chs1 and Cg chit4[J].Frontiers in Physiology,2019,10:1034.
[19] MIGLIOLI A,DUMOLLARD R,BALBI T,et al.Characterization of the main steps in first shell formation in Mytilus galloprovincialis:possible role of tyrosinase[J].Proceedings Biological Sciences,2019,286(1916):20192043.
[20] SCHÖNITZER V,EICHNER N,CLAUSEN-SCHAUMANN H,et al.Transmembrane myosin chitin synthase involved in mollusc shell formation produced in Dictyostelium is active[J].Biochemical and Biophysical Research Communications,2011,415(4):586-590.
[21] FENG D D,LI Q,YU H,et al.Identification of conserved proteins from diverse shell matrix proteome in Crassostrea gigas:characterization of genetic bases regulating shell formation[J].Scientific Reports,2017,7:45754.
[22] WEISS I M,SCHÖNITZER V.The distribution of chitin in larval shells of the bivalve mollusk Mytilus galloprovincialis[J].Journal of Structural Biology,2006,153(3):264-277.
[23] ZHANG Y T,JIAO Y,LI Y P,et al.Comprehensive analysis of microRNAs in the mantle central and mantle edge provide insights into shell formation in pearl oyster Pinctada fucata martensii[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2021,252:110508.
[24] PENG M X,CARDOSO J C R,POWER D M.Evolution of chitin-synthase in molluscs and their response to ocean acidification[J].Molecular Phylogenetics and Evolution,2024,201:108192.
[25] CHEN W,CAO P,LIU Y S,et al.Structural basis for directional chitin biosynthesis[J].Nature,2022,610(7931):402-408.
[26] 韩琦,王铌翔.几丁质合酶在人类致病真菌中的研究进展[J].微生物学报,2024,64(1):98-107. HAN Q,WANG N X.Progress in chitin synthases of human pathogenic fungi[J].Acta Microbiologica Sinica,2024,64(1):98-107.(in Chinese)
[27] WANG Y,FAN H W,HUANG H J,et al.Chitin synthase 1 gene and its two alternative splicing variants from two sap-sucking insects,Nilaparvata lugens and Laodelphax striatellus (Hemiptera:Delphacidae)[J].Insect Biochemistry and Molecular Biology,2012,42(9):637-646.
[28] STENZEL H B.Oysters:composition of the larval shell[J].Science,1964,145(3628):155-156.
[29] BARTON A,HALES B,WALDBUSSER G G,et al.The Pacific oyster,Crassostrea gigas,shows negative correlation to naturally elevated carbon dioxide levels:implications for near-term ocean acidification effects[J].Limnology and Oceanography,2012,57(3):698-710.
[30] WALDBUSSER G G,BRUNNER E L,HALEY B A,et al.A developmental and energetic basis linking larval oyster shell formation to acidification sensitivity[J].Geophysical Research Letters,2013,40(10):2171-2176.
[31] LI L,WANG Y Q,LI G Y,et al.Genomic and transcriptomic analyses of chitin metabolism enzymes in Tenebrio molitor[J].Archives of Insect Biochemistry and Physiology,2022,111(3):e21950.
[32] 郭意龙,赵萧,张天泽,等.西方蜜蜂几丁质合成酶基因的生物信息学分析及功能研究[J].昆虫学报,2023,66(4):469-477. GUO Y L,ZHAO X,ZHANG T Z,et al.Bioinformatic analysis and functional study on chitin synthase gene in Apis mellifera[J].Acta Entomologica Sinica,2023,66(4):469-477.(in Chinese)
[33] 陈宇,唐英琪,王含蕊,等.金针菇几丁质合成酶基因家族鉴定及其表达分析[J].微生物学通报,2024,51(2):612-625. CHEN Y,TANG Y Q,WANG H R,et al.Characterization and expression analysis of chitin synthase family in Flammulina filiformis[J].Microbiology China,2024,51(2):612-625.(in Chinese)
[34] LI M,JIANG C,WANG Q H,et al.Evolution and functional insights of different ancestral orthologous clades of chitin synthase genes in the fungal tree of life[J].Frontiers in Plant Science,2016,7:37.
[35] MERZENDORFER H.Insect chitin synthases:a review[J].Journal of Comparative Physiology B,Biochemical,Systemic,and Environmental Physiology,2006,176(1):1-15.
[36] COUTINHO P M,DELEURY E,DAVIES G J,et al.An evolving hierarchical family classification for glycosyltransferases[J].Journal of Molecular Biology,2003,328(2):307-317.
[37] ZHAO R,TAKEUCHI T,LUO Y J,et al.Dual gene repertoires for larval and adult shells reveal molecules essential for molluscan shell formation[J].Molecular Biology and Evolution,2018,35(11):2751-2761.
[38] ARANDA-BURGOS J A,DA COSTA F,N VOA S,et al.Embryonic and larval development of Ruditapes decussatus (Bivalvia:Veneridae):a study of the shell differentiation process[J].Journal of Molluscan Studies,2014,80(1):8-16.
VOA S,et al.Embryonic and larval development of Ruditapes decussatus (Bivalvia:Veneridae):a study of the shell differentiation process[J].Journal of Molluscan Studies,2014,80(1):8-16.