辽河口位于辽东湾北部,附近有辽河、大辽河、大凌河和小凌河等入海河流,营养盐充足、饵料生物丰富,是重要的渔业生产区域,同时也是辽东湾北部众多渤海经济鱼类的索饵场和产卵场[1]。近年来,受环境污染、过度开发及人类活动等因素的影响,辽河口水域鱼类多样性急剧下降,甚至出现鱼类群体小型化、低龄化等现象[2]。因此,及时准确掌握辽河口海域鱼类生物多样性变化,对维护辽东湾乃至渤海区渔业生物种群的稳定性及渔业生态健康可持续发展均具有重要意义。
目前,中国开展海洋渔业资源调查评估的主要手段有底拖网调查法、声学调查法和潜水观测调查法等,但这些方法均具有一定的局限性[3]。例如,底拖网调查法成本高、对资源环境的破坏性较强、覆盖面局限性大(即无法覆盖礁石区、浅水区等);声学调查法和潜水观测调查法对设备和操作人员的要求高、受天气的局限性强(如风浪天气、水质浑浊等)。而环境DNA(environmental DNA,eDNA)技术作为一种新兴的高通量测序技术,因其采样成本低、普适性强、监测灵敏度高、对生境破坏性小等特点已在水生生态系统鱼类生物多样性调查与监测中得到广泛应用[4-6],在渔业资源调查中具有很大应用潜力,可作为传统渔业资源调查方法的有效补充。eDNA是指生物体的皮肤、黏液、唾液、粪便、尿液和腐烂体等释放到外界环境介质中的游离DNA分子或片段[7]。eDNA技术是指通过定量PCR和高通量测序,对环境样品中提取的eDNA进行定性和定量分析,从而推测和评估目标环境中生物的存在、种类、丰度和分布[8-9]。目前,eDNA技术已广泛应用于海洋鱼类资源调查评估。相关研究中,Stoeckle等[10]同时使用底拖网捕捞和eDNA技术对美国新泽西海岸的鱼类资源进行了调查;Thomsen等[11]运用eDNA技术与底拖网技术结合对极地和深海区域的鱼类进行了种类鉴定和生物量评估;言苛程等[12]运用eDNA技术与底拖网技术相结合的方式对南黄海西部鱼类多样性进行调查;另有诸多学者运用eDNA技术与底拖网调查相结合的方式分别对黄河口、长江口和珠江口的鱼类生物多样性进行了调查[13-15]。
迄今,运用eDNA技术对辽河口及其邻近海域鱼类多样性调查的研究还比较缺乏,仅在崔晓宇等[16]基于eDNA宏条形码对辽东半岛重要河口鱼类多样性研究中有所涉及,但其仅在辽河口区域设置了1个eDNA调查站位且缺乏底拖网调查数据,不能准确全面地反映辽河口及其邻近海域鱼类多样性的真实情况。本研究中在辽河口及其邻近海域同时开展了eDNA采样和拖网调查,通过对拖网调查结果与eDNA分析结果的比较,揭示了该海域鱼类种类、丰度和多样性分布特征,旨在为评价辽河口及其邻近海域鱼类种群动态变化及渔业资源可持续利用提供科学依据。
1 材料与方法
1.1 拖网调查样品采集及处理
拖网调查采样于2022年10月20—23日进行,调查区域位于辽河口及其邻近海域,站位设置如图1所示,调查网具为单船有翼单囊拖网(网口宽度为10 m,网口高度为3 m,囊网网目为20 mm),拖速为3 kn,每站拖网时间为30 min。使用YSI EXO多参数水质检测仪检测每个站位的水深、温度、盐度、pH和溶氧等水环境因子。渔获样品带回实验室进行分析处理,渔获种类鉴定到种,并对每种鱼类进行称量(精确到0.01 g)和数量统计,将数据量化成相对渔获质量(g/h)和相对渔获数量(ind./h)进行分析。采样及样品分析方法参照《海洋调查规范》(GB/T 12763.6—2007)。鱼类鉴定及区系划分参照《辽宁省动物志·鱼类》[17]《中国海洋生物名录》[18]和《中国海洋鱼类》[19]。
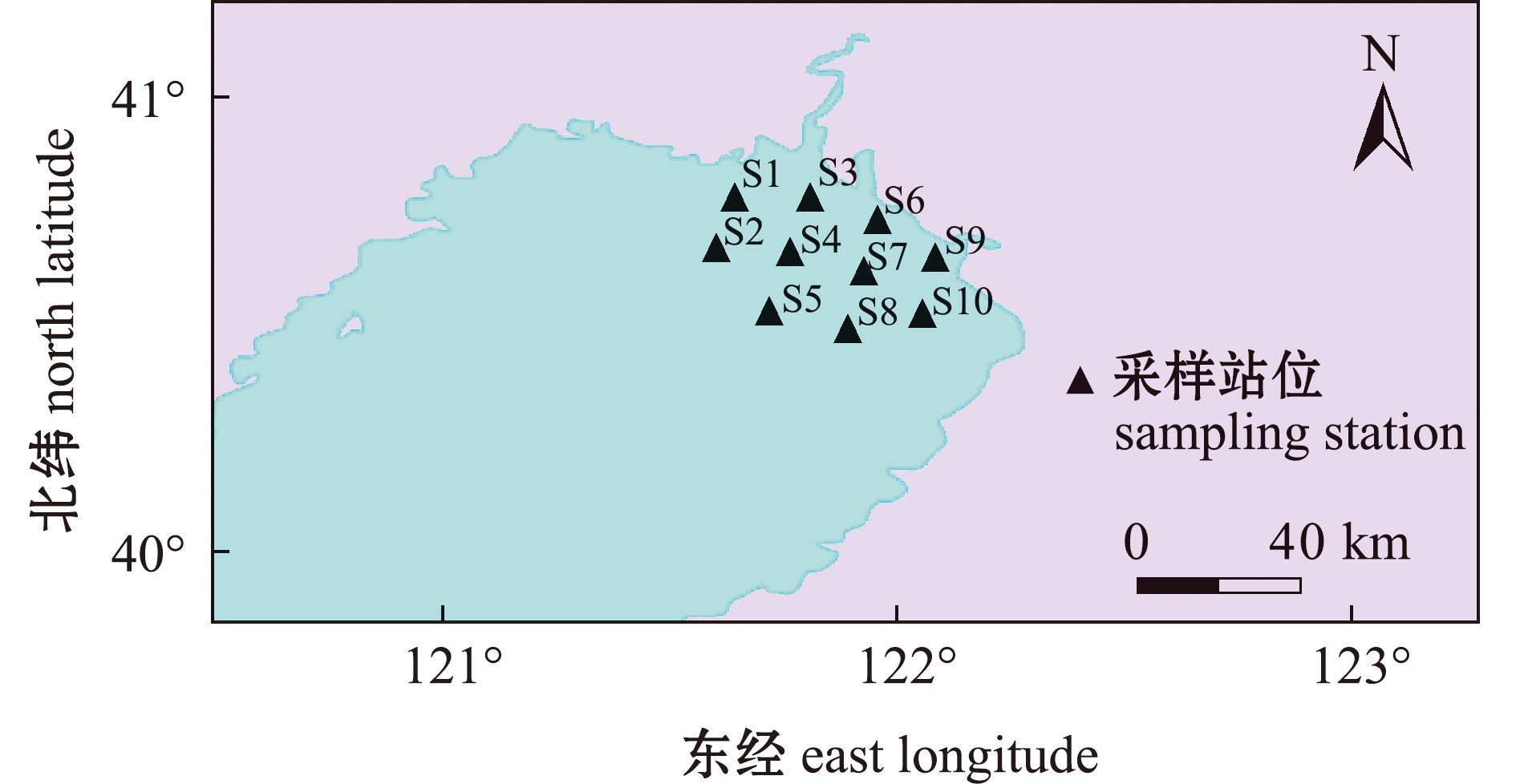
图1 辽河口调查站位
Fig.1 Sampling stations in the Liaohe Estuary
1.2 eDNA样品采集及处理
1.2.1 eDNA样品采集 eDNA样品采集与拖网调查采样同步进行。调查船舶在到达预设调查站位后,先行完成eDNA水样采集工作,然后放网拖拽作业。在10个调查站位共计采集到30个eDNA样本。eDNA采样方法为先用分层采水器在每个采样站位的表层(距海面<2 m)、中层、底层(距海底<2 m)各采集1 L海水,混合储存于容量为3 L的一次性无菌采样袋中。然后从无菌采样袋中取1 L水样作为eDNA样本,每个站位设置3个平行样。水样采集后立即在调查船上选用孔径为0.45 μm的混合纤维无菌滤膜(Sartorius,德国)和津腾无油真空泵(GM-0.33A)进行水样抽滤。为避免交叉污染,每个样品抽滤前后均使用体积分数为10%的次氯酸钠溶液将抽滤容器浸泡消毒5 min,并用超纯水清洗,且每个样品抽滤使用单独的一次性橡胶手套。每次抽滤设置1个阴性对照(过滤1 L超纯水),以评估是否存在交叉污染。抽滤后将滤膜保存于5 mL的无菌离心管中,用锡箔纸包裹离心管后置于液氮中速冻,带回实验室置于-80 ℃冰箱中保存备用。
1.2.2 eDNA提取 选用DNeasy Blood&Tissue kit试剂盒(Qiagen,德国)进行水样eDNA提取,提取好的eDNA用100 μL的AE缓冲液洗脱保存后用20 g/L的琼脂凝胶进行电泳检测。将合格的DNA提取溶液于-20 ℃冰箱中保存用于后续扩增测序。
1.2.3 PCR扩增及高通量测序 选用海洋硬骨鱼类线粒体12S rRNA基因部分序列通用引物(MiFish-U/E)进行eDNA的PCR扩增[20]。PCR扩增采用20 μL反应体系进行:5×FastPfu Buffer 4 μL,dNTPs 2 μL,正、反向引物各0.8 μL,FastPfu Polymerase 0.4 μL,模板DNA 10 ng,补dd H2O至20 μL。扩增条件:95 ℃下预变性 3 min,98 ℃下变性20 s,65 ℃下退火15 s,72 ℃下延伸15 s,共进行40 次循环,72 ℃下终延伸5 min。为保证后续数据分析的准确性及可靠性,需满足两个条件:1) 尽可能使用低循环数扩增;2) 保证每个样本扩增的循环数一致。随机选取具有代表性的样本进行预试验,确保在最低循环数中使绝大多数样本能够扩增出浓度合适的产物,每个样本重复3次,将同一样本的PCR产物混合后用20 g/L琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂(AXYGEN公司)切胶回收PCR产物,Tris_HCl洗脱,20 g/L琼脂糖电泳检测及纯化。参照电泳初步定量结果,将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合。最后将PCR产物送上海凌恩生物科技有限公司,使用Illumina PE250测序平台(美国)对文库进行双端测序。
1.3 数据处理
采用MiFish pipeline对下机原始数据过滤,采用Usearch软件和gold数据库,采用denovo和reference结合的方式去除嵌合体。选用QIIME2生物组分析软件基于DADA2法进一步降噪得到不含扩增与测序错误、不含嵌合体的扩增子序列变体(Amplicon Sequence Variants,ASVs)。采用blastn比对法将测序数据质控后的ASVs与鱼类线粒体数据库(https://mitofish.aori.u-tokyo.ac.jp/)、NCBI数据库进行物种分类注释,并分别在各个分类阶元统计各样本的群落组成。结合渤海水生生物历史分布资料[17-19]对所注释物种进行人工校对,手动剔除非海洋鱼类信息及阴性对照所包含的鱼类扩增子序列信息,以排除对后续分析干扰。基于R语言(4.1.2版本)进行结果可视化分析,采用ggplot2、vegan和ggpubr包进行群落组成多样性分析,采用ade4和ggplot2包完成主成分分析(PCA),采用SPSS 20.0软件进行线性回归分析。
2 结果与分析
2.1 高通量测序及底拖网采样
采样结果表明,10个站位采集的30个水样均成功提取扩增到了eDNA。经过质量控制和筛选,10个采样站位获得的序列数为54 887~65 520 条,序列平均长度为169~172 bp。10个底拖网调查站位中,除S6站位由于网衣破裂未采集到样品,其余9个站位均采集到了鱼类样本。其中,相对渔获数量为83.57~1 057.84 ind./h,相对渔获质量为2 163.21~51 968.10 g/h。eDNA测序及相对渔获量结果见表1。
表1 eDNA测序结果及底拖网相对渔获量
Tab.1 eDNA sequencing results and relative catch of bottom trawl
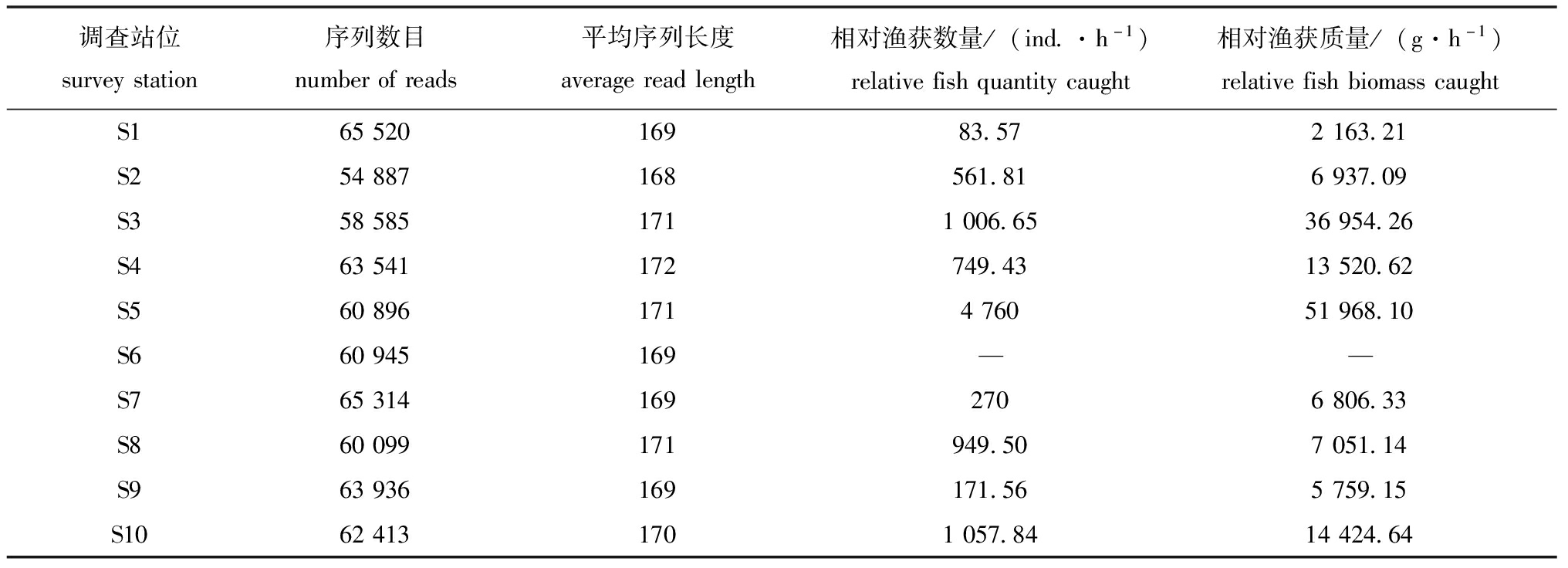
调查站位surveystation序列数目numberofreads平均序列长度averagereadlength相对渔获数量/(ind.·h-1)relativefishquantitycaught相对渔获质量/(g·h-1)relativefishbiomasscaughtS16552016983.572163.21S254887168561.816937.09S3585851711006.6536954.26S463541172749.4313520.62S560896171476051968.10S660945169——S7653141692706806.33S860099171949.507051.14S963936169171.565759.15S10624131701057.8414424.64
2.2 eDNA与底拖网调查鱼类种类组成
两种调查方法共采集到鱼类40种(表2)。ASVs 注释结果表明,调查海域共采集到鱼类37种,隶属于8目21科34属,其中35种注释到了种水平,蛇鲻属和绿鳍鱼属的2种鱼类仅鉴定到属水平;底拖网调查共采集到鱼类16种,隶属于3目7科15属,eDNA技术检测到的鱼类种类数是底拖网调查的2.3倍。底拖网采集到的16种鱼类中除普氏栉虾虎鱼、中华栉孔虾虎鱼和短鳍![]() 3种外,其他13种鱼类均被eDNA技术监测到。从分类来看,eDNA技术监测到的鱼类主要为虾虎鱼科(Gobiidae),占总种类数的24.3%,其次为鳀科(Engraulidae),占总种类数的10.8%;底拖网调查采集到的鱼类也以虾虎鱼科为主,占总种类数的43.75%,其次为
3种外,其他13种鱼类均被eDNA技术监测到。从分类来看,eDNA技术监测到的鱼类主要为虾虎鱼科(Gobiidae),占总种类数的24.3%,其次为鳀科(Engraulidae),占总种类数的10.8%;底拖网调查采集到的鱼类也以虾虎鱼科为主,占总种类数的43.75%,其次为![]() 科(Callionymidae)、鳀科和舌鳎科(Cynoglossidae),均占12.5%。
科(Callionymidae)、鳀科和舌鳎科(Cynoglossidae),均占12.5%。
表2 基于eDNA技术和底拖网调查监测到的鱼类
Tab.2 Fish species detected by eDNA technology and bottom trawl survey
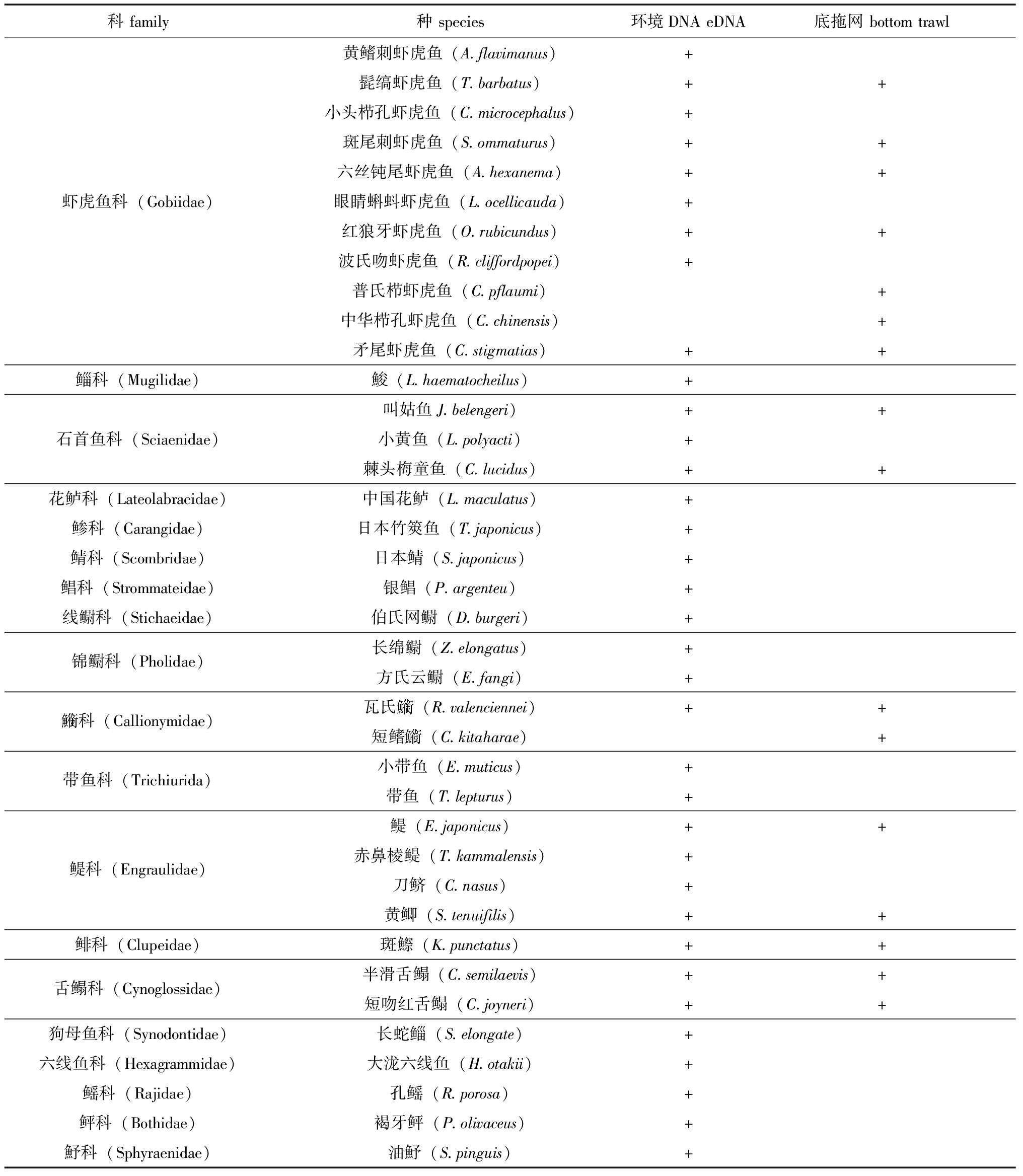
科family种species环境DNAeDNA底拖网bottomtrawl黄鳍刺虾虎鱼(A.flavimanus)+髭缟虾虎鱼(T.barbatus)++小头栉孔虾虎鱼(C.microcephalus)+斑尾刺虾虎鱼(S.ommaturus)++六丝钝尾虾虎鱼(A.hexanema)++虾虎鱼科(Gobiidae)眼睛蝌蚪虾虎鱼(L.ocellicauda)+红狼牙虾虎鱼(O.rubicundus)++波氏吻虾虎鱼(R.cliffordpopei)+普氏栉虾虎鱼(C.pflaumi)+中华栉孔虾虎鱼(C.chinensis)+矛尾虾虎鱼(C.stigmatias)++鲻科(Mugilidae)鮻(L.haematocheilus)+叫姑鱼J.belengeri)++石首鱼科(Sciaenidae)小黄鱼(L.polyacti)+棘头梅童鱼(C.lucidus)++花鲈科(Lateolabracidae)中国花鲈(L.maculatus)+鲹科(Carangidae)日本竹筴鱼(T.japonicus)+鲭科(Scombridae)日本鲭(S.japonicus)+鲳科(Strommateidae)银鲳(P.argenteu)+线鳚科(Stichaeidae)伯氏网鳚(D.burgeri)+锦鳚科(Pholidae)长绵鳚(Z.elongatus)+方氏云鳚(E.fangi)+科(Callionymidae)瓦氏(R.valenciennei)++短鳍(C.kitaharae)+带鱼科(Trichiurida)小带鱼(E.muticus)+带鱼(T.lepturus)+鳀(E.japonicus)++鳀科(Engraulidae)赤鼻棱鳀(T.kammalensis)+刀鲚(C.nasus)+黄鲫(S.tenuifilis)++鲱科(Clupeidae)斑鰶(K.punctatus)++舌鳎科(Cynoglossidae)半滑舌鳎(C.semilaevis)++短吻红舌鳎(C.joyneri)++狗母鱼科(Synodontidae)长蛇鲻(S.elongate)+六线鱼科(Hexagrammidae)大泷六线鱼(H.otakii)+鳐科(Rajidae)孔鳐(R.porosa)+鲆科(Bothidae)褐牙鲆(P.olivaceus)+魣科(Sphyraenidae)油魣(S.pinguis)+
注:+表示该物种在调查站位被监测到。
Note:+ indicates the species was detected in the sampleling area.
2.3 eDNA与底拖网调查鱼类群落结构及多样性
基于eDNA的鱼类群落多样性Alpha多样性分析结果表明,10个调查点观测度指数(observed-species)为16~22,平均值为19;香农指数(Shannon)为0.958~1.897,平均值为1.442;辛普森指数(Simpson)为0.417~0.811,平均值为0.648。差异显著性检验结果表明,各Alpha多样性指数(观测度指数、香农指数和辛普森指数)的组间无显著性差异。基于eDNA的主要优势物种相对丰度如图2所示,相对丰度前五位的鱼类分别为矛尾虾虎鱼、斑尾刺虾虎鱼、短吻红舌鳎、棘头梅童鱼和鳀,出现频率均为100%。基于底拖网调查各站位鱼类组成丰度如图3所示,相对丰度前五位的优势物种依次为矛尾虾虎鱼、短吻红舌鳎、斑尾刺虾虎鱼、棘头梅童鱼和六丝钝尾虾虎鱼,且前五位优势种出现频率也为100%。调查结果表面显示,eDNA与底拖网调查鱼类优势种呈现出较高的一致性。
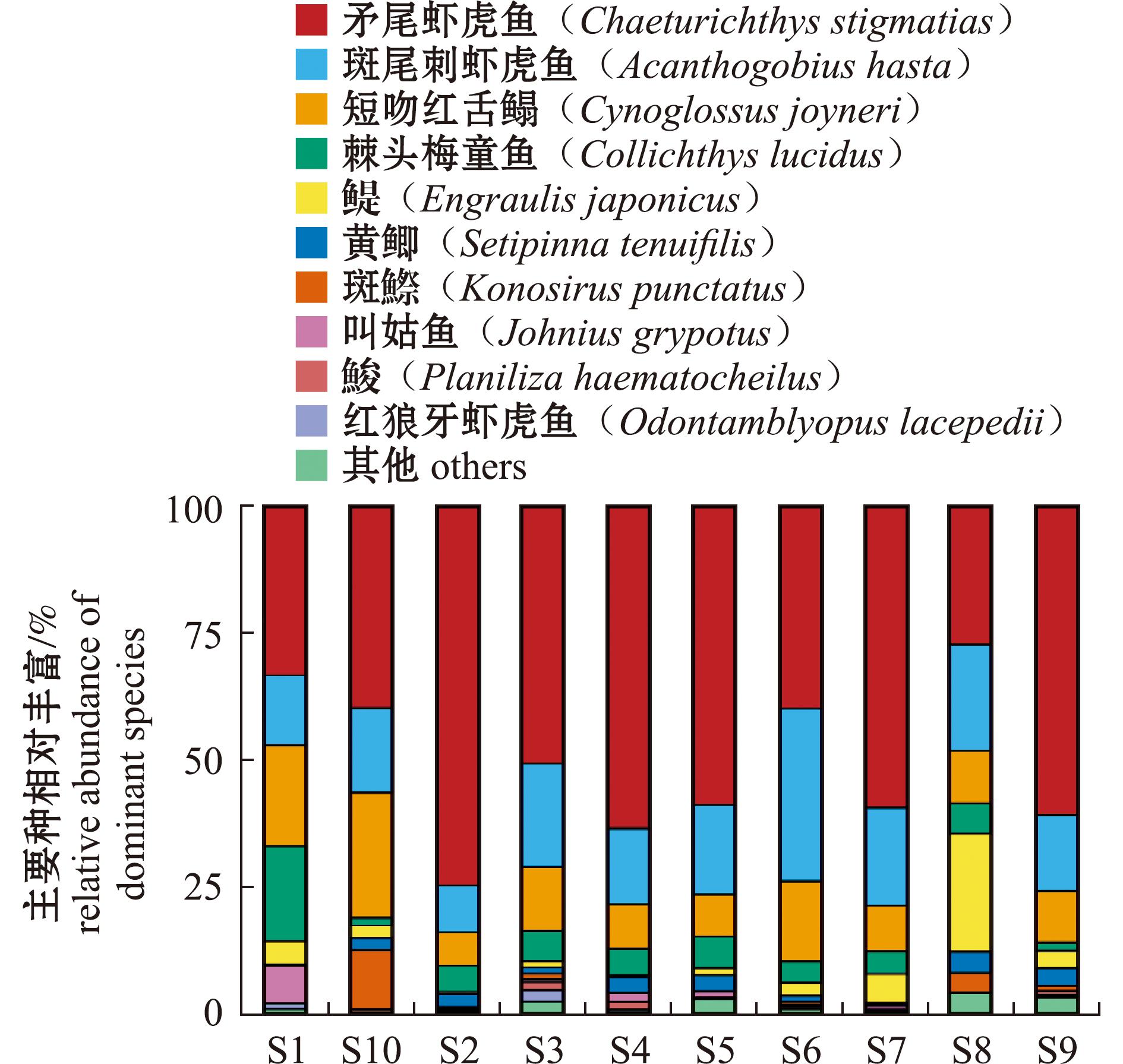
图2 基于eDNA的各站位优势鱼类组成
Fig.2 Composition of dominant fish species at each station based on eDNA
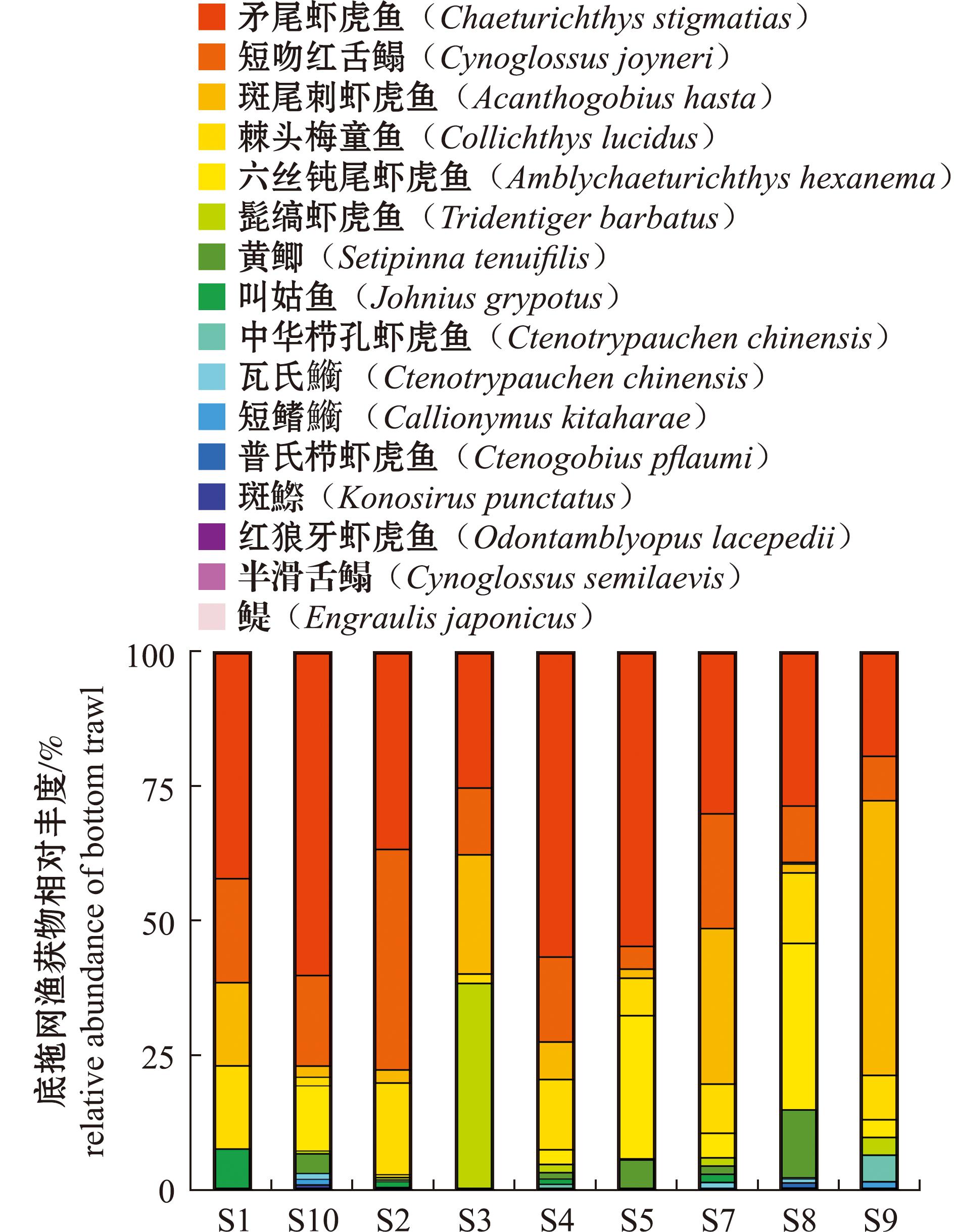
图3 底拖网调查各站位优势鱼类组成
Fig.3 Composition of dominant fish species at each station by bottom trawl survey
主成分分析(principal component analysis,PCA)是一种对数据进行降维排序的特征值分析方法,通过分析不同样品物种和基因组分,将多组数据的差异反映在二维坐标图上,坐标轴取能够最大化反映方差值的两个特征值。本研究PCA结果如图4所示。从图4可见,PC1能够解释43.04%的鱼类群落组成差异,PC2能够解释14.0%的鱼类群落组成差异,前两个主成分能够解释57.04%的鱼类群落差异。10个调查站位的30个样本交错混合分布于PCA的二维坐标平面上,大多样本的组内差异大于组间差异,不存在样本有规律的显著聚集现象,说明10个调查站位间的鱼类群落组成无显著性差异(P>0.05)。组间差异ANOSIM分析结果R值为0.42,统计检验P值为0.09,表明样本组间差异较小,且无显著性差异(P>0.05)。
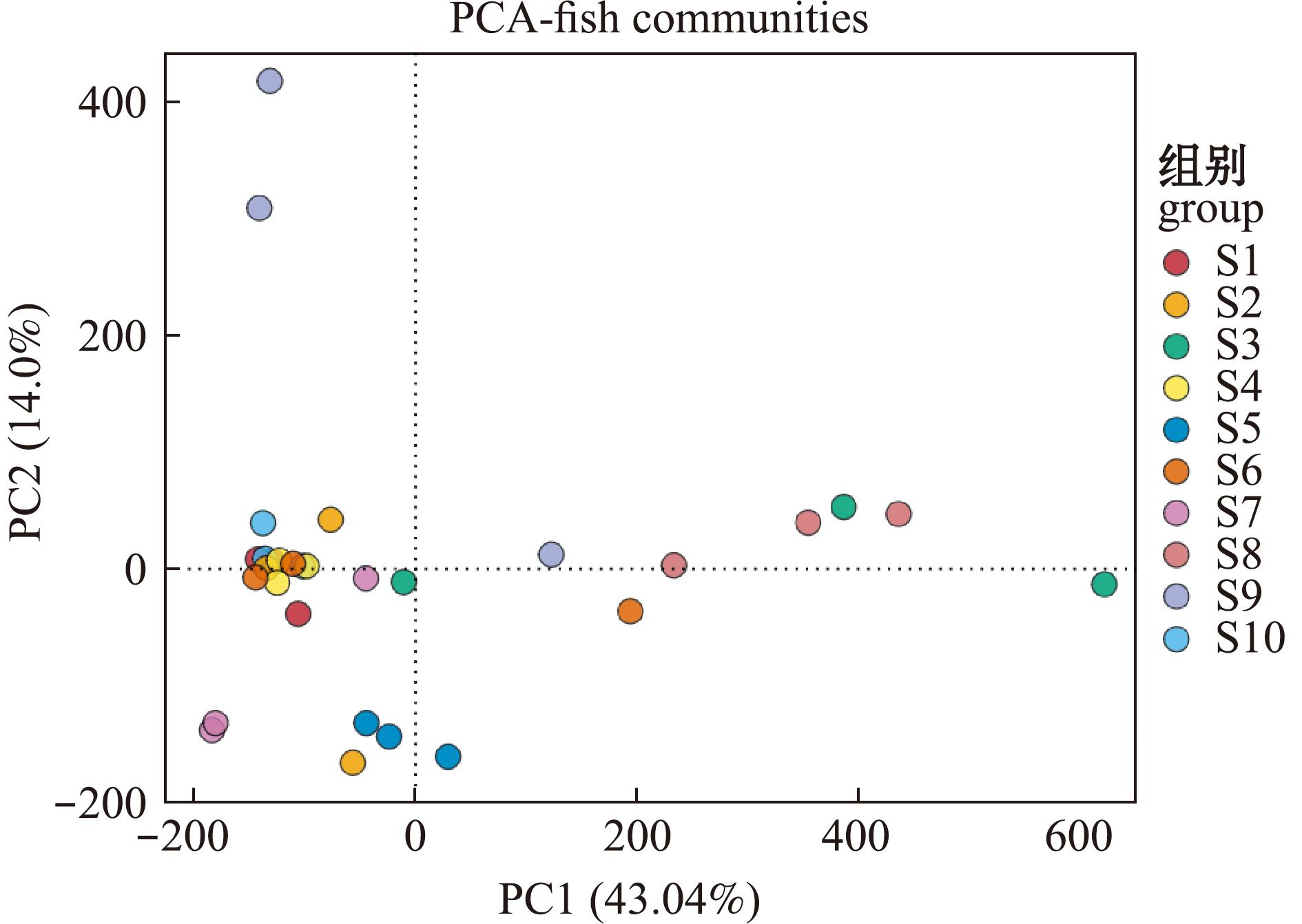
图4 基于ASVs的主成分分析
Fig.4 Analysis diagram of PCA based on ASVs
2.4 底拖网相对渔获量与eDNA相对丰度比较
10个查站位中,有9个站位同时采集到鱼类样本和eDNA样本。对9个站位鱼类样本的平均生物量与eDNA样本的平均相对丰度进行比较分析,两种调查方法的共有物种为13种。13种鱼类的生物量数据与eDNA相对丰度数据见表3。从表3可见,在底拖网调查中,相对渔获质量排前三的物种分别为矛尾虾虎鱼(6 297.77 g/h)、斑尾刺虾虎鱼(4 700.23 g/h)和短吻红舌鳎(1 563.65 g/h);相对渔获数量排前三的物种为矛尾虾虎鱼(507.19 ind./h)、六丝钝尾虾虎鱼(191.21 ind./h)和短吻红舌鳎(114.96 ind./h);eDNA相对丰度排前三的物种分别为矛尾虾虎鱼(254 574)、斑尾刺虾虎鱼(89 512)和短吻红舌鳎(64 539)。
表3 13个物种的eDNA相对丰度与相对渔获量对比
Tab.3 Comparison between eDNA relative abundance and relative catchrate of 13 fish species
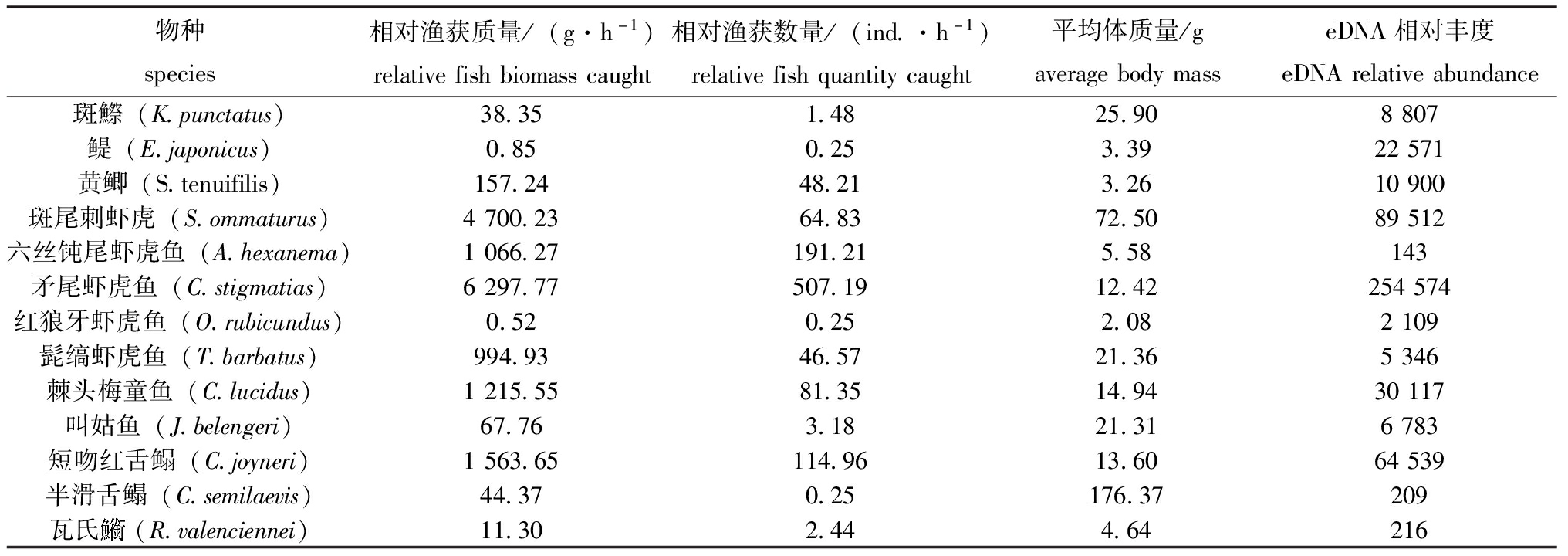
物种species相对渔获质量/(g·h-1)relativefishbiomasscaught相对渔获数量/(ind.·h-1)relativefishquantitycaught平均体质量/gaveragebodymasseDNA相对丰度eDNArelativeabundance斑鰶(K.punctatus)38.351.4825.908807鳀(E.japonicus)0.850.253.3922571黄鲫(S.tenuifilis)157.2448.213.2610900斑尾刺虾虎(S.ommaturus)4700.2364.8372.5089512六丝钝尾虾虎鱼(A.hexanema)1066.27191.215.58143矛尾虾虎鱼(C.stigmatias)6297.77507.1912.42254574红狼牙虾虎鱼(O.rubicundus)0.520.252.082109髭缟虾虎鱼(T.barbatus)994.9346.5721.365346棘头梅童鱼(C.lucidus)1215.5581.3514.9430117叫姑鱼(J.belengeri)67.763.1821.316783短吻红舌鳎(C.joyneri)1563.65114.9613.6064539半滑舌鳎(C.semilaevis)44.370.25176.37209瓦氏(R.valenciennei)11.302.444.64216
为探讨运用eDNA高通量测序技术来评估鱼类群落生物量和数量组成的可行性,本研究中还开展了eDNA相对丰度与相对渔获质量、相对渔获数量及平均个体质量间的皮尔逊(Pearson)相关性分析。从表4可见,eDNA相对丰度与相对渔获质量、相对渔获数量呈极强的相关性,r值分别为0.921和0.888,且相关性检验均达到极显著性水平(P<0.01),说明eDNA相对丰度与底拖网相对渔获量间存在较强的线性相关性。另一方面,eDNA相对丰度与个体平均体质量间相关系数为-0.046,说明eDNA相对丰度与个体平均体质量间的线性相关性极弱。
表4 eDNA相对丰度与相对渔获量及平均体质量相关性
Tab.4 Relationship between relative abundance of eDNA,relative catch rate,and average body mass

eDNA相对丰度eDNArelativeabundance相对渔获质量/(g·h-1)relativefishbiomasscaught相对渔获数量/(ind.·h-1)relativefishquantitycaught平均个体质量/gaveragebodymasseDNA相对丰度eDNArelativeabundance—0.0000.0000.876相对渔获质量/(g·h-1)relativecatchquality0.921∗—0.0000.867相对渔获数量/(ind.·h-1)relativecatchnumber0.888∗0.814∗—0.596平均体质量averagebodymass/g-0.0460.049-0.156—
注:对角线下为相关性指数,对角线上为显著性检验值;*表示在0.01水平相关性极显著(P<0.01)。
Note:below the diagonal is correlation index,and above the diagonal is the significance test value;*indicates the correlation were highly significant at 0.01 level.
2.5 鱼类群落结构与环境因子关系
调查采样区域的水环境因子调查监测结果见表5。从表5可见,采样区域温度分布为13.80 ℃~15.21 ℃;盐度分布为20.00~21.90;水深分布为5.82~9.18 m;pH分布为8.74~9.03;溶氧分布为6.62~7.82 mg/L。基于eDNA高通量测序数据和采样期间测定的环境因子数据进行RDA分析结果如图5所示。从图5可见,RDA1对鱼类群落结构差异的解释量为70.59%,RDA2对鱼类群落结构差异解释量为17.2%。前两个主成分可以解释87.79%的群里差异。温度和深度与鱼类群落分布间的相关性不高(相关系数R分别为0.470和0.487),相关性统计检验均不显著(P>0.05)。
表5 采样站位水环境因子监测结果
Tab.5 Monitoring results of water environmental factor of sampling station
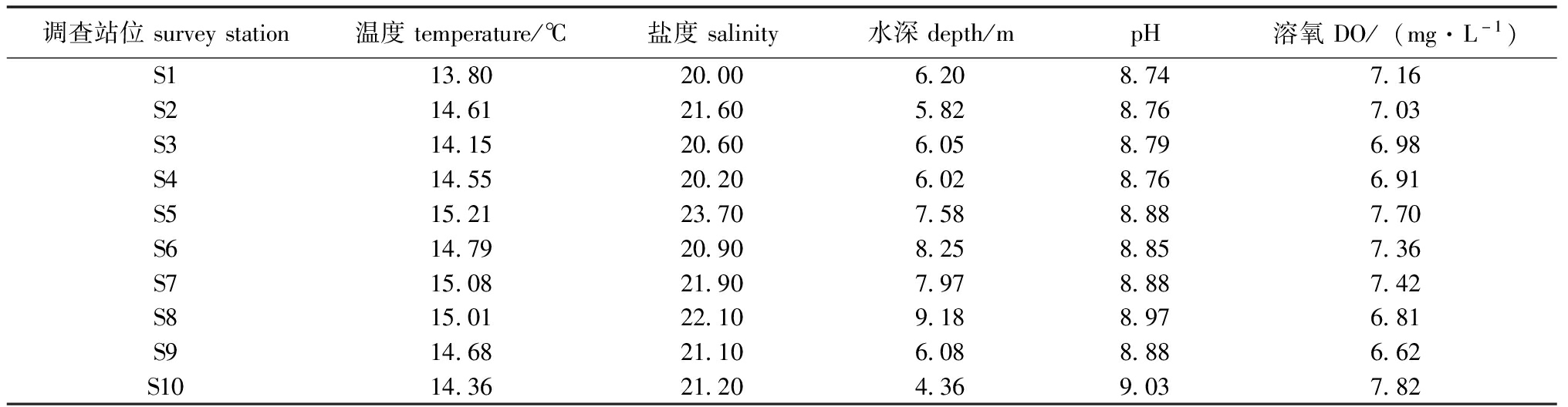
调查站位surveystation温度temperature/℃盐度salinity水深depth/mpH溶氧DO/(mg·L-1)S113.8020.006.208.747.16S214.6121.605.828.767.03S314.1520.606.058.796.98S414.5520.206.028.766.91S515.2123.707.588.887.70S614.7920.908.258.857.36S715.0821.907.978.887.42S815.0122.109.188.976.81S914.6821.106.088.886.62S1014.3621.204.369.037.82
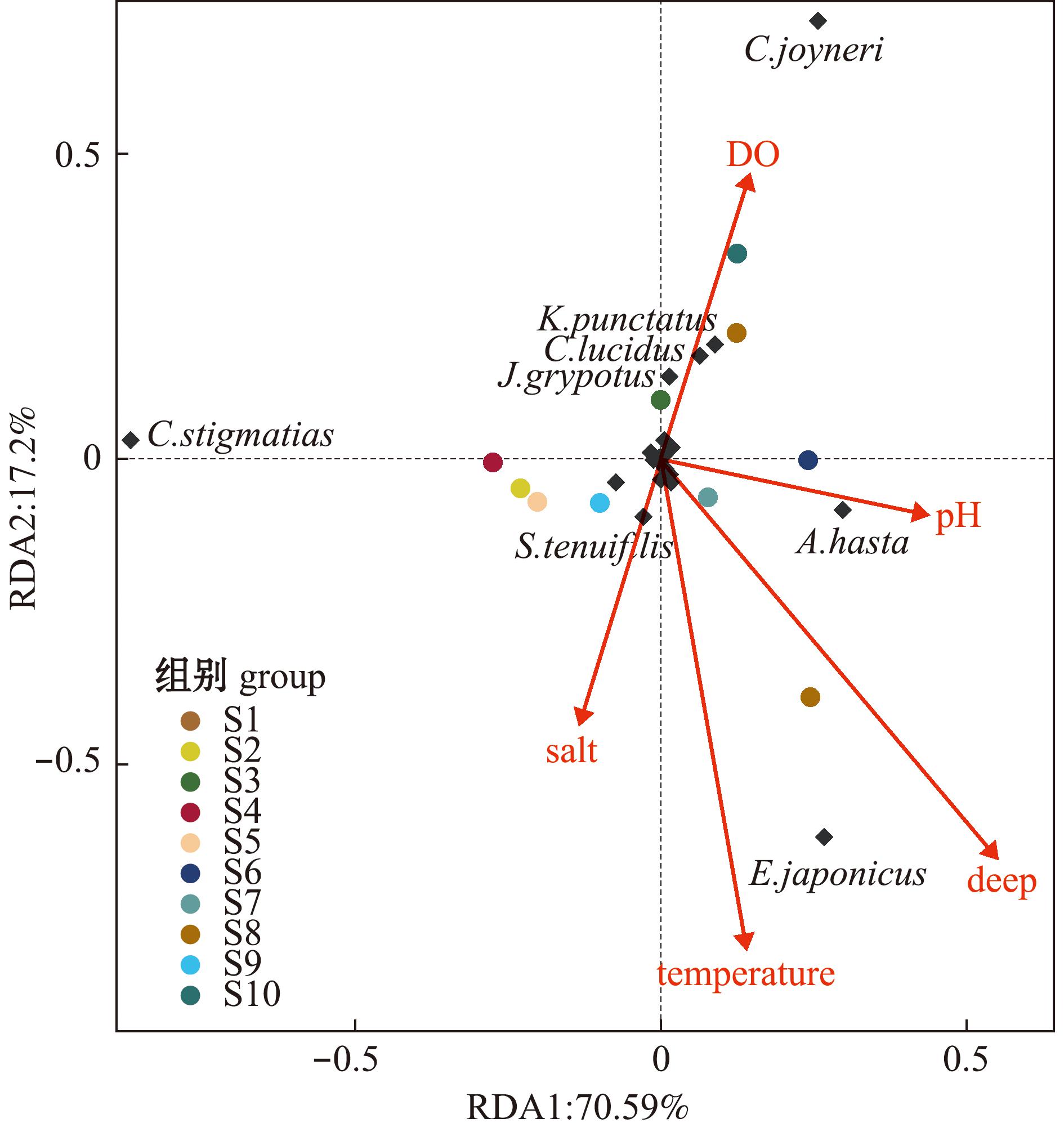
图5 环境因子对鱼类群落组成影响的RDA分析
Fig.5 Influence of environmental factors on fish community based on RDA analysis
3 讨论
3.1 拖网调查与eDNA技术的鱼类种类比较
传统的渔业资源调查采样方法(如底拖网、流刺网)可以第一时间获取调查区域鱼类的种类、丰度和分布等信息。尽管传统方法在调查过程中具有许多局限性,但拖网调查仍为目前渔业资源调查中最常用的方法[21]。近年来,eDNA技术因其成本低、普适性强和监测灵敏高等特点已广泛应用于鱼类生物多样性调查与监测,但由于其固有的不确定性(温度、水流、生命史和代谢对其影响)而不能完全取代传统方法[22]。将拖网调查和eDNA方法相结合,综合对比调查区域历史鱼类分布情况,可以实现更准确全面的渔业资源评价。
本研究中运用eDNA技术监测到的37种鱼类,其中,35种注释到了种水平,另有2种仅注释到属水平(蛇鲻属和绿鳍鱼属)。李晨虹等[23]研究表明,全面准确的比对数据库是eDNA技术高效应用的前提,而有限的DNA参考数据库是目前eDNA技术在水生生物监测中应用面临的挑战之一。本研究中两种鱼类仅定位到属水平,就是缺乏针对调查海域全面系统的eDNA比对数据库所致,如果有参考数据库,这两个物种可能精确定位到种,因此,建立专门的渤海鱼类DNA参考数据库意义重大。本研究中底拖网调查采集到鱼类16种,其中除普氏栉虾虎鱼、中华栉孔虾虎鱼、短鳍![]() 3种外,其他13种鱼类如髭缟虾虎鱼、斑尾刺虾虎鱼、六丝钝尾虾虎鱼、红狼牙虾虎鱼、矛尾虾虎鱼、叫姑鱼、棘头梅童鱼、瓦氏
3种外,其他13种鱼类如髭缟虾虎鱼、斑尾刺虾虎鱼、六丝钝尾虾虎鱼、红狼牙虾虎鱼、矛尾虾虎鱼、叫姑鱼、棘头梅童鱼、瓦氏![]() 、鳀、黄鲫、斑鰶、半滑舌鳎和短吻红舌鳎均被eDNA技术监测到。eDNA技术鉴定到种的35种鱼类中,有22种鱼类仅被eDNA技术检测出。经过与《辽宁省动物志·鱼类》[17]《中国海洋生物名录》[18]和《中国海洋鱼类》[19]等鱼类鉴定及区系划分材料对比,该22种鱼类在辽河口及附近海域均有历史分布记录。22种鱼类包括黄鳍刺虾虎鱼、小头栉孔虾虎鱼、眼睛蝌蚪虾虎鱼、波氏吻虾虎鱼、大泷六线鱼、孔鳐、褐牙鲆、长蛇鲻、小黄鱼、中国花鲈、长绵鳚、方氏云鳚、伯氏网鳚和油魣等14种底栖鱼类,以及鮻、银鲳、日本竹筴鱼、日本鲭、小带鱼、带鱼、赤鼻棱鳀和刀鲚等8种中上层鱼类。造成两种调查方法采样种类数显著差异的主要原因可能有两方面:一是底拖网采样的局限性,底拖网采集底层鱼类具明显优势,但其采集中上层鱼类具有很大的局限性,本研究中仅被eDNA技术检测而未被底拖网采集到的样本大多为中上层鱼类,如日本竹筴鱼、日本鲭、银鲳、赤鼻棱鳀和刀鲚等,此外,底拖网在岩礁区适用性差,导致大泷六线鱼等鱼类采集不到;二是目标鱼种相对稀少。长蛇鲻、孔鳐和褐牙鲆等潜沙性底层鱼类在河口区域分布相对较稀少,虽能被eDNA技术监测到遗传信号,但被底拖网采集到的概率较低。由此可见,eDNA技术可以有效地弥补底拖网采样的不足,可采集到调查海域稀有物种和底拖网难以采集到的鱼类样本信息。
、鳀、黄鲫、斑鰶、半滑舌鳎和短吻红舌鳎均被eDNA技术监测到。eDNA技术鉴定到种的35种鱼类中,有22种鱼类仅被eDNA技术检测出。经过与《辽宁省动物志·鱼类》[17]《中国海洋生物名录》[18]和《中国海洋鱼类》[19]等鱼类鉴定及区系划分材料对比,该22种鱼类在辽河口及附近海域均有历史分布记录。22种鱼类包括黄鳍刺虾虎鱼、小头栉孔虾虎鱼、眼睛蝌蚪虾虎鱼、波氏吻虾虎鱼、大泷六线鱼、孔鳐、褐牙鲆、长蛇鲻、小黄鱼、中国花鲈、长绵鳚、方氏云鳚、伯氏网鳚和油魣等14种底栖鱼类,以及鮻、银鲳、日本竹筴鱼、日本鲭、小带鱼、带鱼、赤鼻棱鳀和刀鲚等8种中上层鱼类。造成两种调查方法采样种类数显著差异的主要原因可能有两方面:一是底拖网采样的局限性,底拖网采集底层鱼类具明显优势,但其采集中上层鱼类具有很大的局限性,本研究中仅被eDNA技术检测而未被底拖网采集到的样本大多为中上层鱼类,如日本竹筴鱼、日本鲭、银鲳、赤鼻棱鳀和刀鲚等,此外,底拖网在岩礁区适用性差,导致大泷六线鱼等鱼类采集不到;二是目标鱼种相对稀少。长蛇鲻、孔鳐和褐牙鲆等潜沙性底层鱼类在河口区域分布相对较稀少,虽能被eDNA技术监测到遗传信号,但被底拖网采集到的概率较低。由此可见,eDNA技术可以有效地弥补底拖网采样的不足,可采集到调查海域稀有物种和底拖网难以采集到的鱼类样本信息。
3.2 基于eDNA的鱼类生物多样性比较
利用eDNA高通量测序技术,不仅可以监测到比拖网采样方法更多的鱼类,而且还可以分析不同站位间物种组成的差异[24]。在本研究中,笔者还根据ASVs的注释结果,对10个调查站位间的物种组成、群落差异进行了对比分析。结果表明,各调查站位观测度指数、香农指数和辛普森指数间的差异不大。PCA结果也表明,大多样本的组内差异大于组间差异,不存在样本有规律的显著聚集现象。如上均表明调查区域调查站位间的鱼类群落组成差异不显著。本次调查同时对调查站位开展的水环境因子与群落结构的RDA分析结果表明,各水环境因子(温度、盐度、pH、溶解氧和水深)与鱼类群落结构的相关性不显著,生境的相似性可能是调查区域调查站位间鱼类群落组成差异不显著的根本原因。Keck等[25]研究表明,eDNA宏条形码与传统的生物群落评估方法具有一致性和互补性。本研究中表明,eDNA技术与底拖网调查分析的优势种结果具有高度一致性,前4种优势种均为矛尾虾虎鱼、斑尾刺虾虎鱼、短吻红舌鳎和棘头梅童鱼。这表明eDNA技术在辽河口海域进行快速、高效的鱼类生物多样性调查监测中具有很高的适用性。
3.3 基于eDNA的生物量评估
准确的渔业资源生物量评估是进行有效渔业管理的前提。近年来,诸多学者运用eDNA高通量测序技术揭示了海洋鱼类eDNA相对丰度与鱼类群落组成之间的相关性。相关研究表明,调查水域目标生物量越高,监测到的eDNA相对丰度也越高[26-28]。Urban等[29]研究探讨了应用eDNA方法评估捕捞渔获物组成的可行性,并建立了eDNA-生物量的广义线性混合模型,结果表明,鲭eDNA相对丰度与渔获物中鲭质量百分比间存在强烈的相关性(r值0.812,P<0.001),eDNA相对丰度越高,渔获物中鲭鱼质量占比也越高。Lyu等[13]和Jiang等[14]分别运用eDNA宏基因组技术与底拖网调查相结合的方法对黄海口和珠江口的秋季渔业资源生物多样性进行调查评估,结果表明,在黄河口及其附近海域鱼类运用两种方法同时采集到的22种鱼类中,eDNA的相对丰度与底拖网相对渔获质量和相对渔获数量间存在较高的线性相关性(r值分别为0.921和0.888),且相关性极显著(P<0.01);珠江口及其附近海域鱼类运用两种方法同时采集到的35种鱼类中,eDNA的相对丰度与底拖网相对渔获质量和相对渔获数量间存在线性相关性(r值分别为0.53和0.56),且相关性极显著(P<0.01),两者均呈现出水环境中eDNA相对丰度越高,拖网采样中相应鱼类的相对渔获质量和相对渔获数量越高的趋势。本研究结果与上述研究结果具有较高的一致性,结果表明,在辽河口及附近海域鱼类eDNA相对丰度与底拖网调查的相对渔获质量和相对渔获数量间存在较强的线性正相关性,相关系数处于较高水平(r值分别为0.921和0.888),且相关性达到极显著水平(P<0.01)。eDNA相对丰度最高的鱼类为矛尾虾虎鱼,其相对渔获质量和数量在底拖网采集的13种鱼类样品中也位居首位。在eDNA相对丰度排名2~4位的斑尾刺虾虎鱼、短吻红舌鳎、棘头梅童鱼中,短吻红舌鳎的相对渔获质量和数量排第3位,棘头梅童鱼的相对渔获质量和数量排第4位,斑尾刺虾虎鱼的相对渔获质量和数量分别排第2位和第5位,两种调查结果均呈现出较高的一致性。虽然本研究中通过底拖网调查法和eDNA技术检测到的13种鱼类中,大多数都证实了eDNA丰度与相对渔获质量和数量之间存在正相关性,但也有eDNA与拖网法检测的差异在少数鱼类中发现。如鳀和红狼牙虾虎鱼的eDNA相对丰度较高但相对渔获数量和相对渔获质量值较低,六丝钝尾虾虎鱼的相对渔获数量和相对渔获质量值较高,但eDNA相对丰度较低(表4)。Thomsen等[11]的研究中也出现了类似的结果,表明拖网捕捞方法可能不适合某些大型或快速游动的中上层鱼类。此外,尽管生物量可能直接影响eDNA浓度,但许多其他因素也可能间接影响eDNA浓度,同一物种的两个个体即使体质量相等,但生长年龄、摄食量及摄食种类的不同,也会导致其eDNA释放量存在差异[30]。在未来研究应用中,为了更好地了解eDNA浓度与生物量的相关性,需综合考虑温度、盐度、发育阶段、降解、饲养和营养水平等诸多影响因子。
4 结论
1)eDNA技术较底拖网调查具有更高的物种检出效率。对调查海域稀有物种和中上层鱼类具有更好的监测效果。
2)eDNA技术可以作为传统渔业资源调查方法的有效补充,运用于辽河口海域鱼类群落生物多样性的监测评估。
3)运用eDNA的相对丰度评估辽河口海域鱼类资源生物量具有较高的可行性。通过底拖网调查法和eDNA技术都监测到的13种鱼类中,大多数都证实了eDNA相对丰度与相对渔获质量和数量之间存在显著的正相关性。
总之,eDNA技术与传统渔业资源调查评估方法具有一致性和互补性,利用eDNA高通量测序不仅可以监测到比拖网捕捞方法更多的鱼种,而且可以分析不同站位间物种组成的差异,在辽河口及附近海域鱼类生物多样性监测评估方面具有较大的应用潜力。
[1] 董婧,刘修泽,王爱勇.辽宁近海与河口区渔业资源[M].北京:中国农业出版社,2018.DONG J,LIU X Z,WANG A Y.Fishery resources in the coastal and estuarine areas of Liaoning Province[M].Beijing:China Agriculture Press,2018.(in Chinese)
[2] 叶金清,柳圭泽,袁秀堂,等.基于生物学特征的辽河口鮻资源开发与保护[J].大连海洋大学学报,2020,35(2):302-307.YE J Q,LIU G Z,YUAN X T,et al.Exploitation and protection of mullet Liza haematocheila stocks in Liaohe Estuary based on biological characteristics[J].Journal of Dalian Ocean University,2020,35(2):302-307.(in Chinese)
[3] 陈刚,陈卫忠.渔业资源评估中声学方法的应用[J].上海水产大学学报,2003,12(1):40-44.CHEN G,CHEN W Z.The application of acoustic method in fishery resources survey[J].Journal of Shanchai Fisheries University,2003,12(1):40-44.(in Chinese)
[4] 秦传新,左涛,于刚,等.环境DNA在水生生态系统生物量评估中的研究进展[J].南方水产科学,2020,16(5):123-128.QIN C X,ZUO T,YU G,et al.Advances in research of environmental DNA(eDNA) in biomass assessment of aquatic ecosystems[J].South China Fisheries Science,2020,16(5):123-128.(in Chinese)
[5] 陈炼,吴琳,刘燕,等.环境DNA metabarcoding及其在生态学研究中的应用[J].生态学报,2016,36(15):4573-4582.CHEN L,WU L,LIU Y,et al.Application of environmental DNA metabarcoding in ecology[J].Acta Ecologica Sinica,2016,36(15):4573-4582.(in Chinese)
[6] 沈梅,肖能文,卢林,等.环境DNA技术及在鱼类监测中的应用[J].水生态学杂志,2022,43(2):133-141.SHEN M,XIAO N W,LU L,et al.Review of environmental DNA detection methods and their application to fish monitoring[J].Journal of Hydroecology,2022,43(2):133-141.(in Chinese)
[7] BOHMANN K,EVANS A,GILBERT M T P,et al.Environmental DNA for wildlife biology and biodiversity monitoring[J].Trends in Ecology &Evolution,2014,29(6):358-367.
[8] ADAMS C I M,KNAPP M,GEMMELL N J,et al.Beyond biodiversity:can environmental DNA (eDNA) cut it as a population genetics tool?[J].Genes,2019,10(3):192.
[9] BARRENECHEA ANGELES I,LEJZEROWICZ F,CORDIER T,et al.Planktonic foraminifera eDNA signature deposited on the seafloor remains preserved after burial in marine sediments[J].Scientific Reports,2020,10(1):20351.
[10] STOECKLE M Y,ADOLF J,CHARLOP-POWERS Z,et al.Trawl and eDNA assessment of marine fish diversity,seasonality,and relative abundance in coastal New Jersey,USA[J].ICES Journal of Marine Science,2021,78(1):293-304.
[11] THOMSEN P F,MØLLER P R,SIGSGAARD E E,et al.Environmental DNA from seawater samples correlate with trawl catches of subarctic,deepwater fishes[J].PLoS One,2016,11(11):e0165252.
[12] 言柯程,李建超,田永军,等.基于环境DNA metabarcoding和底拖网调查的南黄海西部鱼类多样性比较[J].中国海洋大学学报(自然科学版),2023,53(5):71-81.YAN K C,LI J C,TIAN Y J,et al.Comparison of fish biodiversity in western south Yellow Sea based on environmental DNA metabarcoding and trawl survey[J].Periodical of Ocean University of China(Nature Science Edition),2023,53(5):71-81.(in Chinese)
[13] LYU D,QIAN T Y,LI F,et al.Monitoring and assessing the species diversity and abundance of marine teleost around the Yellow River Estuary in June using environmental DNA[J].Frontiers in Marine Science,2023,10:1123831.
[14] JIANG P W,ZHANG S,XU S N,et al.Comparison of environmental DNA metabarcoding and bottom trawling for detecting seasonal fish communities and habitat preference in a highly disturbed estuary[J].Ecological Indicators,2023,146:109754.
[15] 王汝贤,杨刚,耿智,等.环境DNA技术在长江口鱼类多样性分析中的应用[J].水生生物学报,2023,47(3):365-375.WANG R X,YANG G,GENG Z,et al.Application of environmental DNA technology in fish diversity analysis in the Yangtze River Estuary[J].Acta Hydrobiologica Sinica,2023,47(3):365-375.(in Chinese)
[16] 崔晓玉,张云雷,张金勇,等.基于环境DNA宏条形码的辽东半岛重要河口鱼类多样性特征[J].大连海洋大学学报,2024,39(4):578-588.CUI X Y,ZHANG Y L,ZHANG J Y,et al.Fish diversity in important estuaries in Liaodong Peninsula based on environmental DNA metabarcoding[J].Journal of Dalian Ocean University,2024,39(4):578-588.(in Chinese)
[17] 刘蝉馨,秦克静,丁耕芜,等.辽宁省动物志·鱼类[M].沈阳:辽宁科学技术出版社,1987:1-229.LIU C X,QIN K J,DING G W,et al.Zoology of Liaoning Province·Fish[M].Shenyang:Liaoning Science and Technology Press,1987:1-229.(in Chinese)
[18] 刘瑞玉.中国海洋生物名录[M].北京:科学出版社,2008:887-1066.LIU R Y.Checklist of marine biota of China Seas[M].Beijing:Science Press,2008:887-1066.(in Chinese)
[19] 陈大刚,张美昭.中国海洋鱼类[M].青岛:中国海洋大学出版社,2015.CHEN D G,ZHANG M Z.Marine fishes of China[M].Qingdao:China Ocean University Press,2015.(in Chinese)
[20] MIYA M,SATO Y,FUKUNAGA T,et al.MiFish,a set of universal PCR primers for metabarcoding environmental DNA from fishes:detection of more than 230 subtropical marine species[J].Royal Society Open Science,2015,2(7):150088.
[21] HONGO Y,NISHIJIMA S,KANAMORI Y,et al.Fish environmental DNA in Tokyo Bay:a feasibility study on the availability of environmental DNA for fisheries[J].Regional Studies in Marine Science,2021,47:101950.
[22] JERDE C L.Can we manage fisheries with the inherent uncertainty from eDNA?[J].Journal of Fish Biology,2021,98(2):341-353.
[23] 李晨虹,凌岚馨,谭娟,等.环境DNA技术在水生生物监测中的挑战、突破和发展前景[J].上海海洋大学学报,2023,32(3):564-574.LI C H,LING L X,TAN J,et al.Challenge,breakthrough and future perspectives of environmental DNA technology in monitoring aquatic organisms[J].Journal of Shanghai Ocean University,2023,32(3):564-574.(in Chinese)
[24] HALLAM J,CLARE E L,JONES J I,et al.High frequency environmental DNA metabarcoding provides rapid and effective monitoring of fish community dynamics[J].Environmental DNA,2023,5(6):1623-1640.
[25] KECK F,BLACKMAN R C,BOSSART R,et al.Meta-analysis shows both congruence and complementarity of DNA and eDNA metabarcoding to traditional methods for biological community assessment[J].Molecular Ecology,2022,31(6):1820-1835.
[26] WANG X Y,LU G Q,ZHAO L L,et al.Assessment of fishery resources using environmental DNA:the large yellow croaker (Larimichthys crocea) in the East China Sea[J].Fisheries Research,2021,235:105813.
[27] TAKAHARA T,MINAMOTO T,YAMANAKA H,et al.Estimation of fish biomass using environmental DNA[J].PLoS One,2012,7(4):e35868.
[28] KNUDSEN S W,EBERT R B,HESSELSØE M,et al.Species-specific detection and quantification of environmental DNA from marine fishes in the Baltic Sea[J].Journal of Experimental Marine Biology and Ecology,2019,510:31-45.
[29] URBAN P,BEKKEVOLD D,HANSEN B K,et al.Using eDNA to estimate biomass of bycatch in pelagic fisheries[J].Environmental DNA,2024,6(1):e377.
[30] KLYMUS K E,RICHTER C A,CHAPMAN D C,et al.Quantification of eDNA shedding rates from invasive bighead carpHypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix[J].Biological Conservation,2015,183:77-84.