福建省沿海葡萄牙牡蛎遗传多样性和遗传结构分析
张钰霆1,吴镇2,李聪2,陈火荣2,黄泽坤3,何水清1,张智1,穆景利1*
(1.闽江学院 地理与海洋学院,福建 福州 350108;2.福建省渔业资源监测中心,福建 福州 350003;3.厦门大学 海洋与地球学院 近海海洋环境科学国家重点实验室,福建 厦门 361102)
摘要:为研究福建省沿海葡萄牙牡蛎(Crassostrea angulata)天然群体的遗传结构和遗传多样性,根据线粒体COⅠ基因序列对从福建省沿海自然海区(福清、莆田、泉州和东山)采集的373个牡蛎进行鉴定,共发现62个葡萄牙牡蛎,占比16.6%;随后采用GBS(genotyping-by-sequencing)简化基因组测序技术对这4个葡萄牙牡蛎地理群体(福清、莆田、泉州和东山分别为12、12、10、12个)进行遗传多样性、遗传分化及遗传结构分析。结果表明:4个葡萄牙牡蛎群体的观测杂合度(Ho)为0.243~0.271,其中,东山群体的Ho值最高(0.271),福清群体的Ho值最低(0.243);4个群体之间的遗传分化系数(Fst)为0.021 0~0.036 8,其中,东山群体和泉州群体的遗传分化系数最高(0.036 8);遗传结构分析显示,东山群体单独为一个类群,福清、莆田和泉州为另一个类群。研究表明,福建省沿海葡萄牙牡蛎群体的遗传多样性处于较低水平,种质资源有所退化;在种群分化方面,东山群体与福清、莆田和泉州3个群体形成了较明显的遗传分化。
关键词:葡萄牙牡蛎;GBS简化基因组测序;遗传多样性;遗传结构
DOI:10.16535/j.cnki.dlhyxb.2023-214
文章编号:2095-1388(2024)02-0275-06
中图分类号:S 917.4
文献标志码:A
收稿日期:2023-09-28
基金项目:福建省科技厅科研项目(2023N0028)
作者简介:张钰霆(1990—),男,博士,副教授。E-mail:ytzhang@mju.edu.cn
通信作者:穆景利(1979—),男,博士,教授。E-mail:jlmu@mju.edu.cn
葡萄牙牡蛎(Crassostrea angulata),又称“福建牡蛎”,隶属于软体动物门(Mollusca)双壳纲 (Bivalvia)珍珠贝目(Pterioida)牡蛎科(Ostreidae)。因其肉质鲜美、营养价值高,且具有生长快、产量高和环境适应能力强等优点,已成为世界水产养殖单种产量最高的贝类[1-3]。在中国,葡萄牙牡蛎主要分布于长江以南地区,是福建、广东和浙江省沿海的重要养殖贝类[2,4]。近年来,随着葡萄牙牡蛎产业的迅速发展,高密度养殖、近亲繁殖和累代人工育苗等在一定程度上引起了种质退化、遗传多样性降低等问题,对葡萄牙牡蛎的种质资源造成了严重影响[5-6]。在牡蛎人工育苗生产实践中,部分亲本来自自然海区的天然群体[7]。葡萄牙牡蛎天然群体的种质资源退化,反向限制了水产养殖业的健康发展。因此,开展福建省沿海葡萄牙牡蛎天然群体遗传多样性的研究,在分子水平上分析其种群遗传结构,深入了解不同群体的遗传特征和种群分化,对葡萄牙牡蛎的种质资源保护和开发利用具有重要意义。
单核苷酸多态性(SNP)是高密度遗传图谱构建和种质资源保护等领域的理想标记之一[8-9]。相比于AFLP技术和微卫星SSR技术,基于SNP的遗传分析具有更高的覆盖度和准确性[10-12]。GBS (genotyping-by-sequencing)简化基因组测序技术,是指利用合适的限制性内切酶,对全基因组进行特异性酶切,获取具有代表性的基因组片段;然后基于高通量测序平台进行测序,获得单核苷酸多态性SNP分子标记,并进行基因分型[13-14]。随着二代测序平台的发展和测序费用的降低,GBS 技术迅速发展,能够在较短的时间内开发出大量的SNP标记,是目前开发SNP分子标记最为有效、经济的方法之一[15]。
本研究中,利用GBS简化基因组测序技术对福建省沿海葡萄牙牡蛎的4个不同地理群体进行遗传结构和遗传多样性分析,以期为葡萄牙牡蛎种质资源研究提供科学参考。
1 材料与方法
1.1 样品采集
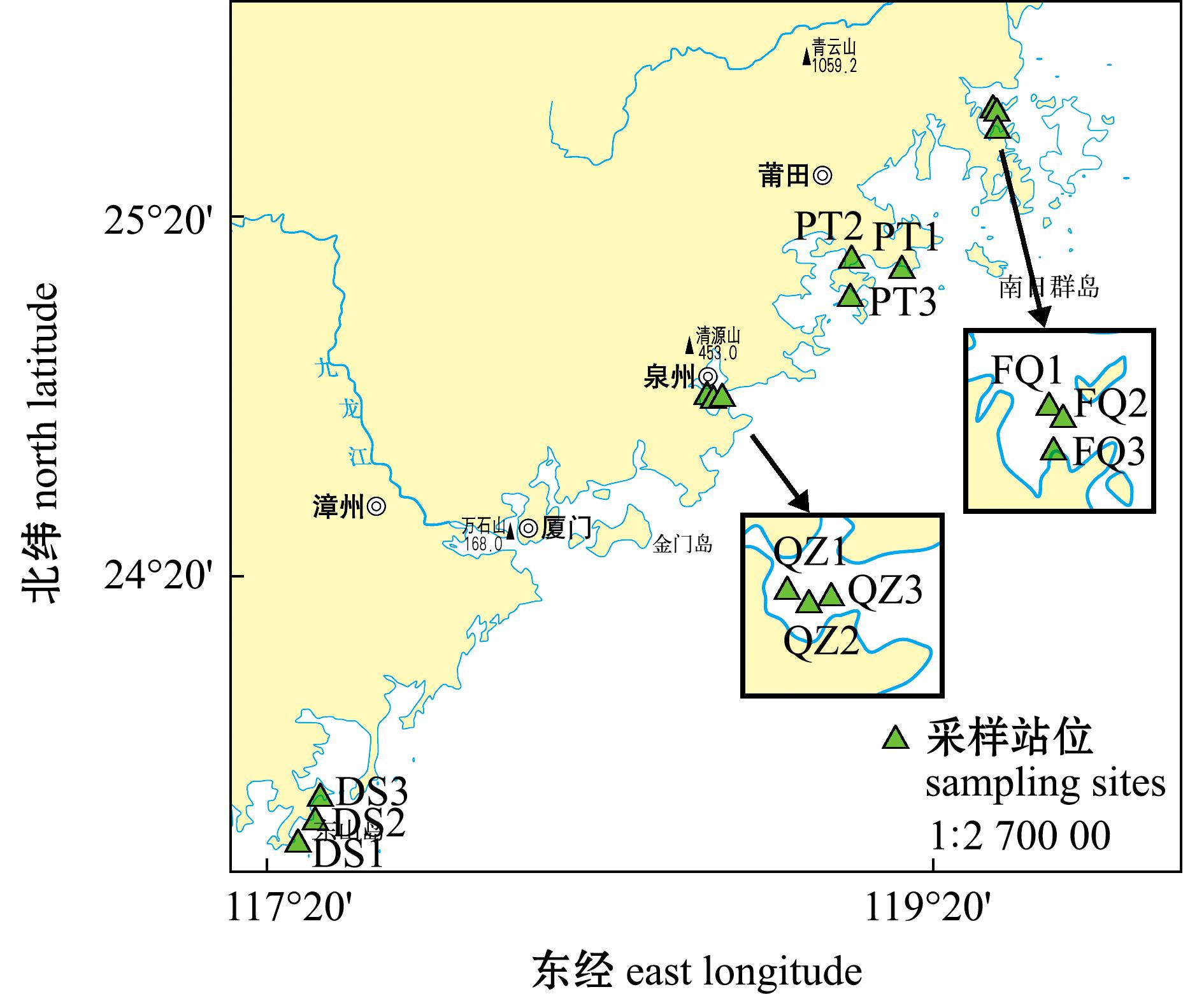
于2020年5—8月在福建省沿海主要自然海区选定4个采集地点,从北向南依次为福清、莆田、泉州和东山,随机采集适量牡蛎样品。采样信息见表1和图1。对样品进行解剖,取其闭壳肌置于-80 ℃超低温冰箱中保存备用。
表1 葡萄牙牡蛎采样信息
Tab.1 Sampling information on Crassostrea angulata
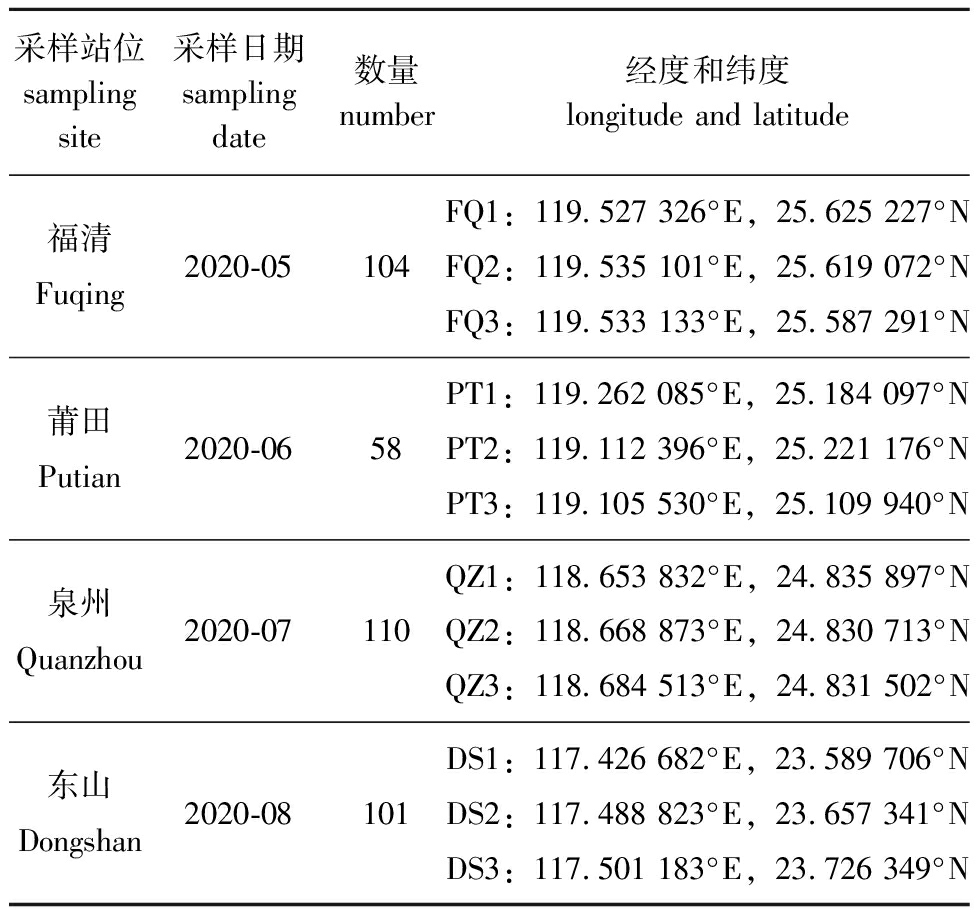
采样站位 sampling site采样日期 sampling date数量 number经度和纬度 longitude and latitude福清 Fuqing2020-05104FQ1:119.527 326°E,25.625 227°NFQ2:119.535 101°E,25.619 072°NFQ3:119.533 133°E,25.587 291°N莆田 Putian2020-0658PT1:119.262 085°E,25.184 097°NPT2:119.112 396°E,25.221 176°NPT3:119.105 530°E,25.109 940°N泉州 Quanzhou2020-07110QZ1:118.653 832°E,24.835 897°NQZ2:118.668 873°E,24.830 713°NQZ3:118.684 513°E,24.831 502°N东山 Dongshan2020-08101DS1:117.426 682°E,23.589 706°NDS2:117.488 823°E,23.657 341°NDS3:117.501 183°E,23.726 349°N
1.2 方法
1.2.1 牡蛎DNA的提取与鉴定 采用天根生化科技(北京)有限公司试剂盒提取牡蛎样品DNA,用琼脂糖凝胶电泳分析DNA的纯度和完整性,使用Nanodrop和Qubit仪器检测DNA的浓度和纯度,选取吸光度比值(A260 nm/A280 nm)为1.8~2.0的基因组DNA进行分装,并于-20 ℃下保存备用。根据巨牡蛎属线粒体 COⅠ序列,基于多重种类特异性PCR技术[14-15],通过PCR扩增产物条带大小初步判断牡蛎种类。委托生工生物工程(上海)股份有限公司进行测序,进一步确定其种类。
1.2.2 GBS文库构建与测序 选择质检合格的基因组DNA样本,选用限制性内切酶MseⅠ和MspⅠ对基因组进行酶切,添加接头后回收长度为220~450 bp的片段,完成测序样本文库的构建。利用二代测序技术,在Illumina HiSeq 测序平台对文库进行双末端PE(Paired-end)150测序。
1.2.3 数据分析与位点检测 对全基因组测序原始数据进行质量控制,去掉含接头序列、低质量碱基和未测出碱基的reads,分析GC含量分布情况,过滤得到有效数据。统计酶切片段,采用SAMTOOLS等软件检测SNP位点,经过滤后获得高质量的SNP位点。
1.2.4 群体遗传多样性分析 基于过滤后的SNP位点,采用Arlequin软件[16]计算各群体的观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)、等位基因数(Na)和有效等位基因数(Ne),并进行群体核苷酸多样性(Pi)比较分析。
1.2.5 群体遗传分化与遗传结构分析 采用Arlequin软件进行分子方差分析(AMOVA),评估群体遗传变异的来源。基于个体基因组SNP差异程度,计算群体之间的遗传距离和遗传分化系数(Fst);运用TreeBeST软件[17]计算距离矩阵,通过邻接法构建系统进化树;运用GCTA软件[18]计算特征向量及特征值,并利用R软件绘制PCA分布图;采用Admixture软件[19]进行群体遗传结构分析,并结合PCA分布图和系统进化树判断最优的分群数(K值)。
2 结果与分析
2.1 葡萄牙牡蛎的鉴定
采用多重种类特异性PCR方法分析了从福建省沿海4个自然海区采集的373个牡蛎样品,鉴定共获得62个葡萄牙牡蛎,其中,福清15个(占采集牡蛎样品的14.42%),莆田16个(占27.59%),泉州13个(占11.82%),东山18个(占17.82%)(表2)。
表2 葡萄牙牡蛎鉴定结果
Tab.2 Results of the identification of Crassostrea angulata

群体population牡蛎样本总数total number of the samples葡萄牙牡蛎数量(占比/%)C.angulata number非葡萄牙牡蛎数量(占比/%) other oyster number熊本牡蛎 C.sikamea囊牡蛎属 Saccostrea sp.福清 FQ10415 (14.42)68 (65.38)21 (20.19)莆田 PT5816 (27.59)42 (72.41)0 (0.00)泉州 QZ11013 (11.82)86 (78.18)11 (10.00)东山 DS10118 (17.82)41 (40.59)42 (41.58)
2.2 群体的遗传多样性
4个群体共46个葡萄牙牡蛎的DNA样品符合上机测序要求,其中,福清12个,泉州10个,莆田12个,东山12个。原始数据过滤后,获得高质量SNPs位点共51 478个。群体遗传多样性分析显示:4个葡萄牙牡蛎群体的Na值为1.788~1.829,Ne值为1.302~1.323;Ho值为0.243~0.271,其中,东山群体Ho值最高(0.271),福清群体Ho值最低(0.243);He值为0.228~0.247,PIC值为0.169~0.177,Pi值为0.213~0.225(表3)。这表明,福建省沿海葡萄牙牡蛎的遗传多样性较低(PIC<0.25),4个站点中,东山群体的遗传多样性程度最高。
表3 葡萄牙牡蛎不同地理群体的遗传多样性指数
Tab.3 Genetic diversity indices of different geographical populations of Crassostrea angulata

群体population等位基因Na有效等位基因Ne观测杂合度Ho期望杂合度He多态信息含量PIC核苷酸多样性Pi福清 FQ1.8241.3020.2430.2300.1690.213莆田 PT1.8291.3020.2560.2280.1690.213泉州 QZ1.7881.3060.2660.2430.1690.216东山 DS1.8201.3230.2710.2470.1770.225
2.3 群体的遗传分化与遗传结构
泉州、福清、莆田和东山4个群体间的Fst值为0.021 0~0.036 8,种间遗传距离为0.014 3~0.022 3(表4)。这表明,群体间遗传分化程度低(Fst<0.05),遗传距离较小。
表4 群体间遗传分化系数和标准遗传距离
Tab.4 Genetic differentiation coefficient and genetic distance between populations
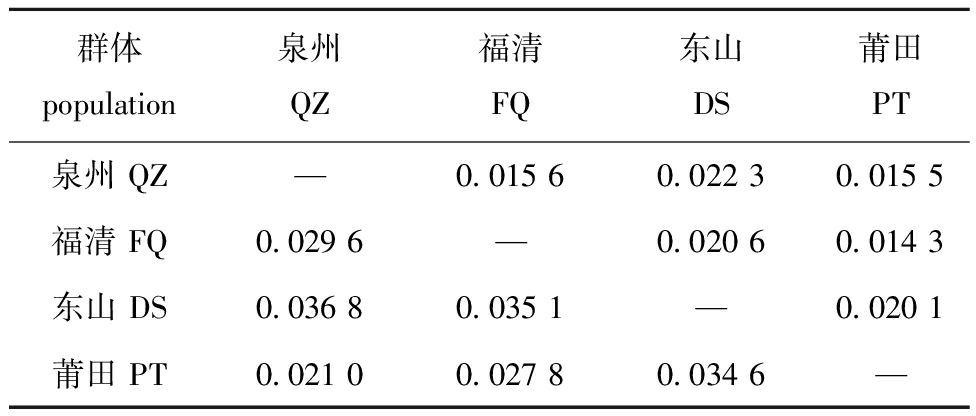
群体population泉州QZ福清FQ东山DS莆田PT泉州 QZ—0.015 60.022 30.015 5福清 FQ0.029 6—0.020 60.014 3东山 DS0.036 80.035 1—0.020 1莆田 PT0.021 00.027 80.034 6—
注:下三角为种群间遗传分化系数(Fst);上三角为种群间遗传距离。
Note:Genetic differentiation coefficient(Fst)between populations is shown by lower triangle,and genetic distance between populations is shown by upper triangle.
AMOVA分析显示,4个群体间无显著性遗传分化(P>0.05),98.62%的遗传变异来自群体内,而群体间的遗传变异仅为1.38%(表5)。系统进化树分析显示,4个群体分为2支,其中,泉州、福清和莆田3个群体聚为一支,东山群体单独为一支(图2)。
表5 群体间分子方差分析
Tab.5 AMOVA analysis between populations
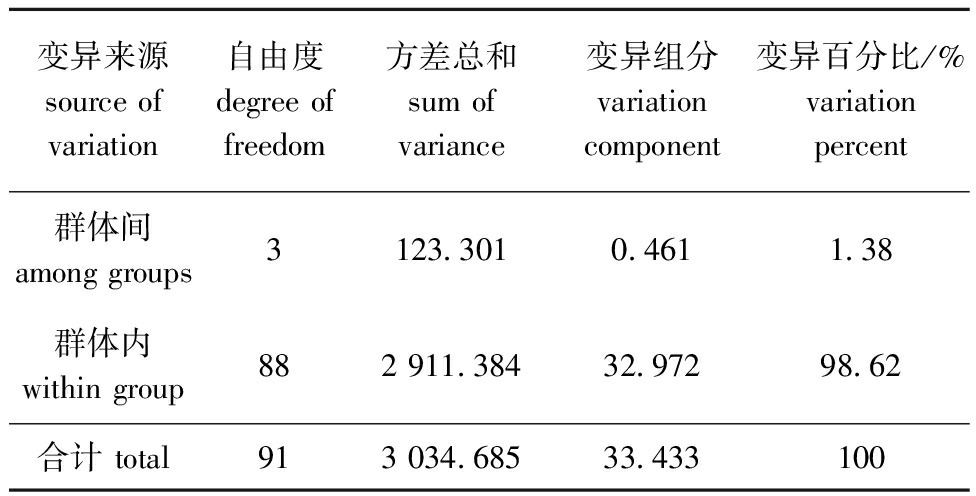
变异来源source of variation自由度degree of freedom方差总和sum of variance变异组分variation component变异百分比/%variation percent群体间among groups3123.3010.4611.38群体内within group882 911.38432.97298.62合计 total913 034.68533.433100
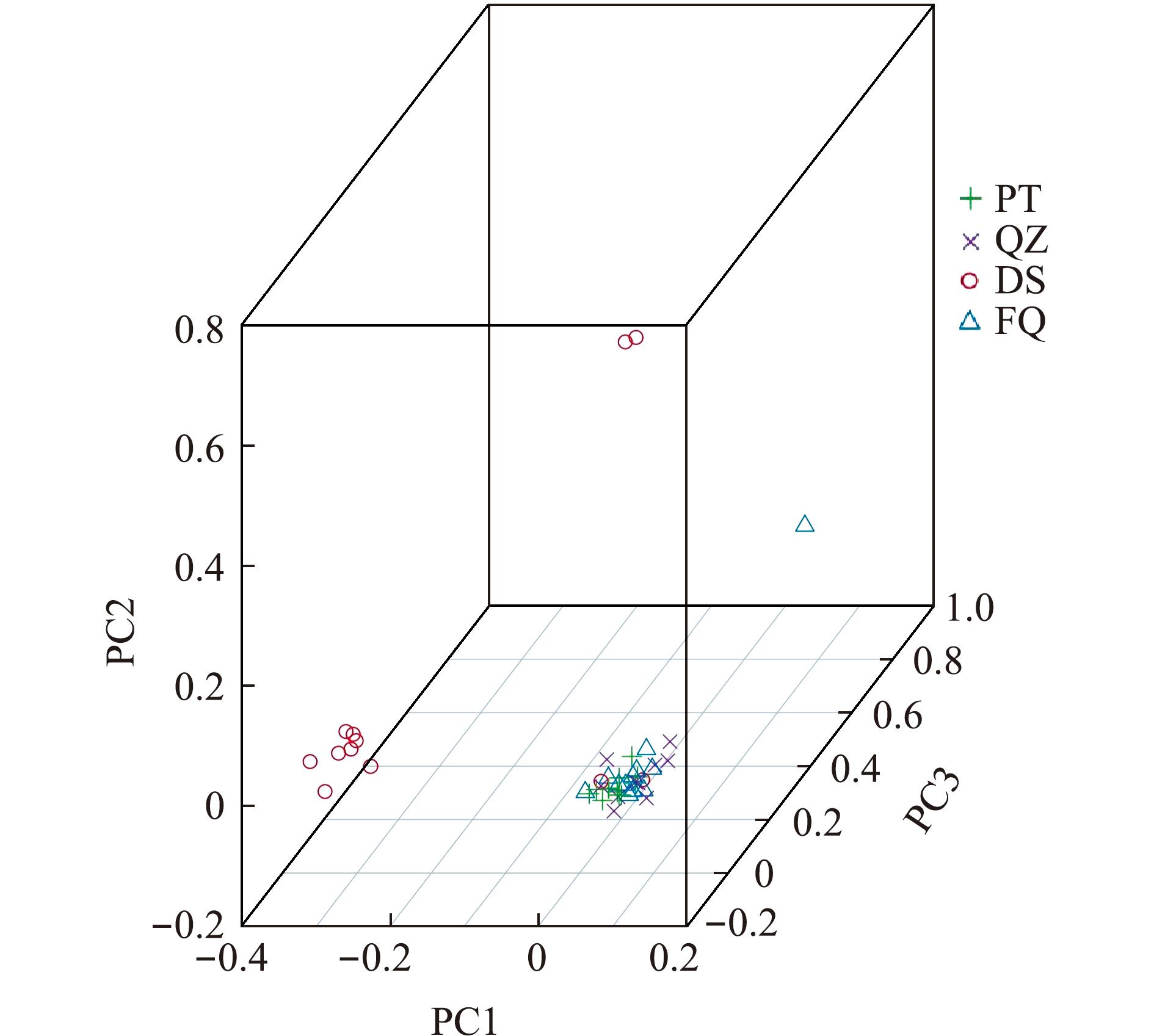
主成分分析(PCA)结果与系统进化树分析结果类似,东山群体的大部分样品聚集成簇,福清、莆田和泉州群体聚为另外一簇且无法相互区分(图3)。以上结果表明,东山群体与其他3个群体的遗传背景差异较大。
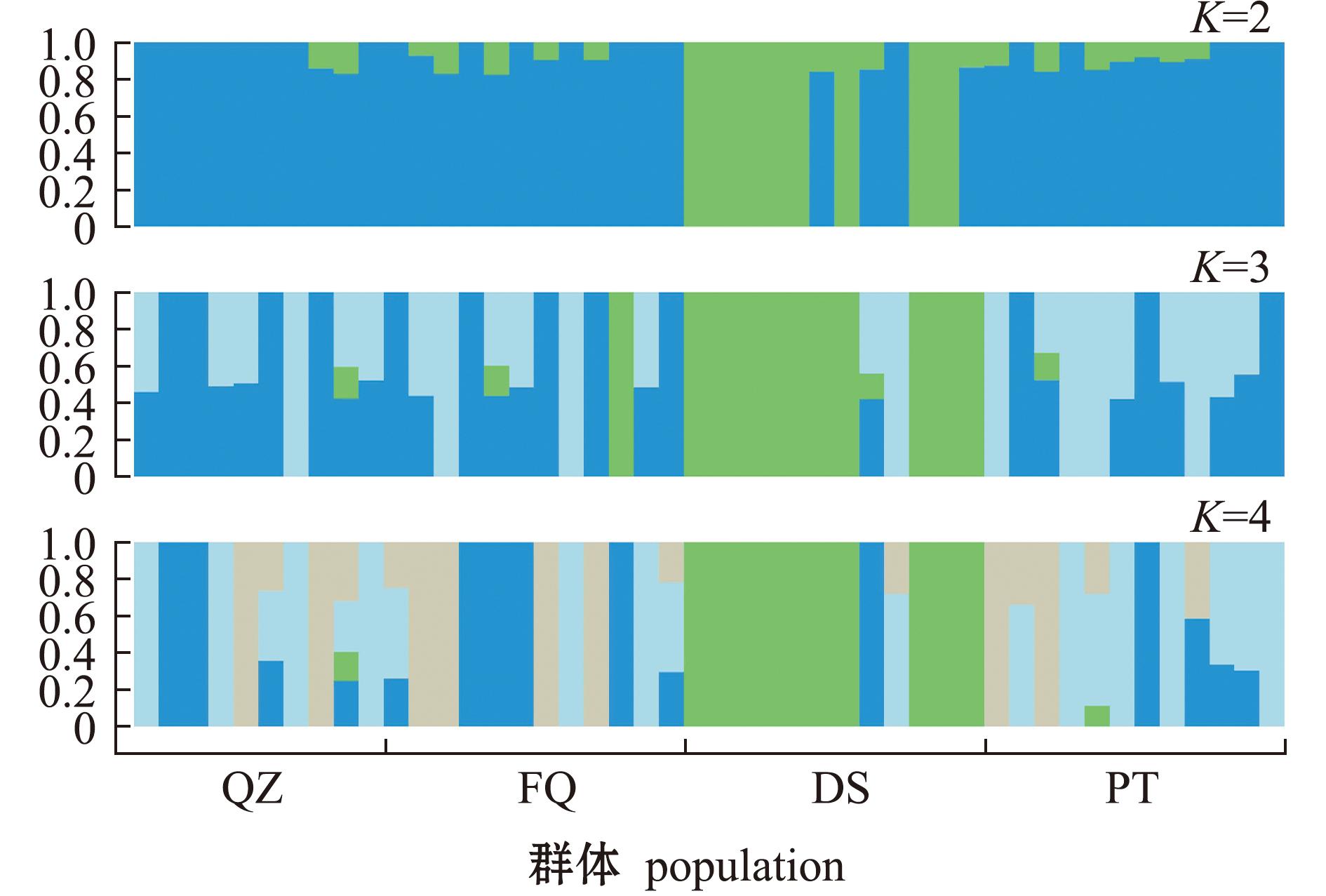
葡萄牙牡蛎遗传结构分析结果如图4所示,结合CV(cross validation error)曲线、PCA和群体进化树分析结果,综合判断最优K值为2,表明4个地理群体可分为2个类群,即东山群体单独为一个类群,福清、莆田和泉州群体为另一个类群。
3 讨论
3.1 福建省沿海葡萄牙牡蛎的占比
本研究中分析了福建省沿海4个自然海区373个牡蛎样品,共鉴定获得62个葡萄牙牡蛎,葡萄牙牡蛎所占比例约为16.6%。2009年,杜玄等[1]随机抽取了福建省沿海327个牡蛎样本进行线粒体COⅠ基因的鉴定,结果发现,有200个为葡萄牙牡蛎,其所占比例约为61.2%。2011年,巫旗生[20]分析了福建海区采集的180个牡蛎个体样本,结果发现,有163个为葡萄牙牡蛎,其所占比例高达90%。2021—2022年,于诗奇等[21]对福建省沿海19个采样点共960个牡蛎样品进行分析,葡萄牙牡蛎样本总体占比为63.23%,且各站点葡萄牙牡蛎所占比例差异较大,其中,葡萄牙牡蛎占比低于10%的站点有4个。本研究中发现较低的葡萄牙牡蛎占比,可能与采样点位置选择有关。
3.2 葡萄牙牡蛎4个群体的遗传多样性比较
杂合度能够反映群体遗传多态性的均匀度,是衡量群体遗传多样性的重要指标。本研究中,采用GBS简化基因组测序技术分析了福建省沿海4个葡萄牙牡蛎地理群体的遗传多样性,观测杂合度为0.243~0.271,期望杂合度为0.228~0.247,该结果与巫旗生等[2]采用AFLP方法分析的期望杂合度(0.230~0.242)相接近,说明福建省沿海葡萄牙牡蛎群体的遗传多样性较低。4个群体的观测杂合度在数值上均大于期望杂合度,说明群体中不存在杂合子缺失现象。此外,4个群体较低的等位基因数(Na<5)和多态信息含量(PIC<0.25)提示福建省沿海葡萄牙牡蛎的遗传多样性处于较低水平,可能存在种质资源衰退的问题,需采取相应的种质管理措施以保护其遗传多样性。福建省是葡萄牙牡蛎养殖大省,人工养殖群系的扩散导致天然群体种质资源被污染,加上牡蛎养殖缺乏系统有效的育种规划[6],长期种质混乱等多因素可能导致了天然群体遗传多样性的降低。
3.3 葡萄牙牡蛎4个群体的遗传分化与遗传结构比较
本研究中,葡萄牙牡蛎4个群体间的遗传分化系数均小于0.05,表明群体间未发生明显的遗传分化现象,群体遗传背景同质性较高。AMOVA分析结果显示,大部分遗传变异来自种群内部,种群间的变异程度极低,表明4个葡萄牙牡蛎地理群体的遗传分化较弱。群体遗传分化结果显示,东山群体与其他群体间的遗传分化系数均大于0.03,说明东山群体与其他群体存在较大的遗传分化。群体遗传结构分析结果也表明,东山群体单独为一个类群,福清、泉州和莆田3个群体为另一个类群,说明东山群体与其他群体形成了地理隔离。Ma等[22]对近江牡蛎(Crassostrea hongkongensis)研究发现,广西与广东的近江牡蛎群体由于雷州半岛的阻隔形成了地理隔离。除了牡蛎,还有其他海洋贝类也出现了这种地理隔离现象,如李成华等[23]发现福建南北部4个泥蚶(Tegillarca granosa)群体存在地理隔离现象;Miller等[24]对澳大利亚南部的三角斧蛤(Donax deltoides)开展群体遗传结构分析时发现,巴斯海峡是阻碍其幼体扩散的物理屏障。葡萄牙牡蛎成体为固着生活的物种,幼体虽然可浮游,但迁移的距离比较有限[25]。因此,笔者推测东山与其他3个站点(福清、泉州和莆田)较大的地理距离(中间隔着九龙江入海口的半咸水区),可能是造成群体间产生遗传分化的一个重要原因。
4 结论
1)福建省沿海4个葡萄牙牡蛎地理群体的等位基因数为1.788~1.829,有效等位基因数为1.302~1.323,观测杂合度为0.243~0.271,期望杂合度为0.228~0.247,多态信息含量为0.169~0.177,核苷酸多样性为0.213~0.225,表明福建省沿海葡萄牙牡蛎群体的遗传多样性较低,存在种质资源退化问题。
2)4个地理群体之间的遗传分化系数为0.021 0~0.036 8,遗传距离为0.014 3~0.022 3,表明群体间遗传分化程度低,遗传距离较小。
3)4个葡萄牙牡蛎地理群体中,东山群体单独为一个类群,福清、莆田和泉州为另一个类群,形成了较明显的遗传分化。
参考文献:
[1] 杜玄,郭希明,钱鲁闽.福建沿海巨蛎属牡蛎的主要种类及其分布[J].台湾海峡,2009,28(3):399-404. DU X,GUO X M,QIAN L M.Distribution of Crassostrea oysters in coastal sea of Fujian[J].Journal of Oceanography in Taiwan Strait,2009,28(3):399-404.(in Chinese)
[2] 巫旗生,宁岳,曾志南,等.福建沿海葡萄牙牡蛎养殖群体遗传多样性的AFLP分析[J].上海海洋大学学报,2013,22(3):328-333. WU Q S,NING Y,ZENG Z N,et al.Genetic diversity analysis of cultivated Crassostrea angulata populations along the coastline of Fujian by AFLP[J].Journal of Shanghai Ocean University,2013,22(3):328-333.(in Chinese)
[3] 章超桦.牡蛎营养特性及功能活性研究进展[J].大连海洋大学学报,2022,37(5):719-731. ZHANG C H.Research progress on nutritional characteristics and bioactivities of oysters:a review[J].Journal of Dalian Ocean University,2022,37(5):719-731.(in Chinese)
[4] WANG H Y,QIAN L M,LIU X A,et al.Classification of a common cupped oyster from southern China[J].Journal of Shellfish Research,2010,29(4):857-866.
[5] 张荣良,王卫军,冯艳微,等.长牡蛎(Crassostrea gigas)壳宽快速生长选育群体遗传多样性及遗传结构的微卫星标记分析[J].渔业科学进展,2016,37(4):90-96. ZHANG R L,WANG W J,FENG Y W,et al.Assessment of genetic variability and microsatellite analysis of Pacific oyster(Crassostrea gigas) after artificial selection of the shell width[J].Progress in Fishery Sciences,2016,37(4):90-96.(in Chinese)
[6] LAZOSKI C,GUSM O J,BOUDRY P,et al.Phylogeny and phylogeography of Atlantic oyster species:evolutionary history,limited genetic connectivity and isolation by distance[J].Marine Ecology Progress Series,2011,426:197-212.
O J,BOUDRY P,et al.Phylogeny and phylogeography of Atlantic oyster species:evolutionary history,limited genetic connectivity and isolation by distance[J].Marine Ecology Progress Series,2011,426:197-212.
[7] 曾志南,宁岳.福建牡蛎养殖业的现状、问题与对策[J].海洋科学,2011,35(9):112-118. ZENG Z N,NING Y.History,status and future of oyster culture in Fujian China[J].Marine Sciences,2011,35(9):112-118.(in Chinese)
[8] LI G,HUBERT S,BUCKLIN K,et al.Characterization of 79 microsatellite DNA markers in the Pacific oyster Crassostrea gigas[J].Molecular Ecology Notes,2003,3(2):228-232.
[9] 赵莲,薛蓓,高焕,等.SNP分子标记技术在经济甲壳动物中的应用进展[J].海洋渔业,2017,39(2):233-240. ZHAO L,XUE B,GAO H,et al.Progress on the SNP molecular markers in economic crustaceans[J].Marine Fisheries,2017,39(2):233-240.(in Chinese)
[10] WU T D,NACU S.Fast and SNP-tolerant detection of complex variants and splicing in short reads[J].Bioinformatics,2010,26(7):873-881.
[11] LI R Q,LI Y R,FANG X D,et al.SNP detection for massively parallel whole-genome resequencing[J].Genome Research,2009,19(6):1124-1132.
[12] NIELSEN R,PAUL J S,ALBRECHTSEN A,et al.Genotype and SNP calling from next-generation sequencing data[J].Nature Reviews Genetics,2011,12(6):443-451.
[13] ELSHIRE R J,GLAUBITZ J C,SUN Q,et al.A robust,simple genotyping-by-sequencing (GBS) approach for high diversity species[J].PLoS One,2011,6(5):e19379.
[14] DAVEY J W,HOHENLOHE P A,ETTER P D,et al.Genome-wide genetic marker discovery and genotyping using next-generation sequencing[J].Nature Reviews Genetics,2011,12(7):499-510.
[15] 边力,王军.简化基因组测序技术及其在海洋生物研究中的应用[J].厦门大学学报(自然科学版),2017,56(1):3-12. BIAN L,WANG J.The reduced-representation sequencing technique and its applications in marine organisms[J].Journal of Xiamen University (Natural Science Edition),2017,56(1):3-12.(in Chinese)
[16] EXCOFFIER L,LISCHER H E.Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J].Molecular Ecology Resources,2010,10(3):564-567.
[17] VILELLA A J,SEVERIN J,URETA-VIDAL A,et al.EnsemblCompara GeneTrees:complete,duplication-aware phylogenetic trees in vertebrates[J].Genome Research,2009,19(2):327-335.
[18] YANG J,LEE S H,GODDARD M E,et al.GCTA:a tool for genome-wide complex trait analysis[J].The American Journal of Human Genetics,2011,88(1):76-82.
[19] ALEXANDER D H,NOVEMBRE J,LANGE K.Fast model-based estimation of ancestry in unrelated individuals[J].Genome Research,2009,19(9):1655-1664.
[20] 巫旗生.福建沿海主要养殖牡蛎种类及其遗传多样性研究[D].长沙:湖南农业大学,2012. WU Q S.Studies on taxonomy and genetic diversity of cultured oysters in coastal sea of Fujian[D].Changsha:Hunan Agricultural University,2012.(in Chinese)
[21] 于诗奇,韩自强,陈燕婷,等.福建沿海巨蛎属(Crassostrea)牡蛎的种类及其分布[J].海洋与湖沼,2023,54(6):1682-1692. YU S Q,HAN Z Q,CHEN Y T,et al.The species and distribution of Crassostrea oyster in Fujian coast[J].Oceanologia et Limnologia Sinica,2023,54(6):1682-1692.(in Chinese)
[22] MA H T,LI L,XIAO S,et al.Microsatellite-based study of population genetics of Crassostrea hongkongensis in Southern China[J].Aquaculture Reports,2021,19:100591.
[23] 李成华,李太武,宋林生,等.福建南北泥蚶种内分化的RAPD分析[J].动物学研究,2003,24(5):362-366. LI C H,LI T W,SONG L S,et al.RAPD analysis on intra-species differentiation of Tegillarca granosa populations to the south and north of Fujian Province[J].Zoological Research,2003,24(5):362-366.(in Chinese)
[24] MILLER A D,VERSACE V L,MATTHEWS T G,et al.Ocean currents influence the genetic structure of an intertidal mollusc in southeastern Australia-implications for predicting the movement of passive dispersers across a marine biogeographic barrier[J].Ecology and Evolution,2013,3(5):1248-1261.
[25] 翁朝红,谢仰杰,肖志群,等.福建和广东南部葡萄牙牡蛎养殖群体和野生群体遗传多样性分析[J].热带海洋学报,2016,35(3):94-98. WENG Z H,XIE Y J,XIAO Z Q,et al.Analysis of genetic diversity in several wild and hatchery populations of Crassostrea angulata from South Fujian and South Guangdong[J].Journal of Tropical Oceanography,2016,35(3):94-98.(in Chinese)
Analysis of genetic diversity and genetic structure of Portuguese oyster (Crassostrea angulata ) from the coast of Fujian Province
ZHANG Yuting1,WU Zhen2,LI Cong2,CHEN Huorong2,HUANG Zekun3,HE Shuiqing1,ZHANG Zhi1,MU Jingli1*
(1.College of Geography and Oceanography,Minjiang University,Fuzhou 350108,China;2.Fujian Fisheries Resources Monitoring Center,Fuzhou 350003,China;3.State Key Laboratory of Marine Environmental Science,College of Oceans and Earth,Xiamen University,Xiamen 361102,China)
Abstract: In order to investigate genetic diversity and genetic structure of Portuguese oyster (Crassostrea angulata) from the coast of Fujian Province, China, 373 samples of the Portuguese oyster were collected from the coastal areas of Fuqing, Putian, Quanzhou and Dongshan in Fujian Province were sampled, and 62 out of the 373 oyster was identified as C.angulata based on their mitochondrial COⅠ sequences, accounting for 16.6%. Then, the genetic diversity, variation and population structure of these C.angulata were analyzed by the genotyping-by-sequencing (GBS) technology. Results showed that the heterozygosity (Ho) value ranged from 0.243 to 0.271, with the maximal Ho value of 0.271 in the Dongshan population, and the minimal Ho value of 0.243 in the Fuqing population. The Fst value among the 4 populations ranged from 0.021 0 to 0.036 8 with the maximal Fst value of 0.036 8 between the Dongshan and Quanzhou populations. In addition, the result of the genetic structure analysis showed that samples from the Dongshan population were separated from others (the Fuqing, Putian and Quanzhou populations), indicating that there was a low genetic diversity and potential germplasm recession of C.angulata in the coast of Fujian Province and a geographic isolation between the Dongshan population and the other three populations.
Key words: Crassostrea angulata; genotyping-by-sequencing (GBS); genetic diversity; genetic structure






 O J,BOUDRY P,et al.Phylogeny and phylogeography of Atlantic oyster species:evolutionary history,limited genetic connectivity and isolation by distance[J].Marine Ecology Progress Series,2011,426:197-212.
O J,BOUDRY P,et al.Phylogeny and phylogeography of Atlantic oyster species:evolutionary history,limited genetic connectivity and isolation by distance[J].Marine Ecology Progress Series,2011,426:197-212.