弗氏柠檬酸杆菌RPA-LFD快速检测方法的建立及应用
王姝然1,2,3,范厚勇4,徐嘉楠4 ,周天琦1,2,3 ,许丹1,2,3,郑跃平4*
(1.上海海洋大学 水产与生命学院,上海 201306;2.上海海洋大学 国家水生动物病原库,上海 201306;3.上海海洋大学 农业农村部淡水水产种质资源重点实验室,上海 201306;4.上海市水生野生动植物保护研究中心,上海 200092)
摘要:为建立一种针对弗氏柠檬酸杆菌(Citrobacter freundii)敏感性高、特异性强的临床快速检测方法,根据该菌定居因子基因cfa的保守序列,设计特异性引物,采用重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA),并结合琼脂糖凝胶电泳(AGE)和侧流层析试纸条(LFD)方法进行反应条件的优化及特异性与灵敏度的检测。结果表明:本研究中所建立的弗氏柠檬酸杆菌重组酶聚合酶扩增结合侧向流动试纸条(RPA-LFD)快速检测方法,在38 ℃条件下扩增20 min后,能特异性地检测到弗氏柠檬酸杆菌,对弗氏柠檬酸杆菌纯培养物和基因组DNA的最小检出限分别为1.5×102 CFU/mL和200 fg/μL,是常规PCR(cfa引物)检测灵敏度(20 pg/μL)的100倍;利用所建立的RPA-LFD法与常规PCR同时检测杂交鲟攻毒试验样本,两种方法的检测结果一致。研究表明,本研究中建立的弗氏柠檬酸杆菌RPA-LFD方法,操作简便、反应迅速、特异性好、灵敏度高,并且不需要昂贵的仪器,该方法可为未来弗氏柠檬酸杆菌细菌性疾病的早期诊断提供更有效的技术支持。
关键词:弗氏柠檬酸杆菌;快速检测;重组酶聚合酶扩增技术;侧向流动试纸条
DOI:10.16535/j.cnki.dlhyxb.2023-200
文章编号:2095-1388(2024)02-0241-09
中图分类号:S 943
文献标志码:A
收稿日期:2023-09-09
基金项目:上海市水生野生动植物保护研究中心委托项目“中华鲟主要细菌性疾病的预警和防控”(沪水野研专项2022-01号)
作者简介:王姝然(1999—),女,硕士研究生。E-mail:wsr11282023@163.com
通信作者:郑跃平( 1981—),男,工程师。E-mail:zyping1107@aliyun.com
柠檬酸杆菌属(Citrobacter)隶属于肠杆菌科(Enterobacteriaceae),普遍分布于自然水资源、污水废物、土壤环境和铝箔食品中[1]。目前,弗氏柠檬酸杆菌(Citrobacter freundii)已经成为影响野生和养殖鱼类最普遍的鱼类病原体之一,可感染野生陆生动物、两栖动物、龟类、水生哺乳动物和养殖鱼类,是一种典型的人-兽-鱼共患条件性致病菌[2]。已有研究表明,弗氏柠檬酸杆菌对团头鲂(Megalobrama amblycephala)[3]、虹鳟(Oncorhynchus mykiss)[4]、花鳗鲡(Anguilla marmorata)[5]和鲫(Carassius auratus)[6]等鱼类均具有致病性。
中华鲟(Acipenser sinensis)是一种洄游性鱼类,主要分布在长江流域,已被列入国际自然保护联盟(International Union for Conservation of Nature and Natural Resource,IUCN)濒危物种红色名录[7]。由于生境改变、污染、过度捕捞和筑坝的原因,野生中华鲟数量目前已大幅下降[8]。近年来,在集约化人工养殖条件下,鲟更容易暴发疾病,其中,细菌性疾病被认为是鲟养殖业的主要威胁,该病的流行对中华鲟养殖业造成了巨大的经济损失。研究人员已从中华鲟体内分离出分枝杆菌(Mycobacterium)[9]、维氏气单胞菌(Aeromonas veronii)[10]、嗜水气单胞菌(Aeromonas hydrophila)[11]和脑膜败血伊丽莎白菌(Elizabethkingia meningoseptica)[12]等致病菌。然而,中华鲟养殖过程中出现了一种由弗氏柠檬酸杆菌诱发死亡的疾病,该病原菌具有很强的致死性,中华鲟感染后会出现行动迟缓、食欲不振、腹部与肝脏充血的症状[13]。目前,针对该病原菌的治疗仍依赖于使用阿奇霉素、头孢噻肟、新霉素、多西环素和左氧氟沙星等抗生素,长期使用抗生素药物容易出现耐药性或药物残留等诸多风险,因此,建立一种快速、高效的弗氏柠檬酸杆菌现场检测技术,对早期预防和控制中华鲟感染弗氏柠檬酸杆菌疾病的发展尤为重要。
目前,生理生化鉴定仍然是检测弗氏柠檬酸杆菌的传统方法,但是使用分子生物学方法从基因层面探索细菌的分类地位和物种关系时具有鉴定速度快、准确性高的特点,在一定程度上弥补了传统方法鉴定周期长、易受培养条件影响和特征反应不明显等缺点。常规PCR、多重PCR及实时荧光定量PCR等分子生物学技术已被应用于检测弗氏柠檬酸杆菌的特异性基因,如cfa、ureD、ureF、viaB和opmA等。然而,上述方法均需要复杂的热循环装置和专业的人员来完成,且反应时间长,无法满足中华鲟养殖现场检测的诉求。目前,重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA)、环介导等温扩增技术(loop-mediated isothermal amplification,LAMP)、多重交叉位移扩增技术(multiple cross displacement amplification,MCDA)和依赖解旋酶的恒温基因扩增技术(helicase-dependent amplification,HAD)等快速检测技术已应用于水产养殖病原体的现场检测中。与LAMP和MCDA相比,RPA技术需要设计的引物较简单,同时具有操作简单、反应时间短和反应温度低等优点。RPA反应主要是通过重组酶打开DNA双链并在37~42 ℃温度内对DNA靶序列进行指数扩增,以便在30 min内完成反应[14]。侧向层析试纸条(lateral flow dipstick,LFD)技术是一种结合免疫层析技术用于DNA分子快速检测的技术,能够在3~5 min内快速检测扩增产物,不需要借助任何信号读取设备,用肉眼即可观察到结果。RPA-LFD技术已成功应用到布鲁氏菌(Brucella)[15]、美人鱼发光杆菌(Photobacterium damselae)[16]及金黄色葡萄球菌(Staphylococcus aureus)[17]等致病菌的检测中。因此,将RPA-LFD技术应用在弗氏柠檬酸杆菌的现场检测中具有重要的研究价值。
本研究中,将RPA扩增技术与LFD技术结合起来,以弗氏柠檬酸杆菌的定居因子cfa(cyclopropane fatty acyl phospholipid synthase)基因为靶基因,建立一种快速、特异和灵敏的弗氏柠檬酸杆菌检测技术,并组装开发相应的试剂盒,以期为未来中华鲟弗氏柠檬酸杆菌细菌性疾病的早期诊断提供更有效的技术支持。
1 材料与方法
1.1 材料
菌株:试验共使用菌株6株,包括嗜水气单胞菌(Ah-221212)、弗氏柠檬酸杆菌(CF1212)、大肠杆菌(Escherchia coli,EC2202)、副溶血弧菌(Vibrio parahaemolyticus,ATCC17802)、金黄色葡萄球菌(Sa220228)和维氏气单胞菌(AV2307)。维氏气单胞菌由上海海洋大学国家水生动物病原库从浙江湖州某黄颡鱼养殖场病鱼分离获得;弗氏柠檬酸杆菌和嗜水气单胞菌由本实验室从上海市水生野生动植物保护研究中心死亡中华鲟体内分离获得;副溶血弧菌由上海海洋大学胡鲲老师馈赠;其余菌株均由本实验室从不同患病水产动物分离获得。
主要试剂与仪器:DNA快速扩增试剂盒(基础型WLB8201KIT)、DNA恒温快速检测试剂盒(胶体金试纸条型WLN8203KIT)和彩虹型侧向流动试纸条(LFD)购自山东潍坊安普未来生物科技有限公司;微量样品基因组DNA提取试剂盒(DP316)、细菌基因组DNA提取试剂盒(DP302-02)购自天根生物技术公司;DNA Marker DL1000、DNA Marker DL2000、6×Loading Buffer和Premix TaqTM(Ex TaqTM version 2.0)购自TaKaRa公司;苯酚(分析纯)和三氯甲烷(分析纯)购自国药集团化学试剂有限公司。
1.2 方法
1.2.1 总DNA提取 挑取弗氏柠檬酸杆菌菌株接种到液体BHI培养基中,30 ℃下振荡培养8 h,取1 mL弗氏柠檬酸杆菌新鲜菌液,使用细菌基因组DNA提取试剂盒提取其基因组DNA,并将得到的DNA于-20 ℃下保存。用平板菌落计数法得到弗氏柠檬酸杆菌菌液浓度为1.5×109 CFU/mL,采用煮沸法提取DNA用于菌液灵敏度的检测,将弗氏柠檬酸杆菌菌液稀释为9个所需要浓度(1.5×108~1.5×101 CFU/mL),分别取1 mL菌液在100 ℃下处理10 min后,以12 000 r/min离心5 min,上清液即为模板。
1.2.2 引物设计及筛选 根据弗氏柠檬酸杆菌的cfa基因设计引物和探针,同时通过对GenBank中的CP055247.1、CP048416.1、CP024672.1、CP035276.1、CP086287.1、CP049015.1和CP056256.1的cfa基因序列进行比对分析,进一步明确弗氏柠檬酸杆菌cfa基因的保守区域。采用Primer Premier 6.0软件,以弗氏柠檬酸杆菌cfa的保守区域为基础,设计RPA引物,RPA引物长度设置为30~35 bp,产物长度设置为100~500 bp,GC含量设置为20%~80%。基于RPA引物对设计探针,探针设计长度不小于46 nt,探针5′端有FAM标记,3′端有C3间隔,探针中间位置有THF碱基置换,THF位点至少在30 nt之前,在15 nt之后,采用Oligo软件对探针进行筛选,以减少探针与引物之间的相互配对。所有引物和探针均由生工生物工程上海股份有限公司合成,具体的引物和探针详情见表1。
表1 试验所用引物和探针
Tab.1 Primers and probes used in the experiment
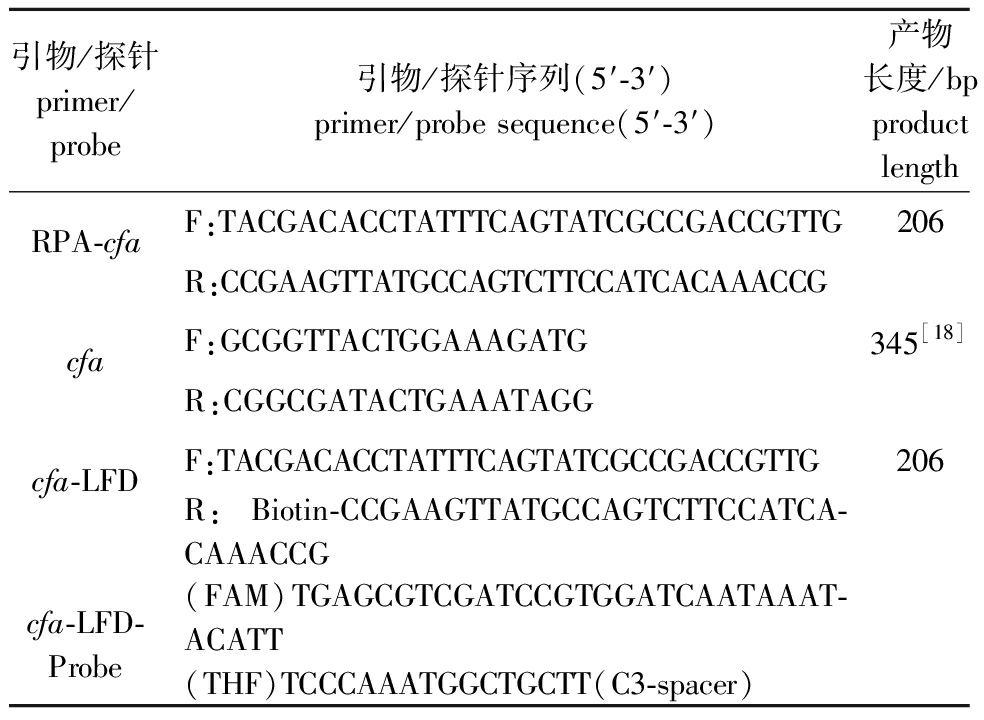
引物/探针 primer/probe引物/探针序列(5'-3') primer/probe sequence(5'-3')产物长度/bpproduct lengthRPA-cfaF:TACGACACCTATTTCAGTATCGCCGAC-CGTTG206R:CCGAAGTTATGCCAGTCTTCCATCA-CAAACCGcfaF:GCGGTTACTGGAAAGATG345[18]R:CGGCGATACTGAAATAGGcfa-LFDF:TACGACACCTATTTCAGTATCGCCGAC-CGTTG206R:Biotin-CCGAAGTTATGCCAGTCTTC-CATCACAAACCG cfa-LFD-Probe(FAM)TGAGCGTCGATCCGTGGAT-CAATAAATACATT(THF)TCCCAAATGGCTGCTT(C3-spacer)
1.2.3 RPA-AGE检测体系 RPA-AGE试验根据DNA 快速扩增试剂盒(基础型WLB8201KIT)说明书进行,在0.2 mg干粉管中加入50 μL的反应体系。反应体系包含:A Buffer 29.4 μL,RPA-cfa上、下游引物(10 μmol/L)各2 μL,灭菌水12.1 μL,弗氏柠檬酸杆菌基因组DNA 2 μL。将上述反应溶液混匀后,再加入2.5 μL B Buffer后启动反应,简短混匀离心,将干粉管放入金属浴中在38 ℃下孵育30 min。待反应结束后,加入等体积的酚氯仿溶液(苯酚与氯仿的体积比为1∶1)以12 000 r/min离心3 min,对RPA扩增产物进行提纯。离心后的上清液即为提纯后的RPA,将其用15 g/L琼脂糖凝胶电泳检测。
1.2.4 RPA-LFD检测体系 为了筛选探针和引物组合,使用DNA恒温快速检测试剂盒进行RPA-LFD反应。在冻干粉管中依次加入A Buffer 29.4 μL,cfa-LFD上、下游引物(10 μmol/L)各2 μL,探针(10 μmol/L)0.6 μL,弗氏柠檬酸杆菌基因组DNA 2 μL,ddH2O 11.5 μL。然后在以上反应混合液中加入2.5 μL B Buffer,充分混匀后离心,放入恒温金属浴中在38 ℃下孵育15~30 min。待反应结束后,取5 μL扩增产物添加到含95 μL纯水中,混匀后将侧向流动试纸条插入反应稀释液中等待约5 min,直至显示出测试线和控制(对照)线。
1.2.5 反应条件的优化 以弗氏柠檬酸杆菌的基因组为模板进行RPA-AGE和RPA-LFD反应,从而进一步确定该菌RPA-LFD反应的最佳条件。为了提高RPA扩增效率,对反应温度进行优化,在不同温度(30、33、35、37、38、40 ℃)下孵育30 min。确定好最佳温度后,将干粉管置于38 ℃恒温金属浴中分别反应10、15、20、25、30、40 min,确定反应所需的最短时间和最佳反应时间。
1.2.6 特异性试验 以弗氏柠檬酸杆菌、嗜水气单胞菌、金黄色葡萄球菌、大肠杆菌、维氏气单胞菌和副溶血弧菌的基因组DNA为模板,在上述优化得到的最佳反应条件下进行 RPA-LFD 和RPA-AGE检测,同时设置阴性对照(以ddH2O为模板),以评估该方法的特异性。
1.2.7 灵敏度试验 基因组检测灵敏度:采用10倍系列稀释法,对弗氏柠檬酸杆菌的纯基因组DNA进行稀释,以获得浓度分别为200 ng/μL、20 ng/μL、2 ng/μL,200 pg/μL、20 pg/μL、2 pg/μL,200 fg/μL、20 fg/μL的弗氏柠檬酸杆菌基因组DNA。以倍比稀释的DNA为模板,使用RPA引物进行PCR、RPA-LFD和RPA-AGE检测,同时使用常规PCR引物(cfa引物)(表1)进行扩增,比较4种不同检测方法之间灵敏度的差异。PCR反应体系(25 μL):Primer F和Primer R各1 μL,ddH2O 9.5 μL,DNA 1 μL,Premix TaqTM (Ex TaqTM version 2.0) 12.5 μL。扩增程序:94 ℃下预变性3 min,94 ℃下变性30 s,60 ℃下退火30 s,72 ℃下延伸30 s,共进行30个循环。将两种方法的敏感性进行对比分析。
纯菌液检测灵敏度:将培养至对数期的弗氏柠檬酸杆菌菌液进行10倍梯度稀释,采用煮沸法提取1.5×108~1.5×101 CFU/mL 8个浓度纯菌液的DNA,然后将其作为模板用于RPA-LFD和PCR检测,并将二者的检测结果进行比较。
1.2.8 攻毒试验样本的检测 鉴于中华鲟为濒危保护动物,本研究中选择与其亲缘关系较近的杂交鲟(Acipenser baerii♀×A.schrencki♂)作为攻毒对象。试验用杂交鲟20尾(体长为20~30 cm,体质量为50~100 g),由上海市水生野生动植物保护研究中心提供,并将其暂养 7 d后用于细菌感染。试验组随机取 10 尾杂交鲟腹腔注射弗氏柠檬酸杆菌菌悬液,注射量为200 μL/尾,菌液浓度为 3×109 CFU/mL,对照组同样选取 10 尾杂交鲟腹腔注射200 μL/尾PBS缓冲液。分别在攻毒后2、7、10、14 d后,取杂交鲟的鳃、肝脏、脾脏、肾脏、胰脏和肠道组织,利用微量样品基因组DNA提取试剂盒对采集的33份杂交鲟攻毒样品进行DNA提取,分别采用RPA-LFD及常规PCR法(cfa引物)进行样本检测,并将二者的检测结果进行比较,从而评估本试验中建立的弗氏柠檬酸杆菌检测方法对鲟样本的适用性,以确保该方法能实际应用于鲟养殖现场检测。
2 结果与分析
2.1 基于弗氏柠檬酸杆菌cfa基因的 RPA 扩增
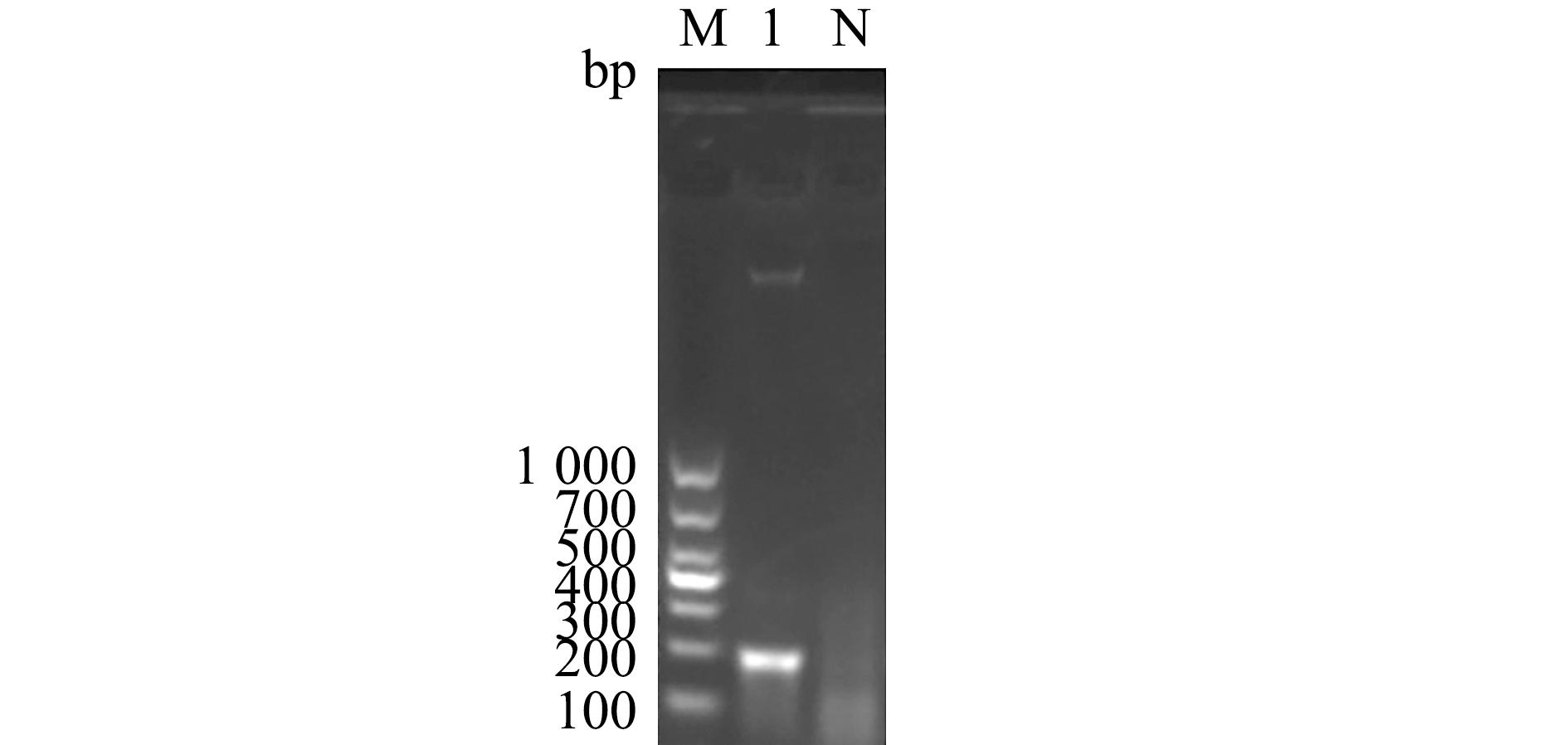
以cfa基因为靶序列设计的特异性引物对弗氏柠檬酸杆菌基因组 DNA 进行 RPA 扩增。结果获得一条约206 bp的目的条带,与预期结果一致(图1)。这表明,该特异性引物可用于检测弗氏柠檬酸杆菌。
2.2 RPA反应最佳温度的筛选
利用RPA引物组合对弗氏柠檬酸杆菌基因组DNA进行RPA-AGE反应,将反应温度分别设置为 30、33、35、37、38、40 ℃,反应时间为30 min。在温度37~40 ℃下,能看到明亮清晰的条带,其扩增效果要明显优于30、33、35 ℃(图2(a))。同时利用上述引物和探针进行RPA-LFD扩增,在30 ℃时试纸条检测线上出现较弱的条带,在33 ℃时出现比较清晰的条带,在35~40 ℃时出现明显清晰的条带,但与35 ℃相比,温度为37~40 ℃时试纸条检测线的颜色更深(图2(b))。这表明,该试验较好的反应温度为37~40 ℃,因此,后续试验选择38 ℃作为最佳反应温度。
2.3 RPA反应最佳时间的筛选
在38 ℃反应温度下设定了6个不同的时间,通过RPA-AGE反应可以看出,反应10、15 min时条带较浅,反应20~40 min时有明显清晰的条带且无差异(图3(a))。通过RPA-LFD反应可以看出,反应时间越长试纸条检测线上的条带越明显,反应10 min时就可以观察到结果,反应20 min时出现的条带比较明显,反应25、30 min时条带效果最好。因此,选择25 min作为后续试验的最佳反应时间(图3(b))。
2.4 特异性试验
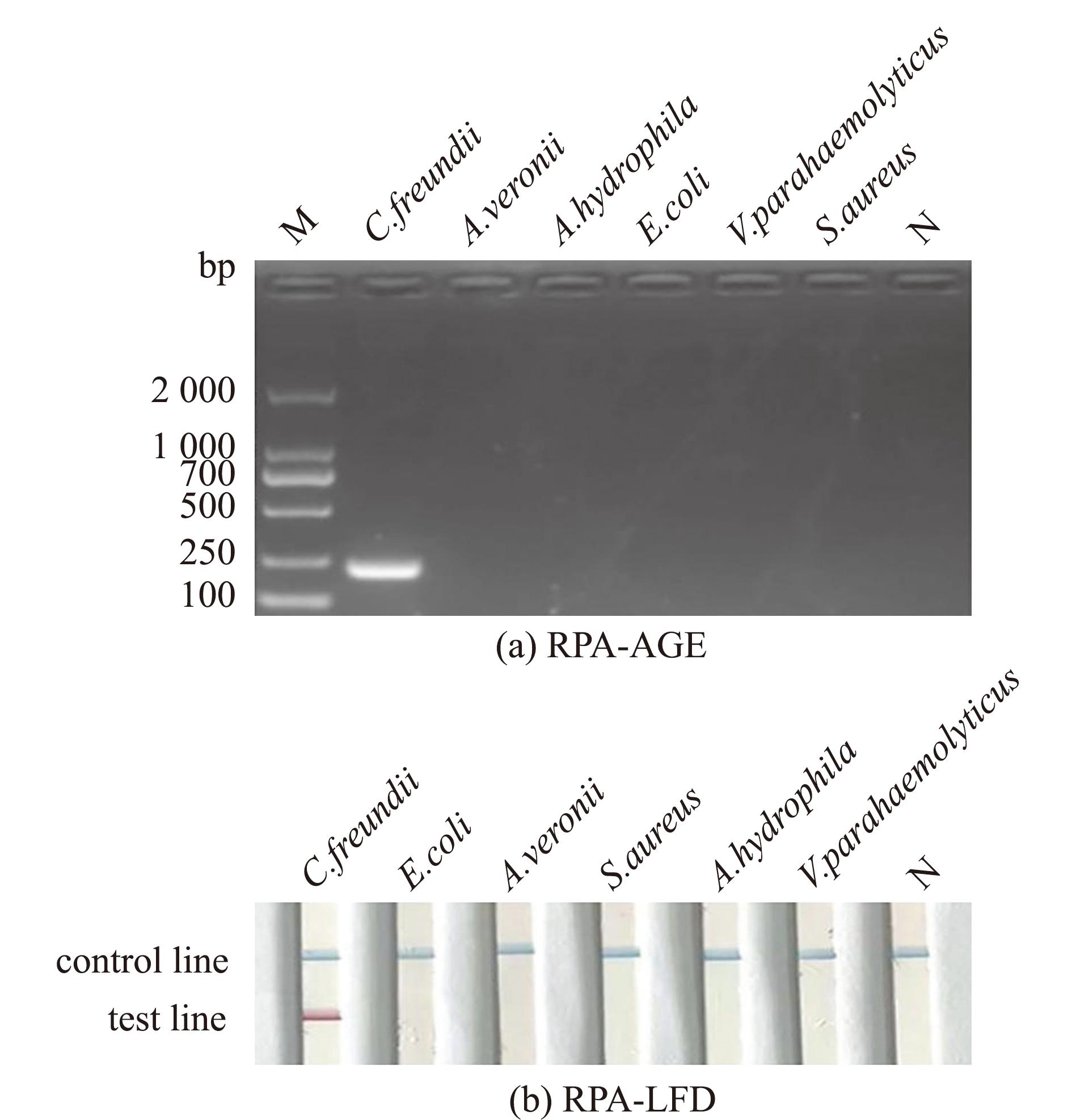
分别以弗氏柠檬酸杆菌、嗜水气单胞菌、金黄色葡萄球菌、大肠杆菌、维氏气单胞菌和副溶血弧菌的基因组DNA为模板进行RPA-LFD和RPA-AGE扩增,阴性对照不加模板,用ddH2O代替。从图4可见,无论是RPA-AGE方法还是RPA-LFD方法,都只有弗氏柠檬酸杆菌能够进行较好的扩增,与其他病原菌并无交叉反应,说明该方法对检测弗氏柠檬酸杆菌具有较强的特异性。
2.5 基因组检测灵敏度
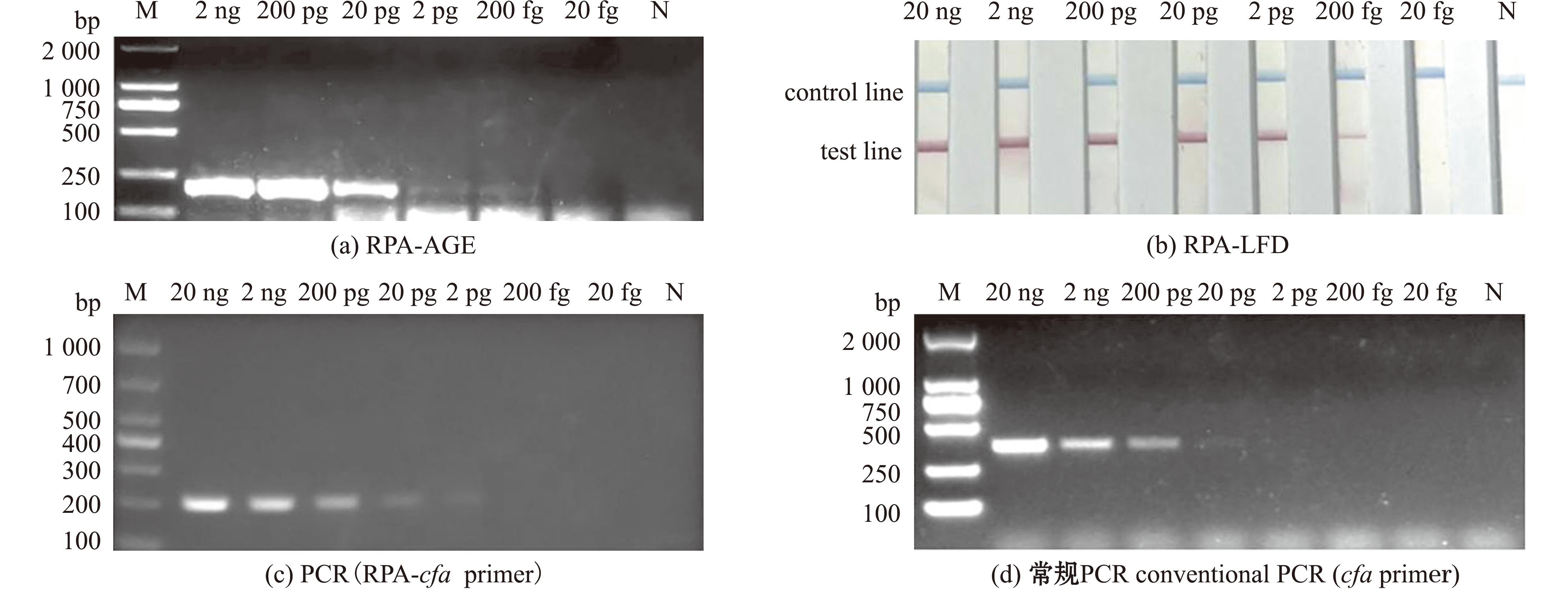
使用RPA-AGE、RPA-LFD及常规PCR方法对稀释后不同浓度的弗氏柠檬酸杆菌基因组总DNA模板进行检测,最低检出浓度分别为200 fg/μL(图 5(a))、200 fg/μL(图5(b))和20 pg/μL(图 5(d)),采用RPA-cfa引物进行PCR扩增的最低检出浓度为2 pg/μL(图5(c))。RPA-AGE法和RPA-LFD法的检测灵敏度一致,均为200 fg/μL,但与RPA-AGE法相比,RPA-LFD法所用时间更短,仅20 min就可观察到结果。这表明,RPA-LFD方法的检测灵敏度最高也最快速。
2.6 纯菌液浓度检测灵敏度
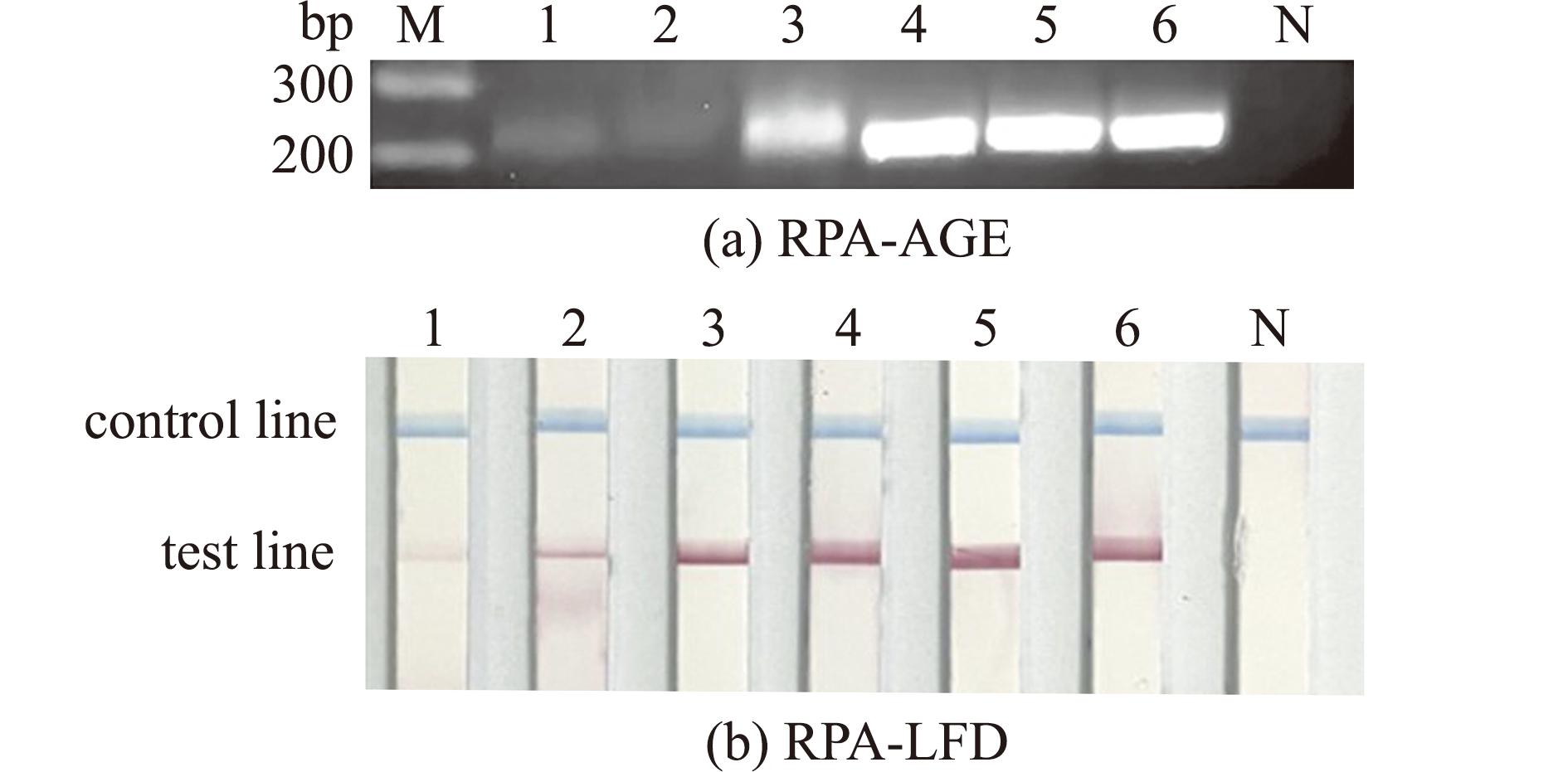
试纸条T线上的条带亮度随着弗氏柠檬酸杆菌纯菌液浓度的递减而减弱,当菌液的浓度低至1.5×102 CFU/mL时,纸条T线上仍然能看到微弱的条带(图6(b))。然而,常规PCR法(cfa引物)只能检测到1.5×104 CFU/mL的弗氏柠檬酸杆菌(图6(a))。这表明,RPA-LFD法检测弗氏柠檬酸杆菌纯菌液是常规PCR法检测灵敏度的100倍。
2.7 攻毒试验样本的检测
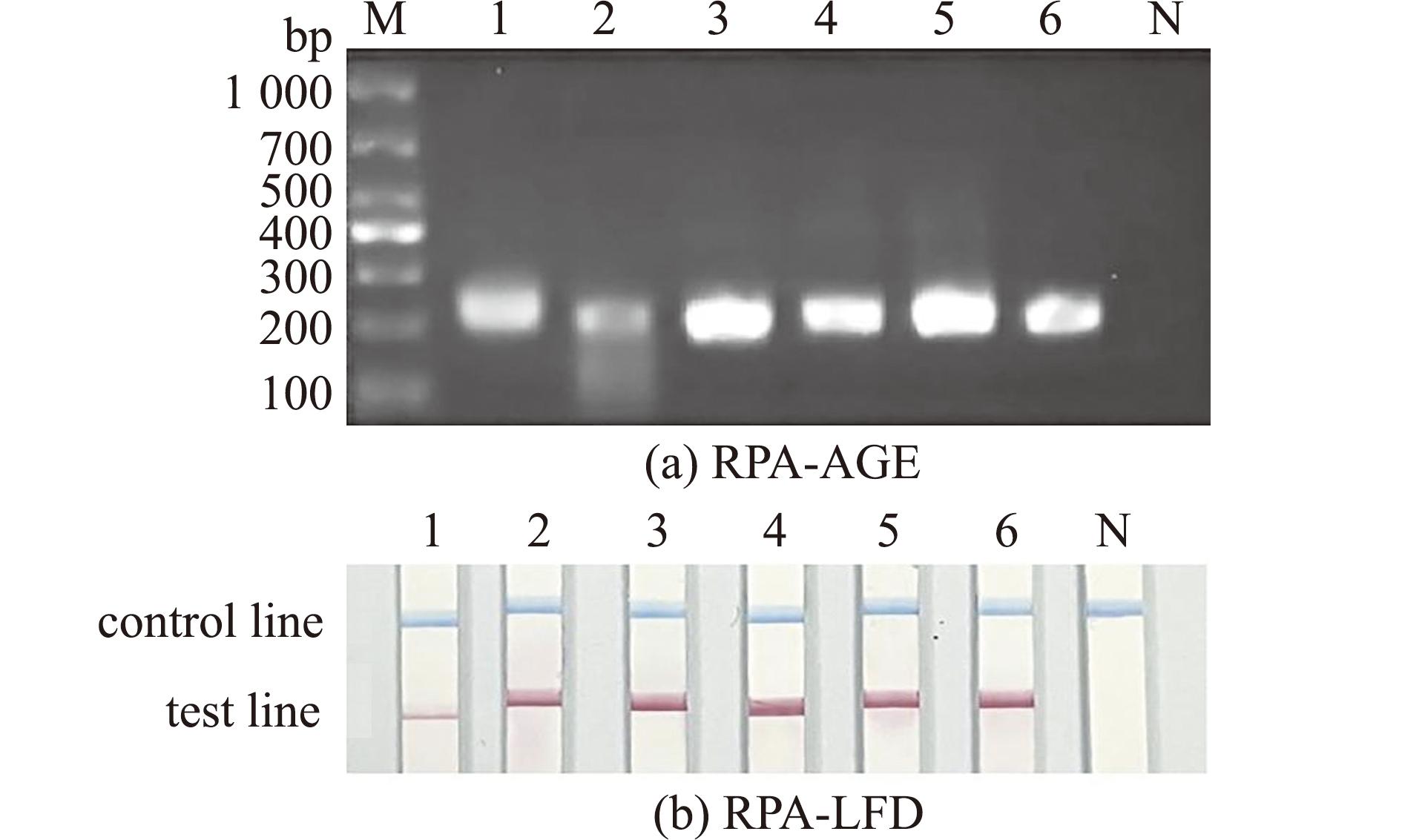
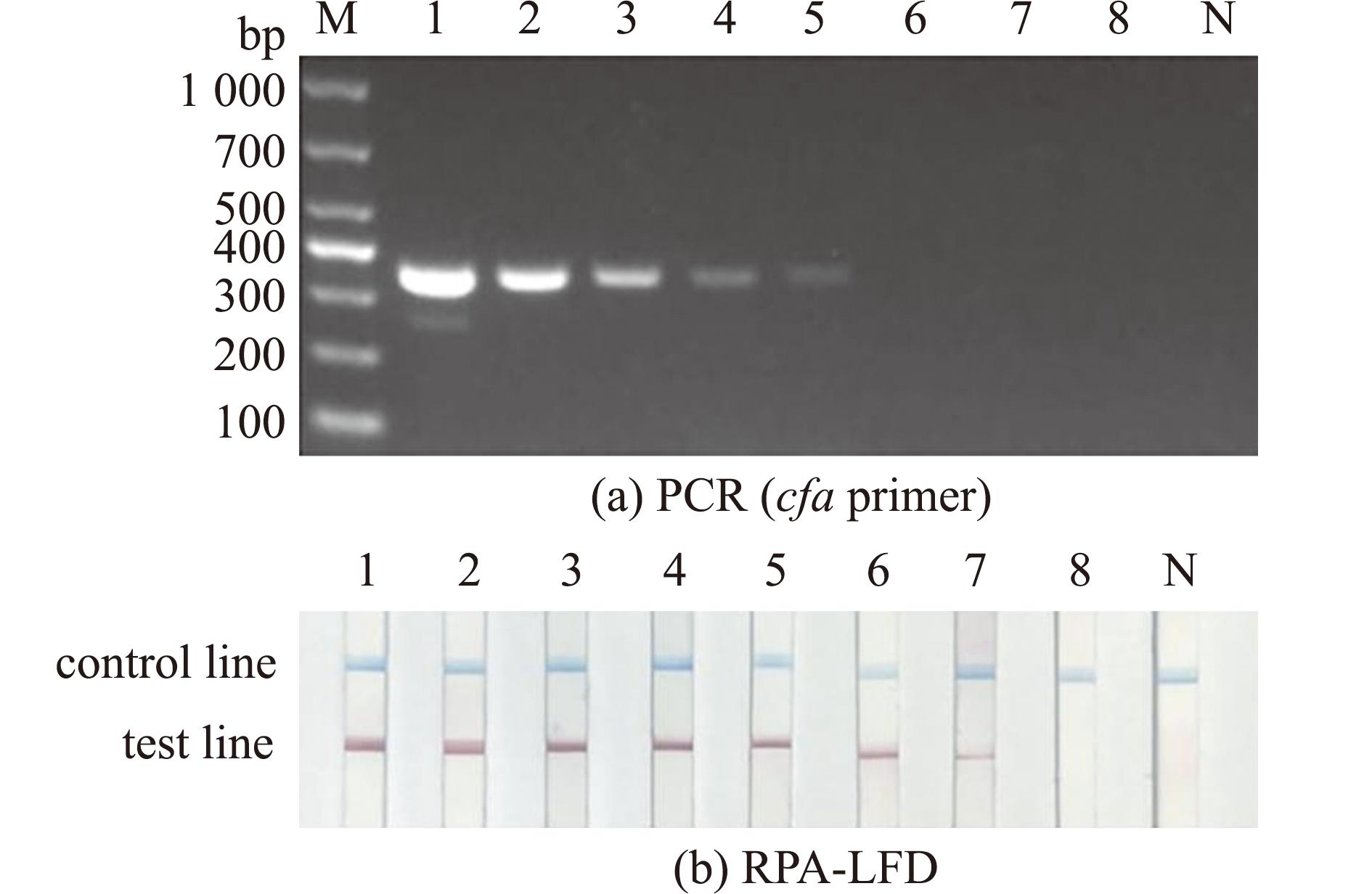
观察腹腔注射弗氏柠檬酸杆菌菌液的杂交鲟状态发现,随着感染时间的延长,杂交鲟游动逐渐缓慢,进食能力也变差。攻毒2 d后发现,有两尾杂交鲟死亡,7 d后再次有两尾杂交鲟死亡,剩余的杂交鲟直至14 d时均未死亡,对照组未观察到鲟死亡或显著变化。死亡杂交鲟口腔红肿,常伴有出血现象,肛门红肿,解剖后发现其肝脏、脾脏和胰脏出血严重。人工感染的杂交鲟与自然感染的中华鲟具有相同的临床疾病症状。使用微量样品基因组DNA提取试剂盒提取33份杂交鲟攻毒组织样本,并将其作为模板进行RPA-LFD检测,同时采用常规PCR引物进行PCR平行试验。采用RPA-LFD方法在攻毒试验样本中检测出6个含有弗氏柠檬酸杆菌的阳性样本,分别集中在肝脏、肾脏、脾脏和胰脏处,且与PCR检测结果一致(图7)。这说明,本研究中建立的RPA-LFD检测方法可有效检测出发病杂交鲟体内的弗氏柠檬酸杆菌。
3 讨论
3.1 弗氏柠檬酸杆菌对鲟的危害
中华鲟是国家一级保护动物,具有极高的营养价值、学术价值和经济价值。然而,随着中华鲟养殖规模的不断扩大,中华鲟病害频发。目前,细菌性疾病的流行已经对中华鲟养殖业造成了巨大的经济损失。弗氏柠檬酸杆菌是近年来被广泛研究的一类病原菌,该菌主要分布在水生生态系统中,当宿主受到高水温、缺氧和有机污染等刺激时,该病原体就会导致其患病[19]。中华鲟是一种冷水鱼类,其最适生长温度为20~22 ℃[20]。夏季持续处于28 ℃左右的高水温是中华鲟的主要应激源,这可能增加了中华鲟对弗氏柠檬酸杆菌的易感性。已有研究指出,弗氏柠檬酸杆菌疾病表现出感染性、传播性和较高的致死率,当水温超过22 ℃时,该菌可能感染施氏鲟(Acipenser schrencki)、西伯利亚鲟(A.baerii)和俄罗斯鲟(A.gueldenstaedti)等多种鲟,鲟感染后的主要症状包括游动活力下降、食欲减退、口腔红肿,并伴有肛门红肿等。中华鲟感染弗氏柠檬酸杆菌后,症状表现为典型的出血性败血症和内脏器官出血[21],这与本研究中杂交鲟感染弗氏柠檬酸杆菌后的症状一致。此外,该疾病的早期临床症状并不明显,导致在疾病早期难以察觉患病鲟,往往在进入疾病中后期时才能被发现,从而造成一定的经济损失。值得注意的是,由于细菌性致病菌种类繁多,充血红肿是多种疾病的共同特征,仅依靠表观症状难以准确对病因进行判断。针对上述情况,建立一种快速、特异和可靠的检测技术对防治弗氏柠檬酸杆菌病是十分必要的,这将为未来中华鲟弗氏柠檬酸杆菌细菌性疾病的早期诊断提供了更有效的技术支持。
3.2 现有弗氏柠檬酸杆菌检测技术比较
近年来,针对弗氏柠檬酸杆菌所开发的检测方法越来越成熟,PCR、qPCR、多重PCR和免疫层析试纸条等多种快检技术已应用于弗氏柠檬酸杆菌的检测中。周昱[22]根据沙门氏菌fimY基因、迟缓爱德华氏菌fimA基因、弗氏柠檬酸杆菌idh基因及奇异变形杆菌idsC基因建立了食品中高灵敏度的多重PCR方法,该方法最少可从7 CFU/mL的弗氏柠檬酸杆菌菌液中检测到目标基因。王芳等[23]基于弗氏柠檬酸杆菌ompA基因成功建立了冷水鱼养殖中弗氏柠檬酸杆菌病的SYBR Green Ⅰ荧光定量 PCR诊断方法,该方法特异性好、灵敏度高,最低检测限可达10 copies/μL。Li等[24]利用PCR 技术建立了快速检测牛肉食品中弗氏柠檬酸杆菌的方法。Kuo等[25]开发了一种可应用于模拟水和生活污水样品中大肠菌群的荧光原位杂交(FISH)方法,该技术利用16S rRNA特异性探针能同时检测水样中的4种大肠菌群,包括大肠杆菌、肺炎克雷伯菌(Klebsiella pneumoniae)、产气肠杆菌(Enterobacter aerogenes)和弗氏柠檬酸杆菌。孙智同等[26]利用胶体金免疫层析技术成功研制出一种快速检测中华鳖弗氏柠檬酸杆菌的试纸条,该试纸条稳定性较好、特异性强且灵敏度高,最低能检测出105 CFU/mL的弗氏柠檬酸杆菌菌液。检测靶点的选择对特定诊断也至关重要,靶向毒力基因是致病菌分子检测的常见做法,除上述基因外,定居因子cfa基因是产肠毒素大肠杆菌的重要致病因子,该基因已被用作弗氏柠檬酸杆菌检测的目标基因。早在2005年,基于弗氏柠檬酸杆菌cfa基因编码环丙烷脂肪酸合成酶的特定区域成功建立了一种新的检测和定量弗氏柠檬酸杆菌的方法,该方法最低检出限为103 CFU/mL弗氏柠檬酸杆菌,且与其他柠檬酸杆菌属及肠道细菌无交叉反应[27]。虽然上述基于聚合酶链反应的扩增被认为是分子诊断的金标准,但其也存在固有的缺点,如耗时、仪器昂贵和扩增程序复杂等,严重限制了该方法在养殖一线的适用性。然而,RPA技术无需PCR仪等特殊仪器,只需通过金属浴进行恒温扩增,具有检测时间短、灵敏度高及操作简单等优点,使用LFD法可以在几分钟内用肉眼观察到RPA扩增结果。因此,相比其他技术,RPA-LFD技术非常适合现场检测。
3.3 弗氏柠檬酸杆菌RPA-LFD 检测效果
目前,已报道的弗氏柠檬酸杆菌的快速检测方法有 PCR、qPCR和多重PCR法,尚未见 RPA技术用于检测弗氏柠檬酸杆菌的报道。与LAMP方法相比,RPA受抑制剂的影响较小且引物设计相对简单[28]。探针在RPA中起着至关重要的作用,通过设计RPA引物/探针组合,可以在更大程度上避免假阳性[29]。本研究中,针对弗氏柠檬酸杆菌的cfa基因保守序列设计了引物和探针,从而提高了RPA-LFD 试验成功的可行性。然而,室外现场使用等温扩增技术的实用性往往受到限制,因此,优化反应条件是获得准确可靠结果的关键。本研究中,确定了38 ℃为反应的最佳温度,RPA阳性扩增可在20 min内目测到结果,而传统的PCR分析通常需要2~3 h才能完成,整个检测时间明显缩短。更值得注意的是,与 PCR、胶体金免疫层析技术和荧光定量PCR 等检测方法相比,本研究中建立的 RPA-LFD检测方法的灵敏度更高,每次反应可检测到低至1.5×102 CFU/mL的弗氏柠檬酸杆菌或200 fg/μL的基因组DNA。本研究中选取6种菌株进行特异性试验,除了弗氏柠檬酸杆菌之外,嗜水气单胞菌、维氏气单胞菌均为能引发鲟患细菌性疾病的致病菌。虽然还未发现鲟感染副溶血弧菌和金黄色葡萄球菌的研究报道,但二者均为人畜共患菌并具有较强的感染性,是未来引发鲟患细菌性疾病的主要威胁。本研究中建立的RPA-LFD技术与上述常见水产病原菌进行交叉反应,结果显示无特异性条带产生,表明建立的RPA-LFD方法具有良好的特异性,将该方法用于杂交鲟攻毒样本检测,成功检测到杂交鲟肝脏、脾脏、肾脏和胰脏中存在弗氏柠檬酸杆菌的6个阳性样本,检测准确率达100%,与常规PCR方法的检测结果一致,但RPA-LFD法具有检测灵敏度和准确性高的特点。因此,本研究中所建立的弗氏柠檬酸杆菌RPA-LFD检测方法是一种可替代PCR的现场检测技术,具有广阔的临床应用前景,为中华鲟弗氏柠檬酸杆菌细菌性疾病的快速诊断、实时监控和有效预防治疗提供了技术支持。
4 结论
1) 根据弗氏柠檬酸杆菌cfa基因保守序列设计出了 RPA 引物/探针组合,引物特异性良好,与其他6株水产养殖过程中常见病原菌无交叉反应。
2) 本研究中所建立的弗氏柠檬酸杆菌RPA-LFD检测方法在38 ℃下20 min内就可观察到结果。与其他检测方法相比,该方法具有快速简便且准确的特点。
3) 与常规PCR、RPA-AGE两种方法相比,本研究中建立的弗氏柠檬酸杆菌RPA-LFD检测方法灵敏度最好,最低检出限为200 fg/μL。
4)对杂交鲟攻毒试验样本检测发现,RPA-LFD方法的检测结果与常规PCR检测结果一致,均检测出6个含有弗氏柠檬酸杆菌的杂交鲟组织阳性样本。这说明,建立的弗氏柠檬酸杆菌RPA-LFD检测方法具有较好的可靠性,可以适用于鲟养殖现场检测。
参考文献:
[1] 薛巧,赵战勤,刘会胜,等.弗氏柠檬酸杆菌对动物和人致病性研究进展[J].动物医学进展,2015,36(7): 81-85. XUE Q,ZHAO Z Q,LIU H S,et al.Progress on Citrobacter freundii pathogenicity in animals and humans[J].Progress in Veterinary Medicine,2015,36(7):81-85.(in Chinese)
[2] BEHERA B K,PARIA P,DAS A,et al.Molecular identification and pathogenicity study of virulent Citrobacter freundii associated with mortality of farmed Labeo rohita (Hamilton 1822), in India[J].Aquaculture,2022,547:737437.
[3] 张冬星,康元环,田佳鑫,等.团头鲂致病性弗氏柠檬酸杆菌的分离鉴定及药敏试验[J].中国兽医科学,2016,46(12): 1589-1595. ZHANG D X,KANG Y H,TIAN J X,et al.Isolation, identification and drug susceptibility of the pathogenic Citrobacter freundii from Megalobrama amblycephala[J].Chinese Veterinary Science,2016,46(12):1589-1595.(in Chinese)
[4] 张明洋,胡安东,程振涛,等.虹鳟弗氏柠檬酸杆菌的分离鉴定与多位点序列分型[J].基因组学与应用生物学,2020,39(6):2521-2528. ZHANG M Y,HU A D,CHENG Z T,et al.Identification of the Citrobacter freundii in Oncorhynchus mykiss by multilocus sequence typing[J].Genomics and Applied Biology,2020,39(6):2521-2528.(in Chinese)
[5] 杨方园,关瑞章,李忠琴,等.花鳗鲡病原菌弗氏柠檬酸杆菌的鉴定[J].集美大学学报(自然科学版),2013, 18(2): 81-87. YANG F Y,GUAN R Z,LI Z Q,et al.Identification of pathogenic Citrobacter freundii isolated from Anguilla marmorata[J].Journal of Jimei University (Natural Science Edition),2013,18(2):81-87.(in Chinese)
[6] 王利,魏勇.鲫鱼弗氏柠檬酸杆菌的鉴定及系统发育分析[J].水产科学,2012,31(8):481-484. WANG L,WEI Y.Identification and phylogenetic analysis of bacterium Citrobacter freundii from crucian carp Carassius auratus by 16S rDNA genes[J].Fisheries Science,2012,31(8):481-484.(in Chinese)
[7] WANG J H,WEI Q W,ZOU Y C.Conservation strategies for the Chinese sturgeon,Acipenser sinensis:an overview on 30 years of practices and future needs[J].Journal of Applied Ichthyology,2011,27(2):176-180.
[8] 张陵,郭文献,李泉龙.长江流域珍稀特有物种中华鲟生态保护措施[J].华北水利水电大学学报(自然科学版),2022,43(1):96-102. ZHANG L,GUO W X,LI Q L.Ecological protection measures of Chinese sturgeon, a rare and endemic species in the Yangtze River Basin[J].Journal of North China University of Water Resources and Electric Power (Natural Science Edition),2022,43(1):96-102.(in Chinese)
[9] 张书环,聂品,舒少武,等.子二代中华鲟分枝杆菌感染及血液生理生化指标的变化[J].中国水产科学,2017,24(1):136-145. ZHANG S H,NIE P,SHU S W,et al.Mycobacteriosis in cultured F2 generation Chinese sturgeon (Acipenser sinensis) and relevant blood physiological and biochemical indices[J].Journal of Fishery Sciences of China,2017,24(1):136-145.(in Chinese)
[10] 田甜,张建明,杜合军.中华鲟源维氏气单胞菌的分离鉴定及其药敏特性[J].安徽农业大学学报,2017, 44(6):1010-1016. TIAN T,ZHANG J M,DU H J.Isolation and identification of Aeromonas veronii from Chinese sturgeon and its antibiotic sensitivity[J].Journal of Anhui Agricultural University,2017,44(6):1010-1016.(in Chinese)
[11] MENG Y,XIAO H B,ZENG L B.Isolation and identification of the hemorrhagic septicemia pathogen from Amur sturgeon, Acipenser schrenckii[J].Journal of Applied Ichthyology,2011,27(2):799-803.
[12] 邸军,张书环,黄君,等.中华鲟脑膜败血伊丽莎白菌的分离鉴定及药敏特性[J].水产学报,2018,42(1):120-130. DI J,ZHANG S H,HUANG J,et al.Isolation, identification and antibiotic sensitivity of Elizabethkingia meningoseptica from Chinese sturgeon (Acipenser sinensis)[J].Journal of Fisheries of China,2018,42(1):120-130.(in Chinese)
[13] YANG J,TIAN T,XIAO K,et al.Pathogenic infection and immune-related gene expression of Chinese sturgeon (Acipenser sinensis) challenged by Citrobacter freundii[J].Developmental and Comparative Immunology,2021,114: 103872.
[14] WANG F,GE D B,WANG L,et al.Rapid and sensitive recombinase polymerase amplification combined with lateral flow strips for detecting Candida albicans[J].Analytical Biochemistry,2021,633:114428.
[15] 张珊珊,李楠,郝镯,等.布鲁氏菌RPA-LFD快速检测方法的建立与应用[J].中国兽医科学,2021,51(10): 1215-1220. ZHANG S S,LI N,HAO Z,et al.Development and evaluation of a rapid visualization detection method for Brucella based on RPA-LFD[J].Chinese Veterinary Science,2021,51(10):1215-1220.(in Chinese)
[16] 余庭,蒋晗,杨宇婷,等.可视化RPA-LFD技术快速检测美人鱼发光杆菌[J].中国计量大学学报,2020,31(4):496-501. YU T,JIANG H,YANG Y T,et al.Rapid detection of Photobacterium damselae with visual RPA-LFD technology[J].Journal of China University of Metrology,2020,31(4):496-501.(in Chinese)
[17] 吴华华,王锦鑫,谷庆花,等.金黄色葡萄球菌重组酶聚合酶扩增方法的建立[J].中国预防兽医学报,2020, 42(8): 797-801. WU H H,WANG J X,GU Q H,et al.Establishment of a recombinase polymerase amplification method for detection of Staphylococcus aureus[J].Chinese Journal of Preventive Veterinary Medicine,2020,42(8):797-801.(in Chinese)
[18] 肖宁,孔令严,周昊,等.克氏原螯虾病原弗氏柠檬酸杆菌的分离鉴定及其药敏与黏附特性[J].水产学报,2016,40(6):946-955. XIAO N,KONG L Y,ZHOU H,et al.Isolation and identification of Citrobacter freundii from diseased Procambarus clarkii and its antibiotics susceptibility and adhesion characteristics[J].Journal of Fisheries of China, 2016, 40(6): 946-955.(in Chinese)
[19] BANDEIRA G Jr,DOS SANTOS A C,DE FREITAS SOUZA C,et al.Citrobacter freundii infection in silver catfish (Rhamdia quelen):hematological and histological alterations[J].Microbial Pathogenesis,2018,125:276-280.
[20] XU J,ZENG X H,JIANG N,et al.Pseudomonas alcaligenes infection and mortality in cultured Chinese sturgeon, Acipenser sinensis[J].Aquaculture,2015,446:37-41.
[21] DENG D,MU Z Z,LV X Y,et al.Pathogenicity of Plesiomonas shigelloides and Citrobacter freundii isolated from the endangered Chinese sturgeon (Acipenser sinensis)[J].Microbial Pathogenesis,2022,173:105818.
[22] 周昱.食品中四种致病菌的多重PCR检测方法的建立和应用[D].福州:福建农林大学,2013. ZHOU Y.Estabilishment and applicaion of multiplex PCR for detecion of four foodborne bacterial pathogens[D].Fuzhou: Fujian Agriculture and Forestry University,2013.(in Chinese)
[23] 王芳,康超,李永霞,等.弗氏柠檬酸杆菌SYBR Green Ⅰ荧光定量PCR诊断方法的建立及耐药性分析[J].江苏农业科学,2016,44(11):286-289. WANG F,KANG C,LI Y X,et al.Development of a SYBR Green Ⅰ real-time quantitative PCR for detection of Citrobacter freundii and its antibiotic sensitivity[J].Jiangsu Agricultural Sciences,2016,44(11):286-289.(in Chinese)
[24] LI Y,MUSTAPHA A.Development of a polymerase chain reaction assay to detect enteric bacteria in ground beef[J].Food Microbiology,2004,21(3):369-375.
[25] KUO J T,CHANG L L,YEN C Y,et al.Development of fluorescence in situ hybridization as a rapid, accurate method for detecting coliforms in water samples[J].Biosensors,2020,11(1):8.
[26] 孙智同,章礼平,李登峰.一种快速检测弗氏柠檬酸杆菌免疫层析试纸条的研制[J].宁波大学学报(理工版),2019,32(3):15-20. SUN Z T,ZHANG L P,LI D F.Development of a colloidal gold immunochromatographic test strip for detection of Citrobacter freundii[J].Journal of Ningbo University(Natural Science &Engineering Edition),2019,32(3):15-20.(in Chinese)
[27] KACL KOV
KOV E,KRASCSENICSOV
E,KRASCSENICSOV K,PANGALLO D,et al.Detection and quantification of Citrobacter freundiiand and C.braakii by 5′-nuclease polymerase chain reaction[J].Current Microbiology,2005,51(4): 229-232.
K,PANGALLO D,et al.Detection and quantification of Citrobacter freundiiand and C.braakii by 5′-nuclease polymerase chain reaction[J].Current Microbiology,2005,51(4): 229-232.
[28] AHMED F A,LARREA-SARMIENTO A,ALVAREZ A M,et al.Genome-informed diagnostics for specific and rapid detection of Pectobacterium species using recombinase polymerase amplification coupled with a lateral flow device[J].Scientific Reports,2018,8(1):15972.
[29] LIU X Q,YAN Q Y,HUANG J,et al.Influence of design probe and sequence mismatches on the efficiency of fluorescent RPA[J].World Journal of Microbiology and Biotechnology,2019,35(6):95.
Establishment and application of a method for rapid detection ofCitrobacter freundii by recombinase polymerase amplification (RPA) combined with lateral flow strips (LFD)
WANG Shuran1,2,3,FAN Houyong4,XU Jianan4,ZHOU Tianqi1,2,3,XU Dan1,2,3,ZHENG Yueping4*
(1.College of Fisheries and Life Science,Shanghai Ocean University,Shanghai 201306,China;2.National Pathogen Collection Center for Aquatic Animals,Shanghai Ocean University,Shanghai 201306,China;3.Key Laboratory of Aquatic Genetic Resources,Ministry of Agriculture and Rural Affairs,Shanghai Ocean University,Shanghai 201306,China;4.Shanghai Aquatic Wildlife Conservation and Research Center,Shanghai 200092,China)
Abstract: In order to establish a rapid detection method for Citrobacter freundii based on recombinase polymerase amplification (RPA) technology, the specific RPA primers were designed based on the conserved sequence of the cfa gene of C.freundii, the reaction conditions were optimized, and the specificity and sensitivity were evaluated by recombinase polymerase amplification (RPA) combined with agarose gel electrophoresis (AGE) and lateral flow chromatography (LFD). The results showed that C. freundii was detected within 20 min of amplification at 38 ℃ by the established RPA-LFD rapid detection method, with the minimum limit of detection (LOD) of the C.freundii pure culture and genomic DNA of 1.5×102 CFU/mL and 200 fg/μL by RPA-LFD method, 100 times higher sensitivity than that by conventional PCR (cfa primer). The consistent results were observed when sturgeon hybrid infected with C.freundii clinical samples was simultaneously detected using both the established RPA-LFD and conventional PCR methods. The research indicated that the RPA-LFD method for C.freundii established in this study is simple, rapid, specific and sensitive, and does not require expensive instrumentation, which can provide more effective technical support for the early diagnosis of bacterial diseases caused by C.freundii in the future.
Key words: Citrobacter freundii; rapid detection; recombinase polymerase amplification technique; lateral flow test strips


 KOV
KOV E,KRASCSENICSOV
E,KRASCSENICSOV