青海湖裸鲤肠道菌群对不同盐度胁迫的响应
赵子丰1,金文杰1,2,赵静1,王蓉1,祁洪芳3,李瑾3,白鹏1,2*,李长忠1,2*
(1.青海大学 生态环境工程学院,高原动物和资源利用研究室,青海 西宁 810016;2.青海大学 生态环境工程学院,基因工程实验室,青海 西宁 810016;3.青海湖裸鲤救护中心 青海湖裸鲤繁育与保护重点实验室,青海 西宁 810016)
摘要:为了探讨盐度胁迫对青海湖裸鲤(Gymnocypris przewalskii)肠道菌群结构的影响,基于三代全长16S扩增子测序技术,对不同盐度(0、5、10、15)下青海湖裸鲤肠道内容物进行微生物测序及信息分析。结果表明:在门水平上,不同盐度组青海湖裸鲤肠道菌群的相对丰度不同,对照组(盐度0)和低盐度组(盐度5、10)肠道菌群中,相对丰度较高的是梭杆菌门(Fusobacteria)和变形菌门(Proteobacteria),其次是厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)和浮霉菌门(Planctomycetes)等也占有一定比例,高盐度组(盐度15)中以变形菌门、拟杆菌门、浮霉菌门和疣微菌门为主要菌群;在属水平上,对照组和低盐度组肠道菌群中鲸杆菌属(Cetobacterium)和气单胞菌属(Aeromonas)相对丰度较高,其次是弧菌属(Vibrio)和希瓦氏菌属(Shewanella),高盐度组中以弧菌属、假红杆菌属(Pseudorhodobacter)和卤蕨属(Haloferula)为主要菌群。研究表明,低盐度(盐度≤10)胁迫对青海湖裸鲤肠道菌群结构的影响不大,而高盐度(盐度≥15)胁迫则会抑制青海湖裸鲤肠道菌群和优势菌群的生长且影响较大。
关键词:青海湖裸鲤;16S扩增子;盐度;肠道菌群
DOI:10.16535/j.cnki.dlhyxb.2023-213
文章编号:2095-1388(2024)02-0225-09
中图分类号:S 917.4
文献标志码:A
收稿日期:2023-09-28
基金项目:青海省重大科技专项(2019-NK-A2);青海省科协中青年科技人才托举工程(2022QHSKXRCTJ34)
作者简介:赵子丰(1997—),男,硕士研究生。E-mail:zzf125808@163.com
通信作者:白鹏(1992—),男,博士,讲师。E-mail:baipeng_6@163.com
李长忠(1966—),男,博士,教授。E-mail:lichangzhong@qhu.edu.cn(并列通信作者)
青海湖裸鲤(Gymnocypris przewalskii)隶属于鲤形目(Cypriniformes )鲤科(Cyprinidae)裂腹鱼亚科(Schizothoracinae)裸鲤属(Gymnocypris)[1],是青海湖中唯一的野生经济性鱼类,其在青海湖“鱼鸟共生”生态系统中起着主要作用,对青海湖水生生态系统的平衡有着重要作用[2]。青海湖地处青藏高原,地理环境独特,是中国最大的内陆咸水湖泊,其水体盐度在15左右[3],温度与溶氧量较低,在此环境下青海湖裸鲤生长发育较为迟缓[2]。其中,盐度是野生型青海湖裸鲤相较于人工淡水养殖条件下生长速度缓慢的重要原因之一[2]。由于青海湖中的盐碱度过高不利于其繁衍后代,每年的5月底到8月期间,性腺发育成熟的青海湖裸鲤从青海湖洄游到布哈河、沙柳河、泉吉河和黑马河等主要的淡水支流,选择水流较缓、河底有沙石的地方进行产卵繁殖,繁殖后的亲鱼及孵化后的幼鱼再从河流中返回到青海湖[4],在洄游过程中其必须面临适应水体盐度等环境因子变化[5]。
水体盐度是影响鱼类生长、代谢和繁育的重要环境因子,盐度变化直接影响鱼类的存活、生长、呼吸代谢、肌肉品质及生殖发育[6]。此外,水体中的盐度还会影响鱼类肠道微生物的多样性及结构[7]。肠道微生物在保持宿主肠道健康、促进肠道正常发育、改善体内能量吸收与脂肪代谢及抵御病原侵入等过程中发挥极其重要的作用[8]。
本研究中,通过三代全长16S扩增子测序技术分析了青海湖裸鲤在不同盐度下肠道菌群的组成及群落结构变化,以期为后续通过筛选青海湖裸鲤在最适盐度下的优势菌群来制作微生物饲料,以及促进青海湖裸鲤的人工生态养殖提供参考。
1 材料与方法
1.1 材料
试验用鱼为青海湖裸鲤救护中心在全淡水中养殖的1龄青海湖裸鲤,共400尾,初始体质量为(16.486±3.530)g,初始体长为(8.65±1.10)cm,初始全长为(12.90±1.50)cm。试验开始前于缸(120 L)内暂养14 d。
1.2 方法
1.2.1 试验设计 试验共设4组,盐度分别为0(对照组)、5、10、15,分别记为QH-0(A)、QH-5(B)、QH-10(C)、QH-15(D)组。盐度用海水晶(购自江西盐通科技有限公司)配制,将暂养14 d后的青海湖裸鲤放入已经调好盐度且充分曝气的水中。试验期间,定时定量投喂水产专用饲料(山东汉业生物科技有限公司),每日投喂两次,每次投喂体质量的3%,日换水两次,以保持养殖水体纯净,试验结束前一天停止投喂。
1.2.2 样品采集 试验共进行7周,取样时从每组随机取9尾鱼,所有样品鱼活力较好,体表完整无擦伤,剖检内脏无病灶。用丁香酚将鱼麻醉后,在无菌条件下取出整条肠道,再用体积分数为75%的乙醇棉球擦拭肠道外壁,用灭菌手术剪剪开肠道,用解剖刀刮取各肠道内容物,将每3尾鱼的肠道内容物混合为1个样品,每组设置3个平行,共12个样品,并将样品置于-80 ℃超低温冰箱中保存待测。取样结束后将样品送至上海凌恩生物科技有限公司进行扩增子高通量测序分析。
1.2.3 总DNA提取和检测
1)DNA提取及质量检测。采用E.Z.N.A.® Soil DNA Kit提取DNA;使用电泳检测DNA浓度(20 g/L琼脂糖凝胶,1×TAE缓冲液,100 V,电泳20 min),以1 μL Loading Buffer、1 μL DNA和4 μL无菌水为混样,检测DNA的浓度。
2)PCR扩增及Illumina Miseq测序。用原组学样本中获得的DNA进行目标菌群的扩增子制备,采用16S rRNA V1~V9区引物27F(上游引物,5′ barcode-AGAGTTTGATCMTGGCTCAG 3′)和1492R(下游引物,5′CRGYTACCTTGTTACGACTT 3′)进行PCR扩增。PCR扩增程序:94 ℃下预变性4 min;94 ℃下变性30 s,55 ℃下退火30 s,72 ℃下延伸1 min,共进行27个循环;最后在72 ℃下保持10 min。扩增产物经20 g/L琼脂糖凝胶电泳后进行核酸纯化。纯化产物送至上海凌恩生物科技有限公司进行Illumina Miseq高通量测序及分析。
1.2.4 物种注释、多样性分析和菌群功能预测 使用UPARSE[9](http://drive5.com/uparse/,version 7.1)将OTU按照98.65%的一致性阈值聚类,并使用UCHIME鉴定并去除嵌合序列,利用RDP classifier[10](http://rdp.cme.msu.edu/,version 2.2)对每条序列进行物种分类注释。为了得到每个OTU对应的物种分类信息,采用Uclust算法[11]对OTU代表序列进行分类学分析,并分别在各个分类水平上统计各样本的群落组成。使用R语言计算Alpha多样性指数,包括Chao 1、ACE、Shannon和Pd-faith等。通过分析不同样本OTU(98.65%相似性)组成反映样本间的差异和距离,如样本组成越相似,反映在PCA图中的距离越近。使用LefSe软件进行微生物差异分析,LDA Score的筛选值设置为4.0。利用PICRUSt 2软件进行样本微生物群落的功能预测。
2 结果与分析
2.1 不同盐度胁迫下青海湖裸鲤的生长情况
从表1可见,5、10盐度组的终末体长、全长、体质量和存活率略高于0、15盐度组,盐度15组最低。
表1 盐度胁迫7周后青海湖裸鲤体质量、体长的变化
Tab.1 Changes in body weight and body length in Gymnocypris przewalskii exposed to salinity stress for 7 weeks
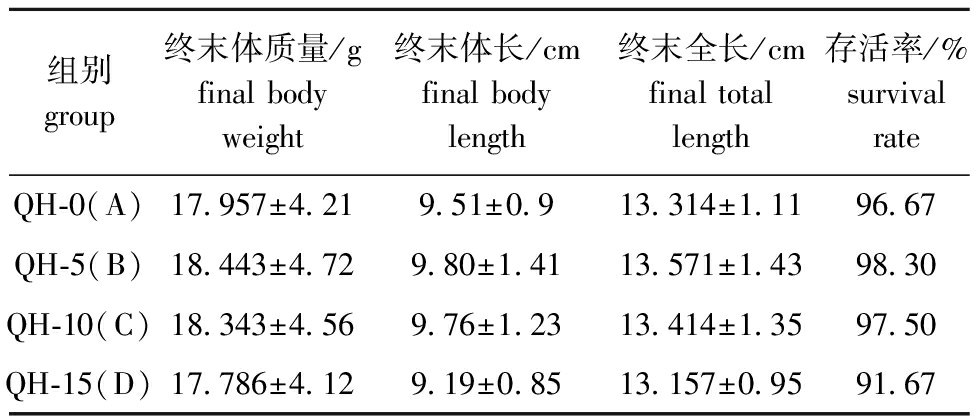
组别group终末体质量/gfinal body weight终末体长/cmfinal body length终末全长/cmfinal total length存活率/%survival rateQH-0(A)17.957±4.219.51±0.913.314±1.1196.67QH-5(B)18.443±4.729.80±1.4113.571±1.4398.30QH-10(C)18.343±4.569.76±1.2313.414±1.3597.50QH-15(D)17.786±4.129.19±0.8513.157±0.9591.67
2.2 样品DNA质量检测及测序质量
利用基因组DNA提取试剂盒,得到了各盐度组青海湖裸鲤肠道菌群总基因组DNA,电泳条带较清晰(图1)。各盐度组PCR扩增结果均较好,可以满足后续测序要求(表2)。
表2 PCR扩增结果
Tab.2 PCR amplification results
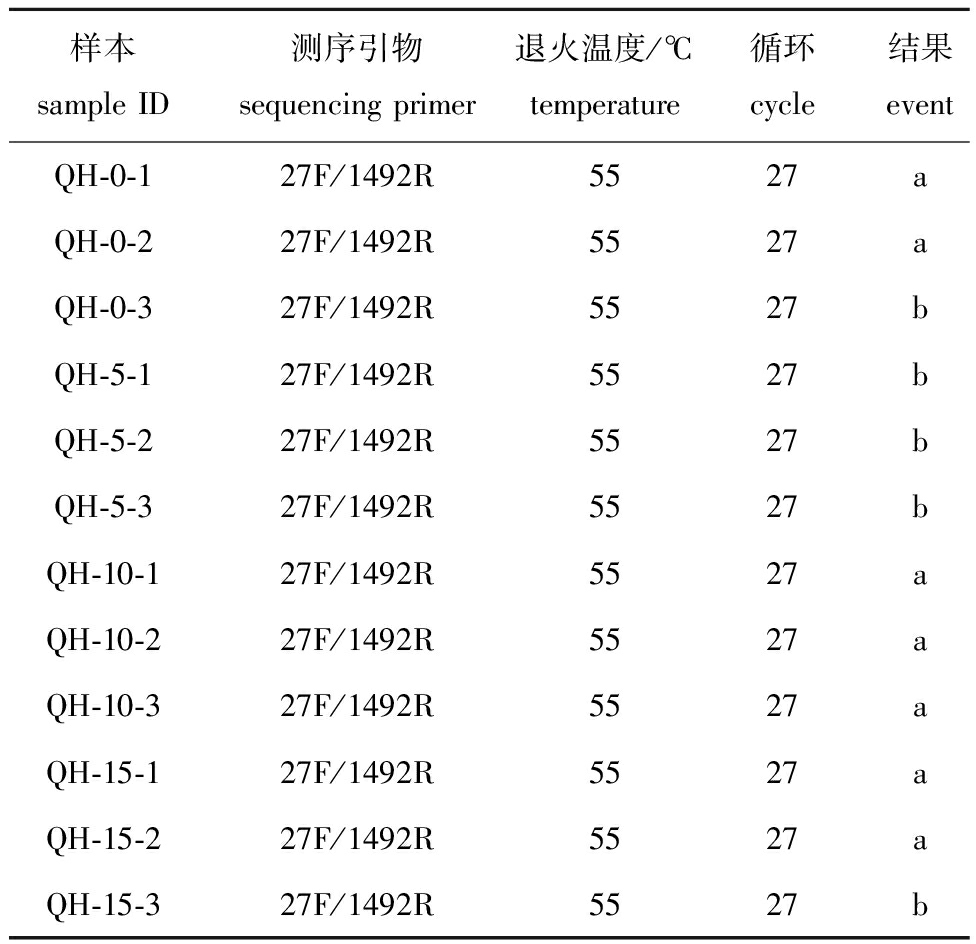
样本sample ID测序引物sequencing primer退火温度/℃temperature循环cycle结果eventQH-0-127F/1492R5527aQH-0-227F/1492R5527aQH-0-327F/1492R5527bQH-5-127F/1492R5527bQH-5-227F/1492R5527bQH-5-327F/1492R5527bQH-10-127F/1492R5527aQH-10-227F/1492R5527aQH-10-327F/1492R5527aQH-15-127F/1492R5527aQH-15-227F/1492R5527aQH-15-327F/1492R5527b
注:a表示PCR产物目的条带大小正确,浓度合适;b表示PCR产物目的条带大小正确,浓度偏低;a或b都可进行后续试验。
Note:a indicates that the target band of the PCR product is correct in size and the concentration is appropriate;b indicates that the target band of the PCR product is correct in size and the concentration is low;Subsequent experiments are therefore allowed for both a and b.
本试验中,从4个盐度组共采集了12个样品,各样品的DNA均提取成功且每个样品的肠道内容物也测序成功。测序所得的序列用Fastp和FLASH软件进行预处理,12个样本共获得399 042条合格的16S rRNA基因序列,每个样本产生的有效序列数目为30 157~38 623,有效序列的平均长度为1 445.79 bp。对所有序列进行98.65%相似水平OTU划分并进行物种注释,可划分为4 318个OTU,分为23门46纲95目197科505属1 274种。
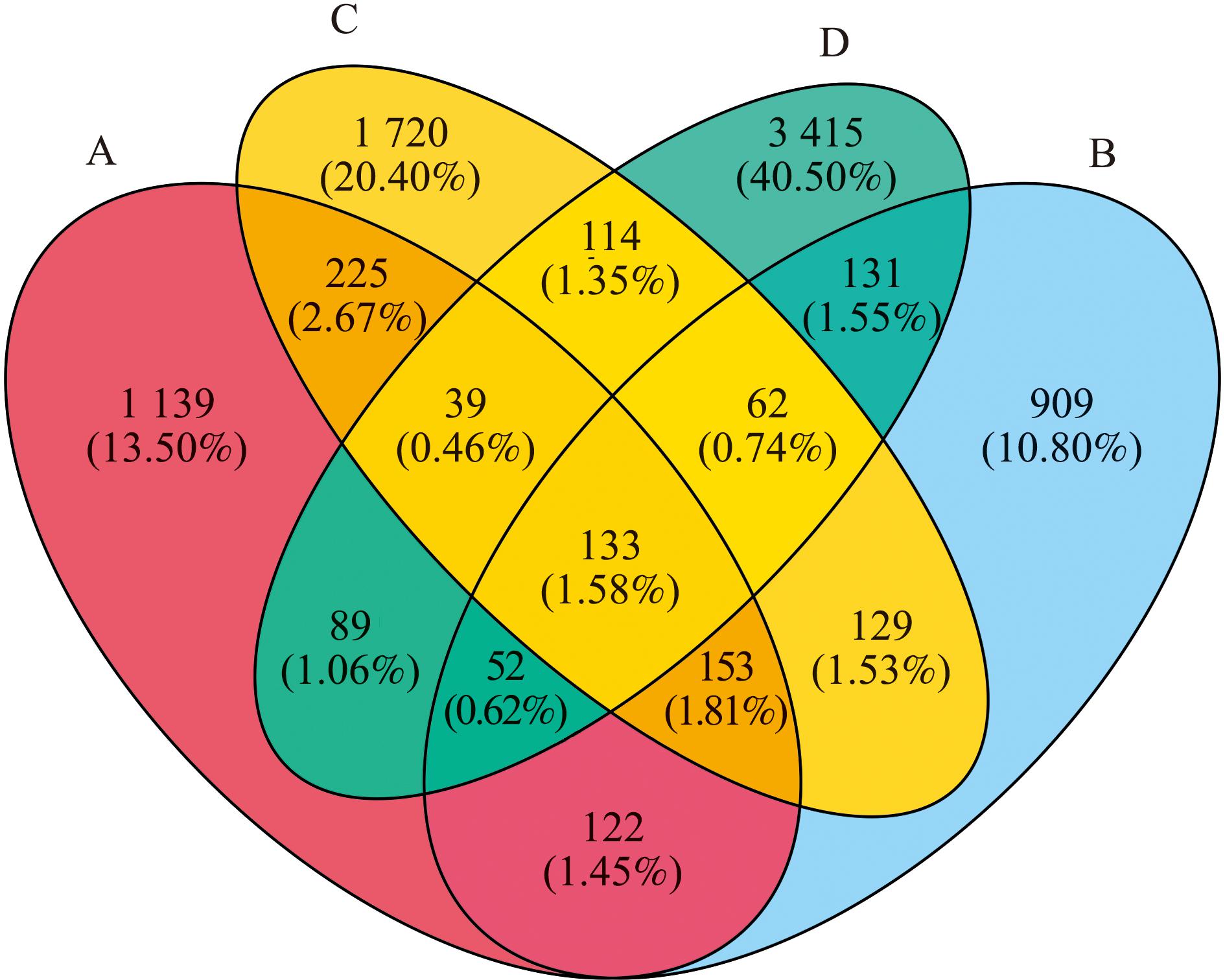
2.3 肠道菌群OTU分布韦恩图
从图2可见:12个样品肠道菌群OTU总数为8 432,4组共有OTU数133个;D组(盐度为15)样本中肠道菌群的OTU数与A(对照组)、B(盐度为5)和C(盐度为10)3组相比显著升高,A、B、C 3组的OTU数相差较小,分别为1 139、909、1 720,除A组外,其他3组随着盐度的上升,OTU数也逐渐升高。
2.4 不同盐度组肠道菌群的Alpha多样性指数
从表3可见:15盐度组肠道菌群的ACE和Chao 1指数显著高于其他3组(P<0.05),说明该组菌群的物种丰度较低盐度组高;15盐度组肠道菌群的Shannon指数显著高于其他3组(P<0.05),说明该组菌群的生物多样性较低盐度组高;全部样品的覆盖度指数为0.94~0.99,反映出各样品序列基本都已被检测出。说明本试验数据真实可靠,能够反映不同盐度下青海湖裸鲤肠道菌群组成的多样性。
表3 不同盐度下青海湖裸鲤肠道菌群的Alpha多样性指数
Tab.3 Alpha diversity index of intestinal flora in Gymnocypris przewalskii under different salinities
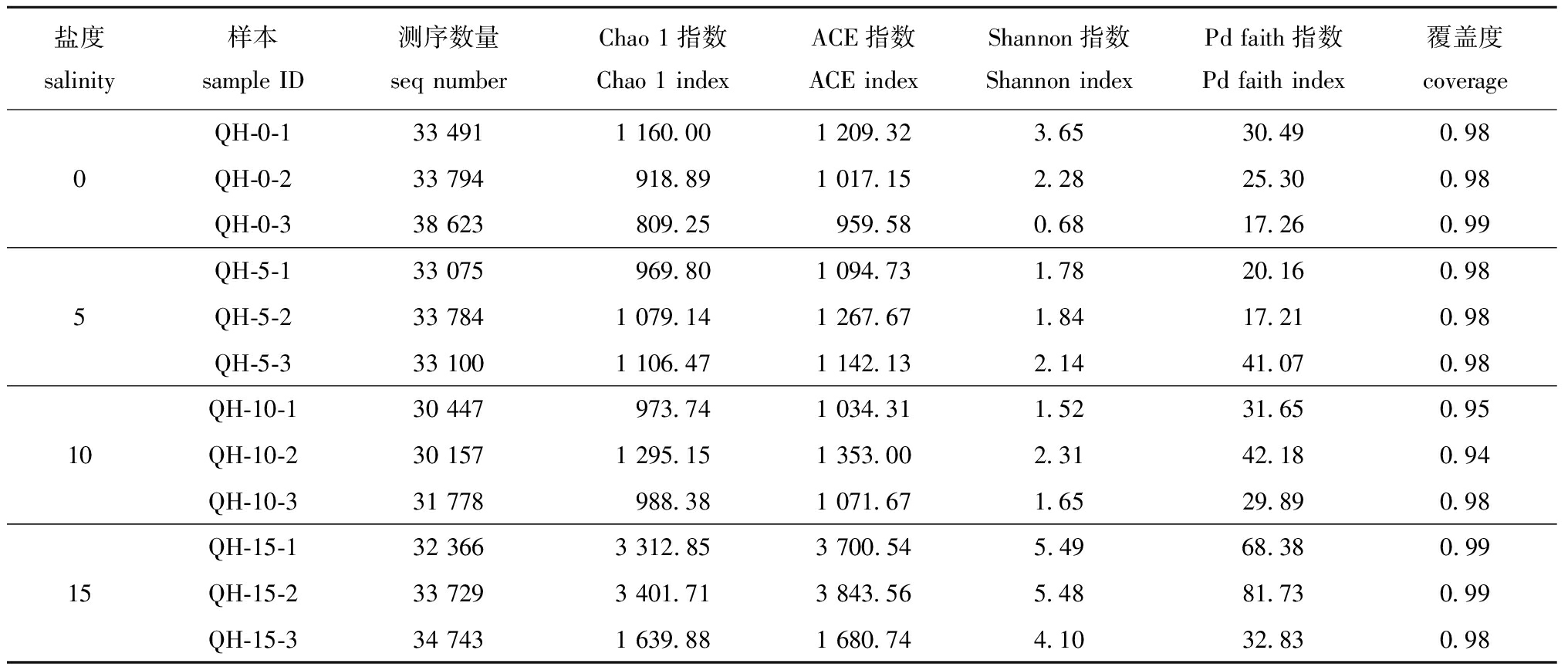
盐度salinity样本sample ID测序数量seq numberChao 1指数Chao 1 indexACE指数ACE indexShannon指数Shannon indexPd faith指数Pd faith index覆盖度coverageQH-0-133 4911 160.001 209.323.6530.490.980QH-0-233 794918.891 017.152.2825.300.98QH-0-338 623809.25959.580.6817.260.99QH-5-133 075969.801 094.731.7820.160.985QH-5-233 7841 079.141 267.671.8417.210.98QH-5-333 1001 106.471 142.132.1441.070.98QH-10-130 447973.741 034.311.5231.650.9510QH-10-230 1571 295.151 353.002.3142.180.94QH-10-331 778988.381 071.671.6529.890.98QH-15-132 3663 312.853 700.545.4968.380.9915QH-15-233 7293 401.713 843.565.4881.730.99QH-15-334 7431 639.881 680.744.1032.830.98
2.5 不同盐度组肠道菌群组成差异
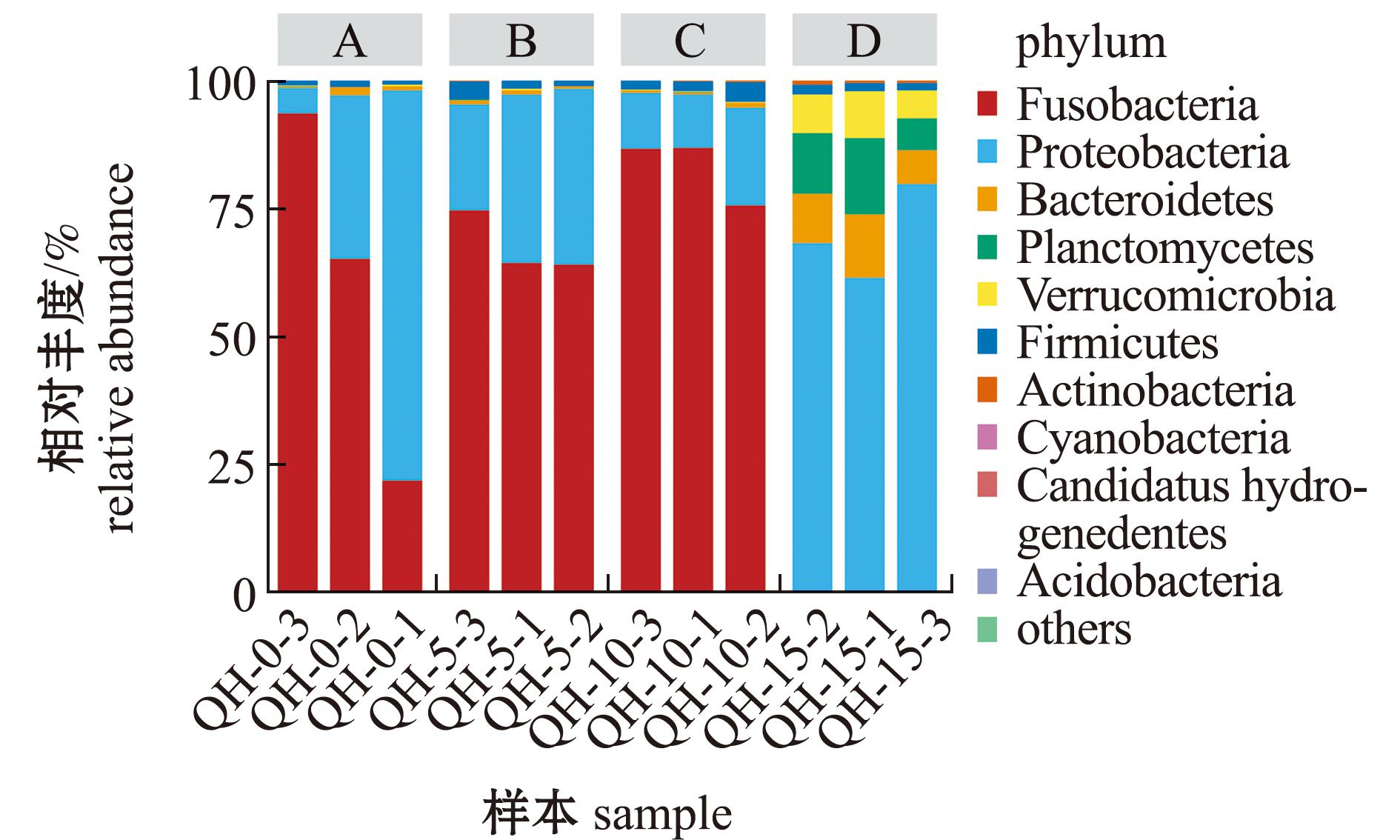
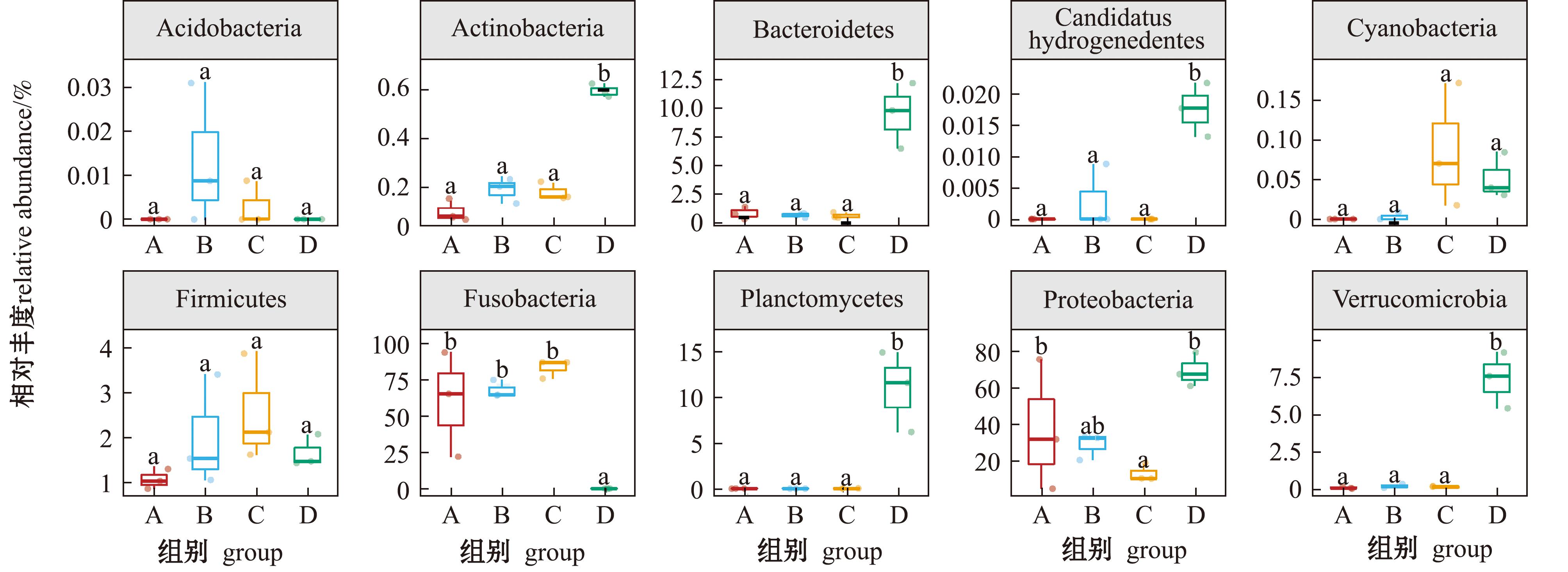
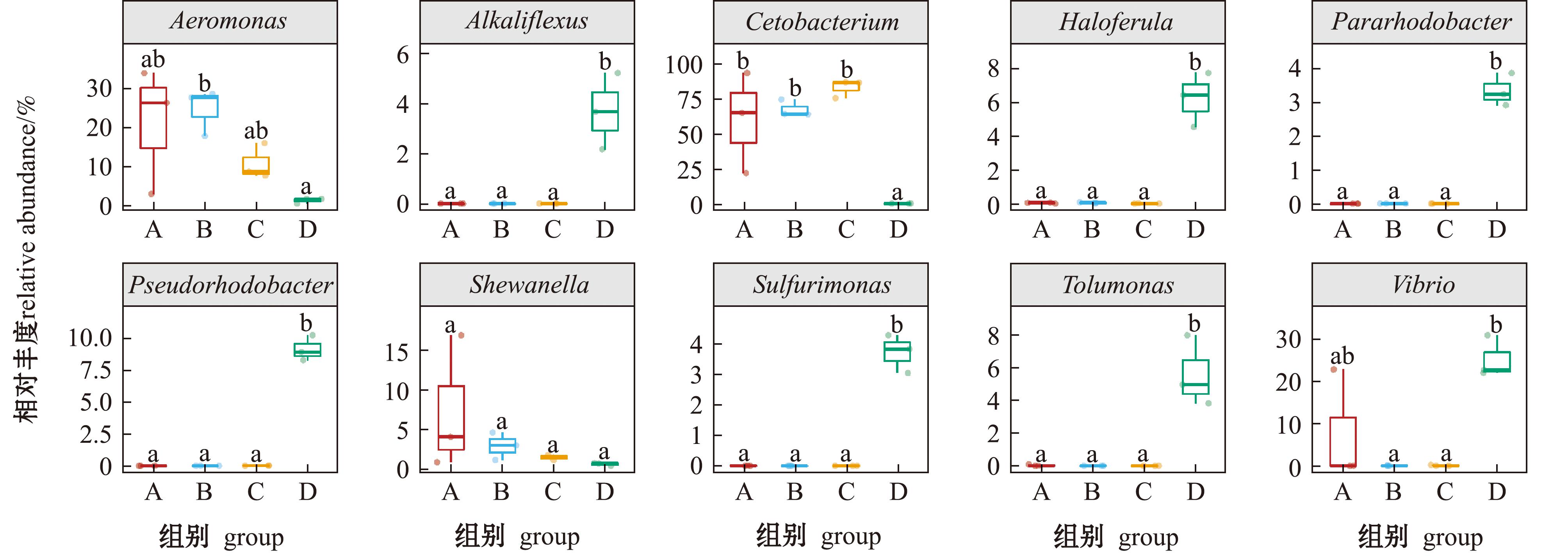
从图3可见,0、5、10盐度组中菌群的分布与15盐度组相比有明显变化。从图4可见:在门水平上,不同盐度组肠道菌群的相对丰度不同,其中,0、5、10盐度组肠道菌群中,相对丰度较高的是梭杆菌门(Fusobacteria)和变形菌门(Proteobacteria),其次是厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)和浮霉菌门(Planctomycetes)等也占有一定比例;15盐度组中以变形菌门、拟杆菌门、浮霉菌门和疣微菌门为主要菌群。从图5可见:在属水平上,0、5、10盐度组肠道菌群中,鲸杆菌属(Cetobacterium)和气单胞菌属(Aeromonas)相对丰度较高,其次是弧菌属(Vibrio)和希瓦氏菌属(Shewanella);15盐度组中以弧菌属、假红杆菌属(Pseudorhodobacter)和卤蕨属(Haloferula)为主要菌群。
2.6 肠道菌群组成结构的主成分分析
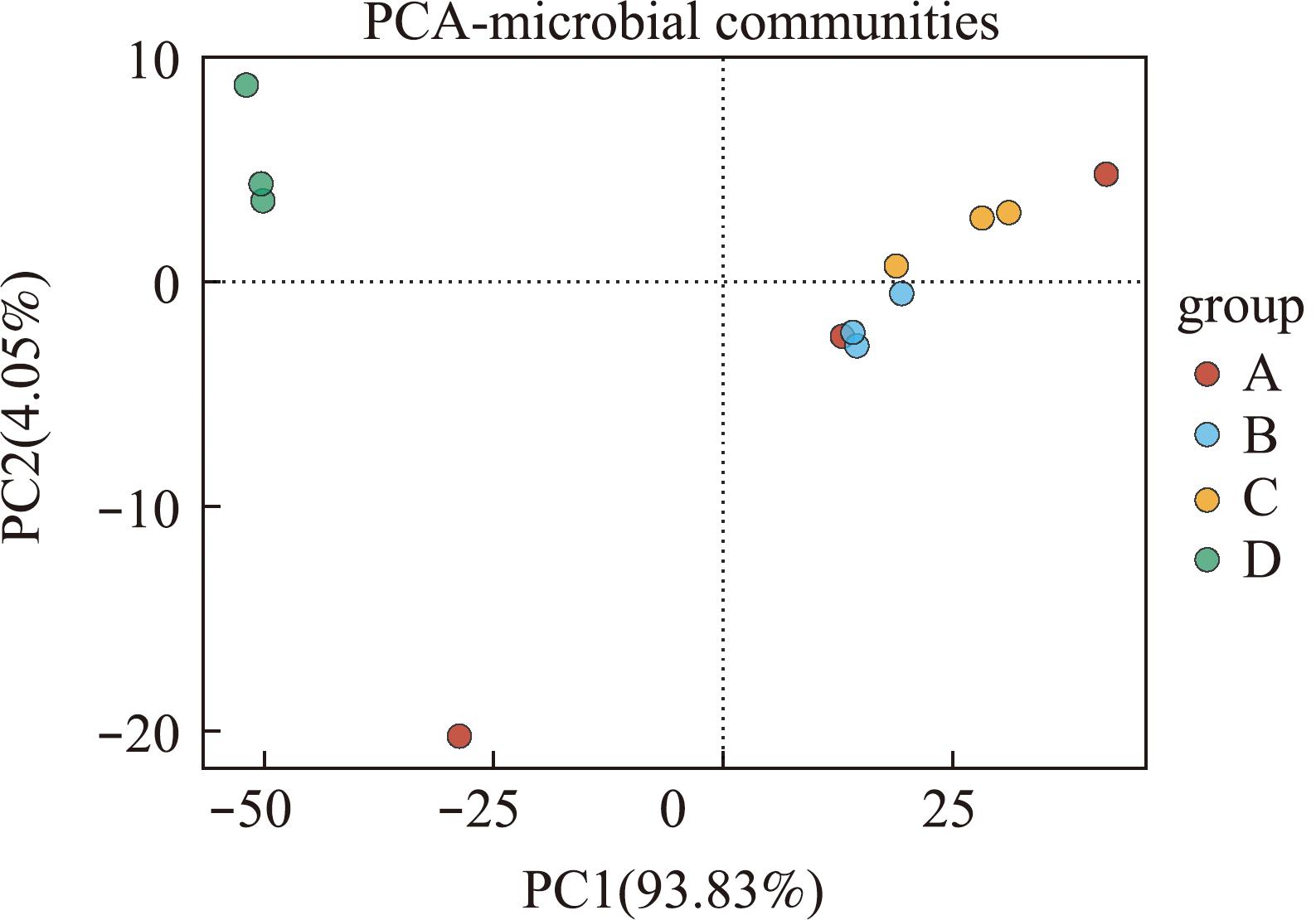
PCA分析显示:青海湖裸鲤高盐度组样品和低盐度样品出现在不同象限,表明高盐度组和低盐度组的肠道菌群组成不同,二者之间存在明显差异;在无盐胁迫的对照组,青海湖裸鲤的肠道菌群呈无规律分布;在同一盐度下,肠道菌群样品之间距离较近,呈明显聚类,这表明在同一盐度下青海湖裸鲤肠道菌群结构组成有相同的变化,个体差异较小(图6)。
2.7 LefSe差异分析
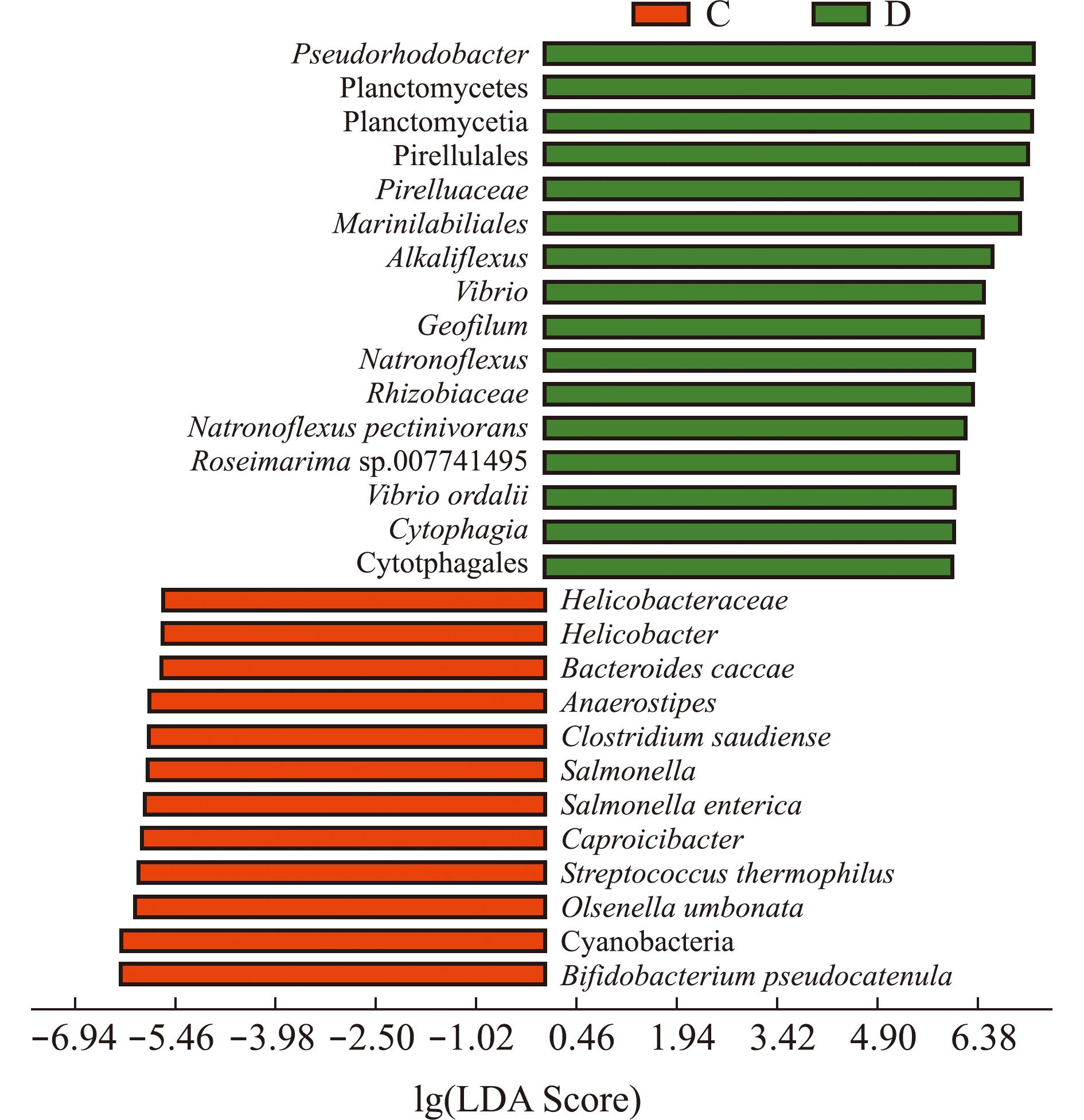
LDA值分布柱状图中展示了LDA Score大于设定值的物种,即组间具有统计学差异的Biomarker代表丰度差异显著的物种(图7)。LefSe分析发现:0、5、10盐度组之间的Group、LDA、P值均为空白,表示该物种在组间无显著性差异,即为无差异物种;15盐度组与其他3组相比存在统计学差异的生物标志物,本研究中共获得28个生物标志物;10盐度组的生物标志物主要为假小链双歧杆菌(Bifidobacterium pseudocatenulatum)和蓝细菌门(Cyanobacteria),15盐度组的生物标志物主要为假红杆菌属(Pseudorhodobacter)和浮霉菌门(Planctomycetes)(图7)。这表明,15盐度组的青海湖裸鲤肠道菌群与其他3组之间差异显著,与上述研究结果一致。
2.8 肠道菌群功能预测
利用PICRUSt2软件对不同盐度下青海湖裸鲤的肠道菌群进行功能预测分析,通过KEGG可以注释到L1、L2、L3 3个水平。在一级功能层面,各盐度组青海湖裸鲤肠道菌群的主要功能包括细胞过程(cellular processes)、环境信息处理(environ-mental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、新陈代谢(metabolism)和有机系统(organismal systems)等。在二级功能层面,共注释到41个信号通路,各样本中的菌群功能基因丰度如图8所示。在细胞过程中,肠道菌群主要在活性(motility)及细胞生长与死亡(cell growth and death)中起作用;在环境信息处理过程中,菌群主要在膜运输(membrane transport)和信号传导(signal transduction)中起作用;在遗传信息传递过程中,菌群主要在翻译(translation)及复制与修复(replication and repair)等中起作用;在人类病方面,主要在神经退行性疾病(neurodegener ative disease)及内分泌与代谢疾病(endocrine and metabolic disease)等中起作用;在新陈代谢方面,菌群主要在碳水化合物代谢(carbohydrate metabolism)、氨基酸代谢(amino acid metabolism)、核苷酸代谢(nucleotide metabolism)及辅助因子与维生素(cofactors and vitamins)等中起着重要作用。
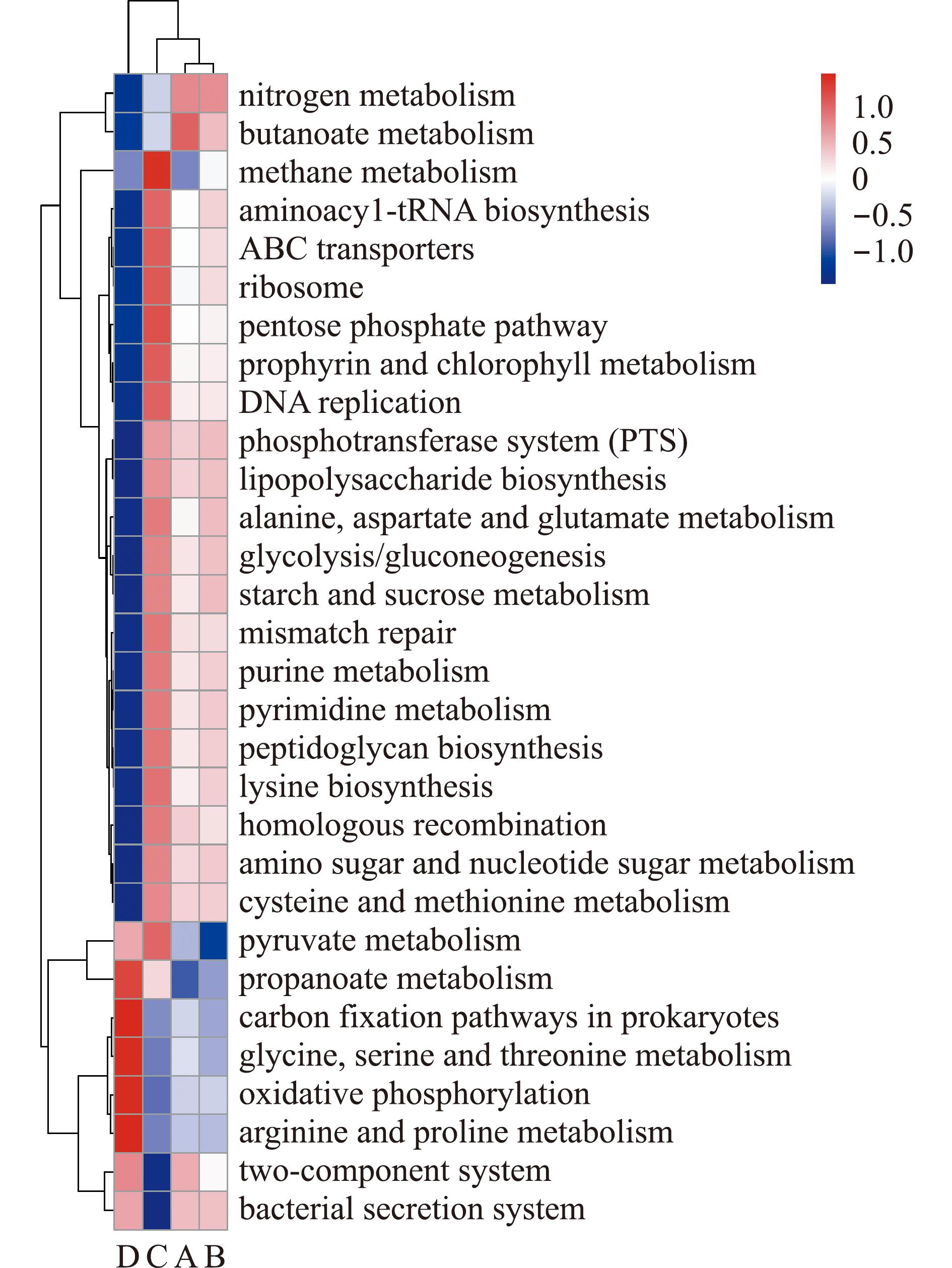
在三级功能层面,对照组与低盐度组(盐度5和10)肠道菌群功能基因主要参与同源重组(homologous recombination)、氨基酸糖与核苷酸糖代谢(amino sugar and nucleotide sugar metabolism)、半胱氨酸与蛋氨酸代谢(cysteine and methionine metabolism)、磷转移酶系统(phosphotransferase system)和脂多糖的生物合成(lipopolysaccharide biosynthesis)等相关通路;高盐度组肠道菌群功能基因主要参与原核生物的碳固定途径(carbon fixation pathways in prokaryotes)、丝氨酸与苏氨酸代谢(serine and threonine metabolism)、氧化磷酸化(oxidative phosphorylation)、丙酸盐代谢(propanoate metabolism)及精氨酸与脯氨酸代谢(arginine and proline metabolism)等相关通路(图9)。
3 讨论
3.1 盐度对青海湖裸鲤肠道菌群多样性和丰度的影响
本研究表明,不同盐度下青海湖裸鲤肠道内容物样品OTU个数具有一定的差别,除对照组外,随盐度增加,肠道菌群的OTU数也随之增多,原因可能是在高盐度下,原本的肠道菌群不足以提供宿主所需的营养物质,随着盐度的升高,更多种类的耐盐菌出现,以促进宿主消化吸收[12]。而对照组与盐度5之间所呈现OTU数的下降趋势可能是盐度抑制了一些致病菌的生长[13]。田璐[14]对3个盐度下的黄姑鱼(Nibea albifora)研究发现,高盐度组下黄姑鱼肠道菌群较对照组与低盐组明显增多,这与本研究结果相似。
3.2 盐度对青海湖裸鲤肠道优势菌群的影响
水体中的盐度变化会影响鱼类肠道菌群的多样性及结构,不同盐度下的优势菌群也有所差异。李改娟等[15]研究发现,鲑科鱼类在由淡水入海的过程中,肠道菌群组成结构会随着环境中盐度的变化而变化,在淡水中的鲑科鱼类肠道优势菌群以单胞菌属和肠杆菌属为主,而在海水中嗜盐性的弧菌则占优势。本研究表明,15高盐度组青海湖裸鲤肠道梭杆菌门数量与其他3个低盐度组相比显著降低,可能是高盐度下抑制了此类细菌的生长,该菌主要与炎症性肠病和肠道感染有关[14];而变形菌门、拟杆菌门、浮霉菌门和疣微菌门相较于其他3组明显增多,可能是在高盐度生境下营养匮乏,这些菌可为宿主分泌多种营养物质[16],并促进宿主消化吸收。此外,在高盐度组中还发现,具有致病性的弧菌属显著高于对照组,可能是此类细菌的喜盐偏好性质决定了在此盐度下组成丰富[17],其也是导致青海湖裸鲤在高盐度下存活率略低的主要原因。本研究中发现,低盐度可以促进青海湖裸鲤的生长发育,在0、5、10低盐度组中,鲸杆菌属为优势菌群,鲸杆菌属数量随着盐度的升高呈现先升高后下降的趋势,并在高盐度15时急剧降低。鲸杆菌属不仅可以合成维生素B12[18],帮助宿主分解营养物质,而且还与宿主抗病原菌感染相关[19],这可能是在低盐度组青海湖裸鲤生长性能优于高盐度组的主要原因之一;气单胞菌属可以引起多种水产养殖动物患病,不仅会给水产养殖业带来严重的经济损失,还通过水生动物和水产品感染人和动物,导致腹泻和食物中毒[20]。田璐[14]研究发现,高盐度组气单胞菌属显著低于对照组,这是因为其最适生存盐度为0~4,所以在高盐度下抑制了该菌的生长,通过16S rRNA基因组测序发现,高盐度组与低盐度组的黄姑鱼肠道菌群组成差异较大,这与本研究结果一致。
3.3 盐度对青海湖裸鲤肠道菌群功能的影响
通过PICRUSt2软件对不同盐度下青海湖裸鲤肠道菌群进行功能预测发现,与新陈代谢功能相关的菌群丰度较高,其中,碳水化合物代谢、氨基酸代谢、核苷酸代谢及辅助因子与维生素相关菌群占比较高,表明青海湖裸鲤肠道菌群对宿主的营养代谢方面起作用。常亚娟等[21]通过对长江口野生日本鳗鲡(Anguilla japonica)幼体肠道菌群进行COG和KEGG功能注释预测发现,其幼体肠道中与新陈代谢功能相关的菌群丰度最高,其中,氨基酸转运与代谢、无机离子运输与代谢、脂质代谢和碳水化合物运输与代谢相关菌群占比较高;粘立等[22]对皱纹盘鲍(Haliotis discus hannai)肠道菌群研究发现,其肠道菌群所编码的基因与代谢功能的相关性增加,包括氨基酸代谢、碳水化合物代谢和酶家族等,说明鲍肠道菌群参与了多种分子代谢;姜之信等[23]对玻璃鳗肠道菌群进行功能预测分析发现,鳗肠道均以氨基酸运输与代谢功能菌群占比最高。综上所述,肠道菌群主要在宿主的代谢方面起作用。后续试验可以针对青海湖裸鲤在营养代谢过程中对消化道中的核心菌群进行分离培养,从幼苗孵育到放流之前的养殖过程中饲喂添加相应益生菌的饵料,以此来提高青海湖裸鲤在增殖放流中的成活率及快速适应盐度环境的能力,有望加速青海湖裸鲤种质资源的恢复。赵园园等[24]在研究赤眼鳟(Squaliobarbus curriculus)时也体现了通过筛选优势菌制作益生菌饲料以促进健康养殖这一思路。
鱼类肠道微生物群落对肠道的正常发育、改善体内能量吸收与脂肪代谢,以及抵御病原侵入等过程中具有极其重要的作用[25-26],同时也受到如宿主的生境(盐度、温度和溶解氧等)、食性、年龄结构及饲料组成成分[27-28]等诸多因素的影响。本研究中,通过对青海湖裸鲤肠道菌群的分析,揭示了不同盐度下青海湖裸鲤肠道菌群的结构组成及优势菌群的功能,为青海湖裸鲤传统的人工养殖模式提供了新的思路(如微生物饲料的使用),并对青海湖裸鲤种质资源的加速恢复具有重要的意义,但不同盐度下的优势菌群在肠道营养中的确切作用还有待进一步探究。
4 结论
1)不同盐度下青海湖裸鲤肠道菌群组成结构有一定差异,对照组与低盐度组(盐度5、10)的肠道菌群组成结构较为接近,而高盐度组(盐度15)与其他3组相比,肠道菌群群落组成结构差异明显。
2)15盐度组与0、5、10盐度组之间的青海湖裸鲤肠道优势菌群及其功能存在差异,对照组和低盐度组中肠道优势菌群的KEGG功能预测主要富集在新陈代谢、碳水化合物代谢、同源重组、氨基酸糖与核苷酸糖代谢等通路,而高盐度组优势菌群的主要功能是新陈代谢、碳水化合物代谢、原核生物的碳固定途径及丝氨酸与苏氨酸代谢氧化等通路。
参考文献:
[1] 韩青青.低氧诱导因子-1(HIF-1)对青海湖裸鲤和鲫鱼碱性环境适应的影响[D].金华:浙江师范大学,2019. HAN Q Q.Effects of hypoxia-inducible factor-1 (HIF-1) on the alkaline environment adaptation of naked carp in Qinghai Lake and crucian carp[D].Jinhua:Zhejiang Normal University,2019.(in Chinese)
[2] 黄屾,李长忠,李梓瑄,等.盐度对青海湖裸鲤生长及渗透调节基因的影响[J].水产科学,2022,41(4):527-536. HUANG S,LI C Z,LI Z X,et al.Effects of different salinities on growth and osmotic regulation gene expression of Gymnocypris przewalskii[J].Fisheries Science,2022,41(4):527-536.(in Chinese)
[3] WU J,PENG Z,GUAN T W,et al.Diversity of Actinobacteria in sediments of Qaidam Lake and Qinghai Lake,China[J].Archives of Microbiology,2021,203(6):2875-2885.
[4] 崔美岩,吕好新,张志霞,等.青海湖裸鲤肠道乳酸菌多样性与抑菌活性[J].微生物学通报,2016,43(9):2028-2039. CUI M Y,LÜ H X,ZHANG Z X,et al.Diversity and antimicrobial activity of intestinal lactic acid bacteria isolated from Gymnocypris przewalskii[J].Microbiology China,2016,43(9):2028-2039.(in Chinese)
[5] 黄建蓉.盐度对青藏高原湖泊微生物群落结构与功能稳定性的影响[D].武汉:中国地质大学,2021. HUANG J R.The influence of salinity on the microbial community structure and functional stability in Qinghai-Tibetan Lakes[D].Wuhan:China University of Geosciences,2021.(in Chinese)
[6] 孙梦蕾,蒋洁兰,王莉苹,等.低盐胁迫下红鳍东方鲀幼鱼鳃、肾和肠组织结构的变化[J].广东海洋大学学报,2016,36(6):38-43. SUN M L,JIANG J L,WANG L P,et al.Structural changes in gill,kidney and intestine of juvenile Takifugu rubripes under low salinity treatment[J].Journal of Guangdong Ocean University,2016,36(6):38-43.(in Chinese)
[7] LIU Y,FU X,ZHOU H,et al.Different types of non-starch polysaccharides alter the growth,intestinal flora and serum metabolite profile of grass carp,Ctenopharyngodon idella[J].Metabolites,2022,12(10):1003.
[8] 郭倩倩,胡军娜,魏金锁,等.水产动物肠道微生物的研究方法[J].河南水产,2019(1):17-20. GUO Q Q,HU J N,WEI J S,et al.Study method of intestinal microflora on aquatic animal[J].Henan Fisheries,2019(1):17-20.(in Chinese)
[9] EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[10] STACKEBRANDT E,GOEBEL B M.Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic and Evolutionary Microbiology,1994,44(4):846-849.
[11] WANG Q,GARRITY G M,TIEDJE J M,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Applied and Environmental Microbiology,2007,73(16):5261-5267.
[12] LIU D R,ZHANG Z W,SONG Y K,et al.Effects of salinity on growth,physiology,biochemistry and gut microbiota of juvenile grass carp (Ctenopharyngodon idella)[J].Aquatic Toxicology,2023,258:106482.
[13] CHEN H,PAN J F,WANG Y Z,et al.Growth,health status and gut microbiota of the scalloped spiny lobster (Panulirus homarus) at different salinities[J].Aquaculture,2023,562:738779.
[14] 田璐.盐度对黄姑鱼生存生长、非特异性免疫及肠道菌群的影响[D].舟山:浙江海洋大学,2019. TIAN L.Effects of salinity on growth,nonspecific immunity and micro-organism of Nibea albiflora[D].Zhoushan:Zhejiang Ocean University,2019.(in Chinese)
[15] 李改娟,赵全东,高娜,等.鱼类健康与肠道微生物关系研究进展[J].河北渔业,2020(8):56-58. LI G J,ZHAO Q D,GAO N,et al.Research progress on the relationship between gut microbes and fish health[J].Hebei Fisheries,2020(8):56-58.(in Chinese)
[16] 油九菊,柳敏海,殷小龙,等.基于高通量测序的东海带鱼肠道菌群结构分析[J].渔业研究,2018,40(6):434-440. YOU J J,LIU M H,YIN X L,et al.Analysis of bacterial communities’ structure in intestinal flora of Trichiurus haumela based on high-throughput sequencing[J].Journal of Fisheries Research,2018,40(6):434-440.(in Chinese)
[17] 冯丹,高小迪,李云凯.海洋鱼类肠道微生物研究进展及应用前景[J].生态学杂志,2021,40(1):255-265. FENG D,GAO X D,LI Y K.Research progress and application prospect of gut microbiota of marine fish[J].Chinese Journal of Ecology,2021,40(1):255-265.(in Chinese)
[18] 孙中石,范艳蕊,吕爱军,等.锦鲤肠道不同部位菌群组成结构及多样性分析[J].南方农业学报,2021,52(2):483-490. SUN Z S,FAN Y R,LÜ A J,et al.Community and diversity analysis of microflora from different intestinal segments of koi carp(Cyprinus carpio var.koi)[J].Journal of Southern Agriculture,2021,52(2):483-490.(in Chinese)
[19] 苟妮娜,钟明智,王开锋.基于16S rRNA高通量测序的野生和养殖多鳞白甲鱼肠道微生物群落组成研究[J].西北农业学报,2021,30(7):963-970. GOU N N,ZHONG M Z,WANG K F.Intestinal microbial community of wild and cultured Onychostoma macrolepi based on 16S rRNA high-throughput sequencing[J].Acta Agriculturae Boreali-Occidentalis Sinica,2021,30(7):963-970.(in Chinese)
[20] WANG F Y,LIU Y,LI G X,et al.16S rRNA gene amplicon sequencing of gut microbiota from naked carp (Gymnocypris przewalskii) in Qinghai Lake,China[J].Microbiology Resource Announcements,2021,10(23):e0037421.
[21] 常亚娟,唐文乔,郭弘艺.基于16S rRNA高通量测序的长江口日本鳗鲡幼体肠道微生物群落研究[J].中国水产科学,2023,30(6):699-710. CHANG Y J,TANG W Q,GUO H Y.Study on the intestinal microbial community of Japanese eel larvae in the Yangtze River Estuary based on 16S rRNA high-throughput sequencing[J].Journal of Fishery Sciences of China,2023,30(6):699-710.(in Chinese)
[22] 粘立,王志鹏,张海涛,等.皱纹盘鲍在转口期阶段肠道菌群结构演替研究[J].渔业科学进展,2023,44(1):156-168. NIAN L,WANG Z P,ZHANG H T,et al.Succession of the intestinal microflora structure of Haliotis discus hannai during the weaning period[J].Progress in Fishery Sciences,2023,44(1):156-168.(in Chinese)
[23] 姜之信,李慷,巴旭冰,等.中国近海洄游日本鳗鲡(Anguilla japonica)玻璃鳗肠道微生物菌群分析[J].海洋与湖沼,2023,54(6):1728-1736. JIANG Z X,LI K,BA X B,et al.Analysis of intestinal microflora of Japanese eel (Anguilla japonica) in migration along China offshore[J].Oceanologia et Limnologia Sinica,2023,54(6):1728-1736.(in Chinese)
[24] 赵园园,杨丽敏,孟金柱,等.赤眼鳟肠道菌群结构及其潜在功能研究[J].山东农业大学学报(自然科学版),2023,54(1):47-54. ZHAO Y Y,YANG L M,MENG J Z,et al.Study on the structure and potential function of intestinal microbiota in Squaliobarbus curriculus[J].Journal of Shandong Agricultural University (Natural Science Edition),2023,54(1):47-54.(in Chinese)
[25] 樊英,王友红,姬广磊,等.基于宏基因组测序技术揭示大泷六线鱼肠道微生物特征[J].微生物学报,2023,63(1):357-375. FAN Y,WANG Y H,JI G L,et al.Metagenomic sequencing reveals the intestinal microbial characteristics of Hexagrammos otakii[J].Acta Microbiologica Sinica,2023,63(1):357-375.(in Chinese) 26] ZUO Z H,SHANG B J,SHAO Y C,et al.Screening of intestinal probiotics and the effects of feeding probiotics on the growth,immune,digestive enzyme activity and intestinal flora of Litopenaeus vannamei[J].Fish &Shellfish Immunology,2019,86:160-168.
[27] 张丽,许国焕,成艳波,等.高糖饲料对黄颡鱼生长、糖脂代谢及肠道代谢物的影响[J].中国饲料,2021(5):67-71. ZHANG L,XU G H,CHENG Y B,et al.Effect of high carbohydrate diet on growth,glucose and lipid metabolism and intestinal metabolites of yellow catfish(Pelteobagrus fulvidraco)[J].China Feed,2021(5):67-71.(in Chinese)
[28] 丰文雯.草鱼消化道微生物影响因素的比较研究[D].北京:中国科学院大学, 2018. FENG W W.Comparative study of microbiological influencing factors in the digestive tract of Ctenopharyngodon idella[D].Beijing:University of Chinese Academy of Sciences,2018.(in Chinese)
Response of intestinal microbiome of Gymnocypris przewalskiito different salinity stresses
ZHAO Zifeng1,JIN Wenjie1,2,ZHAO Jing1,WANG Rong1,QI Hongfang3,LI Jin3,BAI Peng1,2*,LI Changzhong1,2*
(1.Research Laboratory of Plateau Animals and Resource Utilization,College of Ecological and Environmental Engineering,Qinghai University,Xining 810016,China;2.Laboratory of Genetic Engineering,College of Ecological and Environmental Engineering,Qinghai University,Xining 810016,China;3.Laboratory of Breeding and Conservation of Naked Carp in Qinghai Lake,The Rescue and Rehabilitation Center of Naked Carps of Qinghai Lake,Xining 810016,China)
Abstract: In order to explore the effect of salinity stress on the structure of the intestinal flora of Gymnocypris przewalskii, microbial sequencing and information analysis of the intestinal contents of G.przewalskii at different salinity (0, 5, 10, 15) were based on the three-generation full-length 16S amplicon sequencing technology. The relative abundance of the intestinal flora of G.przewalskii differed in different salinity groups, with higher relative abundance of Fusobacteria and Proteobacteria, followed by Firmicutes and Bacteroidetes, in the intestinal flora in the control group (salinity 0) and in the low-salinity group (salinity 5, 10). Verrucomicrobia, Actinobacteria and Planctomycetes were also represented. Proteobacteria, Bacteroidetes, Planctomycetes and Verrucomicrobia were the dominant flora in the high-salinity group (salinity 15). At genus level, the relative abundance of Cetobacterium and Aeromonas followed by Vibrio and Shewanella was higher in the intestinal flora in the control and low-salinity groups, while Vibrio, Pseudorhodobacter and Haloferula were the dominant flora in the high-salinity group. The findings indicated that low-salinity (salinity ≤10) stress had little effect on the structure of the intestinal flora of G.przewalskii, whereas high-salinity (salinity≥15) stress inhibited and had a greater effect on the growth of the intestinal flora and dominant flora of G.przewalskii.
Key words: Gymnocypris przewalskii; 16S amplicon; salinity; intestinal flora