一株盐单胞菌属(Halomonas)细菌趋化因子受体基因挖掘及相关蛋白序列分析
徐爽1,李晨1,姜嘉琳1,谢晓晨1,王博1,霍忠明2,方蕾1*
(1.大连海洋大学 海洋科技与环境学院,辽宁 大连 116023;2.大连海洋大学 水产与生命学院,辽宁 大连 116023)
摘要:为了解一株长牡蛎(Crassostrea gigas)致病性盐单胞菌(Halomonas sp.)7T的趋化性,采用毛细管法研究其趋化行为,并基于全基因组数据,利用KEGG Pathway分析其趋化信号通路,通过与同源物种比对分析趋化因子受体种类,进一步挖掘甲基受体趋化蛋白(methyl-accepting chemotaxis proteins,MCPs)序列,再结合MeMe数据库和TMHMM分析MCPs结构。结果表明:Halomonas sp.7T具备趋化行为,其全基因组含有完整趋化信号通路,共有22个基因编码MCPs,涵盖5种常见趋化因子受体;22个MCPs均含有信号转导结构域,其中,19个含有跨膜结构域,3个不含跨膜结构域;基于信号转导结构域内七肽单位保守序列和存在的对称插入缺失对这22个MCPs进行分类,发现17个为36H型,1个为40H型,其余4个与已知分类均不匹配;跨膜结构分布特征和疏水性分析发现,这22个MCPs中有20个符合拓扑结构,其中Class Ⅰ型、Class Ⅱ型、Class Ⅲ型、Class Ⅳ型分别为10、4、3、3个,另有2个MCPs不符合拓扑结构。研究表明,长牡蛎致病性盐单胞菌7T具备趋化性,其基因组存在完整趋化信号通路,有22个基因编码MCPs,这些MCPs具有独特的结构,可能参与介导与细菌生存和环境胁迫响应相关的趋化行为。
关键词:盐单胞菌;趋化性;趋化因子受体基因;甲基受体趋化蛋白;长牡蛎
DOI:10.16535/j.cnki.dlhyxb.2023-222
文章编号:2095-1388(2024)02-0203-12
中图分类号:S 917.1;Q 735
文献标志码:A
收稿日期:2023-10-17
基金项目:辽宁省应用基础研究计划项目(2022JH2/101300141);山东省重点研发计划项目(2023LZGCQY001);国家重点研发计划项目(2019YFC1407704,2018YFD0901404);国家自然科学基金(41606133);大连市杰出青年科技人才项目(2021RJ09)
作者简介: 徐爽(1997—),男,硕士研究生。E-mail:13263368672@163.com
通信作者:方蕾(1978—),女,博士,副教授。E-mail:fanglei2014@dlou.edu.cn
长牡蛎(Crassostrea gigas)又称太平洋牡蛎、日本真牡蛎,为全球主要养殖牡蛎品种之一,中国主要在山东和广东省沿海养殖,其具有生长速度快、出肉率高和适应性强等特点[1]。然而,长牡蛎在不同生长阶段都有被病原微生物(原生动物、细菌和病毒等)侵染的可能,微生物病害也是导致长牡蛎减产的原因之一[1]。
盐单胞菌属(Halomonas)细菌为革兰氏阴性细菌,其好氧、嗜盐和/或耐盐,主要分布在盐碱地、盐湖、海水及部分海洋生物体表或体内[2]。盐单胞菌多与海洋生物营共生,人们先后从海鞘、虾和海胆中分离出对宿主无致病性的共生盐单胞菌[2]。此外,盐单胞菌对人、大型海藻、海水鱼和扇贝等的致病性也有报道[2]。盐单胞菌属细菌的工业用途广泛,可作为细胞工厂生产生物塑料、聚酯纤维和蛋白质等,且由于其嗜盐或耐盐,大规模培养时污染容易被控制,被认为是下一代工业中生物制造的重要载体[3]。盐单胞菌还可用于去除环境中的重金属、放射性物质,以及制备生物燃料电池[4]。目前,已有多株盐单胞菌属细菌完成了全基因组测序,一些功能基因已被解析,包括趋化信号通路[5]、反硝化信号通路、硫化信号通路[6]和耐盐信号通路[7]等。
细菌趋化性是指具有运动能力的细菌在受到化学刺激时能够做出趋向有利环境同时远离不利环境的定向运动[8]。研究表明,趋化行为在致病性细菌侵染宿主的早期阶段发挥重要作用,能指导细菌进入宿主体内并表达毒力基因[9]。趋化基因与毒力基因协同作用,使细菌的致病性发挥到最大[9]。
细菌中存在由趋化信号接收器和调节蛋白组成的趋化信号通路,以介导趋化行为。趋化信号接收器是指位于细胞外膜的甲基受体趋化蛋白(methyl-accepting chemotaxis proteins,MCPs),其广泛存在于古菌和细菌中[10]。目前,已发现的MCP基因数量逾10万,多源于细菌。研究表明,细菌响应环境因子变化的速度与其基因组中MCP基因的数量呈正相关,而细菌基因组中MCP基因的数量主要由细菌生活的环境和代谢途径决定,与基因组大小无直接相关性[11]。
本课题组从北黄海长牡蛎幼虫养殖环境样本中分离获得一株致病性盐单胞菌(Halomonas sp.)7T,人工感染试验结果显示,其能显著降低长牡蛎D形幼虫和稚贝的存活率,其中,感染后的D形幼虫出现软体组织分解、变红等症状,感染后的稚贝出现外套膜萎缩、变红等症状[12]。本文中,采用毛细管法研究了Halomonas sp.7T的趋化行为,并基于全基因组数据,利用KEGG Pathway分析Halomonas sp.7T的趋化信号通路,通过与同源物种比对分析趋化因子受体种类,进一步挖掘甲基受体趋化蛋白MCPs序列,再结合MeMe数据库和TMHMM分析MCPs的结构,以期为解析盐单胞菌7T的致病机理提供科学参考。
1 材料与方法
1.1 材料
1.1.1 菌种来源 菌种Halomonas sp.7T由本课题组分离自北黄海长牡蛎幼虫养殖环境样本[12]。
1.1.2 趋化试验用培养基及溶液
1)2216E液体培养基。1 L 2216E液体培养基包含5 g蛋白胨、1 g酵母浸粉、0.1 g柠檬酸铁、19.45 g氯化钠、5.98 g氯化镁、3.24 g硫酸钠、1.8 g氯化钙、0.55 g氯化钾、0.16 g碳酸钠、0.08 g溴化钾、0.034 g氯化锶、0.022 g硼酸、0.004 g硅酸钠、0.002 4 g氟化钠、0.001 6 g硝酸铵、0.008 g磷酸氢二钠和适量去离子水,pH为7.6±0.2 (25 ℃),121 ℃高压灭菌15 min。
2)2216E固体培养基。向1 L 2216E液体培养基中添加15 g琼脂,121 ℃高压灭菌15 min,即得固体培养基。
3)无机培养基。1 L无机培养基包含3 g磷酸氢二钠、1 g磷酸二氢钾、5 g硫酸铵、0.02 g氯化镁、0.02 g氯化钙、0.001 5 g对氨基苯甲酸和适量去离子水,pH为7.0 (25 ℃),121 ℃高压灭菌15 min。
4)趋化缓冲液。1 L趋化缓冲液包含2.282 g磷酸氢二钾、0.034 g EDTA、0.12 g硫酸镁和适量离子水,pH为7.0,121 ℃高压灭菌15 min。
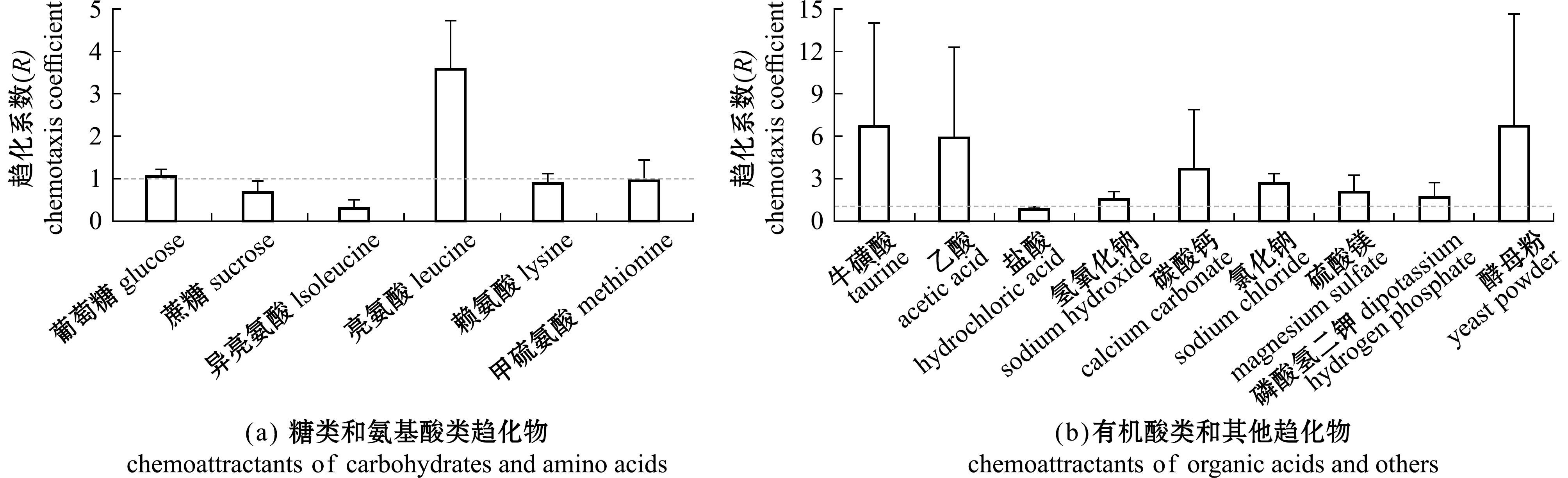
5)趋化待测液。待测趋化剂包括葡萄糖、蔗糖、异亮氨酸、亮氨酸、赖氨酸、甲硫氨酸、乙酸、牛磺酸、碳酸钙、酵母粉、盐酸、氢氧化钠、氯化钠、磷酸氢二钾和硫酸镁等。以各种待测趋化剂为溶质,以液体无机培养基为溶剂分别配制质量浓度为2 g/L的趋化待测液,用孔径为0.22 μm的无菌微孔滤膜过滤除菌后,再加入灭菌液体无机培养基稀释至终浓度为0.2 g/L(有机酸除外)或0.1 g/L(有机酸)。
1.1.3 趋化试验用Halomonas sp.7T菌液 从2216E固体培养基上挑取Halomonas sp.7T单菌落接种至2216E液体培养基,35 ℃、120 r/min条件下培养12 h至OD600 nm 为0.6~0.8,室温下以3 000×g离心10 min收集菌体,用灭菌趋化缓冲液清洗3次后再用灭菌趋化缓冲液调节OD600 nm至0.1。
1.2 方法
1.2.1 趋化试验设计 为了研究Halomonas.sp.7T是否具备趋化性,选取2种碳源(葡萄糖、蔗糖)、4种氨基酸(异亮氨酸、亮氨酸、赖氨酸和蛋氨酸)、2种有机酸(乙酸、牛磺酸)和7种其他物质(盐酸、氢氧化钠、碳酸钙、氯化钠、硫酸镁、磷酸氢二钾和酵母粉)作为趋化剂,采用改良后的毛细管法测定Halomonas sp.7T的趋化性[13]。试验组使用制备好的各种趋化待测液,对照组使用液体无机培养基。玻璃毛细管一端吸入5 μL趋化待测液(或液体无机培养基),另一端用2216E固体培养基封闭,然后插入加有400 μL OD600 nm为0.1的菌液中。28 ℃室温孵育1 h,取出毛细管用无菌海水清洗表面,折断毛细管,用洗耳球将趋化待测液(或液体无机培养基)吹入1.5 mL离心管中,加入1 mL灭菌海水混匀后,取100 μL涂布于2216E平板上,35 ℃倒置培养40 h后进行平板菌落计数。
每一种趋化剂的趋化试验均设置3组重复,数据处理参照刘力铭等[13]的方法。使用Excel 2019软件处理数据,使用SPSS 26软件分析差异的显著性。趋化系数(R)计算公式为
R=试验组趋化待测液中的菌落形成单位/空白对照组液体无机培养基中的菌落形成单位。
当试验组R>1且与空白对照组R存在显著性差异时,可判断细菌对趋化剂具有正趋化响应;当试验组R<1且与空白对照组R存在显著性差异时,可判断细菌对趋化剂具有负趋化响应。
1.2.2 Halomonas sp.7T基因组相似性计算 Halomonas sp.7T全基因组序列由本课题组提供,NCBI登录号为SRR16888456,CNCB登录号为PRJCA007167。用于分析基因组相似性的盐单胞菌属细菌基因组信息如表1所示。
表1 用于平均核苷酸相似度(ANI)分析的同属细菌基因组信息
Tab.1 Information of the genomes from the other bacterial species of Halomonas used in ANI analysis
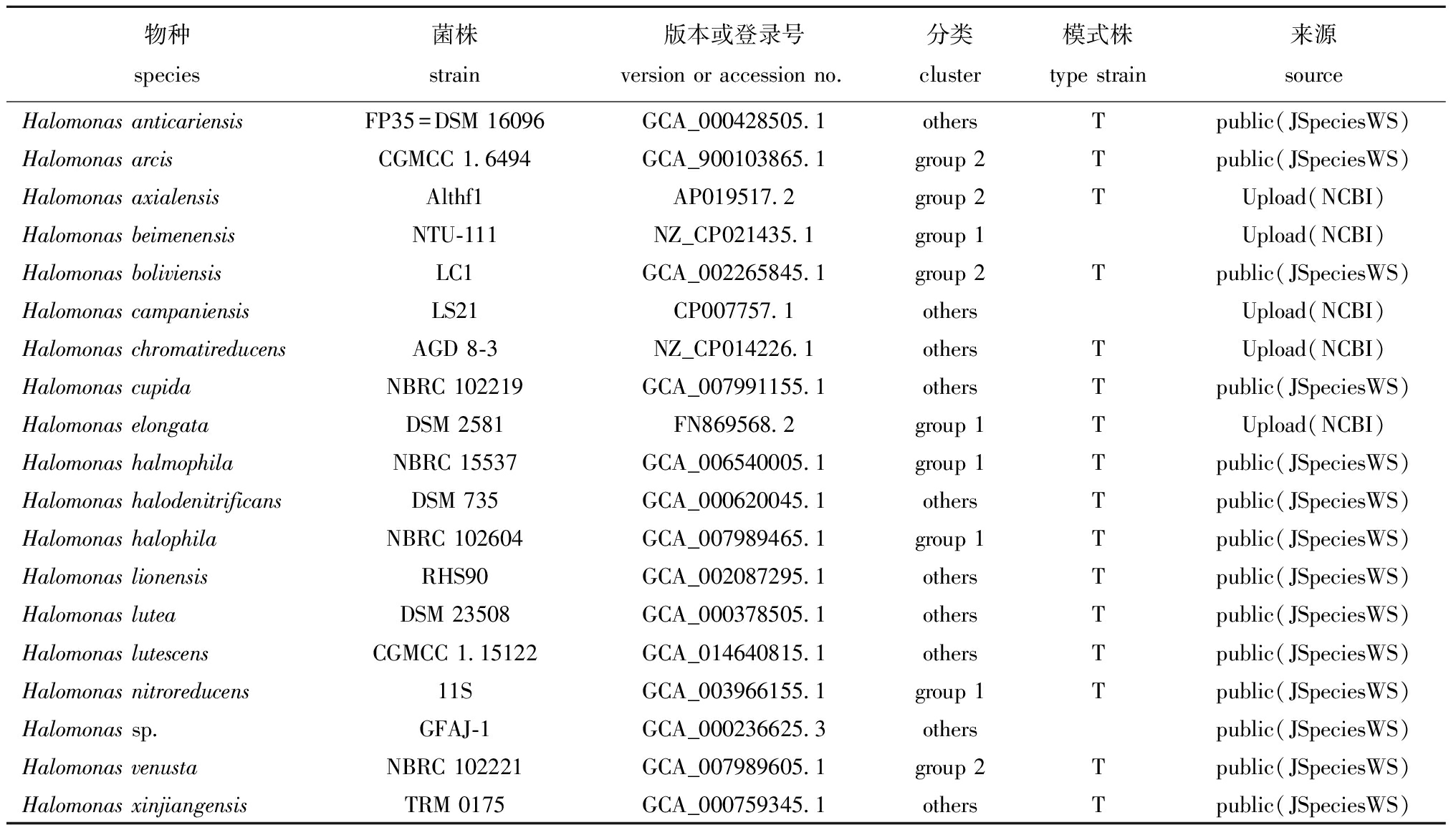
物种 species菌株 strain版本或登录号 version or accession no.分类 cluster模式株 type strain来源 sourceHalomonas anticariensisFP35=DSM 16096GCA_000428505.1othersTpublic(JSpeciesWS)Halomonas arcisCGMCC 1.6494GCA_900103865.1group 2Tpublic(JSpeciesWS)Halomonas axialensisAlthf1AP019517.2group 2TUpload(NCBI)Halomonas beimenensisNTU-111NZ_CP021435.1group 1Upload(NCBI)Halomonas boliviensisLC1GCA_002265845.1group 2Tpublic(JSpeciesWS)Halomonas campaniensisLS21CP007757.1othersUpload(NCBI)Halomonas chromatiredu-censAGD 8-3NZ_CP014226.1othersTUpload(NCBI)Halomonas cupidaNBRC 102219GCA_007991155.1othersTpublic(JSpeciesWS)Halomonas elongataDSM 2581FN869568.2group 1TUpload(NCBI)Halomonas halmophilaNBRC 15537GCA_006540005.1group 1Tpublic(JSpeciesWS)Halomonas halodenitrifi-cansDSM 735GCA_000620045.1othersTpublic(JSpeciesWS)Halomonas halophilaNBRC 102604GCA_007989465.1group 1Tpublic(JSpeciesWS)Halomonas lionensisRHS90GCA_002087295.1othersTpublic(JSpeciesWS)Halomonas luteaDSM 23508GCA_000378505.1othersTpublic(JSpeciesWS)Halomonas lutescensCGMCC 1.15122GCA_014640815.1othersTpublic(JSpeciesWS)Halomonas nitroreducens11SGCA_003966155.1group 1Tpublic(JSpeciesWS)Halomonas sp.GFAJ-1GCA_000236625.3otherspublic(JSpeciesWS)Halomonas venustaNBRC 102221GCA_007989605.1group 2Tpublic(JSpeciesWS)Halomonas xinjiangensisTRM 0175GCA_000759345.1othersTpublic(JSpeciesWS)
采用平均核苷酸相似度(average nucleotide identity,ANI)计算Halomonas sp.7T基因组与表1所列同属不同种细菌基因组的相似性,使用JSpeciesWS中的ANIb[14]工具计算受试基因组平均核苷酸相似度,并对结果进行制图分析。
1.2.3 Halomonas sp.7T趋化信号通路预测 在NCBI数据库中,Halomonas chromatireducens AGD 8-3的完整趋化信号通路已经被注释。因此,为了预测Halomonas sp.7T的趋化信号通路,选择H.chromatireducens AGD 8-3和该属的一个标准株H.axialensis Althf1作为参考菌株。将Halomonas sp.7T基因组编码的全部蛋白序列上传至KEGG数据库(https://www.kegg.jp/kegg/pathway.html)进行注释,以挖掘趋化信号通路基因。其中,H.chromatireducens AGD 8-3基因组数据的NCBI登录号为CP014226.1,KEGG登录号为T04292,H.axialensis Althf1基因组数据的NCBI登录号为AP019517.2,KEGG登录号为T06639。
1.2.4 Halomonas sp.7T甲基受体趋化蛋白的多序列比对与系统发育分析 选择H.chromatireducens作为盐单胞菌属参考菌株,在NCBI数据库中该菌株的MCP基因已完成注释和分类。对完成注释的Halomonas sp.7T甲基受体趋化蛋白序列,首先使用NCBI的Protein BLAST和Position-Specific-Iterated-BLAST(PSI-BLAST)进行同源序列比对[15],选择用于后续系统发育分析的蛋白序列。然后使用MEGA 7软件对H.chromatireducens和Halomonas sp.7T的MCPs进行系统发育分析,其中,采用ClustalW方法进行多序列比对,采用Maximum Likelihood(ML)方法建立系统发育树。ML参数设置:步长检验为500,计算模型为Poisson model,空位处理选择全部删除,其他选型保持默认值。在最终生成的系统发育树中,只对归类在同一节点下且自展值大于70的同源序列给出分类推断。
1.2.5 Halomonas sp.7T甲基受体趋化蛋白结构域和跨膜结构域预测 利用SMART在线数据库(http://smart.embl.de)、NCBI的CDD(Conserved Domain Database)数据库(http://www.ncbi. nlm.nih.gov/Structure/cdd/wrpsb.cgi)及MeMe网站(https://meme-suite.org/meme/index.html)对Halomonas sp.7T的MCPs结构域进行分析,再利用TBtools软件完成分析结果的制图输出。利用TMHMM 2.0[16]对Halomonas sp.7T甲基受体趋化蛋白跨膜区域进行在线分析,预测22个MCPs的跨膜位点和结构。
1.2.6 Halomonas sp.7T的MCP基因启动子预测 对转录起始位点上游2 000 bp范围内的DNA序列进行启动子同源序列比对。利用在线软件Softberry中的BPROM程序[17]进行启动子预测,再利用Bacterial Promoter Prediction[18]和iPromoter-2L[19]对σ因子进行分类,预测潜在启动子位点和类型。
1.2.7 Halomonas sp.7T部分甲基受体趋化蛋白基因序列验证 挑取Halomonas sp.7T单菌落接种至灭菌2216E液体培养基,35 ℃、120 r/min条件下摇床培养至OD600 nm为0.6~0.8 (12 h以内),取适量菌液离心收集菌体,使用TIANGEN细菌基因组DNA提取试剂盒(Cat.#DP302-02,中国)提取总DNA。
对待验证的MCP基因,首先利用GenScript在线工具Primer Design基于其高通量测序序列设计引物,扩增基因全长(表2),引物由华大基因公司合成。以Halomonas sp.7T总DNA为模板,用设计的特异性引物分别扩增不同MCP基因全长。PCR扩增体系:模板DNA 100 ng,10 μmol/L正、反向引物各0.6 μL,2×高保真DNA聚合酶(Cat.#R045,中国)15 μL,用ddH2O补足至30 μL。PCR扩增程序:95 ℃下预变性2 min;95 ℃下变性30 s,58 ℃下退火30 s,72 ℃下延伸1 min 30 s,共进行36个循环;72 ℃下总体延伸5 min,16 ℃下保温。取1 μL PCR产物进行琼脂糖凝胶电泳检测,根据电泳条带数量、长度和亮度判断PCR扩增是否成功。
表2 Halomonas sp.7T部分MCP基因全长扩增引物信息
Tab.2 Information of primers used to amplify the full length of some MCP genes in Halomonas sp.7T
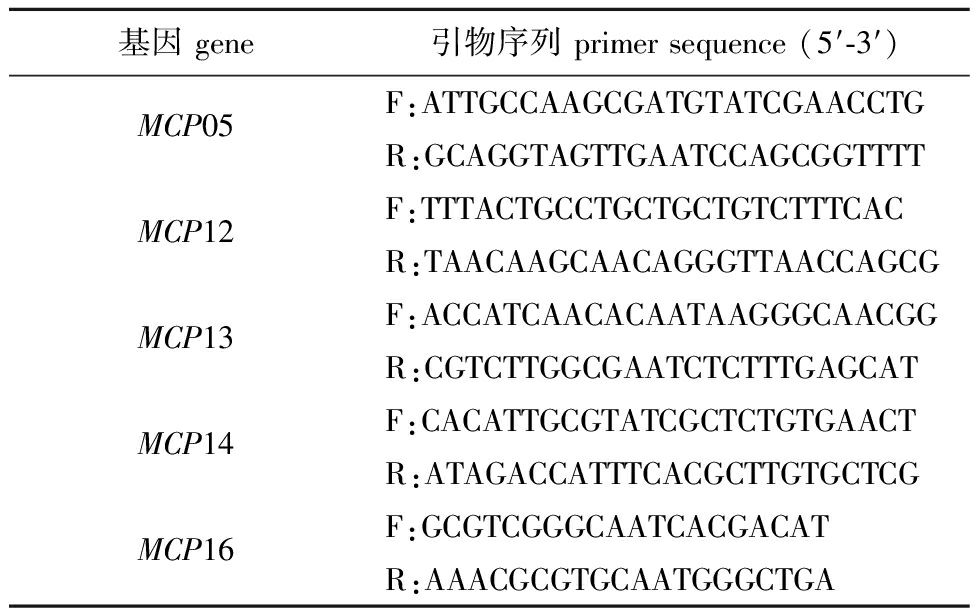
基因 gene引物序列 primer sequence (5'-3')MCP05F:ATTGCCAAGCGATG-TATCGAACCTGR:GCAGGTAGTTGAATCCAGCG-GTTTTMCP12F:TTTACTGCCTGCTGCT-GTCTTTCACR:TAACAAGCAACAGGGTTAAC-CAGCGMCP13F:ACCATCAACACAATA-AGGGCAACGGR:CGTCTTGGCGAATCTCTTT-GAGCATMCP14F:CACATTGCGTATCGCTCTGT-GAACTR:ATAGACCATTTCACGCTTGT-GCTCGMCP16F:GCGTCGGGCAATCACGACATR:AAACGCGTGCAATGGGCTGA
对基因全长扩增成功的PCR产物,直接向反应体系中加入0.2 μL的rTaq DNA聚合酶(5 U/μL),72 ℃下延伸10 min,实现在PCR扩增片段的3′端添加A。然后用AXYGEN AP-GX-250 DNA凝胶回收试剂盒(Cat.# 186,中国)纯化上述PCR产物。使用TaKaRa的TA克隆试剂盒,将纯化后的PCR产物(目的片段)连接到pMD19-T载体上。反应体系及反应条件:pMD19载体 1 μL,PCR 产物1 μL,ddH2O 3 μL,Solution Ⅰ 5 μL;16 ℃下反应30 min。再将TA克隆连接产物通过热击的方法导入大肠杆菌DH5α感受态细胞中,通过蓝白斑筛选白色菌落即阳性转化子,通过菌落PCR验证PCR产物是否插入。将验证成功的白色菌落接种至相应抗性的LB液体培养基,37 ℃、120 r/min条件下摇床培养8~16 h,取适量菌液离心,收集菌体,使用Magen质粒小提试剂盒(P1001-03C)抽提重组质粒并送至华大基因公司测序。测序结果通过CExpress进行校正拼接,再使用SnapGene对一代测序结果和高通量测序结果进行比较分析。
2 结果与分析
2.1 Halomonas sp.7T趋化试验
从图1可见,受试的15种趋化待测物中,呈正趋向性(R>1)的包括亮氨酸和氢氧化钠,呈负趋向性(R<1)的包括异亮氨酸、蔗糖、盐酸,无响应(R=1)的包括葡萄糖、赖氨酸、甲硫氨酸、乙酸、牛磺酸、碳酸钙、酵母粉、氯化钠、磷酸氢二钾和硫酸镁等。对趋化试验数据进行统计分析发现,与对照组存在显著差异的仅有使用亮氨酸作为趋化剂的试验组,表明Halomonas sp. 7T存在趋化行为,亮氨酸是其趋化诱导剂(P<0.05)。
2.2 基于细菌基因组平均核苷酸相似度(ANI)分析的Halomonas sp.7T分类
从图2可见,Halomonas sp.7T基因组(图中用○表示)与其他19种盐单胞菌基因组的ANI值均在69%以上,表明7T确为盐单胞菌属细菌。
基于ANI数值,20种细菌可分为3组,分别是右下角以H.halophila为分界线的第一组、左上角以Halomonas sp.GFAJ-1(图中用①表示)为分界线的第二组及介于二者之间的第三组,组内不同种基因组两两比较的ANI值均在71%以上。Halomonas sp.7T位于第二组,其基因组与该组Halomonas sp.GFAJ-1基因组最为相似,ANI值为84.64%;第二组代表菌株为盐单胞菌属标准菌株H.axialensis(图中用②表示),其基因组与Halomonas sp.7T基因组的ANI值为79.21%;盐单胞菌属模式菌株H.elongata DSM 2581(图中用▲表示)位于第一组,其基因组与Halomonas sp.7T基因组的ANI值仅为70.63%(图2)。这提示,Halomonas sp.7T可能是>盐单胞菌属新发现的一种细菌。
2.3 Halomonas sp.7T趋化信号通路预测
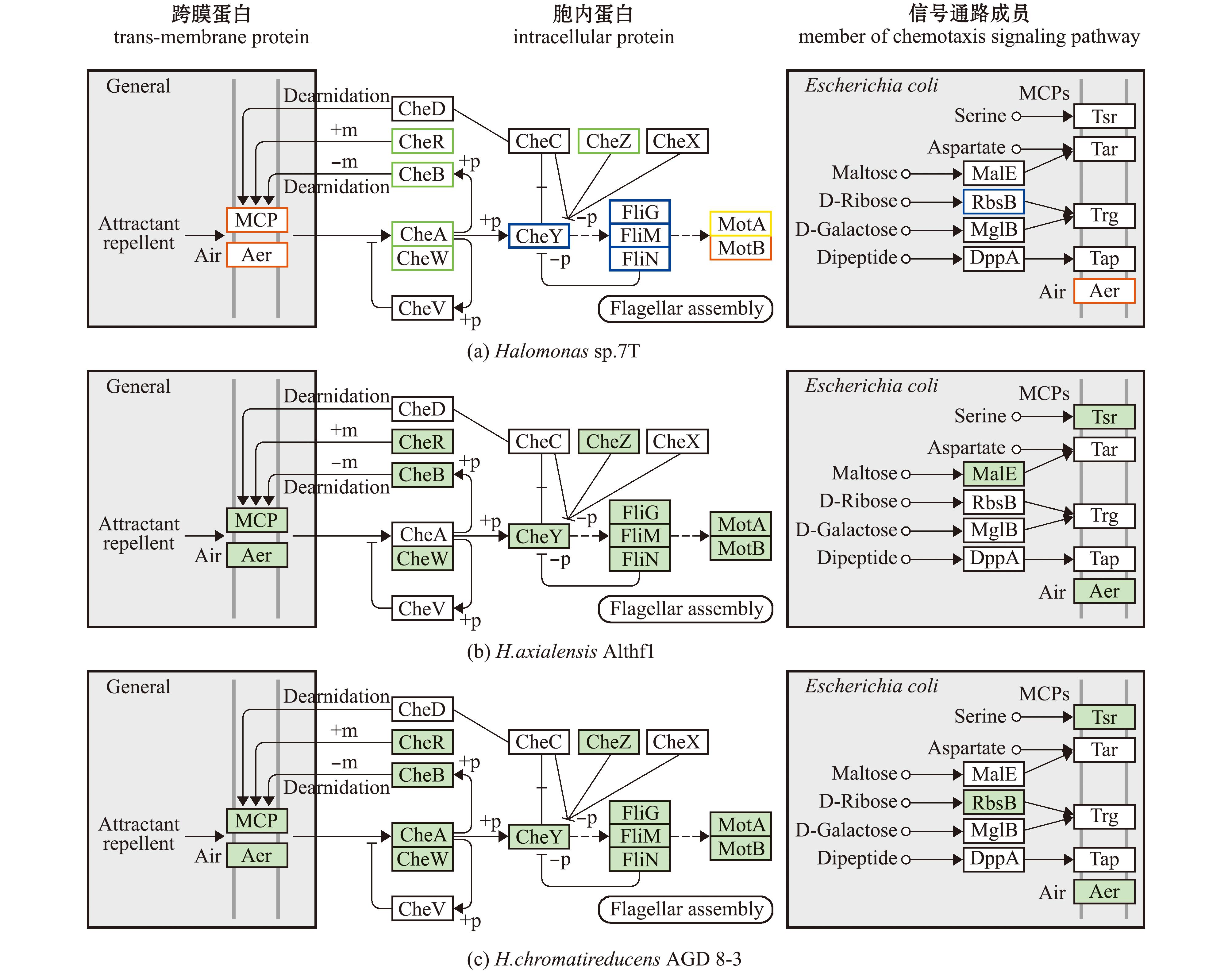
以大肠杆菌为参考,分别通过KEGG Pathway富集分析对Halomonas sp.7T、H.axialensis Althf1和H.chromatireducens AGD 8-3的趋化信号通路进行注释,结果如图3所示。
Halomonas sp.7T趋化信号通路由36个基因组成(图3(a)中用彩色边框标注),分别编码1个底物结合蛋白(RbsB)、9个MCPs、4个趋氧受体(Aer)、2个组氨酸激酶(CheA)、2个MCPs甲基水解酶(CheB)、2个MCPs甲基转移酶(CheR)、2个连接蛋白(CheW)、1个鞭毛运动调节蛋白(CheY)、2个磷蛋白磷酸酶(CheZ)、4个鞭毛马达蛋白(FliG、FliL、FliM、FliN/Y各1个)和7个鞭毛定子(3个MotA、4个MotB)。
H.axialensis Althf1趋化信号通路由26个基因组成(在图3(b)中用绿色填充标注),分别编码1个底物结合蛋白(MalE)、7个MCPs、1个Aer、2个CheB、1个CheR、1个CheW、2个CheY、1个CheZ、4个鞭毛马达蛋白(FliG、FliL、FliM各1个和2个FliN)和6个鞭毛定子(2个MotA、4个MotB)。
H.chromatireducens AGD 8-3趋化信号通路由34个基因组成(在图3(c)中用绿色填充标注),分别编码1个底物结合蛋白rbsB、13个MCPs、2个Aer、1个CheA、3个CheB、3个CheR、1个CheW、1个CheY、1个CheZ、3个鞭毛马达蛋白(FliG、FliL、FliM、FliN各1个)和5个鞭毛定子(2个MotA、3个MotB)。
2.4 基于多序列比对的Halomonas sp.7T甲基受体趋化蛋白系统发育分析
从表3可见,Halomonas sp.7T基因组共有22个基因编码MCPs,涵盖4+1种常见MCPs类别(天冬氨酸感受器受体Tar、丝氨酸感受器受体Tsr、核糖和半乳糖感受器受体Trg、肽感受器受体Tap和趋氧受体Aer),包括KEGG Pathway富集分析预测的13个MCP基因,分别编码3个Tsr、2个Tar、1个Trg、3个Tap、4个Aer,另有9个MCPs未能分类。而H.chromatireducens AGD 8-3的基因组中含16个MCP基因,分别编码3个Tsr、4个Tar、3个Trg、4个Tap、1个Aer和1个未能分类的MCP[20]。
表3 基于Halomonas sp.7T全基因组序列的MCP基因信息
Tab.3 Information of the MCP gene based on the holo-genome data of Halomonas sp.7T
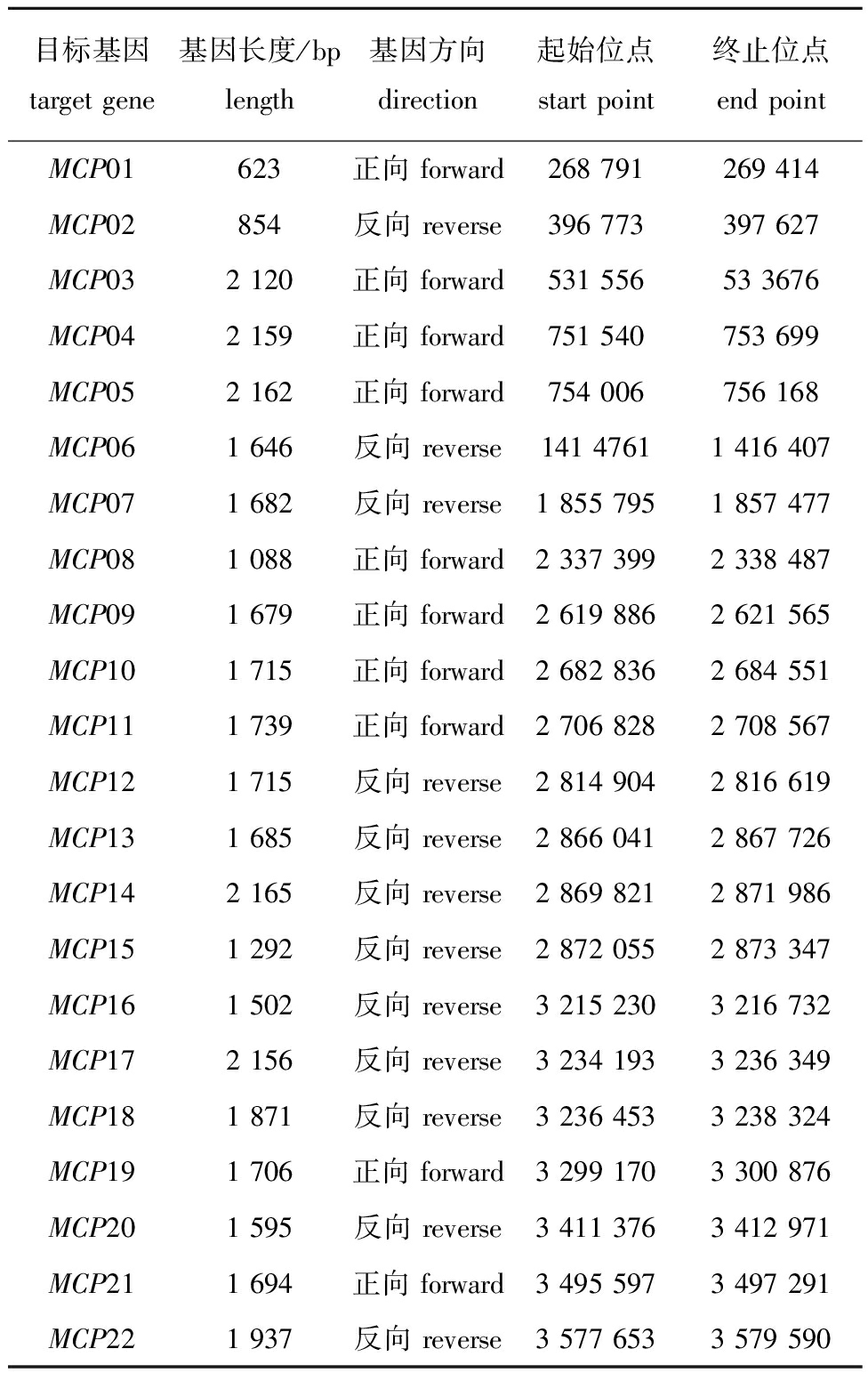
目标基因target gene基因长度/bplength基因方向direction起始位点start point终止位点end pointMCP01623正向 forward268 791269 414MCP02854反向 reverse396 773397 627MCP032 120正向 forward531 55653 3676MCP042 159正向 forward751 540753 699MCP052 162正向 forward754 006756 168MCP061 646反向 reverse141 47611 416 407MCP071 682反向 reverse1 855 7951 857 477MCP081 088正向 forward2 337 3992 338 487MCP091 679正向 forward2 619 8862 621 565MCP101 715正向 forward2 682 8362 684 551MCP111 739正向 forward2 706 8282 708 567MCP121 715反向 reverse2 814 9042 816 619MCP131 685反向 reverse2 866 0412 867 726MCP142 165反向 reverse2 869 8212 871 986MCP151 292反向 reverse2 872 0552 873 347MCP161 502反向 reverse3 215 2303 216 732MCP172 156反向 reverse3 234 1933 236 349MCP181 871反向 reverse3 236 4533 238 324MCP191 706正向 forward3 299 1703 300 876MCP201 595反向 reverse3 411 3763 412 971MCP211 694正向 forward3 495 5973 497 291MCP221 937反向 reverse3 577 6533 579 590
为了解Halomonas sp.7T是否具备功能完整的MCPs,选择H.chromatireducens AGD 8-3的MCPs作为参考,将7T趋化信号通路中的MCPs与AGD 8-3的MCPs进行多序列比对和系统发育分析。从图4可见,Halomonas sp.7T的MCP04、MCP05、MCP11和MCP14与AGD 8-3的Aer氨基酸序列发生聚类,该结果与KEGG Pathway分析结果一致,说明这4个MCPs与趋氧受体Aer同源,可能与Halomonas sp.7T的趋氧能力相关。除此之外,自展值在90以上的还有Tsr组的MCP12、MCP15,Tar组的MCP09、MCP13;Trg组的MCP03,Tap组的MCP10、MCP16和MCP20。MCPs的4组均被覆盖,据此推测,盐单胞菌7T拥有完整的趋化感受器类别。
从图4还可见,这22个MCPs中有11个能与H.chromatireducens AGD 8-3的MCPs单独聚成一个分支,其中有4个Tsr、2个Tar、1个Trg、3个Tap和1个Aer,除MCPⅠ中Tsr 2的自展值为73以外,其余自展值均大于90。据此推测,Halomonas sp.7T具备全部类型的趋化信号受体。
2.5 Halomonas sp.7T甲基受体趋化蛋白结构域分布特征
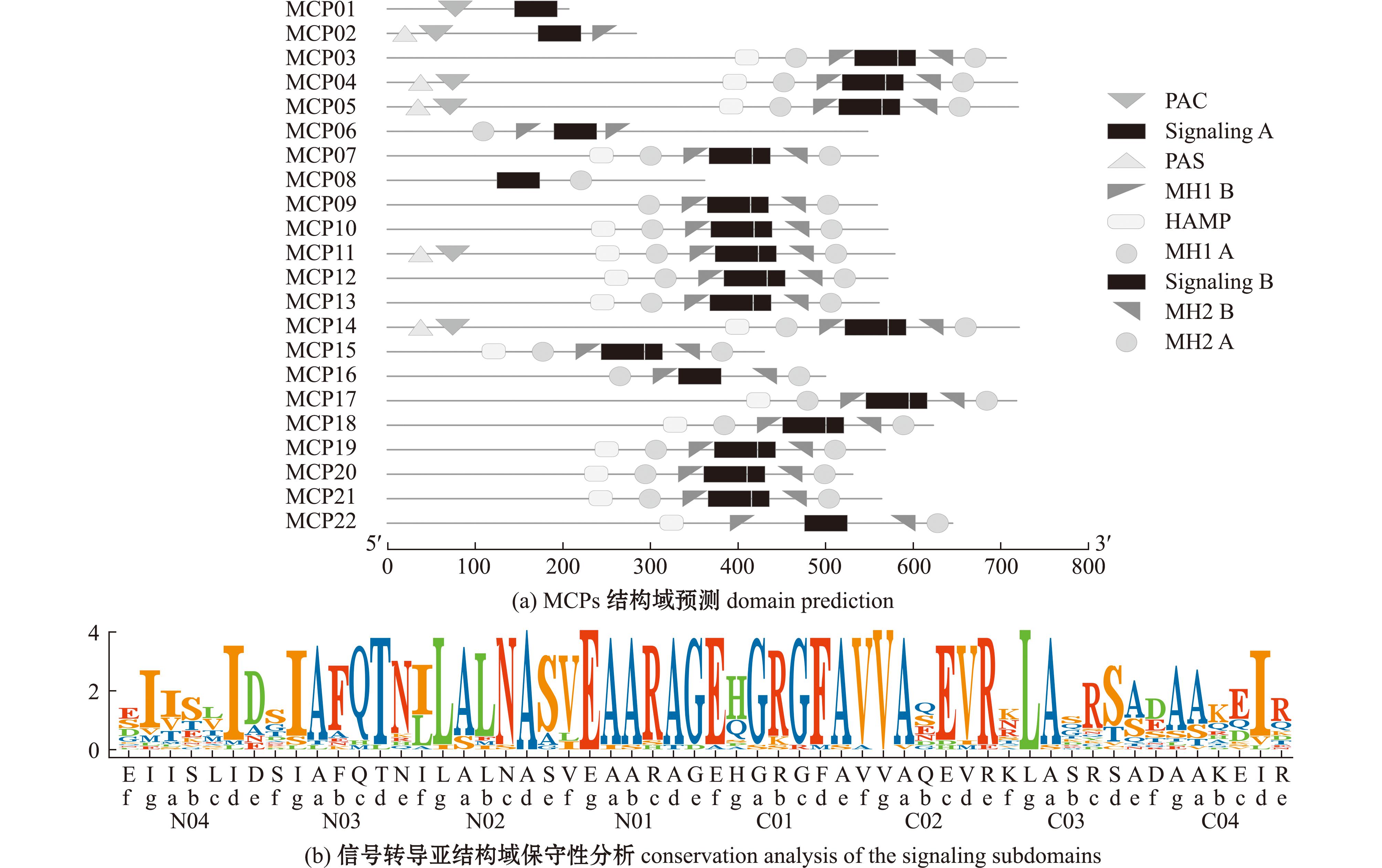
从图5(a)可见,22个MCPs中有6个含有PAS/PAC结构域,分别是MCP01、MCP02、MCP04、MCP05、MCP11和MCP14。22个MCPs均成功预测出信号转导结构域,基于信号转导结构域内七肽单位保守序列和存在的对称插入缺失进行分类,其中17个MCPs属于36H(MCP03、MCP04、MCP05、MCP07、MCP09、MCP10、MCP11、MCP12、MCP13、MCP14、MCP15、MCP16、MCP17、MCP18、MCP19、MCP20和MCP21)、1个属于40H(MCP06),另有4个未能匹配出分类结果,分别是MCP01、MCP02、MCP08和MCP22。MCPs信号转导结构域8个七肽单位的保守性分析显示:在MCPs二聚体间相互作用保守位点N03b,属于36H的17个MCPs为苯丙氨酸(F)残基,而属于40H的MCP06为谷氨酸(E)残基;在MCPs二聚体内相互作用保守位点C02d,属于36H和40H的MCPs均为缬氨酸(V)残基(图5(b))。
Halomonas sp.7T的22个MCPs跨膜结构分析显示,除MCP01、MCP02和MCP08外,其余19个MCPs均含有跨膜结构域,其中,含有1个跨膜结构域的MCPs有2个(MCP09、MCP22),含有2个跨膜结构域的MCPs有16个(MCP03、MCP04、MCP05、MCP06、MCP07、MCP10、MCP11、MCP12、MCP13、MCP14、MCP15、MCP17、MCP18、MCP19、MCP20和MCP21),含有5个跨膜结构域的MCPs有1个(MCP16)。结合图5(a)分析,这些跨膜结构域均位于HAMP(histidine kinases,adenylate cyclases and methyl-accepting chemotaxis proteins)连接结构域前面,与甲基受体趋化蛋白的典型结构特征相符[21]。
对 Halomonas sp.7T的22个MCPs进行跨膜结构分布特征分析,结果表明,有20个MCPs符合拓扑结构,其中,属于ClassⅠ型的MCPs有10个,分别是MCP03、MCP07、MCP10、MCP12、MCP13、MCP17、MCP18、MCP19、MCP20和MCP21;属于Class Ⅱ型的MCPs有4个,分别是MCP04、MCP05、MCP11和MCP14;属于Class Ⅲ型的MCPs有3个,包括含有2个相连跨膜结构域的MCP06、MCP15和含有4个相连跨膜结构域的MCP16;属于Class Ⅳ型的MCPs有3个,分别是MCP01、MCP02和MCP08。另有2个MCPs不符合拓扑结构,分别是MCP09和MCP22。
2.6 Halomonas sp.7T的MCP基因启动子预测
22个MCP基因中有9个使用σ70型启动子(MCP02、MCP03、MCP04、MCP05、MCP11、MCP13、MCP16、MCP17和MCP19),占比为40.9%;有2个基因使用σ24型启动子(MCP10、MCP21),占比9.1%;各有1个基因使用σ32型启动子(MCP12)和σ38型启动子(MCP20),占比均为4.5%;另有9个基因的启动子尚不能明确分型(MCP01、MCP06、MCP07、MCP08、MCP09、MCP14、MCP15、MCP18和MCP22),占比为40.9%。
2.7 Halomonas sp.7T部分MCP基因序列验证
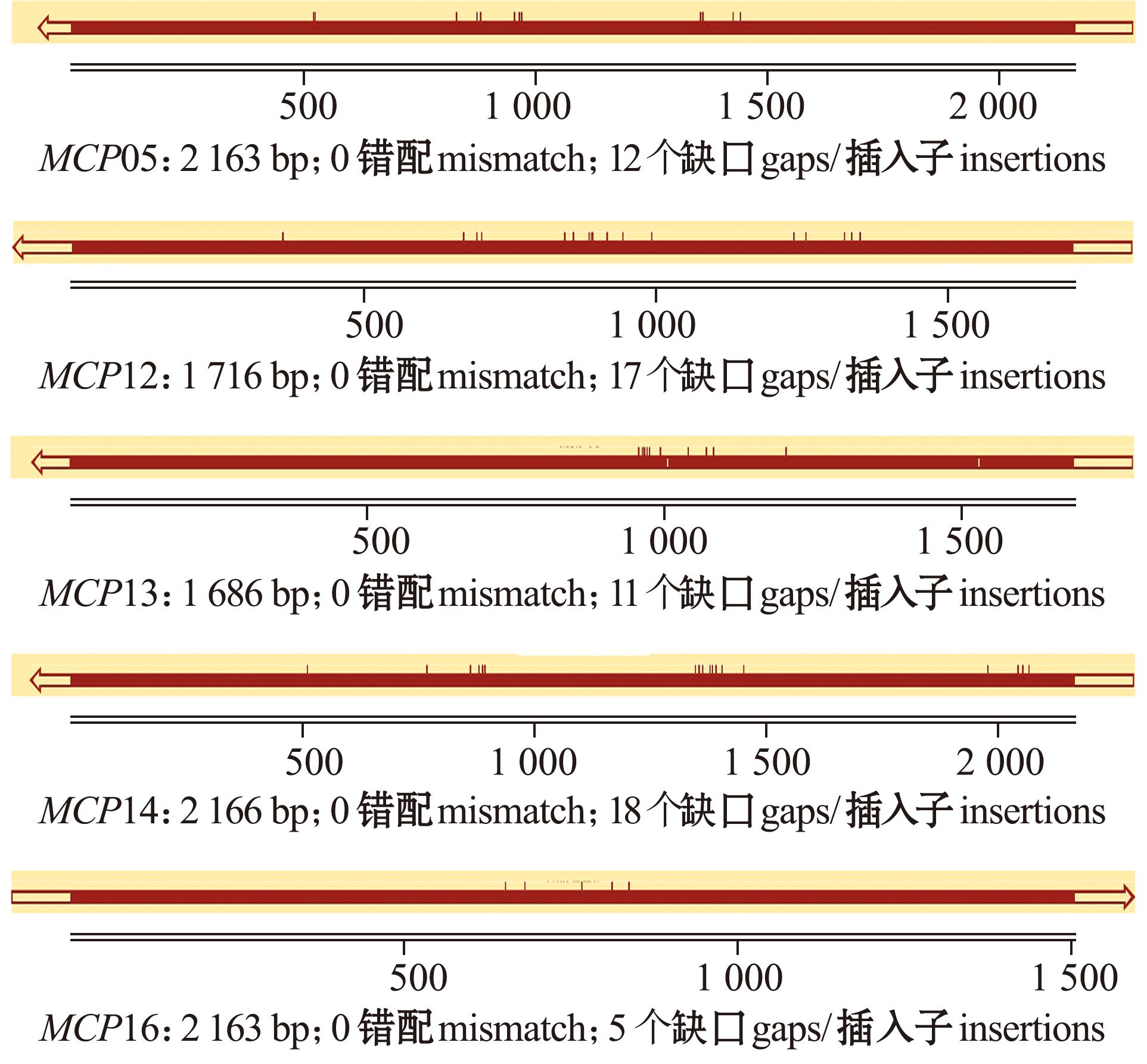
为验证Halomonas sp.7T基因组数据的准确性,从22个MCP基因中随机选择5个基因(MCP05、MCP12、MCP13、MCP14、MCP16)进行验证。基于高通量测序数据分别设计引物扩增这5个基因的全长并进行Sanger法测序,再将一代测序结果与高通量测序结果进行比对分析(图6)。对于验证的5个MCP基因,其一代测序结果与高通量测序结果均高度匹配,错配率均为0,存在少量插入缺失,但频率均低于10个/kb。此外,对这5个基因的一代测序结果进行BLASTx搜索,结果显示,其编码产物均为细菌甲基受体趋化蛋白(图略)。基因序列验证结果表明,基于Halomonas sp.7T基因组高通量测序结果进行的趋化因子受体基因预测是准确的。
3 讨论
3.1 Halomonas sp.7T可能是盐单胞菌属的一个新种
平均核苷酸相似度(ANI)用于比较不同原核生物的遗传相关性,并用95%的ANI数值作为物种界线来定义不同种,高于此数值的物种即认为属于相同种[22]。本研究中,通过ANI计算发现,盐单胞菌7T基因组与盐单胞菌属其他种的基因组一致性为69%~85%,由此推测,Halomonas sp.7T是盐单胞菌属的一个新种[23]。本研究中所使用Halomonas sp.7T基因组数据的准确性,可由部分MCP基因验证试验结果加以证明。
3.2 Halomoans sp.7T的趋化性和完整趋化信号通路
趋化性是生物应对环境变化时趋利避害的生存策略。许多致病性细菌都被证实具备趋化性,趋化性与其毒力的发挥密切相关。本研究中,通过毛细管法研究了长牡蛎致病性Halomonas sp.7T的趋化行为,发现其具备趋化性,亮氨酸为趋化诱导物。
本研究中,对Halomonas sp.7T基因组进行KEGG信号传导通路分析发现,该基因组具备完整的趋化信号通路,编码感受环境变化的受体蛋白,调节、反馈环境信息的信号传递蛋白,以及最终控制运动的鞭毛蛋白。Halomonas sp.7T基因组中胞内编码6种趋化调节蛋白(CheA、CheB、CheR、CheW、CheY和CheZ)的基因和编码5种运动调节蛋白(FliG、FliM、FliN、MotA和MotB)的基因均与大肠杆菌相关基因成功匹配,提示Halomonas sp.7T具备完整的调节自身趋化运动的基因。然而,Halomonas sp.7T基因组中的趋化信号通路与大肠杆菌趋化信号通路不能完全匹配,相似的情况在盐单胞菌属的H.axialensis Althf1和H.chromatireducens AGD 8-3中同样存在。
3.3 Halomonas sp.7T 的MCPs功能和结构特征
细菌生存环境的不同决定了其趋化信号通路的差异,即使是同属不同种的细菌,其趋化信号通路也不尽相同,而不同种细菌的MCPs种类差异尤为明显。本研究中,Halomonas sp.7T基因组中共有22个MCP基因,编码产物涵盖常见的5种MCPs类型。基于大肠杆菌趋化信号通路的映射分析表明,Halomonas sp.7T的5种MCP基因仅Aer与大肠杆菌完全匹配,其余4种MCP基因均未能预测成功。分别将盐单胞菌属标准菌株H.axialensis Althf1和参考菌株H.chromatireducens AGD8-3基因组映射到大肠杆菌趋化信号通路发现,MCP基因的种类同样出现缺失。另据报道,H.anticariensis FP35T有21个MCP基因,仅有部分能映射到大肠杆菌趋化信号通路[24]。本研究结果与此结果一致。
在细菌中,甲基受体趋化蛋白包含1个感受器结构域即配基结合结构域(ligand binding domain,LBD)、2个跨膜螺旋、1个HAMP连接结构域、1个甲基化结构域(含2个甲基化区域MH1、MH2)和1个信号转导结构域[21,25-26]。在趋化过程中,细菌通过MCPs的配基结合结构域感知环境中的不同信号,通过与胞内CheW和CheA蛋白相连的甲基化结构域和信号转导结构域把环境信号传递到胞内。其中,甲基化结构域的氨基酸组成较为保守,被甲基化时产生生化信号。根据配基结合结构域和跨膜结构的拓扑学性质,MCPs被分为Class Ⅰ、Class Ⅱ、Class Ⅲ和Class Ⅳ 4大类型[11]。属于Class Ⅰ型的MCPs含有1个周质配基结合结构域、1个跨膜结构域和1个胞内信号结构域;属于Class Ⅱ型的MCPs在N-端含有1个胞质内配基结合结构域,之后是2个连续跨膜结构域、1个胞质内HAMP区域和1个胞质内信号区域;属于Class Ⅲ型的MCPs跨膜结构域从1~8个不等,感受器或位于细胞膜,或位于最后一个跨膜螺旋之后、细胞质内,同时含有1个胞质内HAMP区域和1个胞质内信号区域;属于Class Ⅳ型的MCPs为胞质内可溶性蛋白,不含跨膜结构域,含有或不含显著配基结合结构域,多存在于古菌。目前,已发现的甲基受体趋化蛋白多属于Class Ⅰ型,比如大肠杆菌的Tar、Tsr、Trg及Tap[25]。大肠杆菌中的趋氧受体Aer属于Class Ⅱ型MCPs[27]。而本研究中,Halomonas sp.7T的4个趋氧受体Aer跨膜结构分析也显示,其属于Class Ⅱ型MCPs。针对Halomonas sp.7T甲基受体趋化蛋白的跨膜结构分析结果与上述研究结果一致。值得注意的是,本研究中有2个MCPs因不符合拓扑学性质,未能归类。
此外,本研究中结构域分析结果显示,Halomonas sp.7T的22个MCPs中6个含有胞外感受器PAS/PAC结构域,包括4个趋氧因子受体。PAS/PAC结构域位于甲基受体趋化蛋白的N端,属于胞外感受器,可通过与小分子辅基结合感受周围化学信号,包括氧气、光、氧化还原电位等能量变化[28]。这4个MCPs的KEGG Pathway分析、系统发育分析、蛋白结构分析结果均一致,提示其可能作为趋氧受体参与Halomonas sp.7T的趋氧运动。
在大多数细菌中,MCPs的信号传导结构域都是由多个七肽单位组成的对称插入缺失对,并形成两股α-螺旋,根据信号传导结构域保守序列和存在的对称插入缺失,可将细菌甲基受体趋化蛋白分为7类,分别是24H、28H、34H、36H、38H、40H和44H[29]。此外,信号转导结构域包含两类保守作用位点,通常位于N04~C04,分别是参与二聚体内部相互作用的adeg位点和二聚体间相互作用的bcf位点[29]。本研究中,Halomonas sp.7T的22个MCPs均含有信号转导结构域,其中17个为36H型,1个为40H型,另有4个未能分类。针对Halomonas sp.7T甲基受体趋化蛋白的信号传导结构域分析结果与上述研究结果也一致。
3.4 Halomonas sp.7T的MCP基因启动子类型
本研究中,Halomonas sp.7T的MCP基因启动子包括组成型和胁迫诱导型,提示其编码的MCPs介导的趋化行为可能与细菌的生存和响应环境胁迫有关。细菌基因的启动子位于转录起点上游,包含两个高度保守区域,即-35区和-10区(pribnow box),为RNA聚合酶结合位点,参与转录起始[30]。RNA聚合酶由核心酶和σ因子组成,其中σ因子负责识别待转录基因的启动子并与之结合。细菌的σ因子有多种类型,分别识别不同基因的启动子。大肠杆菌有7种不同类型的σ因子,其中σ70负责持家基因的转录,σ54负责氮代谢调控基因的转录,σ38负责稳定期基因的转录,σ32负责热休克基因转录,σ28负责鞭毛合成基因的转录,σ24负责极端高温下相关基因的转录,σ19负责调控铁离子转运基因的转录[31]。据报道,与细菌趋化运动相关基因的表达由σ28和σ54共同调节[32]。本研究中,Halomonas sp.7T的22个MCP基因中,有9个基因的启动子为σ70型,表明启动子结合位点相对保守,为持家基因型表达;另有2个基因的启动子为σ24型,2个基因的启动子分别为σ32型和σ38型,这3种类型的启动子均与胁迫相关,推测其可能参与Halomonas sp.7T对环境胁迫的响应。
4 结论
1)通过毛细管法证实长牡蛎致病性盐单胞菌Halomonas sp.7T具备趋化性,亮氨酸是其趋化诱导剂。
2)基于全基因组数据挖掘出Halomonas sp.7T的完整趋化信号通路,发现了大量编码甲基受体趋化蛋白的基因,其编码蛋白涵盖5种常见趋化因子受体。
3)Halomonas sp.7T的MCP基因启动子既有组成型又有胁迫诱导型,表明其参与介导的趋化行为可能与细菌生存及响应环境胁迫相关。
参考文献:
[1] KING W L,JENKINS C,SEYMOUR J R,et al.Oyster disease in a changing environment:decrypting the link between pathogen,microbiome and environment[J].Marine Environmental Research,2019,143:124-140.
[2] KIM K K,LEE J S,STEVENS D A.Microbiology and epidemiology of Halomonas species[J].Future Microbiology,2013,8(12):1559-1573.
[3] YE J W,CHEN G Q.Halomonas as a chassis[J].Essays in Biochemistry,2021,65(2):393-403.
[4] 曾繁锦.三相单室微生物燃料电池高盐下产电与脱氮耦合研究[D].大连:大连海事大学,2020. ZENG F J.Coupling of electricity generation and denitrification in three-phase single-chamber mfcs in high-salt conditions[D].Dalian:Dalian Maritime University,2020.(in Chinese)
[5] GASPEROTTI A F,REVUELTA M V,STUDDERT C A,et al.Identification of two different chemosensory pathways in representatives of the genus Halomonas[J].BMC Genomics,2018,19(1):266.
[6] WANG L P,SHAO Z Z.Aerobic denitrification and heterotrophic sulfur oxidation in the genus Halomonas revealed by six novel species characterizations and genome-based analysis[J].Frontiers in Microbiology,2021,12:652766.
[7] JADHAV K,KUSHWAHA B,JADHAV I,et al.Genomic analysis of a novel species Halomonas shambharensis isolated from hypersaline lake in Northwest India[J].Molecular Biology Reports,2021,48(2):1045-1053.
[8] 杨姗姗,马丽,孙柏欣,等.细菌趋化性研究进展[J].中国农学通报,2015,31(6):121-127. YANG S S,MA L,SUN B X,et al.Progress in bacterial chemotaxis[J].Chinese Agricultural Science Bulletin,2015,31(6):121-127.(in Chinese)
[9] KRUKONIS E S,DIRITA V J.From motility to virulence:sensing and responding to environmental signals in Vibrio cholerae[J].Current Opinion in Microbiology,2003,6(2):186-190.
[10] KORT E N,GOY M F,LARSEN S H,et al.Methylation of a membrane protein involved in bacterial chemotaxis[J].Proceedings of the National Academy of Sciences of the United States of America,1975,72(10):3939-3943.
[11] SALAH UD-DIN A I M,ROUJEINIKOVA A.Methyl-accepting chemotaxis proteins:a core sensing element in prokaryotes and Archaea[J].Cellular and Molecular Life Sciences:CMLS,2017,74(18):3293-3303.
[12] 李晨.长牡蛎幼虫养殖环境中一株致病性盐单胞菌的分离鉴定[D].大连:大连海洋大学,2022. LI C.Isolation and identification of a pathogenic strain of Halomonas from the aquaculture environment breeding the Crassostrea gigas larvae[D].Dalian:Dalian Ocean University,2022.(in Chinese)
[13] 刘力铭,钱爱霞,鄢庆枇,等.哈维氏弧菌TS-628菌株趋化性研究[J].渔业科学进展,2010,31(6):104-109. LIU L M,QIAN A X,YAN Q P,et al.Chemotaxis of Vibrio harveyi TS-628[J].Progress in Fishery Sciences,2010,31(6):104-109.(in Chinese)
[14] GORIS J,KONSTANTINIDIS K T,KLAPPENBACH J A,et al.DNA-DNA hybridization values and their relationship to whole-genome sequence similarities[J].International Journal of Systematic and Evolutionary Microbiology,2007,57(Pt 1):81-91.
[15] ALTSCHUL S F,MADDEN T L,SCH FFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
FFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
[16] SONNHAMMER E L,VON HEIJNE G,KROGH A.A Hidden Markov model for predicting transmembrane helices in protein sequences[C]//Proceedings of the 6th international conference on intelligent systems for molecular biology.Menlo Park,California,USA:The AAAI Press,1998:175-182.
[17] VICTOR S,ASAF S.Automatic annotation of microbial genomes and metagenomic sequences[M].NY,USA:Nova Science Publishers,2011:61-78.
[18] DE AVILA E SILVA S,ECHEVERRIGARAY S S,GERHARDT G J L.BacPP:bacterial promoter prediction:a tool for accurate sigma-factor specific assignment in enterobacteria[J].Journal of Theoretical Biology,2011,287:92-99.
[19] LIU B,YANG F,HUANG D S,et al.iPromoter-2L:a two-layer predictor for identifying promoters and their types by multi-window-based PseKNC[J].Bioinformatics,2018,34(1):33-40.
[20] SHARKO F S,SHAPOVALOVA A A,TSYGANKOVA S V,et al.Draft genome sequence of “Halomonas chromatireducens” strain AGD 8-3,a haloalkaliphilic chromate- and selenite-reducing Gammaproteobacterium[J].Genome Announcements,2016,4(2):e00160-e00116.
[21] FALKE J J,BASS R B,BUTLER S L,et al.The two-component signaling pathway of bacterial chemotaxis:a molecular view of signal transduction by receptors,kinases,and adaptation enzymes[J].Annual Review of Cell and Developmental Biology,1997,13:457-512.
[22] ARAHAL D R.Whole-genome analyses[M]//Methods in Microbiology.Amsterdam:Elsevier,2014:103-122.
[23] JAIN C,RODRIGUEZ L M,PHILLIPPY A M,et al.High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries[J].Nature Communications,2018,9(1):5114.
[24] SAMPEDRO I,PÉREZ-MENDOZA D,TORAL L,et al.Effects of halophyte root exudates and their components on chemotaxis,biofilm formation and colonization of the halophilic bacterium Halomonas anticariensis FP35T[J].Microorganisms,2020,8(4):575.
[25] ZHULIN I B.The superfamily of chemotaxis transducers:from physiology to genomics and back[J].Advances in Microbial Physiology,2001,45:157-198.
[26] WATTS K J,MA Q H,JOHNSON M S,et al.Interactions between the PAS and HAMP domains of the Escherichia coli aerotaxis receptor Aer[J].Journal of Bacteriology,2004,186(21):7440-7449.
[27] BIBIKOV S I,BIRAN R,RUDD K E,et al.A signal transducer for aerotaxis in Escherichia coli[J].Journal of Bacteriology,1997,179(12):4075-4079.
[28] TAYLOR B L,ZHULIN I B.PAS domains:internal sensors of oxygen,redox potential,and light[J].Microbiology and Molecular Biology Reviews,1999,63(2):479-506.
[29] ALEXANDER R P,ZHULIN I B.Evolutionary genomics reveals conserved structural determinants of signaling and adaptation in microbial chemoreceptors[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(8):2885-2890.
[30] PERRON G G,WHYTE L,TURNBAUGH P J,et al.Functional characterization of bacteria isolated from ancient Arctic soil exposes diverse resistance mechanisms to modern antibiotics[J].PLoS One,2015,10(3):e0069533.
[31] BARRIOS H,VALDERRAMA B,MORETT E.Compilation and analysis of sigma(54)-dependent promoter sequences[J].Nucleic Acids Research,1999,27(22):4305-4313.
[32] SHAH D S,PORTER S L,MARTIN A C,et al.Fine tuning bacterial chemotaxis:analysis of Rhodobacter sphaeroides behaviour under aerobic and anaerobic conditions by mutation of the major chemotaxis operons and cheY genes[J].The EMBO Journal,2000,19(17):4601-4613.
Digging the chemokine receptors-encoding genes and analyzing the relative protein sequences of a Halomonas strain
XU Shuang1,LI Chen1,JIANG Jialin1,XIE Xiaochen1,WANG Bo1,HUO Zhongming2,FANG Lei1*
(1.College of Marine Science and Environment,Dalian Ocean University,Dalian 116023,China;2.College of Fisheries and Life Science,Dalian Ocean University,Dalian 116023,China)
Abstract: With an attempt to understand the chemotaxis of Halomonas sp.7T, a pathogenic bacterium to Pacific oyster (Crassostrea gigas), its chemotaxis was investigated by capillary tube method and the holo-genome sequencing data were utilized to analyze its chemosensory signaling pathway. Genes involved in chemotaxis were predicted and the chemosensory pathway of 7T was analyzed using KEGG Pathway. The chemokine receptors-encoding genes were dug out and sequences of the relative proteins were analyzed. The chemokine receptors were further grouped via aligning and comparing with the homologues, and structures of MCPs (methyl-accepting chemotaxis proteins) were analyzed through incorporating MeMe database and TMHMM analysis. The results showed that chemotaxis was observed in Halomonas sp.7T. Across its genome, complete signaling pathways existed and there were 22 MCPs-encoding genes, covering the 5 kinds of most conventioal MCPs. Signal transduction domains were predicted in the 22 MCPs. Additionally, 19 MCPs harbored the trans-membrane domain whereas 3 did not. Based on the differences in the heptapeptide repetitive sequences, the 22 MCPs were further classified into different groups. Seventeen MCPs belonged to the group of 36H and 1 MCP was in the groups of 40H, whereas the other 4 MCPs were not categorized into any known group. Additionally, 20 of the 22 MCPs harbored trans-membrane domains conforming to topology, among which 10 were grouped to Class Ⅰ, 4 to Class Ⅱ, 3 to Class Ⅲ, and 3 to Class Ⅳ. The results showed that chemotaxis was observed in Halomonas sp.7T, a bacterial strain pathogenic to Crossostrea gigas. Complete chemosensory signaling pathways existed in its genome. In total, 22 genes encoded methyl-accepting chemotaxis proteins, which were of specific structural characteristics and might be involved in mediating chemotactic behaviours crucial to the existence as well as responses to environmental stresses of the bacterial strain.
Key words: Halomonas; chemotaxis; chemokine receptor-encoding gene; methyl-accepting chemotaxis proteins (MCPs); Crassostrea gigas





 FFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
FFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.