低眼巨鲶(Pangasianodon hypophthalmus)隶属于鲶形目(Siluriformes)鱼芒鲶科(Pangasiidae)鱼芒鲶属(Pangasianodon),俗称“巴沙鱼”(Basa ),20世纪90年代引进到中国[1],是中国华南地区常见养殖鱼类。低眼巨鲶生长性能优越,蛋白质含量高,常被用作酸菜鱼、冷冻鱼片和预制菜的原材料[2]。低眼巨鲶富含维生素D,也适用于鱼油营养产品的制作[3]。此外,低眼巨鲶也是一种优良的鱼油提取源,具有良好的热氧化稳定性,可作为普通棕榈油的有效替代品[4]。近年来,因其在水产养殖、健康营养品和食品加工等方面的显著经济价值,中国对低眼巨鲶需求日益增长,目前进口量已超过30万t,位列全球之首[5]。以低眼巨鲶为代表的巴沙鱼产业在中国不断壮大,已经形成了个性化产业链[6]。
然而,中国低眼巨鲶养殖业仍处于初级阶段,目前,尽管已经初步掌握了苗种培育技术[7-8],并在海南等地进行了小规模的苗种生产和养殖,但尚未形成规模化发展局面。低眼巨鲶种鱼主要从越南引进,其苗种质量参差不齐,存在种质混乱的问题,这也成为制约低眼巨鲶养殖产业发展原因之一。因此,为解决当前低眼巨鲶市场上种质混乱的问题,需要对其养殖群体开展准确的资源评估,深入研究低眼巨鲶养殖群体的遗传背景,这对于提升中国低眼巨鲶种质资源保育和管理具有重要意义。
微卫星技术(simple sequence repeat,SSR)[9]常用于分析鱼类群体遗传多样性。微卫星等位基因间呈现共显性遗传,具有高度多态性、分布广泛且易于扩增的特点,在分子生物学领域中逐渐被广大研究者所关注。这一分子标记技术广泛应用于鱼[10]、虾[11]和蟹[12]等水生生物群体遗传多样性相关研究,是研究者深入了解各种生物群体遗传结构的强有力工具。从20世纪起,许多常见鱼类的相关研究中已经开始使用微卫星技术[13-15],目前微卫星技术已广泛应用于鲶形目鱼类的群体遗传多样性研究[16-18],为相关鱼类群体遗传多样性研究方面提供了丰富的参考数据。
本研究中选用东南地区4处低眼巨鲶养殖群体作为试验对象,通过10个微卫星分子标记对其进行遗传变异分析,研究了低眼巨鲶养殖群体的遗传多样性及遗传结构。旨在筛选出优质种质材料,理清其间亲缘关系,以期为探明低眼巨鲶养殖群体的遗传背景提供有益参考,也为低眼巨鲶养殖种质资源的保育提供基础数据。
1 材料与方法
1.1 材料
本试验从百荣水产良种有限公司(广东省湛江市)、翔泰渔业股份有限公司(海南省文昌市)、佛山三水白金水产种苗有限公司 (广东省佛山市)和广东罗非鱼良种场(广东省广州市)4处低眼巨鲶养殖场采集商品鱼规格的低眼巨鲶,采集鱼鳍保存在无水乙醇中。最终有效样本113 尾,将4个区域分别标记为广东湛江YJ(n=30)、海南文昌HN (n=30)、 广东佛山GYQB (n=26)和广东广州GYZJ (n=27)(下同)。
1.2 方法
1.2.1 DNA提取 利用MicroElute Genomic DNA Kit(Omega Biotek,Inc.Norcross,Georgia,USA)试剂盒提取样品DNA。通过琼脂糖凝胶电泳检测DNA的纯度及完整性。通过 NanoQTM微型分光光度计检测DNA浓度。
1.2.2 微卫星引物设计及筛选 参考本实验室引物设计及筛选方法[19-20],通过低眼巨鲶转录组数据得到SSR位点40 000多个,针对3、4核苷酸重复引物序列进行引物设计选取得到低眼巨鲶微卫星位点1 920 对SSR引物,并进一步筛选,从中得到96 对引物用于下一步筛选。引物采用接头法合成。
从每个群体中各选取8个样本,用引物扩增样本,反应在Veriti 384 PCR仪上进行。PCR反应体系为10 μL,包括:2×Taq PCR Master Mix 7.5 μL,上下游引物混合物2 μL (10 μmol/L),基因组DNA 1 μL (50~200 ng),去离子水4.5 μL。PCR 扩增程序为96 ℃下预变性3 min,96 ℃下变性30 s,最适温度下退火30 s,72 ℃下延伸1 min,共进行30 个循环,最后在72 ℃下再延伸10 min,在12 ℃下保存。PCR反应结束后,扩增产物经荧光毛细管电泳检测。采用GeneMarkerV2.20软件对结果进行分析,得到10对PIC值在0.59以上,等位基因数大于3的引物用于群体分型。
1.2.3 PCR扩增与检测 在筛选出10 对引物后,对收集样本进行PCR扩增,将PCR产物进行琼脂糖凝胶电泳检测,对照DNA Marker 的浓度进行定量。利用DNA 测序仪ABI 3730xl 进行毛细管电泳检测。
1.3 数据处理析
利用GenAlEx 6.501软件[21]计算观测等位基因(Na)、有效等位基因(Ne)、香农指数(I)、多态性信息指数(PIC)、观测杂合度(Ho)、期望杂合度(He)和近交系数(Fis)。利用GenAlEx version 6.501 软件计算各群体间和群体内的变异、分化并进行显著性检验;利用STRUCTURE软件对113份低眼巨鲶样品进行群体结构分析,设置K=1~20,Burn-in周期为10 000,MarkovChain Monte Carlo(MCMC)设为100 000,每个K值运行20次,并利用在线工具STRUCTURE HARVESTER[22] 算出最佳ΔK值(即为最佳群体分层情况)。根据最佳K值结果,利用DISTRUCT 软件将CLUMMP的结果进行可视化[23]。采用Wright[24]的公式计算遗传分化系数(Fst)和基因流(Nm),基因流(Nm)∶Nm=0.25(1- Fst)/Fst。利用GenAIex软件进行主坐标分析(PCoA)[25]。利用powermarker软件计算各群体间的遗传距离。利用UPGMA方法进行聚类分析,并绘制环状聚类图。
2 结果与分析
2.1 微卫星引物筛选及遗传多样性分析
从备选引物库中得到96 对引物,一共筛选出10 对PIC值在0.59以上、等位基因数大于3的引物用于后续分析(表1)。10 对引物对113个样本进行扩增,共检测出等位基因74个(Na),其中,最小等位基因数目为4,最大等位基因数目为13,平均每个位点等位基因数目为7.4。有效等位基因(Ne)总数为43.225,范围为2.247(Pab013)~8.557(Pab063),平均每个位点有效等位基因数目为4.322。香农指数(I)为1.025(Pab013)~2.251(Pab063),平均值为1.560。观测杂合度(Ho)为0.482(Pab013)~0.841(Pab040),平均值为0.707。期望杂合度(He)为0.555(Pab013)~0.883(Pab063),平均值为0.741。多态信息含量(PIC)为0.504(Pab013)~0.872(Pab063),平均值为0.702(表2)。
表1 低眼巨鲶微卫星引物序列及扩增信息
Tab.1 Microsatellite primer sequence and amplification information of Pangasianodon hypophthalmus

位点 locus重复单元 repeat motif上游引物 forward primer(5′-3′)下游引物 reverse primer(5′-3′)目的片段 primer lengthPab013(TTG)6CATCAGGGCCTGTGACTTCTGCTCGAGTACCAAACAAGCC217Pab022(AAT)17TCATCTCGACCATCTGACCACACTCCTCCACACACCTTCA205Pab040(AGA)9AAGAGGAAACGAAGGAAGGGACCATCCGTCTCTCATCAGC252Pab055(GAG)8TGGACTGTGAGGTGGATGAAAAAGTCGTCCTGGTCTCCCT239Pab063(TAATA)8AGCAAGCCAGAGGTGACAGTTGCTGAATGCAGTGTGACAG143Pab073(AGGA)8TGGATTAAAGCGGGTTGAAGTCAGTGAGGTCAGGATCACG149Pab084(CAT)11GAGAGCCATCAGAGTCCGAGTTGGTGGAGAGGAACAGGAG213Pab088(TAT)8GCTTTCTGGCAATTCTTGGATTGGTCTGAGATGCATTTGG230Pab095(TGT)9AGCGCTGCTGTATAGGGTGTAGCTTGCACACTGTGAGCTG181Pab096(AAAC)6TGGATCCTCTTTGGGAAGTGGGTGCTTTGTTGAGGCAGTT279
表2 低眼巨鲶10个微卫星位点的多样性指数
Tab.2 Diversity index of Pangasianodon hypophthalmus at 10 microsatellite loci
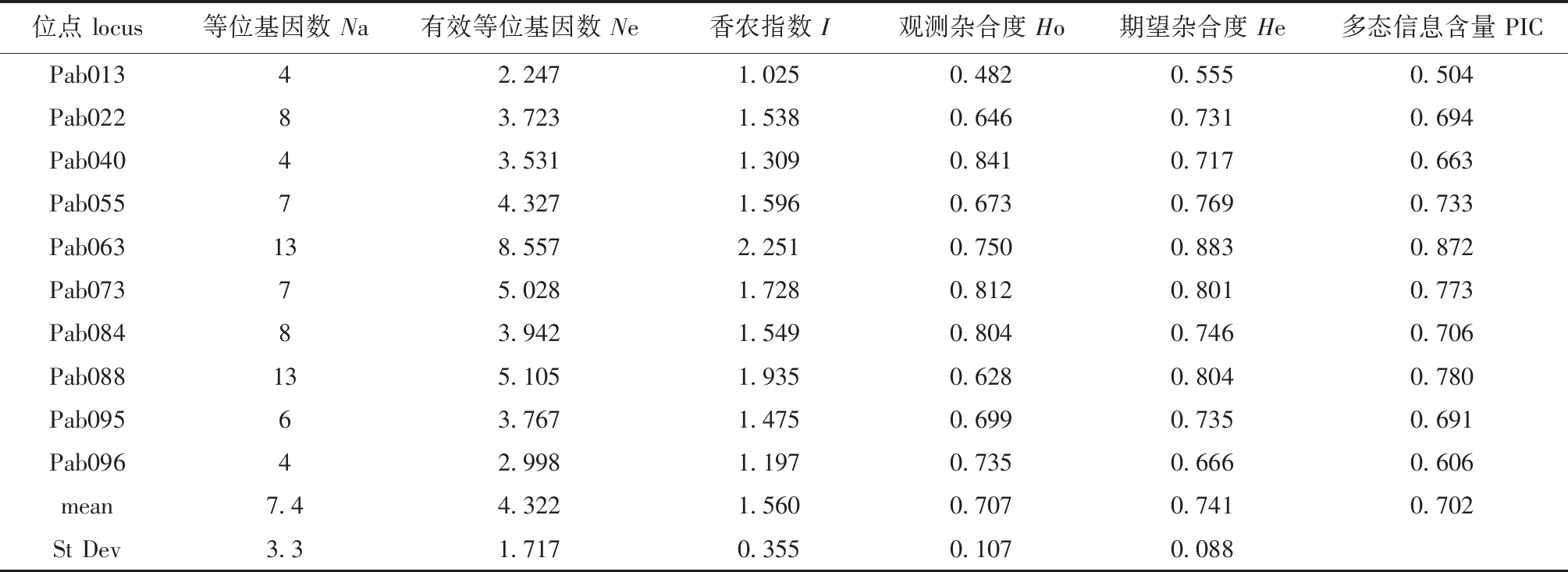
位点 locus等位基因数 Na有效等位基因数 Ne香农指数 I观测杂合度 Ho期望杂合度 He多态信息含量 PICPab01342.2471.0250.4820.5550.504Pab02283.7231.5380.6460.7310.694Pab04043.5311.3090.8410.7170.663Pab05574.3271.5960.6730.7690.733Pab063138.5572.2510.7500.8830.872Pab07375.0281.7280.8120.8010.773Pab08483.9421.5490.8040.7460.706Pab088135.1051.9350.6280.8040.780Pab09563.7671.4750.6990.7350.691Pab09642.9981.1970.7350.6660.606mean7.44.3221.5600.7070.7410.702St Dev3.31.7170.3550.1070.088
用10对引物评估群体的近交系数和基因流,群体内近交系数(Fis)为-0.271(Pab040)~0.008(Pab063)。整体近交系数(Fit)为-0.176(Pab040)~0.207(Pab088),平均值为0.040。遗传分化系数(Fst)为0.071(Pab073)~0.276(Pab088),平均值为0.023。基因流(Nm)为0.655(Pab088)~3.261(Pab073),平均值为0.305(表3)。
表3 10对引物评估群体的近交系数和基因流
Tab.3 Inbreeding coefficients and gene flow in populations evaluated with 10 primers

位点locus群体内近交系数Fis整体近交系数Fit遗传分化系数Fst基因流NmPab013-0.0750.1230.1851.104Pab022-0.0330.1160.1441.485Pab040-0.271-0.1760.0753.096Pab055-0.1890.1120.2530.739Pab0630.0080.1490.1421.505Pab073-0.089-0.0120.0713.261Pab084-0.168-0.0770.0782.973Pab088-0.0960.2070.2760.655Pab095-0.0570.0540.1052.123Pab096-0.241-0.1070.1082.074mean-0.1210.0390.1441.901SE0.0290.0400.0230.305
2.2 低眼巨鲶养殖群体遗传多样性分析
10个微卫星位点在广东佛山、广东广州、海南文昌、广东湛江4个养殖群体中等位基因数(Na)分别为6.3、5.5、3.9、4.3,均值为5.0;有效等位基因数(Ne)分别为3.790、3.716、2.675、2.922,均值为3.276 ;香农指数(I)分别为1.431、1.393、0.981、1.149,均值为1.239;平均观测杂合度(Ho)分别为0.742、0.778、0.613、0.706,均值为0.710;平均期望杂合度(He)分别为0.698、0.690、0.530、0.619,均值为0.634(表4)。
表4 4个养殖群体在10个微卫星位点的遗传参数
Tab.4 Genetic parameters of 4 cultured populations of Pangasianodon hypophthalmus at 10 microsatellite loci
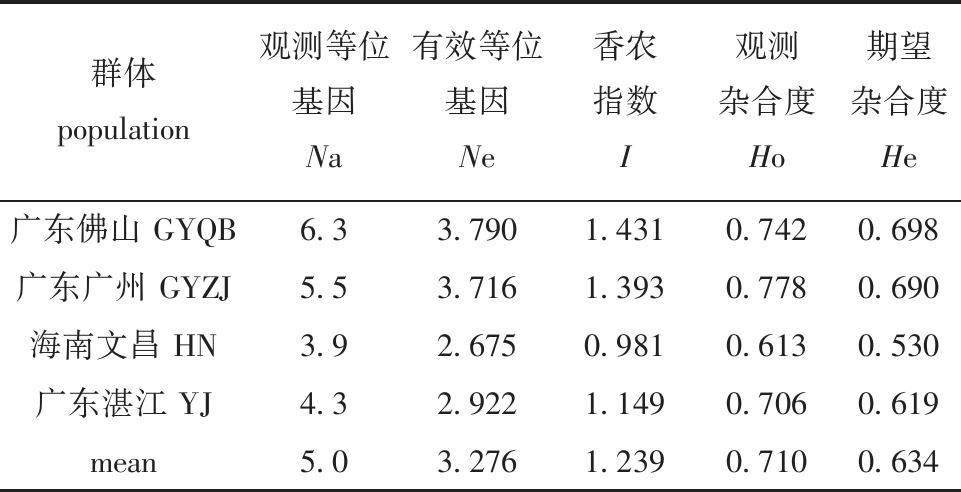
群体population观测等位基因Na有效等位基因Ne香农指数I观测杂合度Ho期望杂合度He广东佛山 GYQB6.33.7901.4310.7420.698广东广州 GYZJ5.53.7161.3930.7780.690海南文昌 HN3.92.6750.9810.6130.530广东湛江 YJ4.32.9221.1490.7060.619mean5.03.2761.2390.7100.634
2.3 低眼巨鲶养殖群体间遗传结构分析
2.3.1 分子方差分析(AMOVA)和基因流估算 分子方差分析表明,16%的遗传变异存在于种群间,84%的遗传变异存在于个体,个体的变异是低眼巨鲶总变异的主要来源(表5)。低眼巨鲶4个养殖群体之间Fst值的范围为0.030~0.130,广州与佛山两个群体间的值最小,为 0.030,小于0.05,说明两处群体间遗传分化程度较小,湛江与海南文昌群体之间的Fst值最高达到0.196,大于0.05,说明两处群体间遗传分化程度较高,其余群体间Fst值均大于0.05,证明广州、佛山群体与湛江、海南文昌群体间均存在一定的遗传分化(表6)。
表5 群体的分子方差分析(AMOVA)
Tab.5 Molecular ANOVA for populations
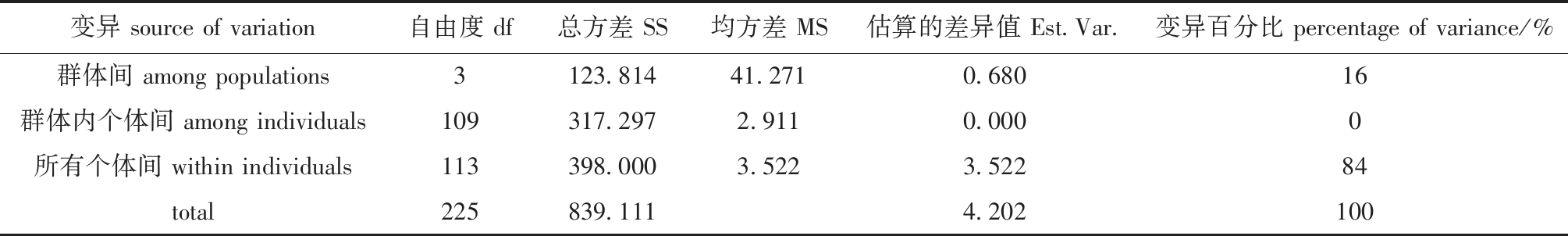
变异 source of variation自由度 df总方差 SS均方差 MS估算的差异值 Est.Var.变异百分比 percentage of variance/%群体间 among populations3123.81441.2710.68016群体内个体间 among individuals109317.2972.9110.0000所有个体间 within individuals113398.0003.5223.52284total225839.1114.202100
表6 4个低眼巨鲶养殖群体遗传分化系数(Fst)及基因流(Nm)
Tab.6 Genetic differentiation coefficient (Fst) and gene flow (Nm) among 4 cultured populations of Pangasianodon hypophthalmus
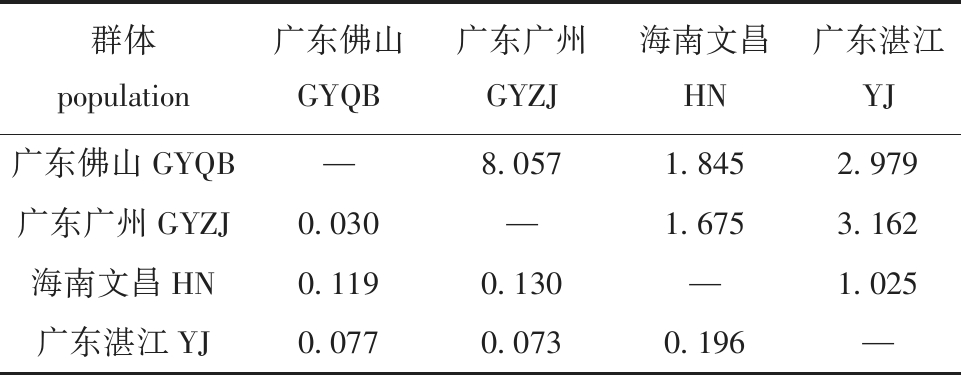
群体population广东佛山GYQB广东广州GYZJ海南文昌HN广东湛江YJ广东佛山GYQB —8.0571.8452.979广东广州GYZJ 0.030—1.6753.162海南文昌HN 0.1190.130—1.025广东湛江YJ 0.0770.0730.196—
注:对角线以下为遗传分化系数,对角线以上为基因流。
Note:The data below the diagonal represent Fst value,and above the diagonal represent genetic distance.
2.3.2 遗传距离与聚类分析 遗传距离计算结果显示,群体间遗传距离中,广东湛江和海南文昌群体间遗传距离最大,为0.476(HN/YJ)。广东广州和广东佛山群体间遗传距离最小,为0.117(GYQB/GYZJ)(表7)。
表7 群体间的遗传距离
Tab.7 Genetic distances between populations
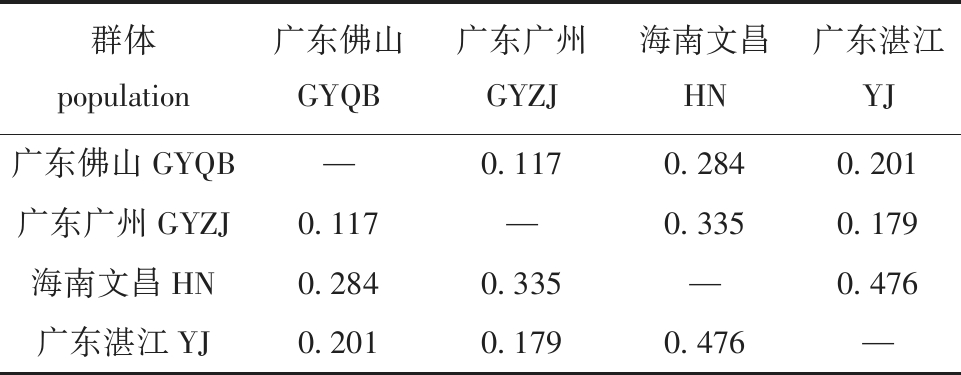
群体population广东佛山GYQB广东广州GYZJ海南文昌HN广东湛江YJ广东佛山GYQB—0.1170.2840.201广东广州GYZJ0.117—0.3350.179海南文昌HN0.2840.335—0.476广东湛江YJ0.2010.1790.476—
2.4 低眼巨鲶个体间遗传结构分析
利用10个分子标记对113份低眼巨鲶样本的结构进行评估。将113份低眼巨鲶样本划分为2个亚群(图1),根据似然值最大原则,判断最佳K值等于2(图2)。
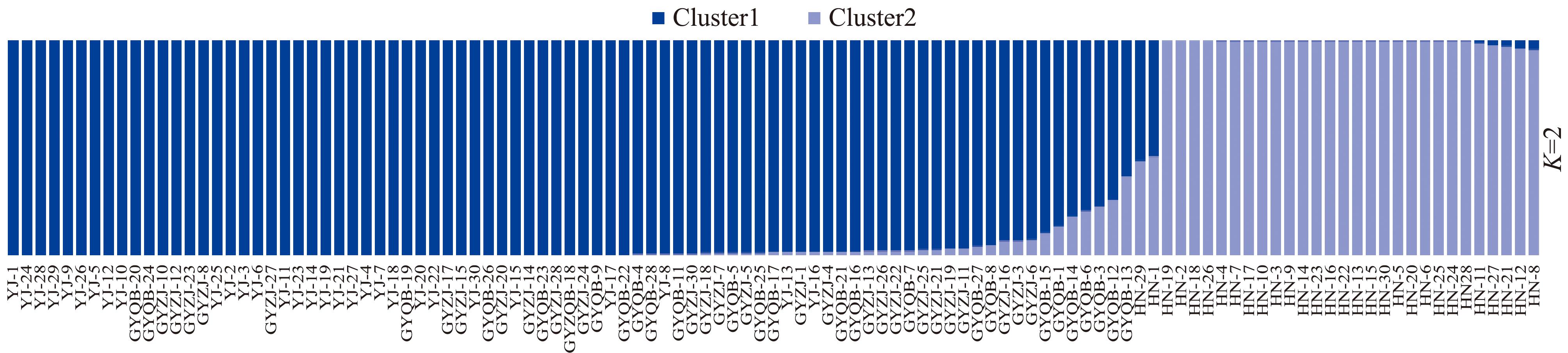
图1 4个低眼巨鲶养殖群体K值图
Fig.1 K-plot for 4 cultured populations of Pangasianodon hypophthalmus
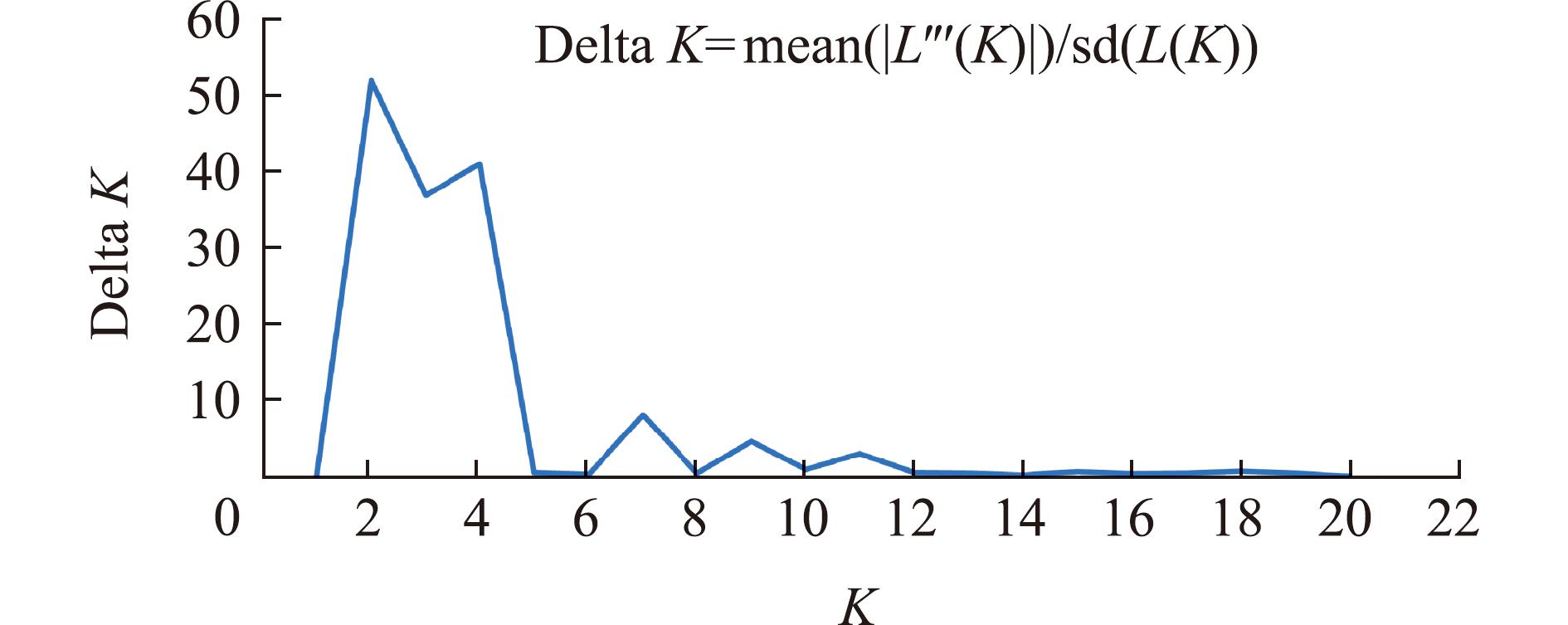
图2 STRUCTURE分析群体聚类结果(K=2)
Fig.2 Population genetic structure based on structure analysis at K=2
主坐标分析(PCoA)结果显示,广州群体(GYZJ)和佛山群体(GYQB)之间并无差异,且和湛江群体(YJ)相距较近。文昌群体(HN)相对其他3个群体遗传关系较远(图3)。个体样本的UPGMA聚类结果与主坐标结果大致相近,海南群体单独聚为一支,部分广州和佛山群体聚为一小支,另一部分广州和湛江群体聚为另一小支,3个广东群体聚为一大支(图4)。
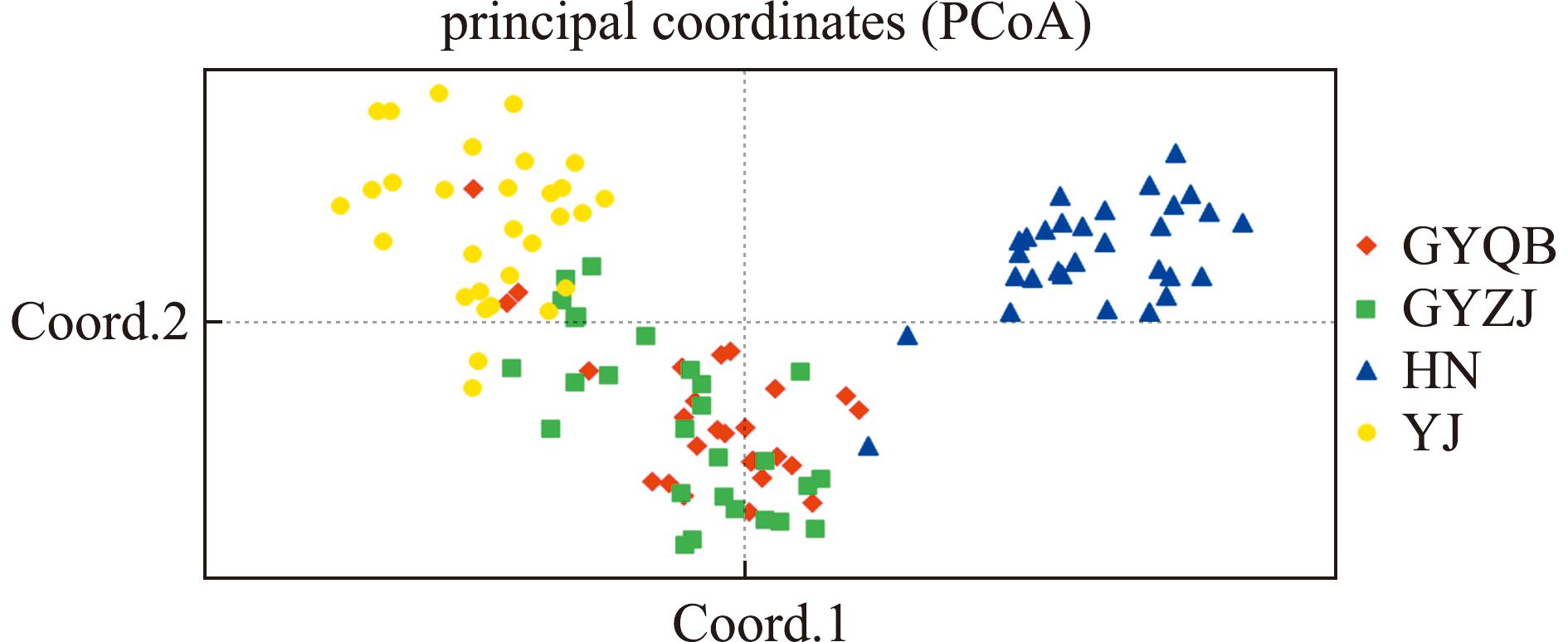
图3 低眼巨鲶样本的主坐标分析
Fig.3 Principal coordinate analysis plot of Pangasianodon hypophthalmus samples
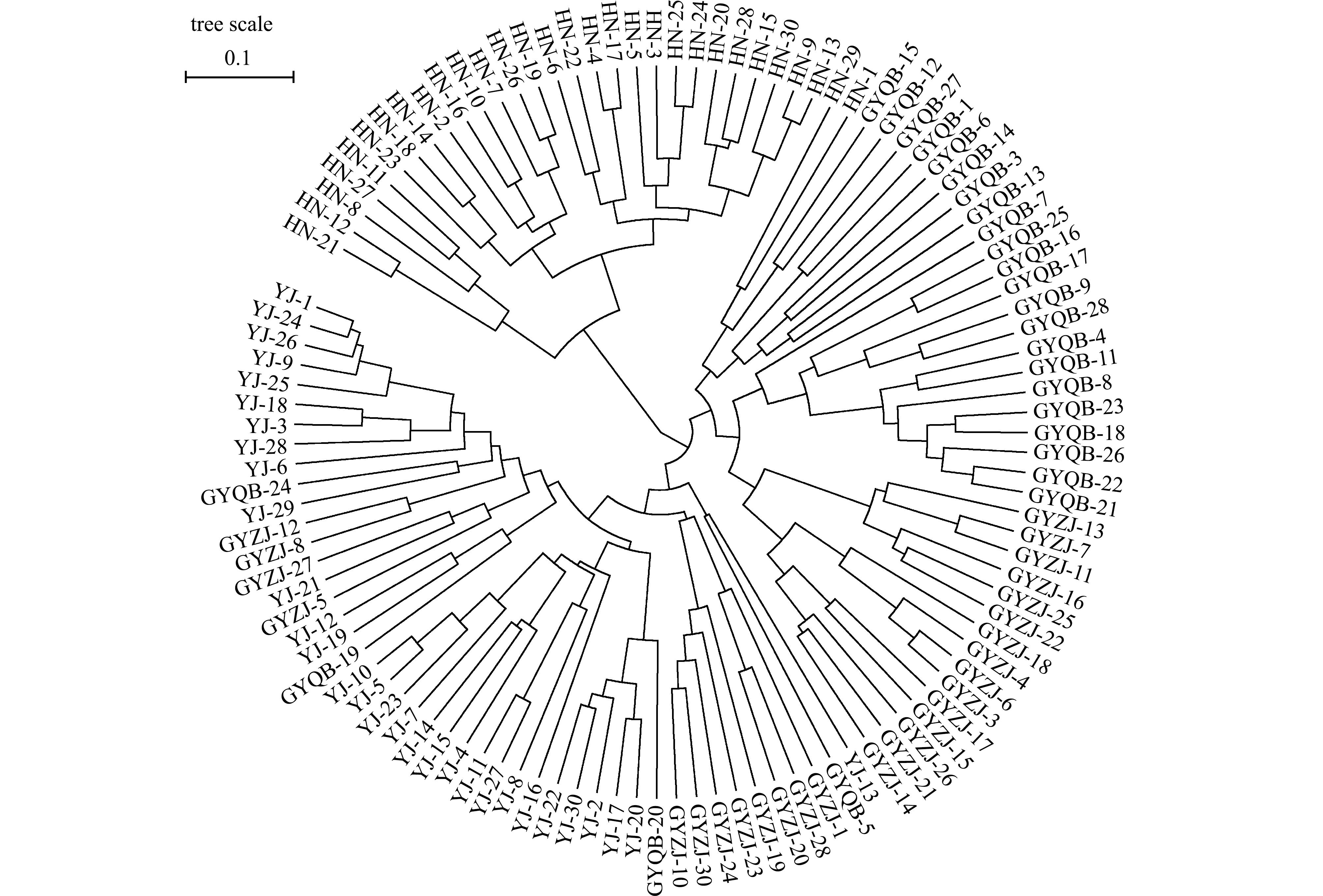
图4 低眼巨鲶样本的UPGMA聚类结果
Fig.4 UPGMA clustering results of Pangasianodon hypophthalmus samples
3 讨论
3.1 微卫星标记多态性及特征分析
本研究中,得到的10对引物多态信息含量(PIC)的数值范围为0.504(Pab013)~ 0.872(Pab063),且均高于0.5,根据Boststein的研究结果[26],PIC>0.5为高度多态性,表明本研究中选择的微卫星位点可作为低眼巨鲶遗传多样性及遗传结构分析的良好评价工具。
本研究中,采用10对引物在4个低眼巨鲶养殖群体中共检测出74个等位基因。其中,最小等位基因数目为4,最大等位基因数目为13,平均每个位点等位基因数目为7.4。有效等位基因总数为43.225,数值变化范围为2.247(Pab013)~8.557(Pab063)。香农指数(I)的数值范围为1.025(Pab013)~2.251(Pab063)。根据香农指数结果,佛山养殖群体的遗传多样性最丰富,其次是广州、湛江、文昌。物种杂合度越高,遗传多样性越高[27]。本研究中,观测杂合度高于期望杂合度,这可能意味着这个群体中的基因座存在一定程度的失衡选择或者基因漂变等现象,导致观测杂合度的数值偏离了期望值。同时,与哈代-温伯格提出的标准(Ho<0.5)相比[28],低眼巨鲶养殖群体多数大于0.5,表示低眼巨鲶养殖群体杂合度较高。
4个养殖群体中广州群体与佛山群体等位基因与杂合度明显高于其他两个群体且数值相近(表4),表明这两个群体种质来源相近,保留了较高的遗传多样性水平,且具有一定的选育潜力。4个养殖群体的有效等位基因(Ne)均小于观测等位基因(Na),这种现象也存在于罗氏沼虾(Macrobrachium rosenbergii)[11]、大口黑鲈(Micropterus salmoides) [29]等物种中。出现这种现象主要是等位基因不均衡导致,有些等位基因可能非常普遍,而其他等位基因则相对稀少。有效等位基因数较低意味着少数几个等位基因可能在种群遗传结构中起着主导作用。这种不均匀程度依次为佛山>广州>湛江>文昌。4个群体测得的观测杂合度(Ho)均高于期望杂合度(He),表明4个群体内部存在较高的遗传多样性,在鳜(Siniperca chuatsi)[30]、鲢(Hypophthalmichthys molitrix)[31] 等物种中也存在相近现象。实际观测到的杂合度比所预测的数量要多,意味着个体之间存在较多的遗传差异,这可能是由于存在选择压力或基因流的影响。表明该4处养殖群体遗传多样性丰富,更具生长优势,有助于群体适应环境变化,提高抗病能力和生存率。
3.2 低眼巨鲶群体遗传多样性分析
遗传分化指数(Fst)是评价群体间遗传分化程度的重要指标。Fst值越大,说明群体间的遗传分化程度越高。4个养殖群体间的Fst值范围为0.030~0.196(表6),这表明群体间存在不同程度的遗传分化。广州与佛山两个养殖群体遗传分化程度最低,该结果与实际情况相符,广州与佛山地理位置相对较近,因此可能出现较多的群体苗种交换,分化程度较低。而海南文昌与广东湛江之间遗传分化程度最高,海南省与广东省由于地域的限制,基因交流较少,因此呈现出较大的遗传分化。从基因流数据看,广州和佛山群体之间基因流程度最低,而湛江养殖群体与文昌养殖群体之间基因流程度最高,说明湛江养殖群体与文昌养殖群体间基因交流相对较少,分化程度较高。Wright[24]指出,当Nm大于1时,Nm>1表示遗传分化不显著。本研究中所有位点Nm均大于1,平均值为4.011,说明研究中的4个群体各个群体间基因交流频繁,遗传分化水平较低。 Fst值较大的群体,基因流数值均偏小,证明了研究结果的准确性。
遗传距离是衡量群体间遗传分化程度的重要指标[32]。4个群体间的遗传距离最大为0.476(海南文昌和广东湛江),最小为0.117(广东佛山和广州)。UPGMA聚类分析结果将4个群体分为了两支,一支为海南文昌,另一支为广东的3个群体。其中,佛山和广州的养殖群体单独聚为一小支。通过主坐标分析(PCoA)对遗传距离进行可视化处理,发现佛山和广州群体之间并无明显的遗传差异,其坐标点在图上几乎完全重叠。这两个群体与湛江群体的距离也相对较近。然而,海南群体在遗传上与其他3个群体的距离较远,其坐标点在PCoA图的右侧单独分布。这说明低眼巨鲶的养殖群体间存在一定的遗传分化,并且在广东省内的3个养殖群体间,遗传关系较为紧密。此外,UPGMA聚类分析结果与PCoA分析大致一致,都将广东和海南的群体分为了两大支。但在广东群体的内部分类上,存在一些差异。主坐标分析结果表明,广州和佛山群体在遗传上较为相近,而UPGMA聚类将广州群体一部分与佛山群体聚为一支,另一部分则与湛江群体为同一支。本研究结果指出,尽管佛山和湛江群体在遗传多样性上存在一定差异,广州群体与这两个群体的具体遗传关系仍需进一步研究和确认。
AMOVA分析表明,4个养殖群体的遗传变异主要是由于个体差异,占总变异的84%,与陈会娟[33]和潘秋芝[34]研究结果一致。不同养殖群体间造成的差异较小,只有16%,说明4处养殖群体之间遗传分化水平较低。通过Structure分析得出4处养殖群体的最佳K值(K=2),群体结构结果表明,113份样本可划分为两个聚类簇:一个是海南群体聚类簇,另一个聚类簇则包括广东省的广州、佛山和湛江群体。这与PCoA分析结果吻合。这种遗传差异可能是中国从越南或东南亚不同地方引种造成的,广东省的广州、佛山和湛江群体亲缘关系较近,可能引自同一区域。此外,气候可能也是造成这种差异的原因[35]。温度是影响鱼类生长的重要因素,不同温度可能会导致群体间的遗传分化。导致低眼巨鲶的4个群体被分两个亚群的原因可能是多种因素共同作用的结果,如遗传漂变、自然选择、环境因素、养殖方式和管理措施、基因交流等。这些因素相互作用,导致这些群体间产生了差异,从而被鉴别为两个亚群。
综上所述,4处低眼巨鲶养殖群体均存在高度的遗传多样性,种质资源良好,有一定的稳定性及选育潜力。海南群体与广东群体亲缘关系较远,广东省内广州与佛山群体亲缘关系最为密切,推测两地种苗来源可能较为接近。本研究对低眼巨鲶养殖群体遗传多样性的研究有助于更好地了解低眼巨鲶的遗传结构,从而为其保护和管理提供更好的科学依据。
4 结论
1)低眼巨鲶4个养殖群体平均观测杂合度(Ho)分别为0.742、0.778、0.613、0.706,均值为0.710,表明低眼巨鲶遗传多样性处于中高水平,在生产实践中具有进一步选育的潜力。
2) 根据遗传距离,4个养殖群体被分为两个遗传群体,海南群体单独聚为一支,其余3个群体聚为一支,4个群体间遗传距离最大为0.476(海南文昌/广东湛江),最小为0.117(广东广州/广东佛山)。海南群体与其他3个群体亲缘关系较远,有一定的遗传分化,要注意种质保存,以维持低眼巨鲶养殖种质资源的丰度。
3)低眼巨鲶4个养殖群体均具有丰富的遗传多样性,种质资源良好,具有较高的选育潜力。通过选育广州和佛山群体,可以提升群体的生长优势和抗病能力,此外,加强海南群体的独立保护,可维持整体遗传多样性和资源稳定性。
[1] 魏于生.巴沙鱼人工繁殖获得成功[J].淡水渔业,1998,28(2):36.
WEI Y S.A success in the artificial propagation of Pangasius hanigtoa[J].Freshwater Fisheries,1998,28(2):36.(in Chinese)
[2] 柯欢,王莉,熊伟.2种酸菜鱼鱼肉营养物质含量比较[J].食品工业,2022,43(12):314-317.
KE H,WANG L,XIONG W.Comparison of nutrient contents of two kinds of fish with pickled cabbage[J].The Food Industry,2022,43(12):314-317.(in Chinese)
[3] WANG Y,HOU H,ZHANG K,et al.Preparation and evaluation of biocompatibility of acellular dermal matrix of Basa fish (Pangasius bocourti)[J].Material Sciences,2018,8(4):346-354.
[4] KHOR Y P,WAN S Y,TAN C P,et al.Potential of using basa catfish oil as a promising alternative deep-frying medium:a thermo-oxidative stability study[J].Food Research International,2021,141:109897.
[5] 冯晓玲,陈茜,黎氏垂玲.越南巴沙鱼出口贸易发展分析[J].经济视角,2017,(3):84-93.
FENG X L,CHEN Q,LIT T L.Analysis of the development of export trade of basa fish in Vietnam[J].Economic Development Studies,2017,(3):84-93.(in Chinese)
[6] 罗乐佳,杨晶晶,聂艺云,等.巴沙鱼养殖与开发研究进展[J].水产养殖,2022,43(9):1-5,42.
LUO L J,YANG J J,NIE Y Y,et al.A review of breeding and application of Pangasianodon hypophthalmus[J].Journal of Aquaculture,2022,43(9):1-5,42.(in Chinese)
[7] 袁宗伟,陈诏,徐鸿飞,等.苏氏圆腹(鱼芒)人工繁殖及杂交子代胚胎发育观察[J].贵州农业科学,2019,47(6):73-78.
YUAN Z Z,CHEN Z,XU H F,et al.Artificial propagation of Pangasias sutchi and observation of hybrid progeny embryo[J].Guizhou Agricultural Science,2019,47(6):73-78.(in Chinese)
[8] 倪未,陈海港,刘晓莉,等.巴沙鱼低眼巨鲶胚胎及仔稚鱼发育观察及其生长模型的确定[J].大连海洋大学学报,2023,38(4):584-592.
NI W,CHEN H G,LIU X L,et al.Embryonic development and growth of larvae and juvenile basa fish striped catfish (Pangasianodon hypophthalmus) and the determination of growth model[J].Journal of Dalian Ocean University,2023,38(4):584-592.(in Chinese)
[9] ZIETKIEWICZ E,RAFALSKI A,LABUDA D.Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J].Genomics,1994,20(2):176-183.
[10] 余成晨,张伟,徐晓雁,等.草鱼选育群体不同世代遗传变异的微卫星分析[J].中国水产科学,2022,29(8):1109-1118.
YU C C,ZHANG W,XU X Y,et al.Genetic variations among different generations of a breeding population of grass carp (Ctenopharyngodon idella) using microsatellite markers[J].Journal of Fishery Sciences of China,2022,29(8):1109-1118.(in Chinese)
[11] 陈雪峰,王春琳,李荣华,等.罗氏沼虾转录组SSR标记筛选及其与体质量相关性分析 [J].水生生物学报,2022, 46 (8):1150-1159.
CHEN X F,WANG C L,LI R H,et al. Screening of transcriptomic SSR markers and their correlation with body weight in Macrobrachium rosenbergii [J].Acta Hydrobiologica Sinica, 2022,46(8):1150-1159.(in Chinese)
[12] 胡玉婷,凌俊,江河,等.中华绒螯蟹4个养殖群体遗传多样性与遗传结构分析[J].江苏农业科学,2022,50(16):54-59.
HU Y T,LING J,JIANG H,et al.Genetic diversity and genetic structure of four cultured populations of the Chinese mitten crab (Eriocheir sinensis) [J].Jiangsu Agricultural Sciences,2022,50 (16):54-59.(in Chinese)
[13] TAGGART J,FERGUSON A. Minisatellite DNA fingerprints of salmonid fishes[J].Animal Genetics,1990,21 (4):377-389.
[14] CARTER R, MAIR G, SKIBINSKI D,et al. The application of DNA fingerprinting in the analysis of gynogenesis in tilapia[J].Aquaculture,1991,95 (1-2):41-52.
[15] DESVIGNES J F,LAROCHE J,DURAND J D,et al. Genetic variability in reared stocks of common carp (Cyprinus carpio L.) based on allozymes and microsatellites[J].Aquaculture,2001,194 (3-4):291-301.
[16] 张世勇,刘洪岩,王江,等.基于微卫星标记的斑点叉尾鮰家系鉴定技术及应用[J].水生生物学报,2021,45(2):327-333.
ZHANG S Y,LIU H Y,WANG J,et al.Establishment and application of pedigree identification technique for channel catfish based on microsatellite markers[J].Acta Hydrobiologica Sinica,2021,45(2):327-333.(in Chinese)
[17] 梁业松,张维炜,宋飞彪,等.虎龙杂交斑养殖群体遗传多样性与遗传结构的微卫星分析[J].水产学报,2022,46(1):31-40.
LIANG Y S,ZHANG W W,SONG F B,et al.Genetic diversity and genetic structure analysis of aquaculture groups of hybrid grouper [Epinephelus fuscoguttatus (♀) × E.lanceolatus (♂)] using microsatellite markers[J].Journal of Fisheries of China,2022,46(1):31-40.(in Chinese)
[18] 郑翔,徐杰杰,张佳佳,等.4个瓦氏黄颡鱼群体遗传多样性的微卫星分析[J].水产科学,2020,39(5):657-668.
ZHENG X,XU J J,ZHANG J J,et al.Genetic diversity analysis in four different geographical populations of yellow catfish Pelteobagrus vachelli by microsatellite markers[J].Fisheries Science,2020,39(5):657-668.(in Chinese)
[19] 陈辰,朱新平,李伟.一种中华鳖基因组微卫星多重PCR体系构建方法 [P].广东省:CN201811111994.9,2022-03-01.
CHEN C,ZHU X P,LI W.A method for constructing a microsatellite multiplex PCR system for the Chinese softshell turtle genome [P].Guangdong Province:CN201811111994.9,2022-03-01.(in Chinese)
[20] 徐昊旸,杨雪莹,倪未,等.乌龟选育世代遗传多样性及遗传结构的微卫星分析[J].大连海洋大学学报,2023,38(5):812-818.
XU H Y,YANG X Y,NI W,et al.Genetic diversity and genetic structure of selected generations of Chinese pond turtle(Mauremys Reevesii) based on microsatellite DNA[J].Journal of Dalian Ocean University,2023,38(5):812-818.(in Chinese)
[21] PEAKALL R,SMOUSE P E.Genalex 6:genetic analysis in Excel.Population genetic software for teaching and research[J].Molecular Ecology Notes,2006,6(1):288-295.
[22] EARL D A,VONHOLDT B M.STRUCTURE HARVESTER:a website and program for visualizing STRUCTURE output and implementing the Evanno method[J].Conservation Genetics Resources,2012,4(2):359-361.
[23] JAKOBSSON M,ROSENBERG N A.CLUMPP:a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure[J].Bioinformatics,2007,23(14):1801-1806.
[24] WRIGHT S.Evolution in mendelian populations[J].Genetics,1931,16(2):97-159.
[25] GOWER J C.Some distance properties of latent root and vector methods used in multivariate analysis[J].Biometrika,1966,53(3/4):325-338.
[26] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[27] QIN Y,SHI G,SUN Y.Evaluation of genetic diversity in Pampus argenteus using SSR markers[J].Genetics and Molecular Research:GMR,2013,12(4):5833-5841.
[28] 于冬梅,马海涛,孙效文.三个虹鳟养殖群体遗传结构的微卫星分析[J].动物学杂志,2007,42(4):8-14.
YU D M,MA H T,SUN X W.Genetic diversity in three rainbow trout populations as revealed by microsatellite markers[J].Chinese Journal of Zoology,2007,42(4):8-14.(in Chinese)
[29] 傅建军,龚雅婷,朱文彬,等.大口黑鲈微卫星多重PCR体系及3个群体的遗传分析[J].水生生物学报,2023,47(9):1514-1522.
FU J J,GONG Y T,ZHU W B,et al.Multiplex PCR sets of microsatellites and their application in genetic analyses for three populations of largemouth bass(Micropterus salmoides)[J].Acta Hydrobiologica Sinica,2023,47(9):1514-1522.(in Chinese)
[30] 范士琦,冯婧昀,苗晓敏,等.重庆养殖场鳜群体微卫星遗传多样性研究[J].水产养殖,2023,44(7):18-23.
FAN S Q,FENG J Y,MIAO X M,et al.Microsatellite genetic diversity of Chinese perch(Siniperca chuatsi)populations in three farms in Chongqing[J].Journal of Aquaculture,2023,44(7):18-23.(in Chinese)
[31] 王亚军.鲢微卫星标记开发及湖北养殖群体遗传多样性分析[D].荆州:长江大学,2023.
WANG Y J.Development of microsatellite markers for silver carp and genetic diversity analysis of breeding population in Hubei province[D].Jingzhou:Yangtze University,2023.(in Chinese)
[32] THORPE J P.The molecular clock hypothesis:biochemical evolution,genetic differentiation and systematics[J].Annual Review of Ecology and Systematics,1982,13:139-168.
[33] 陈会娟.长江中游四大家鱼放流亲本对早期资源和遗传多样性的影响研究[D].重庆:西南大学,2019.
CHEN H J.Study on the influence of parents released from the middle reaches of the Yangtze River on early resources and genetic diversity[D].Chongqing:Southwest University,2019.(in Chinese)
[34] 潘秋芝.斑鳜微卫星标记开发及其遗传多样性评价[D].贵阳:贵州大学,2022.
PAN Q Z.Development of microsatellite markers and evaluation of genetic diversity of Siniperca chuatsi[D].Guiyang:Guizhou University,2022.(in Chinese)
[35] 寿鹿,杜卫国,陆祎玮.北草蜥种群间生活史变异的成因分析:热环境、食物可利用性和体温的岛屿间差异[J].动物学报,2005(5):797-805.
SHOU L,DU W G,LU Y W. The causal analysis of inter-population variation in life histories of the northern grass lizard Takydromus septentrionalis:between-island differences in thermal environment, food availability and body temperature [J].Acta Zoologica Sinica,2005 (5):797-805.