乌鳢(Channa argus)隶属于鲈形目鳢亚目鳢科鳢属,主要分布在中国长江及以北等水系。乌鳢具有生长速度快、耐低氧、抗逆性强等特点,且其肉质鲜美、蛋白质含量高、骨刺较少,还具有抗氧化、促进伤口愈合、调节心血管、治疗骨关节炎和抗抑郁等药用效能,深受消费者的喜爱[1]。据《中国渔业统计年鉴》统计,乌鳢近年来养殖产量不断升高,2023年超55万t,是已审定通过的水产新品种鳢的重要亲本来源。开发乌鳢新品种能提高养殖产量及社会效益,满足市场对优质苗种需求,促进乌鳢养殖产业的可持续发展。目前,由于多数苗种场利用养殖商品鳢作为亲本进行育苗,亲本种质混杂,近亲交配情况难以避免,导致生长速度降低、畸形率升高、抗逆性衰退及饵料系数增加等问题[2],因此,应当鉴定、利用中国地方特色乌鳢种质资源,选育出品质高、抗性好、产量大的乌鳢新品种,以促进鳢养殖产业健康发展。
在分子标记研究领域中,SNP具有数量多分布广、遗传稳定性高、富有代表性和易实现自动化分析等优点。因此,SNP是基因组中最丰富和最具潜力的DNA变异类型。随着高通量测序技术和SNP分型技术的发展,SNP已成为群体遗传分析的重要标记。在简化基因组测序研究领域中,SLAF测序(specific-locus amplified fragment sequencing,SLAF-seq)既是一种双酶切简化基因组测序方法,也是一种低成本高效的全基因组SNP分型方法。与传统技术相比,SLAF-seq具有显著优势,能够有效缩短基因组测序的时间和成本,降低数据处理的复杂性,目前,该方法凭借其高效性与低廉成本已成为水生生物种群遗传学研究领域的可靠工具,如刘亚楠等[3]采用SLAF-seq发现克氏原螯虾(Procambarus clarkii)的14个群体遗传多样性较低。Wanghe等[4]运用SLAF-seq成功界定了物种,为西藏高原鱼类的遗传多样性保护提供了参考依据。
鳢作为重要的经济鱼类,目前在国内外均有其群体的遗传多样性研究。在北美等地采用基于2b-RAD测序SNP标记[5]或微卫星标记[6]评估乌鳢群体的种群规模结构等遗传背景信息,揭示乌鳢的入侵潜力,为有效管理乌鳢种群的扩散及入侵提供了有力支持。在南亚及东南亚的巴基斯坦[7]、印度[8]和菲律宾[9]通过线粒体DNA相关序列及湄公河三角洲[10]地区采用微卫星标记解析了当地鳢群体的遗传多样性水平等信息,为鳢的保护策略提供了参考依据。在中国等地通过线粒体DNA相关序列[11-20]、微卫星标记[21-26]、AFLP标记[27]和基于转录组测序SNP标记[28],以及基于全基因组重测序SNP标记[29]分析了乌鳢群体的遗传多样性及群体遗传结构等遗传背景信息,推动了地方乌鳢种质资源的开发利用和综合保护。本研究中旨在利用SLAF-seq技术对5个地理乌鳢群体的SNP标记进行大规模开发和遗传分析,以期为乌鳢遗传资源的保护及新品种培育提供遗传背景信息。
1 材料与方法
1.1 材料
2017年4月—2018年9月,分别从黑龙江省抚远(HLJ)采集19尾、四川省内江(NJH、NJB)采集35尾、山东省微山湖(SD)采集21尾和浙江省湖州(ZJ)采集6尾,共4个地区乌鳢群体合计81个活体样本(表1),其中四川省内江群体中,乌鳢(NJH)的体色呈灰黑色,而乌鳢(NJB)的体色呈白色。
表1 乌鳢样品采样信息
Tab.1 Sample details of Channa argus
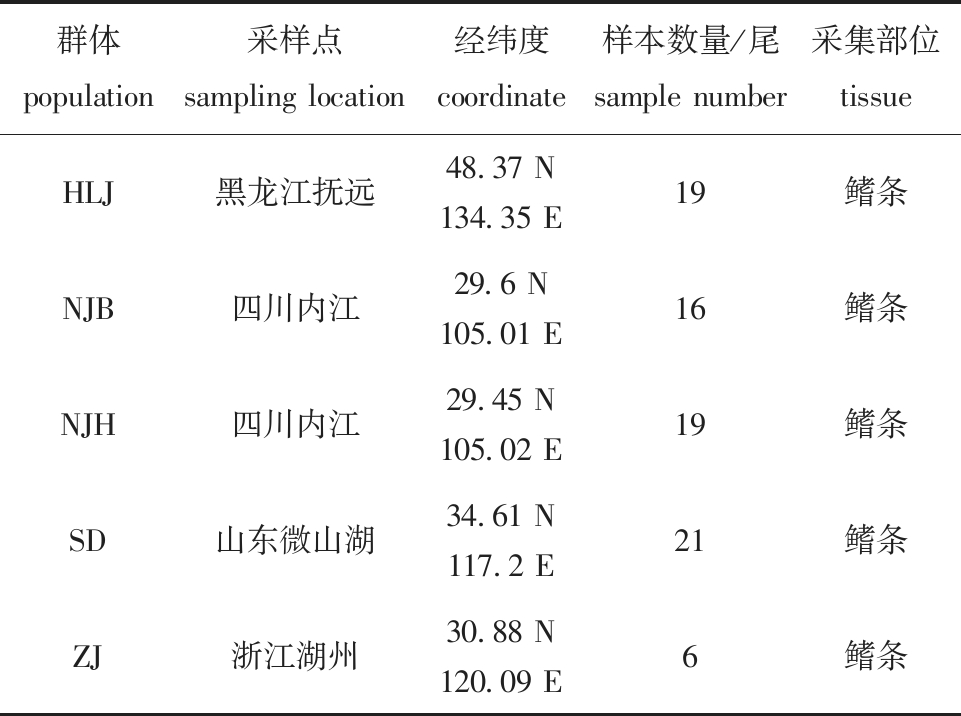
群体population采样点sampling location经纬度coordinate样本数量/尾sample number采集部位tissueHLJ黑龙江抚远48.37 N134.35 E19鳍条NJB四川内江29.6 N105.01 E16鳍条NJH四川内江29.45 N105.02 E19鳍条SD山东微山湖34.61 N117.2 E21鳍条ZJ浙江湖州30.88 N120.09 E6鳍条
1.2 方法
1.2.1 样本来源和DNA提取 采集样本的鳍条组织并存放在无水乙醇溶液中,然后在-20 ℃下保存。利用Qiagen的血液/组织基因组DNA提取试剂盒(DNeasy Blood and Tissue Kit)对81个样品鳍条组织的基因组DNA进行提取。通过NanoDrop One分光光度计(Thermo Scientific,USA)和1 g/L琼脂糖凝胶电泳检查DNA质量和浓度后,委托北京百迈客生物科技有限公司检测。
1.2.2 SLAF-seq文库构建与测序 本研究采用一种基于高通量测序的高效大规模基因分型方法SLAF-seq技术。为使超100 000个SLAF标签均匀分布在乌鳢种群中,利用酶切预测软件SLAF-predict对乌鳢参考基因组(基因组大小为630 M,GC含量为39.93%)进行电子酶切预测,选择最佳酶切方案对各样品基因组DNA开展酶切。在37 ℃下采用Klenow(NEB)片段和dATP将单核苷酸A添加到3′末端中,然后用T4 DNA连接酶将Duplex Tag-labeled测序接头连接到poly A的酶切片段上,对所获得的片段进行PCR扩增,采用Agencourt AMPure XP磁珠(Beckman Coulter,High Wycombe,UK)纯化PCR产物并合并,通过2 g/L琼脂糖凝胶电泳分离。采用QI Aquick Gel Extraction Kit(QIAGEN)试剂盒对切除片段进行纯化。在文库质检合格后采用Illumina Hi SeqTM 2500测序平台(Illumina,Inc;San Diego,CA,US)对凝胶纯化的产物进行读长126 bp×2 测序,以上均委托北京百迈客生物科技有限公司完成。
1.2.3 SLAF标签与SNP分子标记检测 利用Dual-index对测序得到的原始数据进行识别,得到各个样品的序列reads,过滤测序reads的接头、过滤含N多的reads及过滤低质量reads后,根据Q20(read质量得分为20,表示碱基测序错误概率为1%)和GC含量这两个关键质量指标来进行测序质量和数据量的评估。采用BLAT软件根据序列相似性对高质量成对末端reads进行聚类,不同个体间序列的相似度超过90%被确定为一个SLAF标签,以每个SLAF标签中深度最高的序列类型作为参考序列开发SNP分子标记,采用比对软件BWA(v0.7.17)将测序数据与乌鳢参考基因组比对,应用基因组分析工具包GATK(v4.2)和SAMtools(v1.13)进行SNP变异检测构建原始SNP数据集。采用PLINK(v1.9)根据位点信息完整性(INT)≥0.5和次要等位基因频率(MAF)≥0.05的标准过滤高质量的SNP。最后,采用筛选出的高质量SNP进行进一步分析。
1.2.4 群体遗传进化统计分析 基于质控后SNP 信息,采用VCFtools(v0.1.16)计算配对群体遗传分化系数(FST)与基因流(Nm),核苷酸多态性(π),多态信息含量(PIC)。基于邻接法(neighbor-joining methods)采用PHYLIP(v3.697)软件构建样品的系统发育树表示群体间的进化关系,并将发育树文件(newick)采用ggtree(v3.82)以可视化展示群体间的关系。采用PLINK(v1.9)进行主成分分析(PCA),并计算观测杂合度(Ho)和期望杂合度(He)遗传多样性指数。采用ADMIXTURE(v1.30)分析群体结构,并进行聚类,最后根据最低交叉验证错误率(CV error)值判断最优分群数。
2 结果与分析
2.1 建库质量评估与测序结果
对乌鳢参考基因组进行电子酶切预测,根据酶切方案确定两种限制性内切酶Rsa Ⅰ(New England Biolabs,NEB)和Hae Ⅲ(New England Biolabs,NEB),酶切片段长度在364~414的序列定义为SLAF标签。所有测序样品共获得317.66 Mb reads数据,测序样品平均reads数量为3 921 817,测序平均Q20为99.17%,平均GC含量为41.19%,通过SOAP软件与参考基因组比对率平均为97.95%(表2)。上述测序数据表明,所获得数据可靠,可用于后期分析。
表2 乌鳢样品测序数据统计表
Tab.2 Statistics of sequencing data in Channa argus
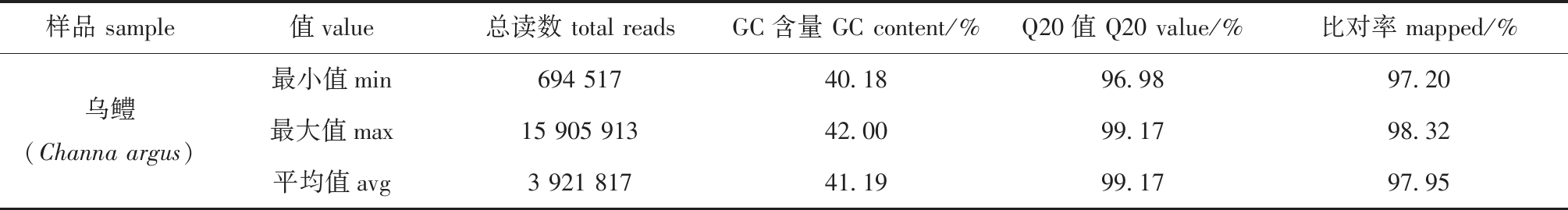
样品 sample值value总读数 total readsGC含量 GC content/%Q20值 Q20 value/%比对率 mapped/%最小值min694 51740.1896.9897.20乌鳢(Channa argus)最大值max15 905 91342.0099.1798.32平均值avg3 921 81741.1999.1797.95
2.2 SLAF标签与SNP分子标记统计
从81份乌鳢材料中共检测到981 780个SLAF标签,平均每份样品SLAF标签数量为210 796,平均测序总深度为3 581 414,平均测序深度为16.33 x。通过BWA(v0.7.17)软件将SLAF标签定位到参考基因组上,其中多态性的SLAF标签共有427 703个。采用GATK(v4.2)和SAMtools(v1.13)两种方法得到的SNP标记交集作为最终SNP标记数据集,共得到1 987 293个群体SNP标记。对群体SNP过滤,共筛选808 976个高质量SNP标记,可用于后续乌鳢群体遗传进化分析。各样品完整度为16.37%~60.07%,平均为34.93%,杂合率为1%~27.03%,平均为3.57%,表明检测样品基因组杂合度较高(表3)。
表3 SLAF标签和SNP标记数据统计
Tab.3 Data statistics of SLAF and SNP
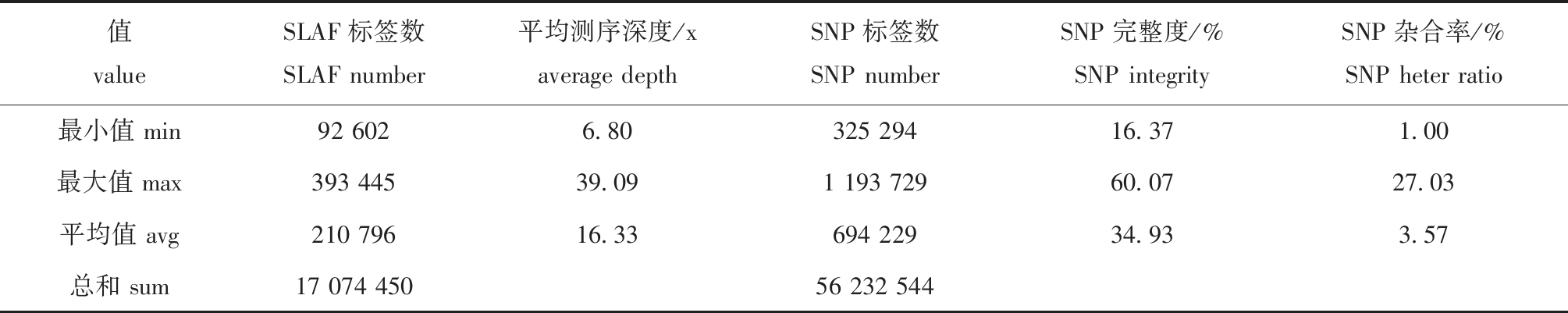
值valueSLAF标签数SLAF number平均测序深度/xaverage depthSNP标签数SNP numberSNP完整度/%SNP integritySNP杂合率/%SNP heter ratio最小值 min92 6026.80325 29416.371.00最大值 max393 44539.091 193 72960.0727.03平均值 avg210 79616.33694 22934.933.57总和 sum17 074 45056 232 544
注:x表示基因组大小倍数;完整度,即每个样品SNP在全部样品中的信息比重;杂合率,即每个样品含两种或两种以上碱基类型的SNP所占比重。
Note:x represents the multiple of genome size;integrity ration,the information proportion of each sample SNP in all samples;Heterozygosity,the proportion of SNPs containing two or more base types in each sample.
2.3 群体遗传多样性分析
群体多样性指数是衡量物种多样性高低的重要标准。通过计算Ho、He、PIC、FIS和π来分析各群体的遗传变异程度,统计结果见表4。 5个群体的观测杂合度(Ho)为0.078~0.134,期望杂合度(He)为0.094~0.183,近交系数(FIS)为0.288~0.579,核苷酸多态性(π)为2.96×10-6~5.73×10-6,多态信息含量(PIC)为0.156~0.250,属于低水平多态性位点(PIC<0.25)(表4)。四川内江两群体(NJH)与(NJB)间Ho与He最相近,近交系数FIS最高,核苷酸多态性π值较低。
表4 乌鳢群体的遗传多样性
Tab.4 Genetic diversity for populations of Channa argus
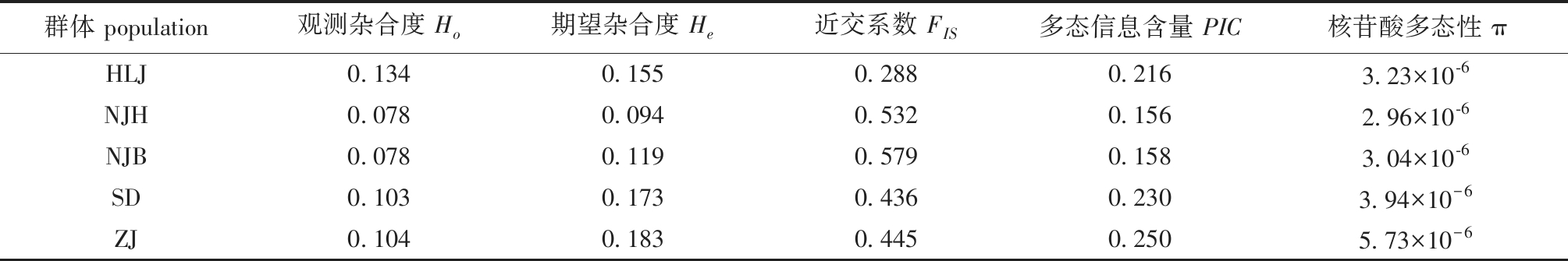
群体 population观测杂合度 Ho期望杂合度 He近交系数 FIS多态信息含量 PIC核苷酸多态性 πHLJ0.1340.1550.2880.2163.23×10-6NJH0.0780.0940.5320.1562.96×10-6NJB0.0780.1190.5790.1583.04×10-6SD0.1030.1730.4360.2303.94×10-6ZJ0.1040.1830.4450.2505.73×10-6
2.4 群体遗传分化分析
计算5个群体之间的Fst值与Nm。结果显示,乌鳢各群体之间基因流为0.503~0.856,均小于1,其中,四川内江群体(NJH)与(NJB)的基因流最大,为0.856。乌鳢各群体间遗传分化系数为0.042~0.247,山东与四川内江、黑龙江群体、浙江群体间遗传分化系数为0.05~0.15,处于中度分化水平,四川内江(NJH)与(NJB)群体间遗传分化系数为0.042,处于低度遗传分化水平,其余群体间遗传分化指数均大于0.15,处于高度遗传分化水平(表5)。
表5 乌鳢群体间的遗传分化系数和基因流
Tab.5 FST values and gene flow between each population of Channa argus
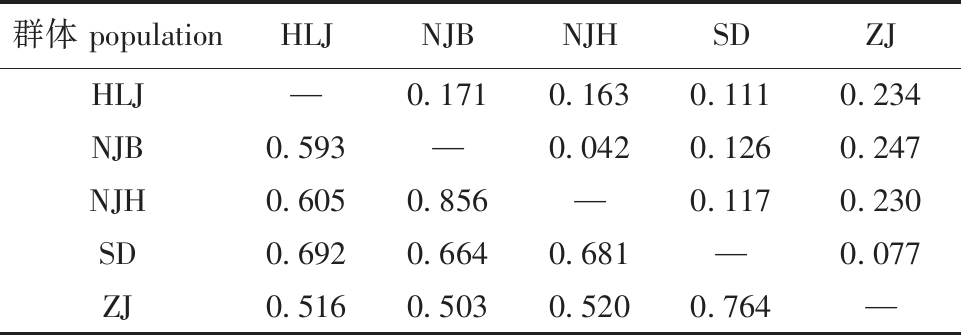
群体 populationHLJNJBNJHSDZJHLJ—0.1710.1630.1110.234NJB0.593—0.0420.1260.247NJH0.6050.856—0.1170.230SD0.6920.6640.681—0.077ZJ0.5160.5030.5200.764—
注:对角线上的部分为Fst,对角线下的部分为Nm。
Note:The part above the diagonal is Fst,and the part below the diagonal is Nm.
2.5 系统发育树分析
基于筛选高质量SNP标记,采用基于邻接法PHYLIP(v3.697)软件构建81尾乌鳢群体的系统发育树(图1)。乌鳢首先以黑龙江抚远群体(HLJ)的样本聚为一支,然后与山东微山湖群体(SD)、 浙江湖州群体(ZJ)及四川内江群体(NJH)与(NJB)聚为一支。其中,黑龙江群体(HLJ)与浙江群体(ZJ)分支距离较长,两者差异较大,亲缘关系较远。四川内江群体(NJB)与(NJH)由于地处环境相近处于同一分支,亲缘关系较近,而浙江群体(ZJ)分离出来聚为一支。
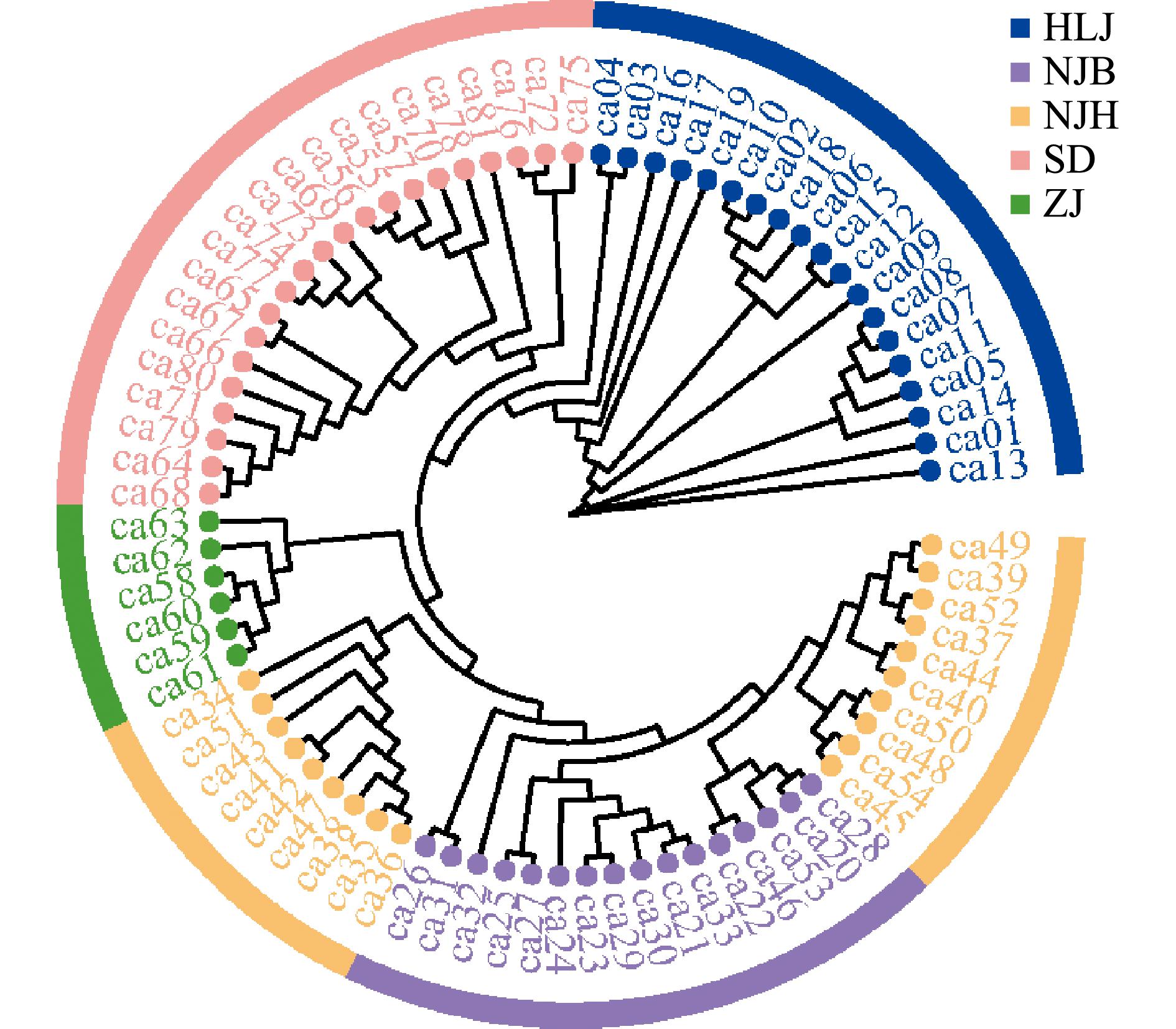
图1 乌鳢群体系统发育树
Fig.1 Phylogenetic tree of the Channa argus populations
2.6 群体结构分析
为进一步表明乌鳢群体之间的遗传关系,基于ADMIXTURE(v1.30)软件分析群体遗传结构结果显示(图2),当K=4时,交叉验证错误率最低,81尾乌鳢个体有最佳聚类效果,此时对群体遗传结构与系统发育树分类结果进行比较,发现这两个分类结果有较高的一致性。
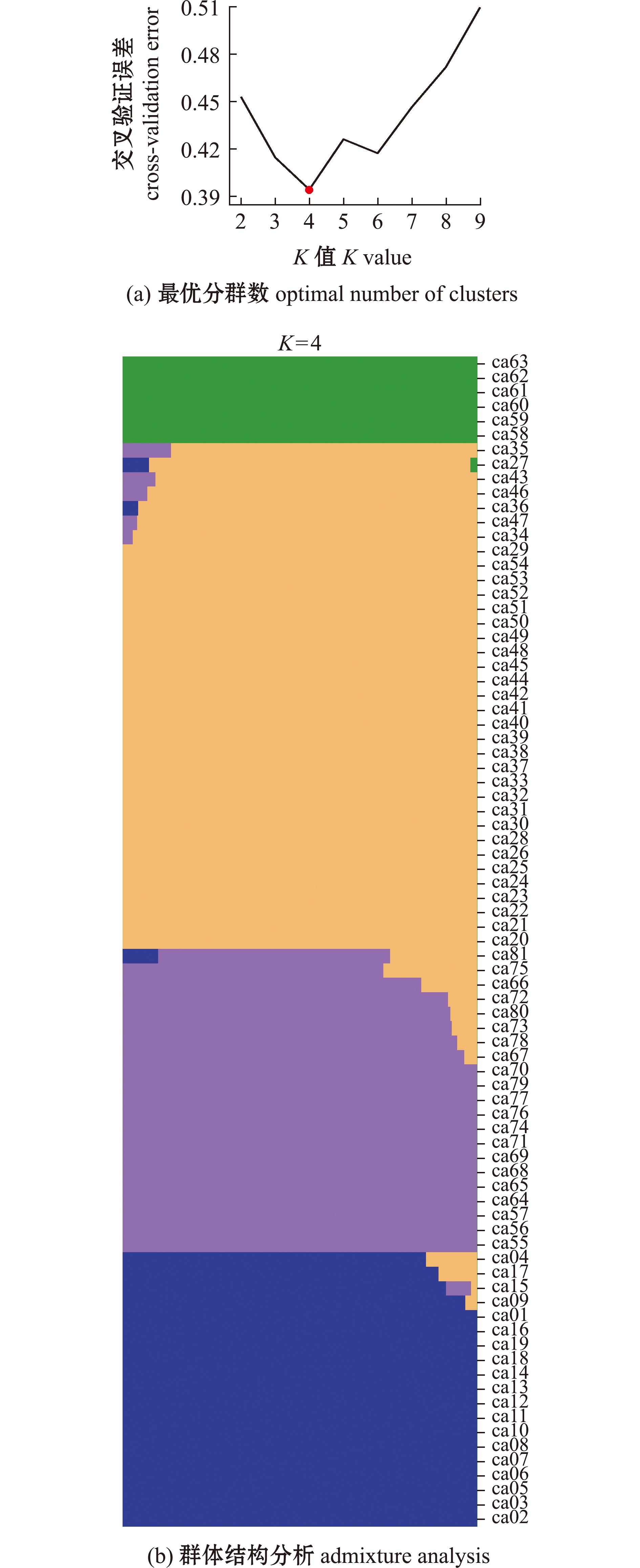
图(a)不同K值所对应的交叉验证错误率,红点显示为最佳群体数。
The admixture validation error rate corresponding to the different K values,red dot represents the optimal number of K clustering.
图2 乌鳢最优分群数与群体结构分析
Fig.2 Optimal number of clusters and Admixture analysis of Channa argus populations
2.7 群体主成分分析
通过主成分分析进一步表明了81尾乌鳢群体的聚类结果(图3),分析结果显示,浙江群体(ZJ)与其他群体间存在相对较远的亲缘关系,而山东群体(SD)和黑龙江群体(HLJ)存在相对较近的亲缘关系,四川内江群体(NJH)与(NJB)存在相近的亲缘关系,这些结果与系统发育树的聚类结果类似。
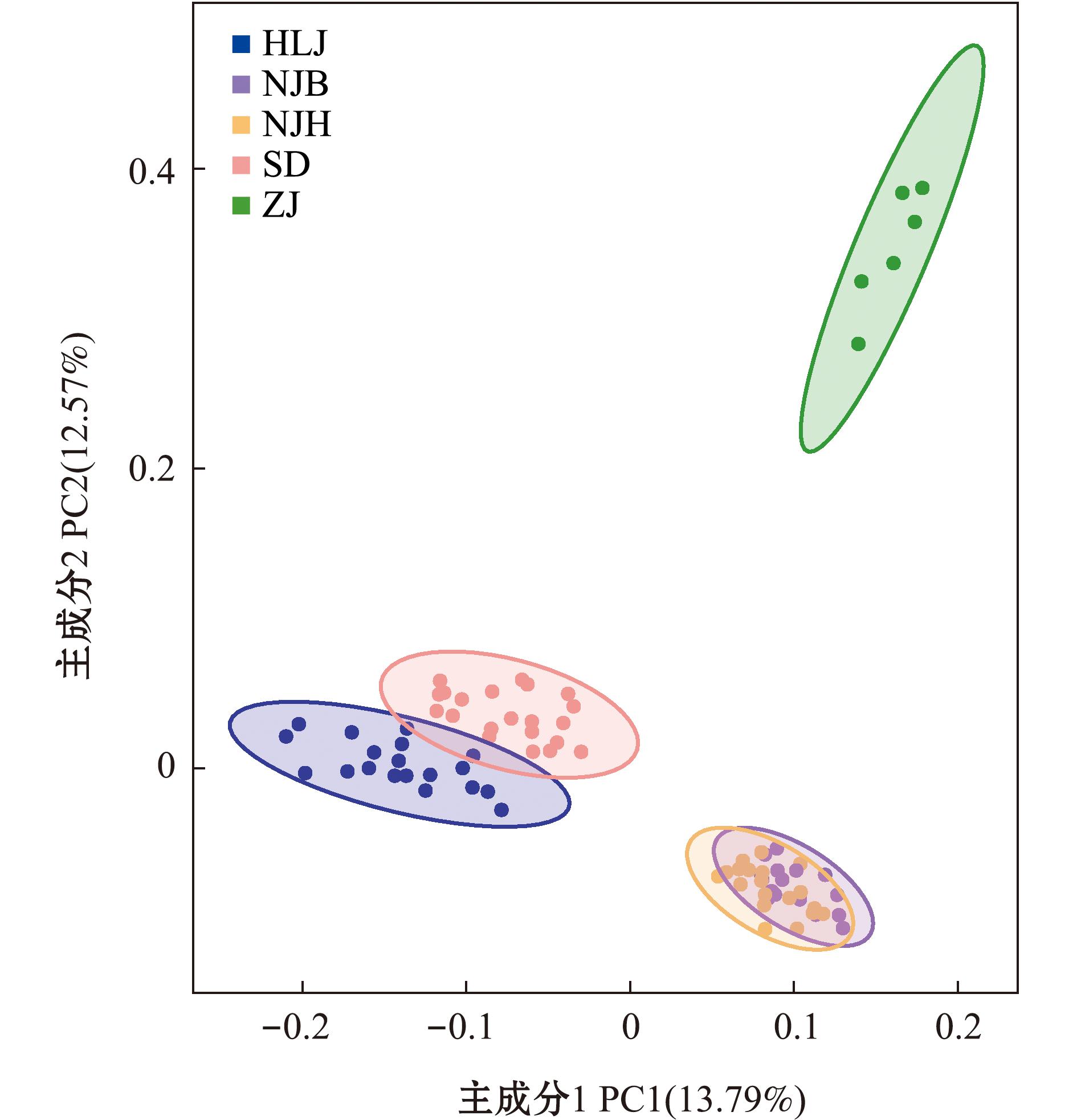
图3 乌鳢群体PCA分析
Fig.3 PCA analysis of Channa argus populations
3 讨论
3.1 5个乌鳢群体的遗传多样性
随着高通量技术发展,SLAF-seq简化基因组测序技术已被应用于多种水生生物,这项技术是实现SNP标记开发的重要手段。在本研究中,开发了1 987 293个乌鳢群体SNP标记,远高于Resh等[5]基于2b-RAD测序得到的23 695个SNP标记,以及张伯池[28]基于转录组测序开发的20 374个SNP标记,但低于郑合家[29]基于全基因组重测序挖掘得到2 962 454个SNP标记,所获SNP标记数量的差异主要是由于所选测序技术手段不同,以及基因组特征及选择的试验群体数量和群体特征不同。本研究中利用SLAF-seq技术对乌鳢5个群体进行简化基因组测序,能够以较低的成本获得高质量的位点信息,从而评估乌鳢群体的遗传多样性。
遗传多样性能够反映出群体间遗传多样性水平,为有效开发和利用种质资源提供理论依据。本研究中5个乌鳢群体的杂合度Ho和He值及群体多态信息含量PIC值均低于乌鳢研究中其他分子标记观察到的结果[23,28-29],推测原因是单个微卫星标记所检测等位基因数量为SNP标记的4~11倍[30],能够提供更丰富的遗传信息,在计算遗传多样性参数方面更佳。如在相同地区乌鳢群体研究中,Yan等[24]采用10个微卫星标记得出的杂合度值高出张伯池[28]用18个SNP标记的杂合度数1.5~2倍,Zhu等[22]与张露等[23]采用微卫星标记研究山东群体的多态信息含量(PIC值)同样远高于郑合家[29]研究的山东群体。但在微卫星分子标记研究中,当样本采集量小于30且分子标记量小于25时,遗传多样性数值变化差异显著[31],如刘改艳[32]在湖南采集的19尾乌鳢,基于微卫星标记测得的杂合度(0.275 7~0.326 2)比其他研究中微卫星标记测得结果低50%左右,然而SNP分子标记在全基因组中大量存在且均匀分布,因此群体样本数量可显著减小[33-34]。本研究中,5个乌鳢群体Ho均低于He,表明该群体存在一定程度的近亲繁殖。这一结果与Zhu等[22]、Yan等[24]、Zhou等[25]结果类似。Zhou等[25]研究的山东微山湖群体、黑龙江群体,以及张露等[23]研究的浙江湖州群体,Ho均低于He,结果与本研究类似。此外,本研究中5个乌鳢群体的杂合度、群体多态信息含量和核苷酸多态性较低,均处于较低水平(表4),表明这些群体的遗传多样性较为匮乏,这一发现与高志远[20]研究中黑龙江抚远和山东微山湖群体的遗传多样性较低的结果一致,也与周伟等[17]发现的乌鳢群体较低的核苷酸多样性水平类似。
3.2 群体遗传分化及结构
根据Wright的遗传分化系数,本研究中四川内江群体(NJH)与(NJB)之间的遗传分化系数为0.042,表明其亲缘关系较近。这与系统发育树和PCA分析的结果一致,显示两群体之间存在基因交流迹象,推测原因是四川内江两群体地理位置相近。Zhou等[12]通过分析线粒体的Cyt b基因、12S rRNA[18]、COI基因[11]和mtDNA[16]确定了内江乌鳢的遗传多样性和两种颜色形态的关系,表明内江群体中的普通乌鳢和白乌鳢在分子水平上应当视为同一物种,白乌鳢的体色呈白色原因可能是鱼类所生活的生态环境改变影响其皮肤黑色素沉着[35]。Fan等[13]和张露等[23]分别采用微卫星和mtDNA技术解析普通乌鳢与白乌鳢的遗传结构,发现远离内江的乌鳢与位于内江的白乌鳢群体间出现了极大的遗传分化水平(0.403>0.25),原因可能是地理位置相隔较远。本研究中,部分乌鳢样本来自远离四川内江水系的地区,如山东、湖南和浙江等地。在群体遗传分化水平上,地理位置相隔较近的四川内江白乌鳢与内江乌鳢群体分化水平较低;地理位置相隔较远的四川内江白乌鳢与其他地区乌鳢群体分化水平较高,这两个组别呈现出较大的差异水平,能够在其他研究中得到相似结果。山东群体(SD)与浙江群体(ZJ)、黑龙江群体(HLJ)、四川内江群体(NJH)和(NJB)之间的遗传分化系数为0.077、0.111、0.117、0.126,处于中度遗传分化水平。浙江群体(ZJ)与黑龙江群体(HLJ)、四川内江群体(NJH)和(NJB),它们的群体间分化系数分别为0.234、0.247、0.230,表明群体间高度遗传分化,亲缘关系较远,在系统发育树和PCA分析中也能观察到类似结果。各地区间的群体遗传分化水平均有不同,地理位置相近的群体分化水平较低,反之则较高[22-23,25],这与本研究结果中各地区群体之间遗传分化情况类似。张露等[23]通过微卫星标记发现,内江白乌鳢群体与内江乌鳢群体间的分化程度较高,与本研究中内江白乌鳢和内江乌鳢的遗传分化水平较低的结果有所差异,可能由于分子标记的方法不同,得到的遗传背景信息也不同。
本研究中群体间Nm均小于1,其中,四川内江(NJH)与(NJB)群体间的Nm最大,为0.856,表明各群体间基因交流较少,与Fst分析结果基本一致。5个乌鳢群体遗传分化水平不一,可能是群体之间存在空间隔离且不具备洄游特性,在一定程度上阻碍了种群间的基因交流。然而,在青鱼(Mylopharyngodon piceus)9个群体[36]的遗传变异分析中发现,群体间的遗传分化主要处于低度和中度分化水平,存在基因交流水平较低,这与本研究结果类似。此外,群体的聚类分析结果表明,除四川内江群体间存在基因交流外,其他群体均单独聚类,各群体分化明显且群体结构单一。在河川沙塘鳢(Odontobutis potamophila)[37]的4个地理群体(太湖、望江、当涂和休宁)遗传多样性和遗传结构研究中发现群体结构单一,与本研究结果相似。当K=4时,交叉验证错误率最低,81尾乌鳢个体有最佳聚类效果,5个乌鳢群体可分为4个亚群。四川内江(NJH)与(NJB)乌鳢群体的系统发育树分支关系与采样地理位置相异,虽然地理位置不同,但白乌鳢与普通乌鳢在形态特征、乳酸脱氢酶、酯酶同工酶和染色体类型进行比较,被认为是普通乌鳢的白化变种[16],因此将群体结构聚类分析划分成4个亚群。浙江群体聚成一簇,表现出一致的遗传背景,未发现基因交流痕迹,这可能是由于浙江群体采样数量较少,需要增加样本数量以进一步确认。其他地区群体中存在来自祖先亚群基因交流。系统发育树分析和主成分分析的聚类结果基本一致,四川内江群体(NJH)与(NJB)存在交叉聚类现象,其他地区均单独聚类。
4 结论
1)5个乌鳢群体的观测杂合度(Ho)为0.078~0.134,期望杂合度(He)为0.094~0.183,近交系数(FIS)为0.288~0.579,核苷酸多态性(π)为2.96×10-6~5.73×10-6,多态信息含量(PIC)为0.156~0.250,属于低水平多态性位点,表明5个乌鳢群体的遗传多样性水平较低。
2)乌鳢各群体之间基因流(Nm)为0.503~0.856,各群体间遗传分化系数(Fst)为0.042~0.247。四川内江(NJB)和(NJH)群体间处于低度遗传分化水平,其他群体间处于中度遗传分化水平以上,表明群体间遗传分化水平处于中低等水平。
3)5个乌鳢群体可分为4个地理群体,四川内江群体中乌鳢与白乌鳢为1个类群,其他地区黑龙江、山东和浙江分别单独为1个类群,形成了较为明显的遗传分化。
[1] 喻志新,张哲溶,张玥,等.乌鳢的药理作用研究进展[J].亚太传统医药,2021,17(1):183-186.
YU Z X,ZHANG Z R,ZHANG Y,et al.Research progress on pharmacological activity of Ophiocephalus argus[J].Asia-Pacific Traditional Medicine,2021,17(1):183-186.(in Chinese)
[2] 张新铖,陈昆慈,朱新平.乌鳢、斑鳢及杂交种养殖研究现状[J].广东农业科学,2011,38(22):132-134.
ZHANG X C,CHEN K C,ZHU X P.Status of research on the culture of Channa argus,Channa maculata and hybrid snakehead [J].Guangdong Agricultural Sciences,2011,38(22):132-134.(in Chinese)
[3] 刘亚楠,刘洁,魏上,等.克氏原螯虾养殖群体的SLAF测序及遗传多样性分析[J].南方农业学报,2021,52(12):3265-3273.
LIU Y N,LIU J,WEI S,et al.Analysis of genetic diversity among the farmed Procambarus clarkii populations using the SLAF-seq technology [J].Journal of Southern Agriculture,2021,52(12):3265-3273.(in Chinese)
[4] WANGHE K Y,FENG C G,TANG Y T,et al.Phylogenetic relationship and taxonomic status of Gymnocypris eckloni (Schizothoracinae) based on specific locus amplified fragments sequencing[J].Frontiers in Ecology and Evolution,2022,10:933632.
[5] RESH C A,GALASKA M P,MAHON A R.Genomic analyses of northern snakehead (Channa argus) populations in North America[J].PeerJ,2018,6:e4581.
[6] WEGLEITNER B,TUCKER A,CHADDERTON L,et al.Identifying the genetic structure of introduced populations of northern snakehead (Channa argus) in Eastern USA[J].Aquatic Invasions,2016,11(2):199-208.
[7] KAMRAN M,YAQUB A,MALKANI N,et al.Identification and phylogenetic analysis of Channa species from riverine system of Pakistan using COI gene as a DNA barcoding marker[J].Journal of Bioresource Management,2020,7(2):88-98.
[8] BAISVAR V S,SINGH M,KUMAR R.Population structuring of Channa striata from Indian waters using control region of mtDNA[J].Mitochondrial DNA Part A,DNA Mapping,Sequencing,and Analysis,2019,30(3):414-423.
[9] ABDULMALIK-LABE O P,QUILANG J P.Genetic diversity of lacustrine populations of the snakehead gudgeon Giuris laglaizei (Gobiiformes:Eleotridae) in the Philippines based on mitochondrial DNA control region[J].Biologia,2023,78(2):515-527.
[10] SAWASAWA W.Genetic diversity of endangered snakehead Channa lucius (cuvier,1831) in the Mekong delta inferred from ISSR markers[J].Asian Fisheries Science,2020,33(3):266-273.
[11] ZHOU A G,XIE S L,LIU S L,et al.Genetic diversity of northern snakehead (Channa argus) based on complete mitochondrial COI gene sequences[J].Mitochondrial DNA Part B,2019,4(1):599-602.
[12] ZHOU A G,XIE S L,WANG Z L,et al.Genetic diversity and geographic differentiation in northern snakehead (Channa argus) based on mitochondrial cyt b gene[J].Pakistan Journal of Zoology,2018,51(1): 359-362.
[13] FAN W,ZHANG L,SU J,et al.Genetic diversity of two color morphs of northern snakehead (Channa argus) unveiled by the mitochondrial DNA D-loop region[J].Mitochondrial DNA Part B,Resources,2022,7(3):515-520.
[14] 肖明松,崔峰,康健,等.淮河乌鳢线粒体DNA控制区结构分析及遗传多样性研究[J].华中师范大学学报(自然科学版),2013,47(1):82-90.
XIAO M S,CUI F,KANG J,et al.Genetic structure and variation of wild Ophicephalus Argus cantor from Huaihe River based on MtDNA D-loop sequences[J].Journal of Central China Normal University (Natural Sciences Edition),2013,47(1):82-90.(in Chinese)
[15] 董新培,穆淑梅,周楠,等.不同地理群体乌鳢线粒体DNA控制区结构分析及遗传多样性[J].水产学报,2014,38(9):1277-1285.
DONG X P,MU S M,ZHOU N,et al.Structure analysis of mtDNA D-Loop region and the genetic diversity of Channa argus in different populations[J].Journal of Fisheries of China,2014,38(9):1277-1285.(in Chinese)
[16] ZHOU A G,SUN D,LIU S L,et al.The species identity of the two color morphs of northern snakehead (Channa argus) based on mtDNA control region sequences[J].Pakistan Journal of Zoology,2021,53(4):1591-1594.
[17] 周伟,高天翔,王俊,等.乌鳢群体遗传多样性和遗传结构分析[J].水产学报,2017,41(10):1521-1532.
ZHOU W,GAO T X,WANG J,et al.Genetic diversity and structure analysis based on the mitochondrial DNA control region of the northern snakehead (Channa argus) [J].Journal of Fisheries of China,2017,41(10):1521-1532.(in Chinese)
[18] ZHOU A G,XIE S L,WANG Z L,et al.Molecular evidence of two colour morphs of northern snakehead (Channa argus) based on mitochondrial 12S rRNA[J].Mitochondrial DNA Part B,Resources,2017,2(1):283-286.
[19] ZHOU A G,ZHUO X L,ZOU Q,et al.Population genetic diversity of the northern snakehead (Channa argus) in China based on the mitochondrial DNA control region and adjacent regions sequences[J].Mitochondrial DNA,2015,26(3):341-349.
[20] 高志远.中国野生乌鳢(Channa argus)遗传多样性分析[D].广州:暨南大学,2013.
GAO Z Y.Genetic diversity analysis of wild Channa argus in China[D].Guangzhou:Jinan University,2013.(in Chinese)
[21] XIAO M,BAO F,ZHAO Y,et al.Development of genetic novel SSR markers by transcriptome sequencing in ophicephalus Argus cantor[J].Russian Journal of Genetics,2020,56(2):253-260.
[22] ZHU S R,LI J L,XIE N,et al.Genetic diversity based on SSR analysis of the cultured snakehead fish,Channa argus(Channidae) in China[J].Genetics and Molecular Research:GMR,2014,13(3):8046-8054.
[23] 张露,樊威,苏建,等.基于微卫星标记的不同地区乌鳢和白化乌鳢群体遗传多样性研究[J].西南农业学报,2022,35(6):1455-1461.
ZHANG L,FAN W,SU J,et al. Genetic diversity of northern snakehead (Channa argus) and Albino northern snakehead from different regions unveiled by the microsatellite markers [J].Southwest China Journal of Agricultural Sciences,2022,35(6):1455-1461.(in Chinese)
[24] YAN R J,ZHANG G R,GUO X Z,et al.Genetic diversity and population structure of the northern snakehead (Channa argus Channidae:Teleostei) in central China:implications for conservation and management[J].Conservation Genetics,2018,19(2):467-480.
[25] ZHUO X L,LIANG R S,CHEN Y F,et al.Genetic characterization of northern snakehead (Channa argus) populations in China using microsatellite markers[J].Biochemical Systematics and Ecology,2012,43:25-31.
[26] ZHOU A,LÜ Z,XIE S,et al.Isolation and characterisation of polymorphic microsatellite markers in the progeny of nine mating groups of snakeheads[J].Indian Journal of Fisheries,2015,62(2):33-38.
[27] ZHOU A,ZHUO X,LUO J,et al.Investigations on genetic diversity of northern snakehead (Channa argus) populations in China using amplified fragment length polymorphism (AFLP) markers[J].Indian Journal of Fisheries,2015,62(3)29-42.
[28] 张伯池.基于转录组测序的乌鳢SNP标记开发及其群体遗传多样性研究[D].武汉:华中农业大学,2017.
ZHANG B C.Development of SNP markers and study on population genetic diversity of Ophiocephalus argus based on transcriptome sequencing[D].Wuhan:Huazhong Agricultural University,2017.(in Chinese)
[29] 郑合家.山东省5个乌鳢地理群体的形态性状和遗传结构分析[D].上海:上海海洋大学,2023.
ZHENG H J.Morphological characters and genetic structure analysis of five geographical populations of snakehead in Shandong Province[D].Shanghai:Shanghai Ocean University,2023.(in Chinese)
[30] VAN INGHELANDT D,MELCHINGER A E,LEBRETON C,et al.Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers[J].TAG Theoretical and Applied Genetics Theoretische Und Angewandte Genetik,2010,120(7):1289-1299.
[31] 戴习林,刘洁,李晶晶,等.罗氏沼虾种群SSR分析中样本量及标记量对遗传多样性指标的影响[J].水产学报,2017,41(7):1083-1095.
DAI X L,LIU J,LI J J,et al.Effects of sample size and loci number on genetic diversity index in Macrobrachium rosenbergii with microsatellite markers[J].Journal of Fisheries of China,2017,41(7):1083-1095.(in Chinese)
[32] 刘改艳.乌鳢、斑鳢性别差异SSR标记的初步筛选及其遗传多样性分析[D].上海:上海海洋大学,2011.
LIU G Y.Gender differences in the preliminary screening with SSR and genetic diversity of Channa argus and Channa maculata [D].Shanghai:Shanghai Ocean University,2011.(in Chinese)
[33] TRASK J A,MALHI R S,KANTHASWAMY S,et al.The effect of SNP discovery method and sample size on estimation of population genetic data for Chinese and Indian rhesus macaques (Macaca mulatta)[J].Primates;Journal of Primatology,2011,52(2):129-138.
[34] NAZARENO A G,BEMMELS J B,DICK C W,et al.Minimum sample sizes for population genomics:an empirical study from an Amazonian plant species[J].Molecular Ecology Resources,2017,17(6):1136-1147.
[35] JIANG Y L,ZHANG S H,XU J,et al.Comparative transcriptome analysis reveals the genetic basis of skin color variation in common carp[J].PLoS One,2014,9(9):e108200.
[36] 鲍生成,包天杰,王沈同,等.基于线粒体COⅠ基因的9个青鱼群体遗传变异分析[J].水生生物学报,2022,46(7):933-938.
BAO S C,BAO T J,WANG S T,et al. Genetic diversity of mitochondrial CO I gene in populations of black carp from the Yangtze River and Pearl River [J].Acta Hydrobiologica Sinica,2022,46(7):933-938.(in Chinese)
[37] 张烨,凌俊,段国庆,等.安徽河川沙塘鳢不同地理群体遗传多样性和遗传结构研究[J].安徽农业大学学报,2019,46(3):383-388.
ZHANG Y,LING J,DUAN G Q,et al.Genetic diversity and population structure analyses of Odontobutis potamophila from different geographic populations in Anhui Province[J].Journal of Anhui Agricultural University,2019,46(3):383-388.(in Chinese)