鰤鱼诺卡氏菌(Nocardia seriolae)是鱼类诺卡氏菌病的主要病原[1],为好气菌、革兰氏阳性菌,大部分无气丝,部分有气生菌丝体,基丝分枝,横隔断裂成杆状体和球状体,分类学上属细菌域(Bacteria)厚壁菌门(Firmicutes)放线菌纲(Actinobacteria)放线菌目(Actinobacterales)诺卡氏菌科(Nocardiaceae)诺卡氏菌属(Nocardia)[2]。鱼类诺卡氏菌病主要的病理变化为内脏器官形成直径为1~5 mm黄色或白色结节(肉芽肿),有的病鱼鳃盖内缘或鳃丝上出现结节、腹腔内有纤维瘤、皮肤溃疡[3]。鰤鱼诺卡氏菌于1968年首次由Kariya等在日本五条鰤(Seriola quinqueradiata)和褐牙鲆(S.purpurascens)身上分离出来[4]。自20世纪90年代以来,鰤鱼诺卡氏菌感染暴发频繁,对全球水产养殖业带来的危害日益严重。据不完全统计,发病鱼种包括乌鳢(Channa argus)[5]、大口黑鲈(Micropterus salmoides)[6]、大黄鱼(Larimichthys crocea)[7]、鳗(Anguilla japonica)[8]和卵形鲳鲹(Trachinotus ovatus)[9]等约42种淡水与海水鱼类,故亟需开展鰤鱼诺卡氏菌相关研究以抑制鱼类诺卡氏菌病的流行。
基于鰤鱼诺卡氏菌不同菌株的形态特征、生理生化特征、抗生素敏感性、基因序列和基因检测等,已有不少报道对鰤鱼诺卡氏菌的表型和基因型多样性开展相关研究。如Shimahara等[10-11]对1970—2005年日本分离的58株鰤鱼诺卡氏菌和1997—2007年台湾分离到的92株鰤鱼诺卡氏菌进行表型和遗传特征研究,将其分为20多个基因型,日本分离株和台湾分离株的基因型和表型具有较大差异。满其蒙等[12]对2006—2012年从华南分离的15株鰤鱼诺卡氏菌进行了聚类分析,将其分成了多个不同亚群,表明鰤鱼诺卡氏菌具有丰富表型多样性。同时,发现其亚群划分与分离地点和宿主无明显的相关性。罗愿等[13]对2013—2017年从华南分离到的9株鰤鱼诺卡氏菌的生长、致病性、毒力基因和耐药性基因等进行了比较分析,结果表明,这9株菌株在生长特性、致病性和耐药性方面具有高度相似性。总的来说,鰤鱼诺卡氏菌菌株的表型和基因型随着分离地区和年份的不同存在较大差异,因此在进行疫苗研究时,应通过菌株分型确定流行株再用于疫苗开发。目前,对鰤鱼诺卡氏菌进行菌株分型尚处于初级阶段,如何划分鰤鱼诺卡氏菌的菌株类型及该菌主要包含哪些菌株类型均有待进一步研究。
多重PCR也称复合PCR,其基本原理与常规PCR相同,多重PCR是一种特殊的PCR技术,与常规PCR的主要区别在于,多重PCR在同一反应体系中会加入两对或以上的引物,因此其可以一次扩增多个靶位点,实现多个位点的基因型鉴别[14]。理论上,只要PCR扩增条件合适,引物对的数量可以不限,且多重PCR通过优化反应体系和反应条件可减小假阳性的现象。目前,尚未见到多重PCR方法对鰤鱼诺卡氏菌进行分型。
目前,菌株分型的鉴定方法主要包括检测血清型、生理生化特征、抗生素敏感性等指标,以及基因组测序和特殊基因的分子检测等。传统的细菌分型方法,如生化分型需要耗费大量的时间与人工成本[15],血清分型存在制备抗血清的步骤复杂、检测过程耗时长、易出现交叉反应等缺陷[16],均无法实现对细菌的快速分型鉴定,一定程度上不利于对病原菌进行及时防治。因此,建立一种快速精准的鰤鱼诺卡氏菌菌株分型方法,对于鱼类诺卡氏菌病防治极为重要。本研究中拟应用多重PCR技术,建立一种高效准确的鰤鱼诺卡氏菌菌株分型方法,以期为鱼类鰤鱼诺卡氏菌病的防控提供参考。
1 材料与方法
1.1 材料
29株鰤鱼诺卡氏菌及其来源见表1。菌株SZ0214、WL-2021-1、WL-2020-1、HB202008、FS201912、JZL-2019、ZJ0503、WL-2023-11由本实验室分离保藏,菌株N-1、N-2、N-3、N-4、N-5、N-6、N-8、N-9、N-11、N-12、N-13、N-14、N-15、N-16、N-17、N-18、N-19、N-20、N-21、N-22、N-23由珠海市现代农业技术发展中心赠送。
表1 29株鰤鱼诺卡氏菌及其来源
Tab.1 29 strains of Nocardia seriolae and their origin
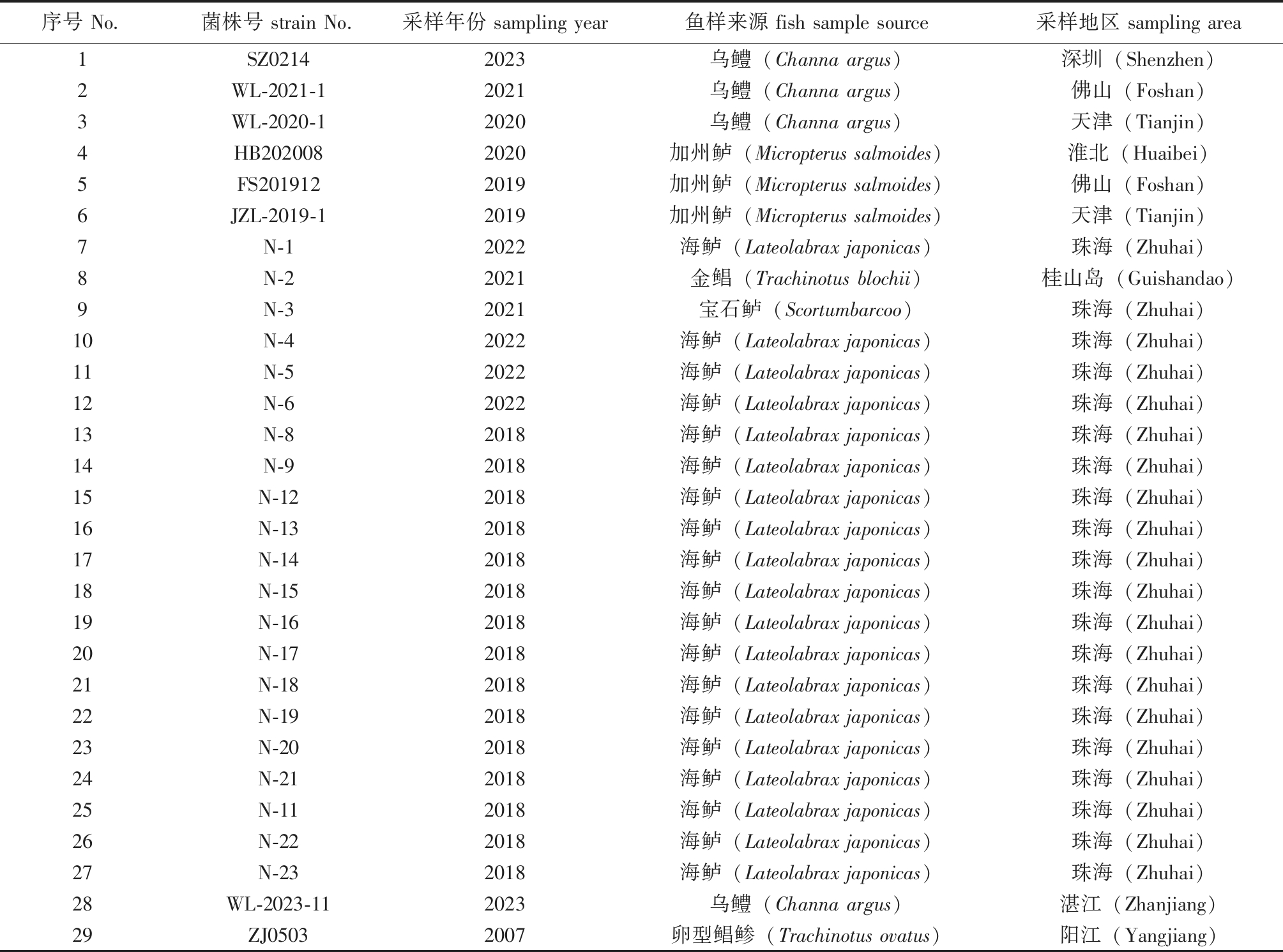
序号 No.菌株号 strain No.采样年份 sampling year鱼样来源 fish sample source采样地区 sampling area1SZ02142023乌鳢(Channa argus)深圳(Shenzhen)2WL-2021-12021乌鳢(Channa argus)佛山(Foshan)3WL-2020-12020乌鳢(Channa argus)天津(Tianjin)4HB2020082020加州鲈(Micropterus salmoides)淮北(Huaibei)5FS2019122019加州鲈(Micropterus salmoides)佛山(Foshan)6JZL-2019-12019加州鲈(Micropterus salmoides)天津(Tianjin)7N-12022海鲈(Lateolabrax japonicas)珠海(Zhuhai)8N-22021金鲳(Trachinotus blochii)桂山岛(Guishandao)9N-32021宝石鲈(Scortumbarcoo)珠海(Zhuhai)10N-42022海鲈(Lateolabrax japonicas)珠海(Zhuhai)11N-52022海鲈(Lateolabrax japonicas)珠海(Zhuhai)12N-62022海鲈(Lateolabrax japonicas)珠海(Zhuhai)13N-82018海鲈(Lateolabrax japonicas)珠海(Zhuhai)14N-92018海鲈(Lateolabrax japonicas)珠海(Zhuhai)15N-122018海鲈(Lateolabrax japonicas)珠海(Zhuhai)16N-132018海鲈(Lateolabrax japonicas)珠海(Zhuhai)17N-142018海鲈(Lateolabrax japonicas)珠海(Zhuhai)18N-152018海鲈(Lateolabrax japonicas)珠海(Zhuhai)19N-162018海鲈(Lateolabrax japonicas)珠海(Zhuhai)20N-172018海鲈(Lateolabrax japonicas)珠海(Zhuhai)21N-182018海鲈(Lateolabrax japonicas)珠海(Zhuhai)22N-192018海鲈(Lateolabrax japonicas)珠海(Zhuhai)23N-202018海鲈(Lateolabrax japonicas)珠海(Zhuhai)24N-212018海鲈(Lateolabrax japonicas)珠海(Zhuhai)25N-112018海鲈(Lateolabrax japonicas)珠海(Zhuhai)26N-222018海鲈(Lateolabrax japonicas)珠海(Zhuhai)27N-232018海鲈(Lateolabrax japonicas)珠海(Zhuhai)28WL-2023-112023乌鳢(Channa argus)湛江(Zhanjiang)29ZJ05032007卵型鲳鲹(Trachinotus ovatus)阳江(Yangjiang)
鰤鱼诺卡氏菌采用脑心浸液肉汤 (BHI) 培养基 28 ℃下培养。细菌全基因组DNA提取试剂盒与动物组织DNA提取试剂盒购自TIANGEN公司。Premix TaqTM PCR预混液购自TaKaRa公司。引物由生工生物工程(上海)股份有限公司合成。PCR仪为耶拿分析仪器公司。
1.2 方法
1.2.1 筛选种内差异的基因片段并设计PCR引物 采用 NCBI (http://www.ncbi.nlm.nih.gov)的BLAST程序对已公布的鰤鱼诺卡氏菌20个菌株的全基因组数据(表2)进行序列比对分析,筛选获得81个候选种内差异基因片段,即存在于部分鰤鱼诺卡氏菌菌株基因组,但不存在于所有鰤鱼诺卡氏菌菌株的基因片段。本研究中对其中的53个候选基因片段进行了单重PCR鉴定,鉴于大部分基因在本实验室24个菌株(HB202008、FS201912、WL-2021-1、N-2,N-1、WL-2020-1、N-4、JZL-2019-1、SZ0214,N-3、N-6、N-5,N-8、N-9、N-12、N-13、N-14、N-15、N-16、N-17、N-18、N-19、N-20、N-21)PCR验证结果均显示为无种内差异,因此在本文中仅展示10个候选种内差异基因片段的筛选过程。针对这10个候选种内差异基因片段(PCCB、TerC、ArsC、AraC、ISOPREN、YbaB、ALAT、dhaK、IclR 、TetR)设计特异性PCR引物(表3)。加上文献报道的在鰤鱼诺卡氏菌不同菌株中具有种内差异的dop毒力基因及其PCR引物(表3),一并由生工生物工程(上海)股份有限公司合成。
表2 20株鰤鱼诺卡氏菌全基因组
Tab.2 Complete genome data of 20 strains of Nocardia seriolae
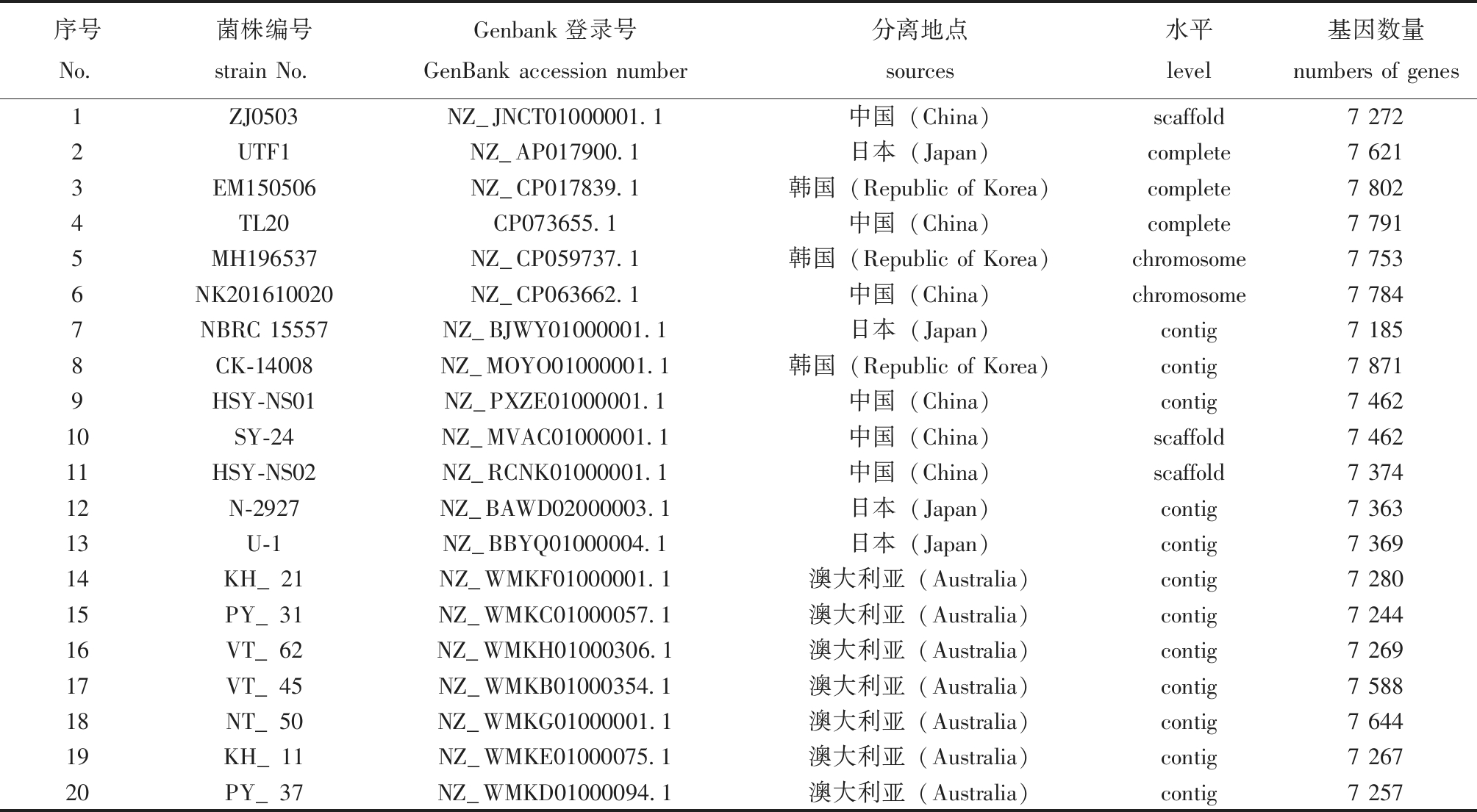
序号No.菌株编号strain No.Genbank登录号GenBank accession number分离地点sources水平level基因数量numbers of genes1ZJ0503NZ_JNCT01000001.1中国(China)scaffold7 2722UTF1NZ_AP017900.1日本(Japan)complete7 6213EM150506NZ_CP017839.1韩国(Republic of Korea)complete7 8024TL20CP073655.1中国(China)complete7 7915MH196537NZ_CP059737.1韩国(Republic of Korea)chromosome7 7536NK201610020NZ_CP063662.1中国(China)chromosome7 7847NBRC 15557NZ_BJWY01000001.1日本(Japan)contig7 1858CK-14008NZ_MOYO01000001.1韩国(Republic of Korea)contig7 8719HSY-NS01NZ_PXZE01000001.1中国(China)contig7 46210SY-24NZ_MVAC01000001.1中国(China)scaffold7 46211HSY-NS02NZ_RCNK01000001.1中国(China)scaffold7 37412N-2927NZ_BAWD02000003.1日本(Japan)contig7 36313U-1NZ_BBYQ01000004.1日本(Japan)contig7 36914KH_21NZ_WMKF01000001.1澳大利亚(Australia)contig7 28015PY_31NZ_WMKC01000057.1澳大利亚(Australia)contig7 24416VT_62NZ_WMKH01000306.1澳大利亚(Australia)contig7 26917VT_45NZ_WMKB01000354.1澳大利亚(Australia)contig7 58818NT_50NZ_WMKG01000001.1澳大利亚(Australia)contig7 64419KH_11NZ_WMKE01000075.1澳大利亚(Australia)contig7 26720PY_37NZ_WMKD01000094.1澳大利亚(Australia)contig7 257
表3 11对目的基因引物
Tab.3 11 pairs of target gene primer
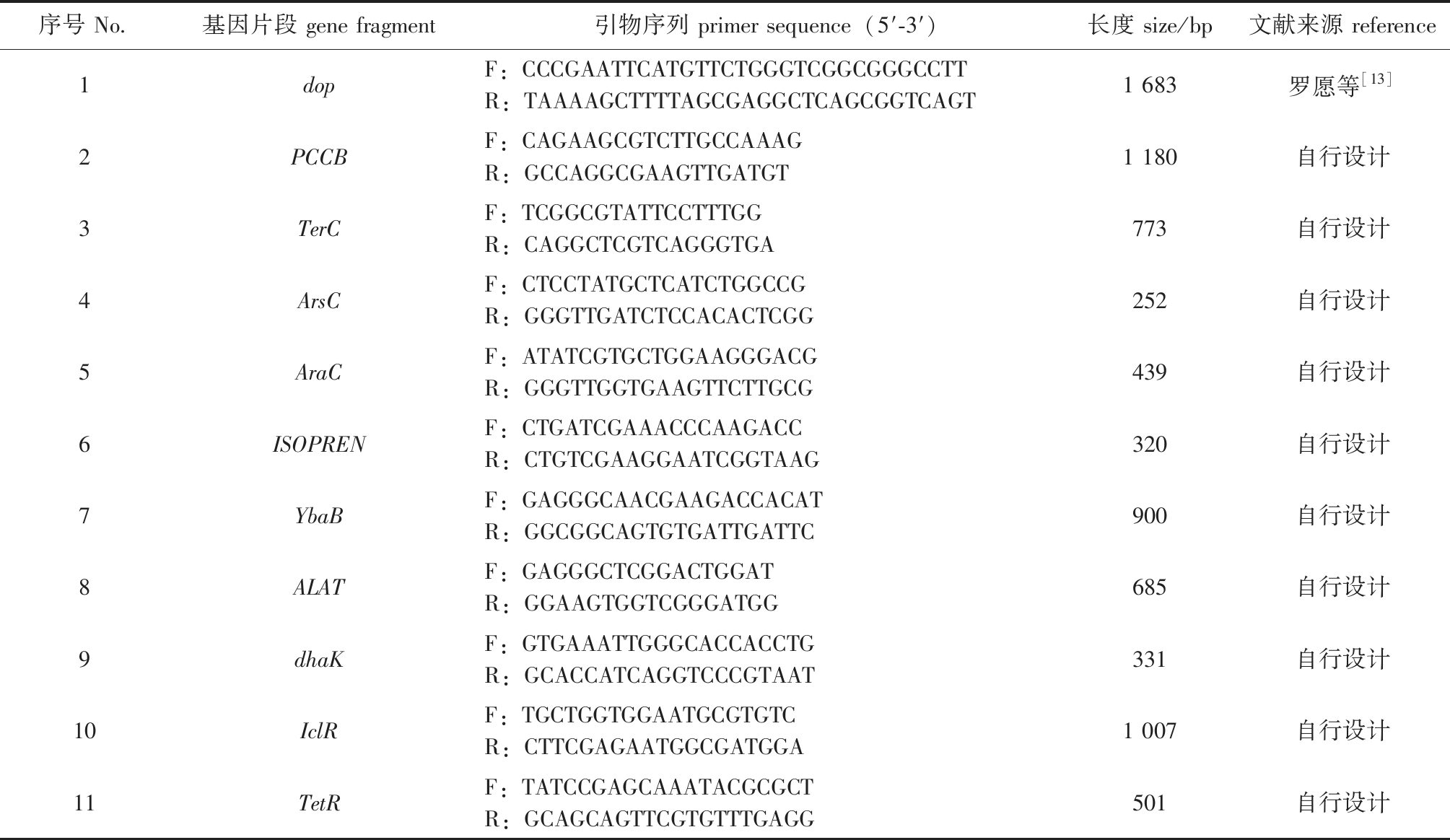
序号 No.基因片段 gene fragment引物序列 primer sequence(5′-3′)长度 size/bp文献来源 reference1dopF:CCCGAATTCATGTTCTGGGTCGGCGGGCCTTR:TAAAAGCTTTTAGCGAGGCTCAGCGGTCAGT1 683罗愿等[13]2PCCBF:CAGAAGCGTCTTGCCAAAGR:GCCAGGCGAAGTTGATGT1 180自行设计3TerCF:TCGGCGTATTCCTTTGGR:CAGGCTCGTCAGGGTGA773自行设计4ArsCF:CTCCTATGCTCATCTGGCCGR:GGGTTGATCTCCACACTCGG252自行设计5AraCF:ATATCGTGCTGGAAGGGACGR:GGGTTGGTGAAGTTCTTGCG439自行设计6ISOPRENF:CTGATCGAAACCCAAGACCR:CTGTCGAAGGAATCGGTAAG320自行设计7YbaBF:GAGGGCAACGAAGACCACATR:GGCGGCAGTGTGATTGATTC900自行设计8ALATF:GAGGGCTCGGACTGGATR:GGAAGTGGTCGGGATGG685自行设计9dhaKF:GTGAAATTGGGCACCACCTGR:GCACCATCAGGTCCCGTAAT331自行设计10IclRF:TGCTGGTGGAATGCGTGTCR:CTTCGAGAATGGCGATGGA1 007自行设计11TetRF:TATCCGAGCAAATACGCGCTR:GCAGCAGTTCGTGTTTGAGG501自行设计
1.2.2 引物筛选、多重PCR引物组合筛选 参考陈国权等[17]筛选获得的种间特异、种内保守的鰤鱼诺卡氏菌特异性基因片段及其设计的特异性659-F/R引物(扩增片段大小为566 bp),分别以鰤鱼诺卡氏菌菌株的DNA为模板进行单重PCR扩增,用以鉴定用于引物筛选的24个菌株(HB202008、FS201912、WL-2021-1、N-2,N-1、WL-2020-1、N-4、JZL-2019-1、SZ0214,N-3、N-6、N-5,N-8、N-9、N-12、N-13、N-14、N-15、N-16、N-17、N-18、N-19、N-20、N-21)为鰤鱼诺卡氏菌。
通过单重PCR,可确定11对引物(表3)的扩增条带是否具备单一性,能否体现不同来源的差异性。PCR反应条件为95 ℃下变性5 min;95 ℃下变性30 s,60 ℃下退火30 S,72 ℃下延伸1 min,共进行30个循环,最后在72 ℃下再延伸10 min。设置20 μL PCR反应体系:Premix TaqTM PCR预混液10 μL、表3中的引物对(浓度100 ng/μL)各1 μL、DNA模板1 μL、用ddH2O补足至20 μL。PCR反应结束后进行琼脂糖凝胶电泳。筛选出可扩增出单一、种内差异的PCR引物对后,设置50 μL PCR反应体系Premix TaqTM PCR预混液25 μL,总引物15 μL(3对候选引物等比例加入),DNA模板为5 μL,用ddH2O补足至50 μL。PCR反应程序为95 ℃下预变性5 min;95 ℃下变性30 s,60 ℃下退火30 s,72 ℃下延伸1 min,共进行30个循环;最后在72 ℃下延伸10 min。将扩增片段长度具有差异的引物进行组合,根据扩增效果进行筛选,既能体现不同来源菌株的差异性,又不能出现不合理的扩增条带缺失。
1.2.3 多重PCR引物对比例优化 根据“1.2.2节”中试验结果,选定扩增效果较好的多重PCR候选引物对组合,设置50 μL PCR反应体系:Premix TaqTM PCR预混液25 μL,总引物为15 μL(引物对1∶引物对2∶引物对3为1∶1∶1),DNA模板为5 μL,用ddH2O补足50 μL。PCR反应程序为95 ℃下预变性5 min;95 ℃下变性30 s,60 ℃下退火30 s,72 ℃下延伸1 min,共进行30个循环;最后在72 ℃下再延伸10 min,检测引物对1∶引物对2∶引物对3为1∶1∶1时的扩增效果。根据等比例引物配比的扩增效果,适当增加弱带的引物配比比例后,再次进行扩增。以目的片段均能扩增出单一清晰条带且不产生非特异性条带作为优化引物配比的指标。
1.2.4 多重PCR的灵敏程度检测 用ddH2O稀释鰤鱼诺卡氏菌DNA,制成8个鰤鱼诺卡氏菌 DNA浓度梯度,该菌 DNA 浓度分别为30 ng/μL、6 ng/μL、1 ng/μL、500 pg/μL、250 pg/μL、125 pg/μL、62.5 pg/μL、31.3 pg/μL。按照优化后的引物配比,采用不同浓度的鰤鱼诺卡氏菌 DNA模板,以ddH2O为空白对照,检测多重PCR的灵敏度。
1.2.5 多重PCR对鰤鱼诺卡氏菌培养菌落的菌株分型应用 29株鰤鱼诺卡氏菌(表1)接种至液体培养基进行扩培,随后使用BHI固体培养基 28 ℃下培养60~72 h后,挑取颗粒状凹起状、表面形成褶皱的单菌落,采用细菌全基因组DNA提取试剂盒(Tiangen)提取DNA,根据“1.2.4节”配比指标的优化多重PCR循环参数,使用引物组合(ISOPREN、ALAT、IclR ),对29株菌株进行分型,采用建立的鰤鱼诺卡氏菌多重PCR菌株分型方法对鰤鱼诺卡氏菌培养菌落进行应用。
1.2.6 多重PCR对鰤鱼诺卡氏菌感染鱼样的菌株分型应用 试验用鱼为杂交鳢,攻毒前适应养殖7 d。用培养的鰤鱼诺卡氏菌ZJ0503菌株菌液腹腔注射健康的杂交鳢,剂量参考王文基等[1]的LD50浓度进行攻毒,注射量为100 μL,菌液浓度为1×108 CFU/mL。攻毒后连续观察10 d,分别取5尾有明显发病症状的杂交鳢和两尾未攻毒健康杂交鳢的肝脏、脾脏和头肾,采用动物组织DNA提取试剂盒(Tiangen)提取组织DNA,采用引物659-F/R验证病鱼为鰤鱼诺卡氏菌感染,采用建立的鰤鱼诺卡氏菌多重PCR菌株分型方法对病鱼组织内的鰤鱼诺卡氏菌进行应用。
2 结果与分析
2.1 鰤鱼诺卡氏菌的PCR引物的鉴定、筛选
采用特异性引物659-F/R对24株鰤鱼诺卡氏菌菌株进行PCR扩增,PCR结束后进行琼脂糖凝胶电泳检测,结果如图1所示。从图1可见,24株菌株均能扩增出清晰条带,且片段大小与预期的片段大小一致(566 bp),表明24株菌株均为鰤鱼诺卡氏菌。
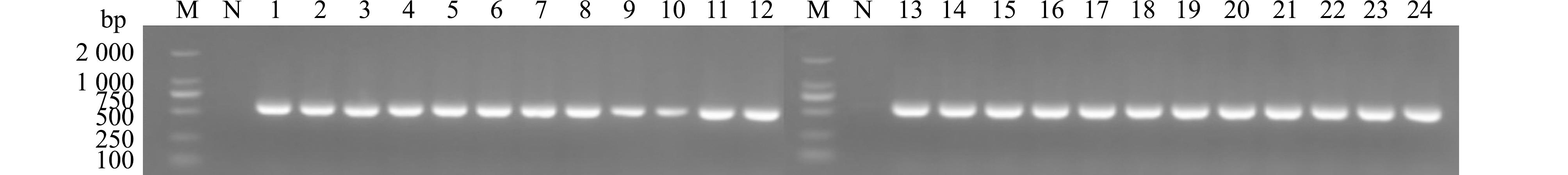
M—DL2000 DNA marker;N—空白对照;1~24对应的鰤鱼诺卡氏菌菌株为HB202008,FS201912,WL-2021-1,N-2,N-1,WL-2020-1,N-4,JZL-2019-1,SZ0214,N-3,N-6,N-5,N-8,N-9,N-12,N-13,N-14,N-15,N-16,N-17,N-18,N-19,N-20,N-21。
M—DL2000 DNA marker;N—blank control;1-24 the corresponding Nocardia seriolae strains are HB202008,FS201912,WL-2021-1,N-2,N-1,WL-2020-1,N-4,JZL-2019-1,SZ0214,N-3,N-6,N-5,N-8,N-9,N-12,N-13,N-14,N-15,N-16,N-17,N-18,N-19,N-20,and N-21.
图1 特异性引物659的PCR扩增
Fig. 1 PCR amplification of specific primers 659
采用引物dop-F/R、PCCB-F/R、TerC-F/R、ArsC-F/R、AraC-F/R、ISOPREN-F/R、YbaB-F/R、ALAT-F/R、dhaK-F/R、IclR-F/R、TetR-F/R,分别以24株鰤鱼诺卡氏菌的DNA为模板进行单重PCR扩增,结果如图2所示。从图2可知,单重PCR检测结果显示,24株鰤鱼诺卡氏菌均携带PCCB、TerC、YbaB、dhaK、TetR和dop基因片段,而ArsC、AraC和ISOPREN基因片段只在HB202008、FS201912及N-14 3个菌株中检测到,ALAT基因片段只在18株菌株中检测到,IclR基因片段只在17株菌株中检测到(表4)。

M—DL2000 DNA分子标准;N—空白对照;1~24对应的鰤鱼诺卡氏菌菌株顺序同图1。
M—DL2000 DNA marker;N—blank control;1-24 are corresponding Nocardia seriolae strains with the rank orders as same as those in Fig.1.
图2 11对引物单重PCR扩增结果
Fig. 2 Simplex PCR amplification for 11 pairs of primers
表4 24株鰤鱼诺卡氏菌菌株单重PCR扩增结果
Tab.4 Results of simplex PCR amplification of 24 Nocardia seriolae strains
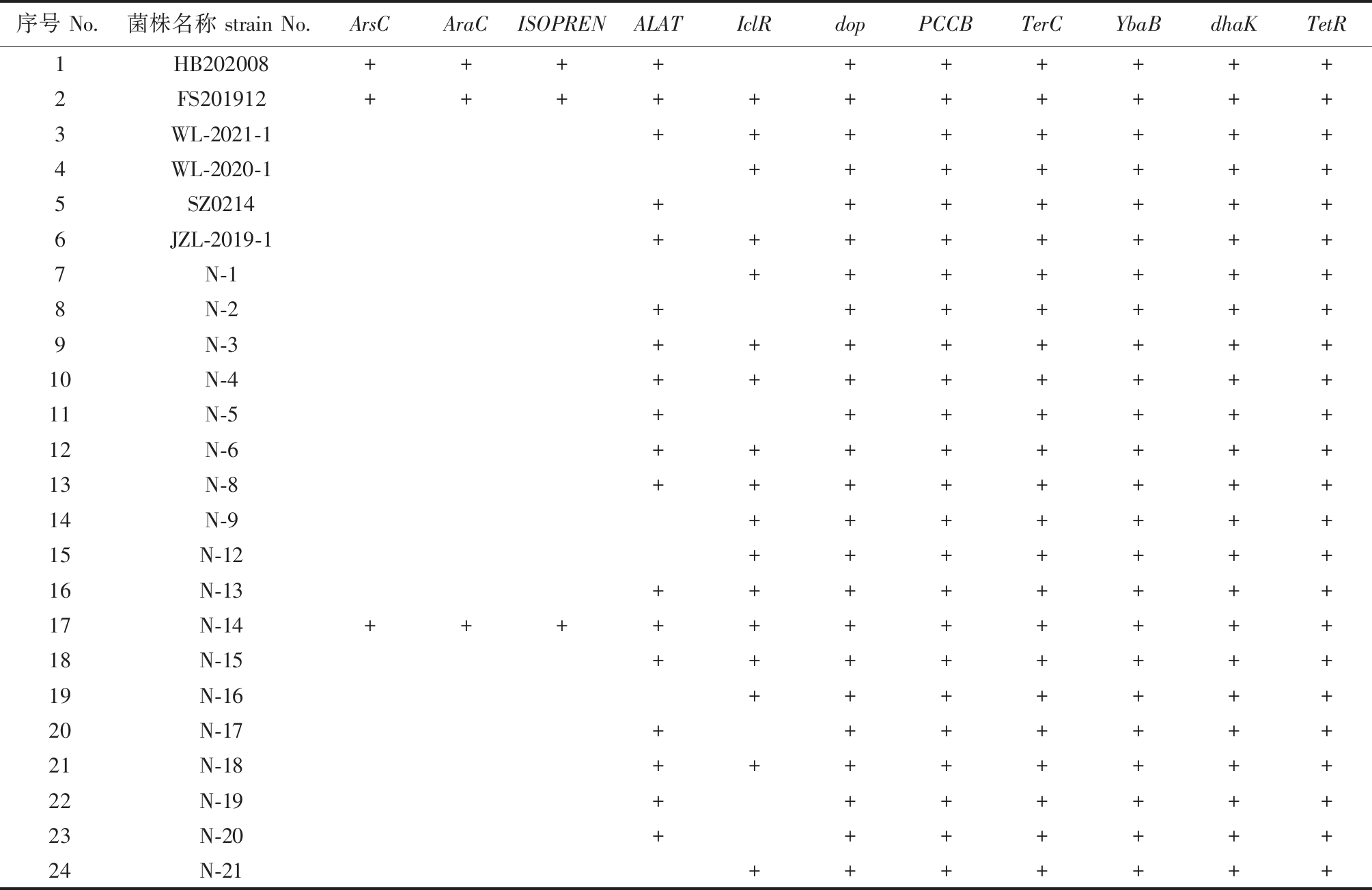
序号 No.菌株名称 strain No.ArsCAraCISOPRENALATIclRdopPCCBTerCYbaBdhaKTetR1HB202008++++++++++2FS201912+++++++++++3WL-2021-1++++++++4WL-2020-1+++++++5SZ0214+++++++6JZL-2019-1++++++++7N-1+++++++8N-2+++++++9N-3++++++++10N-4++++++++11N-5+++++++12N-6++++++++13N-8++++++++14N-9+++++++15N-12+++++++16N-13++++++++17N-14+++++++++++18N-15++++++++19N-16+++++++20N-17+++++++21N-18++++++++22N-19+++++++23N-20+++++++24N-21+++++++
注:+表示目的基因检测阳性。
Notes:+ means target gene which is tested as positive.
2.2 多重PCR体系的引物组合筛选
根据“2.1节”中单重PCR试验结果,ArsC、AraC、ISOPREN、ALAT和IclR 基因片段具有种内差异。根据其引物扩增片段在各菌株的扩增情况及扩增片段长度差异,以引物ISOPREN-F/R、引物ALAT-F/R、引物IclR-F/R作为多重PCR分型方法建立了引物组合Ⅰ。分别以24株鰤鱼诺卡氏菌的DNA为模板进行多重PCR的扩增,检测结果如图3所示。从图3可见,各菌株均能扩增出合理的条带,即在单重PCR体系中检测呈阳性的基因片段在多重PCR体系中均能扩增出相应的条带,在单重PCR体系中目的基因检测阴性的基因片段在多重PCR体系中均未扩增出条带。各菌株对应的菌株分型见表5。由于ArsC、AraC和ISOPREN基因片段的PCR扩增呈现相同的菌株分布,引物组合Ⅱ(AraC、ALAT、IclR )和组合Ⅲ(ArsC、ALAT、IclR)的多重PCR扩增结果与引物组合Ⅰ相同。表明所设计的3组引物组合均可用于建立鰤鱼诺卡氏菌菌株分型的多重PCR。
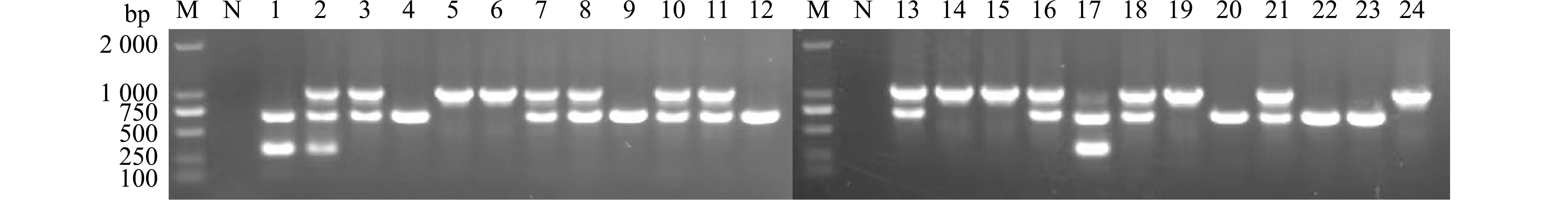
M—DL2000 DNA分子标准;N—空白对照;1~24对应的鰤鱼诺卡氏菌菌株同图1。
M—DL2000 DNA marker;N—blank control;1-24 are corresponding Nocardia seriolae strains with the rank order as same as that in Fig 1.
图3 引物组合Ⅰ(ISOPREN、ALAT、IclR)多重PCR扩增结果
Fig.3 Results of multiplex PCR amplification of primer combination Ⅰ (ISOPREN,ALAT,IclR)
表5 24株鰤鱼诺卡氏菌菌株对应的菌株分型
Tab.5 Corresponding strain typing of 24 Nocardia seriolae strains
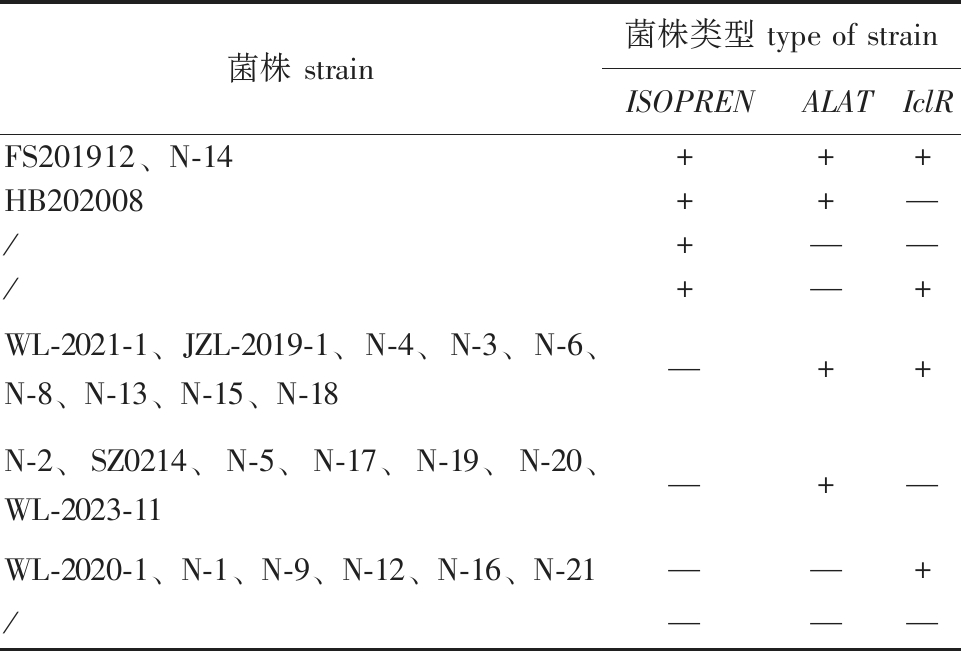
菌株 strain菌株类型 type of strainISOPRENALATIclRFS201912、N-14+++HB202008++—/+——/+—+WL-2021-1、JZL-2019-1、N-4、N-3、N-6、N-8、N-13、N-15、N-18—++N-2、SZ0214、N-5、N-17、N-19、N-20、WL-2023-11—+—WL-2020-1、N-1、N-9、N-12、N-16、N-21——+/———
注:+表示有扩增片段,—表示无扩增片段,下同。
Note:+means amplified fragments;—means no amplified fragments,et sequentia.
2.3 多重PCR引物配比优化及反应条件参数的确立
2.3.1多重PCR引物配比的优化 根据“2.2节”中多重PCR试验结果,使用引物组合Ⅰ(ISOPREN、ALAT、IclR)、组合Ⅱ(AraC、ALAT、IclR)和组合Ⅲ(ArsC、ALAT、IclR),两株鰤鱼诺卡氏菌FS201912和N-14均能扩增出3条目的基因片段,因此选定上述两个菌株作为优化多重PCR体系引物配比的菌株。多重PCR反应体系与反应条件见“2.4节”。结果显示,当引物组合Ⅰ(ISOPREN、ALAT、IclR)总引物浓度比值为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=1∶1∶1时,IclR基因片段只能检测出一条很淡的荧光条带(图4(a))。调整3对引物配比为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=1∶1∶2时,FS201912菌株3个目的基因片段均不能检测出条带(图4(b))。调整3对引物配比为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=1∶2∶2时,3个目的基因片段均出现较清晰条带(图4(c))。调整引物配比为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=2∶1∶2时,3个目的基因片段均出现清晰条带,且3个条带的亮度差别较小(图4(d)),所以选择该配比为最佳引物配比。
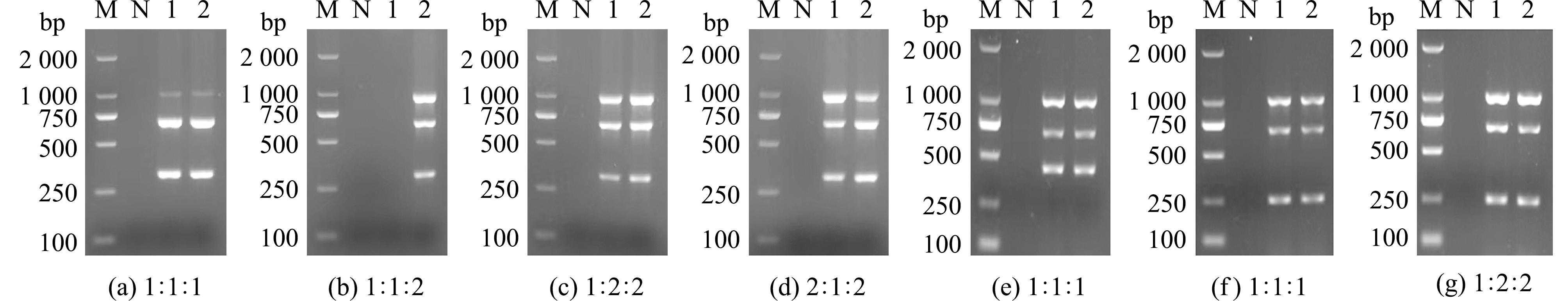
M—DL2000 DNA Marker;N—空白对照;1—鰤鱼诺卡氏菌菌株FS201912;2—鰤鱼诺卡氏菌菌株N-14;(a)~(d) 引物组合Ⅰ ISOPREN-F/R∶ALAT-F/R∶IclR-F/R配比结果;(e) 引物组合Ⅱ AraC-F/R∶ALAT-F/R∶IclR-F/R配比结果;(f)、(g) 引物组合ⅢArsC-F/R∶ALAT-F/R∶IclR-F/R配比结果。
M—DL2000 DNA marker;N—blank control;1—Nocardia seriolae strain FS201912;2—Nocardia seriolae strain N-14;(a)-(d) primer combination Ⅰ ISOPREN-F/R∶ALAT-F/R∶IclR-F/R matching results;(e) primer combination Ⅱ AraC-F/R∶ALAT-F/R∶IclR-F/R matching results;(f),(g) primer combination Ⅲ ArsC-F/R∶ALAT-F/R∶IclR-F/R matching results.
图4 不同引物浓度多重PCR扩增结果
Fig.4 Results of multiplex PCR amplification of different primer concentrations
当引物组合Ⅱ(AraC、ALAT、IclR)总引物浓度比值为AraC-F/R∶ALAT-F/R∶IclR-F/R=1∶1∶1时,3个目的基因片段均出现清晰条带,且3个条带的亮度差别较小(图4(e)),故直接使用该比例。
当引物组合Ⅲ(ArsC、ALAT、IclR)总引物浓度比值为ArsC-F/R∶ALAT-F/R∶IclR-F/R=1∶1∶1时,ALAT、IclR基因片段只能检测出很淡的荧光条带(图4(f))。调整3对引物配比为ArsC-F/R∶ALAT-F/R∶IclR-F/R=1∶2∶2时,3个目的基因片段均出现清晰条带(图4(g)),所以选择该配比为最佳引物配比。
2.3.2 多重PCR反应条件参数的确立 经过多重PCR反应条件参数的优化,确立多重PCR反应体系(50 μL)为Premix TaqTM PCR预混液25 μL、总引物15 μL(组合Ⅰ引物配比ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=2∶1∶2;组合Ⅱ引物配比为AraC-F/R∶ALAT-F/R∶IclR-F/R=1∶1∶1;组合Ⅲ引物配比为ArsC-F/R∶ALAT-F/R∶IclR-F/R=1∶2∶2)、DNA模板5 μL、加ddH2O补足50 μL;多重PCR反应程序为95 ℃下预变性5 min;95 ℃下变性30 s、60 ℃下退火30 s、72 ℃下延伸1 min,共进行30个循环;最后在72 ℃下再延伸10 min。
2.4 多重PCR的灵敏程度检测
根据优化后的多重PCR反应条件和程序,对提取好的FS201912和N-14两株鰤鱼诺卡氏菌DNA进行等比稀释,形成8个浓度梯度,进行多重PCR的灵敏程度检测。对引物组合ISOPREN-F/R(320 bp)、ALAT-F/R(685 bp)、IclR-F/R(1 007 bp)进行多重PCR灵敏度检测,从图5(a)、(b)可见,当鰤鱼诺卡氏菌的DNA模板量为125 ng/μL时仍能同时扩增出合理的目标条带,条带片段大小与预期一致。阴性空白对照无任何扩增条带。表明组合Ⅰ所建立的鰤鱼诺卡氏菌多重PCR的灵敏度可达125 pg/μL。
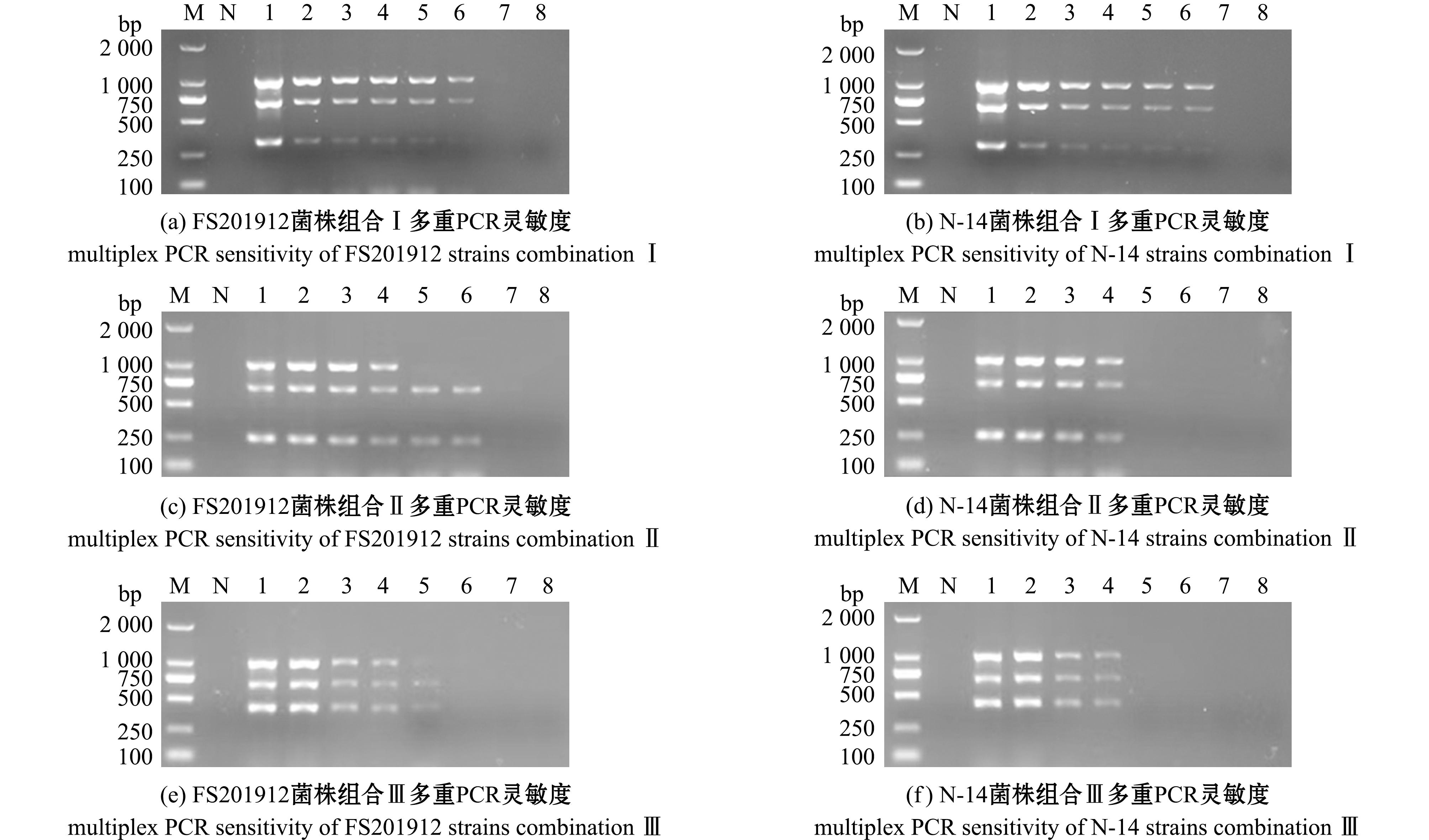
M—DL2000 DNA Marker;N—空白对照;1~8—鰤鱼诺卡氏菌DNA浓度分别为30 ng/μL,6 ng/μL,1 ng/μL,500 pg/μL,250 pg/μL,125 pg/μL,62.5 pg/μL,31.3 pg/μL。
M—DL2000 DNA marker;N—blank control;1-8—Nocardia seriolae DNA concentration are 30 ng/μL,6 ng/μL,1 ng/μL,500 pg/μL,250 pg/μL,125 pg/μL,62.5 pg/μL,and 31.3 pg/μL.
图5 不同浓度下多重PCR的灵敏度
Fig.5 Sensitivity of multiplex PCR under different concentrations
对引物组合AraC-F/R(439 bp)、ALAT-F/R(685 bp)、IclR-F/R(1 007 bp)进行多重PCR灵敏度检测,从图5(c)、(d)可见,鰤鱼诺卡氏菌DNA模板量为500 ng/μL时仍能同时扩增出合理的目标条带,条带片段大小与预期一致。阴性空白对照无任何扩增条带。表明组合Ⅱ所建立的鰤鱼诺卡氏菌多重PCR的灵敏度可达500 pg/μL。
对引物组合ArsC-F/R(252 bp)、ALAT-F/R(685 bp)、IclR-F/R(1 007 bp)进行多重PCR灵敏度检测,从图5(e)、(f)可见,鰤鱼诺卡氏菌DNA模板量为500 ng/μL时仍能同时扩增出合理的目标条带,条带片段大小与预期一致。阴性空白对照无任何扩增条带。表明组合Ⅲ所建立的鰤鱼诺卡氏菌多重PCR的灵敏度可达500 pg/μL。
2.5 多重PCR对鰤鱼诺卡氏菌培养菌落的菌株分型应用
采用优化后的多重PCR参数,采用引物组合Ⅰ(ISOPREN、ALAT、IclR)对从感染鱼类诺卡氏菌病鱼体分离到的29株鰤鱼诺卡氏菌菌株进行多重PCR分型,结果如图6所示,29株鰤鱼诺卡氏菌菌株对应的菌株分型见表6。
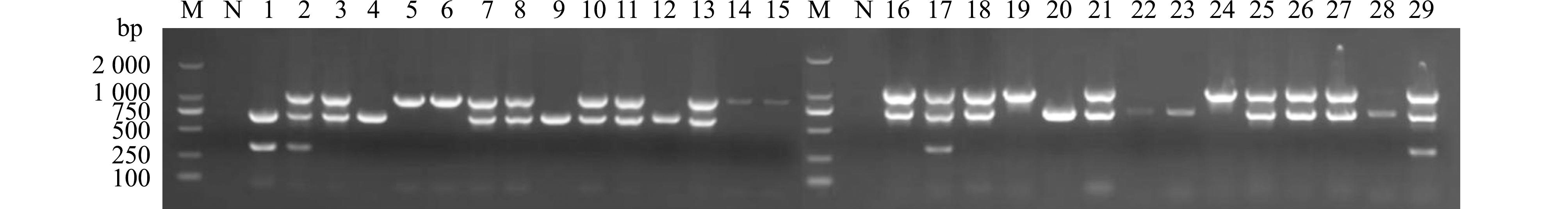
M—DL2000DNA分子标准;N—空白对照;1~24—对应的鰤鱼诺卡氏菌菌株同图1;25~29—对应的鰤鱼诺卡氏菌菌株为N-11,N-22,N-23,WL-2023-11,ZJ0503。
M—DL2000 DNA marker;N—blank control;1-24—the same as Fig.1;25-29— corresponding Nocardia seriolae strains are N-11,N-22,N-23,WL-2023-11,and ZJ0503.
图6 鰤鱼诺卡氏菌菌株多重PCR分型的应用
Fig.6 Application of multiplex PCR typing of Nocardia seriolae strains
表6 29株鰤鱼诺卡氏菌菌株对应的菌株分型
Tab.6 Strains typing of 29 Nocardia seriolae strains
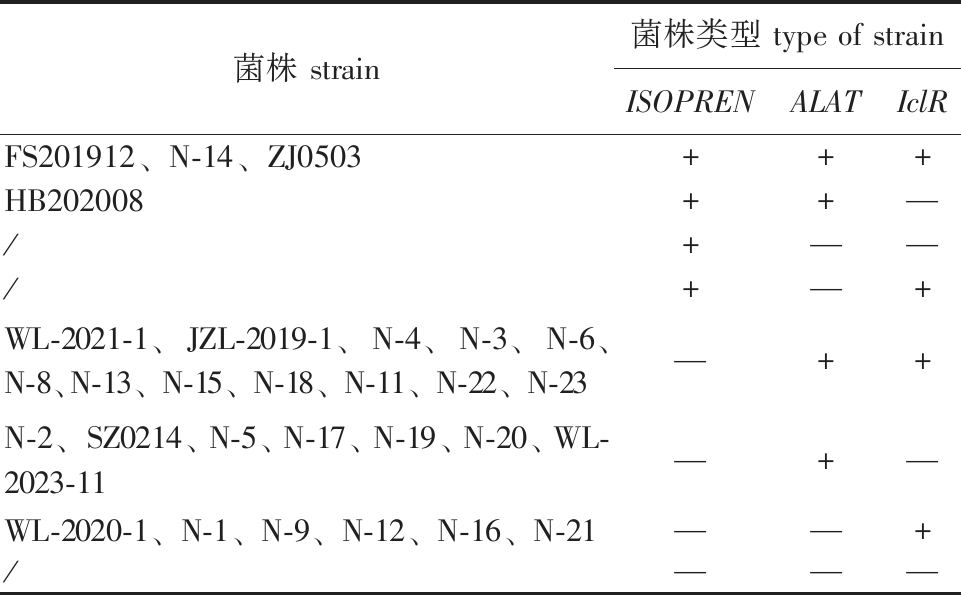
菌株 strain菌株类型 type of strainISOPRENALATIclRFS201912、N-14、ZJ0503+++HB202008++—/+——/+—+WL-2021-1、JZL-2019-1、N-4、N-3、N-6、N-8、N-13、N-15、N-18、N-11、N-22、N-23—++N-2、SZ0214、N-5、N-17、N-19、N-20、WL-2023-11—+—WL-2020-1、N-1、N-9、N-12、N-16、N-21——+/———
2.6 多重PCR对鰤鱼诺卡氏菌感染鱼样的菌株分型应用
采用引物659-F/R分别检测患病与健康杂交鳢的肝脏、脾脏和头肾。结果显示,患病杂交鳢的肝脏、脾脏和头肾均能扩增出目标条带(566 bp),而健康杂交鳢的肝脏、脾脏和头肾均不能检测出目标片段(图7)。表明患病杂交鳢均被鰤鱼诺卡氏菌感染。
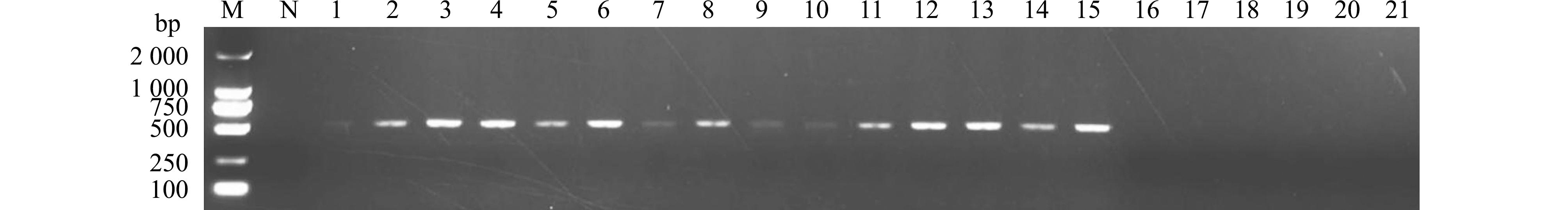
M—DL2000 DNA 分子标位;N—空白对照;1、4、7、10、13—患诺卡氏菌病不同杂交鳢的肝脏;2、5、8、11、14—不同患病杂交鳢的脾脏;3、6、9、12、15—不同患病杂交鳢的头肾;16、19和17、20和18、21—不同健康杂交鳢的肝脏、脾脏与头肾。
M—DL2000 DNA marker;N—blank control;1,4,7,10,13—the liver of the different hybrids of Channa argus infected with Nocardia seriolae;2,5,8,11,14—the spleen of the different hybrids of Channa argus infected with Nocardia seriolae;3,6,9,12,15—the head kidney of the different hybrids of Channa argus infected with N.seriolae;16/19,17/20 and 18/21—the livers,spleens and head kidney of the different healthy Channa argus.
图7 特异性引物659的应用结果
Fig.7 Application results of specific primers 659
采用优化后的PCR程序和条件,对组合ⅠISOPREN-F/R、ALAT-F/R、IclR-F/R进行多重PCR的鱼体应用,结果显示,患鱼类诺卡氏菌病杂交鳢的肝脏、脾脏和头肾均能扩增出组合Ⅰ的3个基因片段(320、685、1 007 bp),而健康杂交鳢的肝脏、脾脏、头肾均未检测出任何条带(图8)。表明建立的鰤鱼诺卡氏菌多重PCR分型方法具有较好的实际应用价值。
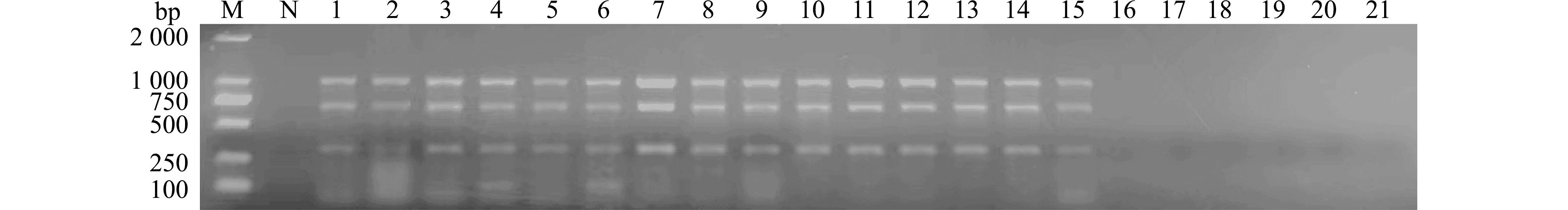
M为DL2000 DNA分子标准;N为空白对照;1~21—同图7。
M—DL2000 DNA marker;N—blank control;1-21—the same as Fig.7.
图8 引物组合Ⅰ(ISOPREN、ALAT、IclR)的应用结果
Fig.8 Application results of the primer combination Ⅰ (ISOPREN,ALAT,and IclR)
3 讨论
3.1 多重PCR分型的建立
鱼类诺卡氏菌病是在中国广泛流行的水产传染病,鰤鱼诺卡氏菌已成为其主要病原菌。鰤鱼诺卡氏菌具有丰富的表型多样性,且其亚群划分与地理环境和宿主无明显相关性。因此,为了对鰤鱼诺卡氏菌的病原分型和防治提供依据,建立一种快速准确的鰤鱼诺卡氏菌菌株分型方法十分重要。本研究中通过对多种鰤鱼诺卡氏菌的全基因数据进行对比分析,筛选并验证获得5个鰤鱼诺卡氏菌具种内差异的基因片段(ArsC、AraC、ISOPREN、ALAT及IclR)并用于多重PCR菌株分型鉴定。利用生物信息学预测上述基因片段功能如表7所示,本研究中选择了PCR产物大小有差异的3组引物组合,用于建立多重PCR技术对鰤鱼诺卡氏菌进行菌株分型的方法。建立的引物组合Ⅰ引物最优配比为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=2∶1∶2,灵敏度可达125 pg/μL;引物组合Ⅱ的引物最优配比为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=1∶1∶1,灵敏度可达500 pg/μL;引物组合Ⅲ的引物最优配比为ISOPREN-F/R∶ALAT-F/R∶IclR-F/R=1∶2∶2,灵敏度可达500 pg/μL。将优化后的多重PCR方法应用于检测鰤鱼诺卡氏菌培养菌落和感染鱼的组织,结果显示,所建立的多重PCR菌株分型方法可应用于鰤鱼诺卡氏菌培养物和感染鱼样检测,能够满足目前对鰤鱼诺卡氏菌进行快速分型鉴定的需要,具有较好的实用价值。
表7 5个鰤鱼诺卡氏菌种内差异基因片段功能的预测
Tab.7 Predicted function of five intraspecies differential gene fragments of Nocardia seriolae

基因片段 gene fragment基因功能 gene functionArsC编码砷还原酶AraC编码转录调节因子ISOPREN编码转录调节因子ALAT编码氨基转移酶IclR编码转录调节因子
多重PCR不仅具有常规PCR的特异性和灵敏度,且相对快捷和经济,已被广泛应用于包括微生物检测与鉴定在内的诸多领域[18]。Cai等[19]根据溶藻弧菌的collagenase、ompK、及toxR基因构建的多重PCR方法,成功鉴定了溶藻弧菌的强毒株与弱毒株;王路才等[20]通过大肠杆菌的9对毒力基因分别建立了两组多重PCR组合,实现了对4种致泻性大肠杆菌的快速诊断与识别。同时,多重PCR技术还做到了在亚种水平上识别和区分同种的不同类型菌株,如Umezawa等[21]根据16S rRNA、rpoB和gyrB基因的部分序列设计多重PCR引物,鉴定了核梭杆菌和坏死梭杆菌的不同亚种;张阁等[22]通过多重PCR技术实现了对禽分枝杆菌3种亚型的鉴定。多重PCR技术能为各种病原微生物的快速诊断及其流行病学的调查提供依据[23]。
3.2 鰤鱼诺卡氏菌有效分型
目前尚未有对鰤鱼诺卡氏菌进行有效分型的报道,其菌株的主要类型也不得而知。相关研究根据对α-葡萄糖苷酶活性的测定,将鰤鱼诺卡氏菌分为α-葡萄糖苷酶阳性与α-葡萄糖苷酶阴性两种类型[11]。α-葡萄糖苷酶在微生物的糖代谢中具有重要生理功能,然而α-葡萄糖苷酶活性与鰤鱼诺卡氏菌的致病性之间的相关性尚不清楚。Davis等[24]用脉冲场凝胶电泳(PFGE)方法对鰤鱼诺卡氏菌进行分子分型,利用Xba I、Ase I两种限制性内切酶分别将鰤鱼诺卡氏菌分成12种与9种菌株类型。PFGE技术需要贵重的仪器且操作步骤较为复杂,缺乏足够的分辨率区分大小几乎相同的谱带,分析结果受人为因素影响较大,容易产生一定的主观性[24]。相较之下,本研究中建立了多重PCR技术应用于鰤鱼诺卡氏菌菌株分型的方法,该方法避免了传统细菌分型需要经过分离培养、生化试验或血清凝集等操作繁杂且过程耗时长的试验,且灵敏度高、操作简单、耗时短,每个样本最少可以只使用一个PCR管,具有快速、实用的优点,实现了对鰤鱼诺卡氏菌更快更精准的分型鉴定。
在后续研究中,本研究团队将进一步丰富鰤鱼诺卡氏菌种内差异基因,并将基因型与菌株表型(如耐药性和毒性等)相结合,应用多重PCR技术构建更全面的鰤鱼诺卡氏菌菌株分型方法,从而快速准确地确定鰤鱼诺卡氏菌流行株的菌株类型,这对鱼类诺卡氏菌病进行针对性治疗或疫苗研制具有重要意义。
4 结论
1)多重PCR技术建立的引物组合Ⅰ(ISOPREN-F/R、ALAT-F/R、IclR-F/R)低至125 pg/μL混合样品仍可被检出;引物组合Ⅱ(AraC-F/R、ALAT-F/R、IclR-F/R)低至500 pg/μL混合样品仍可被检出;引物组合Ⅲ(ArsC-F/R、ALAT-F/R、IclR-F/R)低至500 pg/μL混合样品仍可被检出,说明引物组合对于鰤鱼诺卡氏菌菌株的分型具备良好的效果,且具有较高灵敏度。
2)利用多重PCR技术可在1~2 h内完成鰤鱼诺卡氏菌菌株的分型鉴定,为实践中鰤鱼诺卡氏菌感染病例的菌株分型提供了快速、准确、实用的鉴定方法,对鱼类诺卡氏菌的疫病防治具有一定应用价值。
[1] 王文基,陈建林,侯素莹,等.鰤鱼诺卡氏菌感染乌斑杂交鳢的组织病理学研究[J].基因组学与应用生物学,2019,38(10):4439-4446.
WANG W J,CHEN J L,HOU S Y,et al.A histopathological study of Nocardia seriolae infection in hybrid snakehead[J].Genomics and Applied Biology,2019,38(10):4439-4446.(in Chinese)
[2] 张建丽,刘志恒.诺卡氏菌型放线菌的分类[J].微生物学报,2001,41(4):513-517.
ZHANG J L,LIU Z H.Taxonomy of the nocardioform actinomycetes[J].Acta Microbiologica Sinica,2001,41(4):513-517.(in Chinese)
[3] LIU Y S,CHEN G Q,XIA L Q,et al.A review on the pathogenic bacterium Nocardia seriolae:aetiology,pathogenesis,diagnosis and vaccine development[J].Reviews in Aquaculture,2023,15(1):14-34.
[4] 满其蒙.鰤鱼诺卡氏菌致病机制的研究[D].上海:上海海洋大学,2013.
MAN Q M.Studies on pathogenic mechanism of Nocardia seriolae[D].Shanghai:Shanghai Ocean University,2013.(in Chinese)
[5] 王国良,徐益军,金珊,等.养殖乌鳢诺卡氏菌病及其病原研究[J].水生生物学报,2009,33(2):277-283.
WANG G L,XU Y J,JIN S,et al.Research on the nocardiosis and pathogen in reared snakehead,Ophiocephalus argus Cantor[J].Acta Hydrobiologica Sinica,2009,33(2):277-283.(in Chinese)
[6] BYADGI O,CHEN C W,WANG P C,et al.De novo transcriptome analysis of differential functional gene expression in largemouth bass (Micropterus salmoides) after challenge with Nocardia seriolae[J].International Journal of Molecular Sciences,2016,17(8):1315.
[7] 朱志东,吕莉,邓剑壕,等.鱼类诺卡氏菌病的研究进展[J].水产养殖,2018,39(1):48-52.
ZHU Z D,LÜ L,DENG J H,et al.Research review of fish nocardiosis[J].Journal of Aquaculture,2018,39(1):48-52.(in Chinese)
[8] DOK J,NAM-SIL L,WAND J,et al.Nocardia seriolae infection in the cultured eel Anguilla japonica in Korea[J].Journal of Fish Diseases,2018,41(11):1745-1750.
[9] 黄郁葱,简纪常,吴灶和,等.卵形鲳鲹结节病病原的分离与鉴定[J].广东海洋大学学报,2008,28(4):49-53.
HUANG Y C,JIAN J C,WU Z H,et al.Isolation and identification of the pathogen causing sarcoidosis of Trachinotus ovatus[J].Journal of Guangdong Ocean University,2008,28(4):49-53.(in Chinese)
[10] SHIMAHARA Y,NAKAMURA A,NOMOTOR,et al.Genetic and phenotypic comparison of Nocardia seriolae isolated from fish in Japan[J].Journal of Fish Diseases,2008,31(7):481-488.
[11] SHIMAHARA Y,HUANGY F,TSAIM A,et al.Genotypic and phenotypic analysis of fish pathogen,Nocardia seriolae,isolated in Taiwan[J].Aquaculture,2009,294(3/4):165-171.
[12] 满其蒙,冯娟,区又君,等.15株鱼源致病性鱼诺卡氏菌的聚类分析[J].南方水产科学,2013,9(5):86-92.
MAN Q M,FENG J,OU Y J,et al.Cluster analysis on 15 strains of pathogenic Nocardia seriolae from fish[J].South China Fisheries Science,2013,9(5):86-92.(in Chinese)
[13] 罗愿,邓玉婷,赵飞,等.9株鱼源鰤诺卡氏菌生物学特征和致病性比较[J].微生物学通报,2021,48(8):2733-2749.
LUO Y,DENG Y T,ZHAO F,et al.Comparative on characteristics and pathogenicity of Nocardia seriolae isolated from 9 fishes[J].Microbiology China,2021,48(8):2733-2749.(in Chinese)
[14] 罗伟,高泽霞,曾聪,等.团头鲂微卫星多重PCR体系的建立及应用[J].大连海洋大学学报,2013,28(5):418-423.
LUO W,GAO Z X,ZENG C,et al.Establishment and application of microsatellite multiplex PCR system for bluntsnout black bream Megalobrama amblycephala[J].Journal of Dalian Ocean University,2013,28(5):418-423.(in Chinese)
[15] 叶方钰,刘爽,王权,等.奶牛乳房炎三种主要链球菌多重PCR检测方法的建立[J].中国兽医科学,2023,53(2):182-188.
YE F Y,LIU S,WANG Q,et al.Development of multiplex PCR for detection of Streptococcus in bovine mastitis[J].Chinese Veterinary Science,2023,53(2):182-188.(in Chinese)
[16] BIAN S Z,JIA Y Y,ZHAN Q Y,et al.VPsero:rapid serotyping of Vibrio parahaemolyticus using serogroup-specific genes based on whole-genome sequencing data[J].Frontiers in Microbiology,2021,12:620224.
[17] 陈国权,黄碧艳,张子雯,等.鰤鱼诺卡氏菌特异性PCR检测方法的建立[J].基因组学与应用生物学,2021,40(S2):2656-2665.
CHEN G Q,HUANG B Y,ZHANG Z W,et al.Establishment of specific PCR detection method for Nocardia anchovies[J].Genomics and Applied Biology,2021,40(S2):2656-2665.(in Chinese)
[18] 白菊红,康建平,张星灿,等.多重PCR技术在病原微生物检测中的应用[J].食品工业科技,2019,40(7):322-325,331.
BAI J H,KANG J P,ZHANG X C,et al.Application of multiplex PCR technology on detection of pathogenic microorganism[J].Science and Technology of Food Industry,2019,40(7):322-325,331.(in Chinese)
[19] CAI S H,LU Y S,WU Z H,et al.A novel multiplex PCR method for detecting virulent strains of Vibrio alginolyticus[J].Aquaculture Research,2009,41(1):27-34.
[20] 王路才,苏小艳,张焕容,等.四种致泻性大肠杆菌多重PCR检测方法的建立及在大熊猫上的应用[J].四川畜牧兽医,2023,50(9):31-35.
WANG L C,SU X Y,ZHANG H R,et al.Establishment of four multiplex PCR detection methods for diarrheal Escherichia coli and their application in giant pandas[J].Sichuan Animal &Veterinary Sciences,2023,50(9):31-35.(in Chinese)
[21] UMEZAWA K,HAYASHI S,TSUZUKIBASHI O,et al.One step multiplex PCR for identifications at subspecies level of Fusobacterium nucleatum and Fusobacterium necrophorum[J].Open Journal of Stomatology,2022,12:183-195.
[22] 张阁,彭永,刘国权,等.多重PCR对禽分枝杆菌亚种鉴定的初步应用[J].中国兽药杂志,2017,51(10):11-17.
ZHANG G,PENG Y,LIU G Q,et al.Use of multiplex PCR assay for the identification of Mycobacterium avium sub-species[J].Chinese Journal of Veterinary Drug,2017,51(10):11-17.(in Chinese)
[23] 计若云.鼠伤寒沙门菌及其单相变异体多重PCR检测方法的建立及流行病学调查[D].扬州:扬州大学,2022.
JI R Y.Establishment and epidemiological investigation of a novel multiplex PCR detection method for detection of Salmonella typhimurium and its monophasic variant[D].Yangzhou:Yangzhou University,2022.(in Chinese)
[24] DAVIS M A,HANCOCK D D,BESSER T E,et al.Evaluation of pulsed-field gel electrophoresis as a tool for determining the degree of genetic relatedness between strains of Escherichia coli O157:H7[J].Journal of Clinical Microbiology,2003,41(5):1843-1849.