贝壳基质蛋白是一类存在于贝壳和珍珠矿化结构中的蛋白,在CaCO3沉积和贝壳形成中发挥调节作用[1]。迄今为止,已从长牡蛎(Crassostrea gigas)、马氏珠母贝(Pinctada martensi)和巨海扇蛤(Pecten maximus)等不同贝类中鉴定出数百种贝壳基质蛋白,其中,Nacrein是最早报道的与珍珠质形成紧密相关关系的贝壳基质蛋白之一[2]。Nacrein含有1个碳酸酐酶(carbonic anhydrase,CA)结构域,其主要功能为催化CO2和水反应生成![]() 并促进
并促进![]() 的进一步分解,从而为CaCO3沉积提供
的进一步分解,从而为CaCO3沉积提供![]() 此外,Nacrein还包含1个GXN重复区,该区域主要由Gly-Xaa-Asn(X为Asp、Asn或Glu)组成,通过抑制CaCO3合成调控晶体构型[3]。在CaCO3沉积过程中,Nacrein的CA结构域和GXN重复区可能发挥不同的调控作用。
此外,Nacrein还包含1个GXN重复区,该区域主要由Gly-Xaa-Asn(X为Asp、Asn或Glu)组成,通过抑制CaCO3合成调控晶体构型[3]。在CaCO3沉积过程中,Nacrein的CA结构域和GXN重复区可能发挥不同的调控作用。
贝壳基质蛋白对海洋酸化等环境胁迫下的贝壳形成过程具有重要的调控作用。绝大部分贝类贝壳由约95%的CaCO3晶体和0.1%~5%的有机基质组成,其共同决定了贝壳的结构特性[4]。研究发现,贝壳的形成过程对环境变化极为敏感。例如,当海水pH由8.1降低至7.9时,栉孔扇贝(Chlamys farreri)的贝壳矿化率降低约33%,当pH降至7.3时,矿化率趋近于0,无法形成新的贝壳[5]。在pH为7.4的酸化环境中,长牡蛎幼虫的畸形率高达95%,其中约70%的幼虫不能形成完整的矿化壳,而对照组(pH 8.1)长牡蛎幼虫的畸形率仅为32%[6]。酸化环境下软骨基质蛋白-骨形态发生蛋白(CMP1-BMP7)信号通路的激活可能受到抑制从而影响贝壳形成[7]。酸化环境影响贝壳基质蛋白等矿化相关基因的表达,如pH为7.7的酸化环境会显著影响马氏珠母贝矿化相关基因Pif177和Pearlin的表达[8]。然而,目前关于Nacrein在酸化条件下如何参与CaCO3沉积和贝壳形成的研究仍然较为有限[9]。
长牡蛎作为中国重要的海水养殖贝类,在食物供给和维护生态环境等方面发挥着重要作用[10-12]。近年来,海洋酸化等全球性环境变化对长牡蛎等贝类矿化过程的影响受到广泛关注。目前已在长牡蛎中鉴定到Nacrein、Tyrosinase和Shematrin等数十种贝壳基质蛋白,其在响应酸化胁迫和调控贝壳形成等生物过程中发挥着重要作用[13-14]。从长牡蛎基因组中已鉴定了10余个NacreinL基因(Nacrein-like protein),有学者对CgNacreinLF1、CgNacreinLF2和CgNacreinLF3等基因的结构和进化特征进行了探究,但其在长牡蛎贝壳形成过程中的具体功能尚不明确[3]。本研究中探索了长牡蛎CgNacreinL1的表达模式及在CaCO3沉积中的调控作用,以期明确CgNacreinL1在贝壳形成中的功能,为Nacrein和贝壳生物矿化相关研究提供参考。
1 材料与方法
1.1 材料
试验用长牡蛎采自大连庄河市养殖海区,2龄,壳长为(13±1) cm,置于18 ℃过滤海水中暂养7 d,每日换水1/3并投喂两次175 μm筛绢过滤的商品化螺旋藻粉。试验用Trans 5α克隆感受态细胞和Transetta(DE3)表达感受态细胞均购自北京全式金生物技术有限公司。
1.2 方法
1.2.1 长牡蛎不同组织和细胞采集 随机选取9只长牡蛎,采集外套膜、鳃、血淋巴细胞、闭壳肌、性腺、唇瓣和肝胰腺样品;在体视显微镜下将外套膜分离为外褶、中褶和内褶,采集外套膜不同褶皱部位的样品。每组样品均设置3个平行,取样后向样品中加入1 mL Trizol溶液并液氮速冻,转移至-80 ℃超低温冰箱中保存备用。
1.2.2 CgNacreinL1 RNAi试验及样品收集 试验设为空白(blank)组、negative control (NC) -RNAi组和CgNacreinL1-RNAi组(表1),每组放置18只长牡蛎。siRNA干粉由上海吉玛制药技术有限公司合成,室温4 000 g下离心1 min,每OD(约33 μg)siRNA加入125 μL灭菌海水。blank组不做处理,向NC-RNAi和CgNacreinL1-RNAi组的每只长牡蛎分别注射100 μL siRNA-NC和100 μL siRNA-CgNacreinL1,首次注射2 d后,按相同剂量进行第2次注射,共注射2次。分别在第2次注射后的第3天和第5天,从每组中随机选取9只长牡蛎,每3只为1组取外套膜组织样品并混合,每组样品设置3个平行。在外套膜组织样品中加入1 mL Trizol,于液氮中速冻用于RNA提取。收集去除软体部的长牡蛎贝壳,采用体积分数为5%的NaOH溶液去除贝壳上附着的杂质,采用去离子水清洗并晾干备用。
表1 试验所用引物
Tab.1 Primers used in this study
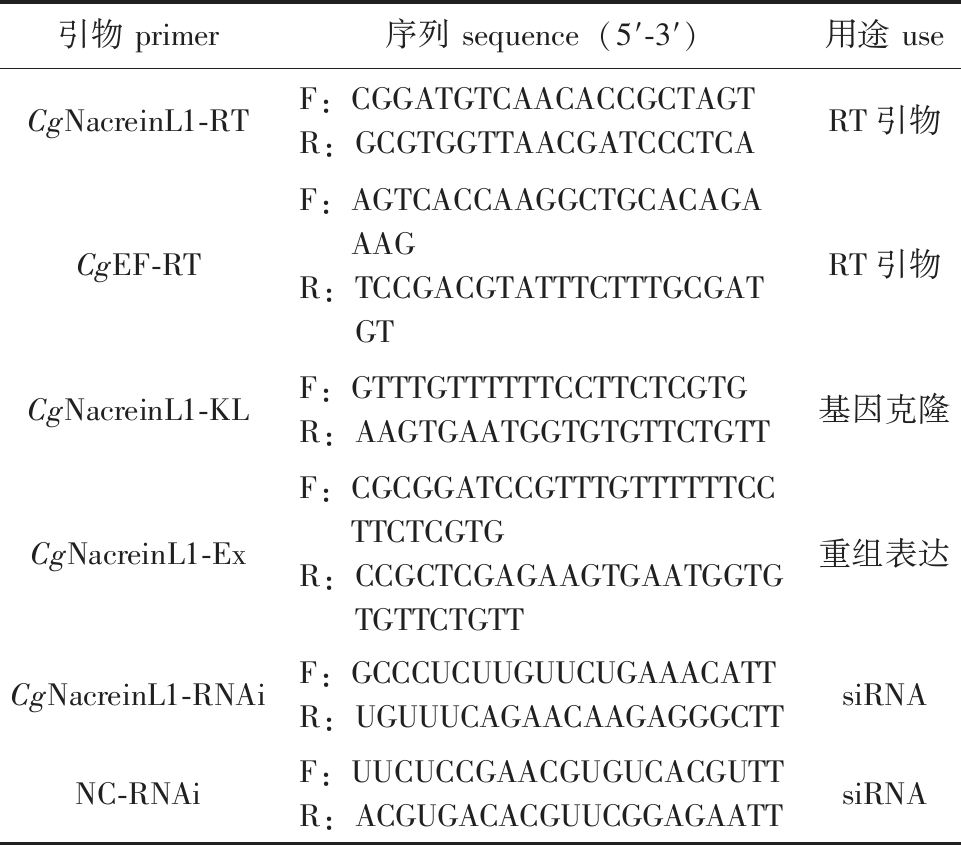
引物 primer序列 sequence(5′-3′)用途 useCgNacreinL1-RTF:CGGATGTCAACACCGCTAGTR:GCGTGGTTAACGATCCCTCART引物CgEF-RTF:AGTCACCAAGGCTGCACAGAAAGR:TCCGACGTATTTCTTTGCGATGTRT引物CgNacreinL1-KLF:GTTTGTTTTTTCCTTCTCGTGR:AAGTGAATGGTGTGTTCTGTT基因克隆CgNacreinL1-ExF:CGCGGATCCGTTTGTTTTTTCCTTCTCGTGR:CCGCTCGAGAAGTGAATGGTGTGTTCTGTT重组表达CgNacreinL1-RNAiF:GCCCUCUUGUUCUGAAACATTR:UGUUUCAGAACAAGAGGGCTTsiRNANC-RNAiF:UUCUCCGAACGUGUCACGUTTR:ACGUGACACGUUCGGAGAATTsiRNA
1.2.3 长牡蛎酸化胁迫与样品收集 酸化胁迫试验分为两组,即pH分别为8.10±0.05和7.80±0.05,其中pH 为8.10±0.05为对照组,pH为 7.80±0.05为酸化胁迫组。对照组海水正常空气曝气,向酸化胁迫组海水中通入空气和CO2的混合物,实时监测pH并调控CO2的浓度,使pH维持在7.80±0.05。分别在胁迫后的第3、7、14天和第28天,从每组中随机选取9只长牡蛎,采集外套膜不同褶皱部位的样品,每3只为1组,每组样品设置3个平行。将样品加入1 mL Trizol溶液后液氮速冻,转移至-80 ℃超低温冰箱中保存备用。
1.2.4 CgNacreinL1生物信息学分析 利用NCBI长牡蛎基因组数据库(GCA_902806645.1)检索确定CgNacreinL1基因(XP_034329950.1)及CDS序列。利用Expasy(https://www.expasy.org/)估算CgNacreinL1蛋白序列的分子量(Mw)和理论等电点(pI)[15];采用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)分析CgNacreinL1开放阅读框(ORF)并推导氨基酸序列[16];利用Prot Param(http://web.expasy.org/protparam/)分析CgNacreinL1氨基酸序列理化性质;采用Uniprot(https://www.uniprot.org/)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)、Novopro(https://novopro.cn/tools/signalp)分析蛋白质结构、跨膜结构域及信号肽[17];通过ExPASy(http://web.expasy.org/vg/index/protein)分析蛋白质亲疏水性;利用GENEDOC进行同源性比对;采用MEGA-X和邻接法(Neighbor-Joining)构建系统进化树[18]。筛选长牡蛎、棉铃虫(Helicoverpa zea)、葡萄牙牡蛎(Crassostrea angulata)、根萤叶甲(Diabrotica virgifera virgifera)、欧洲平牡蛎(Ostrea edulis)、麦红吸浆虫(Sitodiplosis mosellana)、美洲牡蛎(Crassostrea virginica)和大棘穗软珊瑚(Dendronephthya gigantea)等物种的NacreinLs用于以上分析。
1.2.5 总RNA提取和cDNA合成 采用Trizol法提取长牡蛎外套膜、鳃、血淋巴细胞、闭壳肌、性腺、唇瓣和肝胰腺的总RNA。采用TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix反转录试剂盒(北京全式金生物技术有限公司)进行反转录,最终产物保存于-80 ℃超低温冰箱中,用于CgNacreinL1全长cDNA克隆和RT-qPCR试验。
1.2.6 CgNacreinL1基因克隆 采用Primer Premier 5软件设计CgNacreinL1(Genbank ID:XM_034474059.1)特异性引物(表1)。本试验中按照TaKaRa Ex Taq DNA聚合酶说明书配制PCR反应体系,PCR反应条件为94 ℃下预变性5 min;94 ℃下变性30 s,55 ℃下退火30 s,72 ℃下延伸1 min,共进行30个循环;72 ℃下终延伸10 min。PCR产物经凝胶电泳纯化后转入pMD19-T克隆载体(Takara,日本),转化至大肠杆菌Trans 5α感受态细胞进行测序验证。
1.2.7 重组CgNacreinL1蛋白的表达纯化及功能验证 重组CgNacreinL1蛋白的表达和纯化参考已报道方法进行并做适当修改[19],具体步骤如下:首先,采用引物(表1)CgNacreinL1-Ex-F和CgNacreinL1-Ex-R(分别含有BamH Ⅰ和Xho Ⅰ内切酶切点)扩增CgNacreinL1的编码区;其次,将扩增产物经BamH Ⅰ和Xho Ⅰ核酸酶酶切及凝胶纯化后,连接至pET-30a表达载体(TRAN,中国),将构建成功的重组表达载体pET-30a-CgNacreinL1转化至大肠杆菌Transetta(DE3)(TRAN,中国)内,并对携带pET-30a-CgNacreinL1的阳性菌株进行DNA测序;最后,将纯化的重组CgNacreinL1蛋白保存于-80 ℃超低温冰箱中用于后续试验。
试验中向20 mmol/L NaHCO3-CaCl2复合溶液(20 mL)中添加1 mL重组CgNacreinL1蛋白溶液进行孵育,检测溶液pH值变化,确定CgNacreinL1对CaCO3沉积的影响。试验共设置5个处理组,包括1个对照组和4个试验组。对照组中未添加重组CgNacreinL1蛋白,试验组A、B、C和D中分别添加终浓度为0.109、0.357、0.576、1.152 mmol/L的重组CgNacreinL1蛋白,利用酸碱检测仪每隔15 s检测记录pH变化情况,直至溶液pH不再发生明显波动[20-21]。化学反应公式为
2NaHCO3+CaCl2=CaCO3↓+CO2+H2O+2NaCl。
(1)
1.2.8 CgNacreinL1的mRNA表达分析 利用Primer Premier 5软件设计CgNacreinL1的RT-qPCR特异性引物(表1)。采用Quant Studio 6实时荧光定量PCR仪和SYBR premix ExTaq(Thermo Fisher,美国)体系进行RT-qPCR,以CgEF(GenBank ID:NM_001305313)为内参基因,利用2-ΔΔCt法计算基因的相对表达水平[22]。
1.2.9 贝壳结构的扫描电子显微镜(SEM)观察 选取贝壳中间部位制成0.5 cm×0.5 cm小块,使用去离子水清洗断面,置于超声波清洗仪中处理10 min,晾干备用[23]。利用日本电子株式会社JSM-7800F型扫描电子显微镜观察拍照。
1.3 数据处理
试验数据均以平均值±标准差(mean±S.D.)表示,试验均设置3个平行,采用BM SPSS Statistics 26软件进行ANOVA分析及多重比较,显著性水平设为0.05。采用GraphPad Prism 8软件绘制统计图表。
2 结果与分析
2.1 CgNacreinL1基因结构特征
长牡蛎CgNacreinL1 cDNA全长1 037 bp,5′UTR为15 bp,3′UTR为44 bp,ORF为978 bp,可编码325个氨基酸。预测蛋白相对分子质量为36 510,等电点为5.34,含有1个N-糖基化位点(Asn56),总平均亲水性(GRAVY)为-0.306,无规卷曲占54.46%,延伸链占23.69%,α-螺旋结构占19.08%,β-转角结构占2.27%(图1)。预测发现,CgNacreinL1蛋白序列含有1个信号肽(1~29 aa),1个CA结构域及酸性氨基酸残基(D/E)区,无GXN重复区,无跨膜结构域。
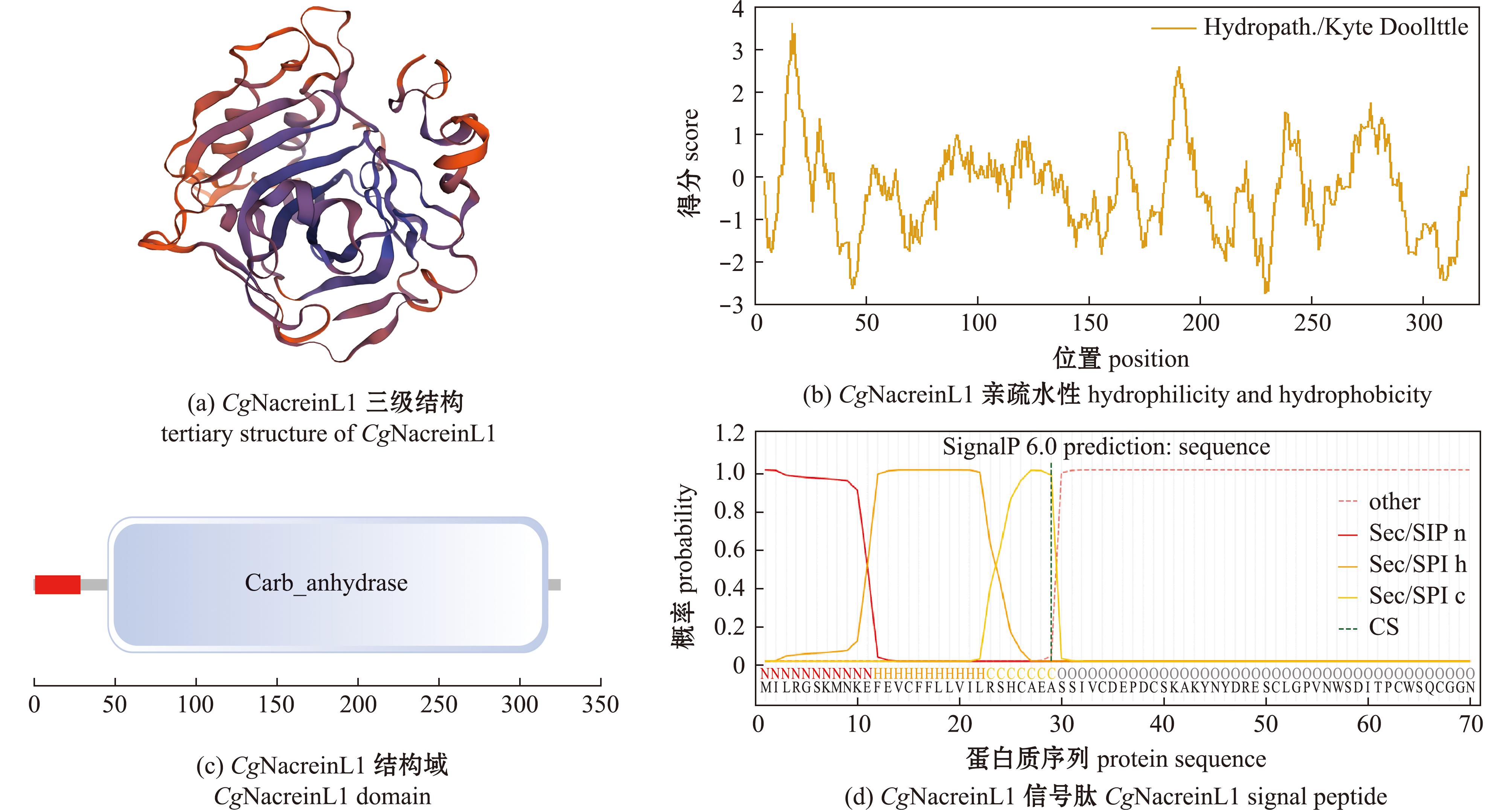
图1 CgNacreinL1结构特征
Fig.1 Structural characteristics of CgNacreinL1
2.2 CgNacreinL1蛋白序列进化特征
将长牡蛎CgNacreinL1的氨基酸序列与其他物种NacreinL进行序列比对发现,CgNacreinL1的氨基酸序列与葡萄牙牡蛎NacreinL具有较高的同源性,序列一致性为97.54%,与其他物种中已鉴定的NacreinL同源性较低,序列一致性为22.15%~41.90%。进一步分析表明,CgNacreinL1含有NacreinL蛋白中保守的GSEHS和YTYE肽段,其CA结构域与其他物种NacreinL表现出较高的保守性(图2)。

绿框标识为蛋白中保守的肽段,紫框标识为CgNacreinL1的CA结构域,黄框标识为酸性氨基酸残基(D/E)区。Crassostrea virginica—美洲牡蛎;Ostrea edulis—欧洲平牡蛎;Crassostrea angulate—葡萄牙牡蛎;Crassostrea gigas—长牡蛎;Dendronephthya gigantea—大棘穗软珊瑚;Helicoverpa zea—棉铃虫;Sitodiplosis mosellana—麦红吸浆虫;Mytilus californianus—紫贻贝。
The green box indicates the conserved peptides in the protein,the purple box indicates the CA domain of CgNacreinL1,and the yellow box indicates the acidic amino acid residue (D/E) region.Crassostrea virginica—Eastern oyster;Ostrea edulis—European flat oyster;Crassostrea angulate—Portuguese oyster;Crassostrea gigas—Pacific oyster;Dendronephthya gigantea—Carnation tree coral;Helicoverpa zea—Corn earworm;Sitodiplosis mosellana—Orange wheat blossom midge;Mytilus californianus—Blue mussel.
图2 CgNacreinL1多序列比对及序列分析
Fig.2 Multi-sequence alignment and sequence analysis of CgNacreinL1
系统进化分析显示,NacreinL系统进化树主要分为软体动物、刺胞动物和节肢动物3个分支。软体动物的NacreinL具有较近的进化关系,其中,长牡蛎CgNacreinL1首先与葡萄牙牡蛎NacreinLC2聚为一支,随后与美洲牡蛎和欧洲平牡蛎(Ostrea edulis)NacreinL聚为一支(图3)。

Crassostrea virginica—美洲牡蛎;Ostrea edulis—欧洲平牡蛎;Crassostrea angulate—葡萄牙牡蛎;Crassostrea gigas—长牡蛎;Dendronephthya gigantea—大棘穗软珊瑚;Helicoverpa armigera—棉铃虫;Diabrotica virgifera virgifera—根萤叶甲;Sitodiplosis mosellana—麦红吸浆虫。
Crassostrea virginica—Eastern oyster;Ostrea edulis-European flat oyster;Crassostrea angulate—Portuguese oyster;Crassostrea gigas—Pacific oyster;Dendronephthya gigantea—Carnation tree coral;Helicoverpa zea—Corn earworm;Diabrotica virgifera virgifera—Western corn rootworm;Sitodiplosis mosellana—Orange wheat blossom midge.
图3 NacreinL系统进化树
Fig.3 Phylogenetic tree of NacreinL
2.3 CgNacreinL1基因在不同组织中的表达特征
CgNacreinL1 mRNA在长牡蛎外套膜、鳃、血淋巴细胞、闭壳肌、性腺、唇瓣和肝胰腺中均有表达,其中在闭壳肌和外套膜中表达水平较高,分别为性腺中表达水平的21.24倍(P<0.05)和11.54倍(P<0.05)(图4)。此外,CgNacreinL1在长牡蛎外套膜外褶、中褶和内褶中均有表达,且在外褶中表达水平最高,为内褶的54.95倍(P<0.05),中褶的40.49倍(P<0.05)(图5)。
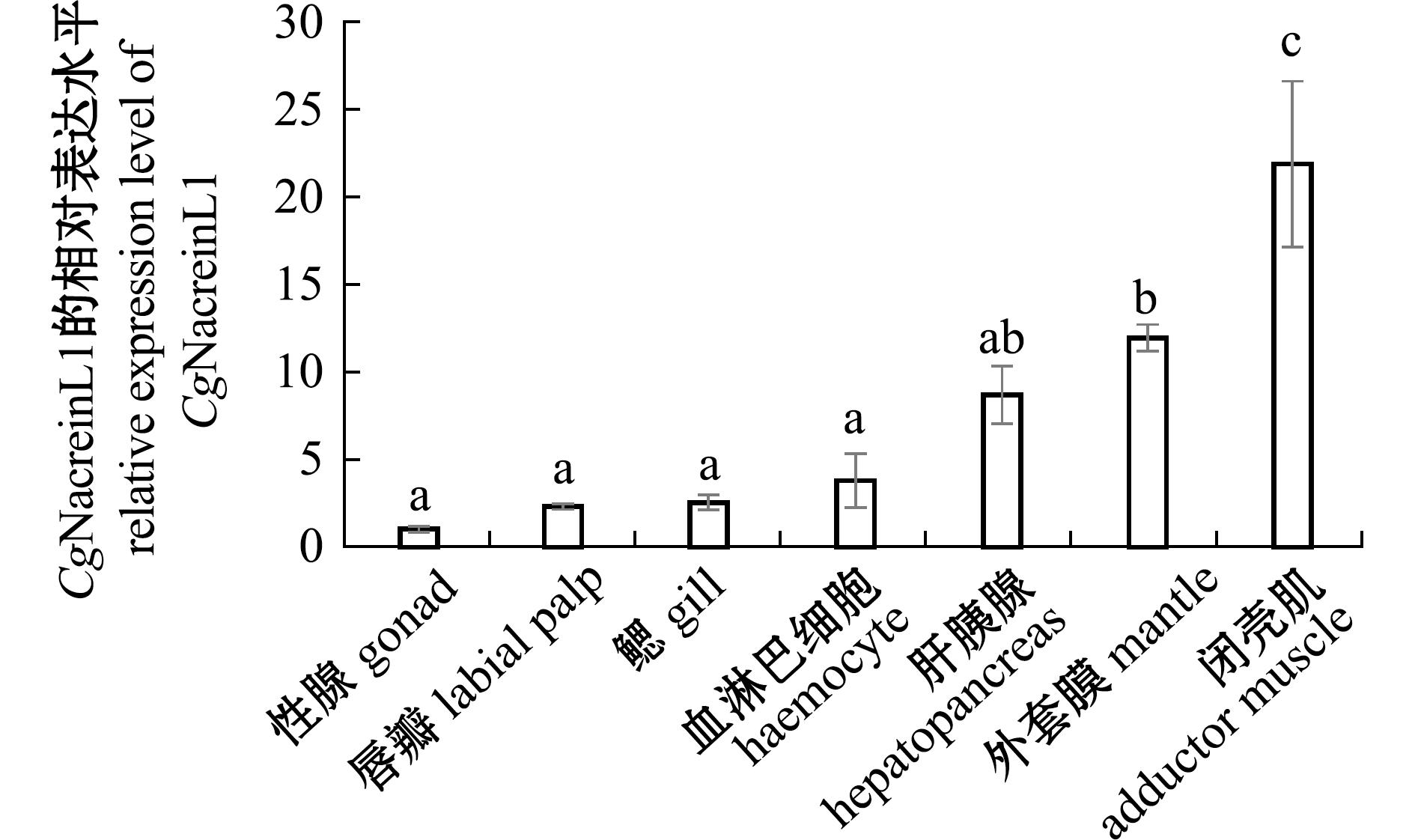
标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05),下同。
The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter are not significant differences,et sequentia.
图4 CgNacreinL1在长牡蛎不同组织中的mRNA表达水平
Fig.4 mRNA expression levels of CgNacreinL1 in different tissues of Crassostrea gigas
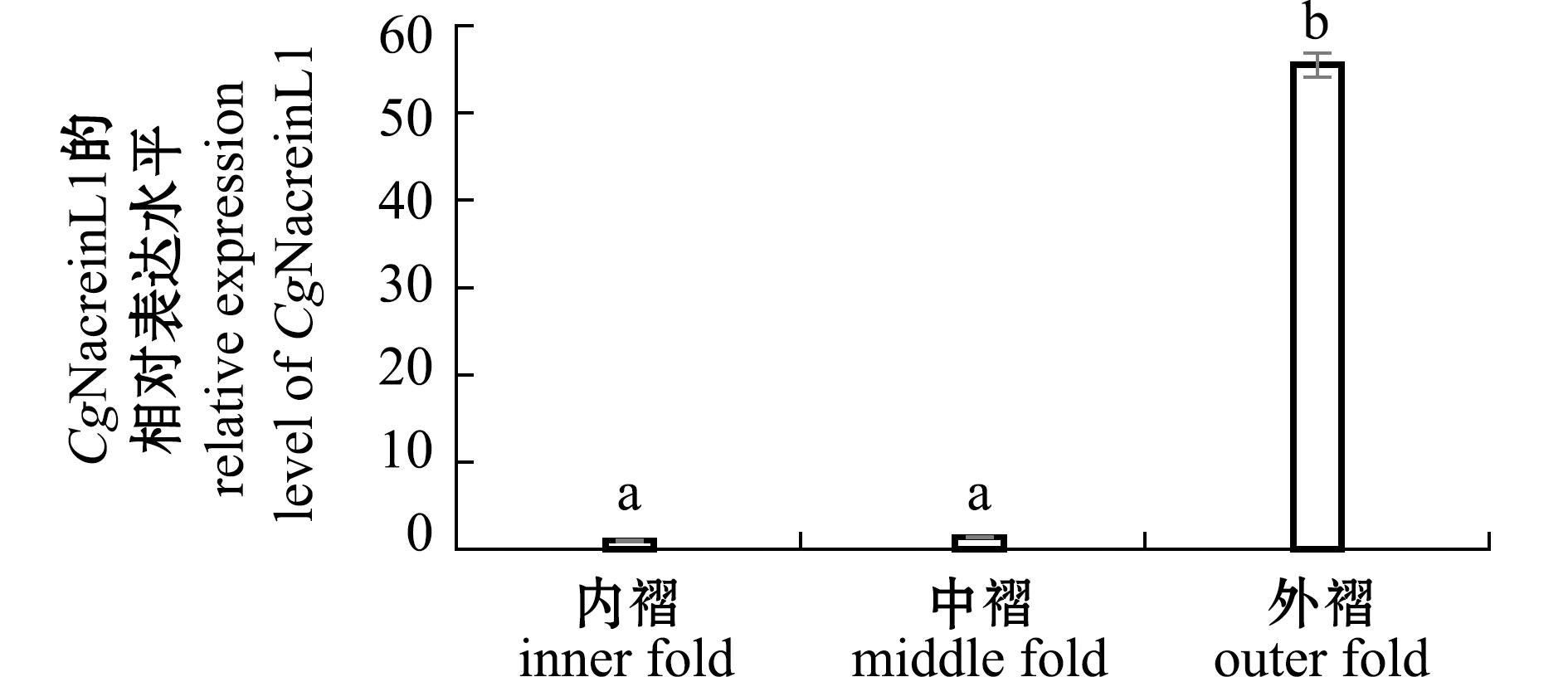
图5 CgNacreinL1在长牡蛎外套膜外褶、中褶和内褶中的mRNA表达水平
Fig.5 mRNA expression levels of CgNacreinL1 in the outer,middle and inner folds of mantle of Crassostrea gigas
2.4 重组CgNacreinL1蛋白孵育对NaHCO3-CaCl2溶液pH的影响
NaHCO3-CaCl2复合溶液初始pH为8.45±0.10,在加入不同浓度重组CgNacreinL1蛋白溶液孵育的15 s内,pH迅速下降至7.75±0.10。对照组pH于15 s后持续下降,下降平均速率为0.01/min。A、B、C和D试验组的pH于15 s后平均下降速率(0.005/min)明显小于对照组,分别为0.008、0.004、0.003、0.000 6/min(图6)。
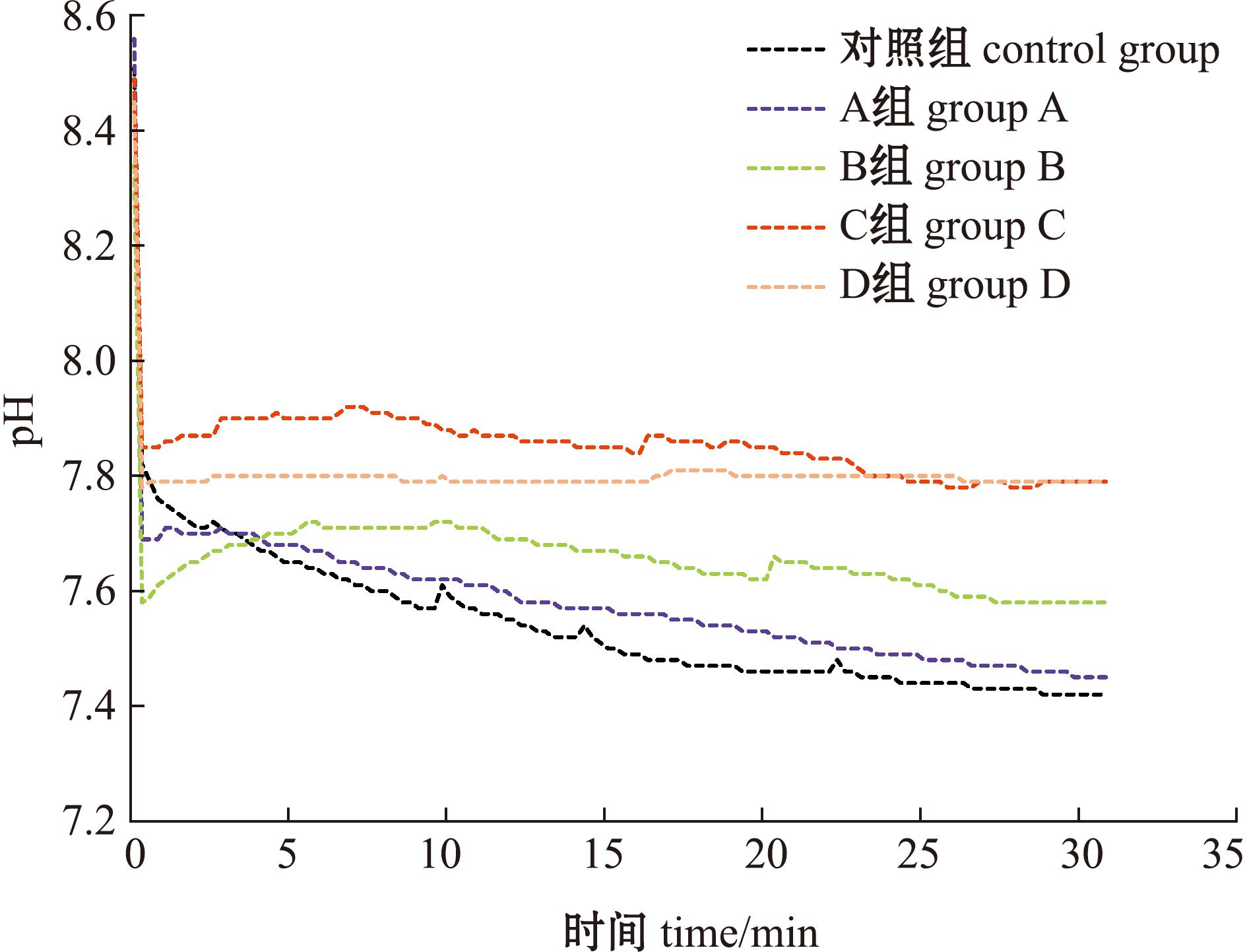
图6 重组CgNacreinL1蛋白孵育对NaHCO3-CaCl2溶液pH的影响
Fig.6 Effects of recombinant CgNacreinL1 protein on the pH of NaHCO3-CaCl2 solution
2.5 干扰CgNacreinL1后贝壳棱柱层结构变化
为探究CgNacreinL1与贝壳形成之间的关系,注射siRNA靶向抑制CgNacreinL1的表达,并检测长牡蛎外套膜中CgNacreinL1的mRNA相对表达水平及贝壳结构变化。RT-qPCR结果显示,在注射siRNA-CgNacreinL1的3 d后,长牡蛎外套膜中CgNacreinL1的mRNA表达水平与NC-RNAi组相比显著下降,为NC-RNAi组的0.056倍(P<0.01),在注射siRNA-CgNacreinL1的5 d后,长牡蛎外套膜中CgNacreinL1的mRNA表达水平与NC-RNAi组相比显著下降,为NC-RNAi组的0.014倍(P<0.05)(图7)。
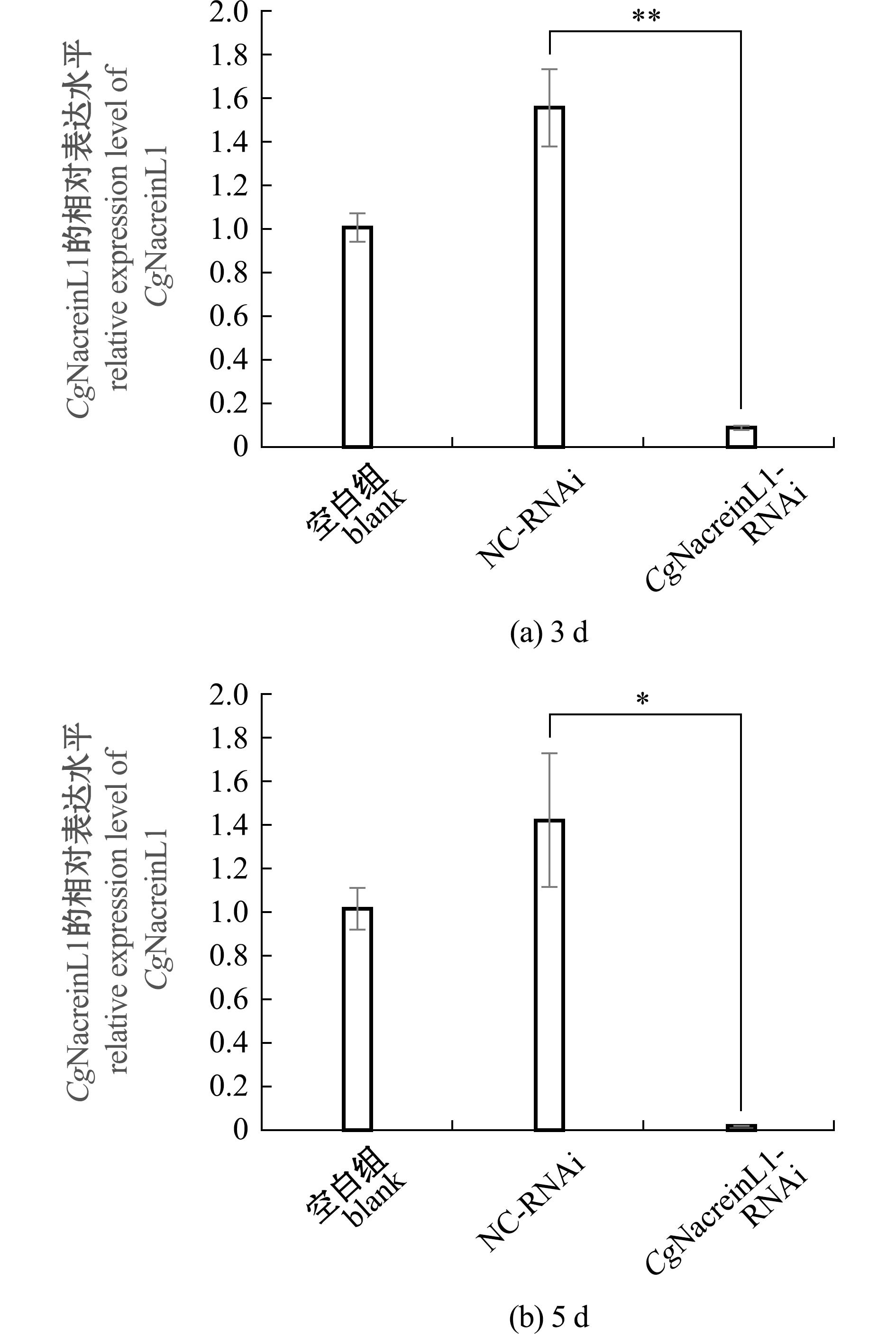
*表示组间有显著性差异(P<0.05),**表示组间有极显著性差异(P<0.01)。
*means significant difference between the groups (P<0.05),**means very significant differences between the groups (P<0.01).
图7 CgNacreinL1基因在RNAi干扰3 d和5 d后的表达特征
Fig.7 Expression characteristics of CgNacreinL1 after 3 d and 5 d of RNAi interference

图8 干扰CgNacreinL1后的贝壳棱柱层结构变化
Fig.8 Structural changes in shell prismatic layer after the interference of CgNacreinL1
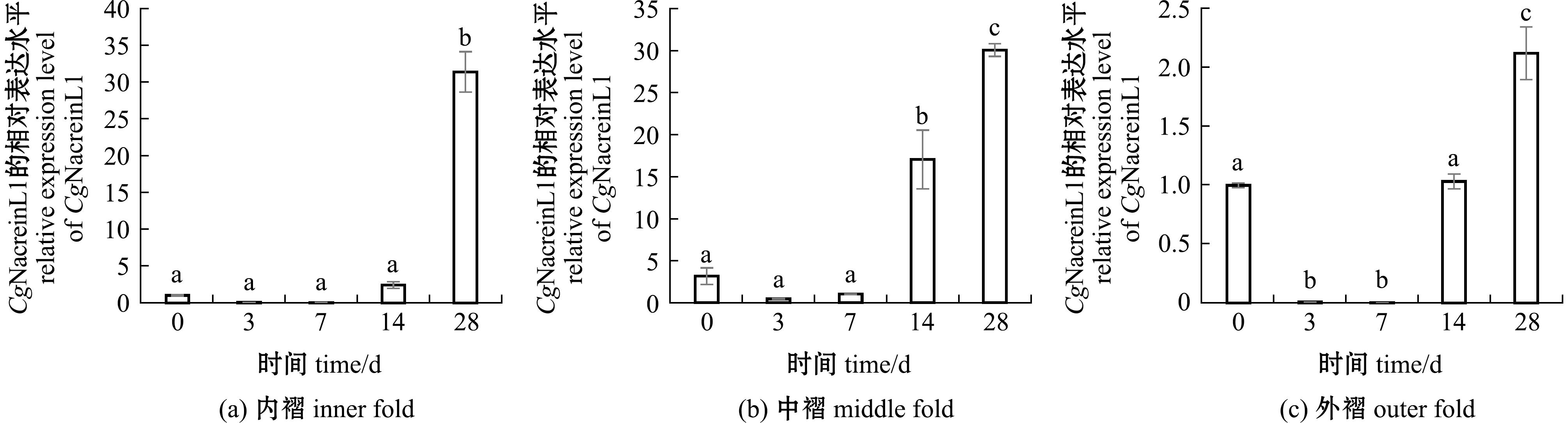
图9 酸化胁迫下CgNacreinL1基因在长牡蛎外套膜内褶、中褶和外褶中的表达水平
Fig.9 Expression levels of CgNacreinL1 in the inner fold,middle fold and outer fold of mantle under acidification stress
利用扫描电子显微镜观察发现,干扰CgNacreinL1后的贝壳结构发生了显著变化。在未注射siRNA-CgNacreinL1时,贝壳的棱柱层结构排列较为松散,表面具有不规则排布的细小孔洞;在注射siRNA-CgNacreinL1 3 d和5 d后,贝壳的棱柱层结构排列变得紧密,表面的细小孔洞和空隙明显减少。
2.6 酸化胁迫下CgNacreinL1在外套膜中的mRNA表达水平变化
在28 d酸化胁迫试验中,CgNacreinL1的mRNA表达水平在长牡蛎外套膜的不同部位均呈现先下降后升高的趋势。在酸化胁迫3、7、14 、28 d后,外套膜内褶中CgNacreinL1的mRNA表达水平分别为对照组长牡蛎的0.07倍(P>0.05)、0.05倍(P>0.05)、2.45倍(P>0.05)和30.97倍(P<0.05);外套膜中褶中CgNacreinL1的mRNA表达水平分别为对照组长牡蛎的0.14倍(P>0.05)、0.32倍(P>0.05)、5.43倍(P<0.05)和9.59倍(P<0.05);外套膜外褶中CgNacreinL1的mRNA表达水平分别为对照组长牡蛎的0.007倍(P<0.05)、0.0013倍(P<0.05)、1.03倍(P>0.05)和2.12倍(P<0.05)。
3 讨论
3.1 CgNacreinL1的序列与进化特征
本研究中克隆得到长牡蛎CgNacreinL1基因,其与前期在长牡蛎中鉴定到的CgNacreinLF1、CgNacreinLF2和CgNacreinLF3具有序列相似性,均具有1个CA结构域,且GXN重复区被酸性氨基酸残基(D/E)区取代;不同之处在于,CgNacreinLF1和CgNacreinLF2具有信号肽和跨膜区域,而CgNacreinLF3则不具有信号肽和跨膜区域[24],相比之下,CgNacreinL1包含信号肽,但无跨膜区域。以上结果对于进一步揭示NacreinL基因家族的结构和进化特征具有一定意义。
CgNacreinL1与葡萄牙牡蛎NacreinL具有较高的同源性,序列一致性为97.54%,与其他生物NacreinL同源性较低,序列一致性为22.15%~41.90%。酸性氨基酸是成年牡蛎壳的主要可溶性有机基质[25-26],且在调控CaCO3矿化过程中发挥重要作用[27]。长牡蛎CgNacreinL1含有CA结构域和酸性氨基酸残基(D/E)区,预示其可能在调控贝壳形成中发挥特定功能。
3.2 CgNacreinL1的组织分布特征
长牡蛎中的CgNacreinL1基因在外套膜外褶区域具有较高的表达水平,显著高于内褶和中褶。外套膜通过分泌有机大分子物质来调控无机矿物质的沉积,进而形成贝壳骨架,其边缘加厚形成三层褶皱:内褶具有放射肌和环肌,能够使外套膜边缘紧贴于贝壳上;中褶含有大量感觉细胞或感觉器,具有响应外界刺激和感知环境变化的功能;外褶具有较强的分泌功能,主要通过合成贝壳基质蛋白参与贝壳形成的调控。目前相关研究已有较多报道,如贝壳基质蛋白LCDP的mRNA于外套膜外褶中存在阳性信号,并在珍珠层与棱柱层的生长阶段发挥关键作用,干扰其表达后会导致文石片沉积无序和棱柱层晶体粗糙等变化[4]。因此,CgNacreinL1在长牡蛎外套膜外褶中的较高表达水平暗示其可能参与了长牡蛎贝壳的形成过程。
3.3 CgNacreinL1对CaCO3沉积的影响
研究发现,重组CgNacreinL1蛋白的孵育能够明显抑制NaHCO3-CaCl2溶液pH的下降速率,提示该蛋白能够降低CaCO3的沉积速率,并抑制CaCO3的形成,这与Nacrein蛋白功能的已有报道一致。在Nacrein蛋白中,GXN重复区在抑制CaCO3形成过程中发挥关键作用。当Nacrein GXN重复区发生突变时,其抑制CaCO3形成的活性显著减弱[22]。此外,当上游转录因子的mRNA表达受到干扰时,Nacrein的表达水平明显降低,导致珍珠层表面出现大量散乱的CaCO3晶体颗粒,并形成不规则的多层堆积,最终导致正常层状结构完全中断[28]。此外,Miyashita等[29]使用牛红细胞CA(BECA)作为对照组,通过EDTA浸提马氏珠母贝珍珠层Nacrein蛋白,进行体外结晶试验以观察其对CaCO3沉积的影响。结果表明,Nacrein蛋白能够显著抑制CaCO3沉积。基于以上研究结果,本研究中推测尽管长牡蛎CgNacreinL1蛋白的GXN重复区被酸性氨基酸残基(D/E)区取代,仍可能发挥着抑制CaCO3沉积的作用。为了验证这一假设,本研究中进一步观察了CgNacreinL1的mRNA表达被抑制后贝壳结构的变化。结果显示,贝壳的棱柱层结构排列变得更加紧密,表面孔洞和空隙明显减少。值得注意的是,CgNacreinL1的CA结构域具有催化CO2水化反应的作用,能够为CaCO3的形成提供![]() 离子,从而间接促进CaCO3的形成。然而,本研究中发现,CgNacreinL1蛋白在贝壳形成过程中主要发挥抑制CaCO3沉积的功能,这表明酸性氨基酸残基(D/E)区与CA结构域在CaCO3沉积过程中存在拮抗作用,且前者的抑制作用占主导地位。然而,关于这一作用机制的具体细节还需进一步研究来阐明。
离子,从而间接促进CaCO3的形成。然而,本研究中发现,CgNacreinL1蛋白在贝壳形成过程中主要发挥抑制CaCO3沉积的功能,这表明酸性氨基酸残基(D/E)区与CA结构域在CaCO3沉积过程中存在拮抗作用,且前者的抑制作用占主导地位。然而,关于这一作用机制的具体细节还需进一步研究来阐明。
3.4 酸化胁迫下CgNacreinL1在长牡蛎外套膜中的表达特征
本研究中进一步分析了CgNacreinL1 mRNA在酸化胁迫下不同时间及在外套膜不同部位(外褶、中褶和内褶)中的表达变化。研究结果显示,在酸化胁迫下,CgNacreinL1在外套膜的不同部位中具有相似的mRNA表达模式,即在3~7 d时表达水平明显下降,在14~28 d时表达水平明显上升。已有研究发现,在酸化胁迫过程中贝壳基质蛋白mRNA水平的变化会对贝壳形成产生显著影响。如长牡蛎在酸化胁迫(pH 7.80±0.05)后,外套膜中褶CgCMP1的mRNA表达水平显著降低,表明酸化可能通过CMP1-BMP7途径影响长牡蛎贝壳生长[7]。本研究中发现,CgNacreinL1表达水平在酸化胁迫早期显著下降,这可能是由于酸化胁迫导致贝壳结构受损,机体通过降低CgNacreinL1表达水平加速CaCO3晶体的形成,以应对外界酸化环境对于贝壳结构的损伤;而在酸化胁迫后期,机体则通过提高CgNacreinL1的表达水平来调控贝壳CaCO3晶体的排布构型,从而确保贝壳结构的完整性。这些发现为理解CgNacreinL1在酸化胁迫下对贝壳形成的调控机制提供了新的见解。
4 结论
1)重组CgNacreinL1蛋白孵育能够显著抑制NaHCO3-CaCl2溶液pH的下降速率,表明重组CgNacreinL1蛋白能够降低CaCO3的沉积速率,抑制CaCO3形成。
2)干扰CgNacreinL1的mRNA表达后,长牡蛎贝壳棱柱层结构排列紧密,表面孔洞和空隙明显减少,表明CgNacreinL1具有调控贝壳CaCO3构型的功能。
3)在酸化胁迫中,CgNacreinL1在外套膜不同部位均呈现先下降后升高的表达变化规律,表明外套膜CgNacreinL1参与酸化胁迫响应过程。
[1] 刘晓军,李家乐.养殖珍珠贝贝壳基质蛋白研究进展[J].上海海洋大学学报,2013,22(2):200-205.
LIU X J,LI J L.Matrix proteins in the shell of cultured pearl bivalves[J].Journal of Shanghai Ocean University,2013,22(2):200-205.(in Chinese)
[2] MIYAMOTO H,YANO M,MIYASHITA T.Similarities in the structure of nacrein,the shell-matrix protein,in a bivalve and a gastropod[J].Journal of Molluscan Studies,2003,69(1):87-89.
[3] 宋小瑞.长牡蛎贝壳形成相关蛋白及其基因的研究[D].青岛:中国科学院研究生院(海洋研究所),2013.
SONG X R.Study on shell formation-related proteins and their genes in oyster,Crassostrea gigas[D].Qingdao:Institute of Oceanology,Chinese Academy of Sciences,2013.(in Chinese)
[4] 袁永斌.三角帆蚌3个贝壳基质蛋白基因在贝壳形成过程中的功能探究[D].上海:上海海洋大学,2023.
YUAN Y B.Function of three shell matrix protein genes in Hyriopsis cumingii during shell formation[D].Shanghai:Shanghai Ocean University,2023.(in Chinese)
[5] 张明亮.栉孔扇贝生理活动对近海碳循环的影响[D].青岛:中国科学院研究生院(海洋研究所),2011.
ZHANG M L.Effects of physiological activities of Chlamys farreri on offshore carbon cycle[D].Qingdao:Institute of Oceanology,Chinese Academy of Sciences,2011.(in Chinese)
[6] PARKER L M,ROSS P M,O’CONNOR W A.Comparing the effect of elevated pCO2 and temperature on the fertilization and early development of two species of oysters[J].Marine Biology,2010,157(11):2435-2452.
[7] 朱婷.软骨基质蛋白(CMP)在调节酸化胁迫下长牡蛎贝壳形成中的作用研究[D].大连:大连海洋大学,2023.
ZHU T.Role of cartilage matrix protein (CMP) in regulating the formation of oyster shells under acidification stress[D].Dalian:Dalian Ocean University,2023.(in Chinese)
[8] 董冰冰,黄荣莲,王庆恒,等.海洋酸化对马氏珠母贝珍珠层形成的影响[J].海洋科学,2015,39(8):39-46.
DONG B B,HUANG R L,WANG Q H,et al.Effects of ocean acidification on nacre formation and expression of related genes in Pinctada martensii[J].Marine Sciences,2015,39(8):39-46.(in Chinese)
[9] SONG X R,WANG X T,LI L,et al.Identification of two novel nacrein-like proteins involved in the shell formation of the Pacific oyster Crassostrea gigas[J].Molecular Biology Reports,2014,41(7):4273-4278.
[10] 刘冉阳,高磊,张兴志,等.广西北海大风江水域环境及养殖香港牡蛎健康状况分析[J].大连海洋大学学报,2024,39(4):551-558.
LIU R Y,GAO L,ZHANG X Z,et al.Analysis on the environment of Dafeng River in Beihai,Guangxi and the health status of cultivated Hong Kong oysters (Crassostrea hongkongensis) [J].Journal of Dalian Ocean University,2024,39(4):551-558.(in Chinese)
[11] 任黎华.桑沟湾筏式养殖长牡蛎及其主要滤食性附着生物固碳功能研究[D].青岛:中国科学院研究生院(海洋研究所),2014.
REN L H.Study on carbon sequestration function of raft culture oyster and its main filter-feeding attached organisms in Sanggou Bay[D].Qingdao:Institute of Oceanology,Chinese Academy of Sciences,2014.(in Chinese)
[12] JONES A R,ALLEWAY H K,MCAFEE D,et al.Climate-friendly seafood:the potential for emissions reduction and carbon capture in marine aquaculture[J].Bioscience,2022,72(2):123-143.
[13] 杜津柘.贝壳基质蛋白及其磷酸化对生物矿化的调控研究 [D].北京:清华大学,2021.
DU J Z.Regulation of shell matrix protein and its phosphorylation on biomineralization [D].Beijing:Tsinghua University,2021.(in Chinese)
[14] 刘雅.马氏珠母贝低温适应性分子机制的初步研究[D].湛江:广东海洋大学,2018.
LIU Y.Preliminary study on the molecular mechanism of low temperature adaptability of Pinctada fucata martensii [D].Zhanjiang:Guangdong Ocean University,2018.(in Chinese)
[15] 浦竞文.三角帆蚌贝壳hic基质蛋白基因的克隆鉴定及功能初探[D].上海:上海海洋大学,2018.
PU J W.Cloning and identification of hic matrix protein gene from Hyriopsis cumingii shell and preliminary study on its function[D].Shanghai:Shanghai Ocean University,2018.(in Chinese)
[16] 林家豪.稻绿蝽和大稻缘蝽化学感受基因鉴定与表达分析[D].广州:仲恺农业工程学院,2020.
LIN J H.Identification and expression analysis of chemosensitive genes in rice green stinkbug and dayuan stinkbug[D].Guangzhou:Zhongkai University of Agriculture and Engineering,2020.(in Chinese)
[17] 胡杰伟.海洋生物基因组数据库中新型降钙素序列挖掘、优化与功能验证[D].上海:中国人民解放军海军军医大学,2018.
HU J W.Mining,optimization and functional verification of novel calcitonin sequences in marine genomedatabase[D].Shanghai:Naval Medical University,2018.(in Chinese)
[18] 郝瑞娟.基于全基因组关联分析解析马氏珠母贝生长和矿化性状关键基因[D].湛江:广东海洋大学,2020.
HAO R J.Analysis of key genes of growth and mineralization traits of Pinctada martensii based on genome-wide association study[D].Zhanjiang:Guangdong Ocean University,2020.(in Chinese)
[19] 卢晓晓.肉鸡谷胱甘肽S-转移酶A3基因的克隆及蛋白的表达和纯化[D].晋中:山西农业大学,2019.
LU X X.Cloning,expression and purification of broilers Glutathione S-Transferase A3 in Escherichia coli[D].Jinzhong:Shanxi Agricultural University,2019.(in Chinese)
[20] 黄荣莲.珍珠贝贝壳蛋白组学及胁迫环境下的应激调控网络[D].青岛:中国科学院研究生院(海洋研究所),2016.
HUANG R L.Proteomics of Pinctada margarita shell and stress regulation network under stress environment[D].Qingdao:Institute of Oceanology,Chinese Academy of Sciences,2016.(in Chinese)
[21] MIYAMOTO H,MIYOSHI F,KOHNO J.The carbonic anhydrase domain protein nacrein is expressed in the epithelial cells of the mantle and acts as a negative regulator in calcification in the mollusc Pinctada fucata[J].Zoological Science,2005,22(3):311-315.
[22] 耿真.新喀里多尼亚弧菌分离鉴定及其对虾夷扇贝的致病性研究 [D].大连:大连海洋大学,2022.
GENG Z.Isolation and identification of Vibrio neocaledonicus and its pathogenicity to Yesso scallop (Patinopecten yessoensis)[D].Dalian:Dalian Ocean University,2022.(in Chinese)
[23] 莫天宝,徐洪强,何京,等.五种双壳贝类贝壳微观结构观察与成分分析[J].海洋科学,2022,46(12):41-49.
MO T B,XU H Q,HE J,et al.Microstructure and composition analysis of five species of economic bivalves[J].Marine Sciences,2022,46(12):41-49.(in Chinese)
[24] 宋小瑞,王晓通,李莉,等.长牡蛎nacrein基因的克隆、结构及进化分析[J].海洋科学,2015,39(10):85-93.
SONG X R,WANG X T,LI L,et al.Molecular cloning and characterization of nacrein gene in Pacific oyster (Crassostrea gigas)[J].Marine Sciences,2015,39(10):85-93.(in Chinese)
[25] ALBECK S,WEINER S,ADDADI L.Polysaccharides of intracrystalline glycoproteins modulate calcite crystal growth in vitro[J].Chemistry-A European Journal,1996,2(3):278-284.
[26] 陈文韬.牡蛎壳组成特性及其综合利用研究[D].福州:福建农林大学,2013.
CHEN W T.Study on composition characteristics and comprehensive utilization of oyster shell[D].Fuzhou:Fujian Agriculture and Forestry University,2013.(in Chinese)
[27] 杨士林,高倩,王海棠.酸性氨基酸调控碳酸钙仿生矿化过程研究[J].黑龙江大学自然科学学报,2010,27(4):495-499,503.
YANG S L,GAO Q,WANG H T.Biomimetic mineralization of calcium carbonate with acidic amino acid[J].Journal of Natural Science of Heilongjiang University,2010,27(4):495-499,503.(in Chinese)
[28] SUN J,XU G R,WANG Z S,et al.The effect of NF-κB signalling pathway on expression and regulation of nacrein in pearl oyster,Pinctada fucata[J].PLoS One,2015,10(7):e0131711.
[29] MIYASHITA T,OKAMOTO A,TAKAGI R.Inhibitory effects of carbonic anhydrase nacrein on calcium carbonate crystallization[J].Kindai University,2004,14:11-16.