鲍属原始腹足目鲍科鲍属的软体动物,可食用部分含有多种生物活性化合物,具有抗氧化、抗菌和抗癌等活性,其味道鲜美、营养丰富,具有较高的经济价值[1]。皱纹盘鲍(Haliotis discus hannai)主要分布于西北太平洋的日本北部、朝鲜半岛和中国辽东与山东半岛水域[2]。其中,产自中国黄渤海的皱纹盘鲍,占中国鲍养殖总量的85%以上,是中国主要的养殖贝类[3]。在自然海域中,皱纹盘鲍多见于水质清澈、海藻繁盛的低潮线及浅海岩礁地带。在鲍的个体发育过程中,伴随着生活方式和营养模式的转变,从担轮幼体至面盘幼虫阶段,鲍营浮游生活,此阶段的幼体不进行摄食活动,所需的代谢能量完全来源于胚胎的卵黄储备;鲍发育至面盘幼虫后期和匍匐幼体前期,历经附着变态过程,逐渐转变为底栖生活,开始刮食硅藻[4];从幼鲍开始,其消化系统和摄食器官发育成熟,食物也逐渐转变为大型海藻。截至目前,已报道了包括皱纹盘鲍在内的约9种鲍的基因组信息[5-6],但鲍对饵料的适应机制,还未得到深入的解析。
皱纹盘鲍养殖常用的几种适口藻类,如海带(Laminaria japonica)、龙须菜(Gracilaria lemaneiformis)和石莼(Ulva pertusa),除含有丰富的蛋白质、维生素、矿物质和微量元素外,还含有高度硫酸化的海藻多糖[7]。自Lyu等[8]、Dodgson等[9]在海洋软体动物中鉴定到硫酸酯酶活性以来,相关学者从生物化学的角度,提出硫酸酯酶在海洋食藻动物的物质代谢及利用中可能发挥着重要功能,然而,在分子生物学水平开展的鲍相关硫酸酯酶研究鲜有报道。
硫酸酯酶基因分布在大多数原核和真核物种中,在结构和功能上高度保守,可以水解如糖胺聚糖、脑硫脂和类固醇硫酸酯等多种底物的硫酸酯键,释放硫酸基[10]。硫酸酯酶除了参与机体代谢外,在一些物种如海胆[11]、 线虫(Pristionchus pacificus)[12]的个体发育中也起着决定性的作用。本研究中以皱纹盘鲍为研究对象,利用转录组测序技术,解析了海带(褐藻)、龙须菜(红藻)及孔石莼(绿藻)3 种藻类喂养条件下内脏团组织差异表达的基因,筛选到众多的硫酸酯酶基因,表明该类基因在鲍对不同饵料的代谢和利用中可能发挥重要作用,并进一步选取硫酸酯酶ids-1(iduronate 2-sulfatase-like)基因,克隆其编码序列,分析了基因的序列信息,并利用大肠杆菌体外表达系统对IDS-1进行了重组表达和纯化。本研究中通过实时荧光定量PCR的方法对硫酸酯酶ids-1基因的组织分布、发育阶段及不同饵料喂养下的表达特征开展了初步研究,以期为探究其在皱纹盘鲍饵料适应中的分子调控机制提供科学参考。
1 材料与方法
1.1 材料
试验用2龄成体皱纹盘鲍来自山东省青岛市前沿海洋种业有限公司,平均壳长为(80±3)mm。随机选取6只健康鲍,分离其消化腺、鳃、外套膜和上足触手组织,液氮冻存,用于基因的组织特异性表达分析。
依据文献方法对皱纹盘鲍进行促熟和繁育[4],并收集早期胚胎至成体阶段的样品于液氮中冻存,用于基因的发育阶段特异性表达分析。本试验中共取6个不同发育时期的皱纹盘鲍样品,分别为受精后0 h(受精卵)、6 h(原肠胚期)、12 h(担轮幼虫期)、60 h(面盘幼虫期)、5 d(匍匐幼虫期)及1龄鲍(消化腺、鳃、外套膜和上足触手组织的混合样品);胚胎及幼体发育阶段在显微镜下观察、鉴定。幼鲍以底栖硅藻为食,成体投喂海带。
饲喂试验所用8月龄幼鲍由育苗场运至中国科学院海洋研究所活体培育室暂养4周。在此期间,投喂配合饲料,海水水温保持在20 ℃~25 ℃,盐度为30±1。随后,选取规格接近的健康幼鲍进行随机分组,每组50只,分别投喂海带(褐藻)、龙须菜(红藻)和孔石莼(绿藻)。养殖6个月后,取消化腺,用于总RNA的提取及转录组测序(每组各6只);另外,每组各选取3只幼鲍,饥饿48 h清空消化道食物后,取其消化腺,用于硫酸酯酶活性检测。取样鲍平均壳长为(32±2) mm。
1.2 方法
1.2.1 总RNA的提取 采用TRIzol试剂盒(Invitrogen)提取各收集样品的总RNA,方法参照使用说明书。使用NanoDrop 2 000检测RNA的浓度及纯度,通过10 g/L琼脂糖凝胶电泳检测RNA的完整性。
1.2.2 皱纹盘鲍消化腺粗提液的制备及硫酸酯酶活性检测 解剖分离皱纹盘鲍的消化腺,称重,置于冰上预冷的玻璃匀浆器内,分别加入6倍体积的柠檬酸钠缓冲液(100 mmol/L,pH 5.6)[13],手动研磨。彻底匀浆后,4 ℃下,12 000 g离心20 min;取上清,即得消化腺粗提液。粗提液的蛋白含量利用BSA标准溶液和考马斯亮蓝G-250测定。将粗提液分别稀释10倍及100倍,用于硫酸酯酶的酶活检测。
利用底物4-硝基邻苯二酚硫酸二钾盐检测硫酸酯酶的酶活特性[14]。具体方法如下,首先制备200 μL反应体系:包含100 mmol/L乙酸钠缓冲液(pH 5.0),1.25 mmol/L底物溶液及一定稀释倍数的消化腺提取液;37 ℃下孵育30 min后,取出40 μL反应液,加入200 μL NaOH溶液,混匀、显色,使用酶标仪(Tecan)检测OD值。
1.2.3 皱纹盘鲍ids-1基因的克隆 采用反转录试剂盒PrimeScriptTM RT reagent Kit (Takara)进行反转录,合成第一链cDNA。从转录组测序结果中获得皱纹盘鲍ids-1的基因片段,参照皱纹盘鲍的基因组序列信息[15],利用NCBI Primer Blast设计5′及3′特异性引物(表1)。以皱纹盘鲍成体cDNA为模板,使用ApexHF HS DNA Polymerase FS(艾科生物科技有限公司)扩增ids-1全长cDNA序列。PCR反应程序:94 ℃下预变性3 min;98 ℃下循环变性10 s,57 ℃下退火复性15 s,72 ℃下延伸60 s,共进行35个循环;在72 ℃下再延伸10 min。PCR产物经10 g/L琼脂糖凝胶电泳分离后,利用PCR产物凝胶回收试剂盒(艾科瑞生物科技有限公司)回收DNA。将回收产物与pMD19-T载体(pMD19-T Vector Cloning Kit,Takara)连接,转化到感受态细胞Trelief 5α(北京擎科生物科技有限公司)中,于LB平板(Amp+)培养,挑选阳性克隆,用于测序分析(北京擎科生物科技有限公司)。
表1 试验所用引物
Tab.1 Primers used in this study

引物 primer引物序列 primer sequence(5′-3′)用途 usageids-1F:CCGGTGTTCCTCGTACAACTR:GTAAGACATAAAACAGTCCGAGTTC基因克隆qPCR ids-1F:AACACCGACGACTACCCCTAR:CTTGACCGTCACTGGACACAqPCRRPS9F:GTCGGCTCGTGCGTATR:GGATGTTCACCACCTGTTTqPCR重组F:TCTCCCGGGACTAGTGAGCTCATGAACAAGTTACTAAGTGCAGR:CTTTGTAGTCCATTTGGATCCGTCATGTTGGTAACACACTCC重组表达
1.2.4 序列分析 采用NCBI(http://www.ncbi.nlm.nih.gov)网站上的BLAST工具,进行基因序列相似性及同源性查找和比较;采用ORF finder工具分析基因编码区。采用https://www.expasy.org网站提供的ProtParam工具进行蛋白质基本物理化学参数分析。采用SignalP 6.0 Server软件查找信号肽。通过NetNGlyc 1.0 Server软件查找糖基化位点。采用InterProScan预测蛋白质功能结构域。
1.2.5 系统演化分析 从NCBI、Uniprot和Ensembl数据库中下载代表物种的硫酸酯酶蛋白序列,用于系统发育分析。采用MAFFT进行蛋白序列比对。采用RAxML构建ML系统发生树,设置最佳模型为LG+Gamma+Invariant,自举法检验(Bootstrap)次数设为1 000。用于构建系统发生树的蛋白序列信息见表2。
表2 用于系统发育分析的硫酸酯酶蛋白序列号
Tab.2 Accession numbers of sulfatase proteins used for phylogenetic analysis

名称 name物种 species序列号 accession numberHsap SULF1Homo sapiensENSP00000431753.1Cgig SULF1Crassostrea gigasXP_11433656.3Pcan SULF1Pomacea canaliculataXP_25089753.1Hruf SULF2Haliotis rufescensXP_46333207.2Drer SULF2Danio rerioXP_5169326.1Hsap ARSAHomo sapiensENSP00000216124.5Drer ARSADanio rerioNP_1013561.2Hsap ARSBHomo sapiensENSP00000426879.2Drer ARSIDanio rerioXP_2664306.1Dmel ARSBDrosophila melanogasterFBpp0074909Osin ARSJOctopus sinensisXP_29635402.1Pcan ARSBPomacea canaliculataXP_25082392.1Cgig ARSJCrassostrea gigasXP_11444079.2Hrub ARSBHaliotis rubraXP_46575335.1Hruf ARSB1Haliotis rufescensXP_46329316.2Hruf ARSB2Haliotis rufescensXP_46368365.2Hsap STSHomo sapiensENSP00000499549.2Drer STSDanio rerioXP_5168454.1Pcan STSPomacea canaliculataXP_25102836.1Cgig STSCrassostrea gigasXP_11446202.2Hrub STSHaliotis rubraXP_46578444.1Hruf STSHaliotis rufescensXP_46355903.2Hsap IDSHomo sapiensENSP00000339801.6Drer IDSDanio rerioNP_1073537.1Dmel IDSDrosophila melanogasterFBpp0072939Osin IDSOctopus sinensisXP_29642168.1Cgig IDSCrassostrea gigasXP_34301733.1Hrub IDSHaliotis rubraXP_46561830.1Hruf IDSHaliotis rufescensXP_46377275.2Hsap SGSHHomo sapiensENSP00000461160.1Pcan SGSHPomacea canaliculataXP_25089513.1Cgig SGSHCrassostrea gigasXP_34312464.1Hruf SGSHHaliotis rufescensXP_46355968.2Hrub SGSHHaliotis rubraXP_46557150.1
1.2.6 皱纹盘鲍IDS-1的重组表达及蛋白纯化 以“1.2.3节”构建的含ids-1全长cDNA的质粒为模板,采用重组F/R引物(表1),扩增ids-1编码区,采用同源重组的方式,通过Exnase Ⅱ(诺唯赞生物同源重组试剂盒C112)连接至表达载体pRSET A,转化大肠杆菌BL21(DE3)感受态细胞(擎科生物TSC-E01)。重组蛋白表达条件为37 ℃下震荡培养至OD600值为0.6左右,加入终浓度1 mmol/L的 IPTG,16 ℃下诱导表达12~16 h。菌液在冰浴条件下经超声破碎后,采用SDS-PAGE检测重组蛋白的表达情况。采用His标签蛋白琼脂糖纯化树脂(翌圣生物,20502ES50)纯化重组蛋白,透析复性后,采用“1.2.2节”所述方法,检测重组蛋白IDS-1的硫酸酯酶活性。
1.2.7 Real-time PCR分析 采用Taq Pro Universal SYBR qPCR Master Mix试剂盒 (南京诺唯赞生物科技股份有限公司),利用QuantStudio 6 Flex Real-Time PCR System (ThermoFisher,USA)进行基因的荧光定量分析,内参基因为RPS9[16]。通过2-ΔΔCt法计算ids-1基因在各检测样本中的相对表达量[17]。相关引物由北京擎科生物科技有限公司合成,序列信息见表1。采用GraphPad Prism 8软件对组间数据进行单因素方差分析(one-way-ANOVA)。
1.3 数据处理
1.3.1 文库构建和测序 提取的RNA库检合格后,随机挑选同组内两只幼鲍的RNA等量混合后建库,每组共计3个生物学重复。采用 Illumina Nova-seq 平台进行高通量测序,测序数据上传至NCBI数据库(BioProject PRJNA1118221)。对原始数据进行质控并过滤,分别去除含接头的序列、含N比例大于10%的序列及质量值 Q≤10的碱基数占整条 Read的 50%以上的 Reads。
1.3.2 差异表达基因筛选 采用HISAT2软件对筛选后的高质量Reads与皱纹盘鲍全基因组进行比对[18],并利用StringTie软件对比对上的Reads进行基因表达定量[19]。通过 DESeq2 R 软件包进行差异基因鉴定[20] 。采用 log2|FoldChange|≥1 且 adjusted P value≤0.05 作为阈值。
1.3.3 富集分析 采用DAVID数据库(https://david.ncifcrf.gov/)对参考基因组预测的蛋白进行功能注释。采用clusterProfiler[21] R 软件包对筛选出的差异表达基因进行富集分析,显著性cut-off值设置为0.05,获得生物学过程(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)上的差异基因富集数。并采用ggplot2 R 软件包对结果进行可视化分析[22]。
2 结果与分析
2.1 不同饵料喂养条件下内脏团转录组的分析结果
将不同藻类喂养的3个试验组两两之间进行差异基因的对比,分别为龙须菜和海带对比组(Long vs Hai)、海带和孔石莼对比组(Hai vs Shi)及孔石莼和龙须菜对比组(Shi vs Long)。表3为不同藻类喂养条件下,皱纹盘鲍内脏团差异基因筛选结果。
表3 皱纹盘鲍内脏团差异基因筛选结果
Tab.3 DEGs in visceral mass of Haliotis discus hannai

分组group上调基因个数up-regulated下调基因个数down-regulatedLong vs Hai104118Hai vs Shi8093Shi vs Long7772
将各组GO term的Pvalue从小到大排序选取前5 个绘制气泡图,结果如图1所示。

图1 皱纹盘鲍内脏团差异表达基因的GO富集分析
Fig.1 GO enrichment analysis of DEGs in visceral mass of Haliotis discus hannai
生物学过程大类的富集结果表明,GO:0006027(glycosaminoglycan catabolic process)在3组差异基因集中均显著富集到,说明采用不同藻类喂养皱纹盘鲍会对分解代谢硫酸化多糖的相关基因造成表达量上的影响。在Hai vs Shi与Shi vs Long两组的富集结果中,差异基因也富集到了GO:0006790(sulfur compound metabolic process)中。此外,差异基因还富集到了一些固醇类物质合成与代谢相关的功能词条,如GO:0006695 (cholesterol biosynthetic process),GO:0016126(sterol biosynthetic process),GO:0008203 (cholesterol metabolic process),GO:0016125 (sterol metabolic process)等,表明投喂不同藻类可能会影响到皱纹盘鲍的某些激素调控通路。
细胞成分大类中,3组差异基因所富集的 GO term 主要有GO:0005764(lysosome),GO:0000323(lytic vacuole),GO:0005775(vacuolar lumen),GO:0043202(lysosomal lunen),GO0005788(endoplasmic reticulum lumen)等,溶酶体和内质网是与蛋白质的合成、加工及降解相关的重要细胞器,差异基因富集到这几类功能词条中,也表明皱纹盘鲍在不同藻类喂养条件下会对一些相关蛋白的表达与分泌造成一定影响。
在分子功能方面,3组差异基因均富集到GO:0004423(iduronate-2-sulfatase activity)和GO:0008484(sulfuric ester hydrolase activity )两个功能词条,表明在皱纹盘鲍内脏团中,硫酸酯水解的相关基因会受到摄食的海藻种类影响。艾杜糖醛酸 2-硫酸酯酶(iduronate-2-sulfatase)属于硫酸酯酶基因家族的一类,GO富集的结果暗示了皱纹盘鲍所摄食的海藻种类与硫酸酯酶基因的表达相关联。此外,在3组分子功能方面的GO富集结果中,GO:0016798(hydrolase activity,acting on glycosyl bonds)均有存在,表明3组藻类在多糖结构和硫酸化结构上的不同会影响一些酶类物质的表达量与活性。
2.2 皱纹盘鲍消化腺粗提液具硫酸酯酶活性
在蜗牛[23]、帽贝[24]、螺[25]、牡蛎[26]及红鲍[27]等多种软体动物消化腺中,已有硫酸酯酶纯化及活性的相关报道,并表明硫酸酯酶可能参与了食物的代谢和利用过程。本研究中分别提取了3组不同藻类饲喂的皱纹盘鲍消化腺粗提液,随后利用4-硝基邻苯二酚硫酸二钾盐检测硫酸酯酶的酶活特性。波长405 nm作为底物特征吸收峰,在515 nm处为产物4-硝基儿茶酚的特征吸收峰。结果如图2所示,将消化腺粗提液分别稀释10倍和100倍后,均呈现出显著的硫酸酯酶活性信号,表明皱纹盘鲍消化腺中硫酸酯酶的存在。在皱纹盘鲍消化腺转录组数据中鉴定到一个保守的硫酸酯酶基因ids-1,该基因与人类的IDS可能是直系同源基因;为了探讨该蛋白的活性特征及其与鲍饵料适应的相关性,对其开展了系统发育和克隆表达相关研究。
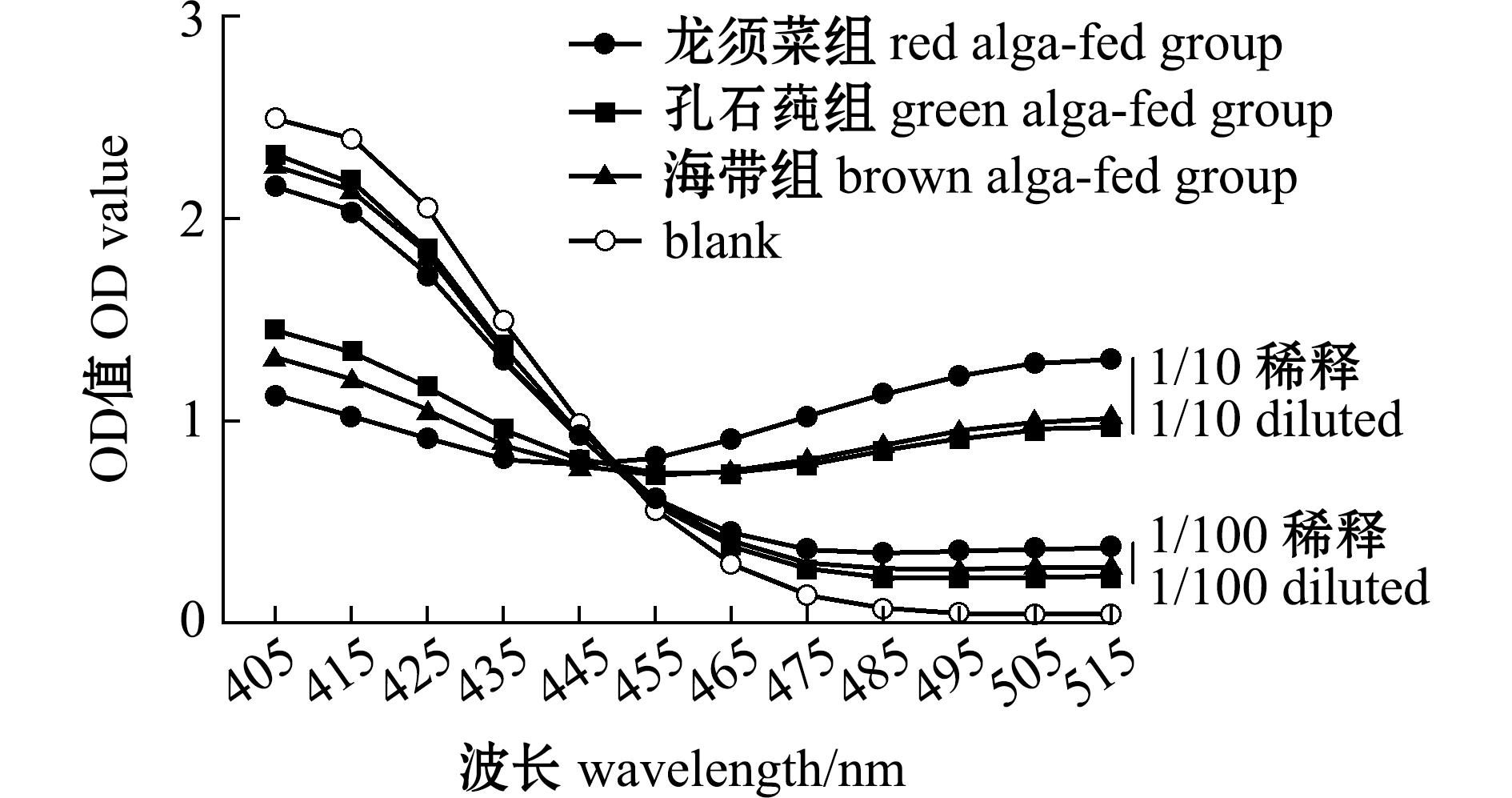
图2 皱纹盘鲍消化腺粗提液的硫酸酯酶活性
Fig.2 Sulfatase activity in the crude digestive-gland extracts of disk abalone
2.3 皱纹盘鲍硫酸酯酶ids-1的基因克隆及序列特征
克隆测序结果显示,皱纹盘鲍ids-1基因的开放阅读框长度为1 767 bp,可编码588个氨基酸,所编码蛋白的理论相对分子质量为6 728 000,理论等电点为8.45。进一步分析发现,IDS-1蛋白具N端信号肽结构(1~23 aa)、保守的硫酸酯酶结构域(28~376 aa)及9个糖基化位点(图3)。同源比对结果显示,该蛋白在皱纹盘鲍中尚无报道,其与红鲍IDS相似性最高,序列一致性达98%;其次为黑唇鲍IDS,相似性为92%。

小写字母为非翻译区(UTR)序列;大写字母为编码区序列;下划线为预测的信号肽;灰色阴影表示硫酸酯酶的保守结构域;黑色圆框为预测的糖基化位点;双下划线为3′UTR多聚腺苷酸化的加尾信号。
Letters represent UTR regions;capital letter represents the coding sequences;the putative signal peptide is underlined;the conserved sulfatase domain is shown in grey;black circle represents the potential glycosylation sites;putative poly(A) signal sequences are underlined doubly.
图3 皱纹盘鲍ids-1基因的核苷酸序列和推导的氨基酸序列
Fig.3 Nucleotide and deduced amino acid sequences of ids-1 from disk abalone
2.4 皱纹盘鲍硫酸酯酶IDS-1的系统发育分析及蛋白多重比对
利用皱纹盘鲍硫酸酯酶IDS-1的蛋白序列,经同源比对后,从NCBI、Uniprot和Ensembl数据库中选取代表性物种的硫酸酯酶蛋白序列,参照人类硫酸酯酶命名方式[10,23]将序列信息汇总(表2),并对其进行了系统发育分析,结果如图4所示。所选的代表性硫酸酯酶在系统发生树上分为4个明显的分支,代表已报道的不同功能的硫酸酯酶蛋白[28];其中亲缘关系最近的皱纹盘鲍、红鲍、黑唇鲍的IDS聚在一小支,三者同源性最高。

Hdis—皱纹盘鲍;Hruf—红鲍;Hrub—黑唇鲍;Cgig—长牡蛎;Pcan—福寿螺;Osin—章鱼;Dmel—黑腹果蝇;Drer—斑马鱼;Hsap—智人;ARS—芳基硫酸酯酶;IDS—艾杜糖醛酸2-硫酸酯酶;SGSH—乙酰肝素N-硫酸酯酶;STS—甾类硫酸酯酶;SULF—硫酸酯酶。
Hdis—Haliotis discus hannai;Hruf—Haliotis rufescens;Hrub—Haliotis rubra;Cgig—Crassostrea gigas;Pcan—Pomacea canaliculata;Osin—Octopus sinensis;Dmel—Drosophila melanogaster;Drer—Danio rerio;Hsap—Homo sapiens;ARS—arylsulfatase;IDS—iduronate 2-sulfatase;SGSH—N-sulfoglucosamine sulfohydrolase;STS—steroid sulfatase;SULF—sulfatase.
图4 硫酸酯酶氨基酸序列的系统演化树
Fig.4 Phylogenetic analysis of amino acid sequence of sulfatases
通过MAFFT软件进一步对所选8个物种的IDS进行了氨基酸序列比对分析[29]。从图5可见,硫酸酯酶IDS在物种间具较高的保守性,均具有硫酸酯酶的典型五肽基序“CXPXR”,而且10个主要的活性位点(D36、D37、C75、R79、K126、H129、H220、D325、H326和K339)在皱纹盘鲍和7个物种中均高度保守。

图5 皱纹盘鲍与其他物种IDS的多序列比对
Fig.5 Multiple alignment of IDS sequences between Haliotis discus hannai and other species
2.5 皱纹盘鲍ids-1基因的表达特征
为了进一步探讨不同藻类饵料与ids-1基因的表达是否有关联,分别检测并比较了其在孔石莼、海带和龙须菜喂养组皱纹盘鲍消化腺中的表达情况。从图6可见:转录组测序结果中ids-1基因表达量的变异程度较大(图6(a)),该基因在3组饵料喂养组中均有表达,其中海带组的表达水平高于其他两组,但无显著性差异。实时荧光定量PCR检测结果显示,在不同藻类喂养组中,ids-1基因的表达水平存在显著性差异。其中,海带喂养组的表达量最高,龙须菜喂养组次之,而孔石莼喂养组的表达量最低(图6(b))。

*表示组间有显著性差异;**表示P<0.01;***表示P<0.001.
*means the difference between groups;**means P<0.01;***means P<0.001.
图6 不同藻类饲喂条件下皱纹盘鲍消化腺ids-1基因表达特征
Fig.6 Expression profiles of ids-1 mRNA in digestive gland of Haliotis discus hannai under different algae feeding conditions
本研究中还通过实时荧光定量PCR分析了ids-1在皱纹盘鲍成体组织中的表达模式。 ids-1在消化腺、鳃、外套膜和上足触手组织中均有表达,其中消化腺表达量最高,其次为鳃和外套膜,上足触手组织中的表达量最低(图7(a))。随后,利用皱纹盘鲍早期胚胎、幼虫及成体样品分析了ids-1在皱纹盘鲍发育过程中的表达特征(图7(b))。总的来说,ids-1在皱纹盘鲍从受精卵至浮游幼虫的早期发育阶段表达量较低;至第5天的匍匐幼虫期,其表达量显著升高;而成体鲍的表达量比匍匐幼虫期又提高了约14倍。

0 h—受精卵时期;6 h—原肠胚时期;12 h—担轮幼虫时期;60 h—面盘幼虫期;5 d—匍匐幼虫期;adult—1龄鲍混合组织;*表示P<0.05;**表示P<0.01;****表示P<0.000 1。
0 h—fertilized eggs;6 h—gastrula stage;12 h—trochophore stage;60 h—veliger larvae stage;5 d—plantigrade stage;adult—mixed tissues of one year-old abalone;*means the difference between groups,P<0.05;**means P<0.01;****means P<0.000 1.
图7 皱纹盘鲍ids-1的表达特征
Fig.7 Expression profiles of ids-1 mRNA in Haliotis discus hannai
2.6 皱纹盘鲍IDS-1的重组表达与酶活检测
经SDS-PAGE检测,含重组质粒的大肠杆菌经IPTG诱导后,在70 000左右有明显的蛋白表达条带,表达形式为包涵体(图8(a))。通过His标签蛋白琼脂糖纯化树脂纯化重组蛋白,透析复性后,采用假底物4-硝基邻苯二酚硫酸二钾盐检测其硫酸酯酶的活性(图8(b)),在515 nm处未出现分解产物的特征吸收峰,表明该重组蛋白无明显的硫酸酯酶活性。

M—标准蛋白marker;1—未诱导的全菌液;2—诱导表达菌液的上清部分;3—诱导表达菌液的沉淀部分;4—纯化的重组蛋白。红色箭头指示目的蛋白条带。
M—standard protein marker;1—uninduced whole bacterial lysate;2—supernatant of induced bacterial lysate;3—pellet of induced bacterial lysate;4—purified recombinant protein.The IDS-1 protein is marked with a red arrow.
图8 IDS-1重组蛋白的SDS-PAGE分析及酶活检测
Fig.8 SDS-PAGE analysis and sulfatase activity analysis of recombinant IDS-1 protein
3 讨论
3.1 硫酸酯酶基因表达受摄食藻类影响
硫酸酯酶基因分布广,在从细菌到真核生物的大多数物种中均有报道。其蛋白在序列和功能上高度保守,蛋白家族成员多,包括芳基硫酸酯酶(ARS)、艾杜糖硫酸酯酶(IDS)等在内的多个亚家族[10]。转录组数据的 GO 富集结果显示,3组藻类饲喂后的差异表达基因在糖胺聚糖分解代谢、溶酶体、内质网等条目均有富集。相关研究表明,硫酸酯酶多存在于细胞的溶酶体中,其主要功能为分解糖胺聚糖类化合物,参与相关生命活动的调节[30],富集分析的结果暗示了皱纹盘鲍所摄食的海藻种类会对硫酸酯酶基因家族的表达造成影响。而在分子功能(molecular function)大类中,差异基因富集到了GO:0004423(iduronate-2-sulfatase activity)中,表明在不同藻类喂养下皱纹盘鲍硫酸酯酶基因会发生差异表达。
3.2 皱纹盘鲍硫酸酯酶ids-1的序列特征
本研究中克隆获得了皱纹盘鲍的硫酸酯酶ids-1基因。皱纹盘鲍IDS-1编码588个氨基酸,具有硫酸酯酶的保守结构域,N端有23个氨基酸的信号肽,并具有广泛的糖基化位点。由于物种亲缘关系较近,皱纹盘鲍与红鲍、黑唇鲍的IDS不仅具有氨基酸序列上的高度一致性,在系统发育分析上也呈现出高度的同源性。通过多序列比对发现,皱纹盘鲍IDS-1与软体动物、节肢动物甚至脊椎动物IDS间的序列保守性均较高,含有硫酸酯酶家族高度保守的五肽基序“C-X-P-X-R”和IDS亚家族高度保守的10个活性位点(D36、D37、C75、R79、K126、H129、H220、D325、H326和K339)。人细胞溶酶体内的IDS基因突变,会使体内的黏多糖代谢发生障碍,从而导致Ⅱ型黏多糖贮积症的发生[31]。但是,IDS-1在皱纹盘鲍中的表达特征及相关功能尚无报道。
3.3 皱纹盘鲍硫酸酯酶ids-1的表达特征
一直以来,软体动物的消化腺是分离、纯化硫酸酯酶的重要来源[19]。在红鲍中,也早有硫酸酯酶获得纯化的报道,表明硫酸酯酶在食藻动物对硫酸多糖的分解代谢中可能发挥重要作用[27]。与上述研究结果相一致,利用皱纹盘鲍的消化腺粗提液,在较低的浓度下依然可以检测到硫酸酯酶的酶活信号,说明皱纹盘鲍消化腺中含有丰富的硫酸酯酶。进一步的组织表达模式分析显示,ids-1基因在皱纹盘鲍消化腺中的表达量显著高于其他组织,也表明该基因与消化腺的代谢功能密切相关。此外,ids-1在鲍的鳃和外套膜等其他组织中也有表达,对于ids-1在这些组织中所发挥的作用,还需进行更深入的研究。
皱纹盘鲍ids-1具有明显的发育阶段表达特异性,从受精卵到浮游幼虫期间表达量较低,至匍匐幼虫期表达量开始升高,在成体鲍中表达量最高,达到匍匐幼虫期的14倍左右。鲍在浮游幼虫时期营养模式为自养型,所需的代谢能量完全来源于胚胎的卵黄储备,幼虫发育不需要外源食物[27]。皱纹盘鲍在个体发育史中发生了从浮游生活到底栖生活的转变,在变态发育过程中会伴随着食性的转换,从以卵黄颗粒为主的营养模式(受精后0 h至60 h)逐渐转换为摄食硅藻、扁藻等底栖单胞藻类[4](受精后5 d至稚贝早期),待消化系统和摄食器官逐渐成熟,其主要饵料逐渐转为大型藻类(稚贝后期至成体)。皱纹盘鲍ids-1在发育过程中开始大量表达的时间与皱纹盘鲍营养模式转换的时间相吻合,这表明ids-1可能参与了皱纹盘鲍对饵料的适应调节过程。
本研究中在对3种藻类喂养下皱纹盘鲍内脏团转录组比较的分析中,发现了差异基因多在与分解和代谢有关的条目中富集,如糖胺聚糖降解、碳水化合物衍生物的分解代谢、硫酸化合物的分解代谢及一些氨基酸的代谢等。这表明,采用不同种类的藻类喂养皱纹盘鲍,会影响消化吸收相关基因的表达量,也证明皱纹盘鲍在基因水平上存在着对不同饵料的适应机制。在已有对皱纹盘鲍摄食的相关研究中,成年皱纹盘鲍对不同种类的藻类表现出偏好性,如对褐藻的喜好程度高于红藻与绿藻[32]。皱纹盘鲍喜欢摄食叶片较大且能在上面爬行的藻类,对海带的摄食率会高于其他形状较细的大型藻类[33]。石莼类海藻由于藻体较薄,会在海水中漂浮,较大的浮动会影响鲍的刮食性摄食,降低鲍的摄食效率[34]。本研究中发现,在不同藻类喂养的皱纹盘鲍消化腺中,ids-1的表达差异十分显著,在海带(褐藻)喂养组中,其表达量显著高于龙须菜(红藻)组和孔石莼(绿藻)组,其中孔石莼喂养组的基因表达量最低。有研究表明,硫酸化结合位点在糖类结构上的不同会使硫酸酯酶的偏好性存在显著差异[35],ids-1在不同饵料条件下的表达差异,可能与皱纹盘鲍对不同藻类的摄食偏好性及摄食量等直接相关。
3.4 皱纹盘鲍硫酸酯酶IDS-1蛋白的功能分析
本研究中利用原核表达系统获得了皱纹盘鲍硫酸酯酶IDS-1重组蛋白,但该重组蛋白并未显示硫酸酯酶的活性。据报道,IDS-1属于甲酰甘氨酸依赖性硫酸酯酶,其蛋白活性依赖于特殊的翻译后修饰,由硫酸酯酶修饰因子(sulfatase modifying factor,SUMF)又称甲酰甘氨酸生成酶(formylglycine-generating enzyme,FGE)所完成。内质网的SUMF特异性识别新合成的硫酸酯酶蛋白N端的保守基序“C-X-P-X-R”,将半胱氨酸转化为甲酰基甘氨酸,此翻译后修饰对硫酸酯酶的活性至关重要[36-37]。据推测,无活性的IDS-1重组蛋白可能是由于缺少必需的翻译后修饰,后续将采用共表达IDS-1与其修饰酶SUMF的策略,以期获得具有活性的IDS-1蛋白,继续探究其在皱纹盘鲍饵料适应过程中的作用。
4 结论
1)在3组不同藻类饲喂的皱纹盘鲍内脏团差异表达基因的筛选中,硫酸酯酶家族得到显著富集,表明此类基因参与了藻类的分解代谢过程。
2)皱纹盘鲍的硫酸酯酶ids-1基因,在氨基酸序列上不仅具有硫酸酯酶家族所特有的“C-X-P-X-R”五肽结构,而且存在IDS亚家族中高度保守的活性位点,表明皱纹盘鲍IDS-1在分子结构上具保守性。但是,利用原核表达系统获得的IDS-1重组蛋白并未显示硫酸酯酶的活性,表明翻译后的修饰作用为其活性所必需。
3)ids-1的表达水平在不同藻类喂养组中有显著性差异,表明该基因的表达受到摄食偏好性及摄食量差异的直接影响;ids-1在鲍不同组织中差异表达,消化腺中表达量较高,表明该基因可能与食物的代谢和利用相关;此外,ids-1还呈现了发育阶段的表达特异性,这与皱纹盘鲍的营养模式及食性转换密切相关,表明该基因可能参与了鲍的饵料适应调节过程。
[1] 王腾,赵新楠,林怡辰,等.鲍营养品质及影响因素研究进展[J].大连海洋大学学报,2024,39(1):172-184.
WANG T,ZHAO X N,LIN Y C,et al.Research progress on nutritional quality and influencing factors related nutrients in abalone: a review[J].Journal of Dalian Ocean University,2024,39(1):172-184.(in Chinese)
[2] DENG Y,LIU X,ZHANG G.Genotype-environment interaction of growth in Haliotis discus hannai[J].Qindao:Marine Sciences,2005,29(12):31.(in Chinese)
[3] 栾淼,杨陆飞,高峰,等.皱纹盘鲍深远海养殖技术研究[J].科学养鱼,2023(5):66-67.
LUAN M,YANG L F,GAO F.Study on culture technique of disk abalone in deep sea[J].Scientific Fish Farming,2023(5):66-67.(in Chinese)
[4] 陈木,卢豪魁,陈世杰,等.皱纹盘鲍人工育苗的初步研究[J].动物学报,1977(1):35-46.
CHEN M,LU H K,CHEN S J,et al.A preliminary study on the artificial rearing of the larvae and juveniles of Haliotis discus hannai Ino[J].Current Zoology,1977(1): 35-46.(in Chinese)
[5] GRIFFITHS J S,SAHASRABUDHE R M,MARIMUTHU M P A,et al.A draft reference genome of the red abalone,Haliotis rufescens,for conservation genomics[J].The Journal of Heredity,2022,113(6):673-680.
[6] BARKAN R,COOKE I,WATSON S A,et al. Chromosome-scale genome assembly of the tropical abalone (Haliotis asinina) [J]. Scientific Data,2024. 11(1): 999.
[7] 刘莉莉,问莉莉,李思东.海藻营养成分及高值化利用的研究进展[J].轻工科技,2012,28(3):10-11,23.
LIU L L,WEN L L,LI S D.Research progress on nutritional components and high-value utilization of seaweeds[J].Guangxi Journal of Light Industry,2012,28(3):10-11,23.(in Chinese)
[8] LYU Q Q,JIAO W Q,ZHANG K K,et al.Proteomic analysis of scallop hepatopancreatic extract provides insights into marine polysaccharide digestion[J].Scientific Reports,2016,6:34866.
[9] DODGSON K S,SPENCER B.Studies on sulphatases.Ⅶ.A preliminary account of the glycosulphatase of Littorina littorea[J].The Biochemical Journal,1954,57(2):310-315.
[10] DIEZ-ROUX G,BALLABIO A.Sulfatases and human disease[J].Annual Review of Genomics and Human Genetics,2005,6(1):355-379.
[11] MITSUNAGA-NAKATSUBO K,AKIMOTO Y,KAWAKAMI H,et al.Sea urchin arylsulfatase,an extra cellular matrix component,is involved in gastrulation during embryogenesis[J].Development Genes and Evolution,2009,219(6):281-288.
[12] RAGSDALE E J,MÜLLER M R,RÖDELSPERGER C,et al.A developmental switch coupled to the evolution of plasticity acts through a sulfatase[J].Cell,2013,155(4):922-933.
[13] FREDERICK A R,LEE A M,WEHRLE B A,et al.Abalone under moderate heat stress have elevated metabolic rates and changes to digestive enzyme activities[J].Comparative Biochemistry and Physiology Part A:Molecular &Integrative Physiology,2022,270:111230.
[14] ROY A B.The sulphatase of ox liver.1.The complex nature of the enzyme[J].Biochemical Journal,1953,53(1):12-15.
[15] NAM B H,KWAK W,KIM Y O,et al.Genome sequence of Pacific abalone (Haliotis discus hannai):the first draft genome in family Haliotidae[J].GigaScience,2017,6(5)[2024-03-12].https://academic.oup.com/gigascience/article/doi/10.1093/gigascience/gix014/3062834.DOI:10.1093/gigascience/gix014.
[16] 李明珠,麦康森,何艮,等.实时荧光定量PCR检测皱纹盘鲍Δ5脂肪酸去饱和酶基因表达方法的建立及应用[J].水生生物学报,2014,38(2):328-334.
LI M Z,MAI K S,HE G,et al.Establishment and application of real-time quantitative pcr reaction system for study in gene expression of δ5 fatty acyl desaturase in abalone (Haliotis discus hannai Ino)[J].Acta Hydrobiologica Sinica,2014,38(2):328-334.(in Chinese)
[17] SCHMITTGEN T D,LIVAK K J.Analyzing real-time PCR data by the comparative CT method[J].Nature Protocols,2008,3:1101-1108.
[18] KIM D,LANGMEAD B,SALZBERG S L.Hisat:a fast spliced aligner with low memory requirements[J].Nature Methods,2015,12(4):357-360.
[19] PERTEA M,KIM D,PERTEA G M,et al.Transcript-level expression analysis of RNA-seq experiments with HISAT,StringTie and Ballgown[J].Nature Protocols,2016,11(9):1650-1667.
[20] LOVE M I,HUBER W,ANDERS S.Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J].Genome Biology,2014,15(12):550.
[21] YU G,WANG L G,HAN Y,et al.ClusterProfiler: an R package for comparing biological themes among gene clusters[J].OMICS: A Journal of Integrative Biology 2012,16(5):284-287.
[22] 袁佳.R语言及ggplot2在环境空气监测数据可视化中的应用[J].中国高新技术企业,2015(11):88-91.
YUAN J.Application of R language and ggplot2 in visualization of ambient air monitoring data[J].China High-Tech Enterprises,2015(11):88-91.(in Chinese)
[23] DODGSON K S,POWELL G M.Studies on sulphatases 26.Arylsulphatase activity in the digestive juice and digestive gland of Helix pomatia[J].The Biochemical Journal,1959,73(4):666-671.
[24] LLOYD P F,LLOYD K O,OWEN O.Carbohydrate sulphatases of marine molluscs assay of glycosulphatases[J].Biochem,1962,85(1):193-198.
[25] HATANAKA H,OGAWA Y,EGAMI F.Arylsulphatase and glycosulphatase of Charonia lampas.substrate specificity towards sugar sulphate derivatives[J].The Biochemical Journal,1976,159(2):445-448.
[26] JANER G,MESIA-VELA S,KAUFFMAN F C,et al.Sulfatase activity in the oyster Crassostrea virginica:its potential interference with sulfotransferase determination[J].Aquatic Toxicology,2005,74(1):92-95.
[27] SPAULDING D C,MORSE D E.Purification and characterization of sulfatases from Haliotis rufescens:evidence for changes in synthesis and heterogeneity during development[J].Journal of Comparative Physiology B,Biochemical,Systemic,and Environmental Physiology,1991,161(5):498-515.
[28] PARENTI G,MERONI G,BALLABIO A.The sulfatase gene family[J].Current Opinion in Genetics &Development,1997,7(3):386-391.
[29] 乔琨,郑春花,陈贝,等.皱纹盘鲍肽聚糖识别蛋白的表达及免疫功能特性[J].大连海洋大学学报,2023,38(6):947-955.
QIAO K,ZHENG C H,CHEN B,et al.Expression and immune function characteristics of peptidoglycan recognition protein gene in disk abalone(Haliotis discus hannai)[J].Journal of Dalian Ocean University,2023,38(6):947-955.(in Chinese)
[30] HUANG Y F,MIZUMOTO S,FUJITA M.Novel insight into glycosaminoglycan biosynthesis based on gene expression profiles[J].Frontiers in Cell and Developmental Biology,2021,9:709018.
[31] 朱翊,傅得兴.艾度硫酸酯酶的药理与临床评价[J].中国新药杂志,2008,17(9):798-799.
ZHU Y,FU D X.Pharmacology and clinical evaluation of idursulfase[J].Chinese Journal of New Drugs,2008,17(9):798-799.(in Chinese)
[32] 胡耿,郭继春,刘德斌.鲍对海藻的摄食选择与利用效果[J].饲料研究,2016(16):8-10,32.
HU G,GUO J C,LIU D B.Feeding choice and utilization effect of abalone on seaweed[J].Feed Research,2016(16):8-10,32.(in Chinese)
[33] 吴永沛,陈昌生,蔡慧农,等.人工饲料及海藻养成九孔鲍营养成分的比较[J].海洋科学,2000,24(9):4-6.
WU Y P,CHEN C S,CAI H N,et al.Comparative studies on the effect of different nutritional compositions on the abalone Haliotis diversi color supertexta fed on artificial diets and natural seaweed diets[J].Marine Sciences,2000,24(9):4-6.(in Chinese)
[34] 任黎华,张继红,王文琪,等.饵料对不同规格皱纹盘鲍能量收支的影响[J].渔业科学进展,2011,32(5):51-57.
REN L H,ZHANG J H,WANG W Q,et al.Effects of three dietary treatments on the energy budget of different sized Haliotis discus hannai Ino[J].Progress in Fishery Sciences,2011,32(5):51-57.(in Chinese)
[35] LOFT K J,BOJAROV P,SL
P,SL MOV
MOV K,et al.Synthesis of sulfated glucosaminides for profiling substrate specificities of sulfatases and fungal beta-N-acetylhexosaminidases[J].Chembiochem,2009,10(3):565-576.
K,et al.Synthesis of sulfated glucosaminides for profiling substrate specificities of sulfatases and fungal beta-N-acetylhexosaminidases[J].Chembiochem,2009,10(3):565-576.
[36] ALM CIGA-D
CIGA-D AZ C J,TOLOSA-D
AZ C J,TOLOSA-D AZ A D,PIMENTEL L N,et al.Anaerobic sulfatase maturase AslB from Escherichia coli activates human recombinant iduronate-2-sulfate sulfatase (IDS) and N-acetylgalactosamine-6-sulfate sulfatase (GALNS)[J].Gene,2017,634:53-61.
AZ A D,PIMENTEL L N,et al.Anaerobic sulfatase maturase AslB from Escherichia coli activates human recombinant iduronate-2-sulfate sulfatase (IDS) and N-acetylgalactosamine-6-sulfate sulfatase (GALNS)[J].Gene,2017,634:53-61.
[37] BERTEAU O,GUILLOT A,BENJDIA A,et al.A new type of bacterial sulfatase reveals a novel maturation pathway in prokaryotes[J].The Journal of Biological Chemistry,2006,281(32):22464-22470.