POU(POU domain transcription factor)家族基因是由POU 结构域和Homeobox结构域共同组成的转录因子,由其家族起始成员Pit-1、Oct-1、Oct-2和Unc-86的首字母缩写POU命名[1-2]。POU家族基因通过同源或异源二聚体的形式与靶基因转录因子结合位点(transcription factor binding site,TFBS)(5′ATGCAAAT 3′或5′ATGCTAAT 3′)结合发挥正向或负向转录调控作用[1,3]。POU 结构域由 POUS和 POUH链段形成,由灵活的接头连接,POUS和 POUH分别可以与ATGC和AAAT/TAAT区域结合[4]。目前,POU家族基因的研究主要集中在脊椎动物,如在人类(Homo sapiens)中共发现了17个POU家族基因,主要在神经系统发育等过程中发挥重要功能[5]。但目前在无脊椎动物中,仅在黑腹果蝇(Drosophila melanogaster)、合浦珠母贝(Pinctada fucata)和中华卤虫(Artemia sinica)等物种中鉴定到POU3、POU5、POU2及POU4等POU家族基因[6-7],对其功能研究还十分有限。
POU家族基因通过调控靶基因的转录过程,在生物响应环境胁迫过程中发挥重要作用。如在高温胁迫下,鸡(Gallus gallus)POU2F1可以通过结合热休克蛋白70(heat shock 70 000 protein,HSP70)启动子区域进而调控HSP70的表达[8]。POU2F2通过促进 Sestrin2表达抑制小鼠(Mus musculus)神经元细胞的氧化应激和铁死亡[9]。近年研究发现,POU家族基因能够通过调控Bcl-2(B-cell lymphoma-2)等凋亡相关分子的表达参与调节细胞凋亡过程。如POU家族基因成员Brn-3a可以刺激小鼠神经突起生长的能力,并通过诱导Bcl-2基因的表达保护神经元细胞免于凋亡[7]。
长牡蛎(Crassostrea gigas)是重要的水产养殖经济贝类,通常生活在河口和潮间带区域,为研究贝类环境胁迫响应机制的模式物种之一[10]。目前养殖长牡蛎存在夏季高温、盐度变化及病原微生物感染等生物与非生物胁迫问题[11]。已有研究表明,细胞凋亡是生物响应环境胁迫的重要生理过程,前期研究发现,长牡蛎基因组中凋亡相关基因出现大量扩张[12]。高温胁迫下,长牡蛎通过调控与能量代谢相关的基因、未折叠蛋白反应(unfolded protein response,UPR)和抗凋亡通路等维持基本的生命活动[13-14]。目前,关于长牡蛎凋亡响应高温胁迫的研究主要集中在凋亡过程发生机制,关于上游转录调控的研究还较少。本研究中,利用生物信息学分析、逆转录定量PCR(reverse transcription quantitative PCR,RT-qPCR)和流式细胞术等分析了转录因子POU家族基因的进化和分子特征,明确了新发现的CgPOU3F4L基因在高温胁迫下的表达模式,揭示了其在长牡蛎血淋巴细胞凋亡过程中的调控作用,以期为进一步了解牡蛎POU家族基因的结构和功能特征提供参考依据。
1 材料与方法
1.1 材料
试验用2龄长牡蛎采集自大连庄河市长牡蛎养殖海区。试验用于大肠杆菌(Escherichia coli)Trans5α化学感受态细胞购自北京全式金生物技术有限公司。
1.2 方法
1.2.1 样品采集 将采集回的长牡蛎于(15±1) ℃海水中暂养7 d后,在靠近闭壳肌壳缘部分钻直径为1 mm的孔,再次暂养7 d[15]。随机选取9 只长牡蛎,取血淋巴以800 g、4 ℃下离心10 min,去上清后加入PBS重悬血淋巴细胞,将重悬后的液体移入1.5 mL EP管中,再次以800 g、4 ℃下离心10 min,去上清,向管中加入1 mL TRIzolTM试剂重悬;采集鳃、肝胰腺、闭壳肌、外套膜、唇瓣和性腺等组织,加入1 mL TRIzolTM试剂,存放于-80 ℃超低温冰箱中。随机选取72只长牡蛎进行28 ℃高温水浴处理,在处理后0、3、6、12、24、36、48、72 h采集试验动物样品,每个时间点随机挑选9只长牡蛎,每3只混合为一个平行,抽取血淋巴,以800 g、4 ℃下离心10 min收集血淋巴细胞并重悬于1 mL TRIzolTM中保存,存放于-80 ℃超低温冰箱中备用。
1.2.2 POU家族基因鉴定 采用本地化BLASTP程序,将进化代表物种的POU氨基酸序列(附表1)比对到长牡蛎基因组蛋白模型数据库中(Genbank Bioproject ID:PRJNA276446和PRJNA598006)(e value<1×10-10),筛选潜在的POU家族基因,进化代表物种包括模式动物斑马鱼(Danio rerio)、热带爪蟾(Xenopus tropicalis)、绿蜥蜴(Anolis carolinensis)、鸡、小鼠和人类,头索动物厦门文昌鱼(Branchiostoma belcheri)、尾索动物玻璃海鞘(Ciona intestinalis)、半索动物柱头虫(Saccoglossus kowalevskii)、棘皮动物紫色球海胆(Strongylocentrotus purpuratus)、软体动物虾夷扇贝(Mizuhopecten yessoensis)和加州海兔(Aplysia californica)、蜕皮动物线虫(Caenorhabditis elegans)。采用CD-hit程序(c=0.99)根据序列的相似度对序列进行聚类和去冗余[16]。采用Batch CDD-Search程序进行结构域比较及去除非目的基因序列[17]。采用ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)查找序列中所有可能的ORF区,去除ORF区不完整的序列。利用本地化tBLASTN程序将筛选得到的POU基因序列比对到基因组序列上,根据比对结果进行POU家族基因筛选。
表1 试验所用引物
Tab.1 Primers used in this study

引物 primer序列 sequence(5′-3′)用途 usageCgPOU3F4LF:TGCCTCTACCTATAATGGR:GTCCTGTCAGTGCTTTTGPCRCgPOU3F4L-RTF:TGTGTTTTCCCTTCTTTGGCATR:TGCTGTGTGAGGGCTATTCGRT-qPCRCgBcl-2-RTF:ATCGTTGCTGTCTACACTTTTGCR:TGGTTCCCAATGTCACGAGART-qPCRCgCaspaase3-RTF:TCGGATACCCAAGAGCCAGAR:AGCTTCCCACACCACTCTTRT-qPCRCgEF-RTF:AGTCACCAAGGCTGCACAGAAAGR:TCCGACGTATTTCTTTGCGATGTRT-qPCRCgPOU3F4L-senseCGGAACCUUAUACGGUAAUTT(硫代修饰)siRNACgPOU3F4L-antisenseAUUACCGUAUAAGGUUCCGTT(硫代修饰)siRNANC-senseUUCUCCGAACGUGUCACGUTT(硫代修饰)siRNANC-antisenseACGUGACACGUUCGGAGAATT(硫代修饰)siRNA
1.2.3 POU基因结构域、系统进化分析、多序列比对及转录结合位点预测 采用SMART(http://www.smart.embl-heidelbe rg.de/)进行基因结构域分析,采用MEME(https://meme-suite.org/meme/tools/meme)工具进行motif位点分析,采用ExPASV Server(https://www.expasy.org/)软件进行蛋白分子理化性质预测。采用最大似然法(LG氨基酸替代模型)和本地化IQ-TREE2软件对POU 氨基酸序列进行系统发育分析,Bootstrap 为1 000[18],对POU氨基酸序列进行系统发育分析,用于分析的序列包括长牡蛎、人、小鼠、鸡和斑马鱼等物种的POU氨基酸序列(附表1)。采用GeneDoc程序(2.7版本)进行多序列比对[19]。采用iTOL(https://itol.embl.de/)网站对系统进化树进行格式调整。采用MATCH(https://gene-regulation.com)在线软件,矩阵组选择所有生物的免疫细胞类群库,以10%假阴性匹配(minFN10)为截取值,进行基因启动子区域TFBS预测[20]。
1.2.4 CgPOU3F4L基因克隆 根据鉴定到的CgPOU3F4L序列,采用Primer primer 5软件设计ORF框的克隆引物CgPOU3F4L-F和CgPOU3F4L-R(表1),采用Takara Ex Taq酶体系通过PCR扩增CgPOU3F4L的ORF序列。PCR反应条件为95 ℃下变性5 min;引物TM值温度退火30 s,72 ℃下延伸1 min 30 s,共进行25个循环,最后在72 ℃下再延伸10 min。PCR产物经凝胶纯化后插入pMD19-T克隆载体(Takara,日本),转化至大肠杆菌Trans5α化学感受态细胞进行测序验证。
1.2.5 总RNA 的提取和cDNA合成 将含有Trizol的组织样品用冷冻研磨仪进行研磨,按照体积比5∶1加入氯仿替代物,在4 ℃,12 000 g条件下离心15 min;吸取上清后加入等体积异丙醇,混匀后于-80 ℃下沉降10 min;4 ℃,12 000 g的条件下离心15 min,去上清后加入体积分数为75%的乙醇,上下混匀,洗涤RNA 沉淀,于4 ℃,12 000 g条件下离心5 min,弃掉上清,重复加入75%乙醇洗涤一次;干燥RNA后采用DEPC水溶解;采用Nanodrop 2000测定RNA浓度,并采用10 g/L的琼脂糖凝胶电泳,检测RNA的完整性;采用一步法gDNA去除和cDNA合成Supermix试剂盒(TransGen,中国)合成cDNA第一链。cDNA合成在42 ℃下恒温15 min,85 ℃下加热5 s及42 ℃下恒温30 min,85 ℃下加热5 s条件下进行反应,用于RT-qPCR和PCR将cDNA模板稀释20倍并保存在-80 ℃超低温冰箱用于后续试验。
1.2.6 siRNA干扰CgPOU3F4L基因试验 随机选取27只牡蛎进行siRNA干扰试验;分别将CgPOU3F4L及NC的siRNA干粉片段(吉玛生物,中国)采用灭菌海水进行稀释,向1 OD干粉中加入100 μL灭菌海水,使浓度达到0.01 OD/μL;试验组(命名为CgPOU3F4L-RNAi组)共9只牡蛎,每3只设为一个平行,向每只牡蛎注射100 μL稀释后的CgPOU3F4L的siRNA片段,在注射后12 h开始进行28 ℃高温处理;对照组(NC组)共9只牡蛎,每3只设为一个平行,向每只牡蛎注射100 μL稀释后的NC的siRNA片段,在注射后12 h开始进行28 ℃高温处理;空白组(blank组)牡蛎未接受任何处理。在高温处理24 h后收集血淋巴样品,步骤同“1.1节”,用于测定血淋巴细胞中凋亡相关基因(CgBcl-2及CgCaspase3)的mRNA表达水平及血淋巴细胞凋亡率。同时设计引物CgPOU3F4L-RT-F和CgPOU3F4L-RT-R(表1)进行RT-qPCR,对干扰效率进行评估。
1.2.7 CgPOU3F4L和凋亡相关基因mRNA表达水平分析 通过RT-qPCR检测长牡蛎CgPOU3F4L和细胞凋亡相关基因(CgBcl-2及CgCaspase3[21])的mRNA表达水平,采用延伸因子基因CgEF(GenBankID:NM_001305313)作为内参基因(表1)。RT-qPCR反应利用ABI PRISM 7500序列检测系统(Thermo Fisher,美国),采用SYBR premix ExTap(Thermo Fisher,美国)体系进行。反应总体积为10 μL,包括SYBR Green Master Mix 5 μL、正反引物各0.2 μL、ROX 0.2 μL、15倍稀释的cDNA 2 μL和RNA free water 2.4 μL。RT-qPCR在95 ℃下恒温10 min,95 ℃下恒温10 s,60 ℃下恒温45 s,共进行40个循环。在65~95 ℃进行熔解分析。采用2-ΔΔCt方法计算mRNA表达水平[22]。
1.2.8 高温胁迫后CgPOU3F4L-RNAi长牡蛎的血淋巴细胞凋亡率测定 采用Annexin V-FITC/PI双标记法,采用Annexin VFITC凋亡检测试剂盒(碧云天,中国),利用FCM(flow cytometry,流式细胞术)测定血淋巴细胞的凋亡率[23]。将采集的血淋巴在800 g,4 ℃下离心10 min,收集血淋巴细胞,采用L-15加盐培养基轻轻吹打。加入195 μL的Annexin V-FITC结合物,轻轻地重新悬浮5×104~105个细胞。加入5 μL 的Annexin V-FITC和10 μL的碘化丙啶(PI),在室温下(20 ℃~25 ℃,避光)孵育15 min,标记凋亡细胞。采用L-15加盐培养基将血淋巴细胞重悬并稀释,过筛后转移到玻璃圆底管中,采用FACS Aria II SORP流式细胞仪(BD,美国)测定细胞凋亡率。
1.3 数据处理
本研究中所有试验均设置 3个样品重复,试验数据采用平均值±标准差(mean±S.D.)表示,试验数据均采用IBM SPSS 24.0 软件进行单因素方差分析(one-way ANOVA)和多重比较(multiple comparisons),显著性水平设为0.05。采用GraphPad Prism 8软件绘制统计图表。
2 结果与分析
2.1 CgPOU家族基因鉴定及分子特征
为了鉴定长牡蛎POU家族基因,根据注释结果筛选候选CgPOU并将其比对到本地化长牡蛎转录组及基因组数据库。分析结果从长牡蛎基因组数据库中鉴定到4个CgPOU家族基因,分别为CgPOU2F1、CgPOU3F4、CgPOU4F3及CgPOU3F4L。CgPOU家族基因的开放阅读框(open reading frame,ORF)长度为967~1 248 bp。其中,CgPOU3F4L的开放阅读框最长为1 248 bp,可编码416个氨基酸,蛋白相对分子质量为46 060,理论等电点为7.31(表2)。通过基因和蛋白结构分析发现,CgPOU2F1、CgPOU3F4、CgPOU3F4L及CgPOU4F3均含有POU家族基因的典型特征结构域:POU结构域及Homeobox结构域(图1)。通过motif序列分析发现,CgPOU2F1、CgPOU3F4、CgPOU3F4L及CgPOU4F3均具有相同的motif1、motif2及motif3基序(图2)。
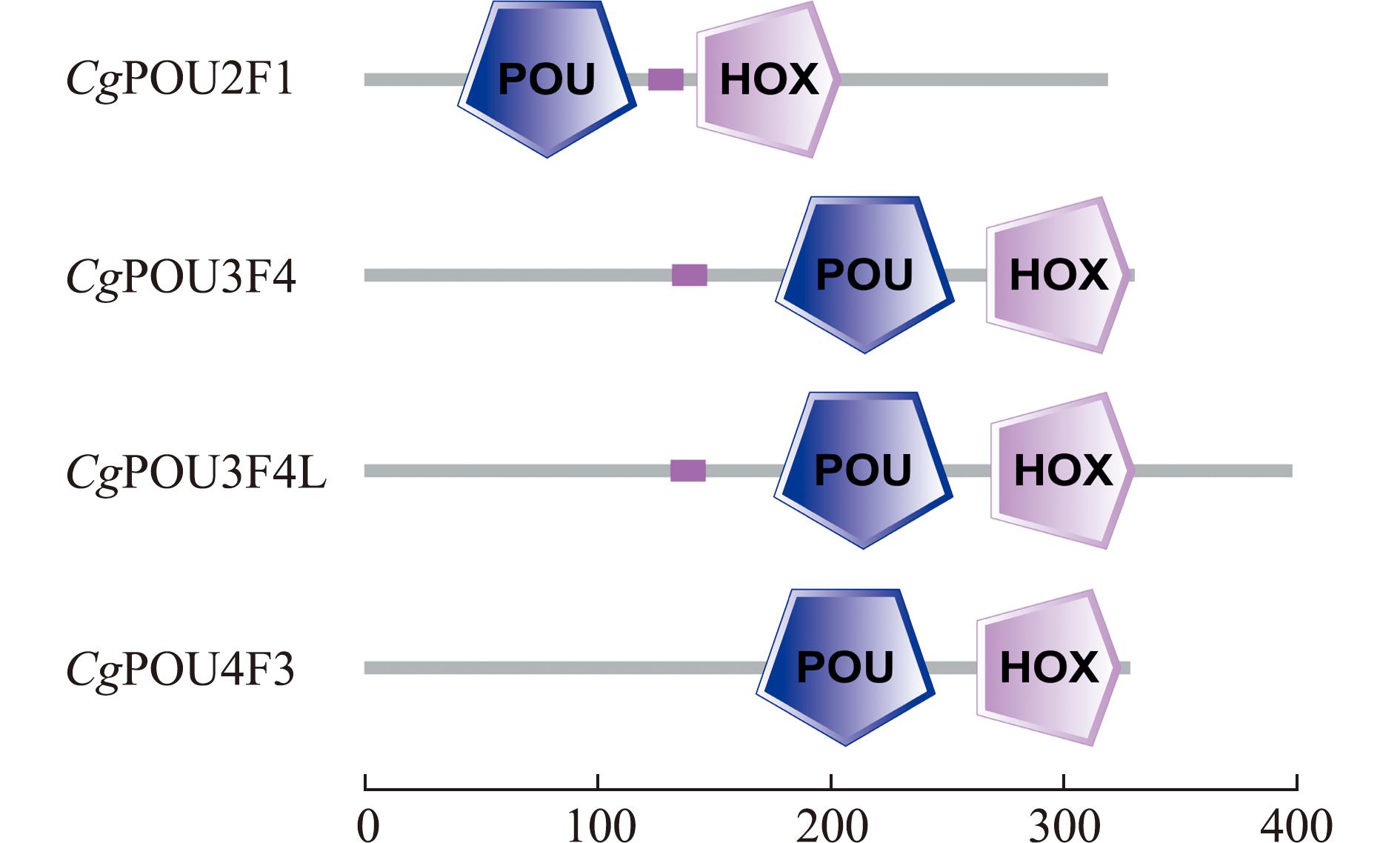
图1 CgPOU家族基因结构域分析
Fig.1 Domain analysis of CgPOU family genes

图2 CgPOU家族基因motif序列分析
Fig.2 Motif sequence analysis of CgPOU family genes
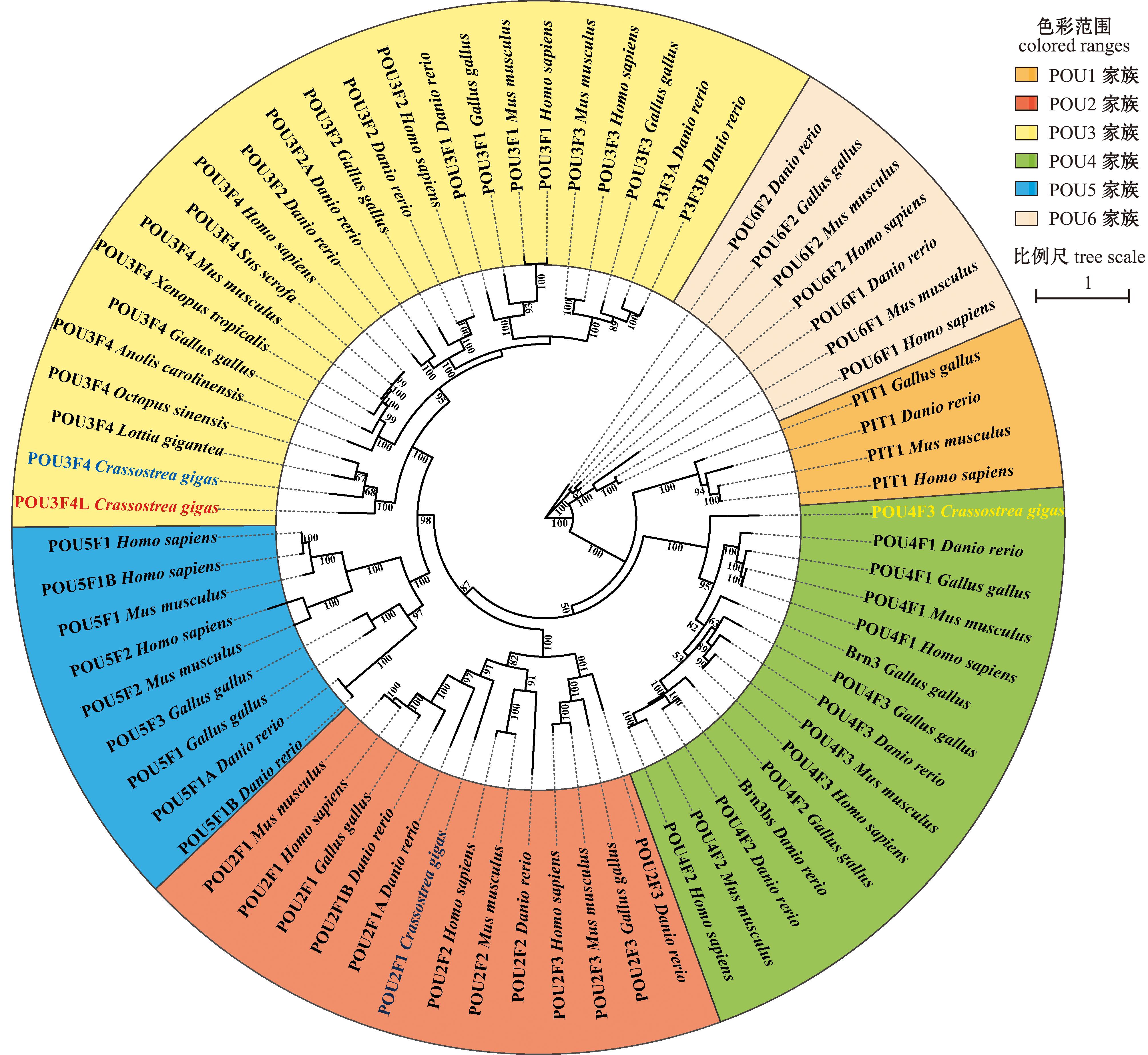
图3 POU家族基因氨基酸序列的系统进化树
Fig.3 Phylogenetic tree of amino acid sequences in POU family gene
表2 CgPOU家族基因及蛋白理化性质
Tab.2 Protein and physicochemical properties of CgPOU family genes
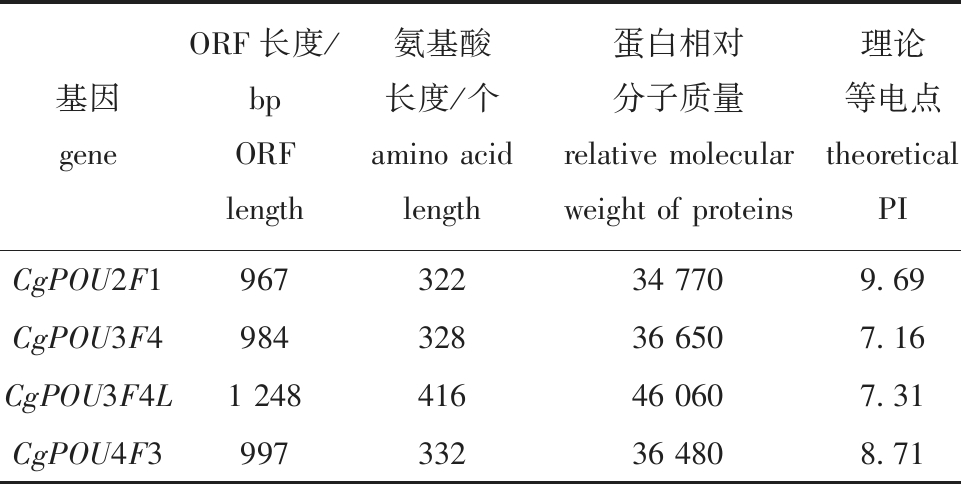
基因geneORF长度/bpORF length氨基酸长度/个amino acid length蛋白相对分子质量relative molecular weight of proteins理论等电点theoreticalPICgPOU2F196732234 7709.69CgPOU3F498432836 6507.16CgPOU3F4L1 24841646 0607.31CgPOU4F399733236 4808.71
2.2 CgPOU家族基因的系统进化分析及POU3F4基因的多序列比对
为了解析POU家族基因的系统进化关系,将获得的目的基因与模式物种POU家族的共67个基因采用本地化Muscle程序及IQ-Tree 2进行氨基酸多序列比对及系统进化分析,不同的基因家族采用不同颜色进行区分(图 3)。结果表明,POU家族基因进化均较为保守。长牡蛎中CgPOU2F1与其他物种的POU2成员共同聚为POU2子家族。CgPOU4F3与其他物种中POU4成员聚为POU4子家族。CgPOU3F4与霸王莲花青螺(Lottia gigantea)及中华真鞘(Octopus sinensis)中POU3F4聚为一支,再与CgPOU3F4L基因聚为一支后与其他物种POU3成员聚为POU3子家族,最后POU3家族与POU5家族聚为一支,进化关系较近。POU6家族与其他家族进化关系较远。多序列比对结果表明,长牡蛎CgPOU3F4和CgPOU3F4L均具有POU结构域和Homeobox结构域,但在CgPOU3F4L氨基酸序列的362~416区间处存在一段特有的氨基酸序列(图4)。

红色框代表POU结构,蓝色框代表Homeobox结构域,虚线框代表CgPOU3F4L的特有氨基酸序列。
The POU domain is shown in red box,the Homeobox domain is shown in blue box,and the specially conserved region is shown in dashed box.
图4 POU3F4基因氨基酸序列的多序列比对
Fig.4 Multiple sequence alignment of POU3F4 amino acid sequences
2.3 CgPOU3F4L基因mRNA在不同组织和高温胁迫后血淋巴细胞中的表达模式
本研究中在长牡蛎中鉴定到了CgPOU3F4和CgPOU3F4L,发现CgPOU3F4L具有一段特有的氨基酸序列,其所涉及的功能分化特征还不清楚,因此本研究中进一步研究了CgPOU3F4L的表达特征及对环胁迫的响应功能。组织分布表达试验结果发现,CgPOU3F4L基因在肝胰腺中的mRNA表达水平最低,在外套膜、闭壳肌和血淋巴细胞中mRNA表达水平较高,分别是肝胰腺中表达水平的91.32倍(P<0.05)、19.24倍(P<0.05)和7.92倍(P<0.05)(图5(a))。血淋巴细胞是长牡蛎响应高温胁迫的主要场所[14],因此检测了高温胁迫下CgPOU3F4L在长牡蛎血淋巴细胞中的表达水平变化,结果显示,高温胁迫下长牡蛎血淋巴细胞中CgPOU3F4L的表达水平呈现出先增加后减少的趋势,在高温胁迫后12、24、36 h显著升高,分别为0 h的2.63倍、3.05倍和3.62倍(P<0.05)(图5(b))。
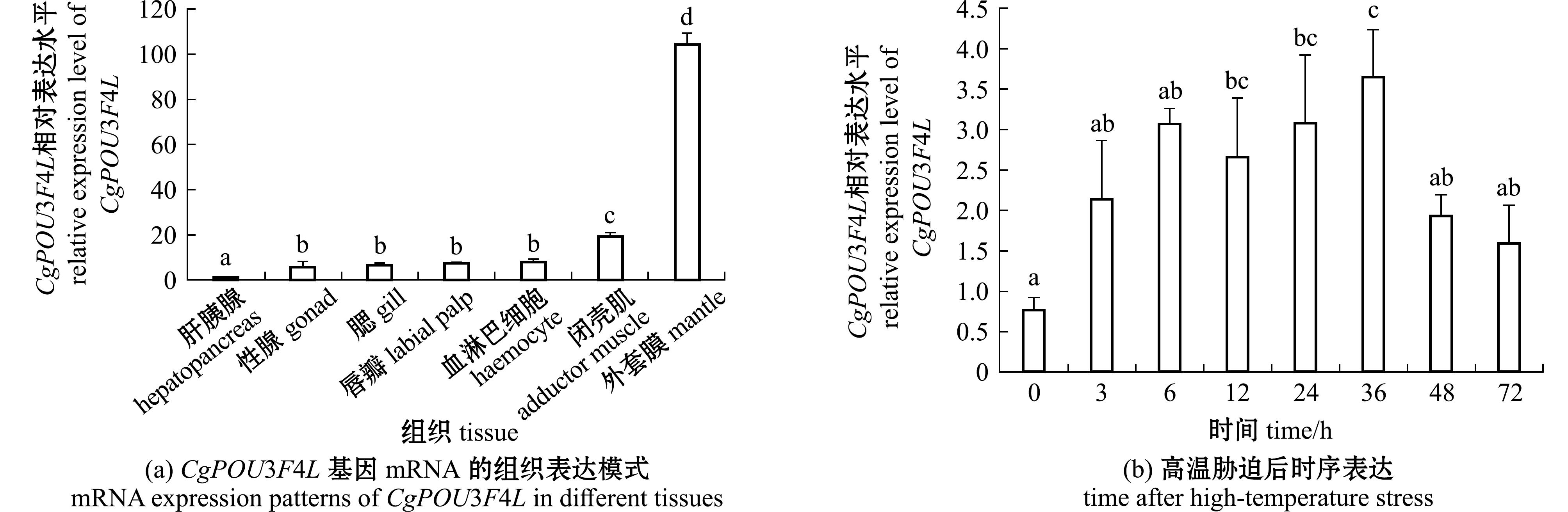
标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05),下同。
The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter are not significant differences,et sequentia.
图5 CgPOU3F4L基因mRNA的组织和高温胁迫后血淋巴细胞中的时序表达模式
Fig.5 mRNA expression patterns of CgPOU3F4L in different tissues and in haemocytes after high-temperature stress
2.4 凋亡相关基因启动子区的POU转录结合位点
采用MATCH软件预测凋亡相关基因CgBcl-2、CgBax、CgCaspase3、CgCaspase9及CgIAP启动子区的POU转录结合位点。转录因子结合位点预测结果显示,在CgBcl-2和CgCaspase3的启动子区域中发现了POU转录结合位点(表3),其中,在CgBcl-2启动子区1 197~1 207区间处发现了正向结合位点ATGCACTAAT,在CgCaspase3的启动子区1 286~1 297区间位点处发现了反向结合位点ATTTGCTGCAT。
表3 转录因子结合位点预测
Tab.3 Prediction of TFBS
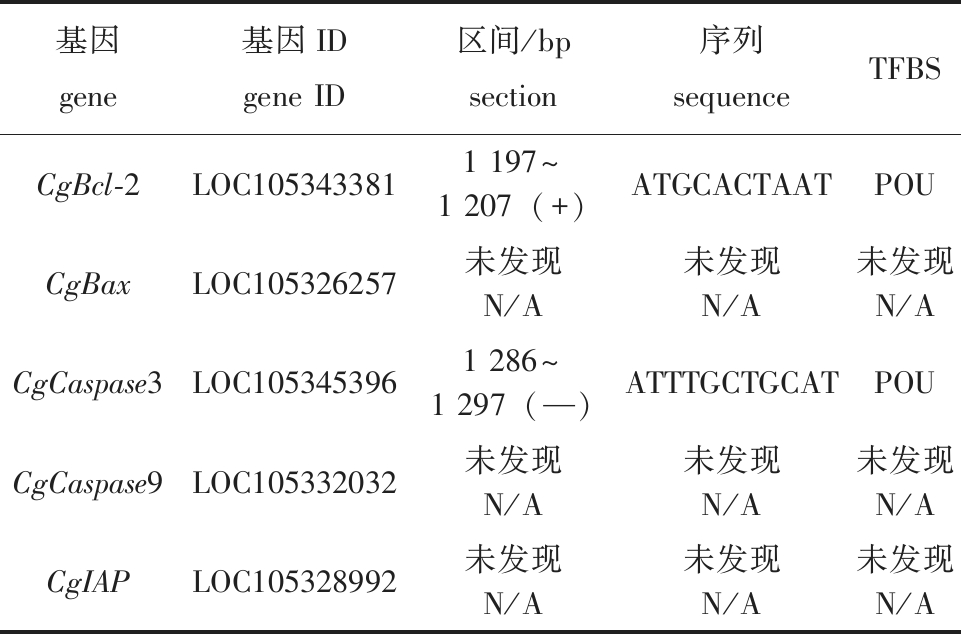
基因gene 基因IDgene ID区间/bpsection序列sequenceTFBSCgBcl-2LOC1053433811 197~1 207(+)ATGCACTAATPOUCgBaxLOC105326257未发现N/A未发现N/A未发现N/ACgCaspase3LOC1053453961 286~1 297(—)ATTTGCTGCATPOUCgCaspase9LOC105332032未发现N/A未发现N/A未发现N/ACgIAPLOC105328992未发现N/A未发现N/A未发现N/A
2.5 高温胁迫后CgPOU3F4L-RNAi组长牡蛎血淋巴细胞中凋亡相关基因mRNA表达水平
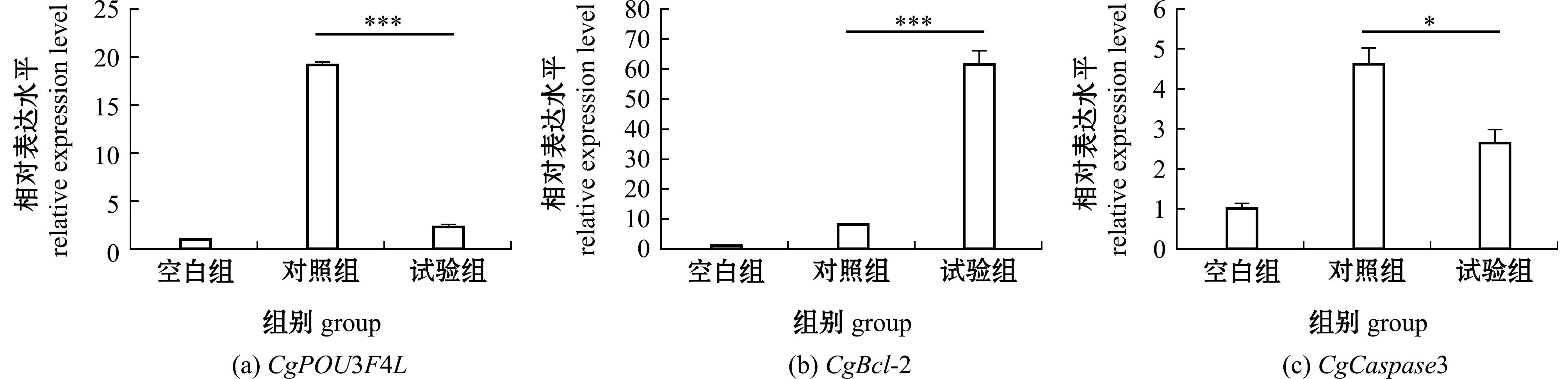
利用RT-qPCR技术对高温胁迫后CgPOU3F4L-RNAi组长牡蛎血淋巴细胞中的CgBcl-2及CgCaspase3的mRNA相对表达水平进行了检测。利用RNAi技术抑制CgPOU3F4L的表达后,在高温胁迫24 h后试验组与对照组长牡蛎CgPOU3F4L基因mRNA表达水平均上升,且试验组长牡蛎中CgPOU3F4L基因的mRNA表达水平极显著低于对照组,为对照组的0.12倍(P<0.001)(图6(a))。进一步检测了启动子区含有POU结合位点的凋亡相关基因(CgBcl-2和CgCaspase3)的mRNA表达水平。结果显示,在高温胁迫24 h后试验组与对照组长牡蛎凋亡相关基因mRNA表达水平均上升,且试验组长牡蛎中CgBcl-2基因的mRNA表达水平极显著高于对照组,为对照组的7.49倍(P<0.001)(图6(b)),同时CgCaspase3基因的mRNA表达水平显著低于对照组,为对照组的0.57倍(P<0.05)(图6(c))。
*表示组间有显著性差异(P<0.05);***表示组间有极显著性差异(P<0.001)。
*means significant difference between the groups (P<0.05);***means very significant differences between the groups (P<0.001).
图6 高温胁迫后CgPOU3F4L-RNAi组长牡蛎的血淋巴细胞中凋亡相关基因的mRNA相对表达水平
Fig.6 Relative mRNA expression levels of apoptosis-related genes in blood lymphocytes in CgPOU3F4L-RNAi of Pacific oyster after high-temperature stress
2.6 高温胁迫后CgPOU3F4L-RNAi组长牡蛎血淋巴细胞的凋亡率
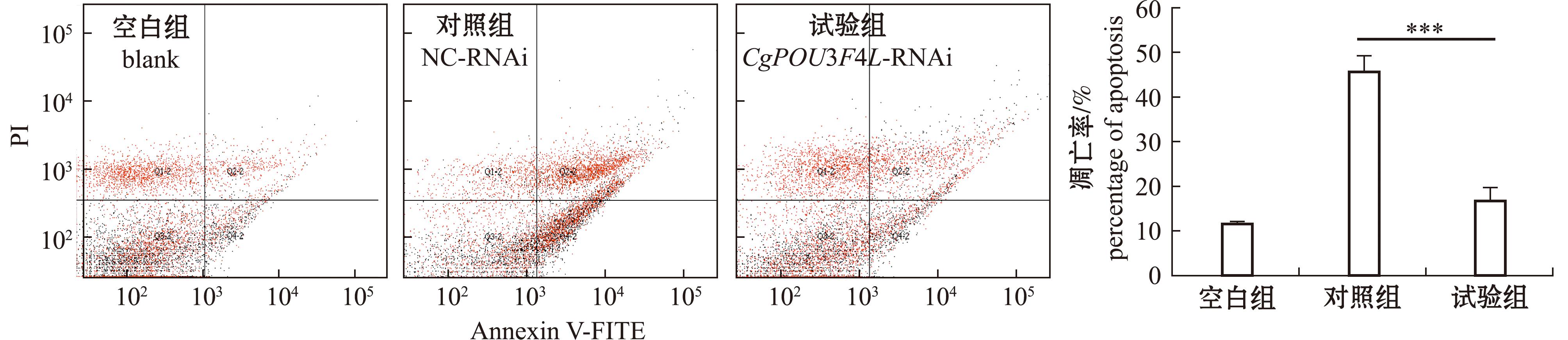
通过Annexin V-FITC细胞凋亡检测试剂盒,采用双染法检测高温胁迫24 h后CgPOU3F4L-RNAi长牡蛎的血淋巴细胞凋亡率(图7)。结果显示,在高温胁迫24 h后,试验组与对照组长牡蛎的血淋巴细胞凋亡率均出现了上升,其中,CgPOU3F4L-RNAi组的细胞凋亡率(16.53%)极显著低于对照组(45.90%,P<0.001)。
***表示组间有极显著性差异(P<0.001)。***means very significant differences between the groups (P<0.001).
图7 高温胁迫后CgPOU3F4L-RNAi长牡蛎的血淋巴细胞凋亡情况及凋亡率统计
Fig.7 Statistics of haemocyte apoptosis and apoptosis rate in CgPOU3F4L-RNAi of Pacific oyster exposed to high-temperature stress
3 讨论
3.1 POU家族基因鉴定及进化分析
POU家族基因具有POU和Homeobox结构域,是一类能够在生长发育、代谢和免疫反应等重要生命过程中发挥转录调控功能的重要转录因子,通过调控靶基因的转录过程参与生物对环境胁迫响应,并可能在细胞凋亡调节中发挥作用[1-2,7]。目前对长牡蛎POU家族基因的功能研究较少,仅在调控褐黑素合成及血红素-过氧化物酶基因参与的色素沉着中有相关研究[24]。本研究中通过长牡蛎基因组数据库,在长牡蛎中共鉴定出4个CgPOU家族基因,分别为CgPOU2F1、CgPOU3F4、CgPOU3F4L及CgPOU4F3,均含有一个POU结构域及一个Homeobox结构域。已有研究表明,POU结构域是POU家族基因进行同源或异源二聚化进而结合下游靶基因启动子区域的转录结合位点的关键结构域,Homeobox结构域在胚胎发育、基因转录调节及细胞分化等方面发挥重要作用,二者是POU家族基因的重要组成部分[6]。因此初步推测CgPOU2F1、CgPOU3F4、CgPOU3F4L及CgPOU4F3属于CgPOU家族,结构域的保守性说明了CgPOU家族基因的生物学功能与高等动物具有一定相似性。系统进化分析结果显示,CgPOU家族基因进化关系均较保守,本研究中从长牡蛎中同时鉴定到了2个POU3F4基因,分别为CgPOU3F4和CgPOU3F4L,这是较为罕见的,此外CgPOU3F4L基因与其他POU3F4基因相比具有一段特有的氨基酸序列,这可能是由基因扩张造成的基因积极进化,常形成更加多样化的环境适应能力,但是其所带来的功能分化特征还不清楚。本研究中POU3家族与POU5家族共同聚为一支,进化关系近,功能具有相似性。已有研究表明,POU3及POU5家族可以响应环境胁迫,如在中华卤虫中AsAPH-1(POU3家族)表达水平随着生存环境中盐度的增加而显著增加[25];在奶牛(Bos taurus)中,季节性和诱导性高温胁迫均可以降低转录因子的mRNA表达水平,包括生长分化因子(GDF9)、3-磷酸甘油醛脱氢酶(GAPDH)和POU5F1等[26]。故本研究中探究了特异性扩张基因CgPOU3F4L在长牡蛎环境胁迫应答过程中作用。
3.2 CgPOU3F4L参与长牡蛎高温胁迫的响应过程
本研究中长牡蛎CgPOU3F4L参与了高温胁迫的应答过程,并利用 RT-qPCR技术检测了CgPOU3F4L的 mRNA 在长牡蛎不同组织中的表达模式,结果显示,在肝胰腺中CgPOU3F4L基因的mRNA表达水平最低,在外套膜、闭壳肌和血淋巴细胞中处于较高表达水平。长牡蛎血淋巴细胞在响应环境胁迫中发挥重要作用[27],因此,本研究中进一步检测了高温胁迫下长牡蛎血淋巴细胞中CgPOU3F4L的时序表达模式。CgPOU3F4L的表达水平呈现出先增加后减少的趋势,与0 h相比,在高温胁迫的12、24、36 h时出现了显著升高。已有研究表明,长牡蛎在遭遇高温胁迫时,首先调控未折叠蛋白反应和抗凋亡系统以维持基本的生命活动。随着胁迫的持续,胁迫损伤逐渐变得不可修复,当胁迫时间延长至24 h时,细胞凋亡被激活[14]。在本研究中,CgPOU3F4L在血淋巴细胞中出现较高表达,同时在高温胁迫的12、24、36 h时表达水平升高,因此推测CgPOU3F4L可能参与长牡蛎高温胁迫响应过程,并在高温胁迫后12~36 h时发挥关键作用。
CgPOU3F4L可以通过调控凋亡相关基因的表达进而促进长牡蛎血淋巴细胞凋亡来响应高温胁迫。本研究结果显示,在高温胁迫24 h后的长牡蛎血淋巴细胞中,试验组CgBcl-2 mRNA的表达水平显著高于对照组,为对照组的7.49倍,CgCaspase3 mRNA的表达水平显著低于对照组,为对照组的0.57倍,同时采用流式细胞分析术发现,试验组的细胞凋亡率(16.53%)极显著低于对照组(45.90%)。已有研究表明,POU家族可通过调节凋亡相关基因的表达来参与凋亡过程,如在小鼠神经元细胞中 POU家族的成员Brn-3a,通过诱导Bcl-2基因的表达保护神经元细胞免于凋亡[7]。在成骨细胞中抑制POU2AF1表达可以促进成骨细胞凋亡[28]。与目前POU家族在脊椎动物中的研究不同,本研究中发现,高温胁迫下CgPOU3F4L-RNAi的长牡蛎中CgBcl-2的表达水平升高,同时CgCaspase3的表达水平降低且细胞凋亡率降低,这些结果表明,CgPOU3F4L可以抑制CgBcl-2且促进CgCaspase3的mRNA表达水平,并通过促进血淋巴细胞凋亡来参与调控长牡蛎高温胁迫应答。
3.3 CgPOU3F4基因可能在长牡蛎中发生扩张
长牡蛎通过基因扩张方式响应反复和极端的环境胁迫。在生物进化过程中,常通过基因扩张等方式构建更加复杂且稳定的调控网络来调节生命活动,以适应外界的环境变化[29]。基因家族扩张增加了基因调控网络的稳定性,对于家族内成员及功能分化具有重要意义[30]。本研究中在长牡蛎中鉴定到了POU家族的4个成员,同时仅在长牡蛎中能同时鉴定到CgPOU3F4及CgPOU3F4L,这可能是由于基因扩张导致的。研究表明,环境胁迫是基因扩张的一个重要触发因素,马齿苋(Portulacineae marked)的极端基因扩张满足了适应恶劣环境压力的要求[31]。硬壳蛤(Mercenaria mercenaria)通过 IAP家族的大规模扩张应对环境胁迫[32]。潮间带区域是长牡蛎的主要栖息地,存在各种剧烈的环境胁迫,如高温、干露及盐度变化等[33]。长牡蛎中基因扩张现象在HSP70、IAP和Bax等基因中发现,并已被证明与长牡蛎对环境胁迫应答机制的完善有关[12]。在本研究中CgPOU3F4L可以在长牡蛎高温胁迫应答过程中发挥重要的凋亡转录调控功能,因此猜测CgPOU家族基因的扩张与长牡蛎对环境胁迫应答机制的完善有关。
4 结论
1)本研究在长牡蛎基因组中鉴定了4个POU家族基因,其中仅在长牡蛎中同时存在CgPOU3F4和CgPOU3F4L,可能与环境胁迫响应和进化有关。
2)CgPOU3F4L的mRNA在外套膜、闭壳肌及血淋巴细胞中显著性高表达,并在28 ℃高温胁迫12、24、36 h后出现了显著高表达,表明该基因参与长牡蛎高温胁迫应答过程。
3)长牡蛎CgPOU3F4L可通过调控CgBcl-2和CgCaspase3的mRNA表达水平及血淋巴细胞凋亡率来参与高温胁迫应答。
[1] PHILLIPS K,LUISI B.The virtuoso of versatility:POU proteins that flex to fit[J].Journal of Molecular Biology,2000,302(5):1023-1039.
[2] HERR W,STURM R A,CLERC R G,et al.The POU domain:a large conserved region in the mammalian pit-1,oct-1,oct-2,and Caenorhabditis elegans unc-86 gene products[J].Genes &Development,1988,2(12A):1513-1516.
[3] REM NYI A,TOMILIN A,POHL E,et al.Differential dimer activities of the transcription factor oct-1 by DNA-induced interface swapping[J].Molecular Cell,2001,8(3):569-580.
NYI A,TOMILIN A,POHL E,et al.Differential dimer activities of the transcription factor oct-1 by DNA-induced interface swapping[J].Molecular Cell,2001,8(3):569-580.
[4] JIN W S,WANG L,ZHU F,et al.Critical POU domain residues confer Oct4 uniqueness in somatic cell reprogramming[J].Scientific Reports,2016,6:20818.
[5] MATHIS J M,SIMMONS D M,HE X,et al.Brain 4:a novel mammalian POU domain transcription factor exhibiting restricted brain-specific expression[J].The EMBO Journal,1992,11(7):2551-2561.
[6] BÜRGLIN T R,AFFOLTER M.Homeodomain proteins:an update[J].Chromosoma,2016,125(3):497-521.
[7] SMITH M D,DAWSON S J,BOXER L M,et al.The N-terminal domain unique to the long form of the Brn-3a transcription factor is essential to protect neuronal cells from apoptosis and for the activation of Bbcl-2 gene expression[J].Nucleic Acids Research,1998,26(18):4100-4107.
[8] KISLIOUK T,CRAMER T,MEIRI N.Methyl CpG level at distal part of heat-shock protein promoter HSP70 exhibits epigenetic memory for heat stress by modulating recruitment of POU2F1-associated nucleosome-remodeling deacetylase (NuRD) complex[J].Journal of Neurochemistry,2017,141(3):358-372.
[9] YANG J H,GUO Q,WANG L,et al.POU domain class 2 transcription factor 2 inhibits ferroptosis in cerebral ischemia reperfusion injury by activating Sestrin2[J].Neurochemical Research,2023,48(2):658-670.
[10] 刘冉阳,高磊,张兴志,等.广西北海大风江水域环境及养殖香港牡蛎健康状况分析[J].大连海洋大学学报,2024,39(4):551-558.
LIU R Y,GAO L,ZHANG X Z,et al.Analysis on the environment of Dafeng River in Beihai,Guangxi and the health status of cultivated Hong Kong oysters (Crassostrea hongkongensis) [J].Journal of Dalian Ocean University,2024,39(4):551-558.(in Chinese)
[11] XING Z,GAO L,LIU R Y,et al.The oxidative stress of the Pacific oyster Crassostrea gigas under high-temperature stress[J].Aquaculture,2023,577:739998.
[12] ZHANG G F,FANG X D,GUO X M,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[13] 高磊,孔宁,刘冉阳,等.夏季北黄海贝类养殖区环境因子、长牡蛎糖原和免疫指标变化的调查研究[J].水产学报,2024,48(4):167-178.
GAO L,KONG N,LIU R Y,et al.Investigation on the variation of environmental factors,glycogen and immune parameters of Pacific oyster (Crassostrea gigas) in the North Yellow Sea shellfish farming area in summer[J].Journal of Fisheries of China,2024,48(4):167-178.(in Chinese)
[14] YANG C Y,GAO Q,LIU C,et al.The transcriptional response of the Pacific oyster Crassostrea gigas against acute heat stress[J].Fish &Shellfish Immunology,2017,68:132-143.
[15] YANG Y H,QIAO X,YU S M,et al.A trace amine associated receptor mediates antimicrobial immune response in the oyster Crassostrea gigas[J].Developmental and Comparative Immunology,2024,156:105171.
[16] FU L M,NIU B F,ZHU Z W,et al.CD-HIT:accelerated for clustering the next-generation sequencing data[J].Bioinformatics,2012,28(23):3150-3152.
[17] LU S N,WANG J Y,CHITSAZ F,et al.CDD/SPARCLE:the conserved domain database in 2020[J].Nucleic Acids Research,2020,48(D1):D265-D268.
[18] MINH B Q,SCHMIDT H A,CHERNOMOR O,et al.IQ-TREE 2:new models and efficient methods for phylogenetic inference in the genomic era[J].Molecular Biology and Evolution,2020,37(5):1530-1534.
[19] AGGARWAL D,WARNE B,JAHUN A S,et al.Genomic epidemiology of SARS-CoV-2 in a UK university identifies dynamics of transmission[J].Nature Communications,2022,13(1):751.
[20] CARTHARIUS K,FRECH K,GROTE K,et al.Matinspector and beyond:promoter analysis based on transcription factor binding sites[J].Bioinformatics,2005,21(13):2933-2942.
[21] WANG L L,SONG X R,SONG L S.The oyster immunity[J].Developmental &Comparative Immunology,2018,80:99-118.
[22] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C(T)) Method[J].Methods,2001,25(4):402-408.
[23] LUO M,YANG Z Q,HUANG J C,et al.Genistein protects ovarian granulosa cells from oxidative stress via cAMP-PKA signaling[J].Cell Biology International,2020,44(2):433-445.
[24] MIN Y,LI Q,YU H.Heme-peroxidase 2 modulated by POU2F1 and SOX5 is involved in pigmentation in Pacific oyster (Crassostrea gigas)[J].Marine Biotechnology,2022,24(2):263-275.
[25] WANG J Q,HOU L,YI N,et al.Molecular analysis and its expression of a Pou homeobox protein gene during development and in response to salinity stress from brine shrimp,Artemia sinica[J].Comparative Biochemistry and Physiology Part A,Molecular &Integrative Physiology,2012,161(1):36-43.
[26] ROTH Z.Physiology and endocrinology symposium:cellular and molecular mechanisms of heat stress related to bovine ovarian function[J].Journal of Animal Science,2015,93(5):2034-2044.
[27] SONG L S,WANG L L,QIU L M,et al.Bivalve immunity[M]//Advances in Experimental Medicine and Biology.Boston,MA:Springer US,2010:44-65.
[28] WANG X P,ZOU C C,HOU C J,et al.POU class 2 homeobox associating factor 1,as a hub candidate gene in OP,relieves osteoblast apoptosis[J].Applied Biochemistry and Biotechnology,2024:1-25.
[29] HOLLAND P W.Evolution of homeobox genes[J].Wiley Interdisciplinary Reviews Developmental Biology,2013,2(1):31-45.
[30] HU Q D,TAN H R,IRWIN D M.Evolution of the vertebrate resistin gene family[J].PLoS One,2015,10(6):e0130188.
[31] WANG N,YANG Y,MOORE M J,et al.Evolution of portulacineae marked by gene tree conflict and gene family expansion associated with adaptation to harsh environments[J].Molecular Biology and Evolution,2019,36(1):112-126.
[32] SONG H,GUO X M,SUN L N,et al.The hard clam genome reveals massive expansion and diversification of inhibitors of apoptosis in Bivalvia[J].BMC Biology,2021,19(1):15.
[33] ZHANG G F,LI L,MENG J,et al.Molecular basis for adaptation of oysters to stressful marine intertidal environments[J].Annual Review of Animal Biosciences,2016,4:357-381.