随着全球气候变暖,许多浅海生物为了躲避高温,开始向深海区域迁徙,静水压力成为影响海洋生物垂直分布的主要环境因素。Brown等[1]研究表明,一些浅水生物能够在其自然分布范围外的深海区域生存。Oger等[2]和Somero等[3]研究发现,高静水压力会影响细胞内的升华反应平衡和反应速率,同时也会影响分子间距离和弱化学键。虽然高压会扰乱神经系统、心脏功能和膜运输,但有研究指出,某些蛋白质在高压环境下相对稳定,且完全变性需要超过百个大气压[3]。这意味着高压环境下仍有一些蛋白质能够保持稳定,从而维持生物体的基本生命活动。其他学者的研究也表明,在经历高静水压处理后,不同生物体内会启动多种相似的生物过程以应对高压应激,包括遗传信息处理、脂肪酸代谢、抗氧化和免疫功能[4-7]等。
虾夷扇贝(Mizuhopecten yessoensis)属于软体动物,广泛分布于俄罗斯远东、日本北海道和朝鲜半岛沿海海域。目前,中国的虾夷扇贝养殖主要采用浮筏养殖和底播养殖两种模式。然而,由于近年来种质退化和病原体侵袭等问题,浮筏养殖的产量并不稳定[8]。为了稳定并增加虾夷扇贝的产量,扩大其底播养殖面积被视为一种可行的方法。
近年来,转录组测序技术已被广泛应用于研究水生生物在特定环境条件下的分子机制。如Brown等[9]利用该技术对挪威石蟹(Lithodes maja)进行研究,发现在高静水压力胁迫下,蟹中与神经传递和代谢相关的基因表达发生变化,这表明高压可能影响石蟹的神经系统并增加其代谢成本。Liang等[10]的研究也显示,刺参(Apostichopus japonicus)在静水压胁迫下,其转录组分析揭示了多个压敏和热敏基因的表达变化,同时泛素化和内吞作用这两个过程也对高压产生响应。Hu等[11]的研究发现,在斑马鱼中,大脑对静水压力尤为敏感,且在适应10 MPa压力后,其转录水平和稳定性均有所降低。这些研究表明,转录组测序技术可以有效地揭示生物在高静水压力下的分子响应。特别是对虾夷扇贝而言,通过分析高压胁迫后的差异表达基因和功能,可以进一步理解其可能的分子途径,并可为研究其如何适应深海环境提供重要依据。
1 材料与方法
1.1 材料
本试验在大连海洋大学农业农村部北方海水增养殖重点实验室进行,试验用虾夷扇贝采自大连市长海县,全部采用浮筏养殖方式,养殖水层深度为5~10 m。试验设计包括3个对照组和3个试验组,每组各暂养10只虾夷扇贝,壳长为(75.64±2.15)mm。试验前,所有扇贝在实验室条件下暂养7 d,水温控制在10 ℃。试验期间每天定时投喂小球藻(Chlorella vulgaris),并每24 h更换一次新鲜海水以保证水质,确保试验个体适应实验室环境。试验开始前3 d停止投食,以减少因消化状态差异导致的潜在反应差异。
试验用设备为容积2 L的FH-100M手动静水压机(青岛海星仪器有限公司),试验开始前2 h,向压力舱中加满10 ℃的过滤海水,以确保海水温度稳定。根据前期预试验结果,虾夷扇贝在25 MPa的高压下存活状态良好,故将试验条件设定为25 MPa(相当于2 500 m水深)。使用液压泵以每分钟2 MPa的速度对试验组施加压力,直至达到25 MPa。为避免水中溶解氧不足对试验结果产生影响,在高压胁迫2 h后[12],迅速释放压力,取出3组虾夷扇贝。随后从每组中随机抽取3个扇贝,取其鳃、外套膜和闭壳肌作为生物学重复样本,并立即在液氮中冷冻。最后,将样品存放于-80 ℃超低温冰箱中,以便后续进行RNA提取。
1.2 方法
1.2.1 RNA分离和文库制备 提取样品总RNA并使用DNase消化DNA后,使用带有Ologo(dT)磁珠富集真核生物mRNA;加入打断试剂将mRNA打断成短片段,以打断后的mRNA为模板,用六碱基随机引物合成一链cDNA,然后配制二链合成反应体系合成双链cDNA,并使用试剂盒纯化双链cDNA;纯化的双链cDNA再进行末端修复、加A尾并连接测序接头,然后进行片段大小选择,最后进行PCR扩增;构建好的文库用Agilent 2100 Bioanalyzer质检合格后,采用Illumina HiSeq X Ten进行测序,产生125 bp的双端数据。
1.2.2 差异表达分析 高通量测序中产生的原始数据为fastq格式序列,为了得到高质量的数据还需进行进一步过滤,采用Trimmomatic软件进行指控并去除接头,过滤掉低质量碱基及N碱基。采用Hisat2将干净的读数与虾夷扇贝参考基因组进行比对。每个差异基因的差异表达量均使用FPKM计算,并通过HTSeqcount获得落到每个样本中基因的reads数目,采用DEGSeq(2012)R软件包对数据进行标准化和差异表达分析。P<0.05和|foldchange|>1.5被设置为显著差异表达。通过差异表达基因(DEGs)的层次聚类分析以证明不同组合样品中基因的表达模式。然后采用Coatools和KOBAS 3.0软件将差异表达基因与GO数据库和KEGG数据库进行分析比对,获得GO功能注释和KEGG富集通路。
1.2.3 RT-qPCR验证 在显著差异基因中随机选取11个基因,包括8个上调基因和3个下调基因进行qRT-PCR验证。以β-actin作为内参基因,由武汉金凯瑞生物工程有限公司合成引物,基因名称和引物序列见表1,按2-ΔΔCt计算基因的相对表达量,与测序结果进行比对。
表1 实时荧光定量引物
Tab.1 Primers used for real-time PCR analysis

基因gene引物 primer正向 forward(5′-3′)反向 reverse(3′-5′)F11CTACAACCACCACAACCACCACTACTTCTTCGTTCACAAAGGGCAGGTCbirc7-aGCTAGTATTGTGTTCCTGCCTTGTGGCCTTGACCGTGCCTCTGATGHSP70B2CACGAGAGCCAGATTTGAGGACTTGATCCGCCAACCAGCACGATTTCNAFICCTGAGCCAGTCTGCCATTATACCGTTTGCTGCTTGTCCCAACTTGTGCyp26bCCCGATGACGAAAGGCTATTGGACGTGAGGTGGTAGATGATGGCGAACInx-19TTCTCCTTCCGACAACGACATTGCGCCTCGCCGATCTCCTTCAATATACDnajb4CACACAGTTTGCGAGGGAAGGTAGGGGCTGTTTGGGAAGTGGAAGTCSCLYAGCGTCTCCCGAACACTTGTAATGTGCGTTCCTTGCCACAGATTCAGSlco2a1GGTTAAACGGGCAACATACGGAATCGCCAGACCAAGTAGCACCCATTGRNASEHGCAGCCTGTCGAGCAATAGAGCAGCGTTGATGAGGAACTGACTGTCbirc7-bGCTCATCTCCTCCAGTTGACACAGGCAACAGGATTTAACACGCCCATCβ-actinCAAACAGCAGCCTCCTCGTCATCTGGGCACCTGAACCTTTCGTT
2 结果分析
2.1 测序结果
测得常压和25 MPa静水压下,虾夷扇贝不同组织的转录组数据总量为133.92 G,经过过滤后有效数据为125.57 G,有效碱基比为93.46%~97.74%,GC含量为41.63%~45.17%,表明测序获得的数据质量符合要求。
2.2 差异基因分析
采用DEGeq2 R软件包(v1.22.2)对AM、M、G 3组不同组织的差异基因新型进行表达分析,以P<0.05,|log2FC|>1作为筛选差异基因的选择标准,在闭壳肌组(AM)组中获得280个差异基因,其中,上调基因130个,下调基因150个;在外套膜(M)组中获得529个差异基因,其中,上调基因有250个,下调基因有279个;在鳃(G)组中共获得704个差异基因,其中,上调基因314个,下调基因390个,将两两对比后进行韦恩分析,共有51个基因同时在3组中显著表达(图1)。
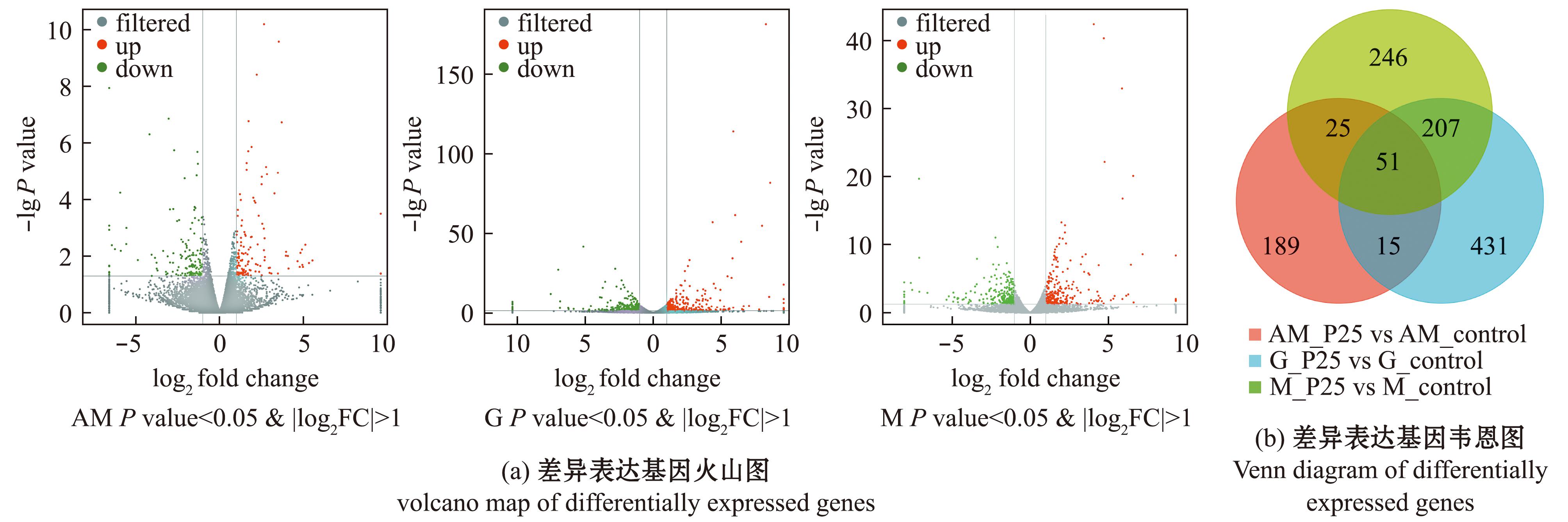
图1 闭壳肌、外套膜、鳃的差异表达基因
Fig.1 Differentially expressed genes in the adductor muscle,mantle,and gills
2.3 差异基因的功能注释
在GO分析中,3组上调基因分别被注释的数量为AM(44)、G(49)、M(48),下调的差异基因分别被注释的术语数量为AM(44)、G(47)、M(47)。DEGs的功能在GO富集注释,基因本体论富集分为三个部分——生物过程、细胞组分和分子功能,在3组的差异富集结果中,分别对这三部分中的前十条注释进行分析。如图2所示,3组共同拥有的术语为生物过程中的半胱氨酸型内肽酶活性在细胞凋亡过程中的抑制作用,分子功能中的半胱氨酸型内肽酶抑制剂活性参与细胞凋亡过程、伴侣结合和泛素蛋白连接酶活性。

图2 差异表达基因的GO富集分析
Fig.2 GO enrichment analysis of differentially expressed genes
2.4 差异表达基因在KEGG中的富集分析
对KEGG前20个通路进行富集分析,以差异基因数量、P值和环境得分作为筛选标准,差异基因越多,环境得分越高,P值越小,通路越显著。从图3可见,共同富集通路包括抗原呈递、弓形虫病、内质网中的蛋白质加工、雌激素信号通路、MAPK信号通路、NF-kB信号通路、TNF信号通路等,主要涉及生物对环境信息处理、细胞增殖、免疫和细胞凋亡等过程。
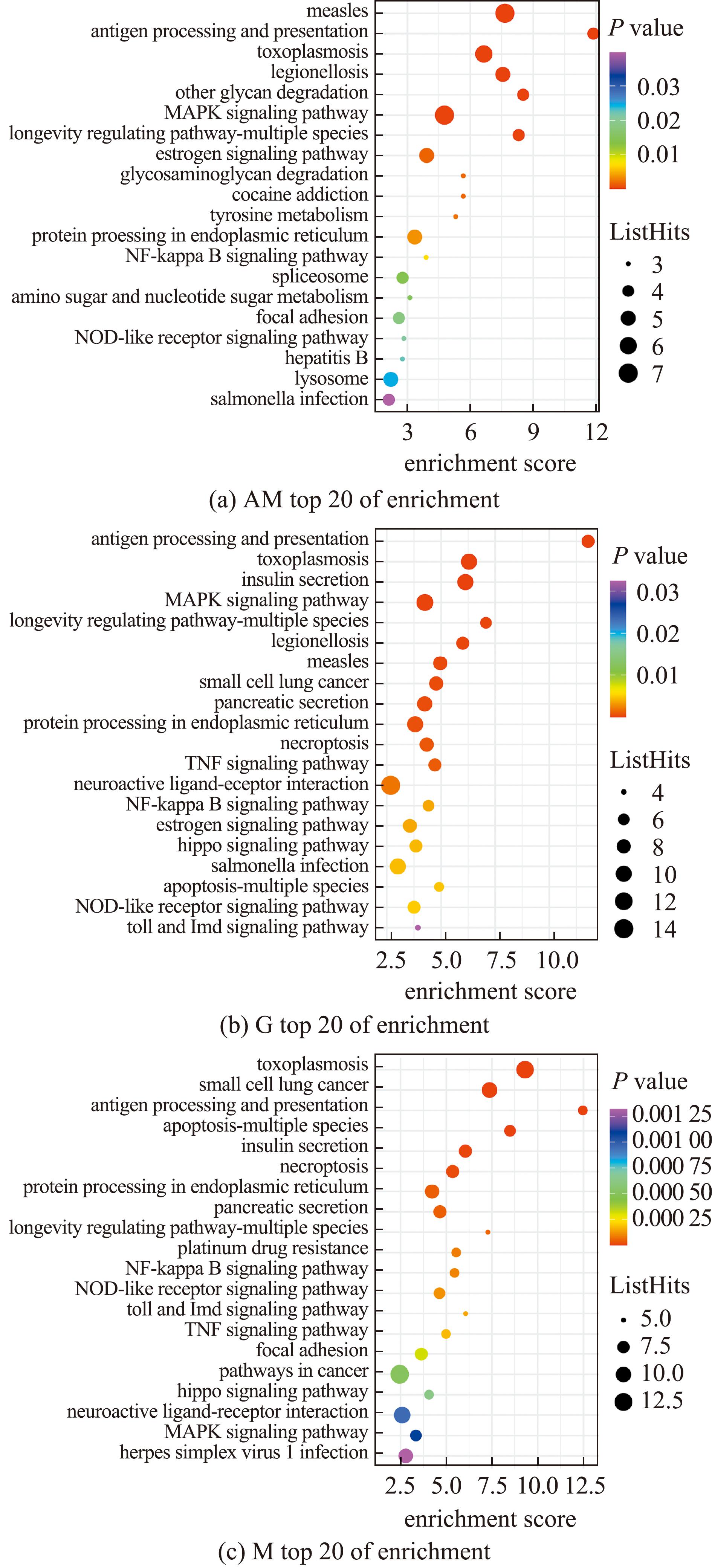
图3 差异表达基因KEGG富集通路气泡图(前20)
Fig.3 Bubble diagram of KEGG enrichment pathway for differentially expressed genes (top 20)
2.5 qRT-PCR验证
通过qRT-PCR技术从闭壳肌、鳃和外套膜的共同表达的差异基因中选出11个基因进行验证,比较结果如图4所示,荧光定量分析的结果与转录组测序的结果基本一致,符合分析结果。
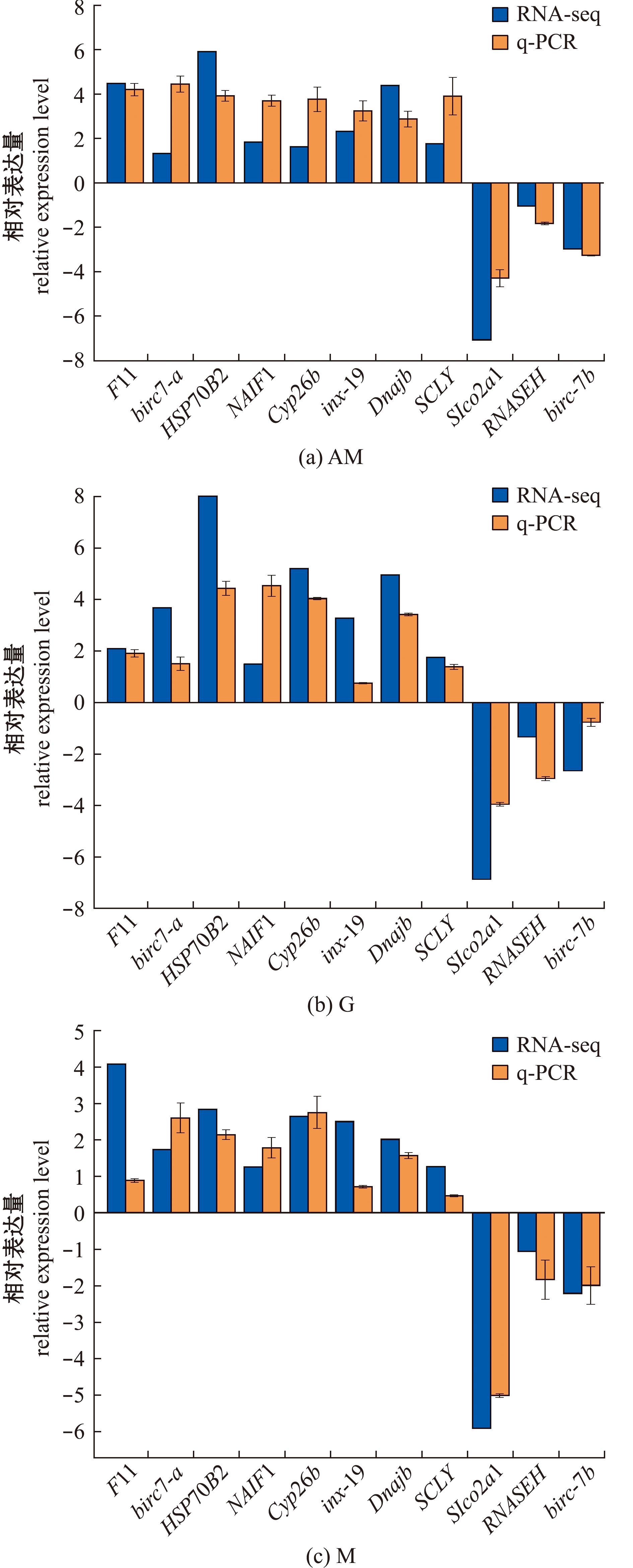
图4 3组差异表达基因的RT-qPCR验证结果分析
Fig.4 Analysis of RT-qPCR verification results of three groups of differentially expressed genes
3 讨论
在1 000~3 000 m深度范围内,生物群体的活动呈现出明显的深度分区。研究表明,一些浅水生物能够在这个区间定居。尽管在深海的极端压力环境中,生物的多样性和生物量逐渐减少[13],但深海海沟中仍然存在多种动物,主要由底栖生物组成,如鱼类、双壳类和腹足类等[14]。通过研究虾夷扇贝对高静水压力的转录组响应,可以更好地理解这些生物对深海环境的适应性,有助于扩大养殖范围。
3.1 高静水压力对虾夷扇贝不同组织之间的影响
在对虾夷扇贝不同组织进行GO和KEGG富集分析后,发现闭壳肌在应激反应中特别富集了与能量代谢相关的通路。具体来说,在GO分析中,质膜和细胞质中差异基因的富集最为显著。质膜对细胞信号传导、跨膜运输、细胞结构和黏附等功能至关重要[15],而细胞质则参与了代谢、信号传导和物质运输等过程[16]。在KEGG分析中,闭壳肌中的其他聚糖降解、糖胺聚糖降解和酪氨酸代谢通路也显著富集,特别是β-己糖胺酶亚基和甘露糖苷酶的表达显著,后者能够促进糖原合成,为生物体提供额外的能量。此外,研究显示,在静水压力胁迫下,生物体内与能量代谢相关的生物过程得到了显著增强,深海生物的信号传导过程也比浅海生物更为复杂和发达[17-19]。在鳃组织中,与基因调控相关的过程特别富集,如DNA结合转录因子活性、序列特异性DNA结合、启动子特异性染色质结合及DNA去甲基化的负调控。FOXl2、Mafk和Runt等转录因子的表达显著,其中FOXl2是叉头盒家族的一员,能够结合DNA并调控基因表达,与多种信号通路相关[20]。通过对斑马鱼的研究表明,更高水平的转录组稳定性有助于深海鱼类适应深海环境[11]。这些发现表明,虾夷扇贝的不同组织对高静水压力的生理反应各不相同。闭壳肌作为开闭贝壳的主要器官,在高压环境下需要更多的能量来维持扇贝的正常运作;而鳃组织则对高压更为敏感,其基因表达和转录过程相较其他组织能更快地响应。
3.2 静水压胁迫对虾夷扇贝内质网的影响
静水压力对生物系统的影响主要表现为“压力增大,体积变小”,这种压力可以改变DNA的氢键,导致DNA损伤,影响其形成的复合物[21]。此外,高压还会破坏蛋白质的稳定性,导致蛋白质变性和错误折叠,从而产生对细胞有害的物质[22],为了抵消这些负面影响,生物体会提高遗传信息处理和蛋白质加工相关基因的表达水平。在应激反应后,HSP蛋白家族的表达水平普遍升高,这在本次试验中也得到了证实。特别是在内质网的蛋白质加工中,HSP68、HSP70B2、HSP90和BAG2等基因的表达显著。HSP蛋白能够与抗原蛋白结合,促进内吞作用中囊泡的运输,以消除错误折叠的蛋白质[23],在中华原钩虾[18]和马里亚纳狮子鱼[5]研究中,热休克蛋白家族被发现可以促进多种蛋白在高压环境下正确折叠和激活,从而保证信号传导和细胞存活等重要的细胞过程。BAG2蛋白通过与HSP70的ATPase结构高亲和力相互作用,加速ATP酶循环,调控折叠、聚集和降解反应,修复错误蛋白[24-25]。在DNA复制和修复通路中,一些通用转录因子如HES和IIH亚基也表现出差异表达。转录组分析显示,泛素化过程显著增强,与内质网蛋白质协同作用,共同消除由高静水压力引起的错误蛋白。
3.3 静水压胁迫对虾夷扇贝免疫系统的影响
研究表明,深海生物由于感知到高压环境带来的新功能和特殊信号,其信号传导基因的表达相较于浅水生物更为显著[26]。在KEGG分析中,研究发现抗原呈递、MAPK信号通路和TNF信号通路等显著上调。在抗原呈递过程中,热休克蛋白与吞噬细胞的抗原结合,调控如Toll样受体、NOD样受体和TNF信号通路等下游信号通路。MNK1在信号传导中通过elF4E磷酸化调控细胞周期蛋白mRNA的释放[27],并控制特定mRNA(如TNF)的翻译[28],从而影响TNF信号通路的表达。MNK1的上调降低了生物对细胞凋亡的敏感性,其作用位于半胱天冬酶调控的细胞凋亡过程下游[29]。高静水压引起的错误蛋白折叠激发了虾夷扇贝体内的多种信号因子,诱导免疫反应[30]。在本试验中,免疫系统有13个上调基因,包括丝氨酸蛋白酶,以及birc-7a、IAP,HSP70、HSP68和TNF受体相关因子等,这些基因参与了多种不同的信号通路。IAP能够抑制炎症反应,减少炎症介质的释放[31]。作为较低等的软体动物,细胞凋亡是其主要的免疫方式之一,有助于维持组织稳态[32]。细胞凋亡可由外源性或死亡受体途径和内源性或线粒体途径激活,不同信号通路间的相互作用可以放大细胞凋亡信号,半胱天冬酶作为细胞凋亡过程的启动子被直接激活。NF受体蛋白和NF-kB转录因子作为外源性信号因子,参与软体动物的凋亡通路,进一步调控溶酶体的释放[7]。软体动物细胞凋亡是一个动态过程[33],凋亡抑制蛋白IAP在半胱天冬酶启动细胞凋亡过程后,通过抑制半胱天冬酶活性和控制TNF受体相关因子调节NF-kB通路,发挥阻断凋亡的作用,维持虾夷扇贝体内的动态平衡。
4 结论
1)通过分析虾夷扇贝不同组织在高压胁迫后的转录组变化情况,发现闭壳肌中能量代谢相关通路显著,鳃中与遗传信息处理相关通路显著,说明虾夷扇贝不同组织对高压胁迫的响应模式不同。
2)高静水压力的影响下,虾夷扇贝体内损伤细胞数量增加,细胞稳态遭到破坏。以热休克蛋白为核心的基因群通过抗原呈递等信号通路激活细胞凋亡,促进内质网对错误蛋白质的吞噬和新蛋白质的合成。说明高压环境对虾夷扇贝生长、免疫具有显著影响。
[1] BROWN A,THATJE S.Explaining bathymetric diversity patterns in marine benthic invertebrates and demersal fishes:physiological contributions to adaptation of life at depth[J].Biological Reviews of the Cambridge Philosophical Society,2014,89(2):406-426.
[2] OGER P M,JEBBAR M.The many ways of coping with pressure[J].Research in Microbiology,2010,161(10):799-809.
[3] SOMERO G N.Adaptations to high hydrostatic pressure[J].Annual Review of Physiology,1992,54:557-577.
[4] LAN Y,SUN J,TIAN R M,et al.Molecular adaptation in the world’s deepest-living animal:Insights from transcriptome sequencing of the hadal amphipod Hirondellea gigas[J].Molecular Ecology,2017,26(14):3732-3743.
[5] WANG K,SHEN Y J,YANG Y Z,et al.Morphology and genome of a snailfish from the Mariana Trench provide insights into deep-sea adaptation[J].Nature Ecology &Evolution,2019,3(5):823-833.
[6] XIE Z,JIAN H H,JIN Z,et al.Enhancing the adaptability of the deep-sea bacterium Shewanella piezotolerans WP3 to high pressure and low temperature by experimental evolution under H2O2 stress[J].Applied and Environmental Microbiology,2018,84(5):e02342-e02317.
[7] SUN J,ZHANG Y,XU T,et al.Adaptation to deep-sea chemosynthetic environments as revealed by mussel genomes[J].Nature Ecology &Evolution,2017,1(5):121.
[8] 张明明,赵文.我国虾夷扇贝死亡原因的探讨及控制对策[J].中国水产,2008(2):65-66,74.ZHANG M M,ZHAO W.Discussion on the causes of death of scallop Patinopecten yessoensis in China and its control countermeasures[J].China Fisheries,2008(2):65-66,74.(in Chinese)
[9] BROWN A,THATJE S,MORRIS J P,et al.Metabolic costs imposed by hydrostatic pressure constrain bathymetric range in the lithodid crab Lithodes maja[J].The Journal of Experimental Biology,2017,220(Pt 21):3916-3926.
[10] LIANG L Y,CHEN J W,LI Y N,et al.Insights into high-pressure acclimation:comparative transcriptome analysis of sea cucumber Apostichopus japonicus at different hydrostatic pressure exposures[J].BMC Genomics,2020,21(1):68.
[11] HU M L,WU B S,XU W J,et al.Gene expression responses in zebrafish to short-term high-hydrostatic pressure[J].Zoological Research,2022,43(2):188-191.
[12] 陈家炜,张海滨.压力及温度对海洋动物生理影响的研究进展[J].海洋通报,2017,36(6):601-610.CHEN J W,ZHANG H B.A research progress of physiological effects of marine animals by hydrostatic pressure and temperature[J].Marine Science Bulletin,2017,36(6):601-610.(in Chinese)
[13] REX M A,ETTER R J,MORRIS J S,et al.Global bathymetric patterns of standing stock and body size in the deep-sea benthos[J].Marine Ecology Progress Series,2006,317:1-8.
[14] JAMIESON A J,FUJII T,MAYOR D J,et al.Hadaltrenches:the ecology of the deepest places on earth[J].Trends in Ecology &Evolution,2010,25(3):190-197.
[15] LAUDE A J,PRIOR I A.Plasma membrane microdomains:organization,function and trafficking[J].Molecular Membrane Biology,2004,21(3):193-205.
[16] LUBY-PHELPS K.The physical chemistry of cytoplasm and its influence on cell function:an update[J].Molecular Biology of the Cell,2013,24(17):2593-2596.
[17] ZHANG W J,ZHANG C,ZHOU S Y,et al.Comparative genomic analysis of obligately piezophilic Moritellaya yanosii DB21MT-5 reveals bacterial adaptation to the challenger deep,Mariana Trench[J].Microbial Genomics,2021,7(7):000591.
[18] CHEN J W,LIUH L,CAI S Y,et al.Comparative transcriptome analysis of Eogammarus possjeticus at different hydrostatic pressure and temperature exposures[J].Scientific Reports,2019,9(1):3456.
[19] BROWN A,THATJE S.Respiratory response of the deep-sea amphipod Stephonyx biscayensis indicates bathymetric range limitation by temperature and hydrostatic pressure[J].PLoS One,2011,6(12):e28562.
[20] FU M J,CHEN H,CAI Z P,et al.Forkhead box family transcription factors as versatile regulators for cellular reprogramming to pluripotency[J].Cell Regeneration,2021,10(1):17.
[21] AERTSEN A,VAN HOUDT R,VANOIRBEEK K,et al.An SOS response induced by high pressure in Escherichia coli[J].Journal of Bacteriology,2004,186(18):6133-6141.
[22] BALNY C,MASSON P,HEREMANS K.High pressure effects on biological macromolecules:from structural changes to alteration of cellular processes[J].Biochimica et Biophysica Acta,2002,1595(1/2):3-10.
[23] MORRIS J P,THATJE S,HAUTON C.The use of stress-70 proteins in physiology:a re-appraisal[J].Molecular Ecology,2013,22(6):1494-1502.
[24] KABBAGE M,DICKMAN M B.The BAG proteins:a ubiquitous family of chaperone regulators[J].Cellular and Molecular Life Sciences:CMLS,2008,65(9):1390-1402.
[25] XU Z,PAGE R C,GOMES M M,et al.Structural basis of nucleotide exchange and client binding by the Hsp70 cochaperone Bag 2[J].Nature Structural &Molecular Biology,2008,15(12):1309-1317.
[26] ASHBY M K.Survey of the number of two-component response regulator genes in the complete and annotated genome sequences of prokaryotes[J].FEMS Microbiology Letters,2004,231(2):277-281.
[27] TOPISIROVIC I,RUIZ-GUTIERREZ M,BORDEN K L B.Phosphorylation of the eukaryotic translation initiation factor eIF4E contributes to its transformation and mRNA transport activities[J].Cancer Research,2004,64(23):8639-8642.
[28] BUXAD M,PARRA J L,ROUSSEAU S,et al.Themnks are novel components in the control of TNFα biosynthesis and phosphorylate and regulate hnRNP A1[J].Immunity,2005,23(2):177-189.
M,PARRA J L,ROUSSEAU S,et al.Themnks are novel components in the control of TNFα biosynthesis and phosphorylate and regulate hnRNP A1[J].Immunity,2005,23(2):177-189.
[29] CHRESTENSEN C A,ESCHENROEDER A,ROSS W G,et al.Loss of MNK function sensitizes fibroblasts to serum-withdrawal induced apoptosis[J].Genes to Cells:Devoted to Molecular &Cellular Mechanisms,2007,12(10):1133-1140.
[30] KOVAC A,ZILKA N,KAZMEROVA Z,et al.Misfolded truncated protein τ induces innate immune response via MAPK pathway[J].Journal of Immunology,2011,187(5):2732-2739.
[31] PARK S,KIM D,LEE W,et al.Discovery of pan-IAP degraders via a CRBN recruiting mechanism[J].European Journal of Medicinal Chemistry,2023,245:114910.
[32] GERDOL M,GOMEZ-CHIARRI M,CASTILLO M G,et al.Immunity in molluscs:recognition and effector mechanisms,with a focus on Bivalvia[M]//COOPERE.Advances in Comparative Immunology.Cham:Springer,2018:225-341.
[33] ROMERO A,NOVOA B,FIGUERAS A.The complexity of apoptotic cell death in mollusks:an update[J].Fish &Shellfish Immunology,2015,46(1):79-87.