三疣梭子蟹(Portunus trituberculatus)隶属于甲壳纲,十足目[1],是中国重要的海水养殖蟹类,年养殖产量高达10万t。盐度是影响三疣梭子蟹人工养殖的重要环境因素之一[2],低盐胁迫下三疣梭子蟹鳃丝增厚,鳃腔扩大,上皮层发生解体,死亡率增加[3]。此外,低盐胁迫还导致梭子蟹体内活性氧(ROS)的积累[4],进而促进梭子蟹体内醛类产生[5],其中,丙二醛(MDA)是脂质氧化后最常见的醛类。过量的醛会与蛋白质和核酸发生反应,破坏其功能,从而导致细胞死亡[6]。
醛脱氢酶属于NAD(P)+依赖性酶家族[7],具有底物特异性,催化各种醛氧化成相应的羧酸,从而减少脂质的过氧化[8]。Aldh基因被认为是消除过剩醛的“醛清除剂”[9-10]。其参与对生物和非生物环境的胁迫适应,并在胁迫条件下调节醛稳态[11]。已有研究发现,Aldh基因可能与三疣梭子蟹低盐适应有一定联系[12],但其具体的功能及调节机制尚不清楚。
DNA甲基化是最常见的表观遗传修饰方式,对细胞分化[13]、组织特异性基因表达[14]、基因组印记[15]、X染色体失活[16]等都具有重要作用。研究发现,盐度胁迫能够引起水产动物的甲基化水平发生变化,如半滑舌鳎的pacap[17]、igf1[18]、igf2基因[19]及罗非鱼渗透压调节相关基因prkaca[20]的DNA甲基化水平在盐度胁迫中发生了显著改变。已有研究表明,低盐养殖环境下,三疣梭子蟹鳃组织中的整体甲基化水平降低与盐度胁迫相关[21]。然而,低盐环境下DNA甲基化如何调控基因表达从而介导组织损伤的机制并不明确。
本研究中,对三疣梭子蟹Aldh基因结构及系统进化进行了探究,并分析了该基因启动子区域的DNA甲基化水平、表达水平及MDA含量间的相关性,以期为揭示三疣梭子蟹在低盐环境中的损伤和适应过程中的表观遗传机制提供参考。
1 材料与方法
1.1 材料
本研究所用三疣梭子蟹采集于昌邑市海丰水产养殖有限责任公司。2022年8月选取100只健康有活力的梭子蟹(体质量为35.5 g±2.8 g),在室内暂养一周。暂养期间,盐度为30 mg/L,温度为(26±2)℃,pH为8.7,定时投喂新鲜饵料,每天换水一次。
1.2 方法
1.2.1 试验设计 暂养一周后,随机选取3只梭子蟹,取其鳃、肝胰腺、肌肉、眼柄、脑、肠、胃和心脏共8个组织,暂存于液氮中,用于组织表达分析。随后在体积为10 m3混凝土池中进行低盐胁迫试验,将蟹分为3组,每组30只,将每组盐度骤降至11 mg/L(预试验得到的72 h半致死浓度),通过在海水中添加淡水来调配到半致死盐度[22]。分别取0 h和低盐胁迫6、12、24、48、72 h的三疣梭子蟹鳃组织,每个时间点取3只个体混为一管,并储存在液氮中,用于低盐胁迫不同时间的甲基化分析和基因表达分析。
1.2.2 生物信息学分析 通过GSDS(http://gsds.gao-lab.org/)绘制Aldh基因结构图,采用Expasy ProtParam tool在线应用分析软件(https://web.expasy.org/protparam/)对Aldh的氨基酸序列进行分析,通过NCBI数据库对其序列进行Blast,采用SignalP-4.1在线应用软件(https://services.healthtech.dtu.dk/services/SignalP-4.1/)对Aldh信号肽进行分析,利用其保守序列分别用DNAMAN 6.0和MEGA 11.0软件进行同源性和构建系统发育进化树分析。
1.2.3 DNA和RNA提取 使用海洋动物DNA提取试剂盒(全式金,中国)从低盐胁迫不同时间的鳃组织中提取全基因组DNA,使用TransZol Up Plus RNA Kit(全式金,中国)提取总RNA。随后,通过10 g/L琼脂糖凝胶电泳和紫外分光光度计检测DNA和RNA的完整性。
1.2.4 三疣梭子蟹Aldh基因的时空表达分析 采用PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒(TaKaRa,日本)将RNA反转录为cDNA。采用Primer Primer 5.0设计qPCR引物 (表1),通过qPCR技术对Aldh基因在三疣梭子蟹不同组织和低盐胁迫不同时间进行表达分析,反应体系为10 μL,包括cDNA模板1 μL、ROX 0.2 μL、TB Green Premix Ex Taq Ⅱ 5μL、上下游引物各0.4 μL、ddH2O 3 μL。反应程序为95 ℃下预变性30 s,95 ℃下变性5 s,60 ℃下退火34 s,共进行40个循环;95 ℃下延伸15 s,60 ℃下退火1 min,95 ℃下复性15 s。使用β-actin作为内参基因,每个样品设置3个生物学重复。
表1 本研究所用引物
Tab.1 Primer used in this research
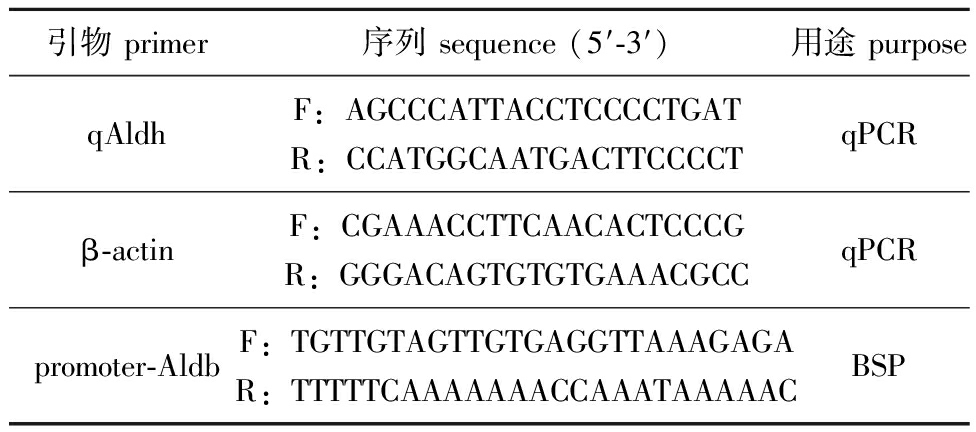
引物 primer序列 sequence (5′-3′)用途 purposeqAldhF:AGCCCATTACCTCCCCTGATR:CCATGGCAATGACTTCCCCTqPCRβ-actinF:CGAAACCTTCAACACTCCCGR:GGGACAGTGTGTGAAACGCCqPCRpromoter-AldbF:TGTTGTAGTTGTGAGGTTAAAGAGAR:TTTTTCAAAAAAACCAAATAAAAACBSP
1.2.5 Aldh基因在三疣梭子蟹低盐胁迫不同时间的甲基化分析 利用NCBI数据库获取三疣梭子蟹Aldh基因启动子序列,利用PROMO在线软件(https://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3)预测启动子区域的转录因子结合区域,使用Methprimer(http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi)设计启动子区域的甲基化引物 (表1)。将DNA亚硫酸氢盐转化试剂盒(Vazyme,中国)修饰后的DNA使用2 × EpiArt HS Taq Master Mix(Vazyme,中国)进行PCR扩增,PCR扩增体系(40 μL):模板4 μL,2×混合液20 μL,上下游引物各1.6 μL,ddH2O 12.8 μL。PCR扩增条件:95 ℃下预变性5 min,95 ℃下变性30 s,55 ℃下退火30 s,72 ℃下延伸30 s,共进行45个循环;72 ℃下复性10 min;4 ℃下保存。PCR产物经琼脂糖检测合格后,切胶回收纯化,经pMD-18T连接2 h后转化至大肠杆菌DH5α感受态细胞中,37 ℃下过夜培养,随机挑选10个单克隆送至上海生工生物工程有限公司测序。利用QUMA(http://quma.cdb.riken.jp/)在线软件进行甲基化水平计算和可视化分析。
1.2.6 MDA含量测定 脂质过氧化是通过硫代巴比妥酸法测定MDA含量评定的[23]。将低盐胁迫下三疣梭子蟹鳃组织样品在0.1 mol/L的磷酸盐缓冲溶液(phosphate buffer saline,PBS)溶液中匀浆,然后使用MDA检测试剂盒(南京建成生物科技有限公司)测定MDA含量。MDA含量测定参照试剂盒说明书。
1.3 数据处理
CG甲基化水平=甲基化CG位点/总CG位点。采用2-ΔΔCt方法计算基因的相对表达水平,并采用SPSS单因素方差分析其表达差异。采用Pearson相关系数分析DNA甲基化水平与表达水平之间的相关性,显著性水平设为0.05。
2 结果与分析
2.1 三疣梭子蟹Aldh基因生物信息学分析
对三疣梭子蟹Aldh基因的基因结构进行分析,观察到Aldh基因由10个CDS和10个内含子组成(图1(a))。采用Expasy ProtParam tool在线软件对Aldh的氨基酸序列进行分析,预测的分子式为C2 558H3 984N700O760S19,相对分子质量为57 313.15,等电点(PI)为6.12,不稳定指数为30.79,GRAVY为-0.227,结果表明,三疣梭子蟹Aldh基因是稳定且亲水的蛋白质。利用NCBI保守结构域预测发现,Aldh基因存在一个ALDH_F1AB_F2_RALDH1结构域,属于NAD结合位点结构域。

图1 三疣梭子蟹Aldh基因结构与其他物种的氨基酸序列比对
Fig.1 Amino acid sequence comparison of the Aldh gene structure of Portunustrituberculatus with that of other species
使用SignalP-4.1在线软件对Aldh信号肽分析,发现其不存在信号肽,推测三疣梭子蟹Aldh蛋白可能为胞内蛋白。同源性分析表明,三疣梭子蟹Aldh氨基酸保守结构域与中华绒螯蟹(Eriocheir sinensis)的氨基酸保守结构域一致性最高(88.13%),与其他物种的相似性分别为凡纳滨对虾(Litopenaeus Vannamei)86.04%、斑节对虾(Penaeus monodon)85.42%、大海鲢(Megalops cyprinoides)71.46%、黑腹果蝇(Drosophila melanogaster)71.46%、虎蛇(Notechis scutatus)70.83%、大黄鱼(Larimichthys crocea)70.83%(图1(b))。使用邻接法构建系统进化树发现,三疣梭子蟹Aldh与中华绒螯蟹聚为一支,再与凡纳滨对虾、斑节对虾和黑腹果蝇聚为一大支。虎蛇、大海鲢和大黄鱼聚为一大支。三疣梭子蟹等无脊椎动物的Aldh与脊椎动物的Aldh间群体互相独立(图2)。
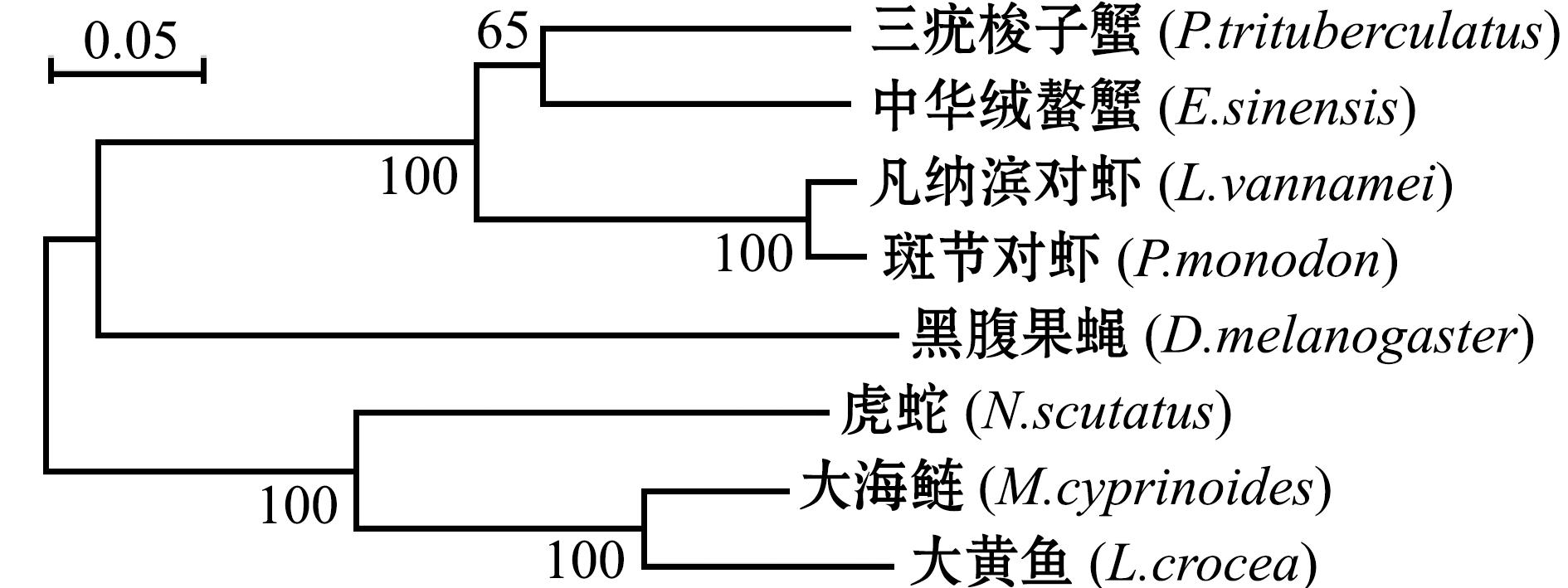
图2 三疣梭子蟹Aldh基因进化树分析
Fig.2 Evolutionary tree analysis of Aldh gene in Portunustrituberculatus
2.2 三疣梭子蟹Aldh基因时空表达模式
Aldh基因在梭子蟹8个组织中的qPCR结果显示,该基因在全部组织中均有表达,其中,在鳃中的表达水平最高,在眼柄中的表达水平最低(图3(a))。低盐胁迫下,三疣梭子蟹Aldh基因的表达水平显著降低,对照组的表达水平为低盐胁迫后表达水平的69.5倍(图3(b))。
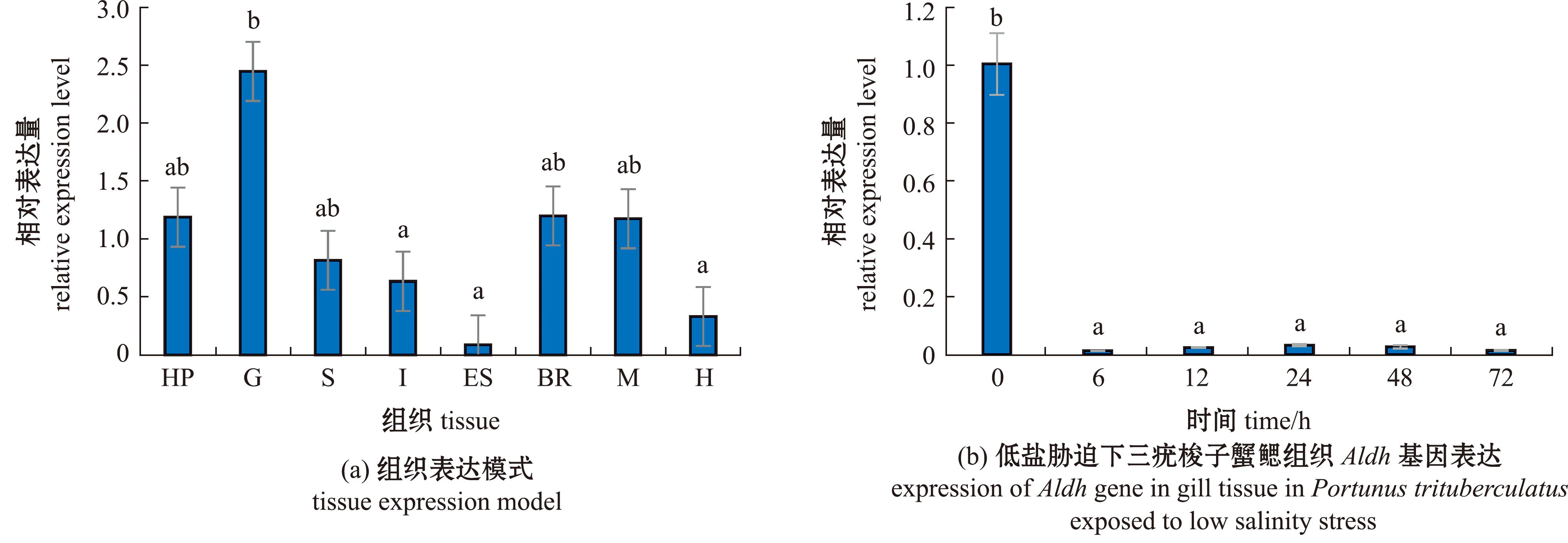
HP—肝胰腺;G—鳃;S—胃;I—肠;ES—眼柄;BR—脑;M—肌肉;H—心脏。标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05),下同。
HP—hepatopancreas;G—gill;S—stomach;I—intestine;ES—eyestalk;BR—brain;M—muscle;H—heart.The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter within the same column are not significant differences,et sequentia.
图3 三疣梭子蟹Aldh基因的时空表达模式
Fig.3 Temporal and spatial expression patterns of the Aldh gene in Portunustrituberculatus
2.3 Aldh基因启动子区域的序列分析及CpG岛预测
通过NCBI将Aldh基因的转录起始位点上游2 000 bp作为启动子序列,采用MethPrimer在线软件预测启动子CpG位点。在启动子中发现1个CpG岛,长度为132 bp(位点1 329~1 460 bp)(图4(a))。采用MethPrimer软件设计亚硫酸氢盐测序PCR引物对,在测序区共检测到4个CG位点(图4(b))。
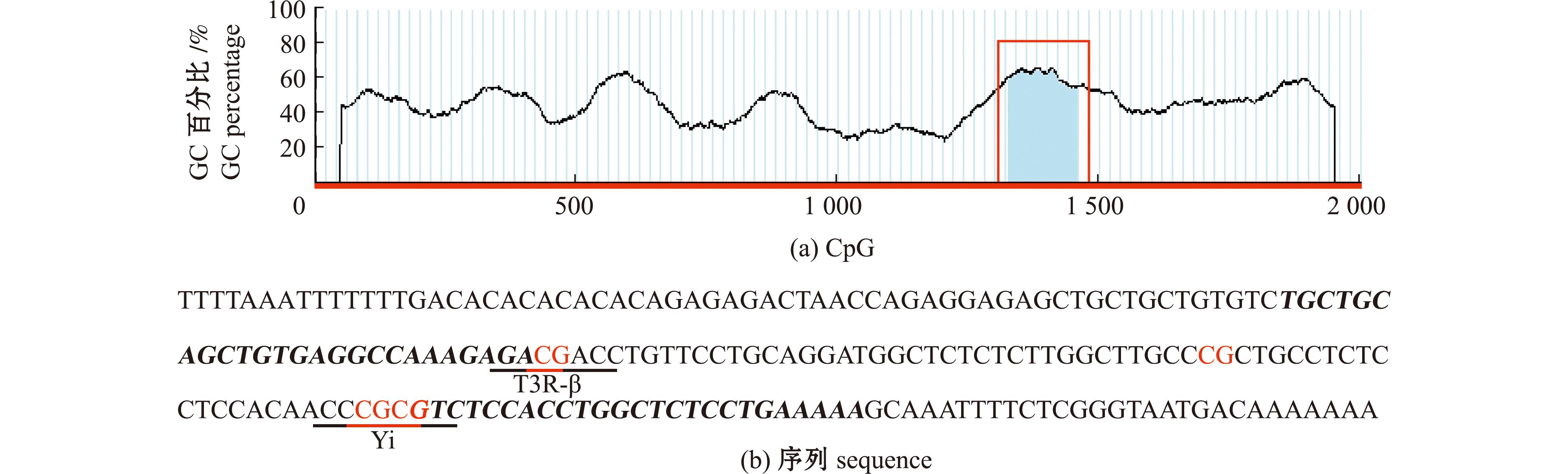
红框中蓝色区域为CpG岛,序列中加粗斜体和红色分别代表引物和CG位点,下划线为预测的转录因子结合位点。
The blue area in the red box is the CpG island,the bolded italics and red color in the sequence represent primers and CG sites,respectively,and the underlines are predicted transcription factor binding sites.
图4 三疣梭子蟹Aldh基因启动子区域CpG岛预测及部分序列
Fig.4 CpG island prediction results and partial sequence of Aldh promoter of Portunustrituberculatus
2.4 低盐胁迫下Aldh基因的甲基化分析
利用QUMA在线软件进行分析,在引物扩增序列中的4个甲基化位点中,1、4位点在低盐胁迫72 h的甲基化水平发生变化。低盐胁迫72 h后Aldh基因启动子的平均甲基化率为85%,0 h的平均甲基化率为72.5%(图5(a))。在1位点中,0 h的甲基化率为0%,而72 h的甲基化率为40%。在2、3位点的甲基化率均为100%。在4位点中,0 h的甲基化率为90%,而72 h的甲基化率为100%(图5(b))。
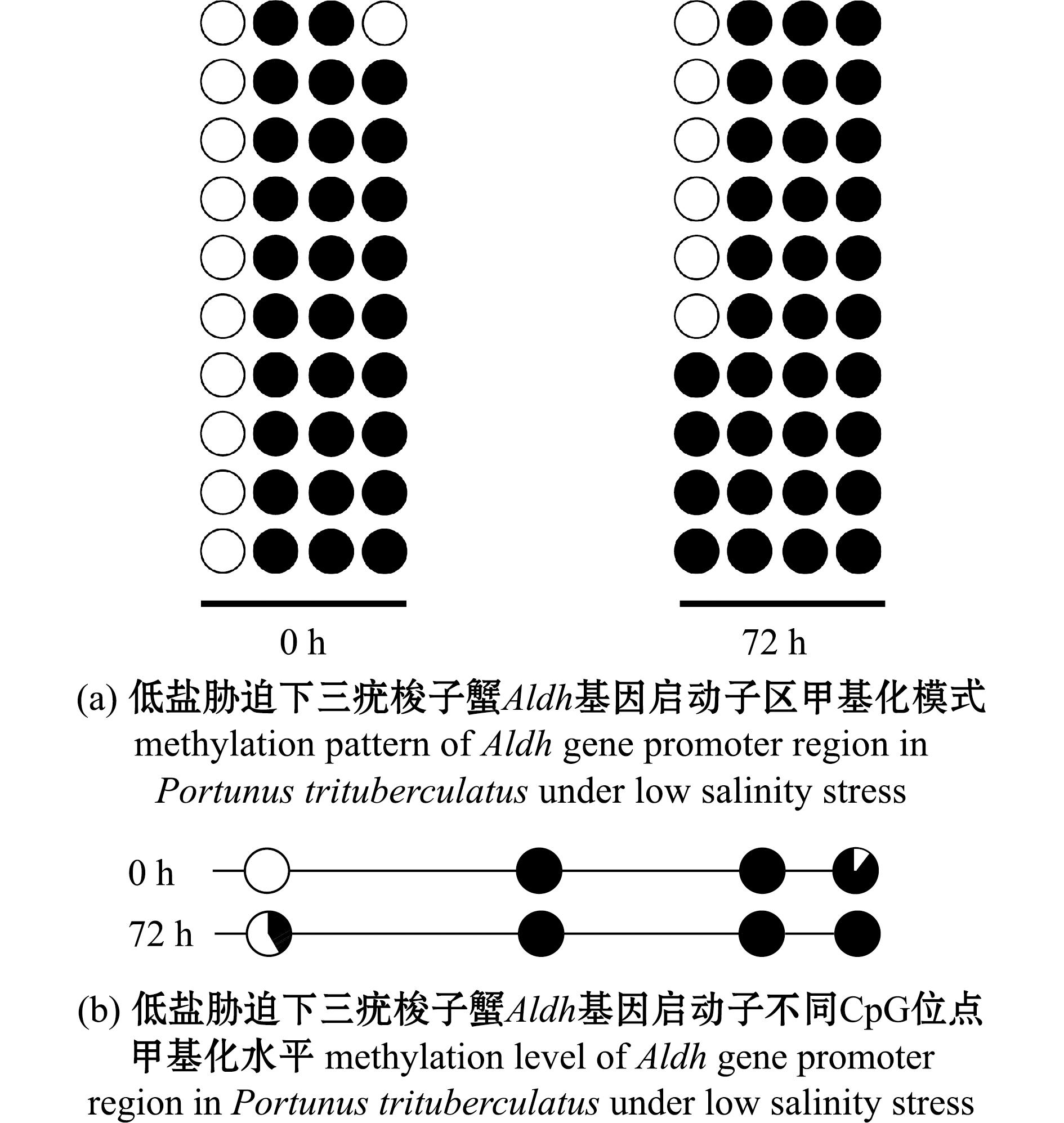
黑色表示发生甲基化,白色表示未发生甲基化。
Methylation is in shown in black color,and no methylation is shown in white color.
图5 三疣梭子蟹Aldh基因在低盐胁迫0 h和72 h甲基化水平
Fig.5 Methylation levels of Aldh gene in Portunustrituberculatus exposed to low salinity for 0 h and 72 h
2.5 MDA含量测定
在低盐胁迫下,三疣梭子蟹鳃组织MDA含量呈现先升高后降低的趋势(图6)。在6 h时,MDA含量达到峰值(25.333 33 μmol/L),与对照组相比具有显著性差异(P<0.05),随后,MDA含量开始下降,虽然与对照组无显著性差异(P>0.05),但仍高于对照组水平。
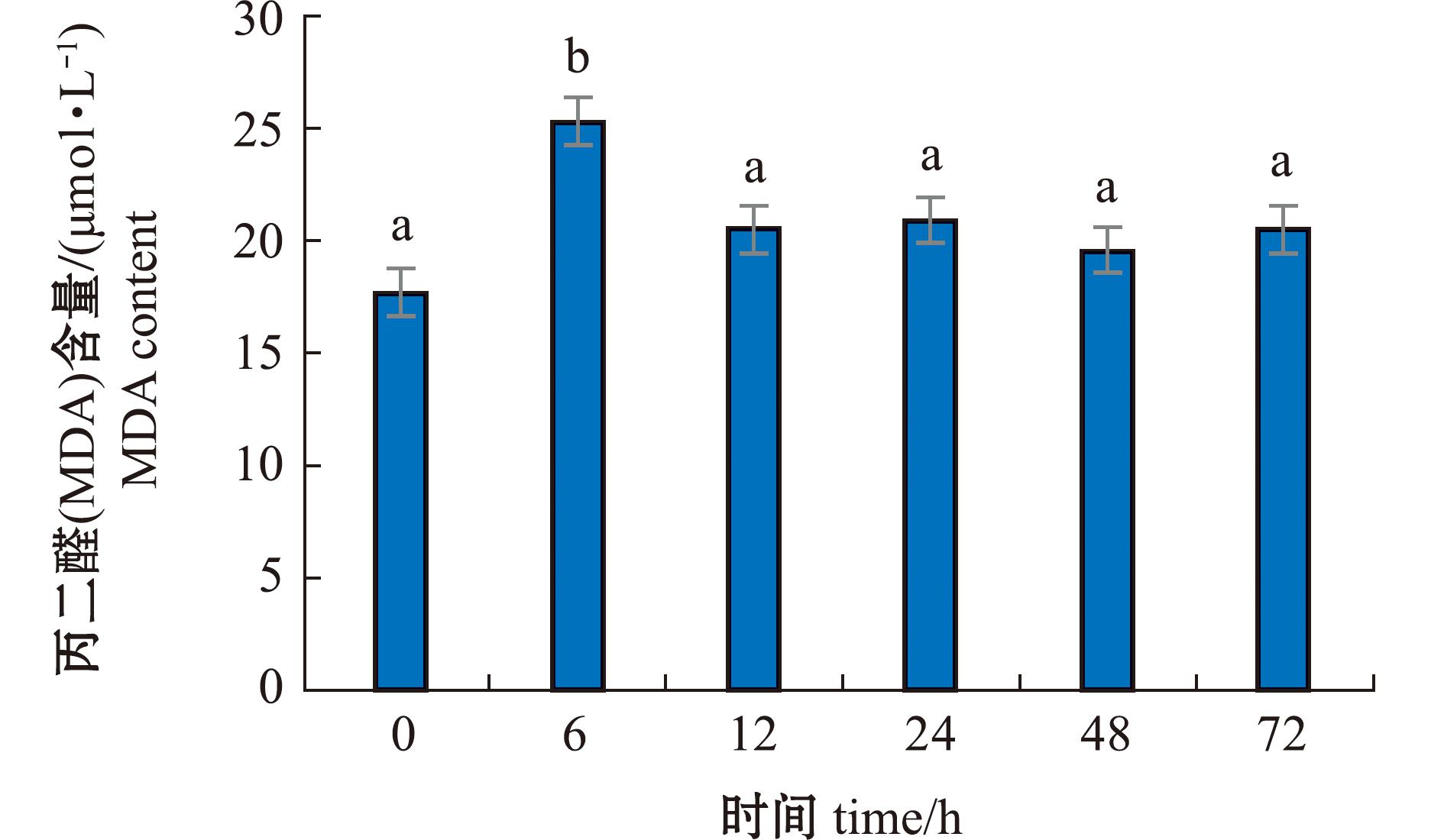
图6 低盐胁迫下三疣梭子蟹鳃组织MDA含量
Fig.6 MDA content in gill tissue of Portunustrituberculatus under low salinity stress
2.6 低盐胁迫下Aldh基因的表达和甲基化的联合分析
采用Pearson相关系数分析Aldh基因在低盐胁迫鳃中的相对表达水平与Aldh启动子区域甲基化水平的相关性。分析表明,Pearson相关系数为-0.992,表明启动子区域的甲基化水平与表达水平存在显著负相关(P<0.05)(图7)。
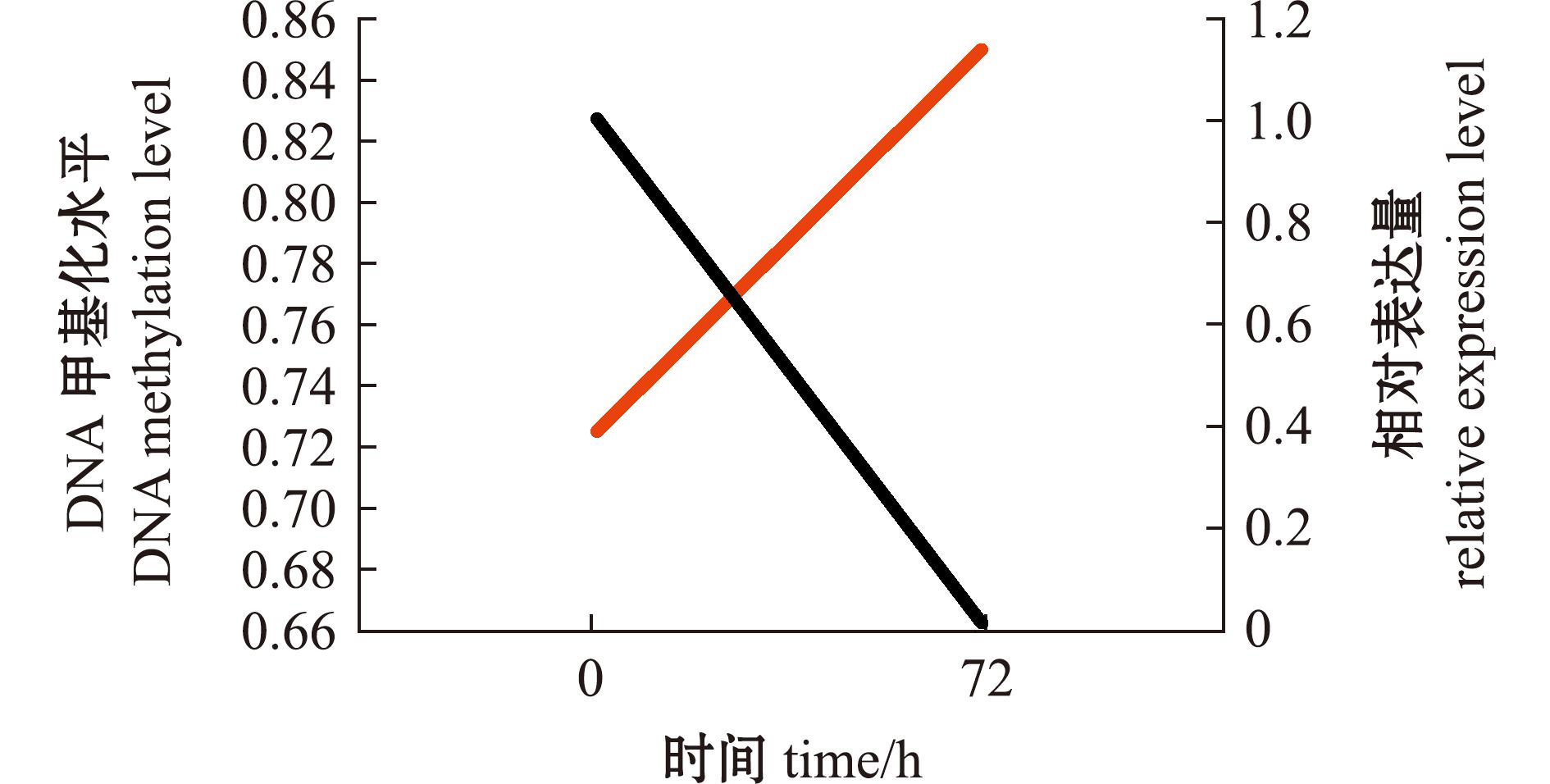
红色为甲基化水平,黑色为表达水平。
Methylation level is shown in red color,and expression level is shown in black color.
图7 低盐胁迫下三疣梭子蟹Aldh基因甲基化和基因表达联合分析
Fig.7 Joint analysis of Aldh gene methylation and expression in Portunustrituberculatus under low salinity stress
3 讨论
3.1 Aldh基因的生物信息学特征
Aldh基因在各种物种的生理活动中都起着至关重要的作用。然而,关于Aldh基因在三疣梭子蟹低盐胁迫方面的研究尚未见报道。首先,信号肽预测结果,显示Aldh基因可能为胞内蛋白,这与大豆中的相关研究相一致[24],表明了其在细胞内的重要作用。其次,通过NAD结合位点的预测,推测Aldh基因在氧化代谢中发挥重要作用[25],这与草鱼的研究结果相符[26],暗示了其在适应低盐胁迫中可能发挥作用。此外,氨基酸同源性分析表明,三疣梭子蟹Aldh基因与同属甲壳纲的中华绒螯蟹的同源性最高,进化树分析符合传统动物分类次序,这为其进化和功能提供了重要线索。
3.2 Aldh基因在三疣梭子蟹中的表达模式及MDA含量
进一步研究表明,Aldh基因在三疣梭子蟹的各组织中普遍表达,这与在斑马鱼[27]和大西洋鲑[28]中的研究结果类似。在本研究中,三疣梭子蟹Aldh基因在鳃组织中的表达量最高。这可能与鳃作为维持三疣梭子蟹渗透压平衡的主要器官有关。鳃组织能够通过主动运输离子和水分子,调节体液中的离子浓度和水分含量,以维持三疣梭子蟹体内外渗透压平衡。研究发现,在低盐胁迫下,Aldh基因表达量显著下降。据报道,在原油胁迫下,眼斑拟石首鱼Aldh基因的表达量也显著降低[29]。MDA含量能够反映低盐胁迫下三疣梭子蟹鳃组织脂质过氧化程度和抗氧化能力,且间接反映细胞受到自由基攻击的严重程度。在低盐胁迫下,三疣梭子蟹鳃组织中MDA含量先增加后降低,但均高于对照组。结果表明,低盐胁迫对三疣梭子蟹机体造成氧化损伤,体内发生脂质过氧化反应,这与低盐驯化下大黄鱼[30]、葛氏长臂虾[31]和凡纳滨对虾[32]的研究结果一致。研究表明,在低盐胁迫下,MDA含量增加,可破坏细胞结构,且鳃组织无法完成正常功能,从而造成Aldh基因在鳃组织中表达量显著降低。
3.3 低盐胁迫下Aldh甲基化水平与基因表达水平的相关性
DNA甲基化是一种生物学过程,其中,甲基基团被添加到DNA分子中的特定位置,通常是在胞嘧啶环上的CpG位点上,从而调节基因表达和细胞功能。在抗逆过程中,DNA甲基化在CpG岛上的调节作用尤为显著。CpG岛是DNA序列中富含CpG二核苷酸的区域,通常位于基因的启动子区域,对基因的表达起着关键调控作用。CpG岛上的DNA甲基化状态可影响基因的表观遗传调控,调节细胞的应激响应和适应能力,有助于维持基因组稳定性和细胞的适应性反应,从而对抗外界环境的挑战。本研究分析了三疣梭子蟹Aldh基因启动子区域的甲基化水平,结果显示,在低盐胁迫条件下,Aldh基因的甲基化水平增加,而其表达水平则出现下降。这与大鼠的研究结果一致[33],这也表明高度保守的基因DNA甲基化调控机制在不同物种间具有相对的一致性。本研究表明,在低盐胁迫下三疣梭子蟹Aldh基因甲基化水平与mRNA呈负相关关系,这与碳酸盐碱胁迫下,拟穴青蟹Sp-CA基因的甲基化水平与表达水平研究结果一致[34]。此外,低盐胁迫下,在大黄鱼鳃[35]、肾脏[36]和肝脏[37]中,启动子区域的差异甲基化基因与基因表达均呈负相关关系。这些结果表明,低盐胁迫可能导致三疣梭子蟹Aldh甲基化水平升高,从而抑制启动子活性,导致Aldh表达水平降低及MDA含量升高,并引起三疣梭子蟹鳃组织损伤。
4 结论
1)三疣梭子蟹Aldh基因显示出稳定的基因结构和亲水的蛋白质特性,表明其在胞内可能具有重要功能。同源性分析显示,三疣梭子蟹与中华绒螯蟹有密切联系,其与无脊椎动物之间具有保守性。
2)Aldh基因在三疣梭子蟹各组织中均有表达,鳃中最高,眼柄最低。低盐胁迫下,Aldh基因表达显著下降,约为对照组的1/70。低盐胁迫导致鳃组织中MDA含量先升后降。结果表明,三疣梭子蟹Aldh基因在低盐环境中发挥着重要作用。
3)低盐胁迫条件下,Aldh基因启动子区域的甲基化水平显著增加,与Aldh基因的表达水平呈现负相关(P<0.05)。这表明甲基化水平可能对Aldh基因的表达起调节作用,并参与三疣梭子蟹对低盐环境的适应性反应。
[1] YAMAUCHI M M,MIYA M U,NISHIDA M.Complete mitochondrial DNA sequence of the swimming crab,Portunus trituberculatus (Crustacea:Decapoda:Brachyura)[J].Gene,2003,311:129-135.
[2] LÜ J J,LIU P,WANG Y,et al.Transcriptome analysis of Portunus trituberculatus in response to salinity stress provides insights into the molecular basis of osmoregulation[J].PLoS One,2013,8(12):e82155.
[3] 韩晓琳,高保全,王好锋,等.低盐胁迫对三疣梭子蟹鳃和肝胰腺显微结构及家系存活的影响[J].渔业科学进展,2014,35(1):104-110.HAN X L,GAO B Q,WANG H F,et al.Effects of low salinity stress on microstructure of gill and hepatopancreas and family survival rate of Portunus trituberculatus[J].Progress in Fishery Sciences,2014,35(1):104-110.(in Chinese)
[4] LU Y L,ZHANG J Y,CAO J W,et al.Long-term ammonia toxicity in the hepatopancreas of swimming crab Portunus trituberculatus:cellular stress response and tissue damage[J].Frontiers in Marine Science,2022,8:757602.
[5] ZANETTE J,DE ALMEIDA E A,DA SILVA A Z,et al.Salinity influences glutathione S-transferase activity and lipid peroxidation responses in the Crassostrea gigas oyster exposed to diesel oil[J].The Science of the Total Environment,2011,409(10):1976-1983.
[6] CAO Y X,WANG J,ZHAO S Q,et al.Overexpression of the aldehyde dehydrogenase AhALDH3H1 from Arachis hypogaea in soybean increases saline-alkali stress tolerance[J].Frontiers in Plant Science,2023,14:1165384.
[7] CHEN Z,CHEN M,XU Z S,et al.Characteristics and expression patterns of the aldehyde dehydrogenase (ALDH) gene superfamily of foxtail millet (Setaria italica L.)[J].PLoS One,2014,9(7):e101136.
[8] HE D H,LEI Z P,XING H Y,et al.Genome-wide identification and analysis of the aldehyde dehydrogenase (ALDH) gene superfamily of Gossypium raimondii[J].Gene,2014,549(1):123-133.
[9] SUNKAR R,BARTELS D,KIRCH H H.Overexpression of a stress-inducible aldehyde dehydrogenase gene from Arabidopsis thaliana in transgenic plants improves stress tolerance[J].The Plant Journal,2003,35(4):452-464.
[10] KIRCH H H,BARTELS D,WEI Y L,et al.The ALDH gene superfamily of Arabidopsis[J].Trends in Plant Science,2004,9(8):371-377.
[11] YANG C L,ZHOU Y,FAN J,et al.SpBADH of the halophyte Sesuvium portulacastrum strongly confers drought tolerance through ROS scavenging in transgenic Arabidopsis[J].Plant Physiology and Biochemistry:PPB,2015,96:377-387.
[12] LÜ J J,SUN D F,YAN D P,et al.Quantitative trait loci mapping and marker identification for low salinity tolerance trait in the swimming crab (Portunus trituberculatus)[J].Frontiers in Genetics,2019,10:1193.
[13] LIANG P,SONG F,GHOSH S,et al.Genome-wide survey reveals dynamic widespread tissue-specific changes in DNA methylation during development[J].BMC Genomics,2011,12(1):231.
[14] ZHANG C W,XUE P,GAO L Z,et al.Highly conserved epigenetic regulation of BOULE and DAZL is associated with human fertility[J].The FASEB Journal,2016,30(10):3424-3440.
[15] WEBER M,HELLMANN I,STADLER M B,et al.Distribution,silencing potential and evolutionary impact of promoter DNA methylation in the human genome[J].Nature Genetics,2007,39(4):457-466.
[16] CARREL L,WILLARD H F.X-inactivation profile reveals extensive variability in X-linked gene expression in females[J].Nature,2005,434(7031):400-404.
[17] SI Y F,HE F,WEN H S,et al.DNA methylation of pacap gene is involved in growth superiority of female half smooth tongue sole (Cynoglossus semilaevis) in different salinities[J].Journal of Ocean University of China,2021,20(1):169-180.
[18] LI S P,HE F,WEN H S,et al.Low salinity affects cellularity,DNA methylation,and mRNA expression of igf1 in the liver of half smooth tongue sole (Cynoglossus semilaevis)[J].Fish Physiology and Biochemistry,2017,43(6):1587-1602.
[19] LI S P,HE F,WEN H S,et al.Half smooth tongue sole (Cynoglossus semilaevis) under low salinity stress can change hepatic igf 2 expression through DNA methylation[J].Journal of Ocean University of China,2020,19(1):171-182.
[20] SU H H,MA D M,SHANG H W,et al.DNA methylation of the prkaca gene involved in osmoregulation in tilapia hybrids (Oreochromis mossambicus × Oreochromis hornorum)[J].Gene,2020,752:144791.
[21] LU S K,LI R H,GAO T L,et al.Analysis of DNA methylation level of Portunus trituberculatus subjected to low salinity with methylation-sensitive amplification polymorphism[J].Journal of Ocean University of China,2019,18(5):1158-1162.
[22] 隋延鸣,高保全,刘萍,等.三疣梭子蟹“黄选1号” 盐度耐受性分析[J].渔业科学进展,2012,33(2):63-68.SUI Y M,GAO B Q,LIU P,et al.Preliminary studies on the salinity tolerance of “Huangxuan No.1” Portunus trituberculatus[J].Progress in Fishery Sciences,2012,33(2):63-68.(in Chinese)
[23] COURTNEY A J,XU J C,XU Y.Responses of growth,antioxidants and gene expression in smooth cordgrass (Spartina alterniflora) to various levels of salinity[J].Plant Physiology and Biochemistry:PPB,2016,99:162-170.
[24] 黄方,迟英俊,何慧,等.大豆醛脱氢酶基因GmALDH3-1的克隆及在生殖器官中的表达[J].遗传,2010,32(5):492-497.HUANG F,CHI Y J,HE H,et al.Cloning and characterization of an aldehyde dehydrogenase gene GmALDH3-1 from soybean[J].Hereditas,2010,32(5):492-497.(in Chinese)
[25] SU W H,ZHANG C,WANG D J,et al.A comprehensive survey of the aldehyde dehydrogenase gene superfamily in Saccharum and the role of ScALDH2B-1 in the stress response[J].Environmental and Experimental Botany,2022,194:104725.
[26] FONG W,CHOY K.Purification and characterization of grass carp mitochondrial aldehyde dehydrogenase[J].Chemico-Biological Interactions,2001,130/131/132(1/2/3):161-171.
[27] LASSEN N,ESTEY T,TANGUAY R L,et al.Molecular cloning,baculovirus expression,and tissue distribution of the zebrafish aldehyde dehydrogenase 2[J].Drug Metabolism and Disposition:the Biological Fate of Chemicals,2005,33(5):649-656.
[28] MINGHETTI M,OLSEN R E,TOCHER D R.Molecular cloning,characterization and nutritional regulation of key enzymes required for the effective utilization of marine wax esters by Atlantic salmon (Salmo salar L.)[J].Aquaculture Nutrition,2010,16(5):483-495.
[29] CA IZARES-MART
IZARES-MART NEZ M A,QUINTANILLA-MENA M,DEL-R
NEZ M A,QUINTANILLA-MENA M,DEL-R O-GARC
O-GARC A M,et al.Acute exposure to crude oil induces epigenetic,transcriptional and metabolic changes in juvenile Sciaenops ocellatus[J].Bulletin of Environmental Contamination and Toxicology,2022,108(1):85-92.
A M,et al.Acute exposure to crude oil induces epigenetic,transcriptional and metabolic changes in juvenile Sciaenops ocellatus[J].Bulletin of Environmental Contamination and Toxicology,2022,108(1):85-92.
[30] 张惠,曾霖,熊逸飞,等.盐度驯化改善大黄鱼盐度胁迫耐受性的作用机制[J].中国水产科学,2023,30(3):334-343.ZHANG H,ZENG L,XIONG Y F,et al.Mechanism of salinity acclimation in Larimichthys crocea improving tolerance to salinity stress[J].Journal of Fishery Sciences of China,2023,30(3):334-343.(in Chinese)
[31] 胡润豪,史文军,王盼,等.温度、盐度变化对葛氏长臂虾存活和抗氧化能力的影响[J].中国水产科学,2022,29(4):535-548.HU R H,SHI W J,WANG P,et al.Effects of temperature and salinity on the survival and antioxidant capacity of Palaemon gravieri[J].Journal of Fishery Sciences of China,2022,29(4):535-548.(in Chinese)
[32] SHOFIATUN S,HANDAYANI K S,SURYANI H,et al.Oxidative stress responses in gills of white shrimp Litopenaeus vannamei Boone,1931 following exposure to cadmium at different salinities[J].Cahiers De Biologie Marine,2017,58(4):461-466.
[33] WANG P,SHEN C,DIAO L,et al.Aberrant hypermethylation of aldehyde dehydrogenase 2 promoter upstream sequence in rats with experimental myocardial infarction[J].Bio Med Research International,2015,2015:503692.
[34] WANG X P,FANG W,LIU L,et al.Molecular characterization and DNA methylation analysis of carbonic anhydrase (Sp-CA) in the mud crab Scylla paramamosain: its potential osmoregulation role under carbonate alkalinity stress[J].Aquaculture Reports,2023,30:101591.
[35] YANG J,LIU M H,ZHOU T T,et al.Transcriptome and methylome dynamics in the gills of large yellow croaker (Larimichthys crocea) during low-salinity adaption[J].Frontiers in Marine Science,2023,10:1082655.
[36] YANG J,LIU M H,ZHOU T T,et al.Genome-wide methylome and transcriptome dynamics provide insights into epigenetic regulation of kidney functioning of large yellow croaker (Larimichthys crocea) during low-salinity adaption[J].Aquaculture,2023,571:739410.
[37] ZHANG Y,ZHU F,TENG J,et al.Effects of salinity stress on methylation of the liver genome and complement gene in large yellow croaker (Larimichthys crocea)[J].Fish &Shellfish Immunology,2022,129:207-220.