蜡样芽孢杆菌群是包含多种表型特征的基因封闭细菌群[1];基于NCBI分类系统至少包括22个种[2],其中以炭疽芽孢杆菌和蜡样芽孢杆菌最为引人注意[3]。2017年,Liu等[4]首次从太平洋沉积物中分离报道了副炭疽芽孢杆菌(Bacillus paranthracis,BP)模式株Mn5T这一蜡样芽孢杆菌群的近缘新种。此后,陆续从各类环境和生物样品,如人粪便[5]、埃博拉病毒感染者血液[6]、书页表面[7]、糠床[8]、蜂蜜[9]、三角羽峭壁蕨[10]、生活垃圾[11]和土壤[12]等中分离到副炭疽芽孢杆菌。副炭疽芽孢杆菌中的某些菌株可以作为动植物的益生菌[9,12],某些菌株可以作为酶制剂的工程菌[13],也有一些菌株是条件致病菌[7]。
目前,致病性副炭疽芽孢杆菌的研究较少,已知其可引起人的呕吐腹泻[14]和菌血症[6],可致中国大鲵的皮肤溃疡和腹水[15],以及奶牛乳腺炎[16],尚未见鱼源致病性副炭疽芽孢杆菌的相关报道。以全基因组测序为基础,从毒力基因和耐药基因角度理解致病菌株特性是细菌病原生物学研究的新趋势。目前致病性副炭疽芽孢杆菌的全基因组信息还比较缺乏,相关报道较少[7]。从书页表面分离的副炭疽芽孢杆菌BCCL01株携带的毒力基因有磷脂酶C(plc)、细胞溶素(cytolysin)、金属蛋白酶A抑制剂(inhA)和非溶血性肠毒素NHE的nheA/B/C,携带的耐药基因包括编码多药耐药外排泵、β-内酰胺酶、磷霉素耐药蛋白(FosB)和万古霉素B型耐药蛋白(VanW)[7]。从奶牛乳腺炎中分离的副炭疽芽孢杆菌4M株、1702株和1710株携带的毒力基因包括非溶血性肠毒素(Nhe)的nheA/B/C、inhA、hblA和cytK2;携带的耐药基因包括vmlR和fosB[16]。从中国大鲵分离的副炭疽芽孢杆菌SHOU-BC01株携带的毒力基因包括鞘磷脂酶(SMase)的sph、细胞毒素(CytK)、非溶血性肠毒素(Nhe)的nheC、胞外多糖(BPS)的bpsC、芽孢杆菌素(bacillibactin)的dhbA/B/C/E/F、表面蛋白(IlsA)、PlcR-PapR群感效应系统(papR和plcR)及Ⅶ型分泌系统的essC、esxB和esxL[15]。本研究中从患病团头鲂(Megalobrama amblycephala)内脏中分离到一株副炭疽芽孢杆菌BC006株,在全基因组测序基础上对其基因结构与功能、毒力基因与耐药基因进行解析,以期能加深对渔源致病性多重耐药副炭疽芽孢杆菌的理解。
1 材料与方法
1.1 材料
患病团头鲂体表和鳃丝发白,腹胀背瘦,眼周、腹部和鳍条充血出血,蛀鳍,肝胰腺溃烂、发白且有暗红色淤斑,脾脏肿胀发黑,体肾肿胀、色黯淡,肠道壁薄如纸,肠道胀气,肠内容物有腐败气味。
将患病鱼肝、脾和肾混合物涂布于MH平板上,获得形态均一的菌落,选6个菌落纯化后,得到一株对团头鲂致死率最高且引起哥伦比亚血琼脂β-溶血的菌株,命名为BC006。
1.2 方法
1.2.1 基于保守基因序列的菌株分子生物学鉴定 由于16S rDNA只能将BC006鉴定为蜡样芽孢杆菌群,进一步采用管家基因gyrB、rpoB与tuf将BC006鉴定到种[17](表1),并以副炭疽芽孢杆菌(Bacillus paranthracis)CP101136株、炭疽芽孢杆菌(Bacillus anthracis)AE017334株、蜡样芽孢杆菌(Bacillus cereus)ATCC14579株、苏云金芽孢杆菌(Bacillus thuringiensis) ATCC10792株、短小芽孢杆菌(Bacillus pumilus)ATCC7061株、枯草芽孢杆菌(Bacillus subtilis)ATCC6051株、地衣芽孢杆菌(Bacillus licheniformis)ATCC14580株和金黄色葡萄球菌(Staphylococcus aureus)ATCC12600株的对应序列为参考进行系统发育分析。
表1 用于BC006分子鉴定的引物
Tab.1 Primers for molecular identification of BC006
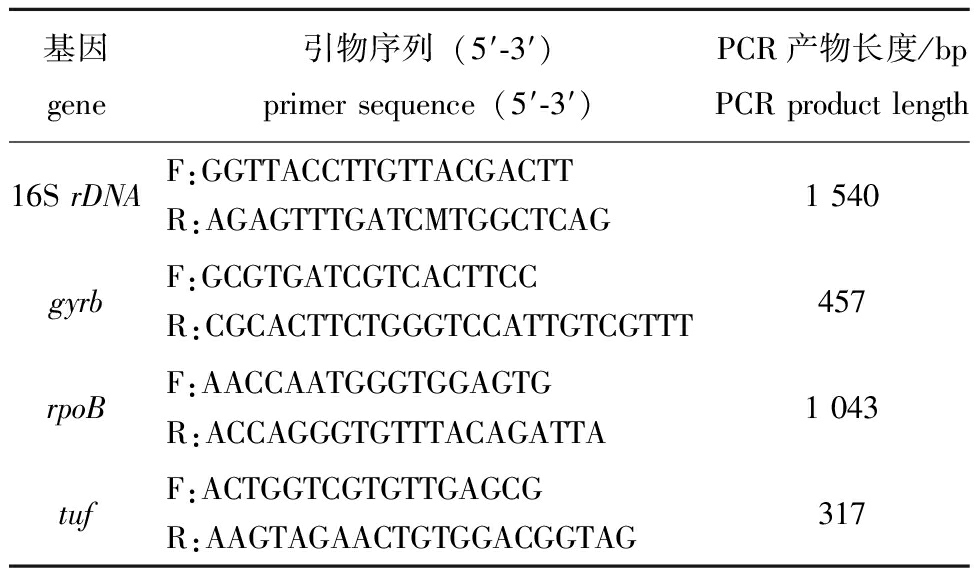
基因gene引物序列(5′-3′)primer sequence(5′-3′)PCR产物长度/bpPCR product length16S rDNAF:GGTTACCTTGTTACGACTTR:AGAGTTTGATCMTGGCTCAG1 540gyrbF:GCGTGATCGTCACTTCCR:CGCACTTCTGGGTCCATTGTCGTTT457rpoBF:AACCAATGGGTGGAGTGR:ACCAGGGTGTTTACAGATTA1 043tufF:ACTGGTCGTGTTGAGCGR:AAGTAGAACTGTGGACGGTAG317
1.2.2 BC006株全基因组测序 BC006株由武汉希望组生物有限公司采用PacBio和Illumina进行全基因组测序。基因组序列提交NCBI,获得登录号。使用Circos(v0.69)软件绘制基因组圈图。
1.2.3 平均核酸相似度 采用EZbiocloud分析副炭疽芽孢杆菌BC006株与8株近缘物种的平均核酸相似度(average nucleotide identity,ANI)[18]。
1.2.4 基因注释 利用COG、KEGG、GO、Refseq、Pfam和TIGRFAMs数据库对编码蛋白进行功能注释。
1.2.5 基因组共线性分析 基因组共线性分析用mauve进行,使用的7株副炭疽芽孢杆菌包括牛源1710株(CP113385.1)、人肠道源AM04S-41株(NZ_JAQDIH010000001.1)、大鲵源SHOU-BC01株(CP089743.1)、人血液源ELWA-3_3298株(NZ_WQMW01000001.1)、爬行动物源PIW23株(NZ_JAKJQA010000001.1)、益生菌株2-2株(NZ_NJQT01000004.1)与团头鲂源BC006株(CP119629)。
1.2.6 菌株致病力与毒力基因分析 健康团头鲂(体质量为5 g±2 g)购自安徽海辉水产养殖有限公司。清洁级昆明白小鼠(体质量为20 g±3 g)购自安徽医科大学试验动物中心。根据动物个体的体质量调整攻毒菌液浓度,攻毒组每只动物腹腔注射菌液0.1 mL、对照组腹腔注射等体积灭菌PBS。每个剂量组放置10只动物,设1010、109、108、107、106 CFU/g 5个攻毒组和1个对照组。菌液浓度由OD625 nm=0.1约为108 CFU/mL标定大致数量级,最终用平板计数法确定准确浓度。攻毒期间,养殖团头鲂水温为(25±1)℃,停食充气;小鼠正常供水供料。观察10 d,记录每天死亡量,解剖濒死动物,观察病理变化,取肝、脾和肾分离菌株并鉴定。采用Reed-Muench法计算LD50。通过毒力因子数据库(virulence factors database,VFDB)分析毒力基因。
1.2.7 药敏试验与耐药基因分析 采用K-B纸片法测定BC006对31种抗生素的敏感性。通过抗生素耐药性数据库(comprehensive antibiotic resistance database,CARD)分析耐药基因。
2 结果与分析
2.1 基于保守基因的BC006株分子生物学鉴定
蜡样芽孢杆菌群的炭疽芽孢杆菌、副炭疽芽孢杆菌、蜡样芽孢杆菌和苏云金芽孢杆菌的16S rDNA序列一致性均在97%以上,无法区分这4个近缘物种。管家基因gyrB、rpoB和tuf分别编码DNA旋酶(DNAgyrase)B亚基、RNA 聚合酶B亚基和延伸因子,根据其中任何一个基因构建的系统发育树,都可区分副炭疽芽孢杆菌BC006与其他3个近缘物种,BC006株的16S rDNA、gyrb、rpoB和tuf系统发育树如图1所示。
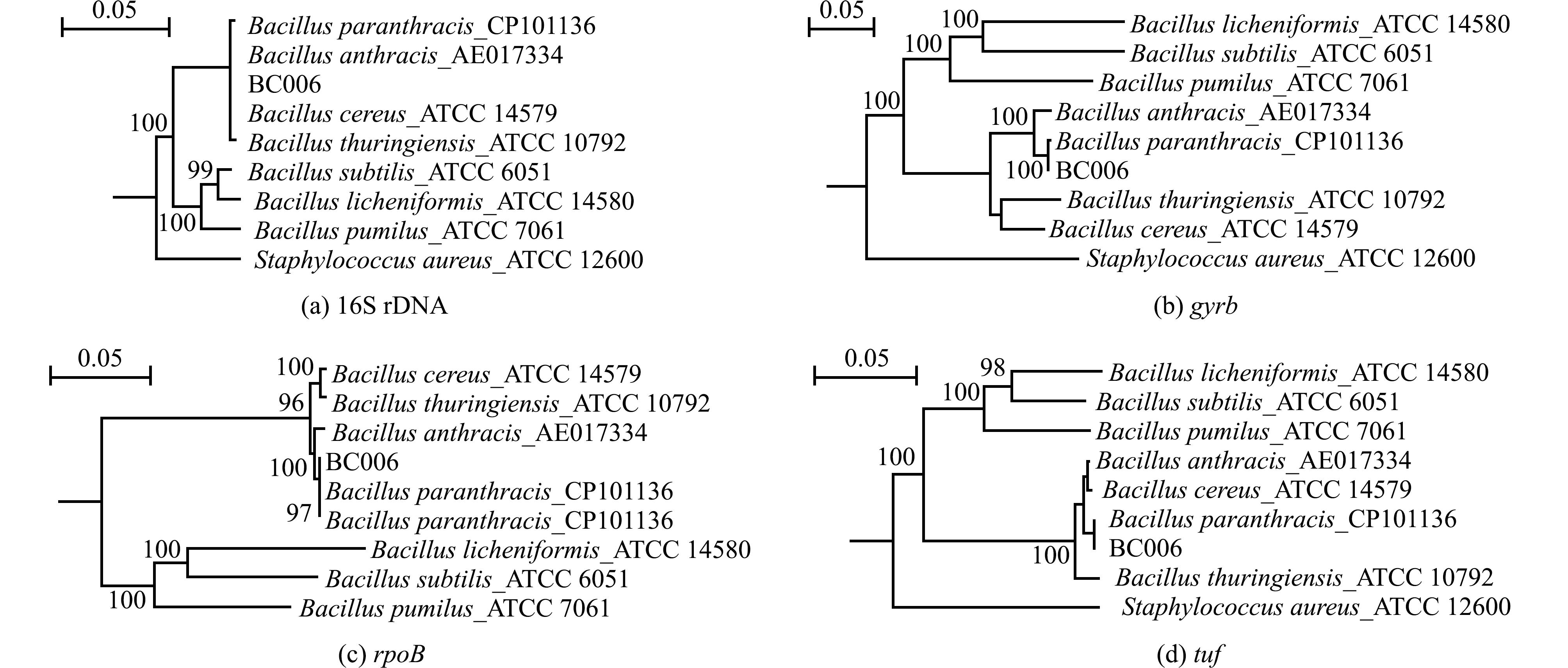
图1 BC006的16SrDNA、gyrb、rpoB和tuf系统发育树
Fig.1 Phylogenetic tree of BC006 based on 16S rDNA,gyrB,rpoB and tuf
2.2 BC006基因组概况
副炭疽芽孢杆菌BC006全基因组5 457 931 bp,GC含量为35.56%,含1个染色体(GenBank ID CP119629)和7个质粒(GenBank ID CP119630~CP119636)。该全基因组共有5 748个基因结构,其中编码区(coding sequence,CDS) 5 539个,tRNA 105个,rRNA 42个,基因组岛(genomic island)61个,原噬菌体区域(prophage region)1个。副炭疽芽孢杆菌BC006染色体测序深度、GC含量、GC-skew及基因组结构注释进行整合,绘制副炭疽芽孢杆菌BC006染色体基因组圈如图2所示。
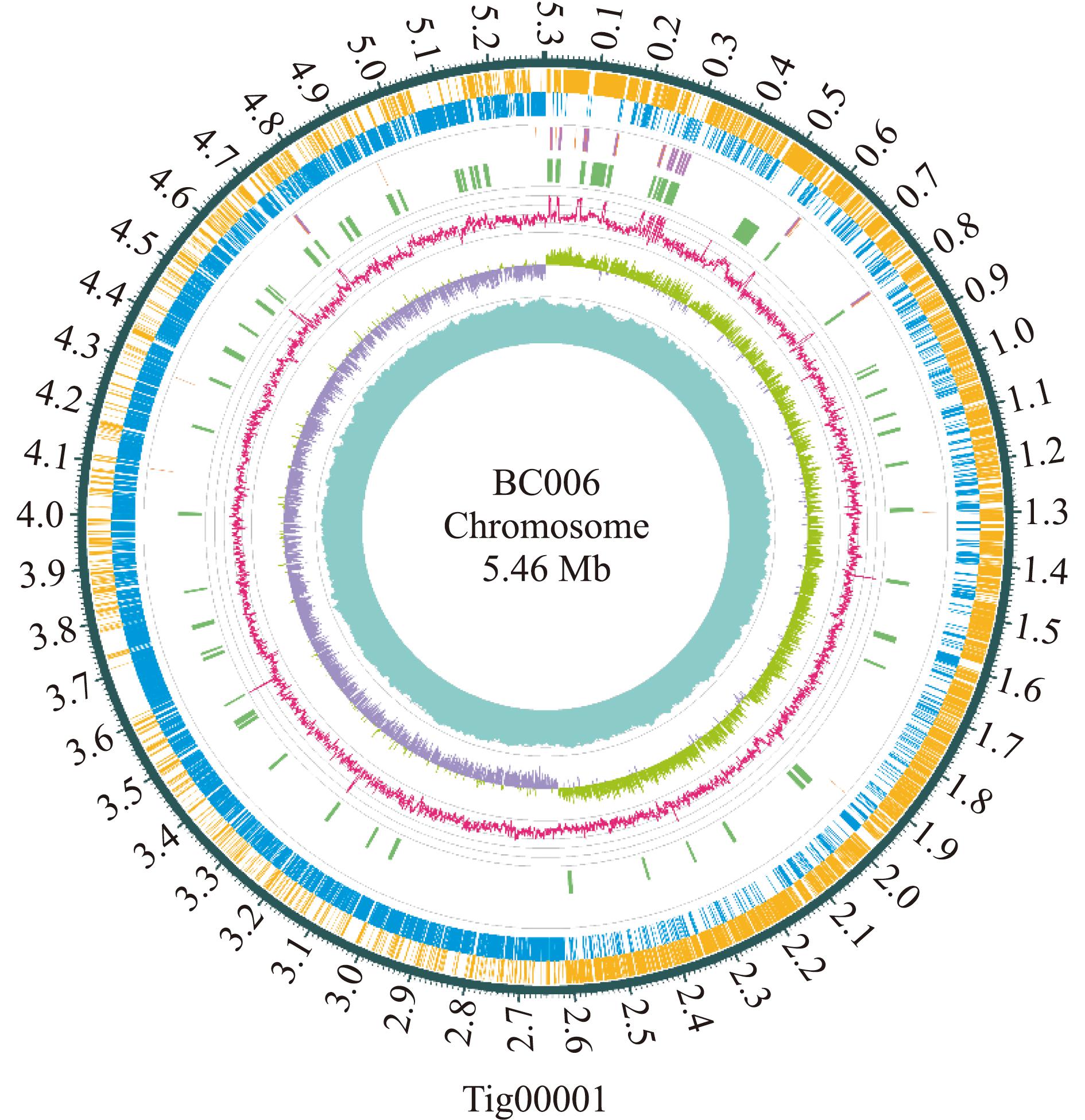
由外到内依次为编码基因(正义链),编码基因(负义链),tRNA(橙色)和rRNA(紫色),CRISPR、原噬菌体和基因岛,GC比(%),GC-skew,测序深度(×)。
From outside to inside,coding gene (sense strand),coding gene (antisense strand),tRNA(orange) and rRNA(purple),CRISPR,prophage region and genomic island,GC content (%),GC-skew,sequencing depth (×).
图2 副炭疽芽孢杆菌BC006染色体基因组圈
Fig.2 Chromosomal genome loop diagram of Bacillusparanthracis strain BC006
2.3 平均核苷酸相似度
基于全基因组序列的两个菌株的平均核苷酸相似度(ANI)大于95%,被定义为同一物种。BC006菌株与副炭疽芽孢杆菌(CP101136)、炭疽芽孢杆菌(AE017334)、蜡样芽孢杆菌(ATCC 14579)、苏云金芽孢杆菌(ATCC 10792)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens ATCC 23350)和短小芽孢杆菌(ATCC 7061)的ANI依次为97.85%、95.38%、91.70%、91.52%、69.11%和69.07%。从ANI角度,BC006株可被鉴定为副炭疽芽孢杆菌。
2.4 基因功能注释分析
副炭疽芽孢杆菌BC006全基因组经同源基因族数据库(database of clusters of orthologous genes,COG)、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)、基因本体数据库(gene ontology,GO)、参考序列数据库(reference sequence database,RefSeq)、同源蛋白家族数据库(homologous protein family,Pfam)和蛋白质序列库(institute for genomic research’s database,TIGRFAMs)注释基因数量分别为3 957、2 502、2 902、5 483、4 546、1 420个。
2.4.1 COG富集分析 COG注释的副炭疽芽孢杆菌BC006的编码基因有3 957个(图3),其中387个参与氨基酸的转运与代谢,占9.78%;386个负责转录,占9.75%;385个参与一般生物功能,占9.73%;303个参与翻译、核糖体结构生物过程,占7.66%;272个参与碳水化合物的运输与代谢,占6.97%;169个参与脂质的转运与代谢,占4.27%;225个参与无机离子的转运与代谢,占5.69%;215个参与能量的生产与转化,占5.43%,262个参与信号转导机制,占6.62%;147个负责防御,占3.71%。
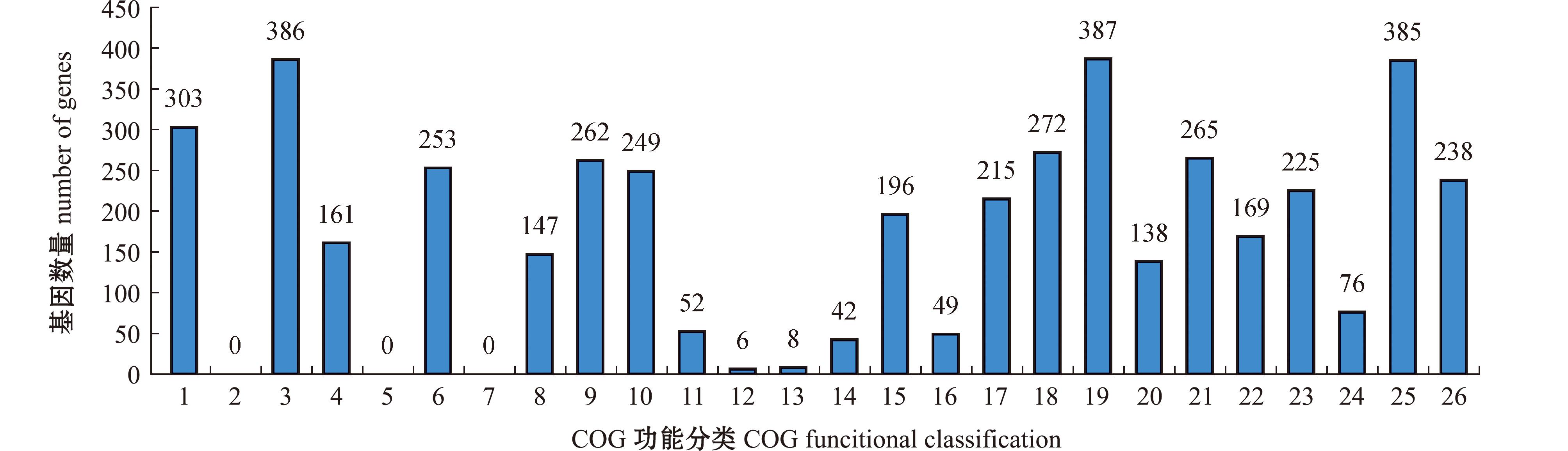
1—translation,ribosomal structure and biogenesis;2—RNA processing and modification;3—transcription;4—replication,recombination and repair;5—chromatin structure and dynamics;6—cell cycle control,cell division,chromosome partitioning;7—nuclear structure;8—defense mechanisms;9—signal transduction mechanisms;10—cell wall/membrane/envelope biogenesis;11—cell motility;12—cytoskeleton;13—extracellular structure;14—intracellular trafficking,secretion,and vesicular transport;15—posttranslational modification,protein turnover,chaperones;16—mobilome:prophages,transposons;17—energy production and conversion;18—carbohydrate transport and metabolism;19—amino acid transport and metabolism;20—nucleotide transport and metabolism;21—coenzyme transport and metabolism;22—lipid transport and metabolism;23—inorganic ion transport and metabolism;24—secondary metabolites biosynthesis,transport and catabolism;25—general function prediction only;26—function unknown.
图3 BC006基因组编码蛋白COG功能分类统计
Fig.3 COG functional classification statistics of genomic coding proteins of BC006
2.4.2 KEGG富集分析 KEGG注释的副炭疽芽孢杆菌BC006的基因有2 502个,被富集到6大类、213个代谢途径(图4)。其中,854个基因参与代谢,占34.13%;215个基因参与遗传信息处理,占8.59%;303个基因参与环境信息处理,占12.11%;202个基因参与细胞过程,占8.07%;50个基因参与生物系统,占2.00%;106个基因参与人类疾病,占4.24%,其中与细菌感染有关的基因18个,与细菌耐药机制有关的基因48个。
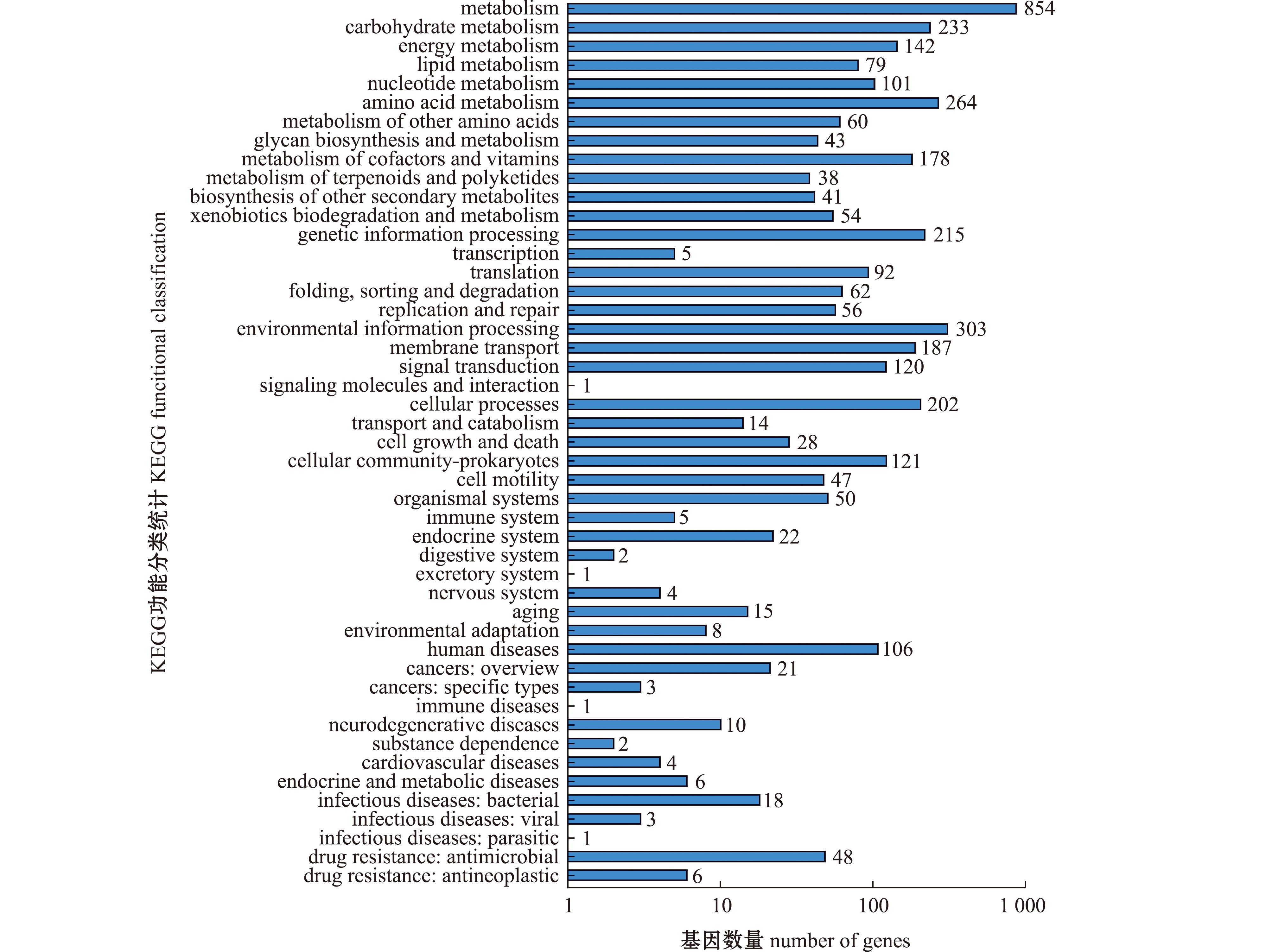
图4 BC006基因组编码蛋白KEGG功能分类统计
Fig.4 Functional classification statistics of genome coding protein KEGG of BC006
2.4.3 GO富集分析 GO 分析描述基因的分子功能(molecular function)、参与的生物过程(biological process)、所处的细胞成分(cellular component)。GO注释的副炭疽芽孢杆菌BC006基因2 902个(图5)。参与分子功能的基因中,2 123个参与催化活性,占73.16%;433个参与转运活性,占14.92%;977个参与分子结合,占33.67%。参与生物过程的基因中,2 433个参与代谢过程,占83.84%;2 123个参与细胞过程,占73.16%;1 296个参与单有机体过程,占44.66%;610个参与细胞过程调节,占21.02%;412个参与刺激应答,占14.20%;673个参与定位,占23.19%;621个参与生物调节,占21.40%。参与细胞组分的基因中,632个参与细胞内组分,占21.78%;600个参与细胞膜组分,占20.68%;329个参与大分子复合体,占11.34%。
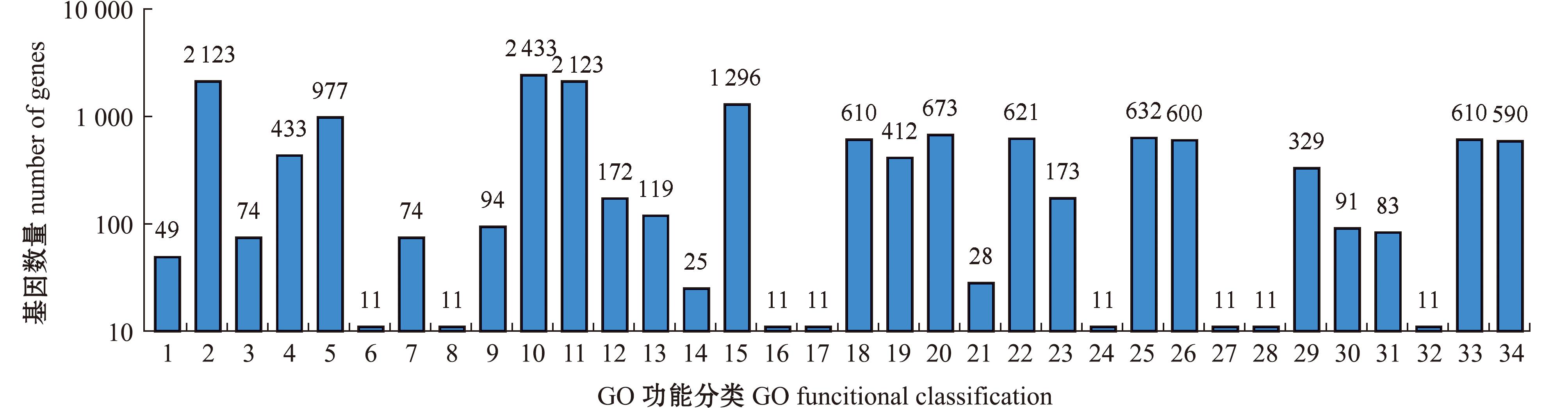
1-8—molecular function;9-23—biological process;24-34—cellular component.1—GO:0000988 transcription factor activity,protein binding;2—GO:0003824 catalytic activity;3—GO:0004871 signal transducer activity;4—GO:0005215 transporter activity;5—GO:0005488 binding;6—GO:0016209 antioxidant activity;7—GO:0060089 molecular transducer activity;8—GO:0098772 molecular function regulator;9—GO:0000003 reproduction;10—GO:0008152 metabolic process;11—GO:0009987 cellular process;12—GO:0023052 signaling;13—GO:0032502 developmental process;14—GO:0040011 locomotion;15—GO:0044699 single-organism process;16—GO:0048518 positive regulation of biological process;17—GO:0048519 negative regulation of biological process;18—GO:0050789 regulation of biological process;19—GO:0050896 response to stimulus;20—GO:0051179 localization;21—GO:0051704 multi-organism process;22—GO:0065007 biological regulation;23—GO:0071840 cellular component organization or biogenesis;24—GO:0005576 extracellular region;25—GO:0005623 cell;26—GO:0016020 membrane;27—GO:0019012 virion;28—GO:0031974 membrane-enclosed lumen;29—GO:0032991 macromolecular complex;30—GO:0043226 organelle;31—GO:0044422 organelle part;32—GO:0044423 virion part;33—GO:0044425 membrane part;34—GO:0044464 cell part.
图5 BC006基因组编码蛋白GO功能分类统计
Fig.5 Functional classification statistics of genome coding protein GO of BC006
2.5 副炭疽芽孢杆菌代表株的基因组共线性分析
基因组共线性分析显示,团头鲂源副炭疽芽孢杆菌BC006与牛源副炭疽芽孢杆菌1 710株共线性程度最高,亲缘最近(图6)。
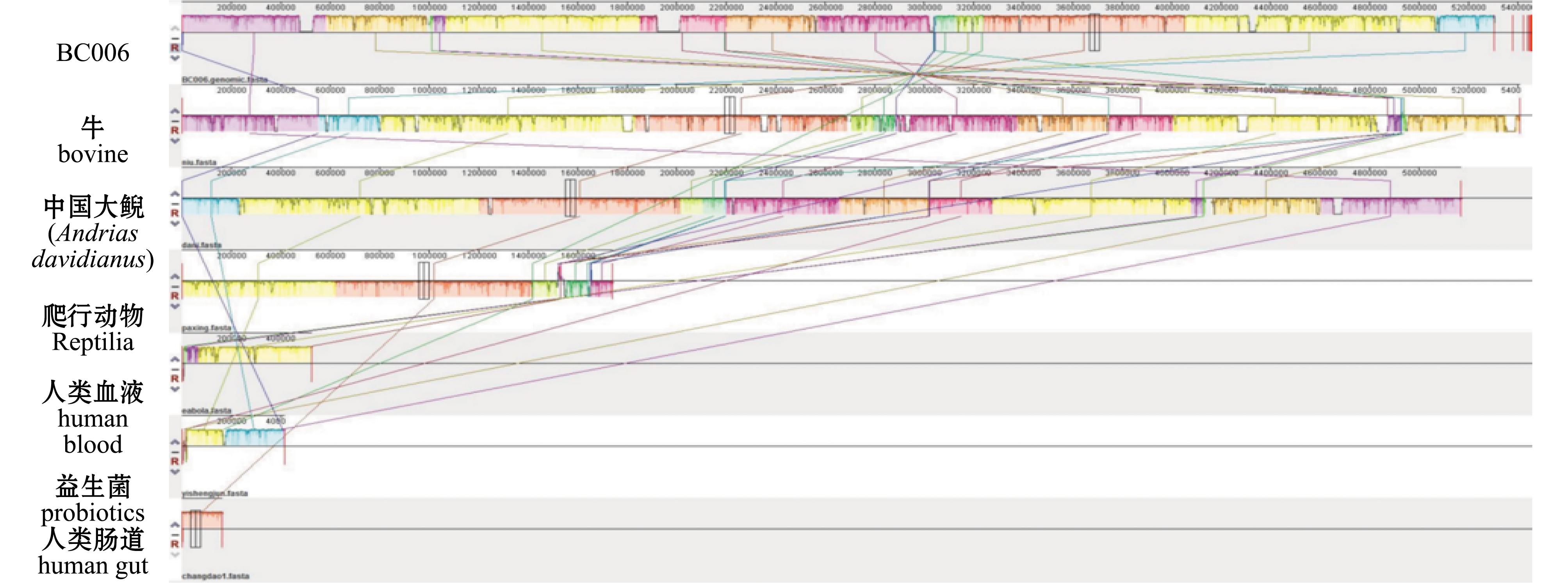
图6 BC006基因组的共线性分析
Fig.6 Collinearity analysis of the BC006 genome
2.6 毒力因子和致病表型分析
2.6.1 毒力基因 副炭疽芽孢杆菌BC006全基因组经毒力因子数据库(virulence factor database,VFDB)预测有6类毒力因子,其中命名明确的基因24个、待定基因19个(表2)。编码酶类的基因,包括编码免疫抑制剂A金属蛋白酶(immune inhibitor A metalloproteinase)的inhA,编码亲磷脂酰胆碱磷脂酶C(phosphatidylcholine-preferring phospholipase C,PC-PLC)的plcA,编码磷脂酰肌醇特异性磷脂酶C(phosphatidylinositol-specific phospholipase C,PI-PLC)的piplc、编码鞘磷脂酶(sphingomyelinase,SMase)的sph。编码铁获取因子基因,包括编码芽孢杆菌素(bacillibactin)的dhbA、dhbB、dhbC、dhbE和dhbF,编码Hal的hal,编码IlsA的ilsA。编码调节因子的基因,包括编码pagR-XO1的pagR-XO1(质粒上),编码plcR-PapR群体感应系统(plcR-PapR quorum sensing)的papR和plcR,编码virR/VirS(listeria)的virR。编码分泌系统的基因,包括编码Ⅶ型分泌系统(type Ⅶ secretion system)的essC和esxB。编码毒素的基因,包括编码溶血素Ⅰ(hemolysin Ⅰ)的alo,编码溶血素Ⅲ(hemolysin Ⅲ)的hlyⅢ,编码非溶血性肠毒素(non-hemolytic enterotoxin,Nhe)组分的nheA、nheB和nheC。
表2 BC006株的毒力基因
Tab.2 Virulence genes of BC006
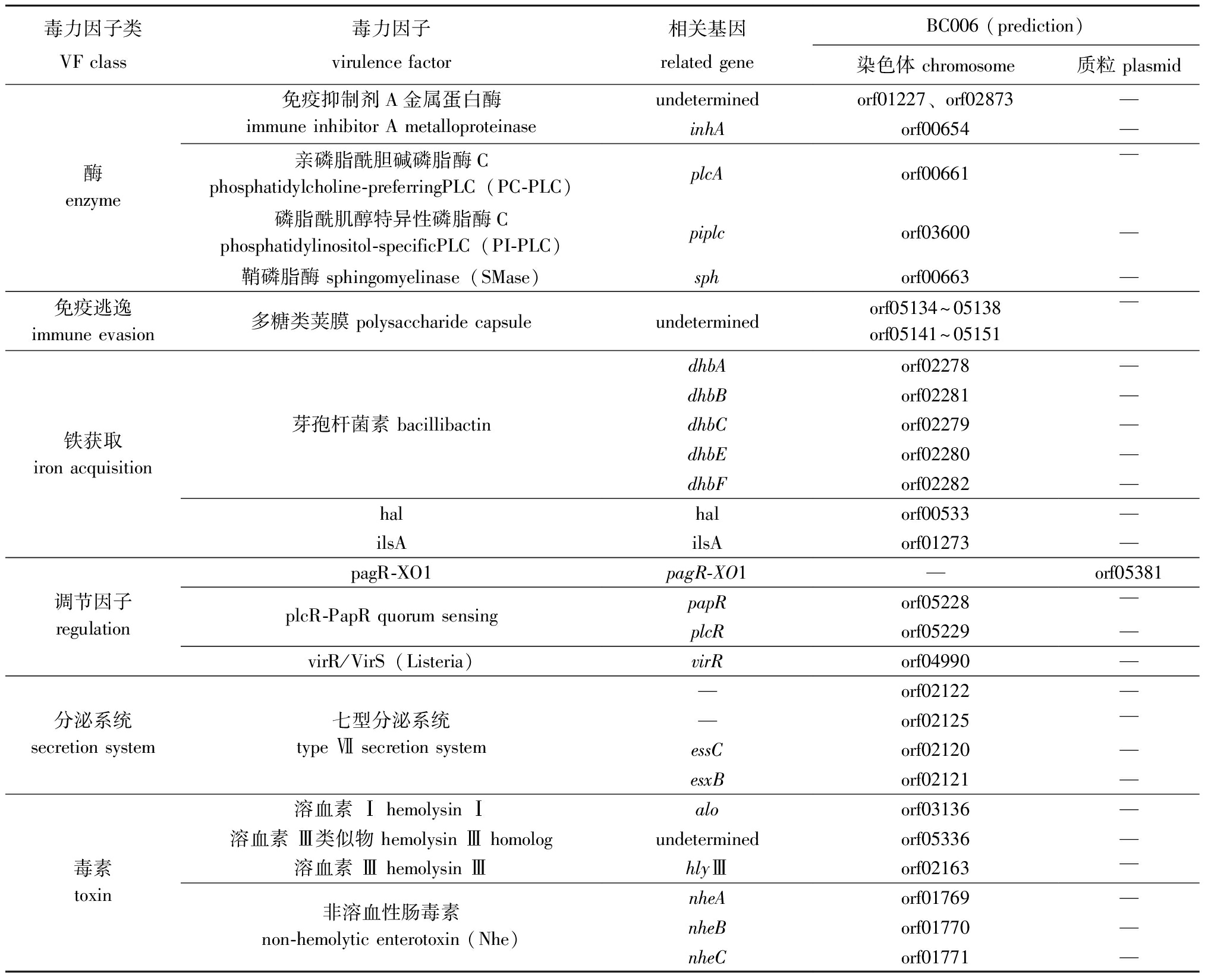
毒力因子类VF class毒力因子virulence factor相关基因related geneBC006 (prediction)染色体 chromosome质粒 plasmid免疫抑制剂A金属蛋白酶immune inhibitor A metalloproteinaseundeterminedorf01227、orf02873—inhAorf00654—酶enzyme亲磷脂酰胆碱磷脂酶Cphosphatidylcholine-preferringPLC(PC-PLC)plcAorf00661—磷脂酰肌醇特异性磷脂酶Cphosphatidylinositol-specificPLC(PI-PLC)piplcorf03600—鞘磷脂酶 sphingomyelinase(SMase)sphorf00663—免疫逃逸immune evasion多糖类荚膜 polysaccharide capsuleundeterminedorf05134~05138orf05141~05151—dhbAorf02278—dhbBorf02281—芽孢杆菌素 bacillibactindhbCorf02279—铁获取iron acquisitiondhbEorf02280—dhbForf02282—halhalorf00533—ilsAilsAorf01273—pagR-XO1pagR-XO1—orf05381调节因子regulationplcR-PapR quorum sensingpapRorf05228—plcRorf05229—virR/VirS(Listeria)virRorf04990——orf02122—分泌系统secretion system七型分泌系统type Ⅶ secretion system—orf02125—essCorf02120—esxBorf02121—溶血素 Ⅰ hemolysin Ⅰaloorf03136—溶血素 Ⅲ类似物 hemolysin Ⅲ homologundeterminedorf05336—毒素toxin溶血素 Ⅲ hemolysin ⅢhlyⅢorf02163—非溶血性肠毒素non-hemolytic enterotoxin (Nhe)nheAorf01769—nheBorf01770—nheCorf01771—
2.6.2 副炭疽芽孢杆菌BC006对团头鲂和小鼠的致死能力 副炭疽芽孢杆菌BC006腹腔攻毒对团头鲂的LD50为4.68×105 CFU/g(图7),对小鼠的LD50为4.22×105 CFU/g(图8)。攻毒鱼腹壁灰褐色、鳞片脱落,鳃盖剐蹭,肛门红肿,腹腔积液;攻毒小鼠呈现眯眼、身体颤抖,腹腔积液,肠道胀气的特征(图9)。
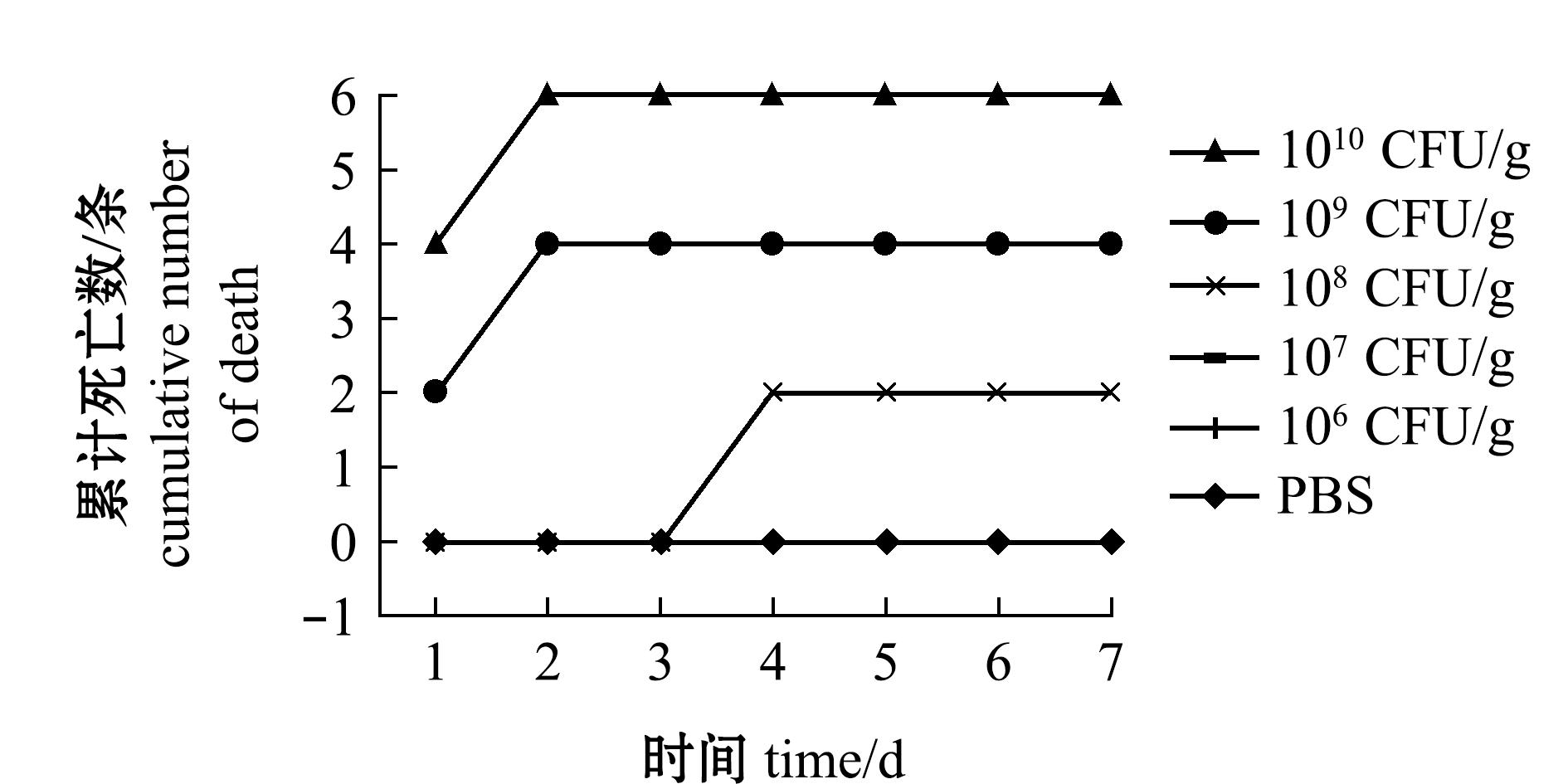
图7 BC006株攻毒团头鲂的剂量死亡曲线
Fig.7 Dose mortality curve of Megalobramaamblycephala challenged with BC006
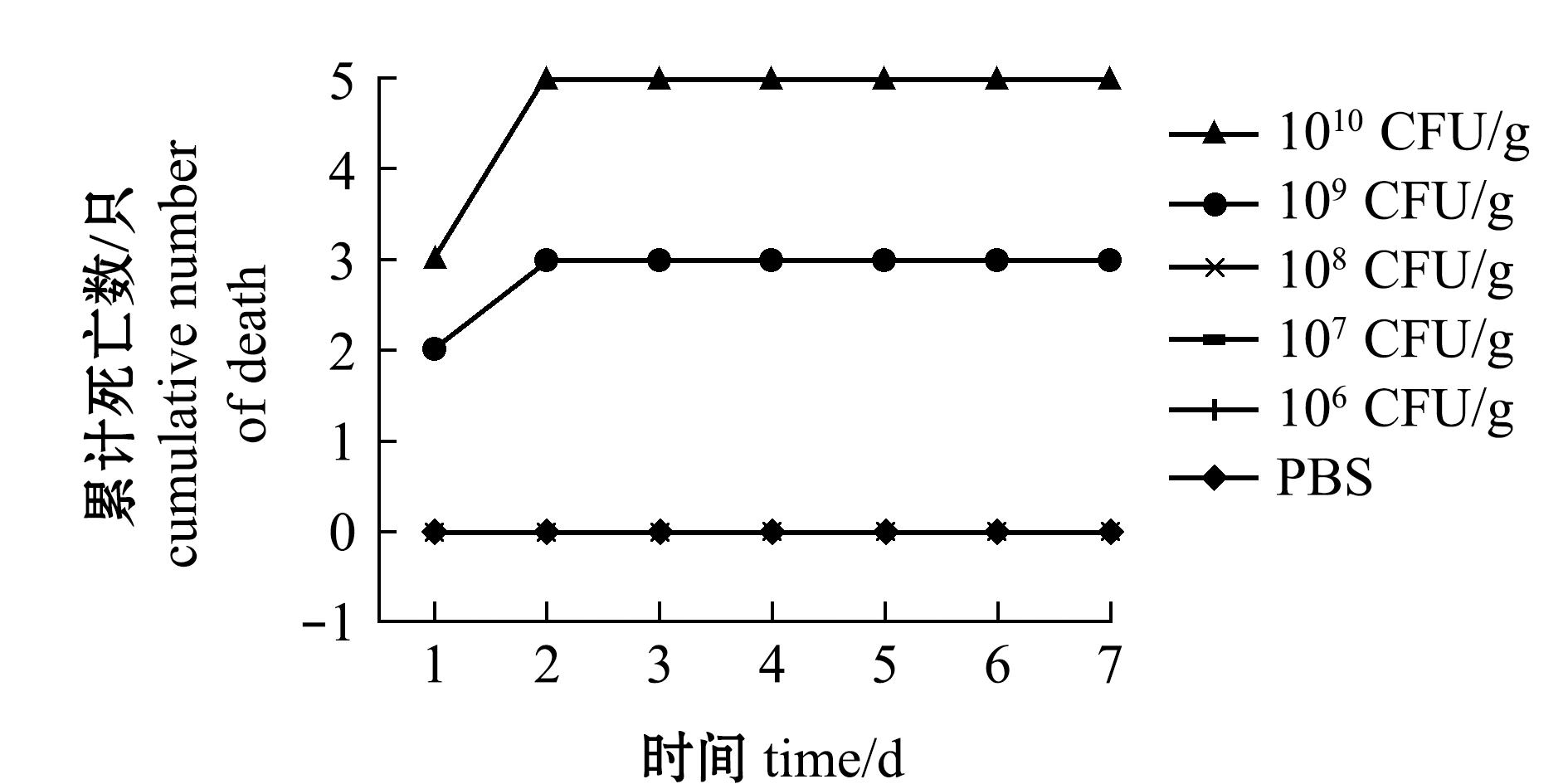
图8 BC006株攻毒小鼠的剂量死亡曲线
Fig.8 Dose mortality curve mice challenged with BC006
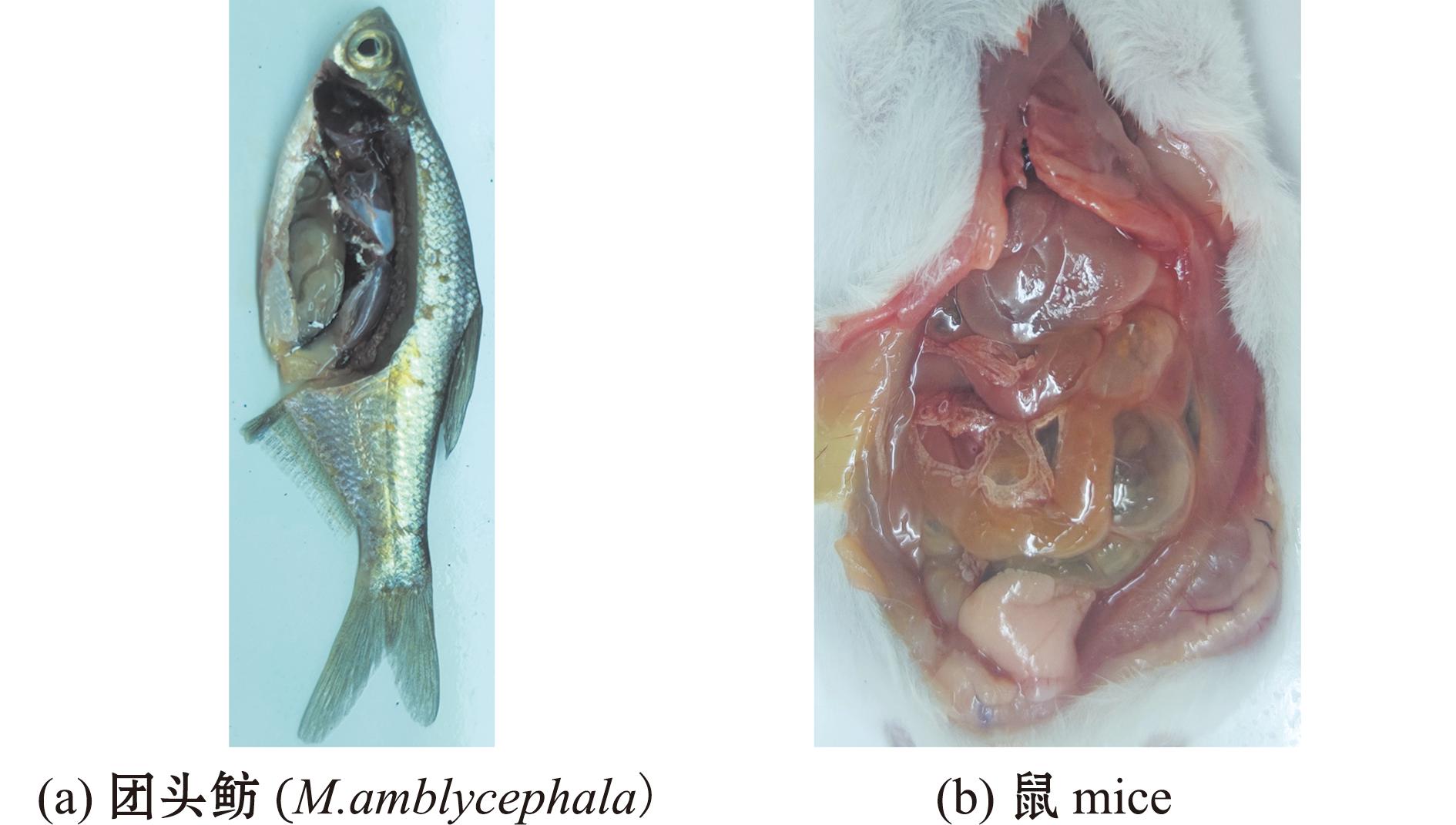
图9 腹腔注射BC006引起团头鲂和小鼠的剖检变化
Fig.9 Anatomical changes of Megalobramaamblycephala and mice injected with BC006 intraperitoneally
2.7 耐药基因与耐药表型
2.7.1 耐药基因型 副炭疽芽孢杆菌BC006全基因组经抗生素耐药性数据库(comprehensive antibiotic resistance database,CARD)预测到16个耐药基因,均分布在染色体上,涉及4种耐药机制:抗生素靶点改变、抗生素外排、抗生素灭活和抗生素靶点保护(表3)。检出抗生素靶点改变基因7个,均属糖肽抗性基因簇(glycopeptide resistance gene cluster),分别属于vanG基因簇中的vanT(2个类似物),vanI基因簇中的vanW(2个类似物),vanM基因簇中的vanY,vanB基因簇中的vanY及vanF基因簇中的vanY。检出抗生素外排基因3个,tet(45)与fexA属于主要易化超家族 (major facilitator superfamily,MFS),qacJ属于小多药耐药家族(small multidrug resistance,SMR)。检出抗生素灭活基因4个,Bc Ⅰ和Bc Ⅱ属于蜡样芽孢杆菌Bc β内酰胺酶(Bacillus cereus Bc β-lactamase),FosB属于磷霉素硫醇转移酶(fosfomycin thiol transferase),mphL属于大环内酯磷酸转移酶(macrolide phosphotransferase,MPH)。检出抗生素靶点保护基因1个,tetB(P)属于四环素抗性核糖体保护蛋白(tetracycline-resistant ribosomal protection protein)。
表3 BC006株的耐药基因
Tab.3 Drug resistance genes of BC006

耐药机制resistance mechanism耐药基因drug resistance gene抗生素分类classification of antibiotics抗生素靶向性改变antibiotic target alterationVanT、VanW、VanY 糖肽类glycopeptide抗生素外排antibiotic effluxtet(45)、fexA、qacJ消毒防腐剂disinfectant and antiseptics、四环素类tetracycline、酰胺醇类phenicol抗生素灭活antibiotic inactivationBcⅠ、BcⅡ、FosB 、mphL大环内酯类macrolide、膦酸类phosphonic acid、头孢菌素类cephalosporin、青霉烷类penem抗生素靶向保护antibiotic target protectiontetB(P)四环素类抗生素tetracycline
2.7.2 耐药性表型 KB药敏结果显示,副炭疽芽孢杆菌BC006对大环内酯类的红霉素,林可酰胺类的林可霉素,硝基咪唑类的甲硝唑,四环素类的多西环素与四环素,头孢菌素类的头孢噻肟,青霉素类的青霉素G,二氨基嘧啶类的甲氧嘧啶,氨基糖苷类的卡那霉素与丁胺卡那,利福霉素类的利福平,喹诺酮类的诺氟沙星、恩诺沙星、氧氟沙星与环丙沙星,磺胺类的复方新诺明等16种抗生素耐药(表4)。
表4 基于KB法的BC006对抗生素的敏感性
Tab.4 Sensitivity of BC006 to antibiotics based on KB method
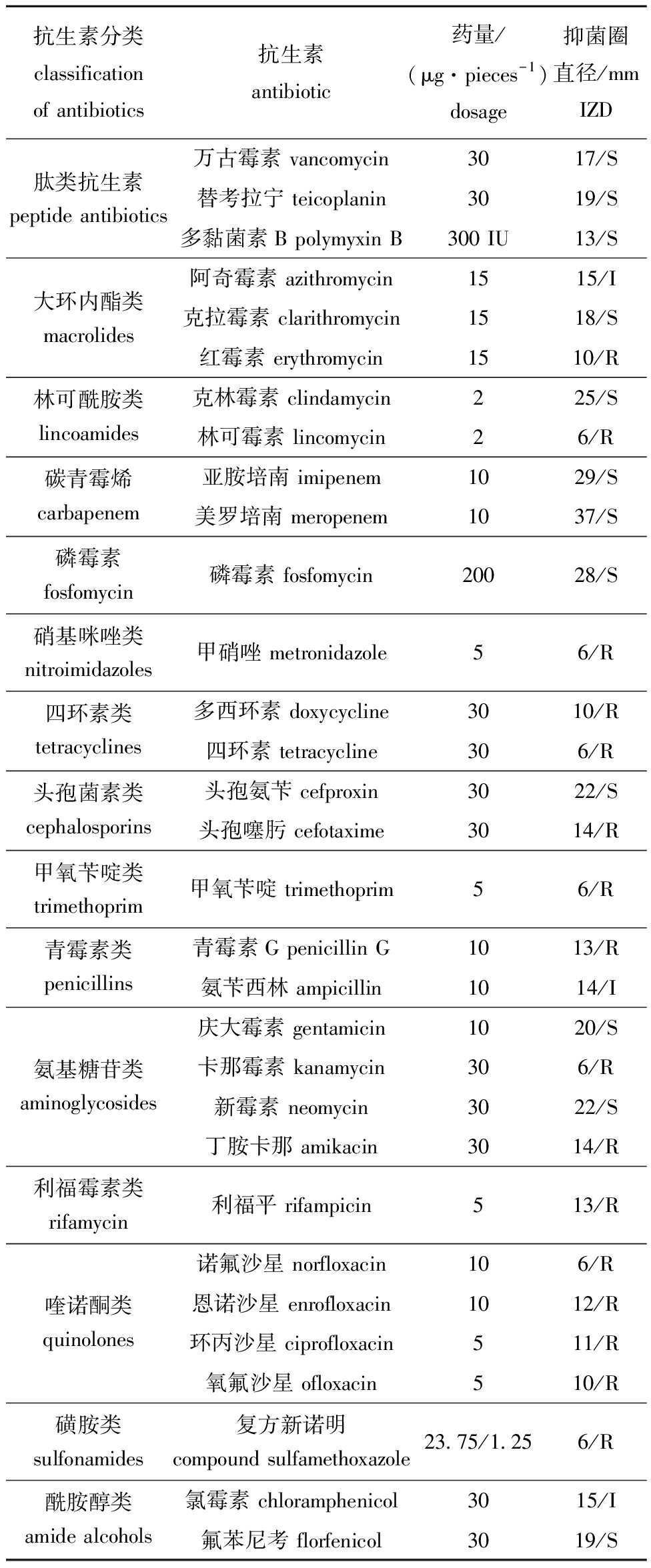
抗生素分类classification of antibiotics抗生素antibiotic药量/(μg·pieces-1)dosage抑菌圈直径/mmIZD万古霉素 vancomycin3017/S肽类抗生素peptide antibiotics替考拉宁 teicoplanin3019/S多黏菌素B polymyxin B300 IU13/S阿奇霉素 azithromycin1515/I大环内酯类macrolides克拉霉素 clarithromycin1518/S红霉素 erythromycin1510/R林可酰胺类lincoamides克林霉素 clindamycin225/S林可霉素 lincomycin26/R碳青霉烯carbapenem亚胺培南 imipenem1029/S美罗培南 meropenem1037/S磷霉素fosfomycin磷霉素 fosfomycin20028/S硝基咪唑类nitroimidazoles甲硝唑 metronidazole56/R四环素类tetracyclines多西环素 doxycycline3010/R四环素 tetracycline306/R头孢菌素类cephalosporins头孢氨苄 cefproxin3022/S头孢噻肟 cefotaxime3014/R甲氧苄啶类trimethoprim甲氧苄啶 trimethoprim56/R青霉素类penicillins青霉素G penicillin G1013/R氨苄西林 ampicillin1014/I庆大霉素 gentamicin1020/S氨基糖苷类aminoglycosides卡那霉素 kanamycin306/R新霉素 neomycin3022/S丁胺卡那 amikacin3014/R利福霉素类rifamycin利福平 rifampicin513/R诺氟沙星 norfloxacin106/R喹诺酮类quinolones恩诺沙星 enrofloxacin1012/R环丙沙星 ciprofloxacin511/R氧氟沙星 ofloxacin510/R磺胺类sulfonamides复方新诺明 compound sulfamethoxazole23.75/1.256/R酰胺醇类amide alcohols氯霉素 chloramphenicol3015/I氟苯尼考 florfenicol3019/S
2.7.3 耐药表型与耐药基因型的比较 BC006株对糖肽类抗生素万古霉素(17 mm/S)和替考拉宁(19 mm/S)敏感;虽然在其基因组上检出糖肽类抗性基因VanT、VanW、VanY,但区域匹配度太低(28.83%~56.01%)。BC006株对磷霉素类抗生素磷霉素(28 mm/S)敏感,但在其全基因组上检出磷霉素类抗性基因类似物磷霉素硫醇转移酶基因家族的FosB(区域匹配度90.58%)。BC006株对酰胺醇类抗生素氯霉素(15 mm/I)与氟苯尼考(19 mm/S)未表现耐药性;但在其中检测出酰胺醇类抗性基因类似物主要易化蛋白超家族fexA基因(区域匹配度99.58%)。
BC006株对四环素类抗生素中的四环素(6 mm/R)和多西环素(10 mm/R)耐药。BC006基因组中携带有抗生素外排泵中的主要易化超家族成员tet(45)(区域匹配度88.62%)和四环素抗性核糖体保护蛋白中的tetB(P)(区域匹配度45.06%),均参与对四环素类的抗性。
BC006株对硝基咪唑类抗生素甲硝唑(6 mm/R),二氨基嘧啶类抗生素甲氧苄啶(6 mm/R),利福平类抗生素利福平(13 mm/R),喹诺酮类抗生素诺氟沙星(6 mm/R)、恩诺沙星(12 mm/R)、环丙沙星(11 mm/R)、氧氟沙星(10 mm/R),磺胺类复方新诺明(6 mm/R)耐药,未检测到相应的特异性耐药基因。BC006株对林可酰胺类的克林霉素(25 mm/S)敏感,对林可霉素(6 mm/R)耐药;对氨基糖苷类的庆大霉素(20 mm/S)和新霉素(22 mm/S)敏感,对卡那霉素(6 mm/R)和丁胺卡那(14 mm/R)耐药;但均未检出特异性的耐药基因。BC006中检测到的小多药耐药性(SMR)抗生素外排泵中的qacJ (两个类似物,区域匹配度39.42%和38.32%),参与多种抗生素耐药。
BC006株对大环内酯类的阿奇霉素(15 mm/I)、克拉霉素(18 mm/S)未呈现耐药性,但对红霉素(10 mm/R)耐药;其携带的大环内酯类磷酸转移酶基因家族的mphL(区域匹配度90.58%)参与对红霉素的耐药。
BC006株对碳青霉烯类抗生素亚胺培南(29 mm/S)和美罗培南(37 mm/S)敏感,对头孢菌素类抗生素头孢氨苄(22 mm/S)敏感、对头孢噻肟(14 mm/R)耐药,对青霉素类的氨苄西林(14 mm/I)中介、对青霉素G(13 mm/R)耐药;其携带的头孢菌素和青霉烯类抗性基因类似物Bc β内酰胺酶家族的Bc Ⅰ与Bc Ⅱ,区域匹配度分别为90.85%和98.44%,参与对头孢噻肟和青霉素G的耐药。
3 讨论
3.1 基因组共线性分析与菌株来源
根据不同副炭疽芽孢杆菌菌株的基因组共线性分析,团头鲂源副炭疽芽孢杆菌BC006株与牛源副炭疽芽孢杆菌1710株共线性最高,即二者在进化上亲缘关系最近。且BC006株与1710株携带了一些相同的毒力基因(如nheA、nheB、nheC,inhA,alo)和耐药基因(如fosB和tetB)[16]。但非常有趣的是,1710株分离自俄罗斯的患乳房炎奶牛,然而在中国安徽省池塘养殖的患病团头鲂内脏中分离到1710株近缘的BC006株,是否与死淘奶牛制造的肉骨粉将副炭疽芽孢杆菌的芽孢带进鱼类饲料原料有关,值得进一步关注和研究。
3.2 毒力因子与致病特性分析
BC006株基因组中存在溶血素 Ⅰ编码基因alo,其编码产物为炭疽溶素O(anthrolysin O,ALO),是含有4个结构域的单肽,属于热稳定性的胆固醇依赖性溶细胞素(CDC)家族。ALO第4个结构域的碳端包含高度保守的11个残基(ECTGLAWEWWR),与富含胆固醇的细胞膜结合,对红细胞和肠上皮细胞的孔形成毒性[19]。溶血毒素Ⅲ(hly Ⅲ)是不耐热的单肽,其溶血活性不受胆固醇抑制,其表达也不受PlcR调控。hly Ⅲ与红细胞表面结合后,hly Ⅲ单体组装形成跨膜孔,最终导致红细胞裂解[20]。
非溶血性肠毒素(non-hemolytic enterotoxin,Nhe)可引起腹泻相关病变(肠炎、肠胀气)和红细胞溶血[20]。Nhe也属于孔形成毒素[21],包含NheA、B和C 3种组分,以独立方式分泌到细胞外,按C、B和A的顺序依次结合到细胞膜上组成穿孔毒素,导致多种上皮细胞和红细胞裂解[22]。NheB和NheC能增强NheA的生物活性。Nhe在细菌生长的指数期产生和分泌,在肠道环境中更容易分泌,并受主要转录调节因子PlcR的调节[23]。
磷脂酶是蜡样芽孢杆菌群的重要毒力因子,包括鞘磷脂酶(SMase)、磷脂酰肌醇特异性磷脂酶C(PI-PLC)、磷脂酰胆碱特异性磷脂酶C(PC-PLC)。磷脂酶可以促进其他毒素的溶血活性。SMase在动物致死中发挥重要作用,阻断SMase活性后,蜡样芽孢杆菌对小鼠的致死能力消失。SMase引起宿主细胞膜损伤,进而引起巨噬细胞功能失调,影响细菌定植,抑制宿主趋化因子生成,干扰营养获得(如铁获取)和溶血素活性[23]。SMase与Nhe协同可引起肠上皮损伤和肠渗漏[24]。PI-PLC与d-肌醇-(α1→6) 氨基葡萄糖形成复合体,促进膜结合糖磷脂酰肌醇(glycolphosphatidylinositol,GPI)锚定蛋白的切割,造成细胞损伤[23]。
金属蛋白酶,主要参与菌株逃逸宿主免疫监测,可以降解宿主的蛋白酶抑制剂、细胞外基质蛋白和上皮屏障蛋白[20]。副炭疽芽孢杆菌的免疫抑制剂A金属蛋白酶(InhA)存在3种同源蛋白,InhA1、 InhA2和InhA3。InhA1为锌依赖性金属蛋白酶,具有锌结合基序和金属蛋白酶常见活性催化中的氨基酸残基(HEXXH)。InhA在细菌感染中发挥核心毒力作用,金属蛋白酶与孔形成毒素协作,通过与宿主蛋白互作促进细菌在宿主体内的增殖,为细菌提供营养、干扰宿主免疫、破坏保护性内皮细胞和上皮屏障[23]。BC006中检测到3个InhA的类似物,其结构和功能还需要进一步研究。
plcR/papR系统是蜡样芽孢杆菌群中比较特异的群体感应系统,其是静止期毒力基因中心转录调节因子。papR信号分子与调控因子plcR 结合,形成四聚体后结合到plcR-box 位点并激活其调控功能;参与调控毒素(如PLC和SMase)、芽孢杆菌素和酶等的表达。papR/plcR还参与细菌生物膜形成[23]。
芽孢杆菌素(bacillibactin)属于儿茶酚型嗜铁素(siderophore,也称为铁载体)其结构特征是以多个2、3-二羟基苯甲酸(dihydroxybenzoate,DHB)相邻的2个羟基作为螯合基团,羧基部分连接不同氨基酸,形成不同结构的儿茶酚型嗜铁素。芽孢杆菌基因组中普遍含有DHB合成所需要的基因dhbA/B/C/E/F,其中dhbC对芽孢杆菌素合成有决定性的调控作用[25]。
3.3 耐药基因型与表型分析
糖肽类抗生素被认为是抵抗革兰氏阳性菌的最后一道防线,但其耐药菌株的报道有增加趋势。糖肽类抗生素耐药细菌能合成突变的肽聚糖前体(其末端为D-Ala-D-Lac或D-Ala-D-Ser),而不是正常的野生型D-Ala-D-Ala。万古霉素和替考拉宁不能和这些突变前体结合,因此细菌产生对万古霉素和替考拉宁的耐药性。以D-Ala-D-Lac结尾的前体由vanA、B、D、F、I和M基因簇编码,而D-Ala-D-Ser结尾的前体由vanC、E、G、L和N基因簇编码。BC006株中虽然检测到了7个抗性基因(2个vanT、2个vanW和3个vanY)类似物,涉及5个基因簇家族(vanG、vanI、vanM、vanB和vanF),但不能构成完整基因簇,无法编码合成突变的肽聚糖前体,这是BC006对糖肽类抗生素万古霉素和替考拉宁呈现敏感的主要原因[26]。虽然BC006上检测到高区域匹配度的FosB和fexA,但未表现出对磷霉素和氟苯尼考的耐药性,此种情况还有待进一步研究。
细菌外排系统有5个主要家族,包括主要易化超家族(MFS)、ATP结合盒(ABC)超家族、小多药耐药性(SMR)家族、耐药结节分化(RND)家族和多药与毒物外排(MATE)家族;其中 ABC 类外排泵是利用 ATP水解释放的能量将抗生素从胞内排出,剩余4类外排泵均是利用质子进入膜内形成的浓度差将抗生素排至胞外[27]。小多药耐药家族(small multidrug resistance,SMR)是广泛分布的一类微生物膜蛋白,属于抗生素外排系统[27],qacJ基因是SMR家族中的药物与消毒剂外运子(drug and antiseptic exporters,Qac)亚类的重要成员[28]。SMR抗生素外排泵,参与细菌对多种抗生素的耐药表型,且赋予细菌对多种抗生素的低水平耐药性,涉及BC006对硝基咪唑类、二氨基嘧啶类、利福平类、喹诺酮类和磺胺类的耐药性[28]。主要易化超家族(MFS)成员的tet (45)基因是引起四环素类耐药性的专一抗生素外排泵[29]。从动物源微生态制剂分离的益生蜡样芽孢杆菌9i株携带tet (45)基因变体,位于可移动遗传元件上,通过外排作用介导对四环素类的耐药性[30]。
抗生素灭活是指细菌通过分泌特异性酶来分解某类抗生素而实现细菌对该抗生素的耐药[31]。根据不同底物谱可将β-内酰胺酶分为青霉素酶、头孢菌素酶和碳青霉烯酶[32]。Bc Ⅰ为蜡样芽孢杆菌A群产的Bcβ-内酰胺酶,Bc Ⅱ为 蜡样芽孢杆菌B1亚群产的Bcβ-内酰胺酶[33],二者均能裂解青霉素类基本结构6氨基青霉烷酸(6-APA)和头孢菌素类基本结构7-氨基头孢霉烷酸( 7-ACA),以实现对青霉素类和头孢菌素类的耐药[34]。
4 结论
1)通过保守基因16S rDNA、gyrB、rpoB与tuf的系统发育树构建、全基因组测序和平均核苷酸相似度比较,鉴定BC006株为副炭疽芽孢杆菌。
2)通过耐药表型与耐药基因分析,副炭疽芽孢杆菌BC006株为多重耐药菌,通过致病力与毒力基因分析,副炭疽芽孢杆菌BC006株对团头鲂和小鼠有致死性,提示副炭疽芽孢杆菌BC006株的多重耐药性和致病性对鱼类和哺乳类健康具有潜在威胁。
[1] CARROLL L M,CHENG R A,WIEDMANN M,et al.Keeping up with the Bacillus cereus group:taxonomy through the genomics era and beyond[J].Critical Reviews in Food Science and Nutrition,2022,62(28):7677-7702.
[2] SORNCHUER P,SANINJUK K,AMONYINGCHAROEN S,et al.Whole genome sequencing reveals antimicrobial resistance and virulence genes of both pathogenic and non-pathogenic B.cereus group isolates from foodstuffs in Thailand[J].Antibiotics,2024,13(3):245.
[3] 吴振超,陈玉珂,高永生,等.一株黄金鲫鱼源蜡状芽孢杆菌的筛选、鉴定及耐性研究[J].大连海洋大学学报,2020,35(6):883-892.WU Z C,CHEN Y K,GAO Y S,et al.Isolation,identification and probiotic properties of a Bacillus cereus strain from golden crucian carp[J].Journal of Dalian Ocean University,2020,35(6):883-892.(in Chinese)
[4] LIU Y,DU J,LAI Q L,et al.Proposal of nine novel species of the Bacillus cereus group[J].International Journal of Systematic and Evolutionary Microbiology,2017,67(8):2499-2508.
[5] BUKHARIN O V,PERUNOVA N B,ANDRYUSCHENKO S V,et al.Genome sequence announcement of Bacillus paranthracis strain ICIS-279,isolated from human intestine[J].Microbiology Resource Announcements,2019,8(44):e00662-e00619.
[6] MATSON M J,ANZICK S L,FELDMANN F,et al.Bacillus paranthracis isolate from blood of fatal Ebola virus disease case[J].Pathogens,2020,9(6):475.
[7] DE SOUSA L P.Genomic and pathogenicity of a Bacillus paranthracis isolated from book page surface[J].Infection,Genetics and Evolution:Journal of Molecular Epidemiology and Evolutionary Genetics in Infectious Diseases,2021,92:104867.
[8] FUKUDA D,NOLASCO-HIPOLITO C.Draft genome sequence of Bacillus paranthracis strain DB-4,isolated from nukadoko,fermented rice bran for Japanese pickles[J].Microbiology Resource Announcements,2021,10(40):e0070521.
[9] ABDELSAMAD N O,ESAWY M A,MAHMOUD Z E,et al.Evaluation of different bacterial honey isolates as probiotics and their efficient roles in cholesterol reduction[J].World Journal of Microbiology &Biotechnology,2022,38(6):106.
[10] TSHISIKHAWE M L,DIALE M O,ABRAHAMS A M,et al.Screening and production of industrially relevant enzymes by Bacillus paranthracis strain MHDS3,a potential probiotic[J].Fermentation,2023,9(11):938.
[11] CHUKWUMA O B,RAFATULLAH M,KAPOOR R T,et al.Isolation and characterization of lignocellulolytic bacteria from municipal solid waste landfill for identification of potential hydrolytic enzyme[J].Fermentation,2023,9(3):298.
[12] CHI Y W,YOU Y M,WANG J C,et al.Two plant growth-promoting bacterial Bacillus strains possess different mechanisms in affecting cadmium uptake and detoxification of Solanum nigrum L[J].Chemosphere,2022,305:135488.
[13] BAEV V,ILIEV I,STEFANOV Y,et al.Exploring the genomic landscape of Bacillus paranthracis PUMB17 as a proficient phosphatidylcholine-specific phospholipase C producer[J].Current Issues in Molecular Biology,2024,46(3):2497-2513.
[14] CARROLL L M,WIEDMANN M,MUKHERJEE M,et al.Characterization of emetic and diarrheal Bacillus cereus strains from a 2016 foodborne outbreak using whole-genome sequencing:addressing the microbiological,epidemiological,and bioinformatic challenges[J].Frontiers in Microbiology,2019,10:144.
[15] 袁璐,张晓妍,吕宇飞,等.一株自中国大鲵的副炭疽芽孢杆菌分离和鉴定[J].微生物学报,2022,62(9):3613-3630.YUAN L,ZHANG X Y,LÜ Y F,et al.Isolation and identification of a Bacillus paranthracis strain from Chinese giant salamander[J].Acta Microbiologica Sinica,2022,62(9):3613-3630.(in Chinese)
[16] SOKOLOV S,BROVKO F,SOLONIN A,et al.Genomic analysis and assessment of pathogenic (toxicogenic) potential of Staphylococcus haemolyticus and Bacillus paranthracis consortia isolated from bovine mastitis in Russia[J].Scientific Reports,2023,13:18646.
[17] 张玲艳,宋丽丽,贾伟娟,等.蜡样芽孢杆菌检测方法的研究进展[J].微生物学通报,2021,48(4):1360-1372.ZHANG L Y,SONG L L,JIA W J,et al.Research progress on detection methods of Bacillus cereus[J].Microbiology China,2021,48(4):1360-1372.(in Chinese)
[18] YOON S H,HA S M,LIM J,et al.A large-scale evaluation of algorithms to calculate average nucleotide identity[J].Antonie Van Leeuwenhoek,2017,110(10):1281-1286.
[19] JOHNSON K A,ENDAPALLY S,VAZQUEZ D C,et al.Ostreolysin A and anthrolysin O use different mechanisms to control movement of cholesterol from the plasma membrane to the endoplasmic reticulum[J].The Journal of Biological Chemistry,2019,294(46):17289-17300.
[20] RAMARAO N,SANCHIS V.The pore-forming haemolysins of Bacillus cereus:a review[J].Toxins,2013,5(6):1119-1139.
[21] 鲁爽.两株细菌的比较鉴定及非溶血性肠毒素基因的表达[D].镇江:江苏大学,2017.LU S.Comparative identification of two bacteria and expression of non-hemolytic enterotoxin genes[D].Zhenjiang:Jiangsu University,2017.(in Chinese)
[22] LINDB CK T,HARDY S P,DIETRICH R,et al.Cytotoxicity of the Bacillus cereus Nhe enterotoxin requires specific binding order of its three exoprotein components[J].Infection and Immunity,2010,78(9):3813-3821.
CK T,HARDY S P,DIETRICH R,et al.Cytotoxicity of the Bacillus cereus Nhe enterotoxin requires specific binding order of its three exoprotein components[J].Infection and Immunity,2010,78(9):3813-3821.
[23] ENOSI TUIPULOTU D,MATHUR A,NGO C,et al.Bacillus cereus:epidemiology,virulence factors,and host-pathogen interactions[J].Trends in Microbiology,2021,29(5):458-471.
[24] DOLL V M,EHLING-SCHULZ M,VOGELMANN R.Concerted action of sphingomyelinase and non-hemolytic enterotoxin in pathogenic Bacillus cereus[J].PLoS One,2013,8(4):e61404.
[25] 刘邮洲,沈佳慧,乔俊卿,等.芽孢杆菌嗜铁素研究进展[J].江苏农业学报,2023,39(1):266-276.LIU Y Z,SHEN J H,QIAO J Q,et al.Research progress of siderophore produced by Bacillus spp[J].Jiangsu Journal of Agricultural Sciences,2023,39(1):266-276.(in Chinese)
[26] AQIB A I,ALSAYEQH A F.Vancomycin drug resistance,an emerging threat to animal and public health[J].Frontiers in Veterinary Science,2022,9:1010728.
[27] DE GAETANO G V,LENTINI G,FAM A,et al.Antimicrobial resistance:two-component regulatory systems and multidrug efflux pumps[J].Antibiotics,2023,12(6):965.
A,et al.Antimicrobial resistance:two-component regulatory systems and multidrug efflux pumps[J].Antibiotics,2023,12(6):965.
[28] BURATA O E,YEH T J,MACDONALD C B,et al.Still rocking in the structural era:a molecular overview of the small multidrug resistance (SMR) transporter family[J].The Journal of Biological Chemistry,2022,298(10):102482.
[29] LI T,WANG Z L,GUO J H,et al.Bacterial resistance to antibacterial agents:mechanisms,control strategies,and implications for global health[J].The Science of the Total Environment,2023,860:160461.
[30] 崔一芳,徐福洲,丁双阳,等.益生蜡样芽孢杆菌可转移四环素耐药基因tet(45)特征分析[J].科学通报,2020,65(32):3619-3625.CUI Y F,XU F Z,DING S Y,et al.Characteristics of a transferable Tet(45) gene conferring resistance to tetracyclines in probiotic Bacillus cereus[J].Chinese Science Bulletin,2020,65(32):3619-3625.(in Chinese)
[31] ROLFF J,BONHOEFFER S,KLOFT C,et al. Forecasting antimicrobial resistance evolution [J].Trends in Microbiology,2024,32 (8):736-745.
[32] BUSH K. Classification for β-lactamases: historical perspectives[J].Expert Review of Anti-infective Therapy,2023,21(5): 513-522.
[33] 李玲,刘耀,魏元苗,等. 食品中蜡样芽孢杆菌耐药性及其机制研究进展[J].微生物学通报,2021,48(12):4943-4953.LI L,LIU Y,WEI Y M,et al. Research progress on antibiotic resistance of Bacillus cereus in the food chain[J].Microbiology China,2021,48(12):4943-4953. (in Chinese)
[34] BUSH K.Past and present perspectives on β-lactamases[J].Antimicrobial Agents and Chemotherapy,2018,62(10):e01076-e01018.