近年来,高通量测序与分子育种技术的迅猛发展及众多水产物种高质量基因组的完成,为水产物种全基因组层面的性状解析和育种值预测提供了可能。中国的水产育种正在从选择育种、杂交育种和倍性操控等传统的育种技术,向现代分子育种技术迅速发展[1]。自2012年完成牡蛎(Crassostrea gigas)基因组测序以来[2],已完成了半滑舌鳎(Cynoglossus semilaevis)[3]、鲤(Cyprinus carpio)[4]、大黄鱼(Larimichthys crocea)[5]、虹鳟(Oncorhynchus mykiss)[6]和大西洋鲑(Salmo salar)[7]等50多种水产养殖生物的全基因组精细图谱绘制。随着全基因组选择(genomic selection,GS)分析方法的不断完善和拓展,水产生物全基因选择技术突破了瓶颈,并在大西洋鲑[8-9]、虾夷扇贝(Patinopecten yessoensis)[10]、大黄鱼[11-13]、牙鲆 (Paralichthys olivaceus)[14-15]和半滑舌鳎[16]等多个物种中取得了良好应用,培育出栉孔扇贝“蓬莱红2号”、牙鲆“鲆优2号”、罗非鱼“壮罗1号”和半滑舌鳎“鳎优1号”等高产抗病新品种[17]。
全基因组选择[18]是一种利用覆盖全基因组的高密度分子标记进行选择育种的新方法,可通过构建预测模型,根据基因组估计育种值进行早期个体的预测和选择,从而缩短世代间隔,加快育种进程,并节约大量成本。统计模型是全基因组选择的核心,极大地影响着基因组预测的准确性和效率。统计模型可分为以全基因组最佳线性无偏预测法(genomic best linear unbiased prediction,GBLUP)为代表的直接法[19]、以贝叶斯(Bayes)为代表的间接法及最近快速发展的机器学习法。GBLUP的实现只需要构建G矩阵,运算速度较快,可减少孟德尔抽样造成的偏差。不同Bayes方法具有不同的先验分布,产生不同程度的收缩,其准确性一般略高于GBLUP,但运算速度较慢。机器学习方法无太多条件限制,适用性广,但部分模型的稳健性较差,预测准确性往往受性状类型的影响。目前,水产物种新品种培育仍以传统的群体选育为主,部分品种培育过程中采用了全基因组选择方法,主要选用的是GBLUP和Bayes模型,使用机器学习模型来做全基因组选择的研究较少。
鲤是世界上淡水鱼类中养殖范围最广的物种之一,目前在100多个国家和地区均有养殖[4]。鲤在中国的养殖驯化历史可以追溯到8 000年前[20],在欧洲也有较长的养殖历史[21]。中国是世界上最大的鲤养殖和消费国,2021年鲤养殖产量约283万t,在水产养殖中占有重要的地位。黄河鲤具有金鳞赤尾、体型梭长和肉质细嫩等特点,“豫选黄河鲤”新品种于2005年通过全国水产原种和良种审定委员会审定后,在黄河流域迅速推广,形成了年产数十万吨的养殖规模,在沿黄河地区渔民增收、产业增效和优质水产品供给等方面贡献显著[22]。然而,由于长期自繁自养造成种质退化、生长变慢等问题,严重影响了其养殖效益。因此,培育生长快速的黄河鲤水产新品种成为产业发展的重要需求。
本研究中,基于黄河鲤的体质量性状评估全基因组选择模型预测的准确性和稳定性,不仅能为今后开展黄河鲤新品种选育提供重要数据依据,也为在更多水产物种中开展全基因组选择模型的优化提供了重要的参考技术路径。
1 材料与方法
1.1 材料
试验用黄河鲤采自河南省水产科学研究院黄河鲤养殖基地,随机采集619尾2龄黄河鲤并记录体质量性状数据。
1.2 方法
1.2.1 样本采集 所有样本取样前先浸泡在MS222溶液中进行麻醉,对每一个样本采集鱼背部肌肉不少于10 g,置于-80 ℃冰箱中保存备用。
1.2.2 基因分型和数据质量控制 取冷藏的鲤背部肌肉用于基因组DNA提取,将样本送到公司,采用鲤250K高通量SNP芯片[23]对同一试验条件下的黄河鲤进行基因分型,使用PLINK 1.90软件[24]对分型结果进行质量控制。
质量控制标准:1)SNP基因分型缺失率(geno)≤0.05;2)最小等位基因频率(maf)≥0.05;3)单个样本的分型缺失率(mind)<0.1。
1.2.3 群体结构分析和全基因组关联分析 基于过滤后的SNP信息进行主成分分析(PCA),获得各个PC的方差解释率及样本在各个PC中的得分矩阵,从SNP中提取的关键信息按照效应从大到小分为PC1、PC2、PC3等,用于表征群体结构。
利用GCTA[25]对鲤分型结果和体质量表型数据进行全基因组关联分析(genome-wide association study,GWAS),以PCA结果进行辅因子校正,GWAS模型为混合线性模型(MLM)。GWAS结果用曼哈顿图和QQ-plot图展示,曼哈顿图显示鲤全基因组中与体质量有关的显著位点及P值,灰色线是全基因组水平上5%的显著性阈值。SNP密度图展示SNP在染色体上的分布。通过鲤注释文件,筛选关联SNP附近的基因。
1.2.4 全基因组选择流程 全基因组选择的具体流程见图1,将有基因分型和表型的参考群体代入模型中评估各模型准确性,然后选出最优的模型方法,把只有基因分型的候选群体代入模型预测育种值,最后根据预测所得的结果决定是否留种培育。
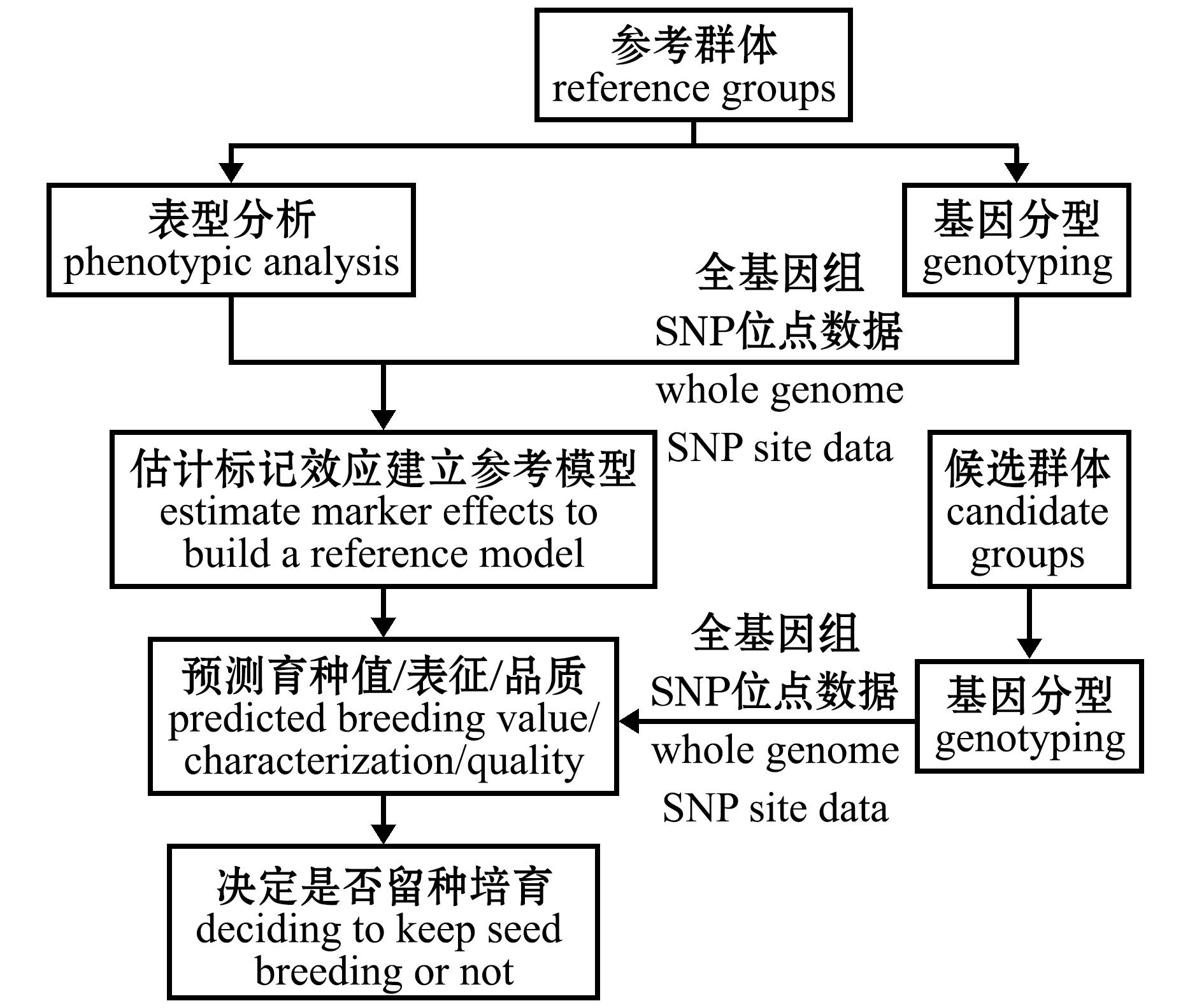
图1 全基因组选择流程
Fig.1 Genome-wide selection process
1.2.5 全基因组选择模型 采用GBLUP、BayesA、BayesB、BayesC、Bayes Lasso、Bayesian Ridge Regression、RKHS、RandomForest、XGBoost和LightGBM 10种模型对训练群体的基因型数据和表型数据进行训练,并通过5倍交叉验证计算预测准确性,然后对预测群体进行表型值预测。
1)GBLUP模型。GBLUP模型于2008年由Vanraden[26]提出,相比于传统BLUP模型中基于系谱计算的A矩阵,GBLUP模型则基于遗传标记计算的G矩阵预测结果更加准确且高效,但仍具有很大的改进空间[27-28]。
2)Bayes类模型(BayesA、BayesB、BayesC、Bayes Lasso、Bayesian Ridge Regression)。最早将贝叶斯方法应用于全基因组选择中的是Meuwisen[18],利用BayesA和BayesB方法解决SNP标记的数目远多于表型记录的问题。在这两种贝叶斯模型的基础上,研究人员又提出了多种贝叶斯模型,如Bayes C和Bayes Lasso等。贝叶斯类方法的统计模型如下:
(1)
式中:y为性状的表型向量;X为b的关系矩阵;b为固定效应;m为标记的数量;Zij为个体i的j标记的基因型(0/1/2);gj为第j个位点贡献的效应值;残差效应e符合正态分布![]()
3)RKHS模型。再生核希尔伯特空间(Reproducing kernel Hilbert space)[29]是一种典型的半参数方法,其使用高斯核函数来拟合模型,即
y=Xb+Khα+ε,
(2)
Kh(xi,xj)=exp(-hdij)。
(3)
式中:α为平均值是0、协方差矩阵是![]() 的多变量正态分布;ε~N(0,Inσ2);Kh为个体相关性的核函数;dij为个体i和j根据基因型计算的欧氏距离的平方;平滑参数h定义为dij平均值的一半。
的多变量正态分布;ε~N(0,Inσ2);Kh为个体相关性的核函数;dij为个体i和j根据基因型计算的欧氏距离的平方;平滑参数h定义为dij平均值的一半。
RKHS模型可采用贝叶斯框架的Gibbs抽样器,或者混合线性模型来求解。使用R语言环境下的BGLR包[30]进行GBLUP模型、Bayes类模型和RKHS模型的计算。
4)机器学习模型。近年来,随着高通量测序技术的快速发展,获得的全基因组数据的标记位点规模愈加庞大,这对全基因组选择方法的发展和完善提出了挑战[31]。研究人员将机器学习相关的理论方法与全基因组选择育种结合,涉及的方法包括支持向量机、随机森林(RandomForest)、XGBoost、LightGBM和神经网络等。
RandomForest是在决策树的基础上提出的一种集成学习方法,组合多个决策树使得结果有较高的精确度和泛化能力;XGBoost是经过优化的分布式梯度提升库,优点是高效、灵活且可移植;GBDT是机器学习中一个长盛不衰的模型,其主要思想是利用弱分类器(决策树)迭代训练以得到最优模型,该模型具有训练效果好、不易过拟合等优点。使用Python环境下的Scikit learn包[32]进行RandomForest、XGBoost、LightGBM模型的计算。
1.2.6 估计准确性标准 5倍交叉验证具体方法为:将训练群体随机分成5份,其中4份作为训练集,1份作为测试集,训练集中每一份抽样均有机会作为测试集,并计算测试集的真实值与预测值间的皮尔森相关系数;重复交叉验证4次,最终的相关系数平均值作为预测精度(r)。
2 结果与分析
2.1 表型和基因型数据的采集与质控
采用250K高密度SNP芯片进行基因分型共得到619尾黄河鲤的138 706个多态性SNP位点,采用PLINK 1.90软件控制基因分型结果的质量,剔除不符合标准的位点和样本,最终获得613个个体的67 506个SNP位点用于后续分析。鲤样本测量的体质量范围为800~2 050 g(图2)。使用R语言的CMplot[33]软件包生成鲤SNP位点的密度图(图3),结果显示,分析用的6万多个位点较均匀地分布在50个染色体上。
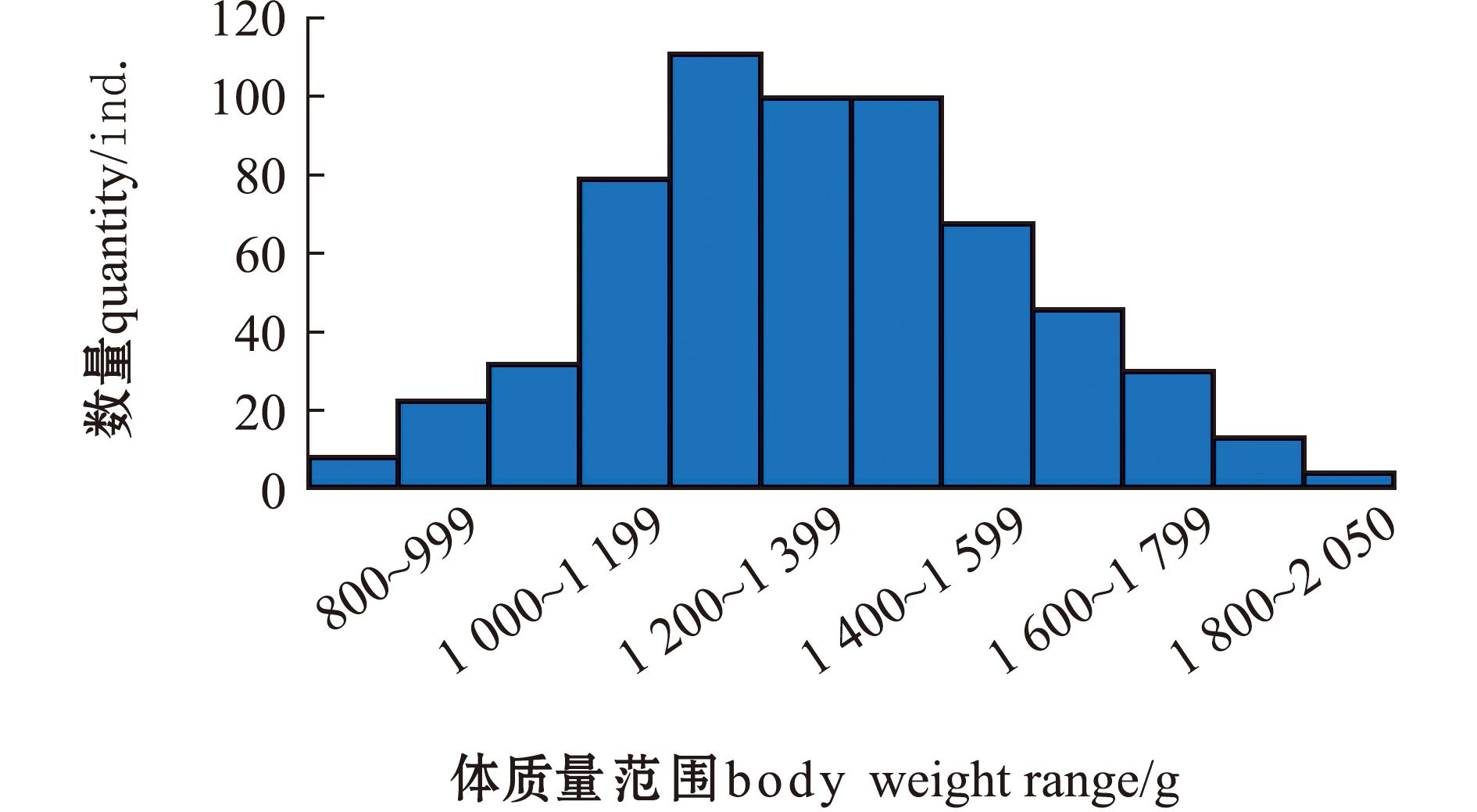
图2 鲤体质量性状分布
Fig.2 Distribution of body weight trait of common carp

图3 鲤体质量性状的SNP密度
Fig.3 SNP density distribution for body weight trait in common carp
2.2 群体结构分析
群体分层因素对GWAS有显著影响,为了研究鲤样本的遗传群体结构,利用质控后的6万多个SNP信息进行主成分分析(PCA),结果显示,鲤群体样本存在一定的群体分层(图4)。考虑到群体分层对GWAS分析的影响,在后续关联分析中利用主成分分析结果作为协变量进行校正。
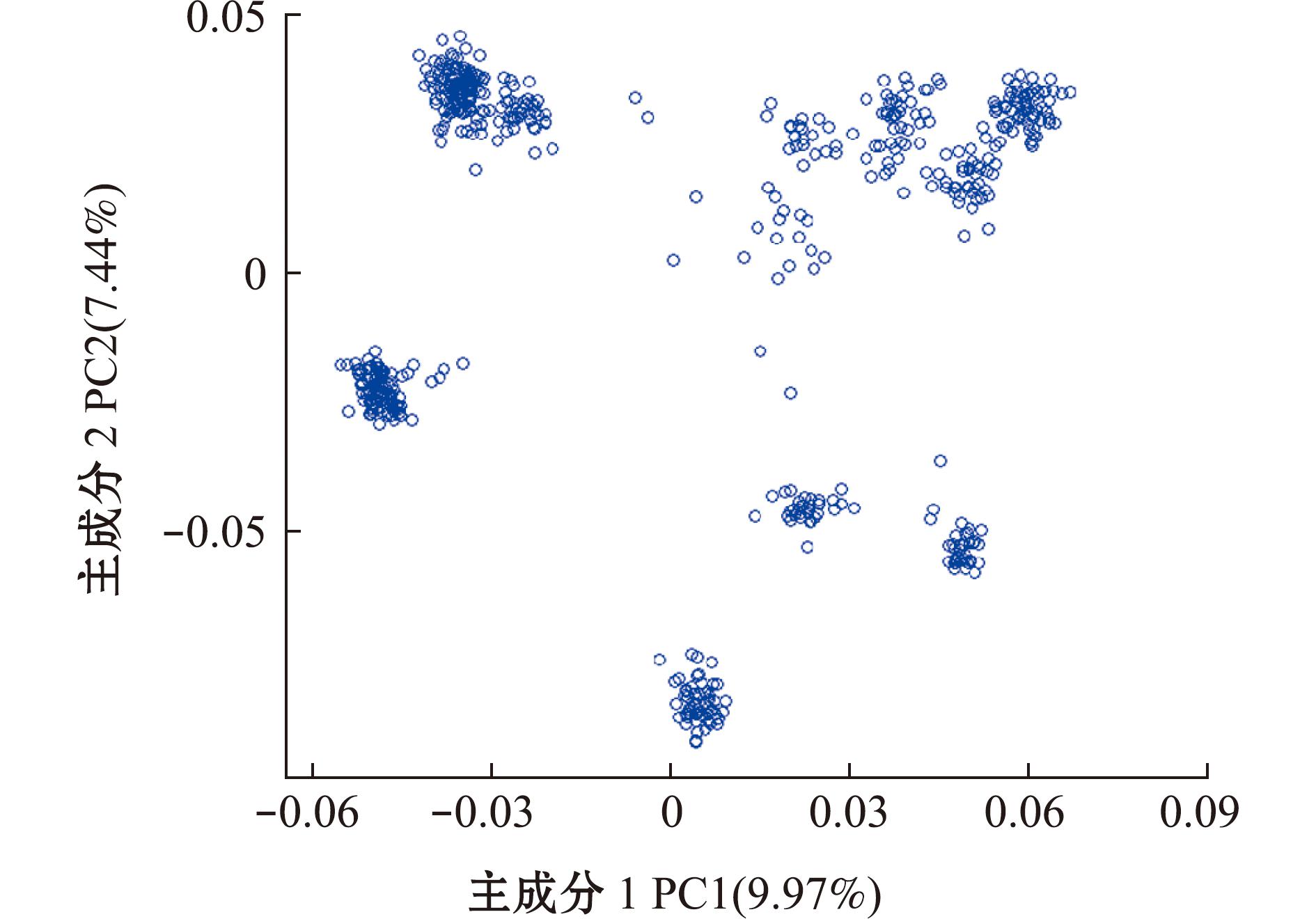
图4 鲤群体主成分分析
Fig.4 Principal component analysis of common carp populations
2.3 全基因组关联分析
使用GCTA软件进行GWAS分析时,由于本研究中的体质量性状为连续性状,所以采用线性回归分析,使用R语言的CMplot软件包生成鲤体质量性状的Q-Q图(图5)和曼哈顿图(图6)。从图5可见,观测值和期望值一致,散点分布与趋势线吻合度较高,基因组膨胀系数较低,可以推测关联分析结果较可靠。从图6可见,显著性关联位点与提示性关联位点成簇分布,大部分位于1号染色体上,表明该基因组区域可能存在与鲤体质量性状密切关联的基因,用Bonferroni校正将设定的显著性水平除以测试次数(SNP数),最终得到一个总的阈值,从而筛选到P值小于7.4×10-7的SNP位点共有5个,分别为carp135405、carp200616、carp200573、carp043762、carp232462,通过SNP附近的基因注释,筛选出WBP1L、GPM6B、TIMMDC1、RCAN1和EOGT 5个基因(表1)。
表1 鲤体质量生长性状相关的全基因组显著SNP详细信息
Tab.1 Details of genome-wide significant SNPs associated with body weight growth trait in common carp
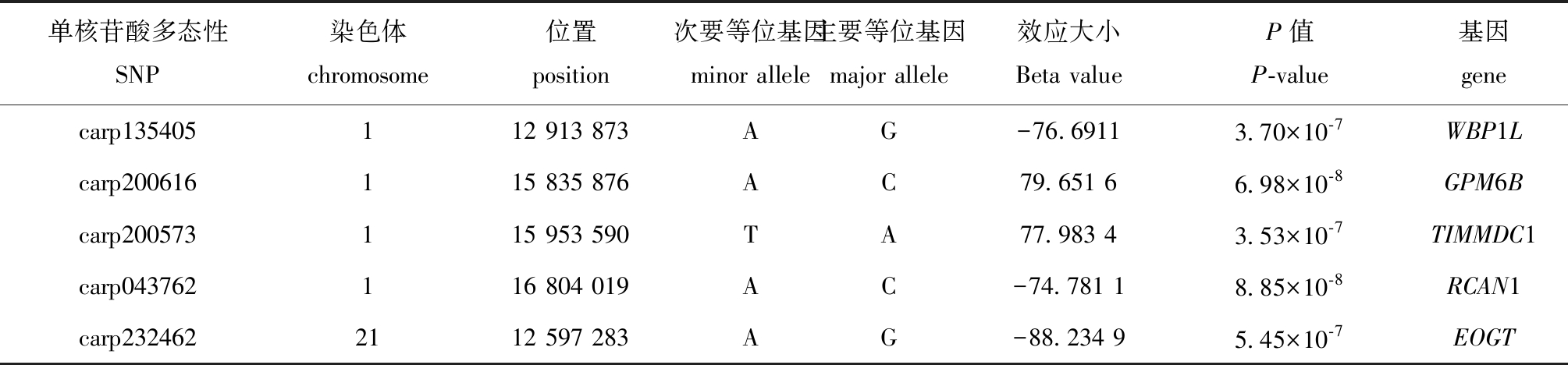
单核苷酸多态性SNP染色体chromosome位置position次要等位基因minor allele主要等位基因major allele效应大小Beta valueP值P-value基因genecarp135405112 913 873AG-76.69113.70×10-7WBP1Lcarp200616115 835 876AC79.651 66.98×10-8GPM6Bcarp200573115 953 590TA77.983 43.53×10-7TIMMDC1carp043762116 804 019AC-74.781 18.85×10-8RCAN1carp2324622112 597 283AG-88.234 95.45×10-7EOGT
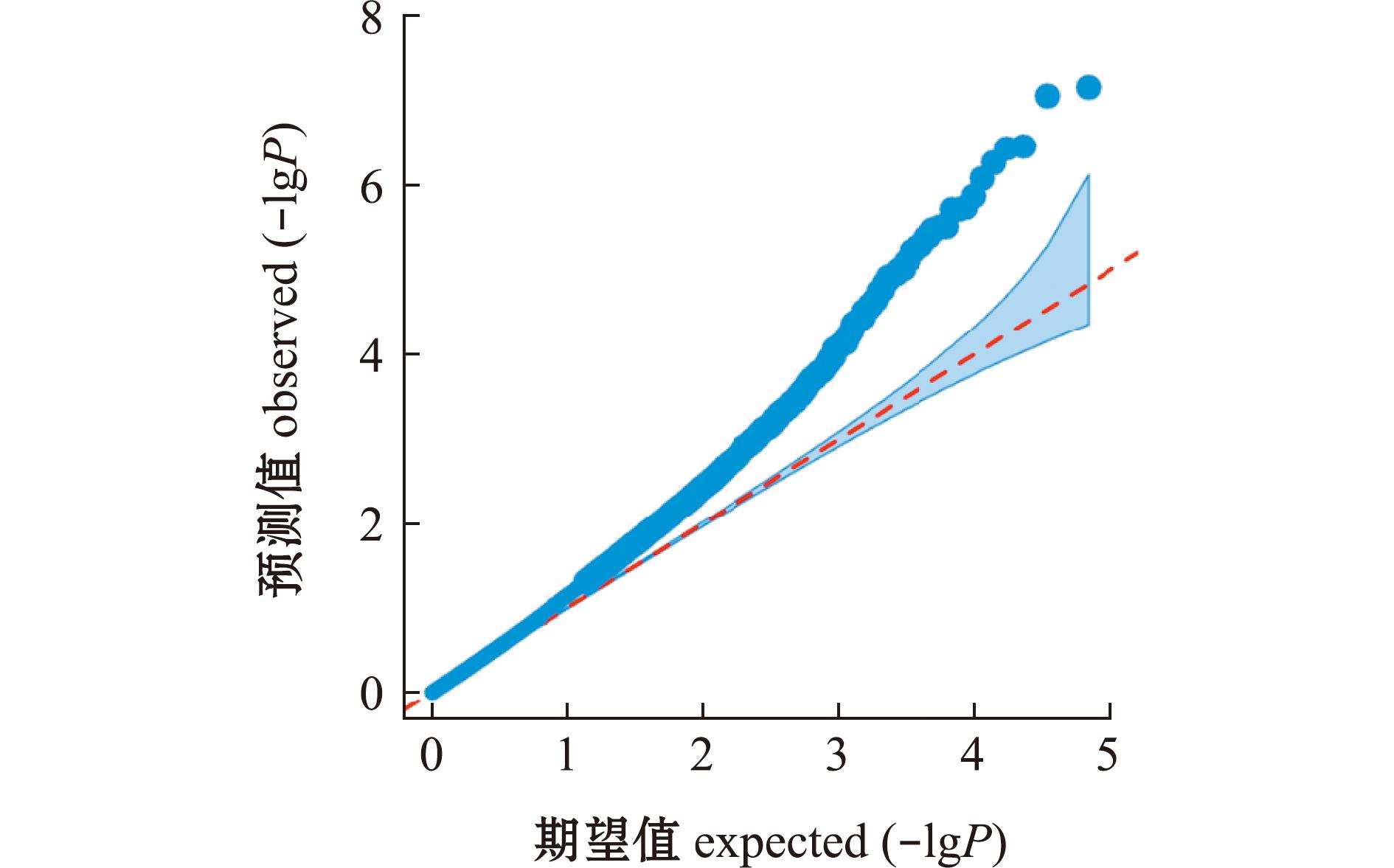
图5 鲤体质量性状GWAS关联分析的Q-Q图
Fig.5 Q-Q plot for GWAS association analysis of body weight trait in common carp
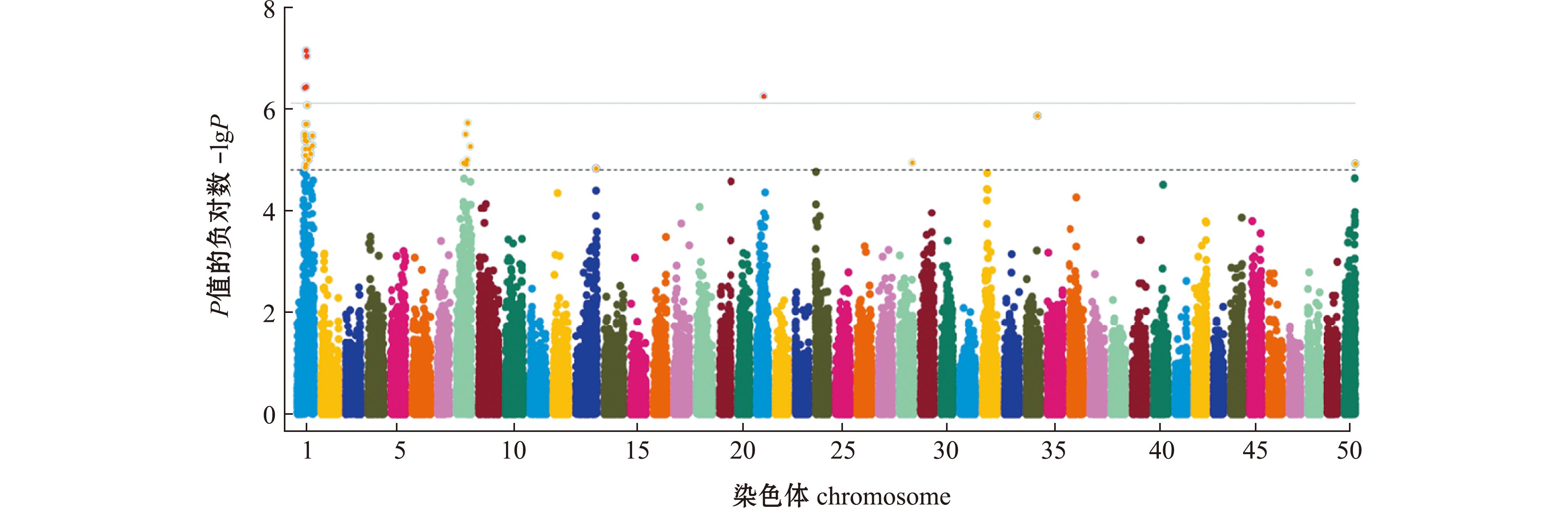
图6 鲤体质量性状GWAS关联分析的曼哈顿图
Fig.6 Manhattan plot for GWAS association analysis of body weight trait in common carp
2.4 鲤体质量性状全基因组选择不同模型的预测准确性比较
按照P值从小到大,分别选择鲤体质量性状关联的前100、500、1 000、3 000、5 000和20 000个SNP位点,使用GBLUP、BayesA等10种全基因组选择模型进行预测,每种模型进行100次5-fold交叉验证重复,并校正评估的准确性。变异系数越小,说明模型预测的稳定性越高。通过图7的变异系数分析可知,除了RandomForest模型外,其他模型的变异系数的值都小于0.4,其中XGBoost、LightGBM和GBLUP模型的变异系数较低,说明这3种模型预测的稳定性比较可靠。

图7 鲤体质量性状的全基因组选择模型准确性变异系数
Fig.7 Variation coefficient of accuracy of genome-wide selection model for body weight trait in common carp
从表2可见,10个模型对鲤体质量性状的预测精度范围为0.052 5~0.452 6,其中,当SNP数量为5 000时,GBLUP模型的预测准确性最高(0.452 6),而SNP数量为20 000时,RandomForest模型的预测准确性最低(0.052 5)。
表2 鲤体质量性状的全基因组选择模型预测准确性
Tab.2 Accuracy of genome-wide selection model for body weight trait in common carp

模型 modelSNP数量 number of SNP100500 1 0003 0005 00020 000GBLUP0.187 40.308 40.344 40.439 30.452 60.400 7Bayesian Ridge Regression0.163 50.246 70.278 80.388 70.412 30.361 1Bayes Lasso0.164 20.254 00.278 50.385 00.402 20.361 7BayesA0.163 40.251 50.278 00.385 20.407 60.363 7BayesB0.154 70.253 00.270 60.376 70.398 40.354 2BayesC0.153 90.255 20.276 20.384 00.405 80.359 0RKHS0.166 80.247 30.278 40.386 00.405 20.353 8RandomForest0.144 20.183 80.157 90.126 90.114 70.052 5XGBoost0.256 40.278 90.246 10.179 70.160 40.109 2LightGBM0.146 30.148 20.135 40.117 50.106 40.083 2
同一模型在不同SNP数量下预测比较发现:在GBLUP和贝叶斯模型中,当SNP数量为100~5 000时,数量越多模型预测准确性越高;当SNP数量为20 000时,所有模型的准确性均有所降低;在3个机器学习模型中,SNP数量为500时,模型的预测准确性均最高,随着SNP数量增加准确性逐渐降低。在同一SNP数量下不同模型比较发现,当SNP为100时,XGBoost模型的预测准确性最高,为0.256 4,当SNP数量为500、1 000、3 000、5 000、20 000时,GBLUP模型的预测准确性均最高,依次为0.308 4、0.344 4、0.439 3、0.452 6和0.400 7(表2)。
3 讨论
3.1 基因组选择模型中最优SNP密度
本研究中,在建立鲤体质量性状最佳GS模型的过程中,通过100次的5倍交叉验证发现,按照GWAS分析结果从P值最小的位点开始筛选,SNP数量为5 000时,GBLUP法、Bayes类及RKHS法基因组选择模型可预测性最高;3种机器学习模型的预测准确性在SNP数量为500时最高。当SNP数量为20 000时,其准确性比5 000时低,说明并不是SNP数量越多,其预测能力越好。这一结果与Zhao等[34]的研究结果一致,Zhao等[34]在大黄鱼抗刺激隐核虫基因组选择育种中发现,选择600个P值最小的SNP参与基因组选择模型时,预测准确性最高。
全基因组选择育种的预测能力受到SNP标记密度和标记间连锁不平衡(linkage disequilibrium,LD)效应的影响,通常SNP标记数量越多,预测精度越高,但是当SNP标记密度处于饱和状态时,增加标记数量对模型预测能力的提升有限[35]。SNP数量越大其准确性越小,可能是其表型和基因型的拟合有偏差,被选到分析的体质量相关联的SNP所占比例更小,线性相关性较小。
3.2 不同模型的预测能力比较
本研究中,基于鲤体质量性状对GBLUP、BayesA、BayesB、BayesC、Bayes Lasso、RKHS、Bayesian Ridge Regression、RandomForest、XGBoost和LightGBM共10种全基因组选择模型的预测准确性进行了比较,结果发现,当SNP数量为100时,XGBoost模型的预测准确性最高(0.2564),当SNP数量为500、1 000、3 000、5 000、20 000时,GBLUP法的预测准确性均优于其他9种模型,最高值为0.452 6;Bayes的5种模型和RKHS模型的预测准确性差值小于0.1,略低于GBLUP法,最高值为0.412 3(Bayesian Ridge Regresson法),最低值为0.153 9(BayesC法);RandomForest模型的预测准确性最低值为0.052 5,最高值为0.183 8;LightGBM模型的预测准确性最低值为0.083 2,最高值为0.148 2,RandomForest和LightGBM模型的准确性最高值均不超过0.2。Wang等[36]对鲤的24个性状进行全基因组选择分析,证实了GBLUP方法在大多数性状上都优于传统的BLUP方法,准确率提高最大为37.5%;Zhao[37]对大黄鱼进行传统模型和机器学习模型的全基因组选择分析,发现机器学习方法并不优于GBLUP和Bayes类方法,在大部分模拟场景下均表现出较低的预测准确性。上述两个研究结果均与本研究结果一致。而Wang等[38]选择4种GS模型对栉孔扇贝的4种性状进行预测,得出RKHS和SNN两种非线性方法的预测效果优于GBLUP和BayesB两种线性方法。上述研究表明,全基因组选择中并不是单一的某一个模型效果最好,模型的准确性与物种、性状及评估方法有关,所以在选择GS模型时都要考虑这些因素。
本研究中,选择RandomForest、XGBoost、LightGBM 3种机器学习模型对黄河鲤体质量性状进行全基因组选择分析,结果发现,机器学习模型的预测效果并没有预想中的突出,可能是在机器学习中一个SNP标记位点相当于一个特征,这意味着5 000个基因数据有着5 000个特征属性。鲤基因分型的高维小样本数据具有以下两个特点:其一,大量无效的噪声位点会干扰模型,降低模型精度;其二,特征矩阵过大致使计算量增加,模型训练时间变长。因此,在机器学习算法中针对基因分型数据高维小样本的特点,需要进行特征降维以减少特征属性的个数[39]。由此可见,全基因组选择在不同物种、不同性状中每种模型的预测能力不一,且受众多因素影响,需要通过不同组合对比分析,选择最适于当前物种和性状的模型。
4 结论
1)通过全基因组关联分析和SNP附近的基因注释,得到WBP1L、GPM6B、TIMMDC1、RCAN1、EOGT共5个基因,说明这5个基因可能与黄河鲤体质量性状相关。
2)通过比较全基因组选择模型的预测准确性发现,GBLUP模型更适合黄河鲤的体质量性状基因组选育。本研究结果为开展鲤经济性状选育及探索鲤经济性状的全基因组选择和品种培育提供了有益参考。
[1] 刘永新,邵长伟,王书,等.简述我国水产种业发展现状、问题与展望[J].中国农村科技,2021(6):62-65.LIU Y X,SHAO C W,WANG S,et al.The present situation,problems and prospects of aquaculture seed industry in China are briefly described[J].China Rural Science &Technology,2021(6):62-65.(in Chinese)
[2] ZHANG G F,FANG X D,GUO X M,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[3] CHEN S L,ZHANG G J,SHAO C W,et al.Whole-genome sequence of a flatfish provides insights into ZW sex chromosome evolution and adaptation to a benthic lifestyle[J].Nature Genetics,2014,46(3):253-260.
[4] XU P,ZHANG X F,WANG X M,et al.Genome sequence and genetic diversity of the common carp,Cyprinus carpio[J].Nature Genetics,2014,46(11):1212-1219.
[5] AO J Q,MU Y N,XIANG L X,et al.Genome sequencing of the perciform fish Larimichthys crocea provides insights into molecular and genetic mechanisms of stress adaptation[J].PLoS Genetics,2015,11(4):e1005118.
[6] BERTHELOT C,BRUNET F,CHALOPIN D,et al.The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates[J].Nature Communications,2014,5:3657.
[7] LIEN S,KOOP B F,SANDVE S R,et al.The Atlantic salmon genome provides insights into rediploidization[J].Nature,2016,533(7602):200-205.
[8] TSAI H Y,HAMILTON A,TINCH A E,et al.Genome wide association and genomic prediction for growth traits in juvenile farmed Atlantic salmon using a high density SNP array[J].BMC Genomics,2015,16:969.
[9] TSAIRIDOU S,HAMILTON A,ROBLEDO D,et al.Optimizing low-cost genotyping and imputation strategies for genomic selection in Atlantic salmon[J].G3-Genes Genomes Genetics,2020,10(2):581-590.
[10] DOU J Z,LI X,FU Q,et al.Evaluation of the 2b-RAD method for genomic selection in scallop breeding[J].Scientific Reports,2016,6:19244.
[11] DONG L S,XIAO S J,CHEN J W,et al.Genomic selection using extreme phenotypes and pre-selection of SNPs in large yellow croaker (Larimichthys crocea)[J].Marine Biotechnology,2016,18(5):575-583.
[12] DONG L S,XIAO S J,WANG Q R,et al.Comparative analysis of the GBLUP,emBayesB,and GWAS algorithms to predict genetic values in large yellow croaker (Larimichthys crocea)[J].BMC Genomics,2016,17:460.
[13] ZHAO J,ZHOU T,BAI H Q,et al.Genome-wide association analysis reveals the genetic architecture of parasite (Cryptocaryon irritans) resistance in large yellow croaker (Larimichthys crocea)[J].Marine Biotechnology,2021,23(2):242-254.
[14] LIU Y,LU S,LIU F,et al.Genomic selection using BayesCπ and GBLUP for resistance against Edwardsiella tarda in Japanese flounder (Paralichthys olivaceus)[J].Marine Biotechnology,2018,20(5):559-565.
[15] LU S,LIU Y,YU X J,et al.Prediction of genomic breeding values based on pre-selected SNPs using ssGBLUP,WssGBLUP and BayesB for Edwardsiellosis resistance in Japanese flounder[J].Genetics,Selection,Evolution,2020,52(1):49.
[16] LU S,ZHOU Q,CHEN Y D,et al.Development of a 38 K single nucleotide polymorphism array and application in genomic selection for resistance against Vibrio harveyi in Chinese tongue sole,Cynoglossus semilaevis[J].Genomics,2021,113(4):1838-1844.
[17] 陈松林,徐文腾,卢昇,等.水产育种生物技术发展战略研究[J].中国工程科学,2023,25(4):214-226.CHEN S L,XU W T,LU S,et al.Development strategy for aquatic breeding biotechnology[J].Strategic Study of CAE,2023,25(4):214-226.(in Chinese)
[18] MEUWISSEN T H,HAYES B J,GODDARD M E.Prediction of total genetic value using genome-wide dense marker maps[J].Genetics,2001,157(4):1819-1829.
[19] ZHANG Z,ZHANG Q,DING X D.Advances in genomic selection in domestic animals[J].Chinese Science Bulletin,2011,56(25):2655-2663.
[20] NAKAJIMA T,HUDSON M J,UCHIYAMA J,et al.Common carp aquaculture in Neolithic China dates back 8,000 years[J].Nature Ecology &Evolution,2019,3(10):1415-1418.
[21] BALON E K.Origin and domestication of the wild carp,Cyprinus carpio:from Roman gourmets to the swimming flowers[J].Aquaculture,1995,129(1/2/3/4):3-48.
[22] 李佩珍,许建,朱优秀,等.黄河鲤生长性状的转录组解析[J].中国渔业质量与标准,2022,12(2):22-31.LI P Z,XU J,ZHU Y X,et al.Transcriptome analysis for growth traits of Cyprinus carpio[J].Chinese Fishery Quality and Standards,2022,12(2):22-31.(in Chinese)
[23] XU J,ZHAO Z X,ZHANG X F,et al.Development and evaluation of the first high-throughput SNP array for common carp (Cyprinus carpio)[J].BMC Genomics,2014,15:307.
[24] PURCELL S,NEALE B,TODD-BROWN K,et al.PLINK:a tool set for whole-genome association and population-based linkage analyses[J].American Journal of Human Genetics,2007,81(3):559-575.
[25] YANG J,LEE S H,GODDARD M E,et al.GCTA:a tool for genome-wide complex trait analysis[J].American Journal of Human Genetics,2011,88(1):76-82.
[26] VANRADEN P M.Efficient methods to compute genomic predictions[J].Journal of Dairy Science,2008,91(11):4414-4423.
[27] REN D Y,AN L X,LI B J,et al.Efficient weighting methods for genomic best linear-unbiased prediction (BLUP) adapted to the genetic architectures of quantitative traits[J].Heredity,2021,126(2):320-334.
[28] YANG J,ZENG J,GODDARD M E,et al.Concepts,estimation and interpretation of SNP-based heritability[J].Nature Genetics,2017,49(9):1304-1310.
[29] GIANOLA D,VAN KAAM J B C H M.Reproducing kernel Hilbert spaces regression methods for genomic assisted prediction of quantitative traits[J].Genetics,2008,178(4):2289-2303.
[30] PEREZ P,DE LOS CAMPOS G.Genome-wide regression and prediction with the BGLR statistical package[J].Genetics,2014,198(2):483-495.
[31] 王琦,朱迪,王宇哲,等.全基因组SNP分型策略及基因组预测方法的研究进展[J].畜牧兽医学报,2020,51(2):205-216.WANG Q,ZHU D,WANG Y Z,et al.Research progress of genomic-wide SNP genotyping and genomic prediction methods[J].Chinese Journal of Animal and Veterinary Sciences,2020,51(2):205-216.(in Chinese)
[32] BISONG E.Introduction to scikit-learn[M]//Building machine learning and deep learning models on google cloud platform.Berkeley,CA:Apress,2019:215-229.
[33] YIN L L,ZHANG H H,TANG Z S,et al.rMVP:a memory-efficient,visualization-enhanced,and parallel-accelerated tool for genome-wide association study[J].Genomics,Proteomics &Bioinformatics,2021,19(4):619-628.
[34] ZHAO J,BAI H Q,KE Q Z,et al.Genomic selection for parasitic ciliate Cryptocaryon irritans resistance in large yellow croaker[J].Aquaculture,2021,531:735786.
[35] ZHONG S Q,DEKKERS J C M,FERNANDO R L,et al.Factors affecting accuracy from genomic selection in populations derived from multiple inbred lines:a Barley case study[J].Genetics,2009,182(1):355-364.
[36] WANG J Y,CHEN L,LI B J,et al.Performance of genome prediction for morphological and growth-related traits in Yellow River carp[J].Aquaculture,2021,536:736463.
[37] 赵吉.大黄鱼(Larimichthys crocea)抗刺激隐核虫(Cryptocaryon irritans)性状的全基因组选择育种研究[D].厦门:厦门大学,2021.ZHAO J.Genome-wide selection breeding of Cryptocaryon irritans resistance in large yellow crocea (Pseudosciaena crocea)[D].Xiamen:Xiamen University,2021.(in Chinese)
[38] WANG Y F,SUN G D,ZENG Q F,et al.Predicting growth traits with genomic selection methods in zhikong scallop (Chlamys farreri)[J].Marine Biotechnology,2018,20(6):769-779.
[39] 张阳.基于机器学习和全基因组选择的长牡蛎选种分类[D].大连:大连理工大学,2021.ZHANG Y.Crassostrea gigas breeding classification based on machine learning and genomic selection[D].Dalian:Dalian University of Technology,2021.(in Chinese)