近年来,随着现代分子生物学技术的发展,简单重复序列标记和单核苷酸多态性标记已逐渐成为动植物遗传多样性分析、分子标记开发和目标性状定位等领域至关重要的工具。简单重复序列(simple sequence repeat,SSR),一般是由1~6个核苷酸为重复单位组成的长达几十个核苷酸的串联重复序列,广泛分布于真核生物基因组中,具有分布广、重复性高、多态性丰富和共显性遗传等优点,目前,基于转录组来源的SSR标记开发已在水生动物中被广泛应用[1-2]。单核苷酸多态性(single nucleotide polymorphism,SNP)是在基因组水平上发生单个核苷酸变异而引起的DNA序列的多态性,包括转换、颠换。由于SNP具有分布广、位点多、易检测、准确率高、遗传稳定性高且在不同物种中具有显著差异等优点,在水生生物中被广泛用于分子辅助育种[3-4]。
传统的分子标记开发方法存在耗时长、效率低、步骤烦琐和通量低等缺点。表达序列标签(expressed sequence tage,EST)测序为基因组测序提供了一个较好的方案,只需要完整染色体的一小部分就可以产生基因转录部分的序列,但这种技术既费时又费力,一般都用于少数模式生物的测序[5]。而二代测序(next-generation sequencing,NGS)技术与传统方法相比,筛选分子标记的成本更低、效益更高[6]。转录组测序(RNA-seq)是基于NGS的一种高通量SSR和SNP标记检测技术,目前已在动植物中被广泛应用。
花斑裸鲤(Gymnocypris eckloni)俗称大嘴鱼、大嘴花鱼,隶属于裂腹鱼亚科(Schizothoracinae)裸鲤属(Gymnocypris),是分布于黄河上游干支流的特有鱼类。20世纪,由于水利工程建设、人为偷捕和水环境污染等,花斑裸鲤资源量大幅度减少[7]。通过软件筛选SSR和SNP标记,可为后续分析黄河上游花斑裸鲤地理群体的遗传多样性和遗传分化提供基础资料,从而确定其地理群体的多样性[8],为其种质资源保护及遗传改良等研究提供理论依据。目前,对于花斑裸鲤的研究主要集中在其系统发育与起源[9]、遗传多样性[10]、人工繁育与驯化养殖[11]、胚胎发育[12]及生物学特性[13]等方面,通过高通量测序技术分析花斑裸鲤SSR和SNP分子标记的分布及特征鲜有报道。
本研究中,将花斑裸鲤3个组织(鳃、肾脏和肝脏)分别进行转录组测序,挖掘测序数据的SSR、SNP和InDel位点,并分析其特点,以期为花斑裸鲤遗传种质资源的保护、遗传多样性分析、遗传图谱构建和分子辅助育种等提供基础资料。
1 材料与方法
1.1 材料
试验用花斑裸鲤于2022年4月采自青海省渔业环境监测站,共采集12尾2+龄的花斑裸鲤,暂养在青海大学冷水鱼养殖基地。
1.2 方法
1.2.1 样本采集 随机选取3尾健康的2+龄人工养殖花斑裸鲤,用MS-222(100 mg/L)麻醉后,分别采集其鳃、肾脏和肝脏组织,将组织样品立即浸入液氮中,运输至青海大学高原动物保护与利用实验室后置于-80 ℃超低温冰箱中保存备用。
1.2.2 RNA提取 使用Trizol法提取样品总RNA。采用10 g/L琼脂糖凝胶电泳和Agilent 2100检测RNA样品的完整性,利用Nanodrop 2000检测RNA样品的纯度(OD260 nm/OD280 nm=1.8~2.0时,判断为合格),利用Qubit 2.0荧光定量仪定量RNA样品的浓度,选择完整性好、纯度高的RNA样品用于后续文库构建。
1.2.3 文库构建和测序 采用NEBNext Ultra RNA Library prep Kit (NEB,USA)进行花斑裸鲤cDNA文库构建。首先通过Qubit 2.0荧光定量仪初测文库浓度,然后再通过Agilent 2100对文库片段分布进行检测,确保插入的片段大小合适、峰型单一、无杂峰、无接头、无引物二聚体,然后通过qPCR对文库有效浓度进行精确定量,达到有效文库浓度后采用Illumina Hiseq 2000平台进行高通量测序。
1.2.4 过滤和组装 利用Cutadapt 1.9.1软件对测序获得的raw date进行过滤,去除含接头、碱基含量>5%、质量评分低的reads后,获得高质量的clean data。使用Trinity 2.4.0拼接获得transcripts,在此基础上进行Corset层次聚类,挑选其中最长的一条参考序列Unigene用于后续分析使用。
1.2.5 SSR、SNP和InDel位点特征分析 获得转录组数据后,利用MISA软件(http://pgrc.ipk-gatersleben.de/misa)对Unigene进行SSR位点搜索,过滤掉单核苷酸重复次数<10、二核苷酸重复次数<6及三、四、五、六核苷酸重复次数<5的SSR,且满足SSR 位点两侧序列长度>100 bp,同时筛选碱基间隔≤100 bp的复合型 SSR。搜索结果采用Excel 2021软件进行统计分析,分析花斑裸鲤SSR的分布特征,包括各类型SSR重复基元的位点数、出现频率、平均分布距离,以及优势重复基元、重复次数和序列长度的分布特征等。采用GATK3软件进行SNP和InDel搜索,过滤掉碱基质量值<40、距离<2的SNP和InDel,分析其位点类型、数量、分布及发生频率等特征。
2 结果与分析
2.1 测序数据和Unigene组装
经Illumina测序后,从花斑裸鲤转录组中共获得56 384 438条原始数据,过滤后得到53 881 712条clean reads,其中,碱基质量值Q20、Q30分别为94.49、86.79,表明测序结果较好。经Trinity 2.4.0软件组装后共得到486 221条Unigenes序列,总长度为483 373 754 bp,平均长度为994 bp,N50为1 480 bp。绝大部分序列的长度分布于200~2 000 bp,占总序列的88.56%。然而Unigene的数量随Unigene序列长度的增加呈下降趋势,长度为200~500 bp的序列数量最多,占总数的38.45%,长度为501~1 000 bp、1 001~2 000 bp和>2 000 bp的序列数量次之,分别占总Unigenes的30.28%、19.83%和11.44%(表1)。
表1 花斑裸鲤基因组数据库长度统计
Tab.1 Statistics of length of Gymnocypris eckloni genome database
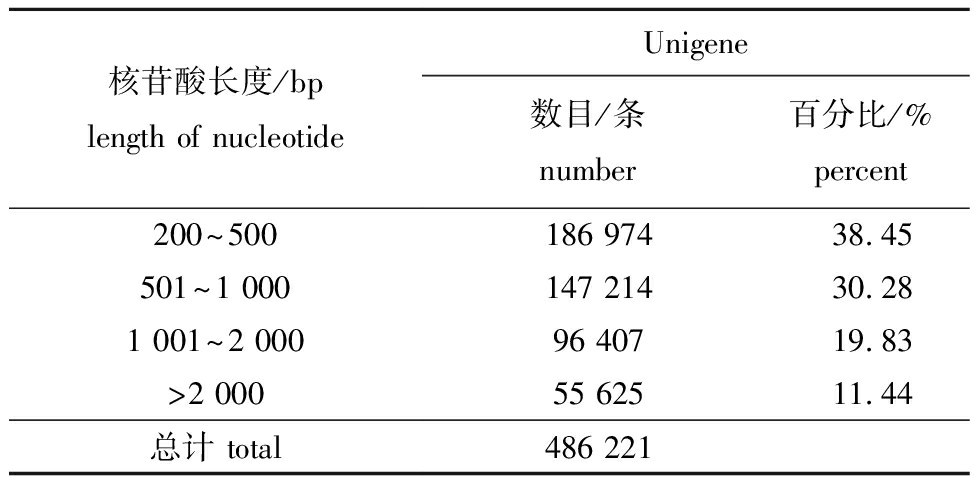
核苷酸长度/bplength of nucleotideUnigene数目/条number百分比/%percent200~500186 97438.45501~1 000147 21430.281 001~2 00096 40719.83>2 00055 62511.44总计 total486 221
2.2 SSR位点特征
2.2.1 SSR位点的分布及密度 利用MISA软件,在花斑裸鲤转录组的486 221条Unigenes序列中共查找到128 727个SSR位点。这些SSR位点分布于105 496条Unigenes上,包含1个以上SSR位点的Unigenes有19 017条,复合型SSR位点的Unigenes有5 438条,且具有完整型SSR(SSR序列中无任何错配或中断)的位点数约为复合型的22.67倍,SSR的发生频率(含有SSR的Unigene数目占Uningene总数的比例,即105 496/486 221)为21.70%,出现频率(SSR位点总数占Unigene总数的比例,即128 727/486 221)为26.47%。SSR的分布密度为0.266 SSR/kb,即平均每3.76 kb序列会出现1个SSR位点。全部SSR总长度为1 741 171 bp,占Unigene总长度的0.36%。
2.2.2 SSR重复类型 通过分析花斑裸鲤转录组SSR数据得知,其SSR重复类型主要有6种类型,在128 727个SSR位点中,主要以单核苷酸重复和二核苷酸重复为主,分别有59 891和54 647个,占总SSR的46.53%和42.45%;其次是三核苷酸重复,共有11 699个,占总SSR的9.09%;四、五、六核苷酸重复占比均较低,分别占总SSR的1.89%、0.03%和0.02%。SSR位点的序列总长度为1 741 171 bp,6种重复类型的SSR位点平均长度为13.53 bp,其中二核苷酸重复序列总长度最长,为804 262 bp,六核苷酸重复序列总长度最短,为1 044 bp(表2)。
表2 花斑裸鲤转录组SSR不同重复类型分布情况
Tab.2 Distribution of different repeat types of SSR in transcriptome of Gymnocypris eckloni
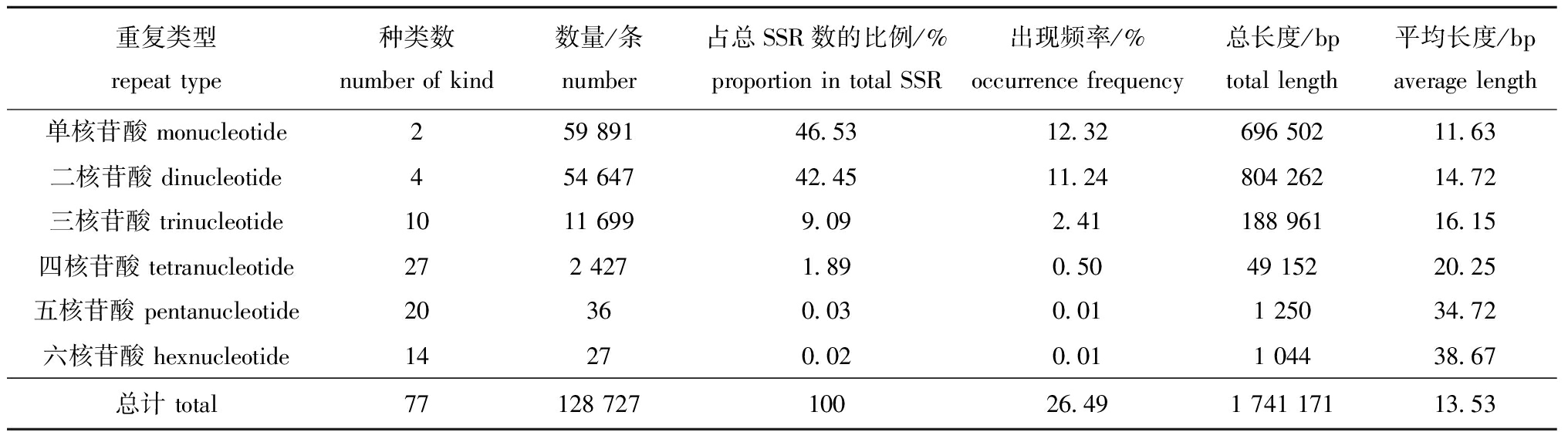
重复类型repeat type 种类数number of kind数量/条number占总SSR数的比例/%proportion in total SSR出现频率/%occurrence frequency总长度/bptotal length平均长度/bpaverage length单核苷酸 monucleotide259 89146.5312.32696 50211.63二核苷酸 dinucleotide454 64742.4511.24804 26214.72三核苷酸 trinucleotide1011 6999.092.41188 96116.15四核苷酸 tetranucleotide272 4271.890.5049 15220.25五核苷酸 pentanucleotide20360.030.011 25034.72六核苷酸 hexnucleotide14270.020.011 04438.67总计 total77128 72710026.491 741 17113.53
2.2.3 SSR重复基元碱基组成 单核苷酸至六核苷酸重复基元分别有2、4、10、27、20、14种。从表3可见:所有SSR重复类型中,单核苷酸重复类型的优势重复基元为A/T,共有53 696个SSR位点,分别占单核苷酸重复SSR和总SSR的89.66%和41.71%,而C/G分别占单核苷酸重复SSR和总SSR的10.34%和4.81%;二核苷酸重复类型的重复基元有AC/GT、AT/AT、AG/CT和CG/CG,分别占总SSR的23.27%、9.83%、9.18%和0.16%;三核苷酸重复类型的主要重复基元为AAT/ATT、ATC/ATG和AGG/CCT,分别占总SSR的3.70%、1.46%和1.05%;重复基元种类最丰富的是四核苷酸重复类型,然而各种类基元包含的SSR位点较少,其重复基元以AGAT/ATCT和AAAG/CTTT为主,分别占总SSR的0.51%和0.40%;五核苷酸重复和六核苷酸重复类型虽然重复基元类型也比较丰富,但各重复基元出现频率均较低(低于0.05%),五核苷酸重复类型的优势重复基元为ACTGC/AGTGC,六核苷酸重复类型的优势重复基元是AACCTG/AGGTTC和AAGGGC/CCCTTG。
表3 转录组SSR各重复基元及比例
Tab.3 SSR repeat motifs and ratio in transcriptome
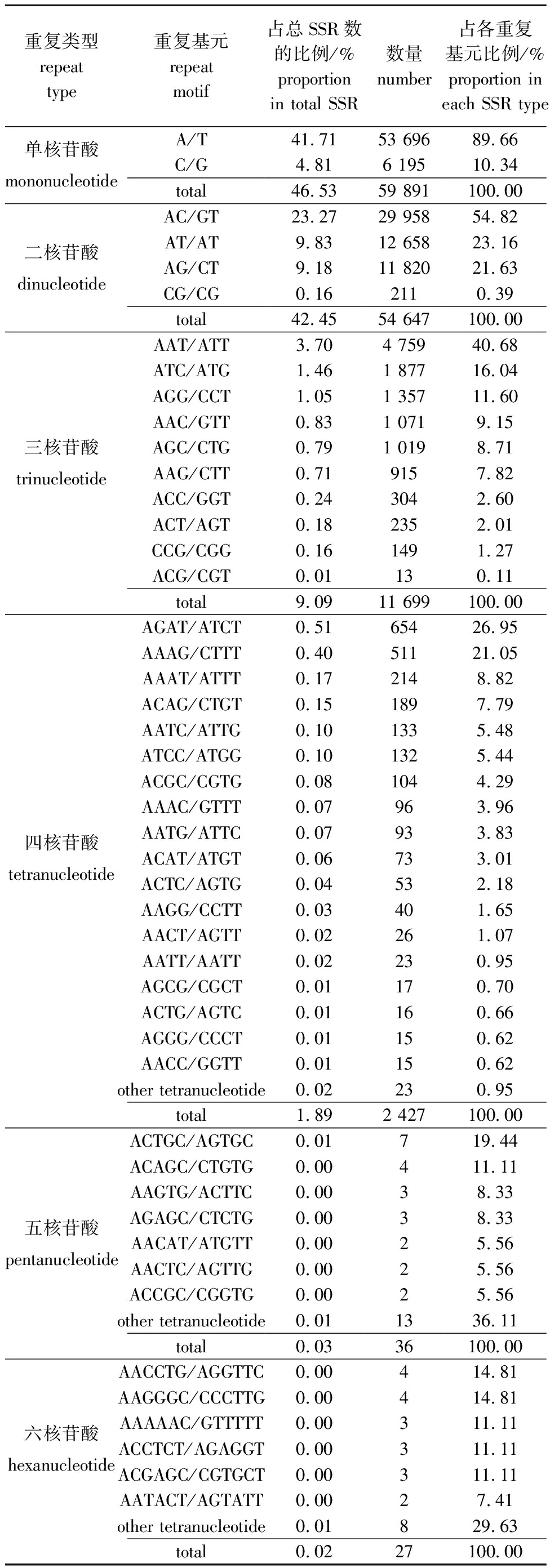
重复类型repeattype重复基元repeat motif占总SSR数的比例/%proportion in total SSR数量number占各重复基元比例/%proportion in each SSR typeA/T41.7153 69689.66单核苷酸 mononucleotideC/G4.816 19510.34total46.5359 891100.00AC/GT23.2729 95854.82AT/AT9.8312 65823.16二核苷酸 dinucleotideAG/CT9.1811 82021.63CG/CG0.162110.39total42.4554 647100.00AAT/ATT3.704 75940.68ATC/ATG1.461 87716.04AGG/CCT1.051 35711.60AAC/GTT0.831 0719.15三核苷酸 trinucleotideAGC/CTG0.791 0198.71AAG/CTT0.719157.82ACC/GGT0.243042.60ACT/AGT0.182352.01CCG/CGG0.161491.27ACG/CGT0.01130.11total9.0911 699100.00AGAT/ATCT0.5165426.95AAAG/CTTT0.4051121.05AAAT/ATTT0.172148.82ACAG/CTGT0.151897.79AATC/ATTG0.101335.48ATCC/ATGG0.101325.44ACGC/CGTG0.081044.29AAAC/GTTT0.07963.96AATG/ATTC0.07933.83四核苷酸 tetranucleotideACAT/ATGT0.06733.01ACTC/AGTG0.04532.18AAGG/CCTT0.03401.65AACT/AGTT0.02261.07AATT/AATT0.02230.95AGCG/CGCT0.01170.70ACTG/AGTC0.01160.66AGGG/CCCT0.01150.62AACC/GGTT0.01150.62other tetranucleotide0.02230.95total1.892 427100.00ACTGC/AGTGC0.01719.44ACAGC/CTGTG0.00411.11AAGTG/ACTTC0.0038.33AGAGC/CTCTG0.0038.33五核苷酸 pentanucleotideAACAT/ATGTT0.0025.56AACTC/AGTTG0.0025.56ACCGC/CGGTG0.0025.56other tetranucleotide0.011336.11total0.0336100.00AACCTG/AGGTTC0.00414.81AAGGGC/CCCTTG0.00414.81AAAAAC/GTTTTT0.00311.11六核苷酸 hexanucleotideACCTCT/AGAGGT0.00311.11ACGAGC/CGTGCT0.00311.11AATACT/AGTATT0.0027.41other tetranucleotide0.01829.63total0.0227100.00
2.2.4 SSR重复次数分布 花斑裸鲤转录组中SSR基元重复次数介于5~35次,随着重复基元次数的增加,SSR位点数呈递减趋势。SSR位点重复集中分布在5~15次,占总SSR位点数的87.52%,其中,重复次数为5~10次的SSR位点共有82 373个,占总SSR位点数的63.99%;其次是重复次数为11~15次的重复基元,共有30 288个SSR位点,占总SSR位点数的23.53%;重复次数为16~20次的SSR位点共有4 192个,重复次数在20次以上的SSR位点仅有176个(图1)。在所有重复基元中,重复次数为10、6、7、11、5次的SSR位点数较多,分别占总SSR位点数的23.00%、18.10%、9.94%、9.94%和8.19%。
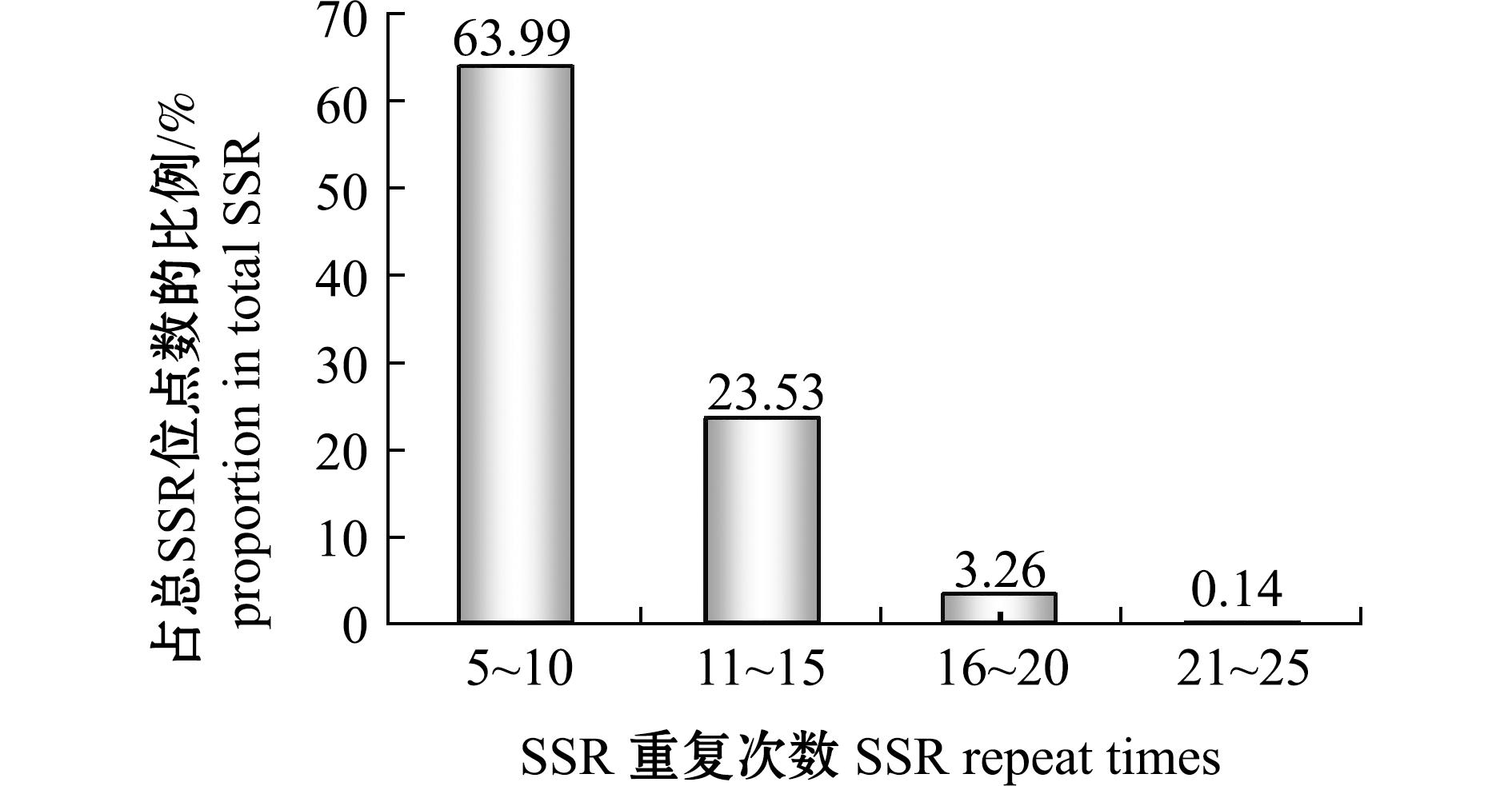
图1 花斑裸鲤转录组SSR不同重复次数分布频率
Fig.1 Frequency of different repeat times of SSR in transcriptome of Gymnocypris eckloni
在不同重复类型SSR间重复次数也存在较大差异。从表4可见:单核苷酸重复基元的重复次数类型最多,共有15种重复次数,集中在10~23次,其中10次重复占单核苷酸SSR位点总数的比例最高(43.07%);二核苷酸重复基元共有9种重复次数,集中在6~12次,其中,6次重复占二核苷酸SSR位点总数的比例最高(37.61%);三核苷酸重复基元共有7种重复次数,集中在5~10次,其中,5次重复占三核苷酸SSR位点总数的比例最高(69.66%);四核苷酸重复基元共有4种重复次数,以5次和6次的重复次数为主,其中,5次重复的SSR占四核苷酸SSR位点总数的比例最高(97.20%);五核苷酸重复基元共有7种重复次数,以5次重复次数的SSR位点为主,占五核苷酸重复SSR位点总数的52.78%;六核苷酸重复基元有7种重复次数,以5次重复次数的SSR 位点为主,占六核苷酸 SSR 位点总数的52.85%。
表4 花斑裸鲤转录组SSR重复次数及数量
Tab.4 Repeat times and number of SSR in transcriptome of Gymnocypris eckloni
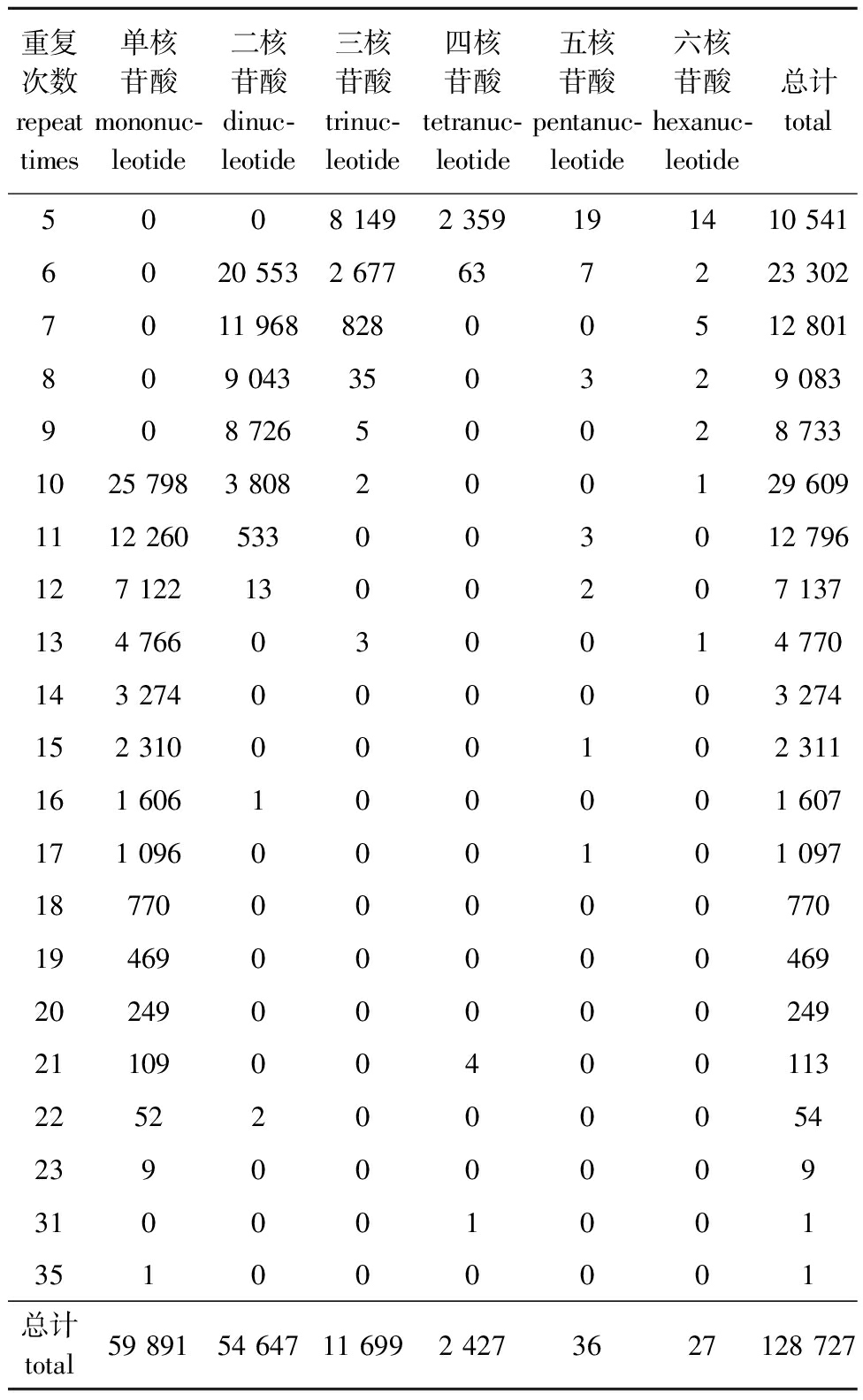
重复次数repeattimes单核苷酸mononuc-leotide二核苷酸dinuc-leotide三核苷酸trinuc-leotide四核苷酸tetranuc-leotide五核苷酸pentanuc-leotide六核苷酸hexanuc-leotide总计total5008 1492 359191410 5416020 5532 677637223 3027011 96882800512 801809 043350329 083908 72650028 7331025 7983 808200129 6091112 260533003012 796127 1221300207 137134 766030014 770143 274000003 274152 310000102 311161 606100001 607171 096000101 097187700000077019469000004692024900000249211090040011322522000054239000009310001001351000001总计total59 89154 64711 6992 4273627128 727
2.2.5 SSR序列长度分布 从表5可见:花斑裸鲤转录组中不同SSR重复基元类型的序列长度差异较大,SSR位点的长度为10~124 bp,平均长度为13.53 bp;SSR序列长度差异最大的是四核苷酸重复类型(20~124 bp),最小的是三核苷酸重复类型(15~39 bp);长度为12 bp的SSR数目最多(27 675个),占总SSR位点数的21.50%,其次是序列长度为10 bp和14 bp的SSR,分别有25 798和15 242个SSR位点,占总SSR位点数的20.04%和11.84%。总体上,花斑裸鲤转录组SSR大部分序列长度为10~20 bp,共有127 003个,占总SSR位点数的98.67%。
表5 花斑裸鲤转录组SSR序列长度分布
Tab.5 Length distribution of SSR repeat sequence in transcriptome of Gymnocypris eckloni
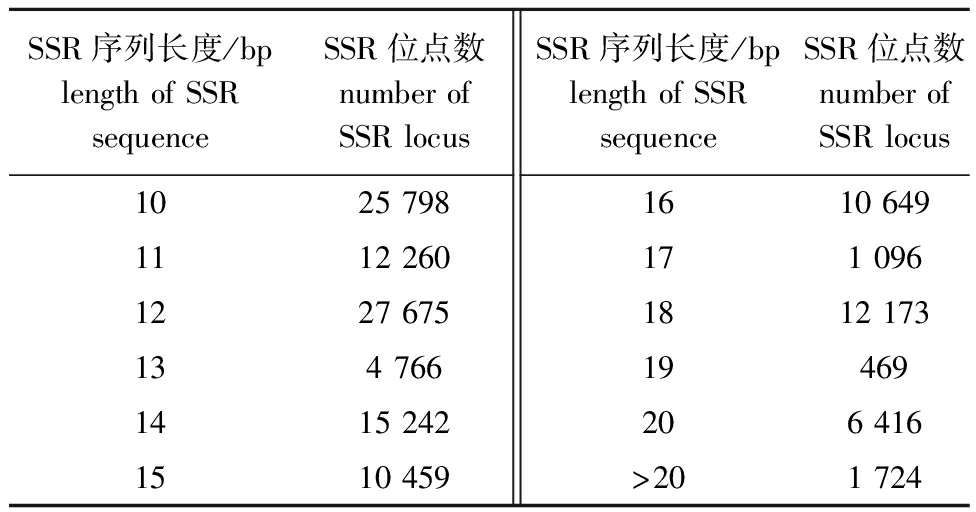
SSR序列长度/bp length of SSR sequenceSSR位点数 number of SSR locusSSR序列长度/bp length of SSR sequenceSSR位点数 number of SSR locus10111213141525 79812 26027 6754 76615 24210 4591617181920>2010 6491 09612 1734696 4161 724
2.3 SNP和InDel位点特征
2.3.1 SNP位点 利用GATK3软件对获得的转录组数据进行SNP位点查找,筛选到含有SNP位点的Unigenes有102 675条,共获得399 080个SNP位点,平均每1 211 bp出现一个SNP位点,在所有的SNP位点中,转换类型的SNP位点有224 662个,颠换类型的SNP位点有174 418个,转换类型发生频率高于颠换类型,转换/颠换比值为1.29。转换类型中A/G略高于C/T,颠换类型中A/T略高于A/C、T/G、C/G(表6)。
表6 花斑裸鲤转录组SNP不同类型分布
Tab.6 Distribution of different repeat types of SNP in transcriptome of Gymnocypris eckloni
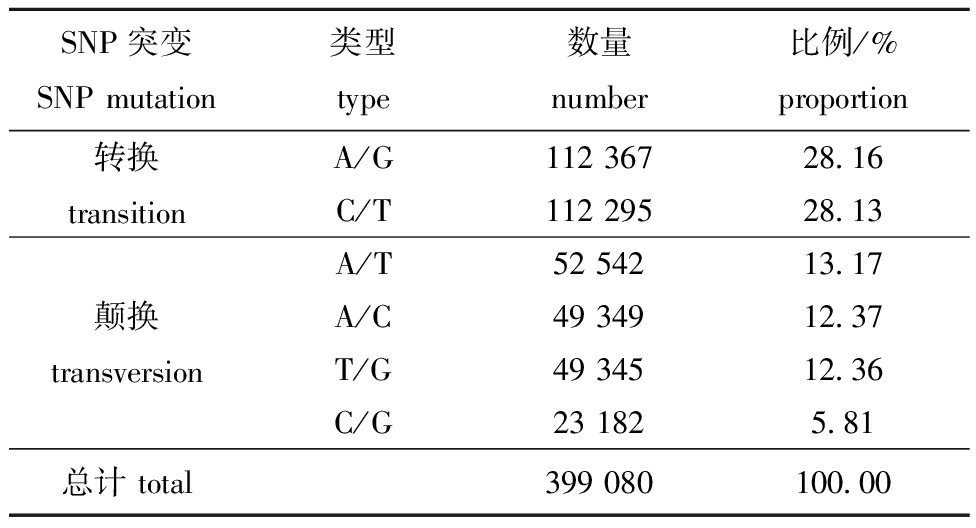
SNP突变SNP mutation类型type数量number比例/%proportion转换 transitionA/G112 36728.16C/T112 29528.13A/T52 54213.17颠换 transversionA/C49 34912.37T/G49 34512.36C/G23 1825.81总计 total399 080100.00
从表7可见,含有1个SNP位点的Unigenes最多(34.54%),其次是含有2个SNP位点的Unigenes(21.21%),随着Unigenes中SNP位点数目的增多,Unigenes数量呈下降趋势,在6 113个Unigenes中发现了10个以上SNP位点。
表7 花斑裸鲤转录组SNP和InDel位点分布
Tab.7 Distribution of SNP/InDel loci in transcriptome of Gymnocypris eckloni
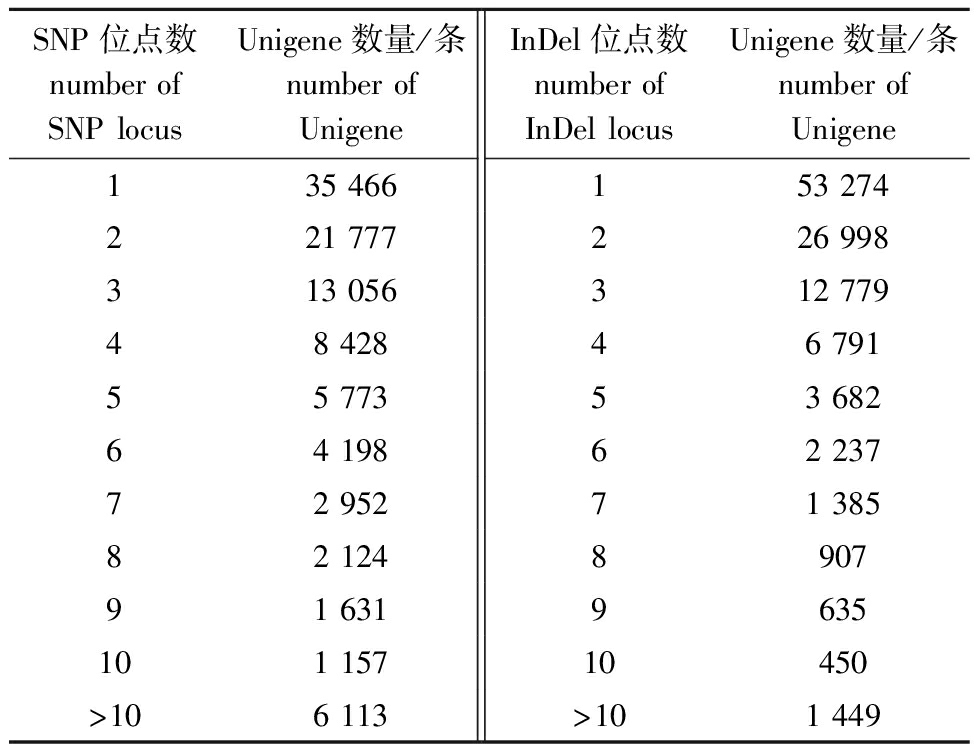
SNP位点数number of SNP locusUnigene数量/条number of UnigeneInDel位点数number of InDel locusUnigene数量/条number of Unigene135 466153 274221 777226 998313 056312 77948 42846 79155 77353 68264 19862 23772 95271 38582 124890791 6319635101 15710450>106 113>101 449
2.3.2 InDel位点 利用GATK3软件对获得的转录组数据进行InDel位点查找,筛选到含有InDel位点的Unigenes有110 587条,共获得254 065个InDel位点,每1 903 bp出现1个InDel位点,InDel位点分布趋势与SNP位点分布趋势大致相同。含有1个InDel位点的Unigenes最多(48.17%),其次是含有2个位点的Unigenes(24.41%),随着Unigenes中InDel位点数目的增多,Unigenes数量呈下降趋势,在1 449个Unigenes上发现了10个以上InDel位点(表7)。
3 讨论
3.1 花斑裸鲤SSR位点特征
3.1.1 SSR重复类型 了解和掌握渔业对象的生物学特征和遗传学背景,是开展渔业资源科学管理及可持续开发利用的前提,花斑裸鲤作为中国黄河上游特有的经济鱼类,线粒体和SSR分子标记仍是其种群遗传学研究的主要手段。随着高通量测序技术的发展和测序成本的降低,基于转录组学数据大量开发具有高多态性的花斑裸鲤SSR位点变得更加高效、便捷。
本研究中,从花斑裸鲤转录组数据中筛选出128 727个SSR位点,SSR的发生频率为21.70%,相较于一些水生动物的SSR发生频率较高,如已报道的红鳍东方鲀(Takifugu rubripes)[14]的SSR发生频率为11.5%,刀鲚(Coilia ectenes)[15]为8.76%,鲤(Cyprinus carpio)[16]为5.55%;而相较于另外一些水生动物SSR的发生频率较低,如已报道的翘嘴鳜(Siniperca chuatsi)[17]SSR发生频率为68.86%,东海带鱼(Trichiurus japanicus)[18]为40.95%,黄姑鱼(Nibea albiflora)[19]为39.30%。上述研究中,SSR发生频率的差异可能与SSR检索标准、数据库有效性及物种差异有关[20]。本研究表明,花斑裸鲤属于转录组中SSR位点较丰富的鱼类,其多态性较高,这些位点的挖掘能为后期花斑裸鲤遗传多样性和种质资源的保护提供充足的序列资源。
本研究中,花斑裸鲤转录组SSR种类丰富,主要以单核苷酸、二核苷酸和三核苷酸为主,这3类分别占总SSR位点数的46.53%、42.45%和9.09%,这与东海带鱼[18]、双须骨舌鱼(Osteoglossum bicirrhosum)[21]和江鳕(Lota lota)[22]等SSR重复类型以单核苷酸和二核苷酸重复为主的研究结果相似,而与牙鲆(Paralichthys olivaceus)[23]、鮸(Miichthys miiuy)[24]等大多数鱼类SSR重复类型以二核苷酸和三核苷酸重复为主的研究结果不同。本研究中,四、五、六核苷酸出现较少,共占1.94%,SSR位点重复单元的不同在一定程度上与物种进化水平有关,短重复单元占比高说明物种进化水平较高[25]。这表明,花斑裸鲤可能具有较长的进化历史并积累了较多的遗传变异。花斑裸鲤的SSR位点中单核苷酸重复、二核苷酸重复和三核苷酸重复中占优势的碱基重复基元分别为A/T、AC/GT和AAT/ATT。在单碱基重复类型中以A/T类型为主,因为富含A/T的序列退火温度较低,有利于DNA解链,通过DNA复制和滑动增加其出现概率[26]。二碱基重复中以AC/GT为主,这一特点与草鱼(Ctenopharyngodon idella)[27]和长江刀鲚[28]的转录组微卫星序列特征相似。相比而言,CG/CG重复类型非常少,仅占二核苷酸重复的0.16%,可能是CG很容易甲基化使得胞嘧啶脱氨基变成胸腺嘧啶的缘故[29]。本研究中发现,10种三核苷酸重复序列中AAT/ATT最丰富,其次是ATC/ATG、AGG/CCT,这与以往研究报道的齐口裂腹鱼(Schizothorax prenanti)[30]和团头鲂(Megalobrama amblycephala)[31]有所不同。五核苷酸和六核苷酸的优势重复基元在不同物种间差异极大,可能与不同物种的种间差异性有关。
3.1.2 SSR多态性特征 SSR多态性可表现为基元重复次数的不同和基元碱基数不同形成的片段长度差异。一般情况下,随着重复次数的增加,微卫星序列的变异性也会增加,其多态性潜力也就越大。本研究中发现,随着核心序列拷贝数的增加,花斑裸鲤基因组中各重复类型SSR的数目呈递减趋势。推测这一现象产生的原因主要有两点:一是SSR重复单元的重复次数增多会导致其长度相应增加,从而引起稳定性降低、突变率增高等情况,并最终导致该重复类型SSR数目减少[32];二是SSR通常会维持一定的长度范围,长等位基因会倾向于变短,使得SSR长度不能无限增长,因而核心序列拷贝数越多的SSR其数目就会越少[33]。本研究中,对花斑裸鲤转录组数据SSR不同重复类型在不同重复次数下的数量分析表明,花斑裸鲤重复次数为5~35,在各重复类型中,以5~10次重复为主,占总数的63.99%;SSR位点数随重复次数的增加而逐渐减少,重复次数达到20次以上的SSR位点仅有176个,且以单核苷酸为主。若不考虑容易发生错配的单碱基重复类型,重复数最高达到31次,表明花斑裸鲤转录组来源的SSR具有较高的多态性潜能。
多态性的高低是判断SSR可用性的一个重要考察指标,而SSR基元序列长度会影响SSR位点的多态性[34],且序列长度与多态性成正比,当SSR长度≥20 bp时,多态性较高,当12 bp≤SSR长度<20 bp时,多态性中等,当SSR长度<12 bp时,呈低多态性[35]。本研究中,花斑裸鲤SSR序列长度为10~124 bp,其中,序列长度为10~20 bp的SSR数量最多,占比为98.67%,12 bp长度的SSR最多,占比为21.50%,最大的片段长度为四核苷酸重复31次(124 bp)。由此推测,花斑裸鲤转录组SSR多态性是中等的,这与Dreisigacker等[25]发现高级基元SSR多态性普遍比低级基元低的结果相似。在6种核苷酸重复类型中,单核苷酸重复基元和二核苷酸重复基元的重复次数类型最多,可能具有更高的多态性,除单核苷酸重复基元的大部分重复次数集中在10~23次外,二、三、四、五、六核苷酸重复基元的大部分重复次数集中在5~12次,相对低于其他鱼类基因组中的微卫星重复次数,这可能与编码区和非编码区所受到的选择压力差异有关。本研究中,序列长度≥20 bp以上的SSR位点共有8 140个,占总SSR位点数的6.32%,这类SSR位点具有潜在较高的多态性,在后续鉴别花斑裸鲤SSR分子标记开发中具有较大的应用潜力。
3.2 花斑裸鲤SNP和InDel位点特征
本研究中,从花斑裸鲤转录组中共检测到399 080个SNP位点,平均每1 211 bp出现1个SNP位点。理论上,转换和颠换的SNP位点数比值应为0.5,而本研究中转换类型的发生频率是颠换类型的1.29倍,这种现象被称为“转换偏差”[36]。表明转换类型的发生并不是随机产生的,而可能与进化过程的选择机制有关。此外,转换位点和颠换位点的总和与SNP位点总数不相等,原因可能是在一个SNP位点同时发生了转换和颠换两种突变。在花斑裸鲤转录组数据中平均每条Unigene上含有3.88个SNP位点,其中,含有1个SNP位点的Unigenes最多(34.54%),其次是含有2个SNP位点的Unigenes(21.21%),随着SNP位点数的增加,相对应的Unigenes数目逐渐减少,这与其他研究结果相似[37-38]。除SNP位点外,本研究中还检测到254 065个InDel位点,平均每1 903 bp出现1个InDel位点。InDel位点的数量分布特征与SNP大致相同,InDel标记是根据等位基因位点处核苷酸序列发生大小不同插入或缺失引起的长度多态性变异而开发的,由于其具有多态性丰富、可重复性高、变异率低且易于检测等优点,可为后续开展花斑裸鲤优异基因挖掘、遗传图谱构建和遗传多样性分析等研究提供基础数据信息。
4 结论
1)从花斑裸鲤转录组中检测到128 727个SSR位点,包括6个重复类型,主要以单碱基和二碱基重复类型为主;大多数SSR基元的重复次数为5~15次。花斑裸鲤的转录组SSR位点丰富且其进化水平较高。
2)从花斑裸鲤转录组中检测到399 080个SNP位点和254 065个InDel位点,其中,转换类型发生频率高于颠换类型,且SNP位点和InDel位点均以含1个位点的Unigenes数最多,随着Unigene所含的SNP位点数和InDel位点数的增加,Unigenne数量呈递减趋势。
3)花斑裸鲤转录组中SSR、SNP和InDel位点数量多,出现频率高,类型丰富,具有较高的多态性潜能,后期可通过进一步的设计、筛选引物,为花斑裸鲤遗传多样性分析、亲缘关系鉴定与遗传资源开发利用等方面提供丰富的基础数据信息。
[1] 李薇,李久煊,荆慧芳,等.基于高通量测序的达氏鲟微卫星标记筛选[J].动物学杂志,2017,52(3):449-457.
LI W,LI J X,JING H F,et al.Development of microsatellite loci for Dabry’s sturgeon(Acipenser dabryanus) using high-throughput sequencing[J].Chinese Journal of Zoology,2017,52(3):449-457.(in Chinese)
[2] 高峰涛,邵长伟,崔忠凯,等.基于高通量测序的青石斑鱼基因组微卫星开发及评价[J].中国海洋大学学报(自然科学版),2017,47(4):52-57.
GAO F T,SHAO C W,CUI Z K,et al.Development and population genetic diversity analysis of microsatellite markers in Epinephelus awoara[J].Periodical of Ocean University of China(Nature Science Edition),2017,47(4):52-57.(in Chinese)
[3] 王朝溪.青海湖裸鲤AFLP指纹图谱的构建以及与生长相关SNP位点的筛选[D].西宁:青海大学,2015.
WANG C X.The establishment of AFLP fingerprinting and SNP markers screening associated with growth traits of Gymnocypris przwalskii[D].Xining:Qinghai University,2015.(in Chinese)
[4] 全迎春,马冬梅,白俊杰,等.大口黑鲈转录组SNPs筛选及其与生长的关联分析[J].水生生物学报,2016,40(6):1128-1134.
QUAN Y C,MA D M,BAI J J,et al.SNPs identification in RNA-Seq data of largemouth bass(Micropterus salmoides) fed on formulated feed and association analysis with growth trait[J].Acta Hydrobiologica Sinica,2016,40(6):1128-1134.(in Chinese)
[5] 曹欣,张弓.超高精度大规模测序Mapping算法FANSe在非模式生物中的应用[J].中国科学:生命科学,2017,47(7):702-707.
CAO X,ZHANG G.Application of the hyper-accurate Mapping algorithm FANSe for next-generation sequencing in non-model organisms[J].Scientia Sinica (Vitae),2017,47(7):702-707.(in Chinese)
[6] 梁晓涵,刘婷婷,张烨,等.基于二代测序的集群分离分析法在木本植物基因定位中的应用[J].世界林业研究,2020,33(2):54-61.
LIANG X H,LIU T T,ZHANG Y,et al.Application of next-generation sequencing based bulked segregant analysis in gene mapping of woody plants[J].World Forestry Research,2020,33(2):54-61.(in Chinese)
[7] 申志新,王思德,王国杰,等.花斑裸鲤在3种环境中的驯化养殖试验[J].水生态学杂志,2011,32(1):149-152.
SHEN Z X,WANG S D,WANG G J,et al.The experiment of domestication and artificial breeding of piebald naked carp in three circumstances[J].Journal of Hydroecology,2011,32(1):149-152.(in Chinese)
[8] 韩永.黄河上游花斑裸鲤Gymnocypris eckloni年龄、生长和种群遗传特性分析[D].上海:上海海洋大学,2016.
HAN Y.Analysis on age,growth,population genetic characters and conservation of Gymnocypris eckloni in upper yellow river[D].Shanghai:Shanghai Ocean University,2016.(in Chinese)
[9] 赵凯,李俊兵,杨公社,等.青海湖及其相邻水系特有裸鲤属鱼类的分子系统发育[J].科学通报,2005,50(13):1348-1355.
ZHAO K,LI J B,YANG G S,et al.Molecular phylogeny of gymnosperms endemic to Qinghai Lake and its adjacent water systems[J].Chinese Science Bulletin,2005,50(13):1348-1355.(in Chinese)
[10] 祁得林.黄河上游花斑裸鲤Cyt b基因的序列变异和遗传多样性[J].动物学研究,2009,30(3):255-261.
QI D L.Genetic variation and diversity of Gymncypris eckloni in the upper Yellow River inferred from mitochondrial Cytochrome b gene[J].Zoological Research,2009,30(3):255-261.(in Chinese)
[11] 唐文家,赵淑梅,袁军,等.花斑裸鲤人工繁育技术[J].科学养鱼,2013(7):9-10.
TANG W J,ZHAO S M,YUAN J,et al.Artificial breeding techniques of naked carp spotted[J].Scientific Fish Farming,2013(7):9-10.(in Chinese)
[12] 董艳珍,邓思红,肖文渊.花斑裸鲤的胚胎发育观察[J].江苏农业科学,2018,46(6):142-144.
DONG Y Z,DENG S H,XIAO W Y.Observation on embryo development of Gymnocypris eckloni Herzensten[J].Jiangsu Agricultural Sciences,2018,46(6):142-144.(in Chinese)
[13] 鄢思利.花斑裸鲤的生物学特性、繁殖特性、胚胎发育及人工培育的研究[D].南充:西华师范大学,2016.
YAN S L.Study on the biology of Gymnocypris eckloni and the research of reproductive property,embryo and artificial cultivation[D].Nanchong:China West Normal University,2016.(in Chinese)
[14] EDWARDS Y J K,ELGAR G,CLARK M S,et al.The identification and characterization of microsatellites in the compact genome of the Japanese pufferfish,Fugu rubripes:perspectives in functional and comparative genomic analyses 1[J].Journal of Molecular Biology,1998,278(4):843-854.
[15] FANG D A,ZHOU Y F,DUAN J R,et al.Screening potential SSR markers of the anadromous fish Coilia nasus by de novo transcriptome analysis using Illumina sequencing[J].Genetics and Molecular Research,2015,14(4):14181-14188.
[16] WANG D,LIAO X L,CHENG L,et al.Development of novel EST-SSR markers in common carp by data mining from public EST sequences[J].Aquaculture,2007,271:558-574.
[17] 袁文成,黄鹤忠,李文龙,等.翘嘴鳜(Siniperca chuatsi)转录组EST-SSR位点的信息分析及其多态性检测[J].海洋与湖沼,2015,46(2):403-409.
YUAN W C,HUANG H Z,LI W L,et al.Analysis of EST-SSRs information in Siniperca chuatsi transcriptome and detection of polymorphism[J].Oceanologia et Limnologia Sinica,2015,46(2):403-409.(in Chinese)
[18] 章霞,柳敏海,李凌刚,等.东海带鱼(Trichiurus japanicus)肝脏转录组SSR和SNP特征分析[J].渔业研究,2019,41(4):269-277.
ZHANG X,LIU M H,LI L G,et al.SSR and SNP analysis based on Trichiurus japanicus transcriptome[J].Journal of Fisheries Research,2019,41(4):269-277.(in Chinese)
[19] 龚诗琦,王志勇,肖世俊,等.黄姑鱼转录组SSR的开发与验证[J].集美大学学报(自然科学版),2016,21(4):241-246.
GONG S Q,WANG Z Y,XIAO S J,et al.Development and verification of SSR based on transcriptome of yellow drum,Nibea albiflora[J].Journal of Jimei University (Natural Science Edition),2016,21(4):241-246.(in Chinese)
[20] DENG K P,DENG R J,FAN J X,et al.Transcriptome analysis and development of simple sequence repeat (SSR) markers in Zingiber striolatum Diels[J].Physiology and Molecular Biology of Plants,2018,24(1):125-134.
[21] 王且鲁,刘奕,宋红梅,等.双须骨舌鱼转录组EST-SSR标记开发与引物筛选[J].淡水渔业,2016,46(6):8-13.
WANG Q L,LIU Y,SONG H M,et al.Development and primer selection of EST-SSR molecular markers based on transcriptome sequencing of Osteoglossum bicirrhosum[J].Freshwater Fisheries,2016,46(6):8-13.(in Chinese)
[22] 孟玮,蒋艳琳,张林,等.基于RNA-Seq技术的江鳕转录组SSR位点信息分析[J].淡水渔业,2019,49(6):10-14.
MENG W,JIANG Y L,ZHANG L,et al.SSR loci information analysis in the transcriptome of burbot(Lota lota) based on RNA-Seq[J].Freshwater Fisheries,2019,49(6):10-14.(in Chinese)
[23] 陈松波,龚丽,刘海金.牙鲆EST资源的SSR信息分析[J].东北农业大学学报,2010,41(10):82-86.
CHEN S B,GONG L,LIU H J.Analysis of SSR information in EST resources of Japanese flounder(Paralichthys olivaceus)[J].Journal of Northeast Agricultural University,2010,41(10):82-86.(in Chinese)
[24] 孙典巧,孙悦娜,王日昕,等.鮸鱼EST序列中微卫星标记的初步筛选及特征分析[J].水生生物学报,2011,35(5):753-760.
SUN D Q,SUN Y N,WANG R X,et al.The characteristic analysis of microsatellites from ESTs in Miichthys miiuy[J].Acta Hydrobiologica Sinica,2011,35(5):753-760.(in Chinese)
[25] DREISIGACKER S,ZHANG P,WARBURTON M L,et al.SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments[J].Crop Science,2004,44(2):381-388.
[26] 倪守胜,杨钰,柳淑芳,等.基于高通量测序的虾夷扇贝基因组微卫星特征分析[J].渔业科学进展,2018,39(1):107-113.
NI S S,YANG Y,LIU S F,et al.Microsatellite analysis of Patinopecten yessoensis using next-generation sequencing method[J].Progress in Fishery Sciences,2018,39(1):107-113.(in Chinese)
[27] WANG D,LIAO X L,CHENG L,et al.Development of novel EST-SSR markers in common carp by data mining from public EST sequences[J].Aquaculture,2007,271(1/2/3/4):558-574.
[28] 于爱清,施永海,徐嘉波,等.长江刀鲚选育群体转录组EST-SSR的分布特征分析[J].渔业科学进展,2019,40(5):101-109.
YU A Q,SHI Y H,XU J B,et al.Characteristic analysis of microsatellites in selected Coilia ectenes using a transcriptome dataset[J].Progress in Fishery Sciences,2019,40(5):101-109.(in Chinese)
[29] YOON J M.Genetic variations between hairtail (Trichiurus lepturus) populations from Korea and China[J].Development &Reproduciton,2013,17(4):363-367.
[30] LUO H,XIAO S J,YE H,et al.Identification of immune-related genes and development of SSR/SNP markers from the spleen transcriptome of Schizothorax prenanti[J].PLoS One,2016,11(3):e0152572.
[31] GAO Z X,LUO W,LIU H,et al.Transcriptome analysis and SSR/SNP markers information of the blunt snout bream (Megalobrama amblycephala)[J].PLoS One,2012,7(8):e42637.
[32] WIERDL M,DOMINSKA M,PETES T D.Microsatellite instability in yeast:dependence on the length of the microsatellite[J].Genetics,1997,146(3):769-779.
[33] ELLEGREN H.Heterogeneous mutation processes in human microsatellite DNA sequences[J].Nature Genetics,2000,24(4):400-402.
[34] MEGL CZ E,N
CZ E,N VE G,BIFFIN E,et al.Breakdown of phylogenetic signal:a survey of microsatellite densities in 454 shotgun sequences from 154 non model eukaryote species[J].PLoS One,2012,7(7):e40861.
VE G,BIFFIN E,et al.Breakdown of phylogenetic signal:a survey of microsatellite densities in 454 shotgun sequences from 154 non model eukaryote species[J].PLoS One,2012,7(7):e40861.
[35] TEMNYKH S,DECLERCK G,LUKASHOVA A,et al.Computational and experimental analysis of microsatellites in rice (Oryza sativa L.):frequency,length variation,transposon associations,and genetic marker potential[J].Genome Research,2001,11(8):1441-1452.
[36] ZHAO H,LI Q Z,LI J,et al.The study of neighboring nucleotide composition and transition/transversion bias[J].Science in China Series C,Life Sciences,2006,49(4):395-402.
[37] 刘华伟,李朝绪,李芬,等.基于转录组测序的椰心叶甲啮小蜂SSR、SNP和InDel位点分析[J].热带作物学报,2021,42(10):2828-2833.
LIU H W,LI C X,LI F,et al.SSR,SNP and InDel analysis based on Tetrastichus brontispae transcriptome[J].Chinese Journal of Tropical Crops,2021,42(10):2828-2833.(in Chinese)
[38] 王艺儒,索玉静,傅建敏.小果甜柿果实转录组的SSR、SNP和InDel特征分析[J].西北农林科技大学学报(自然科学版),2022,50(7):147-154.
WANG Y R,SUO Y J,FU J M.SSR,SNP and InDel analysis based on transcriptome data of Diospyros kaki ‘Xiaoguo-Tianshi’ fruit[J].Journal of Northwest A &F University (Natural Science Edition),2022,50(7):147-154.(in Chinese)