HOX基因属于同源异形盒(Homeobox)家族,是一类高度保守的同源结构域转录因子。HOX蛋白的同源盒结构域HD(homeodomain)可以通过调节靶基因的转录影响多细胞动物胚胎发育过程中身体前后体轴的形成。当该基因表达失去控制时,可能会引起个体发育过程中组织器官形态结构或者功能的异常。HOX蛋白可折叠成3个螺旋,其中,螺旋2和3产生螺旋-转角-螺旋构象,这一构象使其能与DNA稳定结合,发挥转录因子的作用[1-2]。1978年,Lewis[3]在研究果蝇(Drosophila melanogaster)同源异型突变过程中,首次发现了与个体发育有关的HOX基因。后续研究发现,几乎所有真核生物都含有HOX基因,其在染色体上成簇排列,簇的数量与进化过程中的全基因组复制有关[4]。根据其在单一HOX基因簇上的位置,HOX基因被分为4组,Anterior(如脊索动物PG1、PG2)、Group 3(如脊索动物PG3)、Central(如脊索动物PG4~PG8)和Posterior(如脊索动物PG9~PG13)[2,5-6]。
双壳贝类的HOX基因簇通常含有11个HOX基因[2],但在长牡蛎(Crassostrea gigas)中仅有10个,依次为CgHOX1、CgHOX2、CgHOX3、CgHOX4、CgHOX5、CgLOX5、CgLOX4(Lophotrochozoa HOX4)、CgLOX2、CgPOST2和CgPOST1,缺失存在于其他贝类中的ANTP基因[7]。
HOXA7在调节生物体发育中发挥重要作用。如在正常卵母细胞及人类(Homo sapiens)胎儿的肝脏和肾脏中都能检测到HsHOXA7的表达[8-9]。猪(Sus scrofa)的SsHOXA7在胚胎4细胞期和囊胚期表达[10]。进一步研究表明,敲降HsHOXA7表达可以破坏人类胎儿肝脏造血干细胞(FL-HSPCs)的造血功能[11]。小鼠(Mus musculus)的MmHOXA7可以与MmHOXA9、MmRUNX1协同作用促进造血祖细胞的产生[12]。低等动物的LOX4与高等动物的HOXA7高度同源,也参与生物体的发育过程。如环节动物门寡毛纲正颤蚓(Tubifex tubifex)的Ttlab、TtScr1和TtLOX5仅在腹神经索中表达;TtScr2和TtLOX4不仅在幼虫的腹神经索中表达,而且在外侧组织中也有表达;TtDfd和TtLOX2在腹神经索、外侧组织和口前叶都有表达[13]。夏威夷短尾乌贼(Euprymna scolopes)EsLOX4在幼虫的足神经节中表达[14]。腹足类蜗牛(Gibbula varia)的GvLOX4在担轮幼虫的口前纤毛环和面盘幼虫的面盘中表达[15]。在长牡蛎的原肠胚时期,CgLOX4主要分布在壳腺中心区域[16]。以上研究表明,软体动物LOX4可能参与早期幼虫神经细胞、纤毛及壳的发育与分化。
海洋双壳贝类作为沿海地区的重要养殖物种,具有极高的经济价值。但近年来养殖贝类幼虫常出现死亡现象,严重制约了贝类养殖产业的健康发展。据估计,在自然海区中只有不到5%的双壳贝类幼虫能够发育为成体。前期研究发现,高等动物中HOXA7的异常表达会导致多种癌症的发生且与预后不良有关。在低等动物中,LOX4参与幼虫壳、神经系统等的形成,在幼虫发育中起到重要作用。本研究中,利用生物信息学分析、荧光定量PCR和整体免疫荧光技术分析了CgLOX4的结构特征及其在幼虫不同发育时期的mRNA和蛋白表达特征,揭示了CgLOX4在长牡蛎幼虫发育中的作用,以期为深入了解贝类幼虫发育机制提供数据支撑。
1 材料与方法
1.1 材料
试验用50只成体长牡蛎(平均体质量为110 g)购于辽宁省大连市长兴市场,饲养于新鲜海水中,每天换1/2新鲜海水,每日投喂用48 μm筛绢过滤的螺旋藻粉。雌性小鼠购自大连市药检所,平均体质量约25 g,许可证号SCXK(辽)2020-0001,批号20220302031。
1.2 方法
1.2.1 样品的采集与处理 幼虫样本:饲养1周后随机选取20只健康长牡蛎,解剖采集精子和卵细胞,置于新鲜海水中受精。受精卵在25 ℃水温下发育。分别采集发育至受精卵、4细胞期、8细胞期、桑椹胚期、囊胚期、原肠胚期、担轮幼虫期和D型幼虫期的幼虫样品,于-80 ℃超低温冰箱中保存。用于整体免疫荧光的样品在不同梯度甲醇中脱水,并在-20 ℃无水甲醇中保存。
成体样本:随机选取9只长牡蛎,解剖活体取其外套膜、鳃、血淋巴、闭壳肌、性腺、唇瓣和肝胰腺,分别加入500 μL Trizol,摇匀后经液氮速冻后于-80 ℃超低温冰箱中保存。
1.2.2 RNA提取和cDNA合成 使用TransZol Up Plus RNA Kit(北京全式金生物技术股份有限公司)提取牡蛎各组织样品及早期各发育阶段样品的总RNA。通过NanoDrop 2000和10 g/L琼脂糖凝胶电泳对所提取的RNA样品进行定量和定性检测。将质量合格的RNA于-80 ℃超低温冰箱中保存,用于后续cDNA的合成。
参照TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix反转录试剂盒(北京全式金生物技术股份有限公司)说明书配制混合体系,使用PCR仪进行反转录,用于扩增CgLOX4序列。反应体系(20 μL):gDNA Removal 1 μL,Anchored Oligo(dT)18 Primer(0.5 μg/μL)1 μL,2×Ts Reaction Mix 10 μL,TransScript qRT/RI Enzyme Mix 1 μL,Total RNA/mRNA 1 μL,RNase-Free ddH2O 6 μL。若cDNA产物用于qRT-PCR,则在42 ℃下反应15 min;若cDNA产物用于PCR,则在42 ℃下反应30 min。之后在85 ℃下加热5 s,使TransScript® qRT/RI Enzyme Mix和gDNA Removal失活。最终产物保存至-80 ℃超低温冰箱中,合成的cDNA产物用于CgLOX4全长cDNA克隆和qRT-PCR试验。
1.2.3 CgLOX4基因克隆 CgLOX4(CGI_10026562)序列信息来自NCBI数据库(http://www.ncbi.nlm.nih.gov)。利用Primer Premier 5.0软件设计一对基因特异性引物(LOX4-F和LOX4-R)扩增CgLOX4的开放阅读框(ORF)(表1)。PCR反应体系(25 μL):10×PCR Buffer 2.5 μL,dNTP Mix 2 μL,MgCl2 1.5 μL,LOX4-F 1 μL,LOX4-R 1 μL,cDNA 1 μL,DEPC水 15.85 μL,DNA聚合酶 0.15 μL。PCR反应条件:94 ℃下预变性5 min;94 ℃下变性30 s,54 ℃下退火30 s,72 ℃下延伸50 s,共进行35个循环;最后在72 ℃下再延伸10 min。扩增产物经琼脂糖凝胶电泳分离后,使用MiniBEST琼脂糖凝胶DNA提取试剂盒(日本TaKaRa),将切下的琼脂糖凝胶溶解并纯化。纯化产物与pMD19-T载体(日本TaKaRa)连接,连接产物转入 Trans 5α感受态(北京全式金生物技术股份有限公司)中,37 ℃下摇床振荡2 h后,将其均匀涂于含有氨苄抗性的固体培养基上,37 ℃恒温培养箱中正置培养1 h后,将培养皿翻转,倒置培养16 h。在超净台中挑取单菌落于加有DEPC水的离心管中,吹打混匀。吸取1 μL混合均匀的菌液并配制菌落PCR体系,进行菌落PCR扩增。PCR反应体系(11 μL):PCR Master Mix 5 μL,DEPC水3 μL,LOX4-Ex-F 1 μL,LOX4-Ex-R 1 μL,模板1 μL。PCR反应条件:95 ℃下预变性5 min;95 ℃下变性30 s,58 ℃下退火30 s,72 ℃下延伸50 s,共进行25个循环;最后在72 ℃下再延伸10 min。向剩余菌液中加入700 μL含有氨苄抗性的LB培养液,37 ℃下摇床振荡培养4 h。通过琼脂糖凝胶电泳分析菌落PCR产物,筛选呈现阳性信号的菌株送至生工生物工程(上海)股份有限公司进行测序。将测序结果正确的菌液扩培并提取质粒,然后转化至Transetta(DE3)感受态菌株中,37 ℃摇床振荡2 h后将其均匀涂于含有氨苄抗性的固体LB培养基上,后续处理步骤与转入Trans 5α感受态菌株中相同。
表1 试验用引物
Tab.1 Sequences of primers used in this study

引物 primer序列 sequence (5′-3′)功能 functionLOX4-RTF:CACTGACAACTGGAGTCCCCqRT-PCRR:GTGTGCCGAGTTAGGACCAAEF-RTF:AGTCACCAAGGCTGCACAGAAAGqRT-PCRR:TCCGACGTA TTTCTTTGCGATGTLOX4F:ATGAACTCCTACTACCCGGCC基因克隆R:CGGTGACTCGTCACTAGACAGLOX4-ExF:CGCGGATCCATGAACTCCTACTACCCGGCC蛋白表达R:CCGCTCGAGCGGTGACTCGTCACTAGACAG
1.2.4 CgLOX4生物信息学分析 用BLAST(http://www.ncbi.nlm.nih.gov/blast)和Expert Protein Analyst System(http://www. expasy.org)程序对得到的CgLOX4 cDNA序列和推导的氨基酸序列进行分析[17]。用SMART(http://smart.embl-heidelberg. de)预测蛋白结构域[18]。使用ClustalW多重比对程序(http://www.ebi.ac.uk/clustalw)创建多序列比对[18]。采用MEGA 6.0软件构建系统发生树[19]。利用一级蛋白序列数据库Uniprot(https://www.uniprot.org)分析蛋白序列[20]。利用ExPASy(https://web.expasy.org/protscale/)对蛋白序列进行亲疏水性分析[19]。
1.2.5 实时荧光定量(qRT-PCR)分析 根据CgLOX4 cDNA全长序列,使用Primer Premier 5.0软件设计qRT-PCR引物LOX4-RT-F和LOX4-RT-R(表1)。在Quant Studio 6实时荧光定量PCR仪进行qRT-PCR,检测CgLOX4在成体长牡蛎不同组织(鳃、外套膜、血淋巴、闭壳肌、性腺、唇瓣和肝胰腺)和幼虫不同发育阶段(受精卵、4细胞期、8细胞期、桑椹胚期、囊胚期、原肠胚期、担轮幼虫期和D型幼虫期)的表达情况,以EF(NP_001292242.2)为内参基因。qRT-PCR反应体系(10 μL):2×SYBR Green Master Mix 5 μL,ROX Reference Dye(50×) 0.2 μL,LOX4-RT-F 0.2 μL,LOX4-RT-R 0.2 μL,20倍稀释的cDNA模板2 μL,DEPC水2.4 μL。反应程序:95 ℃下预变性10 min,95 ℃下变性10 s,60 ℃下退火45 s,共进行40个循环。来自3只牡蛎的组织混合在一起作为一个样品,每个样品设置3个生物学重复。采用2-ΔΔCt法计算基因的表达量[21]。
1.2.6 CgLOX4的重组蛋白表达和纯化 CgLOX4重组蛋白的表达和纯化按照Jiang等[22]的方法并略做修改。用引物LOX4-Ex-F和LOX4-Ex-R(分别含有BamH Ⅰ和Xho Ⅰ内切酶切点)(表1)扩增CgLOX4的编码区。扩增产物经BamH Ⅰ和Xho Ⅰ核酸酶酶切和凝胶纯化后,连接到含有组氨酸标签的pET-30a表达载体(TRAN,中国)中。将构建的重组表达载体pET-30a-LOX4转化到大肠杆菌Transetta(DE3)(TRAN,中国)。对携带pET-30a-LOX4的阳性菌株DE3进行DNA测序。CgLOX4重组蛋白的诱导表达和纯化步骤参照文献[23]中的方法。将纯化的CgLOX4重组蛋白保存于-80 ℃超低温冰箱中用于后续试验。
1.2.7 CgLOX4抗体制备及蛋白免疫印迹分析 取纯化的重组蛋白(100 μg)用TBS稀释至100 μL,与完全弗氏佐剂(100 μL)混合。用乳化后的混合物皮下注射小鼠4次(每周1次),第4次注射1周后采集并处理小鼠血样,室温下静置4 h获得的血清即为多克隆抗体。利用蛋白免疫印迹分析检测所制备抗体的特异性。蛋白免疫印迹分析方法与前期报道相同[24]。
1.2.8 整体免疫荧光分析 将保种管底部剪掉,与48 μm筛绢黏合,置于24孔板中。分别取1 mL -20 ℃下冷冻保存的担轮幼虫、D型幼虫和壳顶幼虫样品置于剪掉底部的保种管中,并逐步复水至PBST。用PBST浸洗3次,每次5 min,0.5% TritonX-100透膜20 min,0.05%胰蛋白酶液抗原修复20 min;用PBST浸洗3次,每次15 min,Blocking Buffer浸没筛绢,室温封闭2 h,加入800 μL CgLOX4抗体(按照1∶400在Blocking Buffer中稀释),4 ℃下孵育过夜;用PBST浸洗3次,每次30 min,加入800 μL DAPI,染色5 min;用PBST浸洗3次,每次30 min,加入800 μL荧光标记的二抗(按照1∶400在Blocking Buffer中稀释),室温下慢摇孵育1 d;用PBST浸洗3次,每次30 min,依次经1 mL体积分数为70%、85%、95%、100%的异丙醇处理,每次30 s,再用1 mL Murry’s clear(由benzyl benzoate与benzyl alcohol按体积比为2∶1配制而成)处理3次,每次30 s;最后在载玻片上,滴加20 μL 用Murry’s clear处理的样品,封片,并将载玻片置于激光共聚焦显微镜下进行观察[25]。
1.3 数据处理
试验结果均以平均值±标准差(mean±S.D.)表示,并通过IBM SPSS Statistics 20软件进行单因素方差分析(one-way ANOVA)和LSD法多重比较,显著性水平设为0.05。
2 结果与分析
2.1 CgLOX4基因克隆及序列比对
CgLOX4全长为1 862 bp,包括99 bp的5′非编码区(UTR)、834 bp的开放阅读框(ORF)和929 bp的3′非编码区,可编码277个氨基酸,预测蛋白相对分子质量为31 574,等电点为8.36。该序列已录入GenBank,基因号为CGI_10026562。CgLOX4仅包含一个保守的HOX结构域(图1)。ExPASy亲疏水性图谱显示,CgLOX4具有亲水性(图2)。UniProt分析显示,CgLOX4定位于细胞核中(图3(a))。蛋白的三维结构预测显示,CgLOX4蛋白结构与人的HsHOXA7和果蝇的Dmabd-A高度相似(图3(b)、 (c)),CgLOX4蛋白折叠成3个α螺旋(图3(d))。

加粗表示起始密码子(ATG)及终止密码子(TAA),阴影区表示CgLOX4结构域。
Start (ATG) and stop codons (TAA) are shown in bold,and CgLOX4 domain is shaded in light gray.
图1 CgLOX4的cDNA序列及其推导的氨基酸序列和结构域
Fig.1 cDNA sequence,deduced amino acid sequence and domain of CgLOX4
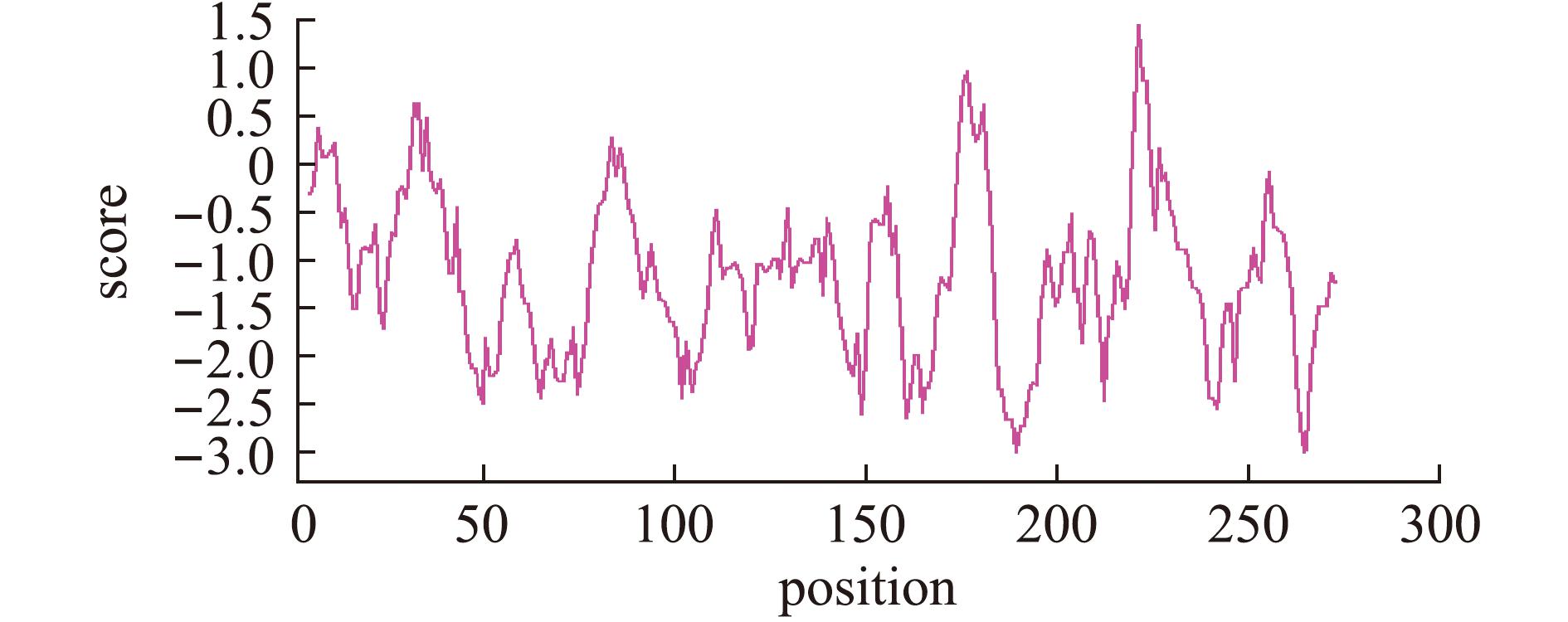
正值表示疏水性,负值表示亲水性。
The positive value indicates hydrophobicity,and negative value indicates hydrophilicity.
图2 CgLOX4亲疏水性分析
Fig.2 Hydrophilicity analysis of CgLOX4
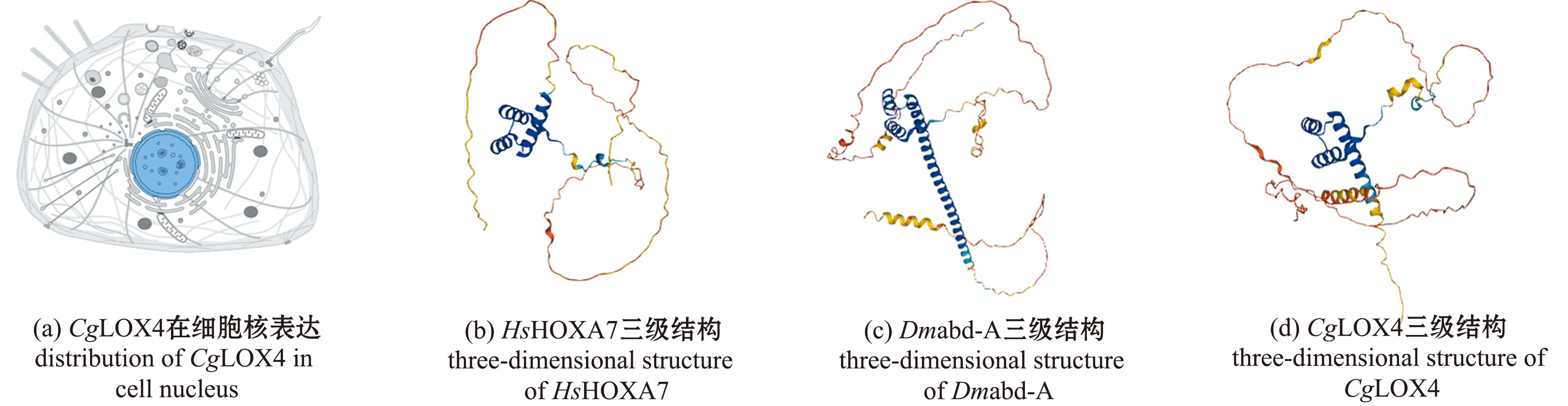
图3 LOX4蛋白序列的UniProt分析
Fig.3 UniProt analysis of LOX4 protein sequence
2.2 CgLOX4的多序列比对和系统进化树
用于多序列比对的17个物种及其LOX4(或HOXA7、abd-A)氨基酸序列在NCBI中的登录号见表2。氨基酸序列比对结果显示,CgLOX4与笠贝(Lottia goshimai)LgLOX4、南美白对虾(Penaeus vannamei)PvHOXA7氨基酸序列的相似性较高(分别为54.2%和38.5%),CgLOX4具有HOX家族特有的HOX结构域(图4)。系统进化分析显示,CgLOX4首先与石鳖(Acanthochitona crinita)和鸭嘴海豆芽(Lingula anatine)的LOX4聚在一起,然后与沙虫(Alitta virens)、海蜗牛(Steromphala varia)和笠贝聚在一起(图5)。

蓝色区域表示同源性氨基酸;粉色区域表示同源性>75%;红色方框表示LOX4保守的HOX结构域。
Blue areas indicate homologous amino acids;Pink indicates >75% homology;Red box indicates the conserved HOX domain of LOX4.
图4 长牡蛎与其他物种LOX4氨基酸的多重序列比对
Fig.4 Multiple sequence alignment of amino acids of LOX4 from Crassostrea gigas and other species
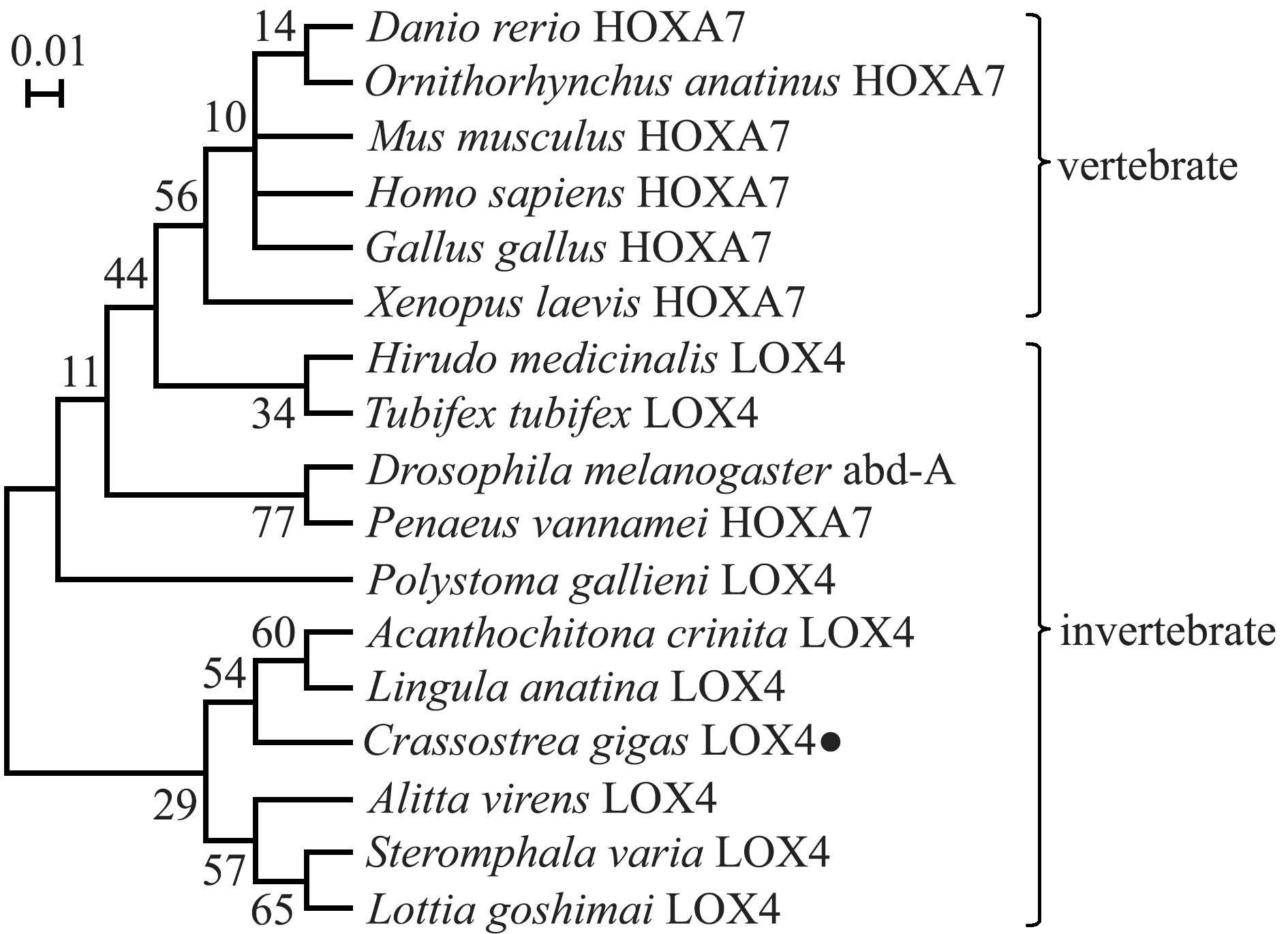
图5 不同物种LOX4氨基酸序列的系统进化树
Fig.5 Phylogenetic tree of the amino acid sequences of LOX4 in different species
表2 用于氨基酸序列同源比对和系统进化树分析的LOX4序列信息
Tab.2 Amino acid sequence homology alignment and phylogenetic analysis of LOX4
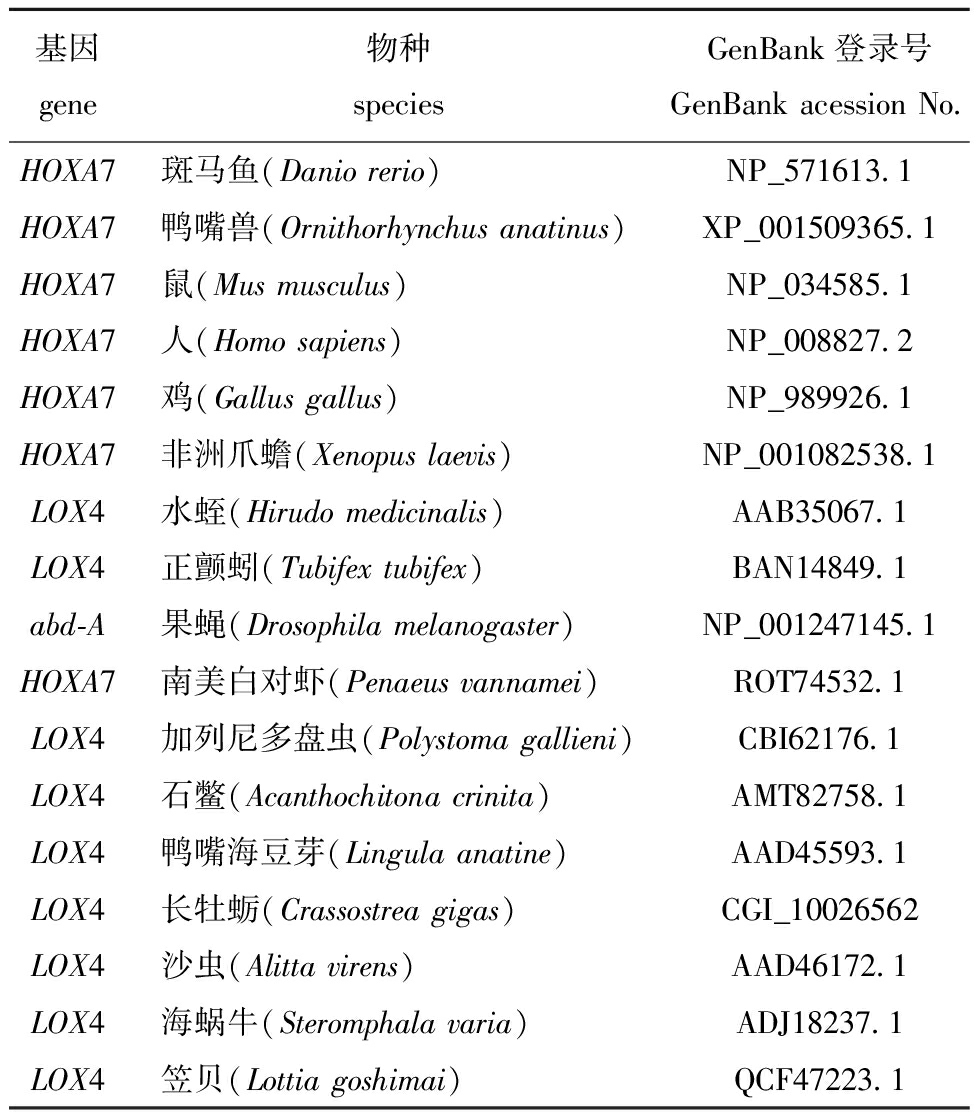
基因gene物种speciesGenBank登录号GenBank acession No.HOXA7斑马鱼(Danio rerio)NP_571613.1HOXA7鸭嘴兽(Ornithorhynchus anatinus)XP_001509365.1HOXA7鼠(Mus musculus)NP_034585.1HOXA7人(Homo sapiens)NP_008827.2HOXA7鸡(Gallus gallus)NP_989926.1HOXA7非洲爪蟾(Xenopus laevis)NP_001082538.1LOX4水蛭(Hirudo medicinalis)AAB35067.1LOX4正颤蚓(Tubifex tubifex)BAN14849.1abd-A果蝇(Drosophila melanogaster)NP_001247145.1HOXA7南美白对虾(Penaeus vannamei)ROT74532.1LOX4加列尼多盘虫(Polystoma gallieni)CBI62176.1LOX4石鳖(Acanthochitona crinita)AMT82758.1LOX4鸭嘴海豆芽(Lingula anatine)AAD45593.1LOX4长牡蛎(Crassostrea gigas)CGI_10026562LOX4沙虫(Alitta virens)AAD46172.1LOX4海蜗牛(Steromphala varia)ADJ18237.1LOX4笠贝(Lottia goshimai)QCF47223.1
2.3 CgLOX4基因在长牡蛎不同组织中的表达
从图6可见,CgLOX4在长牡蛎7个组织中均有表达,其中,在闭壳肌和外套膜中表达量最高,在肝胰腺中表达量最低,且CgLOX4在闭壳肌和外套膜的表达量与肝胰腺相比具有显著性差异(P<0.05),分别为肝胰腺的8.34倍和6.67倍。
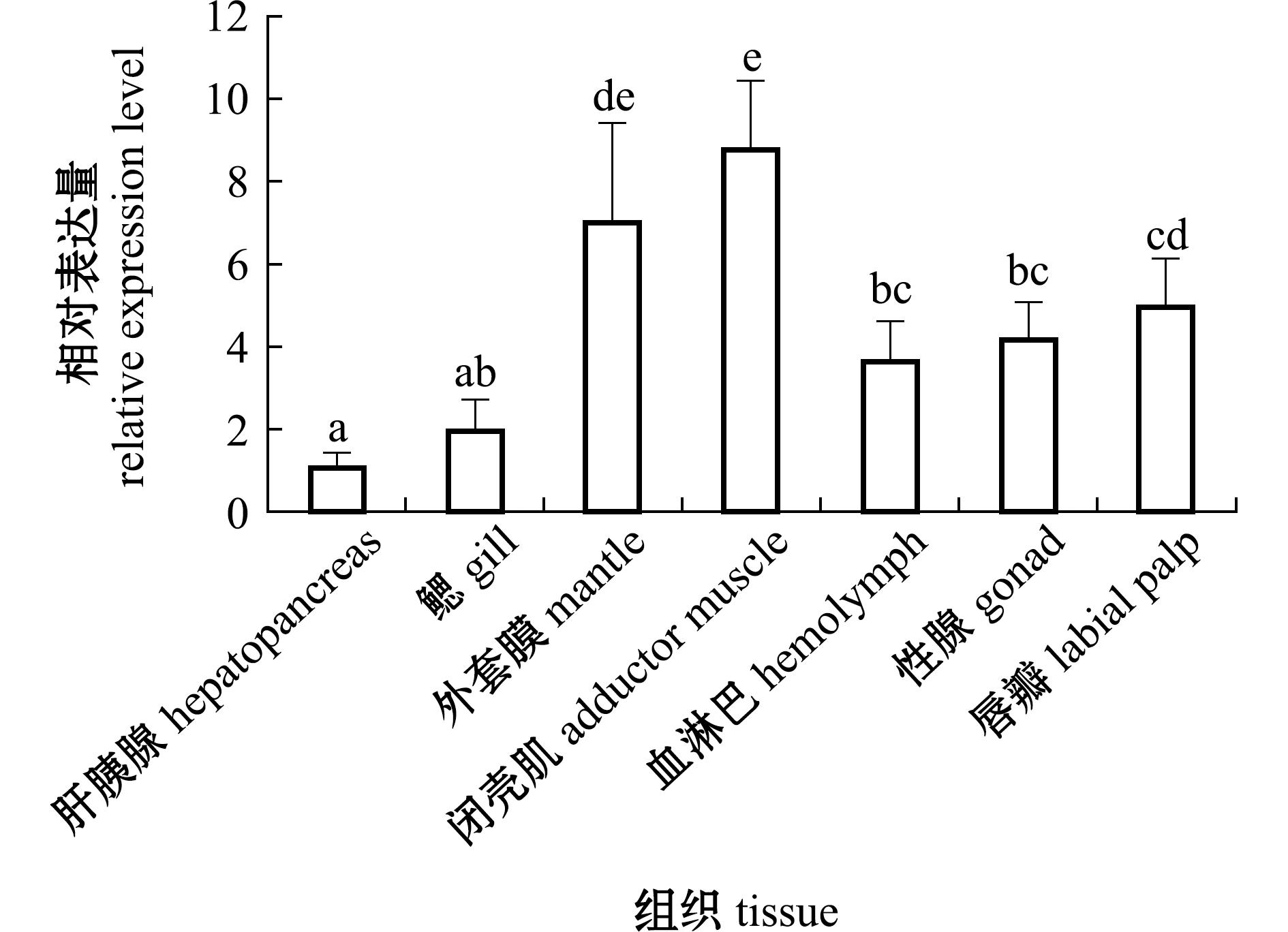
标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P > 0.05),下同。
The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter are not significant differences,et sequentia.
图6 CgLOX4在长牡蛎成体组织中的表达模式
Fig.6 Expressions of CgLOX4 in different tissues
2.4 CgLOX4基因在长牡蛎早期发育过程中的表达
从图7可见,CgLOX4在长牡蛎整个早期发育阶段均有表达,但表达水平具有差异性,其中,CgLOX4在受精卵和4细胞期的表达量显著高于其他时期P<0.05),分别是担轮幼虫期的9.95倍和39.79倍。
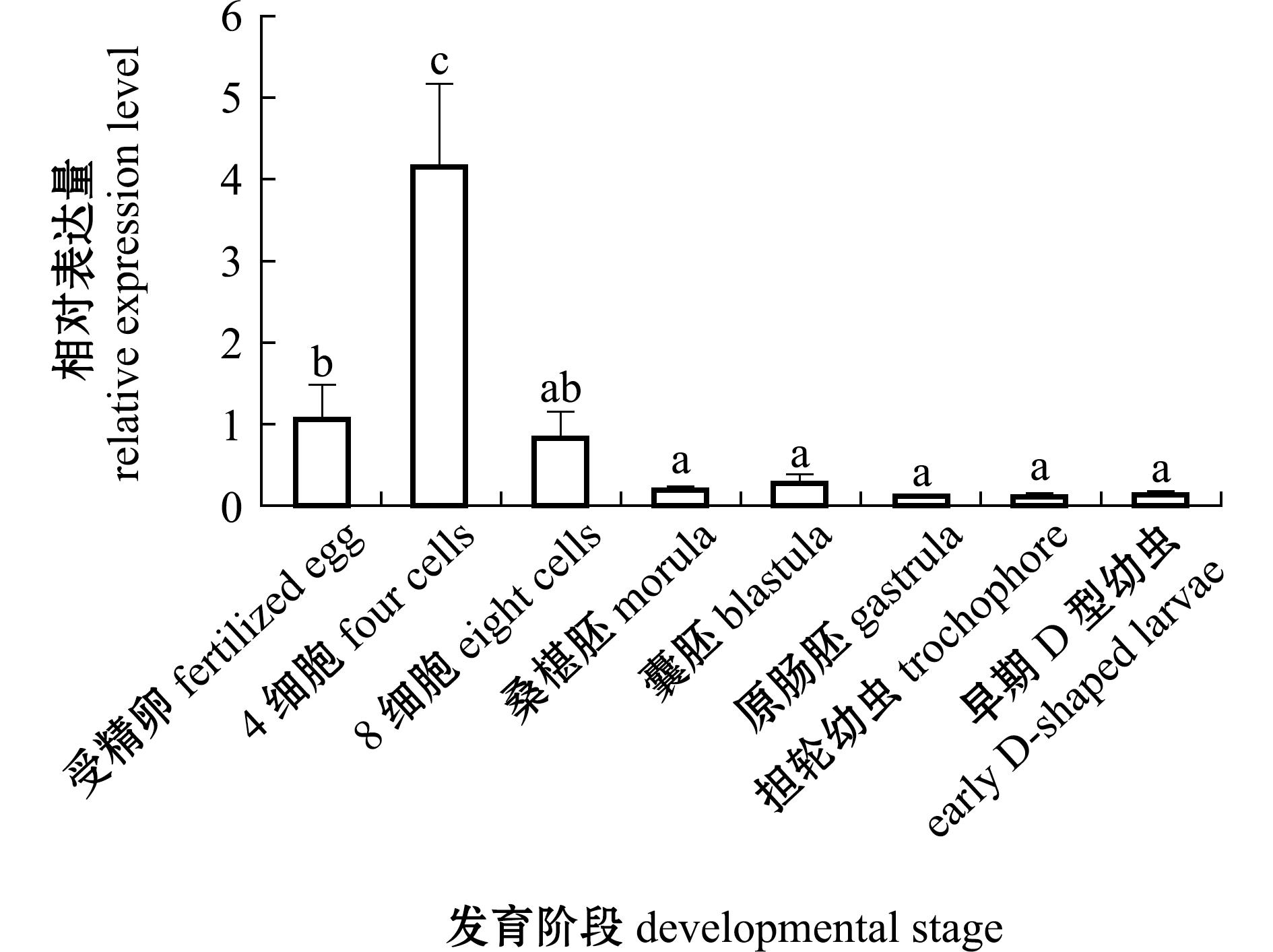
图7 CgLOX4在长牡蛎不同发育阶段的表达
Fig.7 Expressions level of CgLOX4 in the different developmental stages of Crassostrea gigas
2.5 CgLOX4重组蛋白表达、纯化及其多克隆抗体的制备
将克隆得到的CgLOX4基因片段分别连接到pET-30a载体上,同时构建重组质粒pET-30a-LOX4,随后将构建的重组质粒转化到感受态Transetta中。之后对CgLOX4重组蛋白进行诱导表达和纯化,所得到的CgLOX4重组蛋白相对分子质量约为32 000,由于电荷过强,使该蛋白串联了一长串纯化所用的His标签,导致实际电泳结果中蛋白相对分子质量偏大(图8)。使用上述蛋白对小鼠进行注射,得到CgLOX4多克隆抗体,并进行蛋白免疫印迹分析。结果发现,有一条单一的条带,其与预测的CgLOX4蛋白大小一致(图8)。
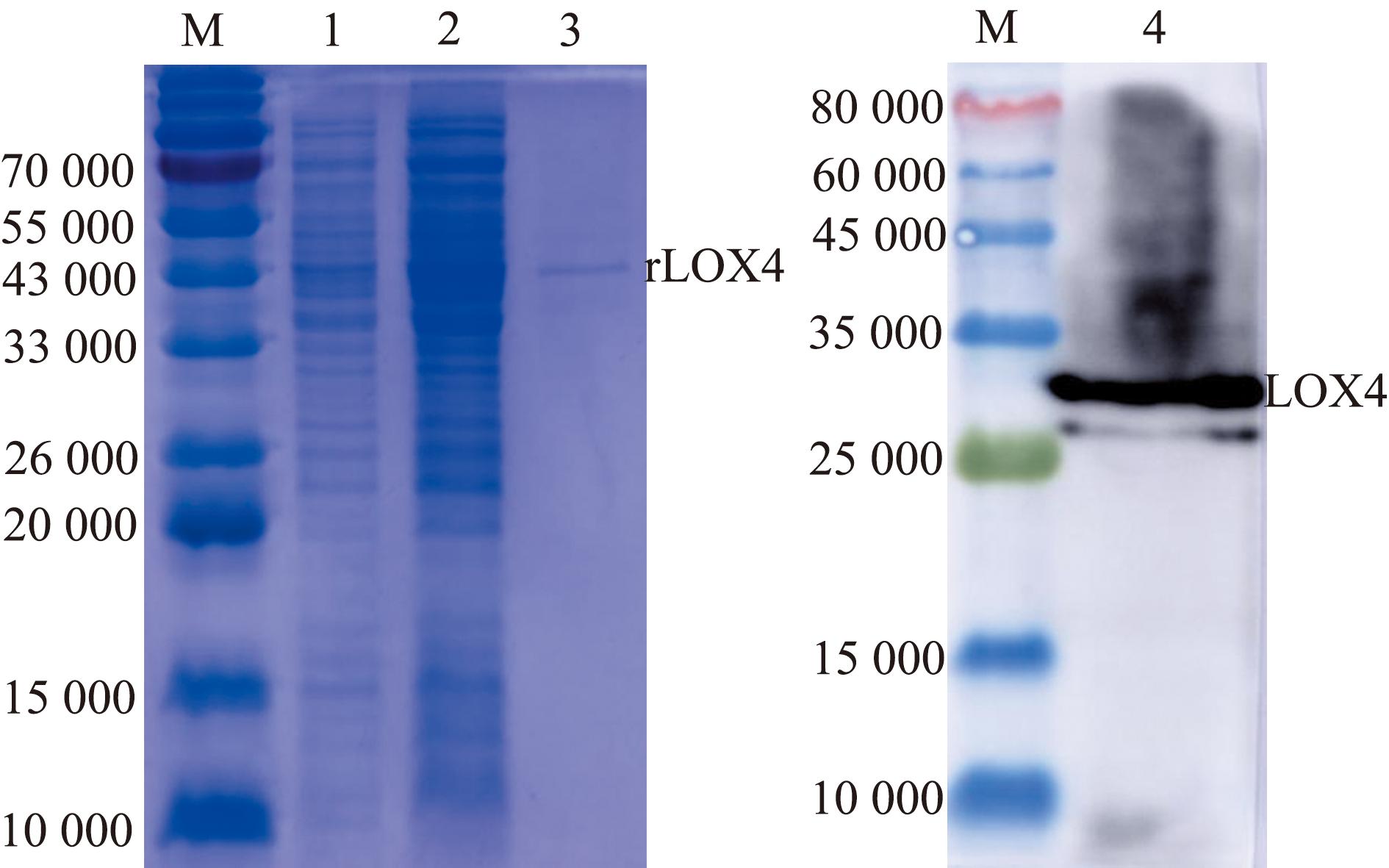
M—蛋白分子量标准;1—诱导前菌液;2—诱导后菌液;3—纯化的CgLOX4重组蛋白;4—CgLOX4抗体。
M—protein molecular weight standard;1—negative control (without IPTG induction);2—IPTG induced;3—purification of LOX4 recombinant protein;4—CgLOX4 antibody.
图8 CgLOX4重组蛋白的表达、纯化和抗体验证
Fig.8 Expression,purification,and antibody verification of CgLOX4 recombinant protein
2.6 CgLOX4在幼虫中的分布
为了探究CgLOX4在长牡蛎幼虫时期的分布情况,对CgLOX4在担轮幼虫、D型幼虫和早期壳顶幼虫时期进行整体免疫荧光试验。CgLOX4被标记为绿色荧光,细胞核被标记为蓝色荧光。结果发现,绿色荧光信号在担轮幼虫时期聚集在壳形成区,呈环状分布;在D型幼虫期观察到绿色荧光信号聚集在内脏团部位,呈点状分布;壳顶幼虫期绿色荧光信号主要聚集在消化腺部位(图9)。
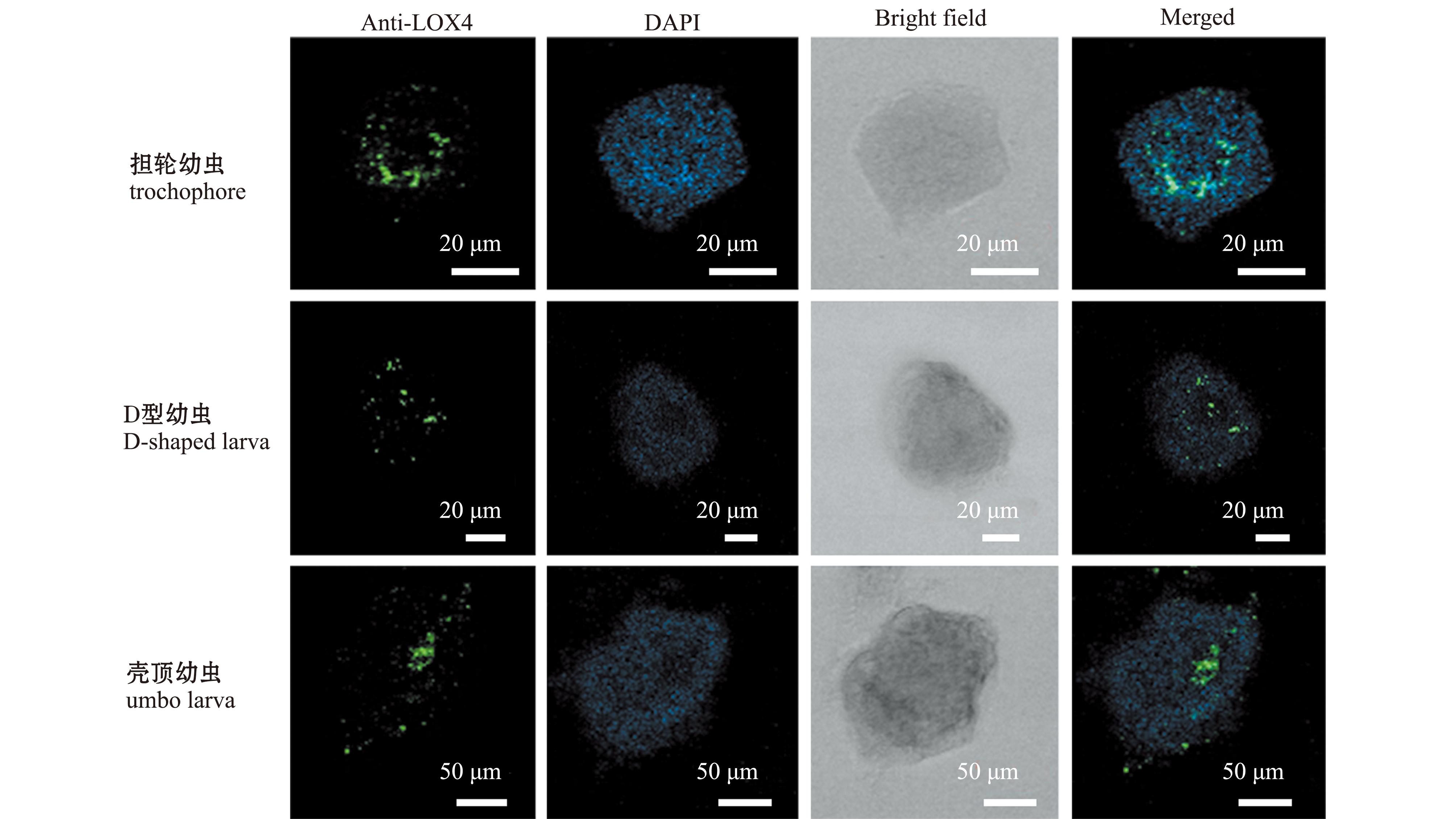
图9 CgLOX4在不同发育时期幼虫中的整体免疫荧光分析
Fig.9 Whole-mount immunofluorescence localization of CgLOX4 in different developmental stages of larvae
3 讨论
3.1 CgLOX4的序列特征与进化分析
本研究中,采用PCR技术扩增得到CgLOX4的全长cDNA序列。SMART蛋白结构域预测显示,CgLOX4只包含一个保守的HOX结构域,符合HOX家族的结构特点,且预测的蛋白三维结构再次印证其在进化上高度保守。通过UniProt预测到CgLOX4蛋白在核内表达,符合其转录因子的特点。CgLOX4整体上亲水性较强,但HOX结构域两端疏水性较强,有利于形成α螺旋,保证其稳定性[26]。CgLOX4氨基酸序列与其他软体动物LOX4和节肢动物HOXA7的相似度较高,CgLOX4与软体动物门石鳖和腕足动物门鸭嘴海豆芽的LOX4亲缘关系最近。这些结果均表明,CgLOX4属于HOX家族,是一个进化保守的基因。
3.2 CgLOX4基因的组织分布特征
鳃、外套膜、血淋巴和闭壳肌等组织是软体动物重要的免疫器官。鳃和外套膜具有滤食功能,可以捕捉和滤食海水中的浮游生物、藻类和悬浮物。LOX4基因已在正颤蚓(Tubifex tubifex)、腹足类蜗牛(Gibbula varia)、加列尼多盘虫(Polystoma gallieni)、石鳖、腕足类动物Terebratalia transversa和水蛭(Hirudo medicinalis)中被鉴定[13,15,27-30]。本研究中,qRT-PCR结果显示,CgLOX4在长牡蛎各组织中均有表达,其中,在闭壳肌和外套膜中表达量较高,在肝胰腺中表达量最低。长牡蛎的闭壳肌通过舒张和收缩控制背侧和腹侧壳板的开合,从而在摄食、提供动力和防御等方面发挥重要作用[31],如遇到刺激时双壳紧闭以抵御刺激。CgLOX4在闭壳肌的表达量最高,表明其可能与长牡蛎的运动和免疫防御有关。外套膜边缘可分为生壳突起、感觉突起和缘膜突起。其中,生壳突起可分泌贝壳角质层;感觉突起可响应外界刺激,感知环境变化;缘膜突起可通过伸展和收缩,控制水流进出,帮助其完成摄食、排精或排卵等过程。CgLOX4在外套膜的表达量较高,表明其可能参与长牡蛎的壳形成、感知内外环境变化和生长繁殖等过程。
3.3 CgLOX4基因在早期发育过程中的表达特征
LOX4在进化上高度保守,其所在的HOX家族被认为起源于双侧动物的最后一个共同祖先[32],是一类重要的发育调控基因,可以控制正常发育组织中的细胞增殖、分化、迁移和死亡[33]。多毛纲动物沙蚕(Nereis virens)NvLOX4在疣足幼虫后期的尾板前部少量细胞中被检测到,并随着年龄的增长其表达水平增加,同时NvLOX4也在新生节段的中胚层呈现低表达[34];掘足纲软体动物角贝(Antalis entails)AeLOX4主要在担轮幼虫期的足中表达[35];单殖亚纲加列尼多盘虫的PgLOX4主要表达于发育的早期[27]。本研究中,CgLOX4在整个胚胎发育过程中均有表达,其中在卵裂期的表达水平显著高于发育后期,且在4细胞期达到最高。正颤蚓TtLOX4也在卵裂期开始表达,到第15阶段开始显著增加[13]。这进一步证实了长牡蛎的10个HOX基因的表达顺序不遵循时间共线性[7]。CgLOX4在4细胞期的表达量达到最高,说明LOX4作为与发育相关的HOX基因家族的一员,是较早激活的转录因子之一。长牡蛎在卵裂期经不完全卵裂形成 A、B、C、D 4个卵裂球,分别对应幼虫的左侧、腹侧、右侧和背侧[36]。双壳类早期胚胎发生中的卵裂模式与其两侧分离的壳板发育密切相关[37],说明CgLOX4不仅对长牡蛎发育初期的螺旋卵裂和由卵裂球产生的特定胚层极为重要,还对壳形成具有一定影响。CgLOX4在卵裂期的高表达提示,LOX4作为胚胎发育过程中较早激活的转录因子之一在调控身体体轴和壳形成中发挥作用。
3.4 CgLOX4蛋白在早期发育过程中的分布特征
HOX属于与个体发育和细胞分化相关的转录因子[38]。对笠贝的研究表明,从原肠胚至面盘幼虫的发育过程中,多个HOX基因表达部位发生迅速变化[39]。这些变化与原肠胚的形成及壳与足的发育等早期发育事件相关[39]。腹足类蜗牛的GvHOX1、GvPOST1和GvPOST2在担轮幼虫的壳形成区表达[40]。耳鲍(Haliotis asinina)的HaHOX1在担轮幼虫期和面盘幼虫期的表达部位都对应于外套膜边缘,表明HOX基因在腹足类壳形成中起作用[41]。笠贝LgLOX4在担轮幼虫后期表达,与背侧壳形成区发育呈正相关[39]。在养殖生产中,长牡蛎在担轮幼虫及之后的发育过程中死亡率逐步增加。本研究中,利用整体免疫荧光试验解析CgLOX4在长牡蛎担轮幼虫、D型幼虫和壳顶幼虫中的分布特征,结果表明,CgLOX4呈环形分布于长牡蛎担轮幼虫的中间部位。长牡蛎与笠贝在担轮幼虫阶段的发育相似。前期有学者利用扫描电镜发现,笠贝担轮幼虫背侧靠近前担轮环的区域首先发生内陷,然后内陷区域细胞表面发育出绒毛状结构,形成贝壳形成区[42]。由于长牡蛎担轮幼虫时期LOX4表达区域与笠贝担轮幼虫贝壳形成区相似,因此,推测CgLOX4在担轮幼虫期参与壳形成,这与CgLOX4的原位杂交结果一致[16]。长牡蛎发育至D型幼虫后,LOX4阳性信号主要分布于D型幼虫中部且靠近直线铰合部。在长牡蛎D型幼虫期,贝壳发生区外翻并逐渐包被胚胎,贝壳发生区细胞分泌贝壳基质形成角质层,碳酸钙在角质层沉积,幼虫背部形成直线铰合部[43]。本研究中还观察到长牡蛎D型幼虫时期LOX4部分阳性信号与内脏团部位重合,推测长牡蛎由担轮幼虫向D型幼虫发育过程中,LOX4从贝壳发生区向内脏团转移。壳顶幼虫期,长牡蛎左壳壳顶突出,右壳生长较慢,LOX4主要分布在消化腺区域。
4 结论
1)CgLOX4具有HOX基因家族的典型特征,包含一个HOX结构域。预测显示,CgLOX4在细胞核中表达,整体上属于亲水性基因,但HOX结构域两端为疏水性。说明CgLOX4属于HOX家族,是一个进化保守的基因。
2)CgLOX4在长牡蛎各组织中均有表达,且在闭壳肌和外套膜表达量较高。说明CgLOX4与长牡蛎的运动、免疫防御、壳形成、感知环境变化和生长繁殖等过程有关。
3)CgLOX4在长牡蛎幼虫各发育时期均有表达,且卵裂期表达量较高,桑椹胚及其后各发育时期CgLOX4表达量维持较低水平。说明CgLOX4在调控身体体轴和壳形成中发挥作用。
4)CgLOX4在担轮幼虫期呈环形分布于背侧中间部位,在D型幼虫期主要表达于中部靠近直线铰合部的位置,在壳顶幼虫期与D型幼虫期的表达部位基本一致。说明CgLOX4在担轮幼虫期参与壳形成,在D型幼虫期参与内脏团形成,在壳顶幼虫期参与消化腺形成。
[1] DE KUMAR B,DARLAND D C.The Hox protein conundrum:the specifics of DNA binding for Hox proteins and their partners[J].Developmental Biology,2021,477:284-292.
[2] 王师,杨志辉,李语丽,等.Hox基因及其在软体动物中的研究进展[J].中国海洋大学学报(自然科学版),2017,47(8):23-30.
WANG S,YANG Z H,LI Y L,et al.Hox genes and their research progresses in molluscs[J].Periodical of Ocean University of China(Natural Science Edition),2017,47(8):23-30.(in Chinese)
[3] LEWIS E B.A gene complex controlling segmentation in Drosophila[J].Nature,1978,276(5688):565-570.
[4] BARUCCA M,CANAPA A,BISCOTTI M A.An overview of Hox genes in Lophotrochozoa:evolution and functionality[J].Journal of Developmental Biology,2016,4(1):12.
[5] FINNERTY J R,MARTINDALE M Q.Ancient origins of axial patterning genes:Hox genes and Para Hox genes in the Cnidaria[J].Evolution &Development,1999,1(1):16-23.
[6] BROOKE N M,GARCIA-FERN NDEZ J,HOLLAND P W H.The ParaHox gene cluster is an evolutionary sister of the Hox gene cluster[J].Nature,1998,392(6679):920-922.
NDEZ J,HOLLAND P W H.The ParaHox gene cluster is an evolutionary sister of the Hox gene cluster[J].Nature,1998,392(6679):920-922.
[7] ZHANG G F,FANG X D,GUO X M,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[8] VERLINSKY Y,MOROZOV G,GINDILIS V,et al.Homeobox gene expression in human oocytes and preembryos[J].Molecular Reproduction and Development,1995,41(2):127-132.
[9] KIM M H,JIN H,SEOL E Y,et al.Sequence analysis and tissue specific expression of human HOXA7[J].Molecular Biotechnology,2000,14(1):19-24.
[10] GAO Y,HYTTEL P,HALL V J.Regulation of H3K27me3 and H3K4me3 during early porcine embryonic development[J].Molecular Reproduction and Development,2010,77(6):540-549.
[11] DOU D R,CALVANESE V,SIERRA M I,et al.Medial HOXA genes demarcate haematopoietic stem cell fate during human development[J].Nature Cell Biology,2016,18(6):595-606.
[12] YU B,WU B Y,HONG P S,et al.Co-expression of Runx1,Hoxa9,hlf,and Hoxa7 confers multi-lineage potential on hematopoietic progenitors derived from pluripotent stem cells[J].Frontiers in Cell and Developmental Biology,2022,10:859769.
[13] ENDO M,SAKAI C,SHIMIZU T.Embryonic expression patterns of Hox genes in the oligochaete annelid Tubifex tubifex[J].Gene Expression Patterns:GEP,2016,22(1):1-14.
[14] LEE P N,CALLAERTS P,DE COUET H G,et al.Cephalopod Hox genes and the origin of morphological novelties[J].Nature,2003,424(6952):1061-1065.
[15] SAMADI L,STEINER G.Expression of Hox genes during the larval development of the snail,Gibbula varia (L.)—further evidence of non-colinearity in molluscs[J].Development Genes and Evolution,2010,220(5):161-172.
[16] LIU G,HUAN P,LIU B Z.Identification of three cell populations from the shell gland of a bivalve mollusc[J].Development Genes and Evolution,2020,230(1):39-45.
[17] 卢锡琴,张丽莉,黄世玉,等.杂色鲍caspase-3基因的克隆及其在发育、弧菌感染、高温和缺氧应激中的表达分析[J].大连海洋大学学报,2022,37(3):411-419.
LU X Q,ZHANG L L,HUANG S Y,et al.Cloning and expression analysis of caspase-3 gene in development,immune and stress of variously colored abalone Haliotis diversicolor[J].Journal of Dalian Ocean University,2022,37(3):411-419.(in Chinese)
[18] 马得友,于莲莲,林威港,等.刺参Wnt9基因克隆、组织分布及在胚胎发育中的表达分析[J].大连海洋大学学报,2022,37(3):394-402.
MA D Y,YU L L,LIN W G,et al.cDNA cloning and expression distribution of Wnt9 gene in adult tissues and embryonic development in sea cucumber Apostichopus japonicas[J].Journal of Dalian Ocean University,2022,37(3):394-402.(in Chinese)
[19] LENG J Y,LI Y N,YANG W W,et al.The involvement of CgCaspase-8-2 in regulating the expressions of cytokines,antibacterial peptide and autophagy-related genes in oysters[J].Fish &Shellfish Immunology,2021,119:145-153.
[20] CONSORTIUM U.UniProt:the universal protein knowledge base in 2023[J].Nucleic Acids Research,2023,51(D1):D523-D531.
[21] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J].Methods,2001,25(4):402-408.
[22] JIANG Q F,ZHOU Z,WANG L L,et al.A scallop nitric oxide synthase (NOS) with structure similar to neuronal NOS and its involvement in the immune defense[J].PLoS One,2013,8(7):e69158.
[23] ZHOU Z,WANG M Q,ZHAO J M,et al.The increased transcriptional response and translocation of a Rel/NF-κB homologue in scallop Chlamys farreri during the immune stimulation[J].Fish &Shellfish Immunology,2013,34(5):1209-1215.
[24] SUN J J,WANG L L,YANG C Y,et al.An ancient BCR-like signaling promotes ICP production and hemocyte phagocytosis in oyster[J].iScience,2020,23(2):100834.
[25] 宋小瑞.长牡蛎免疫系统的发生和发育机制的初步研究[D].青岛:中国科学院大学(中国科学院海洋研究所),2016.
SONG X R.Study on the ontogenesis and development mechanism of immune system in the Pacific oyster Crassostrea gigas[D].Qingdao: University of Chinese Academy of Sciences(Institute of Oceanology,Chinese Academy of Sciences),2016.(in Chinese)
[26] YANG A S,HONIG B.Free energy determinants of secondary structure formation:I.α-helices[J].Journal of Molecular Biology,1995,252(3):351-365.
[27] BADETS M,MITTA G,GALINIER R,et al.Expression patterns of Abd-A/Lox4 in a monogenean parasite with alternative developmental paths[J].Molecular and Biochemical Parasitology,2010,173(2):154-157.
[28] FRITSCH M,WOLLESEN T,WANNINGER A.Hox and ParaHox gene expression in early body plan patterning of Polyplacophoran mollusks[J].Journal of Experimental Zoology Part B:Molecular and Developmental Evolution,2016,326(2):89-104.
[29] SCHIEMANN S M,MART N-DUR
N-DUR N J M,BØRVE A,et al.Clustered brachiopod Hox genes are not expressed collinearly and are associated with lophotrochozoan novelties[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(10):E1913-E1922.
N J M,BØRVE A,et al.Clustered brachiopod Hox genes are not expressed collinearly and are associated with lophotrochozoan novelties[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(10):E1913-E1922.
[30] WONG V Y,AISEMBERG G O,GAN W B,et al.The leech homeobox gene Lox4 may determine segmental differentiation of identified neurons[J].The Journal of Neuroscience:the Official Journal of the Society for Neuroscience,1995,15(8):5551-5559.
[31] 李绘娟,于红,李琪.长牡蛎闭壳肌肌纤维的组织学特性[J].水产学报,2017,41(9):1392-1399.
LI H J,YU H,LI Q.Histological studies on the muscle fibers of Crassostrea gigas adductor[J].Journal of Fisheries of China,2017,41(9):1392-1399.(in Chinese)
[32] GARCIA-FERN NDEZ J.The genesis and evolution of homeobox gene clusters[J].Nature Reviews Genetics,2005,6(12):881-892.
NDEZ J.The genesis and evolution of homeobox gene clusters[J].Nature Reviews Genetics,2005,6(12):881-892.
[33] REZSOHAZY R,SAURIN A J,MAUREL-ZAFFRAN C,et al.Cellular and molecular insights into Hox protein action[J].Development,2015,142(7):1212-1227.
[34] BAKALENKO N I,NOVIKOVA E L,NESTERENKO A Y,et al.Hox gene expression during postlarval development of the polychaete Alitta virens[J].EvoDevo,2013,4(1):13.
[35] WOLLESEN T,RODR GUEZ MONJE S V,DE OLIVEIRA A L,et al.Staggered Hox expression is more widespread among molluscs than previously appreciated[J].Proceedings Biological Sciences,2018,285(1888):20181513.
GUEZ MONJE S V,DE OLIVEIRA A L,et al.Staggered Hox expression is more widespread among molluscs than previously appreciated[J].Proceedings Biological Sciences,2018,285(1888):20181513.
[36] KURITA Y,DEGUCHI R,WADA H.Early development and cleavage pattern of the Japanese purple mussel,Septifer virgatus[J].Zoological Science,2009,26(12):814-820.
[37] KAKOI S,KIN K,MIYAZAKI K,et al.Early development of the Japanese spiny oyster (Saccostrea kegaki):characterization of some genetic markers[J].Zoological Science,2008,25(5):455-464.
[38] 林铃,吴兴中.HOX家族及其在癌症中的研究进展[J].生物化学与生物物理进展,2017,44(9):737-750.
LIN L,WU X Z.Research progress on HOX family and cancer[J].Progress in Biochemistry and Biophysics,2017,44(9):737-750.(in Chinese)
[39] HUAN P,WANG Q,TAN S J,et al.Dorsoventral dissociation of Hox gene expression underpins the diversification of molluscs[J]. BioRxiv,2019:603092.
[40] SAMADI L,STEINER G.Involvement of Hox genes in shell morphogenesis in the encapsulated development of a top shell gastropod (Gibbula varia L.)[J].Development Genes and Evolution,2009,219(9):523-530.
[41] HINMAN V F,O'BRIEN E K,RICHARDS G S,et al.Expression of anterior Hox genes during larval development of the gastropod Haliotis asinina[J].Evolution &Development,2003,5(5):508-521.
[42] 崔梦露,郇聘,刘保忠.笠贝(Lottia goshimai)早期发育过程的扫描电镜观察[J].海洋与湖沼,2021,52(6):1514-1520.
CUI M L,HUAN P,LIU B Z.Early development of Lottia goshimai revealed by scanning electron microscopy[J].Oceanologia et Limnologia Sinica,2021,52(6):1514-1520.(in Chinese)
[43] 刘刚.长牡蛎贝壳发生区细胞群体的鉴定及其调控因子的初步研究[D].青岛:中国科学院大学(中国科学院海洋研究所),2017.
LIU G.Identification of multiple cell populations and the regulatory factors in the oyster shell field[D].Qingdao: University of Chinese Academy of Sciences(Institute of Oceanology,Chinese Academy of Sciences),2017.(in Chinese)