贝类动物缺乏特异性免疫系统,主要依靠先天性免疫防御功能来抵御病原体的入侵,它们的免疫系统由物理防御、细胞免疫和体液免疫共同组成。当生物体受到外源病原微生物入侵时,天然免疫系统通过一系列基因编码的模式识别受体(pattern-recognition receptor,PRR)对微生物病原进行识别。在果蝇和按蚊中发现的PRR分为6个类型[1],包括肽聚糖识别蛋白(PGRP)、革兰阴性细菌结合蛋白(GNBP)、C型凝集素(CTL)、清道夫受体(SCR)、硫脂蛋白(TEP)和半乳糖凝集素(GALE)等。PRR可以识别多种病原体相关分子模式(PAMP),如细菌DNA、脂多糖(LPS)、肽聚糖(PGN)、双链病毒RNA和其他存在于多细胞生物[2]表面的非自身分子。
肽聚糖识别蛋白(PGRP)是先天性免疫系统中一类重要的模式识别受体,可特异性识别细菌细胞壁主要成分肽聚糖。PGRP最初从家蚕的血淋巴中被发现并命名,其可以与革兰氏阳性细菌中Lys型PGN结合[3]。PGRP广泛存在于从昆虫到人类的各种生物体中,哺乳动物中发现了4个PGRPs,即PGLYRP1、PGLYRP2、PGLYRP3和PGLYRP4。PGRP分为PGRP-S、PGRP-I和PGRP-L 3种类型 [4],其中PGRP-S和 PGRP-L主要存在于一些无脊椎动物和脊椎动物中,而PGRP-I仅在哺乳动物中存在[4]。在软体动物中也鉴定出上百种PGRPs,例如大竹蛏(Solen grandis)SgPGRP-S1[5]、刺参(Apostichopus japonicus)AjPGRP-S[6]和皱纹盘鲍(Haliotis discus hannai)HdPGRP[7]等。肽聚糖识别蛋白作为一种主要的模式识别受体,在先天性免疫中发挥着重要的作用。PGRP具有保守的酰胺酶活性结构域,能水解肽聚糖的酰胺键,从而降解病原体[8],也可激活Toll、IMD等信号通路的转录因子,诱导抗菌肽、活性氧的生成,发挥免疫效应[9]。
皱纹盘鲍隶属于软体动物门(Mollusca)腹足纲(Gastropoda)前鳃亚纲(Prosobranchia)原始腹足目(Archaeogastropoda)鲍科(Haliotidae)。近年来,随着水产养殖规模的扩大、集约化程度的提高及沿海水质的日趋恶化,养殖鲍疾病频发,给养殖生产带来巨大的经济损失,严重阻碍了鲍产业的发展[10]。鲍疾病的发生与鲍自身的抗病能力、养殖密度、水质环境及病原菌有着密切的关系[11]。鲍免疫力下降和感染致病菌是鲍死亡的主要原因,因此,寻求免疫防治是保障鲍养殖业健康发展的重要措施。本研究中,从皱纹盘鲍机体自身的免疫防御体系入手,克隆了皱纹盘鲍肽聚糖识别蛋白基因HdhPGRP-SC2-like,通过体外重组表达获得了rHdhPGRP-SC2-like重组蛋白,并探讨了其与PGN的结合活性及结合模式,以期为深入探索鲍抵御病原微生物的免疫应答机制提供科学参考。
1 材料与方法
1.1 材料
试验用皱纹盘鲍体质量为(63±5)g,购自福建晋江福大鲍鱼水产有限公司,暂养于充氧并经沙滤的海水中,每日更换海水并投喂新鲜细基江蓠。
试剂:RNAprep Pure动物组织总RNA提取试剂盒、RNAprep Pure培养细胞/细菌总RNA提取试剂盒、PrimerScriptTM RT-PCR(Perfect Real Time)试剂盒均购自TaKaRa公司;FastStart Essential DNA Green Master购自Roche公司;M-MuLV Reverse Transcriptase、柱式DNA胶回收试剂盒、BL21(DE3)感受态细胞、LB 肉汤琼脂培养基、IPTG和非预染蛋白marker均购自生工生物工程(上海)股份有限公司;Pierce BCA Protein Assay Kit购自Thermo公司;PGRP兔多克隆抗体(bs-7673R)购自Bioss公司;大肠杆菌肽聚糖、藤黄微球菌(Micrococcus luteus)肽聚糖均购自Sigma公司。
1.2 方法
1.2.1 HdhPGRP-SC2-like全长cDNA序列克隆 使用RNAprep Pure总RNA提取试剂盒提取皱纹盘鲍鳃总RNA[12]。利用酶标仪测得RNA的浓度。以特异性引物RT1/RT2逆转录得到cDNA,经RNase H 和TdT处理后,用接头引物5′ adaptor、P1及特异引物R1、R2进行巢氏PCR,扩增5′ RACE序列。以3′ adaptor引物逆转录合成的cDNA为模板,用接头引物P1、P2及基因特异性引物F1、F2进行巢氏PCR,扩增3′ RACE序列,引物序列见表1。PCR反应程序:95 ℃下预变性3 min;95 ℃下变性30 s,58 ℃下退火30 s,72 ℃下延伸60 s,共进行33个循环;最后在72 ℃下再延伸7 min。将PCR产物克隆到pMD-18T载体并送生工生物工程(上海)股份有限公司进行测序。
表1 引物序列
Tab.1 Primer sequence information

引物 primer序列 sequence(5′-3′)用途usage 5′ adaptorGCTGTCAACGATACGCTACGTAACG-GCATGACAGTGCCCCCCCCCCCCCCC5′ RACE3′ adaptorGCTGTCAACGATACGCTACGTAACG-GCATGACAGTGTTTTTTTTTTTTTTTTTT3′ RACEP1GCTGTCAACGATACGCTACGTAAC5′ RACE、3′ RACEP2GCTACGTAACGGCATGACAGTG3′ RACERT1TCTGATAGCTCCTGACTCTCT逆转录RT2TGATGGATGAAGGCGTAT逆转录F1ATCAACACCGTCAAGGCTCTCATTGCT3′ RACEF2AGGTACAGTGTTCTTTTATTAGGACGCCCA5′ RACER1GCAAGATTCAGTCCAGCTACACCCTCAG3′ RACER2GAATGCCTTGTTCCCACCCCAGA5′ RACEQF1GATTGAGCGGGTATTCGGGTRT-qPCRQR1CGTCAACAACCAGGCACTCTRT-qPCRRPL3FTCATTGCACACACCCAGACT内参基因RPL3RCAATGACCTCATCCTGTTCG内参基因
1.2.2 HdhPGRP-SC2-like序列分析 使用DNASIS MAX软件预测HdhPGRP-SC2-like序列的开放阅读框(ORF)。通过ExPASy-Prosite(http://prosite. expasy.org)和SMART在线软件(http://smart. embl-heidelberg.de)预测HdhPGRP-SC2-like蛋白的特征。使用ExPASyprotParam工具(http://web. expasy.org/protparam)预测HdhPGRP-SC2-like蛋白的分子质量和理论等电点。使用NCBI CD(Conserved Domain Search,https://www.ncbi.nlm. nih.gov/Structure/cdd/wrpsb.cgi)分析蛋白的保守结构域。使用NCBI Net WWW的BLASTN2和BLASTP程序进行同源性搜索(http://www.ncbi.nlm.nih.gov/blast)。利用Clustal W创建多序列比对,并用邻接法和MEGA 7.0软件构建HdhPGRP-SC2-like蛋白的系统进化树。
1.2.3 HdhPGRP-SC2-like基因组织表达模式 随机选取3只皱纹盘鲍,分别采集其血细胞、鳃、上足、腹足、闭壳肌、外套膜和消化腺组织样本。使用RNAprep Pure总RNA提取试剂盒提取各组织的总RNA,并按照PrimerScriptTM RT-PCR 试剂盒说明书,将提取的总RNA进行反转录,合成第一链cDNA。采用实时定量PCR(qPCR)测定HdhPGRP-SC2-like mRNA的转录表达,用特异性引物QR1/QF1扩增HdhPGRP-SC2-like mRNA片断,以RPL3为内参基因[13](表1)。反应程序:95 ℃下变性10 min;95 ℃下变性10 s,60 ℃下退火10 s,72 ℃下延伸10 s,共进行45个循环。使用2-ΔΔCt方法计算目的基因相对表达量。
1.2.4 rHdhPGRP-SC2-like蛋白的重组表达 首先合成HdhPGRP-SC2-like pET-28a(+)重组表达载体,并转化至大肠杆菌BL21(DE3)pLysS中表达重组质粒。经IPTG诱导,用质量分数为12% 的SDS-PAGE分析目标蛋白的表达水平。重组蛋白使用HisTrapTM FF Crude镍柱纯化[14],利用含不同浓度咪唑(20、50、500 mmol/L)的磷酸盐洗脱缓冲液,收集蛋白的洗脱峰,最后将收集的样品用尿素梯度透析复性至PBS缓冲液中。使用BCA法测定重组蛋白的浓度。
1.2.5 蛋白免疫印迹(Western blot)分析 重组表达的粗蛋白用质量分数为12%的SDS-PAGE分离,并转移到PVDF膜上。用含5%脱脂奶粉的PBST在37 ℃下封闭2 h,用PGRP兔多克隆抗体(1∶3 000)在37 ℃下孵育3 h。用PBST洗涤4次,在37 ℃下用辣根过氧化物酶标记的山羊抗兔二抗(1∶10 000稀释度)孵育60 min。用PBST洗涤4次后,再用3,3′,5,5′-Tetramethylbenzidine(TMB)显色并拍照。
1.2.6 HdhPGRP-SC2-like结构预测与分子对接 采用AlphaFold2软件[15]对HdhPGRP-SC2-like蛋白的三维结构建模。采用MOE[16]软件对大肠杆菌PGN与HdhPGRP-SC2-like进行分子对接模拟。采用ChemDraw绘制PGN的二维结构,在MOE中通过能量最小化获得PGN的三维结构并作为配体,通过AlphaFold2预测获得HdhPGRP-SC2-like的三维结构并作为受体。参考果蝇PGRP-LB蛋白[17]的PGN结合位点确定对接口袋,包括H108、H109、H132、Y143、H217、 T223和C225等残基。对接流程选择柔性的Induced fit模式,结合口袋氨基酸的侧链并根据配体构象进行优化调整,约束侧链转动的权重设置为10。所有结合模式首先通过London dG打分函数进行排序,前30个构象通过进一步能量优化和GBVI/WSA dG方法对结合自由能再次评价。将最有可能的结合模式使用LigPlus进行分析,并采用PyMOL软件作图。
1.2.7 rHdhPGRP-SC2-like与PGN结合活性测定 通过ELISA试验检测HdhPGRP-SC2-like蛋白与藤黄微球菌PGN、大肠杆菌PGN的结合情况。将藤黄微球菌PGN和大肠杆菌PGN用MilliQ配制质量浓度为1 mg/mL的母液,用包被液分别稀释至质量浓度为80 μg/mL,取50 μL包被液加入微孔板,60 ℃热击2 h,封闭反应2 h,用PBST洗涤3次;加入50 μL不同质量浓度(0、6.25、12.5、25、50、100、200、400 μg/mL)的纯化蛋白,28 ℃下孵育2 h,用PBST洗涤3次;加入PGRP兔多克隆抗体,37 ℃下孵育1 h,用PBST洗涤3次;用辣根过氧化物酶标记的二抗(1∶10 000)37 ℃下孵育1 h,用PBST洗涤3次;TMB显色液避光孵育20 min,用浓度为2 mol/L的H2SO4终止反应。使用酶标仪测定OD450 nm吸光度。
1.2.8 rHdhPGRP-SC2-like的酰胺酶活性测定 参照文献[8]中的方法,测定HdhPGRP-SC2-like的酰胺酶活性。取一定量的藤黄微球菌PGN母液,分别溶解于HEPES缓冲液(20 mmol/L,pH 7.2,含150 mmol/L NaCl)、HEPES-ZnCl2缓冲液(20 mmol/L,pH 7.2,含150 mmol/L NaCl、10 mmol/L ZnCl2 )和HEPES-ZnCl2 EDTA缓冲液(20 mmol/L,pH 7.2,含150 mmol/L NaCl、10 mmol/L ZnCl2、10 mmol/L EDTA)中,并加入50 μg/mL HdhPGRP-SC2-like,使其终浓度为1 mg/mL,以PBS缓冲液作为阴性对照。在120 min内每隔15 min测定一次OD540 nm吸光度。
1.3 数据处理
试验数据均以平均值 ± 标准误(mean± S.E.)表示,采用 SPSS 19.0软件对试验数据进行单因素方差分析(ANOVA),显著性水平设为0.05。
2 结果与分析
2.1 HdhPGRP-SC2-like全长cDNA序列克隆
HdhPGRP-SC2-like基因的ORF从实验室前期建立的Haliotis discus hannai转录组数据库中获得。通过巢氏PCR获得了5′ RACE序列和3′ RACE序列(图1)。经序列拼接最终获得938 bp的HdhPGRP-SC2-like全长cDNA(GenBank登录号OQ621429),其中包含74 bp的5′非翻译区和一个具有典型poly(A)尾巴的81 bp 3′的非翻译区。使用DNASIS MAX软件预测HdhPGRP-SC2-like cDNA的ORF,共编码260个氨基酸组成的多肽链(图2)。预测该蛋白的理论相对分子质量为28 600,等电点为9.62。通过SMART预测HdhPGRP-SC2-like蛋白含有1个信号肽(1~20 aa),1个低复杂性结构域(83~96 aa)、1个PGRP结构域(99~241 aa)和1个Ami_2结构域(111~247 aa)(图2)。

M—DNA marker;1—5′ RACE PCR产物;2—3′ RACE PCR产物。
M—DNA marker;1—5′ RACE PCR product;2—3′ RACE PCR product.
图1 HdhPGRP-SC2-like RACE PCR产物电泳图谱
Fig.1 RACE PCR amplification of the full-length cDNA sequence of HdhPGRP-SC2-like
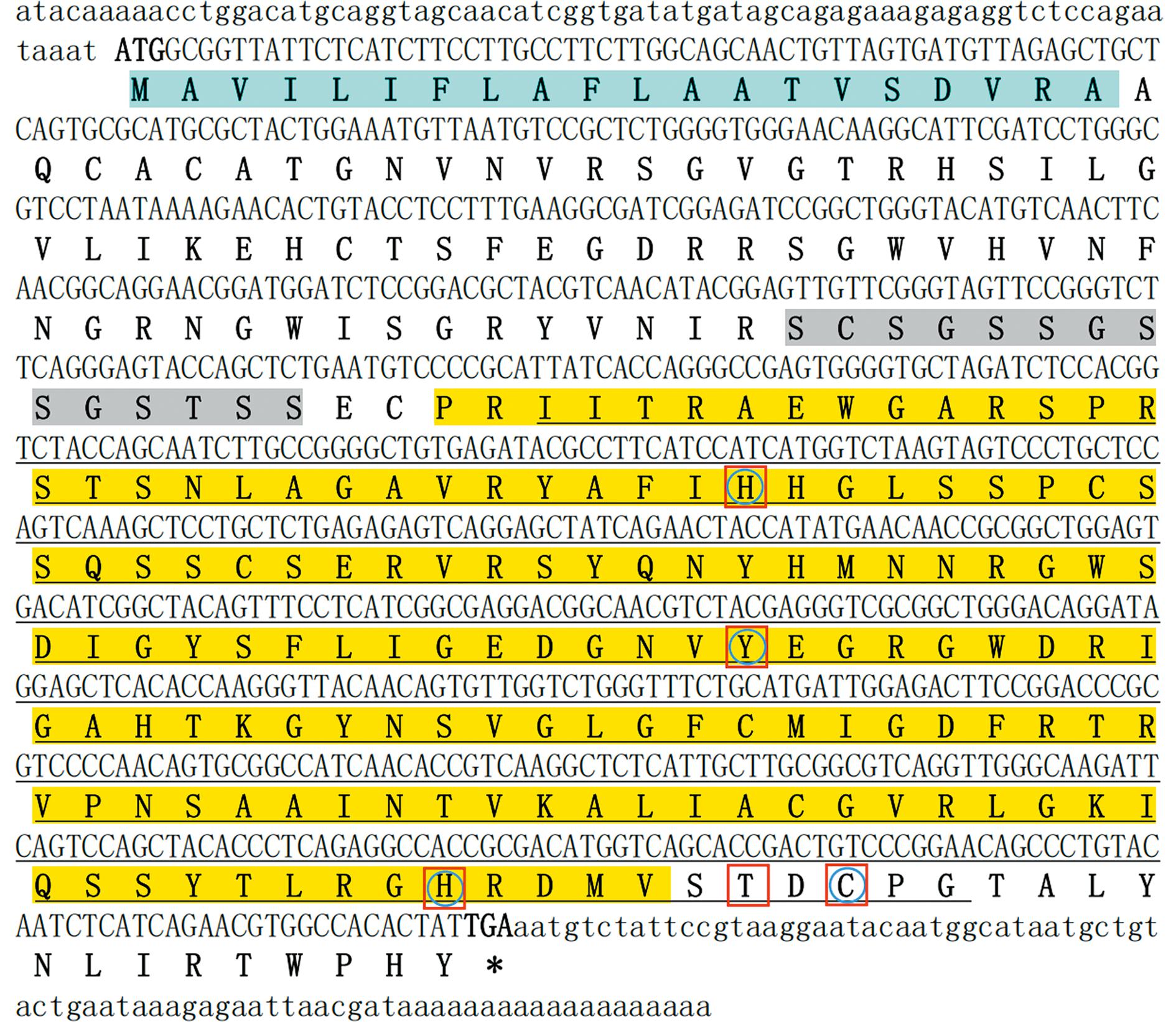
蓝色阴影表示信号肽;灰色阴影表示低复杂性结构域;黄色阴影表示PRGP结构域;下划线表示Ami_2结构域;红色方框表示酰胺酶催化位点;蓝色圆框表示Zn2+结合位点。
Blue shadow indicates signal peptide;Gray shadow indicates low complexity domain;yellow shadow indicates PRGP domain;Underline indicates Ami_2 domain;Red box indicates amidase catalytic site;Blue circle indicates Zn2+ binding site.
图2 HdhPGRP-SC2-like全长cDNA序列及推导的氨基酸序列
Fig.2 Complementary DNA and predicted amino acid sequences of HdhPGRP-SC2-like
2.2 HdhPGRP-SC2-like多序列比对及进化树
通过Clustal W软件对HdhPGRP-SC2-like与其他物种的PGRP结构域进行多序列比对,发现PGRP结构域氨基酸序列具有高度的保守性(图3)。使用MEGA 7.0软件构建HdhPGRP-SC2-like系统进化树(bootstrap 设置为1 000 次)。结果显示,皱纹盘鲍与软体动物分支中黑唇鲍(Haliotis rubra)和红鲍(H.rufescens)的PGRP SC2-like亲缘关系较近(图4)。

Sources and GenBank numbers of PGRPs are as follows:Haliotis discus hannai PGRP(MZ150581),Haliotis discus hannai PGRP-SC2-like(OQ621429),Haliotis discus discus PGRP(KF554145.1),Solen grandis PGRP2(AEW43447.1),Crassostrea gigas PGRP S2(BAG31898.1),Azumapecten farreri PGRP S1 precursor(AAY53765.1),Euprymna scolopes PGRP1(AAY27974.1),Xenopus tropicalis PGLYRP1(AAH91103.1),Drosophila melanogaster PGRP-SD(CAD89198.1),Mus musculus PGLYRP1(AAH05582.1)and Homo sapiens PGRP1(AAI01848.1).
图3 HdhPGRP-SC2-like与部分已知物种PGRPs的氨基酸序列比对
Fig.3 Multiple alignment of amino acid sequences of HdhPGRP-SC2-like and some known species PGRPs

图4 HdhPGRP-SC2-like氨基酸序列的系统进化树
Fig.4 Phylogenetic tree of HdhPGRP-SC2-like amino acid sequence
2.3 HdhPGRP-SC2-like的结构预测
通过AlphaFold2预测HdhPGRP-SC2-like蛋白的3D结构,发现N端由7个β折叠组成,C端由5个ɑ螺旋与5个β折叠组成(图5)。HdhPGRP-SC2-like蛋白具有8个半胱氨酸,可以形成四对二硫键,推测形成二硫键的半胱氨酸为Cys78-Cys201、Cys115-Cys121、Cys3-Cys64 和Cys31-Cys5。
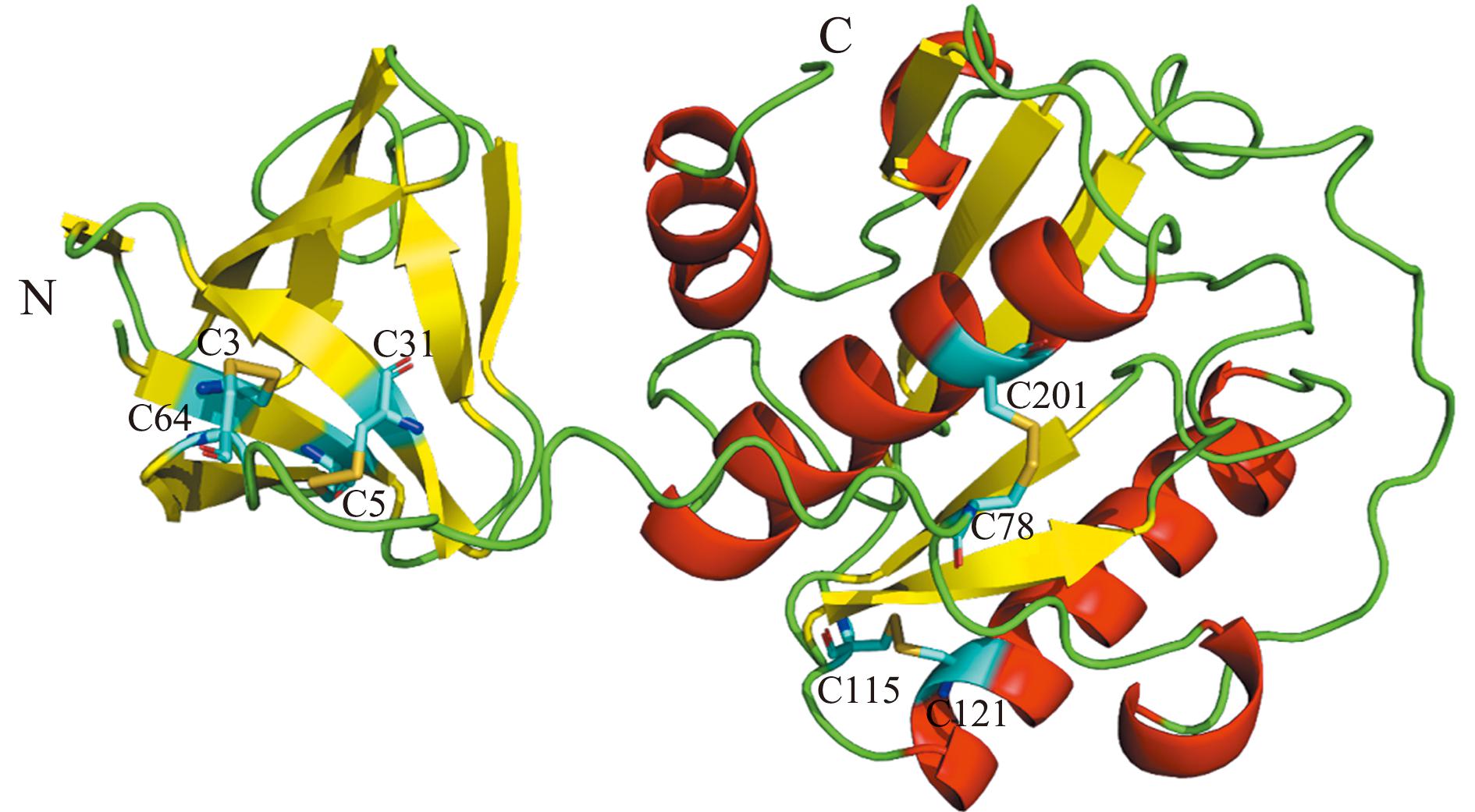
图5 HdhPGRP-SC2-like的三维结构
Fig.5 3D structural diagram of HdhPGRP-SC2-like
2.4 HdhPGRP-SC2-like基因的组织分布
通过qPCR检测HdhPGRP-SC2-like基因在皱纹盘鲍各组织中的表达,结果显示,在各组织中均有一定的表达量,其中,在鳃中表达量最高,其次为外套膜、消化腺、上足、闭壳肌、腹足,血细胞中表达量最低(图6)。
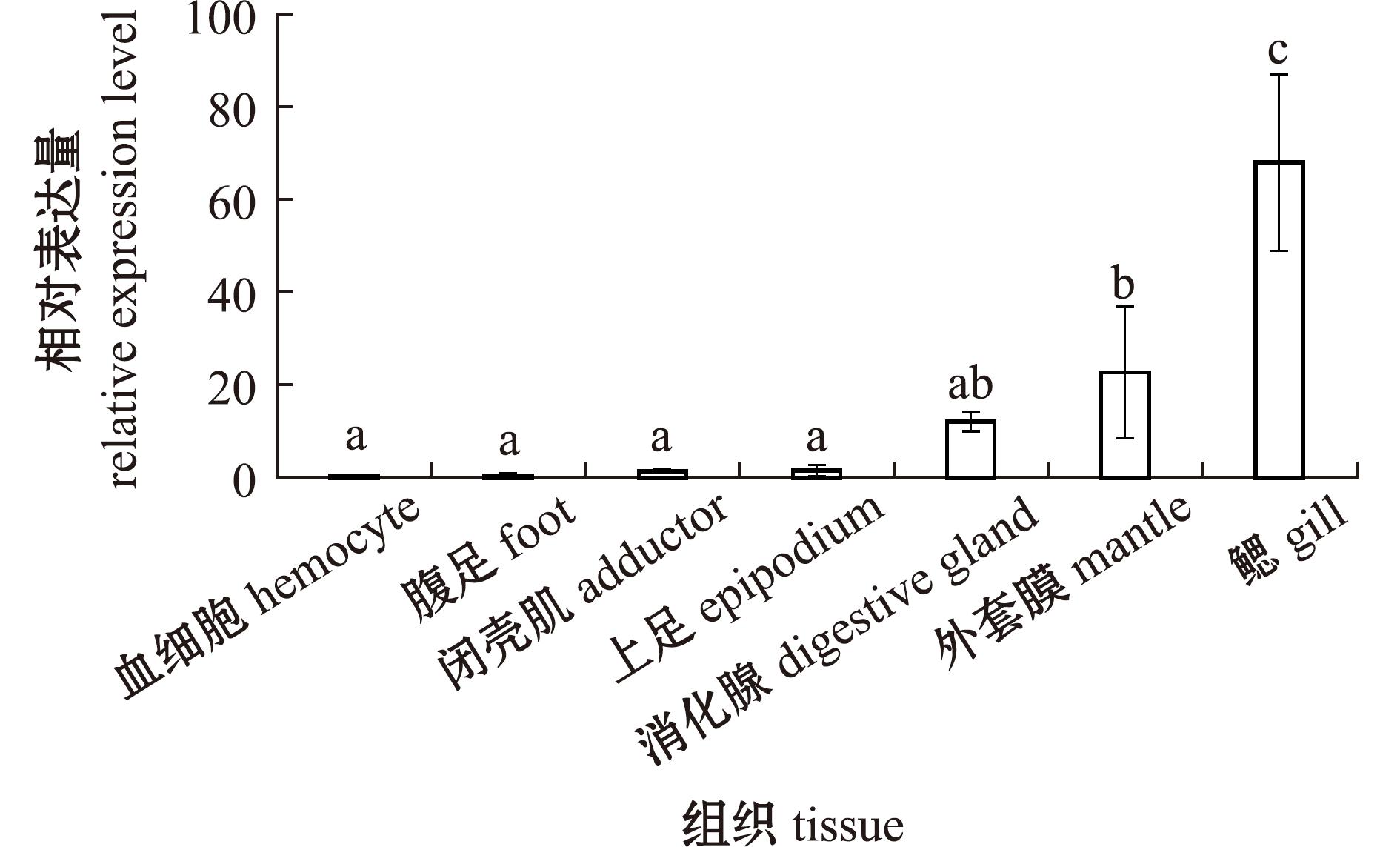
标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。
The means with different letters are significant differences in groups at the 0.05 probability level,and the means with the same letters are not significant differences.
图6 HdhPGRP-SC2-like在皱纹盘鲍组织器官中的表达
Fig.6 Distribution of HdhPGRP-SC2-like transcripts in different tissues of normal Haliotis discus hannai
2.5 rHdhPGRP-SC2-like的载体构建及重组表达
HdhPGRP-SC2-like pET-28a(+)重组表达载体质粒的双酶切结果如图7(a)所示。用0.5 mmol/L IPTG于20 ℃下诱导表达16 h收集菌体,使用超声破碎使其充分溶解后离心,分别对上清和沉淀进行SDS-PAGE电泳,结果如图7(b)所示,rHdhPGRP-SC2-like蛋白在超声破碎的沉淀中表达,上清液中未见目的蛋白。
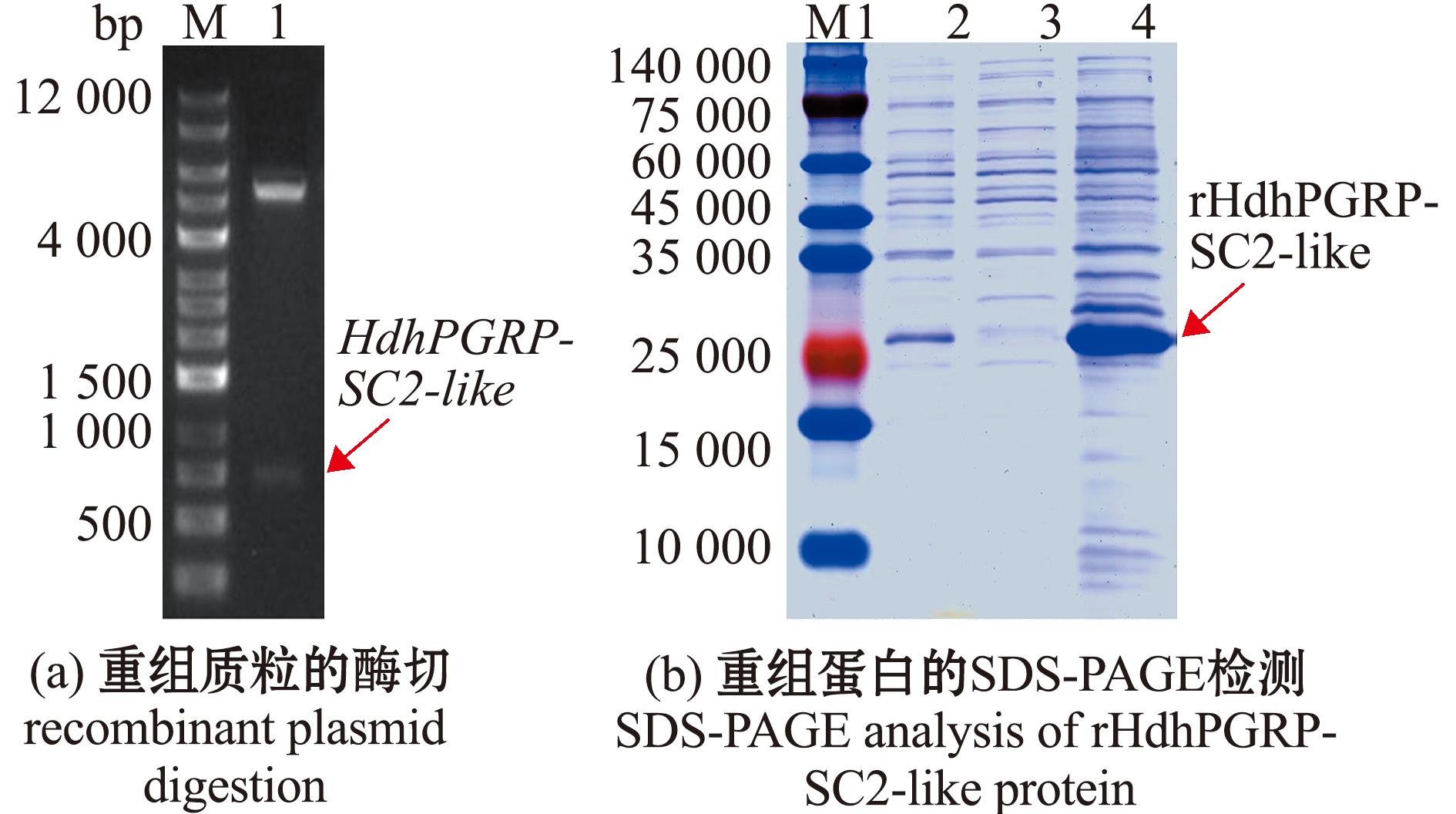
M—DNA marker;M1—蛋白 marker;1—双酶切后的目的基因;2—未诱导;3—上清液;4—沉淀。
M—DNA marker;M1—protein marker;1—the target gene after double digestion;2—pre-induction protein;3—supernatant;4—precipitation.
图7 rHdhPGRP-SC2-like的载体构建及重组表达
Fig.7 Construction of rHdhPGRP-SC2-like vector and recombinant expression
2.6 rHdhPGRP-SC2-like蛋白的纯化及鉴定
融合蛋白的氨基端连接6×His taq标签,通过Ni2+亲和层析可纯化目的蛋白,分别使用浓度为20、50、500 mmol/L的咪唑溶液进行梯度洗脱。对粗蛋白、洗杂流出和洗脱流出分别进行处理,收集样品并进行SDS-PAGE检测,结果如图8(a)所示。利用Western blot对纯化后的蛋白进行验证,结果显示,杂交条带与预期一致(图8(b))。
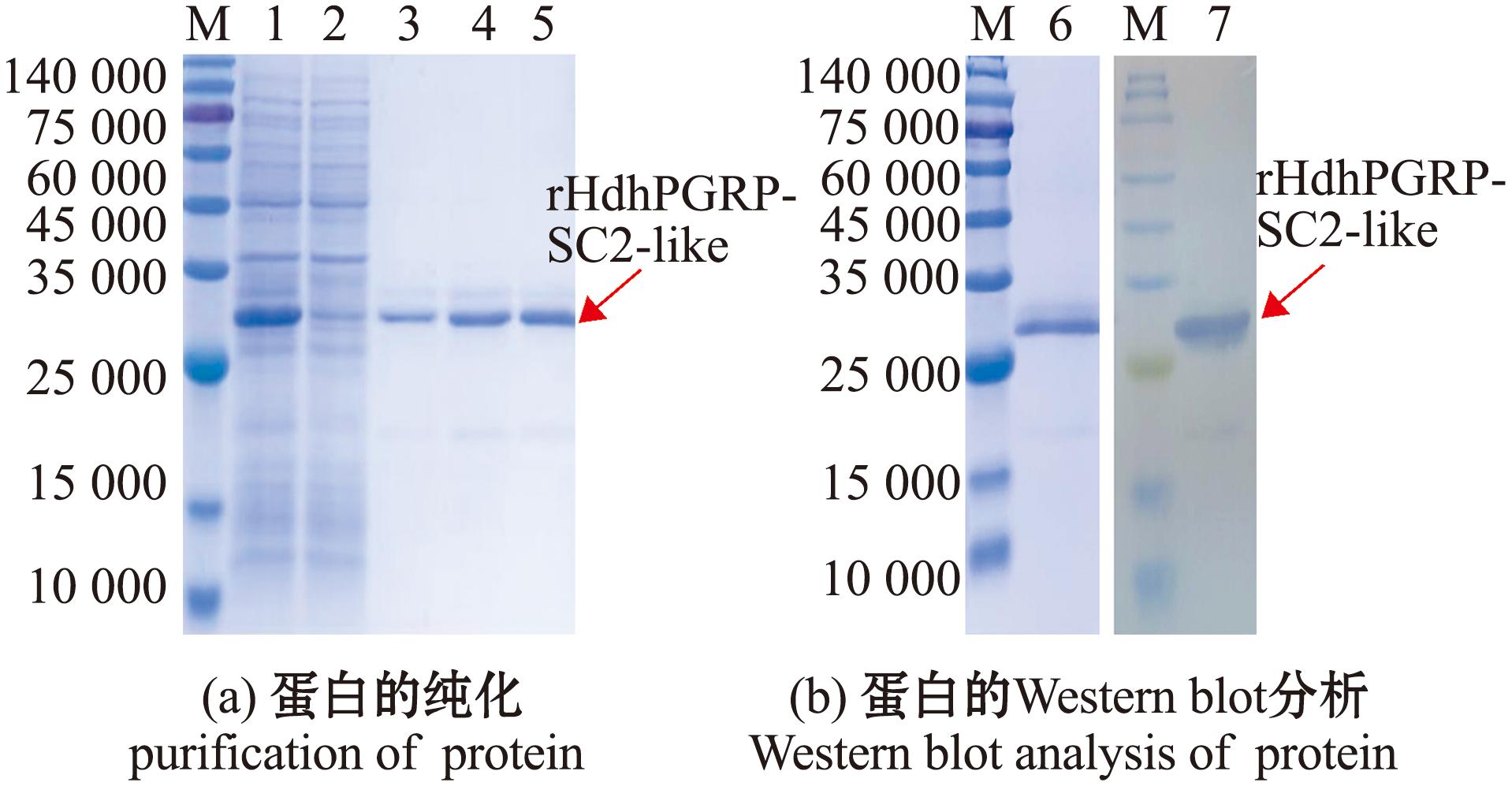
M—蛋白 marker;1—纯化前总蛋白;2—流出组分;3—20 mmol/L 咪唑洗脱组分;4—50 mmol/L咪唑洗脱组分;5、6—500 mmol/L咪唑洗脱组分;7—rHdhPGRP-SC2-like蛋白。
M—protein marker;1—total protein before purification;2—outflow component;3—20 mmol/L imidazole elution component;4— 50 mmol/L imidazole eluting component;5 and 6—500 mmol/L imidazole eluting component;7—rHdhPGRP-SC2-like protein.
图8 rHdhPGRP-SC2-like蛋白的纯化及鉴定
Fig.8 Purification and identification of rHdhPGRP-SC2-like protein
2.7 rHdhPGRP-SC2-like蛋白与PGN结合的活性
通过ELISA法检测rHdhPGRP-SC2-like与肽聚糖结合的活性,结果显示,重组蛋白以浓度依赖的方式与藤黄微球菌PGN、大肠杆菌PGN结合,并且对藤黄微球菌PGN的亲和力更高(图9)。说明rHdhPGRP-SC2-like可以结合革兰氏阳性菌细胞壁。
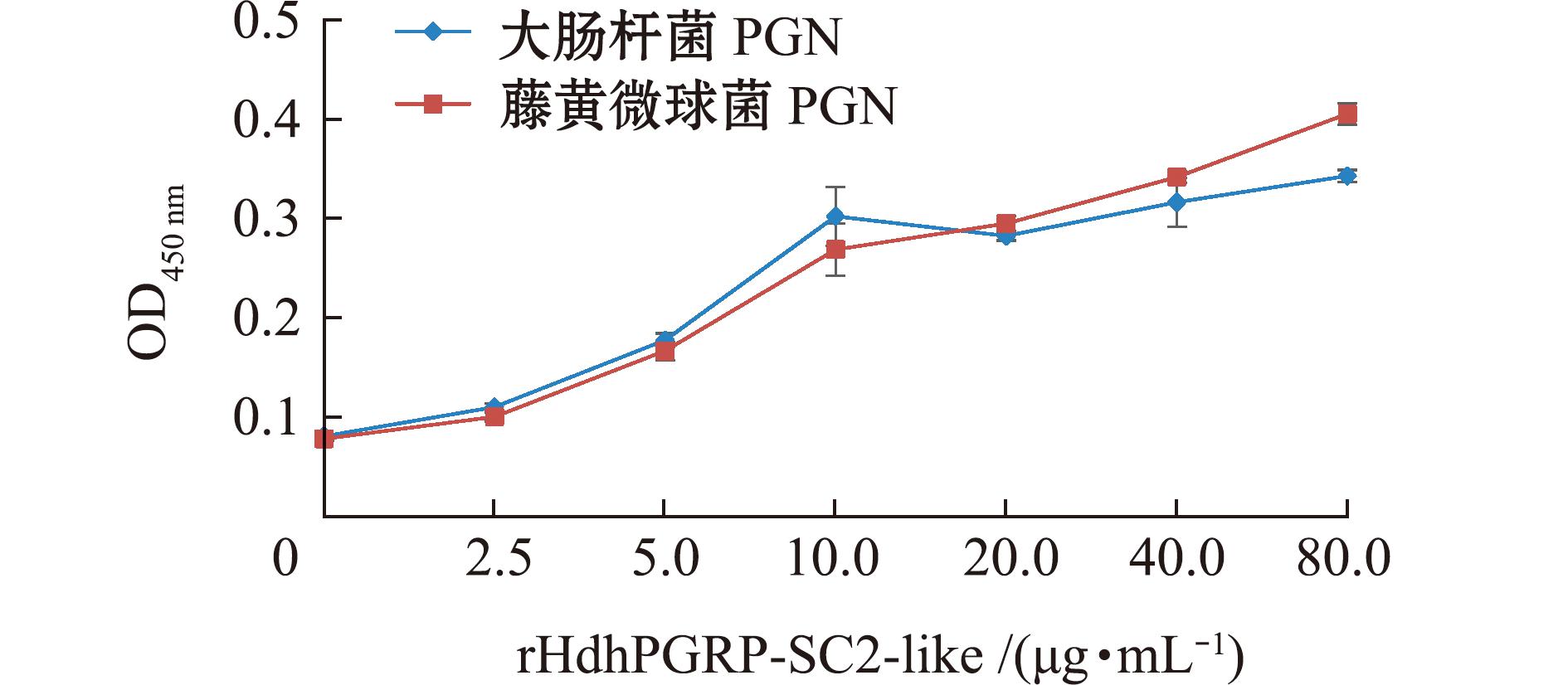
图9 rHdhPGRP-SC2-like与肽聚糖的结合活性
Fig.9 Binding activity of rHdhPGRP-SC2-like with peptidoglycan
2.8 rHdhPGRP-SC2-like蛋白的酰胺酶活性
通过控制缓冲液中Zn2+含量,分析rHdhPGRP-SC2-like的酰胺酶活性,结果显示,随时间延长,藤黄微球菌PGN被重组蛋白逐渐降解,含有Zn2+的试验组OD值下降最显著(图10),表明rHdhPGRP-SC2-like的酰胺酶活性具有Zn2+依赖性。

图10 rHdhPGRP-SC2-like的酰胺酶活性
Fig.10 Amidase activity of rHdhPGRP-SC2-like
2.9 HdhPGRP-SC2-like蛋白与肽聚糖的分子对接
大肠杆菌PGN结构与HdhPGRP-SC2-like的对接打分为-7.9046 kcal/mol,预测的PGN与HdhPGRP-SC2-like的三维结合模式如图11所示,PGN与HdhPGRP-SC2-like的结合位点形成了合适的空间互补,此外,PGN与HdhPGRP-SC2-like还形成了氢键、疏水作用和范德华力相互作用。PGN上的3个氧原子(O40、O42、O68)作为氢键受体,与HdhPGRP-SC2-like上的残基Ser222、Arg183、His217和His108分别形成了4个氢键;PGN上的2个氮原子(N8、N14)作为氢键供体,分别与HdhPGRP-SC2-like上的残基Asn135、Asn134的骨架氧原子形成了2个氢键;PGN还与HdhPGRP-SC2-like有疏水相互作用,涉及的残基有Arg93、Met133、His132、Ser127、Arg136、Arg124、Gly110、Leu111、Cys225、Tyr131、Thr223、Lys167、Trp138、Asp224和Val221。
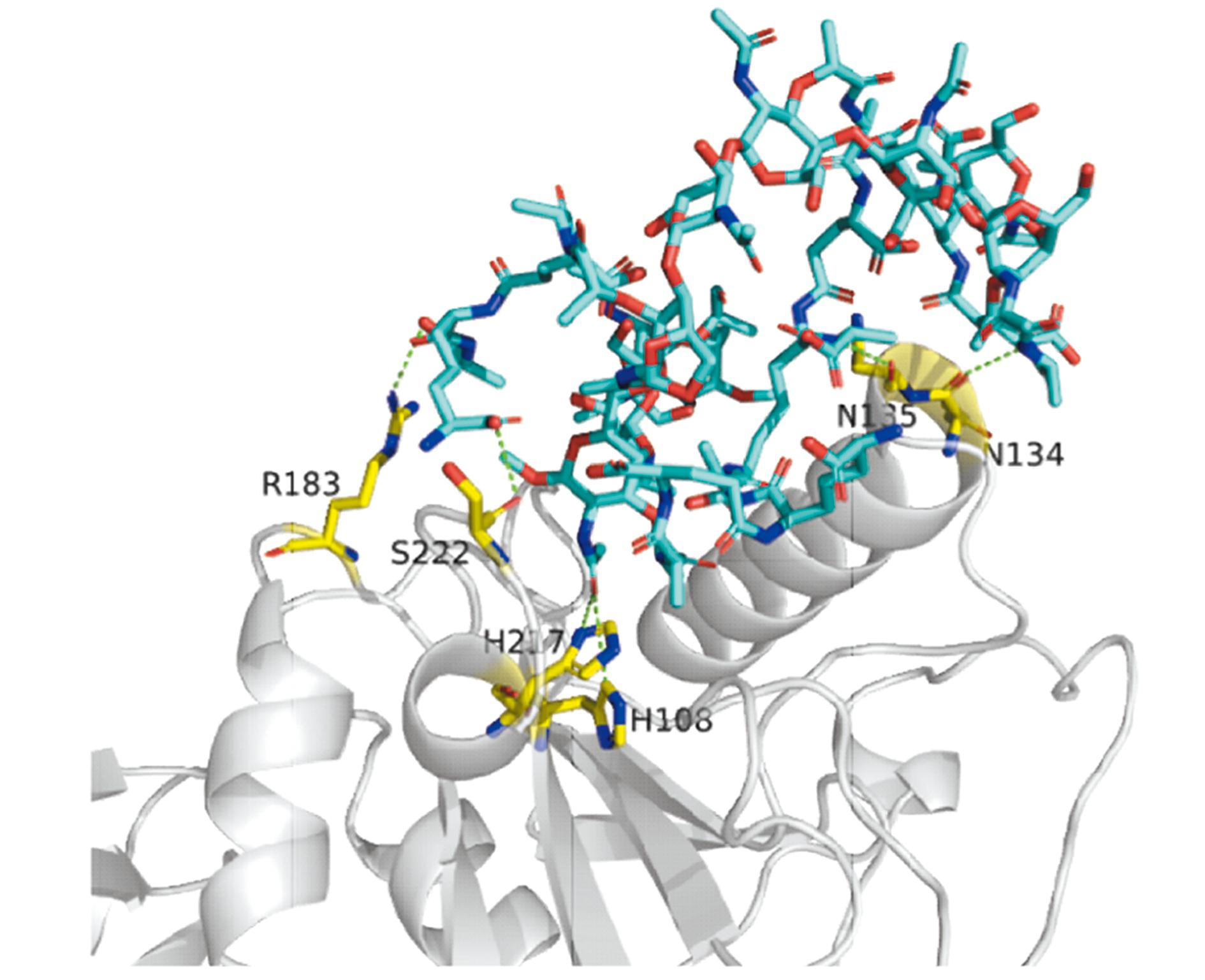
图11 PGN与HdhPGRP-SC2-like的分子对接(三维结合模式)
Fig.11 Molecular docking of PGN with HdhPGRP-SC2-like(predicted 3D binding mode)
3 讨论
3.1 HdhPGRP-SC2-like氨基酸序列特征
本研究中,从皱纹盘鲍克隆获得了一个新的HdhPGRP-SC2-like基因。在HdhPGRP-SC2-like蛋白结构中发现了1个信号肽和1个典型的PGRP结构域,无跨膜结构域,为短型肽聚糖识别蛋白[6,18-19]。通过多序列比对发现,从昆虫到哺乳动物PGRP在结构上高度保守。皱纹盘鲍与黑唇鲍、红鲍的PGRP-SC2-like显示出较高的一致性,这与物种的进化密切相关。在HdhPGRP-SC2-like序列中发现了4个Zn2+结合位点(H128、Y174、H237和C245)和5个酰胺酶催化位点(H128、Y174、H237、T243 和 C245),这些位点在HdhPGRP-SC2-like中高度保守。然而并非每种肽聚糖识别蛋白都具有酰胺酶活性,所有具有酰胺酶活性的 PGRP 在其活性中心中均具有保守的Zn2+结合位点,由2个组氨酸、1个酪氨酸和1个半胱氨酸组成[20]。而在非酰胺酶类 PGRP 中的半胱氨酸被丝氨酸取代。因此,这些关键位点的存在为HdhPGRP-SC2-like具有酰胺酶活性提供了理论基础。
3.2 HdhPGRP-SC2-like基因的组织表达模式
PGRP基因在不同物种中的组织分布表达存在一定差异。许多昆虫的PGRP基因主要在免疫器官中表达。在果蝇中,有13种肽聚糖识别蛋白基因转录形成至少17种肽聚糖识别蛋白,每个基因显示出不同的表达模式[1]。在昆虫中,PGRP-S和其他短型肽聚糖识别蛋白基因在血淋巴、表皮、上皮细胞及肠道中表达,长型肽聚糖识别蛋白基因在血细胞中表达量较高[21]。哺乳动物PGRP-L主要在肝脏中表达,并从肝脏分泌到血液中[22]。三角帆蚌(Hyriopsis cumingi)PGRPS1 mRNA表达量在肝胰腺中最高[23]。海湾扇贝(A.irradians)的PGRP是一种典型的短型PGRP,在其性腺、鳃、血细胞、肾和闭合肌中均有表达[24]。本研究中,HdhPGRP-SC2-like 基因在皱纹盘鲍各组织中呈组成型表达,鳃中表达量最高,其次为外套膜、消化腺,血细胞中表达量最低。鳃作为对各种外源性刺激的屏障,HdhPGRP-SC2-like在消化腺和鳃中的高表达水平暗示其在免疫和抗氧化防御中的重要作用。
3.3 HdhPGRP-SC2-like的免疫活性分析
PGRP对不同类型的PGN表现出不同的结合亲和力。从Manduca sexta中鉴定出的PGRP1只与可溶性Dap型PGN结合[25],来自Bombus ignitus的rPGRP对Lys型PGN具有亲和活性[26]。而rAdPGRP-SC2对Dap型和Lys型PGN均有结合亲和力[27]。本研究中发现,rHdhPGRP-SC2-like对藤黄微球菌PGN、大肠杆菌PGN均有结合能力,且与其他研究者的结论相似,表明该蛋白在革兰氏阳性菌中比在革兰氏阴性菌中具有更强的凝集和结合细菌能力[28]。
酰胺酶在多种生物中发挥重要的免疫作用,一些PGRP结构域与酰胺酶具有同源性,因此,可以水解不溶性PGN的N-乙酰胞苷酸和L-丙氨酸之间的酰胺键,从而降解PGN[4]。在日本盘鲍中发现的一种短型PGRP(AbPGRP),能够结合PGN,具有Zn2+依赖性的酰胺酶活性,且对弧菌具有较强的抗菌活性,这与先前在其他软体动物中报道的PGRP功能特性一致[29]。陈钰莹等[7]从皱纹盘鲍中克隆了肽聚糖识别蛋白(HdPGRP),重组表达产物rHdPGRP对革兰氏阳性菌藤黄微球菌具有显著的抑制作用,且具有Zn2+依赖的酰胺酶活性,可催化降解不溶性肽聚糖。本研究中发现,Zn2+对HdhPGRP-SC2-like发挥酰胺酶活性至关重要,表明PGRP在机体抵御细菌入侵等免疫防御中发挥重要作用。此外,一些研究表明,PGRP蛋白可以增强巨噬细胞对细菌的吞噬作用[30],并参与肠道免疫应答,在维持肠道内微生物稳态中发挥关键作用[31]。HdhPGRP-SC2-like是否也能促进细胞吞噬或具有其他功能,还有待进一步研究。
4 结论
1)从皱纹盘鲍体内克隆并鉴定了HdhPGRP-SC2-like基因,生物信息学分析表明,HdhPGRP-SC2-like蛋白含有4个Zn2+结合位点、5个酰胺酶催化位点和1个PGRP结构域,表明其在进化上高度保守。
2)HdhPGRP-SC2-like mRNA在皱纹盘鲍各组织中均有表达,在鳃、外套膜与消化腺中表达量较高,表明该基因参与机体的抗氧化和免疫防御。
3)重组表达产物rHdhPGRP-SC2-like具有肽聚糖结合活性和酰胺酶活性,在抵御细菌入侵机体的过程中发挥重要作用。
[1] CHRISTOPHIDES G K,ZDOBNOV E,BARILLAS-MURY C,et al.Immunity-related genes and gene families in Anopheles gambiae[J].Science,2002,298(5591):159-165.
[2] ZHANG Z,LONG Q X,XIE J P.Roles of peptidoglycan recognition protein (PGRP) in immunity and implications for novel anti-infective measures[J].Critical Reviews in Eukaryotic Gene Expression,2012,22(3):259-268.
[3] YOSHIDA H,KINOSHITA K,ASHIDA M.Purification of a peptidoglycan recognition protein from hemolymph of the silkworm,Bombyx mori[J].Journal of Biological Chemistry,1996,271(23):13854-13860.
[4] DZIARSKI R,GUPTA D.The peptidoglycan recognition proteins (PGRPs)[J].Genome Biology,2006,7(8):232.
[5] WEI X M,YANG D L,LI H Y,et al.Peptidoglycan recognition protein of Solen grandis (SgPGRP-S1) mediates immune recognition and bacteria clearance[J].Fish and Shellfish Immunology,2018,73:30-36.
[6] HU Z G,CAO X B,GUO M,et al.Identification and characterization of a novel short-type peptidoglycan recognition protein in Apostichopus japonicus[J].Fish and Shellfish Immunology,2020,99:257-266.
[7] 陈钰莹,韩怡静,刘相全,等.皱纹盘鲍肽聚糖识别蛋白在免疫防御中的作用[J].渔业科学进展,2022,43(4):234-242.
CHEN Y Y,HAN Y J,LIU X Q,et al.A peptidoglycan recognition protein(PGRP) from Haliotis discus hannai:possible roles in antibacterial properties[J].Progress in Fishery Sciences,2022,43(4):234-242.(in Chinese)
[8] MELLROTH P,STEINER H.PGRP-SB1:an N-acetylmuramoyl L-alanine amidase with antibacterial activity[J].Biochemical and Biophysical Research Communications,2006,350(4):994-999.
[9] MELLROTH P,KARLSSON J,STEINER H.A scavenger function for a Drosophila peptidoglycan recognition protein[J].Journal of Biological Chemistry,2003,278(9):7059-7064.
[10] ZHANG T,ZHU H,WANG J,et al.Monitoring bacterial community dynamics in abalone (Haliotis discus hannai) and the correlations associated with aquatic diseases[J].Water,2022,14(11):1769.
[11] LIANG S,LUO X,YOU W W,et al.Hybridization improved bacteria resistance in abalone:evidence from physiological and molecular responses[J].Fish and Shellfish Immunology,2018,72:679-689.
[12] QIAO K,WANG C,HUANG L,et al.Molecular characterization of a new tetrodotoxin-binding protein,peroxiredoxin-1,from Takifugu bimaculatus[J].International Journal of Molecular Sciences,2022,23(6):3071.
[13] LEE S Y,NAM Y K.Evaluation of reference genes for RT-qPCR study in abalone Haliotis discus hannai during heavy metal overload stress[J].Fisheries and Aquatic Sciences,2016,19:1-11.
[14] 乔琨,方春华,陈贝,等.皱纹盘鲍HdhTPX2基因在毕赤酵母中的表达及抗氧化活性研究[J].大连海洋大学学报,2019,34(4):463-469.
QIAO K,FANG C H,CHEN B,et al.Expression and antioxidant activity of HdhTPX2 gene from disk abalone Haliotis discus hannai Ino in yeast Pichia pastoris[J].Journal of Dalian Ocean University,2019,34(4):463-469.(in Chinese)
[15] JUMPER J,EVANS R,PRITZEL A,et al.Highly accurate protein structure prediction with AlphaFold[J].Nature,2021,596(7873):583-589.
[16] CCGI M.Molecular operating environment (MOE),2013.08[M].Montreal:Chemical Computing Group Inc,2016:354.
[17] GUAN R,MARIUZZA R A.Peptidoglycan recognition proteins of the innate immune system[J].Trends in Microbiology,2007,15(3):127-134.
[18] ZHANG R N,LI C T,REN F F,et al.Functional characterization of short-type peptidoglycan recognition proteins (PGRPs) from silkworm Bombyx mori in innate immunity[J].Developmental and Comparative Immunology,2019,95:59-67.
[19] HUANG Y,PAN J,LI X,et al.Molecular cloning and functional characterization of a short peptidoglycan recognition protein from triangle-shell pearl mussel (Hyriopsis cumingii)[J].Fish and Shellfish Immunology,2019,86:571-580.
[20] GELIUS E,PERSSON C,KARLSSON J,et al.A mammalian peptidoglycan recognition protein with N-acetylmuramoyl-L[J].Biochemical and Biophysical Research Communications,2003,306(4):988-994.
[21] DIMOPOULOS G,CHRISTOPHIDES G K,MEISTER S,et al.Genome expression analysis of Anopheles gambiae:responses to injury,bacterial challenge,and malaria infection[J].Proceedings of the National Academy of Sciences,2002,99(13):8814-8819.
[22] ZHANG Y,VAN DER FITS L,VOERMAN J S,et al.Identification of serum N-acetylmuramoyl-L-alanine amidase as liver peptidoglycan recognition protein 2[J].Biochimica Et Biophysica Acta-Proteins and Proteomics,2005,1752(1): 34-46.
[23] TAO Y,YANG Z,ZHANG X,et al.Molecular cloning and mRNA expression of the peptidoglycan recognition protein gene HcPGRP1 and its isoform HcPGRP1a from the freshwater mussel Hyriopsis cumingi[J].Genetics and Molecular Biology,2014,37(3):508-517.
[24] NI D,SONG L,WU L,et al.Molecular cloning and mRNA expression of peptidoglycan recognition protein (PGRP) gene in bay scallop (Argopecten irradians,Lamarck 1819)[J].Developmental and Comparative Immunology,2007,31(6):548-558.
[25] SUMATHIPALA N,JIANG H B.Involvement of Manduca sexta peptidoglycan recognition protein-1 in the recognition of bacteria and activation of prophenoloxidase system[J].Insect Biochemistry and Molecular Biology,2010,40(6):487-495.
[26] YOU H,WAN H,LI J,et al.Molecular cloning and characterization of a short peptidoglycan recognition protein (PGRP-S) with antibacterial activity from the bumble bee Bombus ignitus[J].Developmental and Comparative Immunology,2010,34(9):977-985.
[27] YANG H,LI X X,SONG W J,et al.Involvement of a short-type peptidoglycan recognition protein (PGRP) from Chinese giant salamanders Andrias davidianus in the immune response against bacterial infection[J].Developmental and Comparative Immunology,2018,88:37-44.
[28] BAI L,ZHOU Y,SHENG C,et al.Common carp peptidoglycan recognition protein 2 (CcPGRP2) plays a role in innate immunity for defense against bacterial infections[J].Fish and Shellfish Immunology,2023,133:108564.
[29] PREMACHANDRA H K A,ELVITIGALA D A S,WHANG I,et al.Identification of a novel molluscan short-type peptidoglycan recognition protein in disk abalone (Haliotis discus discus) involved in host antibacterial defense[J].Fish and Shellfish Immunology,2014,39(1):99-107.
[30] LI Q,CUI K,XU D,et al.Molecular identification of peptidoglycan recognition protein 5 and its functional characterization in innate immunity of large yellow croaker,Larimichthys crocea[J].Developmental and Comparative Immunology,2021,124:104130.
[31] GAO L,SONG X,WANG J.Gut microbiota is essential in PGRP-LA regulated immune protection against Plasmodium berghei infection[J].Parasites and Vectors,2020,13(1):3.