须鳗虾虎鱼 (Taenioides cirratus)隶属于鲈形目(Perciformes)虾虎鱼科(Gobiidae)近盲虾虎鱼亚科(Amblyopinae),是一种暖水性小型底栖鱼类,摄食小鱼、小虾、桡足类、其他底栖无脊椎动物和有机碎屑,喜穴居,主要栖息于河口半咸淡水水域,在中国主要分布于东海(中国近海)、南海(中国近海),日本、朝鲜、澳大利亚及印度洋北部也有分布[1]。该鱼因体表无鳞片、通体呈深红色,在部分地区被称为血罡鱼、红虫鱼等,是虾虎鱼类中经济价值相对较高的一种[2]。近年来,须鳗虾虎鱼入侵至巢湖、高邮湖、骆马湖和南四湖等内陆湖泊,并成为常见种[3-4],但在其原产地,如长江口、珠江口和南渡江口等水域,须鳗虾虎鱼资源量则不断下降。
近盲虾虎鱼亚科鱼类体型较小,扩散能力较弱,不同地理种群间易形成遗传分化[2,5],近年来,不断有其隐存种(cryptic species)和隐藏多样性(cryptic diversity)的报道。据《中国动物志》[1]记载,中国狼牙虾虎鱼属仅有拉氏狼牙虾虎鱼(Odontamblypus lacepedii)1个种[1],而Tang等[6]基于线粒体ND5基因序列和形态学特征分析认为,中国至少存在分布于东海、黄海、渤海湾沿海的拉氏狼牙虾虎鱼,分布于北部湾、南海北部、东海南部沿海的瑞贝卡狼牙虾虎鱼(O.rebecca),以及分布于东海南部、黄海南部沿海的狼牙虾虎鱼未定种(Odontamblypus sp.)3个有效种。对于须鳗虾虎鱼,据《中国动物志》[1]记载,中国仅有须鳗虾虎鱼1个种[1],而方嘉琪[2]基于形态学分析和线粒体COI和ND2基因序列的系统发育分析,鉴定到3个隐存种,认为海南须鳗虾虎鱼、珠江须鳗虾虎鱼和长江须鳗虾虎鱼可能是3个独立有效种。
线粒体基因组(mitochondrial genome)是一段结构简单、长度较短的 DNA 分子,进化速率较快,以较短的序列片段包含了丰富的遗传信息[7],其广泛应用于鱼类系统发育、种群多态性和种质资源保护等研究中[8-9]。近年来,基因测序技术的发展为研究线粒体全基因组提供了更便捷的方法。本研究中,采集了海南省南渡江口、广东省珠江口、长江水系的太湖与巢湖及淮河水系的南四湖的须鳗虾虎鱼,通过二代测序技术,获得了其线粒体全基因组序列,补充了中国须鳗虾虎鱼不同地理种群线粒体全基因组数据,并结合已有的相关线粒体基因组序列系统发育分析,探究须鳗虾虎鱼不同地理种群间的遗传学差异和隐藏多样性,以期为进一步明确须鳗虾虎鱼的分类地位和资源保护提供参考依据。
1 材料与方法
1.1 采样站位的设置
2017—2020年,使用定置(串联)倒须笼壶(网长为8 m,网高和网宽均为30 cm,网目2a=0.8 cm),在海南省南渡江口(110°26′8.326″E,20°01′39.284″N)、广东省珠江口(113°36′10.861″E,22°54′59.882″N)、长江水系的太湖(120°13′11.057″E,31°30′4.255″N)与巢湖(117°24′57.150″E,31°41′8.628″N)和淮河水系的南四湖(117°13′56.906″E,34°41′4.693″N)采集须鳗虾虎鱼(图1),并获得样本165尾。从样本取少量肌肉组织置于2.0 mL离心管中,用无水乙醇浸泡固定,用于DNA的提取。
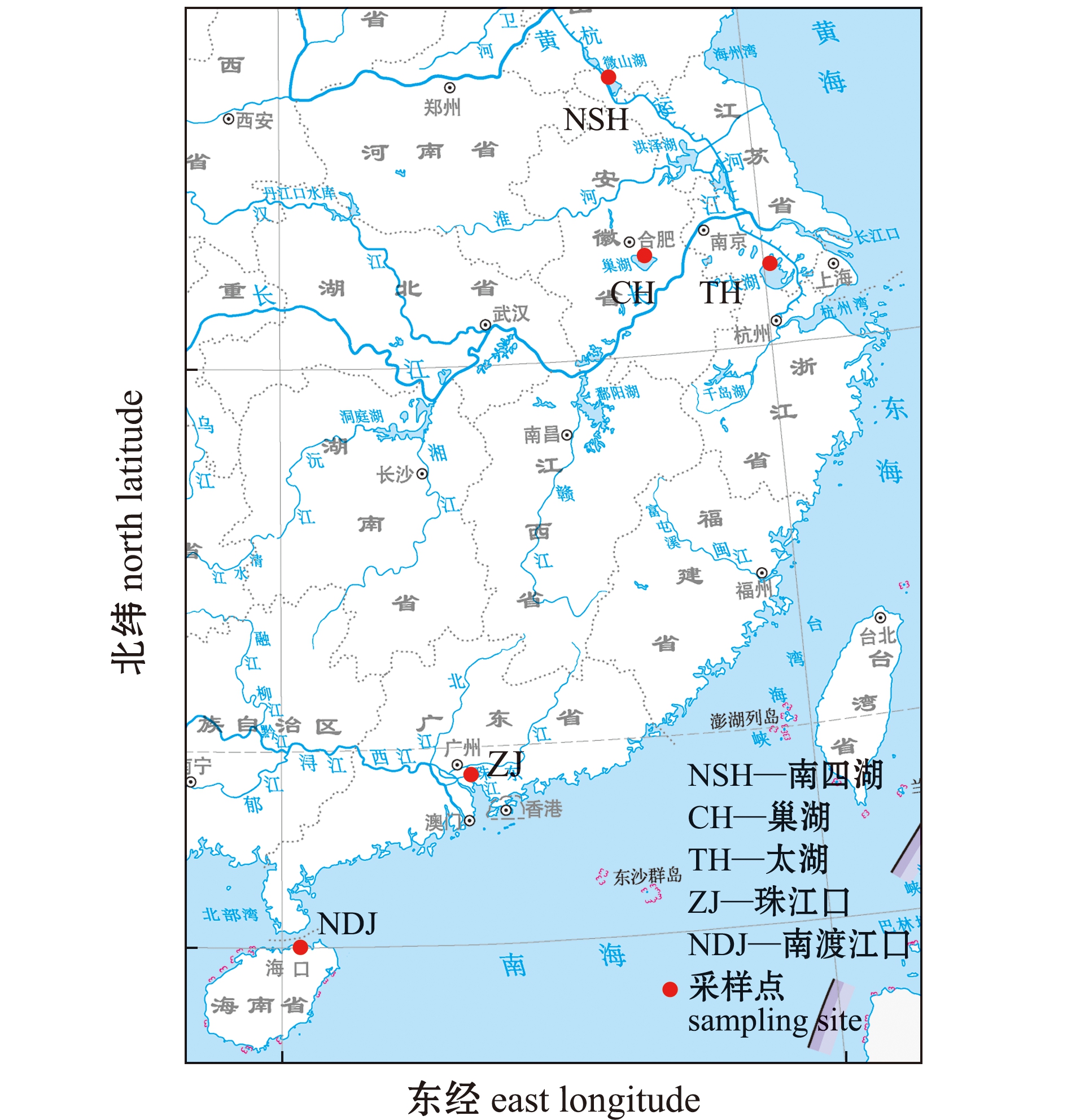
本图基于自然资源部标准地图服务网站GS(2019)1694号标准地图为底图,底图边界无修改。
The figure is based on the standard map GS(2019)1694 in the standard map service website of Ministry of Natural Resources of the People’s Republic of China,with no modifications of the boundaries in the standard map.
图1 须鳗虾虎鱼采样点分布
Fig.1 Sampling sites of Taenioides cirratus
1.2 方法
1.2.1 线粒体基因组测序 采用高盐法提取样本基因组DNA[10],用10 g/L琼脂糖凝胶电泳检测DNA质量,用NanoDrop ND-2000超微量分光光度计测定DNA纯度和浓度。基于线粒体COI和Cyt b基因序列(登录号:OM849673~OM849775)进行群体遗传特征分析,筛选代表性个体进行线粒体全基因组测序。基于COI和Cyt b基因序列构建的系统发育树表明,珠江口样本和南渡江口样本各聚为3个分枝,太湖、巢湖和南四湖样本聚为1个分枝[11]。从珠江口(ZJ1、ZJ2和ZJ3)和南渡江口(NDJ1、NDJ2和NDJ3)种群各选3尾,巢湖(CH1、CH2)、太湖(TH1、TH2)和南四湖(NSH1、NSH2)种群各选取2尾,共筛选12尾样本。
采用Covaris仪超声波将样本基因组DNA打断成350 bp左右片段,对DNA片段末端修复,在3′端加A碱基和测序接头。对连接产物进行PCR扩增,用磁珠回收产物并纯化,构建文库。DNA文库质检合格后使用Illumina Hiseq高通量测序平台进行双末端(paired end)测序,每个样品测序数据量不少于6 Gb。测序由生工生物工程(上海)股份有限公司完成。过滤测序数据中的低质量读数和接头序列,使用NOVOPlasty进行de novo组装,参考已发表的须鳗虾虎鱼线粒体基因组序列[12](登录号:KJ944420.1),使用BioEdit 7.2.5软件对拼接的序列进行校对[13],采用MITOS 2(http://mitos.bioinf.unileipzig.de/index.py)在线软件进行基因注释。将获得的须鳗虾虎鱼完整线粒体基因组序列提交至NCBI(National Center for Biotechnology Information)数据库(表1)。
表1 线粒体系统发育分析所用基因组
Tab.1 Complete mitochondrial genomes used in phylogenetic analysis

注:*表示样本线粒体全基因组序列为本研究获得。
Note:* markes the specimens in present study.
物种species采集地location登录号accession No.须鳗虾虎鱼(T.cirratus)∗巢湖OP326518须鳗虾虎鱼(T.cirratus)∗巢湖OP326520须鳗虾虎鱼(T.cirratus)∗太湖OP326525须鳗虾虎鱼(T.cirratus)∗太湖OP326526须鳗虾虎鱼(T.cirratus)∗南四湖OP326523须鳗虾虎鱼(T.cirratus)∗南四湖OP326524须鳗虾虎鱼(T.cirratus)∗珠江OP326527须鳗虾虎鱼(T.cirratus)∗珠江OP326528须鳗虾虎鱼(T.cirratus)∗珠江OP326529须鳗虾虎鱼(T.cirratus)∗南渡江OP326519须鳗虾虎鱼(T.cirratus)∗南渡江OP326521须鳗虾虎鱼(T.cirratus)∗南渡江OP326522须鳗虾虎鱼(T.cirratus)[11]台湾KJ944420.1须鳗虾虎鱼(T.cirratus)[2]珠江MK541898.1须鳗虾虎鱼(T.cirratus)[2]海南MW682859.1须鳗虾虎鱼(T.cirratus)[2]太湖MK541900.1鳗虾虎鱼属未定种(Taenioides sp.)—MK541899.1鳗虾虎鱼属未定种(Taenioides sp.Ruosonghe)—OL625024.1鲡形鳗虾虎鱼(T.anguillaris)[14]兴化湾KT188772.1红狼牙虾虎鱼(O.rubicundus)[15]舟山JX891626.1钝孔虾虎鱼(A.arctocephalus)—NC_058264.1大弹涂鱼(B.pectinirostris)[16]慈溪NC_016195.1大弹涂鱼(B.pectinirostris)[17]北部湾MN909967.1河川沙塘鳢(O.potamophila)[18]太湖KF305680.1
1.2.2 系统发育分析 通过须鳗虾虎鱼线粒体基因序列在NCBI数据库中进行Blast比对,下载与须鳗虾虎鱼线粒体基因序列同源性较高的鲡形鳗虾虎鱼(Taenioides anguillaris)[14] 、红狼牙虾虎鱼(Odontamblyopus rubicundus)[15][伍汉霖等[1]认为中国的红狼牙虾虎鱼(O.rubicundus)为误鉴,实为拉氏狼牙虾虎鱼(O.lacepedii)的异名]、钝孔虾虎鱼(Amblyotrypauchen arctocephalus)、大弹涂鱼(Boleophthalmus pectinirostris)[16-17]、2个鳗虾虎鱼属未定种个体,以及已发表的须鳗虾虎鱼长江水系、珠江、海南和台湾个体的线粒体基因组序列,使用河川沙塘鳢(Odontobutis potamophila)[18]作为外群(表1),进行系统发育分析。
采用BioEdit 7.2.5截取样本线粒体基因组的13个蛋白编码基因(protein coding genes,PCGs)和2个rRNA 基因序列,拼接成1条序列[19],使用DnaSP 5.0统计样本的单倍型和突变位点[20]。使用Mr Bayes 3.2.7构建贝叶斯法(Bayesian inferences,BI)系统发育树[21],运行4条独立的马尔可夫链(Markov chains),运行50 000 000代,每1 000代抽样1次,丢弃前25%,当分列频率平均标准差小于0.01时认为分析趋于稳定,计算各分枝的后验(posterior)支持率;使用MEGA 7.0构建最大似然法(maximum likelihood,ML)系统发育树,使用 bootstrap(重复次数5 000)检验各分枝的置信度[22]。由于缺乏关于须鳗虾虎鱼线粒体基因序列分歧速率的报道,仅Mukai等[23]报道了虾虎鱼科吻虾虎鱼属未定种(Rhinogobius sp.)线粒体ND5基因序列的分化率为3.89%/Ma,该进化速率以地质事件作为校正点。因此,本研究中使用3.89%/Ma作为须鳗虾虎鱼的线粒体蛋白编码基因和rRNA基因的分歧速率。
2 结果与分析
2.1 线粒体基因组与结构
须鳗虾虎鱼12尾样本线粒体基因组全长为16 641~16 978 bp,均包含13个蛋白编码基因、22个tRNA基因、2个rRNA基因和控制区(D-loop)(图2)。线粒体全基因组长度差异主要是由D-loop长度(975~1 314 bp)差异造成的,各样本的编码基因序列长度较为一致。各样本线粒体基因组序列的碱基组成接近,A、T、C和G平均含量分别为 28.47%、26.65%、29.08%和15.80%,A+T含量(55.12%)大于C+G含量(44.88%)。
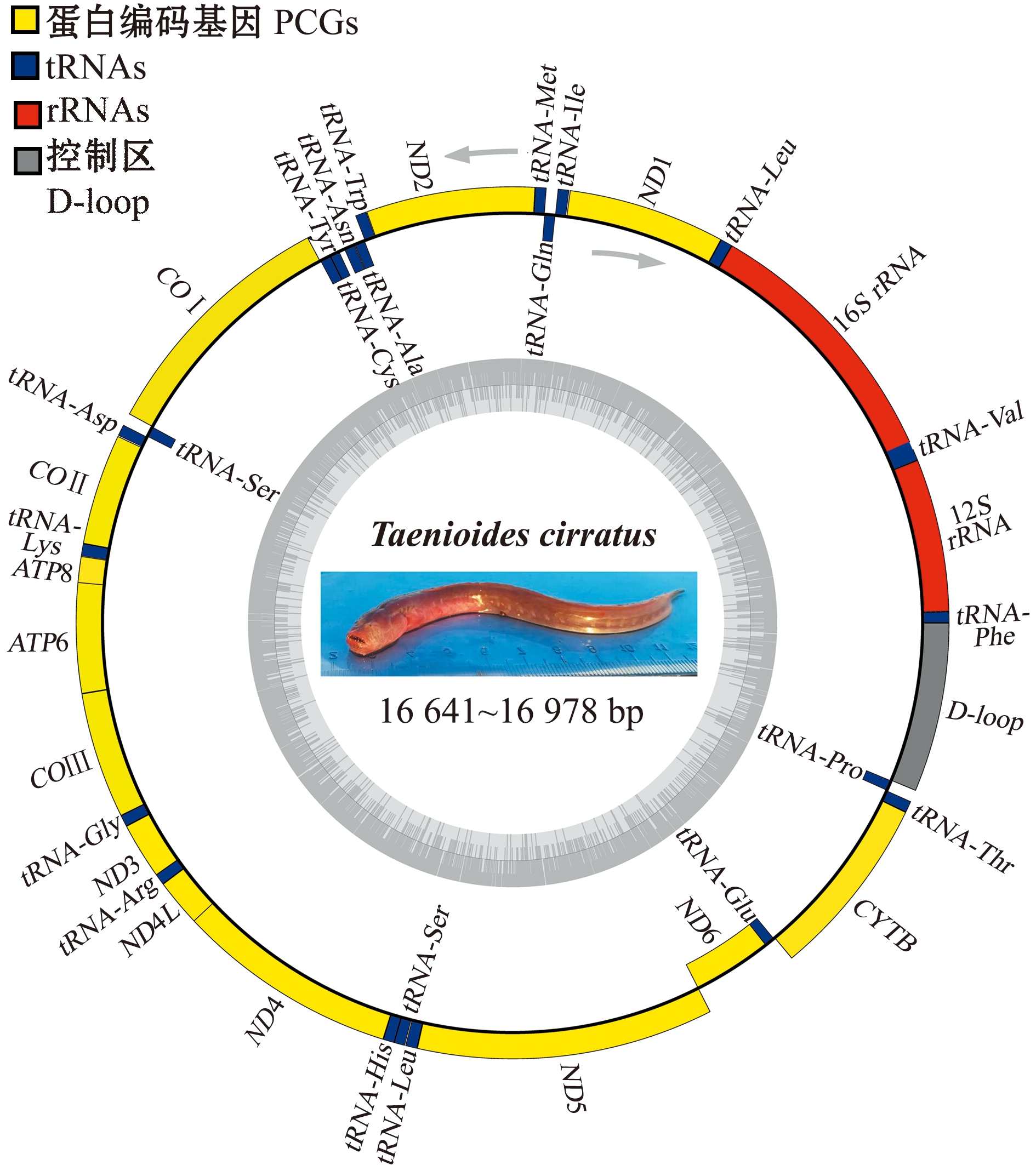
图2 须鳗虾虎鱼线粒体基因组结构
Fig.2 Gene map of the mitochondrial genome of Taenioides cirratus
13个蛋白编码基因中,仅COI基因以GTG为起始密码子,其他基因均以ATG为起始密码子;终止密码子有TAG、TAA和不完全终止密码子TA和T。12尾样本中9个蛋白编码基因的终止密码子一致,4个存在差异:ND1基因,南渡江口3号样本的终止密码子为TAG,其他样本均为TAA;ND2基因,长江和淮河水系样本的终止密码子为TAG,其他样本均为TAA;ND5和ND6基因,珠江口1号样本、长江水系及淮河水系样本的终止密码子为TAG,其他样本均为TAA。以巢湖样本为例,其线粒体基因组结构特征如表2所示,其他样本线粒体基因组结构与巢湖类似。
表2 巢湖须鳗虾虎鱼线粒体基因组结构特征(编号CH1)
Tab.2 Characteristics of complete mitochondrial genome of Taenioides cirratus from the Chaohu Lake (No. CH1)
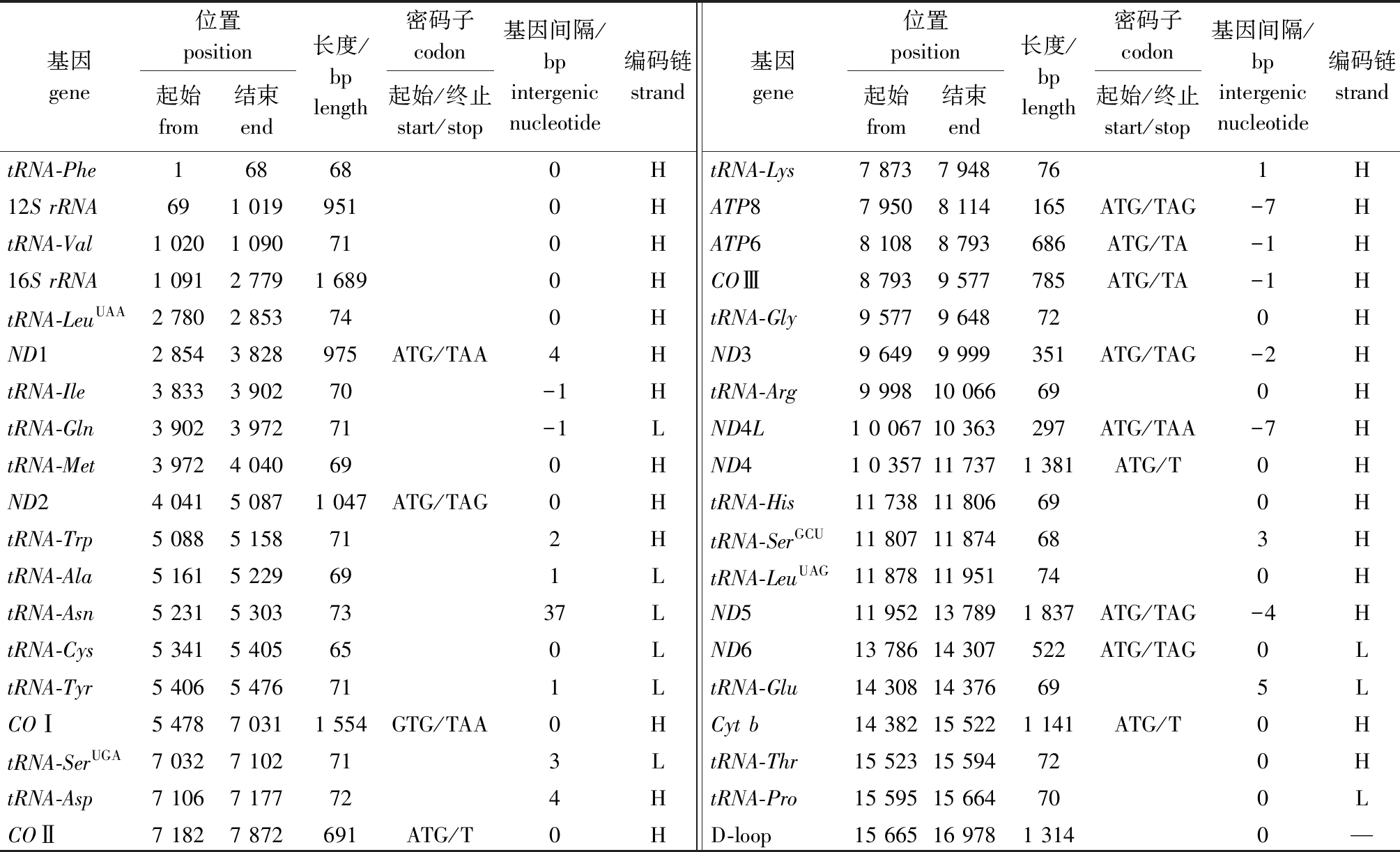
基因gene位置position起始from结束end长度/bplength密码子codon起始/终止start/stop基因间隔/bpintergenic nucleotide编码链strandtRNA-Phe168680H12S rRNA691 0199510HtRNA-Val1 0201 090710H16S rRNA1 0912 7791 6890HtRNA-LeuUAA2 7802 853740HND12 8543 828975ATG/TAA4HtRNA-Ile3 8333 90270-1HtRNA-Gln3 9023 97271-1LtRNA-Met3 9724 040690HND24 0415 0871 047ATG/TAG0HtRNA-Trp5 0885 158712HtRNA-Ala5 1615 229691LtRNA-Asn5 2315 3037337LtRNA-Cys5 3415 405650LtRNA-Tyr5 4065 476711LCOⅠ5 4787 0311 554GTG/TAA0HtRNA-SerUGA7 0327 102713LtRNA-Asp7 1067 177724HCOⅡ7 1827 872691ATG/T0H基因gene位置position起始from结束end长度/bplength密码子codon起始/终止start/stop基因间隔/bpintergenic nucleotide编码链strandtRNA-Lys7 8737 948761HATP87 9508 114165ATG/TAG-7HATP68 1088 793686ATG/TA-1HCOⅢ8 7939 577785ATG/TA-1HtRNA-Gly9 5779 648720HND39 6499 999351ATG/TAG-2HtRNA-Arg9 99810 066690HND4L1 0 06710 363297ATG/TAA-7HND41 0 35711 7371 381ATG/T0HtRNA-His11 73811 806690HtRNA-SerGCU11 80711 874683HtRNA-LeuUAG11 87811 951740HND511 95213 7891 837ATG/TAG-4HND613 78614 307522ATG/TAG0LtRNA-Glu14 30814 376695LCyt b14 38215 5221 141ATG/T0HtRNA-Thr15 52315 594720HtRNA-Pro15 59515 664700LD-loop15 66516 9781 3140—
2.2 系统发育分析
12尾样本的13个蛋白编码基因和2个rRNA基因组合序列长为14 059~14 062 bp,巢湖的2个样本共享1个单倍型,其他样本互为独立单倍型,共得到11个单倍型,检测到2 165个突变位点。基于ML法和基于BI法构建的系统发育树结构完全一致(本研究中仅列出其中1个),11个单倍型聚为3枝,其中,太湖、巢湖和南四湖的5个单倍型、珠江口的1个单倍型和已发表的1个太湖须鳗虾虎鱼单倍型(MK5419001.1)聚成分枝Ⅰ,1个南渡江口单倍型与已发表的1个台湾须鳗虾虎鱼单倍型(KJ9444201.1)、1个福建兴化湾鲡形鳗虾虎鱼单倍型(KT188772.1)、1个舟山红狼牙虾虎鱼单倍型(JX891626.1)和1个鳗虾虎鱼属未定种单倍型(OL625024.1)聚为分枝Ⅱ;2个珠江口单倍型、2个南渡江口单倍型和已发表的1个珠江须鳗虾虎鱼单倍型(MK541898.1)、1个海南须鳗虾虎鱼单倍型(MW682859.1)和1个鳗虾虎鱼属未定种单倍型(MK541899.1)聚成分枝Ⅲ(图3)。
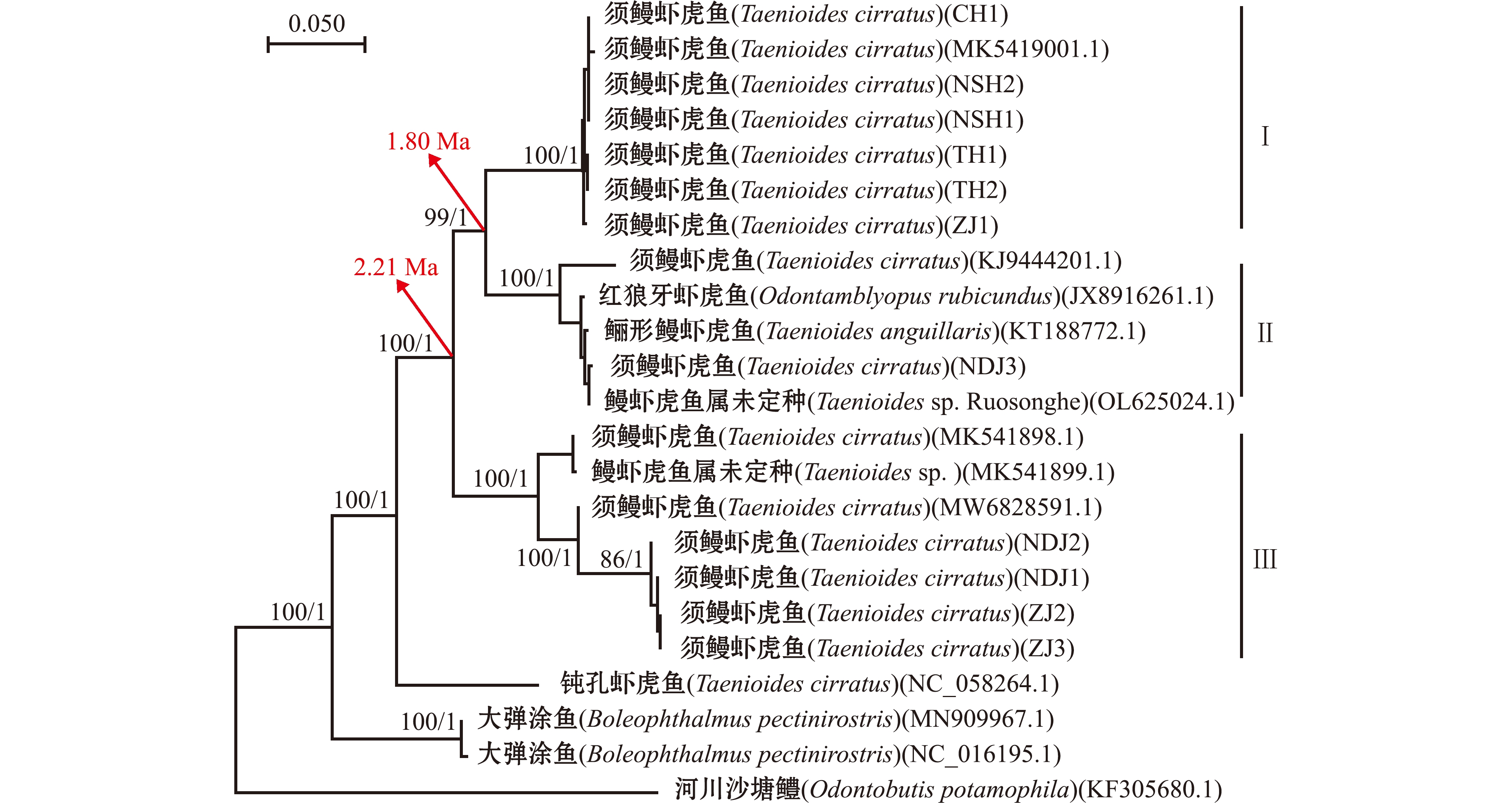
分枝上数值为最大似然分析bootstrap校验百分数和贝叶斯后验概率。
Values on branches indicate the bootstrap proportions from maximum likelihood analysis and the posterior probabilities from Bayesian analysis.
图3 基于13个蛋白编码基因和2个rRNA基因序列构建的最大似然法系统发育树
Fig.3 ML phylogenetic tree based on the 13 PCGs and 2 rRNA genes sequences
3 讨论
3.1 须鳗虾虎鱼线粒体基因组结构特点
本研究中,12个须鳗虾虎鱼样本的线粒体全基因组结构与其他硬骨鱼相似[8],在碱基组成上同样具有明显的A+T偏好。线粒体基因组长度为16 641~16 978 bp,长度的变化主要是因为部分个体D-loop的3′端存在一段143 bp的串联重复序列。D-loop是线粒体DNA中变化最复杂的区域,其进化速率是线粒体DNA其他区段的 5~10倍,近缘种甚至同种不同个体间控制区均会出现较大差异,硬骨鱼类控制区重复序列通常介于几十bp到300 bp之间[8]。D-loop 3′末端的重复序列在虾虎鱼类中普遍存在,如本研究中下载分析的须鳗虾虎鱼(KJ9444201.1)[12]、鲡形鳗虾虎鱼(KT188772.1)[14]、大弹涂鱼(MN909967.1、NC_016195.1)[16-17]和钝孔虾虎鱼(NC_058264.1)等在D-loop均存在一段131~144 bp的重复序列,重复序列的起始位置与本研究中须鳗虾虎鱼类似。D-loop重复序列在进化中的作用有待进一步探究。
3.2 须鳗虾虎鱼的系统发育分析
本研究中构建的系统发育树显示,须鳗虾虎鱼存在3个分歧较深的分枝,其中,分枝Ⅱ的组成最复杂,由海南和台湾的须鳗虾虎鱼与红狼牙虾虎鱼、鲡形鳗虾虎鱼共同组成,表明须鳗虾虎鱼非单系,与红狼牙虾虎鱼、鲡形鳗虾虎鱼构成并系群。但本研究中用于系统发育分析的红狼牙虾虎鱼和鲡形鳗虾虎鱼的样本数量较少,二者与须鳗虾虎鱼更全面的系统发育关系有待进一步研究。方嘉琪[2]根据COI基因和ND2基因序列,构建了近盲虾虎鱼亚科 ML 系统发育树,发现长江水系、珠江口和海南的须鳗虾虎鱼各自聚成1枝,并结合形态学特征分析认为,长江水系须鳗虾虎鱼、海南须鳗虾虎鱼和珠江口须鳗虾虎鱼可能是3个独立物种。本研究表明,须鳗虾虎鱼系统发育树虽有3个分枝,但并不是每个地理种群形成一个独立分枝,如分枝Ⅰ由长江水系样本和部分珠江口样本组成,分枝Ⅲ由部分珠江口样本和部分南渡江口样本组成。Zhang等[11]基于线粒体序列COI和Cyt b基因序列构建的系统发育树表明,长江水系、珠江口和南渡江口种群也未形成独立的系统发育枝,长江水系个体与部分珠江口个体形成一个发育枝,南渡江口部分个体与珠江口部分个体形成一个发育枝,与本研究中结果较吻合。故笔者认为,长江水系须鳗虾虎鱼、海南须鳗虾虎鱼和珠江须鳗虾虎鱼为3个有效种的观点有待商榷。
经测年分析显示,须鳗虾虎鱼3个进化枝的分歧发生在更新世早期(2.2~1.8 Ma)(图3),这可能与这一时期青藏高原的快速隆起有关。青藏高原在2.6~1.7 Ma经历了“青藏运动”的B期和C期,经过这两个阶段的快速隆起,青藏高原平均海拔达到2 000 m以上,当代亚洲季风系统形成,高原边缘河流溯源侵蚀并强烈下切,中国现代水系格局基本形成[24]。水系格局和季风的改变驱动鱼类谱系格局的变化,在中国裂腹鱼类(Schizothoracine)[25]、![]() (Hemiculter leucisculus)[26]和糙隐鳍鲇(Pterocryptis anomala)[27]等鱼类中有广泛报道。笔者推测,更新世早期的青藏高原快速隆起和当代亚洲季风形成带来的中国水系的分化,导致了须鳗虾虎鱼不同地理种群的分离。须鳗虾虎鱼是一种小型底栖鱼类,扩散能力相对较弱,主要栖息在河口盐度较低的水域,盐度较高的海水可能是其扩散的重要地理屏障。须鳗虾虎鱼系统发育树分枝Ⅰ和分枝Ⅲ均包含了2个不同地理种群的个体,各个水系的须鳗虾虎鱼种群均未形成独立分枝,表明相邻水系的须鳗虾虎鱼种群间存在基因交流。
(Hemiculter leucisculus)[26]和糙隐鳍鲇(Pterocryptis anomala)[27]等鱼类中有广泛报道。笔者推测,更新世早期的青藏高原快速隆起和当代亚洲季风形成带来的中国水系的分化,导致了须鳗虾虎鱼不同地理种群的分离。须鳗虾虎鱼是一种小型底栖鱼类,扩散能力相对较弱,主要栖息在河口盐度较低的水域,盐度较高的海水可能是其扩散的重要地理屏障。须鳗虾虎鱼系统发育树分枝Ⅰ和分枝Ⅲ均包含了2个不同地理种群的个体,各个水系的须鳗虾虎鱼种群均未形成独立分枝,表明相邻水系的须鳗虾虎鱼种群间存在基因交流。
3.3 须鳗虾虎鱼资源管理
本研究中,须鳗虾虎鱼具有3个分歧较深的分枝,该结果与基于线粒体序列COI和Cyt b基因序列的系统发育分析结果较为吻合[11]。通常情况下,不同系统发育分枝的地理种群被称为进化重要单位(evolutionary significant unit)和管理单位(management unit)[28-29]。虽然在巢湖、南四湖等内陆湖泊,须鳗虾虎鱼作为本地入侵种,表现出较强的入侵能力,但根据笔者于2018—2020年的调查,须鳗虾虎鱼在原产地资源量不断下降,长江口目前已较难采集到须鳗虾虎鱼,珠江口和南渡江口的须鳗虾虎鱼资源量较以前也大幅下降。考虑到不同系统发育分枝之间显著的遗传学差异,建议未来将南渡江口、珠江口和长江水系的须鳗虾虎鱼种群视为不同管理单位进行资源管理。
4 结论
1)须鳗虾虎鱼线粒体全基因组长为16 641~16 978 bp,包含13个蛋白编码基因、22个tRNA基因、2个rRNA基因和1个D-loop,全基因组长度差异主要是由D-loop长度(975~1 314 bp)差异造成的。
2)须鳗虾虎鱼个体形成3个高支持率进化枝,每个进化枝均包含了至少2个水系的须鳗虾虎鱼个体,太湖、巢湖和南四湖的须鳗虾虎鱼个体聚为1个进化枝,南渡江口和珠江口须鳗虾虎鱼个体均分属2个进化枝。建议未来将南渡江口、珠江口和长江水系的须鳗虾虎鱼种群视为不同管理单位进行资源管理。
[1] 伍汉霖,钟俊生.中国动物志:硬骨鱼纲 鲈形目Ⅴ虾虎鱼亚目[M].北京:科学出版社,2008.
WU H L,ZHONG J S.Fauna sinica:Osteichthyes,Perciformes Ⅴ,Gobioidei[M].Beijing:Science Press,2008.(in Chinese)
[2] 方嘉琪.中国沿海近盲鰕虎鱼亚科鱼类的系统发育及三个种类的分子系统地理学初步研究[M].舟山:浙江海洋大学,2021.
FANG J Q.The phylogeny of Amblyopinae fishes in coastal waters of China and a preliminary study on the molecular phylogeography of three Amblyopinae species[M].Zhoushan:Zhejiang Ocean University,2021.(in Chinese)
[3] QIN J,CHENG F,ZHANG L,et al.Invasions of two estuarine gobiid species interactively induced from water diversion and saltwater intrusion[J].Management of Biological Invasions,2019,10(1):139-150.
[4] LIANG Y Y,FANG T,LI J,et al.Age,growth and reproductive traits of invasive goby Taenioides cirratus in the Chaohu Lake,China[J].Journal of Applied Ichthyology,2020,36(2):219-226.
[5] KURITA T,YOSHINO T.Cryptic diversity of the eel goby,genus Taenioides (Gobiidae:Amblyopinae),in Japan[J].Zoological Science,2012,29(8):538-545.
[6] TANG W,LSHIMATSU A,FU C,et al.Cryptic species and historical biogeography of eel gobies (Gobioidei:Odontamblyopus) along the northwestern Pacific coast[J].Zoological Science,2010,27(1):8-13.
[7] GRAY M W,BURGER G,LANG B F.Mitochondrial evolution[J].Science,1999,283(5407):1476-1481.
[8] 周传江,马爱喆,汪曦,等.鱼类线粒体基因组研究进展[J].河南师范大学学报(自然科学版),2019,47(2):74-82.
ZHOU C J,MA A Z,WANG X,et al.Progress on fish mitochondrial genome[J].Journal of Henan Normal University (Natural Science Edition),2019,47(2):74-82.(in Chinese)
[9] 程佩琳,俞丹,刘焕章,等.基于线粒体基因组全序列的鲟形目鱼类(Pisces:Acipenseriformes)的分子系统发育重建[J].水生生物学报,2021,45(3):487-494.
CHENG P L,YU D,LIU H Z,et al.Molecular phylogeny of Acipenseriformes based on complete mitochondrial genome sequence[J].Acta Hydrobiologica Sinica,2021,45(3):487-494.(in Chinese)
[10] ALJANABI S M,MARTINEZ I.Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques[J].Nucleic Acids Research,1997,25(22):4692-4693.
[11] ZHANG G Q,CHEN C,LU W X,et al.Genetic diversity and phylogeography of Taenioides cirratus in five geographical populations based on mitochondrial COI and Cytb gene sequences[J/OL].Journal of Applied Ichthyology,2023:1-9.https://doi.org/10.1155/2023/4459823.
[12] CHEN I S,WEN Z H.The complete mitochondrial genome of whiskered eel goby Taenioides cirratus (Perciformes,Gobioidei)[J].Mitochondrial DNA Part A:DNA Mapping,Sequencing,and Analysis,2016,27(2):1509-1511.
[13] HALL T.Bioedit:a user-friendly biological sequence alignment editor and analysis program for windows 95/98/nt[J].Nuclc Acids Symposium Series,1999,41(41):95-98.
[14] WEI H,MA H,MA C,et al.Isolation and characterization of the complete mitochondrial genome of Taenioides anguillaris (Gobiidae:Amblyopinae) with phylogenetic consideration [J].Mitochondrial DNA Part A:DNA Mapping,Sequencing,and Analysis,2016,27(6):4681-4682.
[15] 金逍逍,孙悦娜,王日昕,等.虾虎鱼类线粒体全基因组序列结构特征分析及系统发育关系探讨[J].遗传,2013,35(12):1391-1402.
JIN X X,SUN Y N,WANG R X,et al.Characteristics and phylogenetic analysis of mitochondrial genome in the gobies[J].Hereditas,2013,35(12):1391-1402.(in Chinese)
[16] LIU Z Z,WANG C T,MA L B,et al.Complete mitochondrial genome of the mudskipper Boleophthalmus pectinirostris (Perciformes,Gobiidae):repetitive sequences in the control region[J].Mitochondrial DNA,2012,23(1):31-33.
[17] PAN C Y,XIAO S,CHEN X L,et al.The complete mitochondrial genome of the mudskipper,Boleophthalmus pectinirostris (Gobiiformes,Oxudercidae) from Beibu Bay[J].Mitochondrial DNA Part B:Resources,2021,6(4):1337-1338.
[18] MA Z H,YANG X F,ZHANG X J,et al.Complete mitochondrial genome of the freshwater goby Odontobutis potamophila (Perciformes:Odontobutidae)[J].Mitochondrial DNA,2015,26(2):299-300.
[19] YUN S W,KIM H T.The complete mitochondrial genome of Korean endemic loach Iksookimia pacifica (Teleostei,Cypriniformes,Cobitidae)[J].Mitochondrial DNA Part B:Resources,2022,7(7):1257-1259.
[20] LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
[21] RONQUIST F,HUELSENBECK J P.MrBayes 3:Bayesian phylogenetic inference under mixed models[J].Bioinformatics,2003,19(12):1572-1574.
[22] KUMAR S,STECHER G,TAMURA K.MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1870-1874.
[23] MUKAI T,NAKAMURA S,SUZUKI T,et al.Mitochondrial DNA divergence in yoshinobori gobies (Rhinogobius species complex) between the Bonin Islands and the Japan-Ryukyu Archipelago[J].Ichthyological Research,2005,52(4):410-413.
[24] 李吉均,方小敏.青藏高原隆起与环境变化研究[J].科学通报,1998,43(15):1569-1574.
LI J J,FANG X M.Study on uplift of Qinghai-Tibet Plateau and environmental change[J].Chinese Science Bulletin,1998,43(15):1569-1574.(in Chinese)
[25] HE D K,CHEN Y F.Molecular phylogeny and biogeography of the highly specialized grade Schizothoracine fishes (Teleostei:Cyprinidae) inferred from cytochrome b sequences[J].Chinese Science Bulletin,2007,52(6):777-788.
[26] CHEN W T,ZHONG Z X,DAI W,et al.Phylogeographic structure,cryptic speciation and demographic history of the sharpbelly (Hemiculter leucisculus),a freshwater habitat generalist from Southern China[J].BMC Evolutionary Biology,2017,17(1):216.
[27] CHEN W T,LI Y F,HE Y J,et al.Characterization of two complete mitochondrial genomes of Pterocryptis anomala (Siluridae) and its phylogeny and cryptic diversity[J].Biologia,2021,76(2):613-621.
[28] MORISHIMA K,NAKAMURA-SHIOKAWA Y,BANDO E,et al.Cryptic clonal lineages and genetic diversity in the loach Misgurnus anguillicaudatus (Teleostei:Cobitidae) inferred from nuclear and mitochondrial DNA analyses[J].Genetica,2008,132(2):159-171.
[29] SONG Y S,BAE S E,KANG J H,et al.Cryptic diversity in the inshore hagfish,Eptatretus burgeri (Myxinidae,Pisces) from the northwest Pacific[J].Mitochondrial DNA Part B:Resources,2020,5(3):3410-3414.