胰岛素诱导基因(insulin-induced gene,INSIG)是近年来发现的调控脂质代谢的重要因子,该基因通过维持细胞内脂质平衡发挥作用[1]。胰岛素诱导基因包括两个成员INSIG1和INSIG2,这两个基因在人体脂肪代谢、身体质量指数等方面发挥重要作用,其多态性与机体增重、肥胖和糖尿病等性状有关[2-5]。作为反馈调节胆固醇和脂肪合成的重要因子,INSIG1能有效调控脂肪细胞的分化、成熟和脂质合成[6]。INSIG1基因在不同物种、不同组织中的表达量有所差异,关于高等动物INSIG1基因的克隆、分离和表达调控等研究较多[7-10],但该基因在水产动物中的遗传和功能研究方面几乎是空白。
黄河鲤(Cyprinus carpio haematoperus)位居中国四大淡水名鱼之首,具有典型的文化与经济价值,对黄河流域各省水产养殖产业具有重要的经济支撑作用,黄河鲤的品种改良对促进黄河鲤健康养殖及产业发展具有极大的推动作用。鲤(Cyprinus carpio)基因组草图于2014年公布[11],为鲤基因组学的分析提供了重要参考。前期研究发现,INSIG1基因被定位于黄河鲤生长性状QTL区间内[12],暗示INSIG1基因对黄河鲤生长有调控作用,然而关于该基因在鲤中的遗传和功能信息尚未见报道。本研究中,通过对黄河鲤INSIG1基因的序列特征及其表达分析,同时挖掘基因单核苷酸多态性位点并与生长性状进行关联分析,探究了黄河鲤INSIG1基因的功能及其对生长性状的调控作用,以期为黄河鲤的育种工作提供新的候选基因和分子遗传标记,为黄河鲤的品种选育提供科学参考。
1 材料与方法
1.1 材料
从河南省水产科学研究院培育的黄河鲤亲本中挑选雌、雄亲本各26尾,随后采用人工授精的方法对这些亲本随机进行一对一交配,从而得到黄河鲤的26个全同胞家系。待受精卵孵化成幼苗后,随机从各个家系中选择400尾鱼苗置于规格约5 000 m2的鱼塘中养殖。
1.2 方法
1.2.1 黄河鲤生长性状的测定与基因组DNA提取 黄河鲤养殖至8月龄时,从中随机挑选1 000尾黄河鲤子代测定其体长(LB,cm)和体质量(WB,g),并计算肥满度(K),即![]() 剪取1 000尾子代的部分尾鳍组织置于无水乙醇中保存。采用酚-氯仿法进行基因组DNA提取,并利用NanoDrop 2000超微量分光光度计检测DNA的质量和浓度,DNA样品置于-20 ℃冰箱中保存备用。
剪取1 000尾子代的部分尾鳍组织置于无水乙醇中保存。采用酚-氯仿法进行基因组DNA提取,并利用NanoDrop 2000超微量分光光度计检测DNA的质量和浓度,DNA样品置于-20 ℃冰箱中保存备用。
1.2.2 RNA提取与cDNA合成 随机选择3尾黄河鲤进行解剖和组织取样,每尾鱼采集9个组织,包括脑、皮肤、心脏、鳃、脾、肌肉、肾、肠和肝,置于Trizol提取液中并进行RNA提取,然后采用NanoDrop 2000超微量分光光度计检测RNA的质量和浓度,最后利用反转录试剂盒将RNA反转录为cDNA,并检测cDNA的质量和浓度,将cDNA样品置于-20 ℃冰箱中保存备用。
1.2.3 INSIG1生物信息学分析 从NCBI数据库中下载鲤INSIG1基因的DNA序列(GenBank accession No.NC_056603.1)和cDNA序列(GenBank accession No.XM_042728053.1)。对黄河鲤INSIG1蛋白用ProtParam Tool在线程序(https://web.expasy.org/protparam/)进行理化参数分析;采用SignalP 5.0在线程序(https://services. healthtech.dtu.dk/service.php?SignalP-5.0)进行信号肽功能分析;采用DeepTMHMM在线程序(https://dtu.biolib.com/DeepTMHMM)预测跨膜结构;采用Protscale在线程序(https://web.expasy.org/protscale/)进行疏水性分析;采用PRABI(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)在线软件分析蛋白的二级结构和三级结构。
利用NCBI BLAST工具在数据库中获取其他物种的INSIG1氨基酸序列,并通过Clustal X 2.1软件进行物种间氨基酸序列比对,获得黄河鲤INSIG1与其他物种氨基酸序列的一致性。基于MEGA 4.0软件中Bootstrap检验进行1 000次重复,采用Neighbor-Joining(NJ)法对系统进化树进行构建。
1.2.4 INSIG1基因组织表达分析 参照鲤INSIG1基因的cDNA序列(XM_042728053.1),采用Primer Premier 5.0软件设计qRT-PCR引物,以β-actin作为内参基因,送至生工生物工程(上海)股份有限公司进行合成(表1)。以上述3尾黄河鲤的各组织cDNA为模板进行荧光定量PCR检测。qRT-PCR反应体系(20 μL):上、下游引物各0.5 μL,cDNA模板1 μL,2×Universal SYBR qPCR MIX 10 μL,用ddH2O补足至20 μL。PCR反应程序:95 ℃下预变性60 s;95 ℃下变性10 s,60 ℃下退火30 s,共进行40个循环。采用2-ΔΔCt法计算基因的相对表达量。利用SPSS 26.0 软件对各组织的表达量进行单因素方差分析(P<0.05)。
表1 试验用引物信息
Tab.1 Primers used in this study
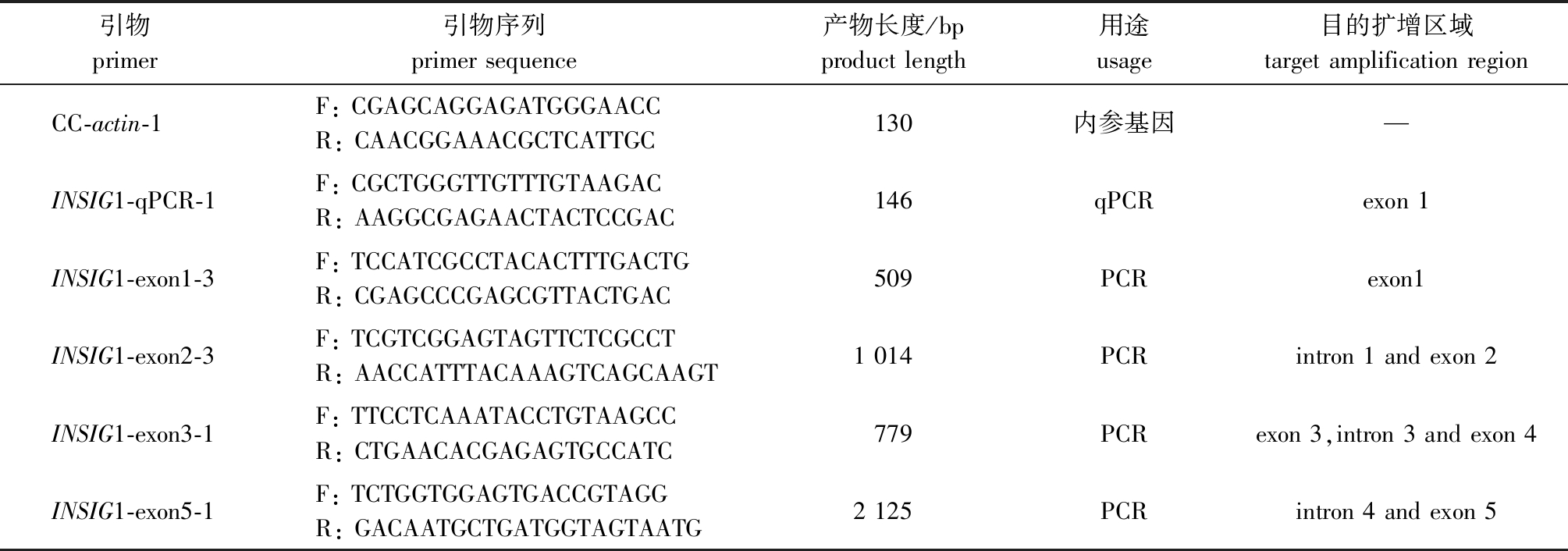
引物 primer 引物序列 primer sequence 产物长度/bpproduct length用途usage目的扩增区域target amplification regionCC-actin-1F: CGAGCAGGAGATGGGAACCR: CAACGGAAACGCTCATTGC130内参基因—INSIG1-qPCR-1F: CGCTGGGTTGTTTGTAAGACR: AAGGCGAGAACTACTCCGAC146qPCRexon 1INSIG1-exon1-3F: TCCATCGCCTACACTTTGACTGR: CGAGCCCGAGCGTTACTGAC509PCRexon1INSIG1-exon2-3F: TCGTCGGAGTAGTTCTCGCCTR: AACCATTTACAAAGTCAGCAAGT1 014PCRintron 1 and exon 2INSIG1-exon3-1F: TTCCTCAAATACCTGTAAGCCR: CTGAACACGAGAGTGCCATC779PCRexon 3,intron 3 and exon 4 INSIG1-exon5-1F: TCTGGTGGAGTGACCGTAGGR: GACAATGCTGATGGTAGTAATG2 125PCRintron 4 and exon 5
1.2.5 INSIG1基因的引物合成及SNP位点挖掘 从上述黄河鲤群体中随机选取10尾黄河鲤子代用于INSIG1基因的SNP位点挖掘。基于鲤INSIG1基因DNA序列(NC_056603.1),利用Primer Premier 5.0软件设计引物(表1),送至生工生物工程(上海)股份有限公司进行合成。PCR反应体系(12.5 μL):基因组DNA 40~50 ng,上、下游引物(5 μmol/L)各0.5 μL,25 mmol/L dNTP 0.4 μL,2.5 U/μL Taq DNA酶0.1 μL (Tiangen),10×Buffer 1.25 μL,用ddH2O补足至12.5 μL。PCR反应条件:94 ℃下预变性4 min;94 ℃下变性35 s,52~62 ℃下退火35 s,72 ℃下延伸40 s,共进行38个循环;最后在72 ℃下再延伸10 min。PCR产物送至生工生物工程(上海)股份有限公司进行测序。利用Clustal X 2.1软件对测序结果进行序列比对分析,挖掘潜在的SNP位点。
1.2.6 INSIG1基因的SNP位点遗传多样性分析 将鉴定筛选到的SNP位点在随机挑选的120尾黄河鲤群体中进行分型,统计每个个体的基因型数据。采用Arlequin软件分析各个SNP位点相应的遗传参数,包括基因型频率、等位基因频率、期望杂合度(expected heterozygosity,He)、观测杂合度(observed heterozygosity,Ho)、多态信息含量(polymorphism information content,PIC)和哈代-温伯格平衡系数(Hardy-Weinberg equilibriums,HWE)。
1.2.7 SNP位点与生长性状的关联分析 采用SPSS 24.0软件中的一般线性模型(general linear model,GLM)对INSIG1基因上各SNP位点的不同基因型与黄河鲤生长性状间的相关性进行方差分析。GLM模型公式为
Y=m+G+e。
式中:Y为生长性状的测量值;m为生长性状测量值的平均值;G为多态位点的基因型的效应值;e为随机误差效应值。显著性水平和极显著性水平的相关性分别设为0.05和0.01。
2 结果与分析
2.1 INSIG1基因序列特征及系统进化树
测序结果显示:黄河鲤INSIG1基因全长为4 853 bp,包含5个外显子和4个内含子;cDNA全长为1 530 bp,5′端UTR长度为231 bp,3′端UTR长度为543 bp,蛋白质编码区(CDS)长度为756 bp,可编码251个氨基酸。氨基酸序列比对显示,黄河鲤INSIG1氨基酸序列与金线鲃(Sinocyclocheilus rhinocerous)、斑马鱼(Danio rerio)和鲫(Carassius auratus)高度相似,一致性分别为97%、97%、98%,而与其他硬骨鱼类的一致性为82%~90%,与哺乳类、鸟类及两栖类动物的一致性为76%~85%(图1)。

浅蓝色底纹和浅绿色底纹分别表示100%和80%的氨基酸残基一致性。
The 100% and 80% of amino acids identity among these sequences are respectively highlighted by light blue and light green shade.
图1 黄河鲤与其他物种的INSIG1氨基酸序列比较
Fig.1 Comparison of deduced amino acid sequences of INSIG1 in Cyprinus carpio with other species
基于26种脊椎动物的INSIG1氨基酸序列,构建INSIG1氨基酸序列的系统进化树。结果显示,该进化树主要分为两支,一支为硬骨鱼类,另一支包括哺乳动物、两栖动物、鸟类和软骨鱼纲,其中,黄河鲤与鲫、斑马鱼、金线鲃的进化关系较近,这与物种进化地位相符合(图2)。
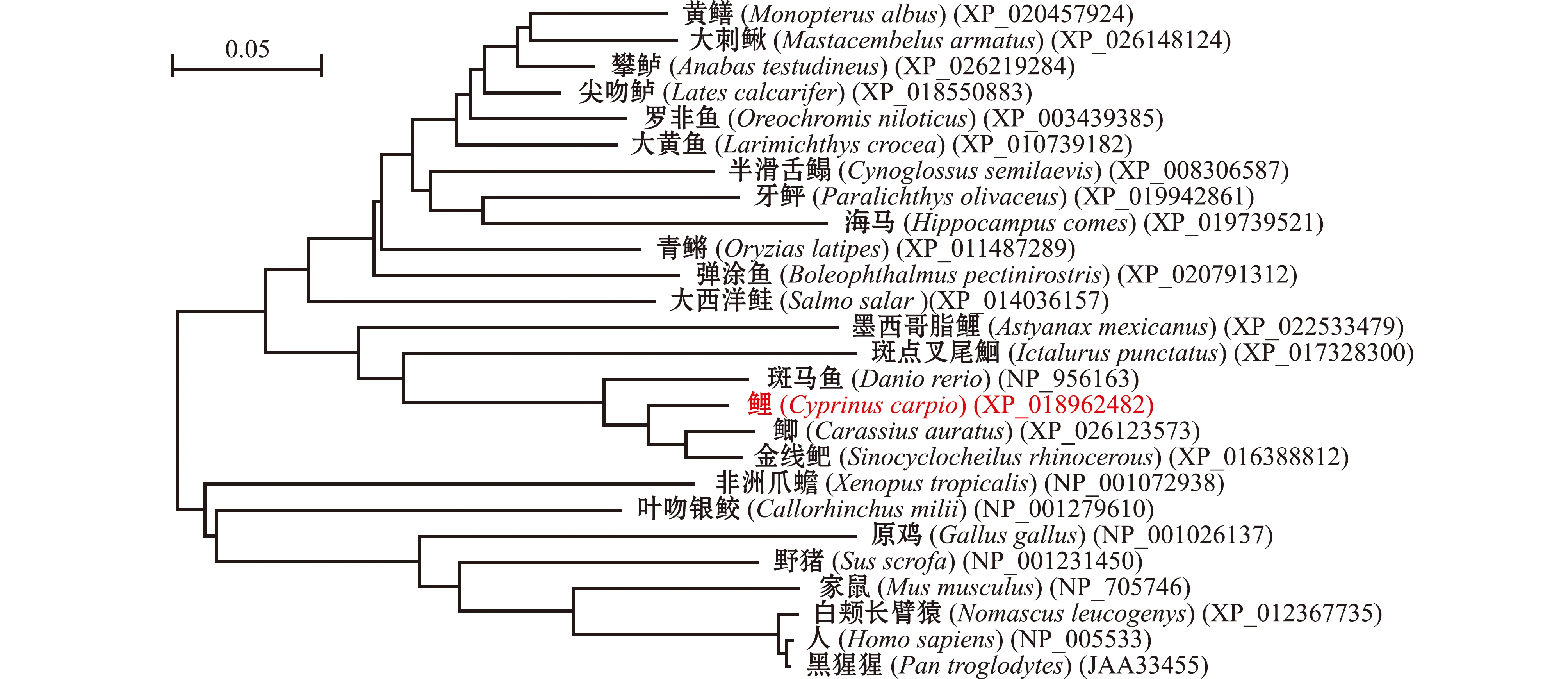
图2 基于26种脊椎动物INSIG1氨基酸序列的Neighbor-Joining进化树
Fig.2 Neighbor-Joining phylogenetic tree based on 26 INSIG1 protein sequences of vertebrates
2.2 INSIG1蛋白结构
2.2.1 INSIG1蛋白理化性质 预测得到黄河鲤INSIG1蛋白相对分子质量为27 674.25,理论等电点pI为8.09,总原子数为3 914,分子式为C1 260H1 966N330O345S13,无信号肽,脂肪系数为110.32,消光系数为53 440,不稳定系数为39.08,表明该蛋白为稳定蛋白。
2.2.2 INSIG1蛋白疏水性 黄河鲤INSIG1蛋白亲水性系数为0.457,表明该蛋白为疏水性蛋白,亲水性最强的是第131位的苯丙氨酸,疏水性最强的是第67位的异亮氨酸和第68位的缬氨酸。
2.2.3 INSIG1蛋白跨膜结构 黄河鲤INSIG1蛋白有6个跨膜螺旋结构,为跨膜蛋白,表明INSIG1与运输调节有关。
2.2.4 INSIG1蛋白高级结构 黄河鲤INSIG1蛋白二级结构预测显示,该蛋白的α-螺旋、延伸链、β-转角和无规则卷曲占比分别为50.6%、13.9%、4.4%和31.1%(图3(a))。INSIG1蛋白的三级结构预测显示,该蛋白结构主要由6个α-螺旋组成,还有部分无规则卷曲(图3(b))。
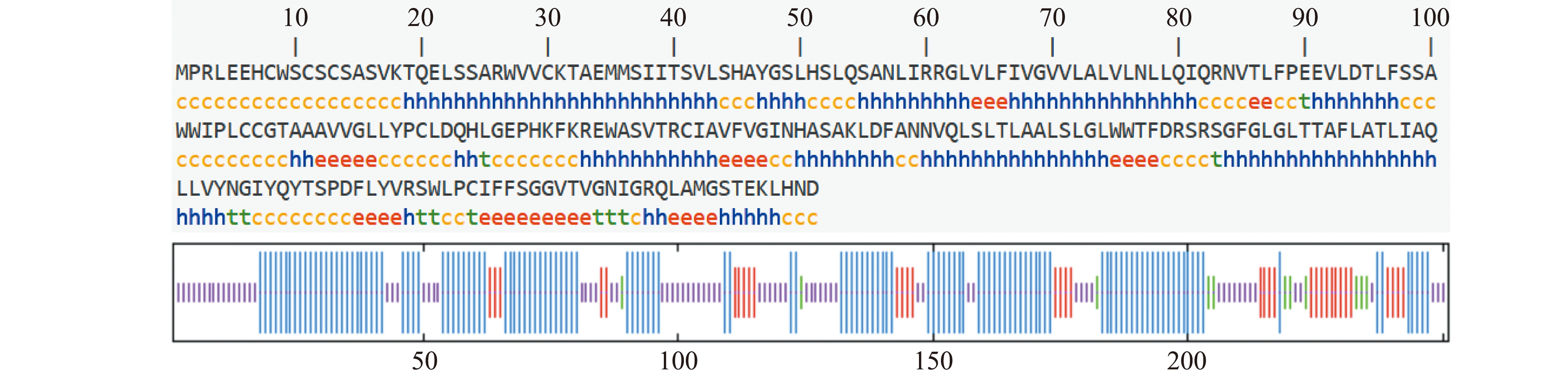
c—无规则卷曲;h—α-螺旋;e—延伸链;t—β-转角。
c—random coil;h—alpha helix;e—extended strand;t—β-turn.
(a)二级结构 secondary structure
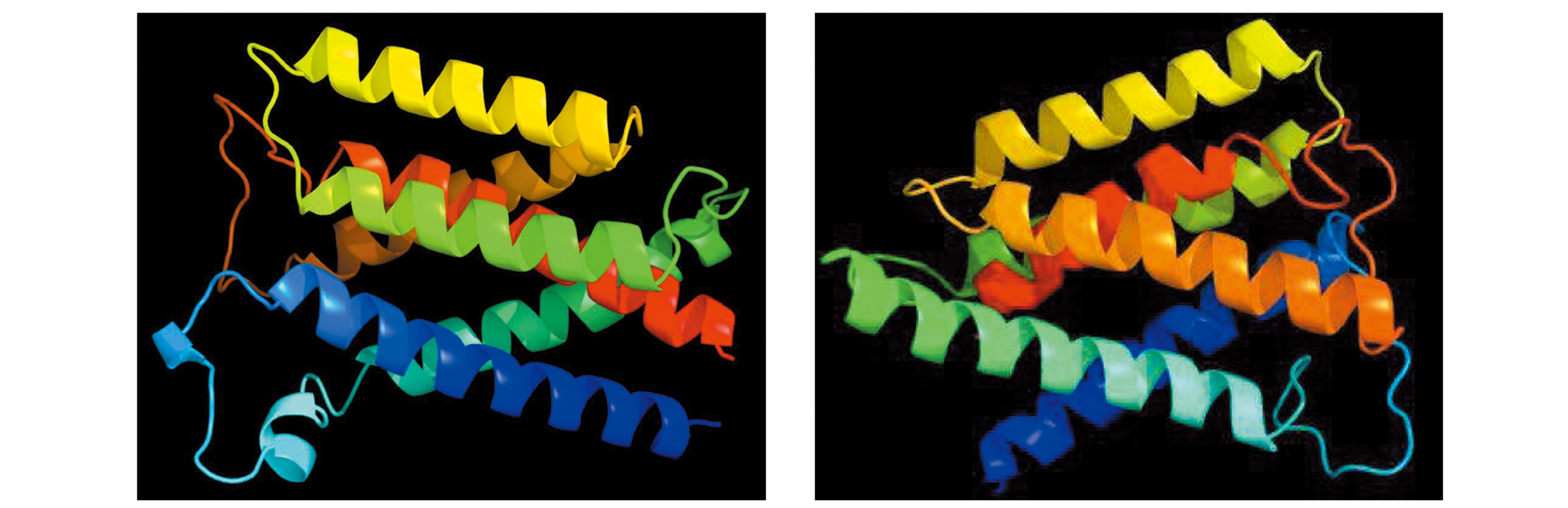
(b)三级结构 tertiary structure
图3 黄河鲤INSIG1蛋白的二级和三级结构预测
Fig.3 Secondary and tertiary structure prediction of INSIG1 protein in Cyprinus carpio haematoperus
2.3 INSIG1基因组织表达谱
INSIG1基因在黄河鲤的肝、脾、肾、肠、心脏、鳃、脑、肌肉和皮肤中均有表达。其中,在肠组织中表达量最高(P<0.05),其次是肝组织,鳃组织中表达量最低(图4)。
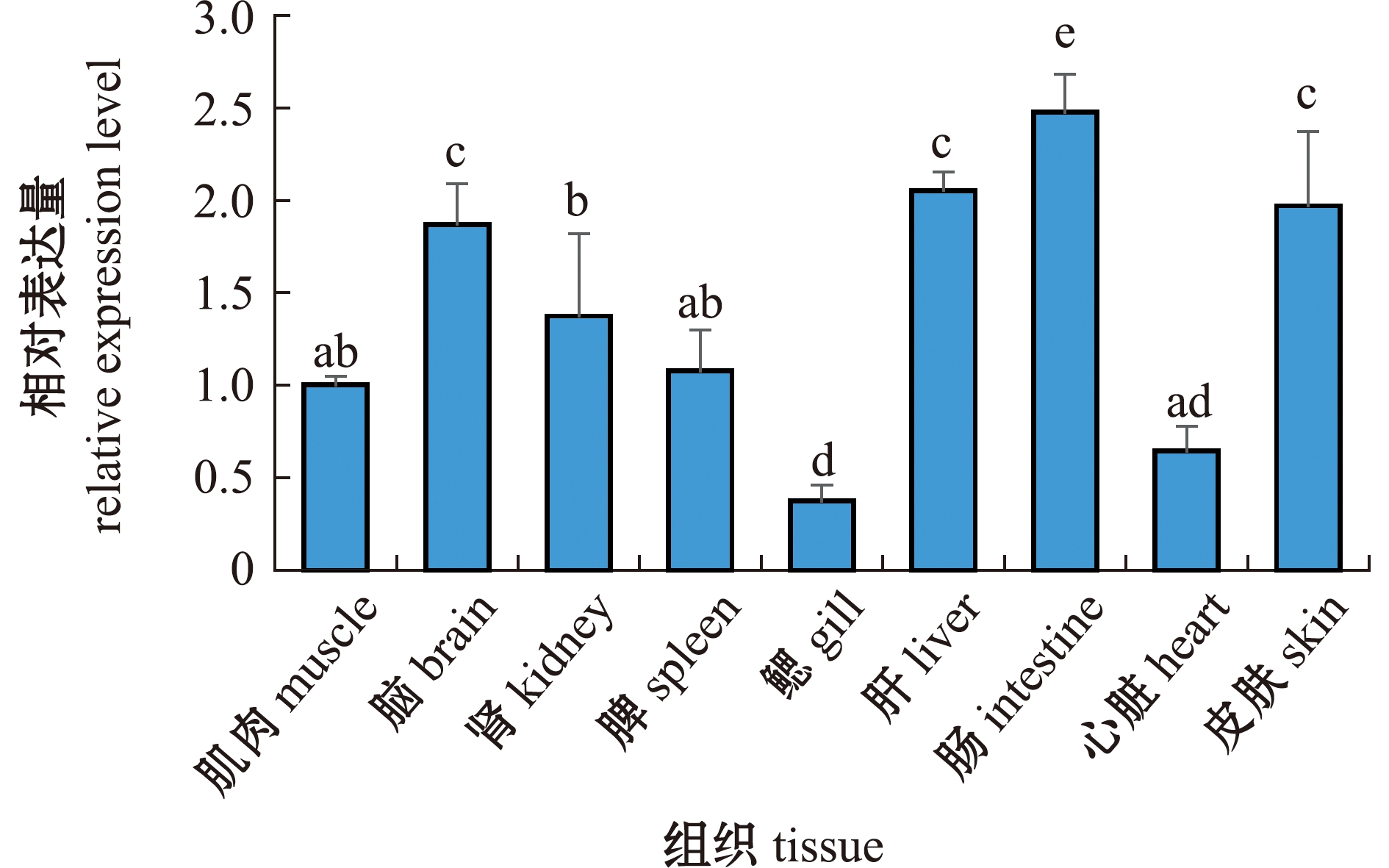
标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。
The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter are not significant differences.
图4 黄河鲤INSIG1基因在组织中的相对表达量
Fig.4 Relative expression level of INSIG1 gene in different tissues of Cyprinus carpio haematoperus
2.4 黄河鲤INSIG1基因SNP位点及其与生长性状的关联分析
通过对10尾黄河鲤INSIG1基因序列比对,共挖掘出10个候选SNP位点。在这10个SNP中,4个分布在外显子区域,6个分布在内含子,其中,仅有一个位于第3个外显子上的单核苷酸突变引起编码氨基酸的改变(亮氨酸Leu到甲硫氨酸Met)(表2)。
表2 黄河鲤INSIG1基因SNP位点信息
Tab.2 SNP locus information of INSIG1 gene in Cyprinus carpio haematoperus
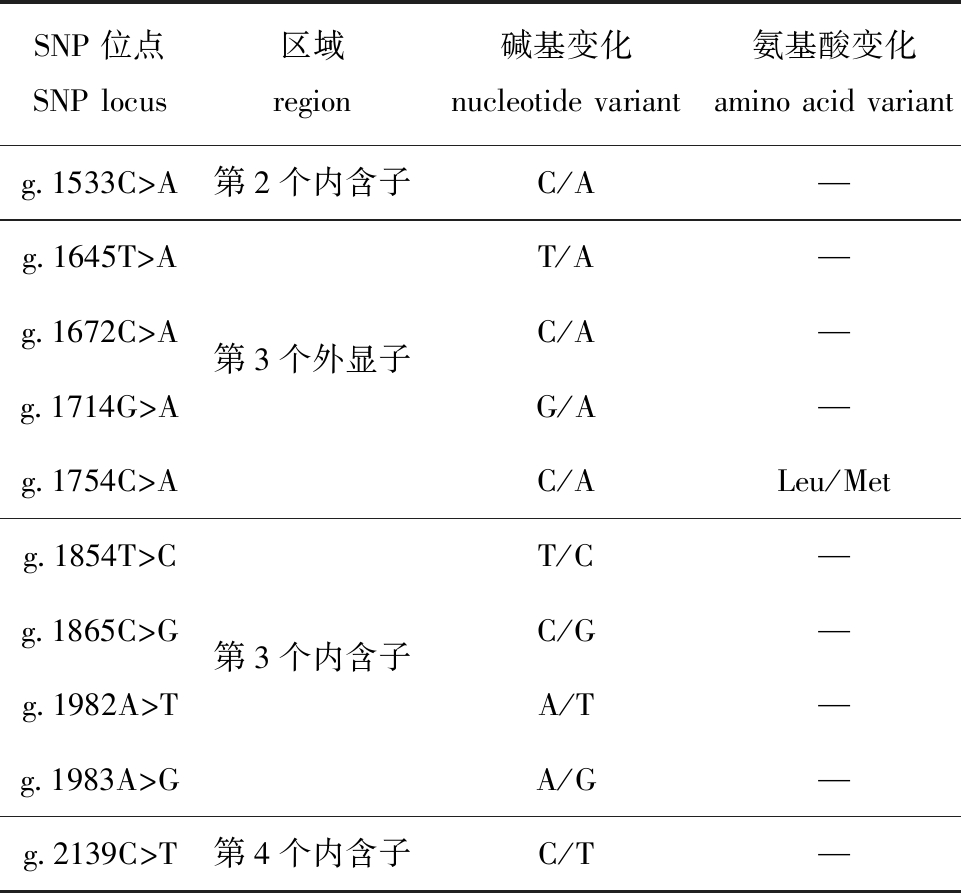
SNP位点SNP locus区域region碱基变化nucleotide variant氨基酸变化amino acid variantg.1533C>A第2个内含子C/A—g.1645T>AT/A—g.1672C>A第3个外显子C/A—g.1714G>AG/A—g.1754C>AC/ALeu/Metg.1854T>CT/C—g.1865C>G第3个内含子C/G—g.1982A>TA/T—g.1983A>GA/G—g.2139C>T第4个内含子C/T—
上述10个SNP位点成功在104尾黄河鲤中进行基因分型,统计分析其遗传参数发现:这10个SNP位点的Ho值为0.019~0.673,平均值为0.265 8;He值为0.019~0.501,平均值为0.240 5;PIC值为0.019~0.374,平均值为0.194 9(表3)。10个位点中,5个为低度多态位点,5个为中度多态位点。卡方检验结果显示,g.1865C>G和g.1982A>T 2个位点极显著偏离哈代-温伯格平衡(P<0.01),其余8个SNP位点均符合哈代-温伯格平衡;g.1983A>G和g.2139C>T 2个位点处于完全连锁状态。在该黄河鲤群体中,g.1672C>A、g.1714G>A和g.1754C>A 3个位点只有1个纯合基因型和1个杂合基因型,其余7个SNP位点均有2个纯合基因型和1个杂合基因型。
表3 黄河鲤群体中INSIG1基因SNP位点的遗传多样性信息
Tab.3 Genetic diversity based on single-nucleotide polymorphisms of INSIG1 gene in the test population of Cyprinus carpio haematoperus

注:括号内为所测个体数;**表示P<0.01,极显著偏离了哈代-温伯格平衡。
Note:The number of individuals measured is described in parentheses;** indicates P<0.01,which deviates significantly from Hardy Weinberg’s balance.
SNP位点SNP locus基因型频率genotypic frequency等位基因频率allelic frequency观测杂合度Ho期望杂合度He多态信息含量PIC哈代-温伯格平衡X2g.1533C>ACC(6)0.062 5C0.197 90.2710.3190.2672.074CA(26)0.270 8A0.802 1AA(64)0.666 7g.1645T>ATA(6)0.058 3T0.029 10.0580.0570.0550.093AA(97)0.941 7A0.970 9g.1672C>ACC(100)0.970 9C0.985 40.0290.0290.0280.022CA(3)0.029 1A0.014 6g.1714G>AGG(96)0.932 0G0.966 00.0680.0660.0630.127GA(7)0.068 0A0.034 0g.1754C>ACC(102)0.980 8C0.990 40.0190.0190.0190.010CA(2)0.019 2A0.009 6g.1854T>CCC(79)0.759 6C0.865 40.2120.2340.2060.882CT(22)0.211 5T0.134 6TT(3)0.028 8g.1865C>GCC(22)0.211 5C0.548 10.6730.4980.37313.383∗∗CG(70)0.673 1G0.451 9GG(12)0.115 4g.1982A>TAA(21)0.201 9A0.528 80.6540.5010.37410.127∗∗AT(68)0.653 8T0.471 2TT(15)0.144 2g.1983A>GAA(63)0.615 4A0.783 70.3370.3410.2820.006AG(36)0.336 5G0.216 3GG(5)0.048 1g.2139C>TCC(63)0.615 4C0.783 70.3370.3410.2820.006CT(36)0.336 5T0.216 3TT(5)0.048 1
将上述INSIG1基因10个SNP位点的不同基因型与黄河鲤生长性状进行关联分析,结果显示:2个SNP位点(g.1854T>C和g.1982A>T)与黄河鲤肥满度性状均显著相关(P<0.05);SNP位点g.1854T>C的CT基因型个体的肥满度显著高于TT基因型个体(P<0.05),SNP位点g.1982A>T的AT基因型个体的肥满度显著高于AA基因型个体(P<0.05);其他位点不同基因型与黄河鲤生长性状均未检测到显著相关性(表4)。
表4 黄河鲤INSIG1基因SNP位点不同基因型与生长性状的关联分析
Tab.4 Multiple comparisons between genotypes of SNP in INSIG1 and growth traits in Cyprinus carpio haematoperus
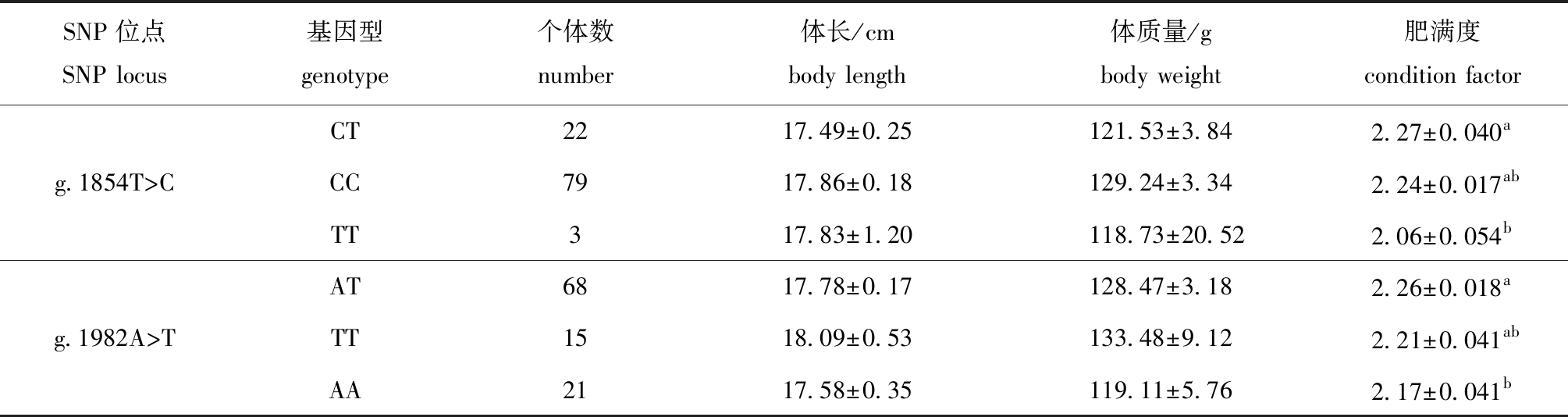
注:同一SNP位点同列中标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。
Note:The means with different letters within the same column for each SNP locus are significantly different in the groups at the 0.05 probability level,and the means with the same letter within the same column are not significant differences.
SNP位点 SNP locus基因型 genotype个体数 number体长/cm body length体质量/g body weight肥满度 condition factorCT2217.49±0.25121.53±3.842.27±0.040ag.1854T>CCC7917.86±0.18129.24±3.342.24±0.017abTT317.83±1.20118.73±20.522.06±0.054bAT6817.78±0.17128.47±3.182.26±0.018ag.1982A>TTT1518.09±0.53133.48±9.122.21±0.041abAA2117.58±0.35119.11±5.762.17±0.041b
3 讨论
3.1 不同物种INSIG1序列和结构的保守性
本研究表明,INSIG1氨基酸序列在不同物种间表现出较高的相似性,这与王京京等[7]、权凯等[14]在家畜中发现的不同物种INSIG1氨基酸序列高度相似的结果一致,序列的保守性预示着该蛋白在不同物种中功能的一致性。黄河鲤INSIG1蛋白为稳定的疏水性蛋白,无信号肽,蛋白三级结构包含6个α-螺旋和部分无规则卷曲,该结果与羊(Capra hircus)[14-15]、猪(Sus scrofa)[16]等INSIG1蛋白结构一致。黄河鲤INSIG1蛋白包含6个跨膜结构,与人(Homo sapiens)INSIG1蛋白跨膜结构数量相同[17],但多于羊[14-15]、猪[16]等INSIG1蛋白的5个跨膜结构数量。INSIG1在不同物种中的跨膜特性表明,该蛋白与物质的跨膜运输调节有关。
3.2 INSIG1基因在黄河鲤组织中的广泛性表达
INSIG1基因在脂质代谢中发挥重要的生理功能,其与机体增重、肥胖、糖尿病、泌乳等性状存在相关性[2-5]。本研究中,通过实时荧光定量发现,INSIG1基因在黄河鲤各组织中呈现广泛性表达,其表达量从高到低依次为肠、肝、皮肤、脑、肾、脾、肌肉、心脏和鳃。该结果与牛(Bos taurus)[18]INSIG1基因的表达结果一致,均表现为肠和肝组织表达量较高,与羊[14-15]、猪[16]和鹅(Ansercygnoides orientalis)[19]等INSIG1基因的表达结果类似。黄河鲤INSIG1基因在肠和肝脏中的高水平表达,可能与人工养殖黄河鲤的肠和肝脏组织脂肪含量较多有关,该基因主要在黄河鲤肠和肝脏中调节脂肪代谢活动,抑制肝内脂质合成和肝脂肪变性[20-21]。
3.3 与生长性状显著相关的INSIG1基因SNP位点的筛选
本研究表明,在黄河鲤INSIG1基因中鉴定得到的10个SNP位点中,有6个位点位于内含子区域,4个位点位于外显子区域,非编码区单核苷酸的突变率较高,推测可能与序列长度及选择压力不同有关[22]。虽然内含子对基因编码蛋白无影响,但它可能在基因转录和mRNA剪接过程中起作用,Fiume等[23]发现,内含子是调节蛋白表达的主要因子,基因内含子位点变异与经济性状的相关性已在多种鱼类中报道。王梓祎等[24]在红鳍东方鲀(Takifugu rubripes)生长激素释放激素基因(GHRH)的内含子区域发现了与红鳍东方鲀体质量、头长等多个生长性状相关的SNP位点;Feng等[25]在鲤胰岛素样生长因子1基因(IGF1)的内含子区域也发现了能显著影响生长性状的SNP位点。本研究中,在黄河鲤INSIG1基因内含子区域也发现2个与黄河鲤肥满度性状显著相关的SNP位点。由此可见,将内含子区域作为性状相关分子标记的开发区域具有广阔的发展前景。
胰岛素诱导基因在脂肪细胞的分化、成熟和脂质合成中起重要的调节作用[6]。有研究表明,INSIG1基因的SNP位点与机体增重、肥胖和生长等性状有关。如Liu等[26]发现,牛INSIG1基因SNP位点与酮体质量、体高等性状显著相关;Smith等[27]发现,人INSIG1基因启动子上的SNP位点与血浆甘油三酯水平有较大关联性。笔者在黄河鲤INSIG1基因中也检测到2个单核苷酸多态性位点与黄河鲤肥满度性状显著相关,虽然INSIG1基因的功能还未在其他鱼类中得到验证,但结合人类及家畜INSIG1基因的研究结果,可以推测,INSIG1基因在机体脂质合成中具有重要的调控作用,通过调控机体生长发育过程,显著影响体质量、肥满度等重要经济性状。因此,在黄河鲤育种工作中,可对黄河鲤进行优势基因型选择育种,以加快黄河鲤选择育种进程,提高黄河鲤群体生产性能。
4 结论
1)黄河鲤INSIG1基因cDNA全长为1 530 bp,蛋白编码区(CDS)全长为756 bp,可编码251个氨基酸。不同物种的INSIG1氨基酸序列及蛋白结构保守性较高,表明不同物种INSIG1基因功能的一致性。
2)INSIG1基因在黄河鲤组织中呈现广泛性表达,其中在肠和肝组织中表达量较高,揭示了INSIG1基因在黄河鲤各组织中均发挥重要调控作用。
3)黄河鲤INSIG1基因的2个SNP位点与黄河鲤的肥满度性状显著相关,表明INSIG1基因可能是黄河鲤生长发育调控的候选基因。该结果为黄河鲤生长性状的分子标记辅助育种提供了可用的分子标记,为进一步开展黄河鲤品种选育提供了科学参考。
[1] DONG X Y,TANG S Q.Insulin-induced gene:a new regulator in lipid metabolism[J].Peptides,2010,31(11):2145-2150.
[2] TIWARI A K,ZAI C C,MELTZER H Y,et al.Association study of polymorphisms in insulin induced gene 2 (INSIG2) with antipsychotic-induced weight gain in European and African-American schizophrenia patients[J].Human Psychopharmacology,2010,25(3):253-259.
[3] LIU F H,SONG J Y,SHANG X R,et al.The gene-gene interaction of INSIG-SCAP-SREBP pathway on the risk of obesity in Chinese children[J].BioMed Research International,2014,2014:538564.
[4] 刘尧吉.利拉鲁肽对2型糖尿病大鼠脂肪组织胰岛素诱导基因表达的影响[D].太原:山西医科大学,2020.
LIU Y J.Effect of liraglutide on insulin-induced gene expression in adipose tissue of type 2 diabetic rats[D].Taiyuan:Shanxi Medical University,2020.(in Chinese)
[5] 张怡宁,宋洁云,刘芳宏,等.胰岛素诱导基因蛋白2多态性及饮食运动行为与儿童肥胖的关系[J].中国学校卫生,2016,37(2):167-170.
ZHANG Y N,SONG J Y,LIU F H,et al.Polymorphism in insulin induced gene 2 and its relation with diet,physical activity and obesity phenotypes in children[J].Chinese Journal of School Health,2016,37(2):167-170.(in Chinese)
[6] SEVER N,YANG T,BROWN M S,et al.Accelerated degradation of HMG CoA reductase mediated by binding of INSIG-1 to its sterol-sensing domain[J].Molecular Cell,2003,11(1):25-33.
[7] 王京京,陈斌.胰岛素诱导基因及其在猪育种中应用的研究进展[J].猪业科学,2014,31(4):92-94.
WANG J J,CHEN B.Research progress of insulin-induced gene and its application in pig breeding[J].Swine Industry Science,2014,31(4):92-94.(in Chinese)
[8] 罗建椿,邱立华,范新阳,等.牛科物种胰岛素诱导基因1(INSIG1)研究进展[J].中国牛业科学,2017,43(3):31-35.
LUO J C,QIU L H,FAN X Y,et al.Research advances of insulin induced gene 1(INSIG1)in bovine species[J].China Cattle Science,2017,43(3):31-35.(in Chinese)
[9] AZZU V,VACCA M,KAMZOLAS I,et al.Suppression of insulin-induced gene 1 (INSIG1) function promotes hepatic lipid remodelling and restrains NASH progression[J].Molecular Metabolism,2021,48:101210.
[10] FAN X Y,QIU L H,TENG X H,et al.Effect of INSIG1 on the milk fat synthesis of buffalo mammary epithelial cells[J].The Journal of Dairy Research,2020,87(3):349-355.
[11] XU P,ZHANG X F,WANG X M,et al.Genome sequence and genetic diversity of the common carp,Cyprinus carpio[J].Nature Genetics,2014,46(11):1212-1219.
[12] WANG X,FU B,YU X,et al.Fine mapping of growth-related quantitative trait loci in Yellow River carp (Cyprinus carpio haematoperus)[J].Aquaculture,2018,484:277-285.
[13] NASH R D M,VALENCIA A H,GEFFEN A J.The origin of Fulton’s condition factor—setting the record straight[J].Fisheries 2006,31:236-238.
[14] 权凯,宋飞虎,李君.豫西脂尾羊INSIG1基因克隆、序列分析及组织表达[J].中国畜牧兽医,2020,47(6):1781-1790.
QUAN K,SONG F H,LI J.Cloning,sequence analysis and tissue expression of INSIG1 gene in Yuxi fat-tailed sheep[J].China Animal Husbandry &Veterinary Medicine,2020,47(6):1781-1790.(in Chinese)
[15] 王慧,罗军,胡仕良,等.西农萨能奶山羊INSIG-1编码区的克隆、序列特征分析和组织表达[J].西北农林科技大学学报(自然科学版),2013,41(8):24-30.
WANG H,LUO J,HU S L,et al.Cloning,sequence characters and tissue expression pattern of INSIG-1 in Saanen Dairy Goat[J].Journal of Northwest A &F University (Natural Science Edition),2013,41(8):24-30.(in Chinese)
[16] 王京京,魏麟,陈斌.猪胰岛素诱导1基因的cDNA克隆和差异表达及蛋白质序列分析[J].湖南农业大学学报(自然科学版),2016,42(5):505-510.
WANG J J,WEI L,CHEN B.Cloning and differential expression of insulin induced gene 1 in pig and the analysis on its protein sequence[J].Journal of Hunan Agricultural University (Natural Sciences Edition),2016,42(5):505-510.(in Chinese)
[17] FEEAMISCO J D,GOLDSTEIN J L,BROWN M S.Membrane topology of human INSIG-1,a protein regulator of lipid synthesis[J].Journal of Biological Chemistry,2004,279(9):8487-8496.
[18] 庞坤,石科,杨亮,等.奶牛胰岛素诱导基因1的克隆、组织分布及稳定表达细胞系的建立[J].基因组学与应用生物学,2016,35(7):1681-1685.
PANG K,SHI K,YANG L,et al.Cloning,tissue distribution and establishment of stable expression cell line of bovine insulin-induced gene 1[J].Genomics and Applied Biology,2016,35(7):1681-1685.(in Chinese)
[19] 郭锚,陈红霞,杨丹丹,等.鹅INSIG1基因的组织表达规律及差异表达分析[J].养禽与禽病防治,2021(8):35-40.
GUO M,CHEN H X,YANG D D,et al.Tissue expression regularity and differential expression analysis of goose INSIG1 gene[J].Poultry Husbandry and Disease Control,2021(8):35-40.(in Chinese)
[20] GONG Y I,LEE J N,LEE P C W,et al.Sterol-regulated ubiquitination and degradation of Insig-1 creates a convergent mechanism for feedback control of cholesterol synthesis and uptake[J].Cell Metabolism,2006,3(1):15-24.
[21] ENGELKING L J,KURIYAMA H,HAMMER R E,et al.Overexpression of Insig-1 in the livers of transgenic mice inhibits SREBP processing and reduces insulin-stimulated lipogenesis[J].The Journal of Clinical Investigation,2004,113(8):1168-1175.
[22] ZHAO Z M,FU Y X,HEWETT-EMMETT D,et al.Investigating single nucleotide polymorphism (SNP) density in the human genome and its implications for molecular evolution[J].Gene,2003,312:207-213.
[23] FIUME E,CHRISTOU P,GIAN S,et al.Introns are key regulatory elements of rice tubulin expression[J].Planta,2004,218(5):693-703.
S,et al.Introns are key regulatory elements of rice tubulin expression[J].Planta,2004,218(5):693-703.
[24] 王梓祎,苟盼盼,杨金,等.红鳍东方鲀生长激素释放激素(GHRH)基因多态性及其与生长性状的关联分析[J].大连海洋大学学报,2022,37(1):34-41.
WANG Z Y,GOU P P,YANG J,et al.Correlation analysis between single nucleotide polymorphisms (SNPs) of GHRH gene and growth traits of tiger puffer Takifugu rubripes[J].Journal of Dalian Ocean University,2022,37(1):34-41.(in Chinese)
[25] FENG X,YU X M,TONG J G.Novel single nucleotide polymorphisms of the insulin-like growth factor-I gene and their associations with growth traits in common carp (Cyprinus carpio L.)[J].International Journal of Molecular Sciences,2014,15(12):22471-22482.
[26] LIU Y,LIU X L,HE H,et al.Four SNPs of insulin-induced gene 1 associated with growth and carcass traits in Qinchuan cattle in China[J].Genetics and Molecular Research,2012,11(2):1209-1216.
[27] SMITH E M,ZHANG Y,BAYE T M,et al.INSIG1 influences obesity-related hypertriglyceridemia in humans[J].Journal of Lipid Research,2010,51(4):701-708.