大竹蛏(Solen grandis),又称蛏子王,属软体动物门(Mollusca)瓣鳃纲(Lamellibranchia)帘蛤目(Veneroida)竹蛏科(Solenida),在菲律宾、朝鲜、日本及中国南北沿海潮间带均有分布,是常见的食用贝类之一[1]。大竹蛏体大壳薄,出肉率高,蛏肉中富含蛋白质、钙、铁、硒和维生素 A 等营养元素,为贝类中的上品,其市场价格高达140元/kg。大竹蛏在繁育、养殖及运输过程中频繁出现水管自切再生现象,水管发生自切后大竹蛏会出现伏沙不潜底,有的出现疲软甚至死亡,导致亲贝催产利用率降低,给育苗企业带来种质浪费和经济损失。在养殖生产中,发生自切后的大竹蛏生长缓慢,出池时间延长,养殖风险加大,如果大竹蛏频繁发生自切再生会导致所谓的“长老”现象。在销售过程中,商品蛏由于受装卸运输的应激影响,大竹蛏普遍发生水管自切,因掉落水管的大竹蛏无人购买,只能丢弃,而水管一般占到个体总质量的20%~25%,对其收益影响较大。因此,大竹蛏水管自切再生问题已成为制约其养殖及销售的关键因素之一。
再生是指生物体的器官甚至整体受到自然环境中外力作用后导致创伤或部分丢失,在剩余部分的基础上长出与损伤或缺失部分相同的形态和功能组织的修复过程。组织损伤的修复和再生是生命科学研究的重要课题之一,国内外对再生的研究主要集中在一些模式动物上,如涡虫(Schmidtea mediterranea)、蝾螈(Ambystoma mexicanum)、小鼠(Mus musculus)和斑马鱼(Danio rerio)等[2-5]。再生通常包括已经存在组织的重组,成体干细胞的利用,细胞的去分化和转分化等,进而实现形态、结构和功能的重建[6]。目前,国内外关于水生动物再生的研究主要有蝾螈的肢体再生[7]、斑马鱼的心脏和尾鳍再生[8-9]、海盘车腕部再生[10]、水螅躯体再生[11]和海参内脏团再生[12]等。研究中发现,再生过程对动物的摄食能力、生长和发育等有较大影响,如刺参出现体质量负生长现象[13],蟹断肢后会出现生长减缓、发育时间(蜕皮或蜕皮周期)延长[14]。再生过程同时还受到包括生长因子、细胞因子等生物活性分子,以及神经、体液和信号通路等因素调控,涉及基因、蛋白和细胞等的作用[15]。在贝类中,仅见关于菲律宾蛤仔(Ruditapes philippinarum)、缢蛏(Sinonovacula constricta)水管再生的研究[16-18],而有关大竹蛏组织再生研究仅见笔者所在课题组发表的利用代谢组学方法评估水管自切对大竹蛏代谢特性影响的论文[19]。本研究中,以人为切除水管组织的大竹蛏为试验对象,通过二代测序技术对大竹蛏组织再生进行转录组测序研究,并分析水管再生期间转录组的变化情况,以期为深入研究大竹蛏水管再生机制提供科学参考。
1 材料与方法
1.1 材料
试验在江苏省海洋水产研究所吕四试验基地进行。以项目组自繁养殖8~10 cm壳长的大竹蛏为试验材料,选取无破损、活力好的大竹蛏于室内1 000 L圆缸中暂养7 d,圆缸内底铺20 cm厚的细沙,加注沙滤海水600 L。暂养期间,海水盐度为28,pH为8.3,水体溶氧量为7.6~8.0 mg/L,水温为26~30 ℃。暂养期间缸内海水保持连续充气,每日上午换水一次,换水量为总水量的50%;选择密度为(15~20)×105 ind./mL 的球等鞭金藻(Isochrysis galbana),早、中、晚定时往暂养缸内加入120 L藻水,保证大竹蛏处于正常养殖状态。
1.2 方法
1.2.1 试验设计及样品采集 以预试验中大竹蛏水管自切后的生长情况为参考,分别设置自切前对照组,以及自切后7 h、7 d和20 d 3个选择组。由于在pH、水压等外部因素刺激下大竹蛏会发生水管自切的随意性,水管断裂位置的不同造成断裂水管长短不一,因此,在正式试验开始时,采用人工切除水管的方法统一将水管切除至水管基部(图1)。从暂养缸中逐一取出大竹蛏,切除水管后的大竹蛏共计60条,置于重新准备的1 000 L圆缸中饲养,饲养条件与暂养期间一致;饲养期间观察缸中大竹蛏存活情况,发现不钻沙个体及时清除。分别取自切前,以及自切后7 h、7 d和20 d 4个时间点的大竹蛏水管基部组织样品,每组设置3个重复,样品对应标号记为BC、ATa、ATb和ATc,所取组织样本用液氮速冻后置于-80 ℃超低温冰箱中保存备用。
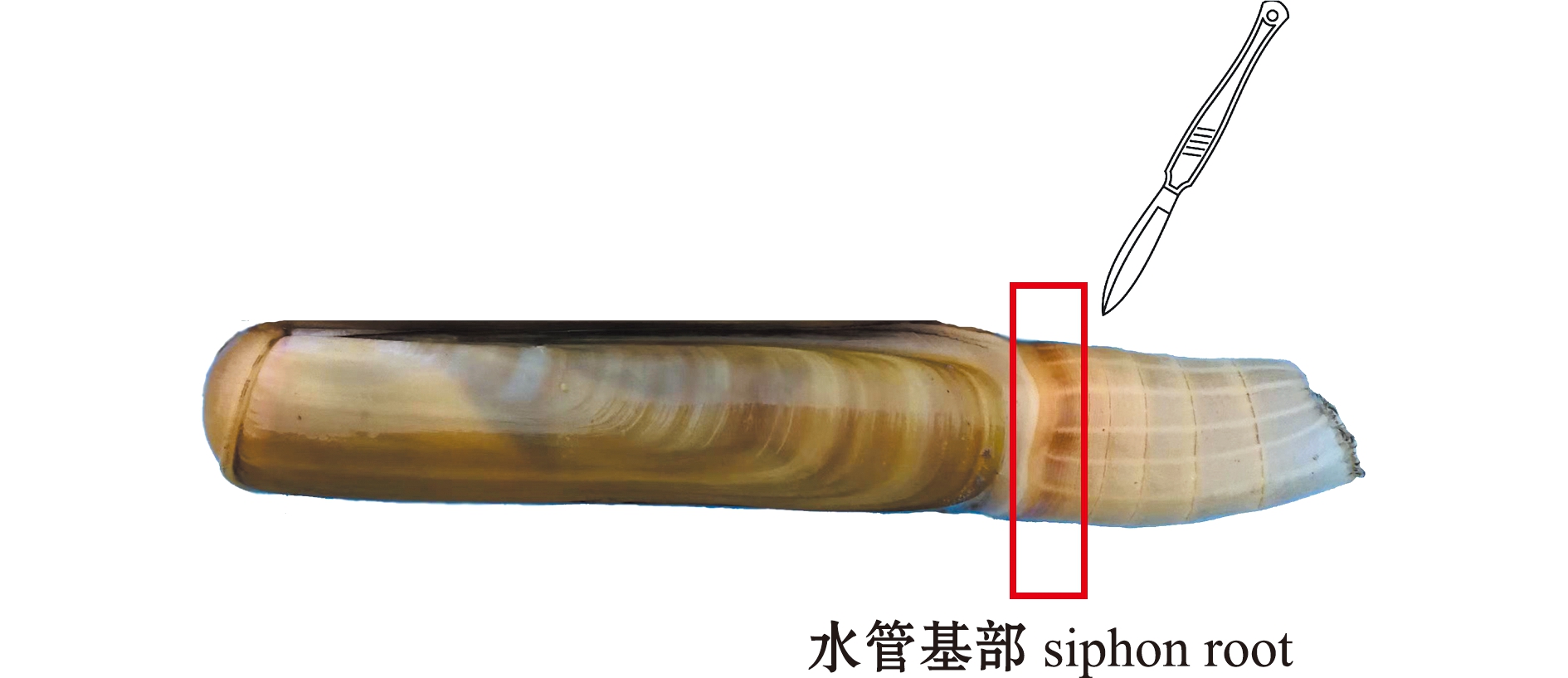
图1 大竹蛏水管组织的切除
Fig.1 Removed siphon of Solen grandis
1.2.2 RNA提取、cDNA文库构建和高通量测序
采用Trizol试剂盒(Invitrogen公司)提取总RNA,采用10 g/L琼脂糖凝胶电泳检测RNA的完整性,采用NanoDrop微量分光光度计测定RNA纯度。RNA检测合格后使用带有Oligo(dT)的磁珠富集mRNA,再使用片段缓冲剂将得到的mRNA裂解为片段,在ProtoScript Ⅱ逆转录酶体系下用随机引物反转录合成cDNA的第一条链,随后用RNase H降解mRNA,在DNA聚合酶Ⅰ体系下以dNTPs为原料合成cDNA的第二条链。采用QiaQuick PCR 提取试剂盒纯化cDNA,再加上polyA末端并连接测序接头。利用琼脂糖凝胶电泳筛选200 bp左右的连接产物并进行PCR扩增,经AMPure XP beads纯化后的PCR产物用以构建cDNA文库,并通过IlluminaNova Seq 6000测序平台进行高通量测序。
1.2.3 测序数据组装与Unigenes注释 测序得到的原始序列(raw reads)通过Fastp 0.18.0软件过滤低质量序列后获得有效序列(clean reads),采用Trinity软件[20]对clean reads进行组装,并挑出每个基因中最长的转录本作为Unigene,将得到的Unigenes与COG、GO、KEGG、KOG、Pfam、Swiss-Prot和NR等数据库进行比对,获得基因功能注释。
1.2.4 差异基因(differentially expressed genes,DEGs)的表达及时间趋势分析 以FPKM(fragments per kilobase of transcript per million mapped reads)值作为基因表达量高低的指标,采用DESeq软件进行组间差异分析,筛选FDR(false discovery rate)<0.05且|log2(fold change)|>1的基因作为差异基因[21],再将得到的差异基因通过GO、KEGG数据库进行富集分析。
采用STEM软件[22]对3个比较组中的并集差异基因进行时间趋势分析,模块数量设定为20,获得显著的基因表达模式,并选取模式中的基因集进行GO富集分析。
1.2.5 RT-qPCR验证 根据测序结果,以β-actin为内参基因[23],随机挑选6个差异基因进行荧光定量(表1)。具体步骤:基于选择基因的CDS序列,采用Primer 5软件设计引物;以反转的cDNA为模板,采用荧光定量试剂盒(SYBR Green),在ABI公司的7 300 plus荧光定量PCR仪上进行操作。PCR反应体系为(20 μL):2×SuperReal PreMix Plus 10 μL,正、反向引物各0.6 μL,cDNA模板1 μL,50×ROX Reference Dye 1 μL,用RNase-free ddH2O补足至20 μL。扩增程序:95 ℃下预变性15 min;95 ℃下变性10 s,56 ℃下退火30 s,72 ℃下延伸31 s,共进行40个循环。使用2-ΔΔCt法计算基因相对表达量。
表1 引物信息
Tab.1 Primer information

基因 ID gene ID基因 gene NR注释 NR annotated引物序列 primer sequence (5′-3′)TRINITY_DN38613_c0_g1BMP10bone morphogenetic protein 10F:AGGCTTGAAGTGAGAATCR:ATGAAACCCACGGCTACGTRINITY_DN23814_c0_g1GPR84G-protein coupled receptor 84-likeF:GGGTCGGGAAATCGTCACTTR:TGAGCAGTTCGTGTCGGATGTRINITY_DN43265_c4_g1Tyrp-1tyrosinase-related protein-1F:TCCAAAGCACTAAGCGTTATR:TTCCATTCGGTCTTCCAGTRINITY_DN13992_c0_g5TLR4toll-like receptor 4F:TGTAAGCGGGAGAAAGTGAATR:TCCTCGTAAGGCTGGAAGTAGTRINITY_DN35676_c0_g2ENO4enolase 4-like isoform X4F:GATTACAGGGCAACAGAAGGTR:ATGTCAGTCAGTTCCTCGTTCTRINITY_DN24272_c2_g1MyomyostatinF:GCGGAACCTTCTTTGACTTGCCR:TCCTCGGTTGGTTTCTGGTCGTβ-actin—F:TGTACGCCAACACTGTCCTGTCR:CATCGTATTCCTGCTTGCTGATC
2 结果与分析
2.1 大竹蛏水管再生的形态特征及再生长情况
从图2可见:试验开始后5 h时,除5条大竹蛏未钻沙外,其余大竹蛏均钻沙;7 h后观察到沙面有气孔,部分大竹蛏水管伸出沙面滤食,发现被切除水管的基部增厚,外缘处新生多个凸起;7 d后清晰可见大竹蛏水管增长,平均增长(6.52±1.02)mm;20 d后大竹蛏水管增长明显,平均增长(38.67±2.11)mm。在此期间,除10、15 d时分别有2、4条大竹蛏死亡外,最终大竹蛏存活率为81.67%。

S—水管;SR—水管基部。
S—siphon;SR—siphon root.
图2 大竹蛏水管恢复生长
Fig.2 Siphon regrowth of Solen grandis
2.2 转录组测序结果和组装
从表2可见,原始序列经质控后共得到241 840 528条有效序列,Q20、Q30分别高于97%和92%,说明测序结果良好。有效序列经Trinity组装后共获得235 719条Transcripts和103 909条Unigenes,Transcripts与Unigenes的N50分别为1 523、1 377 bp。
表2 转录组测序结果
Tab.2 Results of transcriptome sequencing
样本 sample原始碱基长度 raw base length/bp有效序列数 clean readsGC/%Q20/%Q30/%BC16 539 079 90021 796 93345.1597.4393.12BC26 780 994 80022 603 31643.4397.7093.48BC35 468 324 10018 227 74743.2697.5293.03ATa15 510 471 40018 368 23842.5697.3392.85ATa26 868 087 50022 893 62542.7897.4793.17ATa35 478 682 20018 262 27441.3297.0992.32ATb16 047 238 60020 157 46245.6797.4093.08ATb26 045 579 00020 151 93045.3797.5993.40ATb36 161 697 60020 538 99244.7397.4493.16ATc16 411 422 40021 371 40842.4597.5793.16ATc25 120 500 80017 068 33642.3597.2692.73ATc36 120 080 10020 400 26742.5897.5693.15
2.3 Unigenes功能注释
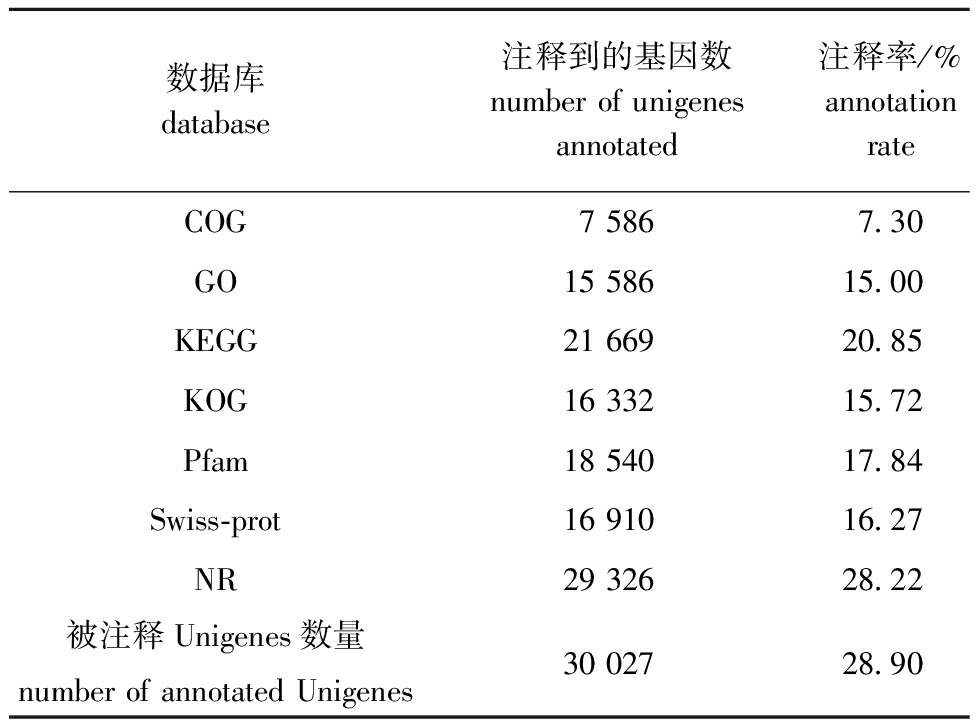
分别在COG、GO、KEGG、KOG、Pfam、Swiss-Prot和NR数据库中对Unigenes进行注释分析。从表3可见,在COG数据库中注释到7 586条Unigenes,在GO、KEGG、KOG、Pfam、Swiss-Prot和NR数据库中分别注释到15 586、21 669、16 332、18 540、16 910和29 326条Unigenes,累计被注释的Unigenes为30 027条,注释率为28.90%,表明依旧有大量的未知基因有待研究。
表3 Unigenes功能注释分析
Tab.3 Analysis of Unigenes functional annotation
数据库database注释到的基因数number of unigenes annotated注释率/%annotation rateCOG7 5867.30GO15 58615.00KEGG21 66920.85KOG16 33215.72Pfam18 54017.84Swiss-prot16 91016.27NR29 32628.22被注释Unigenes数量number of annotated Unigenes30 02728.90
2.4 差异基因的筛选及GO、KEGG分析
差异基因筛选结果显示,BC vs ATa组共筛选出7 098个差异基因,其中,3 343个基因上调,3 755个基因下调;BC vs ATb组共筛选出5 075个差异基因,其中,3 172个基因上调,1 903个基因下调;BC vs ATc组共筛选出4 245个差异基因,其中,2 561个基因上调,1 684个基因下调;862个基因在3组中共表达(图3)。
GO功能注释结果显示,差异基因被分到生物过程(biological process)、细胞组分(cellular component)和分子功能(molecular function)3个类别中。Padj值为多重假设检验校正后的P值,Padj值越小,表明富集程度越高,为了进一步了解差异基因所行使的分子功能,本研究中筛选了分子功能类别中显著富集(Padj<0.05)的GO条目。从表4可见,BC vs ATa组显著富集到4个GO条目,其中,钙离子结合(calcium ion binding)条目的富集程度最高且富集的基因数目也最多,171个差异基因富集到该条目;BC vs ATb、BC vsATc两组中富集程度最高的条目均为几丁质结合(chitin binding)。
表4 3组差异基因显著富集的GO条目
Tab.4 GO Terms with significant enrichment of three groups of DEGs

组别 groupGO条目 GO term基因数 gene numberIDPadjBC vs ATacalcium ion bindingsterol transporter activitymicrotubule motor activityATPase activity171174154GO:0005509GO:0015248GO:0003777GO:00168870.000 110.000 470.000 700.003 28BC vs ATbchitin bindinggap junction channel activitychitinase activitygap junction hemi-channel activityG-protein coupled peptide receptor activity1488811GO:0008061GO:0005243GO:0004568GO:0055077GO:00085280.000 120.000 580.007 670.011 500.011 99BC vs ATcchitin bindingcoenzyme bindingmetalloendopeptidase activitysequence-specific double-strandeddna bindingheme bindingserine-type endopeptidase inhibitor activity1462343715GO:0008061GO:0050662GO:0004222GO:1990837GO:0020037GO:00048670.000 010.000 710.009 580.036 810.040 490.044 01
KEGG通路富集结果显示,BC vs ATa、BC vs ATc两组无通路被显著富集(Padj>0.05),BC vs ATb组共有5条通路被显著富集(Padj<0.05),依次为氨基糖与核苷酸糖代谢(amino sugar and nucleotide sugar metabolism)、氮素代谢(nitrogen metabolism)、黏着斑(focal adhesion)、溶酶体(lysosome)和MAPK信号通路(MAPK signaling pathway)。图4展示了富集程度前20的KEGG通路。
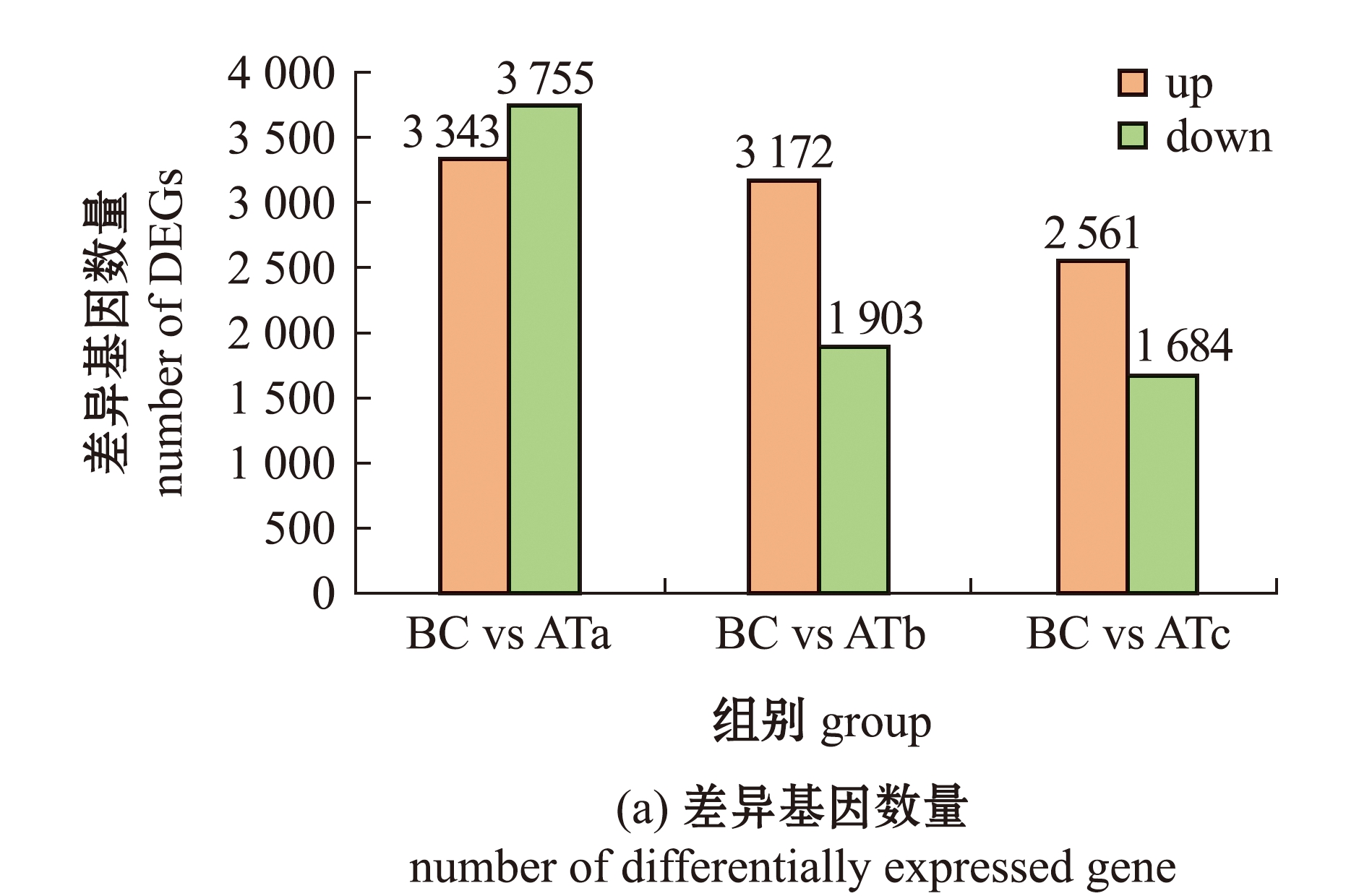
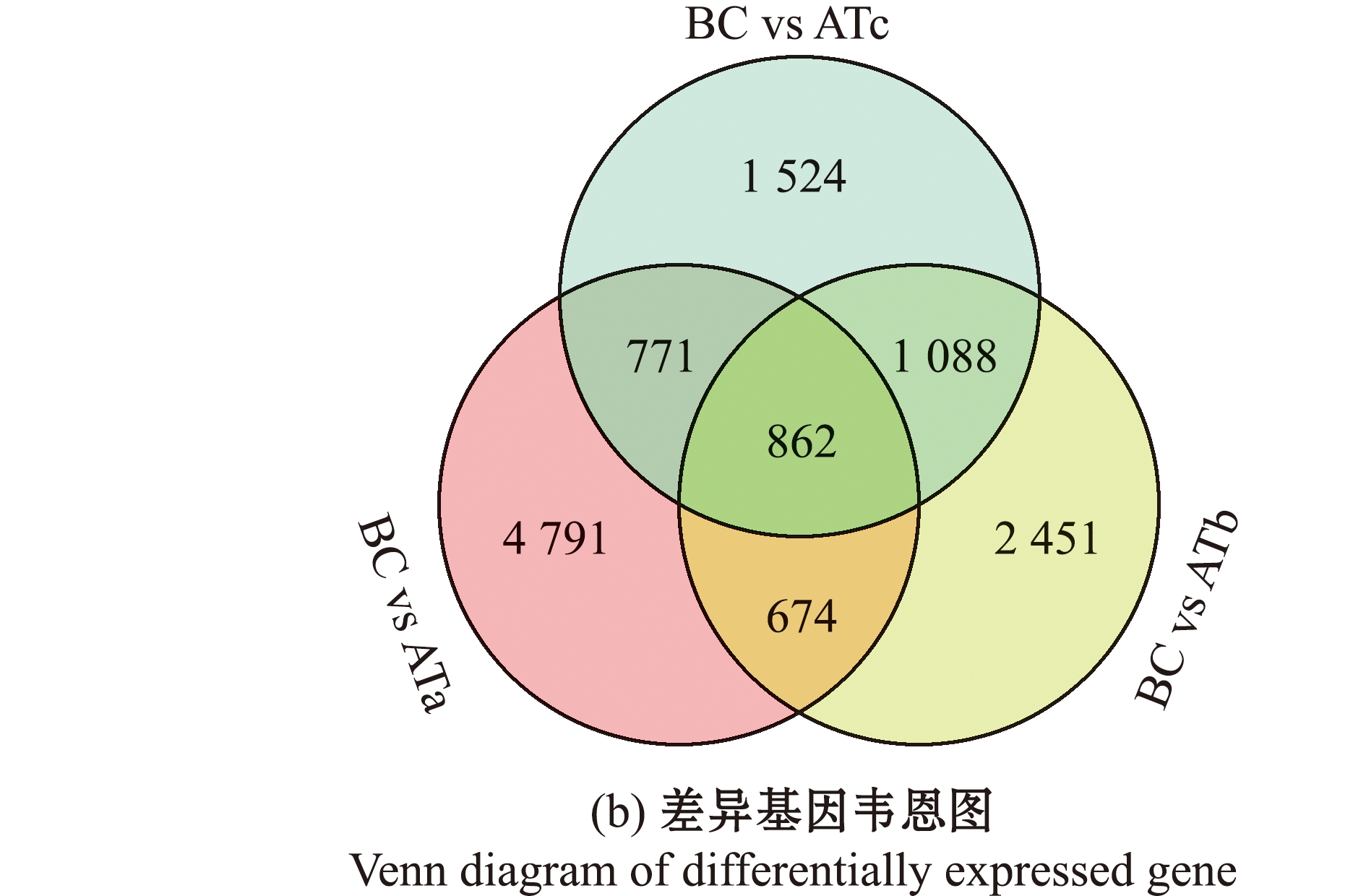
图3 差异基因的筛选
Fig.3 Screening of differentially expressed genes
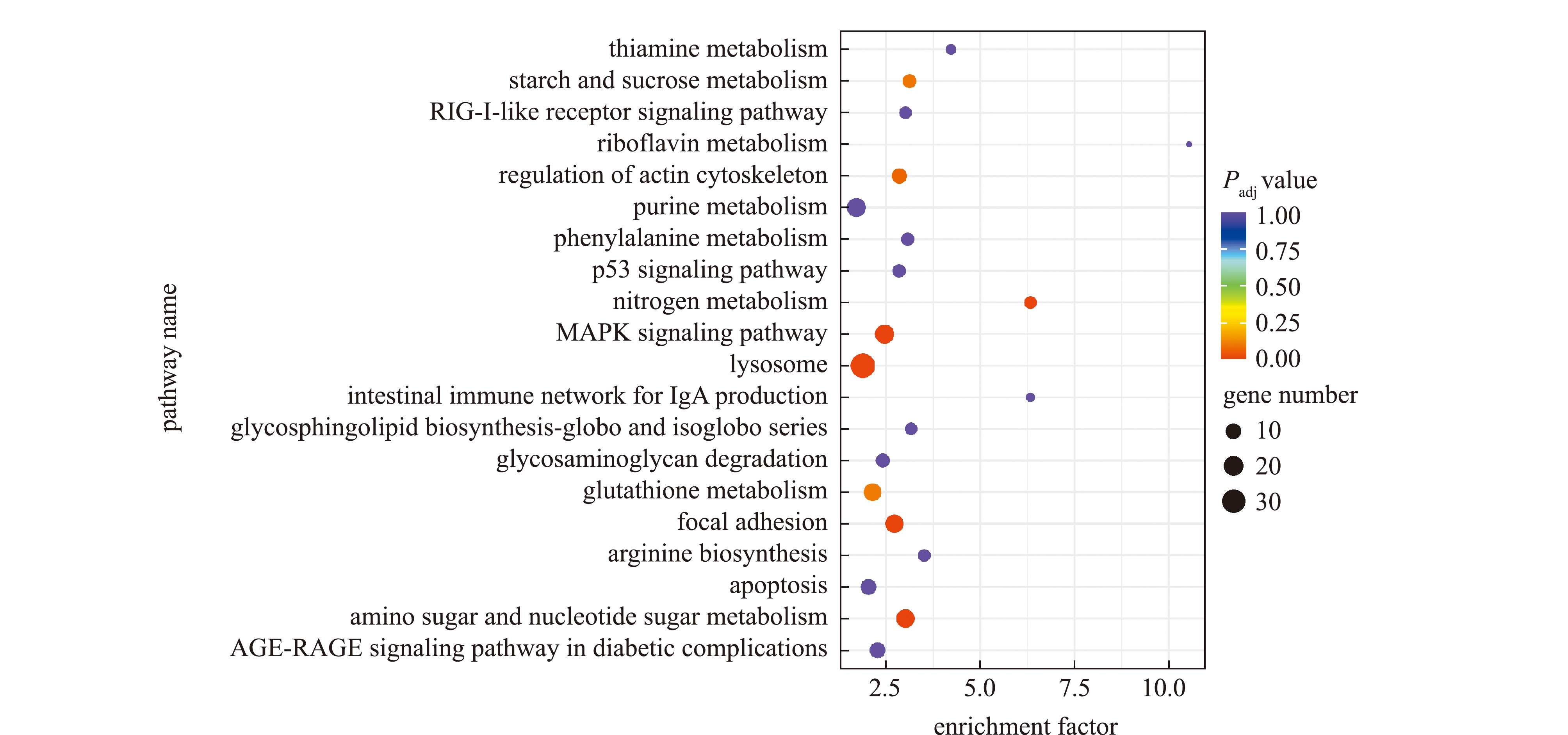
图4 BC vs ATb组差异基因的KEGG富集通路(前20)
Fig.4 KEGG enrichment pathway of differentially expressed genes (top 20)
2.5 差异基因的时间趋势分析
为了解差异基因随时间的变化情况,取3个比较组中差异基因的并集,采用STEM软件进行时间趋势分析,模块数量设定为20。从图5可见,富集基因数前3的模式依次为Profile5、Profile17、Profile12,分别富集到1 966、1 093、981条基因。Profile5的基因表达量在7 h时呈下降趋势,在7、20 d时表达量恢复并保持自切前水平,Profile17的基因表达量自切后高于自切前,Profile12的基因表达量在7 h时保持不变,随后上升并保持不变。
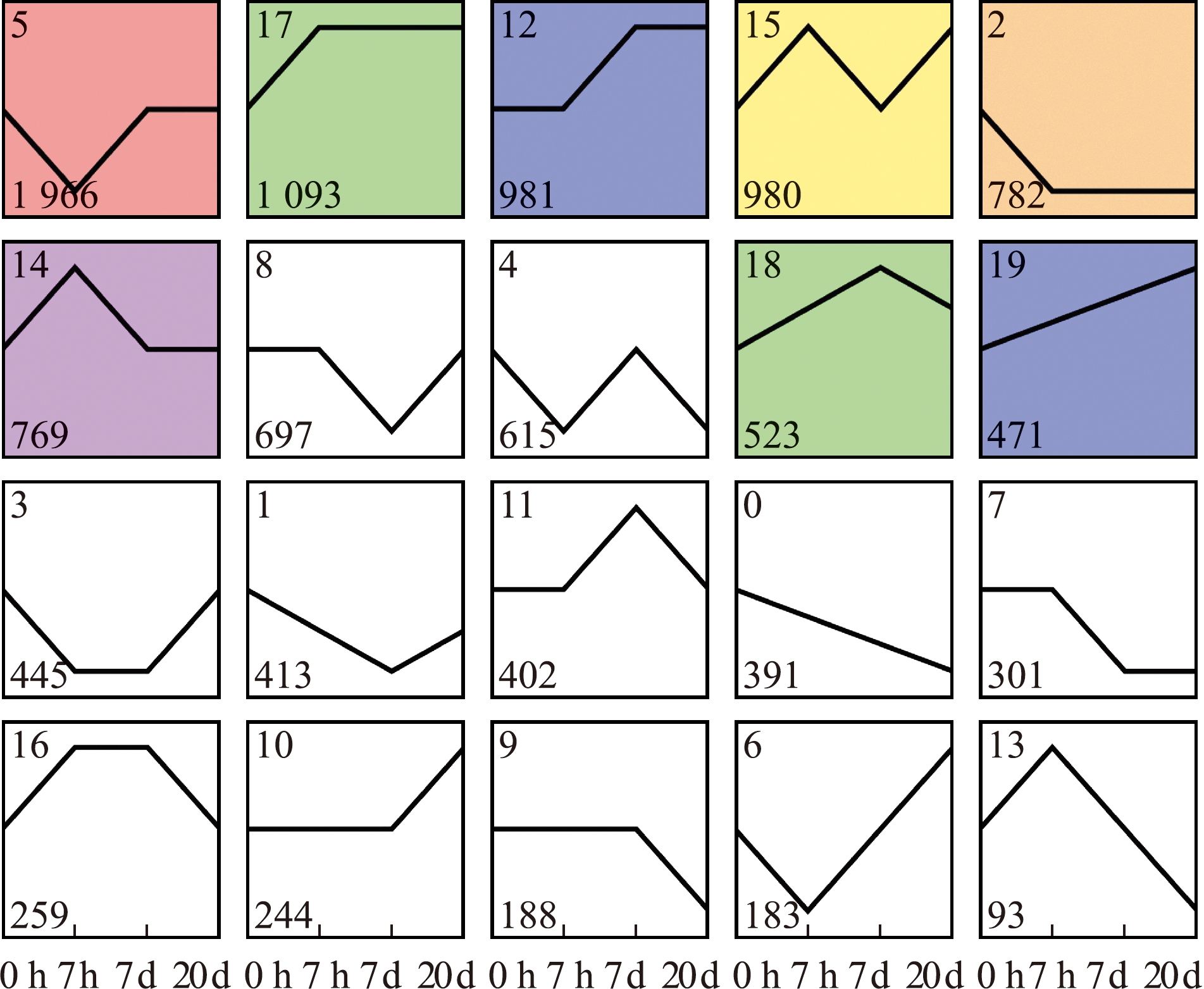
方框左上角数字代表不同基因表达趋势模式,如Profile5;左下方数字代表差异基因个数;彩色方框表示具有显著性。
The number on the upper left of the box denotes different trends,as Profile5.The number on the lower left represents the number of differential genes. Colored box representation is significant.
图5 差异基因的时间趋势分析
Fig.5 Time trend analysis of differentially expressed genes
选取Profile5、Profile17和Profile12 3个模式的基因集进行GO功能富集分析。结果显示,Profile5中共有529条基因注释到离子结合(ion binding)条目,占总注释基因数的53.77%,其次注释到基因数较多的条目为蛋白质结合(protein binding)、钙离子结合(calcicium ion binding)等(图6);Profile17富集基因数较多的条目包括受体活性(receptor activity)和分子传导活性(molecular transducer activity)等,富集程度最高的条目为G蛋白偶联受体活性(G-protein coupled receptor activity);Profile12中基因主要显著富集到氨基糖代谢(amino sugar metabolic process)和几丁质代谢(chitin metabolic process)等条目。

柱形图数值代表差异基因数。The numbers on the bar chart represent the number of differentially expressed genes.
图6 Profile5显著富集的GO条目(前20)
Fig.6 GO terms with significant enrichment of genes in Profiles5(Top 20)
2.6 RT-qPCR验证
为了验证测序结果的可靠性,随机挑选了NR数据库注释下的6条显著差异基因进行荧光定量PCR(RT-qPCR)验证,结果表明(图7),相对定量趋势与RNA-Seq的趋势总体一致,表明测序结果可靠。
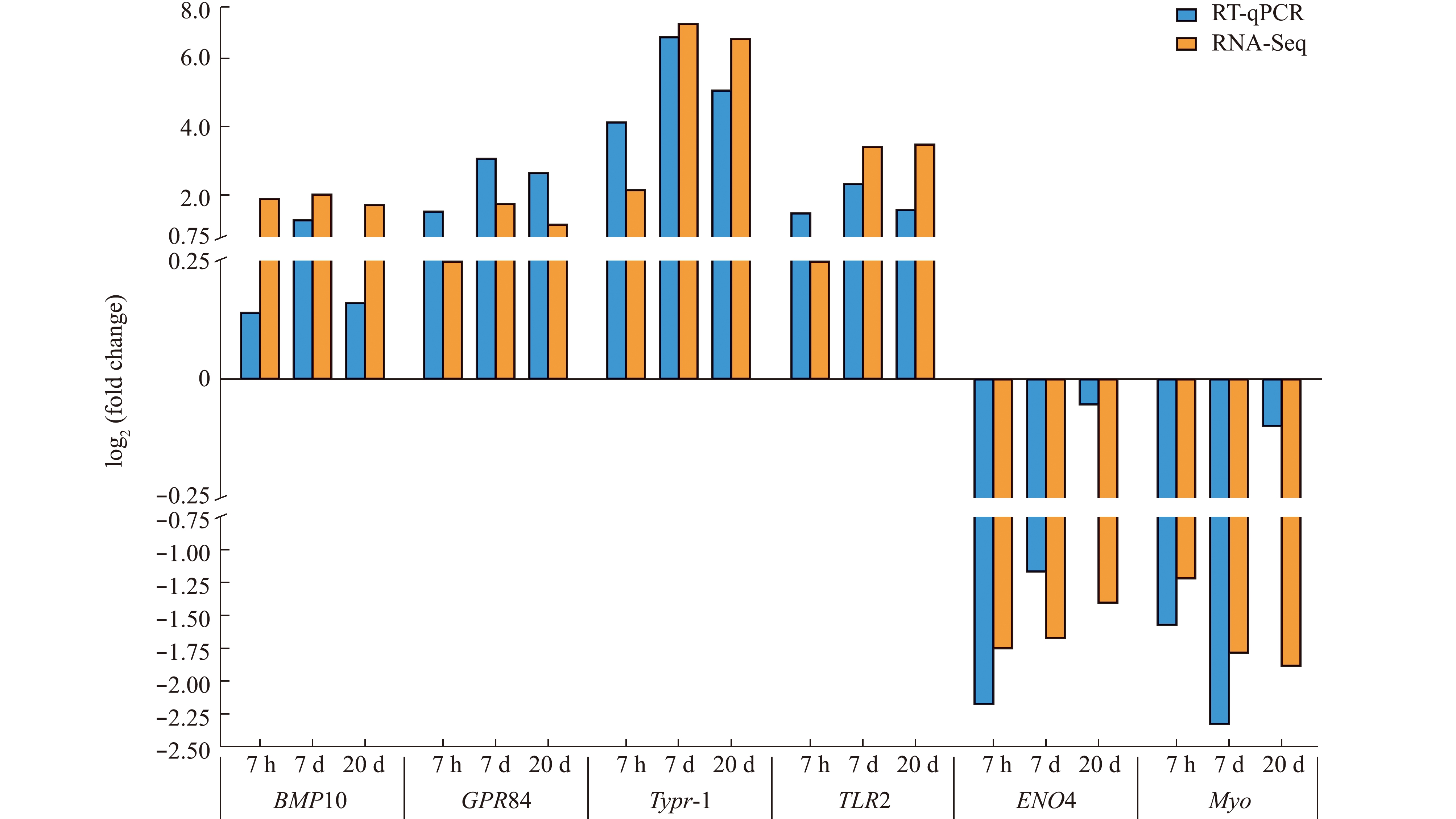
图7 实时荧光定量PCR验证结果
Fig.7 Verification results by real-time quantitative PCR
3 讨论
3.1 Unigenes功能注释信息分析
相关研究中,杨旻珉[24]获得了罗氏沼虾(Macrobrachium rosenbergii)肝胰腺转录组142.38 Gb clean data,Q30在94.49%以上,组装得到252 123条转录本和135 239条Unigenes,N50为1 737 bp,注释率为29.75%。杨轶博[25]基于高通量测序对日本三角涡虫(Dugesia japonica)再生过程进行转录组分析,组装获得163 329条转录本和101 183条Unigenes,Q30在90.5%以上,转录本的N50为1 346 bp,注释率为30.78%。聂鸿涛等[26]开展了大竹蛏高通量转录组测序数据组装和分析,得到305 485条转录本,去冗组装获得190 856条Unigenes,N50为1 875 bp,Q30为91.89%,注释率为33.18%。殷丽坤等[27]对低氧胁迫下中华圆田螺(Cipangopaludina cathayensis)肝脏组织进行高通量测序,发现只有11.46% Unigenes得到注释。本研究中,基于高通量测序技术对大竹蛏水管自切前后的水管基部组织进行转录组测序,获得235 719个转录本和103 909条Unigenes,Q30达92%以上,将其与多个数据库比对共有30 027个Unigenes得到注释,注释率为28.90%,部分基因未得到注释可能是数据库中近源物种基因注释信息较少,还有大量的未知基因有待研究。因此,加强大竹蛏全基因的分析研究,将有助于提高基因的注释率。
3.2 大竹蛏水管组织受损的早期响应
生物体内具有不依赖于转录水平的快速响应损伤的保守分子机制,研究发现,多种小分子物质可以作为信号分子对组织损伤做出响应(如Ca2+、ATP、ROS等)[6],这些信号分子将损伤信号传递给细胞或组织,从而引起一系列响应损伤的生物学过程[28]。Ca2+作为生物体内含量最高的金属离子,在组织受损的快速应答中发挥重要作用,其通过受损细胞膜内流进入细胞与多种蛋白结合[29],提高多种酶活性继而引导组织修复和再生[30],同时Ca2+借助ATP还可以将受损信号向受损组织更远的方向传播[31]。此外,ROS也对钙离子传播受损信号具有重要影响[32]。本研究中,Profile5基因集的GO功能注释发现,自切后7 h大量差异基因显著富集于离子结合条目(图6),其中,钙离子结合条目有98条基因富集到该分子功能条目且富集程度最高。这表明,在大竹蛏水管再生早期或大竹蛏对受损组织的早期响应过程中,钙离子很可能充当信号分子,在传递受损信号中具有重要作用。
3.3 大竹蛏水管再生对受体活性的影响
G蛋白偶联受体(GPCR)是真核生物中最大的一类膜蛋白家族,具有接受并传导多种细胞外信号分子的功能,其在受损组织再生过程中发挥重要作用。周晓春[15]从转录组水平发现,蝾螈肢体再生过程中GPCR介导的信号传导活动可促进细胞分化及组织再生。郭会萍[33]在斑马鱼性腺和心脏再生研究中发现,GPCR可以调控p-PKC/p-Akt活性,影响受损组织芽基生成及可塑性。本研究中,Profile17基因集GO功能注释结果显示,以GPCR活性为代表的受体活性条目被显著富集,其自切后表达量均高于自切前,RT-qPCR验证了GPR84在自切后表达量显著高于自切前。这表明,在大竹蛏水管再生过程中,GPR84很可能充当接收和传导各种再生相关信号的信号分子,并能激活下游信号通路。
3.4 大竹蛏水管再生期间的代谢响应
组织再生是一个耗能过程,DNA合成、细胞有丝分裂和细胞迁移均会消耗大量的能量[34],糖类代谢是生物体内能量的主要来源,途径包括糖酵解和三羧酸循环等。本研究中,BC vs ATb组GO功能注释和KEGG通路富集分析显示,多个与糖代谢相关的条目与通路被显著富集,如几丁质代谢、氨基糖代谢、核苷酸糖代谢和氮素代谢等。Profile12中基因集GO富集结果同样显示,几丁质代谢、氨基糖代谢等代谢过程被显著富集,并且其基因表达量在水管再生早期(自切后7 h)无明显变化,但随着再生的继续,7 d和20 d时基因表达明显上调。这与Wang等[17]在菲律宾蛤仔水管再生过程的研究中发现,再生中后期大量基因被富集到能量代谢和生物合成的结果类似。这表明,大竹蛏水管在响应早期损伤后,物质代谢及生物合成过程可能大量增加以满足组织再生的需求。
4 结论
1)通过高通量测序技术分析大竹蛏水管再生不同时间点的转录组变化情况发现,再生不同时间点基因表达有所差异,差异基因主要富集在离子结合、受体活性和能量代谢等过程中。
2)初步推测大竹蛏水管再生早期,以钙离子为代表的信号分子在传递受损信号过程中发挥重要作用,同时多种受体活性增强响应水管再生过程,随着再生的进行,能量代谢和生物合成过程大量增加以满足组织再生的需求。本研究中初步分析了大竹蛏水管再生过程中的转录组变化情况,为深入研究水管再生机制提供了有益参考。
[1] 吴杨平,陈爱华,姚国兴,等.大竹蛏生物学特性的初步研究[J].水产科学,2011,30(12):764-767.
WU Y P,CHEN A H,YAO G X,et al.Biological characteristics of Solen grandis dunker[J].Fisheries Science,2011,30(12):764-767.(in Chinese)
[2] IVANKOVIC M,HANECKOVA R,THOMMEN A,et al.Model systems for regeneration:planarians[J].Development,2019,146(17):dev167684.
[3] JOVEN A,ELEWA A,SIMON A.Model systems for regeneration:salamanders[J].Development,2019,146(14):dev167700.
[4] MADEN M,VARHOLICK J A.Model systems for regeneration:the spiny mouse,Acomys cahirinus[J].Development,2020,147(4):dev167718.
[5] MARQUES I J,LUPI E,MERCADER N.Model systems for regeneration:zebrafish[J].Development,2019,146(18):dev167692.
[6] 孟曦男,许静秀,徐素宏.组织器官损伤修复和再生研究进展[J].中国细胞生物学学报,2019,41(9):1677-1689.
MENG X N,XU J X,XU S H.Advances in tissue repair and regeneration[J].Chinese Journal of Cell Biology,2019,41(9):1677-1689.(in Chinese)
[7] KAWAKAMI Y,RODRIGUEZ ESTEBAN C,RAYA M,et al.Wnt/beta-catenin signaling regulates vertebrate limb regeneration[J].Genes &Development,2006,20(23):3232-3237.
[8] JOPLING C,SLEEP E,RAYA M,et al.Zebrafish heart regeneration occurs by cardiomyocyte dedifferentiation and proliferation[J].Nature,2010,464(7288):606-609.
[9] WEHNER D,CIZELSKY W,VASUDEVARO M,et al.Wnt/β-catenin signaling defines organizing centers that orchestrate growth and differentiation of the regenerating zebrafish caudal fin[J].Cell Reports,2014,6(3):467-481.
[10] VICKERY M C L,VICKERY M S,MCCLINTOCK J B,et al.Utilization of a novel deuterostome model for the study of regeneration genetics:molecular cloning of genes that are differentially expressed during early stages of larval sea star regeneration[J].Gene,2001,262(1/2):73-80.
[11] GALLIOT B.Hydra,a fruitful model system for 270 years[J].The International Journal of Developmental Biology,2012,56(6/7/8):411-423.
[12] ZHANG X J,SUN L N,YUAN J B,et al.The sea cucumber genome provides insights into morphological evolution and visceral regeneration[J].PLoS Biology,2017,15(10):e2003790.
[13] 檀永凯,李霞,段晶晶.仿刺参内脏再生过程的能量代谢及生化组成变化[J].中国水产科学,2008,15(4):683-688.
TAN Y K,LI X,DUAN J J.Energy metabolism and biochemical composition of Apostichopus japonicus during the regeneration of viscera[J].Journal of Fishery Sciences of China,2008,15(4):683-688.(in Chinese)
[14] WRINN K M,UETZ G W.Impacts of leg loss and regeneration on body condition,growth,and development time in the wolf spider Schizocosa ocreata[J].Canadian Journal of Zoology,2007,85(7):823-831.
[15] 周晓春.从转录组和蛋白组水平研究蝾螈肢体再生中细胞分化的途径和方式[D].新乡:河南师范大学,2014.
ZHOU X C.Study on the posttranscriptional and posttranslational ways and means of differentiation in newt limb regeneration[D].Xinxiang:Henan Normal University,2014.(in Chinese)
[16] BAI Y,NIE H,WANG Z,et al.Genome-wide identification and transcriptome-based expression profiling of Wnt gene family in Ruditapes philippinarum[J].Comparative Biochemistry and Physiology Part D:Genomics and Proteomics,2020,35:100709.
[17] WANG Z,BAI Y,NIE H,et al.Molecular mechanisms of wound healing and regeneration of siphon in the Manila clam Ruditapes philippinarum revealed by transcriptomic analysis[J].Genomics,2021,113(3):1011-1025.
[18] 熊加奉.缢蛏水管发育和水管组织再生分子机制的研究[D].上海:上海海洋大学,2021.
XIONG J F.Study on the molecular mechanism of siphon development and siphon tissue regeneration in the Sinonovacula constricta[D].Shanghai:Shanghai Ocean University,2021.(in Chinese)
[19] WU Y P,CHEN A H,ZHANG Y,et al.Metabolomics approach to assess the effect of siphonal autotomy on metabolic characteristics of razor clam Solen grandis[J].Scientific Reports,2022,12:5528.
[20] GRABHERR M G,HAAS B J,YASSOUR M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome[J].Nature Biotechnology,2011,29(7):644-652.
[21] ANDERS S,HUBER W.Differential expression analysis for sequence count data[J].Nature Precedings,2010,11(10):R106.
[22] ERNST J,BAR-JOSEPHZ.STEM:a tool for the analysis of short time series gene expression data[J].BMC Bioinformatics,2006,7:191.
[23] XIUME I,WE I.Molecular cloning and mRNA expression of two peptidoglycan recognition protein (PGRP) genes from mollusk Solen grandis[J].Fish &Shellfish Immunology,2012,32(1):178-185.
[24] 杨旻珉.急性低温应激下罗氏沼虾相关生化响应及基因表达模式研究[D].湖州:湖州师范学院,2022.
YANG M M.Biochemical indices and expression patterns of related functional genes under acute low temperature stress in Macrobrachium rosenbergii[D].Huzhou:Huzhou University,2022.(in Chinese)
[25] 杨轶博.基于高通量测序的日本三角涡虫再生过程转录组分析[D].新乡:河南师范大学,2018.
YANG Y B.Transcriptome analysis of the regeneration process of Dugesia japonica based on high-throughput sequencing [D].Xinxiang:Henan Normal University,2018.(in Chinese)
[26] 聂鸿涛,姜力文,郑梦鸽,等.大竹蛏高通量转录组测序数据组装和分析[J].大连海洋大学学报,2017,32(6):658-663.
NIE H T,JIANG L W,ZHENG M G,et al.Transcriptome data assembly and analysis of grand jackknife clam Solen grandis through high-throughput sequencing[J].Journal of Dalian Ocean University,2017,32(6):658-663.(in Chinese)
[27] 殷丽坤,黄凯,于凯,等.低氧胁迫下中华圆田螺的肝脏转录组学分析[J/OL].渔业科学进展,2022.DOI:110.19663/j.issn2095-9869.20220418005.
YIN L K,HUANG K,YU K,et al.Transcriptome analysis of liver tissue of Cipangopaludina cathayensis under hypoxic stress[J/OL].Progress in Fishery Sciences,2022.DOI:110.19663/j.issn2095-9869.20220418005.(in Chinese)
[28] HORN A,JAISWAL J K.Cellular mechanisms and signals that coordinate plasma membrane repair[J].Cellular and Molecular Life Sciences,2018,75(20):3751-3770.
[29] POTEZ S,LUGINBÜHL M,MONASTYRSKAYA K,et al.Tailored protection against plasmalemmal injury by annexins with different Ca2+sensitivities[J].Journal of Biological Chemistry,2011,286(20):17982-17991.
[30] XU S,CHISHOLM ANDREW D.A Gαq-Ca2+signaling pathway promotes actin-mediated epidermal wound closure in C.elegans[J].Current Biology,2011,21(23):1960-1967.
[31] COVIAN-NARES J F,KOUSHIK S V,PUHL H L 3rd,et al.Membrane wounding triggers ATP release and dysferlin-mediated intercellular calcium signaling[J].Journal of Cell Science,2010,123(Pt 11):1884-1893.
[32] RECZEK C R,CHANDEL N S.ROS-dependent signal transduction[J].Current Opinion in Cell Biology,2015,33:8-13.
[33] 郭会萍.斑马鱼性腺和心脏可塑性再生的表观遗传调节机制研究[D].上海:上海海洋大学,2019.
GUO H P.The epigenetic mechanisms of gonadal and cardiac plastic regeneration in zebrafish[D].Shanghai:Shanghai Ocean University,2019.(in Chinese)
[34] 杨晖,徐存拴.大鼠肝再生启动阶段能量代谢物靶向定量分析[J].解剖学报,2021,52(3):377-383.
YANG H,XU C S.Targeted quantitative analysis of energy metabolites in the priming phase during rat liver regeneration[J].Acta Anatomica Sinica,2021,52(3):377-383.(in Chinese)