康吉鳗属(Conger Cuvier,1817)隶属于鳗鲡目(Anguillformes)康吉鳗科(Congrinae)。据2023年Eschmeyer’s Catalog of Fishes数据库记载,该属有效种类有16种;据《中国动物志:硬骨鱼纲 鳗鲡目 背棘鱼目》[1]和《中国海洋鱼类》[2]记载,中国沿海康吉鳗属有标本的有3种,即星康吉鳗(C.myriaster)、日本康吉鳗(C.japonicus)和灰康吉鳗(C.cinereus);据《中国海洋及河口鱼类系统检索》[3]记载,中国沿海康吉鳗属为4种,即星康吉鳗、灰康吉鳗、乔氏康吉鳗(C.jordani)和大头康吉鳗(C.macrocephalus)。Smith等[4-5]和Hatooka[6]的研究均表明,日本康吉鳗为乔氏康吉鳗的同物异名。因此,在中国沿海康吉鳗属鱼类有标本的有效种为4种,即星康吉鳗、灰康吉鳗、乔氏康吉鳗和大头康吉鳗,该属最大的形态特征为头中等大,后部侧扁,头部感觉孔少,颌齿呈圆锥状,口裂平直且向后延伸到眼中部下方或后下方[1-3]。
2021年于辽宁省大连市长海县獐子岛海域垂钓得到1尾体色呈不均匀暗褐色、鱼鳍呈黑色、体延长、前部近圆筒形,背鳍起点位于胸鳍末端正上方,鱼体背部无白色点列的鱼类标本,其全长为168.50 cm,体质量为15.25 kg。初步判断其隶属于鳗鲡目康吉鳗科康吉鳗属。根据鳗鲡目鱼类志等历史文献鉴定发现,该标本的形态特征与中国沿海有记录的康吉鳗属4种康吉鳗均有不同。与该属其他12种鱼类的原始描述比对鉴定后发现,该标本符合Jordan和Snyder 1901年定名的暗康吉鳗(C.erebennus)形态特征[7]。该种目前仅在日本海域有报道,且对报道海域底层渔业资源构成了一定威胁[7-9]。本研究中,结合暗康吉鳗种类的原始描述及其相关文献[4,6-9],对采集到的标本形态特征进行了详细再描述,并进行了基于线粒体COⅠ和16S rRNA基因的遗传学分析,首次测定了其COⅠ分子条形码,研究结果不仅填补了中国对暗康吉鳗生物学研究方面的空白,也为监测和防控其对渔业生产存在的潜在风险研究提供了参考。
1 材料与方法
1.1 材料
标本(编号:LHYT2021-021)于2021年11月在辽宁省大连市长海县獐子岛海域(122°01′E,39°10′N)采集,标本为垂钓所得,全长为168.50 cm,体质量为15.25 kg(图1)。鲜活标本用冰块保存后迅速运输至实验室,于-20 ℃下冷冻保存。取腹部肌肉保存于体积分数为95%的乙醇中,用于全基因组DNA提取。
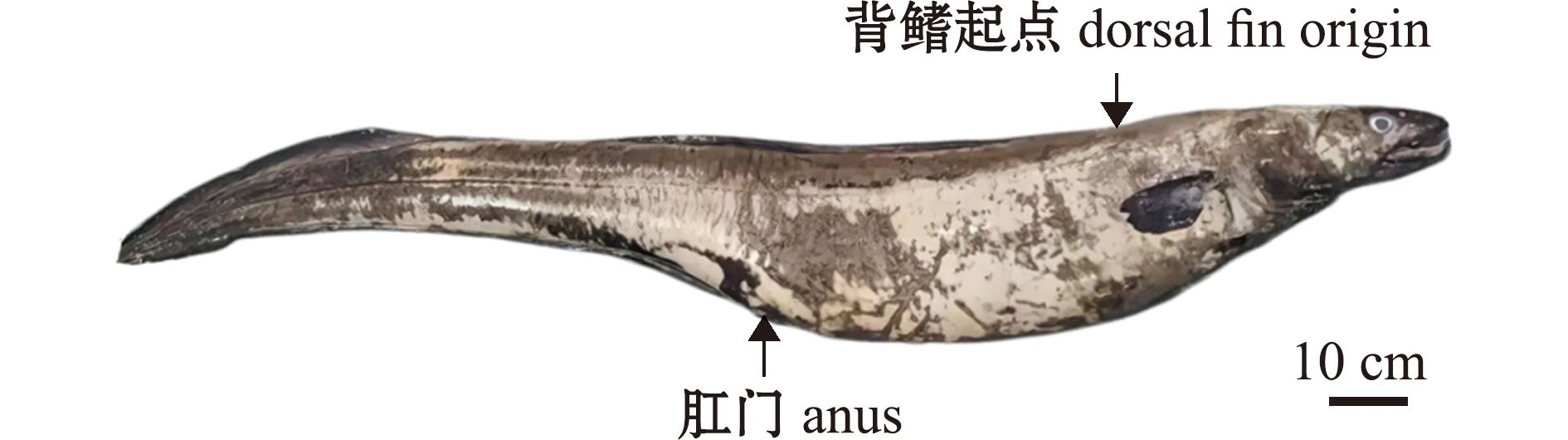
图1 獐子岛海域暗康吉鳗标本
Fig.1 Specimen of Conger erebennus collected from Zhangzidao Island
1.2 方法
1.2.1 形态学测量 以《中国动物志:硬骨鱼纲 鳗鲡目 背棘鱼目》[1]为参考,对标本的17个可量性状、2个可数性状和体色进行描述。由于标本个体较大,采用最小刻度值为1 cm卷尺进行形态学测量。可量性状包括全长、体长、头长、体高、肛前距、肛后距、臀鳍前距、臀鳍起点至肛门距离、背鳍前距、胸鳍长度、上颌长、下颌长、吻长、眼径、前鼻孔间距、后鼻孔间距和前后鼻孔间距。可数性状包括胸鳍条数和脊椎骨数。
1.2.2 DNA条形码分析 取保存的肌肉组织样本,采用酚-氯仿抽提法提取全基因组DNA,将DNA溶解后于4 ℃下保存,送上海凌恩生物科技有限公司进行测序。以已发布的乔氏康吉鳗线粒体基因组(GenBank登录号NC_027186.1)为参考,采用Illumina NovaSeq 6000平台进行线粒体全基因组二代测序,采用GetOrganelle 1.6.4和SPA des-3.13.1软件包对线粒体基因组测序原始数据进行比对组装。用在线MITOS工具对线粒体基因进行注释后,所得的线粒体基因组全序列NCBI登录号为NC0623731,后续系统发育分析中,选取的样本COⅠ和16S rRNA基因序列号也用NC0623731表示。
1.2.3 胃含物组成分析 解剖后通过目测鉴定摄食等级后取出胃含物样品,用吸水纸吸干胃含物表面水分后移至精确度为0.01 g的电子天平上测量胃含物总质量;再利用生物解剖镜(奥林巴斯SZ 61)对胃含物进行种类鉴定,尽可能鉴定到最低分类阶元。将分类后的饵料生物转移到电子天平上进行称量。由于胃含物消化程度不同,部分胃含物的种类识别难度较大,首先将胃含物大致分为鱼类、甲壳类、软体类及其他4类进行统计,然后对能够识别到科水平的种类另进行统计。
1.2.4 遗传学分析 从GenBank下载已发布的3种康吉鳗属鱼类的线粒体16S rRNA和COⅠ序列,并与本研究标本测定的相应序列进行遗传学对比分析。采用MEGA 5.0软件,以小头鸭嘴鳗(Nettastoma parviceps)和斑点长犁齿鳗(Hoplunnis punctata)为外群,构建康吉鳗属的最大似然(maximum-likelihood)系统进化树,并基于Kimura2-Parameter核苷酸最佳替代模型计算遗传距离,以Hebret等[10]提出的物种界定的遗传距离阈值为0.02为标准,确认物种之间的系统发育关系。
采用数量百分比(%)和质量百分比(%)对饵料重要性进行评价。
2 结果与分析
2.1 暗康吉鳗形态特征再描述
Conger erebennus Jordan &Snyder 1901:849,Fig.3[7];Nakabo 2002:229[11];Smith et al.2016:735,Fig.2[4];Moramura et al.2018:27[12];Guitai et al.2020:66[13];Koeda et al.2021:8,Fig.3C[14]。
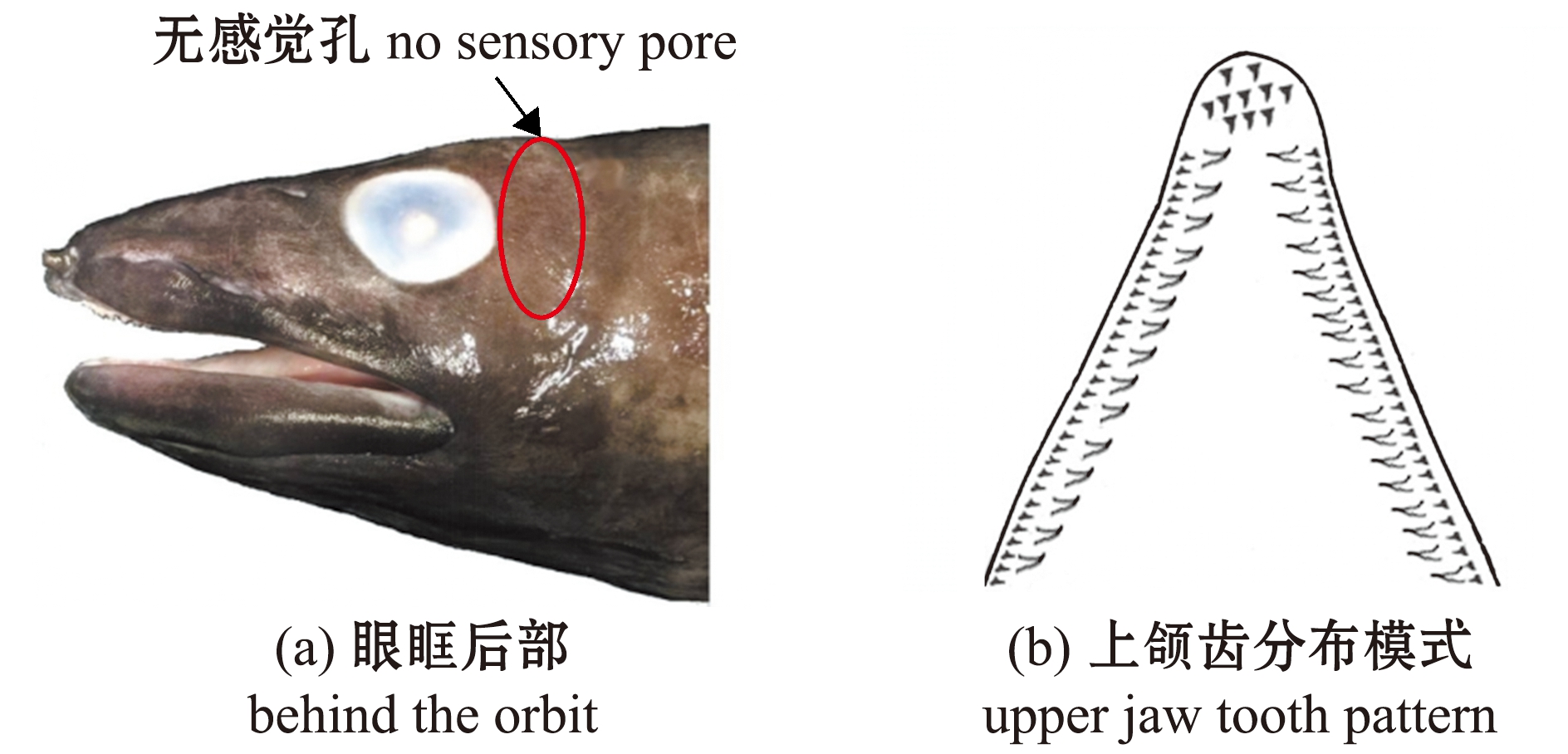
图2 暗康吉鳗眼后部及上颌齿分布模式
Fig.2 Behind the orbit and upper jaw teeth pattern of Conger erebennus
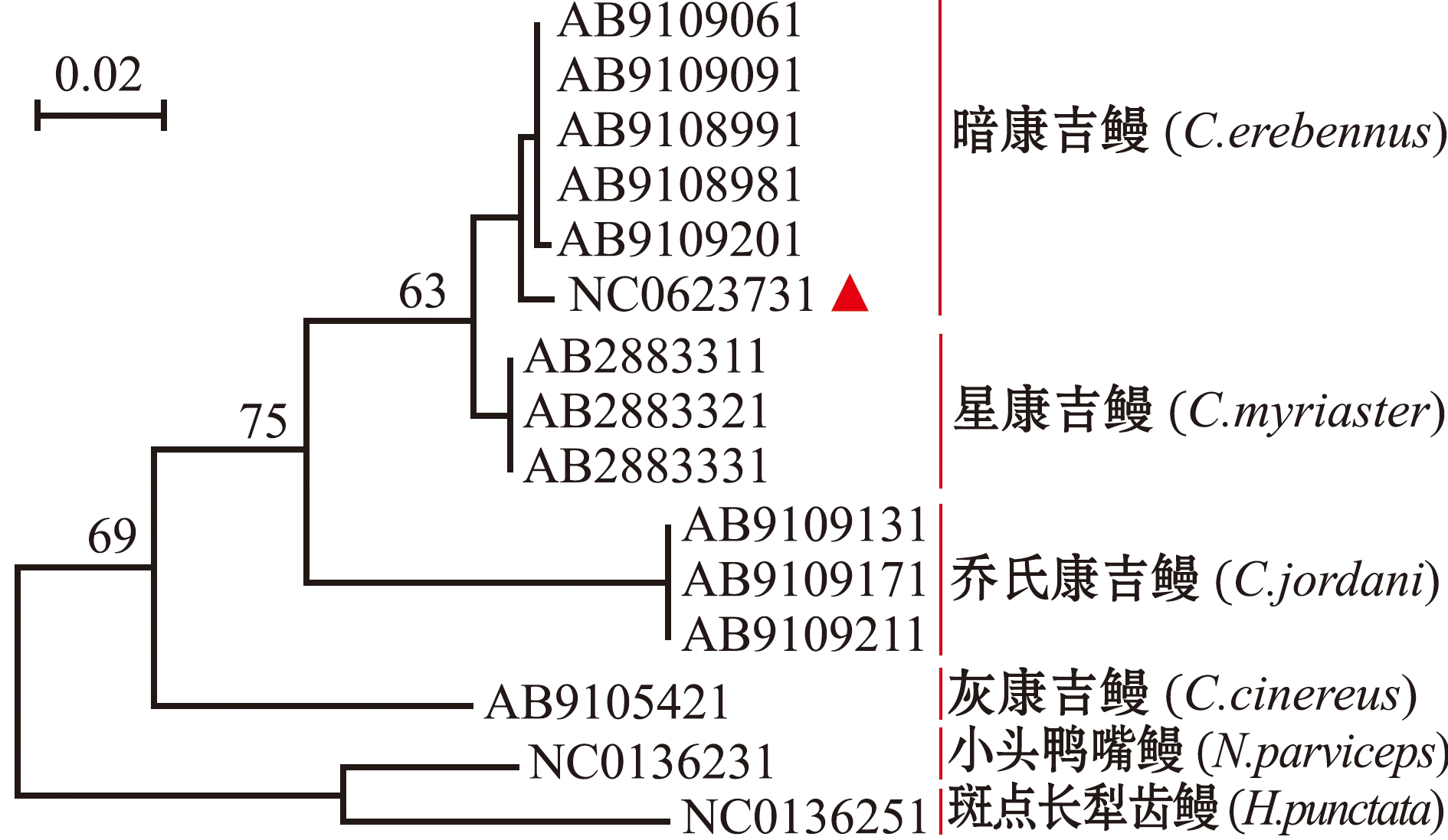
节点的数值为支持率,下同。
The value of the node is the support rate,et sequentia.
图3 基于16S rRNA的最大似然系统发育树
Fig.3 Maximum likelihood tree of Conger elebennus based on 16S rRNA gene
鉴别特征:背鳍起点始于胸鳍后缘正上方;眼后无感觉孔;鱼体背部无白色点列。
特征描述:体延长,前部近圆筒形,后部侧扁。头中等大,呈圆锥形,前部稍平扁。胸鳍发达,背鳍起点始于胸鳍后缘正上方。背鳍向后延伸与尾鳍连接。肛门位于体中部前方。胸鳍软条数19,脊椎骨数149。
上颌略长于下颌,唇发达,左右不相连。唇边缘具扩展的肉质瓣,口闭合时颌齿不外露。上颌齿呈尖锐的圆锥状,前上颌齿不规则分布,后上颌齿2列规则排列,外侧1列细小致密,内侧1列粗大稀疏;舌较大,前部游离。口大,口裂平直向后延伸达瞳孔后下方,未超过眼后缘;眼大,略呈椭圆形,眼被透明膜覆盖,眼后无感觉孔。鼻孔每侧2对,分离;前鼻孔位于吻端近前缘,具短管,短管长度大于前鼻孔孔径;后鼻孔呈圆孔状,位于眼前上缘水平线之下,距离眼前缘较近(小于眼直径的1/2),不具管状,边缘具皮瓣(图1、图2)。
参照Smith等[5]的方法,对该标本形态数据进行标准化处理,即头长、背鳍前距、肛前距、躯干长、肛门后距与全长的比值分别为13.7%、21.4%、39.8%、23.2%、58.5%;头长、背鳍前距、躯干长与肛门前距的比值分别为41.7%、53.7%、58.2%;眼径、眼间距、上颌长、鳃间距、胸鳍长、鳃孔长与头长的比值分别为11.5%、18.2%、32.1%、27.3%、32.1%、14.2%;躯干与头长的比值为1.91。
体色:体无鳞,刚出水时体色呈不均匀暗褐色,腹部具淡褐色斑块;死后通体呈暗褐色,体背侧无白色点列;鱼鳍呈黑色,鱼鳍边缘黑色加深。
2.2 DNA条形码鉴定
2.2.1 16S rRNA基因序列鉴定 将GenBank下载的灰康吉鳗、吉氏康吉鳗和星康吉鳗的16S rRNA基因序列与标本长度为475 bp的16S rRNA序列(NC0623731)比较发现:本研究中标本与Akiyama等[9]发布的采集自日本的5条暗康吉鳗序列相似度极高,在长度为475 bp核苷酸序列上共有9个位点存在核苷酸差异,其中,转换位点6个,颠换位点3个,转换和颠换位点比为2∶1,且遗传距离非常小(0.001~0.003),达到种内水平(<0.02);与星康吉鳗(0.105~0.108)、乔氏康吉鳗(0.123)、灰康吉鳗(0.173)的遗传距离均较大,达到种上水平(>0.02)。系统发育分析显示,本研究中标本的16S rRNA基因序列首先与已发布的暗康吉鳗16S rRNA序列聚为一支,然后与星康吉鳗、乔氏康吉鳗和灰康吉鳗聚类(图3)。
2.2.2 COⅠ基因序列鉴定 将GenBank下载的灰康吉鳗、吉氏康吉鳗和星康吉鳗的COⅠ基因序列与标本长度为582 bp的COⅠ序列(NC0623731)比较发现,本研究中标本与已发布的星康吉鳗(0.123~0.129)、灰康吉鳗(0.153~0.182)、乔氏康吉鳗(0.193~0.221)的遗传距离较大,均达到种上水平(>0.02)。系统发育分析显示,本研究中标本的COⅠ基因序列单独成一支,然后依次与星康吉鳗、灰康吉鳗和乔氏康吉鳗聚类(图4)。
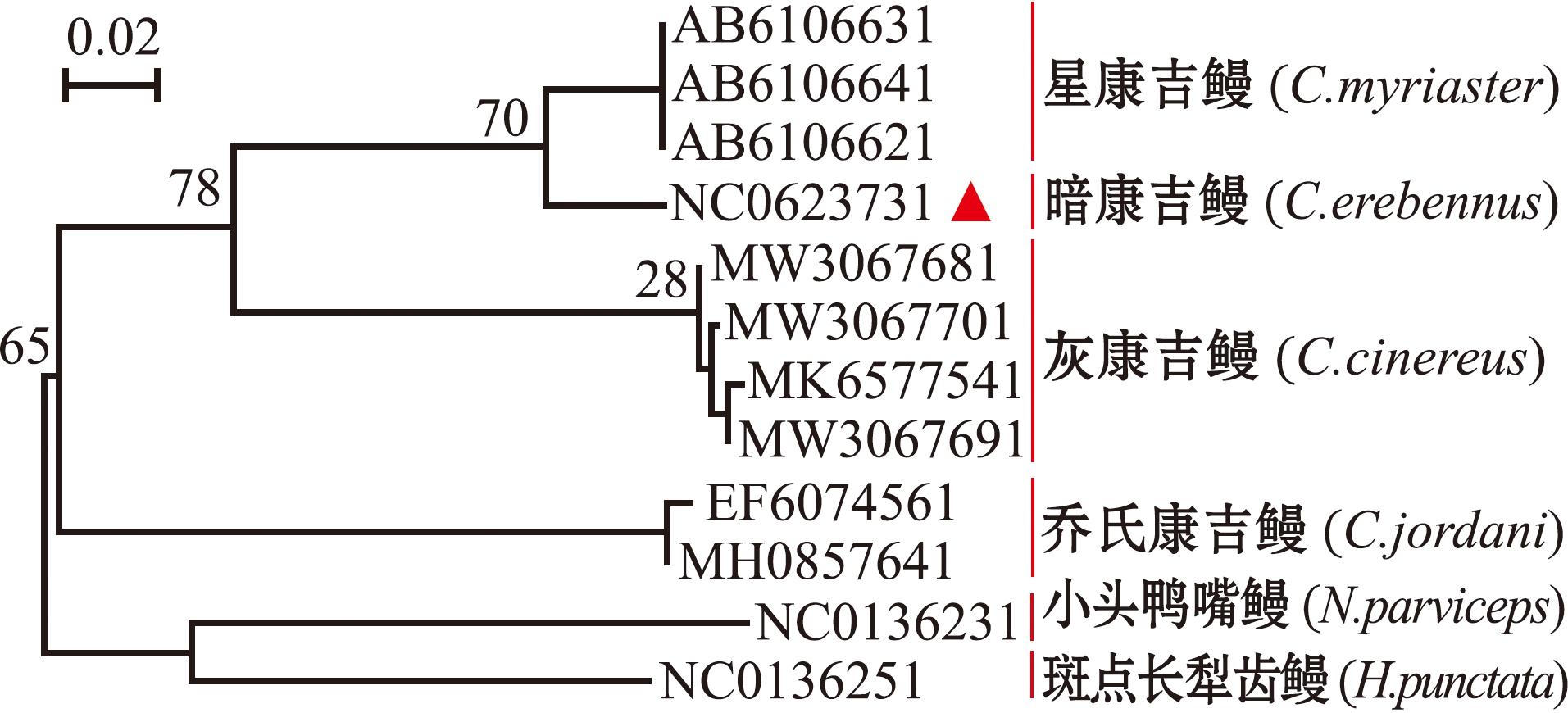
图4 基于COⅠ的最大似然系统发育树
Fig.4 Maximum likelihood tree of Conger elebennus based on COⅠ gene
2.3 胃含物及食性
解剖后对标本的胃含物观察发现,胃内容物占胃总体积的80%(3级胃)。分类鉴定结果显示,胃中含有动物个体总数为16,总质量为35.88 g,类群数量和质量百分比呈现相同趋势,从高到低依次为鱼类、软体类、甲壳类和其他(表1)。利用生物解剖镜对胃含物进行鉴定表明,能鉴定到科水平的生物有8种,其中,鱼类3种(舌鳎、鱼銜和平鲉)、头足类2种(乌贼、章鱼)、棘皮类1种(海参)和甲壳类2种(鼓虾、虾蛄),鱼类数量和质量百分比均为最高,尤其是舌鳎(表1)。
表1 獐子岛海域暗康吉鳗样品的胃含物分析
Tab.1 Stomach contents of Conger erebennus caught in Zhangzidao Island
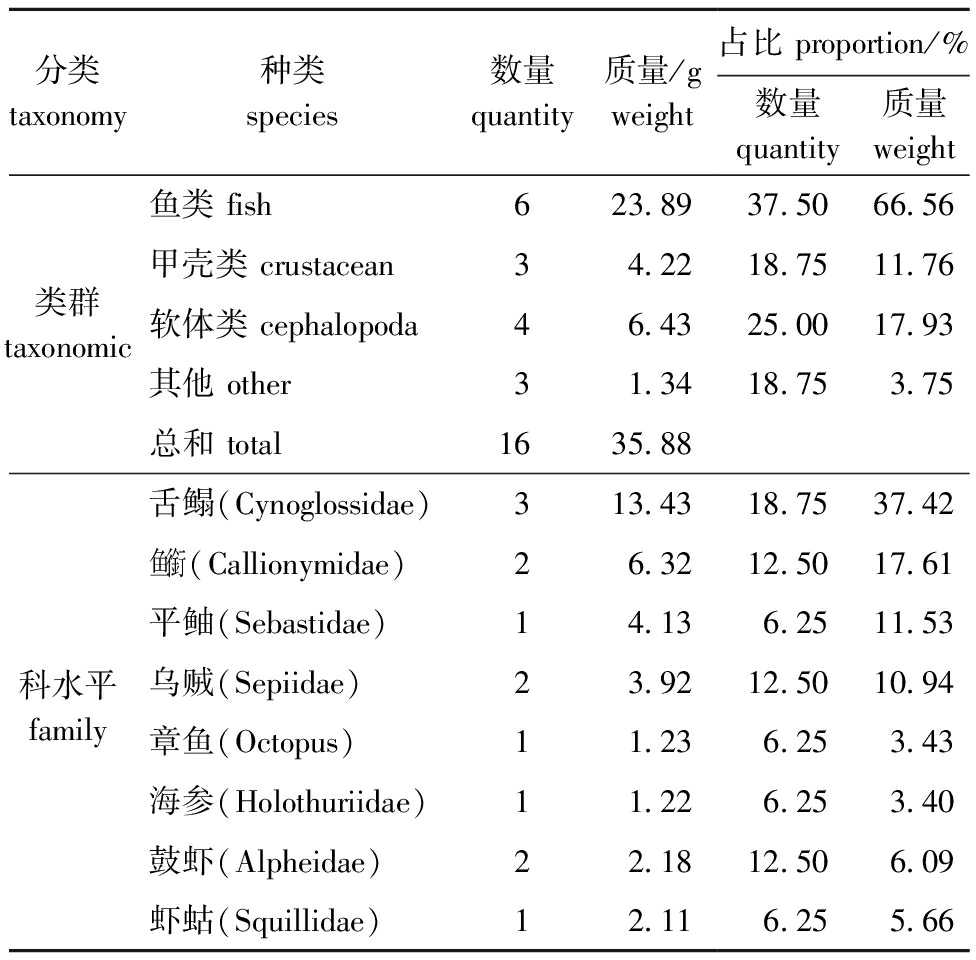
分类taxonomy种类species数量quantity质量/gweight占比 proportion/%数量 quantity 质量weight鱼类 fish623.8937.5066.56甲壳类 crustacean34.2218.7511.76类群taxonomic软体类 cephalopoda46.4325.0017.93其他 other31.3418.753.75总和 total1635.88舌鳎(Cynoglossidae)313.4318.7537.42鱼銜(Callionymidae)26.3212.5017.61平鲉(Sebastidae)14.136.2511.53科水平family乌贼(Sepiidae)23.9212.5010.94章鱼(Octopus)11.236.253.43海参(Holothuriidae)11.226.253.40鼓虾(Alpheidae)22.1812.506.09虾蛄(Squillidae)12.116.255.66
3 讨论
3.1 暗康吉鳗的形态学特征及鉴定
本研究中标本的形态特征与Jordan和Snyder(1901)[7]对暗康吉鳗的原始描述基本一致,标本呈现出全身暗褐色,躯干长与头长的比值为1.91,主要形态特点是背鳍起点始于胸鳍末端正上方,眼后无感觉孔,鱼体背部无白色点列等。本研究中灰康吉鳗头长/全长的比值为13.7%,与Smith等[4]认为暗康吉鳗头长/全长的比值(16%~18%)可以作为鉴定暗康吉鳗形态标准的结论不一致。但Akiyama等[9]对采集自日本东京湾的大量暗康吉鳗(368尾)的形态测定表明,暗康吉鳗的头长与全长的比值为13.4%~24.7%,本研究中标本的比值在其范围之内。这可能是由于Smith等[4]的研究数据来自对少量标本的测定,随着标本量增加,形态测量值范围会有所扩大。Akiyama等[9]研究结果还表明,暗康吉鳗头长与全长的比值范围与乔氏康吉鳗(11.5%~16.9%)有较大重叠。因此,头长与全长的比值能否作为鉴定暗康吉鳗的形态分类依据有待商榷。此外,众多学者在暗康吉鳗形态及分类研究中均一致认为,背鳍起点始于胸鳍末端正上方是其最主要的形态鉴定依据[4,7-9,11-14]。本研究中标本的背鳍起点始于胸鳍末端正上方(图5),与学者们的研究记录完全一致。

A—本研究标本;B—引自Nakabo[11](2002);C—引自Guitai等[13](2020);D—引自Jordan等 [7](1901);E—引自Moramura等[12](2018)。
A—from this study;B—from Nakabo[11](2002);C—From Guitai et al.[13](2020);D—from Jordan et al.[7](1901);E—from Moramura et al.[12](2018).
图5 相关研究中暗康吉鳗的照片
Fig.5 Images of Conger erebennus from related studies
3.2 暗康吉鳗DNA条形码与康吉鳗属其他种类遗传学比较
遗传差异分析显示,本研究中标本的16S rRNA基因序列与Akiyama等[9]研究中测定发布的暗康吉鳗16S rRNA高度一致,在系统发育树上二者聚为一支,且与星康吉鳗、乔氏康吉鳗和灰康吉鳗等其他3种康吉鳗的遗传距离为0.105~0.173,远大于Hebert等[10]提出的种间遗传距离通常大于0.02的鉴定标准。本研究中首次发布了暗康吉鳗的COⅠ序列,且基于COⅠ序列的遗传距离分析表明,暗康吉鳗与其他3种康吉鳗的遗传距离为0.123~0.221,遗传距离达到种上水平;基于COⅠ序列的系统发育分析也显示,暗康吉鳗独成一支。基于COⅠ和16S rRNA构建的系统发育树均支持这4个有效种,且均为暗康吉鳗与星康吉鳗率先聚为一支,之后与灰康吉鳗和乔氏康吉鳗聚类,但与这两种康吉鳗的聚类顺序在两种发育树上有所不同。这种聚类顺序不一致的情况,在石斑鱼亚科的系统发育研究中也存在[10,15],可能是由于16S rRNA较COⅠ更为保守,具有更低的进化速率所致。总之,从遗传差异分析来看,该标本确为暗康吉鳗。
3.3 暗康吉鳗食性分析及其在中国分布范围推测
Akiyama等[9〗和Katayama等[16]对日本东京湾的暗康吉鳗和乔氏康吉鳗的食性、年龄组成和生活史等进行了全面的生物学分析,结果表明,按照大类区分,暗康吉鳗胃含物中数量百分比从高到低依次为鱼类(47.70%)、甲壳类(38.80%)、软体类(9.80%);质量百分比依次为鱼类(63.90%)、软体类(18.00%)和甲壳类(12.40%)。本研究中,采自北黄海獐子岛海域标本胃含物分大类分析种类组成和质量百分比与Akiyama等[9]研究结果一致,主要包含鱼类、甲壳类、软体类和其他,且质量百分比从高到低依次为鱼类、软体类和甲壳类,但胃含物的数量组成有差异,即数量百分比从高到低依次为鱼类、软体类和甲壳类。Akiyama等[9〗和Katayama等[16]的研究表明,暗康吉鳗的胃含物鉴定到科水平时,数量百分比从高到低依次为虾虎鱼、虾蛄、海参、岩礁性鱼、舌鳎、鱼銜、乌贼、鼓虾、章鱼和刺鲀,质量百分比从高到低依次为虾蛄、海参、岩礁性鱼、乌贼、章鱼、刺鲀、鱼銜、舌鳎、虾虎鱼和鼓虾。但本研究中暗康吉鳗胃含物数量百分比依次为舌鳎、鱼銜、乌贼、鼓虾、平鲉、章鱼、海参和虾蛄,质量百分比依次为舌鳎、鱼銜、平鲉、乌贼、鼓虾、虾蛄、章鱼和海参,本研究中胃含物数量组成和占比跟日本学者的研究结果存在一定的差异。造成胃含物种类、数量及质量占比差异可能有两方面因素:一方面,不同海域暗康吉鳗的饵料生物群落结构和组成不同,会导致其胃含物种类结构的差异;另一方面,本研究中仅对1尾标本进行了胃含物分析,分析标本量较少,存在一定偶然性。Akiyama等[9]和Katayama等[16]认为,暗康吉鳗的存在已对东京湾海参资源、底栖鱼类和甲壳类的生存和捕捞生产构成重大威胁,有必要对其进行定期调查,并根据情况采取驱除对策。从本研究中标本的胃含物分析来看,黄海獐子岛海域的暗康吉鳗主要饵料也是舌鳎、平鲉、乌贼、鼓虾、虾蛄、章鱼和海参等重要的经济渔业资源,且该标本的个体较大,提示有必要运用环境DNA、水下观测等技术对暗康吉鳗的生物学和分布规律开展详细研究,以预防其对渔业生产的不利影响。
日本学者研究表明,暗康吉鳗和乔氏康吉鳗通常混居,是一种生活在水深为18~600 m的底层大型罕见凶猛性鱼类[9,14]。自1901年Jordan等[7]在日本东京湾发现暗康吉鳗以来,相继在日本北海道近海、与论岛东部海域、鹿儿岛内之浦湾和博多深海发现该物种。本研究中标本采自中国北黄海长海县獐子岛海域,以前在中国未见报道,为首次记录。据赵永波[17]于1996年记录,黄海北部有乔氏康吉鳗生存。结合日本学者对乔氏康吉鳗和暗康吉鳗的生物学分析,以及本研究中标本也采集自黄海北部,推测暗康吉鳗可能在中国北黄海水深18 m以上的深水海域有所分布。此外,根据线粒体16S rRNA基因序列的比对结果来看,采集自獐子岛海域的暗康吉鳗与日本群体间的遗传距离非常小(0.001~0.003),提示本研究中采集的暗康吉鳗可能起源于日本海域,今后有必要采集更多的样品,以进一步研究其转移机制。
4 结论
1)暗康吉鳗为中国沿海鱼类新记录种,可能分布于中国黄海北部深水区域,属于底层大型凶猛性鱼类,主要对底栖型鱼类、甲壳类、棘皮类和头足类等海洋生物资源的生存构成巨大威胁,应该加强对其监测和生物学研究。
2)暗康吉鳗区别于康吉鳗属其他种鱼类的主要形态学特征是背鳍起点恰位于胸鳍末端正上方,眼后无感觉孔,鱼体背部无白色点列。
3)本研究中标本的16S rRNA基因序列与已发布的暗康吉鳗16S rRNA高度一致;首次发布了暗康吉鳗的COⅠ序列DNA条形码,其16S rRNA和COⅠ序列与康吉鳗属其他种鱼类的遗传差异远达到种上水平。
[1] 张春光.中国动物志:硬骨鱼纲 鳗鲡目 背棘鱼目 [M].北京:科学出版社,2010:258.
ZHANG C G. Zoology of China:Osteichthyes,Anguilliformes,Dorsalis [M].Beijing:Science Press,2010:258. (in Chinese)
[2] 陈大刚,张美昭.中国海洋鱼类[M].青岛:中国海洋大学出版社,2015:412.
CHEN D G,ZHANG M Z.Marine fishes of China[M].Qingdao:China Ocean University Press,2015:412.(in Chinese)
[3] 伍汉霖,钟俊生.中国海洋及河口鱼类系统检索[M].北京:中国农业出版社,2021:161-162.
WU H L,ZHONG J S.Key to marine and estuarial fishes of China[M].Beijing:China Agriculture Press,2021:161-162.(in Chinese)
[4] SMITH D G,SCHWARZHANS W W,POGONOSKI J J.The identity of Conger japonicus(Anguilliformes:Congridae)[J].Copeia,2016,104(3):734-737.
[5] SMITH D G,HO H C.Review of the congrid eel genus Conger (Anguilliformes:Congridae) in Taiwan [J].Zootaxa,2018,4454(1):168-185.
[6] HATOOKA K.Recent revision of the congrid eels of Japan[J].Nippon Suisan Gakkaishi,2019,85(1):77.
[7] JORDAN D S,SNYDER J O.A review of the apodal fishes or eels of Japan,with descriptions of nineteen new species[J].Proceedings of the United States National Museum,1901,23(1239):837-890.
[8] ODANI K,NOBETSU T,MITSUHASHI M,et al.Records of a conger eel,Conger erebennus (Actinopterygii, Anguilliformes,Congridae),from Hokkaido,Japan[J].Bulletin of the Biogeographical Society of Japan,2005,60(3): 21-24.
[9] AKIYAMA S,MOTOMURA D,SHIMOMURA Y,et al.Feeding habits of beach Conger japonicus and Formosa conger Conger erebennus in Tokyo Bay[J].Nippon Suisan Gakkaishi,2015,81(1):130-132.
[10] HEBERT P D,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes[J].Proceedings Biological Sciences,2003,270(1512):313-321.
[11] NAKABO T.with pictorial keys to the species[M].Tokyo,Japan:Tokai University Press,2002.
[12] MORAMURA H,SEIJI H,NENOHIRO S.Identification guide to fishes of the Amami Islands,Japan[M].Kagoshima:Kagoshima University Museum of Comprehensive Studies,2018:27.
[13] GUITAI X Z,YAN Q L,YAMADA S Y,et al.Picture book of fish in dayu market[M].Kagoshima: Kagoshima University Museum of Comprehensive Studies,2020:66.
[14] KOEDA K,TAKASHIMA S,YAMAKITA T,et al.Deep-sea fish fauna on the seamounts of southern Japan with taxonomic notes on the observed species[J].Journal of Marine Science and Engineering,2021,9(11):1294.
[15] 梁日深,陈铭,廖国威,等.基于16S rRNA与COⅠ基因40种石斑鱼亚科鱼类分子系统进化关系[J].海洋学报,2020,42(6):9-19.
LIANG R S,CHEN M,LIAO G W,et al.Molecular phylogenetic relationships of 40 species of Epinephelinae based on the partial sequences of 16S rRNA and COⅠ genes[J].Haiyang Xuebao,2020,42(6):9-19.(in Chinese)
[16] KATAYAMA S,AKIYAMA S,SHIMOMURA Y,et al.Growth patterns and sex ratios of Conger japonicus and C.erebennus in Tokyo Bay[J].Nippon Suisan Gakkaishi,2015,81(4):688-693.
[17] 赵永波.黄海北部(辽宁)鱼类一新记录:日本康吉鳗(Conger japonicus Bleeker)[J].大连水产学院学报,1996,11(3):54-56.
ZHAO Y B.A new record of fish in the northern Yellow Sea (Liaoning) Conger japonicus Bleeker[J].Journal of Dalian Fisheries College,1996,11(3):54-56.(in Chinese)