刀鲚(Coilia nasus)隶属于鲱形目(Clupeiformes)鳀科(Engraulidae)鲚属(Coilia),在中国的渤海、黄海、东海海域及其通海的江河中均能发现其踪迹,其中以长江下游的捕获量最高。长江刀鲚鱼体丰腴肥满,肉鲜味美,曾与鲥、河豚并称为“长江三鲜”[1]。近年来,由于栖息地生存环境受到污染、恶化,以及酷渔滥捕、沿岸工程建设等人类活动的影响,野生长江刀鲚数量急剧下降,甚至不能形成优势种群[1-4],亟须对其自然种质资源进行有效保护、开发和利用。自2019年2月1日开始,中国农业农村部就已停止发放长江刀鲚专项捕捞许可证,禁止对其自然资源进行生产性捕捞,次年又开始实施长江“十年禁渔”政策。同期长江刀鲚全人工繁育技术和苗种规模化生产技术亦获得了突破性进展,这些举措不仅促进了长江刀鲚产业化养殖的发展,而且有效地缓解了长江刀鲚野生种质资源锐减的状况。
分子标记不仅可以探究整个动物基因组的遗传变异,且遗传相对稳定,常用于水产经济动物的群体遗传分析和分子育种研究,其中,微卫星标记(microsatellite)的应用最为广泛[5]。微卫星标记,亦称短链重复序列(short tandem repeats,STRs)或简单重复序列(simple sequence repeat,SSR),是在真核生物基因组中均匀分布的、由1~6个核苷酸串联重复片段构成的序列,这些序列的重复次数和重复单位在不同个体间呈现高度变异性和多态性,被广泛应用于水产经济动物的群体遗传分析、种质鉴定、分子标记辅助良种选育及构建遗传图谱等方面[5-7]。近年来,研究人员利用线粒体基因D-loop[8]、RAPD[9]和AFLP[10]等不同类型的分子标记,开展了长江刀鲚不同群体的遗传变异研究,这些类型的标记试验成本较高、操作复杂且数量相对较少,而微卫星标记则操作相对简单,成本较低且数量多,但对长江刀鲚SSR开发利用的报道较少。Ma等[11]基于FIASO法,从微卫星富集文库开发鉴定到12对多态性SSR标记,用于长江刀鲚和凤鲚的遗传分析。Chen等[12]基于磁珠富集法,从微卫星富集文库开发鉴定到34对多态性SSR标记,用于长江刀鲚及其近缘物种的通用性研究。Rong等[13]基于FIASO法,从微卫星富集文库开发鉴定到20对多态性微卫星标记,用于长江刀鲚的群体遗传研究。基于磁珠富集和FIASO等方法,虽然从水产动物基因组规模化开发的SSR标记多态性较高,但成本相对较高且费时、低效,已不能满足长江刀鲚群体遗传分析及分子标记辅助良种选育对SSR的需求,亟待大规模挖掘开发[5]。
与传统的基因组SSR(genomic SSR,G-SSR)开发技术相比,基于高通量转录组测序技术所开发的SSR,则被定义为表达序列标签-SSR(expressed sequence tag SSR,EST-SSR),虽然标记多态性相对G-SSR较低,但由于其数量丰富、开发成本低和开发周期短,且常常与相关性状的功能基因关联[5,14],而被广泛应用于水产经济动物多态性EST-SSRs的规模化开发与应用。近年来,陆续出现的海量高通量测序数据,亦为水产动物微卫星标记的开发提供了便利条件,同时,一大批针对高通量转录组数据挖掘EST-SSRs的软件,如MISA(MIcroSAtellite Identification Tool)[15]、Candi-SSR[16]、SSRMMD(Simple Sequence Repeat Molecular Marker Developer)[17]等多态性SSR位点筛选软件陆续被开发出来,极大地加快了水产经济动物多态性EST-SSRs的开发和利用研究进程。
本研究中,基于长江刀鲚雌、雄个体的脑转录组数据,采用MISA软件深入挖掘长江刀鲚脑转录组Unigenes中的EST-SSRs,采用SSRMMD软件规模化地开发多态性EST-SSRs,并利用筛选获得的18个多态性较好的EST-SSRs,评估了3个长江刀鲚养殖群体的遗传多样性,以期为长江刀鲚的群体遗传分析、分子标记规模化开发与利用及分子标记辅助良种选育提供科学参考。
1 材料与方法
1.1 材料
用于转录组测序的12尾2龄长江刀鲚(雌、雄各6尾),取自上海市水产研究所奉贤科研基地2018年的繁育群体;用于EST-SSRs多态性分析和有效性验证的长江刀鲚,分别取自该基地2018、2019和2020 年获得的繁育子代,每年各取24尾,分别记为2018、2019、2020养殖群体。
1.2 方法
1.2.1 长江刀鲚脑转录组测序 利用Illumina转录组文库构建试剂盒分别构建长江刀鲚雌、雄个体脑组织的转录组cDNA测序文库,并基于Illumina HisSeq 4000高通量测序平台进行测序。测序原始数据(Raw Reads)采用SeqPrep和Sickle软件进行质控和过滤处理,获得质量较高的纯净数据(Clean Reads);采用Trinity软件对所获得的Clean Reads进行从头组装和去冗余后,获得高质量的雌性脑转录组Unigenes(以下简称“CEF.fa”)和雄性脑转录组Unigenes(以下简称“CEM.fa”)。
1.2.2 长江刀鲚EST-SSRs的识别和特征分析 在Linux操作系统下,利用MISA软件(http://pgrc.ipk-gatersleben.de/misa /misa.html)分别扫描和检索CEF.fa和CEM.fa中的EST-SSRs。单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复基序的最少重复次数分别设置为10、6、5、5、5和5次,经筛选和计算后,统计雌性和雄性刀鲚转录组中这6类核苷酸重复基序的重复次数及各重复类型的比例;复合型EST-SSRs两个位点间最大间隔碱基数设置为100。
1.2.3 长江刀鲚多态性EST-SSRs的快速识别和检测 在Linux操作系统下,采用SSRMMD软件检索CEF.fa和CEM.fa中完美的EST-SSRs位点和候选的多态性EST-SSRs。软件运行参数如下:perl SSRMMD.pl -f1 CEFM/CEF.fa -f2 CEFM/CEM.fa -p1 -me NW -st 0 -t 2;从筛选出的多态性EST-SSRs中随机选取20条序列,并结合长江刀鲚参考基因组序列和EST-SSRs两侧保守侧翼序列的保守性特征,通过Primer Premier 5.0软件,设计、筛选出评分较高的EST-SSRs PCR扩增引物,委托生工生物工程(上海)股份有限公司进行引物合成与纯化,用于后续EST-SSRs有效性和多态性的检测。
从不同年份养殖长江刀鲚的每个样本,分别剪取鳍条组织约5 mg,用灭菌双蒸水清洗,剪碎后转移至1.5 mL离心管中,使用海洋动物组织基因组DNA提取试剂盒(DP324-03)[天根生化科技(北京)有限公司)],提取每个样本的基因组DNA;用15 g/L琼脂糖凝胶电泳评估DNA质量,利用NanoVue Plus紫外可见分光光度计测量浓度后,将符合要求的DNA样品稀释到50 ng/μL,于-20 ℃下保存备用。
随机选取10个DNA样本等量混合成PCR扩增的模板,在Eppendorf Mastercycler X50s梯度PCR仪(ABI,美国)上筛选各引物的最适退火温度。PCR反应体系(10 μL):模板DNA 1 μL,正、反向引物(10 μmol/L)各0.25 μL,2×Taq PCR MasterMix(天根生化,KT201)5 μL,用ddH2O补足至10 μL。PCR反应程序:94 ℃下预变性5 min;94 ℃下变性 0.5 min,48~68 ℃下退火0.5 min,72 ℃下延伸0.5 min,共进行35个循环;最后在72 ℃下再延伸7 min,4 ℃下保存备用。PCR产物用15 g/L的琼脂糖凝胶电泳进行检测,利用紫外凝胶成像系统拍照、保存,确定各EST-SSRs引物的最佳退火温度和有效性。
1.2.4 长江刀鲚不同年份养殖群体的种质资源评估 以2018、2019和2020年的3个长江刀鲚养殖群体共72个DNA样本为试验材料,利用筛选获得的18对多态性EST-SSRs引物(表1),在Eppendorf Mastercycler X50s梯度PCR仪器上进行PCR扩增。PCR反应体系和反应程序参照引物最佳退火温度的筛选过程,在各引物的最佳退火温度(表1)下获取PCR产物。利用10%非变性聚丙烯酰胺凝胶电泳对PCR产物进行电泳检测,通过银染法进行显色定影,利用数码相机对显色清晰的条带拍照保存,用于后续长江刀鲚不同养殖群体的遗传差异分析。
表1 长江刀鲚多态性EST-SSRs引物信息和参数
Tab.1 Primer sequences and parameters of polymorphic EST-SSRs in Coilia nasus

位点 locus重复基序 repeat motif引物序列(5'-3') primer sequence(5'-3')产物长度/bp product退火温度/℃ annealing temperatureEST-SSR01(ATC)7F: TCGTTGTTTCACTTTCATTTR: AGAACTCTCTGTGTTTGGGT15450 EST-SSR02(TGC)8F: TCGCTTCGCTCTCCCATCTCGR: AGGCATGCTGGTGGCCCTCTG14768EST-SSR03(AGAGG)5F: CACGGTCATAAATAAGGCR: TTTTGTTTGCAAGTCAAA16448EST-SSR04(GAG)8F: GTCATTGGTCTGAGCACGCR: GAGCCCACTGGTCCTTTAT12355EST-SSR05(ATC)6F: TCCTTTTTTTTCTGCGACTR: AACCATCCTGACCTTGTGA14552EST-SSR06(ATTC)5F: GTATGGAGGAAAATA-AAAGAAR: ATAATAAATGACAAATAA-CAA15745EST-SSR07(TCTG)6F: CACGACCTTCAAATAGGCTGTR: GGGCTGCAATTATGACTCTCA17757EST-SSR08(ACAGG)5F: TGGCTGGCGTATGTGCTGGGR: TCGGGGGTCTGATGGGGGTC12267EST-SSR09(ACAT)8F: CCCCTTAATCTCTGGTGACATR: GCTAACTGGATACCTGCCCTA11155EST-SSR10(CTG)7F: AGAGGGAAAGAAGAAGTGAGR: TGACATTTATCTGTGCTTGG18450EST-SSR11(CCAT)7F: TACAAGCAGAGGAG-GAGAAGGR: TGAGTGTGTGTGGAGAAAT-GG14756EST-SSR12(TGTT)8F; AAGCGCCCCAGACCCATTTR: TCTCGTGACGTATAACCCC11357EST-SSR13(CAC)13F: AGGGAGGGGGATTTTGTAR: TAGGGGTTGTGTTGTGCT15054EST-SSR14(ACAG)7F: AATAATGCAATGTCACACCGAR: AAATGAAATGAACAAAAAC-CC14955EST-SSR15(CTG)7F: TTTCCTTCAGTCCCGTGCR: TTGAGGGGTTGCGAGTAG12655EST-SSR16(AGGGGT)6F: AAATGAGAAGAAACCCGCAAR: ATCCATCAACTCCCAACCCT18058EST-SSR17(TGTT)11F: ATTTGTTATGCTGTTATGATGR: AACTTAAAGGTAACTCCACTG12148EST-SSR18(GCACT)6F: AAAGAAAAGAATGGGAAAAR: CTGAGCAGCTACTGTGGAG11950
1.2.5 遗传多样性参数的计算与分析 采用GenAlEx 6.0软件计算长江刀鲚不同养殖群体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、遗传相似度、遗传距离和遗传分化指数(FST)等遗传多样性参数,并进行哈迪-温伯格平衡检测(Hardy-Weinberg Equilibrium,HWE);采用MEGA 6.0软件构建不同群体间的UPGMA聚类分析树。
基于Botstein等[18]的公式,计算长江刀鲚不同群体和位点的多态信息含量(polymorphism information content,PIC):
其中:CPI为多态信息含量;pi、pj分别为群体中第i、j个等位基因的频率;N为等位基因数。
2 结果与分析
2.1 长江刀鲚脑转录组序列特征
从雌性长江刀鲚脑转录组中共获得49.69 Gbp Clean Data,6个样品的Clean Data均达到了 7.79 Gbp以上,Q30碱基百分比在95.11%以上,表明测序结果良好;经Trinity软件组装和优化后,共获得N50为1 488 bp的Unigenes 143 374个(表2)。
从雄性长江刀鲚脑转录组中共获得50.15 Gbp Clean Data,6个样品的Clean Data均达到了8.19 Gbp以上,Q30碱基百分比在95.30%以上,表明测序结果良好;经Trinity软件组装和优化后,获得N50为1 569 bp的Unigenes 135 215个(表2)。
表2 雌、雄长江刀鲚个体脑转录组序列信息
Tab.2 Sequence characteristics of the brain transcriptome in the female and male Coilia nasus

性别gender总Unigenes数量/条total Unigenes number总序列长度/bptotal sequence base最长序列长度/bplongest sequence base最短序列长度/bpshortest sequence base平均序列长度/bpaverage lengthN50/bpGC含量/%GC percent雌性 female143 374161 894 38417 903201882.061 48847.73雄性 male135 215154 489 66418 027201904.611 56947.84
2.2 长江刀鲚脑转录中EST-SSRs特征分析
雌性长江刀鲚143 374条Unigenes中总共获得95 905个EST-SSRs,其中,33.99%的Unigenes包含EST-SSRs,11.30%的Unigenes含有1个以上的EST-SSRs,平均每隔1 688 bp有一个EST-SSR;雄性长江刀鲚135 215条Unigenes中共获得88 774个EST-SSRs,其中,33.17%的Unigenes包含EST-SSRs,11.03%的Unigenes含有1个以上的EST-SSRs,平均每隔1 740 bp有一个EST-SSR(表3)。
表3 雌、雄长江刀鲚个体脑转录组EST-SSRs搜索结果
Tab.3 EST-SSRs search results in the brain transcriptome of female and male Coilia nasus
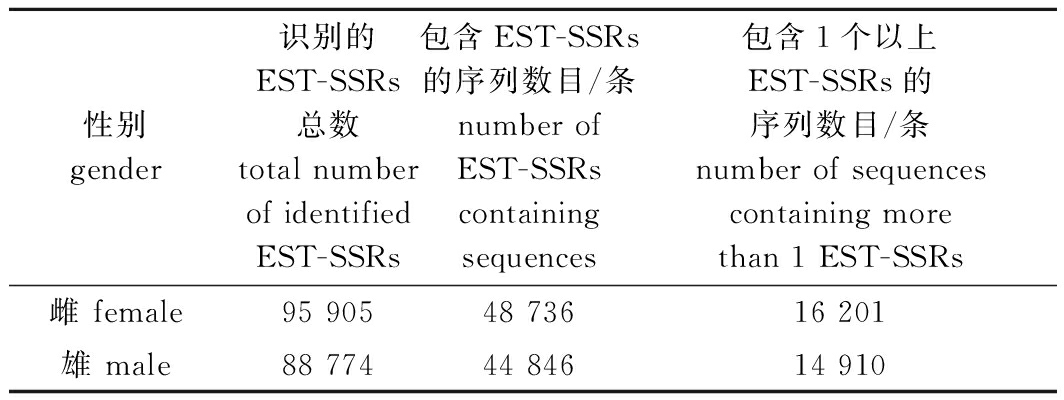
性别gender识别的EST-SSRs总数total number of identified EST-SSRs包含EST-SSRs的序列数目/条number of EST-SSRscontaining sequences包含1个以上EST-SSRs的序列数目/条number of sequences containing more than 1 EST-SSRs雌 female95 90548 73616 201雄 male88 77444 84614 910
长江刀鲚雌、雄个体脑转录组EST-SSRs具有相似的类型特征,从单核苷酸重复EST-SSRs到六核苷酸重复EST-SSRs均有分布,且具有不同程度的数量分布差异,均以二核苷酸重复EST-SSRs数量最多,单核苷酸重复EST-SSRs数量次之,三核苷酸重复EST-SSRs的数量亦较为丰富,四核苷酸重复EST-SSRs的数量相对较少,五核苷酸和六核苷酸重复EST-SSRs的数量最少(表4)。
表4 雌、雄长江刀鲚脑转录组主要EST-SSRs重复基序的数量
Tab.4 Number of main repeat motif of EST-SSRs in the brain transcriptome of female and male Coilia nasus
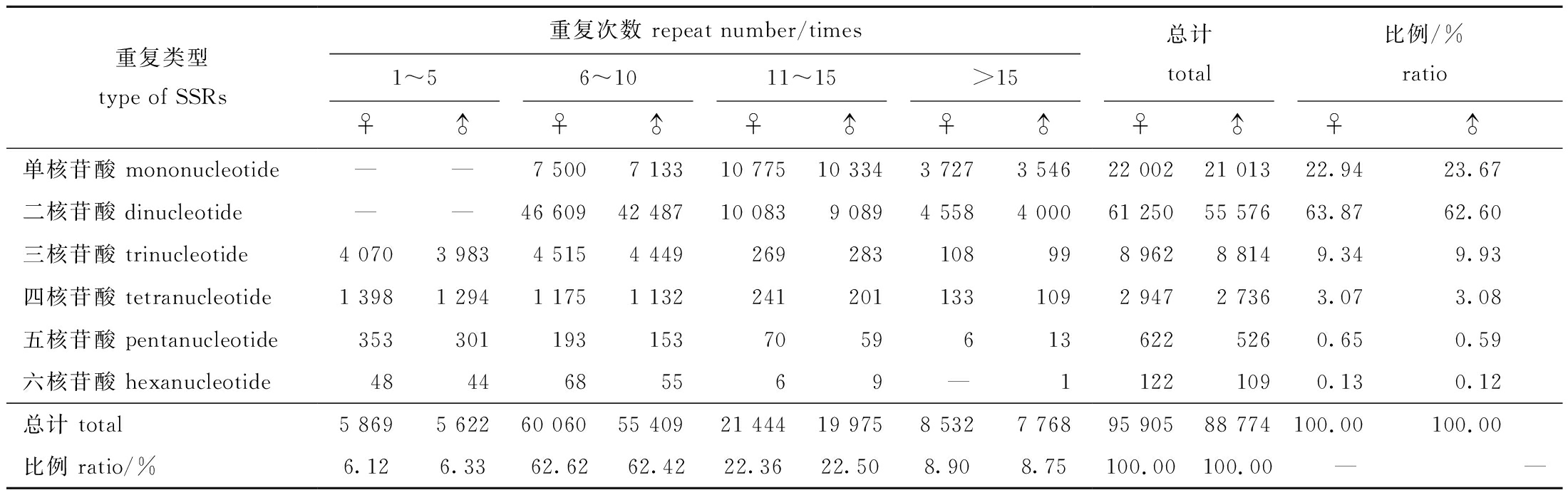
重复类型type of SSRs重复次数 repeat number/times1~56~1011~15>15♀♂♀♂♀♂♀♂总计total比例/%ratio♀♂♀♂单核苷酸 mononucleotide——7 5007 13310 77510 3343 7273 54622 00221 01322.9423.67二核苷酸 dinucleotide——46 60942 48710 0839 0894 5584 00061 25055 57663.8762.60三核苷酸 trinucleotide4 0703 9834 5154 449269283108998 9628 8149.349.93四核苷酸 tetranucleotide1 3981 2941 1751 1322412011331092 9472 7363.073.08五核苷酸 pentanucleotide35330119315370596136225260.650.59六核苷酸 hexanucleotide4844685569 —11221090.130.12总计 total5 8695 62260 06055 40921 44419 9758 5327 76895 90588 774100.00100.00比例 ratio/%6.126.3362.6262.4222.3622.508.908.75100.00100.00 — —
长江刀鲚雌、雄个体脑转录组EST-SSRs主要优势核苷酸重复基序亦有相似的数量分布特征,其中,单核苷酸重复EST-SSRs的优势重复基序均为A/T,二核苷酸均为AC/GT,三核苷酸均为AGG/CCT,四核苷酸均为ACAG/CTGT(表5)。
表5 雌、雄长江刀鲚脑转录组EST-SSRs主要重复基序的类型和数量
Tab.5 Type and number of main repeat motif of EST-SSRs in the brain transcriptome of female and male Coilia nasus
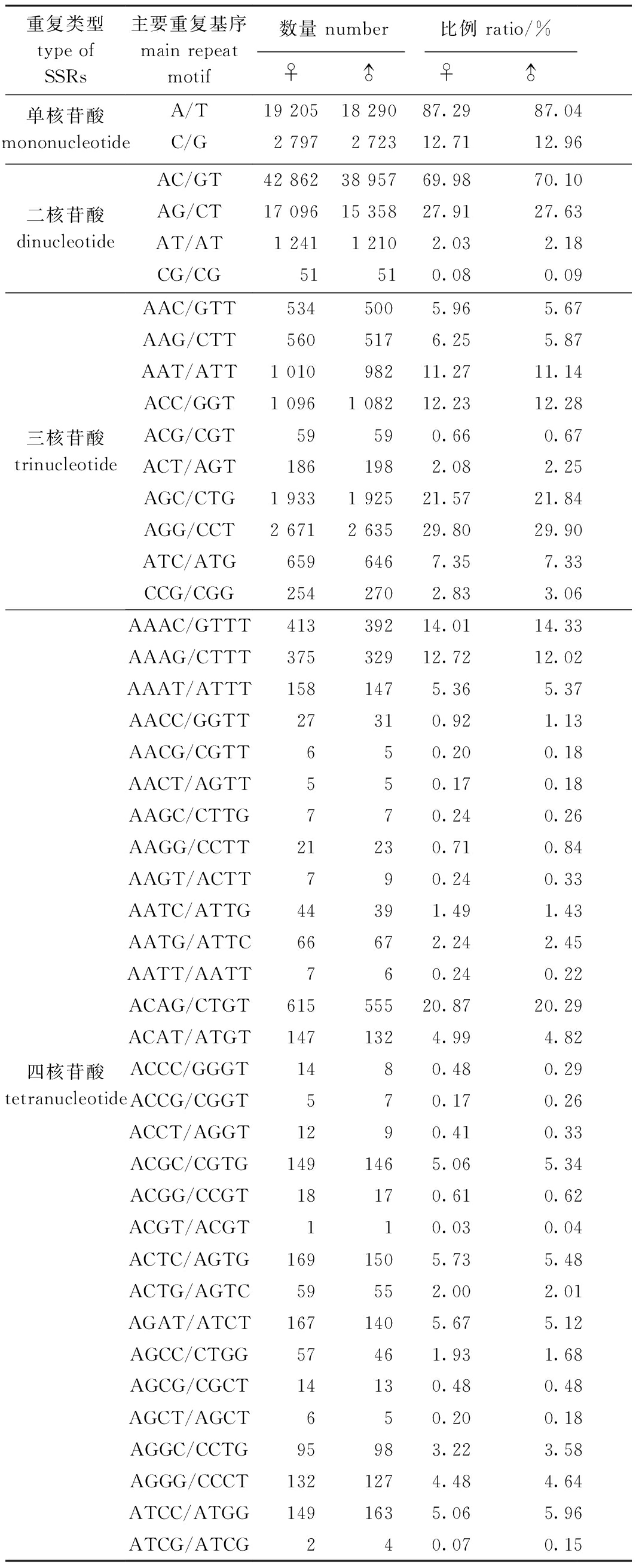
重复类型type of SSRs主要重复基序main repeat motif数量 number比例 ratio/%♀♂♀♂单核苷酸mononucleotideA/T19 20518 29087.2987.04C/G2 7972 72312.7112.96二核苷酸dinucleotideAC/GT42 86238 95769.9870.10AG/CT17 09615 35827.9127.63AT/AT1 2411 2102.032.18CG/CG51510.080.09三核苷酸trinucleotideAAC/GTT 534 5005.965.67AAG/CTT5605176.255.87AAT/ATT1 01098211.2711.14ACC/GGT1 0961 08212.2312.28ACG/CGT59590.660.67ACT/AGT1861982.082.25AGC/CTG1 9331 92521.5721.84AGG/CCT2 6712 63529.8029.90ATC/ATG6596467.357.33CCG/CGG2542702.833.06四核苷酸tetranucleotideAAAC/GTTT 413 39214.0114.33AAAG/CTTT37532912.7212.02AAAT/ATTT1581475.365.37AACC/GGTT27310.921.13AACG/CGTT650.200.18AACT/AGTT550.170.18AAGC/CTTG770.240.26AAGG/CCTT21230.710.84AAGT/ACTT790.240.33AATC/ATTG44391.491.43AATG/ATTC66672.242.45AATT/AATT760.240.22ACAG/CTGT61555520.8720.29ACAT/ATGT1471324.994.82ACCC/GGGT1480.480.29ACCG/CGGT570.170.26ACCT/AGGT1290.410.33ACGC/CGTG1491465.065.34ACGG/CCGT18170.610.62ACGT/ACGT110.030.04ACTC/AGTG1691505.735.48ACTG/AGTC59552.002.01AGAT/ATCT1671405.675.12AGCC/CTGG57461.931.68AGCG/CGCT14130.480.48AGCT/AGCT650.200.18AGGC/CCTG95983.223.58AGGG/CCCT1321274.484.64ATCC/ATGG1491635.065.96ATCG/ATCG240.070.15
2.3 长江刀鲚脑转录组多态性EST-SSRs特征分析
基于SSRMMD软件的搜索结果显示,从长江刀鲚雌、雄脑转录组EST-SSRs序列中筛选获得了5 675 条雌、雄个体共有的EST-SSRs,并从中鉴定出1 082 个多态性EST-SSRs。从图1可见,多态性EST-SSRs的数量随着碱基重复基序类型次数的增加而逐级减少,表明长江刀鲚转录组中多态性EST-SSRs的类型极为丰富,从单核苷酸到六核苷酸均有分布,且主要以单核苷酸重复和二核苷酸重复为主,这两类EST-SSRs的数量约占88.5%,多碱基重复基序的EST-SSRs相对保守,不易累积遗传变异,获得的多态性位点相对较少。
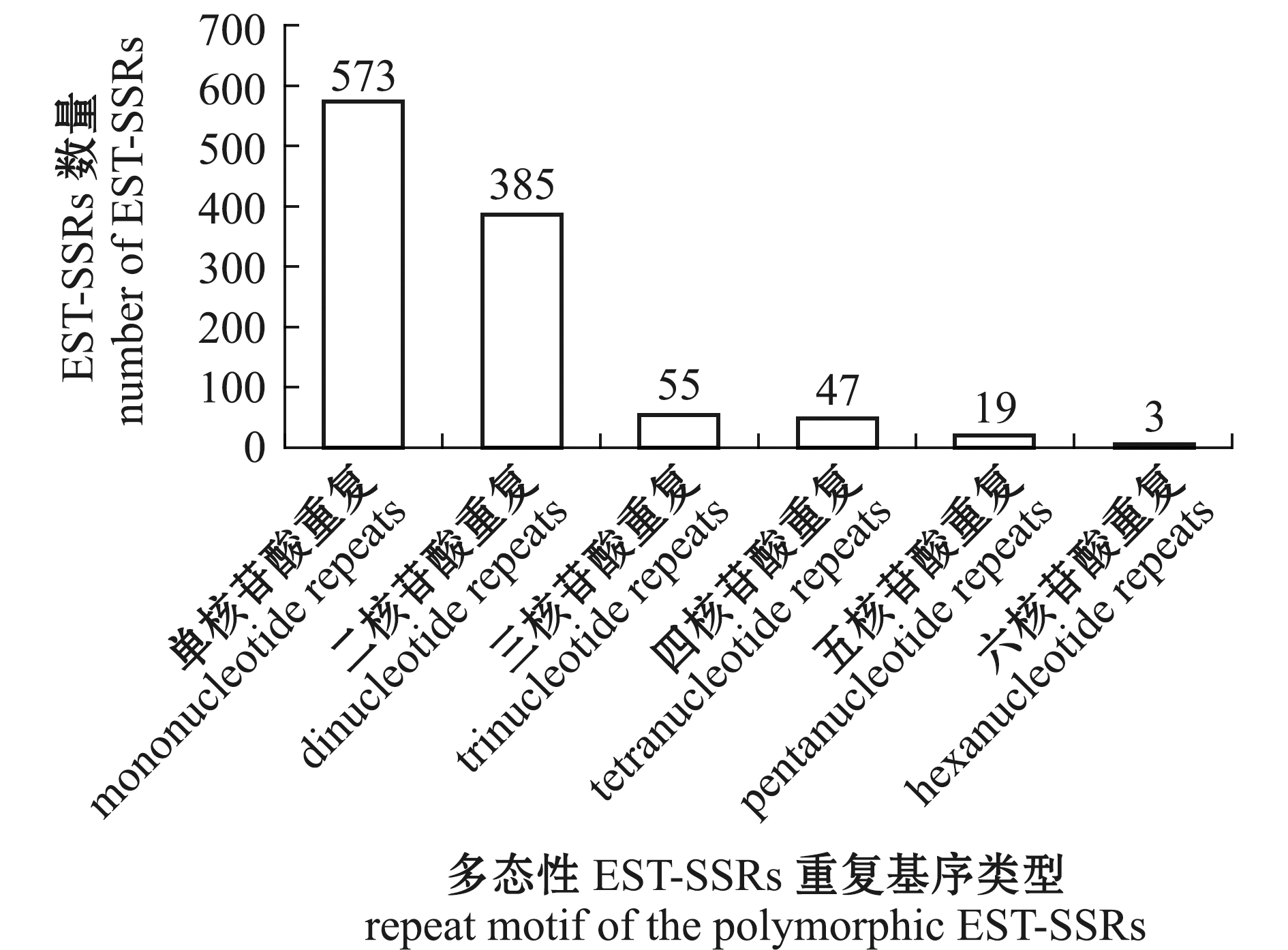
图1 长江刀鲚多态性EST-SSRs的类型和数量特征
Fig.1 Type and quantities characteristics of polymorphic EST-SSRs in Coilia nasus
2.4 多态性EST-SSRs的有效性和多态性验证
以上海市水产研究所奉贤科研基地随机选取的2018、2019和2020群体72尾养殖长江刀鲚的基因组DNA为模板,利用20对EST-SSRs引物进行PCR扩增和检测显示,除1对无扩增产物外,其余19对EST-SSRs引物扩增得到预期大小的PCR产物,有效扩增率为95%。用10%非变性聚丙烯凝胶电泳检测显示,有18对EST-SSRs引物具有较好的多态性,占有效扩增引物的94.7%(引物信息见表1),部分多态性EST-SSRs引物(EST-SSR02、EST-SSR04、EST-SSR08和EST-SSR09)在24个试验样本(2018群体)的PCR扩增电泳图谱见图2。

M—DNA marker(pBR322/MspI);1~24—24个长江刀鲚样品(2018群体)。
M—represents the DNA marker (pBR322/MspI);1-24—denote the 24 samples of Coilia nasus in 2018 population.
图2 长江刀鲚部分EST-SSRs引物的PCR产物电泳图谱
Fig.2 Electrophoretic map of PCR products with partial EST-SSRs primers of Coilia nasus
2.5 长江刀鲚养殖群体的遗传多样性分析
遗传多样性分析结果显示,长江刀鲚2018、2019、2020养殖群体的平均观测杂合度(Ho)、平均期望杂合度(He)和多态信息含量(PIC)分别为0.445 2、0.485 4和0.420 9,表明3个养殖群体的遗传多样性较为丰富,但伴随着繁育年份的增加,3个养殖群体的遗传多样性参数呈现逐年降低的趋势,特别是2020养殖群体的遗传多样性降低幅度相对较大(表6)。哈迪-温伯格平衡检测结果显示,2018、2019、2020养殖群体中分别有4、6、7个位点显著偏离哈迪-温伯格平衡(P<0.05),表明人工选择压力已经对长江刀鲚养殖群体的遗传结构产生了不同程度的影响(表7)。
表6 长江刀鲚不同养殖群体的遗传多样性参数
Tab.6 Genetic variation parameter of different cultured populations of Coilia nasus
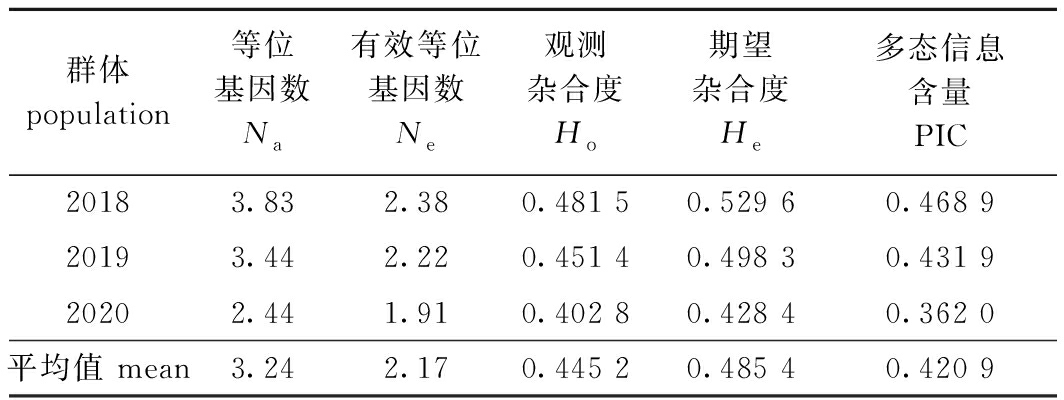
群体 population等位 基因数Na有效等位基因数Ne观测杂合度Ho期望杂合度He多态信息含量 PIC20183.832.380.481 50.529 60.468 920193.442.220.451 40.498 30.431 920202.441.910.402 80.428 40.362 0平均值 mean3.242.170.445 20.485 40.420 9
表7 长江刀鲚不同EST-SSRs的遗传多样性参数
Tab.7 Genetic variation parameter of different EST-SSRs loci in Coilia nasus

位点locus等位基因数Na有效等位基因数Ne观测杂合度Ho期望杂合度He平均多态信息含量PIC哈迪-温伯格平衡(HWE)201820192020EST-SSR012.001.830.444 40.447 30.344 50.391 70.392 80.736 1EST-SSR023.671.680.527 80.394 10.339 50.047 5*0.810 90.001 3*EST-SSR032.001.780.305 60.428 80.334 40.290 00.015 4*0.290 0EST-SSR042.001.150.111 10.127 90.118 50.744 00.105 70.831 3EST-SSR052.671.830.555 60.446 20.361 20.959 20.129 50.231 1EST-SSR063.672.210.541 70.511 00.449 30.691 50.412 90.006 3*EST-SSR073.332.410.263 90.570 90.492 20.241 30.046 6*0.000 0*EST-SSR082.331.750.138 90.403 90.325 50.011 7*0.001 1*0.000 0*EST-SSR094.332.280.500 00.526 60.467 30.946 80.815 60.958 9EST-SSR103.002.740.652 80.633 10.561 80.105 40.033 0*0.154 5EST-SSR112.001.550.194 40.305 00.239 80.917 00.223 10.006 5*EST-SSR123.672.000.416 70.436 60.397 20.003 2*0.977 90.868 3EST-SSR134.333.460.375 00.704 30.651 40.000 2*0.000 0*0.000 1*EST-SSR145.002.950.763 90.658 60.597 80.664 10.981 70.385 0EST-SSR153.332.240.486 10.497 70.437 70.482 10.912 30.422 4EST-SSR163.332.240.444 40.546 30.481 00.091 50.009 3*0.233 6EST-SSR174.672.620.541 70.545 70.510 10.186 30.586 20.402 9EST-SSR183.002.290.750 00.553 80.467 80.052 60.250 60.007 2*平均值 mean3.242.170.445 20.485 40.420 9
注:*表示位点显著偏离哈迪-温伯格平衡(P<0.05)。
Note:*,it indicates that the site significantly deviates from Hardy Weinberg Equilibrium (P<0.05).
遗传距离和遗传相似系数分析结果显示,长江刀鲚2018养殖群体与2020养殖群体的遗传距离最大(0.148 7),遗传相似性最低(0.861 8);2018养殖群体与2019养殖群体的遗传距离最小(0.104 7),遗传相似性最高(0.900 6)(表8)。基于Nei’s遗传距离构建的长江刀鲚不同养殖群体的UPGMA聚类分析显示,长江刀鲚2018养殖群体先与2019养殖群体聚为一支,然后再与2020养殖群体聚为一支(图3)。
表8 长江刀鲚不同养殖群体的遗传相似系数(对角线下)和遗传距离(对角线上)
Tab.8 Genetic similarity index (below diagonal)and genetic distance (above diagonal)of selected populations in Coilia nasus
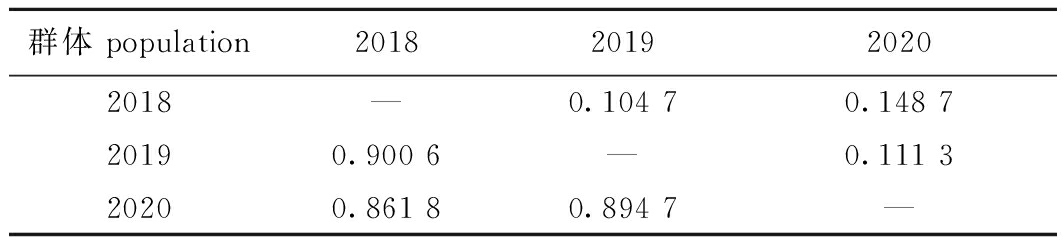
群体 population2018201920202018—0.104 70.148 720190.900 6—0.111 320200.861 80.894 7—
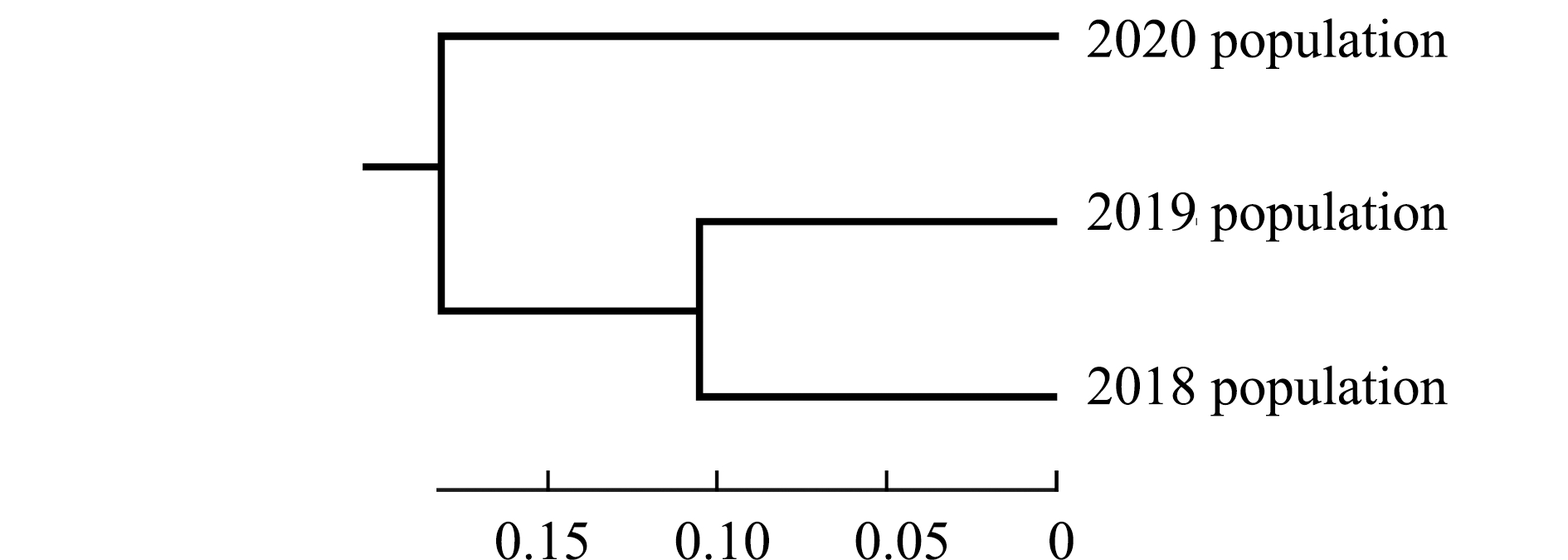
图3 不同养殖群体间的UPGMA聚类分析
Fig.3 UPGMA analysis among different cultured populations
遗传分化指数分析结果显示,长江刀鲚2018、2019、2020养殖群体间存在不同程度的遗传分化,两两群体间均处于中等程度的遗传分化(0.05<FST<0.15),且遗传分化程度呈现随繁育年份增大的趋势(表9)。AMOVA分子方差分析结果显示,长江刀鲚2018、2019、2020养殖群体的大部分遗传变异来源于不同群体内个体间或个体内的遗传差异(约89.57%),仅有10.43%的遗传变异来源于群体间的遗传差异(表10)。
表9 长江刀鲚两两群体间的遗传分化指数(FST)
Tab.9 Index of genetic differentiation between selected populations in Coilia nasus
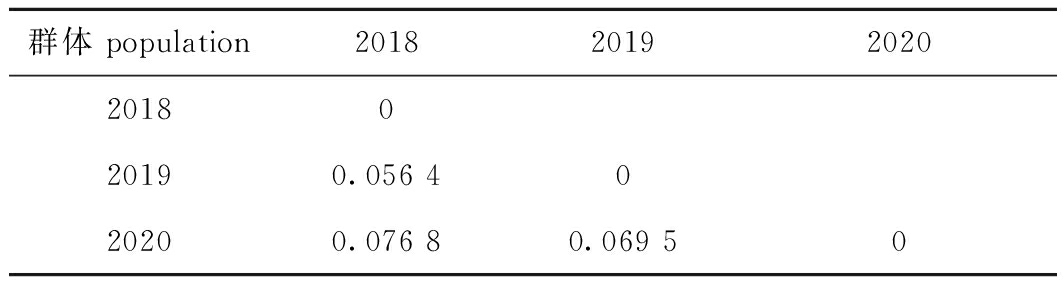
群体 population2018201920202018020190.056 4020200.076 80.069 50
表10 长江刀鲚不同养殖群体间的AMOVA分子方差分析
Tab.10 AMOVA analysis of selected populations in Coilia nasus
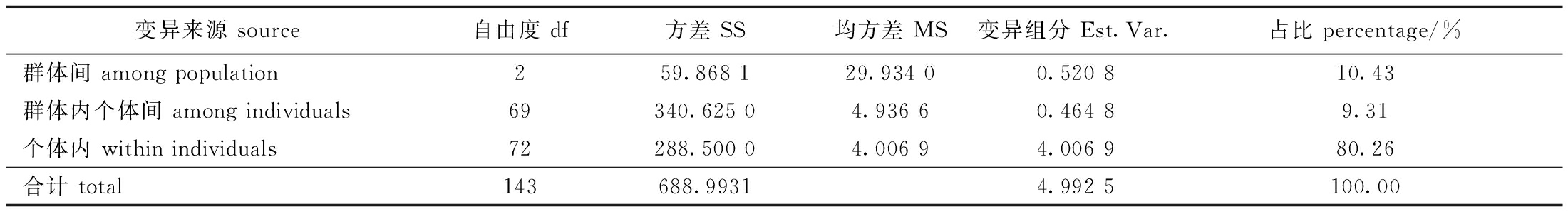
变异来源 source自由度 df方差 SS均方差 MS变异组分 Est.Var.占比 percentage/%群体间 among population259.868 129.934 00.520 810.43群体内个体间 among individuals69340.625 04.936 60.464 89.31个体内 within individuals72288.500 04.006 94.006 980.26合计 total143688.99314.992 5100.00
3 讨论
DNA分子因复制、缺失、滑移或错配及姐妹染色单体的不均等交换等原因而具有高度变异性[5],故微卫星分子标记在水产动物进化过程中被广泛应用于水产动物群体遗传结构分析、亲子鉴定、遗传图谱构建、数量性状定位和分子标记辅助育种[19-20]等相关研究。对于具有参考基因组信息的物种而言,微卫星标记的大规模开发相对方便,而对于目前还无参考基因组信息的物种,则可利用高通量转录组测序技术大规模开发EST-SSRs,以深入开展相关物种基因的等位基因变异、重要经济性状相关基因的挖掘和精细定位研究。长江刀鲚作为中国具有较高经济、营养和养殖价值的江海洄游性鱼类,其可用于分子标记的资源相对匮乏,鉴于全基因组测序的成本相对较高,因此,亟需开展长江刀鲚的转录学研究以获得大量的可用分子标记。
3.1 长江刀鲚脑转录组EST-SSRs特征分析
本研究中,基于长江刀鲚雌、雄个体的脑转录组数据,利用生物信息学方法挖掘其脑转录组中的EST-SSRs,结果在雌、雄长江刀鲚脑转录组中分别获得了95 905条和88 774条EST-SSRs,这些EST-SSRs的种类较为丰富,且6类核苷酸重复基序均有不同程度的数量分布差异,均以二核苷酸重复基序为主(63.87%和62.60%),其次是单核苷酸重复基序(22.94%和23.67%),三核苷酸重复基序亦较为丰富(9.34%和9.93%),其他类型的核苷酸重复基序相对较少。研究表明,多数水产物种EST-SSRs中的重复基序均以二核苷酸重复基序为主[21],本研究中亦获得了与此相一致的结果;而其余类型的优势重复基序则有较大的差异,这可能与不同物种转录组文库构建的组织来源、不同物种的种间特异性、位点突变频率及选择性进化机制有关[5]。本研究中,不同重复基序的碱基优势重复类型表现出不同程度的偏倚性,单核苷酸重复基序以A/T类型为主,可能是由于富含A/T的微卫星退火温度相对较低,有利于双链DNA的解链,通过DNA复制的重组和滑动机制而增加A/T重复基序的概率[5,21];二核苷酸重复基序以AC/GT为主,这与鱼类基因组微卫星中以AC/GT重复为主相符;其余重复基序的优势重复碱基类型在不同物种间差异较大,可能与不同物种的种间差异性有关。
3.2 水产动物多态性EST-SSRs的开发
近年来,陆续有学者利用高通量转录组测序技术在水产经济养殖物种中成功鉴定到多态性较好的EST-SSRs。蔡磊等[22]利用高通量转录组测序技术,从诸氏鲻虾虎鱼(Mugilogobius chulae)肝脏转录组中获得了6 225个EST-SSRs,并从76对引物中筛选获得了32对多态性较好的EST-SSRs,多态率为42.10%。龚诗琦等[23]利用高通量转录组测序技术,从黄姑鱼(Nibea albiflora)转录组中获得了12 254个EST-SSRs,并从80对引物中筛选获得了18个多态性较好的EST-SSRs,多态率为22.5%。岳华梅等[24]利用高通量转录组测序技术,从兴国红鲤(Cyprinus carpio)的垂体和性腺转录组中获得了13 652个微卫星标记,并从30对引物中筛选获得了9个多态性较好的EST-SSRs,多态率为30%。本研究中,从20对微卫星引物中筛选获得了18对多态性较好的EST-SSRs,多态率为90%,引物多态率显著高于其他鱼类的研究结果,本课题组前期从长江刀鲚肌肉和肝脏转录组中随机筛选的EST-SSRs,多态率仅为16%,而转录组中通常具有数万个EST-SSRs,如果利用传统方法将多态性的位点全部筛选出来,往往需要花费大量的人力、物力和财力。本研究中所使用的方法效果较好,特别是针对基因组信息比较匮乏的水产经济动物较为实用,不仅能够借助相关水产动物研究中已公开发表的转录组数据,且随着高通量测序成本的降低,建立转录组测序文库的成本也相对低廉,因此,能够在短时间内迅速获得多态性较好的EST-SSRs,用于相关水产经济物种的种质资源评估。
3.3 刀鲚养殖群体与野生群体遗传多样性比较
群体遗传多样性是评估水产动物种质资源现状的重要理论依据,亦是水产动物在分子水平上开展遗传改良的重要遗传基础。遗传多样性越高,适应能力越强,遗传改良的潜力越大,而观测杂合度和期望杂合度则是评估水产动物不同群体遗传多样性高低的重要参考指标[11]。基于微卫星分子标记技术,一些学者陆续对不同水域刀鲚的野生群体开展了遗传多样性研究。Ma等[11]利用12对基因组SSR引物,对30尾长江水域(武汉)野生长江刀鲚的遗传多样性评估显示,该群体的平均Ho和He分别为0.675和0.650;Chen等 [12]利用34对基因组SSR引物,对30尾鸭绿江水域(丹东)野生刀鲚的遗传多样性评估显示,该群体的平均Ho和He分别为0.53和0.77;Rong等[13]利用20对基因组SSR引物,对60尾黄河流域野生刀鲚的遗传多样性评估显示,该群体的平均Ho和He分别为0.49和0.68;Xuan等[25] 利用11对基因组SSR引物,对31尾长江江口野生刀鲚的遗传多样性评估显示,该群体的平均Ho和He分别为0.807 4和0.854 2;本课题组前期利用12对基因组SSR引物,对30尾靖江水域野生长江刀鲚的遗传多样性评估显示,该群体的平均Ho和He分别为0.516 7和0.738 1[26]。以上结果表明,中国不同水域刀鲚野生群体的遗传多样性差异较大,而刀鲚养殖群体遗传多样性的报道相对较少。本课题组前期利用12对G-SSR引物,评估了长江刀鲚选育世代(F1~F3)的遗传多样性,F1群体的平均Ho和He分别为0.480 6和0.711 2、F2群体为0.444和0.693 3,F3群体为0.400 0和0.684 4;之后又利用16对四核苷酸重复的EST-SSRs引物,评估了长江刀鲚选育世代F3的遗传多样性,获得其平均Ho和He分别为0.341 4和0.397 7。本研究中,2019养殖群体样本为前期研究中选育世代F3的繁育子代(即F4),其平均Ho和He分别为0.451 4和0.498 3,均低于其他野生刀鲚群体的遗传多样性。究其原因,一方面是由于本研究中主要是针对所开发的EST-SSRs进行多态性和有效性的初步验证,所选用的试验样本相对较少,且EST-SSRs相对于基因组SSR更加保守,多态性相对较低;另一方面是由于试验群体经过数年的人工养殖,已经受到了较大的人工选择压力,有效繁育群体的数量亦远小于野生群体,因此,养殖群体的遗传多样性相对于野生群体有所降低。
4 结论
1)利用MISA软件分别从雌、雄长江刀鲚脑转录组数据中成功鉴定到95 905个和88 774个EST-SSRs,利用SSRMMD软件成功鉴定到1 082个多态性EST-SSRs;随机选取20个EST-SSRs进行有效性和多态性验证,获得了18个多态性较好的EST-SSRs,表明本研究中所用方法能够显著提高养殖长江刀鲚多态性EST-SSRs的检出效率。
2)基于18个多态性EST-SSRs评估不同年份长江刀鲚养殖群体的种质资源现状,结果显示,随着繁育年份的增加,3个养殖群体的遗传多样性参数呈逐年降低的趋势,暗示在长江刀鲚后续生产繁育过程中,在增加有效繁育亲本数量的同时,应挑选亲缘关系和遗传分化适宜的繁育亲本,以保证繁育后代仍然具有丰富的遗传多样性。本研究结果不仅丰富了长江刀鲚分子标记的数量,而且对于长江刀鲚后续选育群体的育种效果评估、分子标记辅助育种实践及通用性长江刀鲚微卫星分子标记的开发具有重要的指导意义。
[1] 施永海,张根玉,张海明,等.刀鲚的全人工繁殖及胚胎发育[J].上海海洋大学学报,2015,24(1):36-43.
SHI Y H,ZHANG G Y,ZHANG H M,et al.Complete artificial propagation and embryonic development of Coilia nasus[J].Journal of Shanghai Ocean University,2015,24(1):36-43.(in Chinese)
[2] 袁传宓.长江中下游刀鲚资源和种群组成变动状况及其原因[J].动物学杂志,1988,23(3):12-15.
YUAN C M.Changes and its causes in resources and population composition of Coilia nasus in the middle and lower reaches of the Yangtze River[J].Chinese Journal of Zoology,1988,23(3):12-15.(in Chinese)
[3] 施永海,张根玉,张海明,等.配合饲料和活饵料喂养刀鲚肌肉营养品质分析与比较[J].动物营养学报,2014,26(2):427-436.
SHI Y H,ZHANG G Y,ZHANG H M,et al.Analysis and comparison of nutritional quality of muscle from Coilia nasus fed formulated feed and live feed[J].Chinese Journal of Animal Nutrition,2014,26(2):427-436.(in Chinese)
[4] 魏广莲,徐钢春,顾若波,等.刀鲚的生物学及人工养殖研究进展[J].长江大学学报(自然科学版),2012,9(7): 31-35,41.
WEI G L,XU G C,GU R B,et al.Biology and artificial breeding progress in Coilia nasus[J].Journal of Yangtze University(Natural Science Edition),2012,9(7):31-35,41.(in Chinese)
[5] 于爱清,施永海,徐嘉波,等.长江刀鲚选育群体转录组EST-SSR的分布特征分析[J].渔业科学进展,2019,40(5):101-109.
YU A Q,SHI Y H,XU J B,et al.Characteristic analysis of microsatellites in selected Coilia ectenes using a transcriptome dataset[J].Progress in Fishery Sciences,2019,40(5):101-109.(in Chinese)
[6] 张敏莹,方弟安,周彦锋,等.基于微卫星标记的钱塘江中下游三角鲂亲本群体和自然捕捞群体遗传多样性及遗传结构[J].大连海洋大学学报,2022,37(5):775-783.
ZHANG M Y,FANG D A,ZHOU Y F,et al.Preliminary study on genetic diversity and genetic structure of four parent populations and one fishing population of triangular bream Megalobrama terminalis in the middle and lower reaches of the Qiantang River by microsatellite marker[J].Journal of Dalian Ocean University,2022,37(5):775-783.(in Chinese)
[7] 王鹤,李战军,王亮,等.金乌贼微卫星标记开发及两个野生群体遗传多样性比较[J].大连海洋大学学报,2019,34(4):482-491.
WANG H,LI Z J,WANG L,et al.Isolation of microsatellite markers and genetic diversity of wild populations in golden cuttlefish Sepia esculenta[J].Journal of Dalian Ocean University,2019,34(4):482-491.(in Chinese)
[8] CHENG Q Q,ZHANG Q Y,MA C Y,et al.Genetic structure and differentiation of four lake populations of Coilia ectenes (Clupeiformes:Engraulidae) based on mtDNA control region sequences[J].Biochemical Systematics and Ecology,2011,39(4/5/6):544-552.
[9] 马春艳,刘敏,马凌波,等.长江口刀鲚遗传多样性的随机扩增多态DNA(RAPD)分析[J].海洋水产研究,2004,25(5):19-24.
MA C Y,LIU M,MA L B,et al.Genetic diversity in Coilia ectenes by RAPD analysis[J].Marine Fisheries Research,2004,25(5):19-24.(in Chinese)
[10] 葛家春,曹廷,陈婵娟,等.利用扩增片段长度多态性技术分析长江刀鲚的遗传多样性[J].南京大学学报(自然科学版),2008,44(3):332-338.
GE J C,CAO T,CHEN C J,et al.Analysis of genetic diversity based on amplified fragment length polymorphism fingerprint of Coilia nasus from Yangtze River[J].Journal of Nanjing University (Natural Sciences Edition),2008,44(3): 332-338.(in Chinese)
[11] MA C Y,CHENG Q Q,ZHANG Q Y.Development of 12 polymorphic microsatellite markers in Coilia ectenes Jordan and Seale,1905 (Clupeiformes:Engraulidae) and cross-species amplification in Coilia mystus Linnaeus,1758[J].Environmental Biology of Fishes,2011,91(2):243-249.
[12] CHEN F,ZENG L Y,CHENG Q Q.Development of thirty-four novel polymorphic microsatellite markers in Coilia ectenes (Clupeiformes:Engraulidae) and cross-species amplification in two closely related taxa[J].Journal of Genetics,2012,91(1):e37-e43.
[13] RONG X J,XU Y J,WANG Q Y,et al.Isolation and characterization of polymorphic microsatellite markers from Coilia ectenes[J].Genetics and Molecular Research:GMR,2013,12(4):6011-6017.
[14] 李东明,杨爱国,吴彪,等.栉江珧(Atrina pectinata)EST-SSR标记的开发与应用[J].渔业科学进展,2017,38(2): 137-142.
LI D M,YANG A G,WU B,et al.Development and application of the EST-SSR markers in Atrina pectinata[J]. Progress in Fishery Sciences,2017,38(2):137-142.(in Chinese)
[15] THIEL T,MICHALEK W,VARSHNEY R,et al.Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.)[J].Theoretical and Applied Genetics, 2003,106(3):411-422.
[16] XIA E H,YAO Q Y,ZHANG H B,et al.CandiSSR:an efficient pipeline used for identifying candidate polymorphic SSRs based on multiple assembled sequences[J].Frontiers in Plant Science,2016,6:1171.
[17] GOU X J,SHI H R,YU S F,et al.SSRMMD:a rapid and accurate algorithm for mining SSR feature loci and candidate polymorphic SSRs based on assembled sequences[J].Frontiers in Genetics,2020,11:706.
[18] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[19] 张丹,傅建军,张利德,等.鳙基于10个微卫星标记的亲子鉴定分析[J].基因组学与应用生物学,2019,38(7): 2949-2957.
ZHANG D,FU J J,ZHANG L D,et al.The parentage analysis of bighead carp (Hypophthalmichthys nobilis) based on ten microsatellite markers[J].Genomics and Applied Biology,2019,38(7):2949-2957.(in Chinese)
[20] 郭香,李琪,孔令锋,等.基于微卫星标记整合长牡蛎遗传图谱[J].水产学报,2013,37(6):823-829.
GUO X,LI Q,KONG L F,et al.Microsatellite-based consensus map of the Pacific oyster(Crassostrea gigas)[J]. Journal of Fisheries of China,2013,37(6):823-829.(in Chinese)
[21] 倪守胜,杨钰,柳淑芳,等.基于高通量测序的虾夷扇贝基因组微卫星特征分析[J].渔业科学进展,2018,39(1): 107-113.
NI S S,YANG Y,LIU S F,et al.Microsatellite analysis of Patinopecten yessoensis using next-generation sequencing method[J].Progress in Fishery Sciences,2018,39(1):107-113.(in Chinese)
[22] 蔡磊,余露军,陈小曲,等.诸氏鲻虾虎鱼转录组序列中微卫星标记的初步筛选及特征分析[J].生物技术通报, 2015,31(9):146-151.
CAI L,YU L J,CHEN X Q,et al.A preliminary screening and characteristic analysis of microsatellite markers from transcriptome sequences in Mugilogobius chulae[J].Biotechnology Bulletin,2015,31(9):146-151.(in Chinese)
[23] 龚诗琦,王志勇,肖世俊,等.黄姑鱼转录组SSR的开发与验证[J].集美大学学报(自然科学版),2016,21(4): 241-246.
GONG S Q,WANG Z Y,XIAO S J,et al.Development and verification of SSR based on transcriptome of yellow drum, Nibea albiflora[J].Journal of Jimei University (Natural Science Edition),2016,21(4):241-246.(in Chinese)
[24] 岳华梅,翟晴,宋明月,等.基于转录组测序的兴国红鲤微卫星标记筛选[J].淡水渔业,2016,46(1):24-28.
YUE H M,ZHAI Q,SONG M Y,et al.Development of microsatellite markers in Cyprinus carpio var. singuonensis using next-generation sequencing[J].Freshwater Fisheries,2016,46(1):24-28.(in Chinese)
[25] XUAN Z Y,JIANG T,LIU H B,et al.Mitochondrial DNA and microsatellite analyses reveal strong genetic differentiation between two types of estuarine tapertail anchovies (Coilia) in Yangtze River Basin,China[J]. Hydrobiologia,2021,848(6):1409-1431.
[26] 于爱清,施永海,邓平平.长江刀鲚选育和野生群体遗传多样性的微卫星分析[J].水产科技情报,2019, 46(3):121-125.
YU A Q,SHI Y H,DENG P P.Genetic diversity analysis of cultured and wild populations of Coilia ectenes by microsatellite markers[J].Fisheries Science & Technology Information,2019,46(3):121-125.(in Chinese)