鰤鱼诺卡氏菌(Nocardia seriolae),隶属于厚壁菌门(Firmicutes)放线菌纲(Actinobacteria)放线菌目(Actinobacterales)棒杆菌亚目(Corynebacterineae)诺卡氏菌科(Nocardiaceae)诺卡氏菌属(Nocardia)。鰤鱼诺卡氏菌是鱼类诺卡氏菌病的主要致病菌[1],该菌可感染40余种鱼类,既能感染海水养殖鱼种,如卵形鲳鲹(Trachinotus ovatus)[2]、大黄鱼(Larimichthys crocea)[3]、海鲈(Lateolabrax japonicus)[4]、石斑鱼(Epinephelus spp.)[5]和鳗(Anguilla japonica)[6],也能感染淡水鱼种,如乌鳢(Channa argus)[7]和大口黑鲈(Micropterus salmoides)[8]。鱼体感染鰤鱼诺卡氏菌后的发病症状为肝脏、脾脏和肾脏组织形成大量白色结节,体表出血性腐烂,游动迟缓[9]。鰤鱼诺卡氏菌自然感染发病率可达到30%~60%,患病鱼死亡率高,因此,鰤鱼诺卡氏菌病的暴发会对水产养殖业造成较大的经济损失[10]。
鰤鱼诺卡氏菌感染早期为隐性感染,通常在中晚期才发现明显症状,因此,需要对鰤鱼诺卡氏菌进行早期检测。但从患病鱼体分离出鰤鱼诺卡氏菌的分离率较低(4%~55%)[5,11],通过传统的细菌分离和生化鉴定准确率较差。分子检测方法具有灵敏度高、时间短、操作简单等优点,基于分子手段检测鰤鱼诺卡氏菌的方法已有报道,如聚合酶链反应(polymerase chain reaction,PCR)[12]、环介导等温扩增技术(loop-mediated isothermal amplification,LAMP)[13]和实时荧光定量PCR反应(real-time quantitative PCR,qPCR)[14]。用于鰤鱼诺卡氏菌分子检测方法的片段多采用16S~23S rRNA内转录间隔区(internal transcribed spacers,ITS)和16S rRNA。重组酶介导扩增(recombinase-aided amplification,RAA)技术是一种在恒温下能够实现核酸快速扩增的分子检测方法[15]。RAA检测方法不同于传统的分离检测手段,其在较低温度条件下就可进行体内核酸扩增[16],对试验设备要求低,可满足基层单位对病原菌、病毒的快速检测。本研究中,选取了鰤鱼诺卡氏菌的种内保守、种间变异的特异性基因组片段设计引物,采用RAA技术检测鰤鱼诺卡氏菌,并对各项检测内容进行验证,以期在37 ℃恒温条件下建立一种快速检测鰤鱼诺卡氏菌的方法。
1 材料与方法
1.1 材料
鰤鱼诺卡氏菌的11个不同菌株及其来源见表1。其中,鰤鱼诺卡氏菌ZJ0503、FS201912、HB202008由本实验室从不同患病鱼中分离获得并保存,而鰤鱼诺卡氏菌NS201300047、NS201300048、NS201300049、NS201300050、NS201300051、NK- 201610020、NS201400048和NS201500141由中国水产科学研究院珠江水产研究所水产病害与免疫研究室馈赠。
表1 鰤鱼诺卡氏菌不同菌株及其来源
Tab.1 Different strains and sources of Nocardia seriolae
菌株号 strain No.分离年份 isolated year分离地 isolated location动物来源 source from fishZJ05032013湛江 Zhanjiang卵形鲳鲹 (Trachinotus ovatus)FS2019122019佛山 Foshan大口黑鲈 (Micropterus salmoides)HB2020082020淮北 Huaibei大口黑鲈 (Micropterus salmoides)NS2013000472013珠海 Zhuhai七星鲈 (Lateolabrax japonicus)NS2013000482013南海 Nanhai宝石鲈 (Scortum barcoo)NS2013000492013广州 Guangzhou 革胡子鲶 (Clarias lazera)NS2013000502013重庆 Chongqing杂交鳢 (Channa maculata♀×C.argus♂)NS2013000512013广州 Guangzhou大口黑鲈 (Micropterus salmoides)NK2016100202013佛山 Foshan杂交鳢 (Channa maculata♀×C.argus♂)NS2014000482014中山 Zhongshan杂交鳢(Channa maculata♀×C.argus♂)NS2015001412015珠海 Zhuhai金鲳 (Trachinotus blochii)
7株鰤鱼诺卡氏菌近缘菌,即杀鲑诺卡氏菌(Nocardia salmonicida) 4.5220、河流诺卡氏菌(N.fluminea) 4.5153、褐色诺卡氏菌(N.fusca) 4.1160、星状诺卡氏菌(N.asteroids) 4.1165、黄粉色诺卡氏菌(N.flavorosea) 4.1175、绛红色诺卡氏菌(N.purpurea) 4.1182和紫褐诺卡氏菌(N.violaceofusca) 4.1188,从中国普通微生物菌种保藏管理中心(CGMCC)购买并由本实验室保种;9种水产养殖过程中常见细菌性疾病的病原菌,即无乳链球菌(Streptococcus agalactiae) ZQ0910、溶藻弧菌(Vibrio alginolyticus) HY9901、迟缓爱德华氏菌(Edwardsiella tarda) ATCC15947、嗜水气单胞菌(Aeromonas hydrophila) SZ20180916、舒伯特气单胞菌(Aeromonas schubertii) GIM1.892、美人鱼发光杆菌杀鱼亚种(Photobacterium damselae subsp. piscicida) 1.1032、豚鼠气单胞菌(Aeromonas caviae) BNCC139095、荧光假单胞菌(Pseudomonas fluorescens) ATCC13525和副溶血弧菌(Vibrio parahemolyticus) ATCC17802由本实验室保种。
主要试剂:海洋动物组织基因DNA提取试剂盒DP324和细菌基因组DNA核酸提取试剂盒AG21007购自天根生化科技有限公司;RAA基础型核酸扩增试剂盒(S001ZC)购自杭州众测生物科技有限公司;DNA凝胶回收纯化试剂盒Thermo Scientific Gene JET购自Thermo公司;DH5α感受态购自武汉转导生物实验室。引物由生工生物工程(上海)股份有限公司合成。
1.2 方法
1.2.1 菌株培养与基因组DNA提取 将11株鰤鱼诺卡氏菌,以及杀鲑诺卡氏菌、河流诺卡氏菌、褐色诺卡氏菌、星状诺卡氏菌、黄粉色诺卡氏菌、绛红色诺卡氏菌和紫褐诺卡氏菌活化后,用脑心浸液肉汤(BHI)培养基28 ℃下培养7 d;将美人鱼发光杆菌杀鱼亚种活化,用胰蛋白胨大豆肉汤(TSB)培养基28 ℃下培养24 h;将舒伯特气单胞菌、嗜水气单胞菌、豚鼠气单胞菌和荧光假单胞菌活化后,用LB液体培养基30 ℃下培养20 h;迟缓爱德华氏菌、无乳链球菌、副溶血弧菌和溶藻弧菌活化后,用 LB 液体培养基37 ℃下培养20 h。对以上细菌在平板上挑取单菌落至液体培养基培养,取培养至对数生长后期的细菌培养物,离心收集菌体后,用细菌基因组DNA 提取试剂盒提取各菌株的基因组DNA,于-20 ℃下保存备用。
1.2.2 特异性基因片段的筛选和引物设计 通过分析鰤鱼诺卡氏菌不同菌株的全基因组数据,筛选种内保守、种间变异的特异性片段,选择ORF4001(GenBank登录号:NZ_JNCT01000001.1)的特异性片段为靶序列,设计出鰤鱼诺卡氏菌RAA检测的特异性引物RAA-F/RAA-R(表2),并对特异性序列片段进行生物信息学分析。
表2 RAA和PCR检测使用的引物
Tab.2 Primers used for RAA and PCR assay

引物 primer引物序列(5'-3') primer sequence(5'-3')长度/bp size来源 sourceRAAF:GGTTACGCCTCGCCCTACCCGT-CCATCAR:CCCGGTGTCCCAGTTGACCAT- GATCTCCC250本研究设计659F:ATCTGGAGCCGATTCGGACTGR:ACGCCACTGATCACTCGCATTT566陈国权等[17]
1.2.3 特异性检测 以提取的鰤鱼诺卡氏菌ZJ0503基因组DNA为模板,其他菌株的DNA为对照,RAA-F/RAA-R为特异性引物,使用RAA基础型核酸扩增试剂盒构建RAA扩增体系(共50 μL):向含有冻干酶粉(S001ZC)的0.2 mL TwistAmp反应管中分别加入A buffer 41.5 μL,浓度为10 μmol/L的RAA-F和RAA-R引物各2 μL,DNA模板2 μL,浓度为280 mmol/L的醋酸镁溶液2.5 μL,利用振荡器将反应液混合均匀。将以上反应体系于恒温37 ℃下反应30 min。向扩增产物中加入50 μL由苯酚和氯仿按体积比1∶1混合而成的溶液,充分混匀后,以12 000 r/min离心2 min,取5 μL上清液,用15 g/L琼脂糖凝胶电泳检测产物中是否含有250 bp的片段。扩增产物送生工生物工程(上海)股份有限公司进行测序。
1.2.4 灵敏度检测 利用特异性引物 RAA-F/RAA-R扩增鰤鱼诺卡氏菌ZJ0503的全基因组DNA,将PCR产物进行胶回收,连接T载体后转化至DH5α感受态细胞中,挑取单克隆送至生工生物工程(上海)股份有限公司进行测序,将测序结果正确的菌液扩大培养,利用OMEGA质粒提取试剂盒提取重组质粒。利用Nanodrop 2000超微量分光光度计检测含有靶片段的重组质粒浓度,然后用ddH2O将重组质粒进行梯度稀释后进行RAA恒温检测,反应体系同“1.2.3节”。
1.2.5 患病杂交鳢与健康杂交鳢的检测 健康杂交鳢(Channa maculate♀×C.argus♂)20尾购自广州鸿顺水产养殖基地,将杂交鳢在实验室暂养7 d,随机取10尾用鰤鱼诺卡氏菌悬液进行腹腔注射,注射量为100 μL,菌液浓度为1×106 CFU/mL。在攻毒后连续观察10 d,分别取3尾有明显发病症状的杂交鳢和3尾健康杂交鳢的鳃、肝脏、脾脏、体肾和肌肉组织,使用海洋动物组织基因组DNA提取试剂盒提取各组织的基因组DNA,并分别用RAA和PCR方法扩增。RAA扩增的反应体系和条件同“1.2.3节”。PCR扩增反应体系(共20 μL):Taq DNA Polymerase Mix(TaKaRa)10 μL,引物659-R和659-F各1 μL,ddH2O 7 μL,DNA模板1 μL。PCR反应扩增条件:95 ℃下预变性5 min;95 ℃下循环变性30 s,64 ℃下退火复性30 s,72 ℃下延伸1 min,共进行30个循环;最后在72 ℃下再延伸10 min。将得到的扩增产物用15 g/L琼脂糖凝胶电泳检测并记录结果。
1.2.6 检测应用效果 使用分离自不同鱼种、时间和地点的11株鰤鱼诺卡氏菌(表1)提取基因组DNA模板,按照“1.2.3节”反应体系和条件进行RAA检测,用15 g/L琼脂糖凝胶电泳检测产物,评价RAA检测鰤鱼诺卡氏菌的覆盖度和可靠性。
2 结果与分析
2.1 特异性基因片段
通过分析鰤鱼诺卡氏菌的不同菌株全基因组数据,筛选出种内保守、种间变异的特异性基因片段 ORF4001,长度为825 bp,编码274个氨基酸,其核苷酸序列如图1所示。

图1 鰤鱼诺卡氏菌特异性基因片段的核苷酸序列
Fig.1 Nucleotide sequence of specific gene fragment from Nocardia seriolae
2.2 RAA特异性检测结果
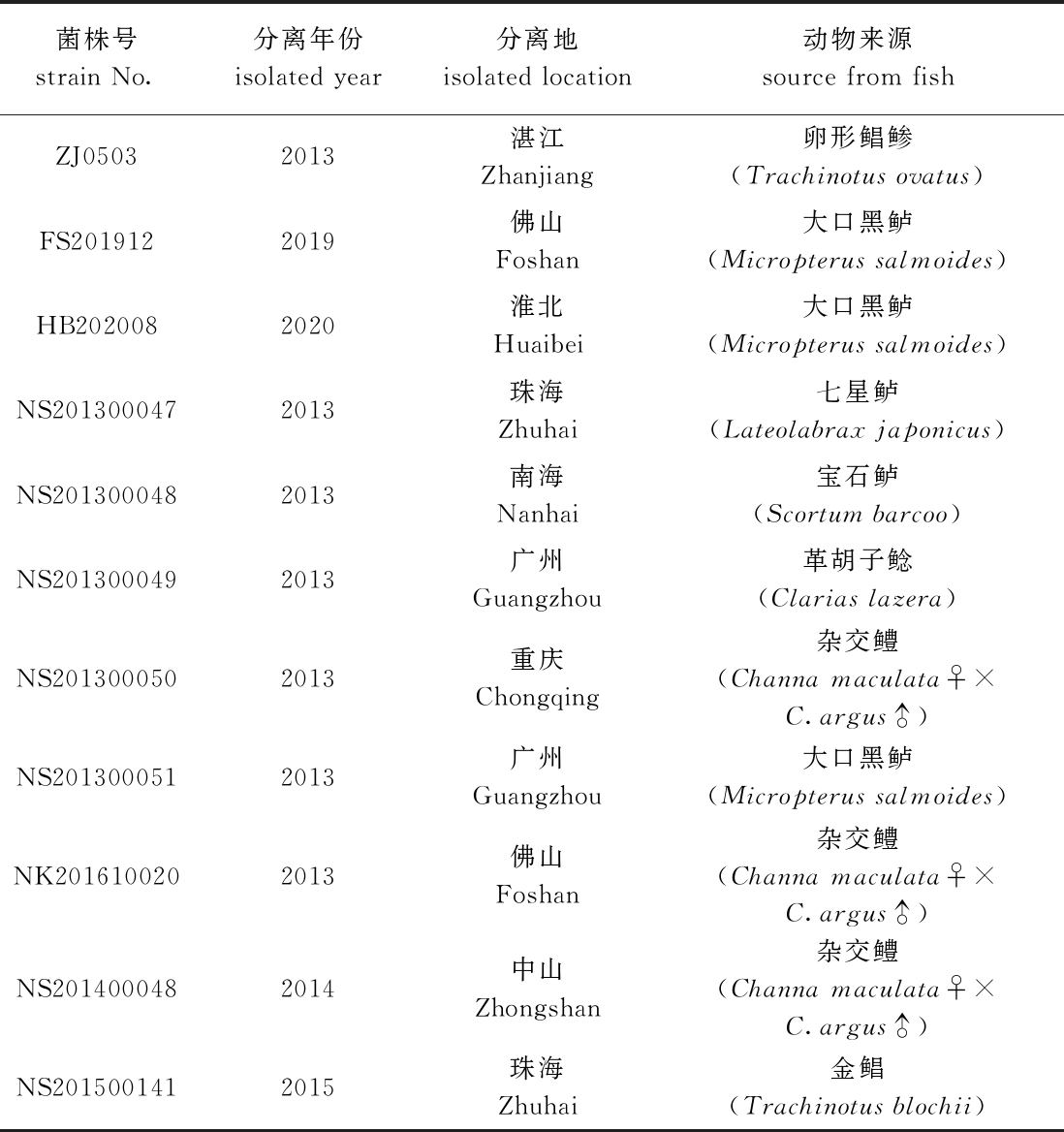
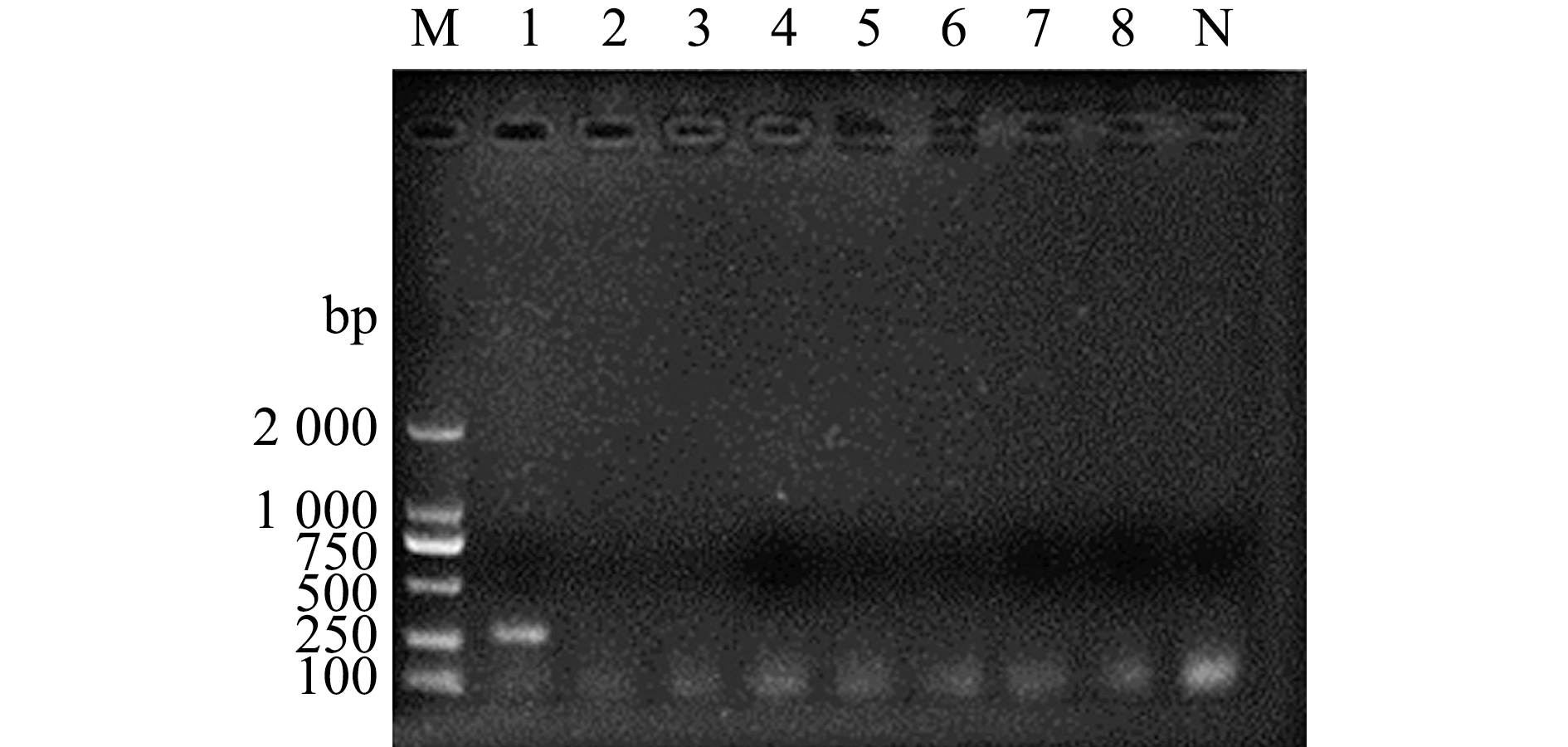
对鰤鱼诺卡氏菌ZJ0503和7个鰤鱼诺卡氏菌ZJ0503近缘菌的基因组DNA进行RAA检测,结果显示,鰤鱼诺卡氏菌ZJ0503的RAA产物有一条约为250 bp的条带且呈阳性,而其他7株近缘诺卡氏菌和阴性对照的RAA产物检测结果均呈阴性(图2)。对鰤鱼诺卡氏菌的扩增产物进行测序,结果表明,扩增片段序列与设计引物的相应片段序列完全一致。对9种水产养殖过程中常见病原菌基因组DNA进行RAA检测,结果显示,仅鰤鱼诺卡氏菌ZJ0503基因组DNA呈阳性,其他病原菌均呈阴性(图3)。说明本研究中所设计的RAA检测引物特异性良好,可以特异性地检测鰤鱼诺卡氏菌。
M—DL2000 DNA分子标准; 1—鰤鱼诺卡氏菌ZJ0503;2—杀鲑诺卡氏菌;3—褐色诺卡氏菌;4—星状诺卡氏菌;5—黄粉色诺卡氏菌;6—绛红色诺卡氏菌;7—紫褐诺卡氏菌;8—河流诺卡氏菌;N—阴性对照。M—DL2000 marker; 1—N.seriolae strain ZJ0503; 2—N.salmonicida; 3—N.fusca; 4—N.asteroids; 5—N.flavorosea; 6—N.purpurea; 7— N.violaceofusca; 8—N.fluminea; N—negative control.
图2 鰤鱼诺卡氏菌及近缘菌的RAA检测结果
Fig.2 RAA assay of Nocardia seriolae and relative Nocardia spp.
M—DL2000 DNA分子标准;1—鰤鱼诺卡氏菌ZJ0503;2—无乳链球菌;3—溶藻弧菌;4—迟缓爱德华氏菌;5—嗜水气单胞菌;6—舒伯特气单胞菌;7—美人鱼发光杆菌杀鱼亚种;8—豚鼠气单胞菌;9—荧光假单胞菌;10—副溶血弧菌;N—阴性对照。M—DL2000 marker; 1—N.seriolae strain ZJ0503; 2—S.agalactiae;3—V.alginolyticus; 4—E.tarda; 5—A.hydrophila; 6—A.schubertii; 7—P.damselae subsp.Piscicida; 8—A.caviae; 9—P.fluorescens;10—V.parahemolyticus;N—negative control.
图3 常见水产病原菌的RAA检测结果
Fig.3 RAA detection results of common pathogenic bacteria in aquaculture
2.3 灵敏度检测结果
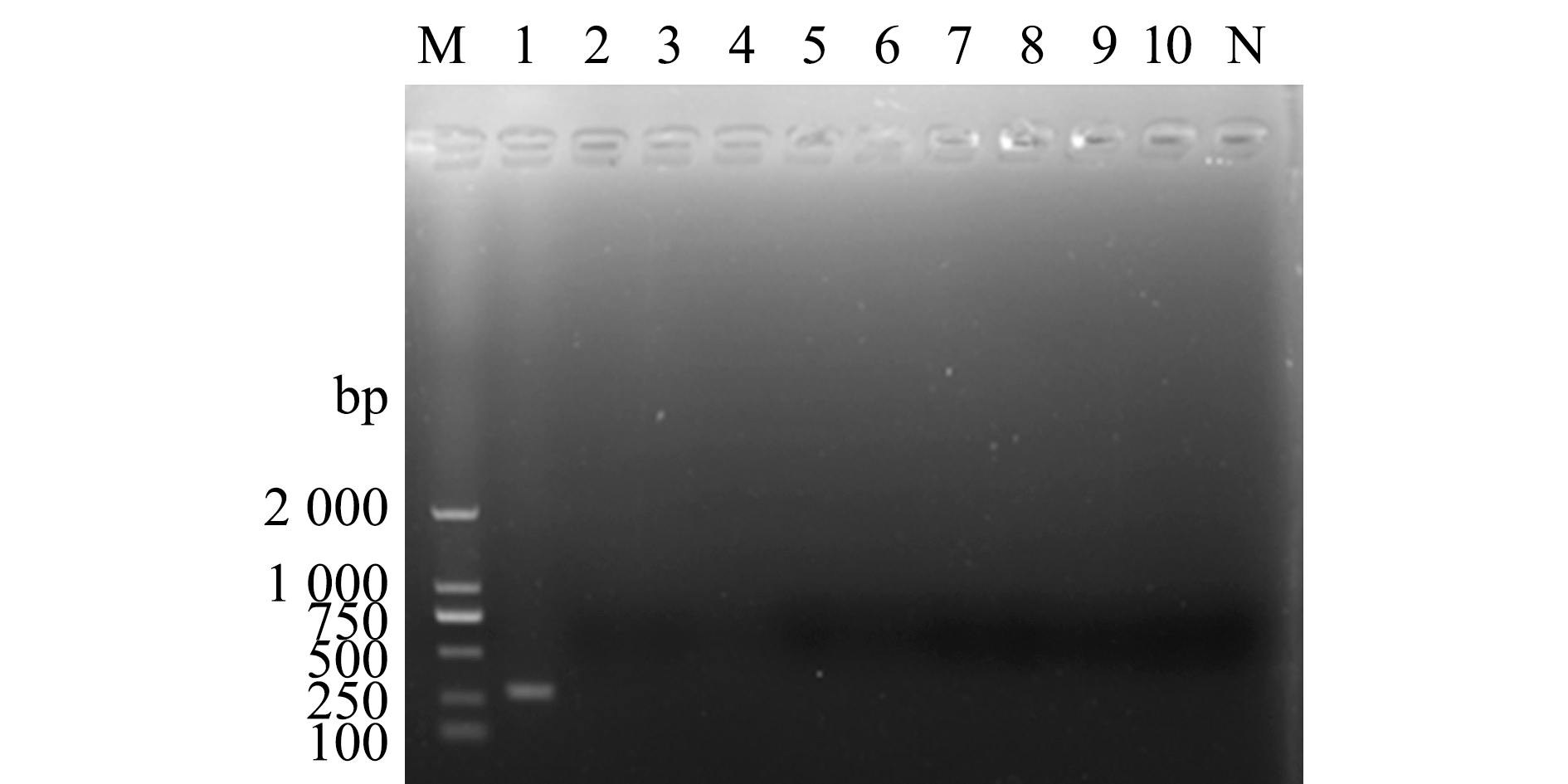
使用RAA特异性引物扩增不同浓度的重组质粒结果如图4所示,重组质粒浓度为100 ng/μL、10 ng/μL、1 ng/μL和100 pg/μL时均可扩增出一致的约为250 bp的靶片段条带,阴性对照组和10 pg/μL重组质粒未能扩增出目的条带。由此可知,本试验中所建立的鰤鱼诺卡氏菌RAA检测方法的灵敏度可以达到100 pg/μL。
M—DL2000 DNA分子标准;N—阴性对照;1—100 ng/μL;2—10 ng/μL;3—1 ng/μL;4—100 pg/μL;5—10 pg/μL。M—DL2000 marker; N—negative control; 1—100 ng/μL; 2—10 ng/μL; 3—1 ng/μL; 4—100 pg/μL; 5—10 pg/μL.
图4 鰤鱼诺卡氏菌RAA检测的灵敏度
Fig.4 Sensitivity of the RAA assay to detect Nocardia seriolae
2.4 患病杂交鳢与健康杂交鳢的检测结果
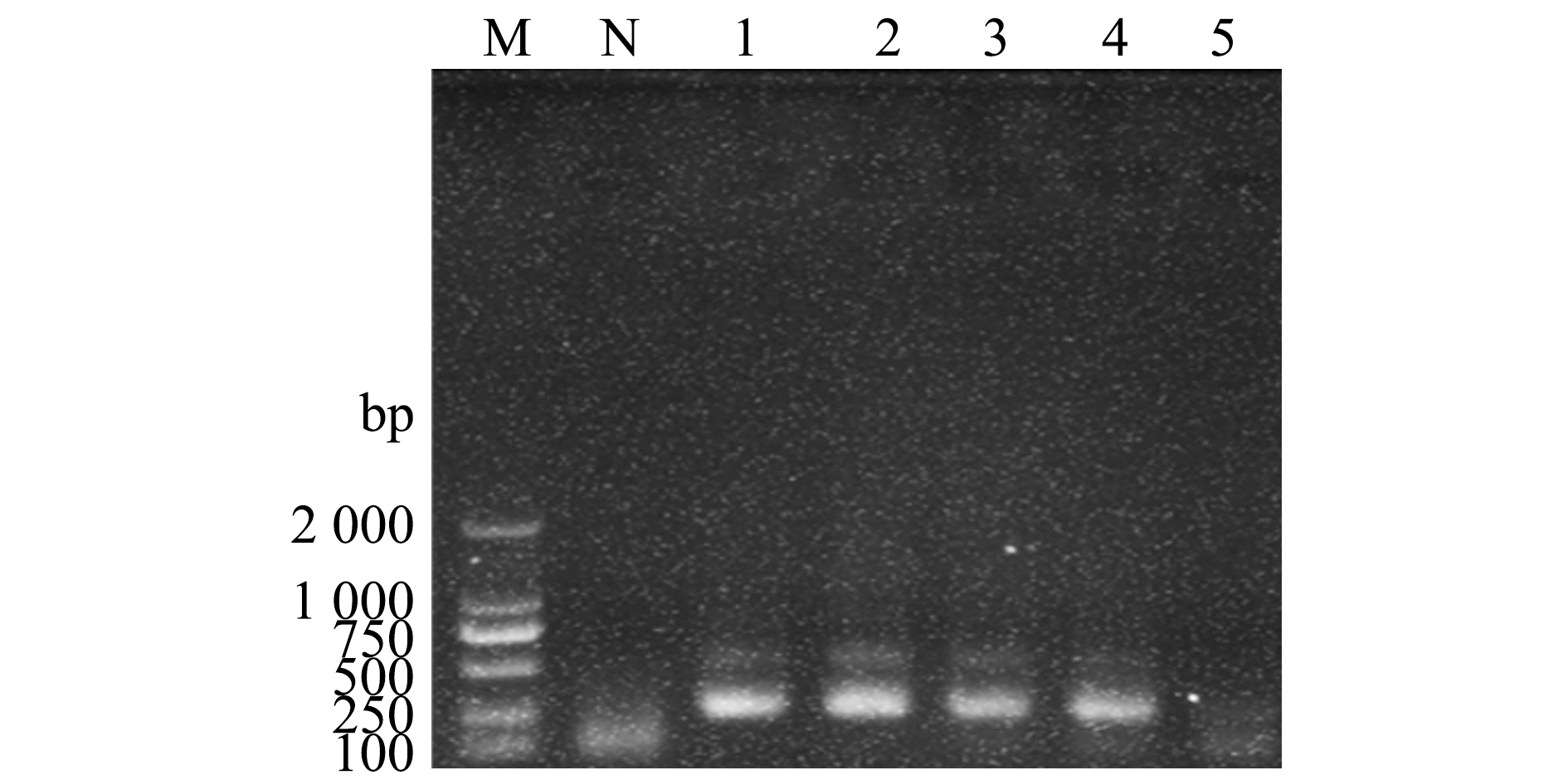
通过腹腔注射鰤鱼诺卡氏菌液的杂交鳢,10 d后可观察到杂交鳢反应愚钝、浮于水表及食欲下降;经解剖后可见,内脏器官有明显白色结节,肝脏有糜烂现象(图5)。取发病鱼的肝脏、体肾、脾脏、鳃和肌肉组织的基因组DNA,并分别使用RAA特异性引物和PCR特异性引物进行扩增,结果显示,均可扩增出相应的目的条带,且呈现阳性(图6)。而健康杂交鳢各组织基因组DNA均未扩增出目的条带(图7)。
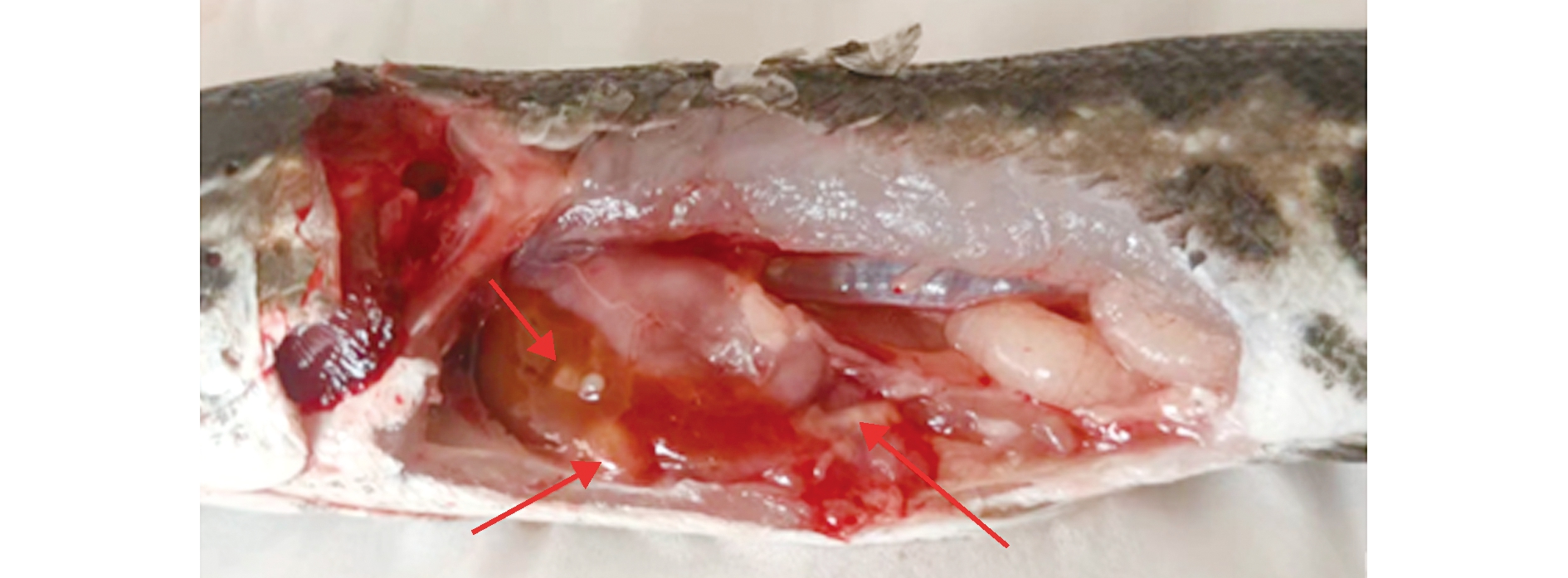
图5 鰤鱼诺卡氏菌感染杂交鳢的腹腔解剖图
Fig.5 Abdominal anatomy of hybrid snakehead infected with Nocardia seriolae
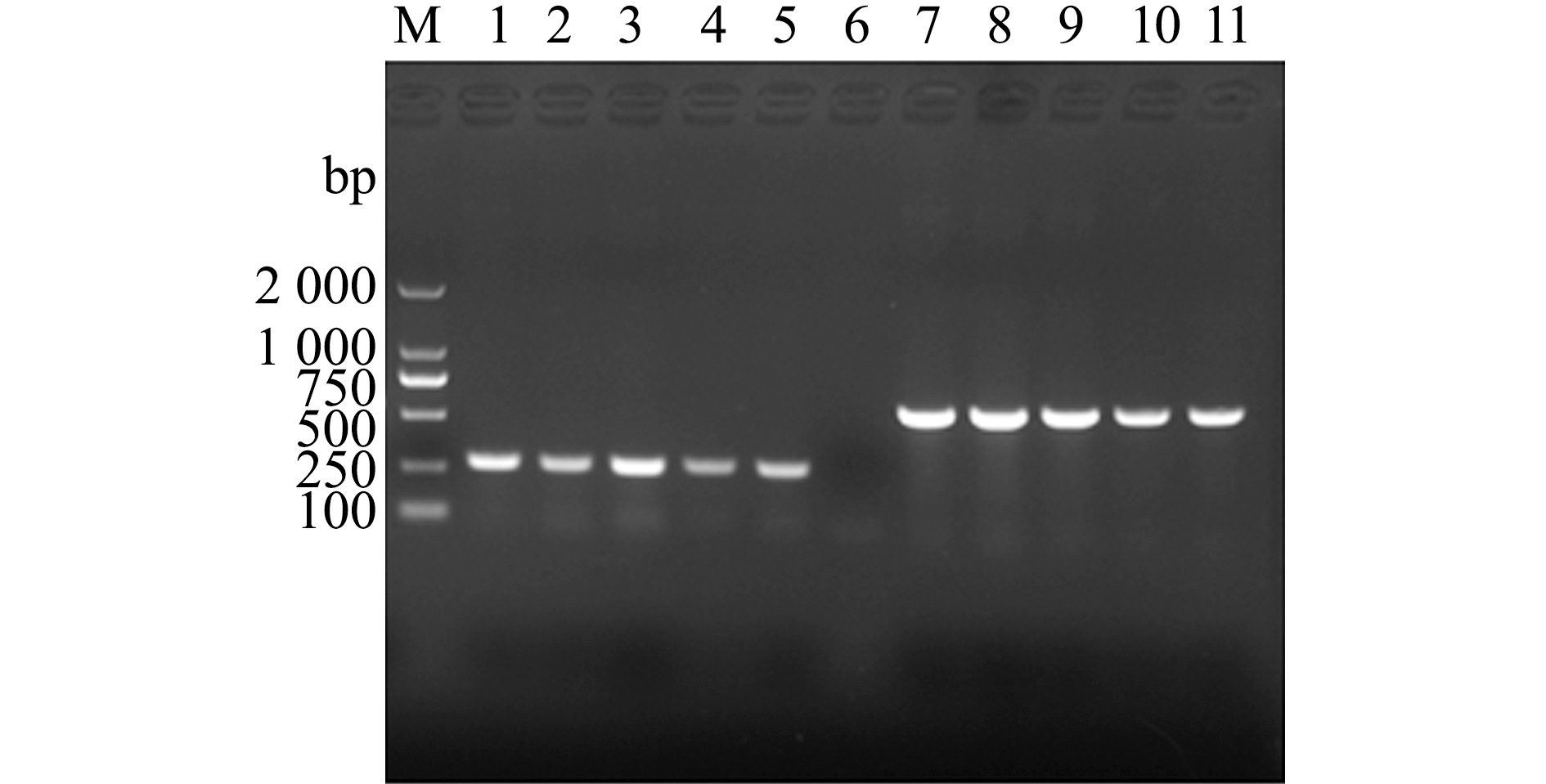
M—DL2000 DNA分子标准;RAA检测,1~5—肝脏、体肾、脾脏、鳃和肌肉,6—阴性对照;PCR检测,7~11—肝脏、体肾、脾脏、鳃和肌肉。M—DL2000 marker; RAA detection,1-5—liver,body kidney,spleen,gills,and muscles,6—negative control; PCR detection,7-11—liver,body kidney,spleen,gills,and muscle.
图6 鰤鱼诺卡氏菌感染杂交鳢的RAA和PCR检测结果
Fig.6 RAA and PCR detection results of hybrid snakehead infected with Nocardia seriolae
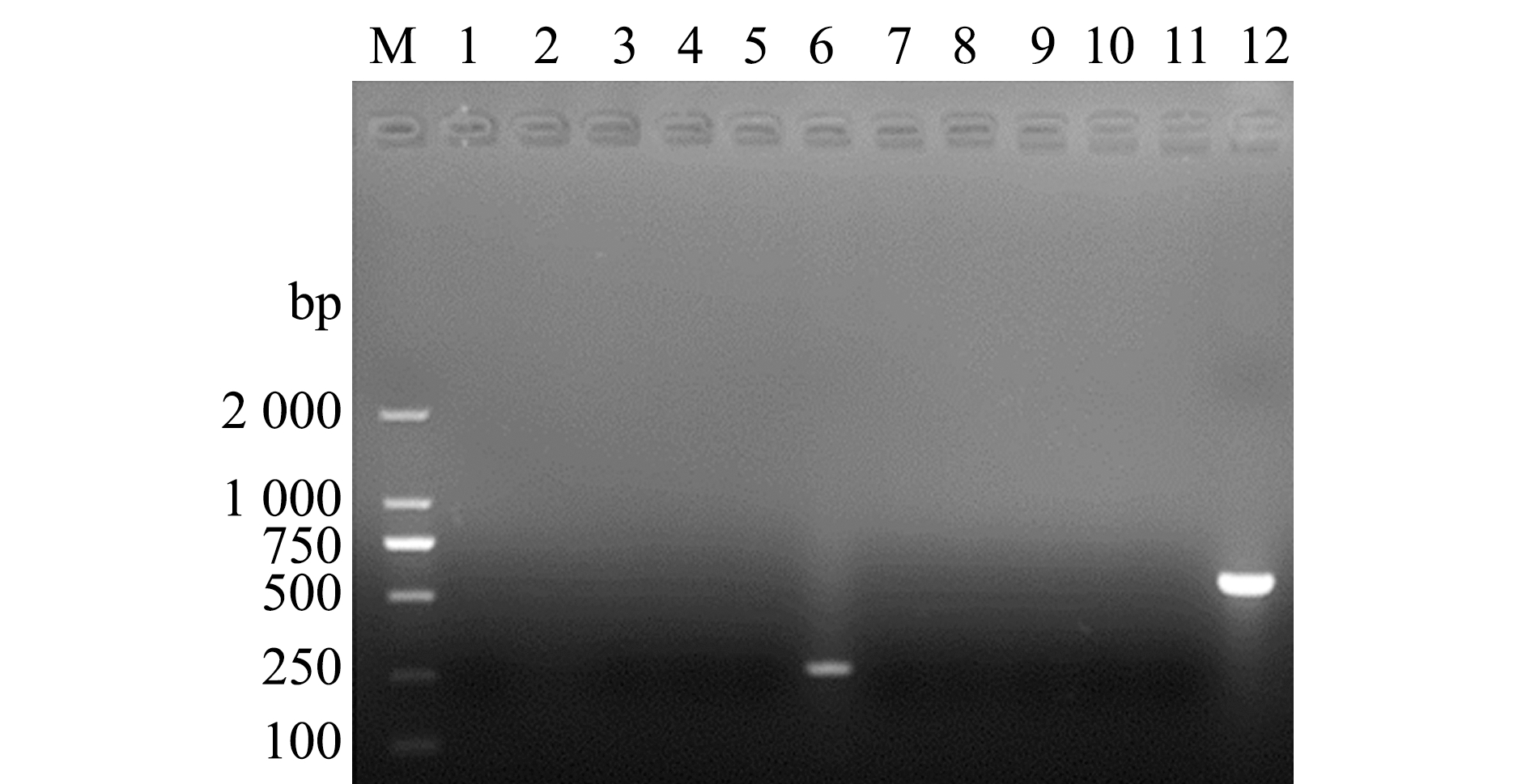
M—DL2000 DNA分子标准;RAA检测,1~5—肝脏、体肾、脾脏、鳃和肌肉,6—阳性对照;PCR检测,7~11—肝脏、体肾、脾脏、鳃和肌肉,12—阳性对照。M—DL2000 marker; RAA detection,1-5—liver,body kidney,spleen,gills,and muscles,6—positive control; PCR detection,7-11—liver,body kidney,spleen,gills,and muscles,12—positive control.
图7 健康杂交鳢的RAA和PCR检测结果
Fig.7 RAA and PCR detection results of healthy hybrid snakehead
2.5 检测应用效果
本研究中使用分离自不同鱼种、时间和地点的11株鰤鱼诺卡氏菌的基因组DNA为模板进行RAA检测。结果显示,不同来源鰤鱼诺卡氏菌的菌株均可扩增出与目的片段大小一致的条带(图8)。这表明,此方法可以应用于检测不同患病鱼种分离到的鰤鱼诺卡氏菌,证实本研究所建立的鰤鱼诺卡氏菌RAA检测技术具有可靠性,可应用于不同来源的鰤鱼诺卡氏菌检测。
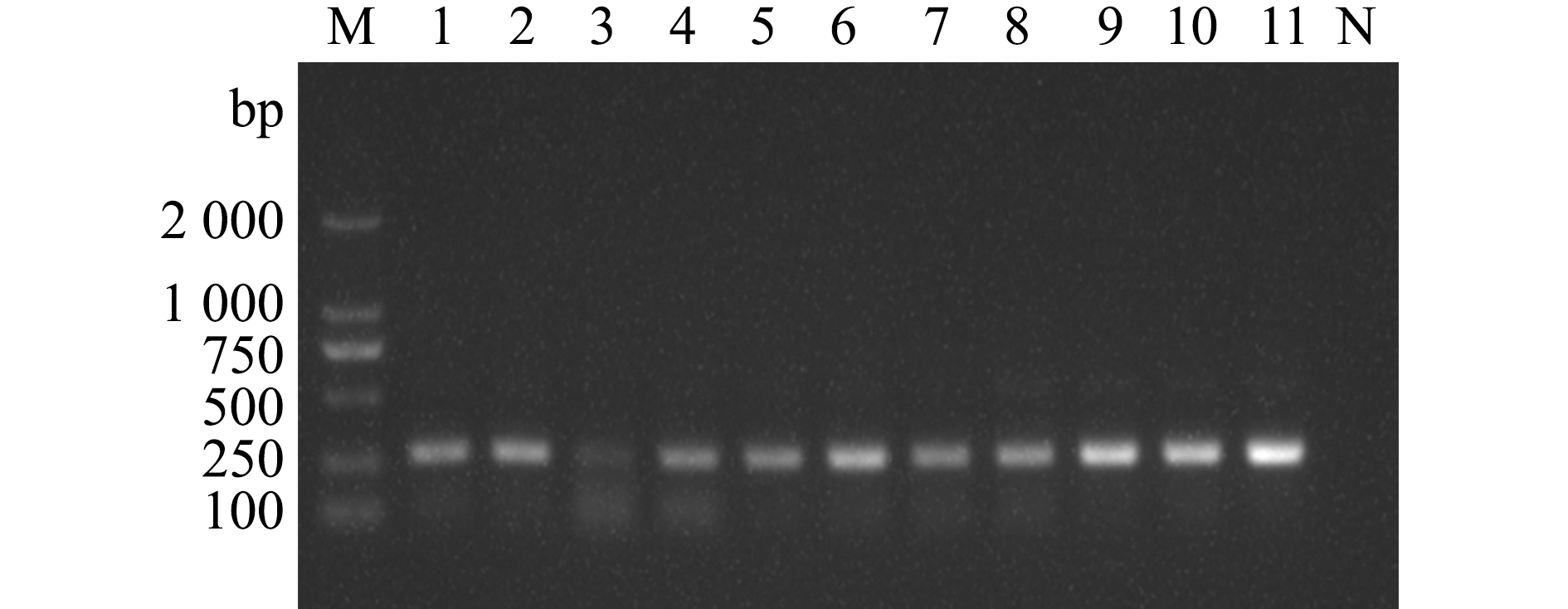
M—DL2000 DNA分子标准;1—NS201300047株;2—NS201400048株;3—NS201300048株;4—NS201300049株;5—NS201500141株;6—NS201300050株;7—NK201610020株;8—NS201300051株;9—FS201912株;10—HB202008株;11—ZJ0503株;N—阴性对照。M—DL2000 marker; 1—strain NS201300047; 2—strain NS201400048; 3—strain NS201300048; 4—strain NS201300049; 5—strain NS201500141; 6—strain NS201300050; 7—strain NK201610020; 8—strain NS201300051; 9—strain FS201912; 10—strain HB202008; 11—strain ZJ0503; N—negative control.
图8 不同来源鰤鱼诺卡氏菌株的RAA检测结果
Fig.8 RAA detection of Nocardia seriolae strains from different sources
3 讨论
3.1 RAA检测方法的选择
鰤鱼诺卡氏菌在侵染鱼体过程中菌体能逃避宿主巨噬细胞杀伤,感染初期引起的内脏病症或炎症不明显,而感染后期巨噬细胞及其演变细胞与鰤鱼诺卡氏菌一起构成了肉芽肿,才具有明显的病症[18-19]。从鰤鱼诺卡氏菌感染致病机理来看,由于肉芽肿成了鰤鱼诺卡氏菌在宿主体内的庇护所和栖息地,故使用药物治疗的效果并不理想。如在一例诺卡氏菌病防治方案中表明,治疗阶段药物见效慢,常用的抗菌药物并不能快速治愈诺卡氏菌病[20]。另外,同样能感染鱼体并形成肉芽肿这一重要病理特征的致病菌有不少,如杀鱼巴斯德氏菌、嗜水气单胞菌和哈维氏弧菌等[21-22],导致在检查患病鱼过程中即使剖检发现患病鱼内脏器官有白色结节,也不能准确诊断为鱼类诺卡氏菌病[23]。针对上述情况,选择一种精准的分子诊断方法对鰤鱼诺卡氏菌病防治至关重要。目前,RAA检测技术已经在多领域病原微生物检测中得以应用,如在食品安全检测中建立了RAA技术,检测水产品的副溶血弧菌(Vibrio parahaemolyticus)[24];在疾病诊断中利用荧光RAA法成功检出细粒棘球绦虫(Echinococcus granulosus)包囊的DNA[25];在农作物病害检测中建立了荧光RAA,检测玉米细菌性枯萎病菌(Pantoeastewartii subsp.stewarii)[26]。因此,本研究中选取种内保守、种间变异的特异性片段作为靶基因,建立RAA检测方法,试验结果显示,近缘菌和相似病症的病原均扩增不出目的条带,做到了对鰤鱼诺卡氏菌的准确识别,从而为鱼类鰤鱼诺卡氏菌病的预防与早期诊疗提供了一种生物技术方面的验证方法,未来还可结合前期样品处理技术和荧光探针的使用,构建更加系统、简便的检测体系。
3.2 鰤鱼诺卡氏菌RAA检测效果
目前,已报道的鰤鱼诺卡氏菌快速检测方法有PCR、qPCR和LAMP[12-14,27-29],尚未见RAA技术用于检测鰤鱼诺卡氏菌的报道。与PCR、LAMP和qPCR等检测方法相比(表3),RAA检测鰤鱼诺卡氏菌具有特异性好、实际应用检测准确率高的特点,由于扩增温度为恒温37 ℃,扩增时间为30 min,且扩增程序简单,也无需使用PCR仪、荧光定量PCR仪等精密仪器,对检测设备要求较低。此外,由于LAMP检测对引物设计要求更高且所需引物较多,RAA检测与之相比也具有一定优势。值得注意的是,可能由于扩增原理、使用的反应酶和试验方法等的差异,使用各种分子检测方法获得的鰤鱼诺卡氏菌检测限不尽相同,PCR[27]、qPCR[14]和LAMP[28]的检测限分别为5×10-6 μg/μL、初始模板浓度的1×10-6 μg/μL和初始模板浓度的1×10-7 μg/μL,而本研究中建立的RAA检测方法在灵敏度(1×10-4 μg/μL)方面并未体现出明显优势,提示需要通过均质化试验进行比较,并进一步优化RAA检测条件。同时在检测成本上也未体现出优势,RAA检测法在检测成本上要高于其他检测方法。这可能是由于目前RAA技术受限于只能使用RAA试剂盒进行恒温扩增反应,而市售的RAA试剂盒受自主知识产权保护价格较高[30]。此外,用本试验中建立的方法进行应用性评估时,仅选取了11个样本进行检测,与多数RAA检测方法建立的研究相比样本量偏少,今后可增加样本量以进一步验证本方法的应用性和重复性。
表3 已报道的鰤鱼诺卡氏菌分子检测方法
Tab.3 Previously reported molecular detection methods of Nocardia seriolae
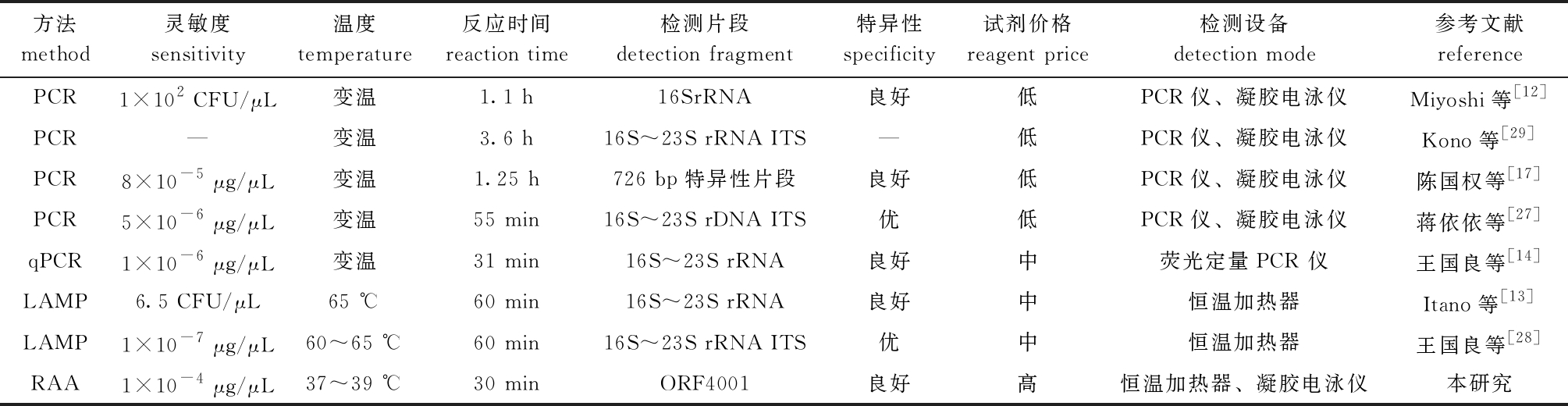
方法method灵敏度sensitivity温度temperature反应时间reaction time检测片段detection fragment特异性specificity试剂价格reagent price检测设备detection mode参考文献referencePCR1×102 CFU/μL变温1.1 h16SrRNA良好低PCR仪、凝胶电泳仪Miyoshi等[12]PCR—变温3.6 h16S^23S rRNA ITS—低PCR仪、凝胶电泳仪Kono等[29]PCR8×10-5 μg/μL变温1.25 h726 bp特异性片段良好低PCR仪、凝胶电泳仪陈国权等[17]PCR5×10-6 μg/μL变温55 min16S^23S rDNA ITS优低PCR仪、凝胶电泳仪蒋依依等[27]qPCR1×10-6 μg/μL变温31 min16S^23S rRNA良好中荧光定量PCR 仪王国良等[14]LAMP6.5 CFU/μL65 ℃60 min16S^23S rRNA良好中恒温加热器Itano等[13]LAMP1×10-7 μg/μL60^65 ℃60 min16S^23S rRNA ITS优中恒温加热器王国良等[28]RAA1×10-4 μg/μL37^39 ℃30 minORF4001良好高恒温加热器、凝胶电泳仪本研究
4 结论
1)根据种内保守、种间变异的基因片段设计出了RAA特异性引物,引物特异性良好,7株鰤鱼诺卡氏菌近缘菌和9种水产养殖过程中常见病原菌均未扩增出目的条带,这说明本研究中所建立的鰤鱼诺卡氏菌重组酶介导等温检测方法具有准确、简便的特点。
2)根据梯度稀释重组质粒的测定试验,本研究中建立的RAA检测方法灵敏度可达到100 pg/μL。
3)从人工感染鰤鱼诺卡氏菌的杂交鳢5个组织基因组DNA和11株不同来源鰤鱼诺卡氏菌的基因组DNA中均可以扩增出目的条带,说明本研究中所建立的鰤鱼诺卡氏菌重组酶介导等温检测方法具有适用性和可靠性。
[1] WANG P C,TSAI M A,LIANG Y C,et al.Nocardia seriolae,a causative agent of systematic granuloma in spotted butterfish,Scatophagus argus,Linn[J].African Journal of Microbiology Research,2014,8(38):3441-3452.
[2] 黄郁葱,简纪常,吴灶和,等.卵形鲳鲹结节病病原的分离与鉴定[J].广东海洋大学学报,2008,28(4):49-53.
HUANG Y C,JIAN J C,WU Z H,et al.Isolation and identification of the pathogen causing sarcoidosis of Trachinotus ovatus[J].Journal of Guangdong Ocean University,2008,28(4):49-53.(in Chinese)
[3] WANG G L,YUAN S P,JIN S.Nocardiosis in large yellow croaker,Larimichthys crocea (Richardson)[J].Journal of Fish Diseases,2005,28(6):339-345.
[4] CHEN S C,LEE J L,LAI C C,et al.Nocardiosis in sea bass,Lateolabrax japonicus,in Taiwan[J].Journal of Fish Diseases,2000,23(5):299-307.
[5] LABRIE L,NG J,TAN Z,et al.Nocardial infections in fish: an emerging problem in both freshwater and marine aquaculture systems in Asia[C]//BONDAD-REANTASO M G,MOHAN V,CRUMLISH M,et al.Asian fisheries society.Diseases in Asian aquaculture VI.Manila:Fish Health Section,Asian Fisheries Society,2008: 297-312.
[6] KIM J D,LEE N S,DO J W,et al.Nocardia seriolae infection in the cultured eel Anguilla japonica in Korea[J].Journal of Fish Diseases,2018,41(11):1745-1750.
[7] 王国良,徐益军,金珊,等.养殖乌鳢诺卡氏菌病及其病原研究[J].水生生物学报,2009,33(2):277-283.
WANG G L,XU Y J,JIN S,et al.Research on the nocardiosis and pathogen in reared snakehead,Ophiocephalus argus Cantor[J].Acta Hydrobiologica Sinica,2009,33(2):277-283.(in Chinese)
[8] 何晟毓,魏文燕,刘韬,等.大口黑鲈致死性结节病病原的分离、鉴定及组织病理学观察[J].水产学报,2020,44(2):253-265.
HE S Y,WEI W Y,LIU T,et al.Isolation,identification and histopathological study on lethal sarcoidosis of Micropterus salmoides[J].Journal of Fisheries of China,2020,44(2):253-265.(in Chinese)
[9] 王文基,陈建林,侯素莹,等.鰤鱼诺卡氏菌感染乌斑杂交鳢的组织病理学研究[J].基因组学与应用生物学,2019,38(10):4439-4446.
WANG W J,CHEN J L,HOU S Y,et al.A histopathological study of Nocardia seriolae infection in hybrid snakehead[J].Genomics and Applied Biology,2019,38(10):4439-4446.(in Chinese)
[10] 夏立群,汪美,赖杰彬,等.鰤鱼诺卡氏菌感染斑马鱼模型的建立与组织病理学研究[J].热带生物学报,2016,7(4):409-416.
XIA L Q,WANG M,LAI J B,et al.Establishment of a zebrafish model for Nocardia seriolae and histopathological study[J].Journal of Tropical Biology,2016,7(4):409-416.(in Chinese)
[11] DEL RIO-RODRIGUEZ R E,RAMIREZ-PAREDES J G,SOTO-RODRIGUEZ S A,et al.First evidence of fish nocardiosis in Mexico caused by Nocardia seriolae in farmed red drum (Sciaenops ocellatus,Linnaeus)[J].Journal of Fish Diseases,2021,44(8):1117-1130.
[12] MIYOSHI Y,SUZUKI S.A PCR method to detect Nocardia seriolae in fish samples[J].Fish Pathology,2003,38(3):93-97.
[13] ITANO T,KAWAKAMI H,KONO T,et al.Detection of fish nocardiosis by loop-mediated isothermal amplification[J].Journal of Applied Microbiology,2006,100(6):1381-1387.
[14] 王国良,刘璐,李思源.鰤鱼诺卡氏菌SYBR GreenⅠ实时荧光定量PCR检测方法的建立与应用[J].水产学报,2012,36(4):509-513.
WANG G L,LIU L,LI S Y.Development of a SYBR GreenⅠreal-time PCR assay for detection of Nocardia seriolae and its application[J].Journal of Fisheries of China,2012,36(4):509-513.(in Chinese)
[15] 吕蓓,程海荣,严庆丰,等.用重组酶介导扩增技术快速扩增核酸[J].中国科学:生命科学,2010,40(10):983-988.
LÜ B,CHENG H R,YAN Q F,et al.Recombinase-aid amplification:a novel technology of in vitro rapid nucleic acid amplification[J].Scientia Sinica(Vitae),2010,40(10):983-988.(in Chinese)
[16] 马巧妮,王萌,朱兴全.重组酶介导扩增技术及其在病原微生物快速检测中的应用进展[J].中国生物工程杂志,2021,41(6):45-49.
MA Q N,WANG M,ZHU X Q.Research advances in recombinase-aided amplification technology and its application in rapid detection of pathogenic microorganisms[J].China Biotechnology,2021,41(6):45-49.(in Chinese)
[17] 陈国权,黄碧艳,张子雯,等.鰤鱼诺卡氏菌特异性PCR检测方法的建立[J].基因组学与应用生物学,2021,40(sup 2):2656-2665.
CHEN G Q,HUANG B Y,ZHANG Z W,et al.Establishment of a specific PCR method for detection of Nocardia seriolae[J].Genomics and Applied Biology,2021,40(sup 2):2656-2665.(in Chinese)
[18] 满其蒙.鰤鱼诺卡氏菌致病机制的研究[D].上海:上海海洋大学,2013.
MAN Q M.Studies on pathogenic mechanism of Nocardia seriolae[D].Shanghai:Shanghai Ocean University,2013.(in Chinese)
[19] MART NEZ-LARA P,MART
NEZ-LARA P,MART NEZ-PORCHAS M,GOLLAS-GALV
NEZ-PORCHAS M,GOLLAS-GALV N T,et al.Granulomatosis in fish aquaculture:a mini review[J].Reviews in Aquaculture,2021,13(1):259-268.
N T,et al.Granulomatosis in fish aquaculture:a mini review[J].Reviews in Aquaculture,2021,13(1):259-268.
[20] 陈文华,赵俊胜,周晓东,等.一例流水槽养殖加州鲈诺卡氏菌病的防治与思考[J].科学养鱼,2020(9):53-54.
CHEN W H,ZHAO J S,ZHOU X D,et al.Prevention and treatment of Nocardia disease of California perch cultured in flume[J].Scientific Fish Farming,2020(9):53-54.(in Chinese)
[21] 李安兴.警惕鱼类诺卡氏菌病[J].海洋与渔业·水产前沿,2011(7):45-48.
LI A X.Beware of Nocardia disease in fish[J].Ocean and Fishery,2011(7):45-48.(in Chinese)
[22] 邱杨玉,郑磊,毛芝娟,等.大黄鱼(Larimichthys crocea)内脏白点病的病原分离和组织病理学观察[J].微生物学通报,2012,39(3):361-370.
QIU Y Y,ZHENG L,MAO Z J,et al.Isolation and identification of the causative agent and histopathology observation of white-spots disease in internal organs of Larimichthys crocea[J].Microbiology China,2012,39(3):361-370.(in Chinese)
[23] 陈海新,朱宇嘉,董碧莲,等.鱼类诺卡氏菌病的研究进展[J].科学养鱼,2021(3):48-51.
CHEN H X,ZHU Y J,DONG B L,et al.Research progress of Nocardia disease in fish[J].Scientific Fish Farming,2021(3):48-51.(in Chinese)
[24] 郝林慧,梁莹,罗纪军,等.重组酶介导扩增技术快速检测水产品中副溶血性弧菌[J].食品安全质量检测学报,2021,12(13):5266-5272.
HAO L H,LIANG Y,LUO J J,et al.Rapid detection of Vibrio parahaemolyticus in aquatic products by recombinase aid amplification[J].Journal of Food Safety & Quality,2021,12(13):5266-5272.(in Chinese)
[25] 丁昕,刘燕红,倪碧娴,等.基于重组酶介导等温扩增技术的细粒棘球绦虫核酸检测方法的建立[J].中国血吸虫病防治杂志,2020,32(4):340-344.
DING X,LIU Y H,NI B X,et al.Establishment of a nucleic acid assay for detection of Echinococcus granulosus based on recombinase-aided isothermal amplification assay[J].Chinese Journal of Schistosomiasis Control,2020,32(4):340-344.(in Chinese)
[26] 单长林,周圆,李孝军.玉米细菌性枯萎病菌荧光重组酶介导等温扩增检测方法的建立与应用[J].广东农业科学,2021,48(1):111-118.
SHAN C L,ZHOU Y,LI X J.Establishment and application of fluorescent recombinase-aided amplification method for Pantoea stewartii subsp. stewarii detection[J].Guangdong Agricultural Sciences,2021,48(1):111-118.(in Chinese)
[27] 蒋依依,李安兴.诺卡菌特异性PCR快速检测方法的建立[J].南方水产科学,2011,7(6):47-51.
JIANG Y Y,LI A X.Establishment of a specific PCR assay to detect Nocardia seriolae[J].South China Fisheries Science,2011,7(6):47-51.(in Chinese)
[28] 王国良,刘璐,徐益军.鱼类致病鰤鱼诺卡氏菌(Nocardia seriolae)的LAMP检测技术建立与应用[J].海洋与湖沼,2011,42(1):27-31.
WANG G L,LIU L,XU Y J.Development and application of loop-mediated isothermal amplification for detection of fish pathogenic Nocardia seriolae[J].Oceanologia et Limnologia Sinica,2011,42(1):27-31.(in Chinese)
[29] KONO T,OOYAMA T,CHEN S C,et al.Sequencing of 16S-23S rRNA internal transcribed spacer and its application in the identification of Nocardia seriolae by polymerase chain reaction[J].Aquaculture Research,2002,33(14):1195-1197.
[30] 孙晓红,后来旺,李达容,等.重组酶等温扩增技术在分析检测中的应用研究进展[J].食品与发酵工业,2020,46(24):265-270.
SUN X H,HOU L W,LI D R,et al.Research progress on the application of isothermal recombinase amplification in analytical detection[J].Food and Fermentation Industries,2020,46(24):265-270.(in Chinese)