副溶血性弧菌(Vibrio parahaemolyticus)是一种革兰氏阴性菌,广泛分布于世界各地温暖的河口和海洋环境中。近年来,由于海鲜消费的增加,生食或未煮熟海鲜中的副溶血性弧菌正成为急性胃肠炎的主要病因[1]。2010年,由副溶血性弧菌引起的凡纳滨对虾(Penaeus vannamei)急性肝胰腺坏死病(AHPND)在中国首次被发现[2-3],随后在越南、马来西亚、泰国和墨西哥等国家的对虾养殖区域也相继发现,严重威胁着全球的对虾养殖业健康发展[4-5]。
在最近的一项研究中,通过对导致出现凡纳滨对虾AHPND症状的副溶血性弧菌菌株进行基因组测序,揭示了与PirAB(PirA、PirB)同源的毒力基因[6-7]。Tran等[8]研究发现,AHPND病原体是一种特异性的副溶血性弧菌,这种变异菌株因其获得了表达致命毒素(PirAB)的质粒而具有较高的致病性[9-10]。这些结果表明,PirAB是导致出现AHPND症状的主要毒力因子[10]。除此之外,副溶血性弧菌还拥有许多其他的毒力因子,包括黏附素、热稳定直接溶血素(TDH)及其相关溶血素(TRH)、MAM7、两种Ⅲ型分泌系统(T3SS1和T3SS2)和两种Ⅵ型分泌系统(T6SS1和T6SS2)等[11-12]。
湛江位于中国大陆最南端,因其丰富的海洋资源已成为中国最大的对虾养殖基地,其产量约占全国的1/5[13]。然而,对该地区对虾养殖池塘内的副溶血性弧菌遗传多样性一直缺乏系统性研究。邓传燕等[14]对广东、广西沿海4地的鱼苗场取样进行了耐药性分析,发现有66.7%的菌株同时对30种以上抗生素具耐药性,并发现了高耐药菌株。高玉龙等[15]通过对分离自广东省阳江市养殖区的88株弧菌进行鉴定和药物敏感试验发现,88株弧菌属于不同的13个种,并且所有菌株均对阿莫西林、四环素等抗生素耐药,同时也显示出多重耐药的性质。马聪等[16]研究了2008—2010年广东地区23起副溶血性弧菌暴发患者临床样本和当餐食品分离株的基因型和耐药性,发现98.6%的分离菌株均携带tdh毒力基因。本研究中,通过2018—2020年对广东省湛江市和茂名市对虾养殖池塘进行取样,从中分离副溶血性弧菌,并对其进行多位点序列分型(MLST)、毒力基因携带检测情况及耐药性等病原学特性表征,以期为该地区副溶血性弧菌的防治提供科学依据。
1 材料与方法
1.1 材料
样品:湛江地区凡纳滨对虾养殖池塘的表层水和沉积物。
试剂:青霉素、环丙沙星、克拉霉素、氨苄西林、四环素、卡那霉素、红霉素、利福平、庆大霉素、复方新诺明、氧氟沙星和左氟沙星12种抗生素药敏纸片购自杭州微生物试剂公司;TCBS培养基、LB琼脂培养基、副溶血性弧菌显色培养基和PCR扩增试剂均购自青岛海博生物技术有限公司。
1.2 方法
1.2.1 患病对虾池塘水样和底泥的采集 分别于2018—2020年每年的5月和11月,在湛江的两个凡纳滨对虾养殖池塘(S1和S2)和茂名的一个养殖池塘(Z1) 进行取样。采样点坐标分别为S1(110°23′97″E,21°02′82″N)、S2(110°59′72″E,21°12′43″N)和Z1(110°90′07″E,21°53′83″N)。分别采集发生急性肝胰腺坏死病的对虾养殖池塘中表层水及沉积物样本。其中,采样点S1和S2养殖对虾为3个月,在养殖期间未换水,水中盐度分别为1.2 和1.9,人工投喂饲料,养殖池体积为24 m3;Z1为全年养殖,不定期换水,水中盐度为13.98,人工投喂饲料。使用水体采集器在池塘四角及中央采集水面表层0.5 m处水样各1.5 L;同时在相同位置,使用采集器采集沉积物样本200 g装入采集袋中。将采集的所有样品置于4 ℃冰盒中保存,带回实验室后对水样和底泥样本进行处理。
1.2.2 菌株分离纯化 在超净工作台上,将表层水样用生理盐水进行梯度稀释后,取200 μL均匀涂布于TCBS琼脂培养基上;取沉积物样本25 g,加入225 mL体积分数为3%的氯化钠碱性蛋白胨水(36±1)℃中增菌8~18 h,取200 μL涂布于TCBS培养基上。将经过处理后的表层水样、沉积物样本置于37 ℃恒温培养箱内培养20~24 h,观察菌落形态。将培养出的单菌落进行划线纯化,挑取TCBS平板上的疑似副溶血性弧菌的蓝绿色单菌落接种到LB培养基上,过夜培养后,于-80 ℃超低温冰箱中保存。
1.2.3 菌株鉴定 挑取TCBS平板上疑似副溶血性弧菌的蓝绿色单菌落,按照《食品安全国家标准 食品微生物学检验 副溶血性弧菌检验》GB 4789.7—2013对其进行生化鉴定,生化试验呈阳性的菌株利用细菌基因组DNA提取试剂盒(天根生化科技有限公司)提取基因组DNA。对菌株16S基因进行PCR扩增。PCR反应体系(共25 μL):DNA模板2.5 μL,上、下 游引物各0.5 μL,Premix Taq 12.5 μL,无菌水9 μL。本试验中用引用信息见表1,PCR扩增产物经脉冲场凝胶电泳分析后,选取条带明亮的样品送至生工生物工程(上海)股份有限公司进行测序,测序结果在NCBI(http://www.ncbi. nlm.nih.gov)上进行同源性比对。
表1 本试验中的引物信息
Tab.1 Primer information in this experiment

目标基因 target引物序列 primer sequence(5'-3')16S rDNAF:GAGTTTGATCCTGGCTCAGR:TACGGTTACCTTGTTACGACTTtdhF:CTAAATAGGGCTGTTCGGCTR:GAAAAGGGACAGATGGAAGCtrhF:CTTTTCCTTCTCCAGGTTCGR:CAGAAAGAGCAGCCATTGTGtlhF:AAAGCGGATTATGCAGAAGCACTGR:GCTACTTTCTAGCATTTTCTCTGCmam7F:CTTAGAAAGCATGAGCGCCCTACR:GATGATGGAACCGACCACAATACvcrD1F:ACGCTATTTCGTTTGTCCCTGTCR:GCTTTCTGATTCGGTTTGCTTTTvcrD2F:TATGGCATGTGGTGTCTATTTGR:CGCTACGCAGTGTAGTTTGTACpirAF:TGACTATTCTCACGATTGGACTGL:CACGACTAGCGCCATTGTTA
1.2.4 药物敏感性试验 采用纸片扩散法,对副溶血性弧菌进行12种常见的抗生素药物敏感性试验。挑取纯化培养18~24 h的副溶血性弧菌单菌落制成菌悬液,用移液枪吸取200 μL菌悬液,涂布于LB培养基上,将药敏片均匀贴于表面,28 ℃恒温培养箱内培养18~24 h后测量抑菌圈直径。记录各类药物对副溶血性弧菌菌株产生的抑菌圈直径,并根据CLSI第三版标准[17]对结果进行判定。
1.2.5 副溶血性弧菌毒力基因检测 采用细菌基因组DNA提取试剂盒提取副溶血性弧菌基因组DNA,对分离的58株副溶血弧菌进行毒力基因检测,采用PCR对tdh、trh、tlh、mam7、vcrD1、vcrD2和pirAB等毒力基因进行检测。引物由生工生物工程(上海)股份有限公司合成,引物信息见表1。PCR产物于凝胶电泳15 min后,使用凝胶成像系统分析结果。将明亮条带送至生工生物工程(上海)股份有限公司进行测序。
1.2.6 多位点序列分型 对分离的58株副溶血弧菌进行MLST分型,首先选择副溶血性弧菌的7个管家基因(dnaE、gyrB、recA、dtdS、pntA、pyrC和tnaA)作为本试验的目的基因,7个管家基因位点与参数见表2。引物来源参考PubMLST网络数据库,7对PCR引物序列由生工生物工程(上海)股份有限公司合成。PCR反应体系(共25 μL)为Premix Taq 12.5 μL,上、下游引物各0.5 μL,模板2 μL,无菌水9.5 μL。PCR扩增反应条件参照PubMLST网络数据库公布的副溶血性弧菌的标准方案进行:96 ℃下预变性5 min;96 ℃下变性1 min,58 ℃下退火1 min,72 ℃下延伸 1 min,共进行30个循环;最后在72 ℃下再延伸10 min[18]。PCR产物送测序公司测序后,将序列提交到PubMLST官网数据库(http://pubmlst.org/vparahaemolyticus/)中进行比对,确定菌株的ST型。采用 MEGA 6.0软件[19]中的最大似然法进行系统发育分析。
表2 MLST分型管家基因位点与参数
Tab.2 Locus and parameters of housekeeping gene in MLST typing

基因位点gene locus引物序列 (5'-3')primer sequence (5'-3')测序片段长度/bplength of sequenced fragmentsrecAF:GAAACCATTTCAACGGGTTCR:CCATTGATAGCTGTTATACCAAGCACCC767gyrBF:GAAGGBGGTATTCAAGCCAAGCR:GAGTCTACCCTCCACWATGTATA624dnaEF:CGRATMACCGCTTTCGCCCGCCCR:GAKATGTGTGAGCTGTTTGCAT589dtdSF:TGGCCATAACGACATTCTGAGATCCR:GAGCCACCAACGTGTTTATGA478pntAF:ACGGCTACGCAAAGAAAATGCATR:TTGAGGCTGAGCTATACTTGC465pyrCF:AGCAACCGGTAAAATTGTCGR:CAGTGTAAAGAAACCGGCACATTA534tnaAF:TGTACGAAATTGCCACCAAAR:AATATTTTCGCCCGATTCAATCGGA454
1.2.7 基因组de novo测序、组装和比较基因组分析 在58株分离株中,通过药敏试验发现了4株多重耐药菌株,基因型均为ST1740。对其中的1株进行基因组de novo测序、组装和比较基因组分析。采用TIANamp Bacteria DNA Kit(天根生化科技有限公司)提取基因组DNA,将电泳合格的ST1740基因组DNA送至北京诺和基因生物信息技术有限公司,在Illumina HiSeq 2500平台进行双末端测序;对FASTQ测序文件使用Trimmomatic 0.36软件进行质量检测,并将尾端删除得分<30的碱基;使用SPAdesv 3.2软件从头组装染色体的重叠群来获得基因组草图,采用在线软件RAST(http://rast.nmpdr.org/rast.cgi)对基因组进行注释并归类其功能类别[20];利用在线工具Resfinder (https://cge.cbs.dtu.dk/services/) 确定耐药基因[21],采用IslandPath-DIOMB程序预测基因组岛[22]。
2 结果与分析
2.1 副溶血性弧菌的检出结果
2018—2020年,从广东省湛江和茂名两个地区受AHPND影响的患病对虾养殖池水体及沉积物中共分离出58株副溶血性弧菌菌株,2018、2019和2020年分别获得副溶血性弧菌30株、7株和21株。其中,38株分离自湛江,20株分离自茂名。
2.2 MLST分型
通过对分离出的58株副溶血性弧菌菌株的7个管家基因进行MLST分型,共鉴定出34个ST型。将每个菌株的7个管家基因序列合并,用MEGA 6.0软件构建系统发育树显示,共分为4个cluster(图1)。其中,2018年分离得到副溶血性弧菌30株,MLST分型得到17种ST型,包括ST1740、ST1256、ST433、ST2193、ST452、ST2013、ST2192、ST299、ST917、ST1734、ST424、ST215、ST1742、ST2204、ST1513、ST970 和ST2208;2019年分离的7株菌株,可分为6种ST型,包括ST558、ST2171、ST180、ST1229、ST1910和ST888;2020年分离的21株菌株,可分为16种ST型,包括ST1740、ST558、ST1799、ST180、ST1166、ST1229、ST415、ST1042、ST413、ST2013、ST1160、ST2198、ST414、ST1061、ST1481和ST1985。
不同ST型包含菌株数量各不相同。ST917和ST1740均包含4个菌株,ST180、ST2013、ST1734、ST424和ST1513均包含3个菌株,剩余ST型只包含1~2个菌株;34个ST型相对分散,且菌株的基因型多样,同一个ST型在不同年份出现,如ST1740、ST2013在2018、2020年中均有出现,而ST558、ST180、ST1229在2019、2020年中均有出现(图1)。MLST分型结果显示出湛江地区副溶血性弧菌的遗传多样性。

图1 58株副溶血性弧菌的聚类分析、分型和毒力基因检测
Fig.1 Cluster analysis, typing results and virulence gene detection of 58 strains of Vibrio parahaemolyticus
2.3 毒力基因检测
tdh和trh是副溶血性弧菌的主要毒力因子,通过PCR对tdh、trh、tlh、mam7、vcrD1和vcrD2等毒力基因的携带情况进行检测,并将明亮的样品进行测序。结果显示,58株菌株均包含毒力基因tlh、mam7和vcrD1,未发现tdh、trh和vcrD2的存在(图1)。其中,8株菌株(ZJ-141/2018、ZJ-142/2018、ZJ-143/2018、ZJ20-3/2020、ZJ20-10/2020、ZJ-124/2018、ZJ-125/2018和ZJ-126/2018)携带284 bp的pirA(图2)。
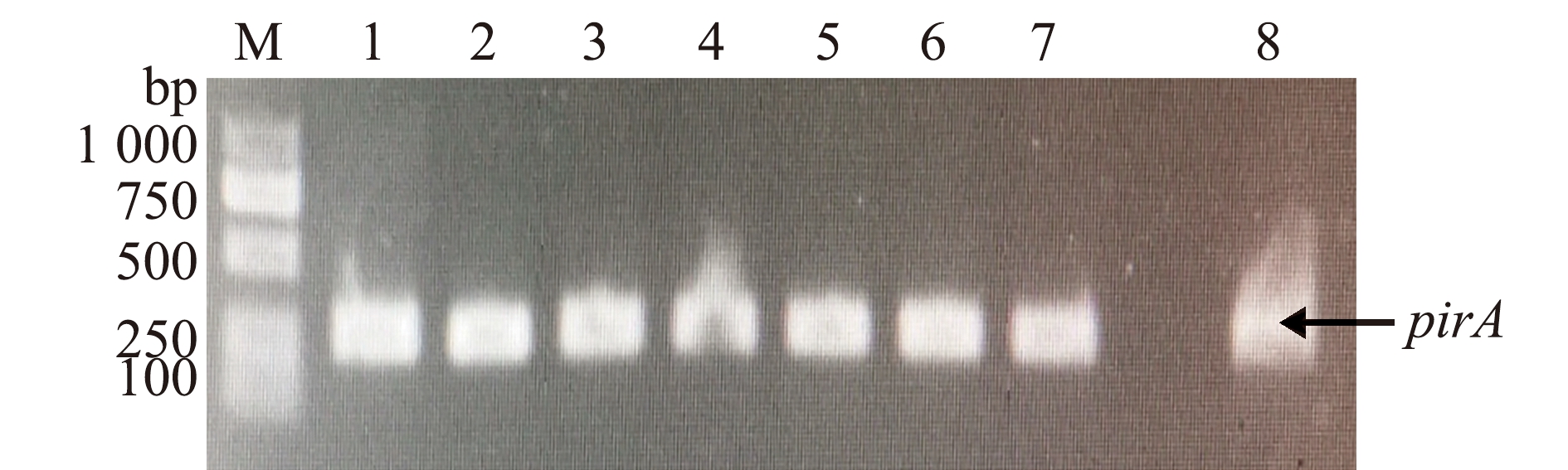
M—DL2000 marker; 1—ZJ-141/2018; 2—ZJ-142/2018; 3—ZJ-143/2018; 4—ZJ20-3/2020; 5—ZJ20-10/2020; 6—ZJ-124/2018; 7—ZJ-125/2018; 8—ZJ-126/2018.
图2 8株携带pirA的副溶血性弧菌琼脂糖凝胶电泳图
Fig.2 Agarose gel electrophoresis of 8 strains of Vibrio parahaemolyticus carrying pirA
2.4 副溶血性弧菌的药物敏感性分析
从表3可见,58株副溶血性弧菌对青霉素的耐药率为100%,对环丙沙星、克拉霉素、氨苄西林、四环素、卡那霉素、红霉素、利福平、庆大霉素和复方新诺明9种抗生素的耐药率为5.1%~15.5%。其中,对氨苄西林、克拉霉素和庆大霉素的耐药率较高,分别为15.5%、13.7%和13.7%。分析表明,64.2%的菌株(包含ST1740、ST558、ST1166、ST1042、ST2013、ST1160、ST414、ST1061、ST1481和ST1958)同时对2种以上的抗生素产生耐药性,其中4株ST1740菌株为多重耐药菌株,对青霉素、氨苄西林、红霉素和复方新诺明4种抗生素耐药。
表3 58株副溶血性弧菌药敏试验结果
Tab.3 Drug susceptibility test results of 58 strains of Vibrio parahaemolyticus
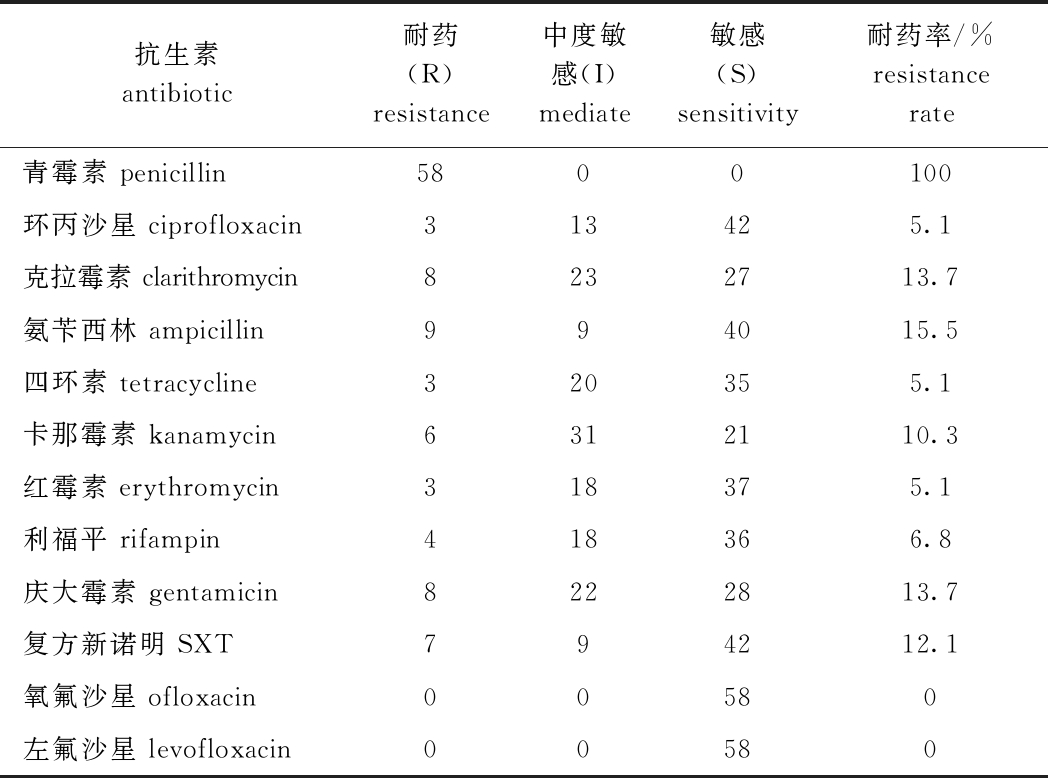
抗生素antibiotic耐药(R)resistance中度敏感(I)mediate敏感(S)sensitivity耐药率/%resistance rate青霉素 penicillin5800100环丙沙星 ciprofloxacin313425.1克拉霉素 clarithromycin8232713.7氨苄西林 ampicillin994015.5四环素 tetracycline320355.1卡那霉素 kanamycin6312110.3红霉素 erythromycin318375.1利福平 rifampin418366.8庆大霉素 gentamicin8222813.7复方新诺明 SXT794212.1氧氟沙星 ofloxacin00580左氟沙星 levofloxacin00580
2.5 1株多重耐药菌株的测序和比较基因组学分析
药敏试验发现,4株ST1740菌株均为多重耐药,且耐药谱相同。因此,本研究中选择其中1株菌株ZJ20-3/2020进行全基因组测序。结果表明,该基因组为5.24 Mbp,总的蛋白编码序列(CDs)数目为5 372,平均GC含量为44.8%。采用在线工具Resfinder查找耐药基因,共发现8个耐药基因,分别为blaCARB-41(青霉素抗性基因)、aadA16(卡那霉素抗性基因)、aph(6)-Id(卡那霉素抗基因性)、aph(3″)-Ib(卡那霉素抗性基因)、tet(A)(四环素抗性基因)、sul1(利福平抗性基因)、dfrA27(利福平抗性基因)和ARR-3(利福平抗性基因)。
通过IslandPath-DIOMB程序发现,ZJ20-3/2020(ST1740)菌株存在4个基因岛。BLAST检索发现,4个基因岛均与希瓦氏菌NCTC12093中的相应序列具有99%的DNA序列一致性。通过比较基因组学分析表明,ZJ20-3/2020菌株共有40个contigs,4个基因岛分别位于基因组片段9、23、25和28位置,其中,两个基因岛含有耐药基因,基因岛2编码aadA16、aph(6)-Id、aph(3″)-Ib、tet(A)和tet(R),基因岛3编码sul1、dfrA27和ARR-3等基因(图3)。

在ZJ20-3/2020菌株中发现了4个额外外源性的基因岛,如方框图所示。图中绿线表示存在同源关系。基因岛4中的数字表示DNA相似性。Four additional genomic islands are found in ST1740 strain which is indicated in the box panel. The green line in the figure indicates the existence of a homologous relationship. The number in gene island 4 indicates DNA similarity.
图3 副溶血性弧菌ZJ20-3/2020(ST1740)菌株及其携带的4个来自希瓦氏菌的基因岛
Fig.3 Vibrio parahaemolyticus ZJ20-3/2020(ST1740) and its four gene islands from Shewanella
3 讨论
3.1 MLST在副溶血性弧菌流行病学中的应用
多位点序列分型(MLST)是一种基于所选管家基因序列多态性对病原菌进行分型的通用方法。在这种分型方案中,每个独特的基因片段被分配到一个不同的等位基因号,每个管家基因座的等位基因组合被分类为序列类型(ST)[23]。Hossain等[24]利用该方法研究发现,从美国鲇中获得的嗜水气单胞菌菌株与中国患病草鱼中分离出的嗜水气单胞菌具有高度相似的基因组,表明美国鲇分离株来自亚洲起源的嗜水气单胞菌菌株。因此,这项技术有可能提高研究者对病原体传播的认识,并确定病原体的来源。
Fu等[25]通过对2008—2017年分离的233株副溶血性弧菌菌株的全基因组分析,发现233株菌株可分为84种ST型,并发现AHPND的传播模式包括两个步骤,即副溶血性弧菌在养殖区域的传播和其携带的AHPND相关质粒在弧菌属间的水平转移,同时也表明副溶血性弧菌菌株具有高度的遗传多样性。孙明玉等[9]对12株致病性副溶血性弧菌MLST分型发现,从广东分离的致病性菌株的新序列型ST1740与ST975的遗传关系最为相近。王瑶华等[26]通过MLST分型从16株AHPND致病菌中发现了9个ST型,其中包含了2个未知型和2个未报道过致病性副溶血性弧菌的相关序列型。陈伟冰等[27]通过对茂名市涉水产品中分离出的44株副溶血性弧菌菌株进行PFGE、MLST分型,发现44株副溶血性弧菌菌株分为44种不同的 PFGE 带型,其中30个ST型,呈现一定的遗传多样性。本研究中,通过对湛江和茂名地区58株副溶血性弧菌菌株进行MLST分型,发现了34个ST型,其中ST1256、ST433、ST2193、ST2192、ST299、ST2198、ST215、ST2204和ST970等9个ST型菌株在陈伟冰等[27]的研究结果中也存在,表明这些ST型可能是该地区的主要流行亚型。
3.2 副溶血性弧菌毒力基因鉴定
副溶血性弧菌可产生几种类型的溶血素,包括耐热直接溶血素(TDH)、TDH相关溶血素(TRH)和不耐热溶血素(TLH)。TDH在副溶血性弧菌的致病性中起着至关重要的作用[28]。然而,TRH和TLH也是副溶血弧菌菌株重要的毒力因子。tlh基因与tdh基因不具有同源性,被认为是副溶血弧菌的特异性基因[29],所有的副溶血弧菌都会产生tlh基因。为验证对虾养殖池塘是否也存在致病性副溶血性弧菌菌株,本研究中对湛江与茂名地区分离出的副溶血性弧菌进行了毒力基因检测,结果发现,58株菌株均包含毒力基因tlh、mam7和vcrD1,未发现tdh、trh和vcrD2的存在。
本研究结果与Song等[30]和檀利军等[31]的研究结果一致,这些研究者从患者中分离的大多数临床菌株均携带tdh基因,且携带tdh基因的环境菌株的检出率远低于临床菌株。近年来的研究也表明,临床来源的副溶血性弧菌分离株中90%为tdh或trh阳性,而环境来源的分离株中只有0.2%~10%含有tdh和trh基因[30,32]。
值得注意的是,本研究中pirAB阳性检出率为13.8%,其中,ST415、ST424和ST1740型菌株均为阳性(图1)。孙明玉[33]通过荧光原位杂交探针法检测到12株AHPND 致病性副溶血弧菌菌株均携带pirAB。贾丹[34]对2013—2016年分离自中国沿海地区发病对虾养殖场的菌株进行了分析,发现pirAB阳性检出率为33.11%,出现最多的血清型为O1∶K25(23.33%),其余类型较为分散,无明显优势型。本研究中,pirAB阳性检出率稍低于前人研究,可能原因是近年来pirAB毒力基因正在向亲缘关系较远的非副溶血性弧菌中转移。陈营等[35]通过对江苏、福建和海南等省的对虾养殖基地样品进行分析,发现副溶血性弧菌中的pirAB毒力基因已经向哈维弧菌(Vibrio harveyi)、创伤弧菌(Vibrio vulnificus)转移,也证明了这一推断。
3.3 副溶血性弧菌基因组中首次发现来自其他属的基因岛
本研究中药敏试验结果显示,58株副溶血性弧菌对青霉素耐药率为100%,对氨苄西林、克拉霉素和庆大霉素的耐药率较高,对环丙沙星、四环素和红霉素的耐药率较低,64.2%的菌株同时对2种以上的抗生素产生耐药性,其中,4株ST1740型菌株为多重耐药菌株。对其中1株ST1740型菌株进行了全基因组测序发现,该菌株共含有8个耐药基因。
本研究中,进一步解析了耐药基因的分布,发现其存在4个来自希瓦氏菌的基因岛,其中2个基因岛携带了多个耐药基因。这些耐药基因的存在也验证了其多重耐药的表型主要是由基因组中携带的耐药基因所决定。基因的水平转移,特别是耐药基因的水平转移是副溶血性弧菌在养殖环境中进化的重要驱动因素。副溶血性弧菌O3∶K6血清型大流行的出现与副溶血性弧菌密切相关,其通过连续获得7个来自霍乱弧菌(Vibrio cholera)、哈维弧菌(Vibrio harveyi)的基因岛,最终演变为能适应人体环境的大流行基因型[36]。致病性弧菌中存在大量来自溶珊瑚弧菌(Vibrio coralliilyticus)、哈维弧菌(Vibrio harveyi)、鳗弧菌(Vibrio anguillarum)和其他未知来源的基因岛[37],但来自希瓦氏菌的基因岛并不常见,仅有少数研究对此进行了报道,如Efimov等[38]通过对以色列鳗鱼养殖场分离的创伤弧菌进行基因组测序,发现其存在biotype 2型和biotype 3型两种基因型。系统发育分析表明,这两种基因型在进化上同源,但biotype 3型获得了一个来自希瓦氏菌的基因岛,毒力得到增强。本研究中,在1株多重耐药副溶血性弧菌基因组中同时发现其含有4种来自希瓦氏菌的基因岛,这是首次在副溶血弧菌中发现来自其他属的基因岛。这可能与养殖过程中大量使用抗生素有关。抗生素的使用提供了一个较强的选择压力,迫使副溶血性弧菌从其他物种获得耐药基因岛,以适应投加不同抗生素的养殖环境。
4 结论
1)通过对湛江和茂名地区的对虾养殖池塘中分离的58株副溶血性弧菌菌株进行分子分型和表型表征,表明该地区对虾养殖池塘中的副溶血性弧菌具有丰富的遗传多样性。
2)在4株多重耐药菌株中发现了一个携带pirAB的新ST型(ST1740),随后的基因组测序发现,该基因组中携带4种来自希瓦氏菌的基因岛,这也是首次在副溶血性弧菌中发现来自希瓦氏菌属的基因岛。耐药基因岛的发现,揭示了菌株获得耐药性的原因。
[1] LETCHUMANAN V,YIN W F,LEE L H,et al.Prevalence and antimicrobial susceptibility of Vibrio parahaemolyticus isolated from retail shrimps in Malaysia[J].Frontiers in Microbiology,2015,6:33.
[2] DE LA PE A L D,CABILLON N A,CATEDRAL D D,et al.Acute hepatopancreatic necrosis disease (AHPND) outbreaks in Penaeus vannamei and P.monodon cultured in the Philippines[J]. Diseases of Aquatic Organisms,2015,116(3):251-254.
A L D,CABILLON N A,CATEDRAL D D,et al.Acute hepatopancreatic necrosis disease (AHPND) outbreaks in Penaeus vannamei and P.monodon cultured in the Philippines[J]. Diseases of Aquatic Organisms,2015,116(3):251-254.
[3] JOSHI J,SRISALA J,TRUONG V H,et al.Variation in Vibrio parahaemolyticus isolates from a single Thai shrimp farm experiencing an outbreak of acute hepatopancreatic necrosis disease (AHPND)[J].Aquaculture,2014,428/429:297-302.
[4] GOMEZ-GIL B,SOTO-RODR GUEZ S,LOZANO R,et al.Draft genome sequence of Vibrio parahaemolyticus strain M0605,which causes severe mortalities of shrimps in Mexico[J].Genome Announcements,2014,2(2):e00055-e00014.
GUEZ S,LOZANO R,et al.Draft genome sequence of Vibrio parahaemolyticus strain M0605,which causes severe mortalities of shrimps in Mexico[J].Genome Announcements,2014,2(2):e00055-e00014.
[5] RESTREPO L,BAYOT B,BETANCOURT I,et al.Draft genome sequence of pathogenic bacteria Vibrio parahaemolyticus strain Ba94C2,associated with acute hepatopancreatic necrosis disease isolate from South America[J].Genomics Data,2016,9:143-144.
[6] YANG Y T,CHEN I T,LEE C T,et al.Draft genome sequences of four strains of Vibrio parahaemolyticus,three of which cause early mortality syndrome/acute hepatopancreatic necrosis disease in shrimp in China and Thailand[J].Genome Announcements,2014,2(5):e00816-e00814.
[7] HAN J E,TANG K F J,TRAN L H,et al.Photorhabdus insect-related (Pir) toxin-like genes in a plasmid of Vibrio parahaemolyticus,the causative agent of acute hepatopancreatic necrosis disease (AHPND) of shrimp[J].Diseases of Aquatic Organisms,2015,113(1):33-40.
[8] TRAN L,NUNAN L D,REDMAN R M,et al.Determination of the infectious nature of the agent of acute hepatopancreatic necrosis syndrome affecting penaeid shrimp[J].Diseases of Aquatic Organisms,2013,105(1):45-55.
[9] 孙明玉,冯博,张昭寰,等.引起凡纳滨对虾急性肝胰腺坏死病的副溶血弧菌MLST新序列型[J].水产学报,2018,42(3):410-418.
SUN M Y,FENG B,ZHANG Z H,et al.New sequence type isolates of AHPND-causing Vibrio parahaemolyticus from Litopenaeus vannamei by multilocus sequence typing[J].Journal of Fisheries of China,2018,42(3):410-418.(in Chinese)
[10] LEE C T,CHEN I,YANG Y,et al. The opportunistic marine pathogen Vibrio parahaemolyticus becomes virulent by acquiring a plasmid that expresses a deadly toxin[J].Proceedings of the National Academy Sciences,2015,112(34):10798-10803.
[11] MAKINO K,OSHIMA K,KUROKAWA K,et al.Genome sequence of Vibrio parahaemolyticus:a pathogenic mechanism distinct from that of V cholerae[J].The Lancet,2003,361(9359):743-749.
[12] ZHANG L L,ORTH K.Virulence determinants for Vibrio parahaemolyticus infection[J].Current Opinion in Microbiology,2013,16(1):70-77.
[13] 李易珊.在美国人的餐桌上,每4条虾中就有一条来自湛江[J].海洋与渔业,2018(10):68-69.
LI Y S.One out of every four shrimps on the American table comes from Zhanjiang[J].Ocean and Fishery,2018(10):68-69.(in Chinese)
[14] 邓传燕,李色东,范敏萍,等.2010—2012年对虾育苗水体副溶血弧菌耐药性分析[J].科学养鱼,2013(9):59.
DENG C Y,LI S D,FAN M P,et al.Analysis of drug resistance of Vibrio parahaemolyticus in shrimp breeding water from 2010 to 2012[J].Scientific Fish Farming,2013(9):59.(in Chinese)
[15] 高玉龙,冯娟,苏友禄,等.广东阳江市闸坡网箱养殖区弧菌耐药性分析[J].海洋环境科学,2014,33(1):32-35.
GAO Y L,FENG J,SU Y L,et al.Analysis of the drug-resistance of the vibrios isolated from net-cage areas in Zhapo-Yangjiang,Guangdong Province[J].Marine Environmental Science,2014,33(1):32-35.(in Chinese)
[16] 马聪,何冬梅,邓小玲,等.广东地区副溶血性弧菌暴发分离优势血清型菌株的分子特征[J].中华微生物学和免疫学杂志,2011,31(12):1093-1098.
MA C,HE D M,DENG X L,et al.Molecular characteristic of dominant serotypes of Vibrio parahaemolyticus isolated from foodborne disease outbreaks in Guangdong Province[J].Chinese Journal of Microbiology and Immunology,2011,31(12):1093-1098.(in Chinese)
[17] 李翠苹,翟倩倩,王想,等.对虾养殖池副溶血弧菌的分离鉴定及其耐药特征、毒力基因分析[J].渔业科学进展,2020,41(6):174-180.
LI C P,ZHAI Q Q,WANG X,et al.Isolation and identification of Vibrio parahaemolyticus from shrimp culture ponds and analysis of its drug resistance and virulence genes[J].Progress in Fishery Sciences,2020,41(6):174-180.(in Chinese)
[18] 闫鹏,高红,林文凭,等.副溶血性弧菌多位点序列分型方法的建立与应用[J].中国预防医学杂志,2016,17(2):103-107.
YAN P,GAO H,LIN W P,et al.Genotyping analysis of Vibrio parahamolyticus by using multi-locus sequence typing technique[J].Chinese Preventive Medicine,2016,17(2):103-107.(in Chinese)
[19] 李司宇,刘雪,王文婧,等.微生物进化树构建方法[J].现代农业科技,2019(19):249-250,253.
LI S Y,LIU X,WANG W J,et al.Construction of microbial evolutionary trees[J].Modern Agricultural Science and Technology,2019(19):249-250,253.(in Chinese)
[20] AZIZ R K,BARTELS D,BEST A A,et al.The RAST Server:rapid annotations using subsystems technology[J].BMC Genomics,2008,9:75.
[21] ZANKARI E,HASMAN H,COSENTINO S,et al.Identification of acquired antimicrobial resistance genes[J].Journal of Antimicrobial Chemotherapy,2012,67(11):2640-2644.
[22] HSIAO W,WAN I,JONES S J,et al.IslandPath:aiding detection of genomic islands in prokaryotes[J].Bioinformatics (Oxford,England),2003,19(3):418-420.
[23] MIYAMOTO Y,KATO T,OBARA Y,et al.In vitro hemolytic characteristic of Vibrio parahaemolyticus:its close correlation with human pathogenicity[J].Journal of Bacteriology,1969,100(2):1147-1149.
[24] HOSSAIN M J,SUN D W,MCGAREY D J,et al.An Asian origin of virulent Aeromonas hydrophila responsible for disease epidemics in United States-farmed catfish[J].mBio,2014,5(3):e00848-e00814.
[25] FU S Z,WEI D W,YANG Q,et al.Horizontal plasmid transfer promotes the dissemination of Asian acute hepatopancreatic necrosis disease and provides a novel mechanism for genetic exchange and environmental adaptation[J].mSystems,2020,5(2):e00799-e00719.
[26] 王瑶华,於俊琦,刘亚南,等.浙江省致急性肝胰腺坏死病副溶血弧菌的MLST分型及耐药性分析[C]//2018年(第十三届)浙江渔业科技论坛论文摘要集.温州:浙江省科学技术协会,2018:33-34.
WANG Y H,YU J Q,LIU Y N,et al.Multilocus sequence typing and antimicrobial resistance analysis of Vibrio parahaemolyticus isolated from shrimp with acute hepatopancreatic necrosis disease in Zhejiang[C]//Proceedings of 2018(the thirteenth) Zhejiang fishery science & technology forum.Wenzhou:Zhejiang Association for Science and Technology,2018:33-34.(in Chinese)
[27] 陈伟冰,李柏生,卢向明,等.广东省茂名市副溶血弧菌涉水产品分离株病原学特征[J].中国热带医学,2020,20(4):351-355.
CHEN W B,LI B S,LU X M,et al.Pathogenic characteristics of Vibrio parahaemolyticus strains isolated from aquatic products in Maoming,Guangdong[J].China Tropical Medicine,2020,20(4):351-355.(in Chinese)
[28] HONDA T,IIDA T.The pathogenicity of Vibrio parahaemolyticus and the role of the thermostable direct haemolysin and related haemolysins[J].Reviews in Medical Microbiology,1993,4(2):106-113.
[29] FLORES-PRIMO A,PARD O-SEDAS V,LIZ
O-SEDAS V,LIZ RRAGA-PARTIDA L,et al.Seasonal abundance of total and pathogenic Vibrio parahaemolyticus isolated from American oysters harvested in the Mandinga Lagoon System,Veracruz,Mexico:implications for food safety[J].Journal of Food Protection,2014,77(7):1069-1077.
RRAGA-PARTIDA L,et al.Seasonal abundance of total and pathogenic Vibrio parahaemolyticus isolated from American oysters harvested in the Mandinga Lagoon System,Veracruz,Mexico:implications for food safety[J].Journal of Food Protection,2014,77(7):1069-1077.
[30] SONG X Y,MA Y J,FU J J,et al.Effect of temperature on pathogenic and non-pathogenic Vibrio parahaemolyticus biofilm formation[J].Food Control,2017,73:485-491.
[31] 檀利军,王敬敬,石千黛,等.副溶血性弧菌耐热性直接溶血素(TDH)的研究进展[J].微生物学报,2020,60(8):1563-1573.
TAN L J,WANG J J,SHI Q D,et al.Advances in research on thermostable direct hemolysin (TDH) of Vibrio parahaemolyticus[J].Acta Microbiologica Sinica,2020,60(8):1563-1573.(in Chinese)
[32] 张婷,杨梦华.副溶血弧菌的毒力基因表达调控的分子机制[J].微生物学报,2020,60(7):1345-1357.
ZHANG T,YANG M H.Molecular mechanisms of virulence genes expression in Vibrio parahaemolyticus[J].Acta Microbiologica Sinica,2020,60(7):1345-1357.(in Chinese)
[33] 孙明玉.引起急性肝胰腺坏死病的副溶血性弧菌MLST及耐药性研究[D].上海:上海海洋大学,2018.
SUN M Y.Multilocus sequence typing and antibiotic resistance anlysis of AHPND-causing Vibrio parahaemolyticus[D].Shanghai:Shanghai Ocean University,2018.(in Chinese)
[34] 贾丹.2013—2016年沿海地区虾源副溶血弧菌的特性分析及致病性研究[D].上海:上海海洋大学,2017.
JIA D.Characterization and pathogenicity analysis of Vibrio parahaemolyticus isolated from penaeid shrimps in China(2013—2016)[D].Shanghai:Shanghai Ocean University,2017.(in Chinese)
[35] 陈营,李云冰,张文文,等.PirAB毒力正在向亲缘关系较远的非副溶血弧菌中转移[J].当代水产,2016,41(9):76-77,79.
CHEN Y,LI Y B,ZHANG W W,et al.PirAB virulence is being transferred to distantly related non-V.parahaemolyticus[J].Current Fisheries,2016,41(9):76-77,79.(in Chinese)
[36] ESPEJO R T,GARC A K,PLAZA N.Insight into the origin and evolution of the Vibrio parahaemolyticus Pandemic strain[J].Frontiers in Microbiology,2017,8:1397.
A K,PLAZA N.Insight into the origin and evolution of the Vibrio parahaemolyticus Pandemic strain[J].Frontiers in Microbiology,2017,8:1397.
[37] FU S Z,WANG Q Y,ZHANG Y X,et al.Dynamics and microevolution of Vibrio parahaemolyticus populations in shellfish farms[J].mSystems,2021,6(1):e01161-e01120.
[38] EFIMOV V,DANIN-POLEG Y,RAZ N,et al.Insight into the evolution of Vibrio vulnificus biotype 3′s genome[J].Frontiers in Microbiology,2013,4:393.