鱼类的品种繁多,体色丰富多彩。观赏鱼类的体色主要由遗传基因和环境调控因子共同作用决定[1]。观赏鱼类的皮肤、鳞片大多呈现出独特的表型,不同群体、不同个体、不同性别或不同发育时期,鱼类体色均表现出显著差异[2]。随着经济社会的长期向好和人民生活水平的提高,人们对观赏鱼类的养殖需求日渐旺盛,且对高品质观赏性状的需求越来越高。通常人们肉眼看到的鱼类体色是由体表鳞片和皮肤组织含有的不同色素细胞及不同数量分布形成的。鱼类色素细胞广泛存在着基因组复制的现象,且数量远多于哺乳类和鸟类。目前,已获得成熟研究的鱼类色素细胞有6种,包含黑色素细胞(melanophores)、红色素细胞(erythrophores)、虹彩细胞(iridophores)、黄色素细胞(xanthophores)、蓝色素细胞(cyanophores)和白色素细胞(leucophores)[3]。
自20世纪中后期开始,国内外学者对鱼类体色的变异开展了大量遗传学基础和分子机制研究[4-7]。如Wang等[4]研究了尼罗罗非鱼(Oreochromis niloticus)体色模型,鉴定出其鳞片中包含4种色素细胞,并发现了参与黑素合成、蝶啶代谢、类胡萝卜素吸收和裂解途径的25个突变基因,其中13个突变基因在F0代和F2代均有表型,但在F1代杂合体均无表型。史东杰等[5-6]对红白锦鲤(Cyprinus carpio)和三色锦鲤皮肤组织样本基因表达谱与差异表达基因功能进行了深入研究。徐伟等[7]对黄色、蓝色和红色鲤的遗传特性研究发现,黄色鲤为杂合体,但红色鲤和蓝色鲤为纯合体。鱼类品种繁多,体色多样,为更好地掌握鱼类肤色变异的遗传机制,需在鱼类中进行广泛探索。
鲤广泛分布于淡水水域,对环境耐受能力强,大、小水体均适宜养殖,且具有营养价值和观赏价值,尤其是鲤的一个突变种——锦鲤,具有红、白、黄、黑、紫、蓝等多种色彩或混合色彩,是研究鱼类体色的理想模型。目前,锦鲤产业处于快速上升期,对渔业经济效益提升具有重要贡献,但因其种质高度杂合,定向培育进程缓慢,研究工作处在传统选育和养殖技术阶段,对其体色变异分子机制的研究尚少。遗传信息缺乏是导致符合分级标准的子代商品率较低的“卡脖子”问题。RNA-Seq技术基于高通量测序平台,可快速获取试验特定状态或特定时期下待测样本的基因表达谱,目前已成功应用于多种鱼类组织样品相关基因的转录本预测、分子标记的筛选及调控机制的研究[8]。本研究中,对红色鲤和黄色鲤皮肤转录组进行了测序,分析表达谱特征,对差异表达基因进行功能注释,以期为鱼类体色基因资源的开发提供重要参考。
1 材料与方法
1.1 材料
试验鱼取自北京博兴养殖技术有限公司工厂化温室循环池。实验室暂养2个月后,于2021年4月29日,随机捞取健康状况良好的全红鲤3尾(体长为33.42 cm±0.24 cm)和全黄鲤3尾(体长为38.75 cm±0.43 cm)进行正式试验。
1.2 方法
1.2.1 皮肤总RNA提取 将试验鱼用25 μg/L MS222麻醉后在冰盘上解剖取样,每尾鱼皮肤组织为一个样本,全红鲤皮肤组织样本为R1、R2、R3,全黄鲤皮肤组织样本为Y1、Y2、Y3,每个样本设置3个生物学重复。-80 ℃下保存,用于皮肤总RNA的提取。使用Trizol试剂盒(TaKaRa,北京)对皮肤组织总RNA进行提取,用Nano Photometer分光光度计检测RNA纯度,使用安捷伦2100 RNA Nano 6000 Assay试剂盒(Agilent Technologies,CA,USA)检测RNA的完整性和浓度。
1.2.2 文库构建及测序 全红鲤和全黄鲤皮肤组织cDNA文库构建及转录组测序由安诺优达基因科技(北京)有限公司完成,测序平台为Illumina PE150。总RNA样品使用磁珠富集法进行纯化、筛选和回收,利用随机引物进行逆转录获得cDNA第一条链,通过2nd Strand/End Repair Enzyme mix和2nd Strand Marking Buffer(均购自北京兆仪世纪科技有限公司)合成cDNA第二条链。
1.2.3 数据组装和基因功能注释 对测序raw reads进行过滤,通过去除低质量reads、接头污染碱基数>5 bp的reads及含N>5%的reads等过程,得到干净的高质量序列(clean reads)。使用鲤参考基因组(http://www.fishbrowser.org/database/commoncarp_genome/index.php/home/index/down/oads)进行序列比对,获得单色鲤样本特异序列信息。计算Q30值及饱和度分析,后续分析均使用高质量数据的序列。使用7大数据库对拼接所得的Unigene 序列进行功能注释,包括Nt在线数据库(NCBI Nucleotide Sequences)、Nr数据库(http://www.ncbi.nlm.nih.gov)、COG数据库(Clusters of Orthologous Groups of Proteins)、Pfam数据库(Protein Family)、GO数据库(Gene Ontology)、KEGG数据库(Kyoto Encyclopedia of Genes and Genomes)和Uniprot数据库(Universal Protein)。
1.2.4 基因表达与差异分析 基因表达量通过其转录的mRNA值进行衡量,转录本丰度越高,说明基因表达水平越高。获得的clean reads 序列比对组装好后,参考基因组注释信息,以转录本在样品中的FPKM(Fragments per Kilobaseper Million Map per Fragments)[9]值作为转录本的表达量。通过对不同试验组间进行显著性差异基因的筛选,可获得差异表达的基因,并区分上调基因和下调基因。使用DEseq[10]软件进行差异表达分析,以|log2(fold change)|≥1和q<0.005作为筛选阈值。使用Goseq和KOBAS软件分别对差异基因进行富集分析(corrected P value<0.05)。差异表达基因通过GO数据库进行聚类分析,通过KEGG数据库进行通路富集分析。采用Samtools软件对全红鲤、全黄鲤皮肤转录组拼接获得的所有基因进行单核苷酸多态性标记(single nucleotide polymorphism,SNP)检测。
1.2.5 qRT-PCR验证 从转录组数据分析结果中随机选取差异表达基因9个,进行qRT-PCR检测(表1)。选择转录组相同批次的RNA经过反转录获得 cDNA,每个样本设置3个重复试验。使用鲤β-actin[11]在荧光定量PCR仪(ABI,Stepone)进行qRT-PCR验证。反应体系(15 μL):2×SYBR Premix Ex Taq Ⅱ 7.5 μL,上、下游引物各0.6 μL,LcDNA 1.5 μL,ddH2O 4.8 μL。反应程序:95 ℃下预变性150 s;95 ℃下变性5 s,58 ℃下退火35 s,72 ℃下延伸30 s,共进行40个循环。采用 2-ΔΔCt法计算基因相对表达量。
表1 qRT-PCR引物序列信息
Tab.1 Primer sequence information required for qRT-PCR
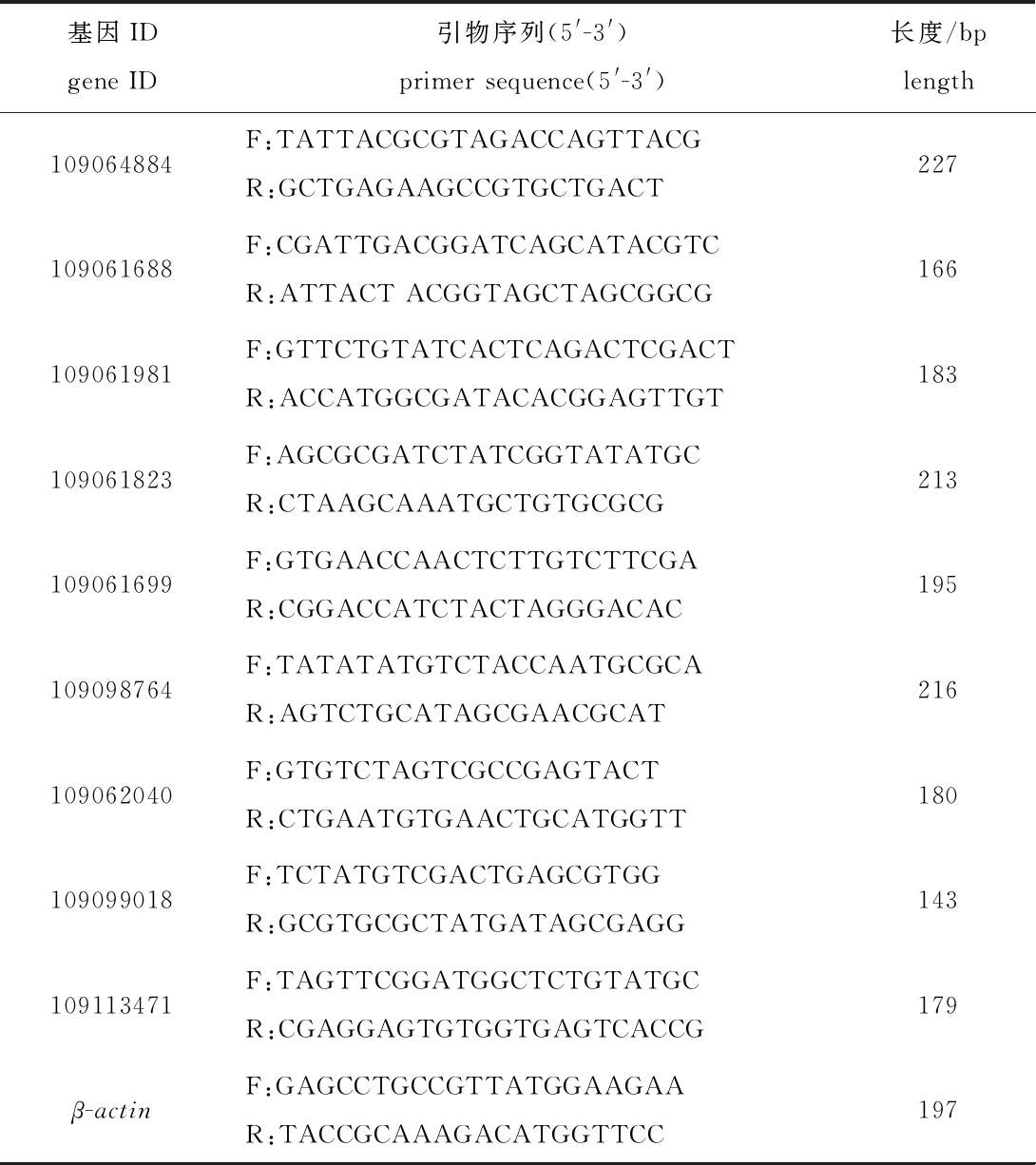
基因ID gene ID引物序列(5'-3')primer sequence(5'-3')长度/bplength109064884F:TATTACGCGTAGACCAGTTACGR:GCTGAGAAGCCGTGCTGACT227109061688F:CGATTGACGGATCAGCATACGTCR:ATTACT ACGGTAGCTAGCGGCG166109061981F:GTTCTGTATCACTCAGACTCGACTR:ACCATGGCGATACACGGAGTTGT183109061823F:AGCGCGATCTATCGGTATATGCR:CTAAGCAAATGCTGTGCGCG213109061699F:GTGAACCAACTCTTGTCTTCGAR:CGGACCATCTACTAGGGACAC195109098764F:TATATATGTCTACCAATGCGCAR:AGTCTGCATAGCGAACGCAT216109062040F:GTGTCTAGTCGCCGAGTACTR:CTGAATGTGAACTGCATGGTT180109099018F:TCTATGTCGACTGAGCGTGGR:GCGTGCGCTATGATAGCGAGG143109113471F:TAGTTCGGATGGCTCTGTATGCR:CGAGGAGTGTGGTGAGTCACCG179β-actinF:GAGCCTGCCGTTATGGAAGAAR:TACCGCAAAGACATGGTTCC197
1.3 数据处理
采用SPSS 17.0软件进行差异分析,差异显著性水平设为0.05。
2 结果与分析
2.1 转录组测序质控和组装
通过提取全红鲤和全黄鲤皮肤组织样本的总RNA,分别构建了全红和全黄皮肤cDNA文库。Illumina平台测序结果显示:3个全红鲤和全黄鲤样品中,分别有133 227 070个和134 924 280个raw reads,129 222 850个和130 869 572 个clean reads;所有净数据比率均大于96%,Q30值均大于93%(表2),说明组装与测序质量较好,可用于后续分析。
表2 样品测序数据质量
Tab.2 Sample sequencing data quality
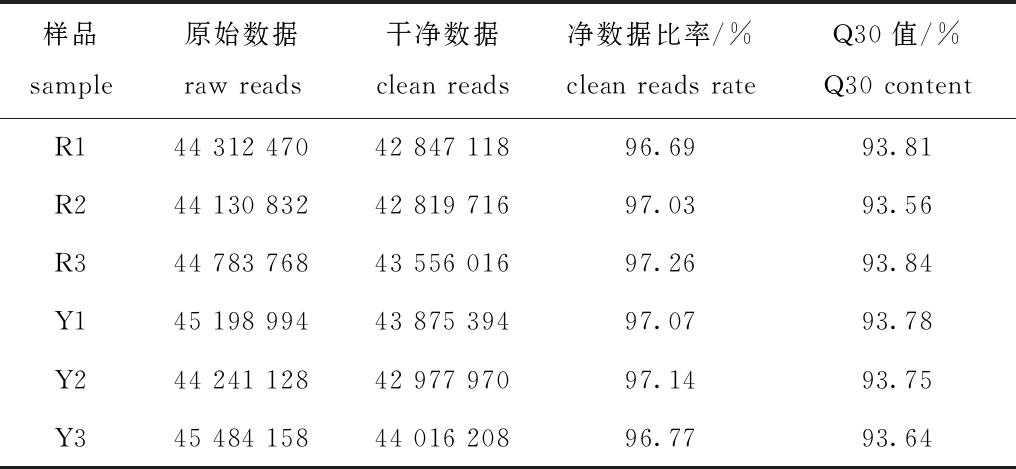
样品 sample原始数据 raw reads干净数据 clean reads净数据比率/% clean reads rateQ30值/% Q30 contentR144 312 47042 847 11896.6993.81R244 130 83242 819 71697.0393.56R344 783 76843 556 01697.2693.84Y145 198 99443 875 39497.0793.78Y244 241 12842 977 97097.1493.75Y345 484 15844 016 20896.7793.64
2.2 功能注释和分类
在Nt、Nr、Pfam、Uniprot、COG、GO和KEGG这7大公共数据库中对获得的61 491个genes进行搜索和功能注释。其中,通过Nt数据库和Pfam数据库比对的单基因数最高,分别为61 490个和61 385个,占比分别为99.99%和99.83%;其次是58 782个单基因匹配至Nr数据库,占比为95.59%;通过COG数据库比对成功的单基因数最少,为11 468个,占比最低(18.65%);在Nr数据库中比对发现,有56.48%(34 729个)的单基因与鲤高度同源,有28.29%(17 394个)的单基因与斑马鱼高度同源,但也发现有15.23%的单基因未能与鲤或斑马鱼比对成功;所有genes均至少注释到一个数据库,但全部数据库都获得注释的基因数为8 378个,占总基因数的13.62%(表3)。
表3 基因功能注释统计
Tab.3 New gene function annotation
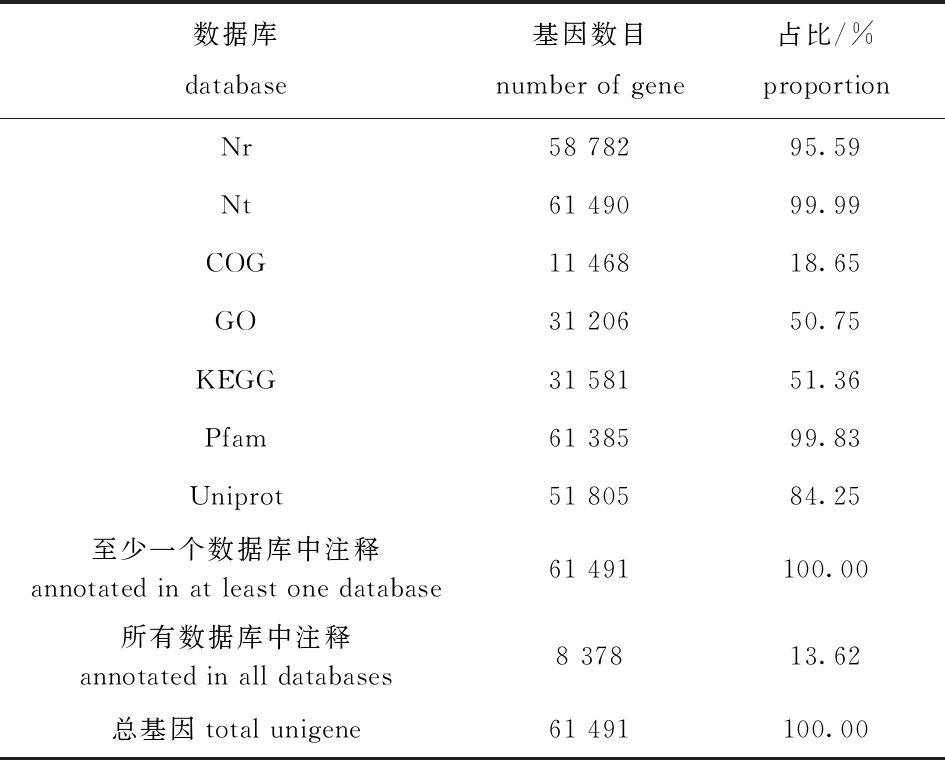
数据库database基因数目number of gene占比/%proportionNr58 78295.59Nt61 49099.99COG11 46818.65GO31 20650.75KEGG31 58151.36Pfam61 38599.83Uniprot51 80584.25至少一个数据库中注释annotated in at least one database61 491100.00所有数据库中注释annotated in all databases8 37813.62总基因total unigene61 491100.00
2.3 差异表达基因的GO富集分析
共获得全红鲤和全黄鲤皮肤组织差异表达基因4 822个,其中有1 929个显著上调,2 893个显著下调(图1)。多个与鱼类黑色素合成、红色素合成、黄色素合成、酪氨酸代谢及喋啶合成相关的基因(mitfa、ednrA、wnt7、agouti和mif等)被鉴定(表4)。
表4 部分体色相关差异表达基因分析
Tab.4 Analysis of some differentially expressed genes
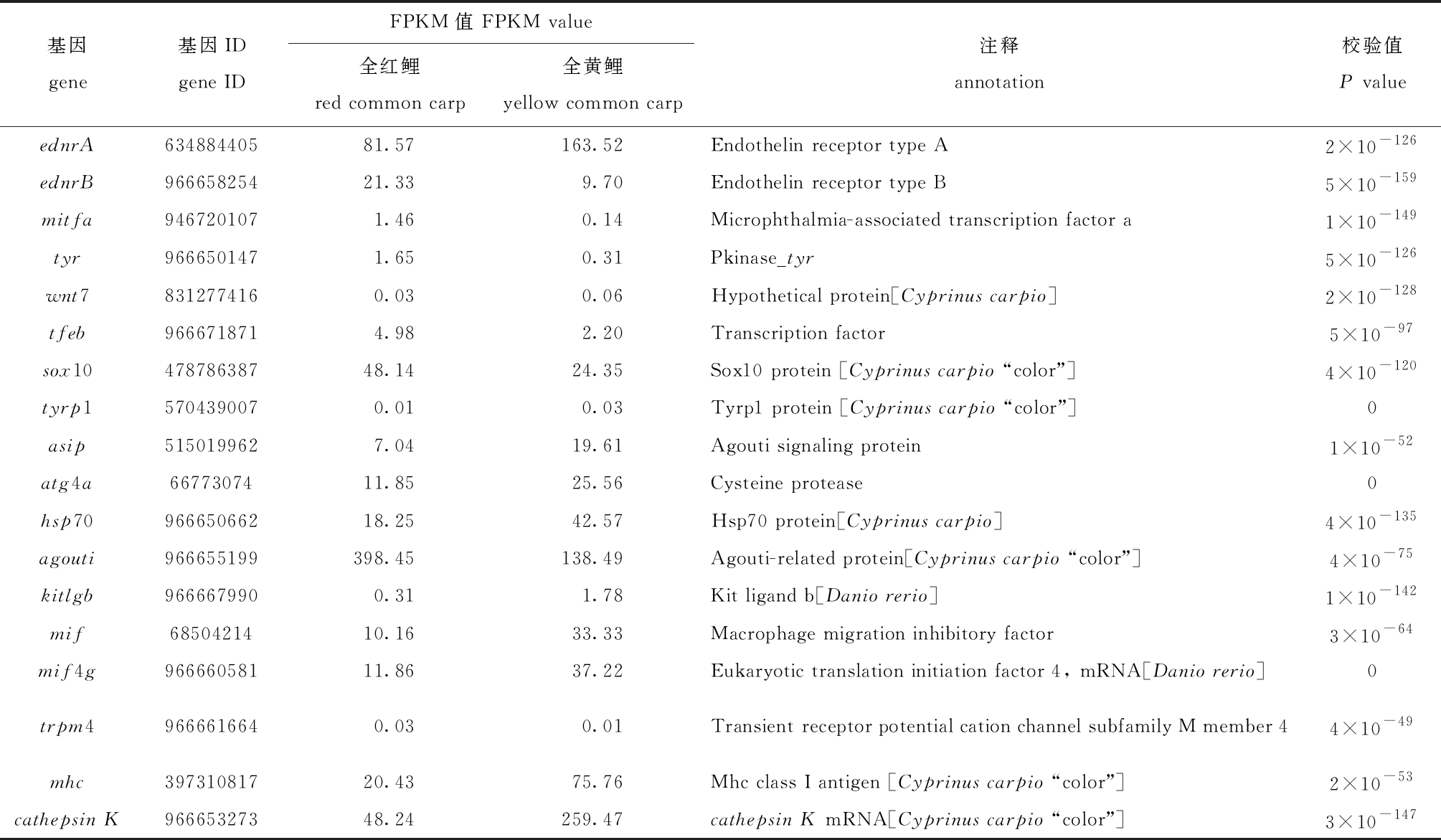
基因gene基因IDgene IDFPKM值 FPKM value全红鲤red common carp全黄鲤yellow common carp注释annotation校验值P valueednrA63488440581.57163.52Endothelin receptor type A2×10-126ednrB96665825421.339.70Endothelin receptor type B5×10-159mitfa9467201071.460.14Microphthalmia-associated transcription factor a1×10-149tyr9666501471.650.31Pkinase_tyr5×10-126wnt78312774160.030.06Hypothetical protein[Cyprinus carpio]2×10-128tfeb9666718714.982.20Transcription factor 5×10-97sox1047878638748.1424.35Sox10 protein [Cyprinus carpio “color”]4×10-120tyrp15704390070.010.03Tyrp1 protein [Cyprinus carpio “color”]0asip5150199627.0419.61Agouti signaling protein1×10-52atg4a6677307411.8525.56Cysteine protease0hsp7096665066218.2542.57Hsp70 protein[Cyprinus carpio]4×10-135agouti966655199398.45138.49Agouti-related protein[Cyprinus carpio “color”]4×10-75kitlgb9666679900.311.78Kit ligand b[Danio rerio] 1×10-142mif6850421410.1633.33Macrophage migration inhibitory factor3×10-64mif4g96666058111.8637.22 Eukaryotic translation initiation factor 4, mRNA[Danio rerio]0trpm49666616640.030.01Transient receptor potential cation channel subfamily M member 44×10-49mhc39731081720.4375.76Mhc class I antigen [Cyprinus carpio “color”]2×10-53cathepsin K96665327348.24259.47cathepsin K mRNA[Cyprinus carpio “color”] 3×10-147

图1 全红鲤和全黄鲤皮肤组织差异表达基因火山图
Fig.1 Volcano map of different transcriptome genes in skin of red common carp and yellow common carp
对差异表达基因GO进行功能注释分析,在细胞组分方面,大部分富集在细胞部分、细胞器、细胞器部分、膜和膜部分等;在生物学过程中,主要注释在生物调节、细胞进程、代谢过程和发育过程等;在分子功能方面,集中富集在结合、催化活性、分子功能活性、分子传感调控和信号转导活性等(表5)。
表5 差异表达基因的GO数据库功能注释
Tab.5 GO assignment of assembled different transcriptome genes
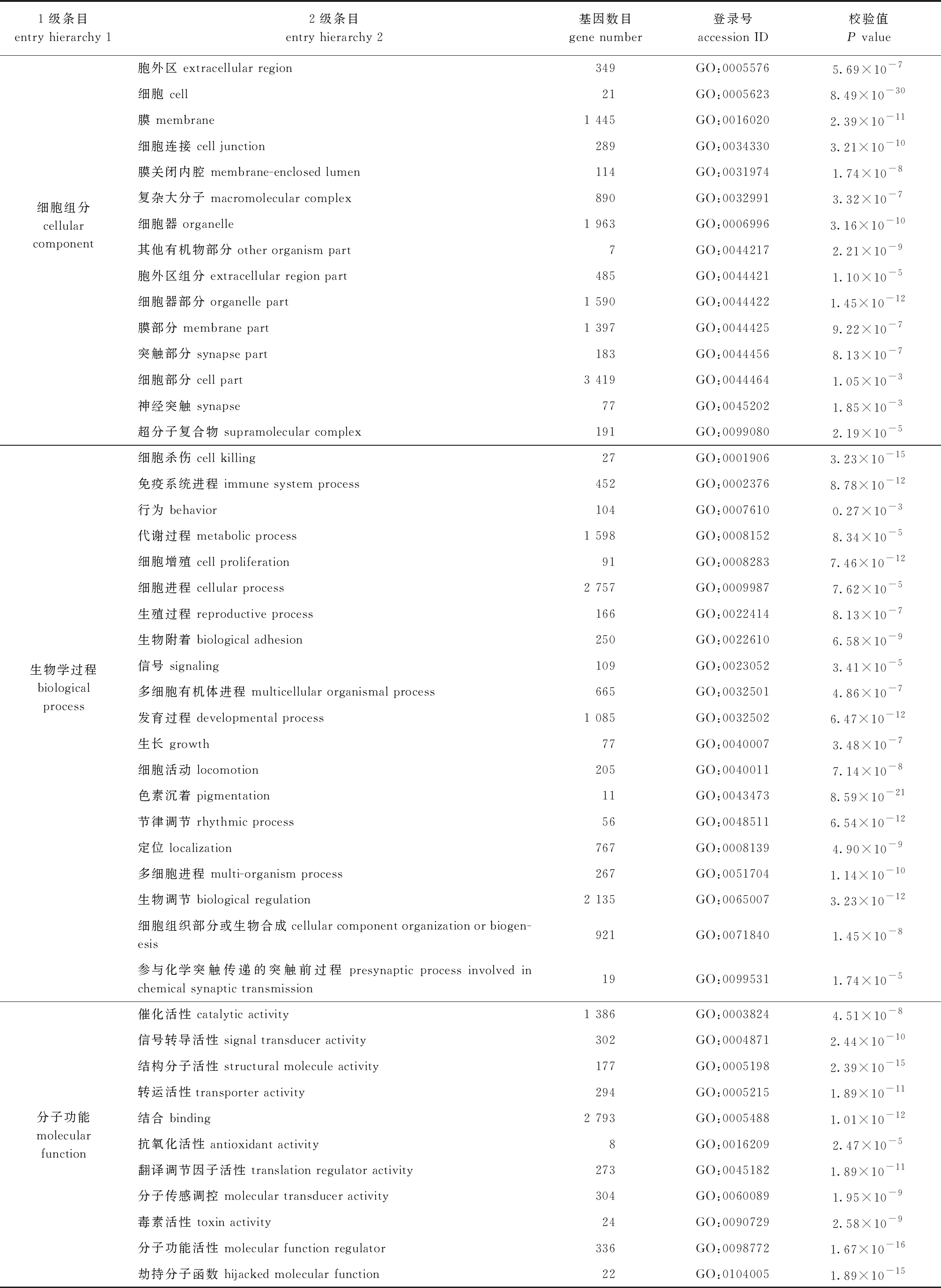
1级条目entry hierarchy 12级条目entry hierarchy 2 基因数目gene number 登录号accession ID校验值P value细胞组分cellular component胞外区 extracellular region349GO:00055765.69×10-7细胞 cell21GO:00056238.49×10-30膜 membrane1 445GO:00160202.39×10-11细胞连接 cell junction289GO:00343303.21×10-10膜关闭内腔 membrane-enclosed lumen114GO:00319741.74×10-8复杂大分子 macromolecular complex890GO:00329913.32×10-7细胞器 organelle1 963GO:00069963.16×10-10其他有机物部分 other organism part7GO:00442172.21×10-9胞外区组分 extracellular region part485GO:00444211.10×10-5细胞器部分 organelle part1 590GO:00444221.45×10-12膜部分 membrane part1 397GO:00444259.22×10-7突触部分 synapse part183GO:00444568.13×10-7细胞部分 cell part3 419GO:00444641.05×10-3神经突触 synapse77GO:00452021.85×10-3超分子复合物 supramolecular complex191GO:00990802.19×10-5生物学过程biological process细胞杀伤 cell killing27GO:00019063.23×10-15免疫系统进程 immune system process452GO:00023768.78×10-12行为 behavior104GO:00076100.27×10-3代谢过程 metabolic process1 598GO:00081528.34×10-5细胞增殖 cell proliferation91GO:00082837.46×10-12细胞进程 cellular process2 757GO:00099877.62×10-5生殖过程 reproductive process166GO:00224148.13×10-7生物附着 biological adhesion250GO:00226106.58×10-9信号 signaling109GO:00230523.41×10-5多细胞有机体进程 multicellular organismal process665GO:00325014.86×10-7发育过程 developmental process1 085GO:00325026.47×10-12生长 growth77GO:00400073.48×10-7细胞活动 locomotion205GO:00400117.14×10-8色素沉着 pigmentation11GO:00434738.59×10-21节律调节 rhythmic process56GO:00485116.54×10-12定位 localization767GO:00081394.90×10-9多细胞进程 multi-organism process267GO:00517041.14×10-10生物调节 biological regulation2 135GO:00650073.23×10-12细胞组织部分或生物合成 cellular component organization or biogen-esis921GO:00718401.45×10-8参与化学突触传递的突触前过程 presynaptic process involved in chemical synaptic transmission19GO:00995311.74×10-5分子功能molecular function催化活性 catalytic activity1 386GO:00038244.51×10-8信号转导活性 signal transducer activity302GO:00048712.44×10-10结构分子活性 structural molecule activity177GO:00051982.39×10-15转运活性transporter activity294GO:00052151.89×10-11结合 binding2 793GO:00054881.01×10-12抗氧化活性 antioxidant activity8GO:00162092.47×10-5翻译调节因子活性 translation regulator activity273GO:00451821.89×10-11分子传感调控 molecular transducer activity304GO:00600891.95×10-9毒素活性 toxin activity24GO:00907292.58×10-9分子功能活性 molecular function regulator336GO:00987721.67×10-16劫持分子函数 hijacked molecular function22GO:01040051.89×10-15
2.4 差异表达基因的KEGG通路富集分析
对所有差异表达基因进行KEGG通路富集分析,全红鲤和全黄鲤皮肤差异表达基因主要在323个KEGG通路中富集,其中显著富集到67个通路;最显著富集的有甘露糖与果糖代谢、糖酵解/糖原异生途径、碳代谢、戊糖磷酸途径、PPAR信号通路、脯氨酸与精氨酸代谢、氨基酸的生物合成、肥厚型心肌病、扩张性心肌病、脂肪酸降解、细胞外基质受体相互作用、色氨酸代谢、脂肪酸代谢和酪氨酸代谢等43条代谢通路,部分极显著富集通路见表6。另外,通过对KEGG富集获得的酪氨酸代谢和黑色素合成通路分析发现,mif、mif4g、agouti、ednrA和ednrB等与红色素和黄色素合成相关的基因显著上调,而tyr、tyrp1、mitfa和wnt7等与黑色素合成相关的基因显著下调。
表6 全红鲤和全黄鲤皮肤组织差异表达基因KEGG极显著富集前23条通路
Tab.6 Analysis of top 23 KEGG enrichment pathways of different transcriptome genes in skin of red common carp and yellow common carp
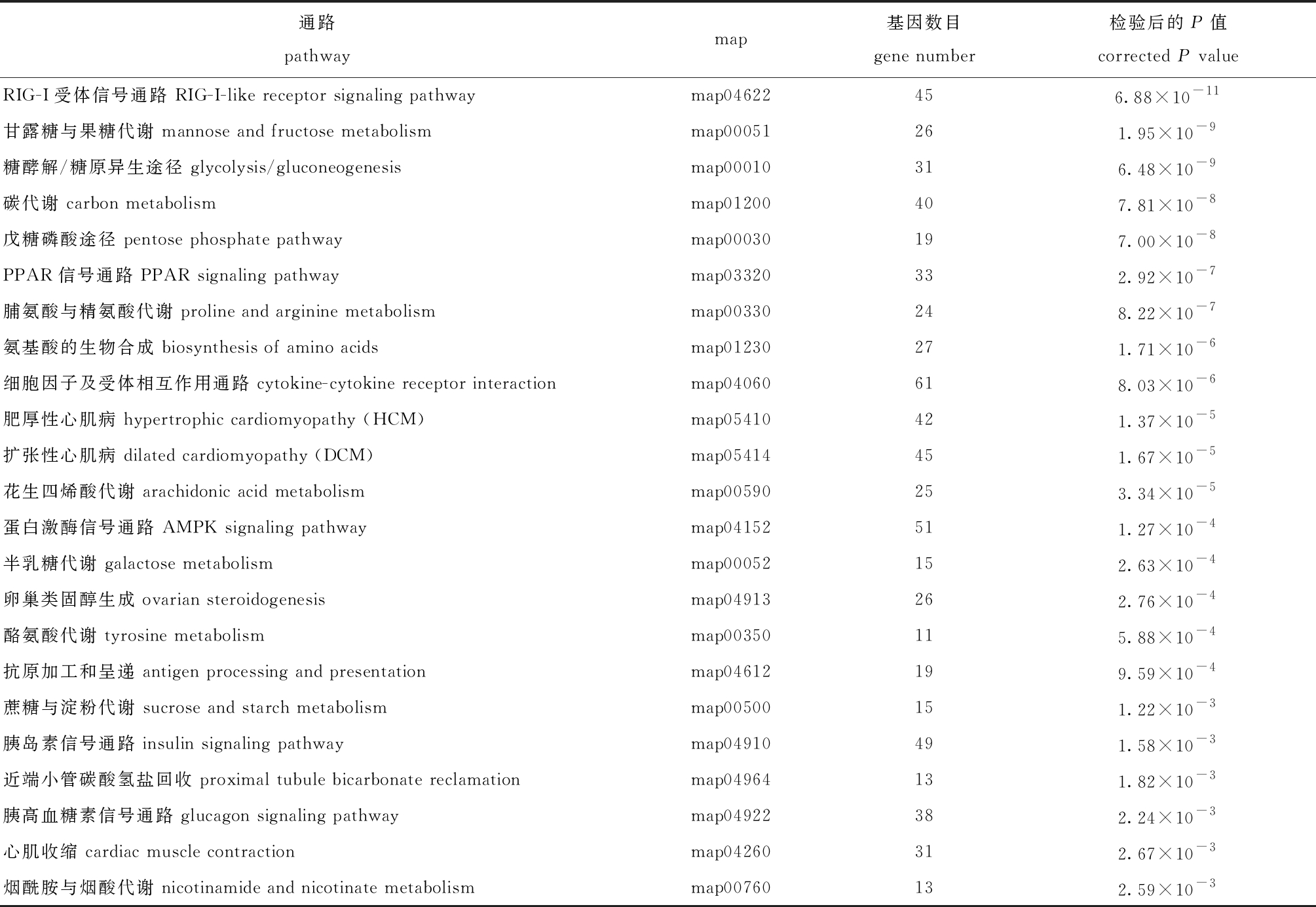
通路 pathwaymap基因数目 gene number检验后的P值 corrected P valueRIG-I受体信号通路 RIG-I-like receptor signaling pathwaymap04622456.88×10-11甘露糖与果糖代谢 mannose and fructose metabolismmap00051261.95×10-9糖酵解/糖原异生途径 glycolysis/gluconeogenesismap00010316.48×10-9碳代谢 carbon metabolismmap01200407.81×10-8戊糖磷酸途径 pentose phosphate pathwaymap00030197.00×10-8PPAR信号通路 PPAR signaling pathwaymap03320332.92×10-7脯氨酸与精氨酸代谢 proline and arginine metabolismmap00330248.22×10-7氨基酸的生物合成 biosynthesis of amino acidsmap01230271.71×10-6细胞因子及受体相互作用通路 cytokine-cytokine receptor interactionmap04060618.03×10-6肥厚性心肌病 hypertrophic cardiomyopathy (HCM)map05410421.37×10-5扩张性心肌病 dilated cardiomyopathy (DCM)map05414451.67×10-5花生四烯酸代谢 arachidonic acid metabolismmap00590253.34×10-5蛋白激酶信号通路 AMPK signaling pathwaymap04152511.27×10-4半乳糖代谢 galactose metabolismmap00052152.63×10-4卵巢类固醇生成 ovarian steroidogenesismap04913262.76×10-4酪氨酸代谢 tyrosine metabolismmap00350115.88×10-4抗原加工和呈递 antigen processing and presentationmap04612199.59×10-4蔗糖与淀粉代谢 sucrose and starch metabolismmap00500151.22×10-3胰岛素信号通路 insulin signaling pathwaymap04910491.58×10-3近端小管碳酸氢盐回收 proximal tubule bicarbonate reclamationmap04964131.82×10-3胰高血糖素信号通路 glucagon signaling pathwaymap04922382.24×10-3心肌收缩 cardiac muscle contractionmap04260312.67×10-3烟酰胺与烟酸代谢 nicotinamide and nicotinate metabolismmap00760132.59×10-3
2.5 qRT-PCR验证
用qRT-PCR检测转录组数据中9个差异表达基因,其结果与转录组测得数据的结果一致,说明转录组结果真实可靠(图2)。

FC—差异倍数; RQ—相关定量。FC—fold change; RQ—relative quantification.
图2 qRT-PCR检测结果
Fig.2 Validation of data using qRT-PCR
2.6 SNP检测
从表7可见:全红鲤和全黄鲤中均存在SNP变异位点,共预测到388 673个,发生频率为1/(426 bp),表示平均每426 bp核酸片段有1个变异位点;在SNP位点中,碱基转换位点为256 325个,碱基颠换位点为132 348个,转换颠换比为1.94;6个SNP变异类型中,C/T的发生频率最高,占SNP位点总数的34.14%,其次是A/G,占SNP位点总数的31.81%,其他A/T、A/C、T/G和C/G 4种类型分别占比为9.97%、8.97%、6.83%和8.28%。
表7 碱基替换情况
Tab.7 Summary of the base substitution
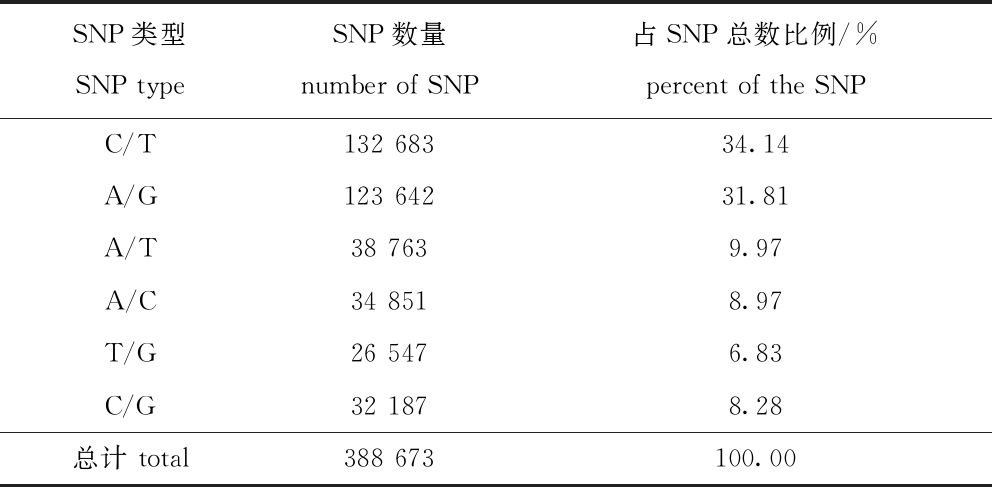
SNP类型SNP typeSNP数量number of SNP占SNP总数比例/%percent of the SNPC/T132 68334.14A/G123 64231.81A/T38 7639.97A/C34 8518.97T/G26 5476.83C/G32 1878.28总计 total388 673100.00
3 讨论
体色是动物伪装逃避敌害、信号交流及求偶繁衍的重要性状,其主要由体表皮肤所含色素细胞的颗粒种类、形态、结构、数量及分布所决定的,色素细胞形成过程又受多种酶、生理刺激物和基因等共同调控。尽管人们对昆虫、斑马鱼(Danio rerio)等体色形成机制已基本了解,但不同生物体在体色形成上有较大不同,而对于动物体色形成机理和代谢规律的认识还较缺乏。鲤具有丰富多彩的颜色,是研究鱼类体色的良好试验材料。然而,有关鲤体色分子形成机制的科学问题目前还所知甚少。
3.1 单色鲤转录组组装与注释
本研究中通过转录组测序分析发现,全红鲤和全黄鲤皮肤组织中至少含有4 822个显著差异表达基因。通过 qRT-PCR对9个基因进行检测,证实了转录组数据结果的可靠性。在Nr数据库中比对和注释的结果显示,全红鲤和全黄鲤转录组数据可比对到58 782条,占总基因数的95.59%,说明全部基因基本可以与已知基因比对成功。在Nr数据库中比对,有56.48%(34 729个)的单基因与鲤基因高度一致,有28.29%(17 394个)的单基因与斑马鱼高度一致,分析原因可能是全红鲤、全黄鲤、斑马鱼和鲤同属于鲤形目(Cypriniformes)鲤科(Cyprinidae)。但也发现有15.23%的单基因未能与鲤或斑马鱼比对成功,这可能是因为全红鲤和全黄鲤属于观赏鱼类,是经自然条件和人工选育获得的鲤的变种,基因高度杂合,而Nr数据库中鲤基因组数据全部来自食用鲤,这部分未能成功比对上的基因可能是鲤基因间的序列,或者是lncRNA,或者是重复序列的表达产物。
3.2 单色鲤体色相关基因及通路
鱼类具有黑色素细胞、红色素细胞、虹彩细胞、黄色素细胞、蓝色素细胞和白色素细胞等6种色素细胞,每种色素细胞含有不同的色素颗粒,色素颗粒的颜色、含量、迁移过程及反光作用,均可使鱼体呈现出多种颜色。田雪等[12]研究表明,mitfa和tyr基因在红色锦鲤体色发生不同阶段的表达水平呈现降低趋势,具有一定量的表达,这与本研究中全红鲤和全黄鲤检测出mitfa和tyr基因表达量的结果一致。Liu等[13]在黄金锦鲤中检测到mitfa基因表达量;Parichy等[14]在瓯江彩鲤(Cyprinus carpio var.)、纯色及黑色板块锦鲤皮肤中检测到tyr表达量。上述研究说明,mitfa和tyr基因不仅在黑色细胞中起作用,也在其他色素细胞中发挥部分功能,但具体作用途径有待进一步研究。田雪等[12]通过观察1~48日龄红色锦鲤体色发生过程,发现1日龄红色锦鲤鱼体表现为无色透明,随后鱼体颜色逐渐增黑,48日龄鱼体呈现红色,可见锦鲤红色皮肤与黑色皮肤退去有密切关系。
动物体色形成机制以哺乳动物和鸟类的研究较多,主要集中在色素调控通路、色素细胞迁移及相互作用等方面,其中,以黑色素合成与运输的分子调控网络的研究最为成熟。研究发现,哺乳动物黑色素合成通路极为复杂且十分保守[15]。黑色素主要在黑色素小体中的膜结构细胞器合成,并沉着在动物的皮毛和眼睛,使其呈现出颜色。黑色素存在褐黑素和真黑素两种类型,只有真黑素影响黑色和褐色的表型,而褐黑素则控制形成红色、黄色和棕色。动物皮毛和眼睛呈现的颜色主要取决于以上两种色素的分布数量与比例[16]。Rees[17]研究发现,黑素皮质素受体1(mc1r)基因由扩展位点(extension locus,E)编码,为G蛋白耦合受体-黑素皮质素受体(mcrs)家族成员中最小的一个。Lu等[18]在信号蛋白ASP纯化过程中发现,小鼠(Mus musculus)的刺鼠信号蛋白基因agouti与多黏菌素耐药基因MCR高度亲和。Kobayash等[19]研究agouti重组体在毛发中引起优黑色素合成褐黑素的过程中,对部分已报道的黑色素合成相关基因具有下调作用,如tyr、trp和mitf等。Millar等[20]研究发现,转基因小鼠的表皮细胞表达的agouti基因可使皮毛呈现黄色,并提示agouti基因通过旁分泌途径对鼠的皮毛进行肤色调控。进一步的研究表明,agouti相关蛋白在大鲵(Andrias davidianus)[21]、虹鳟(Oncorhynchus mykiss)[22]和瓯江彩鲤[23]的皮肤组织中相对表达量较高,且在黄色或红色皮肤中的相对表达量明显高于其他肤色,并具有保守性。本试验中,转录组数据显示,agouti基因在全红鲤皮肤(FPKM=245.07)和全黄鲤皮肤(FPKM=138.49)中均处于高表达水平,而黑色素合成过程中tyr、tyrp1、mitfa和wnt7等关键基因的FPKM值均处于较低的表达水平。由此推测,鲤agouti基因的高表达可拮抗黑色素形成过程中相关基因的表达,抑制黑色素细胞内cAMP水平的升高,使黑色素细胞从其合成的优黑色素转向褐黑素合成,从而增加了褐黑素的数量与比例,与鱼体呈现红色或者黄色有一定的关系。
已有研究发现,两栖动物和硬骨鱼类的黑化抑制因子(mif)可抑制腹部皮肤黑色素细胞发育并导致反噬[20,22]。本研究中,对全红鲤和全黄鲤差异表达基因的KEGG极显著富集的酪氨酸代谢通路进一步分析发现,mif基因在全红和全黄皮肤组织中的FPKM值分别为187.32和138.22,呈现高表达且上调,试验还获得mif的一个多肽表征基因mif4g,推测该基因的高表达与鲤皮肤呈现红色或者黄色有关。迁移肿瘤干细胞McSCs中的糖蛋白wnt信号被认为是由表皮干细胞EpScs分泌的wnt配体产生[24],当wnt配体与其受体结合,β-catenin转移到细胞核,并与转录因子TCF/LEF形成复合物,进而诱导McSCs中色素产生关键基因酪氨酸tyr和小眼畸形相关转录因子mitf的表达[25]。对小鼠的研究发现,皮内注射wnt抑制剂IWR-1、β-catenin激活抑制剂及原癌基因wnt7a干扰siRNA,可阻碍表皮McSCs数量的增加[26]。皮质素(edn)在促进McSCs增殖和分化中具有重要作用,但其只有在活性wnt信号激活下,才能发挥作用[27]。为了研究wnt7、ednrA/ednrB的表达模式是否与小鼠的研究结论一致,本研究中对这3个基因进行定量分析发现,在全红鲤和全黄鲤皮肤中尽管ednrA/ednrB处于高表达,FPKM值分别为81.57/163.52和21.33/9.70,但wnt7表达量极低,FPKM值只有0.03和0.06,并相对下调,低表达的wnt7无法激活McSCs的增殖与分化,这也进一步丰富了全红鲤和全黄鲤体色呈现红色和黄色的分子机制。
3.3 单色鲤的SNP
本研究中,单色鲤转录组SNP的发生频率[1/(426 bp)]高于大黄鱼(Larimichthys crocea)[27][1/(506 bp)]和波纹唇鱼(Cheilinus undulatus)[28][1/(490 bp)],低于美洲牡蛎(Crassostrea virginica)[29][1/(60 bp)];单色鲤SNP转换比例明显高于颠换,这与对波纹唇鱼、大黄鱼、黄尾鲴(Xenocypris dacidi)[30]和大口黑鲈(Micropterus salmoides)[31]等的研究结果相似,Zhao等[32]认为,出现这种情况可能与鱼类进化中所承受的选择压力不同有关。此外,单色鲤SNP转换变异中,C/T比A/G的比例高2.33%,这种结果可能与SNPs在CG序列上出现的频率比AG高密切相关,单色鲤高比例的C/T类型变异,或许更有助于在分子水平上开展重要经济性状分析及品质改良的研究。
4 结论
1)本研究构建了两种鲤皮肤的cDNA文库,通过转录组测序对全红和全黄两种单色鲤皮肤mRNA表达进行比较分析,共检测到4 822个差异表达基因,对差异表达基因进行GO功能分类,并从差异表达基因中检测到388 673个SNP标记。
2)KEGG通路分析发现,酪氨酸代谢通路和黑色素合成通路中,与黑色素合成相关的基因tyr、tyrp1、mitf和wnt7等显著下调,与红色素和黄色素合成相关的基因mif、mif4g、agouti和ednr等显著上调。推测全红和全黄两种体色是由黑色素退掉或被抑制有关,且受多个基因共同作用,其中,tyr、tyrp1、mitfa和wnt7等基因低表达是体色呈红色或黄色的直接因素,而mif、mif4g、agouti和ednr等基因高表达是体色呈红或黄的重要因素。本研究结果可为今后锦鲤分子标记的开发及功能基因的克隆、编辑和功能分析等提供基础数据。
[1] NILSSON SKÖLD H,ASPENGREN S,WALLIN M.Rapid color change in fish and amphibians—function,regulation,and emerging applications[J].Pigment Cell & Melanoma Research,2013,26(1):29-38.
[2] 王成辉.鱼类体色变异的遗传基础研究进展简述[J].上海海洋大学学报,2012,21(5):737-742.
WANG C H.Brief summary on genetic basis of pigmentation in fish[J].Journal of Shanghai Ocean University,2012,21(5):737-742.(in Chinese)
[3] PARICHY D M.Evolution of pigment cells and patterns:recent insights from teleost fishes[J].Current Opinion in Genetics & Development,2021,69:88-96.
[4] WANG C X,LU B Y,LI T,et al.Nile tilapia:a model for studying teleost color patterns[J].Journal of Heredity,2021,112(5):469-484.
[5] 史东杰,陈晓璇,魏东,等.三色锦鲤皮肤转录组测序与功能分析[J].基因组学与应用生物学,2021,40(1):117-127.
SHI D J,CHEN X X,WEI D,et al.The transcriptome squencing and functional analysis of skin tissue of tricolor Cyprinus carpio[J].Genomics and Applied Biology,2021,40(1):117-127.(in Chinese)
[6] 史东杰,胡金有,王赛赛,等.红白锦鲤皮肤转录组测序分析[J].大连海洋大学学报,2019,34(4):475-481.
SHI D J,HU J Y,WANG S S,et al.Transcriptome analysis of skin of red and white koi carp Cyprinus carpio haematopterus[J].Journal of Dalian Ocean University,2019,34(4):475-481.(in Chinese)
[7] 徐伟,李池陶,曹顶臣,等.黄色鲤、蓝色鲤、红色鲤杂交的体色及鳞被遗传特性[J].动物学杂志,2010,45(5):9-17.
XU W,LI C T,CAO D C,et al.Body color and scale type in progenies from different cross combinations among yellow carp,blue carp and red carp[J].Chinese Journal of Zoology,2010,45(5):9-17.(in Chinese)
[8] 董忠典,黎学友,黄承勤,等.湖栖鳍虾虎鱼性腺转录组比较分析[J].水产学报,2021,45(3):365-380.
DONG Z D,LI X Y,HUANG C Q,et al.Comparative transcriptome analysis of the gonad of the lacustrine goby(Gobiopterus lacustris)[J].Journal of Fisheries of China,2021,45(3):365-380.(in Chinese)
[9] TRAPNELL C,WILLIAMS B A,PERTEA G,et al.Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J].Nature Biotechnology,2010,28(5):511-515.
[10] ANDERS S,HUBER W.Differential expression analysis for sequence count data[J].Genome Biology,2010,11(10):R106.
[11] MURAKAMI M,OHI M,ISHIKAWA S,et al.Adaptive expression of uncoupling protein 1 in the carp liver and kidney in response to changes in ambient temperature[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2015,185:142-149.
[12] 田雪,庞小磊,王良炎,等.MITFa及TYR基因在红色锦鲤体色发生不同阶段的表达分析[J].水产科学,2017,36(2):197-201.
TIAN X,PANG X L,WANG L Y,et al.Expression of MITFa and TYR gene in body color formation in red color koi carp Cyprinus carpio at different stages[J].Fisheries Science,2017,36(2):197-201.(in Chinese)
[13] LIU J H,WEN S,LUO C,et al.Involvement of the mitfa gene in the development of pigment cell in Japanese ornamental (Koi) carp (Cyprinus carpio L.)[J].Genetics and Molecular Research:GMR,2015,14(1):2775-2784.
[14] PARICHY D M.Evolution of danio pigment pattern development[J].Heredity,2006,97(3):200-210.
[15] YU F F,LU Y S,ZHONG Z M,et al.Mitf involved in innate immunity by activating tyrosinase-mediated melanin synthesis in Pteria penguin[J].Frontiers in Immunology,2021,12:626493.
[16] HALLSSON J H,HAFLIDAD TTIR B S,SCHEPSKY A,et al.Evolutionary sequence comparison of the Mitf gene reveals novel conserved domains[J].Pigment Cell Research,2007,20(3):185-200.
TTIR B S,SCHEPSKY A,et al.Evolutionary sequence comparison of the Mitf gene reveals novel conserved domains[J].Pigment Cell Research,2007,20(3):185-200.
[17] REES J L.Genetics of hair and skin color[J].Annual Review of Genetics,2003,37:67-90.
[18] LU D S,WILLARD D,PATEL I R,et al.Agouti protein is an antagonist of the melanocyte-stimulating-hormone receptor[J].Nature,1994,371(6500):799-802.
[19] KOBAYASHI T,VIEIRA W D,POTTERF B,et al.Modulation of melanogenic protein expression during the switch from eu- to pheomelanogenesis[J].Journal of Cell Science,1995,108 ( Pt 6):2301-2309.
[20] MILLAR S E,MILLER M W,STEVENS M E,et al.Expression and transgenic studies of the mouse agouti gene provide insight into the mechanisms by which mammalian coat color patterns are generated[J].Development,1995,121(10):3223-3232.
[21] 姜维,邓捷,王启军,等.大鲵Agouti基因的克隆、表达及多态性分析[J].四川动物,2019,38(2):121-129.
JIANG W,DENG J,WANG Q J,et al.Cloning and polymorphism of Agouti gene and its expression in different skin color phenotypes in Andrias davidianus[J].Sichuan Journal of Zoology,2019,38(2):121-129.(in Chinese)
[22] YOON J M.Characterization and tissues distribution of vinculin,Agouti-relating protein and melanocortin 4 receptor genes in rainbow trout,Oncorhynchus mykiss[J].Development & Reproduction,2010,14(4):261-268.
[23] 李康乐.瓯江彩鲤体色相关基因Sox10、Agouti、Tyrp1、Dct的分子克隆与表达分析[D].上海:上海海洋大学,2014.
LI K L.Molecular cloning and expression analysis of pigmentation-related genes,Sox10,Agouti,Tyrp1 and Dct,in Oujiang color common carp,Cyprinus carpio var.color[D].Shanghai:Shanghai Ocean University,2014.(in Chinese)
[24] YAMADA T,HASEGAWA S,INOUE Y,et al.Wnt/β-catenin and kit signaling sequentially regulate melanocyte stem cell differentiation in UVB-induced epidermal pigmentation[J].The Journal of Investigative Dermatology,2013,133(12):2753-2762.
[25] BARKER N.The canonical Wnt/β-catenin signalling pathway[M]//Methods in Molecular Biology.Totowa,NJ:Humana Press,2008:5-15.
[26] MYUNG P S,TAKEO M,ITO M,et al.Epithelial wnt ligand secretion is required for adult hair follicle growth and regeneration[J].Journal of Investigative Dermatology,2013,133(1):31-41.
[27] WANG P P,XIAO S J,HAN Z F,et al.SNP discovery in large yellow croaker (Larimichthys crocea) using Roche 454 pyrosequencing sequencing platform[J].Conservation Genetics Resources,2015,7(4):777-779.
[28] 刘洪涛,刘金叶,杨明秋,等.基于转录组测序的波纹唇鱼SSR和SNP多态特征分析[J].基因组学与应用生物学,2020,39(6):2451-2461.
LIU H T,LIU J Y,YANG M Q,et al.SSR and SNP polymorphic feature analysis based on Cheilinus undulatus transcriptome[J].Genomics and Applied Biology,2020,39(6):2451-2461.(in Chinese)
[29] ZHANG L S,GUO X M.Development and validation of single nucleotide polymorphism markers in the eastern oyster Crassostrea virginica Gmelin by mining ESTs and resequencing[J].Aquaculture,2010,302(1/2):124-129.
[30] 张燕萍,章海鑫,崔璀,等.基于RNA-seq的黄尾鲴肝脏转录组测序与分析[J].水生态学杂志,2018,39(6):87-94.
ZHANG Y P,ZHANG H X,CUI C,et al.Transcriptome analysis of Xenocypris davidi bleeker based on RNA sequencing[J].Journal of Hydroecology,2018,39(6):87-94.(in Chinese)
[31] 李胜杰,白俊杰,赵荦,等.大口黑鲈EST-SNP标记开发及其与生长性状的相关性分析[J].海洋渔业,2018,40(1):38-46.
LI S J,BAI J J,ZHAO L,et al.Development of EST-SNPs in largemouth bass(Micropterus salmoides) and analysis of their correlation with growth traits[J].Marine Fisheries,2018,40(1):38-46.(in Chinese)
[32] ZHAO Z M,BOERWINKLE E.Neighboring-nucleotide effects on single nucleotide polymorphisms:a study of 2.6 million polymorphisms across the human genome[J].Genome Research,2002,12(11):1679-1686.