大菱鲆Scophthalmus maximus俗称多宝鱼,是中国北方地区鲆鲽类的主要养殖品种。近年来,随着养殖集约化、规模化的不断发展,以及种质资源的退化,养殖大菱鲆病害频发,限制了该产业的可持续发展[1]。细菌性疾病在大菱鲆养殖过程中发生的频次最高,其中杀鱼爱德华氏菌Edwardsiella piscicida是大菱鲆养殖中的主要细菌病原之一[2]。
杀鱼爱德华氏菌分布范围广,在世界许多国家和地区均有报道,该菌生存能力强,在温度为10~45 ℃条件下都能生长,最适于温度为28~37 ℃、pH中性的环境中生长[3]。杀鱼爱德华氏菌可感染鱼类的种类较多,能导致罗非鱼Oreochomis niloticus、斑马鱼Danio rerio、大菱鲆、大嘴鲈鱼Micropterus salmiodes、鳗鲡Anguilla japonica等多种鱼类患腹水病、出血性败血症等[4],而大菱鲆对该菌易感性较高且不分季节性,不同月龄个体均能被感染,发病率和死亡率均较高[1]。目前,已发现杀鱼爱德华氏菌携带与感染密切相关的多种毒力因子,如用于黏附在宿主细胞上的菌毛蛋白编码基因(fimA和fliC)、效应蛋白基因(eseJ)、过氧化氢酶基因(katB和katG)、超氧化物歧化酶基因(SodB和SodC)等[2-3]。关于杀鱼爱德华氏菌耐药菌株的研究,毛灿等[5]分析了花鲈源杀鱼爱德华氏菌耐药谱与毒力的相关性,但国内尚未见关于杀鱼爱德华氏菌耐药菌株、耐药基因的研究。杀鱼爱德华氏菌是从迟缓爱德华氏菌中分出的致病性更强的菌株,与迟缓爱德华氏菌表型特征有很多相同之处,目前,仍有一些菌株被误归为迟缓爱德华氏菌[5]。对杀鱼爱德华氏菌的鉴定,除用传统的生理生化方法外,还有通过测定16S rRNA、gyrB基因全序列同源性比对[5]及合成该菌特异性引物EP进行PCR检测[6]等方法的研究报道。
2019年,山东省烟台市某海水养殖场的养殖大菱鲆出现摄食能力减弱、行动迟缓的症状,发病鱼体腹部膨大,吻部、鳍条及鳍基部充血发红,眼睛红肿突出,上颌红肿。车间患该症状大菱鲆约占养殖总量的4%,10 d内患病个体死亡率为70%。本研究中,对患病大菱鲆进行了细菌分离,从具有典型症状病鱼的肝脏、脾脏、肾脏和心脏等组织中分离到1株致病力较强的菌株,对其进行了形态学观察,生理生化性状分析,16S rRNA、gyrB和rpoB基因序列比对,爱德华氏菌特异性引物扩增,以及毒力基因和耐药性分析,以期为该病的致病机理研究及有效防治提供科学参考。
1 材料与方法
1.1 材料
试验用鱼:患病大菱鲆采自山东省烟台市某海水养殖公司,体长为(27±2) cm,体质量为(350±5) g;用于回感试验的健康大菱鲆,体长为(28±1) cm,体质量为(380±5) g,于曝气人工海水中暂养7 d,每天虹吸换水30%,暂养期间水温控制在(17±1) ℃。
主要试剂:脑心浸液琼脂培养基(BHIA)、营养肉汤培养基(NB)、MH培养基(Muller Hinton)、沙门氏志贺氏琼脂培养基(SS)和革兰氏染色试剂盒均购自北京陆桥技术股份有限公司;生理生化鉴定管购自杭州滨和微生物试剂有限公司;细菌基因组提取试剂盒、PCR扩增相关试剂均购自宝生物(大连)有限公司;试验用引物由生工生物工程(上海)股份有限公司合成。
1.2 方法
1.2.1 病原筛查和细菌分离 选取患病大菱鲆,用体积分数为75%的乙醇棉球对体表进行消毒,无菌条件下解剖,取其肝脏、肾脏、脾脏和心脏,用灭菌磷酸盐缓冲液(PBS)清洗3次后充分研磨,用无菌环蘸取研磨组织匀浆液于BHIA平板上划线分离,28 ℃下恒温培养,48 h后观察结果。选取优势菌落进行纯化培养,将体积分数为20%的甘油-NB菌悬液于-80 ℃超低温冰箱中保存备用。
1.2.2 腹腔注射人工感染 选取从腹水病症典型的患病大菱鲆中分离的菌株H4-S18,接种于BHIA培养基上,28 ℃下恒温培养24 h,采用pH 7.4 的灭菌PBS制成浓度为7.8×1010、7.8×109、7.8×108、7.8×107 CFU/mL的菌悬液备用。选择与患病大菱鲆个体质量相近的健康个体50尾,在实验室中暂养一周后,随机分成5组,每组10尾,4个试验组每尾腹腔注射0.1 mL的菌悬液,对照组每尾腹腔注射0.1 mL的灭菌PBS缓冲溶液。试验养殖水箱保持充气,温度控制在(17±1) ℃,每天虹吸换水20%。感染期间不投喂,每天记录鱼的发病和死亡情况。
1.2.3 菌落形态观察 将菌株H4-S18分别接种于BHIA和SS培养基上,28 ℃下培养48 h后观察菌落形态。
1.2.4 生理生化鉴定 选取纯化后致病力较强的菌株H4-S18制成菌悬液,用革兰氏染色法对菌株染色观察,初步判断细菌种类与形态特征;应用生理生化鉴定试剂盒检测菌株的生理生化特性,进一步判断细菌种属。
1.2.5 分子生物学鉴定 应用细菌基因组DNA提取试剂盒提取菌液DNA后,送至生工生物工程(上海)股份有限公司进行16S rRNA、gyrB和rpoB测序,引物序列见表1。将目标菌株的16S rRNA、gyrB和rpoB扩增序列通过NCBI网站进行BLAST同源性分析,采用MEGA 6.0软件进行序列比对并构建系统发育树。
表1 引物序列
Tab.1 Primer sequence used in the experiment
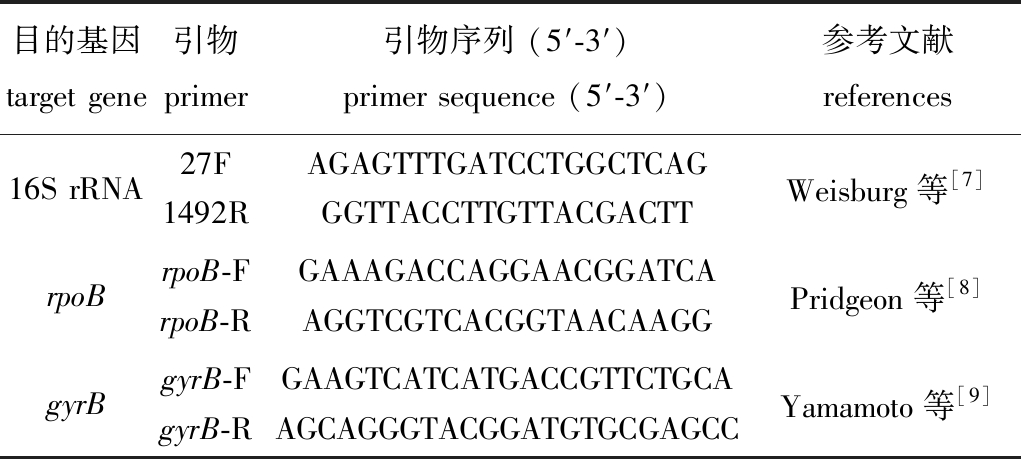
目的基因target gene引物primer引物序列 (5′-3′)primer sequence (5′-3′)参考文献references16S rRNA27F1492RAGAGTTTGATCCTGGCTCAGGGTTACCTTGTTACGACTTWeisburg等[7]rpoBrpoB-FrpoB-RGAAAGACCAGGAACGGATCAAGGTCGTCACGGTAACAAGGPridgeon等[8]gyrBgyrB-FgyrB-RGAAGTCATCATGACCGTTCTGCAAGCAGGGTACGGATGTGCGAGCCYamamoto等[9]
1.2.6 菌株H4-S18的爱德华氏菌种特异性引物检测 取纯化后的优势菌落接种于新鲜的NB培养基中,32 ℃下以140 r/min振荡培养 20 h。取1 mL菌液,按照细菌全基因组DNA抽提试剂盒操作说明书提取DNA,用于特异性引物检测。参考文献[10]中的方法,由生工生物工程(上海)股份有限公司合成爱德华氏菌属内种特异性引物,用于对 H4-S18的种鉴别。各种特异性引物分别为迟缓爱德华氏菌E.tarda(ET)、杀鱼爱德华氏菌(EP)、鮰爱德华氏菌E.ictaluri(ESC)和类杀鱼爱德华氏菌E.piscicida-like(EPL)[10](表2)。PCR扩增条件:95 ℃下预变性5 min;95 ℃下变性15 s,58 ℃下退火15 s,72 ℃下延伸15 s,共进行35个循环;72 ℃下终延伸5 min[10]。PCR扩增后用15 g/L琼脂糖电泳检测,并用紫外凝胶成像拍照。
表2 种类鉴定特异性引物序列
Tab.2 Specific primers for species identification

引物primer序列(5′-3′)sequence (5′-3′)参考文献referencesET-FET-RCAGTGATAAAAAGGGGTGGACTACACAGCAACGACAACGGriffin等[10]ESC-FESC-RACTTATCGCCCTCGCAACGCCTCTGATAAGTGGTTCTCGGriffin等[10]EP-FEP-RCTTTGATCATGGTTGCGGAAGCCTCTGATAAGTGGTTCTCGGriffin等[10]EPL-FEPL-RTTTGATCGGGTACGCTGTAATTGCTCTATACGCACGCGriffin等[10]
1.2.7 菌株H4-S18的致病基因检测 检测菌株H4-S18侵袭相关的菌毛基因fimA、fimB、fimC、fimD和毒力相关基因esrB、mukF、katB、sodB、citC和gadB的携带情况,各致病基因引物见表3,引物由生工生物工程(上海)股份有限公司合成,PCR扩增后用15 g/L琼脂糖电泳检测,并用紫外凝胶成像拍照。
表3 致病基因引物序列
Tab.3 Primer sequence of virulence pathogenic genes

引物primer引物序列 (5′-3′)primer sequence (5′-3′)参考文献referencesfimA-FfimA-RCGGTAAAGTTGAGTTTACGGGTGTGTAACCGTGTTGGCGTAAGSakai等[11]fimB-FfimB-RCTATATGGTGCAGACCTGGCTGAAGGAGACTGTATTGSakai等[11]fimC-FfimC-RAACACCGGTATCAGCGGAACGTTGAATCGGTATGGCGTCSakai等[11]fimD-FfimD-RGGTAACCTGATTTGGCGTTCGGATCACCTGGATCTTATCCSakai等[11]esrB-FesrB-RGATCATGCCTTGCTAGCCTCGGCGACCAGCTTGAGATan等[12]mukF-FmukF-RTTTGGACGGTGAAATGAGCCGTTGCGGTGCCAGTGAA程俊茗等[13]katB-FkatB-RGATGCGATCAAGTTCCCGGAACCTGGATGTACAGATCCCATHan等[14]sodB-FsodB-RATGTCATTCGAATTACCTGCTCGATGTAATAAGCGTGTTCCCAYamada等[15]citC-FcitC-RTTTCCGTTTGTGAATCAGGTCAATGTTTCGGCATAGCGTTG江云等[16]gadB-FgadB-RATTCCCGCTTTGGTTCAGAGGAGGAGCCGATAGTATTGGTA程俊茗等[13]
1.2.8 菌株H4-S18的耐药性检测 按照美国临床检验标准委员会(CLSI)推荐的K-B药敏纸片扩散法操作标准,检测菌株H4-S18对23种抗菌药物的敏感性,按照CLSI标准对结果进行判定。
检测菌株H4-S18的β-内酰胺类TEM基因、氨基糖苷类ant(3″)-Ⅰ基因、喹诺酮类oqxA基因、磺胺类Sul2与Sul3基因、四环素类tet(A)基因和多肽类mcr-1基因等7种耐药基因携带情况,耐药基因引物序列见表4,引物由生工生物工程(上海)股份有限公司合成,PCR扩增后用15 g/L琼脂糖电泳检测,并用紫外凝胶成像拍照。
表4 耐药基因引物序列
Tab.4 Primer sequence of drug resistance gene
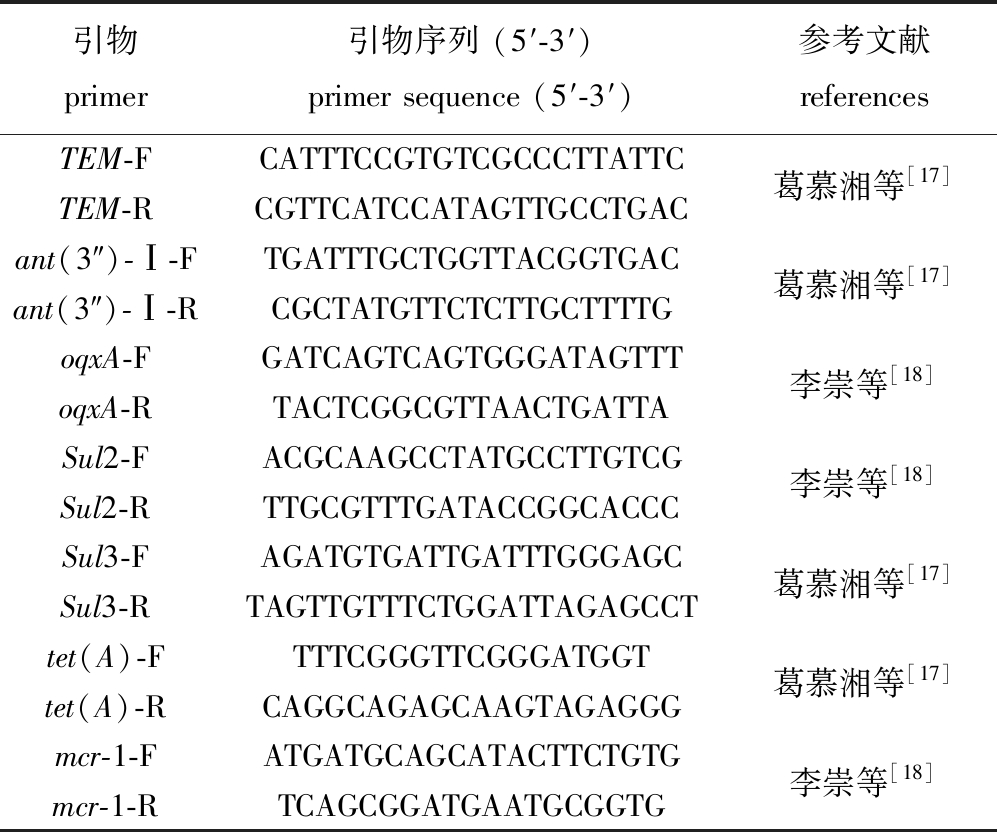
引物primer引物序列 (5′-3′)primer sequence (5′-3′)参考文献referencesTEM-FTEM-RCATTTCCGTGTCGCCCTTATTCCGTTCATCCATAGTTGCCTGAC葛慕湘等[17]ant(3″)-Ⅰ-Fant(3″)-Ⅰ-RTGATTTGCTGGTTACGGTGACCGCTATGTTCTCTTGCTTTTG葛慕湘等[17]oqxA-FoqxA-RGATCAGTCAGTGGGATAGTTTTACTCGGCGTTAACTGATTA李崇等[18]Sul2-FSul2-RACGCAAGCCTATGCCTTGTCGTTGCGTTTGATACCGGCACCC李崇等[18]Sul3-FSul3-RAGATGTGATTGATTTGGGAGCTAGTTGTTTCTGGATTAGAGCCT葛慕湘等[17]tet(A)-Ftet(A)-RTTTCGGGTTCGGGATGGTCAGGCAGAGCAAGTAGAGGG葛慕湘等[17]mcr-1-Fmcr-1-RATGATGCAGCATACTTCTGTGTCAGCGGATGAATGCGGTG李崇等[18]
2 结果与分析
2.1 解剖观察
选取腹部膨大,肛门红肿突出,吻部、鳍条及鳍基部充血发红,眼睛红肿突出,上颌红肿等典型症状的患病鱼进行解剖。剖检可见腹腔内有淡黄色腹水,肠肿大、肠壁薄,内有淡黄色液体或黄便,肝充血发红,脾肿大等症状(图1)。
2.2 人工感染试验
将病原菌分离纯化,获得1株优势菌,命名为H4-S18,用该菌株腹腔注射健康大菱鲆,注射后随感染时间延长,试验鱼伏于池底、反应迟缓,受强烈刺激后反应剧烈,出现无方向性窜游甚至撞壁现象。最高浓度组(7.8×1010 CFU/mL)第8天时开始出现死亡,发病大菱鲆腹腔内有淡黄色腹水,肠内有白便,肠壁充血;感染第9天时,死亡大菱鲆出现腹部膨胀、肛门红肿、鳍条基部充血发红等典型症状,解剖可见肠壁膨大充血发红且内有透明液体;感染第11天时,大菱鲆眼睛红肿突出,鳍条充血发红,注射点红肿呈肉芽状糜烂,肝充血发红(图2)。从人工感染发病死亡鱼体分离的菌株,菌体形态、生理生化鉴定和菌毛基因均与菌株H4-S18相同,这表明,菌株H4-S18可引起健康大菱鲆发病并出现自然发病症状。采用改良寇氏法计算菌株H4-S18对大菱鲆的半致死浓度LD50为1.2×108 CFU/mL。

A—鳍条充血发红;B—腹胀;C—肝脏出血;D—脾脏肿大。
A—red and congested fins; B—abdominal distension; C—liver hemorrhage; D—spleen enlargement.
图1 自然发病大菱鲆主要症状
Fig.1 Main symptoms of naturally diseased turbot

A—鳍条充血发红;B—上颌红肿充血、眼球突出;C—肠道膨大;D—肝脏出血;E—脾脏肿大。
A—red and congested fins; B—maxillary swelling and ulceration and exophthalmos; C—intestinal expansion; D—liver hemorrhage; E—spleen enlargement.
图2 人工感染发病大菱鲆主要症状
Fig.2 Main symptoms of artificially infected turbot
最高浓度组(7.8×1010 CFU/mL)注射后第8天时开始出现死亡,死亡高峰期在第11~14天,第14天时全部死亡;次高浓度组(7.8×109 CFU/mL)第11天时开始出现死亡,第14天时累计死亡率为60%,之后无死亡;对照组整个试验过程中无死亡(图3)。
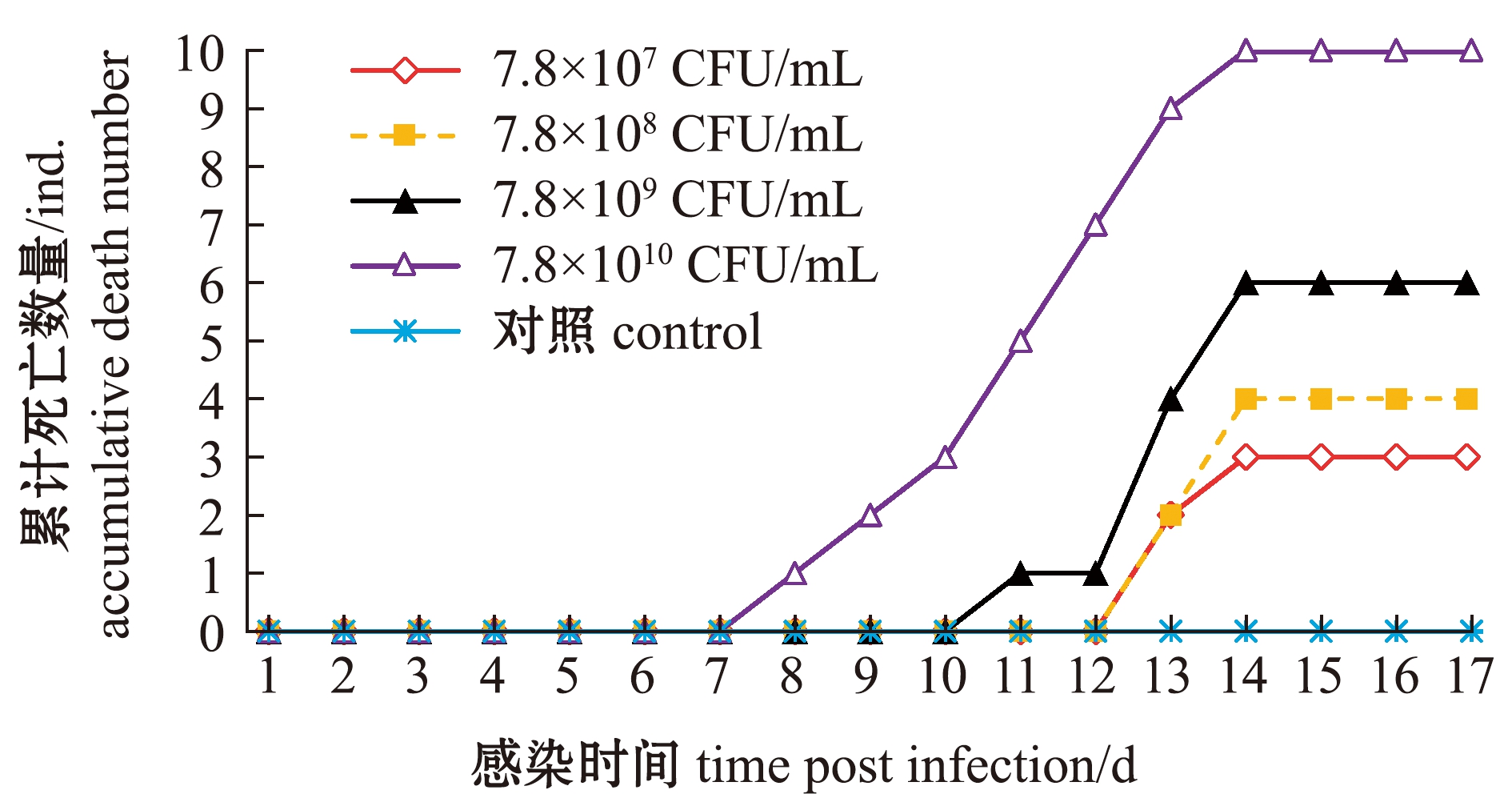
图3 回归感染累计死亡曲线
Fig.3 Accumulative death number of turbot experimentally infected with Edwardsiella piscicida
2.3 菌株H4-S18的鉴定
2.3.1 菌落和菌体形态 菌株H4-S18在BHIA培养基上培养24 h(32 ℃),形成圆形、光滑、温润、微隆起、边缘整齐透明、中间乳白色及直径为0.2~0.5 mm的小菌落(图4A);在SS琼脂培养基上形成黑色小菌落(图4B);菌体呈短杆状,无夹馍,不形成芽孢,革兰氏染色呈红色,属革兰氏阴性菌(图4C)。
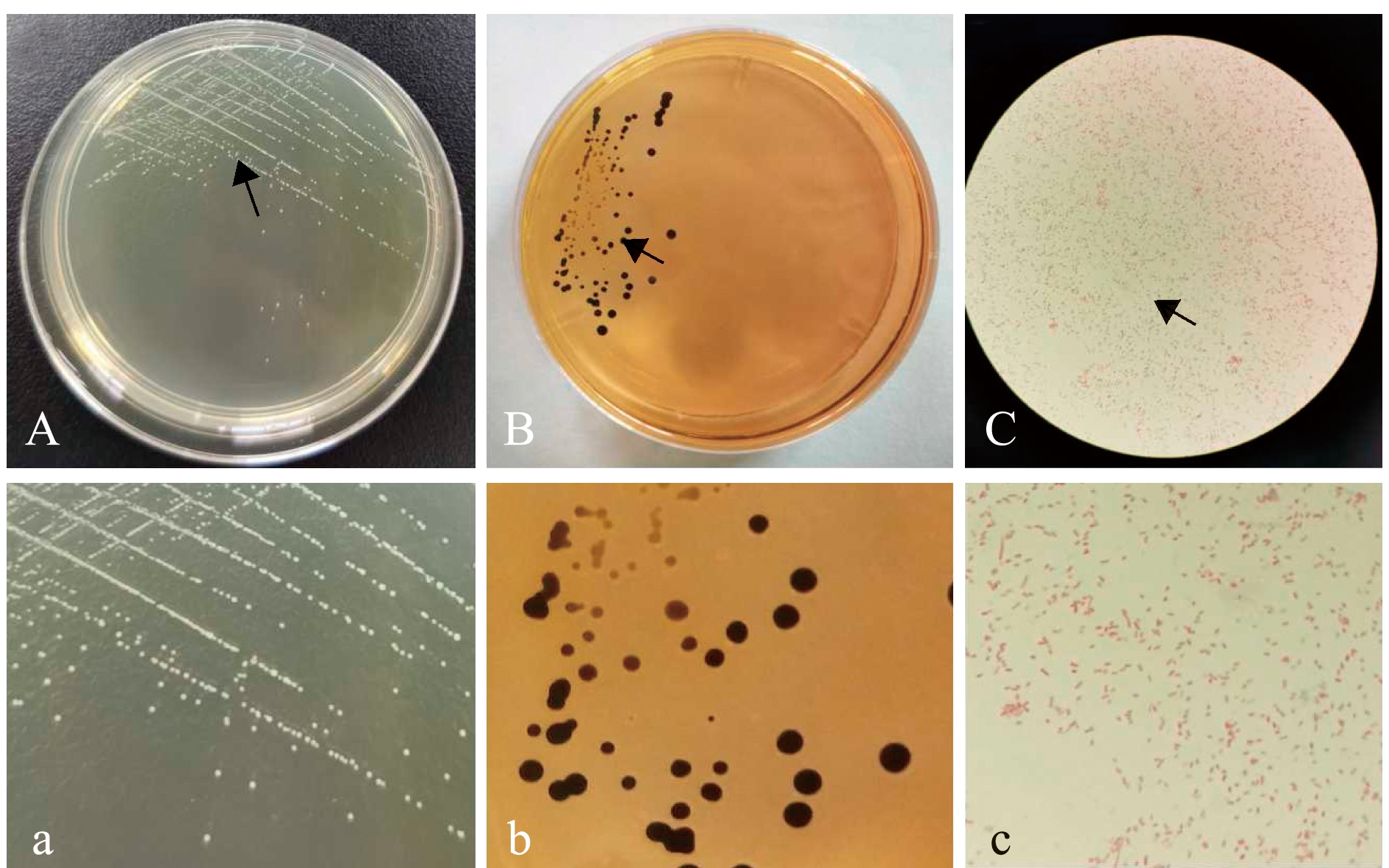
A—BHIA培养基菌落形态;B—SS琼脂培养基菌落形态;C—革兰氏染色显微观察;a—图A箭头处放大;b—图B箭头处放大;c—图C箭头处放大。
A—isolation and culture with BHIA; B—culture with SS agar medium; C—observation with microscope; a—magnification of partial A; b—magnification of partial B; c—magnification of partial C.
图4 菌株H4-S18的菌落形态及革兰氏染色形态
Fig.4 Colony morphology and micro-morphology of strain H4-S18 stained by Gram
2.3.2 生理生化鉴定 生理生化反应显示:赖氨酸、鸟氨酸、葡萄糖产酸、甲基红和SS琼脂等呈阳性;枸橼酸、精氨酸、苯丙氨酸、蔗糖、蜜二糖、鼠李糖、阿拉伯糖、甘露醇、山梨醇、肌醇、苦杏仁苷、半乳糖苷、尿素、硝酸盐还原和氧化酶等呈阴性(表5)。
2.3.3 菌株H4-S18的系统进化分析 提取分离菌株的DNA后,用不同引物进行PCR扩增和测序,分别得到了1 500 bp(16S rRNA)、950 bp(rpoB)和1 200 bp(gyrB)的基因片段(图5),纯化回收PCR产物经生工生物工程(上海)有限公司测序。将菌株H4-S18的16S rRNA序列进行BLAST比对,一致性为99%的有杀鱼爱德华氏菌、迟缓爱德华氏菌。
表5 菌株H4-S18的生化特性
Tab.5 Biochemical identification of strain H4-S18
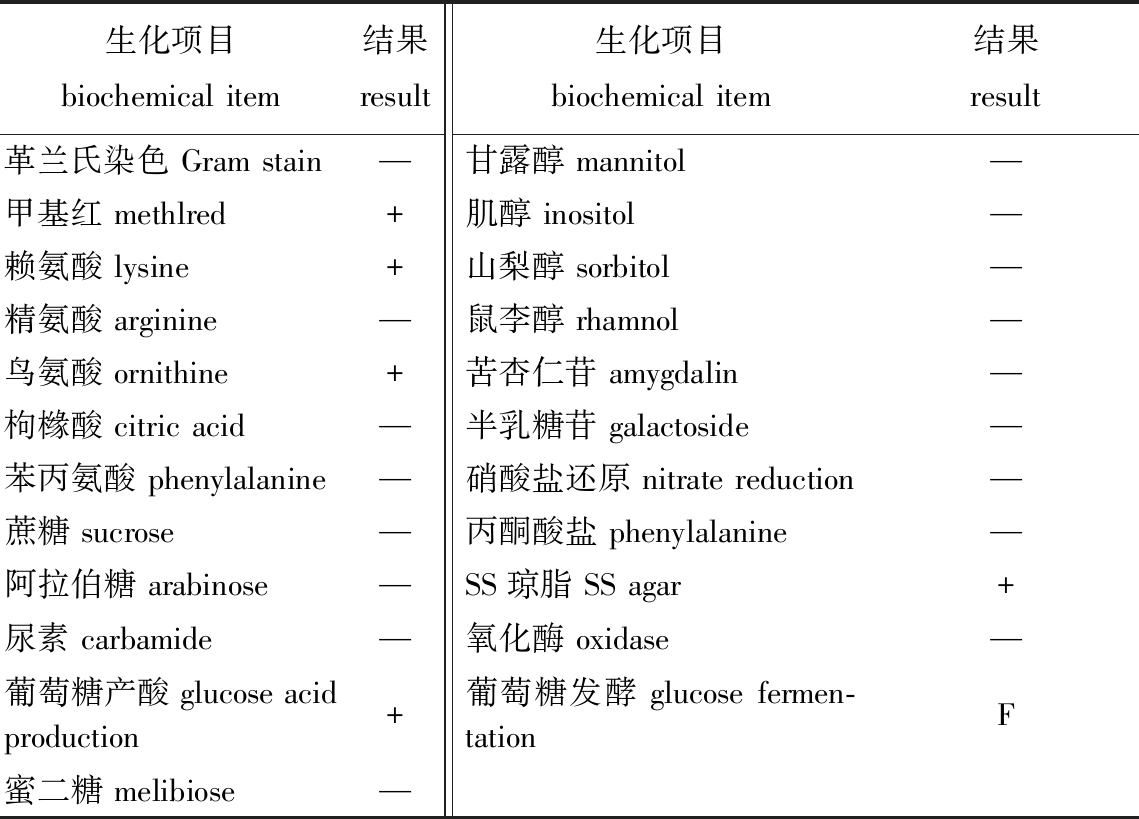
生化项目biochemical item结果result生化项目biochemical item结果result革兰氏染色 Gram stain—甘露醇 mannitol—甲基红 methlred+肌醇 inositol—赖氨酸 lysine+山梨醇 sorbitol—精氨酸 arginine—鼠李醇 rhamnol—鸟氨酸 ornithine+苦杏仁苷 amygdalin—枸橼酸 citric acid—半乳糖苷 galactoside—苯丙氨酸 phenylalanine—硝酸盐还原 nitrate reduction—蔗糖 sucrose—丙酮酸盐 phenylalanine—阿拉伯糖 arabinose—SS琼脂 SS agar+尿素 carbamide—氧化酶 oxidase—葡萄糖产酸 glucose acid production+葡萄糖发酵 glucose fermen-tationF蜜二糖 melibiose —
注:+,阳性; —,阴性;F—发酵。
Note: +, positive; — ,negative; F—fermentation.
为进一步确定其分类地位,采用16S rRNA基因测序与gyrB、rpoB管家基因序列分析相结合的方法进行致病菌H4-S18的系统进化分析(图6~图8),将测序结果与GenBank中已知核苷酸序列进行BLAST比对,结果显示,菌株H4-S18的16S rRNA基因序列与杀鱼爱德华氏菌ET883(NR_125649.1)、SNU1(MN_203719)和SS 01(KC_309472)的一致性分别为99.90%、99.65%和99.65%;菌株H4-S18的gyrB基因序列与杀鱼爱德华氏菌WFE(MG_225486.1)的一致性达99.83%;菌株H4-S18的rpoB基因序列与杀鱼爱德华氏菌ETW41(CP_019440.1)的一致性达99.58%。在16S rRNA、gyrB和rpoB 3个基因系统进化树中,菌株H4-S18均与E.piscicida首先聚在一起,表明其亲缘关系最近。
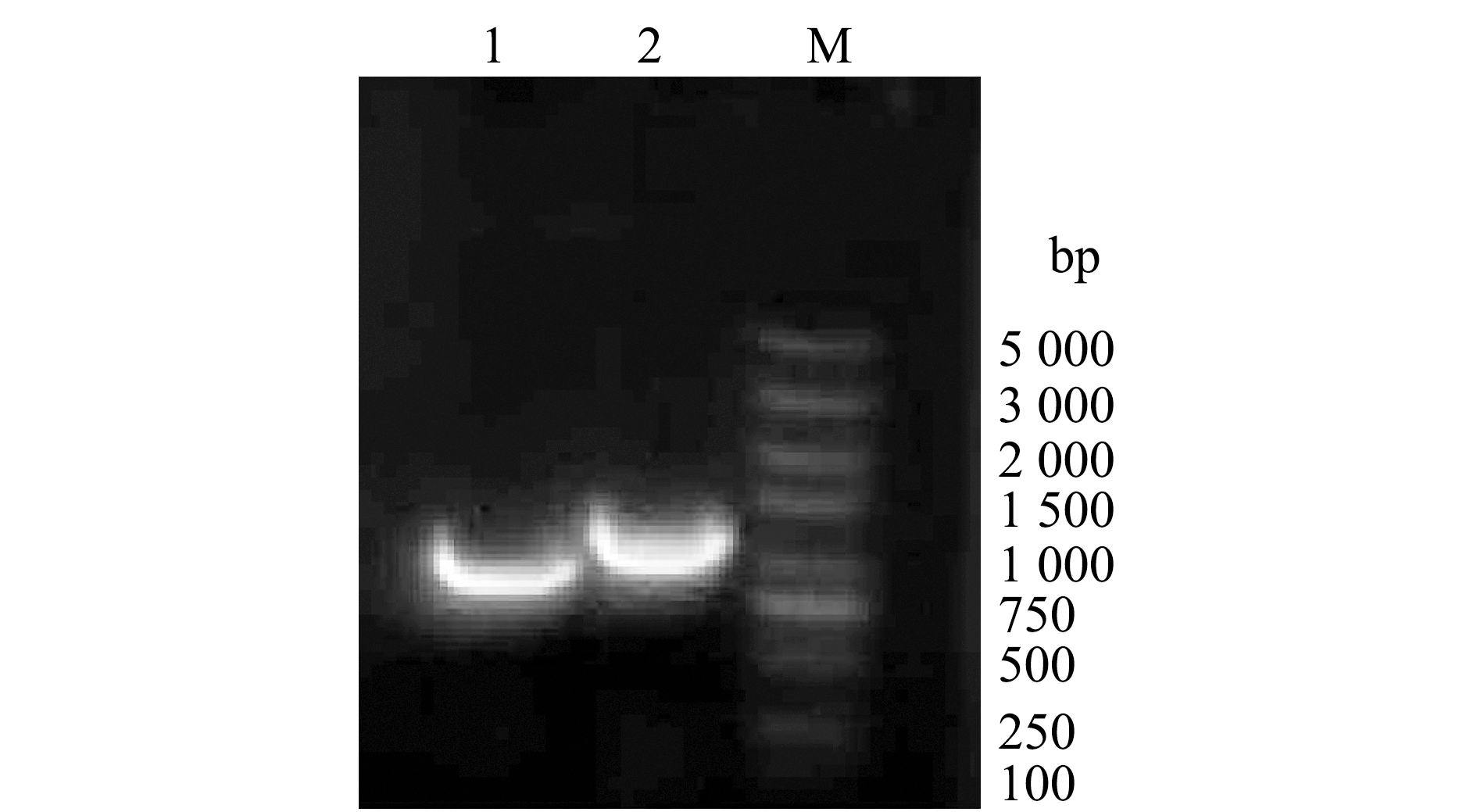
1—rpoB 基因;2—gyrB基因;M—DNA分子标准质量。
1—rpoB gene;2—gyrB gene;M—DNA marker.
图5 菌株H4-S18管家基因PCR检测结果
Fig.5 PCR products of the housekeeping genes from the isolated strain H4-S18
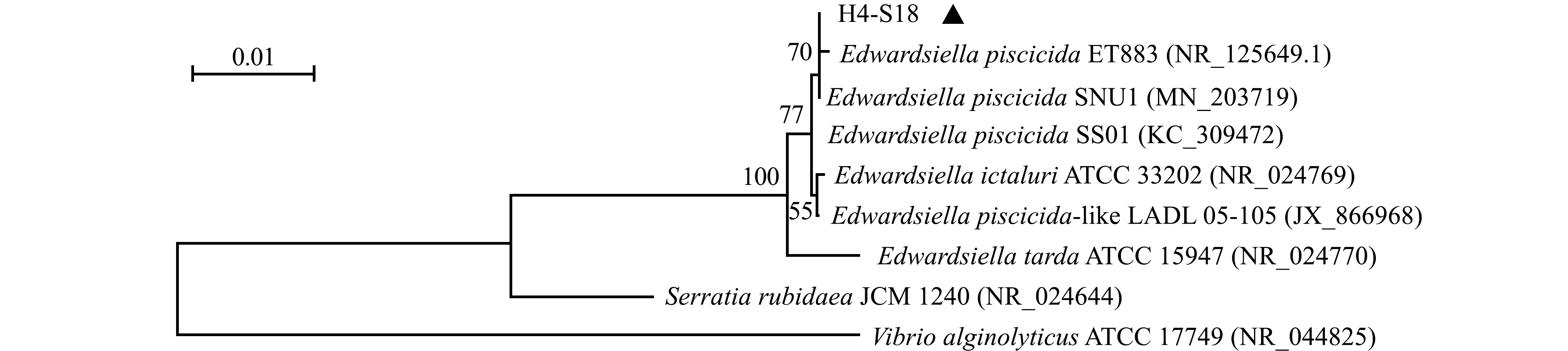
图6 菌株H4-S18的16S rRNA基因序列与近源菌株的系统进化树
Fig.6 Phylogenetic tree of strain H4-S18 based on 16S rRNA gene sequence and its relatives

图7 菌株H4-S18的gyrB基因系统进化树
Fig.7 Phylogenetic tree of strain H4-S18 based on gyrB gene sequences

图8 菌株H4-S18的rpoB基因系统进化树
Fig.8 Phylogenetic tree of strain H4-S18 based on rpoB gene sequences
2.3.4 菌株H4-S18经爱德华氏菌种特异性引物的PCR检测 选用迟缓爱德华氏菌、杀鱼爱德华氏菌、鮰爱德华氏菌和类杀鱼爱德华氏菌种特异性引物,对菌株H4-S18进行特异性PCR鉴定,结果显示,用杀鱼爱德华氏菌EP特异性引物扩增出大小为130 bp的特异性条带,而其他3种引物均未扩增出特异性条带(图9)。结果证实本次分离的菌株H4-S18为杀鱼爱德华氏菌。

M—DNA分子标准质量; 1—迟缓爱德华氏菌引物;2—杀鱼爱德华氏菌引物;3—鮰爱德华氏菌引物;4—类杀鱼爱德华氏菌引物。
M—DNA marker; 1—primer of E.tarada; 2—primer of E.piscicida; 3—primer of E.ictaluri; 4—primer of E.piscicida-like.
图9 种特异性引物对菌株H4-S18的鉴别
Fig.9 Identification of strain H4-S18 by species-specific primers
2.4 菌株H4-S18的致病基因检测
对分离菌株H4-S18的致病基因进行了检测,结果显示,H4-S18的菌毛基因fimA、fimB、fimC和fimD,以及毒力基因esrB、mukF、katB、sodB、citC和gadB均为阳性(图10)。
2.5 菌株H4-S18的耐药性检测
菌株H4-S18对10类23种测试抗生素的敏感性存在差异,对磺胺类2种、多肽类2种和利福霉素类1种在内的5种抗生素类药物耐药;对β-内酰胺类5种、氨基糖苷类3种、喹诺酮类5种、四环素类2种、大环内酯类1种、氯霉素类1种、呋喃类1种在内的18种抗生素敏感(表6)。
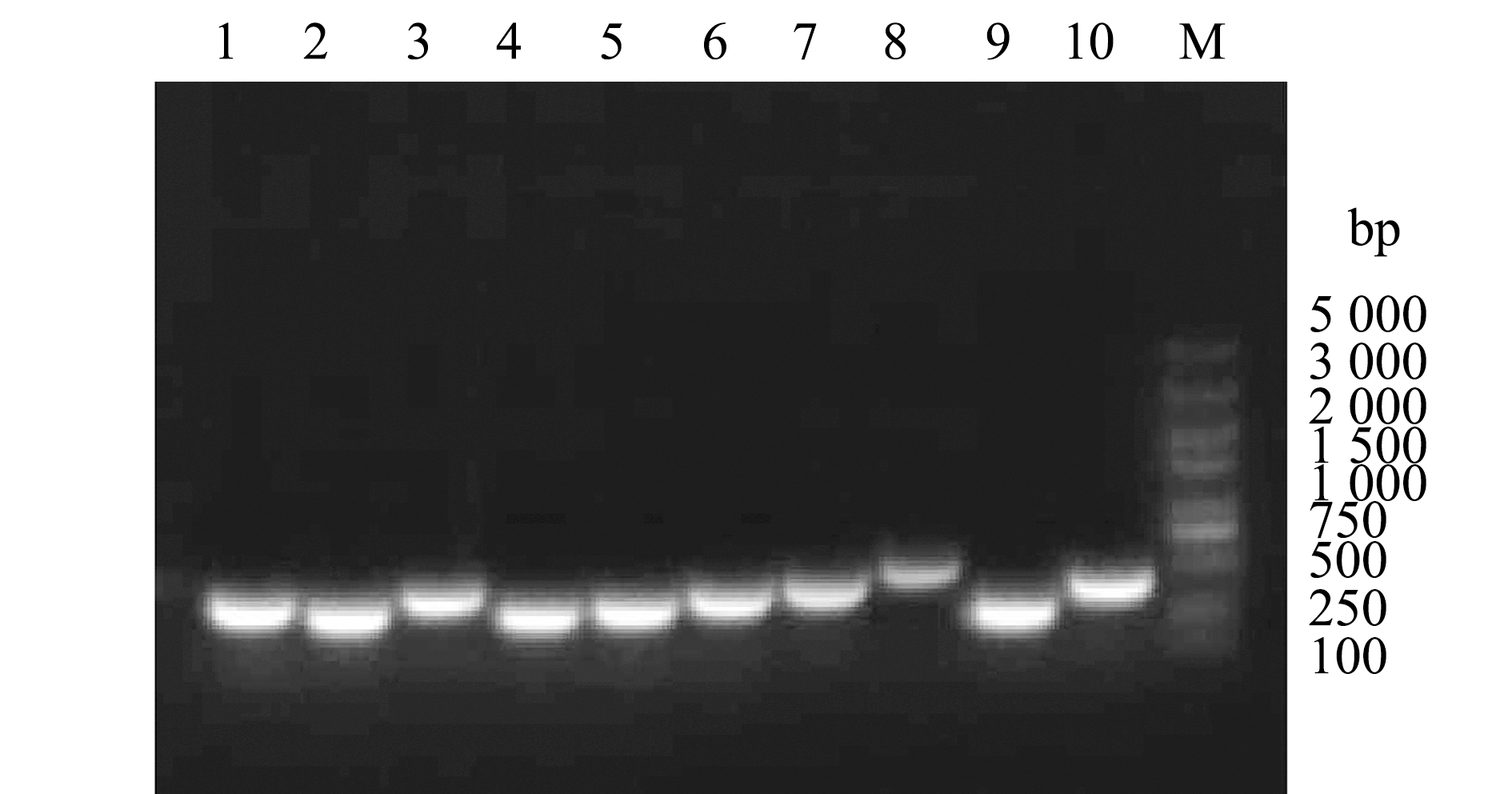
1—fimA基因;2—fimC基因;3—esrB基因;4—mukF基因;5—katB基因;6—fimD基因;7—sodB基因;8—citC基因;9—gadB基因;10—fimB基因;M—DNA分子质量标准。
1— fimA gene; 2—fimC gene; 3—esrB gene;4—mukF gene;5—katB gene; 6—fimD gene; 7—sodB gene; 8—citC gene; 9—gadB gene; 10—fimB gene; M—DNA marker.
图10 菌株H4-S18毒力基因PCR检测结果
Fig.10 PCR products of the virulence-related genes from the isolated strain H4-S18
耐药基因检测结果显示,检测到菌株H4-S18携带β-内酰胺类TEM、氨基糖苷类ant(3″)-Ⅰ、磺胺类tet(A)、四环素类mcr-1和喹诺酮类Sul2等5种耐药基因(图11)。
3 讨论
3.1 杀鱼爱德华氏菌的鉴定
爱德华氏菌是一种危害严重的人鱼共患传染病病原,传统上分为迟缓爱德华氏菌、鮰爱德华氏菌和保科爱德华氏菌3个种[19],其中,迟缓爱德华氏菌的感染宿主和分布范围最广,能引发人类严重的脑膜炎、败血症、肠胃炎和伤口感染等[20],并可导致水产动物全身细菌性败血症[21]。2012年,有学者发现,迟缓爱德华氏菌有两个不同毒力的基因型EdwGⅠ型和EdwG Ⅱ型[22];2013年,Abayneh等[23]根据表型和遗传表征不同进行重新分类,将致病性更强的EdwGⅠ型划为新种,命名为杀鱼爱德华氏菌,与迟缓爱德华氏菌同属于爱德华氏菌属;2016年,Ucko等[24]根据序列差异又将杀鱼爱德华氏菌分为杀鱼爱德华氏菌和类杀鱼爱德华氏菌两个种。
表6 菌株H4-S18对10类23种抗生素的敏感性(n=4)
Tab.6 Sensitivity of strain H4-S18 to 23 kinds of antimicrobial drugs(n=4)
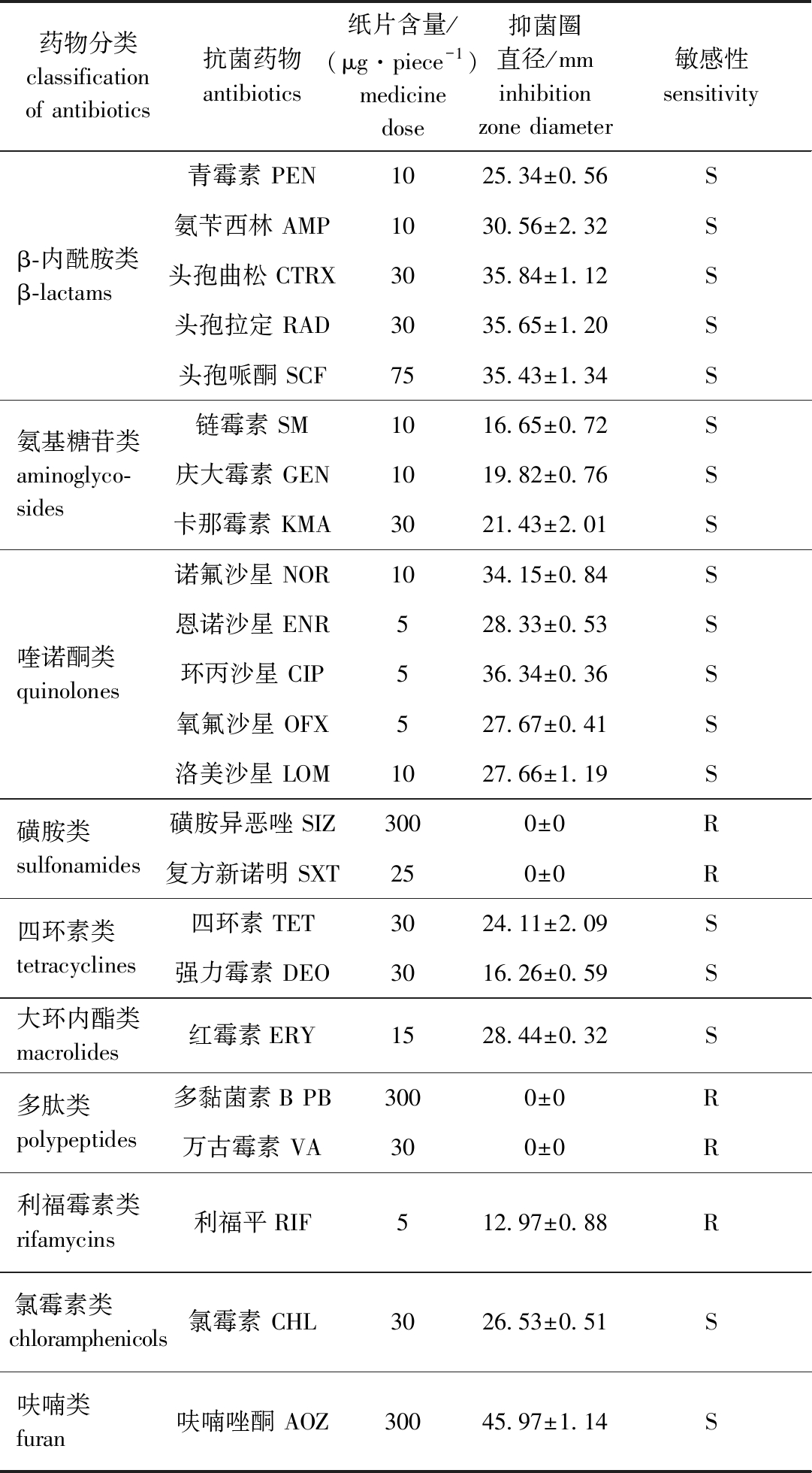
药物分类classification of antibiotics抗菌药物antibiotics纸片含量/(μg·piece-1)medicine dose抑菌圈直径/mminhibition zone diameter敏感性sensitivityβ-内酰胺类β-lactams青霉素 PEN1025.34±0.56S氨苄西林 AMP1030.56±2.32S头孢曲松 CTRX3035.84±1.12S头孢拉定 RAD3035.65±1.20S头孢哌酮 SCF7535.43±1.34S氨基糖苷类aminoglyco-sides链霉素 SM1016.65±0.72S庆大霉素 GEN 1019.82±0.76S卡那霉素 KMA3021.43±2.01S喹诺酮类quinolones诺氟沙星 NOR1034.15±0.84S恩诺沙星 ENR528.33±0.53S环丙沙星 CIP 536.34±0.36S氧氟沙星 OFX527.67±0.41S洛美沙星 LOM1027.66±1.19S磺胺类sulfonamides磺胺异恶唑 SIZ3000±0R复方新诺明 SXT250±0R四环素类tetracyclines四环素 TET3024.11±2.09S强力霉素 DEO3016.26±0.59S大环内酯类macrolides红霉素ERY1528.44±0.32S多肽类polypeptides多黏菌素B PB3000±0R万古霉素 VA300±0R利福霉素类 rifamycins利福平RIF512.97±0.88R氯霉素类 chloramphenicols氯霉素 CHL 3026.53±0.51S呋喃类 furan呋喃唑酮 AOZ30045.97±1.14S
注:R—耐药;S—敏感。
Note:R—resistant; S—sensitive.
本研究中,患病大菱鲆出现了全身出血等败血症现象,通过传统的生理生化鉴定,结果与爱德华氏菌属相似,其中,枸橼酸反应为阴性。前期研究发现,迟缓爱德华氏菌和杀鱼爱德华氏菌的枸橼酸反应多为阴性,而类杀鱼爱德华氏菌的枸橼酸反应多为阳性。初步判定分离菌株H4-S18不是类杀鱼爱德华氏菌。为更准确地鉴定H4-S18的种类,本研究中运用Weisburg等[7]设计的16S rRNA序列分析比对,发现该菌与杀鱼爱德华氏菌、鮰爱德华氏菌、类杀鱼爱德华及迟缓爱德华氏菌等的一致性均在99%以上,无法准确鉴定到种,研究结果与前人一致[1],爱德华氏菌属内的迟缓爱德华氏菌、鮰爱德华氏菌、杀鱼爱德华氏菌和类杀鱼爱德华氏菌的16S~23S rRNA区间基因序列有99%的相似率,仅通过16S rRNA基因序列分析、构建系统发育树的方法,仅能鉴定到爱德华氏菌属,并不能鉴定到种[1]。
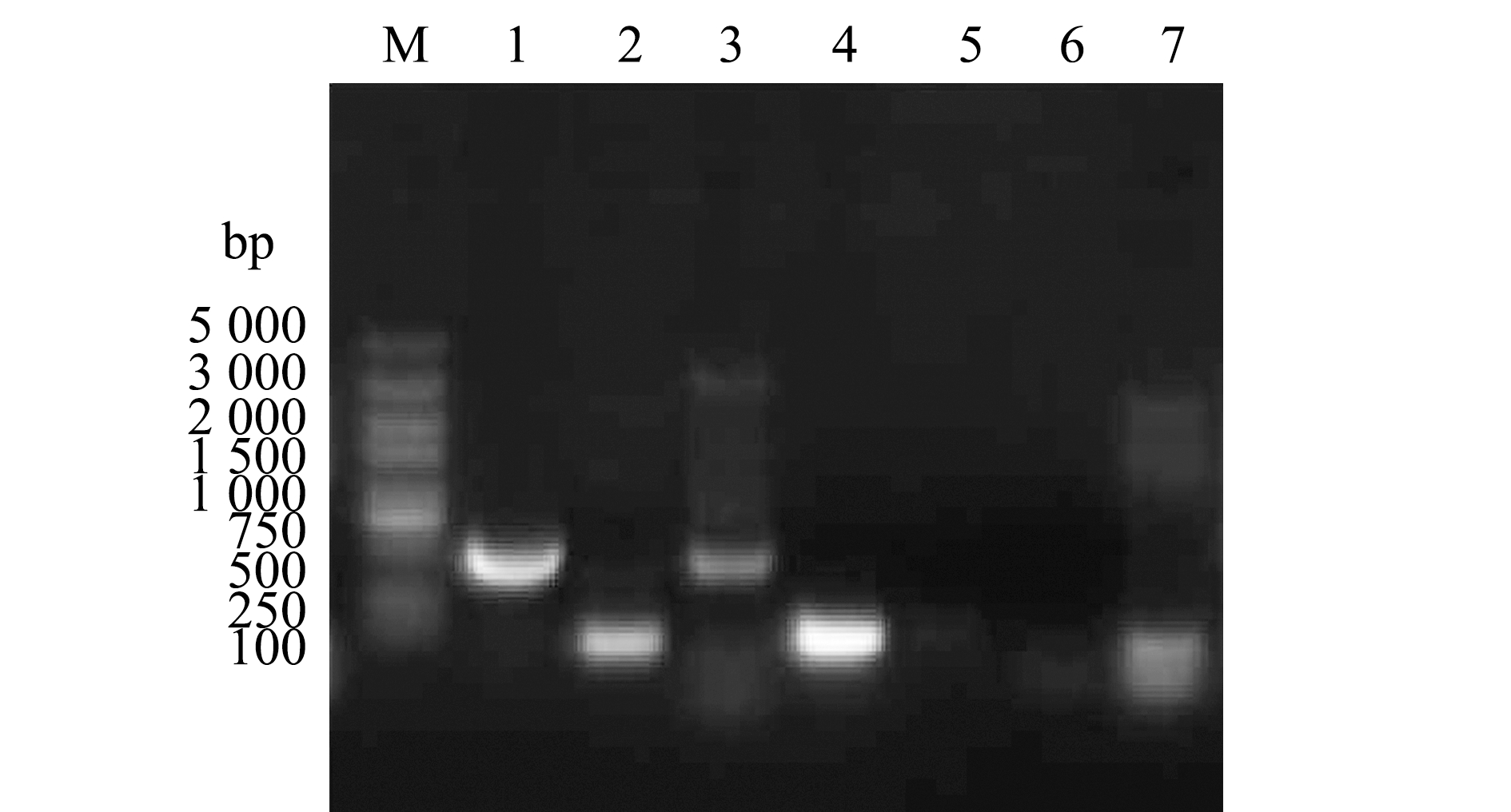
M—DNA分子质量标准;1—TEM基因;2—tet(A)基因;3—mcr-1基因;4—ant(3″)-Ⅰ基因;5—Sul3基因;6—oqxA基因;7—Sul2基因。
M—DNA marker, 1—TEM gene; 2—tet(A) gene; 3—mcr-1 gene;4—ant(3″)-Ⅰgene;5—Sul3 gene; 6—oqxA gene; 7—Sul2 gene.
图11 菌株H4-S18耐药力基因PCR检测结果
Fig.11 PCR products of the drug resistance genes from the isolated strain H4-S18
Soler[25]研究发现, 管家gyrB基因表达水平高于16S rRNA基因,不易在水平间转移,而毛灿等[5]用gyrB 基因进行了杀鱼爱德华氏菌鉴定。本研究中,根据Pridgeon等[8]、Yamamoto等[9]的方法,PCR扩增得到950 bp(rpoB)和1 200 bp(gyrB)的基因片段,序列比对发现,菌株H4-S18的gyrB基因序列与杀鱼爱德华氏菌WFE(MG_225486.1)的一致性为99.83%,菌株H4-S18的rpoB基因序列与杀鱼爱德华氏菌CK41(CP_047671.1) 的一致性为99.58%。为更准确地确定菌株H4-S18的分类地位,本研究中同时参考Griffin等[10]合成爱德华菌属种特异性引物,扩增出杀鱼爱德华氏菌(EP)特异性引物条带。
综合生理生化鉴定、序列比对分析及特异性引物扩增三方面结果,确定本研究中的分离株H4-S18为杀鱼爱德华氏菌。结合本研究及相关报道[26],在杀鱼爱德华氏菌鉴定时,不能仅通过生理生化或16S rRNA序列直接鉴定到种,应结合生理生化鉴定、特异性引物扩增及多个管家基因系统进化关系等结果综合判定,以降低误判,提高鉴定准确率。
3.2 杀鱼爱德华氏菌的致病因子
杀鱼爱德华氏菌能够导致鱼类患皮肤溃烂暴露、骨头坏死、器官肉芽肿病变和败血症等疾病[27-28]。Esteve等[29]从患败血症的野生欧洲鳗中分离出杀鱼爱德华氏菌,本研究中从患出血症大菱鲆中同样分离出该菌。为深入探讨分离株的致病能力,找到更加安全有效的防治方法,本研究中还开展了致病因子的研究。
目前,研究人员已经鉴定出多种与杀鱼爱德华氏菌致病过程有关的毒力因子,包括皮肤毒素、细胞侵袭因子、溶血素、软骨素酶、三型分泌系统(T3SS)和六型分泌系统(T6SS)等[30-32]。esrB是T3SS分泌系统的调节基因,可分泌至胞外形成针筒状结构外鞘,用来刺破宿主细胞膜[33-34]。查阅杀鱼爱德华氏菌、爱德华氏菌属及肠杆菌科相关毒力因子研究发现,mukF基因编码杀伤蛋白可直接杀伤细胞[35];katB基因编码过氧化氢酶,抵抗H2O2对细胞膜、DNA和酶等细胞组成的损伤[36];sodB基因作为氧应激相关蛋白基因,在抗氧化防御中具有重要作用[15];citC基因可帮助细胞分解柠檬酸[37];gadB基因参与抵抗酸性条件,能够减弱胃肠道的酸性物质和吞噬细胞的杀伤作用[37];菌毛基因与爱德华氏菌的致病性强弱有关,fimA毒力基因能够编码鞭毛蛋白,帮助病原黏附到宿主细胞表面[38]。基于此,本研究中选择了毒力启动基因(esrB)、杀伤因子基因(mukF)、过氧化氢酶基因(katB)、抵抗因子基因(sodB)、柠檬酸裂解酶基因(citC)、谷氨酸脱羧酶同工酶基因(gadB)等6种毒力基因和4种菌毛基因(fimA、fimB、fimC和fimD)进行了检测,结果表明,10种毒力基因均被检出,且与上述相关研究中关于毒力基因的研究结果基本一致,但目前这种毒力组合尚未见报道。
毒力基因研究不仅可用于该菌致病机理的研究和毒力判断,还为疫苗的研究提供了重要依据。esrB等T3SS元件与诱导大菱鲆产生免疫应答有密切关系,T3SS不仅是杀鱼爱德华氏菌的重要毒力因子,还是重要的减毒疫苗开发靶点[39]。本研究中,在杀鱼爱德华氏菌分离株H4-S18中检测到了esrB毒力基因,为后续减毒疫苗的研发提供了有益参考。
3.3 杀鱼爱德华氏菌的药物敏感性与耐药基因
抗菌药物在治疗由爱德华氏菌引起的疾病中起到了非常重要的作用,但长期不科学的滥用抗生素,致使许多致病菌产生了耐药性,抗菌药物的防治效果越来越差,因此,科学研究致病菌的药物敏感性,对健康养殖和科学用药具有重要的指导意义。本研究中发现,菌株H4-S18对10类23种测试抗生素的敏感性存在差异,对磺胺异恶唑、复方新诺明2种磺胺类,多黏菌素、万古霉素2种多肽类,利福平1种利福霉素类在内的5种抗生素类药物耐药;对5种β-内酰胺类、3种氨基糖苷类、5种喹诺酮类、1种大环内酯类、1种氯霉素类、2种四环素类和1种呋喃类在内的18种抗生素敏感。尚琨等[40]对大菱鲆源杀鱼爱德华氏菌分离株研究发现,其对杆菌肽、庆大霉素、萘啶酸、磺胺异恶唑、苯唑西林、复方新诺明和头孢呋辛等7种抗菌药物耐药。毛灿等[5]研究发现,花鲈源杀鱼爱德华氏菌87株分离株共有32种耐药谱型,且均为多重耐药菌株,对麦迪霉素、红霉素和利福平的耐药率超过了90%,对青霉素、磺胺异恶唑耐药率为50%~80%。Ma等[41]研究发现,利福平耐药情况普遍存在于环境中。本研究中,菌株H4-S18对利福平耐药,与水体中的抗生素检测结果相同。菌株H4-S18对5种抗生素耐药,与前人研究结果基本相符,同样为多重耐药菌株,且经检测携带了5种耐药基因。菌株H4-S18对头孢曲松、头孢拉啶、头孢哌酮、诺氟沙星、氧氟沙星、洛美沙星和红霉素等类型药物均敏感,近年来,相关部门提倡科学用药,已明令禁止该类药物在水产养殖中使用。根据H4-S18对恩诺沙星敏感的研究结果,推荐在水产养殖中使用恩诺沙星粉防治大菱鲆杀鱼爱德华氏菌病,该药还可用于防治大菱鲆副乳房链球菌病[42]。
目前,关于病原菌耐药基因的检测研究较多,但未见杀鱼爱德华氏菌耐药基因的检测报道。本研究中,参考其他耐药菌的相关研究,筛选了部分耐药基因进行检测,结果显示,菌株H4-S18携带β-内酰胺类TEM、氨基糖苷类ant(3″)-Ⅰ、磺胺类tet(A)、四环素类mcr-1和喹诺酮类Sul2等5种耐药基因,未检测到携带磺胺类Sul3基因及喹诺酮类oqxA基因。研究表明,细菌对抗生素的耐药性与频繁过度使用药物、抗生素的选择性压力等有关[43],而中国海域检出率较高的抗生素有四环素类和喹诺酮类[44-45]。本试验中,杀鱼爱德华氏菌菌株H4-S18同样测出了四环素类mcr-1和喹诺酮类Sul2耐药基因,与前人环境监测研究结果相符,这说明烟台地区海域曾频繁或过度使用四环素类、喹诺酮类药物。本研究结果发现,菌株H4-S18虽携带了β-内酰胺类TEM、氨基糖苷类ant(3″)-Ⅰ、喹诺酮类Sul2等耐药基因,但未表现出相关的耐药性,与葛慕湘等[17]的研究结果一致,证实了耐药基因与耐药表型并不一一对应。本研究中耐药基因及药物敏感性的研究结果,为杀鱼爱德华氏菌耐药菌株的进一步研究及病害防治提供了科学依据。
4 结论
1)从山东省烟台市某养殖场患病大菱鲆中分离到一株优势菌株H4-S18,经生理生化,16S rRNA、gyrB基因、rpoB基因序列比对,以及爱德华氏菌特异性引物扩增等,判定该菌株为杀鱼爱德华氏菌。
2)杀鱼爱德华氏菌H4-S18对大菱鲆的半致死浓度LD50为1.2×108 CFU/mL,说明其具有较强致病性。
3)H4-S18具有fimA、fimB、fimC、fimD等4种菌毛基因和esrB、mukF、katB、sodB、citC、gadB等6种毒力基因,说明其属于强毒株型。
4)H4-S18携带TEM、ant(3″)-Ⅰ、tet(A)、mcr-1、Sul2等5种耐药基因,对2种磺胺类、2种多肽类、1种利福霉素类在内的5种抗生素类药物耐药,说明其属于耐药型菌株。
5)对杀鱼爱德华氏菌耐药型菌株引起的腹水、出血症病防治中,推荐使用恩诺沙星粉(水产用),并严格执行500度日的休药期。
[1] 黄华,王鹤,徐晓莹,等.养殖大菱鲆病原菌杀鱼爱德华氏菌的快速检测模型构建思路[J].齐鲁渔业,2020,37,(7):11-13.HUANG H,WANG H,XU X Y,et al. Construction of rapid detection model of Edwardsiella piscicida from cultured turbot Scophthalmus maximus[J]. Shandong Fisheries,2020,37,(7):11-13.(in Chinese)
[2] 孙永灿.杀鱼爱德华氏菌fucR基因缺失株构建及其生物学特性分析[D].山东农业大学,2021.
SUN Y C.Construction and biological characteristics of fucR gene deletion strain of Edwardsiella piscicida[D].Shandong Agricultural University,2021.(in Chinese)
[3] 刘洋.杀鱼爱德华氏菌中全局调控因子EsrB调控毒力因子表达的分子机制[D].上海:华东理工大学,2017.
LIU Y.Molecular mechanism of global regulator EsrB regulating the expres virulence determinats in Edwardsiella piscicida[D].Shanghai:East China University of Science and Technology,2017.(in Chinese)
[4] BUJAN N,MOHAMMED H,BALBOA S,et al.Genetic studies to re-affiliate Edwardsiella tarda fish isolates to Edwardsiella piscicida and Edwardsiella anguillarum species[J].Systematic and Applied Microbiology,2018,41(1):30-37.
[5] 毛灿,苏友禄,李勇,等.花鲈源杀鱼爱德华菌耐药谱及毒力相关性分析[J].中国水产科学,2020,27,(7):846-857.
MAO C,SU Y L,LI Y,et al.Analysis of the relationship between antibiotic resistance and virulence of Edwardsiella piscicida strains isolated from Lateolabrax maculatus[J].Journal of Fishery Sciences of China,2020,27(7):846-857. (in Chinese)
[6] GRIFFIN M J,WARE C,QUINIOU S M,et al.Edwardsiella piscicida identified in the Southeastern USA by gyrB sequence,species-specific and repetitive equencemediated PCR[J].Diseases of Aquatic Organisms,2014,108(1):23-35.
[7] WEISBURG W G,BARNS S M,PELLETIER D A,et al.16S ribosomal DNA amplification for phylogenetic study[J].Journal of Bacteriology,1991,173(2):697-703.
[8] PRIDGEON J W,KLESIUS P H,YILDIRIM-AKSOY M.Attempt to develop live attenuated bacterial vaccines by selecting resistance to gossypol,proflavine hemisulfate,novobiocin,or ciprofloxacin[J].Vaccine,2013,31(8):2222-2230.
[9] YAMAMOTO S,HARAYAMA S.PCR amplification and direct sequencing of gyrB genes with universal primers and their application to the detection and taxonomic analysis of Pseudomonas putida strains[J].Applied and Environmental Microbiology,1995,61(3):1104-1109.
[10] GRIFFIN M J,WARE C,QUNINIOU S M,et al.Edwardsiella piscicida identified in the Southeastern USA by gyrB sequence,species-specific and repetitive sequencemediated PCR[J].Diseases of Aquatic Organisms,2014,108(1):23-35.
[11] SAKAI T,LIDA T,OSATOMI K,et al.Detection of type 1 fimbrial genes in fish pathogenic and non-pathogenic Edwardsiella tarda strains by PCR[J].Fish Pathology,2007,42(2):115-117.
[12] TAN Y P,ZHENG J,TUNG S L,et al.Role of type Ⅲ secretion in Edwardsiella tarda virulence[J].Microbiology,2005,151(7):2301-2313.
[13] 程俊茗,万明月,周晋扬,等.鲫源迟缓爱德华氏菌的分离鉴定及其毒力基因的检测[J].微生物学通报,2017,44(10):2380-2390.
CHENG J M,WAN M Y,ZHOU J Y,et al. Identification and virulence genes detection of Edwardsiella tarda isolated from Carassius auratus gibelio[J].Microbiology China,2017, 44(10): 2380-2390. (in Chinese)
[14] HAN H J,KIM D H,LEE D C,et al.Pathogenicity of Edwardsiella tarda to olive flounder,Paralichthys olivaceus (Temminck & Schlegel)[J].Journal of Fish Diseases,2006,29(10):601-609.
[15] YAMADA Y,WAKABAYASHI H.Identification of fish-pathogenic strains belonging to the genus Edwardsiella by sequence analysis of sodB[J].Fish Pathology,2009,34(3):145-150.
[16] 江云,李寿崧,王寿昆,等.致病性迟钝爱德华氏菌毒力基因的PCR检测[J].中国食品学报,2008,8(4):123-129.
JIANG Y,LI S S,WANG S K,et al. Detection of the virulence genes of pathogenic Edwardsiella tarda by PCR assay[J].Journal of Chinese Institute of Food Science and Technology, 2008, 8(4): 123-129. (in Chinese)
[17] 葛慕湘,靳晓敏,张艳英,等.迟钝爱德华氏菌耐药表型及4种耐药基因检测[J].水产科学,2015,34(5):300-304.
GE M X,JIN X M,ZHANG Y Y,et al.Detection of drug resistance and four drug resistance genes in bacterial pathogen Edwardsiella tarda[J].Fisheries Science,2015,34(5):300-304. (in Chinese)
[18] 李崇,陈韵如,李沁媚,等.广西某地区猪源大肠杆菌耐药性及部分菌株耐药基因调查[J].中国畜牧兽医,2019,46(12):3749-3758.
LI C,CHEN Y R,LI Q M,et al.Analysis of drug resistance of porcine Escherichia coli and drug resistance genes of some strains in a region of Guangxi[J].China Animal Husbandry and Veterinary Medicine,2019,46(12):3749-3758. (in Chinese)
[19] JANADA J M,ABBOTT S L,KROSKE-BYSTROM S,et al.Pathogenic properties of Edwardsiella species[J].Journal of Clinical Microbiology,1991,29(9):1997-2001.
[20] JANDA J M,ABBOTT S L.Expression of an iron-regulated hemolysin by Edwardsiella tarda[J].FEMS Microbiology Letters,1993,111(2/3):275-280.
[21] MOHANTY B R,SAHOO P K.Edwardsiellosis in fish:a brief review[J].Journal of Biosciences,2007,32(sup 3):1331-1344.
[22] YANG M J,LV Y Z,XIAO J F,et al.Edwardsiella comparative phylogenomics reveal the new intra/inter-species taxonomic relationships,virulence evolution and niche adaptation echanisms[J].Plos One,2012,7(5):e36987.
[23] ABAYNEH T,COLOQUHOUN D J,SORUM H.Edwardsiella piscicida sp.nov.,a novel species pathogenic to fish[J].Journal of Applied Microbiology,2013,114(3):644-654.
[24] UCKO M,COLORNI A,DUBYTSKA L,et al.Edwardsiella piscicida-like pathogen in cultured grouper[J].Diseases of Aquatic Organisms,2016,121(2):141-148.
[25] SOLER L.Phylogenetic analysis of the genus Aeromonas based on two housekeeping genes[J].International Journal of Systematic and Evolutionary Microbiology,2004,54(5):1511-1519.
[26] 张鸿鹄,袁娜,傅超英,等.黑棘鲷内脏结节病病原的分离、鉴定及致病性研究[J].水产学报,2019,43(12):2554-2566.
ZHANG H H,YUAN N,FU C Y,et al. Isolation,identification and pathogenicity studies on visceral granulomaous disease in Acanthopagrus schlegelii[J]. Journal of Fisheries of China, 2019,43(12):2554-2566. (in Chinese)
[27] REICHLEY S R,WALDBIESER G C,TEKEDAR H C,et al.Complete genome sequence of Edwardsiella piscicida isolate S11-285 recovered from channel catfish (Ictalurus punctatus) in Mississippi,USA[J].Genome Announc,2016,4(6):e01259-16.DOI:10.1128/genomeA.01259-16.
[28] FOGELSON S B,PETTY B D,REICHLEY S R,et al.Histologic and molecular characterization of Edwardsiella piscicida infection in largemouth bass (Micropterus salmoides)[J].Journal of Veterinary Diagnostic Investigation,2016,28(3):338-344.
[29] ESTEVE C,ALCAIDE E.Seasonal recovery of Edwardsiella piscicida from wild European eels and natural waters:isolation methods,virulence and reservoirs[J].Journal of Fish Diseases,2018,41(11):1613-1623.
[30] ULLAH M A,ARAI T.Pathological activities of the naturally occurring strains of Edwardsiella tarda[J].Fish Pathology,1983,18(2):65-70.
[31] LING S H M,WANG X H,XIE L,et al.Use of green fluorescent protein (GFP) to study the invasion pathways of Edwardsiella tarda in vivo and in vitro fish models[J].Microbiology,2000,146(1):7-19.
[32] HIRONO I,TANGE N,AOKI T.Iron-regulated haemolysin gene from Edwardsiella tarda[J].Molecular Microbiology,1997,24(4):851-856.
[33] WANG B,MO ZL,XIAO P,et al.A putative T3SS translocon component of Edwardsiella tarda,contributes to virulence in fish and is a candidate for vaccine development[J].Mar Biotechnol,2010,12:378-685.
[34] ZHENG J,LI N,TAN Y.P,et al.EscC is a chaperone for the Edwardsiella tarda type III secretion system putative translocon components EseB and EseD[J].Microbiology,2007(153):1953-1962.
[35] JIN F,KUNITOSHI Y,HIRNONORI N,et al.New killing system controlled by two genes located immediately upstream of the mukB gene in Escherichia coli[J].Molecular and General Genetics,1994,243(2):136-147.
[36] MATTHEW J A,TAN Y P,SRINIVASA RAO P S,et al.Edwardsiella tarda mutants defective in siderophore production,motility,serum resistance and catalase activity[J].Microbiology,2001,147(2):449-457.
[37] CASTERINE-CORNET M P,PENFOUND T A,SNITH D,et al.Control of acid resistance in Escherichia coli[J].Journal of Biosciences,1999,181(11):3525-3535.
[38] 朱文进,陈翠珍,苏咏梅,等.牙鲆致病性迟钝爱德华菌毒力基因的检测及序列分析[J].动物医学进展,2014,35(11):20-24.
ZHU J W,CHEN C Z,SU Y M,et al. Detection and sequencing of virulence genes of pathogenic Edwardsiella tarda in Paralichthys olivaceus[J].Progress in Veterinary Medicine, 2014, 35(11): 20-24. (in Chinese)
[39] WANG X,WANG Q,XIAO J,et al.Hemolysin EthA in Edwardsiella tarda is essential for fish invasion in vivo and in vitro and regulated by two-component system EsrA-EsrB and nucleoid protein HhaEt[J].Fish Shellfish Immunol,2010,29:1082-1091.
[40] 尚琨,王斌,刘欣,等.大菱鲆源杀鱼爱德华氏菌(Edwardsiella piscicida)的分离鉴定[J].河北渔业,2019(1):21-25.
SHANG K,WANG B,LIU X,et al.Isolation and identification of Edwardsiella piscicida from turbot Scophthalmus maximus[J].Hebei Fisheries,2019(1):21-25. (in Chinese)
[41] MA L,LI B,ZHANG T.Abundant Rifampin resistance genes and significant correlations of antibiotic resistance genes and plasmids in various environments revealed by metagenomic analysis[J].Applied Microbiology and Biotechnology,2014,98(11):5195-5204.
[42] 王鹤,李战军,黄华,等.大菱鲆源副乳房链球菌的分离鉴定及其毒力基因型[J].大连海洋大学学报,2021,36(4):563-572.
WANG H,LI Z J,HUANG H,et al. Isolation,identification and virulence gene of Streptococcus parauberis from cultured turbot Scophthalmus maximus[J].Journal of Dalian Ocean University,2021,36(4):563-572. (in Chinese)
[43] CARVALHO M J,MARTINEZ-MURCIA A,ESTEVES A C,et al.Phylogenetic diversity,antibiotic resistance and virulence traits of Aeromonas spp.from untreated waters for human consumption[J].International Journal of Food Microbiology,2012,159(3):230-239.
[44] TAMMINEN M,KARKMAN A,LOHMUS A,et al.Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure[J].Environmental Science & Technology,2011,45(2):386-391.
[45] HE X,DENG M,WANG Q,et al.Residues and health risk assessment of Quinolones and Sulfonamides in cultured fish from Pearl River Delta,China[J].Aquaculture,2016,458:38-46.