台湾白甲鱼Onychostoma barbatulum Pellegrin[1],又名台湾铲颌鱼,是鲤科Cyprinidae白甲鱼属Onychostoma的一种淡水鱼类,主要分布在长江及长江以南水系中,也有少数种类分布在淮河、海河及渭河[2]。台湾白甲鱼喜好栖息在水质清澈且有砾石的山间溪流底层或江河中上游的湍急水流中,其肌肉中富含不饱和脂肪酸、必需氨基酸等,营养丰富、味道鲜美,是受消费者喜爱的重要经济鱼类[3]。近年来,受水体污染、栖息环境破坏及过度捕捞等因素的影响,台湾白甲鱼资源日益匮乏,现已被列为濒危鱼类[4]。因此,加强台湾白甲鱼鱼种的繁殖、保护和研究具有重要意义。
对白甲鱼的研究主要集中在稀有白甲鱼Onychostoma rata[5-6]、多鳞白甲鱼O.macrolepis[7]和长江白甲鱼O.sima[8]等。研究主要涉及白甲鱼的ITS2序列结构与遗传多样性[8]、线粒体DNA特征[5,9]、生物学特征与繁殖技术[10]、肌肉营养成分[11-12]和肠道微生物群落组成[13]等。然而,对台湾白甲鱼的研究相对较少,对其微生物群落构成的研究尚未见报道。本研究中,采用Illumina公司的Novaseq测序平台,对采自不同环境(地点)的台湾白甲鱼皮肤细菌16S rRNA基因进行测序,比较了其皮肤细菌群落特征及其多样性,以期为台湾白甲鱼的健康养殖提供有益参考。
1 材料与方法
1.1 材料
1.1.1 样本 2020年11月,分别于率水河流域的安徽省黄山市休宁县溪口镇生态养殖场采集人工养殖1龄台湾白甲鱼样品6尾和安徽省黄山市休宁县流口镇水产种质保护区采集野生1龄台湾白甲鱼样品6尾。采用符合生态捕鱼法的撒网法捕鱼,台湾白甲鱼生物学信息见表1。
表1 台湾白甲鱼样品的生物学信息
Tab.1 Biological characteristics of Onychostoma barbatulum Pellegrin
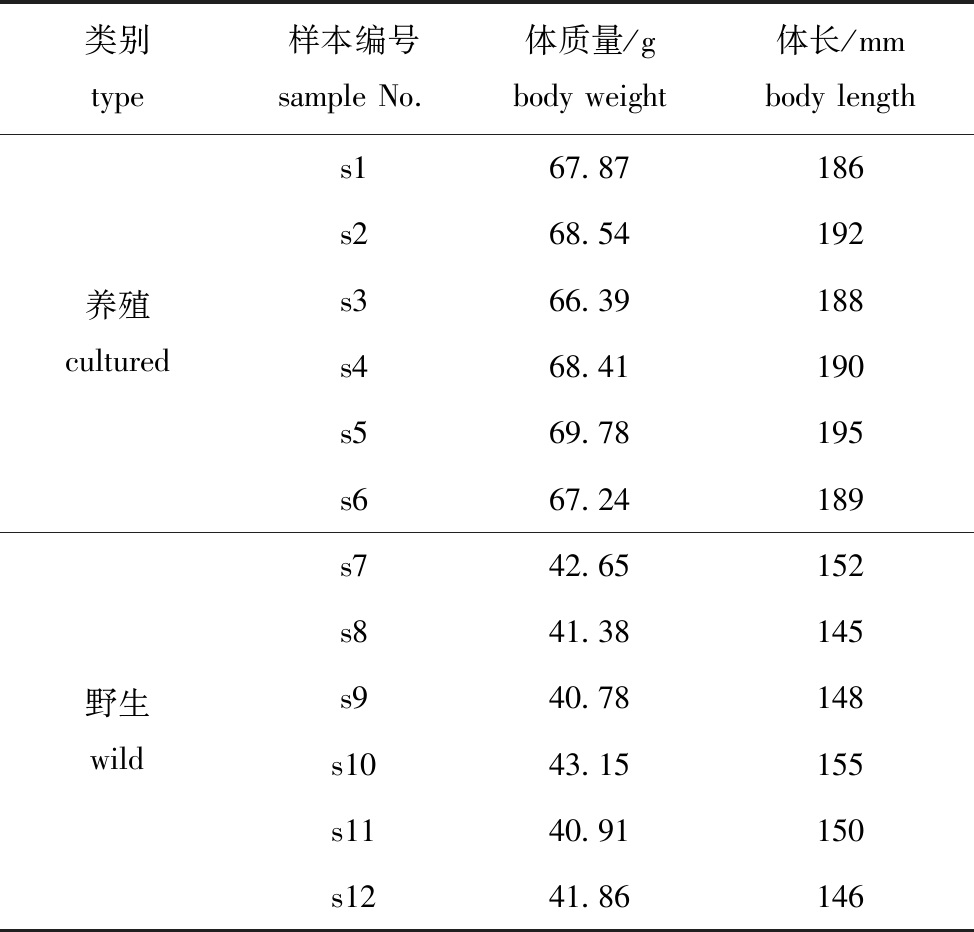
类别type样本编号sample No.体质量/gbody weight体长/mmbody length养殖cultureds167.87186s268.54192s366.39188s468.41190s569.78195s667.24189野生wilds742.65152s841.38145s940.78148s1043.15155s1140.91150s1241.86146
1.1.2 仪器 AC2-4S1型生物安全柜(新加坡Esco公司)、SQ510C型高压灭菌锅(重庆雅马拓公司)、MiniSpin plus型高速离心机(德国艾本德公司)、Tanon-1600型数码凝胶成像系统(上海天能公司)、DS-11型超微量紫外/可见光分光光度计(美国DeNovix公司)。
1.1.3 试剂 dNTPs、Trans 2K plus DNA maker和100 bp DNA marker购自北京全式金生物技术有限公司;十六烷基三甲基溴化铵(CTAB)、三羟甲基氨基甲烷、乙二胺四乙酸(EDTA)、醋酸和无水乙醇等购自国药集团化学试剂有限公司;Agarose琼脂糖(Biowest,西班牙)购自北京索莱宝科技有限公司。
1.2 方法
1.2.1 样品的制备 从采集的样本中分别随机挑选健康无损伤的白甲鱼,在无菌条件下将捕获的白甲鱼颈椎脱臼后牺牲,用无菌钳子钳住鱼嘴并用口腔拭子在鱼的两侧擦拭整个鱼身体(除了头部)以获取皮肤微生物;再将该口腔拭子放入预先准备好的无菌样品管中,放入冰盒内带回实验室。同时,将采完皮肤微生物样品的鱼编号后也放入冰盒内带回实验室,称重和测量长度后,于-80 ℃超低温冰箱中保存备用。
1.2.2 白甲鱼皮肤微生物总DNA的提取 参照赵裕栋等[14]的方法,提取白甲鱼皮肤微生物总DNA。简述如下:用无菌剪刀将含有白甲鱼皮肤微生物的口腔拭子剪下并放入无菌EP管中,向其中加入1.0 mL CTAB裂解液振荡混匀,静置15 min后加入溶菌酶和蛋白酶K,并于56 ℃水浴锅上反应30 min(每10 min混匀一次);离心收集上清液,并向其中加入酚、氯仿、异戊醇混合溶液(三者的体积比为25∶24∶1)450 μL后混合均匀,以12 000 r/min离心10 min;吸取上清液至新EP管中,加入2倍体积的异丙醇,混匀后于4 ℃冰箱中过夜沉淀;以12 000 r/min离心10 min后弃去上清液,再用1 mL体积分数为75%的乙醇洗涤沉淀两次;再次离心弃去上清液,并于超净工作台上挥发残留乙醇,最后用无菌双蒸水洗脱抽提的总DNA,用电泳检测试验结果。
1.2.3 白甲鱼皮肤微生物16S rRNA基因V3~V4序列的扩增 以提取的白甲鱼皮肤微生物基因组DNA为模板,利用引物341F:5′CCTAYGGGRBGCASCAG 3′和806R:5′GGACTACNNGGGTATCTAAT 3′扩增16S rRNA基因的V3~V4可变区。PCR扩增体系(共50 μL):5×PrimerStar buffer 10 μL,dNTPs 2 μL,341F引物2 μL,806R引物2 μL,DNA模板2 μL,PrimerStar polymerase 1 μL,无菌ddH2O 31 μL。PCR扩增程序:95 ℃下预变性10 min;95 ℃下循环变性30 s,52 ℃下退火复性30 s,72 ℃下延伸30 s,共进行30个循环;最后在72 ℃下再延伸10 min;电泳检测扩增结果。
1.2.4 白甲鱼皮肤细菌的高通量测序 上述扩增产物经20 g/L琼脂糖电泳检测后,取电泳条带明显、清晰的样品用超微量紫外/可见光分光光度计检验合格后,用AxyPrep DNA凝胶回收试剂盒回收扩增产物,并委托北京诺禾致源科技股份有限公司利用Illumina Novaseq测序技术平台构建文库和高通量测序。
1.2.5 测序数据的处理与分析 将测序获得的序列中的条码和引物去除,并用软件FLASH[15]对匹配的序列进行组装,获得Raw Tags序列;再用QIIME 2软件[16]对Raw Tags序列进行进一步分析。简述如下:将序列分值Q≤19或序列长度经处理后小于原始序列长度75%的序列舍去;参考数据库the Gold Database中已有的结果,利用USEARCH软件包中的UCHIME算法[17],将测序序列中的嵌合体找出并除去,以获得高质量的序列。
利用UPARSE软件[18]将上述高质量序列中相似度≥97%的序列划分到相同的操作单元(operational taxonomic units,OTUs);随后,用RDP classifier[19]和SILVA数据库[20]对上述OTUs中的代表序列进行注释和分析。利用Chao1指数(http://www.mothur.org/wiki/Chao)、Shannon指数(http://www.mothur.org/wiki/Shannon)和Simpson指数(http://www.mothur.org/wiki/Simpson)等对白甲鱼皮肤细菌多样性进行分析。
2 结果与分析
2.1 白甲鱼皮肤微生物总DNA提取结果
采用改进的CTAB法对采自白甲鱼皮肤的微生物总DNA进行提取。从图1(a)可见,绝大多数的白甲鱼皮肤微生物总DNA基本可成功提取,个别质量不佳的经重新抽提后基本满足试验要求。从图1(b)可见,利用引物341F和806R扩增的16S rRNA基因V3~V4可变区条带均一、清晰,回收后可用于后续测序试验。
2.2 白甲鱼皮肤细菌16S rRNA V3~V4可变区测序结果
采用Illumina Novaseq测序技术平台对上述微生物总DNA进行测序分析。从表2可见:测序获得的原始序列条数普遍在18.4万条以上,最多的可达29.9万条;通过校正测序序列、剔除错误序列和嵌合体外,获得的质控后序列也普遍在3.0万条以上,其中最多的可高达14.5万条。这表明,白甲鱼皮肤存在着大量细菌。

M—DNA分子标准质量; 1~12分别对应样本s1~s12。
M—DNA marker; 1 to 12 correspond to samples s1 to s12, respectively.
图1 台湾白甲鱼皮肤微生物DNA提取结果
Fig.1 Skin microbial DNA extracted from Onychostoma barbatulum Pellegrin
表2 样本测序结果质控统计
Tab.2 Quality control statistics of sequencing results in samples
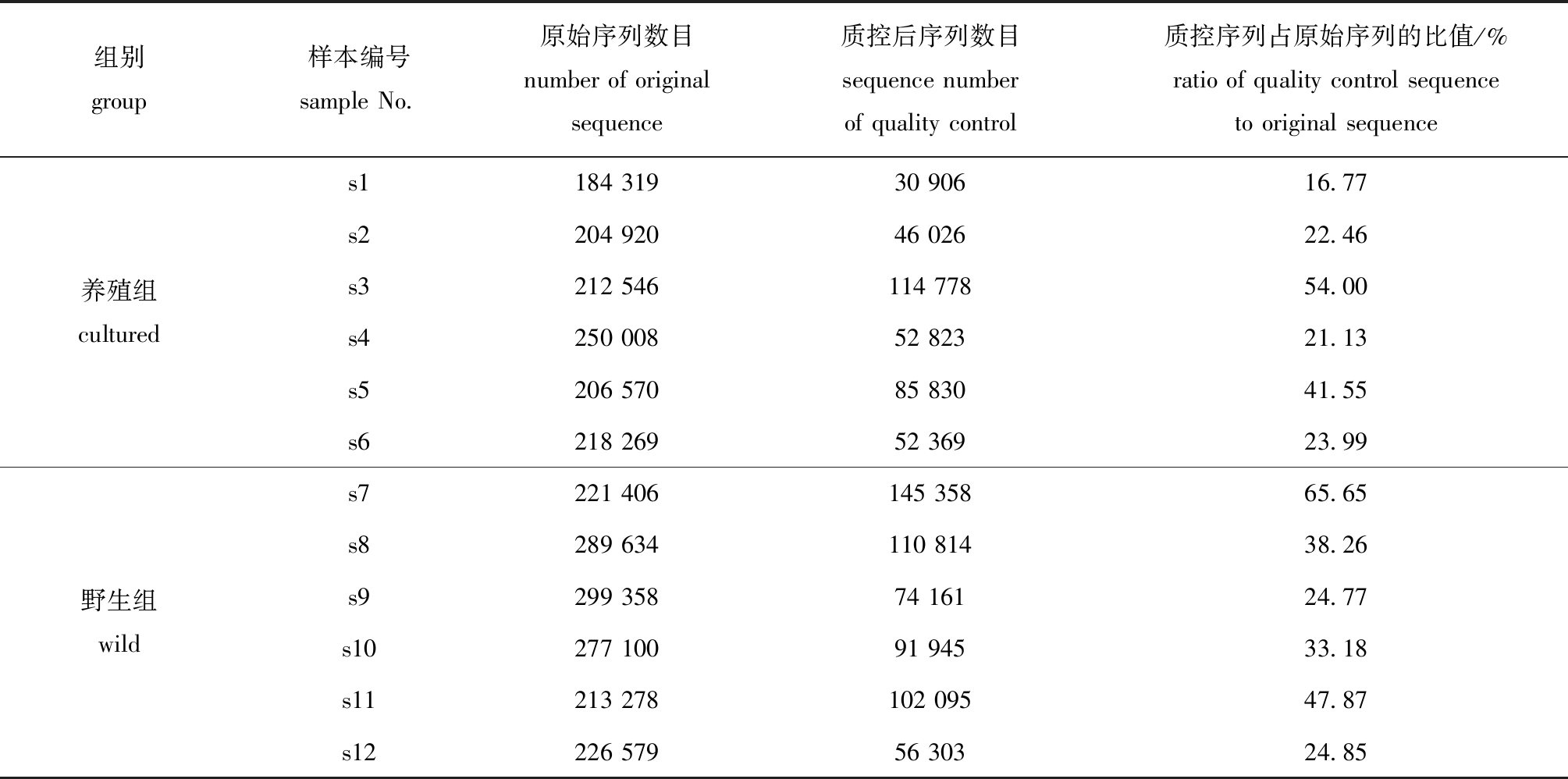
组别group样本编号sample No.原始序列数目number of original sequence质控后序列数目sequence number of quality control质控序列占原始序列的比值/%ratio of quality control sequence to original sequence 养殖组cultureds1184 31930 90616.77s2204 92046 02622.46s3212 546114 77854.00s4250 00852 82321.13s5206 57085 83041.55s6218 26952 36923.99野生组wilds7221 406145 35865.65s8289 634110 81438.26s9299 35874 16124.77s10277 10091 94533.18s11213 278102 09547.87s12226 57956 30324.85
利用稀释曲线(rarefaction curve)对白甲鱼皮肤细菌菌群的测序深度和丰富度进行分析。从图2(a)可见,当测序序列条数达到2.0万条以上时,随着测序深度的加深,台湾白甲鱼皮肤细菌的稀释度曲线趋近饱和,这说明本次测序结果能客观、真实地反映白甲鱼皮肤细菌的真实组成。从图2(b)可见,台湾白甲鱼皮肤细菌测序获得的序列条数在门phylum、纲class、目order、科family和属genus这5个水平上差别不大,但在种species的水平上测序所获得的序列条数有差异。
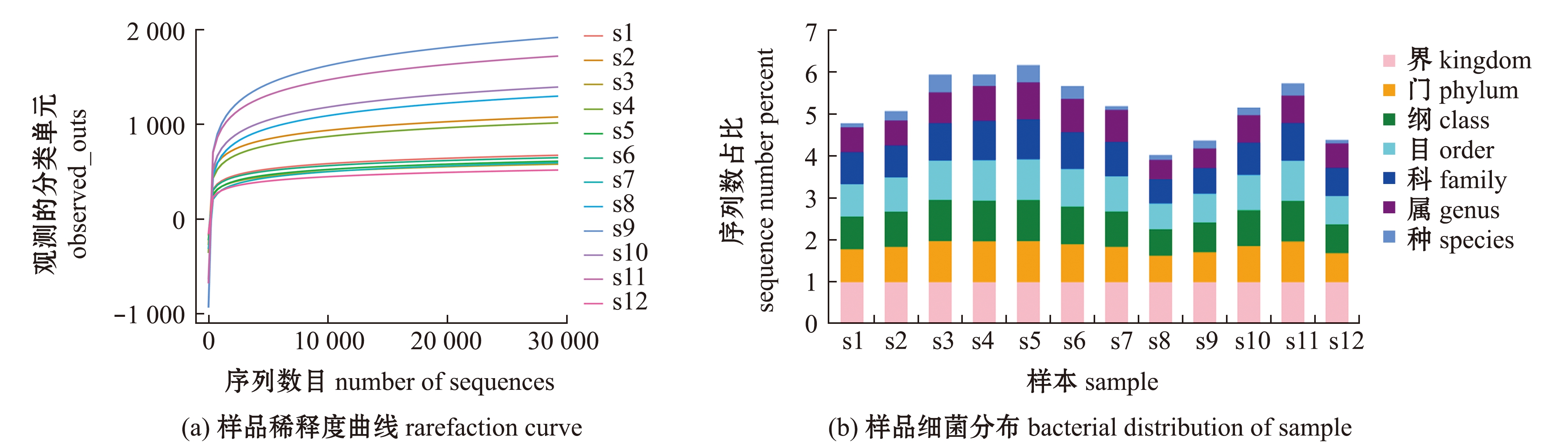
图2 台湾白甲鱼皮肤细菌测序数据特征
Fig.2 Characteristics of skin bacterial sequencing data of Onychostoma barbatulum Pellegrin
2.3 白甲鱼皮肤细菌组成
将测序获得的序列与SILVA数据库中的微生物进行比对和注释,获得台湾白甲鱼皮肤微生物样本中细菌在门和属水平的组成情况。从门水平上分析显示,台湾白甲鱼样本皮肤细菌主要集中在变形菌门Proteobacteria、厚壁菌门Firmicutes、拟杆菌门Bacteroidetes和放线菌门Actinobacteria,还有少量菌分布在疣微菌门Verrucomicrobia、梭杆菌门Fusobacteria和酸杆菌门Acidobacteria等;尽管台湾白甲鱼样本中细菌在门水平的组成种类差别不大,但不同白甲鱼个体和不同环境的白甲鱼皮肤细菌的比例在门水平上有明显的差别(图3)。
从属水平上分析显示,台湾白甲鱼皮肤样本细菌主要集中在嗜氢菌属Hydrogenophilus、芽孢杆菌属Bacillus、普雷沃菌属Prevotella、变形杆菌属Proteus、鳗利斯顿氏菌属Limnohabitans、水栖菌属Enhydrobacter和纤维弧菌属Cellvibrio等,不同白甲鱼个体和不同环境的白甲鱼皮肤细菌的比例在属水平上也有明显的差别(图4)。
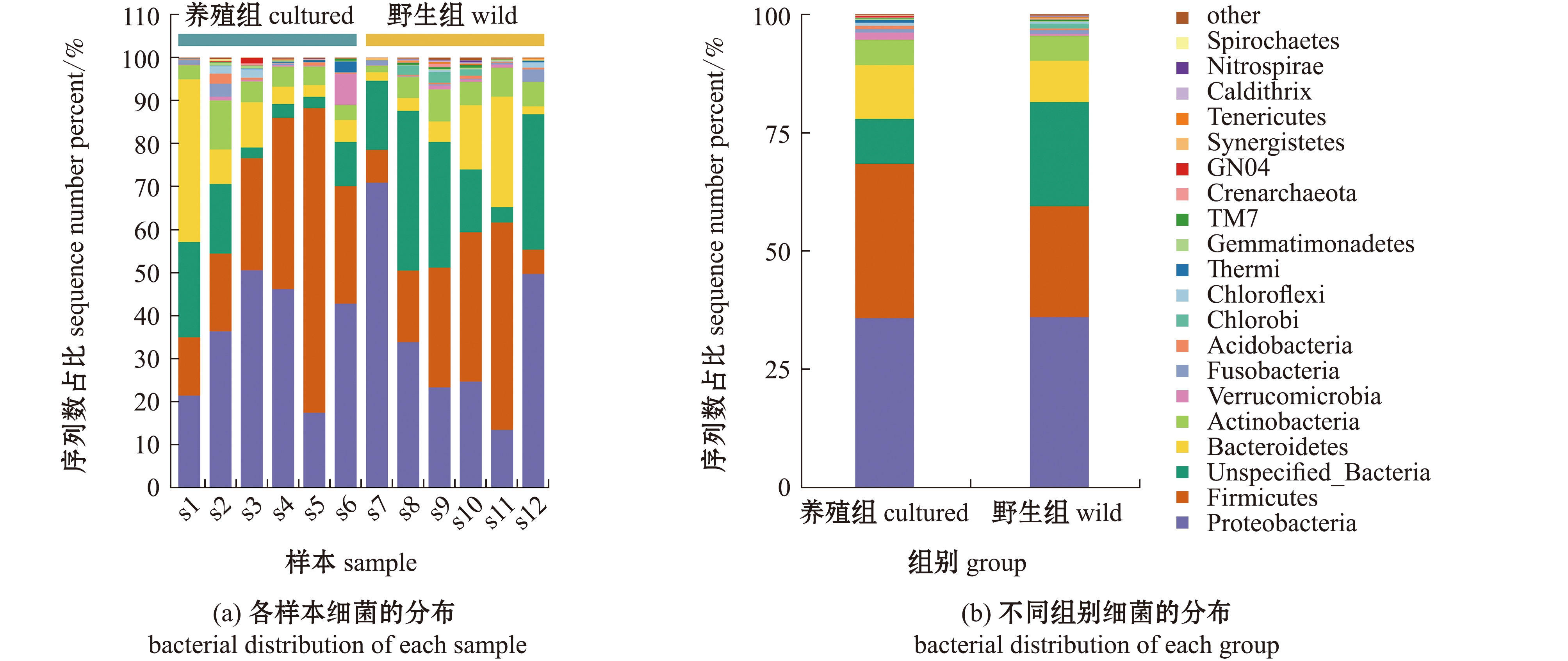
图3 台湾白甲鱼皮肤细菌基于门水平的分布
Fig.3 Distribution of skin bacteria in Onychostoma barbatulum Pellegrin based on the phylum level
2.4 白甲鱼皮肤细菌的优势菌分析
采用热图(heatmap)法对台湾白甲鱼皮肤样本中含量前20的菌进行分析,发现这些细菌在门水平上主要集中在变形菌门、厚壁菌门、拟杆菌门和放线菌门(图5(a))。
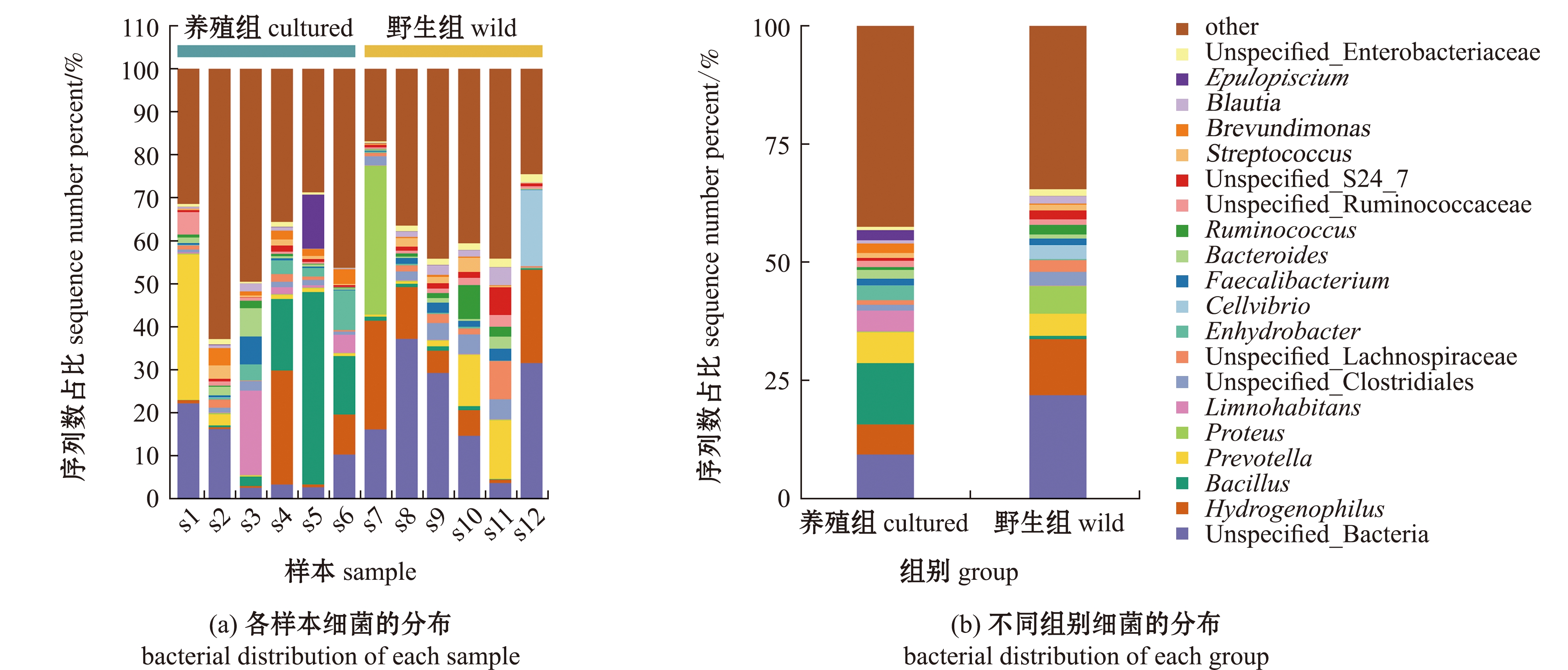
图4 台湾白甲鱼皮肤细菌基于属水平的分布
Fig.4 Distribution of skin bacteria in Onychostoma barbatulum Pellegrin based on the genus level
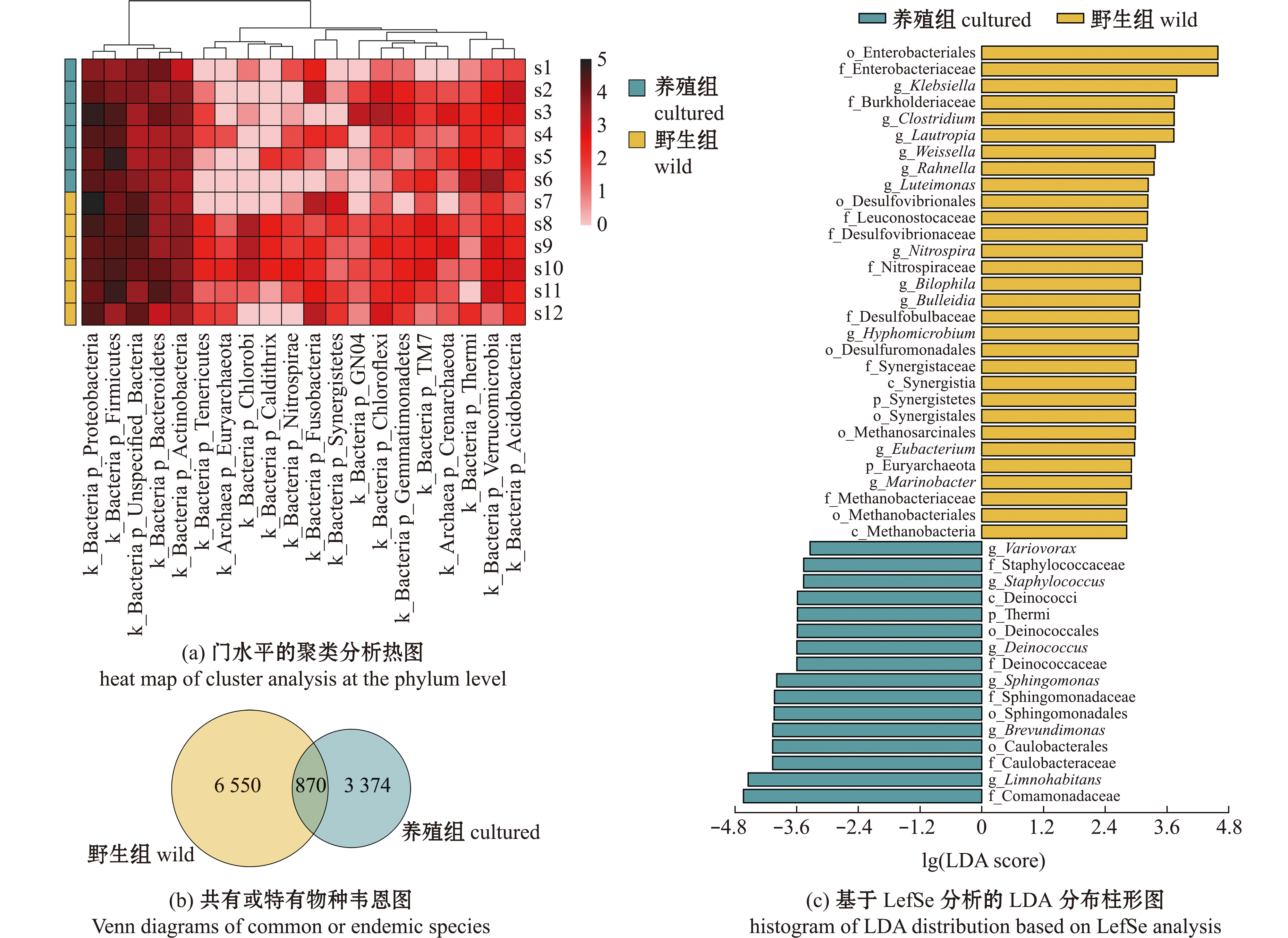
图5 台湾白甲鱼皮肤细菌中优势菌分析
Fig.5 Heatmap analysis of bacterial community in skin microbiome of Onychostoma barbatulum Pellegrin
细菌韦恩图分析显示:养殖白甲鱼皮肤中独有3 374种细菌,野生组白甲鱼皮肤中独有6 550种细菌;它们共有870种细菌,其中,含量最丰富的前8种细菌集中于嗜氢菌属、变形杆菌属、芽孢杆菌属、水栖菌属、普雷沃菌属、刺骨鱼菌属Epulopiscium、纤维弧菌属和雷尔氏菌属Ralstonia(图5(b))。
进一步分析发现:野生组中细菌在互养菌门Synergistetes和广古菌门Euryarchaeota也较多(图5(c))。此外,养殖组中白甲鱼细菌在属水平上集中在贪食菌属Variovorax、葡萄球菌属Staphylococcus、异常球菌属Deinococcus、鞘脂单胞菌属Sphingomonas、短波单胞菌属Brevundimonas和鳗利斯顿氏菌属Limnohabitans;野生组白甲鱼细菌在属水平上集中在克雷伯氏菌属Klebsiella、梭菌属Clostridium、口动菌属Lautropia、魏斯氏菌属Weissella、拉恩氏菌属Rahnella、藤黄单胞菌Luteimonas、硝化螺菌属Nitrospira、嗜胆菌属Bilophila和生丝微菌属Hyphomicrobium(图5(c))。
2.5 白甲鱼皮肤细菌α多样性分析
利用Chao1指数、Shannon指数和Simpson指数对上述台湾白甲鱼皮肤细菌的测序数据进行α多样性分析。从图6可见:野生组的Chao1指数平均值为1 750,大于养殖组,表明野生组物种数较多;养殖组的Shannon指数和Simpson指数分别为7.0和0.975,野生组的Shannon指数和Simpson指数分别为7.5和0.981,表明野生组白甲鱼皮肤细菌的丰富度较高且均匀度较好。Wilcox检验计算发现,养殖组和野生组的Chao1指数、Shannon指数和Simpson指数均不存在显著性差异(P>0.05)。

图6 台湾白甲鱼皮肤细菌的α多样性分析
Fig.6 α diversity of skin bacteria of Onychostoma barbatulum Pellegrin
2.6 白甲鱼皮肤细菌β多样性分析
基于白甲鱼皮肤细菌β多样性进行上述台湾白甲鱼样本间的聚类分析发现,养殖组和野生组的白甲鱼皮肤细菌除个别样本(如s2和s7)外,同一组的样本基本聚在一起(图7(a))。利用样本的OTU丰度信息计算Bray Curtis,并用主坐标分析(PCoA)法分析以上样本的细菌群落结构差异,发现两个组别的白甲鱼皮肤细菌群落具有一定相似性(图7(b))。
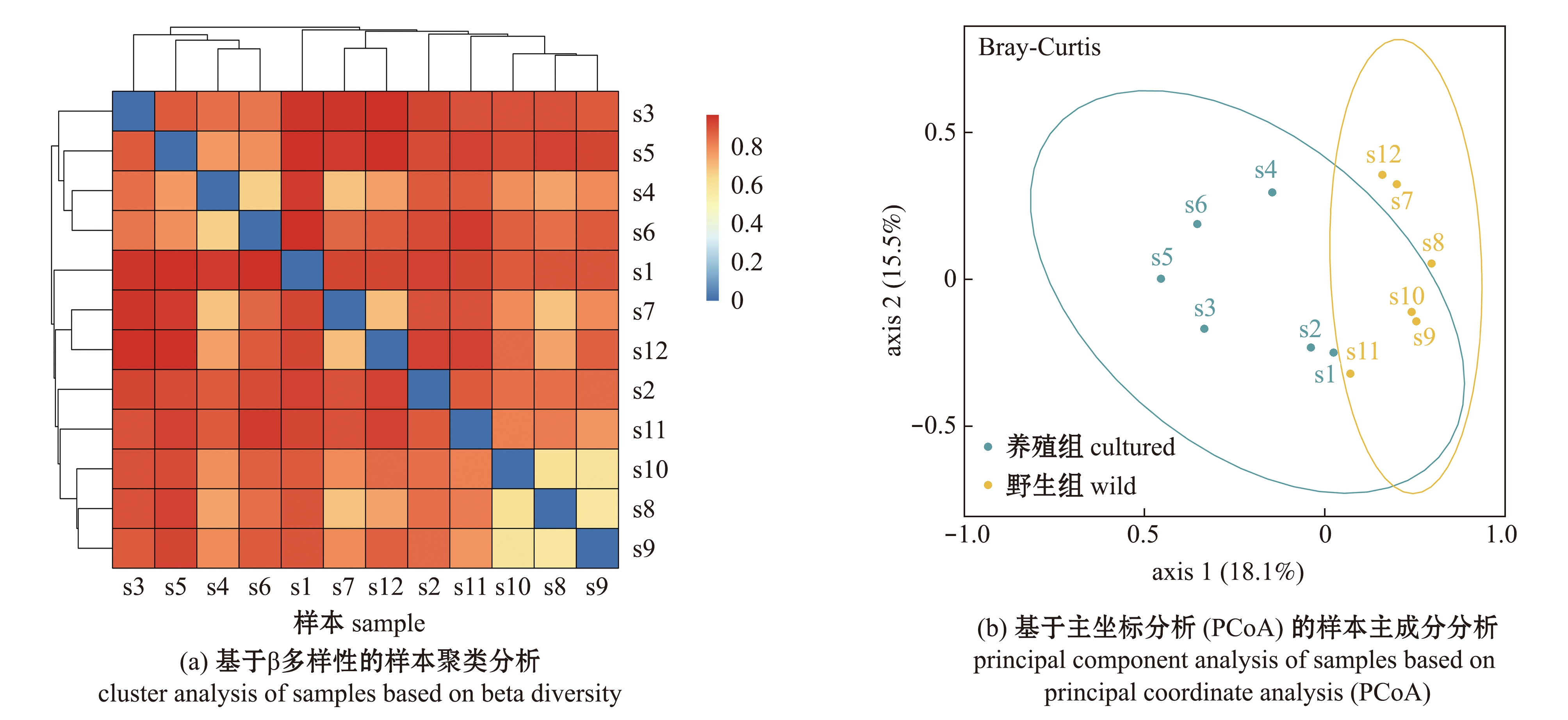
图7 台湾白甲鱼皮肤细菌的β多样性分析
Fig.7 β diversity of skin bacteria of Onychostoma barbatulum Pellegrin
2.7 白甲鱼皮肤细菌功能分析
将测序获得的台湾白甲鱼样本细菌信息与KEGG数据库的代谢通路结合,预测白甲鱼皮肤细菌群落功能。由预测结果可知,以上样本细菌群落中的细菌主要参与氨基酸代谢、碳水化合物代谢、辅因子与维生素代谢、脂代谢、次级代谢产物合成、能量代谢和异生物质代谢等方面。有趣的是,无论是个体水平(图8(a)),还是野生和养殖组的平均水平(图8(b)),参与上述代谢过程的微生物数量差别均不显著,暗示这些细菌对白甲鱼生长是重要的。

图8 台湾白甲鱼皮肤细菌功能分析
Fig.8 Functional analysis of skin bacteria of Onychostoma barbatulum Pellegrin
3 讨论
3.1 台湾白甲鱼皮肤细菌的多样性
白甲鱼是一种重要的经济鱼类,对其进行研究可为加强该鱼种的繁殖、保护等提供科学依据。本研究中,首次采用高通量测序技术对台湾白甲鱼皮肤细菌群落组成进行分析,结果表明,变形菌门、厚壁菌门、拟杆菌门、放线菌门、疣微菌门、梭杆菌门和酸杆菌门是台湾白甲鱼皮肤细菌中丰度最高的菌群。苏月华等[13]报道梭杆菌门和变形菌门是白甲鱼肠道中丰富度最高的菌群,而Wu等[21]通过高通量测序方法也报道梭杆菌门和变形菌门是草鱼Ctenopharyngodon idella肠道内重要的菌群。由此推测,梭杆菌门和变形菌门在鱼的生长中发挥了非常重要的功能。同时,本研究中从台湾白甲鱼皮肤细菌中鉴定的厚壁菌门、拟杆菌门、放线菌门、疣微菌门和酸杆菌门在白甲鱼的肠道中也广泛存在[13,22],这表明以上菌群是白甲鱼中广泛存在的菌群。
3.2 台湾白甲鱼皮肤细菌的特有菌群
从菌群属水平上看,本研究中首次利用高通量测序技术在两个不同环境中生活的白甲鱼皮肤上鉴定出含量丰富的共有菌群,如嗜氢菌属、变形杆菌属、芽孢杆菌属、水栖菌属、普雷沃菌属、刺骨鱼菌属、纤维弧菌属和雷尔氏菌属,这是台湾白甲鱼皮肤细菌群落属水平的首次报道。现有文献中,仅见南极鱼皮肤微生物中存在芽孢杆菌属[23],这说明鱼类皮肤存在芽孢杆菌属菌落。与已报道的白甲鱼肠道中存在的属水平菌落[13,22]相比,本研究中发现的嗜氢菌属、芽孢杆菌属、水栖菌属、普雷沃菌属、刺骨鱼菌属、纤维弧菌属和雷尔氏菌属均不存在于肠道中,暗示这些菌仅在白甲鱼皮肤上定植。此外,养殖组白甲鱼皮肤细菌主要分布于贪食菌属、葡萄球菌属、异常球菌属、鞘脂单胞菌属和短波单胞菌属,而野生组白甲鱼皮肤细菌主要分布于口动菌属、魏斯氏菌属、拉恩氏菌属、藤黄单胞菌属和硝化螺菌属等,表明这些细菌的分布可能与白甲鱼生活环境差异有关。
3.3 台湾白甲鱼皮肤细菌群落的功能
本研究中,利用Chao1指数、Shannon指数和Simpson指数对两个不同环境的台湾白甲鱼皮肤细菌组成进行α多样性分析发现,尽管两个不同环境的白甲鱼皮肤细菌组成、丰富度及均匀度存在差异,但在统计学上并不具有显著性差异,这说明白甲鱼皮肤细菌组成相对稳定。利用KEGG数据库对样本中细菌群落功能进行分析发现,细菌群落丰富度高的菌群主要集中于氨基酸代谢、碳水化合物代谢、辅因子与维生素代谢、脂代谢、次级代谢产物合成和能量代谢等通路,这说明细菌菌群广泛参与白甲鱼的生长、代谢活动,对其生长是非常重要的。
本研究中,充分利用16S rRNA高通量测序技术对台湾白甲鱼皮肤细菌组成进行分析,最大限度地还原其皮肤细菌群落结构与丰度;同时,结合KEGG数据库对细菌群落功能进行预测,研究结果可为后续白甲鱼的繁育、保护提供数据支持。同时,鉴于该测序技术的局限性,后续可通过宏基因组技术对台湾白甲鱼皮肤细菌进行更深入的功能分析。
4 结论
1)养殖和野生台湾白甲鱼皮肤细菌菌群主要分布于18个门,其中,变形菌门、厚壁菌门、拟杆菌门和放线菌门的菌落数最多;从属水平上看,菌落含量丰富度排名前8的菌主要分布在嗜氢菌属、变形杆菌属、芽孢杆菌属、水栖菌属、普雷沃菌属、刺骨鱼菌属、纤维弧菌属和雷尔氏菌属,这是白甲鱼皮肤细菌组成的首次报道。
2)从台湾白甲鱼皮肤细菌中鉴定出的厚壁菌门、拟杆菌门、放线菌门、疣微菌门和酸杆菌门也存在于白甲鱼的肠道中,说明这些菌群是白甲鱼中广泛存在的菌群。
3)嗜氢菌属、芽孢杆菌属、水栖菌属、普雷沃菌属、刺骨鱼菌属、纤维弧菌属和雷尔氏菌属仅存在于白甲鱼皮肤中,暗示其是皮肤特有菌群。
4)结合KEGG数据分析发现,白甲鱼皮肤细菌菌群主要参与氨基酸代谢、碳水化合物代谢、辅因子与维生素代谢、脂代谢、次级代谢产物合成和能量代谢等通路,说明这些细菌对白甲鱼的生长是非常重要的。
[1] JOANNIS J D.Description d’une nouvelle espèce de la famille des Cyprinidae[J].Bulletin DU Museum Nationald Historie Natrellesection B Adansonia,1908,14(6):262-275.
[2] 单向红,乐佩琦,林人瑞.中国动物志—硬骨鱼纲—鲤形目:下卷[M].北京:科学出版社,2000.
SHAN X H,LE P Q,LIN R R.Zoology of China—Bonefish—Cyprinids:Volume 2[M].Beijing:Science Press,2000.(in Chinese)
[3] 尹邦一,代应贵.白甲鱼属鱼类研究进展[J].水产科学,2014,33(1):63-68.
YIN B Y,DAI Y G.A review of research progress in fish Onychostoma[J].Fisheries Science,2014,33(1):63-68.(in Chinese)
[4] 吕彤,郭乔羽,王丁.为长江上游的珍稀和特有鱼类保持生态流[J].世界环境,2009(1):78-79.
LÜ T,GUO Q Y,WANG D.Maintaining the ecological flow for the rare and special fishes in the upper reaches of the Yangtze River[J].World Environment,2009(1):78-79.(in Chinese)
[5] 冉光鑫,代应贵,岳晓烔.稀有白甲鱼西江种群mtDNA D环区的结构及遗传多样性[J].上海海洋大学学报,2012,21(2):176-182.
RAN G X,DAI Y G,YUE X T.Structure and genetic diversity of mtDNA D-loop in the population of Onychostoma rara from the Xijiang River[J].Journal of Shanghai Ocean University,2012,21(2):176-182.(in Chinese)
[6] 代应贵,岳晓烔,尹邦一.濒危鱼类稀有白甲鱼外周血细胞特征[J].动物学杂志,2014,49(6):875-885.
DAI Y G,YUE X T,YIN B Y.Characteristics of peripheral hematocytes in endangered fish Onychostoma rara[J].Chinese Journal of Zoology,2014,49(6):875-885.(in Chinese)
[7] 董武子,王涛,马力,等.秦巴山区多鳞白甲鱼人工繁殖试验[J].畜牧兽医杂志,2016,35(3):27-30,35.
DONG W Z,WANG T,MA L,et al.Artificial reproduction of Varicorhinus macrolepis in Qin-Ba Mountains[J].Journal of Animal Science and Veterinary Medicine,2016,35(3):27-30,35.(in Chinese)
[8] 马跃岗,朱杰,袁万安.长江白甲鱼ITS2序列结构和群体遗传多样性[J].生态学杂志,2012,31(3):670-675.
MA Y G,ZHU J,YUAN W A.ITS2 structure and population genetic diversity of Onychostoma sima (Sauvage) in Yangtzi River[J].Chinese Journal of Ecology,2012,31(3):670-675.(in Chinese)
[9] 彭珊,代应贵.濒危鱼类稀有白甲鱼清水江种群mtDNA D-loop序列多态性[J].水产学报,2009,33(2):196-200.
PENG S,DAI Y G.Sequence polymorphism of mtDNA D-loop in the population of the endangered species Onychostoma rara from the Qingshui River[J].Journal of Fisheries of China,2009,33(2):196-200.(in Chinese)
[10] 陈先均,周剑,李孟均.白甲鱼生物学特征与繁殖技术初探[J].江苏农业科学,2008,36(6):222-223.
CHEN X J,ZHOU J,LI M J.A preliminary study of biological characters and propagation technique for Onychostoma sima[J].Jiangsu Agricultural Sciences,2008,36(6):222-223.(in Chinese)
[11] 代应贵,王晓辉.稀有白甲鱼含肉率及肌肉营养成分分析[J].水产科学,2007,26(1):7-11.
DAI Y G,WANG X H.Dressed ratio and nutritive composition of Onychostoma rara[J].Fisheries Science,2007,26(1):7-11.(in Chinese)
[12] 周兴华,郑曙明,吴青,等.白甲鱼肌肉营养成分与品质的评价[J].西南大学学报(自然科学版),2007,29(8):123-128.
ZHOU X H,ZHENG S M,WU Q,et al.Evaluation of nutritive quality and nutritional components in Ongchostoma sima[J].Journal of Southwest University (Natural Science Edition),2007,29(8):123-128.(in Chinese)
[13] 苏月华,陈少威,吴程,等.基于高通量测序技术研究白甲鱼肠道微生物群落组成[J].福建师范大学学报(自然科学版),2018,34(6):63-71.
SU Y H,CHEN S W,WU C,et al.Microbial community in the gut of Onychostoma sima studied by high-throughput sequencing[J].Journal of Fujian Normal University (Natural Science Edition),2018,34(6):63-71.(in Chinese)
[14] 赵裕栋,周俊,何璟.土壤微生物总DNA提取方法的优化[J].微生物学报,2012,52(9):1143-1150.
ZHAO Y D,ZHOU J,HE J.Optimization of soil microbial DNA isolation[J].Acta Microbiologica Sinica,2012,52(9):1143-1150.(in Chinese)
![]() T,SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
T,SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[16] BOLYEN E,RIDEOUT J R,DILLON M R,et al.Reproducible,interactive,scalable and extensible microbiome data science using QIIME 2[J].Nature Biotechnology,2019,37(8):852-857.
[17] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[18] EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[19] WANG Q,GARRITY G M,TIEDJE J M,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Applied and Environmental Microbiology,2007,73(16):5261-5267.
[20] QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucleic Acids Research,2013,41:D590-D596.
[21] WU S G,WANG G T,ANGERT E R,et al.Composition,diversity,and origin of the bacterial community in grass carp intestine[J].PLoS One,2012,7(2):e30440.
[22] 蔡斌斌.白甲鱼肠道微生物多样性及其功能分析[D].福州:福建师范大学,2017.
CAI B B.Diversity and function analysis of the intestine microbes of Onychostoma sima[D].Fuzhou:Fujian Normal University,2017.(in Chinese)
[23] 赵文静,许强华.伯氏肩孔南极鱼和革首南极鱼皮肤微生物多样性的研究[J].大连海洋大学学报,2019,34(6):792-797.
ZHAO W J,XU Q H.Microbial diversity on skins in two species of Antarctic fish,Trematomus bernacchii and Notothenia coriiceps[J].Journal of Dalian Ocean University,2019,34(6):792-797.(in Chinese)