三角鲂Megalobrama terminalis隶属于鲤形目鲂属,俗称三角鳊、钱塘江塔鳊和法罗等,其生长快、食性广,最大个体可达5 kg。三角鲂自古被视为鱼中上品,是中国重要的土著经济鱼类之一,主要分布在中国的长江水系、黄河水系和黑龙江水系等淡水区域[1]。历史上钱塘江水系具有丰富的三角鲂野生资源,三角鲂产黏性卵,产卵场位于富春江中上游的毕家滩、施家滩及坝下的舒湾滩等处,据不完全统计,1956年三角鲂捕捞量高达100 t之多[2]。多年来,由于非法挖沙对产卵场的破坏,以及生活和工业废水排放、电站桥梁等水工建筑的修建造成的水域环境破坏,钱塘江渔业资源急剧衰退,特别是鲴Xenocypris、三角鲂等土著性经济鱼类资源衰退严重[3]。郝雅宾等[4]于2014年11月—2016 年11 月对钱塘江下游段鱼类调查显示,优势种为光泽黄颡鱼Tachysurus nitidus、鲫Carassius auratus、鲢Hypophthalmichthys molitrix和鳙Hypophthalmichthys nobilis,渔获物中三角鲂占比无论是数量还是质量均不在前五位优势种之列,钱塘江三角鲂野生资源日益衰退,资源保护迫在眉睫。
水产资源保护的核心是种质保护,有效措施之一是增殖放流。为实现对钱塘江三角鲂种质资源的有效保护,1998年,原浙江省水产局和原杭州市水产科学研究所联合申请,在三角鲂主要原产地杭州市建立了国家级钱塘江三角鲂原种场。该原种场重点开展了原种亲本的收集、保存和后备亲本的培育,拥有占地6.67 hm2的保种、培育池塘,以及位于千岛湖铜山水库面积约133.4 hm2的后备亲本保存基地[5-6],原种经过人工选育,繁育生产的后备亲本主要服务于周边良种场。良种场通过扩繁殖,生产苗种用于增殖放流或池塘养殖。三角鲂的规模化增殖放流始于2003年,据杭州市渔政渔港监督管理总站不完全统计,仅2018 年钱塘江中下游就放流了三角鲂冬片54 000 kg。因此, 检测良种场亲本群体遗传多样性现状及其种群间的遗传分化对指导科学增殖放流具有重要意义。
目前,关于三角鲂的相关研究较多,主要集中在不同地理种群的形态差异分析[7-8]、染色体核型分析[9]、指纹图谱构建[10]、鲂属种间遗传关系分析[11]等方面。多位学者对线粒体基因和线粒体基因组进行了测序和比较分析[12-13],也有少量关于转录组[14]、增殖放流效果评估[15]及基于COⅠ序列研究种群遗传多样性[16]的研究报道。
遗传多样性及遗传结构是衡量种质优劣的重要指标。微卫星标记(microsatellite markers) 具有易扩增、共显性及多态性高等优点,被广泛应用于遗传连锁图谱绘制、群体遗传学研究、种质鉴定与亲权分析等方面[17]。本研究中,利用微卫星分子标记结合毛细管电泳基因分型技术,以钱塘江中下游4个良种场亲本群体和1个钱塘江自然捕捞群体为研究对象,对三角鲂不同群体的遗传多样性及遗传结构进行分析,客观评价良种场三角鲂亲本及自然捕捞群体遗传多样性现状,以期为三角鲂科学增殖放流及从源头上控制放流苗种种质质量提供科学依据和数据支撑。
1 材料与方法
1.1 材料
试验用5个群体768尾样本来源于钱塘江中下游的良种场和自然江段。其中,4个亲本群体于2018年4—6月采集于各良种场,采集对象为当年参与繁殖的亲本;自然捕捞群体样本采自钱塘江中下游的桐庐、新桐乡、富阳、一桥和六桥等常规渔业资源监测网点,采样时间为2018年7月—2019年4月(放流之后采集);样本取样部位均为尾鳍,所有样本编号保存于分析纯乙醇中带回实验室备用。各群体样本的采集信息详见表1。
表1 三角鲂样本采集信息
Tab.1 Sample collecting information on black bream Megalobrama terminalis

群体 population采集地 collecting site样本数量/ind. sample number采集日期 date景山 JS湖州市东林镇景山良种场 Huzhou Donglin Jingshan Hatchery1962018-04-22瓜沥镇 GLZ杭州市萧山瓜沥镇柏松良种场 Hangzhou Xiaoshan Guali Baisong Hatchery752018-05-09萧乡 XX杭州市萧乡良种场 Hangzhou Xiaoxiang Hatchery142018-05-19明兴 MX湖州市明兴稀有野生水产良种场 Huzhou Mingxing Rare Wild Fishery Hatchery162018-06-01自然捕捞 BL钱塘江中下游 Middle and Lower Qiantang River4672018-07—2019-04
1.2 方法
1.2.1 DNA提取与引物设计 采用TIANamp Marine Animals DNA Kit进行DNA提取,用经Nared染色的质量分数为0.8%的琼脂糖凝胶电泳进行检测,检测合格的DNA置于-20 ℃下保存备用。微卫星引物位点、序列及其上游引物的5′端荧光素标记参照文献[15]并稍做改进,去掉无效等位基因频率大于0.2的4个微卫星位点(SJ4、SJ9、SJ13、SJ21),合成8对高效荧光标记引物。引物由生工生物工程(上海)股份有限公司合成,引物详细信息见表2。
1.2.2 PCR扩增与测序 PCR反应试剂盒(TaqTM Version 2.0)购自宝生物工程(大连)有限公司。PCR反应体系(共10 μL):Premix Taq 5 μL,双蒸水3.8 μL,10 μmol/L正、反向引物各0.1 μL,20 μmol/L DNA模板1 μL。PCR反应条件:94 ℃下预变性3 min;94 ℃下变性30 s,50~60 ℃下退火30 s,72 ℃下延伸40 s,共进行30个循环;最后在72 ℃下延伸8 min,4 ℃下保存。PCR扩增产物经质量分数为2%的琼脂糖凝胶电泳检验,用全自动凝胶成像分析系统(Syngene)观察目的条带的扩增效果。扩增效果好的产物送生工生物工程(上海)股份有限公司在ABI3730 全自动测序仪上进行毛细管电泳测序和基因分型。采用GeneMapperv 4.1软件读取等位基因大小,最终确定其基因型。
1.2.3 参数统计及分子方差分析 将检测到的微卫星分型结果,利用Excel 2010 软件整理成个体对应的等位基因型数据。采用GenAlEx 6.501软件[18]分析8个微卫星位点的等位基因数(Na)、有效等位基因数(N e)、Shannon’s信息指数(I)、观测杂合度(Ho)、期望杂合度(He)、特有等位基因数(private alleles)和固定指数(fixation index,F)等遗传多样性参数,计算群体间的Nei’s遗传距离,基于群体和个体间遗传距离进行主坐标分析(principal coordinates analysis,PCoA);采用Arlequin 3.5软件[19]进行分子方差分析(AMOVA),并统计群体间遗传分化指数(FST)。相关计算公式为
F=(He-Ho)/He=1-(Ho/He),
I=-∑(pi×ln pi)。
其中,pi为第i个等位基因的频率。
表2 三角鲂8个微卫星引物基本信息
Tab.2 Basic information of eight microsatellite primers from black bream Megalobrama terminalis

位点 locus荧光标记 dye label引物序列 primer sequence(5'-3')重复序列 repeat motif产物范围/bp product range退火温度/℃ annealing temperatureSJ14FAMF: AAAAAAAGAACGAGAGGGR:TGATGATTTGGAGGAAGT(GA)15210~25050.0SJ18HEXF: CCATCCAACACGCCGACTCR: GCGGGAAATCACTTCATA-ACTCAA(AC)15290~38061.5SJ22FAMF: ATGTATTGGGTTGAGGTTR: GAGCTATGGACTCCGTTAT(TG)14250~30653.0SJ23FAMF: CTTACAGACTCCGACAGGR: ATCCACGACTTCCAGAAC(AC)12206~29857.0SJ25HEXF: GGTACTGTTTGTGATGGGCR: CTGCTCACTCAACTTATTG-TAGGTC(GT)16117~15160.0SJ27HEXF: GGCTATTCTCACCA-CATTTCGTGR: GGCTGCACCATATGATA-AAAACAC(TTG)8120~16054.0SJ28TAMF: TGGAGTTAGTGTCCGCTTGTR: AGGATACGGGTGAGTTCG(TG)13320~36656.0SJ31TAMF: GAACCTGAATGAGGACATA-AAGGTR: TAACGCACAGAATTTCA-CATAACC(TG)8212~25853.0
1.2.4 遗传结构分析 将整理好的基因型数据导入Structure 2.3.4软件[20],设置参数,将类群数K设置为1~9, 每个K重复5次,采用Structure harvester软件[21]分析最佳K值。参照Evanno等[22]的方法,选取ΔK最大值对应的K作为最佳K值,对Structure运算结果进行重复抽样分析,得到最佳K值对应的Q值(表示某个体基因变异来源于某一亚群的概率),依据Q值进行Structure图形的绘制,构建5个群体三角鲂参试个体的遗传结构图。
2 结果与分析
2.1 SSR位点多态性及群体遗传多样性
筛选的8对微卫星引物在优化条件下扩增效果良好, PCR产物经毛细管电泳基因分型谱带清晰,杂峰少,最终5个群体共检测出349个等位基因,其中,明兴(MX)群体检测到的等位基因最少(83个),自然捕捞群体(BL)检测到的等位基因最多(312个)。部分位点的基因分型结果见图1。
8个微卫星位点的多态性信息含量(PIC)为0.815~0.967,均为高度多态性位点。从表3可见:三角鲂各群体单个位点检测到的Na平均值为(10±1)~(39±7),Ne平均值为(6.60±0.98)~(14.22±3.08),其中,MX群体的Na和Ne最小,BL群体的这两项指标明显高于其他群体;除极少数位点外(如GLZ、MX和XX群体在SJ23位点,JS群体在SJ14位点,BL群体在SJ31位点),5个群体在绝大多数位点的Ho均大于He(其中,GLZ、MX和XX 3个群体在6个位点上的Ho为1),5个群体的Ho平均值为(0.96±0.02)~(0.98±0.01);绝大多数位点的固定指数为负值,表现出杂合子过剩;5个群体Shannon’s信息指数平均值为(2.02±0.14)~(2.85±0.21);GLZ、JS、MX、XX、BL群体的特有等位基因数依次为5、21、2、0和122个,XX群体未检测到特有等位基因,BL群体检测到的特有等位基因数最多。
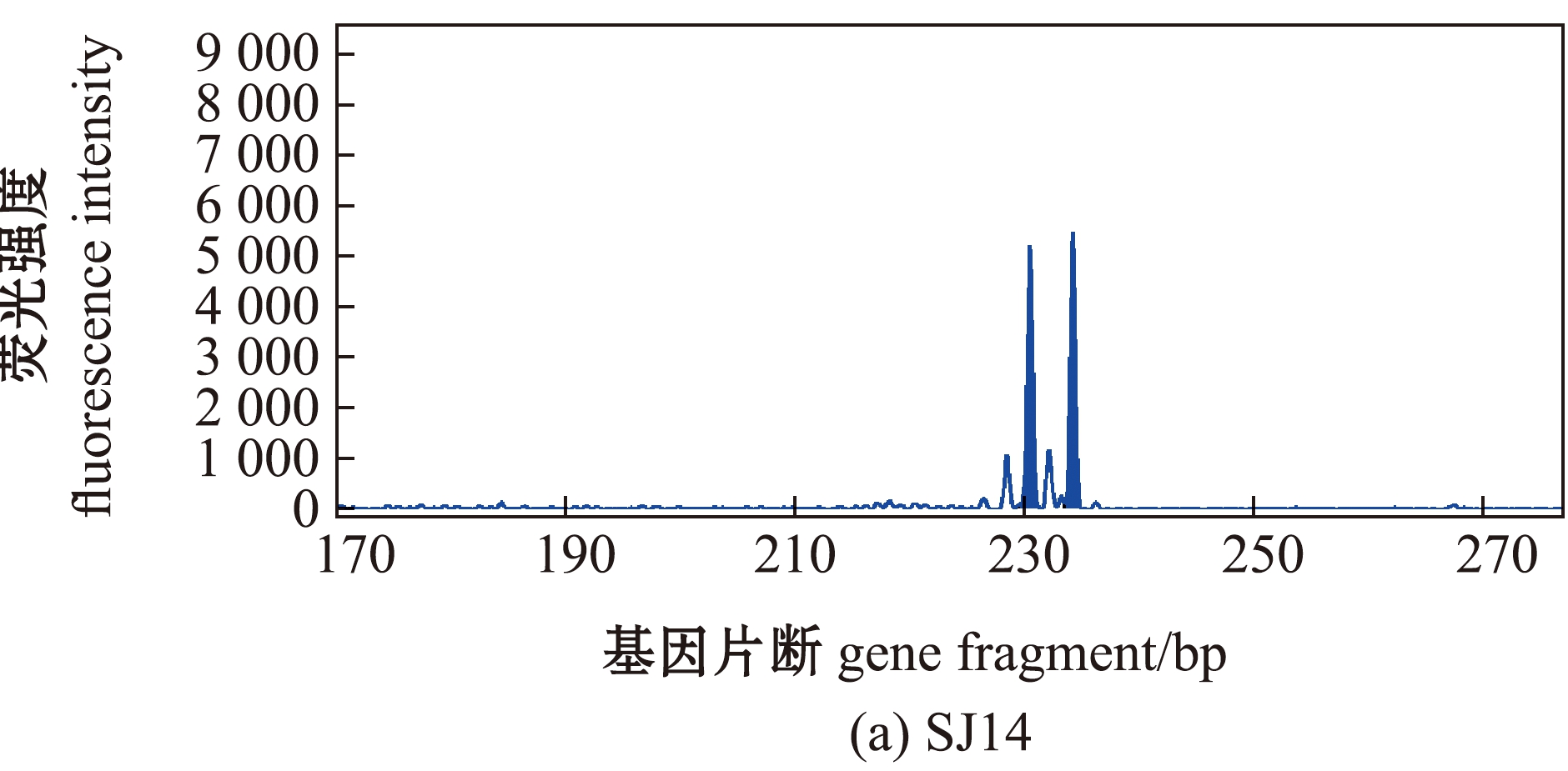
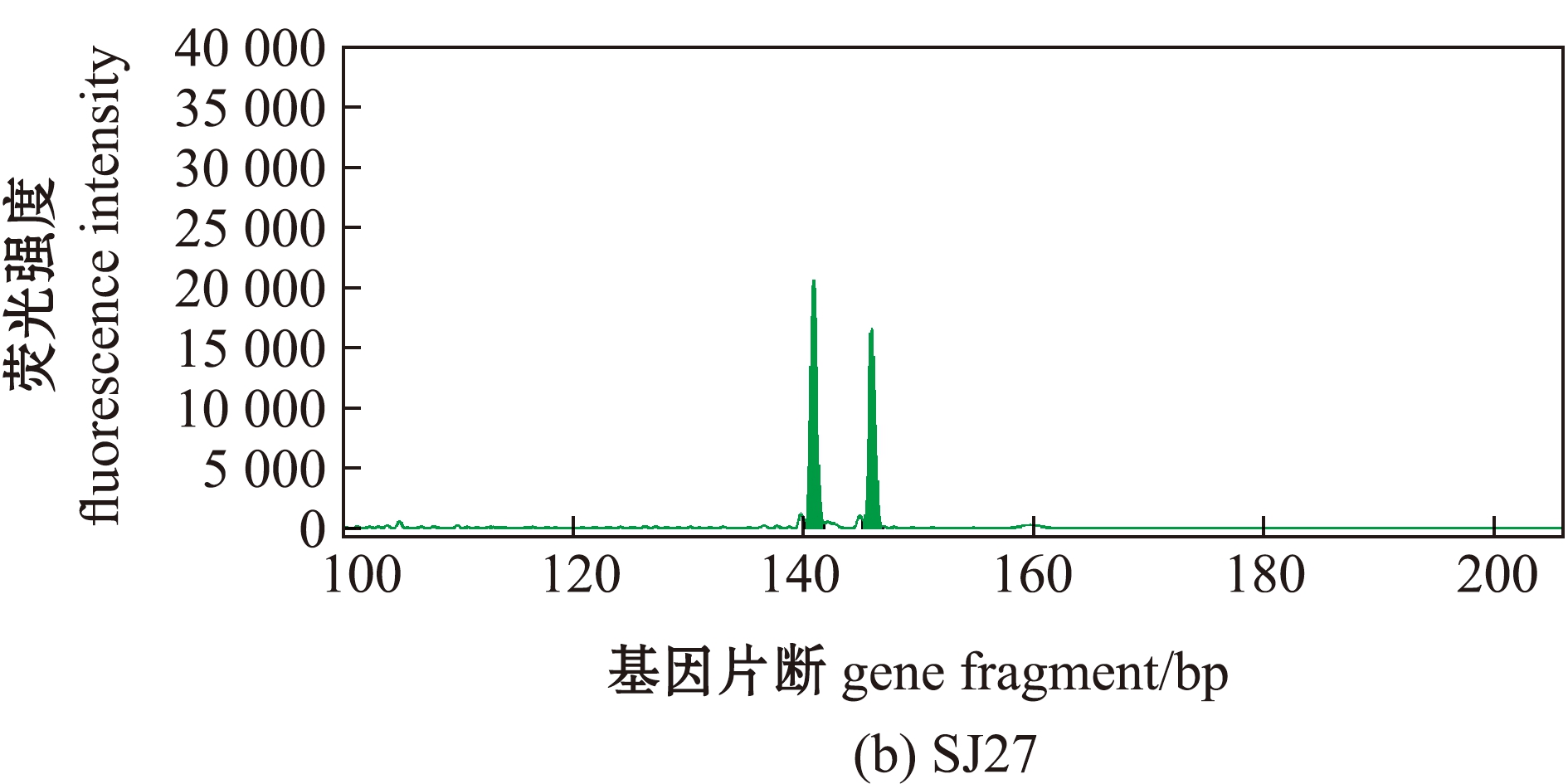
图1 三角鲂微卫星位点SJ14和SJ27的毛细管电泳基因分型结果
Fig.1 Genotyping results of the microsatellite loci SJ14 and SJ27 using capillary electrophoresis in black bream Megalobrama terminalis
表3 三角鲂群体基于8个微卫星位点的遗传多态性参数
Tab.3 Genetic diversity parameter among populations of black bream Megalobrama terminalis base on eight microsatellite loci
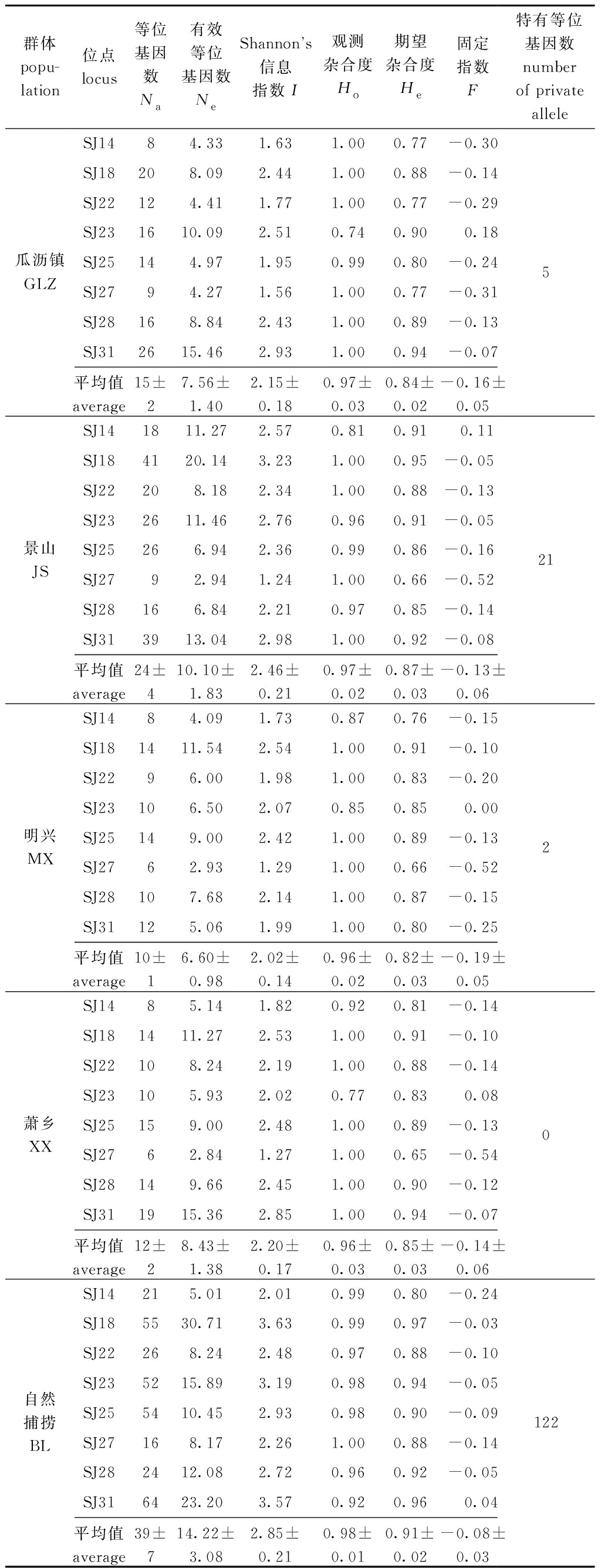
群体popu-lation位点locus等位基因数Na有效等位基因数NeShannons信息指数I观测杂合度Ho期望杂合度He固定指数F特有等位基因数number of privateallele瓜沥镇GLZSJ1484.331.631.000.77-0.30SJ18208.092.441.000.88-0.14SJ22124.411.771.000.77-0.29SJ231610.092.510.740.900.18SJ25144.971.950.990.80-0.24SJ2794.271.561.000.77-0.31SJ28168.842.431.000.89-0.13SJ312615.462.931.000.94-0.07平均值average15±27.56±1.402.15±0.180.97±0.030.84±0.02-0.16±0.055景山JSSJ141811.272.570.810.910.11SJ184120.143.231.000.95-0.05SJ22208.182.341.000.88-0.13SJ232611.462.760.960.91-0.05SJ25266.942.360.990.86-0.16SJ2792.941.241.000.66-0.52SJ28166.842.210.970.85-0.14SJ313913.042.981.000.92-0.08平均值average24±410.10±1.832.46±0.210.97±0.020.87±0.03-0.13±0.0621明兴MXSJ1484.091.730.870.76-0.15SJ181411.542.541.000.91-0.10SJ2296.001.981.000.83-0.20SJ23106.502.070.850.850.00SJ25149.002.421.000.89-0.13SJ2762.931.291.000.66-0.52SJ28107.682.141.000.87-0.15SJ31125.061.991.000.80-0.25平均值average10±16.60±0.982.02±0.140.96±0.020.82±0.03-0.19±0.052萧乡XXSJ1485.141.820.920.81-0.14SJ181411.272.531.000.91-0.10SJ22108.242.191.000.88-0.14SJ23105.932.020.770.830.08SJ25159.002.481.000.89-0.13SJ2762.841.271.000.65-0.54SJ28149.662.451.000.90-0.12SJ311915.362.851.000.94-0.07平均值average12±28.43±1.382.20±0.170.96±0.030.85±0.03-0.14±0.060自然捕捞BLSJ14215.012.010.990.80-0.24SJ185530.713.630.990.97-0.03SJ22268.242.480.970.88-0.10SJ235215.893.190.980.94-0.05SJ255410.452.930.980.90-0.09SJ27168.172.261.000.88-0.14SJ282412.082.720.960.92-0.05SJ316423.203.570.920.960.04平均值average39±714.22±3.082.85±0.210.98±0.010.91±0.02-0.08±0.03122
2.2 群体遗传分化水平
分子方差分析表明,5个鲂群体的遗传变异主要来自群体内的个体间,占94.47%,群体间的遗传变异仅占5.53%,群体间的遗传分化极显著(P<0.01)(表4)。
表4 三角鲂群体基于微卫星标记的分子方差分析结果
Tab.4 Analysis results of molecular variance by microsatellite markers in populations of black bream Megalobrama terminalis
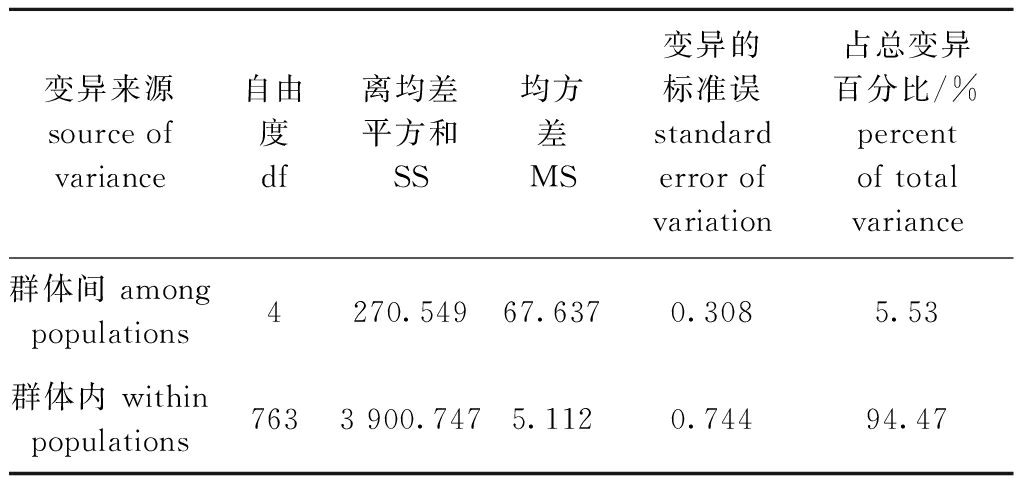
变异来源source of variance自由度df离均差平方和SS均方差MS变异的标准误standard error of variation占总变异百分比/%percent of total variance群体间 among populations4270.54967.6370.3085.53群体内 within populations7633 900.7475.1120.74494.47
鲂群体间遗传分化指数(FST) 分析显示,三角鲂群体间遗传分化指数为0.033~0.076,其中,大多数小于或略大于0.05,只有BL群体与JS和XX两个亲本群体间的遗传分化指数较大,分别为0.064和0.076(表5)。Balloux等[23]以0.05、0.15和0.25为临界值将种群遗传分化划分为很小、中等、较大和很大4个等级。参照此等级划分依据,可知三角鲂4个亲本群体间遗传分化程度较小(0<FST<0.05),BL群体与JS和XX亲本群体间存在中等遗传分化(0.05<FST<0.15)。Nei’s遗传距离分析显示,三角鲂群体间遗传距离为0.216~0.661,4个亲本群体间遗传距离较小,BL群体和亲本群体间遗传距离较大,遗传距离最大值0.661出现在BL群体和XX亲本群体间(表5)。
取前3个主坐标构建主坐标分析图,图2为三角鲂5个群体基于主坐标1(Coord.1)和主坐标2(Coord.2)的聚类图,在主坐标1(Coord.1)上,BL群体和其他亲本群体存在明显分化,5个群体在主坐标1上大致被划分为2个类群,即绝大部分BL群体的个体自成一个类群,BL群体的少数个体和其他4个亲本群体的个体聚集成另一个类群。
表5 三角鲂群体间的遗传分化指数 FST(对角线下)和Nei’s遗传距离(对角线上)
Tab.5 Genetic differentiation FST (below diagonal) and Nei’s genetic distance (above diagonal) among black bream Megalobrama terminalis populations
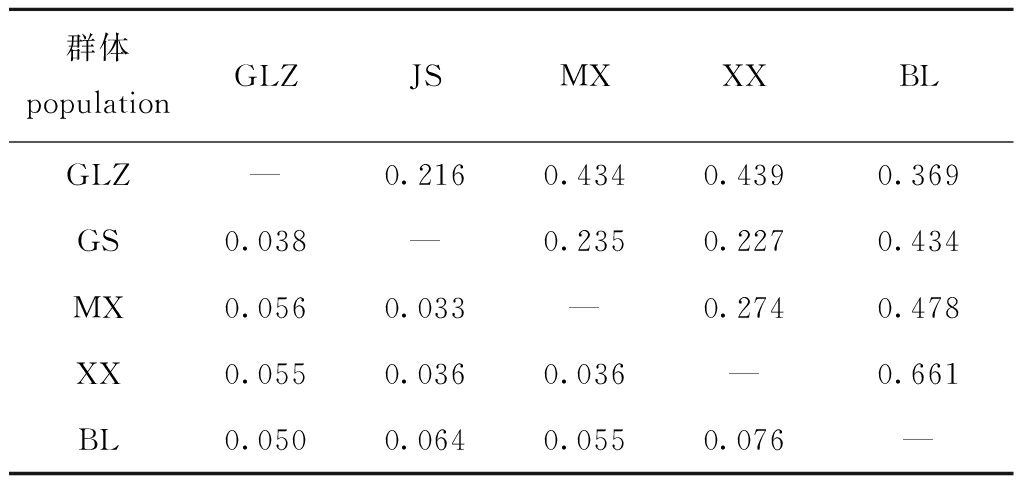
群体populationGLZJSMXXXBLGLZ—0.2160.4340.4390.369GS0.038—0.2350.2270.434MX0.0560.033—0.2740.478XX0.0550.0360.036—0.661BL0.0500.0640.0550.076—
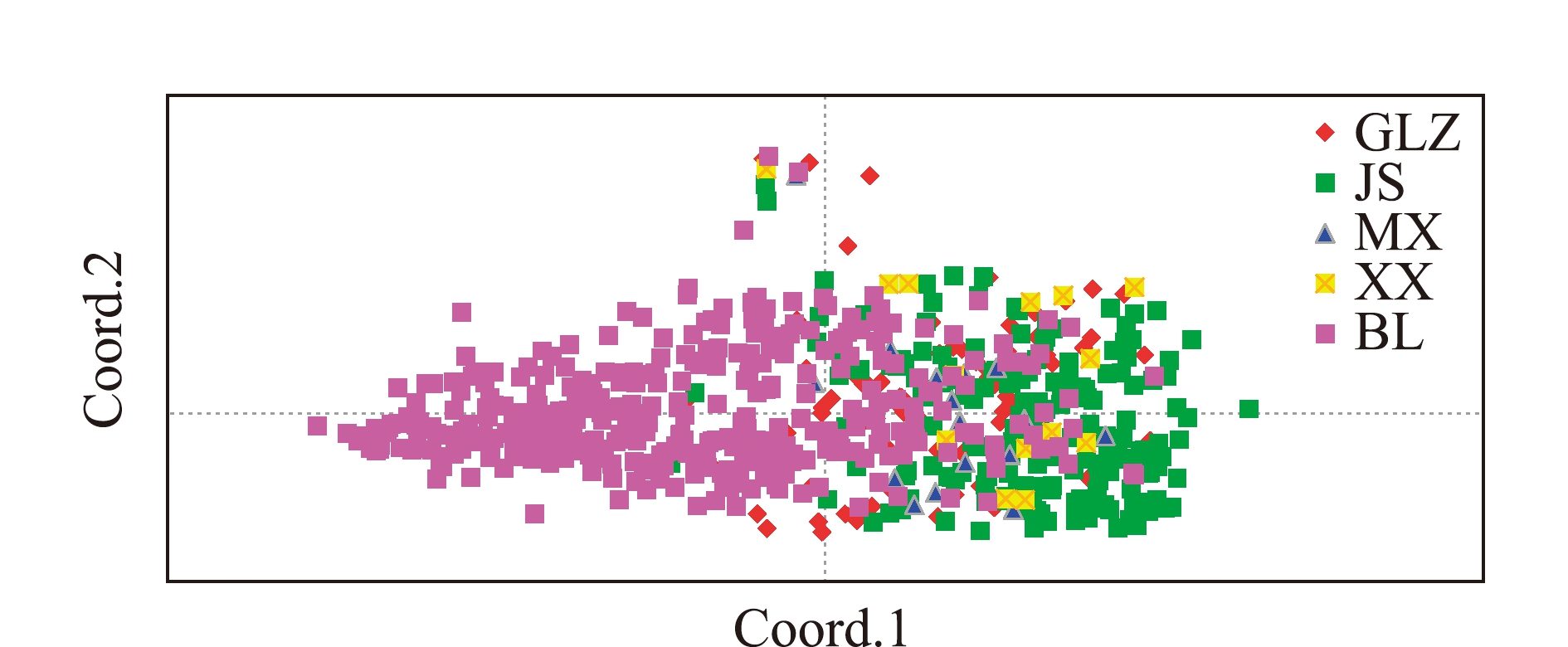
图2 三角鲂5个群体基于Nei’s遗传距离的主坐标分析
Fig.2 Principal coordinates analysis results based on Nei’s genetic distance among five populations of black bream Megalobrama terminalis
2.3 三角鲂群体的遗传结构分析
应用Structure软件分析5个三角鲂群体的遗传结构,根据ΔK和不同假设K值的变化趋势发现,当K=2时,ΔK变化率最大并出现明显拐点(图3), 推断本研究中所有参试个体最佳分组为2个理论类群。取K=2,计算每个样本对应的Q值,根据Q值将所有参试个体分配到可能性最大的类群中,形成反映遗传变异来源的遗传结构图。图4为最佳假设K=2时5个三角鲂群体的遗传结构图,可以看出, 4个亲本群体和1个BL群体被划分成2个大的基因池,其中,绿色基因池代表的类群1(cluster 1)包含GLZ、JS、MX和XX 4个亲本群体的个体,红色基因池代表的类群2(cluster 2)包含绝大多数的BL群体个体和少量来自亲本群体的个体。这与图2显示的在主坐标1(Coord.1)上5个群体被大致分为两大类群的主坐标分析结果一致。
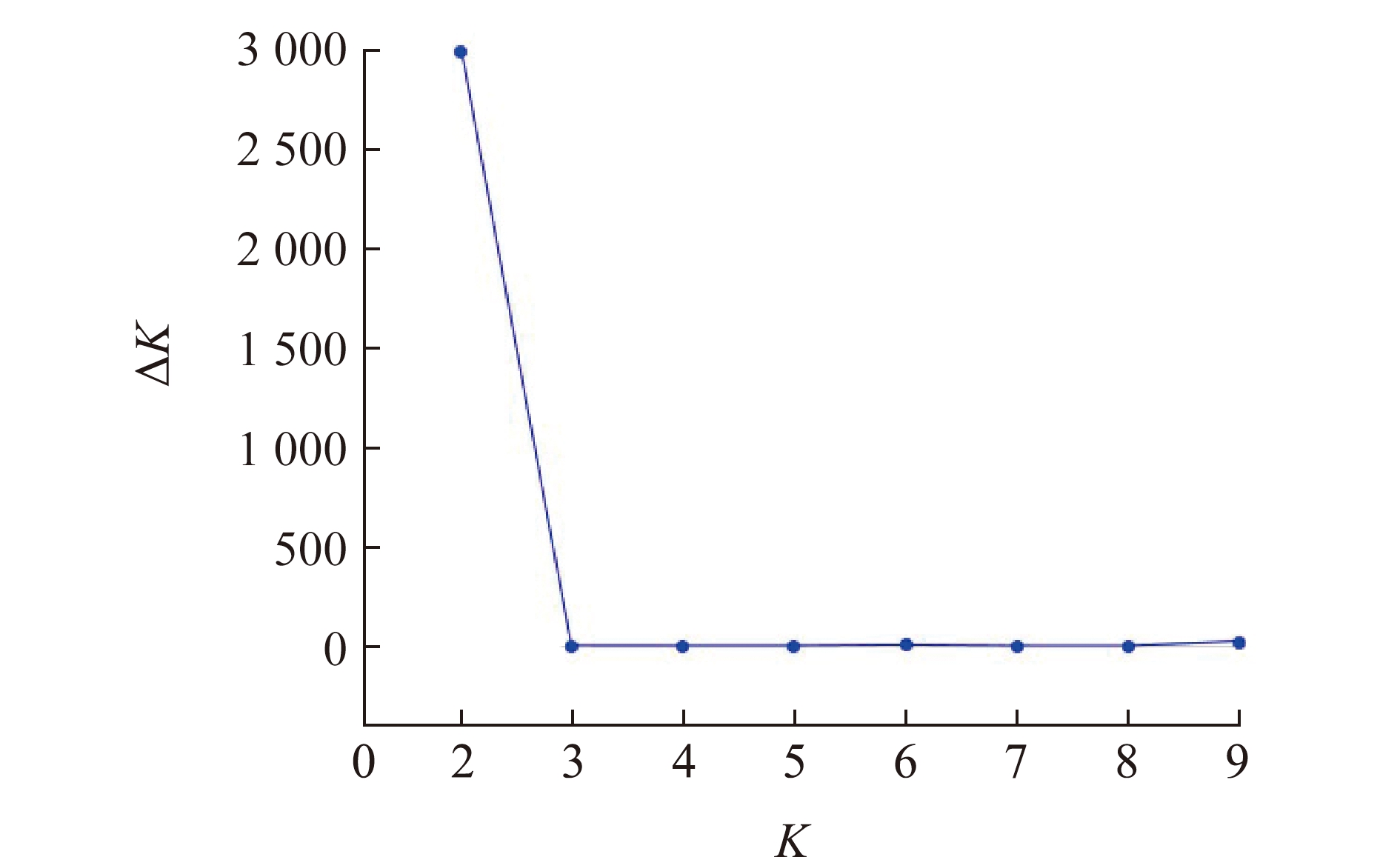
图3 三角鲂5个群体Δ K和不同假设类群数 K值的关系图
Fig.3 Relation between Δ K and different hypothetical cluster number K of five populations in black bream Megalobrama terminalis
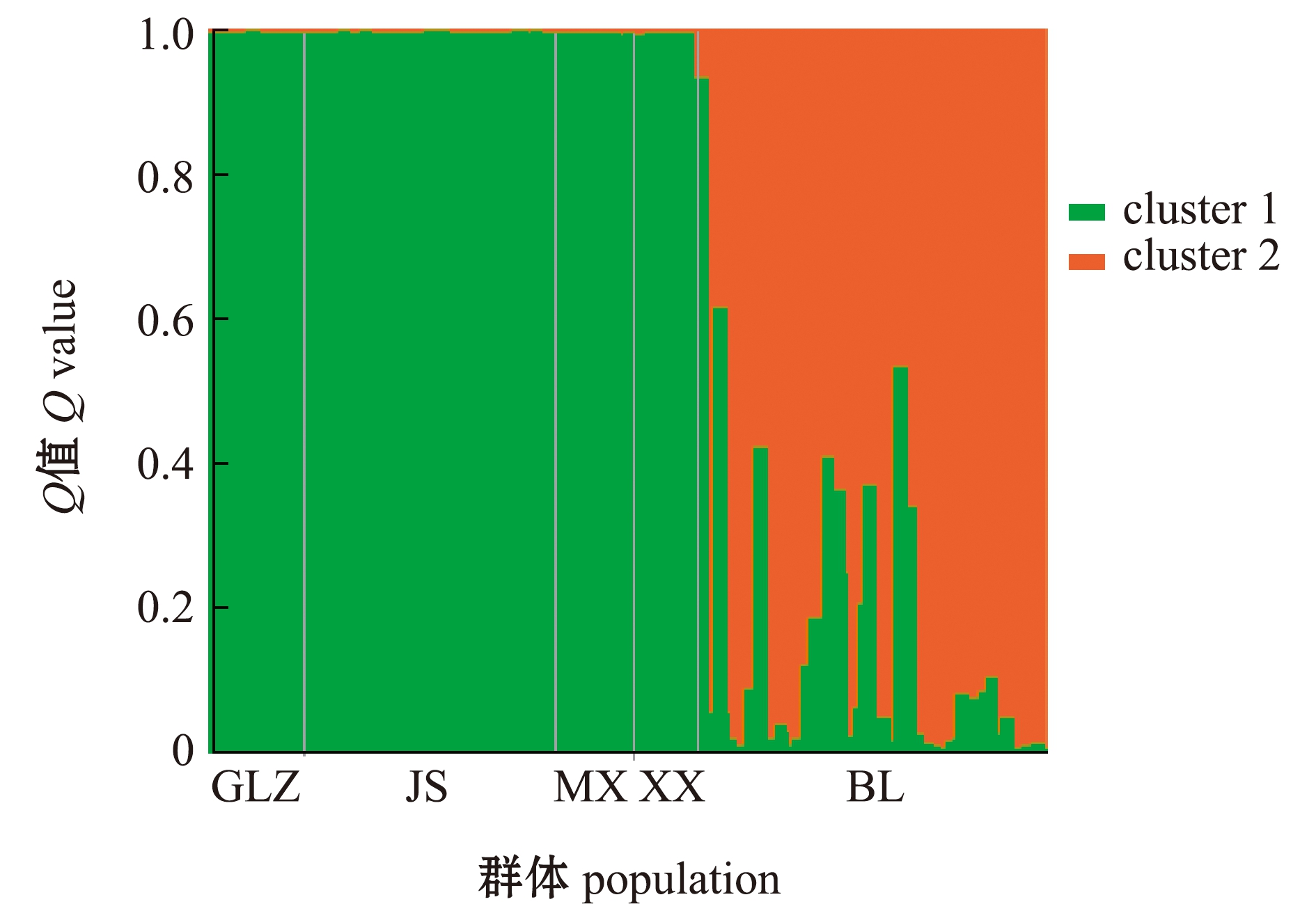
图4 三角鲂5个群体个体在 K=2时的遗传结构图
Fig.4 Genetic structure diagram in five populations of black bream Megalobrama terminalis ( K=2)
3 讨论
3.1 三角鲂群体的遗传多样性及杂合子过剩
观测杂合度是衡量种群遗传变异程度的重要参数,与群体遗传结构变异程度呈正相关,观测杂合度越高,种群的遗传多样性就越丰富[24]。Dewoody等[25]基于微卫星标记对13种淡水鱼类的遗传多样性进行了研究,结果显示,13种淡水鱼类的观测杂合度平均值为0.460 0,认为其遗传多样性较丰富。聂竹兰[14]用13个微卫星位点分析了黑龙江、钱塘江、北江及金沙河水库三角鲂群体的遗传结构及种群遗传多样性,结果表明,钱塘江群体的平均有效等位基因数为4.227 9,观测杂合度为0.556 7~0.931 0,平均Shannon’s指数为1.557 2。本研究中,钱塘江中下游5个三角鲂群体平均有效等位基因数为(6.60±0.98)~(14.22±3.08),观测杂合度平均值为(0.96±0.03)~(0.98±0.01),平均Shannon’s信息指数为(2.02±0.14)~(2.85±0.21)。由此可见,本研究中遗传多样性参数高于聂竹兰等[14]的研究结果,特别是观测杂合度平均值明显高于上述13种淡水鱼类的观测杂合度平均值,表明本研究中钱塘江中下游三角鲂5个群体的遗传多样性丰富,种质提纯及选育空间较大。
固定指数反映了群体内观测杂合度和期望杂合度间的平衡关系。当F<0时,说明杂合子过剩;当F>0时,说明杂合子缺失;当F越接近于零,则基因型的分布越接近平衡状态[26]。张守都等[27]对中国海湾扇贝Argopecten irradians混交家系研究表明,在优先受精及自然选择压力下,扇贝群体不断淘汰自交个体并保留杂交个体,群体表现出显著的杂合子过剩。这种杂合子过剩现象同样发生在两个吉富罗非Oreochromis niloticus群体[28]和中国明对虾Fenneropenaeus chinensis群体中[29]。与上述研究结果一致,本研究中,5个三角鲂群体绝大多数位点的F<0,特别是GLZ、MX和XX 3个亲本群体在6个位点上的观测杂合度为1,出现杂合子过剩现象明显。
Balloux[30]研究表明,杂合子过剩现象通常发生在参与繁殖个体数较少的小种群,而有足够繁殖个体的大种群不容易发生杂合子过剩;Cornuet等[31]研究表明,处于突变-漂移平衡的群体, 其基因位点出现杂合子过剩及不足的概率大致均等; Nei等[32]研究表明,一旦发生瓶颈效应, 就会造成杂合子过剩的现象。因此,杂合子过剩被用于衡量群体数量下降的瞬间效应。综合上述研究结果,推测本研究中钱塘江中下游三角鲂群体,特别是GLZ、MX和XX 3个亲本群体在养殖过程中,因参与繁殖群体个体较少或遭受了人工定向选择压力,发生了某种程度的遗传漂变和瓶颈效应。
李鸥等[33]对草鱼Ctenopharyngodon idella种群的SSR分析表明,等位基因数、各位点平均等位基因数与样本量大小呈显著正相关,绝大多数位点(占位点总数的86.7%)的有效等位基因数随样本容量的增大而增大,但增幅逐渐降低;在样本量达到40以上时,各位点平均等位基因数和平均有效等位基因数趋于稳定;期望杂合度与观测杂合度基本不受样本量大小的影响。闫路娜等[34]开展了微卫星DNA分析中样本量对遗传多样性度量指标影响的研究,结果表明,样本大小与所观测到的每个位点的等位基因数、平均等位基因数均呈显著正相关;对同一种群来说, 当样本量变动时, 尽管直接影响到稀有基因的检出, 但对期望杂合度几乎无影响;30~50个个体是微卫星DNA 分析所需要的最小样本量。参考上述研究结果,本研究中有2个亲本群体(XX群体和MX群体)采样数量偏少(不足30尾),可能导致本研究中得出的这2个群体的部分遗传多样性参数存在偏差,这2个群体的部分遗传多样性参数值(如等位基因数、平均等位基因数)可能低于实际水平。
3.2 三角鲂群体遗传分化及遗传结构
本研究中,从分子方差分析及群体间的遗传分化指数来看,5个三角鲂群体的遗传变异均主要来自群体内的个体间(占94.47%),群体间的遗传变异占5.53%,这与聂竹兰[14]得出的96.4%的遗传变异源于个体间,仅3.6%的变异来自群体间的研究结果一致。三角鲂群体间的遗传分化极显著(P<0.01),三角鲂4个亲本群体间遗传分化程度较小(0<FST<0.05),BL群体与JS和XX两个亲本群体间存在中等遗传分化(0.05<FST<0.15)。不少学者对野生群体和人工养殖群体的遗传分化进行了比较研究。Li等[35]对鲍Haliotis discus 3个养殖群体和2个野生群体进行了微卫星位点分析,结果发现,3个养殖群体等位基因数减少了76%,推测原因,可能是繁殖亲本量太少导致的遗传漂变。张志伟等[36]对3个草鱼群体(1个野生群体和 2个养殖群体)基于微卫星标记的研究结果显示, 2个养殖群体的等位基因数显著低于野生群体,并认为建群者效应是引起养殖群体等位基因丢失的主要原因。唐首杰等[37〗对尼罗罗非鱼群体的研究显示,5个驯养群体在1 个微卫星位点受到了显著的正向选择压力,说明种群(群体) 在选择压力下可能经历更快的遗传进化;与自然选择过程相比,推测人工干预程度和人工选择过程对位点等位基因频率改变存在重要的影响。本研究中,各群体的等位基因数和特有等位基因数也支持上述结论:同一个位点上BL群体检测出的等位基因数或有效等位基因数明显高于4个亲本群体;同时,XX亲本群体未检测到特有等位基因,GLZ、JS、MX 3个亲本群体的特有等位基因数分别为5、21和2个,BL群体检测到的特有等位基因数最多(122个)。推测本研究中,在每年的淘汰、更新留种过程中,三角鲂亲本群体遭受了某种程度的人工定向选择压力,部分个体所携带的遗传信息被淘汰,导致某些亲本群体原有的部分等位基因丢失。
结合5个群体的主坐标散点聚类及遗传结构分析不难看出,BL群体的遗传结构相对独立,并与部分亲本群体呈现出明显的差异。这与分子方差分析得到的群体间遗传分化极显著的结果一致。
3.3 三角鲂种质资源的保护与利用建议
为保护和恢复三角鲂种质资源,在保护其产卵场和改善水域环境条件的同时,应严格控制其野生亲本/后备亲本的捕捞,以维持天然水体三角鲂资源的自我修复潜力。在钱塘江水系,虽然有关部门早在2002年就建立了三角鲂国家级原种场[5],但是由于近年来钱塘江野生亲本数量急剧减少,加之野生亲本在捕捞、运输途中的死亡,必然导致原种亲本数量不足;原种场一般通过繁殖F1代留作后备亲本。由于原种场F1代后备亲本强化培育成本高、周期长(2~3年)及苗种培育规模有限等原因,导致原种场后备亲本供应数量有限、价格偏高。一些养殖场/良种场为了节约成本,往往引进少量原种场后备亲本或苗种后自繁子代留作亲本,加上养殖规模较小,群体数量不够大,这样很容易发生瓶颈效应和近交衰退现象,这可能也是造成了养殖场/良种场亲本群体等位基因缺失的重要原因之一。目前,增殖放流苗种的采购一般采取政府招标的形式。因此,为了更好地保护和利用三角鲂种质资源,提出建议如下:
1)建议相关职能部门在放流前必须对投标苗种场的资质、养殖规模及苗种亲本来源进行严格审查,力争做到苗种来源可追溯,从源头上控制苗种质量。
2)在放流前重点对放流群体遗传背景进行评估,确保天然种群遗传结构的稳定。
3)在放流现场要加强对放流苗种的规格、健康状况及种质状况进行抽样调查和监督。
4)放流后要定期对天然水体自然群体的生长特点、生物学特性及遗传多样性和遗传结构进行跟踪监测,根据监测结果调整放流计划。
4 结论
1)三角鲂5个群体观测杂合度的平均值为(0.96±0.03)~(0.98±0.01),表明钱塘江中下游三角鲂群体遗传多样性处于较高水平。
2)自然捕捞群体与景山、萧乡群体的遗传分化指数为0.05<FST<0.15,表明自然捕捞群体和部分亲本群体间出现了中等遗传分化。
3)大部分位点的观测杂合度大于期望杂合度,说明亲本群体普遍出现了杂合子过剩和部分等位基因丢失现象。
[1] 陈宜瑜.中国动物志:硬骨鱼纲鲤形目(中卷)[M].北京:科学出版社,1998:200-208.
CHEN Y Y.Fauna sinica:Osteichthyes Cypriniformes(middle volume) [M].Beijing:Science Press,1998:200-208.(in Chinese)
[2] 陈马康,童合一,俞泰济,等.钱塘江鱼类资源[M].上海:上海科学技术文献出版社,1990:108-112.
CHEN M K,TONG H Y,YU T J,et,al.Fishery resources of Qiantang River[M].Shanghai:Shanghai Scientific and Technological Literature Publishing House,1990:108-112.(in Chinese)
[3] 童勇义.钱塘江下游渔业资源开发利用现状及制约因素浅析[J].中国水产,2001(4):18-19.
TONG Y Y.Status analysis and restricting factors of exploitation and utilization of fishery resources in the lower reaches of the Qiantang River[J].China Fisheries,2001(4):18-19.(in Chinese)
[4] 郝雅宾,刘金殿,张爱菊,等.钱塘江下游江段鱼类资源现状[J].浙江农业学报,2017,29(10):1620-1629.
HAO Y B,LIU J D,ZHANG A J,et al.Current status of fishery resources in downstream section of Qiantang River[J].Acta Agriculturae Zhejiangensis,2017,29(10):1620-1629.(in Chinese)
[5] 冯晓宇.浙江省三角鲂研究进展及示范推广情况[J].科学养鱼,2009,25 (10):40-41.
FENG X Y.Research progress and demonstration of Megalobrama terminalis in Zhejiang Province[J].Scientific Fish Farming,2009,25 (10):40-41.(in Chinese)
[6] 戴杨鑫,冯晓宇,储忝江,等.铜山水库人工繁殖三角鲂种群的生长情况[J].浙江农业科学,2020,61(2):365-367.
DAI Y X,FENG X Y,CHU T J,et al.Growth of artificial breeding Megalobrama amblycephala in Tongshan Reservoir[J].Journal of Zhejiang Agricultural Sciences,2020,61(2):365-367.(in Chinese)
[7] 宋文,王艺舟,祝东梅,等.三个地理种群鲂的形态差异与判别分析[J].水产学杂志,2013,26(4):1-7.
SONG W,WANG Y Z,ZHU D M,et al.Morphological variation and discriminant analysis of three geographical populations of freshwater bream Megalobrama skolkovii[J].Chinese Journal of Fisheries,2013,26(4):1-7.(in Chinese)
[8] 陈杰,李福贵,黄创新,等.不同鳊鲂属鱼类群体的形态差异分析[J].上海海洋大学学报,2014,23(3):388-394.
CHEN J,LI F G,HUANG C X,et al.Morphological variations of genera Parabramis and Megalobrama teleost populations[J].Journal of Shanghai Ocean University,2014,23(3):388-394.(in Chinese)
[9] 吴成宾,李福贵,陈杰,等.鳊鲂鱼类的染色体核型及DNA含量分析[J].上海海洋大学学报,2015,24(6):801-809.
WU C B,LI F G,CHEN J,et al.Karyotype and DNA contents analysis of Parabramis and Megalobrama[J].Journal of Shanghai Ocean University,2015,24(6):801-809.(in Chinese)
[10] 张倩倩,陈杰,蒋霞云,等.不同鳊鲂鱼类群体微卫星DNA指纹图谱的构建和遗传结构分析[J].水产学报,2014,38(1):15-22.
ZHANG Q Q,CHEN J,JIANG X Y,et al.Establishment of DNA fingerprinting and analysis on genetic structure of different Parabramis and Megalobrama populations with microsatellite[J].Journal of Fisheries of China,2014,38(1):15-22.(in Chinese)
[11] 李思发,朱泽闻,邹曙明,等.鲂属团头鲂、三角鲂及广东鲂种间遗传关系及种内遗传差异[J].动物学报,2002,48(3):339-345.
LI S F,ZHU Z W,ZOU S M,et al.Interspecific phylogenesis and intraspecific genetic differences of genus megalobrama:bluntnose black bream (M.amblycephala),Guangdong black bream (M.hoffmanni) and black bream (M.terminalis)[J].Acta Zoologica Sinica,2002,48(3):339-345.(in Chinese)
[12] 谢楠,刘新轶,冯晓宇,等.鲂属鱼类细胞色素b片段序列分析[J].现代农业科技,2012(1):290-292.
XIE N,LIU X Y,FENG X Y,et al.Sequences analysis on mitochondrial cytochrome b gene fragment of Megalobrama spp.[J].Modern Agricultural Science and Technology,2012(1):290-292.(in Chinese)
[13] HU X S,LUAN P X,CAO C H,et al.Characterization of the mitochondrial genome of Megalobrama terminalis in the Heilong River and a clearer phylogeny of the genus Megalobrama[J].Scientific Reports,2019,9(1):8509.
[14] 聂竹兰.三角鲂转录组分析与不同地理种群遗传多样性研究[D].武汉:华中农业大学,2014.
NIE Z L.Transcriptome analysis and study on genetic diversity of different geographic populations in Megalobrama terminalis[D].Wuhan:Huazhong Agricultural University,2014.(in Chinese)
[15] 张敏莹,王邢艳,张婉平,等.基于微卫星亲权鉴定对钱塘江中下游三角鲂增殖放流效果的研究[J].农业生物技术学报,2020,28(7):1260-1268.
ZHANG M Y,WANG X Y,ZHANG W P,et al.Study on enhancement and releasing effect of triangular bream(Megalobrama terminalis) in the middle and lower reaches of the Qiantang River by microsatellite paternity testing[J].Journal of Agricultural Biotechnology,2020,28(7):1260-1268.(in Chinese)
[16] 王邢艳,徐东坡,张婉平,等.基于COⅠ基因研究钱塘江三角鲂亲本、放流和自然捕捞群体的遗传多样性与遗传分化[J].大连海洋大学学报,2021,36(4):603-611.
WANG X Y,XU D P,ZHANG W P,et al.Genetic diversity and genetic differentiation in broodstock,released populations and natural fishing population of black bream Megalobrama terminalis in the Qiantang River based on COⅠ gene[J].Journal of Dalian Ocean University,2021,36(4):603-611.(in Chinese)
[17] 孙效文,张晓锋,赵莹莹,等.水产生物微卫星标记技术研究进展及其应用[J].中国水产科学,2008,15(4):689-703.
SUN X W,ZHANG X F,ZHAO Y Y,et al.Development and application of microsatellite markers in aquatic species[J].Journal of Fishery Sciences of China,2008,15(4):689-703.(in Chinese)
[18] PEAKALL R,SMOUSE P E.Genalex 6:genetic analysis in excel population genetic software for teaching and research[J].Molecular Ecology Notes,2006,6(1):288-295.
[19] EXCOFFIER L,LISCHER H E L.Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J].Molecular Ecology Resources,2010,10(3):564-567.
[20] PRITCHARD J K,STEPHENS M,DONNELLY P.Inference of population structure using multilocus genotype data[J].Genetics,2000,155(2):945-959.
[21] EARL D A,VONHOLDT B M.Structure Hharvester:a website and program for visualizing structure output and implementing the Evanno method[J].Conservation Genetics Resources,2012,4(2):359-361.
[22] EVANNO G,REGNAUT S,GOUDET J.Detecting the number of clusters of individuals using the software structure:a simulation study[J].Molecular Ecology,2005,14(8):2611-2620.
[23] BALLOUX F,LUGON-MOULIN N.The estimation of population differentiation with microsatellite markers[J].Molecular Ecology,2002,11(2):155-165.
[24] NEI M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics,1978,89(3):583-590.
[25] DEWOODY J A,AVISE J C.Microsatellite variation in marine,freshwater and anadromous fishes compared with other animals[J].Journal of Fish Biology,2000,56(3):461-473.
[26] WEIR B S,COCKERHAM C C.Estimation F-statistics for the analysis of population structure[J].Evolution,1984,38:1358-1370.
[27] 张守都,许飞,李莉,等.海湾扇贝混交家系中优先受精以及自然选择压力下杂合子过剩的研究[J].海洋科学,2013,37(3):1-5.
ZHANG S D,XU F,LI L,et al.Investigation of preferential fertilization and heterozygote excess in mixed family of bay scallop (Argopecten irradians)[J].Marine Sciences,2013,37(3):1-5.(in Chinese)
[28] 李建林,李红霞,唐永凯,等.利用微卫星标记分析两个吉富罗非鱼群体的遗传差异[J].南方农业学报,2015,46(1):138-143.
LI J L,LI H X,TANG Y K,et al.Genetic difference analysis of two genetically improved farmed tilapia populations by using microsatellite marker[J].Journal of Southern Agriculture,2015,46(1):138-143.(in Chinese)
[29] 王军,王清印,孔杰,等.中国明对虾人工选育群体与野生群体遗传多样性的SSR分析[J].渔业科学进展,2018,39(2):104-111.
WANG J,WANG Q Y,KONG J,et al.SSR analysis on genetic diversity in breeding and wild populations of Fenneropenaeus chinensis[J].Progress in Fishery Sciences,2018,39(2):104-111.(in Chinese)
[30] BALLOUX F.Heterozygote excess in small populations and the heterozygote-excess effective population size[J].Evolution,2004,58(9):1891-1900.
[31] CORNUET J M,LUIKART G.Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data[J].Genetics,1996,144(4):2001-2014.
[32] NEI M,MARUYAMA T,CHAKRABORTY R.The bottleneck effect and genetic variability in populations[J].Evolution,1975,29(1):1.
[33] 李鸥,赵莹莹,郭娜,等.草鱼种群SSR分析中样本量及标记数量对遗传多度的影响[J].动物学研究,2009,30(2):121-130.
LI O,ZHAO Y Y,GUO N,et al.Effects of sample size and loci number on genetic diversity in wild population of grass carp revealed by SSR[J].Zoological Research,2009,30(2):121-130.(in Chinese)
[34] 闫路娜,张德兴.种群微卫星DNA分析中样本量对各种遗传多样性度量指标的影响[J].动物学报,2004,50(2):279-290.
YAN L N,ZHANG D X.Effects of sample size on various genetic diversity measures in population genetic study with microsatellite DNA markers[J].Acta Zoologica Sinica,2004,50(2):279-290.(in Chinese)
[35] LI Q,PARK C,ENDO T,et al.Loss of genetic variation at microsatellite loci in hatchery strains of the Pacific abalone (Haliotis discus Hannai)[J].Aquaculture,2004,235(1/2/3/4):207-222.
[36] 张志伟,曹哲明,杨弘,等.草鱼野生和养殖群体间遗传变异的微卫星分析[J].动物学研究,2006,27(2):189-196.
ZHANG Z W,CAO Z M,YANG H,et al.Microsatellites analysis on genetic variation between wild and cultured populations of Ctenopharyngodon idella[J].Zoological Research,2006,27(2):189-196.(in Chinese)
[37] 唐首杰,杨洁,王成辉,等.驯养、选育条件下尼罗罗非鱼群体的选择压力分析[J].中国水产科学,2016,23(4):900-913.
TANG S J,YANG J,WANG C H,et al.Analysis of selective pressure on Nile tilapia(Oreochromis niloticus) populations during domestication/selective breeding[J].Journal of Fishery Sciences of China,2016,23(4):900-913.(in Chinese)