病害是影响水产养殖产业发展的主要原因之一[1-3],病害诊治是提升水产养殖质量的关键[4],有效的病害诊治需要充分掌握并利用水产医学知识,而水产医学知识又被保存于水产医学文本中,因此,为了提升病害诊治效率,促进水产养殖产业快速发展,需要对水产医学文本中的命名实体进行有效的识别。
传统医学命名实体识别方法在人类疾病诊治、农业等领域已经取得了较好的研究进展[5-8],由于领域内实体特征明显、结构简单,实体的识别准确率较高[9-10],而水产医学文本中的实体多以嵌套的形式存在,且实体的特征不明显,不能直接用传统医学命名实体识别方法对水产医学命名实体进行识别[11-12],为了提高水产医学命名实体识别准确率,需要研究一种针对水产医学嵌套实体的有效识别方法。
目前,针对嵌套实体识别方法的研究已经取得了一定进展[13-15]。Li等[16]提出用MGNER模型对嵌套实体进行识别,采用双向长短时记忆网络(bidirectional long short-term memory,BiLSTM)提取嵌套实体特征,并将预测实体标签序列枚举,以实现对嵌套实体的识别,该方法对结构简单的嵌套实体可取得较好的识别效果,但对于结构复杂的嵌套实体,由于不能枚举所有可能的标签序列,导致识别准确率较低;Tan等[17]通过预测实体间相关度的方法对嵌套实体进行识别,当相关度大于阈值时判别为嵌套实体,并进行分步识别,对复杂嵌套实体也能有效识别,但该方法未考虑嵌套实体间的特征交互,对于实体间相关度的预测会存在偏差,最终影响实体识别效果;Katiyar等[18]提出一种基于递归网络的嵌套命名实体识别方法,通过递归网络提取实体特征,构建嵌套实体的超图表示,并运用超图增加嵌套实体间的信息交互,以提升嵌套实体特征表示,实现对嵌套实体的有效识别,但针对长序列嵌套实体识别时,构建的超图结构复杂且优化困难,导致对长序列嵌套实体识别的准确率不高;Xia等[19]提出用Multi-Grained模型对嵌套实体进行识别,利用多粒度思想,对嵌套实体由内而外分不同粒度进行识别,同时,将不同粒度内嵌套实体特征进行传递,提升整段嵌套实体的特征表示,从而解决长序列嵌套实体识别准确率不高的问题。目前,有关水产医学命名实体识别的研究鲜见报道。本研究中,提出一种基于多核卷积的命名实体识别方法(BERT+Multi-CNN+CRF),针对水产医学命名实体识别过程中,因嵌套实体而导致的识别准确率不高的问题,采用BERT(bidirectional encoder representations from transformers)与Multi-CNN相结合的方法,提高嵌套实体的特征表示,以期实现对水产医学嵌套命名实体的有效识别。
1 基于多核卷积的实体识别模型构建
1.1 模型总体架构
针对水产医学命名实体识别任务中存在的嵌套实体识别准确率不高的问题,本研究中提出的BERT+Multi-CNN+CRF模型(图1),主要由BERT[20]、CNN[21]和判别层3部分组成。BERT层实现实体的词嵌入过程;CNN层提取实体特征,并根据获得的特征表示预测实体可能的标签序列;判别层收集预测实体标签序列,并对标签序列进行筛选,删除重叠的预测标签,同时给出当前实体序列所有可能的预测标签,从而实现对长序列嵌套实体的有效识别。
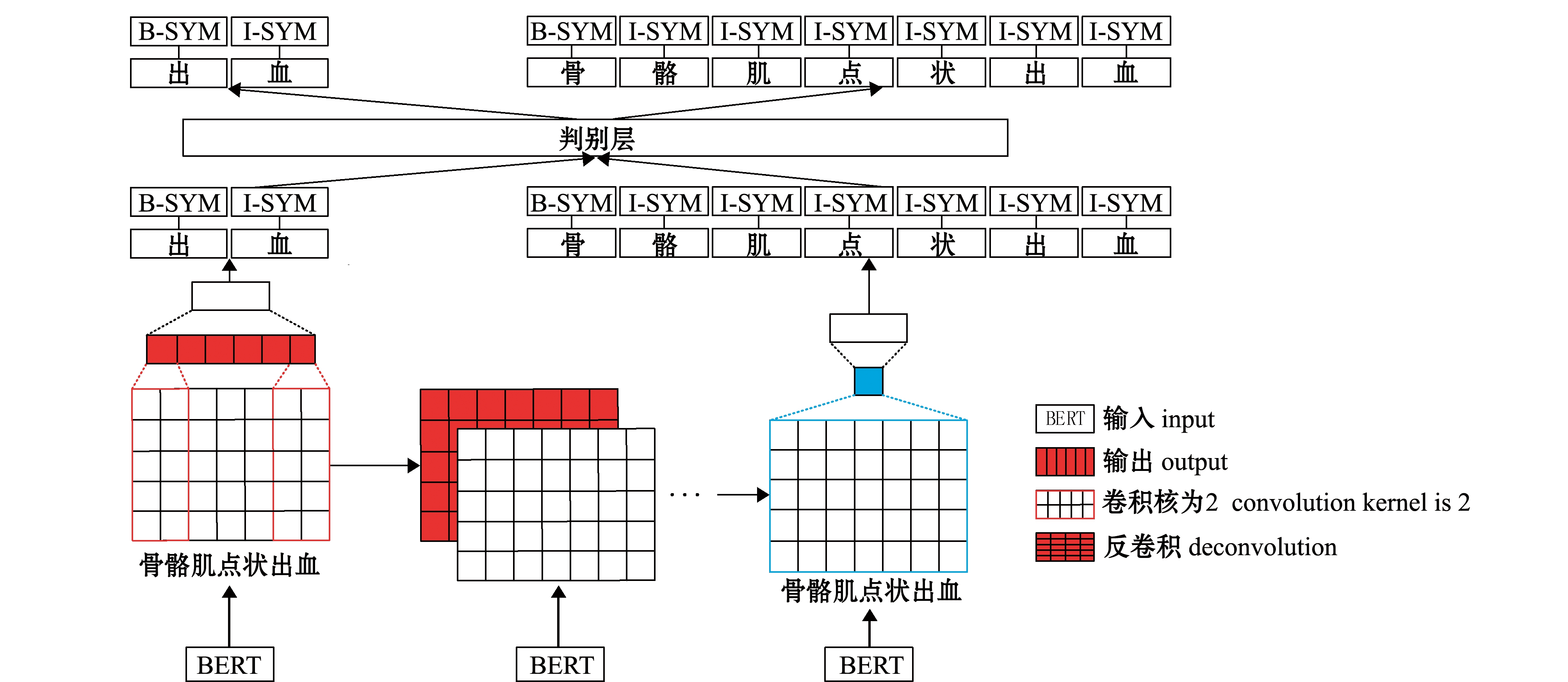
图1 基于多核卷积的实体识别模型结构图
Fig.1 Structure diagram of entity recognition model based on multi-kernel convolution
1.2 BERT层
传统词嵌入方法Word2Vec[22]未考虑词的位置信息,在词嵌入过程中,由于词在不同位置中表达的含义可能不同,易出现一词多义的问题,因此,不能忽略词的相对位置信息。本文中的模型选用BERT作为词嵌入过程,由于BERT中添加了对词位置信息的描述,能够有效解决因位置信息而导致的一词多义问题,从而提高实体的识别准确率。
1.3 CNN层
在特征提取层,本文中的模型在卷积神经网络(convolutional neural networks,CNN)中设置大小为2~15的多个卷积核,对长序列嵌套实体进行特征提取。首先,将BERT输入的向量矩阵进行卷积以提取特征,并预测输出标签序列;然后,将获得的特征矩阵反卷积,得到输入同维矩阵,再将反卷积后特征矩阵与输入特征向量融合,作为下一个卷积核的输入,使下一个卷积过程能够充分利用上一个卷积提取的有效特征,以增强嵌套实体的特征表示,提升嵌套实体的识别效果(图2);最后,为保留实体位置信息,设计时删除了CNN中的池化过程。
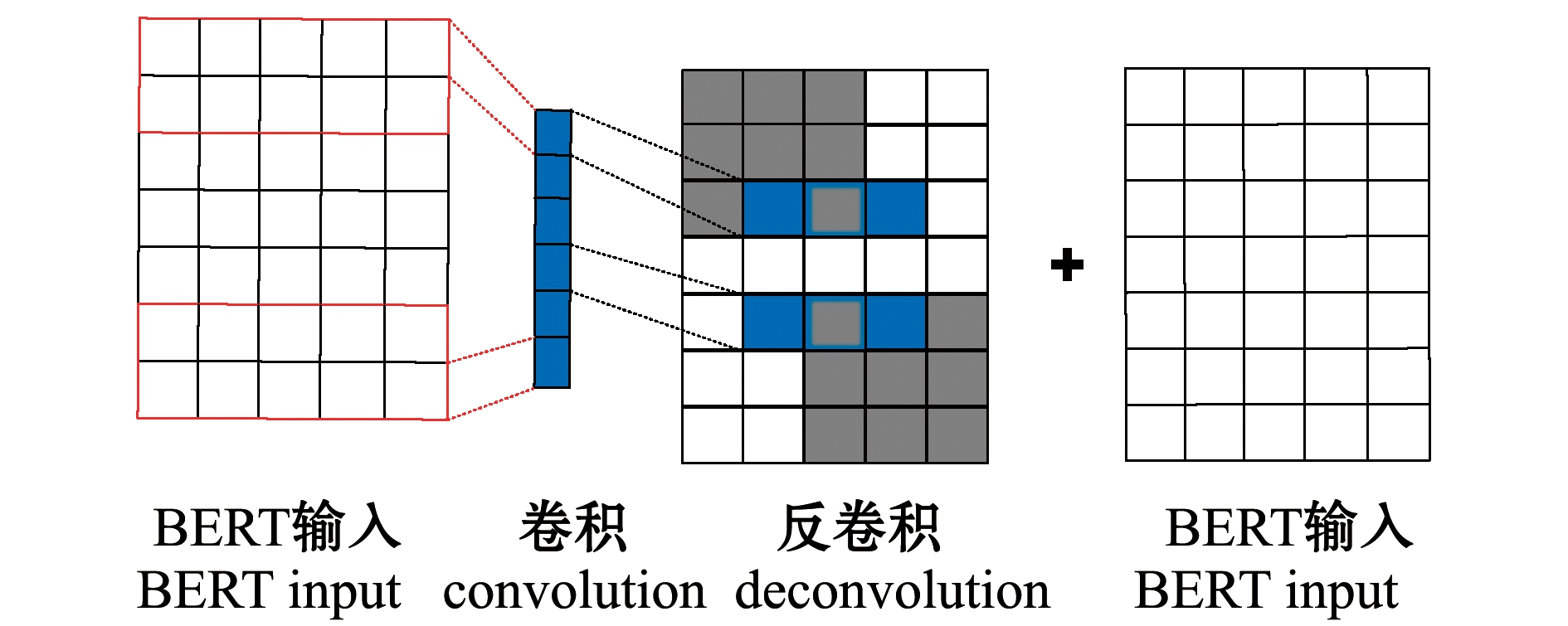
图2 反卷积过程图
Fig.2 Deconvolution process diagram
1.4 判别层
判别层收集了多个预测的标签序列,需要将获得的不同预测标签进行处理,以找到预期的标签序列。从图3可见,当存在两个一致的预测标签序列时,删除卷积核较小的预测标签序列,并将剩余预测标签序列预存;当存在两个不一致的预测标签序列时,同时预存两个预测标签序列,并将所有预存的预测标签输出,得到长序列嵌套实体所有可能预测标签。通过判别层能有效解决长序列嵌套实体识别过程中存在的识别混淆问题,保证长序列嵌套实体中所有嵌套组合实体均被准确识别,从而实现对长序列嵌套实体的识别。
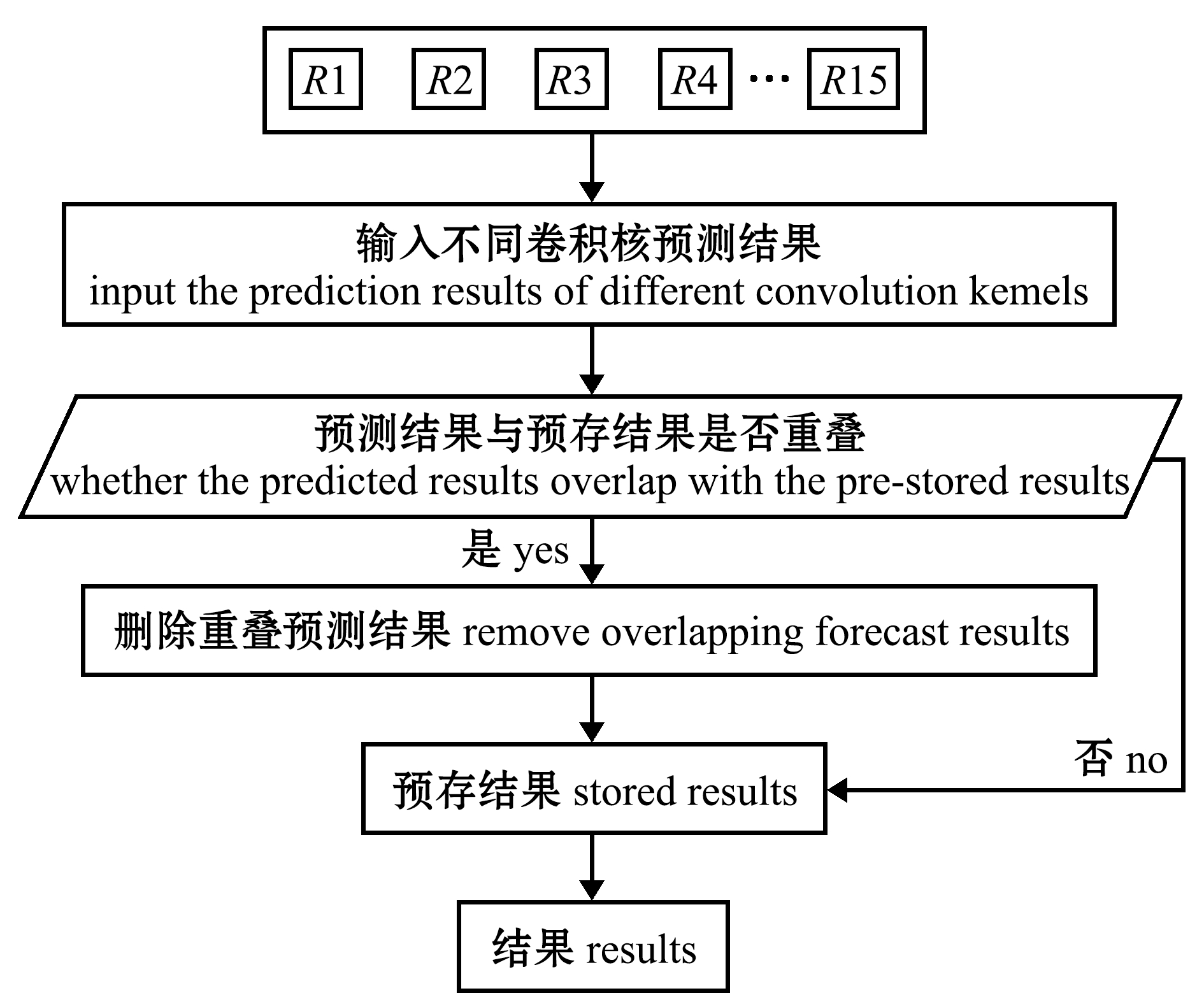
图3 判别流程图
Fig.3 Discrimination flow chart
2 试验
2.1 数据采集和预处理
中国水产养殖方式主要以小型企业为主,未形成大型规模化养殖,也没有形成有效的水产医学相关数据,因此,为保证数据的全面性和多样性,从水产动物病害相关书籍(《鱼病快速防治》《鱼病防治1000问》等)中采集水产动物病害数据,并收集水产养殖网(http://www.shuichan.cc/)2020—2021年里发表的水产动物疾病相关文章200篇,共计50万余字,组成水产医学语料库。采集的数据内容繁杂不能用于模型训练,需要将数据进行预处理,去掉其中包括作者署名、出版地、出版日期、空格、停用词等与水产医学无关的文本,并将语料库送入到Ul+raEdit文本编辑器,用Ul+raEdit正则表达式获取规范的语料库文本。语料库中主要包含水产动物病害检测方法、水产动物疾病治疗方法、水产动物发病时期,以及发病的主要表现、引起水产动物发病的病原体介绍等内容。
2.2 数据库实体类别
构建的水产医学语料库中包含了几十类水产医学实体,由于只针对水产动物病害相关实体进行识别,故需要将语料库的数据进行分类,通过参考医学语料库划分标准及咨询水产领域专家发现,水产动物病害相关实体主要分为6类,分别是水产动物名称、引起疾病的病原体名称、治疗的药品名称、水产动物疾病名称、水产动物患病的身体部位和水产动物患病的临床症状。这6类实体几乎涵盖了从水产动物患病到治疗的全过程,但分析这6类实体发现,实体间联系紧密,不易于区分,为了对实体进行严格区分,还需制定相关的实体标注规则。
2.3 实体标注规则
为了准确区分不同实体,保证同一实体在不同结构下标注方式不同,提高人工标注质量,该语料库制定了水产动物病害实体标注规则,其中6个典型的标注规则如表1所示,具体的标注细节如下:
表1 实体结构特征
Tab.1 Entity structure feature

结构特征structurefeature实例instance标注方法labelingmethod水产动物名称+疾病名称aquaticanimalname+diseasename草鱼出血病单独标注发病部位+临床症状lesionlocation+clinicalsymptom体表发黑,眼突出分两次标注病原体+病原体身体部位pathogen+pathogenbodypart成螨体黑色,背部有花纹病原体的身体部位不标注病原体+发病部位pathogen+lesionlocation柱状杆菌寄生鳃部单独标注水产动物名称+生命体征aquaticanimalname+vitalsign鲔大面积死亡死亡不进行标注形容词+临床症状adjective+clinicalsymptom垂死前表现呆滞分两次标注
1)水产动物名称+疾病名称结构类型,单独标注,如“草鱼出血病”,将“草鱼”标为水产动物名称,“出血病”标为疾病名称。
2)发病部位+临床症状结构类型,分两次标注,如“体表发黑,眼突出”,先将“体表,眼”标为发病部位,再将“体表发黑”“眼发黑”标为临床症状。
3)病原体+病原体身体部位结构类型,病原体的身体部位不进行标注,避免与发病部位重合,如“成螨体黑色,背部有花纹”,只将“成螨”标为病原体。
4)病原体+发病部位结构类型,单独标注,如“柱状杆菌寄生鳃部”,将“柱状杆菌”标为病原体,“鳃部”标为发病部位。
5)水产动物名称+生命体征结构类型,当出现生命体征为水产动物生命终止时,如“死亡”不视为临床部症状实体,不进行标注。
6)形容词+临床症状结构类型为嵌套结构,分两次标注,如“垂死前表现呆滞”,先将“表现呆滞”标注为临床症状,再将“垂死前表现呆滞”整体标注为临床症状。
2.4 标注方法
在命名实体识别任务中,BIO是主流标注方法,对实体识别和关系抽取任务中的实体和关系具有较好的区分效果。采用实体对应的英文缩写代表实体标签名称,与BIO标注方法相结合作为实体的标注方法。具体标签定义及各类实体在语料库中的分布情况如表2所示,其中,水产动物名称类实体数量较多,占实体总数的32.5%,疾病名称类实体相对较少,占实体总数的7.6%。
表2 标签的类别定义
Tab.2 Label category definition
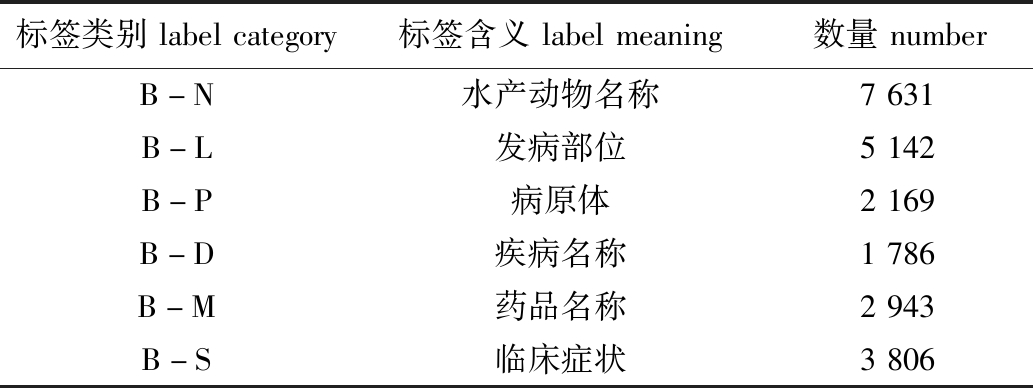
标签类别labelcategory标签含义labelmeaning数量numberB-N水产动物名称7631B-L发病部位5142B-P病原体2169B-D疾病名称1786B-M药品名称2943B-S临床症状3806
2.5 试验环境及评价指标
试验过程将数据按7∶3比例划分为训练集和测试集,训练集35万数据,测试集15万数据,两数据集间无重叠,且测试集的特征分布及类别比例与训练集相近,因此,可以作为实体识别的数据分布。试验运行环境:64 GB 内存,Intel Core i5-4200U CPU@2.30 GHz,GPU GEFORCE GTX 2080ti,1 TB 硬盘;Tensorflow 1.12框架加Python 3.6,Ubuntu 18.04系统。
试验结果采用准确率(precision,P)、召回率(recall,R)和二者的调和平均值(F1)值作为评价指标,当且仅当实体类别和实体边界均正确时才认定为识别正确。准确率、召回率和F1值的计算公式分别为
(1)
(2)
(3)
其中:TP为预测的实体是正确实体的个数;FP为预测的实体是非正确实体的个数;FN为未预测出来的实际实体个数。
2.6 试验参数
模型的参数设置如表3所示,其中,参数通过反复试验确定。对模型效果影响较大的参数,如模型迭代次数,模型效果会随着迭代次数的增加而提升,但训练次数过多会浪费训练时间,同时模型会趋于拟合,导致模型效果下降。在一定范围内数据量越大,数据多样性越丰富,模型识别效果就越好;批处理尺寸较大,容易使模型收敛在局部最优点,但批处理尺寸较小容易使模型跳出鞍点;学习率较高,可能导致Loss值无法收敛,但学习率较低可能导致收敛较慢或梯度损失;不同卷积核的大小影响模型的识别效果,卷积核太小不能完全包含实体的范围,不能准确获取完整的实体特征,但卷积核太大则会增加卷积核的个数,从而影响模型的训练时间[23]。通过控制变量法找到这5种参数的模型最适值。
表3 模型参数设置
Tab.3 Parameter settings

参数parameter数值value学习率learningrate0.0001批处理batchsize32迭代次数epoch200梯度裁剪clip5衰减度dropout0.5词嵌入维度dimension300卷积核kernel15
对各参数进行预试验,结果表明:迭代次数在200次时模型F1值得分最高;测试数据达到15万字符时F1值提升相对较少,这说明此时数据量增加对模型的影响相对较小,能够满足模型学习的基本要求;批处理大小取32时F1得分最高,此时批处理大小符合文本序列尺寸;学习率在10-4时模型的F1值仍有提升,但学习率过小会导致训练时间过长且容易损失梯度,故选取10-4作为模型的学习率;当卷积核大小为15时,能够包含90%以上的实体,且训练速度较快,故将最大卷积核设为15。
2.7 试验设计与结果分析
为验证BERT+Multi-CNN+CRF模型对水产医学命名实体识别的有效性,设计了2组对比试验,与现有识别准确率较高的BERT+BiLSTM+ATT+CRF[15]模型相比,以验证本文中模型能更准确有效识别水产医学文本中的命名实体;与同样采用分步思想识别嵌套实体的Multi-Grained模型[19]相比,以验证本文中模型能够更好地解决水产医学嵌套实体识别时出现的嵌套实体识别准确率不高的问题。
试验1:与目前常用的命名实体识别模型相比,本文中提出的BERT+Multi-CNN+CRF模型的准确率、召回率、F1值均有明显提升,与识别准确率较高的BERT+BiLSTM+ATT+CRF模型相比,BERT+Multi-CNN+CRF模型的准确率、召回率、F1值分别提高了2.25%、3.23%和2.74%(表4)。这表明,本文中提出的BERT+Multi-CNN+CRF模型对水产医学命名实体具有较好的识别效果,是一种有效的水产医学命名实体识别方法。
表4 不同模型的试验结果对比
Tab.4 Comparison of experimental results of different models
%
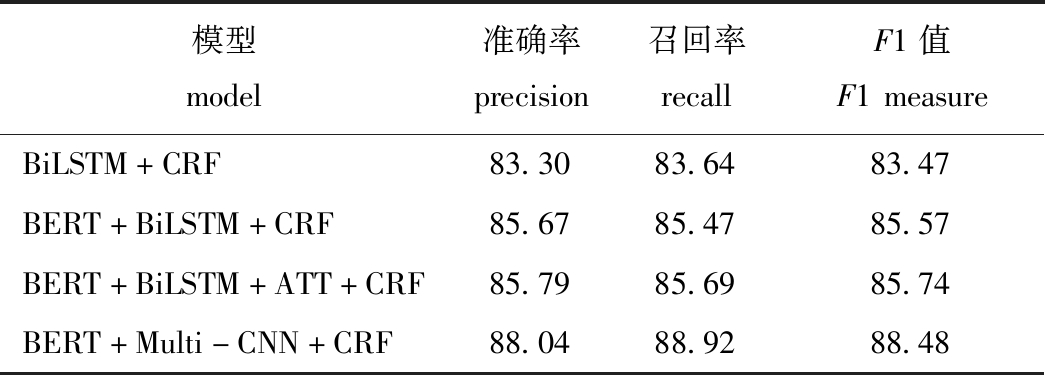
模型model准确率precision召回率recallF1值F1measureBiLSTM+CRF83.3083.6483.47BERT+BiLSTM+CRF85.6785.4785.57BERT+BiLSTM+ATT+CRF85.7985.6985.74BERT+Multi-CNN+CRF88.0488.9288.48
试验2:与嵌套命名实体识别HIT模型[24]和Multi-Grained模型[19]进行对比试验,结果表明,本文中提出的BERT+Multi-CNN+CRF模型具有更好的识别效果,其中与识别准确率较高且同样使用分步识别的Multi-Grained模型相比,BERT+Multi-CNN+CRF模型的准确率、召回率、F1值分别提高了0.83%、1.99%和1.41%(表5)。这表明,这种分步识别方法能够有效处理嵌套命名实体,提高嵌套命名实体识别的准确率,本文中提出的BERT+Multi-CNN+CRF模型是一种针对长序列嵌套实体的有效识别方法。
表5 不同嵌套模型试验结果对比
Tab.5 Comparison of experimental results of different nested models
%
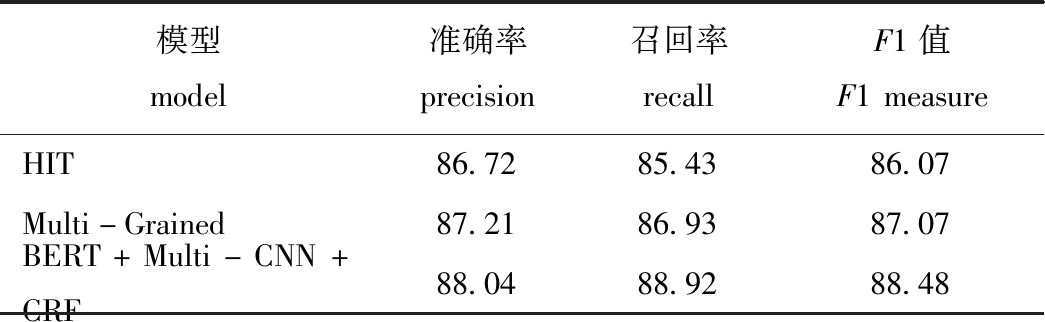
模型model准确率precision召回率recallF1值F1measureHIT86.7285.4386.07Multi-Grained87.2186.9387.07BERT+Multi-CNN+CRF88.0488.9288.48
3 讨论
3.1 模型选取
随着深度学习的快速发展,在命名实体识别任务中也得到了广泛应用。目前,实体识别效果较好的神经网络主要有CNN及LSTM,如赵丰等[25]用CNN对军事领域实体进行识别,取得较好的识别效果。羊艳玲等[26]提出用LSTM对中医药命名实体进行识别,取得了较高的准确率。LSTM与CNN相比,可设置记忆单元,能更好地处理长序列实体,但LSTM是顺序结构,对嵌套实体进行识别时易产生错误传播问题,而且LSTM的模型参数数量也远大于CNN,网络的效率相对较低,故本试验中提出使用CNN对水产医学嵌套实体进行识别。针对CNN不能处理长序列实体的问题,引入BERT模型,BERT模型中包含自注意力机制,增加了长序列实体的权重,能有效弥补CNN自身缺陷。此外,针对长序列水产医学嵌套实体识别准确率不高问题,本试验中设计使用多个卷积核分步提取嵌套实体特征,以提高嵌套实体识别准确率,最终提出了基于多核卷积神经网络的水产医学嵌套命名实体识别模型BERT+Multi-CNN+CRF。
3.2 模型效果分析
本研究中提出的BERT+Multi-CNN+CRF水产医学命名实体识别模型,与任媛等[15]提出的BERT+BiLSTM+ATT+CRF模型相比,本文中的模型更关注实体特征,通过分步卷积策略提升实体特征表示,模型的准确率、召回率、F1值分别达到了88.04%、88.92%和88.48%,实体识别效果有明显提升。与Xia等[19]提出的Multi-Grained模型相比,本文中模型的准确率、召回率、F1值分别提高了0.83%、1.99%和1.41%。本文中的模型通过设置判别层,实现预测结果的多个输出,从而可以更有效地识别水产医学命名实体中的嵌套实体,提升对嵌套实体的识别效果。
综上所述,BERT+Multi-CNN+CRF方法是一种有效的水产医学嵌套实体识别方法,为提高水产医学领域命名实体识别的准确率提供了一种新方法,也为其他领域的命名实体识别提供了一种新思路。
4 结论
1)针对水产医学实体特征不明显的问题,采用多卷积核分步卷积策略及引入BERT添加实体位置信息,以增强实体的特征表示,提高了实体识别的准确率,与传统的命名实体识别模型相比,本文提出的BERT+Multi-CNN+CRF命名实体识别模型的准确率、召回率、F1值分别提高了2.25%、3.23%和2.74%,表明本文中的方法可有效提升实体的特征表示。
2)针对水产医学命名实体识别时出现的长序列嵌套实体识别准确率不高的问题,通过设置判别层使内外层嵌套实体均能被有效识别,与传统的嵌套实体识别方法相比,本文提出的BERT+Multi-CNN+CRF命名实体识别模型的准确率、召回率、F1值分别提高了0.83%、1.99%和1.41%,表明本文中的方法能够提升嵌套实体识别的准确率,是一种有效的水产医学嵌套命名实体识别方法。
[1] 胡金有,王靖杰,张小栓,等.水产养殖信息化关键技术研究现状与趋势[J].农业机械学报,2015(7):251-263.
HU J Y,WANG J J,ZHANG X S,et al.Research status and development trends of information technologies in aquacultures[J].Transactions of the Chinese Society for Agricultural Machinery,2015(7):251-263.(in Chinese)
[2] AYALEW A,FUFA A.Maintenance of fish health in aquaculture:review of epidemiological approaches for prevention and control of infectious disease of fish[J].Veterinary Medicine International,2018,26(2):1-10.
[3] FROEHLICH H E,SAND J N,ESSINGTON T E,et al.Avoiding the ecological limits of forage fish for fed aquaculture[J].Nature Sustainability,2018,1(6):298-303.
[4] PATAI C,SAUL P,VANVIMON S.Applications of microalgal biotechnology for disease control in aquaculture[J].Biology,2018,7(2):24.
[5] 杨锦锋,于秋滨,关毅,等.电子病历命名实体识别和实体关系抽取研究综述[J].自动化学报,2014,40(8):1537-1562.
YANG J F,YU Q B,GUAN Y,et al.An overview of research on electronic medical record oriented named entity recognition and entity relation extraction[J].Acta Automatica Sinica,2014,40(8):1537-1562.(in Chinese)
[6] ZHANG L,ZHOU W D,JIAO L C.Wavelet support vector machine[J].IEEE Transactions on Systems,Man,and Cybernetics Part B:Cybernetics,2004,34(1):34-39.
[7] BENAJIBA Y,ROSSO P.Arabic named entity recognition using conditional random fields[C]//Proceedings of workshop on HL-T &NLP within the Arabic world.Arabic:Arabian Journal of Geosciences,2008:143-153.
[8] 曹依依,周应华,申发海,等.基于CNN-CRF的中文电子病历命名实体识别研究[J].重庆邮电大学学报(自然科学版),2019,31(6):869-875.
CAO Y Y,ZHOU Y H,SHEN F H,et al.Research on named entity recognition of Chinese electronic medical record based on CNN-CRF[J].Journal of Chongqing University of Posts and Telecommunications (Natural Science Edition),2019,31(6):869-875.(in Chinese)
[9] 张晗,郭渊博,李涛.结合GAN与BiLSTM-Attention-CRF的领域命名实体识别[J].计算机研究与发展,2019,56(9):1851-1858.
ZHANG H,GUO Y B,LI T.Domain named entity recognition combining GAN and BiLSTM-Attention-CRF[J].Journal of Computer Research and Development,2019,56(9):1851-1858.(in Chinese)
[10] MCCALLUM A,LI W.Early results for named entity recognition with conditional random fields,feature induction and web-enhanced lexicons[J].Association for Computational Linguistics,2003,4(9):188-191.
[11] 程名,于红,冯艳红,等.融合注意力机制和BiLSTM+CRF的渔业标准命名实体识别[J].大连海洋大学学报,2020,35(2):296-301.
CHENG M,YU H,FENG Y H,et al.Fishery standard named entity recognition with integrated attention mechanism and BiLSTM+CRF[J].Journal of Dalian Ocean University,2020,35(2):296-301.(in Chinese)
[12] 孙娟娟,于红,冯艳红,等.基于深度学习的渔业领域命名实体识别[J].大连海洋大学学报,2018,33(2):265-269.
SUN J J,YU H,FENG Y H,et al.Recognition of nominated fishery domain entity based on deep learning architectures[J].Journal of Dalian Ocean University,2020,33(2):265-269.(in Chinese)
[13] 余诗媛,郭淑明,黄瑞阳,等.嵌套命名实体识别研究进展[J].计算机科学,2021,48(sup 2):1-10,29.
YU S Y,GUO S M,HUANG R Y,et al.Overview of nested named entity recognition[J].Computer Science,2021,48(sup 2):1-10,29.(in Chinese)
[14] 杨鹤,于红,孙哲涛,等.基于双重注意力机制的渔业标准实体关系抽取[J].农业工程学报,2021,37(14):204-212.
YANG H,YU H,SUN Z T,et al.Fishery standard entity relation extraction using dual attention mechanism[J].Transactions of the Chinese Society of Agricultural Engineering,2021,37(14):204-212.(in Chinese)
[15] 任媛,于红,杨鹤,等.融合注意力机制与BERT+BiLSTM+CRF模型的渔业标准定量指标识别[J].农业工程学报,2021,37(10):135-141.
REN Y,YU H,YANG H,et al.Recognition of quantitative indicator of fishery standard using attention mechanism and the BERT+BiLSTM+CRF model[J].Transactions of the Chinese Society of Agricultural Engineering,2021,37(10):135-141.(in Chinese)
[16] LI P,YUAN Z,TU W,et al.Medical knowledge extraction and analysis from electronic medical records using deep learning[J].Chinese Medical Sciences Journal,2019,34(2):133-139.
[17] TAN C Q,QIU W,CHEN M S,et al.Boundary enhanced neural span classification for nested named entity recognition[J].Proceedings of the AAAI Conference on Artificial Intelligence,2020,34(5):9016-9023.
[18] KATIYAR A,CARDIE C.Nested named entity recognition revisited[C]//Proceedings of the 2018 conference of the North American Chapter of the Association for Computational Linguistics:human language technologies.Louisiana,Stroudsburg:Association for Computational Linguistics,2018:861-871.
[19] XIA C Y,ZHANG C W,YANG T,et al.Multi-Grained named entity recognition[EB/OL].2019.(2019-10-20)[2022-02-15].https://arxiv.org/abs/1906.08449.
[20] 杨鹤,于红,刘巨升,等.基于BERT+BiLSTM+CRF深度学习模型和多元组合数据增广的渔业标准命名实体识别[J].大连海洋大学学报,2021,36(4):661-669.
YANG H,YU H,LIU J S,et al.Fishery standard named entity recognition based on BERT+BiLSTM+CRF deep learning model and multivariate combination data augmentation[J].Journal of Dalian Ocean University,2021,36(4):661-669.(in Chinese)
[21] COLLOBERT R,WESTON J,BOTTOU L,et al.Natural language processing (almost) from scratch[J].Journal of Machine Learning Research,2011,12(1):2493-2537.
[22] DEVLIN J,CHANG M,LEE K,et al.Bert:pre-training of deep bidirectional transformers for language understanding[J].Journal of Biomedical Informatics,2020,107(5):1-15.
[23] LIU B,TANGR M,CHEN Y Z,et al.Feature generation by convolutional neural network for click-through rate prediction[C]//The world wide web conference.New York:ACM,2019:1119-1129.
[24] WANG Y,LI Y,TONG H,et al.HIT:nested named entity recognition via head-tail pair and token interaction[C]//Proceedings of the 2020 conference on empirical methods in natural language processing(EMNLP).Punta Cana:ACL,2020:6027-6036.
[25] 赵丰,黄健,张中杰.LAC-DGLU:基于CNN和注意力机制的命名实体识别模型[J].计算机科学,2020,47(11):212-219.
ZHAO F,HUANG J,ZHANG Z J.LAC-DGLU:named entity recognition model based on CNN and attention mechanism[J].Computer Science,2020,47(11):212-219.(in Chinese)
[26] 羊艳玲,李燕,钟昕妤,等.基于BiLSTM-CRF的中医医案命名实体识别[J].中医药信息,2021,38(11):15-21.
YANG Y L,LI Y,ZHONG X Y,et al.Named entity recognition of TCM medical recordsbased on BiLSTM-CRF[J].Information on Traditional Chinese Medicine,2021,38(11):15-21.(in Chinese)