Kelch样基因(Kelch-like)是指包含Kelch重复元件的一系列基因的集合,是存在于动物体中具有重要功能的基因家族。Kelch超家族蛋白除含有Kelch结构域外,通常还包括BTB/POZ结构域和BACK结构域[1]。目前,在哺乳动物中已经有40多种Kelch蛋白被报道[2]。Kelch家族蛋白可以控制肌动蛋白微丝的组装和解聚,参与细胞骨架形态的调节及伪足的形成,进而参与癌细胞转移的过程[3]。在无脊椎动物中,通过对马氏珠母贝Pinctada fucata martensii[4]、长牡蛎Crassostrea gigas[5]、栉孔扇贝Azumapecten farreri[6]和双斑蛸Octopus bimaculatus[7]等基因组分析,均鉴定到了多个Kelch-like基因,但其具体功能及序列变异信息的相关报道相对较少。本课题组前期分析马氏珠母贝基因组Kelch-like基因在组织转录组的表达时发现,其在生物矿化相关组织珍珠囊和外套膜的表达量相对较高,暗示其可能参与马氏珠母贝生物矿化过程。
马氏珠母贝又称合浦珠母贝,是培育海水珍珠的重要贝类,目前已成为中国广东、海南和广西部分沿海地区特色产业之一,其生物矿化过程一直是研究者关注的热点[4]。当贝壳受到损伤时,软体动物会启动贝壳的修复机制,在这一过程中,与生物矿化相关基因的表达量通常会有明显的变化[8-9]。本研究中,分析了马氏珠母贝Kelch样基因序列及其在缺壳损伤后不同时期的表达模式,探究了其在珍珠贝贝壳形成中的作用和功能,以期为研究珍珠贝生物矿化过程提供理论依据。
1 材料与方法
1.1 材料
试验用马氏珠母贝取自广东省湛江市徐闻县大井村海区。取规格一致、鳞片旺盛、生长良好的个体用于试验。
1.2 方法
1.2.1 Kelch-like基因序列分析 利用马氏珠母贝基因组序列[4]获得Kelch-like CDS(coding sequence)序列,利用CDS序列对全长转录组数据(未发表)进行分析,获得Kelch-like基因序列全长。采用ORF Finder在线预测基因开放式阅读框(open reading frame,ORF)和氨基酸序列,并与基因组氨基酸序列进行比对验证;采用DNAMAN软件和Primer Premier 5.0软件对预测的氨基酸序列进行验证,采用NCBI在线比对,鉴定基因所属的蛋白家族;采用ProtParam软在线分析氨基酸序列的理化性质,采用SMART软件预测目的蛋白的结构域,采用TMHMM Server 2.0软件在线预测蛋白序列的跨膜结构域,采用ClustalW2对氨基酸序列进行同源性比对;采用SOPMA预测蛋白的二级结构,采用Phyre2在线预测蛋白的三级结构,并利用Chimera 1.8.1对蛋白的高级结构及位点进行分析。
1.2.2 组织样品的采集 随机选取10只马氏珠母贝,在闭壳肌处抽血,经离心获得血细胞(B),并剪取马氏珠母贝的中央膜(MC)、套膜区(MP)、边缘膜(ME)、闭壳肌(A)、足(F)、肝胰腺(HE)、性腺(GO)、珍珠囊(PS)和鳃(GI),经液氮速冻后,于-80 ℃超低温冰箱中保存备用。
1.2.3 贝壳损伤修复试验 取35只生长良好的马氏珠母贝(壳高为5~7 cm),在其贝壳边缘的同一位置剪一个“V”形缺口,在贝壳受损后的0、6、12、24、36、48、120 h分别剪取5只马氏珠母贝的外套膜,提取总RNA,进行荧光定量PCR,以检测Kelch-like基因在贝壳损伤后的表达。
1.2.4 总RNA 提取、第一链cDNA的合成及基因表达模式分析 采用Trizol法提取马氏珠母贝全组织的总RNA。采用琼脂糖凝胶电泳检测提取RNA的完整性,并利用NanoDropND 1000紫外分光光度计测量A260 mm/A280 mm 值,并分析其浓度及纯度。采用Reverse Transcriptase M-MLV(RNaseH) 进行反转录获得所需的cDNA模板,然后进行实时荧光定量PCR检测。以GAPDH作为实时荧光定量PCR的内参基因[10],利用软件Primer Premier 5.0设计Kelch-like的荧光定量引物(表1)。采用2-△Ct 法对目的基因的实时荧光定量数据进行计算(n=10)。
表1 引物序列
Tab.1 Primer sequences

基因gene引物序列primersequence用途functionKelch-like-STCTTGGGTTTGCCACTGTTAT荧光定量Kelch-like-AGGCACTAAAGTATTGGTTTGACG荧光定量GAPDH-SGCAGATGGTGCCGAGTATGT荧光定量(内参)GAPDH-ACGTTGATTATCTTGGCGAGTG荧光定量(内参)
1.2.5 单核苷酸多态性(SNP)位点分析 选取马氏珠母贝“海选1号”220个个体进行靶区重测序,获得Kelch-like外显子区SNP位点。利用Popgene 32软件计算等位基因数、期望杂合度(He)、观测杂合度(Ho)和哈迪-温伯格平衡(HWE)等遗传参数。采用PIC软件计算SNP位点的多态性信息含量(PIC)。多态性分为3种:高度多态(PIC≥0.50)、低度多态(PIC≤0.25)和中度多态(0.25 利用SPSS 17统计软件对基因表达量数据进行单因素方差分析,若差异显著,采用Tukey法进行组间多重比较,显著性水平设为0.05。 马氏珠母贝Kelch-like基因序列全长为2 127 bp,其中,5′UTR为58 bp,3′UTR为44 bp,其开放式阅读框长度为2 025 bp,可编码674个氨基酸(图1)。通过SMART软件分析得出,Kelch-like氨基酸序列含有1个BTB结构域、1个BACK结构域和2个Kelch结构域。利用TMHMM Server 2.0软件预测其跨膜结构域,结果显示,Kelch-like无跨膜螺旋区,不属于跨膜蛋白。 采用ProtParam软件预测马氏珠母贝Kelch-like蛋白的理论相对分子质量为76 210,等电点为6.74。其中,含量较高的氨基酸为Leu (11.1%)、Ser (9.5%)、Lys (6.4%)和Val (6.1%),负电荷残基为74个,正电荷残基为71个。预测该蛋白的脂溶指数(aliphatic index)为87.92,总平均亲水性(grand averageofhydropathy,GRAVY) 为-0.215,属于亲水性蛋白。采用SOPMA软件对Kelch-like蛋白的二级结构进行预测,发现α螺旋结构占整体的42.14%,β转角结构占整体的5.49%,延伸链占整体的18.55%,无规则折叠占整体的33.82%。 5′和3′非编码区用小写字母表示;编码区及推导的氨基酸序列用大写字母表示;方框内为起始密码子(ATG)和终止密码子(TAA);灰色阴影部分为BTB结构域;黄色阴影部分为BACK结构域;绿色阴影部分为Kelch结构域。 采用ClustalW2软件,将马氏珠母贝、美洲牡蛎Crassostrea virginica (XP_022313917.1)、虾夷扇贝Mizuhopecten yessoensis (XP_021342105.1)的Kelch-like氨基酸序列进行同源性比对,发现马氏珠母贝Kelch-like与其他物种的Kelch-like同源性较高,其中,与美洲牡蛎Kelch-like氨基酸序列的一致性最高(35%),其次为虾夷扇贝,一致性为32.05%。另外,分别对马氏珠母贝、美洲牡蛎、虾夷扇贝Kelch-like氨基酸序列的BTB、BACK和Kelch结构域进行多序列比对,发现马氏珠母贝Kelch-like氨基酸序列的BTB和BACK保守性较高,且Kelch具有保守的GG位点(图2)。 利用Phyre 2程序对马氏珠母贝Kelch-like蛋白进行三维结构预测,并与美洲牡蛎、虾夷扇贝的Kelch-like进行比较(图3),发现马氏珠母贝Kelch-like的BTB、BACK和Kelch结构域在高级结构上高度保守,进一步说明其功能的保守性。 从图4可见,Kelch-like在马氏珠母贝中央膜、套膜区、边缘膜、闭壳肌、足、肝胰腺、性腺、珍珠囊、鳃和血液中均有表达,其中,在边缘膜的表达量显著高于其他组织 (P<0.05),其他组织间的表达水平无显著性差异(P>0.05)。 A、B和C分别表示BTB domain、BACK domain和Kelch domain的多序列比对;深蓝色阴影为保守的氨基酸;粉色阴影为强相似的氨基酸;浅蓝色为弱相似的氨基酸;右边数字为序列氨基酸的个数。 橙色为β折叠,绿色为α螺旋。 为探究Kelch-like基因在生物矿化过程中的作用,本研究中,对该基因在贝壳损伤修复过程中的表达模式进行分析。结果显示,马氏珠母贝外套膜组织中的Kelch-like基因分别在贝壳受损后6、12、120 h时表达量显著上调(P<0.05)(图5)。 图5 Kelch-like在马氏珠母贝损伤修复过程中的表达变化 对马氏珠母贝Kelch-like基因外显子区SNP位点进行分析,共获得56个SNP位点。使用PopGen 32软件分析发现,各位点观测杂合度为0.109 1~0.622 7,平均值为0.363 3,期望杂合度为0.103 4~0.500 1,平均值为0.330 2;39个SNP位点符合哈迪-温伯格平衡;各位点多态性信息含量PIC为0.097 7~0.374 5,其中,25个SNP属于低度多态,31个SNP属于中度多态(表2)。 ME—边缘膜;MP—套膜区;MC—中央膜;A—闭壳肌;F—足;GO—性腺;GI—鳃;HE—肝胰腺;B—血液;PS—珍珠囊。标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05),下同。 表2 Kelch-like基因的SNP位点信息 SNP位点locus观测杂合度Ho期望杂合度He多态性信息含量PIC哈迪-温伯格平衡HWESNP192515778G>T0.59090.49700.37290.0050SNP292515872G>A0.54090.49240.37060.1433SNP392515873A>G0.47270.41350.32750.0331SNP492515924A>T0.12730.12740.11900.9922SNP592515976A>G0.18180.16570.15160.1434SNP692516009T>A0.52270.49240.37060.3604SNP792516021A>G0.58640.49810.37350.0085SNP892516030G>A0.55000.49470.37180.0963SNP992516079A>G0.18640.21240.18950.0669SNP1092516109A>G0.10910.10340.09770.4027SNP1192516165C>T0.17270.15820.14540.1670SNP1292516224T>A0.26820.25880.22490.5867SNP1392516234A>C0.16820.20540.18390.0067SNP1492516258G>A0.51820.49410.37150.4695SNP1592516261C>T0.51360.49360.37120.5460SNP1692516289C>A0.18180.20190.18110.1365SNP1792516303T>C0.18180.20190.18110.1365SNP1892516308A>G0.59550.49810.37350.0037SNP1992516342C>T0.51820.45600.35150.0427SNP2092516377C>T0.32270.30680.25920.4390SNP2192516417G>T0.33640.31520.26510.3174SNP2292516420C>T0.18180.16570.15160.1434SNP2392516492A>G0.55450.49410.37150.0692SNP2492516508C>A0.55270.46260.35510.0533SNP2592516540A>C0.59550.49880.37380.0040SNP2692516579G>A0.56820.48850.36860.0152SNP2792516582A>T0.53180.46000.35370.0203SNP2892516672T>C0.18180.16570.15160.1434SNP2992516702T>A0.54090.49120.37000.1323SNP3092516705A>G0.50450.46000.35370.1502SNP3192516711A>C0.17730.16190.14850.1549SNP3292516777T>C0.12730.12740.11900.9922SNP3392516795T>G0.15910.14680.13560.2069SNP3492516807C>A0.16360.16570.15160.8542SNP3592516865T>C0.20910.19480.17550.2728SNP3692516892C>A0.12270.11500.10860.3418SNP3792516918A>T0.52730.46390.35570.0421SNP3892516989A>G0.18640.16940.15470.1326SNP3992517173T>C0.15910.14680.13560.2069SNP4092517174A>G0.16360.20890.18670.0012SNP4192517245T>C0.55450.49180.37030.0579SNP4292517258C>T0.16360.15820.14540.6038SNP4392517290A>C0.62270.49570.37220.0001SNP4492517320T>C0.10910.10340.09770.4027SNP4592517350C>T0.15000.13910.12910.2368SNP4692517401A>T0.33640.32070.26880.4675SNP4792517482A>G0.41820.41730.32970.9741SNP4892517502A>C0.14090.13910.12910.8419SNP4992517534C>T0.56820.49940.37410.0406SNP5092517538A>G0.57270.49850.37400.0283SNP5192517546C>A0.56360.46870.35830.0026SNP5292517572A>C0.57270.49850.37370.0268SNP5392517591A>G0.18180.16570.15160.1434SNP5492517604A>G0.57730.49990.37440.0214SNP5592517620A>G0.40450.41910.33070.6048SNP5692517647T>C0.55450.50010.37450.1056 对SNP位点进行单倍块分析发现,有10个SNP位点形成了一个单倍块,为4种单倍型(表3)。结合表型分析发现,单倍型AATCTGTCGG为优异单倍型,其壳质量显著大于单倍型AGCCTACCGA和AGCCTACTTA(P<0.05)(图6)。 表3 Kelch-like基因SNP位点单倍型分析 单倍型haplotypeSNP13SNP14SNP15SNP16SNP17SNP18SNP19SNP20SNP21SNP23频率frequencyHap1AATCTGTCGG0.323Hap2AGCCTACCGA0.232Hap3AGCCTACTTA0.184Hap4CGCACGCCGA0.094 图6 Kelch-like单倍型与表型的关系 Kelch-like蛋白通常含有1个BTB/POZ结构域、1个BACK结构域和5~6个Kelch结构域,但各个成员的构成均不相同[1]。本研究中,马氏珠母贝Kelch-like蛋白序列包括1个BTB结构域、1个BACK结构域和2个Kelch结构域,这与上述的结构域分析结果类似。BTB蛋白是E3泛素连接酶的桥头蛋白,在果蝇Drosophila melanogaster、小鼠Mus musculus及人类Homo sapiens的进化过程中均较为保守[11];BTB结构域具有促进蛋白质结合及二聚化反应的功能,BTB结构域的正确折叠和形成的蛋白复合体是BTB蛋白具有正常转录调控功能的必要条件[12-13]。BTB蛋白具有抑制转录的功能,还可以通过对局部核小体的解旋和重新构造而达到转录激活的作用[14-17]。BACK结构域中保守的氨基酸包括N端的Asn-Cys-Leu-Gly-Lle、Val-Arg-[Leu/Met/Phe]-Pro-Leu-Leu和一些疏水氨基酸,BACK结构域在介导蛋白的功能中发挥重要作用[18]。Kelch重复元件由4~7个Kelch构象组成,每个Kelch构象是由4条链组成的β折叠,包含44~56个氨基酸残基[19],具有8个关键的保守位点,包括4个疏水氨基酸,紧临的是2个连续的Gly(GG),隔开一段序列后是2个有固定间隔的芳香族氨基酸Y和W[20-21]。进化保守的结构意味着Kelch结构域在细胞间相互作用、细胞骨架形态、基因表达调控及蛋白质结合方面具有重要的生物学功能[1,20]。本研究中,对马氏珠母贝Kelch-like的蛋白序列进行高级结构分析,发现其BTB、BACK和Kelch结构域在高级结构上高度保守,且与美洲牡蛎和虾夷扇贝的Kelch-like序列结构高度相似。 软体动物外套膜为贝壳矿化的主要组织,许多与矿化过程直接相关的分泌蛋白均在外套膜中大量表达并分泌到体外时行使其矿化功能[9]。软体动物外套膜在功能上存在区域性分工,一般认为,外套膜边缘区负责方解石结晶的棱柱层的形成,而套膜区和中央区则负责文石结晶的珍珠层的形成[22-23]。本研究中发现,马氏珠母贝Kelch-like基因在外套膜边缘区高表达,说明其可能参与贝壳棱柱层的形成过程。科研工作者常用缺壳损伤试验研究参与贝壳形成过程中的蛋白等大分子物质。研究发现,对贝壳进行缺壳损伤后,栉孔扇贝会立刻启动修复程序,再生损伤部位的贝壳,在此过程中参与贝壳矿化的蛋白表达量会产生明显的变化[9];马氏珠母贝贝壳受到损伤后,CREB3L2基因和基质蛋白的表达量会迅速上升,且CREB3L2基因的峰值出现更早,推测其通过影响基质蛋白转录,参与马氏珠母贝生物矿化的调控过程[8];贝壳损伤修复过程中几丁质酶基因表达水平呈现下调趋势,暗示其作为负调控因子参与贝壳修复过程[24]。本研究中,在马氏珠母贝贝壳损伤后6 h,Kelch-like基因出现高表达,暗示其在贝壳损伤修复过程中发挥了作用,进一步说明其在贝壳矿化中的作用。 单核苷酸多态性(SNP)主要是指基因组水平同一位置碱基变异而引起的DNA序列多态性,且SNP遗传稳定性好,通常被认为是二等位基因分子标记,有利于对其进行基因分型[25]。Salem等[26]利用转录组获得50 k的SNP芯片,鉴定获得虹鳟Oncorhynchus mykiss肌肉含量相关数量性状位点(QTLs),其中3个SNP位点位于候选基因Kelch protein 21,该基因通过调控细胞繁殖或分化在骨骼肌发育过程中发挥重要作用。Gonzalez-Pena等[27]对虹鳟出肉率性状进行全基因组关联分析(GWAS),发现出肉质量相关联位点注释到Kelch domain-containing protein 8b基因。Jin等[28]利用250 k SNP芯片对鲇耐热性进行全基因组关联分析,获得了14个性状相关候选基因,其中TRAF2、FBXW5、ANAPC2、UBR1和Kelch-like 29 5个基因通过参与泛素化过程在蛋白质降解过程中发挥重要作用。单倍型是SNP沿染色体的物理排列,其提供的遗传变异信息比SNP更能代表基因的多样性[29]。本研究中,马氏珠母贝Kelch-like基因单倍型AATCTGTCGG的壳质量显著大于单倍型AGCCTACCGA和AGCCTACTTA,说明该单倍型具有作为以壳质量性状为目标的育种标记的潜力,为进一步进行马氏珠母贝优良经济性状的选育提供了参考依据。 1)马氏珠母贝Kelch-like基因在外套膜边缘区显著高表达,并参与贝壳损伤修复过程,说明其在贝壳矿化中发挥着重要作用。 2)马氏珠母贝Kelch-like的单倍型AATCTGTCGG为优异单倍型,该单倍型有望作为以壳质量为目标的育种标记,该结果对于选育具有优良性状的马氏珠母贝具有重要的理论意义。 [1] DHANOA B S,COGLIATI T,SATISH A G,et al.Update on the Kelch-like (KLHL) gene family[J].Human Genomics,2013,7(1):13. [2] 肖枫,宋宏涛,魏群.哺乳动物中Kelch超家族蛋白的结构与功能[J].生物化学与生物物理进展,2011,38(3):210-217. XIAO F,SONG H T,WEI Q.Structure and function of Kelch proteins in mammals[J].Progress in Biochemistry and Biophysics,2011,38(3):210-217.(in Chinese) [3] XUE F Y,COOLEY L.Kelch encodes a component of intercellular bridges in Drosophila egg chambers[J].Cell,1993,72(5):681-693. [4] DU X D,FAN G Y,JIAO Y,et al.The pearl oyster Pinctada fucata martensii genome and multi-omic analyses provide insights into biomineralization[J].Gigascience,2017,6(8):gix059.DOI:10.1093/gigascience/gix059. [5] ZHANG G F,FANG X D,GUO X M,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54. [6] LI Y L,SUN X Q,HU X L,et al.Scallop genome reveals molecular adaptations to semi-sessile life and neurotoxins[J].Nature Communications,2017,8(1):1721. [7] ALBERTIN C B,SIMAKOV O,MITROS T,et al.The octopus genome and the evolution of cephalopod neural and morphological novelties[J].Nature,2015,524(7564):220-224. [8] 鲁冰,郑向南,刘阳嘉,等.合浦珠母贝转录因子PF-CREB3L2基因的克隆和鉴定[J].海洋科学,2018,42(3):77-83. LU B,ZHENG X N,LIU Y J,et al.Cloning and mineralization-related functions of the PF-CREB3L2 gene in Pinctada fucata[J].Marine Sciences,2018,42(3):77-83.(in Chinese) [9] 王俊,高静,谢军,等.栉孔扇贝几丁质酶基因的克隆及功能鉴定[J].水产学报,2017,41(2):189-197. WANG J,GAO J,XIE J,et al.Cloning and mineralization-related functions of the chitinase gene in Chlamys farreri[J].Journal of Fisheries of China,2017,41(2):189-197.(in Chinese) [10] HAO R J,ZHENG Z,WANG Q H,et al.Molecular and functional analysis of PmCHST1b in nacre formation of Pinctada fucata martensii[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2018,225:13-20. [11] SIGGS O,BEUTLER B.The BTB-ZF transcription factors[J].Cell Cycle,2012,11(18):3358-3369. [12] ALBAGLI O,DHORDAIN P,DEWEINDT C,et al.The BTB/POZ domain:a new protein-protein interaction motif common to DNA- and actin-binding proteins[J].Cell Growth &Differentiation:The Molecular Biology Journal of the American Association for Cancer Research,1995,6(9):1193-1198. [13] AHMAD K F,ENGEL C K,PRIV [14] LI X,PENG H,SCHULTZ D C,et al.Structure-function studies of the BTB/POZ transcriptional repression domain from the promyelocytic leukemia zinc finger oncoprotein[J].Cancer Research,1999,59(20):5275-5282. [15] PHAN R T,DALLA-FAVERA R.The BCL6 proto-oncogene suppresses p53 expression in germinal-centre B cells[J].Nature,2004,432(7017):635-639. [16] KELLY K F,OTCHERE A A,GRAHAM M,et al.Nuclear import of the BTB/POZ transcriptional regulator Kaiso[J].Journal of Cell Science,2004,117(25):6143-6152. [17] TAKENAGA M,HATANO M,TAKAMORI M,et al.Bcl6-dependent transcriptional repression by BAZF[J].Biochemical and Biophysical Research Communications,2003,303(2):600-608. [18] STOGIOS P J,PRIV [19] BORK P,DOOLITTLE R F.Drosophila Kelch motif is derived from a common enzyme fold[J].Journal of Molecular Biology,1994,236(5):1277-1282. [20] ADAMS J,KELSO R,COOLEY L.The Kelch repeat superfamily of proteins:propellers of cell function[J].Trends in Cell Biology,2000,10(1):17-24. [21] PRAG S,ADAMS J C.Molecular phylogeny of the Kelch-repeat superfamily reveals an expansion of BTB/Kelch proteins in animals[J].BMC Bioinformatics,2003,4(1):42. [22] 傅郁.海湾扇贝(Argopecten irradians)外套膜组织学组织化学研究[D].青岛:中国海洋大学,2007. FU Y.Histological and histochemical study of the mantle of Argopecten irradians[D].Qingdao:Ocean University of China,2007.(in Chinese) [23] 田群莉.马氏珠母贝外套膜不同区域差异microRNA的筛选和功能研究[D].湛江:广东海洋大学,2015. TIAN Q L.Identification and functional studies of the microRNAs from the marginal and central zone of Pinctada martensii[D].Zhanjiang:Guangdong Ocean University,2015.(in Chinese) [24] MOUNT A S,WHEELER A P,PARADKAR R P,et al.Hemocyte-mediated shell mineralization in the eastern oyster[J].Science,2004,304(5668):297-300. [25] 雷超.马氏珠母贝类胡萝卜素代谢相关基因的筛选及SNP分析[D].湛江:广东海洋大学,2018. LEI C.Screening and SNP analysis on carotenoid metabolism related genes in Pinctada fucata martensii [D].Zhanjiang:Guangdong Ocean University,2018.(in Chinese) [26] SALEM M,AL-TOBASEI R,ALI A,et al.Genome-wide association analysis with a 50K transcribed gene SNP-chip identifies QTL affecting muscle yield in rainbow trout[J].Frontiers in Genetic,2018,9:387. [27] GONZALEZ-PENA D,GAO G T,BARANSKI M,et al.Genome-wide association study for identifying loci that affect fillet yield,carcass,and body weight traits in rainbow trout (Oncorhynchus mykiss)[J].Frontiers in Genetics,2016,7:203. [28] JIN Y,ZHOU T,GENG X,et al.A genome-wide association study of heat stress-associated SNPs in catfish[J].Animal Genetics,2017,48(2):233-236. [29] DUFFY D L,MONTGOMERY G W,CHEN W,et al.A three-single-nucleotide polymorphism haplotype in intron 1 of OCA2 explains most human eye-color variation[J].American Journal of Human Genetics,2007,80(2):241-252.1.3 数据处理
2 结果与分析
2.1 Kelch-like基因的序列和结构
2.2 Kelch-like蛋白的理化性质

5′UTR and 3′UTR are shown by letters;open reading fragment and the deduced amino acid sequences are indicated with capital letters;nucleotides in a box are the initiation codon (ATG) and stop codon (TAA);the sequence in grey background represents the BTB domain;the sequence in yellow background represents the BACK domain;the sequence in green background represents the Kelch domain.
图1 马氏珠母贝Kelch-like核苷酸序列及推导的氨基酸序列
Fig.1 Nucleotide sequence and the deduced amino acid sequence of Kelch-like in pearl oyster Pinctada fucata martensii 2.3 Kelch-like氨基酸同源性分析及三级结构预测
2.4 Kelch-like基因的组织表达模式
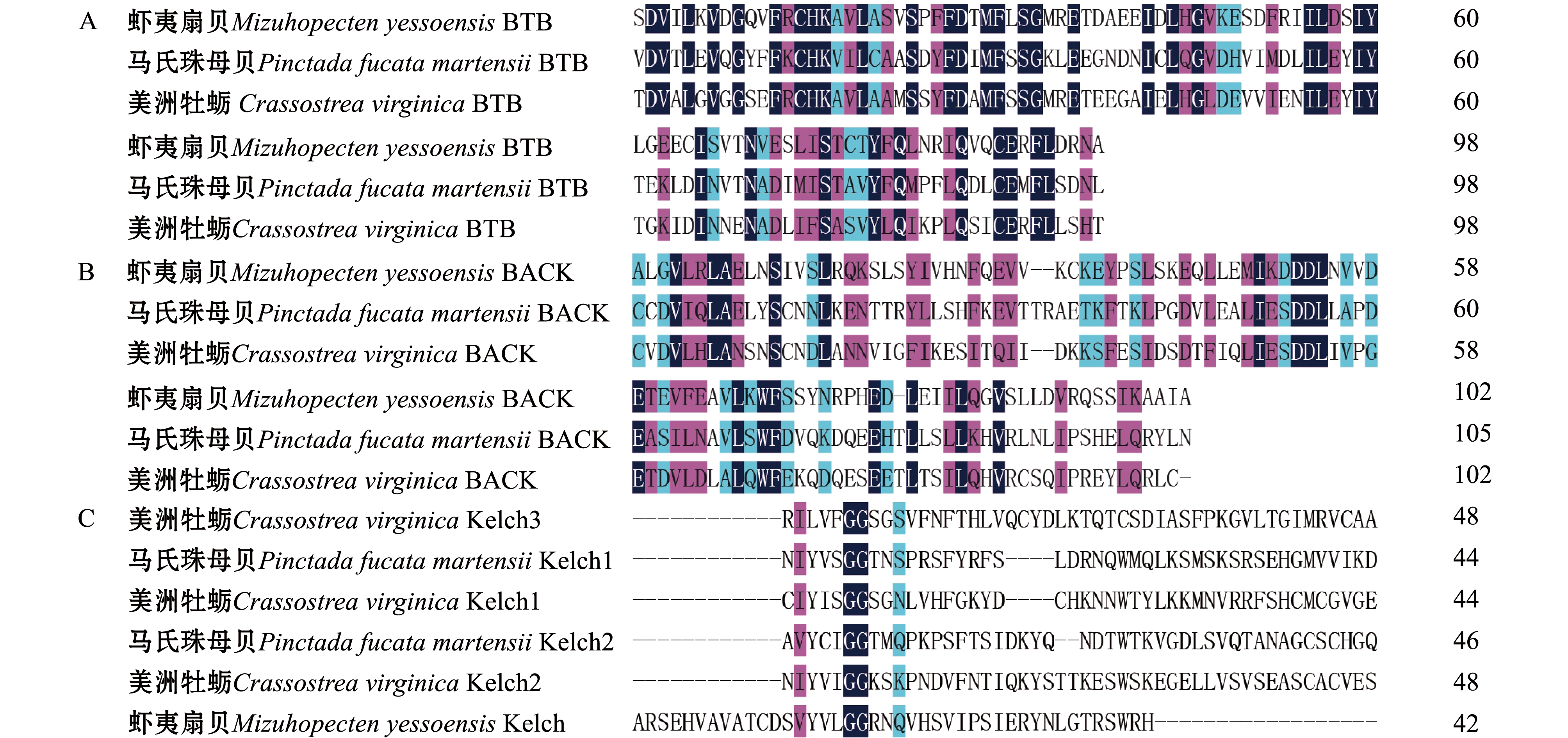
A,B and C represent the multisequence alignment of BTB domain,BACK domain and Kelch domain respectively;dark blue background indicates the conserved amino acid;pink background indicates amino acid with strong similarity;light blue indicates amino acid with weak similarity;the numbers in the right show the number of the sequence alignment amino acid.
图2 马氏珠母贝与其他物种Kelch-like氨基酸的多序列比对
Fig.2 Multiple alignment of amino acid sequence of Kelch-like between the pearl oyster Pinctada fucata martensii and other species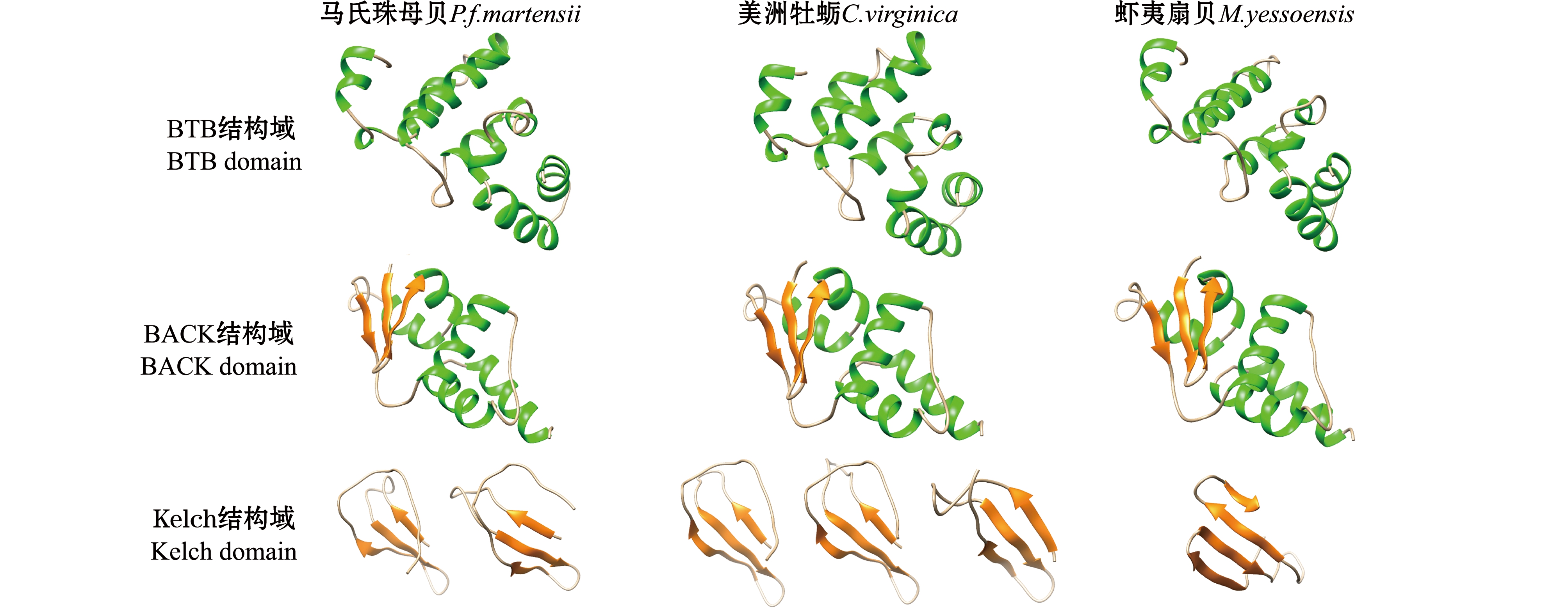
α helix and β sheet are showed in green and orange color,respectively.
图3 马氏珠母贝、美洲牡蛎和虾夷扇贝Kelch-like结构域的三维结构分子模型
Fig.3 Molecular model of the three-dimensional structure of the Kelch-like domain of pearl oyster Pinctada fucata martensii,America oyster Crassostrea virginica and Yesso scallop Mizuhopecten yessoensis 2.5 Kelch-like在贝壳损伤修复过程中的表达变化
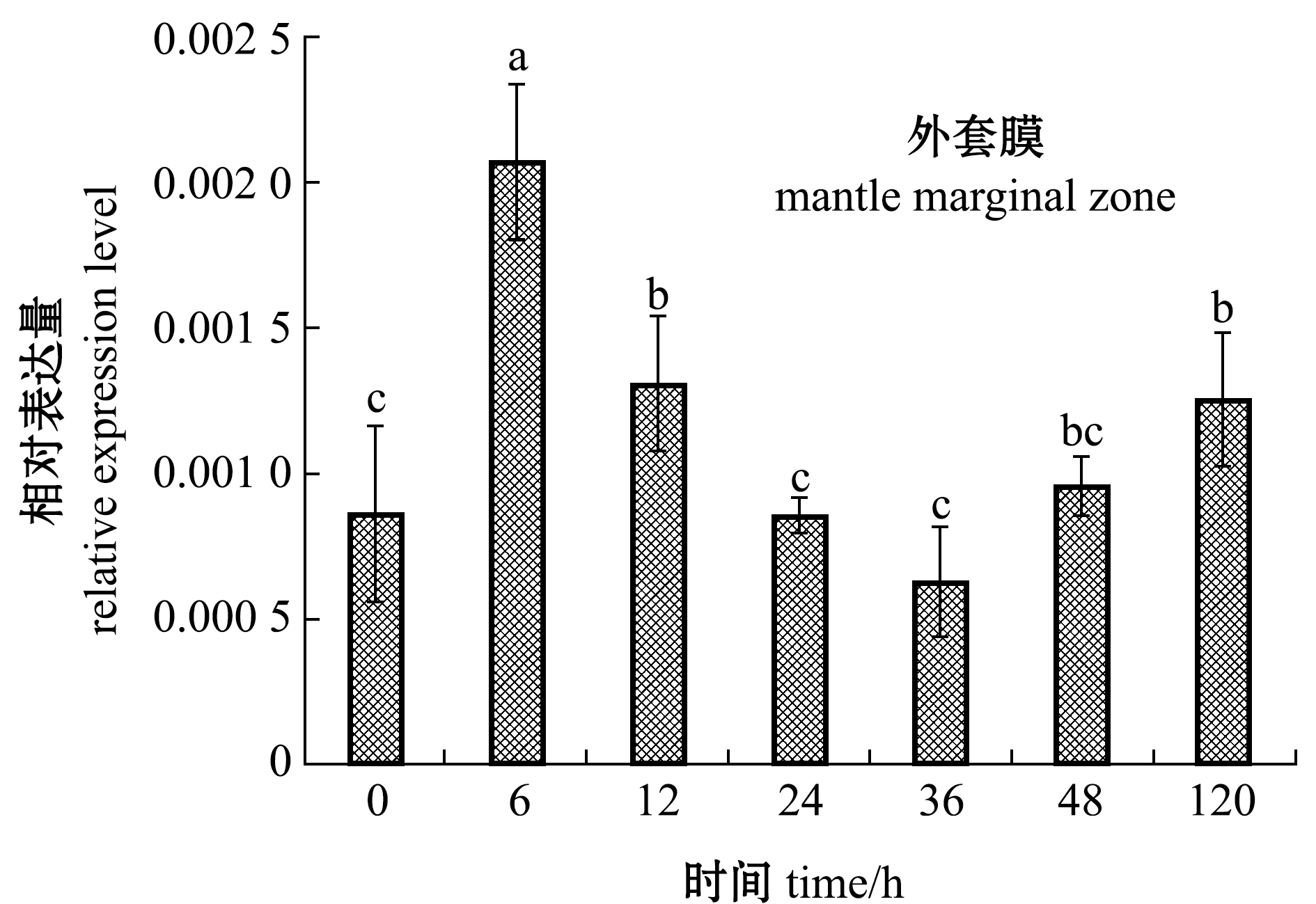
Fig.5 Change in expression level of Kelch-like gene in shell notching from pearl oyster Pinctada fucata martensii2.6 Kelch-like基因SNP位点及单倍型
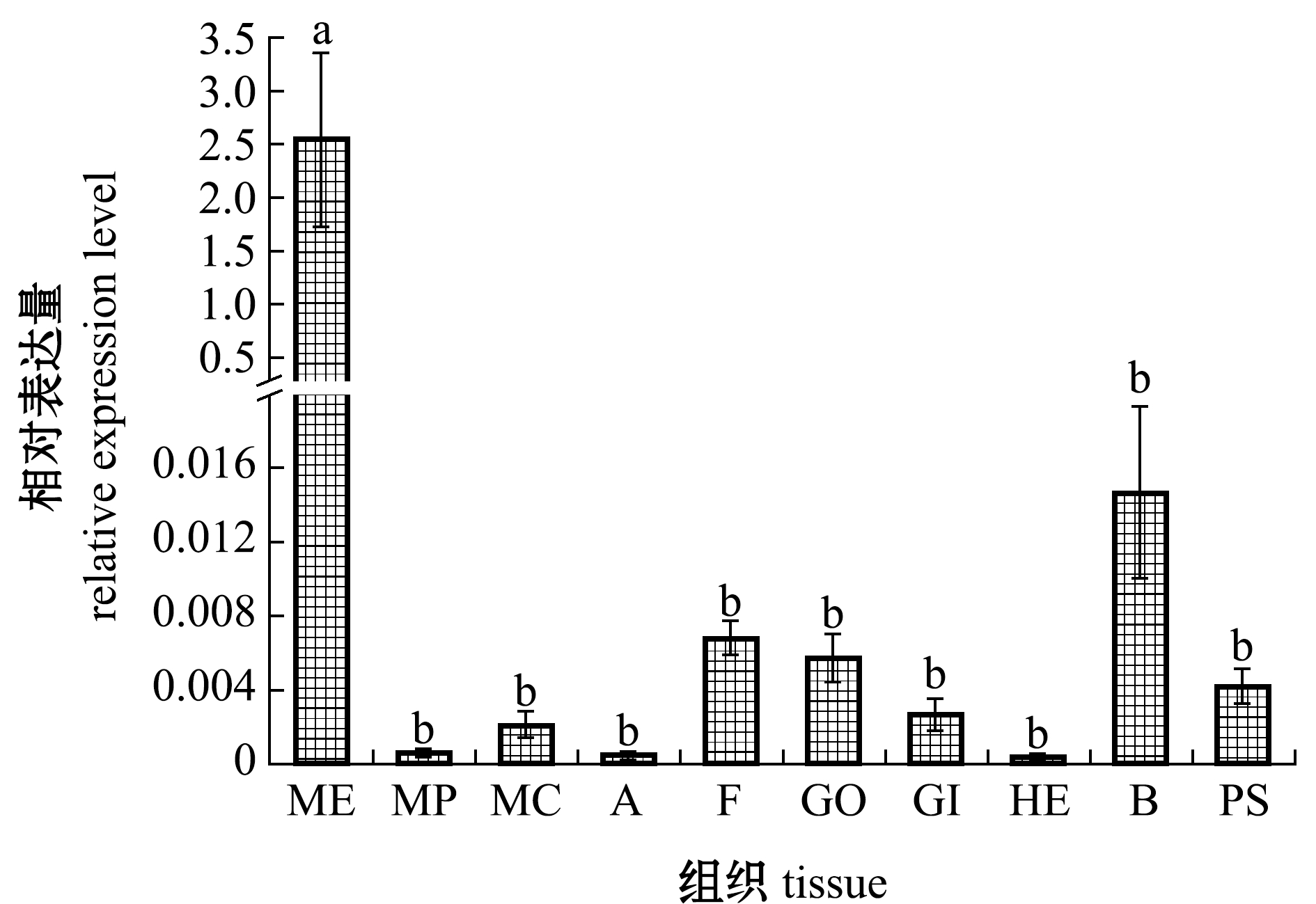
ME—marginal zone;MP—mantle zone;MC—central mantle zone;A—adductor muscle;F—foot;GO—gonad;GI—gill;HE—hepatopancreas;B—hemocyte;PS—pearl sac.The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter are not significant differences,et sequentia.
图4 Kelch-like在马氏珠母贝不同组织中的相对表达量
Fig.4 Relative expression level of Kelch-like gene in different tissues of pearl oyster Pinctada fucata martensii
Tab.2 SNP information of Kelch-like gene 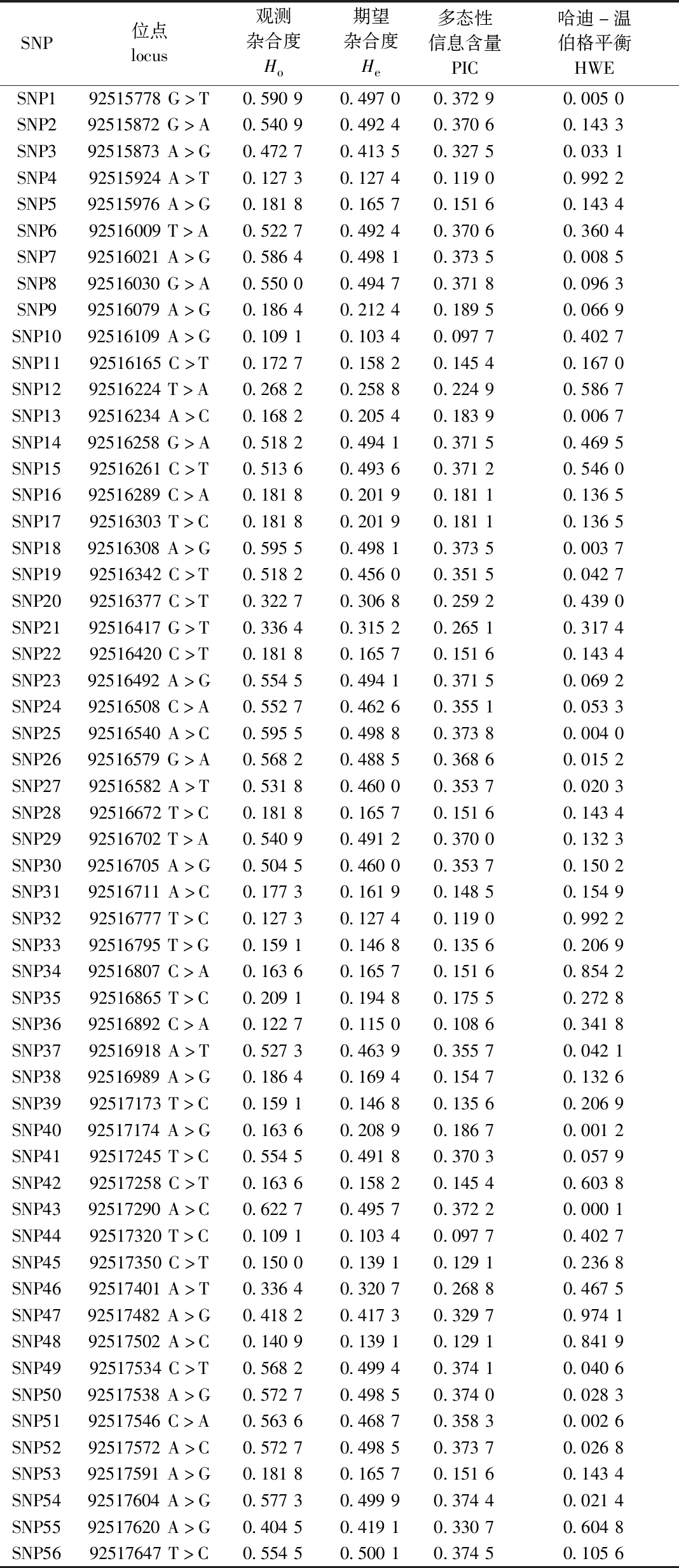
Tab.3 SNP haplotype analysis of Kelch-like gene 
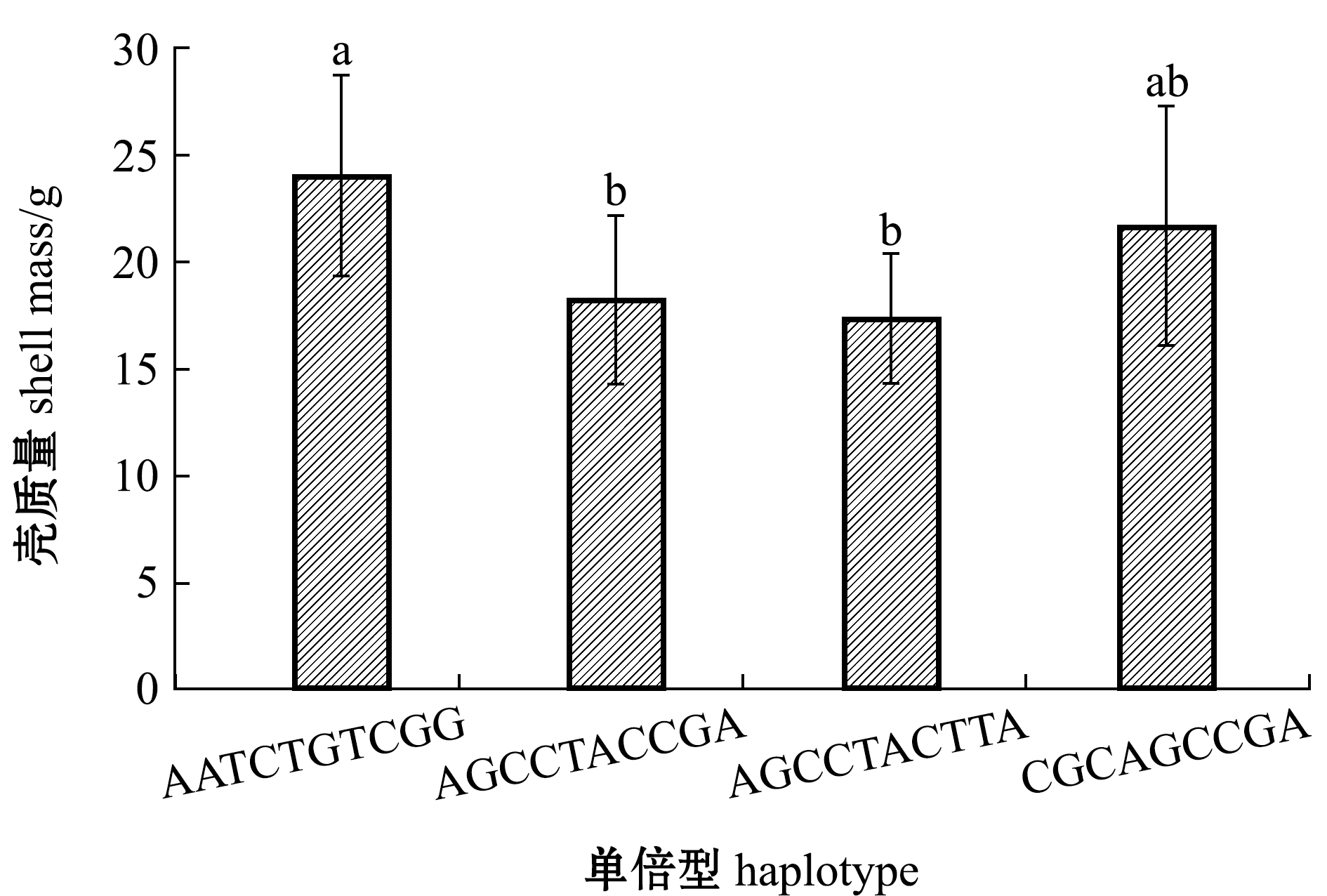
Fig.6 Relationship between Kelch-like haplotype and phenotype3 讨论
3.1 马氏珠母贝Kelch-like基因的序列特征
3.2 马氏珠母贝Kelch-like基因的表达模式
3.3 马氏珠母贝Kelch-like的SNP位点分析
4 结论
 G G.Crystal structure of the BTB domain from PLZF[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(21):12123-12128.
G G.Crystal structure of the BTB domain from PLZF[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(21):12123-12128. G G.The BACK domain in BTB-Kelch proteins[J].Trends in Biochemical Sciences,2004,29(12):634-637.
G G.The BACK domain in BTB-Kelch proteins[J].Trends in Biochemical Sciences,2004,29(12):634-637.