虹鳟Oncorhynchus mykiss是一种具有较高经济价值的冷水性鱼类[1],其生长过程不仅需要较低的水温(12~18 ℃),还需要较高的溶氧(通常在7 mg/L以上)[2-3]。因此,中国虹鳟的主要养殖方式是利用冷水湖泊或水库的网箱养殖,以及利用冷溪流、冷泉水、水库底排冷水的流水养殖,且受地理环境制约较大[4]。
Ep300/Crebbp家族是脊椎动物中普遍存在的一类重要的乙酰转移酶,其丰富的结合结构域使得其可以参与到诸多调控网络中,调节上千个基因的表达,从而保持细胞的稳态,帮助有机体应对外界环境的变化[5]。有研究表明,Ep300/Crebbp也是HIF-1α通路的关键成分[6-7]:低氧条件下HIF-1α蛋白的稳定性比常氧条件下显著增强,累积的HIF-1α会首先通过其C-TAD结构域募集Ep300/Crebbp,再通过bHLH结构域与HIF-1β形成二聚体,变成有功能的转录复合体,最后通过Ep300/Crebbp蛋白上的启动子结合结构域与靶基因上的低氧反应元件(hypoxia response elements,HREs)结合,最终启动下游100~200个基因的表达。HIF-1α已被证明是真核细胞维持氧平衡最主要的调节因子之一[7],因此,Ep300/Crebbp事实上也处于将低氧信号转化为转录调节信号的枢纽位置。
真骨鱼类共同祖先在与其他脊椎动物的祖先分枝发生分歧后经历过全基因组重复(whole genome duplication,WGD)事件[8],虽然多数基因都很快丢失了其中1个拷贝,但笔者前期研究发现,ep300的双拷贝在不同真骨鱼类群中均得到了普遍保留[9]。虹鳟则更为特殊,其较近的一个祖先物种在大约1亿年前还经历过一次额外的全基因组重复事件[10],合理推测其ep300/crebbp家族成员应该更多。一般而言,基因的拷贝数增多至少在早期会伴随剂量效应,从而导致其功能增强[11]。但虹鳟反而不耐低氧,表明其相关功能基因可能丢失了大量拷贝,或者大量拷贝的功能发生了缺失。本研究中,对虹鳟ep300/crebbp家族成员进行拷贝数、组织表达和结构域分析,探讨其不耐低氧的分子机制,以期为虹鳟耐低氧性能改良提供有效的候选基因。
1 材料与方法
1.1 材料
选择斑马鱼Danio rerio作为虹鳟的近缘物种,选择小鼠Mus musculus和原鸡Gallus gallus作为远缘物种。3个物种已被注释的ep300/crebbp家族成员均通过NCBI的基因数据库检索获取,相应的基因及蛋白质序列见表1。
表1 代表性物种的基因信息
Tab.1 Genes of representative species
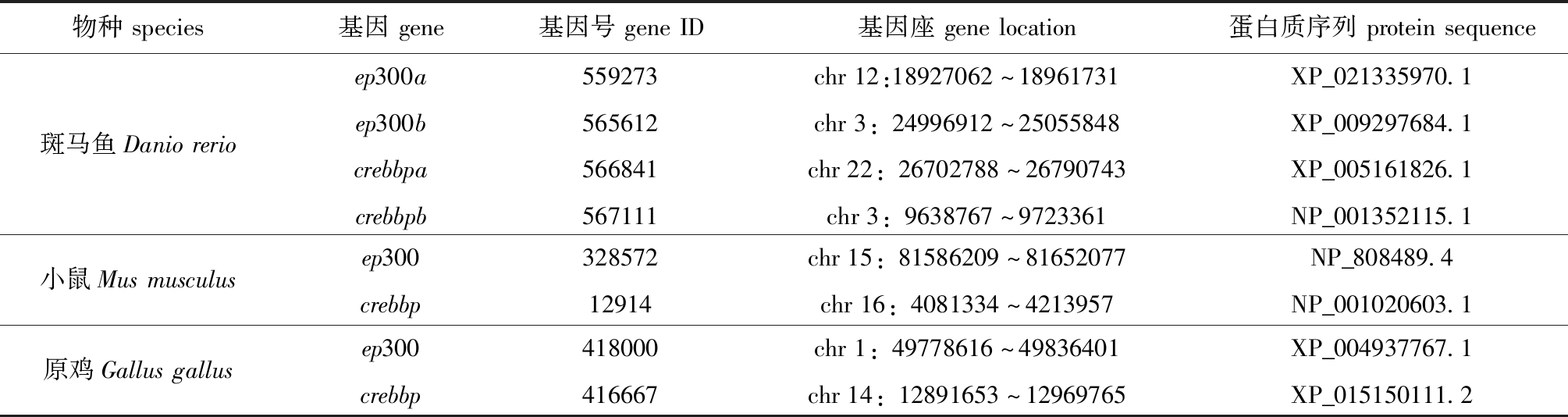
物种species基因gene基因号geneID基因座genelocation蛋白质序列proteinsequence斑马鱼Daniorerioep300a559273chr12:18927062~18961731XP_021335970.1ep300b565612chr3:24996912~25055848XP_009297684.1crebbpa566841chr22:26702788~26790743XP_005161826.1crebbpb567111chr3:9638767~9723361NP_001352115.1小鼠Musmusculusep300328572chr15:81586209~81652077NP_808489.4crebbp12914chr16:4081334~4213957NP_001020603.1原鸡Gallusgallusep300418000chr1:49778616~49836401XP_004937767.1crebbp416667chr14:12891653~12969765XP_015150111.2
1.2 方法
1.2.1 BLAST搜索及结果提取 使用表1中所列的4条斑马鱼蛋白质序列作为查询序列,分别参考虹鳟的非冗余蛋白质数据库(refseq_protein)和基因组数据库(refseq_genomes)进行在线BLASTP搜索和TBLASTN搜索,max target sequences均设置为5 000,e-value设置为1×10-5。编写python脚本提取比对结果,过滤掉覆盖度低于30%的Hits,同时从gene2 accession文件中(下载自https://ftp.ncbi.nlm.nih.gov/gene/DATA/)提取出BLASTP搜索结果中Hits所对应的基因在染色体上的位置信息。
1.2.2 系统发育分析 基于对BLASTP搜索结果的筛选,下载相应的虹鳟蛋白质序列,与斑马鱼、小鼠和原鸡的Ep300/Crebbp蛋白质序列合并,采用MAFFT[12]进行多重序列比对(L-INS-i模式)。采用Gblocks[13]对多重比对结果进行修剪,去除保守性较低的列(参数为-t=protein,-b2=10,-b3=20,-b4=2,-b5=All)。对修剪后的多重比对结果,采用RAxML 8.2.8[14]进行最大似然树的重构,采用GAMMA速率异质性模型,氨基酸替代模型自动选择,自展抽样500次。
1.2.3 基因表达及功能富集分析 使用NCBI的Run Selector(https://www.ncbi.nlm.nih.gov/Traces/ study/)对BioProject号PRJEB37848进行检索,下载该BioProject所包含的虹鳟及斑马鱼的原始转录组测序数据,使用Sratoolkit中的Fasterq-dump程序将压缩数据转换为fastq格式。从NCBI的FTP服务器(https://ftp.ncbi.nlm.nih.gov/genomes/refseq/vertebrate_other/)下载虹鳟和斑马鱼非冗余的转录本数据及最新版本的基因组拼接数据,采用Salmon[15]建立2个物种以基因组为诱饵的索引(SAF genome index)后,对转录本进行定量。用于衡量转录本组织表达水平的单位是TPM (transcripts per million),即每100万个reads里有多少个来自某个特定的转录本。定量完成后,根据gene2 accession文件将同一基因不同转录本的表达数据相加,得到虹鳟和斑马鱼不同基因在不同组织中的表达量。
使用Python语言下Scipy.stats模块[16]的pearsonr函数,计算ep300/crebbp家族不同成员间,以及它们与其所属物种其他基因间表达谱的皮尔逊相关系数。设置合理阈值(r>0.9或r>0.95,同时要求P<0.01)筛选各ep300/crebbp成员的共表达基因,虹鳟上的共表达基因需要先通过本地BLASTP搜索找到它们在斑马鱼上的直系同源基因,然后使用GOATOOLs[17]对转换后的共表达基因进行功能富集分析;斑马鱼上的共表达基因则可以直接进行功能富集分析。
1.2.4 保守结构域分析 基于上述基因表达分析结果,提取出各ep300/crebbp成员表达量最高的转录本所对应的蛋白质序列,使用CDD search[18-19]搜索序列中的保守结构域(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)。根据CDD search搜索结果确定每条序列上各保守结构域的区间,提取并保存各结构域的序列,采用MAFFT分别进行多重序列比对(L-INS-i模式),最后再将所有的多重比对文件按照结构域的顺序重新合并。对于PHD和KAT结构域的原始序列,同时采用I-TASSER 5.1[20]对它们逐个进行三维结构建模,最后采用PyMOL[21]对最佳模型进行可视化。
2 结果与分析
2.1 虹鳟ep300/crebbp家族的拷贝数
TBLASTN搜索结果显示,4条斑马鱼Ep300/Crebbp序列在虹鳟染色体序列上有8个较长的匹配区间,这些匹配区间的长度为18 620~64 649 nt,且对查询序列的覆盖度均在60%左右,可以初步认为这8个匹配区间是8个基因座(表2)。另一方面,BLASTP搜索得到了8条覆盖度非常高的蛋白质序列(Hits),且这些蛋白质序列所对应基因的座位刚好与TBLASTN搜索得到的8个匹配区间相符合(表3)。
表2 斑马鱼Ep300/Crebbp序列参考虹鳟基因组的TBLASTN搜索结果
Tab.2 TBLASTN search results of Ep300/Crebbp sequences of zebrafish Danio rerio against rainbow trout Oncorhynchus mykiss genome
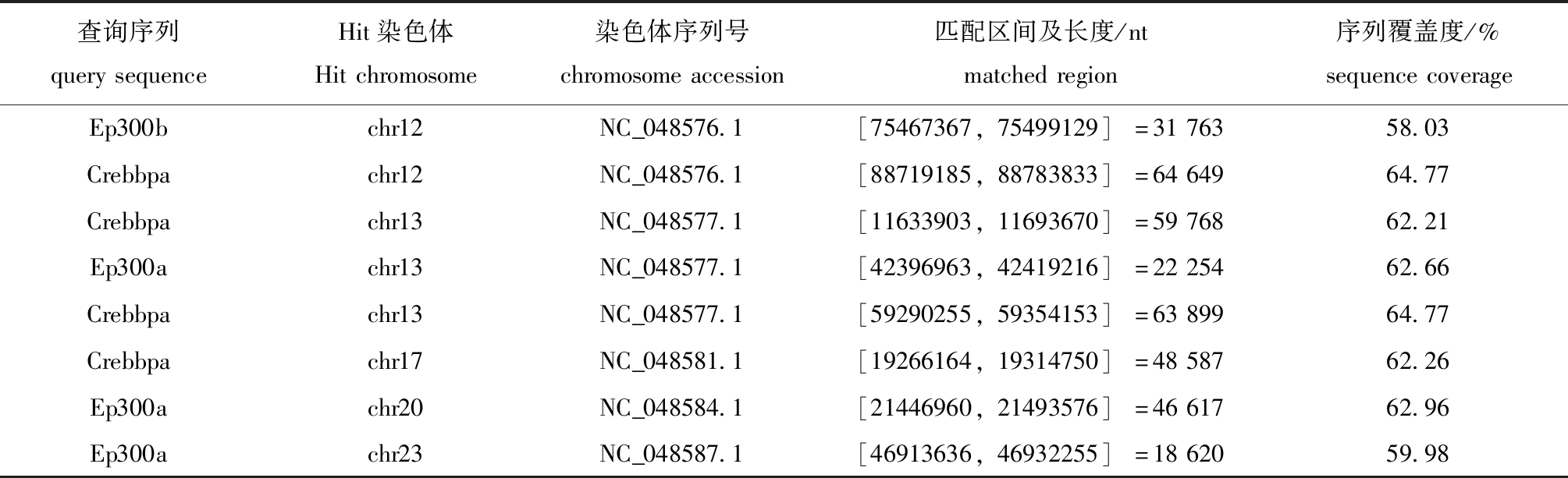
查询序列querysequenceHit染色体Hitchromosome染色体序列号chromosomeaccession匹配区间及长度/ntmatchedregion序列覆盖度/%sequencecoverageEp300bchr12NC_048576.1[75467367,75499129]=3176358.03Crebbpachr12NC_048576.1[88719185,88783833]=6464964.77Crebbpachr13NC_048577.1[11633903,11693670]=5976862.21Ep300achr13NC_048577.1[42396963,42419216]=2225462.66Crebbpachr13NC_048577.1[59290255,59354153]=6389964.77Crebbpachr17NC_048581.1[19266164,19314750]=4858762.26Ep300achr20NC_048584.1[21446960,21493576]=4661762.96Ep300achr23NC_048587.1[46913636,46932255]=1862059.98
注:查询序列与匹配区间的对应关系根据对序列覆盖度的筛选而来,下同。
Note:The correspondence between a query sequence and a matched region is screened by query coverage,et sequentia.
表3 斑马鱼Ep300/Crebbp序列参考虹鳟蛋白质数据库的BLASTP搜索结果
Tab.3 BLASTP search results of Ep300/Crebbp sequences of zebrafish Danio rerio against rainbow trout Oncorhynchus mykiss protein database
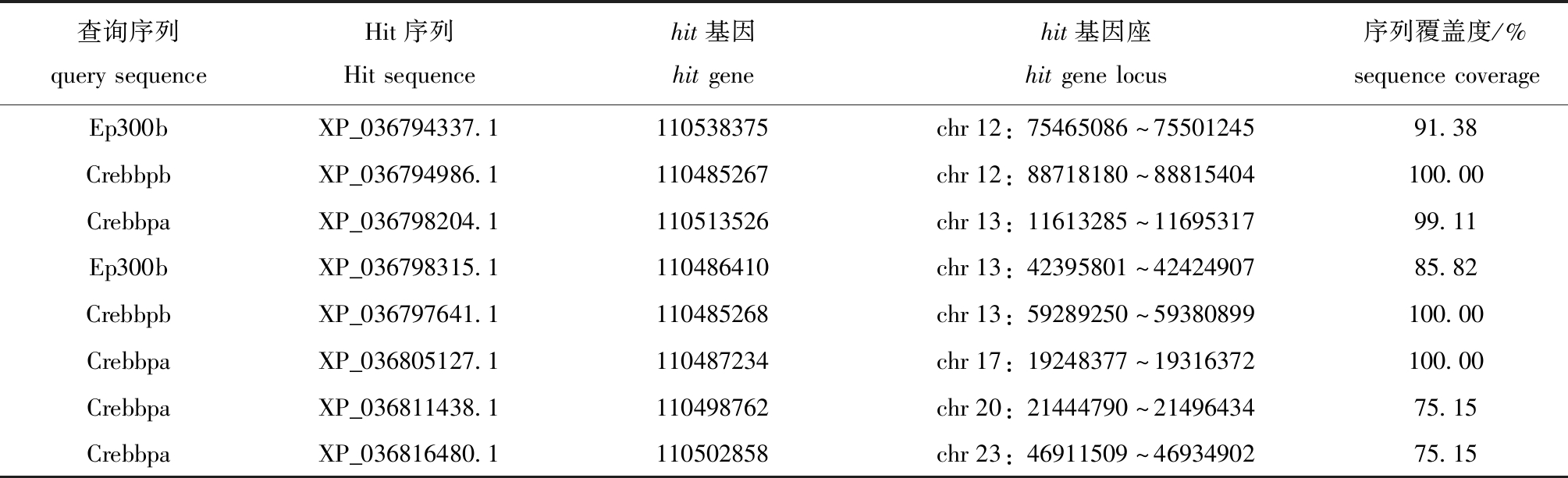
查询序列querysequenceHit序列Hitsequencehit基因hitgenehit基因座hitgenelocus序列覆盖度/%sequencecoverageEp300bXP_036794337.1110538375chr12:75465086~7550124591.38CrebbpbXP_036794986.1110485267chr12:88718180~88815404100.00CrebbpaXP_036798204.1110513526chr13:11613285~1169531799.11Ep300bXP_036798315.1110486410chr13:42395801~4242490785.82CrebbpbXP_036797641.1110485268chr13:59289250~59380899100.00CrebbpaXP_036805127.1110487234chr17:19248377~19316372100.00CrebbpaXP_036811438.1110498762chr20:21444790~2149643475.15CrebbpaXP_036816480.1110502858chr23:46911509~4693490275.15
通过BLAST搜索可以看出,虹鳟很可能存在8个ep300/crebbp基因。重构的最大似然树显示,虹鳟和斑马鱼的ep300/crebbp基因存在2∶1的关系(图1),表明虹鳟的8个ep300/crebbp基因均来自全基因组重复事件,而非小规模的基因重复。由全基因组重复产生的旁系同源基因被称作ohnologs[22],因此,这8个ep300/crebbp成员可以分成4对ohnologs,分别与斑马鱼的4个成员存在直系同源的关系。根据通行的命名规则,将这8个成员命名为ep300aa~crebbpbb(表4)。
表4 虹鳟8个ep300/crebbp成员的命名
Tab.4 Names of eight ep300/crebbp members of rainbow trout Oncorhynchus mykiss
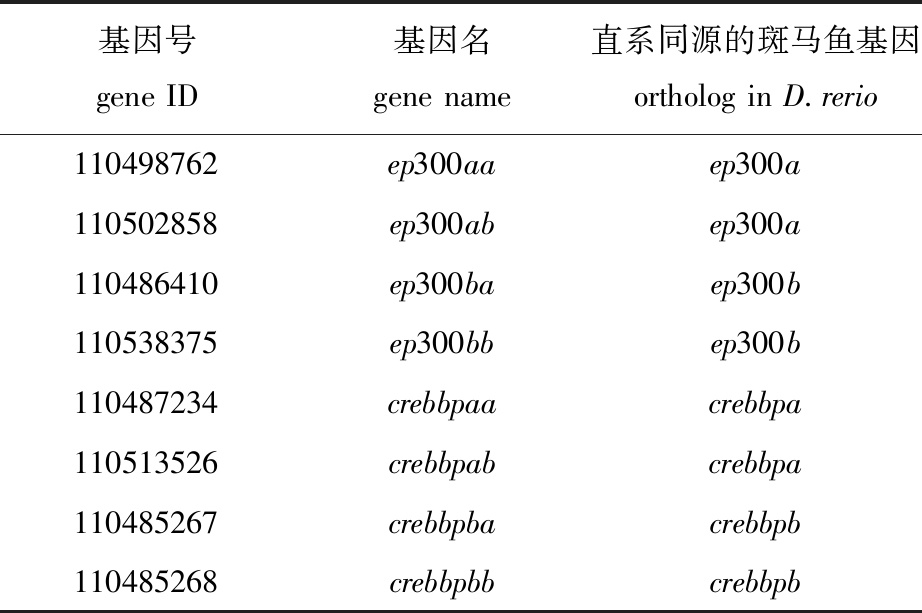
基因号geneID基因名genename直系同源的斑马鱼基因orthologinD.rerio110498762ep300aaep300a110502858ep300abep300a110486410ep300baep300b110538375ep300bbep300b110487234crebbpaacrebbpa110513526crebbpabcrebbpa110485267crebbpbacrebbpb110485268crebbpbbcrebbpb
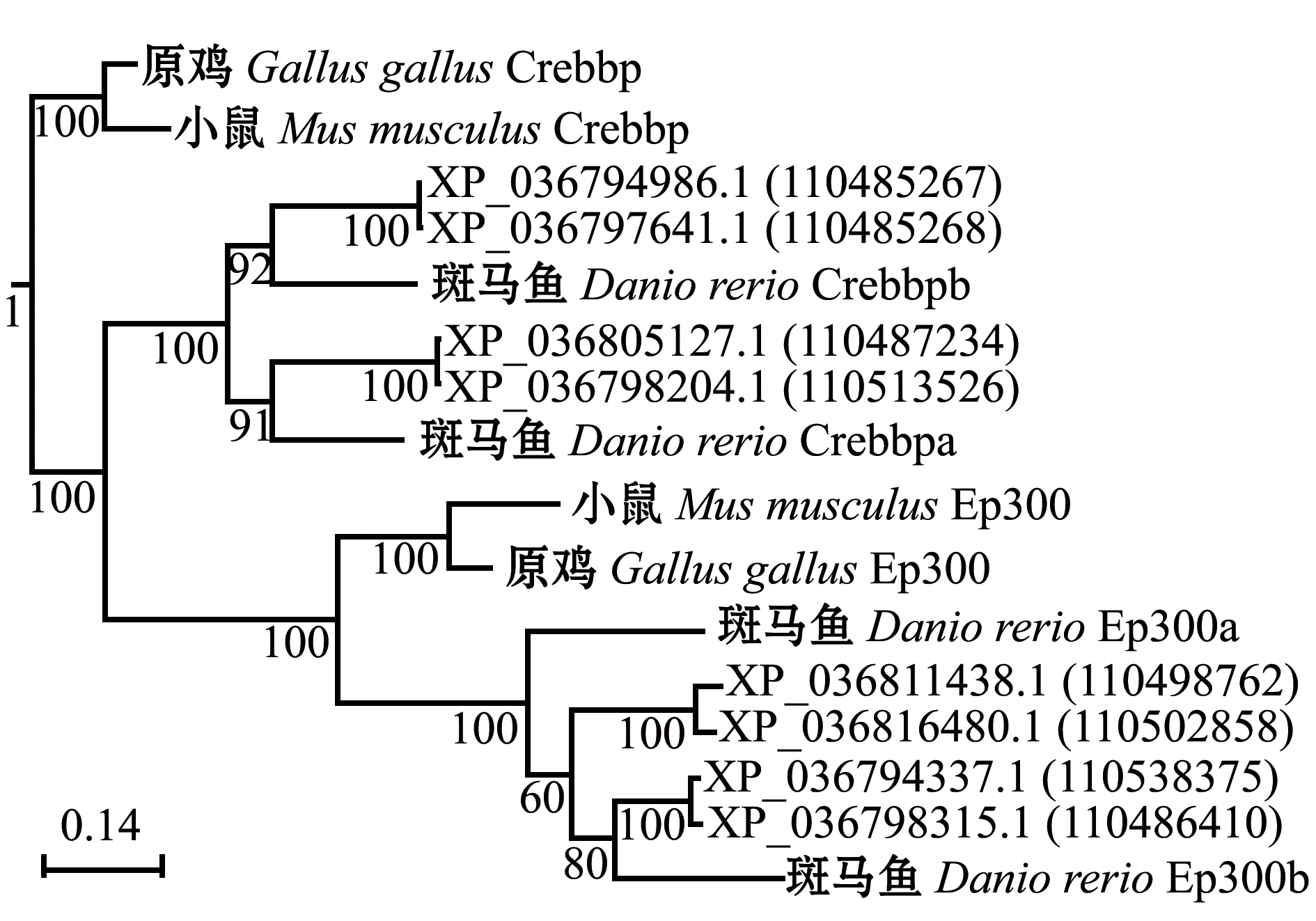
图1 虹鳟8条Ep300/Crebbp候选序列与相关序列的最大似然树
Fig.1 Maximum likelihood tree of eight candidate Ep300/Crebbp sequences of rainbow trout Oncorhynchus mykiss and related sequences
2.2 虹鳟ep300/crebbp家族不同成员的表达特征
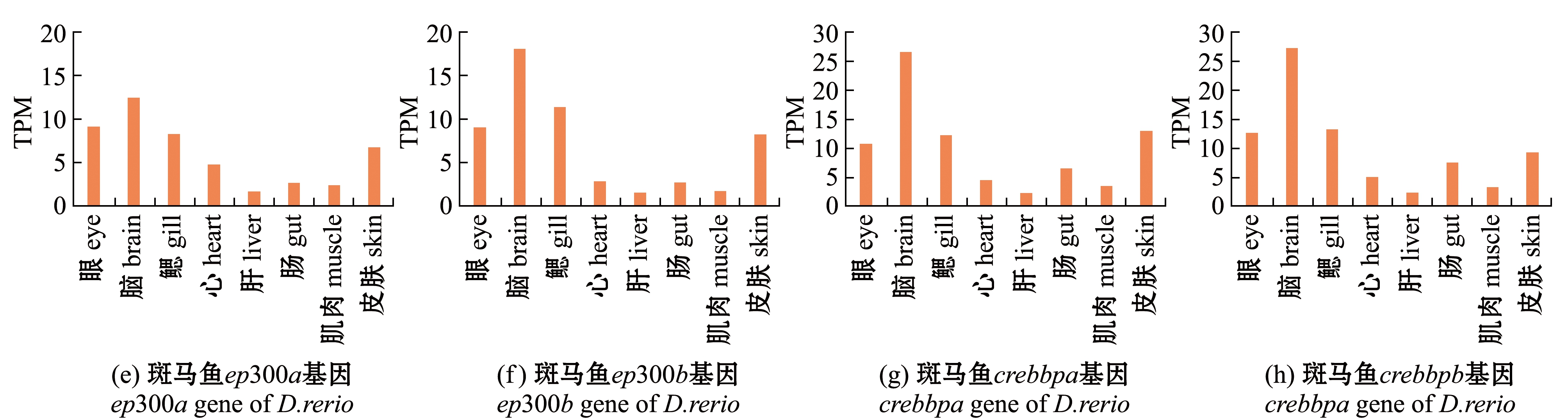
基于对PRJEB37848测序项目原始数据的统计,得到虹鳟8个ep300/crebbp成员及斑马鱼4个ep300/crebbp成员的组织表达水平(图2)。虹鳟的4对ohnologs与各自对应的斑马鱼直系同源基因的表达谱存在不同程度的分化,其中,ep300ba和ep300bb与斑马鱼ep300b的分化程度最低,无论是单个基因还是2个基因的平均值和表达谱的相似度均达到了显著或极显著水平(P<0.05或P<0.01)(图2(b));而crebbpba和crebbpbb与斑马鱼crebbpb的分化程度最高,无论是单个基因还是2个基因的平均值和表达谱的相似度均未达到显著水平(P>0.05)(图2(d))。斑马鱼的4个拷贝两两之间的表达谱相关系数均在0.9以上,而虹鳟的8个拷贝两两之间的表达谱相关系数仅有不到一半大于0.9(用与斑马鱼相同的8个组织的数据计算,相关系数大于0.9的则略多于一半),其中还包括了4对ohnologs内部相关系数大于0.9的情况(表5、表6)。综合分析,虹鳟的4对ohnologs之间功能分歧的程度要大于斑马鱼的4个拷贝,其中,crebbpba和crebbpbb与原始功能的分歧最大,而ep300ba和ep300bb则更接近原始的状态。
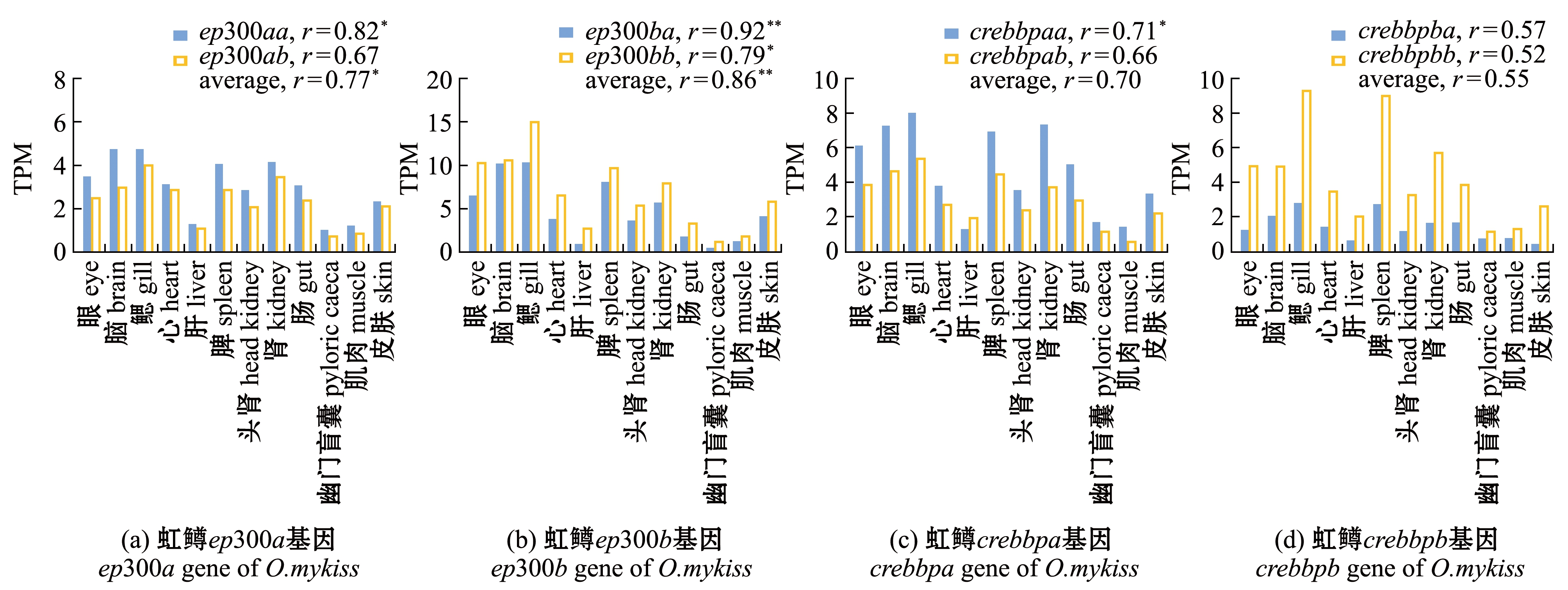
*表示虹鳟和斑马鱼直系同源基因表达谱的相关性达到了显著水平(P<0.05);** 表示相关性达到了极显著水平(P<0.01)。
* indicates significant correlation between the expression profiles of O.mykiss and D.rerio orthologous genes (P<0.05);** indicate very significant correlation(P<0.01).
图2 虹鳟和斑马鱼ep300/crebbp家族成员的组织表达谱
Fig.2 Tissue expression profile of ep300/crebbp members of rainbow trout Oncorhynchus mykiss and zebrafish Danio rerio
表5 虹鳟8个拷贝之间的组织表达谱相关系数
Tab.5 Correlation coefficient of tissue expression profile between eight copies of rainbow trout Oncorhynchus mykiss
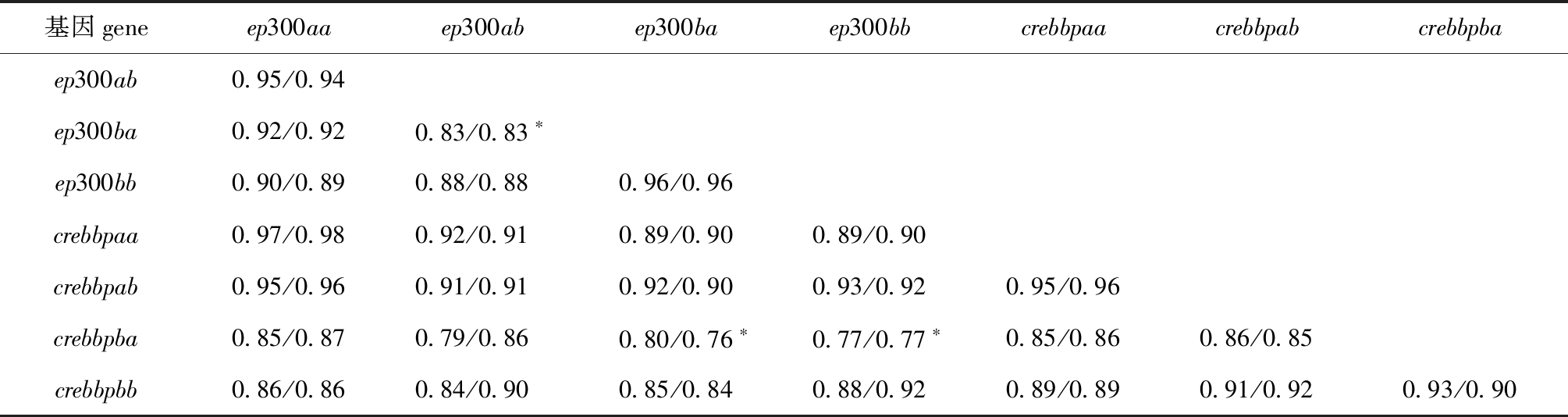
基因geneep300aaep300abep300baep300bbcrebbpaacrebbpabcrebbpbaep300ab0.95/0.94ep300ba0.92/0.920.83/0.83∗ep300bb0.90/0.890.88/0.880.96/0.96crebbpaa0.97/0.980.92/0.910.89/0.900.89/0.90crebbpab0.95/0.960.91/0.910.92/0.900.93/0.920.95/0.96crebbpba0.85/0.870.79/0.860.80/0.76∗0.77/0.77∗0.85/0.860.86/0.85crebbpbb0.86/0.860.84/0.900.85/0.840.88/0.920.89/0.890.91/0.920.93/0.90
注:每2个拷贝间均有2个相关系数,第一个用12个组织的数据计算得到,第二个用与斑马鱼相同的8个组织的数据计算得到。*表示对应的相关系数达到了显著性水平(P<0.05),其余均达到极显著水平(P<0.01)。
Note:There are two correlation coefficient values for each copy pair,with the first one calculated using expression data of 12 tissues and the second one calculated using expression data of 8 tissues (the same as D.rerio).* means the significant correlation coefficient value (P<0.05),and the rest all being very significant(P<0.01).
表6 斑马鱼4个拷贝间的组织表达谱相关系数
Tab.6 Correlation coefficient of tissue expression profile between four copies of zebrafish Danio rerio
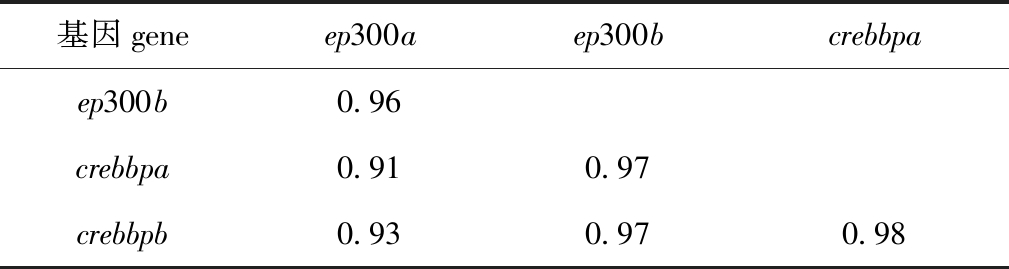
基因geneep300aep300bcrebbpaep300b0.96crebbpa0.910.97crebbpb0.930.970.98
注:所有相关系数均达到了极显著水平(P<0.01)。
Note:All correlation coefficient values are very significant(P<0.01).
2.3 虹鳟ep300/crebbp家族不同成员共表达基因的功能富集分析
通过设置合理的阈值发现,无论是虹鳟还是斑马鱼,其ep300/crebbp不同拷贝均有数量不等的特有共表达基因,即某个基因只与某一特定ep300/crebbp拷贝的表达谱相关系数大于阈值,与其他拷贝的表达谱相关系数则低于阈值(对虹鳟的基因,阈值设为0.9;对斑马鱼的基因,阈值设为0.95)。从表7可以看出,虹鳟crebbpab和crebbpba的共表达基因数量远少于其他拷贝,这些共表达基因所富集的GO条目也非常少,表明它们的功能很可能与其他拷贝发生了较大分化。除这2个拷贝外,其他的虹鳟拷贝和斑马鱼的全部拷贝普遍都能富集到数量较多与结合活性或蛋白质/染色体修饰(主要是乙酰化)相关的GO条目,这与Ep300/Crebbp蛋白的基本功能是相符合的,表明它们的核心功能尚未发生明显退化。无论是虹鳟还是斑马鱼的拷贝,它们的共表达基因能富集到与应对压力/刺激相关的GO条目总体上比较少,其中虹鳟的crebbpbb基因和斑马鱼的ep300a基因属于例外,它们所富集到的此类条目中,均有11个是与应对某些具体化学物质(如酮、甾类激素和皮质醇等)的刺激有关。与低氧应答有关的GO条目只有虹鳟ep300bb和斑马鱼ep300a的共表达基因能富集到。可见,在具体的信号通路里,不同ep300/crebbp拷贝所发挥的功能存在较大不同,表明它们的功能也发生了相应的分化。此外,从共表达基因的功能富集分析结果上来看,虹鳟8个ep300/crebbp拷贝间的功能分化程度要大于斑马鱼的4个拷贝。
表7 虹鳟和斑马鱼ep300/crebbp家族成员共表达基因的功能富集结果
Tab.7 GO enrichment analysis results of co-expressed genes of ep300/crebbp members of rainbow trout Oncorhynchus mykiss and zebrafish Danio rerio
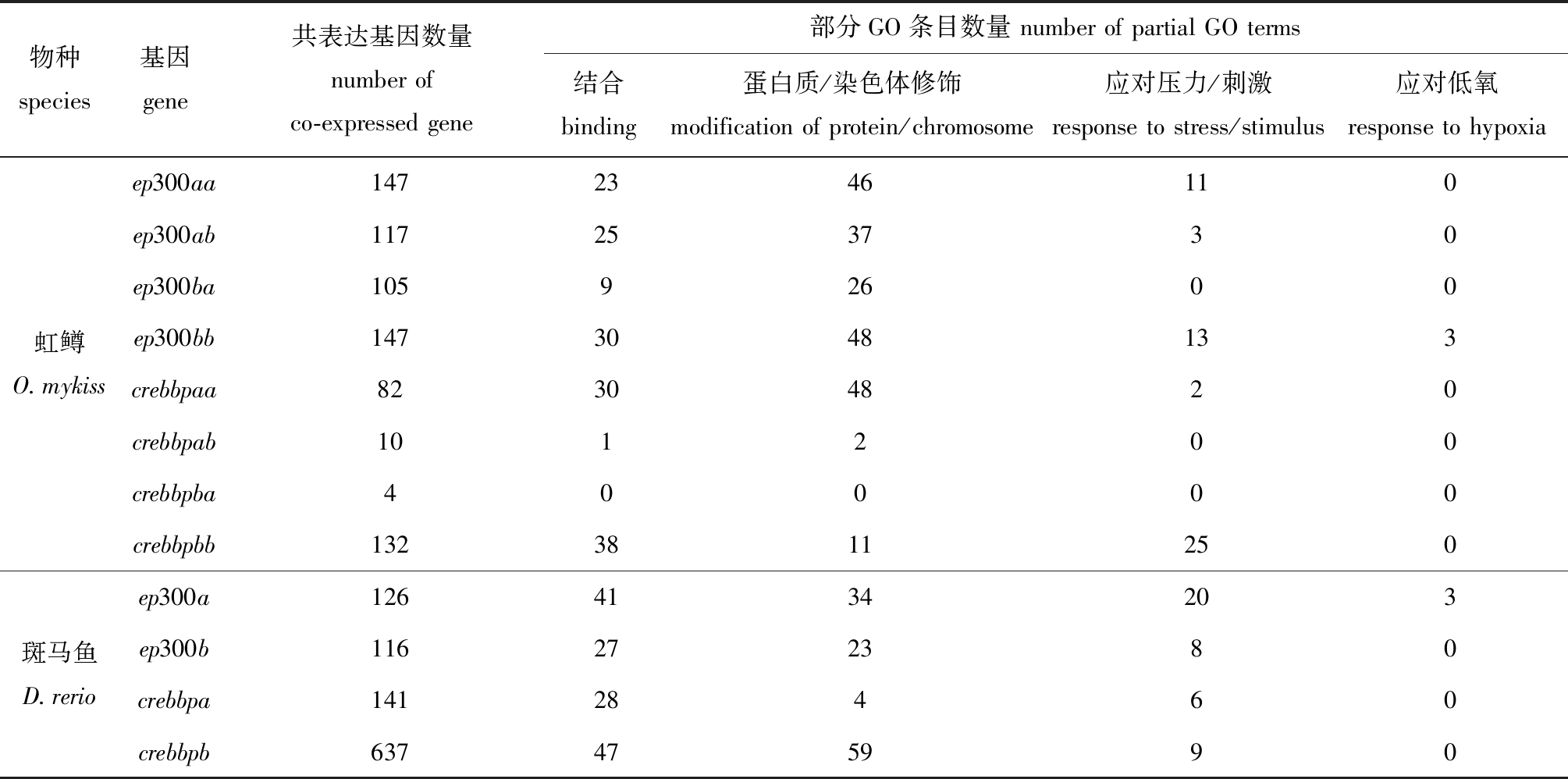
物种species基因gene共表达基因数量numberofco-expressedgene部分GO条目数量numberofpartialGOterms结合binding蛋白质/染色体修饰modificationofprotein/chromosome应对压力/刺激responsetostress/stimulus应对低氧responsetohypoxia虹鳟O.mykissep300aa1472346110ep300ab117253730ep300ba10592600ep300bb1473048133crebbpaa82304820crebbpab101200crebbpba40000crebbpbb1323811250斑马鱼D.rerioep300a1264134203ep300b116272380crebbpa14128460crebbpb637475990
注:斑马鱼共表达基因筛选的阈值是相关系数r>0.95。
Note:For D.rerio the threshold for selection of co-expressed genes is r>0.95.
2.4 虹鳟Ep300/Crebbp家族不同拷贝的保守结构域的序列及结构特征
为进一步探究虹鳟8个Ep300/Crebbp家族成员功能分化的基础,对8个基因各自表达量最高的转录本所对应的蛋白质序列进行CDD搜索,发现8个拷贝均存在该家族所特有的9个结构域(图3)。
将这9个结构域分别进行序列比对后发现,4对ohnologs内部的序列变异程度存在较大的差别:Crebbpaa与Crebbpab间,以及Crebbpba与Crebbpbb间,保守结构域完全一致;Ep300aa与Ep300ab间存在1处插入缺失突变和6处非同义突变,其中的6处非同义突变中,有2处导致了氨基酸残基侧链基团极性的改变;Ep300ba与Ep300bb的保守结构域有35处变异,其中的32处属于插入缺失突变,3处属于非同义突变,32处插入缺失突变分布于KIX、Bromo、RING、KAT及Creb_binding结构域,在前4个结构域是Ep300bb相对于Ep300ba的插入突变,而在Creb_binding结构域则是Ep300ba相对于Ep300bb的插入突变,3处非同义突变中,有2处改变了氨基酸残基侧链基团的极性(图4)。

结构域1~9依次为zf-TAZ、KIX、Bromo_cbp_like、RING_CBP-p300、PHD_p300(或PHD_SF super family)、HAT_KAT11(或HAT_KAT11 super family)、ZZ_CBP、ZnF_TAZ和Creb_binding。
Domains 1-9 are zf-TAZ,KIX,Bromo_cbp_like,RING_CBP-p300,PHD_p300(or PHD_SF super family),HAT_KAT11(or HAT_KAT11 super family),ZZ_CBP,ZnF_TAZ and Creb_binding,respectively.
图3 虹鳟8个Ep300/Crebbp成员的CDD搜索结果
Fig.3 CDD search results of eight Ep300/Crebbp members of rainbow trout Oncorhynchus mykiss
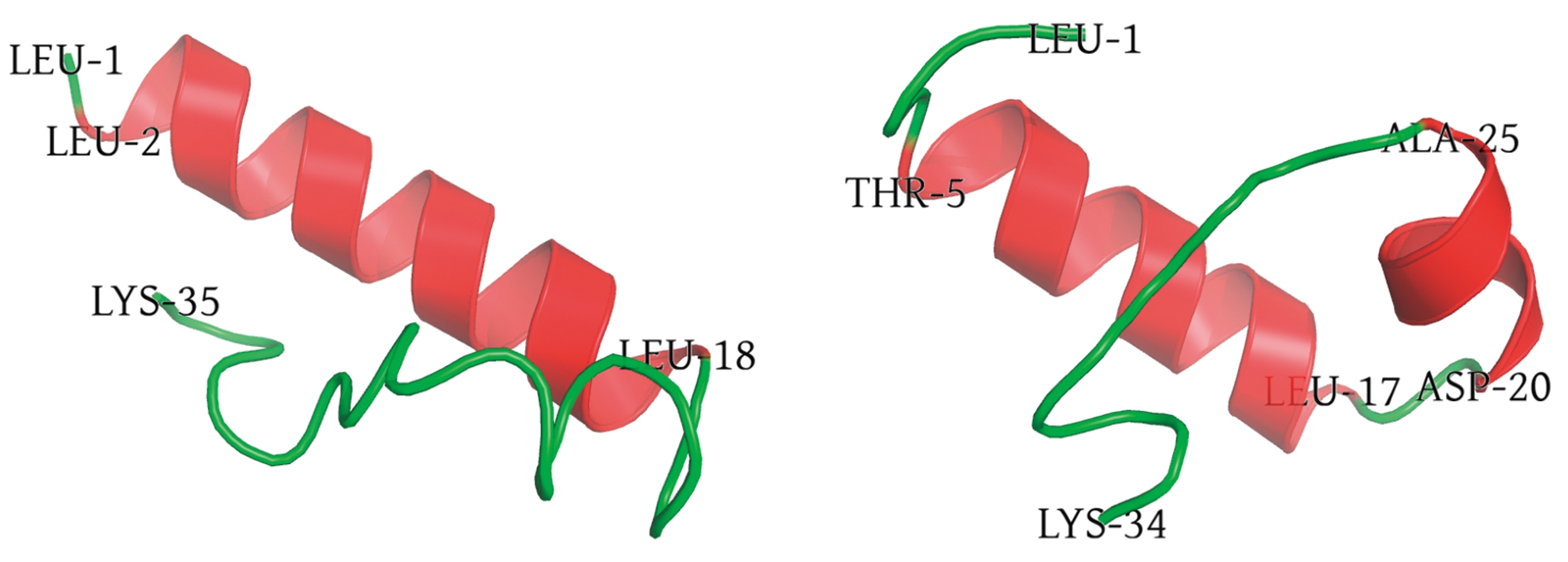
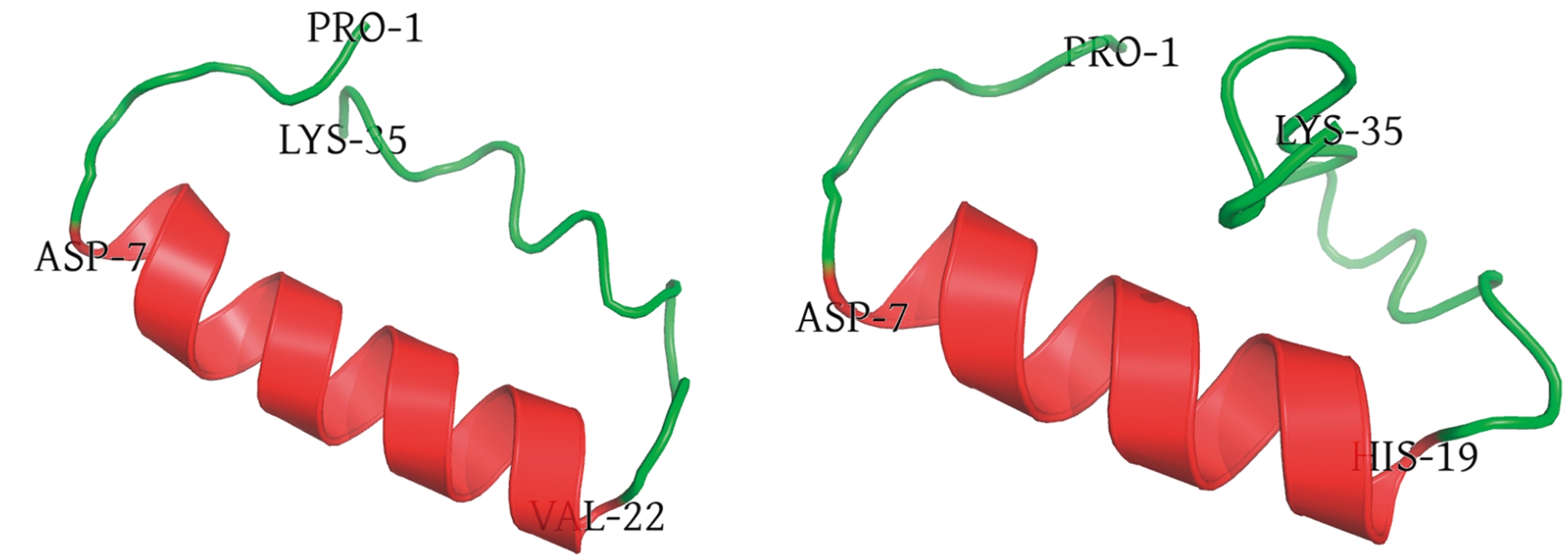
从图3可见,8条序列的PHD和KAT结构域存在较大区别,有4条序列的PHD结构域特征不够明显,4条序列的KAT结构域特征不够明显。使用I-TASSER对这2个结构域进行结构建模,发现KAT结构域过于复杂且高相似度的模板较少,导致即使是最佳模型也接近于随机水平(TM-score通常在0.2左右);相比之下,PHD结构域建模最佳模型的TM-score均在0.44以上,结果较为可信。由于这8条序列中同一对ohnolog内部的PHD结构域序列均完全一致,因此,它们的最佳模型结构也完全一致。
从图5可以看出,PHD结构域的主体是一个由十几个氨基酸形成的螺旋。虽然CDD search结果显示,4条Ep300序列的PHD结构域特征不够明显,但Ep300aa和Ep300ab的螺旋最长,而Ep300ba和Ep300bb除了有一条较长的螺旋外,还有一个较短的螺旋。

天蓝色、红色、黄色和蓝绿色分别代表该氨基酸残基是非极性、中性、碱性和酸性氨基酸。
Sky blue,red,yellow and cyan colors indicate nonpolar,neutral,basic and acidic amino acid residue,respectively.
图4 虹鳟8个Ep300/Crebbp成员保守结构域的序列比对
Fig.4 Sequence alignment of conserved domains of eight Ep300/Crebbp members of rainbow trout Oncorhynchus mykiss
(a)Ep300aa and Ep300ab (b)Ep300ba and Ep300bb (c)Crebbpaa and Crebbpab (d)Crebbpba and Crebbpbb
图5 虹鳟8个Ep300/Crebbp成员PHD结构域的最佳模型
Fig.5 Best models of PHD domain of eight Ep300/Crebbp members of rainbow trout Oncorhynchus mykiss
3 讨论
3.1 ep300/crebbp家族成员的扩张
基因家族的扩张可通过2种形式来实现,即全基因组重复和小规模重复(small-scale duplications,SSDs)。那些在细胞调控网络中处于较为核心位置,编码的蛋白质与大量其他蛋白质存在互作的基因,往往倾向于通过全基因组重复事件增加拷贝数;而那些在调控网络中处于较为边缘位置,即使缺失也不易产生较大危害的基因,则倾向于通过小规模重复来增加拷贝数[11]。ep300/crebbp所编码的蛋白质能够对细胞内不同蛋白质的上千个位点进行乙酰化[23],实现对基因表达及蛋白质的细胞内定位、酶活性等功能的调控。显然,它们的家族扩张更有可能是通过全基因组重复事件实现的。事实上,脊椎动物中普遍存在的ep300和crebbp这2个家族成员本身就是由4.5亿年前的全基因组重复事件所产生[24-25]。笔者在前期研究中发现,不同的真骨鱼类群普遍存在2个ep300基因拷贝,而那些经历过额外全基因组重复事件的物种则存在4个拷贝,这显然也是全基因组重复事件后多拷贝得到保留的结果。本研究中发现,虹鳟的ep300/crebbp家族有8个拷贝,其中,ep300和crebbp各有4个,与前期研究结果及虹鳟祖先物种所经历的全基因组重复事件次数相符合,这表明历次全基因组重复事件所产生的多拷贝被完全保留了下来。
3.2 虹鳟ep300/crebbp家族不同成员的功能分化
全基因组重复事件后能够在较长时期内保留2个拷贝的基因,最初可能只是为了维持剂量平衡;2个拷贝的进化速率通常情况下并不一致,即使它们受到了强大的净化选择压力,随着时间的推移,2个拷贝仍会以新功能化或亚功能化的形式发生分化[11,26]。与同样经历过额外全基因组重复事件的鲤和鲫相比[27],虹鳟对恶劣环境的适应能力要低得多,鉴于ep300/crebbp家族在生物应对环境变化时的重要作用,有理由怀疑虹鳟现有8个成员的功能很可能已发生了较大的分歧,进而导致剂量效应被大大弱化。本研究中,这8个成员表达谱的分离,以及它们的共表达基因所富集GO条目的分离,都能够证明功能分歧的存在。虹鳟crebbpaa与crebbpab,以及crebbpba与crebbpbb间共表达基因数量和富集GO条目的巨大差异,进一步证明这种功能的分歧不仅存在于不同的ohnologs间,同样也存在于ohnologs内部。对保守结构域序列的分析表明,ohnologs内部的变异总体而言是比较小的,大量变异发生在不同的ohnologs间,表明这些成员间的功能分歧在最近一次的全基因组重复事件之前就已经存在[28-29]。本研究中,虹鳟的Crebbpaa与Crebbpab,以及Crebbpba与Crebbpbb间的保守结构域序列完全相同,这表明由共表达基因功能富集所推测出的功能分歧并不是由于这两对ohnologs内部所编码蛋白质的结构发生了显著变化。推测一种可能的原因是这两对ohnologs内部某条序列在非保守结构域的序列变异导致了一定程度的结构创新,从而形成了新功能化。笔者对斑马鱼Ep300蛋白结构的研究就表明,TAZ1结构域侧翼的低复杂度区域有可能存在较短的螺旋结构[9]。由于结构域间的连接序列也可以结合转录因子[30],那些新进化出的螺旋结构很有可能会促进这种结合,最终导致该基因产生新的功能。
3.3 ep300bb在低氧应答中的作用
虹鳟的8个ep300/crebbp家族成员中,只有ep300bb的共表达基因富集到了应对低氧胁迫的GO条目(1/8);相比之下,斑马鱼的4个成员中则是ep300a的共表达基因能富集到低氧胁迫相关的GO条目(1/4)。拷贝数增加反而降低了虹鳟ep300/crebbp家族应对低氧胁迫的效率,这或许可以部分解释为什么虹鳟对溶氧有更高的要求。另一方面,由于ep300bb基因在绝大多数组织中的表达水平均高于另外7个拷贝,即使其他拷贝也在低氧应答中发挥一定作用,它们的重要性也不如前者。虹鳟的ep300bb基因和斑马鱼的ep300a基因是两个物种中与应对低氧胁迫关系最密切的ep300/crebbp成员,但它们并不是直系同源关系,这与笔者前期研究发现的不同真骨鱼类群对ep300拷贝的选择偏好[9]是一致的。Ep300ba和Ep300bb在PHD结构域中所存在的额外较短螺旋很可能与功能创新相关[31],而这2个拷贝在其他保守结构域中甚至存在一级结构上的较大变异,这表明它们拥有非常快的进化速度,且Ep300bb进化的方向很可能有利于提高虹鳟的耐低氧性能。
4 结论
1)虹鳟的ep300/crebbp家族有8个成员,参考斑马鱼的4个ep300/crebbp成员,这8个成员可被分为4对ohnologs,说明全基因组重复事件为虹鳟ep300/crebbp基因家族的分歧进化提供了丰富的遗传材料。
2)不同的ohnologs对之间在组织表达谱、编码蛋白质的一级结构和三级结构上存在较大差异,而同一对ohnologs内部的两个拷贝在这些方面总体上差异较小,可以推测它们的功能也发生了不同程度的分化。
3)ep300bb基因进化速度较快,且与虹鳟耐低氧性能相关性更大,可作为虹鳟遗传改良的重要候选基因。
[1] 柳鹏,高春山,杜晓燕,等.中国东北山区虹鳟三倍体幼鱼生长特性研究[J].大连海洋大学学报,2020,35(2):213-217.
LIU P,GAO C S,DU X Y,et al.Growth performance of juvenile triploid rainbow trout Oncorhynchus mykiss in mountain area of northeast China[J].Journal of Dalian Ocean University,2020,35(2):213-217.(in Chinese)
[2] STILLER K T,VANSELOW K H,MORAN D M,et al.The effect of diet,temperature and intermittent low oxygen on the metabolism of rainbow trout[J].British Journal of Nutrition,2017,117(6):784-795.
[3] WELKER T L,OVERTURF K,ABERNATHY J.Effect of aeration and oxygenation on growth and survival of rainbow trout in a commercial serial-pass,flow-through raceway system[J].Aquaculture Reports,2019,14:100194.
[4] 于振海,丁建华,靖莹,等.山西冷水性鱼类养殖发展现状调研[J].湖南农业科学,2011(21):112-115.
YU Z H,DING J H,JING Y,et al.Investigation of developmental status of cold water fishes aquaculture in Shanxi Province[J].Hunan Agricultural Sciences,2011(21):112-115.(in Chinese)
[5] SHEIKH B N,AKHTAR A.The many lives of KATs—detectors,integrators and modulators of the cellular environment[J].Nature Reviews Genetics,2019,20(1):7-23.
[6] MASOUD G N,LI W.HIF-1α pathway:role,regulation and intervention for cancer therapy[J].Acta Pharmaceutica Sinica B,2015,5(5):378-389.
[7] ZIMNA A,KURPISZ M.Hypoxia-inducible factor-1 in physiological and pathophysiological angiogenesis:applications and therapies[J].Biomed Research International,2015.DOI:10.1155/2015/549412.
[8] PASQUIER J,CABAU C,NGUYEN T,et al.Gene evolution and gene expression after whole genome duplication in fish:the PhyloFish database[J].BMC Genomics,2016,17:368.
[9] WANG X Z,YAN J L.Directional divergence of Ep300 duplicates in teleosts and its implications[J].BMC Evolutionary Biology,2020,20(1):140.
[10] BERTHELOT C,BRUNET F,CHALOPIN D,et al.The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates[J].Nature Communications,2014,5:3657.
[11] CONANT G C,WOLFE K H.Turning a hobby into a job:how duplicated genes find new functions[J].Nature Reviews Genetics,2008,9(12):938-950.
[12] KATOH K,STANDLEY D M.MAFFT multiple sequence alignment software version 7:improvements in performance and usability[J].Molecular Biology and Evolution,2013,30(4):772-780.
[13] CASTRESANA J.Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis[J].Molecular Biology and Evolution,2000,17(4):540-552.
[14] STAMATAKIS A.RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J].Bioinformatics (Oxford,England),2014,30(9):1312-1313.
[15] PATRO R,DUGGAL G,LOVE M I,et al.Salmon provides fast and bias-aware quantification of transcript expression[J].Nature Methods,2017,14(4):417-419.
[16] VIRTANEN P,GOMMERS R,OLIPHANT T E,et al.SciPy 1.0:fundamental algorithms for scientific computing in Python[J].Nature Methods,2020,17(3):261-272.
[17] KLOPFENSTEIN D V,ZHANG L S,PEDERSEN B S,et al.GOATOOLS:a Python library for gene ontology analyses[J].Scientific Reports,2018,8:10872.
[18] LU S N,WANG J Y,CHITSAZ F,et al.CDD/SPARCLE:the conserved domain database in 2020[J].Nucleic Acids Research,2019,48(D1):D265-D268.
[19] MARCHLER-BAUER A,DERBYSHIRE M K,GONZALES N R,et al.CDD:NCBI’s conserved domain database[J].Nucleic Acids Research,2015,43(Database issue):D222-D226.
[20] YANG J Y,YAN R X,ROY A,et al.The I-TASSER suite:protein structure and function prediction[J].Nature Methods,2015,12(1):7-8.
[21] JANSON G,PAIARDINI A.PyMod 3:a complete suite for structural bioinformatics in PyMOL[J].Bioinformatics,2021,37(10):1471-1472.
[22] SINGH P P,ISAMBERT H.Ohnologs v2:a comprehensive resource for the genes retained from whole genome duplication in vertebrates[J].Nucleic Acids Research,2019,48(D1):D724-D730.
[23] WEINERT B T,NARITA T,SATPATHY S,et al.Time-resolved analysis reveals rapid dynamics and broad scope of the CBP/p300 acetylome[J].Cell,2018,174(1):231-244.e12.
[24] BREEN M E,MAPP A K.Modulating the masters:chemical tools to dissect CBP and p300 function[J].Current Opinion in Chemical Biology,2018,45:195-203.
[25] HARA Y,YAMAGUCHI K,ONIMARU K,et al.Shark genomes provide insights into elasmobranch evolution and the origin of vertebrates[J].Nature Ecology &Evolution,2018,2(11):1761-1771.
[26] BRUNET F G,CROLLIUS H R,PARIS M,et al.Gene loss and evolutionary rates following whole-genome duplication in teleost fishes[J].Molecular Biology and Evolution,2006,23(9):1808-1816.
[27] CHEN Z L,OMORI Y,KOREN S,et al.De novo assembly of the goldfish (Carassius auratus) genome and the evolution of genes after whole-genome duplication[J].Science Advances,2019,5(6):eaav0547.
[28] FAUQUIER L,AZZAG K,PARRA M A M,et al.CBP and p300 regulate distinct gene networks required for human primary myoblast differentiation and muscle integrity[J].Scientific Reports,2018,8:12629.
[29] LIPINSKI M,DEL BLANCO B,BARCO A.CBP/p300 in brain development and plasticity:disentangling the KAT’s cradle[J].Current Opinion in Neurobiology,2019,59:1-8.
[30] CONTRERAS-MARTOS S,PIAI A,KOSOL S,et al.Linking functions:an additional role for an intrinsically disordered linker domain in the transcriptional coactivator CBP[J].Scientific Reports,2017,7:4676.
[31] MENDIRATTA S S,SEKULIC N,HERNANDEZ-GUZMAN F G,et al.A novel alpha-helix in the first fibronectin type Ⅲ repeat of the neural cell adhesion molecule is critical for N-glycan polysialylation[J].Journal of Biological Chemistry,2006,281(47):36052-36059.