纳木错裸鲤Gymnocypris namensis隶属于鲤形目Cypriniformes鲤科Cyprinidae裂腹鱼亚科Schizothoracinae纳木错裸鲤属Gymnocypris,仅分布于中国海拔4 718 m的西藏纳木错湖及入湖河流各支流中,是纳木错湖内唯一的经济鱼类[1],被称为中国海拔最高的高原裂腹鱼,其对高原恶劣环境的适应能力较强。在中国脊椎动物红色名录中纳木错裸鲤被列为近危(NT)物种[2]。
西藏土著鱼类主要由3大类群组成,包括鲤形目鲤科裂腹鱼亚科、鳅科条鳅亚科和鲶形目鮡科[3]。据统计,世界上裂腹鱼类有12属100余种,但部分种类存在同物异名的情况,中国约产11属76种[4],主要分布在青藏高原及其周边水域。西藏裂腹鱼亚科鱼类存在染色体多态性这一现象[5],不仅表现在染色体数目多样性,而且还表现在染色体倍性多样性[6-9]。已有学者发现,尖裸鲤Oxygyrnnocypris stewartii存在同种不同个体间的染色体差异,拉萨裂腹鱼Schizothorax waltoni和异齿裂腹鱼Shizothorax oconnori也存在不同地区同种染色体的差异情况[5]。此外,已报道核型的22种裂腹鱼亚科鱼类大多数为多倍体类型,余祥勇等[10]推测,多倍体是高原裂腹鱼亚科核型进化最主要的特征,裂腹鱼亚科鱼类可能具有共同的祖先。由此推测,高原裂腹鱼亚科鱼类为适应高原恶劣多变的地理环境,每个种或种间染色体数目及倍性发生了较大变化。因此,裂腹鱼亚科鱼类是研究多倍体鱼类和高原适应性机制的重要材料。西藏裂腹鱼亚科鱼类染色体的多态性,更有利于其适应西藏地区恶劣多变的高原环境[11],并成为青藏高原水域的优势鱼种。
裂腹鱼亚科鱼类已经确定核型的种类不足全部种类的1/3[12]。有关纳木错裸鲤染色体数目、核型及带型等方面的研究尚未见报道。本研究中,采用吉姆萨(Giemsa)、银染(Ag-NORs)和CMA3/DAPI双重荧光染色方法对纳木错裸鲤的染色体数目、核型和带型等进行分析,以确定纳木错裸鲤的染色体数目、核型和带型特点,并对其倍性进行分析,以补充纳木错裸鲤细胞遗传学方面研究的空白,并为后续开展裂腹鱼亚科鱼类分类和进化地位等相关研究提供科学参考。
1 材料与方法
1.1 材料
试验用健康纳木错裸鲤雄鱼3尾,取自中国西藏自治区纳木错湖,体长为31.1~36.1 cm,体质量为276.1~682.5 g(表1),空运至大连海洋大学水产与生命学院实验室水族箱中暂养,暂养温度为(12±1)℃。
表1 纳木错裸鲤样本规格
Tab.1 Sample size of Gymnocypris namensis
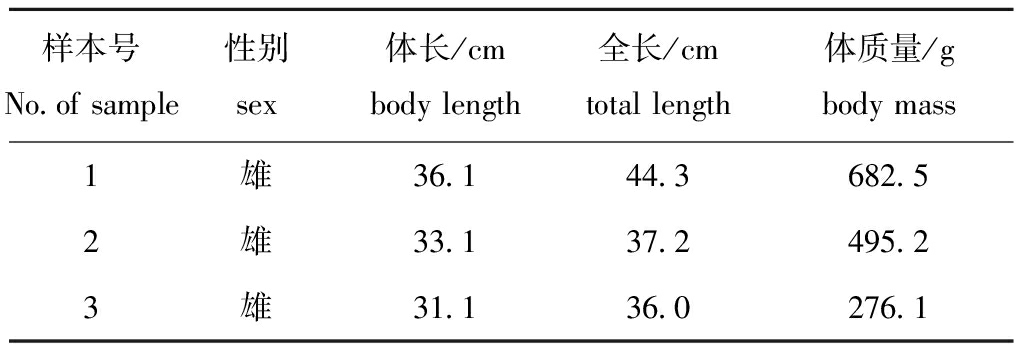
样本号No.of sample性别sex体长/cmbody length全长/cmtotal length体质量/gbody mass1雄 36.144.3682.52雄 33.137.2495.23雄 31.136.0276.1
1.2 方法
1.2.1 肾细胞染色体标本的制备 采用两次植物血球凝集素(PHA)[13]及一次秋水仙素腹腔注射进行样品处理,注射剂量均为6 μg/g(鱼体质量),PHA两次作用时间分别为18~20 h和4~6 h,秋水仙素作用时间为2~3 h。取出鱼体完整肾脏,置于浓度为0.075 mol/L KCl低渗液中低渗45 min,用卡诺固定液(甲醇与冰醋酸的体积比为3∶1)固定3次,每次15 min,于-20 ℃冰箱中冷冻过夜。采用常规冷滴片法进行染色体标本制备,风干后用质量分数为10%的吉姆萨(Giemsa)染色液处理45 min,在普通光学显微镜(Leica DM2000)下镜检并拍照。
1.2.2 染色体数目统计及核型分析 从3尾纳木错裸鲤染色体中期分裂相中各选取30个分散良好、形态清晰完整的分裂相进行计数统计,以确定染色体数目。每尾鱼分别选取5个染色体数目完整、形态清晰的分裂相,参照Levan等[14]提出的标准进行核型分析。
1.2.3 银染(Ag-NORs) 采用Howell等[15]的快速银染法。将纳木错裸鲤肾细胞染色体载玻片置于已预热至65 ℃的加热器平面上,取100 μL质量分数为50%的硝酸银溶液与50 μL质量分数为2%的明胶溶液混匀后,均匀滴到染色体载玻片上,盖上盖玻片及遮光盒,黑暗条件下处理1~2 min,至染色体被染成金黄色,用超纯水(65 ℃)冲洗载玻片后,风干、封片,在普通光学显微镜(Leica DM2000)下镜检并拍照。
1.2.4 CMA3/DAPI双重荧光染色 参照Schweizer等[16-17]和Amoyes等[18]的方法并稍加修改。将纳木错裸鲤肾细胞染色体用0.5 mg/mL CMA3染色60 min,再用0.1 mg/mL DAPI染色15 min。每次染色后均用MI缓冲液(pH 7.0)漂洗2 次,用甘油和MI缓冲液(pH 7.0)的混合液(二者体积比为1∶1)处理后封片,于4 ℃冰箱保存并水平放置,一周内在荧光显微镜(Leica DM2000)下观察拍照。
2 结果与分析
2.1 肾细胞的染色体数目及核型分析
从3尾纳木错裸鲤肾细胞中期分裂相中各选取30个形态清晰完整、分散良好的分裂相进行统计。从表2可见:纳木错裸鲤染色体数目为84~94条,1号个体中92条染色体中期分裂相占比70%,2号个体中92条染色体中期分裂相占比73%,3号个体中92条染色体中期分裂相占比67%;3尾鱼合计有90个染色体中期分裂相,染色体数目为92条的分裂相占比70%。因此,纳木错裸鲤染色体数目为92条,且3个个体间染色体众数一致。
表2 纳木错裸鲤不同个体染色体数目统计
Tab.2 Chromosome number of different individuals of Gymnocypris namensis
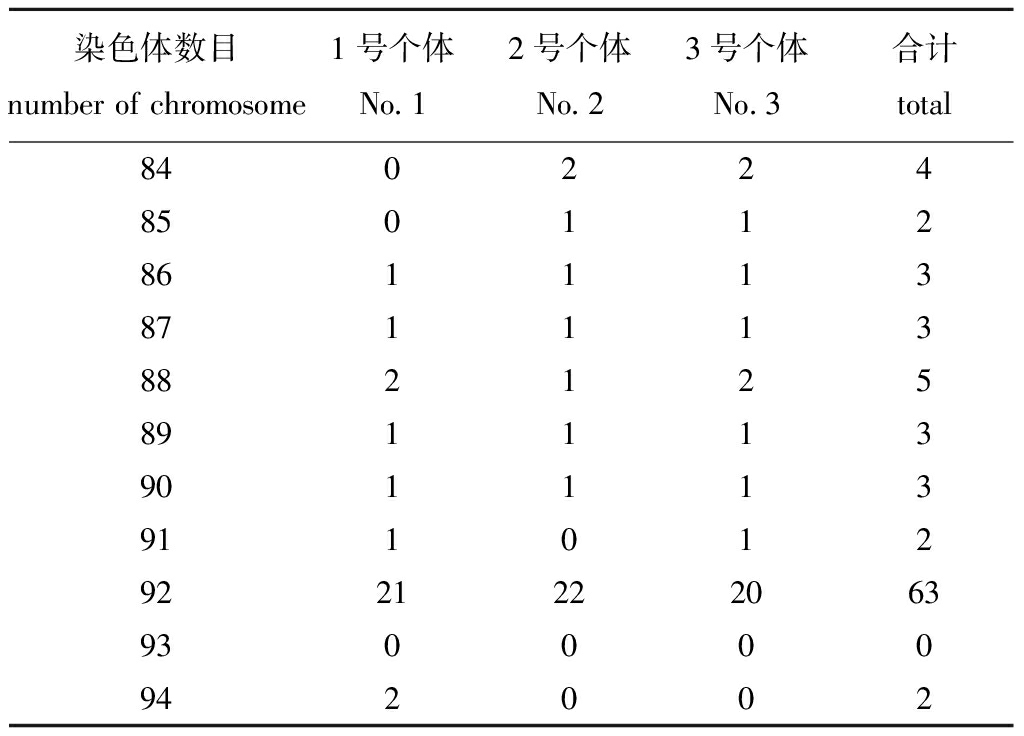
染色体数目number of chromosome1号个体No.12号个体No.23号个体No.3合计total8402248501128611138711138821258911139011139110129221222063930000942002
从3尾纳木错裸鲤中选取染色体数目完整、形态良好的5个中期分裂相,测量每条染色体的臂长(长臂及短臂),计算染色体相对长度及臂比,结果如表3所示。根据Levan等[14]提出的标准,确定染色体类型并进行配对。结果显示,中部着丝点染色体(M)共计40条,亚中部着丝点(SM)染色体共计20条,端部着丝点染色体(T)共计32条,其臂数为152。纳木错裸鲤核型公式为40m+20sm+32t,NF=152(图1)。
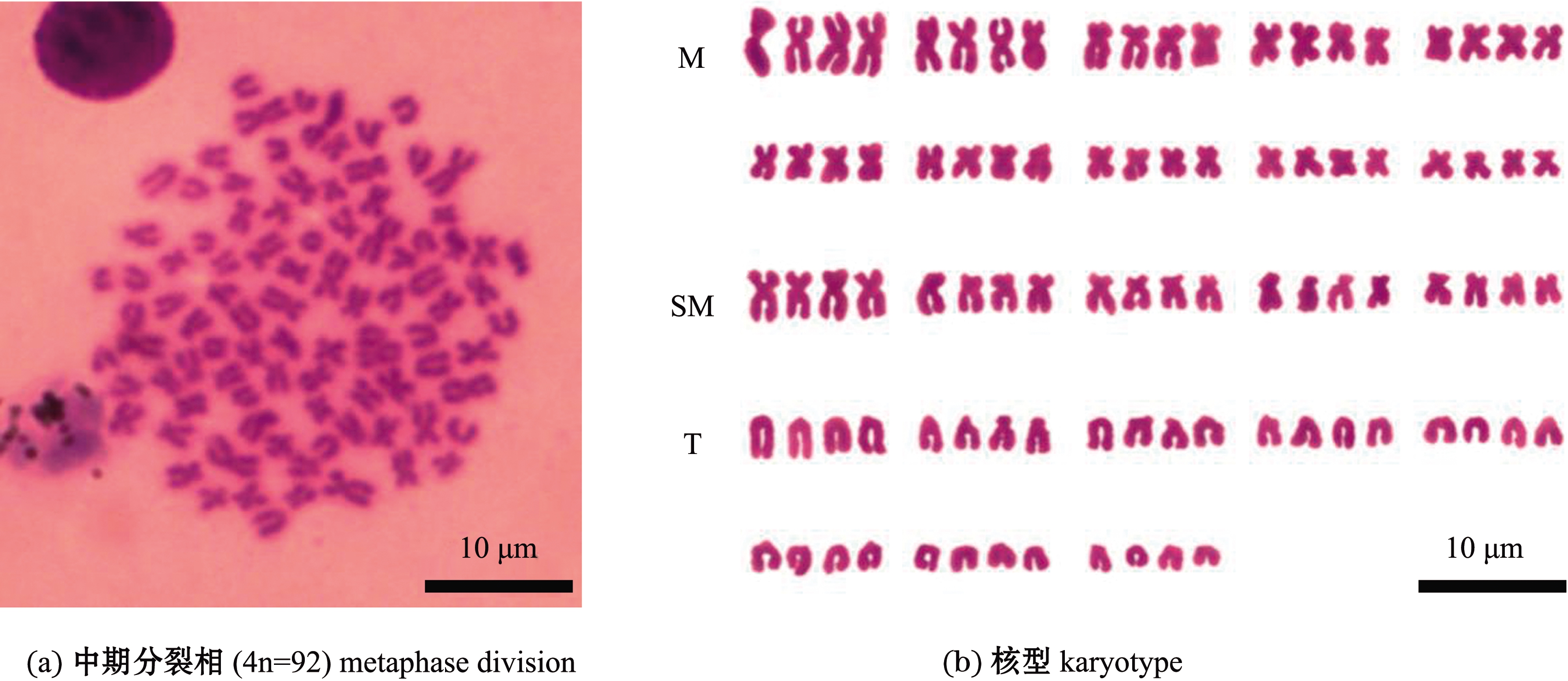
图1 基于Giemsa染色的纳木错裸鲤染色体中期分裂相及核型
Fig.1 Metaphase chromosome and karyotype of Gymnocypris namensis based on Giemsa
表3 纳木错裸鲤的染色体核型数据
Tab.3 Indices of karyotype analysis in Gymnocypris namensis

染色体组序号No.of karyotype 相对长度relative length臂比arm ratio染色体类型classification17.998±0.3051.332±0.105m26.638±0.2981.274±0.077m35.504±0.1821.388±0.091m45.074±0.2941.282±0.039m54.766±0.2121.373±0.080m64.442±0.1771.378±0.188m74.136±0.1721.373±0.127m83.910±0.1441.385±0.100m93.626±0.1501.406±0.036m103.198±0.3011.338±0.075m116.410±0.5371.957±0.156sm125.276±0.4432.028±0.198sm134.608±0.2842.128±0.191sm144.254±0.2032.068±0.156sm153.788±0.3051.599±0.875sm163.952±0.945∞t173.718±0.417∞t183.730±0.274∞t193.446±0.179∞t203.286±0.242∞t213.060±0.225∞t222.774±0.245∞t232.330±0.101∞t
2.2 肾细胞的Ag-NORs分析
银染点数目在间期核和染色体中期分裂相中均表现出多态性。观察统计100个纳木错裸鲤肾细胞银染间期核,呈现出的银染点数目为1~4个(图2),含有2个银染点的频率最高(46%)(表4),并能清晰地观察到最多4个银染点。观察统计30个银染位点清晰、染色体数目完整的中期分裂相,呈现出银染点的数目为1~4个,含有2个银染点的频率最高(47%)(表4),并能够清晰地观察到最多4个银染点(图3(a))。核型分析显示,银染点位于亚中部着丝粒染色体(SM2)短臂的端部区域(图3(b))。这表明,纳木错裸鲤是含有4套染色体的遗传四倍体。
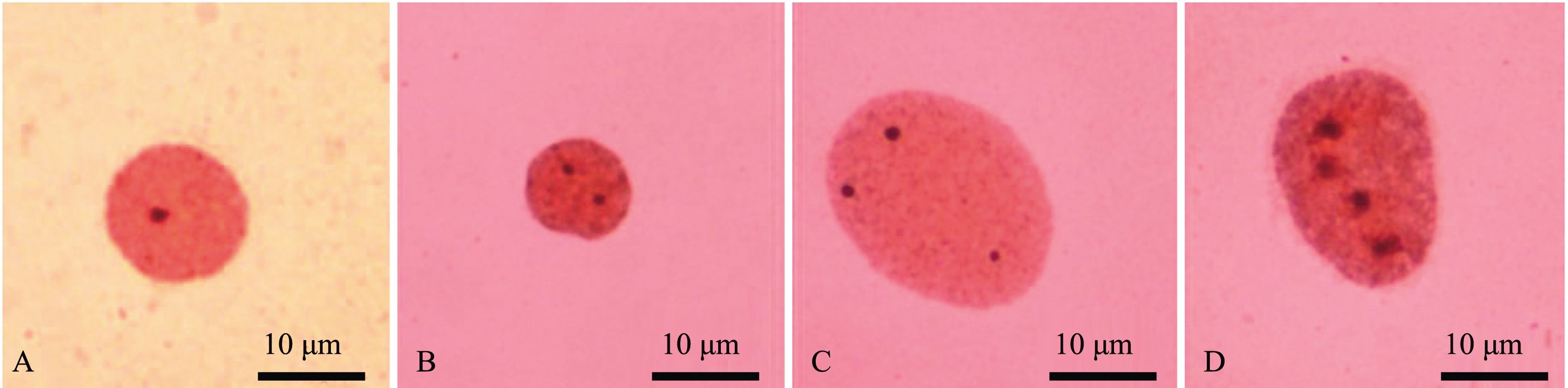
图2 纳木错裸鲤间期核的硝酸银染色
Fig.2 Ag-NORs staining of nuclei at interphase in Gymnocypris namensis
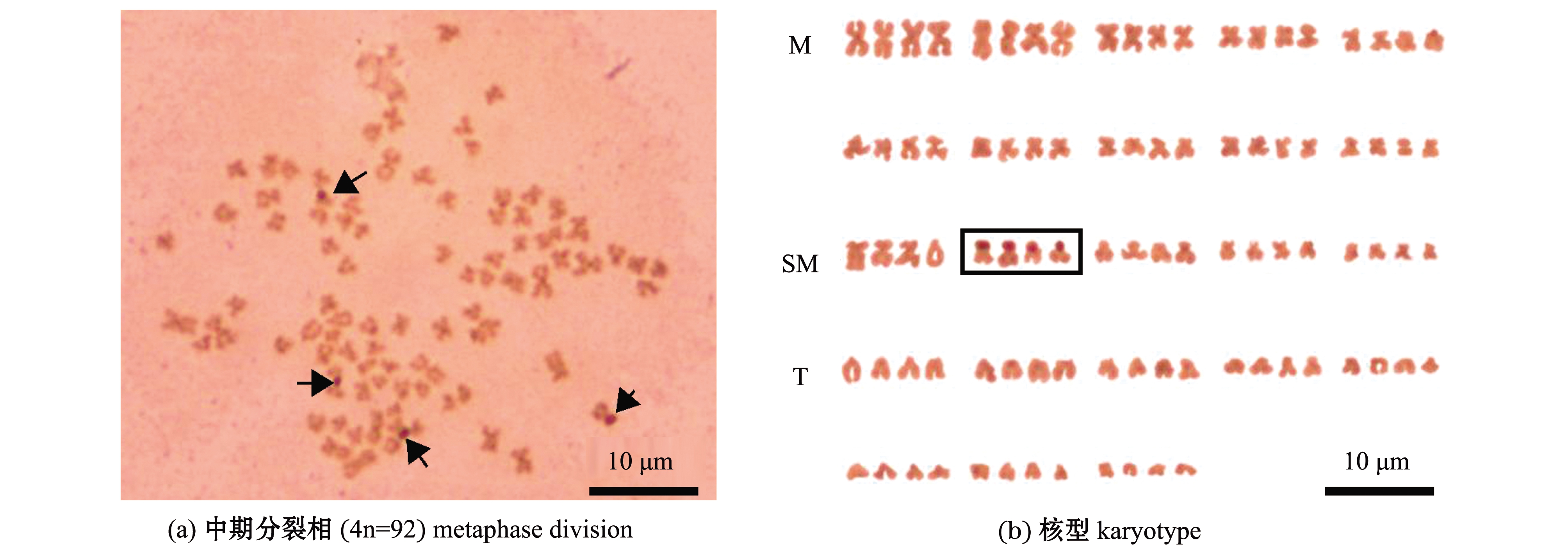
图3 基于Ag-NORs染色的纳木错裸鲤染色体中期分裂相及核型(箭头示银染点)
Fig.3 Metaphase division chromosome and karyotype of Gymnocypris namensis based on Ag-NORs staining(the arrow shows the Ag-NORs site)
表4 纳木错裸鲤中期分裂相银染点频率及间期核中核仁频率
Tab.4 Frequency of silver graining sites at each metaphase and nucleolus in nucleus at interphase
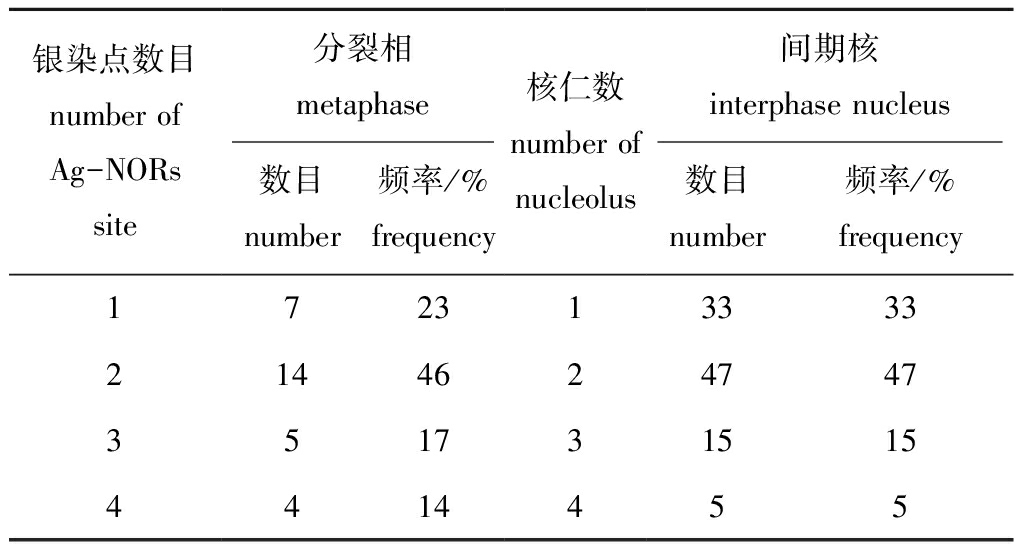
银染点数目number of Ag-NORs site 分裂相metaphase数目number频率/%frequency核仁数number of nucleolus间期核interphase nucleus数目number频率/% frequency17231333321446247473517315154414455
2.3 肾细胞的CMA3/DAPI分析
使用经CMA3/DAPI双重荧光染色处理过的纳木错裸鲤染色体标本制片,置于Leica DM2000荧光显微镜下拍照。当用U激发(蓝光λ=360~400 nm)时,在纳木错裸鲤荧光带型中可观察到4个淡染的DAPI荧光带(图4(a));当用B激发(蓝光λ=440~480 nm)时,在纳木错裸鲤分裂相中显示有4个明亮的CMA3荧光带(图4(b)),经核型分析可知,CMA3阳性位点位于亚中部着丝粒染色体(SM2)短臂的端部区域(图4(c)),与银染点的位置相同,为核仁组织区(NORs)。这表明,纳木错裸鲤是含有4套染色体的遗传四倍体。
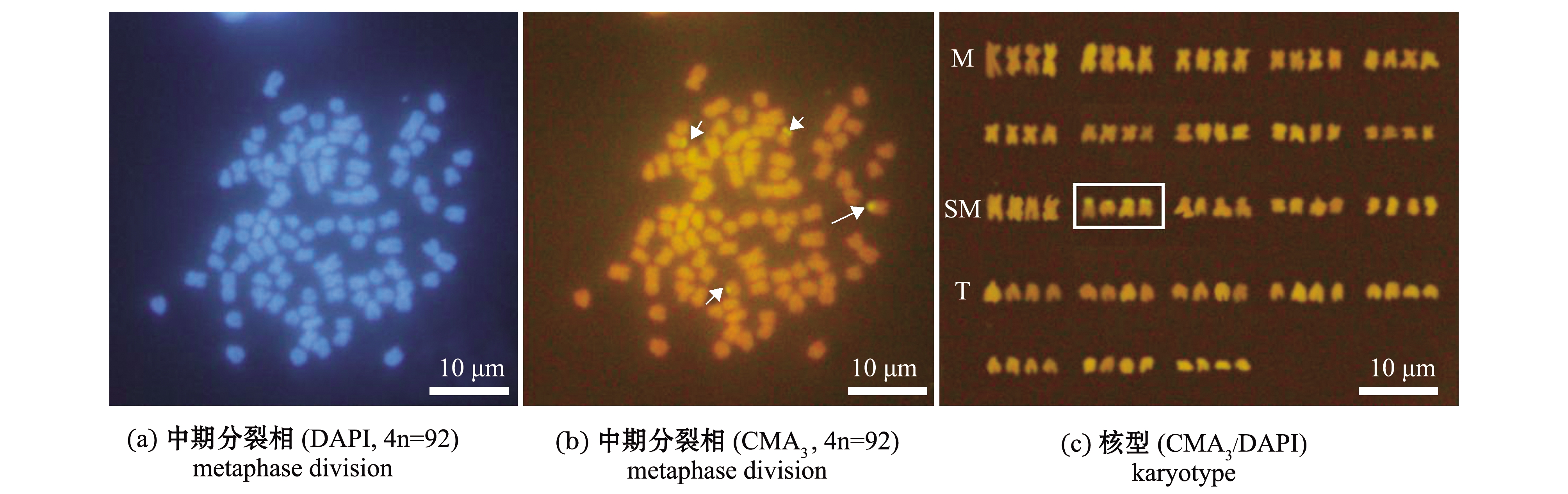
图4 基于CMA3/DAPI染色的纳木错裸鲤染色体中期分裂相及核型(箭头示阳性部位)
Fig.4 Metaphase division and karyotype of Gymnocypris namensis based on CMA3/DAPI(the arrow shows the positive site)
3 讨论
3.1 染色体数目及核型
染色体多态性不仅表现在不同类群、种属间存在明显的差异,如鮡科鱼类染色体数目为2n=48,鳅科高原鳅类染色体数目为2n=50[19],还表现在同种间不同居群间存在明显的差异,如洞庭湖大鳍鳠Mystus macropterus染色体数目为2n=60,核型公式为20m+12sm+16st+12t,NF=92[20],嘉陵江大鳍鳠染色体数目为2n=56,核型公式为20m+16sm+10st+10t,NF=92[21]。已有学者研究发现,拉萨裂腹鱼和异齿裂腹鱼等西藏地区裂腹鱼亚科鱼类存在同种间不同居群染色体数目差异的情况[5,10],尖裸鲤存在同种间不同个体染色体为2n=86[5]和2n=92[10]两种现象。这一特点体现了西藏裂腹鱼亚科鱼类染色体的多态性,也体现出高原裂腹鱼亚科鱼类具有较强的生命进化和适应高原恶劣环境的能力[22-23]。武云飞等[5]推测,西藏裂腹鱼亚科鱼类染色体多态性的特点,可能与青藏高原地区曾经的地质大变动导致的气温骤变、高原气候恶劣,以及各地海拔上升变化明显等不稳定环境因素有关。裂腹鱼亚科鱼类分布较为集中,青藏高原及周边水域是裂腹鱼亚科鱼类的主要生活区域,其中,许多裂腹鱼亚科鱼类仅存在于西藏地区,是西藏地区特有鱼种[24-25],具有地区代表性及进化特点,其染色体多样性在自然界中构成十分独特的系统,具有重要研究意义。但目前西藏裂腹鱼亚科鱼类染色体的研究相对滞后,70%的裂腹鱼亚科鱼类核型尚未见报道。
纳木错裸鲤是西藏地区的特有鱼种[1],但较长时间内对其缺乏系统的生物学研究,在染色体组成方面尚未有报道。本研究结果表明,纳木错裸鲤染色体总数为92条,核型公式为40m+20sm+32t,NF=152,且不同个体间染色体数目及核型相同。纳木错裸鲤的染色体数目与已报道的青藏高原裂腹鱼亚科鱼类染色体数目超过1/3均为92条的研究结果一致[12],与异齿裂腹鱼[8]、拉萨裂腹鱼[8]和尖裸鲤[5]等裂腹鱼亚科鱼类染色体数目相同,均为92条。纳木错裸鲤数目及核型的确定为探究纳木错裸鲤与其他西藏裂腹鱼亚科鱼类的亲缘关系,以及西藏裂腹鱼亚科鱼类的演化和起源奠定了良好的基础。
3.2 倍性分析
目前,已报道的22种裂腹鱼亚科鱼类染色体的组成表明,裂腹鱼亚科鱼类的进化方式以多倍体化为主且多数为四倍体[12]。如花斑裸鲤Gymnocypris eckloni、异齿裂腹鱼和尖裸鲤等裂腹鱼亚科鱼类均为四倍体[10],昆明裂腹鱼Shizothorax grahami和大理裂腹鱼Shizothorax daliensis均为六倍体[8]。余祥勇等[10]对8种裂腹鱼亚科鱼类的染色体核型特征、NF值、红细胞核大小和核DNA含量进行比较,确认其中4种西藏裂腹鱼亚科鱼类为四倍体且染色体变化明显。裂腹鱼亚科鱼类的进化特点与鲃亚科相似[26]。根据化石及考古地质学研究,青藏高原未形成前气候温暖,随着青藏高原隆起,气温骤降,大部分喜暖的原始裂腹鱼被淘汰,少数适应高原环境的鱼活了下来[27]。余祥勇等[10]推测,青藏高原环境变化过程中,在核型演化上很可能主要表现在多倍化,推测青藏高原地区裂腹鱼亚科鱼类可能是多倍体起源的。
NORs特征是研究物种间染色体变异、物种进化和亲缘关系的重要参考依据[28]。银染法是指核仁组织区(NORs)的酸性蛋白成分被硝酸银特异地染成黑色的方法。但如果NORs失活将不能染上色,而CMA3染色是指核仁组织区的rDNA被染色,rDNA并不是酸性蛋白成分,因此,NORs的活性不影响CMA3染色结果。这两种方法均已在生物领域广泛应用。本研究中,采用不同方法对纳木错裸鲤肾细胞染色体进行带型分析,发现染色体经银染及CMA3/DAPI双重染色后最多具有4个信号位点,CMA3阳性部位和位点数目与银染点位置和数目相同,均位于亚中部着丝粒染色体短臂的端部区域,证明纳木错裸鲤是含有4套染色体组的遗传四倍体,这与余祥勇等[10]推测的染色体总数为90~100条的西藏裂腹鱼亚科鱼类均为多倍体的观点一致,且染色体数目为92条的西藏裂腹鱼亚科鱼类均为四倍体,进而推测西藏裂腹鱼亚科鱼类可能具有共同的祖先,在适应青藏高原恶劣多变的生态环境过程中,发生了多倍化[29]。这可能与染色体的着丝点断裂、融合、缺失和易位等情况相关[30]。
综上所述,本研究中确认了纳木错裸鲤染色体数目为92条,核型公式为40m+20sm+32t,NF=152,且未发现有同种不同个体间染色体数目的多样性现象。同时,通过Ag-NORs和CMA3/DAPI双重荧光染色对其倍性进行分析,确定了纳木错裸鲤为含有4套染色体组的遗传四倍体。本研究结果为纳木错裸鲤的染色体组成提供了细胞遗传学证据,并为今后开展西藏裂腹鱼亚科鱼类的分类和演化关系研究提供了科学依据。
4 结论
1)确定了纳木错裸鲤的染色体数目为4n=92,染色体核型公式为40m+20sm+32t,NF=152,与已报道的青藏高原裂腹鱼亚科鱼类染色体数目超过1/3均为92条的研究结果一致。
2)纳木错裸鲤是含有4套染色体组的遗传四倍体,符合裂腹鱼亚科的进化方式以多倍化为主且染色体总数为90~100条的西藏裂腹鱼亚科鱼类均为多倍体的推测。
[1] 何德奎,陈毅峰,蔡斌.纳木错裸鲤性腺发育的组织学研究[J].水生生物学报,2001,25(1):1-13.
HE D K,CHEN Y F,CAI B.Histological studies on the gonadal development of an endemic Tibet fish Gymnocypris namensis[J].Acta Hydrobiologica Sinica,2001,25(1):1-13.(in Chinese)
[2] 蒋志刚,江建平,王跃招,等.中国脊椎动物红色名录[J].生物多样性,2016,24(5):500-551.
JIANG Z G,JIANG J P,WANG Y Z,et al.Red list of China’s vertebrates[J].Biodiversity Science,2016,24(5):500-551.(in Chinese)
[3] 赵亚辉,张洁,张春光.青藏高原鱼类的多样性[J].生物学通报,2008,43(7):8-10.
ZHAO Y H,ZHANG J,ZHANG C G.Fish diversity in the Tibetan Plateau[J].Bulletin of Biology,2008,43(7):8-10.(in Chinese)
[4] 王金林,牟振波,王且鲁,等.西藏裂腹鱼亚科鱼类研究进展[J].安徽农业科学,2018,46(24):16-19.
WANG J L,MOU Z B,WANG Q L,et al.Research progress on Schizothoracinae fishes in Tibet[J].Journal of Anhui Agricultural Sciences,2018,46(24):16-19.(in Chinese)
[5] 武云飞,康斌,门强,等.西藏鱼类染色体多样性的研究[J].动物学研究,1999,20(4):258-264.
WU Y F,KANG B,MEN Q,et al.Chromosome diversity of Tibetan fishes[J].Zoological Research,1999,20(4):258-264.(in Chinese)
[6] 马凯,佟广香,张永泉,等.尖裸鲤染色体核型分析及进化地位研究[J].西北农林科技大学学报(自然科学版),2021,49(12):28-33.
MA K,TONG G X,ZHANG Y Q,et al.Karyotype analysis and evoutionary status of Oxygymnocypris stewartii[J].Journal of Northwest A & F University(Natural Science Edition),2021,49(12):28-33.(in Chinese)
[7] 周贤君.拉萨裂腹鱼个体生物学和种群动态研究[D].武汉:华中农业大学,2014:11.
ZHOU X J.Study on the biology and population dynamics of Schi-zothorax waltoni[D].Wuhan: Huazhong Agricultural University,2014:11.(in Chinese)
[8] 余先觉,周暾,李渝成,等.中国淡水鱼类染色体[M].北京:科学出版社,1989:152-153.
YU X J,ZHOU T,LI Y C,et al.Chromosomes of freshwater fishes from China[M].Beijing:Science Press,1989:152-153.(in Chinese)
[9] 祁得林.青海湖裸鲤染色体核型及多倍性的初步研究[J].青海大学学报(自然科学版),2004,22(2):44-47.
QI D L.Preliminary studies on chromosome karyotype and polyplo-idy of Qinghai Lake naked carp[J].Journal of Qinghai University(Natural Science Edition),2004,22(2):44-47.(in Chinese)
[10] 余祥勇,李渝成,周暾.中国鲤科鱼类染色体组型研究——8种裂腹鱼亚科鱼类核型研究[J].武汉大学学报(自然科学版),1990(2):97-104.
YU X Y,LI Y C,ZHOU T.Karyotpe studies of cyprinid fishes in China——comparative study of the karyotypes of 8 species of Schizothoracinae fishes[J].Journal of Wuhan University(Natural Science Edition),1990(2):97-104.(in Chinese)
[11] 施立明.遗传多样性及其保存[J].生物科学信息,1990,2(4):158-164.
SHI L M.Genetic diversity and its preservation[J].Chinese Bulletin of Life Sciences,1990,2(4):158-164.(in Chinese)
[12] 代应贵,肖海.裂腹鱼类种质多样性研究综述[J].中国农学通报,2011,27(32):38-46.
DAI Y G,XIAO H.Review of studies on the germplasm resources of the Schizothoracinae fishes[J].Chinese Agricultural Science Bulletin,2011,27(32):38-46.(in Chinese)
[13] 林义浩.快速获得大量鱼类肾细胞中期分裂相的PHA体内注射法[J].水产学报,1982,6(3):201-208.
LIN Y H.A PHA injection method in vivo for the rapid obtainment of large numbers of metaphase figures from kideny cells of teleosts[J].Journal of Fisheries of China,1982,6(3):201-208.(in Chinese)
[14] LEVAN A,FREDGA K,SANDBERG A A.Nomenclature for centromeric position on chromosomes[J].Hereditas,1964,52(2):201-220.
[15] HOWELL W M,BLACK D A.Controlled silver-staining of nucleolus organizer regions with a protective colloidal developer:a 1-step method[J].Experientia,1980,36(8):1014-1015.
[16] SCHWEIZER D.Simultaneous fluorescent staining of R bands and specific heterochromatic regions(DA-DAPI bands)in human chromosomes[J].Cytogenetic and Genome Research,1980,27(2/3):190-193.
[17] SCHWEIZER D,AMBROS P,ANDRLE M.Modification of DAPI banding on human chromosomes by prestaining with a DNA-binding oligopeptide antibiotic,distamycin A[J].Experimental Cell Research,1978,111(2):327-332.
[18] AMORES A,MARTINEZ G,REINA J,et al.Karyotype,C-banding,and Ag-NOR analysis in Diplodus bellottii(Sparidae,Perciforms).Intra-individual polymorphism involving heterochromatic regions[J].Genome,1993,36(4):672-675.
[19] 马秀慧.中国鮡科鱼类系统发育、生物地理及高原适应进化研究[D].重庆:西南大学,2015:22.
MA X H.The phylogeny,biogeograph and adaptive evolution of Chinese Sisoridae fishes[D].Chongqing:Southwest University,2015:22.(in Chinese)
[20] 文永彬,史怡雪,刘良国,等.洞庭湖水系3种鲿科鱼的染色体核型分析[J].江苏农业科学,2013,41(12):235-238.
WEN Y B,SHI Y X,LIU L G,et al.Karyotype analysis of three kinds of Bagridae in Dongting Lake[J].Jiangsu Agricultural Sciences,2013,41(12):235-238.(in Chinese)
[21] 马跃岗,张峰,牟平,等.嘉陵江大鳍鳠染色体组型分析[J].现代农业科技,2013(2):260-261.
MA Y G,ZHANG F,MOU P,et al.Analysis of the karotype of Mystus macropterus Bleeker in the Jialin River[J].Modern Agricultural Science and Technology,2013(2):260-261.(in Chinese)
[22] 赵新全,祁得林,杨洁.青藏高原代表性土著动物分子进化与适应研究[M].北京:科学出版社,2008:1-70.
ZHAO X Q,QI D L,YANG J.Molecular evolution and adaptation of representative indigenous animals on the Tibetan Plateau[M].Beijing:Science Press,2008:1-70.(in Chinese)
[23] 许静.雅鲁藏布江四种特有裂腹鱼类早期发育的研究[D].武汉:华中农业大学,2011:8.
XU J.Studies on the early development of four endemic Schizothorax species from the Yarlung Zangbo River[D].Wuhan: Huazhong Agricultural University,2011:8.(in Chinese)
[24] 马宝珊,谢从新,霍斌,等.裂腹鱼类生物学研究进展[J].江西水产科技,2011(4):36-40.
MA B S,XIE C X,HUO B,et al.Advances in biology of Sizothoracicus[J].Jiangxi Fishery Science and Technology,2011(4):36-40.(in Chinese)
[25] 李雷,马波,金星,等.西藏雅鲁藏布江中游裂腹鱼类优先保护等级定量评价[J].中国水产科学,2019,26(5):914-924.
LI L,MA B,JIN X,et al.Studies on the early development of four endemic Schizothorax species from the Yarlung Zangbo River[J].Journal of Fishery Sciences of China,2019,26(5):914-924.(in Chinese)
[26] 昝瑞光,刘万国,宋峥.裂腹鱼亚科中的四倍体—六倍体相互关系[J].遗传学报,1985,12(2):137-142,167-168.
ZAN R G,LIU W G,SONG Z.Tetraploid-hexaploid relationship in Schizothoracinae[J].Journal of Genetics and Genomics,1985,12(2):137-142,167-168.(in Chinese)
[27] 曹文宣,陈宜瑜,武云飞,等.裂腹鱼类的起源和演化及其与青藏高原的隆起关系[M].北京:科学出版社,1981:118-130.
CAO W X,CHEN Y Y,WU Y F,et al.Origin and evolution of Schizothorax and its relationship with uplift of Qinghai-Tibet Plateau[M].Beijing:Science Press,1981:118-130.(in Chinese)
[28] 梁雨婷,隋燚,庄子昕,等.杂交三倍体泥鳅×二倍体泥鳅杂交后代胚胎染色体组构成的研究[J].大连海洋大学学报,2018,33(4):477-480.
LIANG Y T,SUI Y,ZHUANG Z X,et al.Formation of embryonic genomes in hybrid triploid loach×diploid loach[J].Journal of Dalian Ocean University,2018,33(4):477-480.(in Chinese)
[29] 陈毅峰.裂腹鱼类的系统进化及资源生物学[D].武汉:中国科学院水生生物研究所,2000:21-82.
CHEN Y F.Systematics and resource biology of the Schizothoracine fishes[D].Wuhan: Institute of Hydrobiology,Chinese Academy of Scienc,2000:21-82.(in Chinese)
[30] 高文.鱼类染色体研究进展[J].宁德师专学报(自然科学版),2005,17(1):15-18.
GAO W.Advances in study of fish chromosome[J].Journal of Ningde Teachers College(Natural Science Edition),2005,17(1):15-18.(in Chinese)