刺参Apostichopus japonicus是中国北方的重要经济棘皮物种,具有较高的食用价值和药用价值。盐度是影响刺参生理生态学的重要非生物因素,其变化对机体的生理过程具有重大影响[1]。目前,有关盐度对刺参生理生态等影响的研究较多。王玉凤等[2]研究发现,在盐度为 25、27、29、31时,刺参的吞噬活力随着盐度升高而升高,当盐度升高至33时,吞噬活力随之降低。赵斌等[3]研究发现,盐度为31时,刺参的成活率最高且体长增长最大,当盐度升高至35、39时,成活率下降,当盐度降低至23、27时,体长显著下降。袁秀堂等[4]研究发现,当盐度为31.5时,刺参的呼吸和排泄速率较低,而当盐度升高至36或降低至27、22时,呼吸和排泄速率则增加。王吉桥等[5]研究发现,随着盐度下降,刺参幼参体腔液中超氧化物歧化酶活力降低,溶菌酶活力升高。Geng等[6]研究发现,低盐胁迫下在刺参的肠、肌肉和管足中皆可观察到不同程度的损伤,其中,低盐胁迫时的管足结缔组织肿胀且纵向肌肉破裂,高盐胁迫时结缔组织收缩,与低盐胁迫相比高盐胁迫对刺参管足的影响更加显著。这些结果表明,盐度对刺参的存活、生长行为、能量代谢及组织学等都有着极大的影响。
microRNA(miRNA)是一类长度为18~22 nt且进化相对保守的非编码RNA,通过与靶基因特异性结合在转录后调节基因表达。近年来,miRNA及其对应的靶基因参与生物体环境胁迫响应机制已成为研究热点。将尼罗罗非鱼暴露在不同盐度水平下,其肾脏中miR-30c的相对表达量与盐度存在负相向表达趋势,当miR-30c受到低盐胁迫后,HSP70 mRNA的水平显著提高,同时也影响鱼体内的离子浓度和血浆渗透压[7]。三疣梭子蟹Portunus trituberculatus在4个盐度水平胁迫下,鳃组织中有12个差异表达的miRNAs参与盐度胁迫响应[8]。太平洋牡蛎Crassostrea gigas和香港牡蛎Crassostrea hongkongensis在低盐胁迫下,鳃组织中的miR-10a呈现上调表达趋势,推测miRNAs可能在牡蛎的盐度响应中发挥重要功能[9]。鳗鲡Anguilla japonica鳃组织中的miR-122、miR-140-3p和miR-10b-5p在盐度变化过程中参与其渗透调节过程,这3个miRNA在渗透调节中可能发挥不同的作用[10]。尼罗罗非鱼Oreochromis niloticus经高盐胁迫后,在鳃组织中抑制miR-429会影响罗非鱼的渗透调节,并发现miR-429作为OSTF1表达的内源性调控因子,参与尼罗罗非鱼的渗透感应信号传导过程[11]。这些研究结果为水产生物响应低盐胁迫的分子机制提供了数据基础,并为水产养殖中合理设定盐度水平提供了理论参考。
miRNA通过与靶基因3′-非翻译区(UTR)的碱基互补配对从而形成靶向RNA诱导的沉默复合物,其中互补配对会导致转录后靶基因的降解,并能有效调节外界环境中的不利因素。目前,有关miRNA及靶基因参与刺参的盐度响应研究相对较少。蒋亚男[12]对低盐胁迫刺参进行体内和体外试验,验证了3个miRNA(miR-2008、miR-278-3p和miR-92a)与其靶基因在刺参盐度适应调节机制中发挥的作用。郭然等[13]发现,miR-22和miR-27及其靶基因在刺参的盐度适应调节机制中也发挥作用。Tian等[14]发现,miR-10通过靶向基因TBC1D5参与刺参的盐度响应过程。Huo等[15]发现,3个miR-1、miR-2和miR-3 在刺参盐度胁迫中上调表达,这些结果为刺参其他miRNA调节盐度响应机制提供了重要信息。
本研究在课题组前期试验的基础上,测定了低盐胁迫不同时间段后刺参体腔液中的离子浓度和钠钾ATP酶活力,通过对盐度胁迫后的miRNA和靶基因的测序结果进行分析,选择与盐度胁迫相关的3个差异表达的miRNA(miR-2011、miR-2010和miR-124)进行研究,并获得与其互作的靶基因,探究了其在刺参盐度响应中的表达情况,以期为了解刺参盐度响应机制提供科学参考。
1 材料与方法
1.1 材料
试验用刺参取自河北唐山市海域,试验前期暂养于农业农村部北方海水增养殖重点实验室养殖水槽,暂养7 d左右,期间保证24 h充足氧气,每天投食、吸底一次,并用自然海水与曝气自来水混合配制低盐海水(盐度18),连续3 d测量盐度,使海水盐度稳定。
选择状态良好、体质量为(16.38±1.27)g的刺参进行试验。
1.2 方法
1.2.1 试验设计 以低盐胁迫0 h 作为对照组,胁迫6、24、48 h 作为试验组,每组设3个重复,每个重复10头刺参,于胁迫不同时间点,用一次性针头吸取刺参体腔液,将体腔液以3 000 r/min离心5 min,弃上清液获得体腔细胞,用于后续试验。
1.2.2 低盐胁迫下相关离子浓度及钠钾ATP酶活力的测定 在低盐胁迫的0、6、24、48 h取刺参体腔液,用南京建成生物工程研究所的试剂盒测定刺参体腔液中的钠、氯、钾离子浓度,以及钠钾ATP酶活力。
1)钠离子浓度。钠离子与6-氢氧化锑钾在分散剂和除干扰剂的存在下形成均一的浊度。在620~630 nm 波长测定其吸光度值,并计算样本中钠离子浓度(mmol/L)。
钠离子浓度=(样本OD值/标准OD值)×标准品浓度(140 mmol/L)×样品稀释倍数。
2)氯离子浓度。氯离子与硫氰酸汞会形成有色的络合物,络合物颜色的深度与氯离子的浓度成正比。在480 nm 处测定其吸光度值,并计算氯离子浓度(mmol/L)。
氯离子浓度=(样本OD值-空白OD值)/(标准OD值-空白OD值)×标准品浓度(20 mmol/L)×样品稀释倍数。
3)钾离子浓度。先将样本用蛋白沉淀剂处理,而样本中的钾离子与NA-TPB产生混浊且稳定的悬浮液,钾离子的浓度与混浊度成正比。在440 nm处测定其吸光度,并计算样本中钾离子浓度(mmol/L)。
钾离子浓度=(样本OD值-空白OD值)/(标准OD值-空白OD值)×标准品浓度(0.4 mmol/L)×样品稀释倍数。
4)钠钾ATP酶活力。ATP经过ATP酶的催化分解可产生ADP和无机磷,再通过测定无机磷的含量判断ATP酶活力的高低。在636 nm处测定各管吸光度值,并计算样本中ATP酶活力(U/mg prot)。
ATPase活力=(样本OD值-对照OD值)/(标准OD值-空白OD值)×标准品浓度(0.02 μmol/mL)×6×7.8/样本蛋白浓度。
1.2.3 低盐胁迫下渗透压 利用Gonotec Osmomat 03冰点渗透压仪对低盐胁迫后的体腔液进行测定,每个样品测定3次,取其平均值。
1.2.4 差异表达miRNA的筛选及靶基因的生物信息学预测与分析 基于盐度胁迫后刺参转录组和miRNA的高通量测序结果,利用BLAST将刺参转录组的序列同miRNA数据库中的miRNA进行比对,比对过程中设定miRNA序列与转录组序列错配不超过3。将成熟 miRNA序列比对到转录组序列上,在其位置上下游各取100个碱基作为可能的miRNA前体,设置前体最短为55 nt。将获得的miRNA前体序列同蛋白质数据库进行比对,将比对到蛋白质数据库中的序列去掉,经过筛选得到的序列用RNAfold软件预测二级结构,最后根据比对的位置得到最终成熟的miRNA。
经过上述比对预测与分析,从刺参低盐胁迫后的转录组数据库表达谱中筛选获得miR-2011、miR-2010和miR-124这3个miRNA。利用WebLOGO(http://weblogo.berkeley.edu/logo.cgi)对miRNAs前体序列和成熟体序列的碱基保守性进行分析。
利用miRanda 3.01 软件进行3个miRNA靶基因的预测。检测分数(单残基对分数)S>90且最小自由能低于-17 kcal/mol 的候选目标作为首选的靶基因,最终选择得分较高、热力学稳定性较强的基因作为候选基因。针对3个miRNA进行了前体序列分析,并从miRNA Base中选择海胆Lytechinus variegatus、红鳍东方鲀Takifugu rubripes、鲤Cyprinus carpio、斑马鱼Danio rerio、蝠海星Patiria miniata、黑青斑河鲀Tetraodon nigroviridis和柯瓦列夫斯基橡胶虫Saccoglossus kowalevskii进行分析,获得miRNA的进化关系。
1.2.5 总RNA 提取及mRNA反转录 收集体腔细胞离心后加入1 mL Trizol,室温放置15 min,加入200 μL氯仿,震荡后放置15 min,以12 000 r/min离心15 min,吸取上清液放入另一离心管中;加入0.5 mL异丙醇混匀后放置5~10 min,相同转速下离心10 min,弃上清液,加入1 mL乙醇,震荡悬浮,以8 000 g离心5 min,弃上清液,室温晾干5~10 min,加入适量的H2O。mRNA反转录体系(20 μL):RNA 100 ng,oligo(Dt)18 1 μL,Dntp Mixture 0.5 μL,RNase Inbibitor(40 U/μL)0.25 μL,RTase M-MLV(200 U/μL)0.25 μL,用无RNase-free ddH2O补足至20 μL。反应程序:42 ℃下变性退火30 min,95 ℃下酶失活5 min。
1.2.6 miRNA cDNA合成 cDNA合成反应体系(20 μL):RNA 2 000 ng,2×miRNA RT Reaction Buffer 10 μL,miRNA RT Enzyme Mix 2 μL,用无 RNase ddH2O补足至20 μL。反应条件:42 ℃下退火延伸 60 min,95 ℃下酶失活 3 min后反应终止。
1.2.7 miRNA的表达量分析 利用软件Primer 3.0软件在线设计引物,并进行引物特异性检测。试验所用引物均由生工生物工程(上海)股份有限公司合成。3个miRNA及内参基因引物序列见表1。试验miRNA qPCR的反应体系:2×miRcute Plus miRNA Premix(含 SYBR 和ROX)10 μL,Forward primers 0.2 μL(表 1),Reverse primer(100 mmol/L)0.2 μL,50×ROX Reference Dye 0.2 μL,无RNase ddH2O 7.6 μL。qPCR反应程序:95 ℃下预变性10 min;95 ℃下循环变性15 s,60 ℃下退火复性60 s,共进行45个循环。采用 2-△△Ct方法计算基因相对表达量。
表1 引物序列
Tab.1 Primer sequence
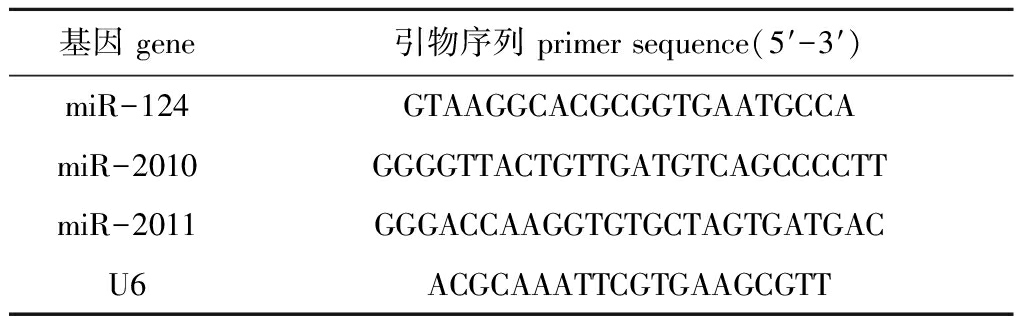
基因 gene 引物序列 primer sequence(5′-3′)miR-124GTAAGGCACGCGGTGAATGCCAmiR-2010GGGGTTACTGTTGATGTCAGCCCCTTmiR-2011GGGACCAAGGTGTGCTAGTGATGACU6ACGCAAATTCGTGAAGCGTT
1.3 数据处理
试验数据均以平均值±标准差(mean±S.D.)表示。采用SPSS 22.0 软件进行单因素方差分析(one-way ANOVA),采用T检验法进行组间比较,显著性水平设为0.05,极显著性水平设为0.01。
2 结果与分析
2.1 低盐胁迫后离子浓度、渗透压及钠钾ATP酶活力的变化
从表2可见:刺参体腔液中的钠离子浓度在低盐度(18)胁迫6、24 h时均显著高于对照组(0 h)(P<0.05),且在6 h 时最高,而在48 h与对照组无显著性差异(P>0.05);氯离子、钾离子浓度在整个低盐胁迫期间均极显著低于对照组(P<0.01),且在 6 h 时出现最低浓度;钠钾ATP酶活力在低盐胁迫 6 h 时开始显著降低(P<0.05),在 24 h 时出现最低值;渗透压在低胁迫后极显著低于对照组(P<0.01),且在24 h 时达到最低值。
表2 不同胁迫时间下刺参体腔液中离子浓度、渗透压和钠钾ATP酶活力的变化
Tab.2 Sodium, potassium, and chloride ion concentrations, osmotic pressure and Na+/K+-ATPase(NKA)activity in coelomic fluid of sea cucumber Apostichopus japonicus under different stress time

胁迫时间/hstress timeNa+/(mmol·L-1)sodiumCl-/(mmol·L-1)chlorideK+/(mmol·L-1)potassium钠钾ATP酶活力/(U·mg-1 prot)activity of Na+/K+-ATPase渗透压/(mol·L-1)osmotic pressure0(对照)139.256 432.130 515.850 90.461 71.036 36168.203 1∗15.436 1∗∗13.589 0∗∗0.386 6∗0.742 7∗∗24156.069 4∗20.602 0∗∗14.173 5∗∗0.186 8∗0.521 0∗∗48144.089 919.527 8∗∗14.117 8∗∗0.443 40.643 2∗∗
注:*表示与对照组有显著性差异(P<0.05);**表示与对照组有极显著性差异(P<0.01)。
Note:*means significant difference compared with the control(P<0.05);**means very significant difference compared with the control(P<0.01).
2.2 3个差异表达miRNA的前体序列分析
基于测序结果比较分析后,获得差异表达的3个miRNA(miR-2011、miR-124和 miR-2010),其前体的茎环结构如图1所示,miR-2011、miR-124和 miR-2010均能形成稳定的茎环结构。其中,miR-2011属于MIPF00007家族,预测其前体长度为100 nt,前体的最小自由能为-219.95 kJ/mol;miR-124 属于MIPF0000021家族,预测其前体长度为101 nt,最小自由能为-144.47 kJ/mol;miR-2010属于MIPF0002019家族,预测其前体长度为79 nt,最小自由能为-139.16 kJ/mol。
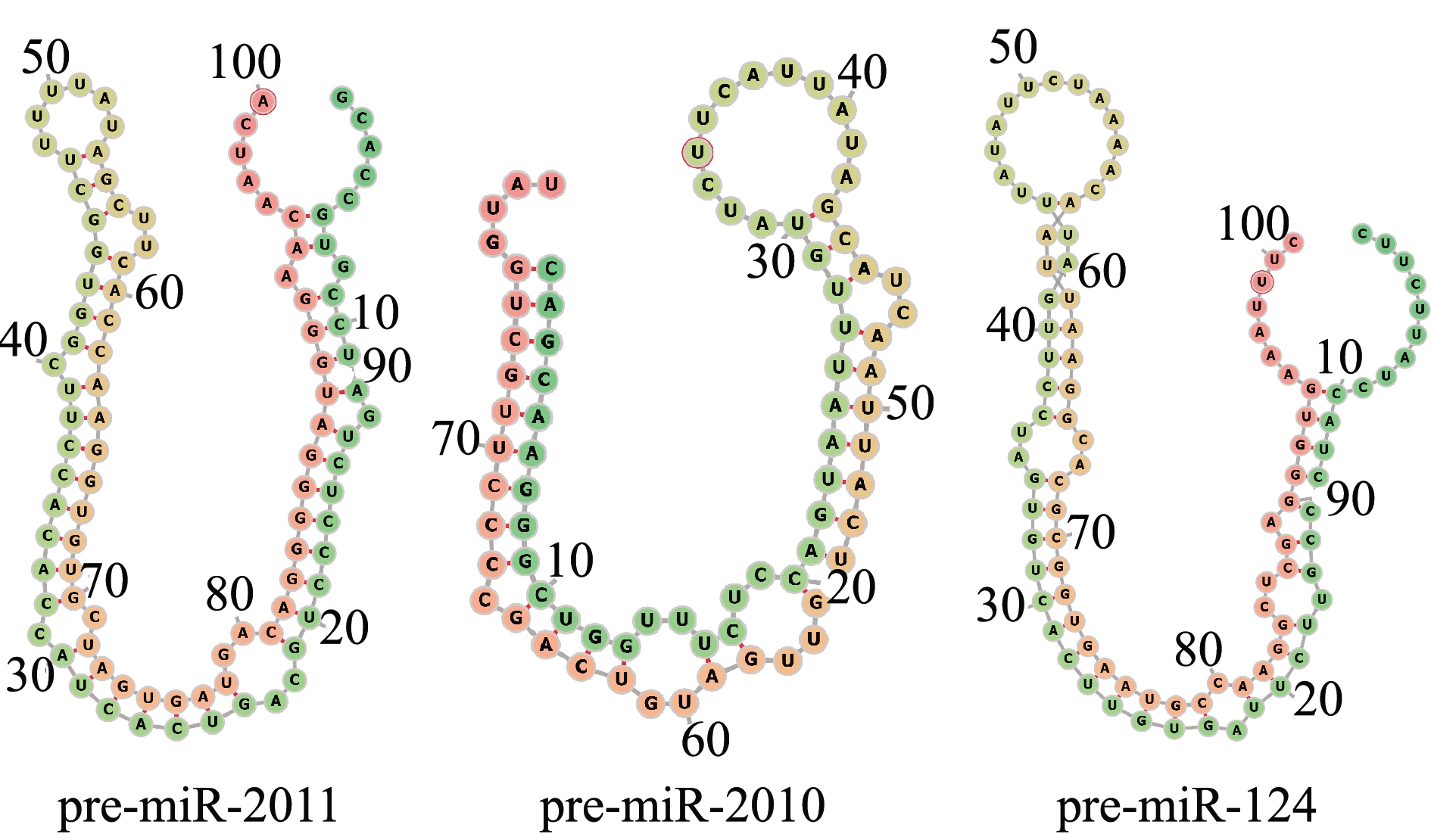
图1 miRNA前体茎环结构
Fig.1 miRNA precursor stem-loop structure
3个miRNA的前体序列与其他物种的聚类分析结果如图2所示,刺参的miR-124、miR-2011和miR-2010分别与海胆的miR-124、miR-2011和miR-2010的前体序列亲缘关系较近,在进化树中归属于同一支。
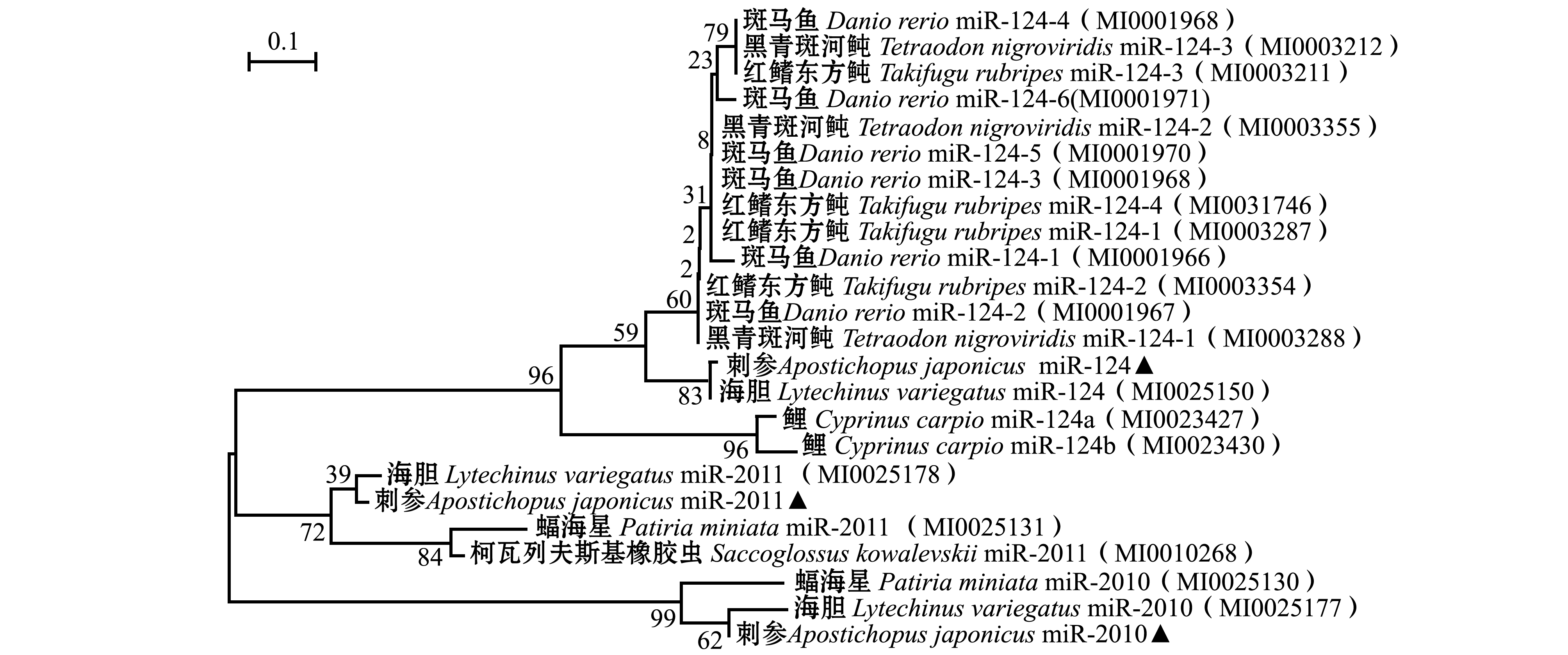
图2 miRNA前体序列结构进化树
Fig.2 Phylogenetic tree of miRNA precursor structure
将miR-124的前体序列和成熟体分别与其对应家族的其他物种进行序列比对分析,结果如图3所示。从图3A可见:miR-124的前体序列中有33个碱基相同,存在两个重叠区,可见miR-124家族成员在进化过程中可能存在高度保守区,这个高度保守区域在26~44碱基位点,另一个在58~76碱基位点;miR-124的前体序列中也存在诸多不保守区域,这些不保守区域可能是在形成的过程中其前体长期自然选择导致了突变的积累并固定的结果。从图3B可见,miR-124的成熟序列中也有保守碱基和存在差异的碱基。从图3C、D可见,miR-2011的前体序列和成熟序列中也有保守碱基和存在差异的碱基。从图3E、F可见,miR-2010的前体序列中存在不一致的碱基,而miR-2010的成熟序列的碱基基本一致。

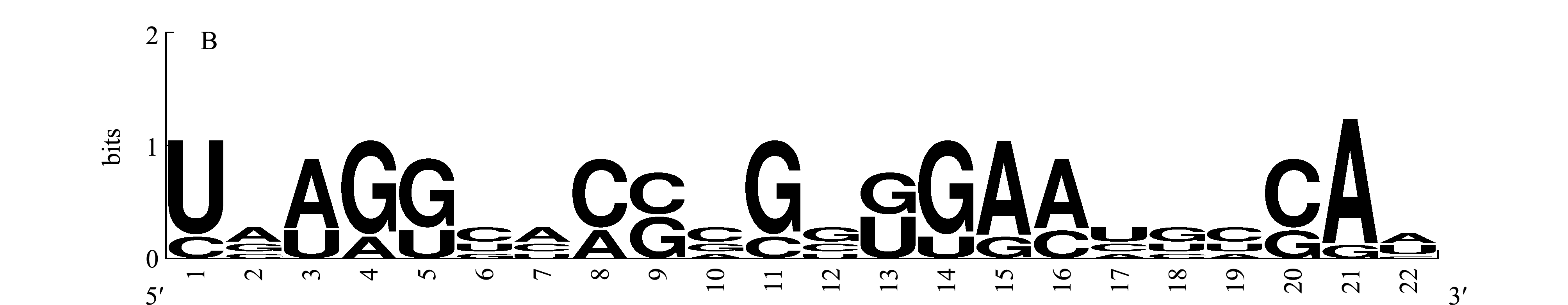
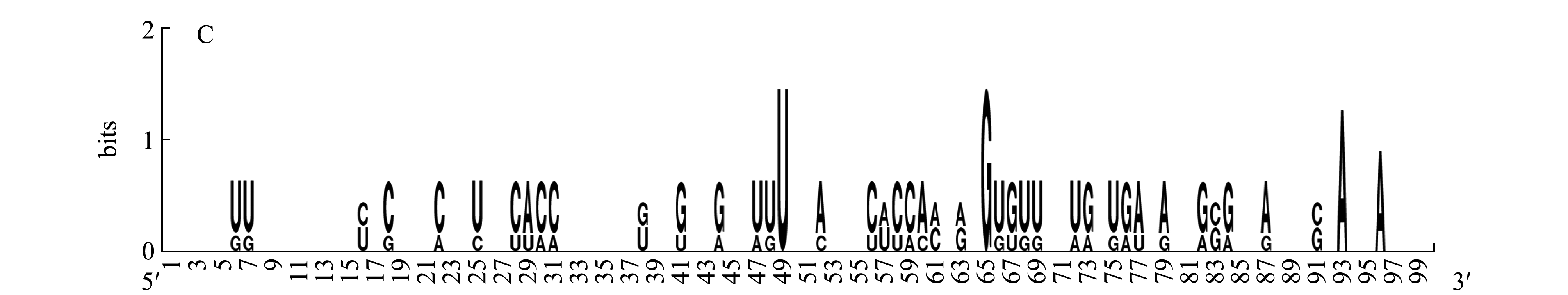



图3 miR-124、miR-2011和miR-2010与其他物种序列的比对
Fig.3 Sequence alignment comparison of miR-124,miR-2011 and miR-2010 with sequences of other species
2.3 3个miRNA靶基因预测结果
利用miRanda 3.01 软件进行靶基因的预测,结果发现,miR-124的2~19位点与EGF3基因的496~517碱基位点能够结合,序列比对分值为145,高于阈值参数90,最小自由能为-79.50 kJ/mol,说明miR-124与靶基因EGF3结合能力强,预测的靶基因EGF3可靠。miR-124的种子区与IMPA1基因的454~474 位点特异性结合,序列比对分值为142,最小自由能为-71.13 kJ/mol,说明IMPA1也是miR-124的靶基因。PPM1L基因3′-UTR 区域序列与miR-2010、miR-2011种子区碱基均能匹配,序列比对得分和最小自由能分别为153、-83.26 kJ/mol和149、-84.94 kJ/mol,预测结果说明,PPM1L基因为miR-2010和miR-2011的共有靶基因。miR-2011的2~21位点与靶基因PBK的655~676位点序列比对分值为164,最小自由能为-88.28 kJ/mol,预测结果说明,PBK基因为miR-2011的另一个靶基因(图4)。

图4 miRNA与靶基因的结合靶点
Fig.4 Binding sites of miRNA to target genes
2.4 差异表达的3个miRNA的验证
在差异表达的miRNA中选择3个miRNA进行分析,测序结果显示,miR-124和miR-2010在盐度胁迫中下调表达,而miR-2011在盐度胁迫中上调表达(表3)。定量PCR验证3个miRNA在刺参盐度胁迫下的表达情况,结果发现,miR-2010和miR-2011的表达情况与测序结果一致,而miR-124的表达与测序结果存在差异(图5)。
表3 低盐胁迫下试验组与对照组差异表达的miRNA调节情况
Tab.3 Differentially expressed miRNAs of sea cucumber in control and treat groups during salinity acclimation

miRNA 试验组表达量 expressed level in test group 对照组表达量expressed level in control group 差异倍数 fold changeP值 P value上升/下降 up/downmiR-12411.290 52033.266 28-1.558 90.001 693downmiR-20102.185 26115.103 66-2.7890.001 606downmiR-201132 165.7715 990.371.008 30up
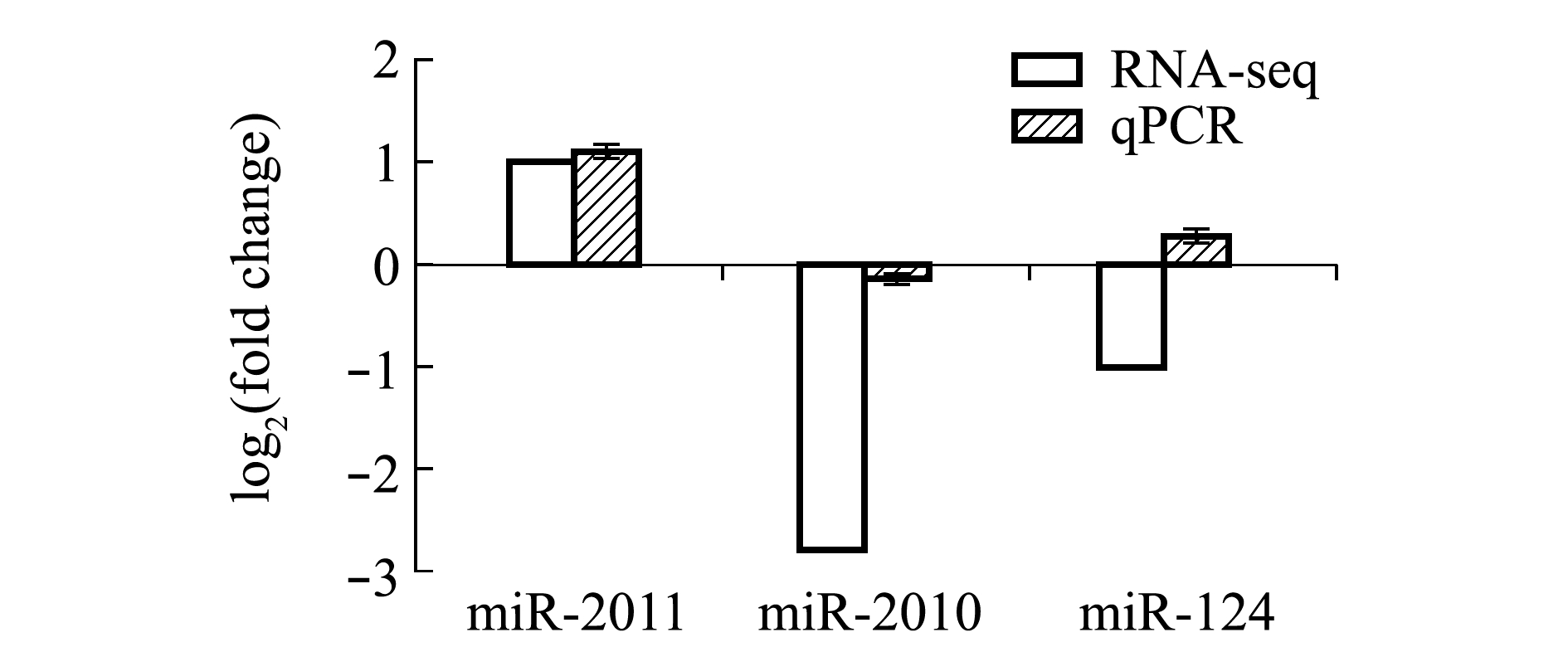
图5 miR-2011、miR-2010和miR-124 RNA的测序与定量表达比较
Fig.5 Comparison of quantification expression and sequencing of miR-2011, miR-2010 and miR-124 RNA
2.5 3个miRNA在盐度胁迫下的表达变化
从图6可见:低盐胁迫下,miR-124的表达量呈先上升后下降的趋势,24 h时其表达量较对照组显著升高(P<0.05),且表达量是对照组的3.4倍,在48 h时表达量逐渐下降并与对照组无显著性差异(P>0.05);miR-2011表达量在各个胁迫时间点均有不同程度的上调,胁迫24、48 h时其表达量与对照组有极显著性差异(P<0.01),且表达量分别为对照组的14倍和80倍;miR-2010表达量在盐度胁迫过程中呈上调表达,胁迫48 h时达到最大值,除胁迫6 h外,其余时间点其表达量均与对照组均有极显著性差异(P<0.01)。
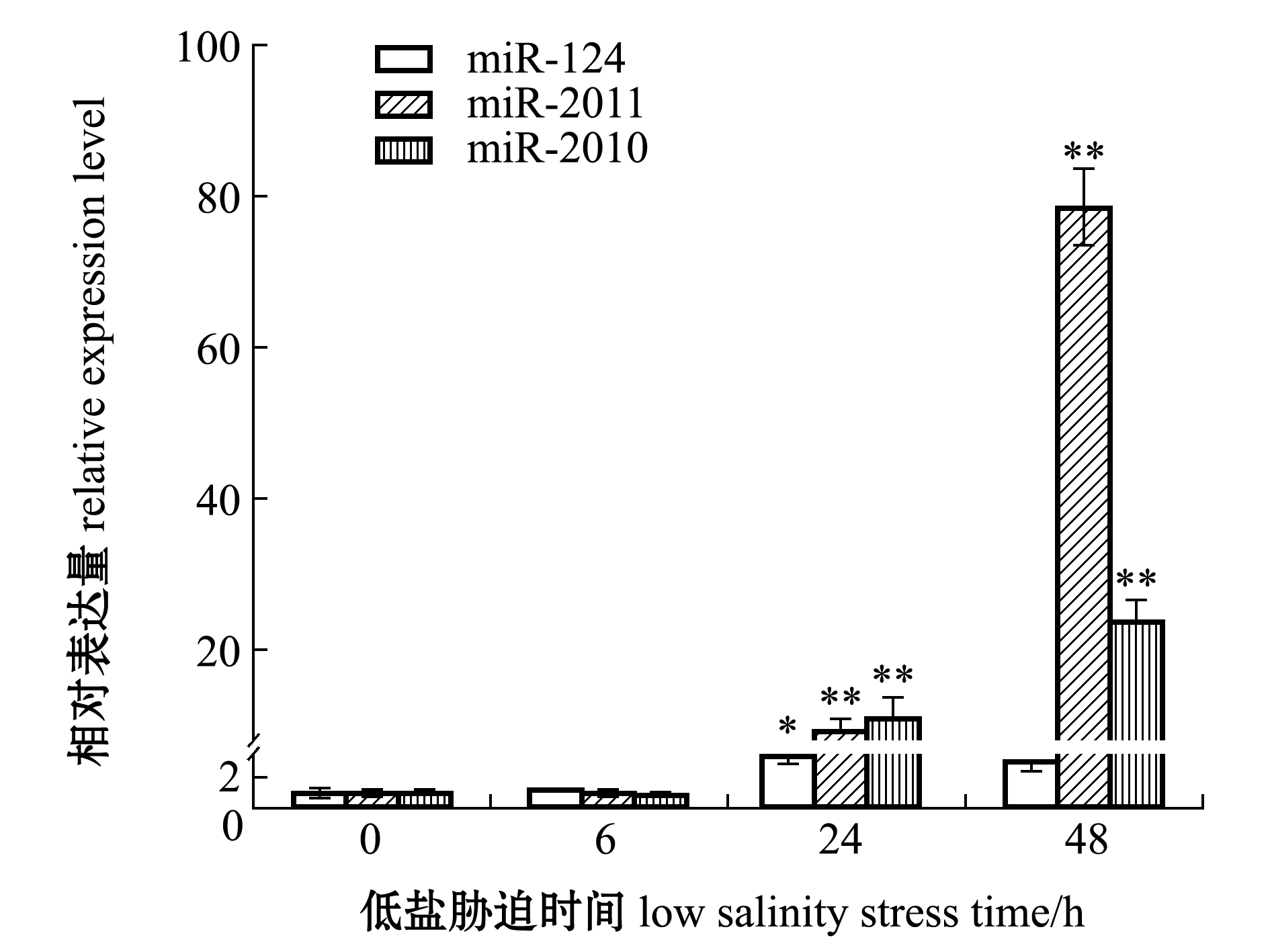
*表示与对照组(0 h)有显著性差异(P<0.05);**表示与对照组有极显著性差异(P<0.01)。
*means significant difference compared with the control(P<0.05);**means very significant difference compared with the control(P<0.01).
图6 不同胁迫时间下miR-124、miR-2011和miR-2010的相对表达量
Fig.6 Relative expression levels of miR-124,miR-2011 and miR-2010 under different stress time
3 讨论
3.1 低盐胁迫下离子浓度、渗透压及酶活力变化
研究表明,刺参经过盐度胁迫后体内的钠、钾、氯离子浓度、渗透压及钠钾ATP酶活力均会随之发生变化[16]。本试验中,钠离子浓度在盐度胁迫后6 h时最高,而氯离子和钾离子浓度在此时则最低,钠钾ATP酶也在6 h时开始变化并在24 h时出现最低值,渗透压在24 h时达到最低值。基于本试验结果推断,6~24 h可能是刺参盐度响应的关键时间点。前期研究结果也发现,刺参在6 h时为其盐度变化的关键时间点,这与本试验结果一致[6]。王茂林等[17]研究结果也发现,钠钾ATP酶在24 h后趋于稳定,认为刺参可能已适应了盐度胁迫的环境。本研究结果为后期试验提供了基础,为了更好地了解刺参的盐度响应过程,可以选取6、24 h作为盐度响应的重要时间节点。
3.2 刺参3个miRNA的序列分析
miRNA通过碱基互补原则切割靶基因mRNA、介导DNA甲基化、抑制翻译等方式在生物生长发育中起着调控功能[18],通过miRNA的序列分析能够说明其进化特性,进而在一定程度上有助于对miRNA功能的理解。近年来,随着高通量测序技术的不断发展,miRNA的生物信息学分析及序列特征取得了一定的进展。研究发现,大多数miRNA家族序列在发育过程中呈现出保守性的特点。如let-7、miR-4和miR-60 等在脊椎动物和无脊椎动物间高度保守,这说明它们可能识别多个位点或靶标,在不同生物中发挥相同或相似的作用[19-20]。miR-183基因簇作为在感觉器官组织中特异性表达的基因簇高度保守,对感觉器官的发育和功能具有重要作用[21-24]。在文昌鱼的研究中,通过对miR-216家族的进化特性分析也发现,miR-216家族均具有较高的保守性[25]。本试验中,miR-2010的成熟序列中存在较多的保守区,miR-124家族成员也在26~44、58~76碱基位点有两个保守区域,这说明miR-2010家族在生物进化过程中高度保守(图3)。这种序列的高度保守性,推测可能是与物种进化过程中生物体基本的共同功能有关。高度的保守性被认为与其功能的重要性有着密切关系,同时也为生物早期进化的同源性提供了某种证据[26-28]。目前,在已经发现的miRNA家族中,miRNA前体序列中和成熟序列中均存在序列的差异。本研究中,miR-124和miR-2011的前体序列中存在诸多变异的区域,且不同miRNA家族中其序列的保守程度存在不一致(图3)。研究认为,这可能是与物种在长期自然选择中出现基因缺失或基因的插入突变等有关,也可能与物种在某些方面的特异性或者特殊功能有关[29]。
3.3 3个miRNA在刺参盐度响应过程的表达分析
研究表明,miRNAs 在介导应激反应中发挥关键作用[30]。如刺参的miR-10在低盐胁迫后被诱导表达,并通过靶向基因TBC1D5参与盐度响应过程[14]。在斑马鱼中miR-8通过靶向基因nherf1进而调节Na+/H+交换器的作用,参与斑马鱼的渗透调节过程[31]。在鳗鲡中,miR-122、miR-140-3p和miR-10b-5p在盐度胁迫后均被诱导表达,并参与其盐度响应过程[10]。在罗非鱼中,miR-429作为OSTF1的调节因子直接调节OSTF1的表达,在罗非鱼的渗透调节过程中发挥重要作用[11]。本研究中,miR-124、miR-2011和miR-2010在盐度胁迫后均被诱导表达(图6),说明这3个miRNA可能参与了刺参的盐度响应过程。随胁迫时间的延长,miR-124表达量先升高后降低,而miR-2011、miR-2010表达量在胁迫各个时间点均有所上调,这3个miRNA的不同表达模式,可能说明在盐度响应过程中,需要3个miRNA不同的表达丰度来适应盐度胁迫的变化。具体的协同作用机制尚需进一步的试验来验证。
3个miRNA预测获得的靶基因PPM1L是锰离子、镁离子依赖的丝氨酸、苏氨酸蛋白磷酸酶,这些基因在物质转运、生物合成等一系列生理功能中发挥重要功能。这些靶基因与3个miRNA间在盐度胁迫响应中的互作关系是下一步研究的重点,需要更多的试验来验证其在刺参盐度响应机制中的作用。
4 结论
1)低盐胁迫下刺参体腔液中的离子浓度均发生变化,钾及氯离子浓度呈先下降后趋于稳定,而钠离子浓度呈先上升后下降的趋势,钾ATP酶活力均下降,说明刺参可能通过调节体内的离子浓度来应对外界环境中盐度的变化。
2)在miR-124、miR-2011和miR-2010的前体序列结构进化中,海参和海胆在进化树上位于同一分支,说明其序列相对保守。
3)通过预测分析发现,miR-124与EGF3、IMPA1能够结合,miR-2011与PPM1L、PBK有靶向位点,miR-2010与PPM1L能够结合,说明miR-124、miR-2011和miR-2010转录后可能通过调节靶基因EGF3、IMPA1、PPM1L、PBK的表达,从而参与刺参盐度胁迫响应过程。
[1] 蒋亚男,田燚,李晓雨,等.盐度应激下刺参4个转运相关基因的适应表达研究[J].大连海洋大学学报,2018,33(6):696-702.
JIANG Y N,TIAN Y,LI X Y,et al.Adaptative expression of four transporter-related genes in sea cucumber Apostichopus japonicus exposed to salinity stress[J].Journal of Dalian Ocean University,2018,33(6):696-702.(in Chinese)
[2] 王玉凤,杨金,胡雅潇,等.盐度对仿刺参免疫指标的影响[J].广东海洋大学学报,2020,40(3):22-29.
WANG Y F,YANG J,HU Y X,et al.Effects of salinities on immune-related indicators of sea cucumber(Apostichopus japonicus)[J].Journal of Guangdong Ocean University,2020,40(3):22-29.(in Chinese)
[3] 赵斌,胡炜,李成林,等.温度和盐度对紫刺参(Apostichopus japonicus Selenta)稚参存活、生长和着色的影响[J].海洋与湖沼,2018,49(3):700-706.
ZHAO B,HU W,LI C L,et al.Effects of temperature and salinity on survival,growth,and coloration of juvenile(Apostichopus japonicus Selenta)[J].Journal of Oceanology and Limnology,2018,49(3):700-706.(in Chinese)
[4] 袁秀堂,杨红生,周毅,等.盐度对刺参(Apostichopus japonicus)呼吸和排泄的影响[J].海洋与湖沼,2006,37(4):348-354.
YUAN X T,YANG H S,ZHOU Y,et al.Salinity effect on respiration and excretion of sea cucumber Apostichopus japonicus(Selenka)[J].Journal of Oceanology and Limnology,2006,37(4):348-354.(in Chinese)
[5] 王吉桥,张筱墀,姜玉声,等.盐度骤降对幼仿刺参生长、免疫指标及呼吸树组织结构的影响[J].大连水产学院学报,2009,24(5):387-392.
WANG J Q,ZHANG X C,JIANG Y S,et al.Effects of abrupt decline in salinity on growth, immunity and histological structure of respiratory trees in juvenile sea cucumber Apostichopus japonicas[J].Journal of Dalian Fisheries University,2009,24(5):387-392.(in Chinese)
[6] GENG C F,TIAN Y,SHANG Y P,et al.Effect of acute salinity stress on ion homeostasis,Na+/K+-ATPase and histological structure in sea cucumber Apostichopus japonicus[J].Springer Plus,2016,5(1):1977.
[7] YAN B,GUO J T,ZHAO L H,et al.MiR-30c:a novel regulator of salt tolerance in tilapia[J].Biochemical and Biophysical Research Communications,2012,425(2):315-320.
[8] LV J J,LIU P,GAO B Q,et al.The identification and characteristics of salinity-related microRNAs in gills of Portunus trituberculatus[J].Cell Stress and Chaperones,2016,21(1):63-74.
[9] ZHAO X L,YU H,KONG L F,et al.High throughput sequencing of small RNAs transcriptomes in two Crassostrea oysters identifies microRNAs involved in osmotic stress response[J].Scientific Reports,2016,6:22687.
[10] WANG X L,YIN D Q,LI P,et al.MicroRNA-sequence profiling reveals novel osmoregulatory microRNA expression patterns in catadromous eel Anguilla marmorata[J].PLoS One,2015,10(8):e0136383.
[11] YAN B,ZHAO L H,GUO J T,et al.miR-429 regulation of osmotic stress transcription factor 1(OSTF1)in tilapia during osmotic stress[J].Biochemical and Biophysical Research Communications, 2012,426(3):294-298.
[12] 蒋亚男.3个microRNA及靶基因在刺参盐度适应中的作用[D].大连:大连海洋大学,2019.
JIANG Y N.Analysis of 3 microRNAs and target genes in salinity adaptation of sea cucumber[D].Dalian:Dalian Ocean University, 2019.(in Chinese)
[13] 郭然,吴玮杰,商艳鹏,等.Aja-miR-22和Aja-miR-27及其靶基因在刺参盐度胁迫后的表达模式[J].中国水产科学,2020,27(4):375-382.
GUO R,WU W J,SHANG Y P,et al.Expression profiles of Aja-miR-22 and Aja-miR-27, and their target genes, in sea cucumber under salinity stress[J].Journal of Fishery Sciences of China, 2020,27(4):375-382.(in Chinese)
[14] TIAN Y,SHANG Y P,GUO R,et al.miR-10 involved in salinity-induced stress responses and targets TBC1D5 in the sea cucumber,Apostichopus japonicas[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2020,242:110406.
[15] HUO D,SUN L N,LI X N,et al.Differential expression of miRNAs in the respiratory tree of the sea cucumber Apostichopus japonicus under hypoxia stress[J].G3(Bethesda),2017,7(11):3681-3692.
[16] 庚宸帆.刺参盐度调节相关基因的定量表达及组织学分析[D].大连:大连海洋大学,2016.
GENG C F.Analysis on salinity related genes expression and histological structure of Apostichopus japonicus[D].Dalian:Dalian Ocean University,2016.(in Chinese)
[17] 王茂林,李岑,杨敏,等.盐度对仿刺参存活和Na+-K+-ATP酶活性的影响[J].渔业现代化,2014,41(1):6-9.
WANG M L,LI C,YANG M,et al.Effects of salinity on osmotic adjustment of sea cucumber[J].Fishery Modernization,2014,41(1):6-9.(in Chinese)
[18] BARTEL D P.MicroRNAs:genomics,biogenesis,mechanism,and function[J].Cell,2004,116(2):281-297.
[19] LAGOS-QUINTANA M,RAUHUT R,YALCIN A,et al.Identification of tissue-specific microRNAs from mouse[J].Current Biology,2002,12(9):735-739.
[20] LEE Y,JEON K,LEE J T,et al.MicroRNA maturation:stepwise processing and subcellular localization[J].The EMBO Journal,2002,21(17):4663-4670.
[21] XU S B,WITMER P D,LUMAYAG S,et al.MicroRNA(miRNA)transcriptome of mouse retina and identification of a sensory organ-specific miRNA cluster[J].Journal of Biological Chemistry,2007,282(34):25053-25066.
[22] KLOOSTERMAN W P,WIENHOLDS E,De BRUIJN E,et al.In situ detection of miRNAs in animal embryos using LNA-modified oligonucleotide probes[J].Nature Methods,2006,3(1):27-29.
[23] WESTON M D,PIERCE M L,ROCHA-SANCHEZ S,et al.MicroRNA gene expression in the mouse inner ear[J].Brain Research,2006,1111(1):95-104.
[24] WIENHOLDS E,KLOOSTERMAN W P,MISKA E,et al.MicroRNA expression in zebrafish embryonic development[J].Science,2005,309(5732):310-311.
[25] 张群.文昌鱼miR-183和miR-216基因簇的表达模式和保守性研究[D].青岛:中国海洋大学,2012.
ZHANG Q.The expression pattern, conservation of miR-183 and miR-216 micorRNA cluster in amphioxus(Branchiostoma belcheri Tsingtauense)[D].Qingdao:Ocean University of China,2012.(in Chinese)
[26] LAGOS-QUINTANA M,RAUHUT R,MEYER J,et al.New microRNAs from mouse and human[J].RNA,2003,9(2):175-179.
[27] LAU N C,LIM L P,WEINSTEIN E G,et al.An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans[J].Science,2001,294(5543):858-862.
[28] LEE R C,AMBROS V.An extensive class of small RNAs in Caenorhabditis elegans[J].Science,2001,294(5543):862-864.
[29] HOUBAVIY H B,DENNIS L,JAENISCH R,et al.Characterization of a highly variable eutherian microRNA gene[J].RNA,2005,11(8):1245-1257.
[30] LEUNG A K L,CALABRESE J M,SHARP P A.Quantitative analysis of Argonaute protein reveals microRNA-dependent localization to stress granules[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(48):18125-18130.
[31] BIGGAR K K,STOREY K B.Insight into post-transcriptional gene regulation:stress-responsive microRNAs and their role in the environmental stress survival of tolerant animals[J].The Journal of Experimental Biology,2015,218(9):1281-1289.