Scara3(scavenger receptor class a member 3)是清道夫受体家族的一员,该基因编码巨噬细胞清道夫受体样蛋白[1]。该蛋白已被证明能消耗活性氧且氧化应激能诱导Scara3的表达,因此,Scara3也被称为细胞应激反应蛋白(CSR)[2]。1998年,从人类胎儿脑中克隆得到Scara3全长基因,并确定该基因位于8p21染色体[3]。人Scara3 cDNA编码606个氨基酸,含有3个不同结构域,即胶原(Collagen)结构域、α-螺旋线圈结构域和跨膜区,其N端跨膜区与4个亮氨酸拉链重叠,还有多个磷酸化和糖基化位点[3-4]。对哺乳动物研究表明,Scara3与脂质代谢有关,通过与聚阴离子配体结合,有助于树突细胞和巨噬细胞获取脂肪酸[4-5]。Scara3与骨髓瘤[1]、手足口病[6]、肥胖症[7]、肝癌[8]和前列腺癌[9]等的诊断和治疗有关。对肝癌研究发现,Scara3导致HPIP蛋白失活并抑制PI3K/AKT通路的激活,从而抑制细胞增殖、迁移和侵袭[8]。对前列腺癌研究发现,Scara3的C末端可与裂解和聚腺苷酸化特异性因子3(CPSF3)结合,抑制CPSF3活性和成熟的mRNA,诱导细胞死亡[9]。在人类癌症中,Scara3可由紫外线、X光照射或活性氧物质诱导,在诱导时以高亲和力结合XIAP,干扰XIAP与半胱天冬酶的相互作用,抑制其活性,从而导致Scara3表达的细胞凋亡[10]。
目前,已鉴定出Scara3的硬骨鱼类有大黄鱼Larimichthys crocea [11]、鲤Cyprinus carpio[12]、大鳞大马哈鱼Oncorhynchus tshawytscha[13]和虹鳟O.mykiss[14],其中,大黄鱼被溶藻弧菌感染后Scara3表达上调,鲤在整个发育阶段Scara3表现出高表达且被嗜水气单胞菌感染后表达显著上调,Scara3作为大鳞大马哈鱼CHSS细胞表面受体促进其与dsRNA结合介导的先天免疫,Scara3在虹鳟上皮细胞系结合LDL和dsRNA(Poly I:C)。这些研究均表明,Scara3在鱼类的机体免疫中具有重要作用。
目前,尼罗罗非鱼已成为重要的养殖经济水产品,2019年中国罗非鱼产量约占全球总产量的四分之一,位居全球第一,国际市场需求量也逐年增加[15]。罗非鱼蛋白质含量高,必需氨基酸种类丰富,是营养全面的优质蛋白来源。近年来,由于链球菌病的暴发,造成了罗非鱼较高的死亡率,并严重威胁到罗非鱼产业的发展[16]。Scara3作为一种重要的模式识别受体(PRRs),在机体免疫中发挥重要功能,但有关罗非鱼Scara3基因研究目前尚未见报道。为了更好地控制罗非鱼疾病,本研究中通过对尼罗罗非鱼Scara3基因的克隆、序列比对和系统进化分析,比较该基因在健康尼罗罗非鱼组织,以及在细菌和病毒感染后的表达情况,并进行亚细胞定位分析,以期为探究Scara3在尼罗罗非鱼病菌防控及免疫反应中的作用提供参考。
1 材料与方法
1.1 材料
试验用尼罗罗非鱼购自广东省湛江市某鱼类养殖场,体质量为(100±10)g。在广东省水产经济动物病原生物学及流行病学重点实验室(28±2)℃、24 h循环水系统中暂养备用。
试验试剂:克隆感受态细胞Trans1-T1,以及RNA提取试剂盒(TransZol Up Plus RNA)、cDNA合成试剂盒、qRT-PCR试剂盒(TransStart Green qPCR SuperMix)均购自北京全式金生物技术有限公司;Primer STAR Max、克隆质粒 pMD18-T载体、HindIII、XhoI限制性内切酶和反转录试剂盒(Reverse Transcriptase M-MLV)均购自TaKaRa公司(大连);PCR 产物纯化试剂盒和RNAlater购自Thermo公司。HEK-293T来自广东省水产经济动物病原生物学及流行病学重点实验室。引物由生工生物工程(上海)股份有限公司广州分公司合成。
1.2 方法
1.2.1 引物设计 根据本实验室的罗非鱼转录组数据,预测尼罗罗非鱼Scara3基因序列,利用Premier 5.0软件设计Scara3引物,并根据Scara3的开放阅读框(ORF)区设计荧光定量PCR和亚细胞定位特异性引物。试验所用引物见表1。
表1 试验引物
Tab.1 Primers used in the experiment

引物 primer序列 sequence(5′-3′)Scara3-475FACAAGTTAGCAACACTGCAGACGScara3-2380RCGTACGAGACAAATTACATCTGACTqRT-Scara3-742FCGGGTTCTGAGTGCTATCAAGTCqRT-Scara3-935RTTCTCCTCGTGTTCATCCGTGβ-actin-SAACAACCACACACCACACATTTCβ-actin-ATGTCTCCTTCATCGTTCCAGTTTScara3-XhoI-FCTCGAGATGAAAGACAACTATGGScara3-HindIII-RCGACTGCAGAATTCGAAGCTT
1.2.2 尼罗罗非鱼总RNA的提取及cDNA合成 尼罗罗非鱼在实验室暂养4周后,取健康罗非鱼6尾,收集其血液、头肾、脑、胸腺、鳃、脾脏、肝脏、皮肤、肠道和肌肉共10个组织样品各10~20 mg,用液氮速冻后置于-80 ℃冰箱中保存备用。
选取规格较大的罗非鱼,取头肾组织,分离尼罗罗非鱼头肾白细胞后立即开始刺激,将细胞分为4组,第1组添加终浓度为5 μg/mL的无乳链球菌Streptococcus agalactiae,第2组添加终浓度为5 μg/mL的脂多糖(LPS),第3组添加终浓度为5 μg/mL的聚肌胞苷酸(Poly I:C),第4组添加等量磷酸盐缓冲液(PBS)作为对照组,每组设3个平行。分别在刺激后 6、12、24、48 h 时收集细胞培养液于1.5 mL离心管中,离心后弃上清液;加入1 mL Trizol UP,吹打混匀后,于-80 ℃下保存备用。
取健康尼罗罗非鱼6尾,2个试验组分别通过腹腔注射浓度为1×107 cfu/mL的无乳链球菌0.1 mL和浓度为0.2 mg/mL的Poly I:C 0.1 mL,对照组注射灭菌的PBS 0.1 mL,每组设3个平行。分别在感染后6、12、24、48、72、96 h时,取其肠道、脾脏和头肾组织各10~20 mg,迅速置入2.0 mL去RNA离心管中,于RNAlater中浸泡,用液氮速冻后于-80 ℃冰箱中保存备用。
按照RNA提取试剂盒(TransZol Up Plus RNA)说明书提取罗非鱼各组织RNA,参照反转录试剂盒(Reverse Transcriptase M-MLV)说明书进行反转录,再按照cDNA合成试剂盒(TransScript First-Strand cDNA Synthesis super mix)说明书合成cDNA。
1.2.3 尼罗罗非鱼Scara3基因克隆 以罗非鱼肠道cDNA为模板,利用引物Scara3-475F与Scara3-2380R(表1)对罗非鱼Scara3基因的编码序列片段进行扩增。PCR反应体系(20 μL):5×Primer STAR Max(Premix)10 μL,cDNA 模板(肠道组织)0.5 μL,Scara3基因上、下游引物各1 μL,ddH2O 7.5 μL。PCR反应条件:94 ℃下预变性5 min;94 ℃下循环变性15 s,55 ℃下退火复性30 s,72 ℃下延伸1 min 50 s,共进行33个循环;最后在72 ℃下再延伸5 min,4 ℃下保存。反应结束后,将PCR产物用凝胶电泳检测,获得单一目的条带大小的片段后,扩大扩增体系,用电泳检测,PCR产物纯化回收后连接pMD-18T载体,然后转化到Trans1-T1感受态细胞中,涂板挑取单菌落进行菌落PCR检测,挑取阳性菌送生工生物工程(上海)股份有限公司广州分公司进行测序。
1.2.4 生物信息学分析 利用NCBI的ORFfinder查找ORF,采用PSITE进行蛋白功能位点分析,采用ExPASy进行蛋白理化性质及亲疏水性分析,采用Phobius和SMART分别进行氨基酸序列的跨膜结构域分析和结构域预测,采用SOPMA和SWISS-MODEL分别预测蛋白的二级结构和三级结构,并用PyMOL软件进行分析,采用ClustalX和Genedoc软件进行多序列比对分析,采用MEGA 6软件构建系统进化树。
1.2.5 亚细胞定位试验 构建 pEGFP-N1-Scara3真核表达载体,限制性酶切位点为XhoI和HindIII。真核表达去内毒素质粒提取参见E.Z.N.A.Endo-Free Plasmid DNA Mini Kit说明书。
将HEK-293T细胞接种到24孔板,培养18 h后分别转染空质粒pEGFP-N1和重组质粒pEGFP-N1-Scara3。孵育48 h 后,经 PBS 洗涤2次的细胞采用体积分数 4%的多聚甲醛固定,用PBS洗涤2次。滴加1 μg/mL 4’,6-二脒基-2-苯基吲哚(4’,6-diamidino-2-phenylindole,DAPI)进行细胞核染色。最后用PBS 清洗 2次,加入抗荧光猝灭封片剂封片,于激光共聚焦显微镜下观察拍照。
1.2.6 实时荧光定量分析 根据尼罗罗非鱼Scara3序列的保守区设计特异性引物(表1),以β-actin作为内参基因,每个样本设置3个平行复孔。用qRT-PCR分析Scara3在健康尼罗罗非鱼各组织的分布,以及经不同刺激物刺激头肾白细胞后的相对表达量和经无乳链球菌和PolyI:C感染后的肠道、头肾和脾脏组织表达情况。
PCR反应体系(10 μL):Super Mix 5 μL,cDNA 模板(原始模板稀释10倍)0.3 μL,正、反向引物各0.2 μL,ddH2O 4.3 μL。PCR反应条件:95 ℃下预变性300 s;95 ℃下循环变性10 s,60 ℃下退火复性15 s,72 ℃下延伸15 s,共进行40个循环。反应结束后用2-△△Ct法计算基因的相对表达量,组织表达以肝脏为基线(1.0),病原微生物刺激后的基因表达以对照组为基线(1.0)。使用 GraphPad Prism进行数据分析及图表处理。
2 结果与分析
2.1 尼罗罗非鱼Scara3基因的克隆及生物信息学分析
2.1.1 Scara3基因克隆及蛋白理化性质预测 克隆得到尼罗罗非鱼Scara3基因(GenBank登录号:XM_005475068.4)cDNA全长3 889 bp,ORF长度为1 827 bp,可编码608个氨基酸(图1)。预测编码的Scara3蛋白理论相对分子质量为66 740,等电点为6.69,分子式为C2883H4622N852O922S24,其中,甘氨酸(Gly)含量最高(10.2%),亮氨酸(Leu)次之(8.9%),色氨酸(Trp)含量最低(0.5%),带正电荷的氨基酸(Arg+Lys)68个,带负电荷的氨基酸(Asp+Glu)71个。该蛋白的不稳定指数为35.22,归为不稳定蛋白;该蛋白的脂溶指数(aliphatic index)为73.27,平均亲水值(GRAVY)为-0.660,属亲水性蛋白。用SoftBerry-Psite预测该蛋白的功能位点,表明其氨基酸序列具有11个N-糖基化位点、9个酪蛋白激酶Ⅱ磷酸化位点、9个微体细胞C端靶信号、7个蛋白激酶C磷酸化位点、5个N-豆蔻酰化位点、1个酪氨酸激酶磷酸化位点、1个cAMP和cGMP依赖的蛋白激酶磷酸化位点和2个亮氨酸拉链。通过SMART分析保守结构域,发现其存在跨膜结构域、螺旋结构域和胶原(Collagen)结构域。

* 终止子 terminator。
图1 罗非鱼Scara3基因核苷酸序列及推导的氨基酸序列
Fig.1 Nucleotide sequence and deduced amino acid sequences of Scara3 gene in Nile tilapia Oreochromis niloticus
2.1.2 蛋白的二级结构和三维结构预测 分析发现,Scara3蛋白的二级结构中,由206个氨基酸组成无规则卷曲,占33.88%;由386个氨基酸组成α-螺旋,占63.49%;由11个氨基酸组成β折叠结构,占1.81%;由5个氨基酸组成延伸链,占0.82%。该蛋白的二级结构中有超过半数的氨基酸参与形成α-螺旋结构,说明该蛋白整体处于一个以α-螺旋为主的二级结构中。利用SWISS-MODLE对Scara3进行同源建模,然后使用PyMOL进行3D结构可视化,并和人的Scara3三维结构图进行比较,发现罗非鱼与哺乳动物的Scara3蛋白三维结构相似(图2)。
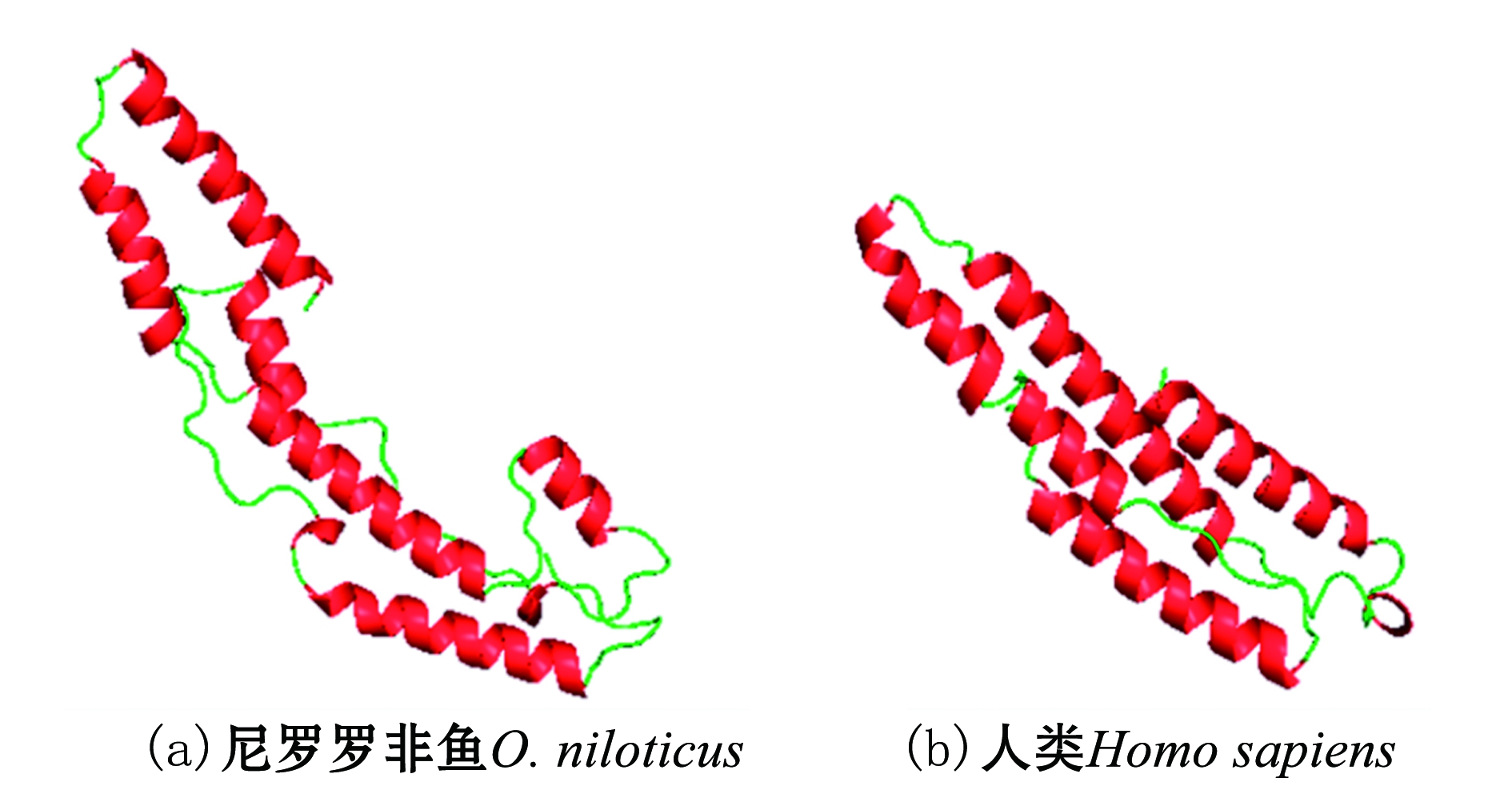
图2 尼罗罗非鱼与人类Scara3蛋白的三维结构
Fig.2 Three-dimensional structures of Scara3 in Nile tilapia Oreochromis niloticus and Homo sapiens
2.1.3 氨基酸序列的同源性及系统进化分析 采用ClustalX软件将尼罗罗非鱼Scara3与其他物种进行多序列比对,结果显示,尼罗罗非鱼与大黄鱼、鲤、河鲈、大鼠及人类的Scara3氨基酸序列一致性分别为79.87%、67.38%、81.59%、47.54%、47.01%,其中与人类的同源性最低(图3)。利用MEGA 6软件构建尼罗罗非鱼与其他物种Scara3氨基酸序列的系统进化树,包括人类、家鼠、斑马鱼、罗非鱼、鹦鹉、矛隼、蜥蜴、蛙等19个物种,结果显示,尼罗罗非鱼与其他多数硬骨鱼类聚为一支,而鸟类、爬虫类、两栖动物和哺乳动物各聚为一支(图4)。

图3 尼罗罗非鱼与其他物种Scara3氨基酸的多重序列比对
Fig.3 Comparison of multi-alignment of Scara3 amino acid sequence of Nile tilapia Oreochromis niloticus with other species
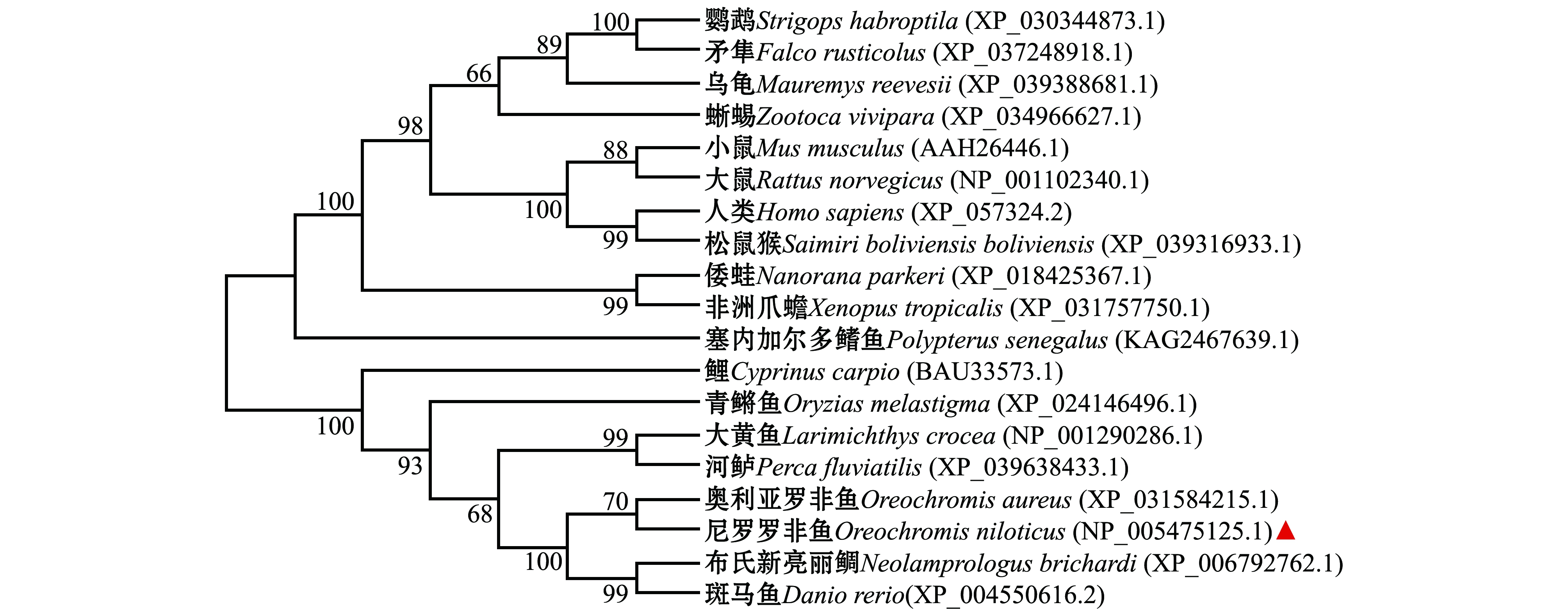
图4 尼罗罗非鱼与其他物种Scara3氨基酸序列的NJ系统进化树
Fig.4 NJ Phylogenetic tree of Scara3 amino acid sequences of Nile tilapia Oreochromis niloticus and other species
2.2 Scara3蛋白的亚细胞定位
构建真核表达重组质粒pEGFP-N1-Scara3,通过在HEK-293T细胞中表达pEGFP-N1-Scara3质粒,产生该质粒的融合蛋白,确定Scara3蛋白的亚细胞定位,结果发现,Scara3蛋白在HEK-293T细胞的全细胞中均分布有绿色荧光,这表明Scara3蛋白在全细胞中表达(图5)。
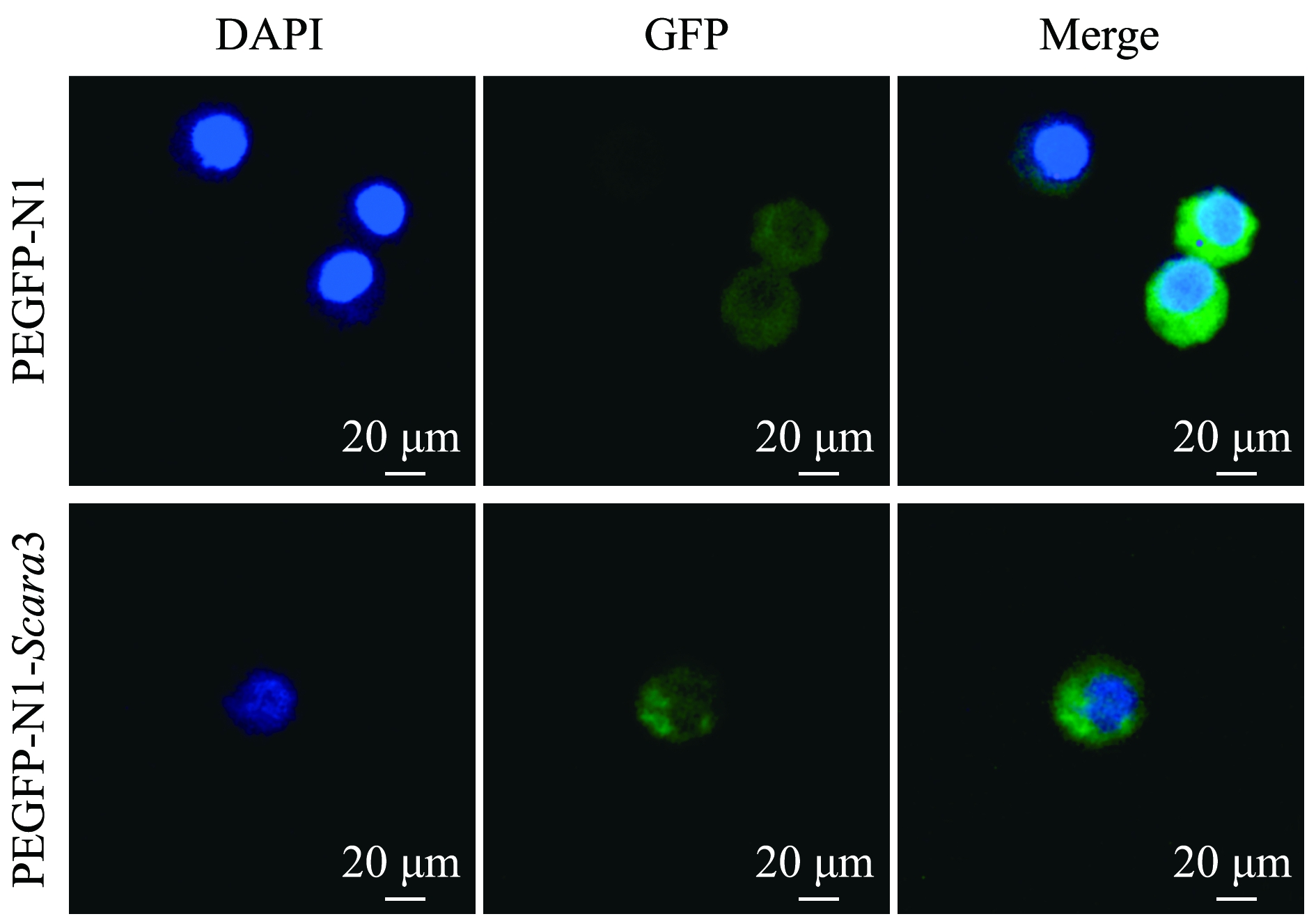
图5 Scara3蛋白在HEK-293T中的亚细胞定位
Fig.5 Subcellular localization of Scara3 in HEK-293T cells by fluorescence microscopy
2.3 尼罗罗非鱼Scara3基因的组织分布
通过罗氏LightCycler96定量PCR仪进行逆转录聚合酶链反应(qRT-PCR)分析,检测健康尼罗罗非鱼各个组织中Scara3基因的表达量,结果表明,Scara3基因在罗非鱼各个组织中均能广泛表达,在血液组织中表达量最高(P<0.05),其次是肠道、皮肤、胸腺、脑、肝脏和肌肉,在脾脏、鳃和头肾中表达量较少(P<0.05)(图6)。
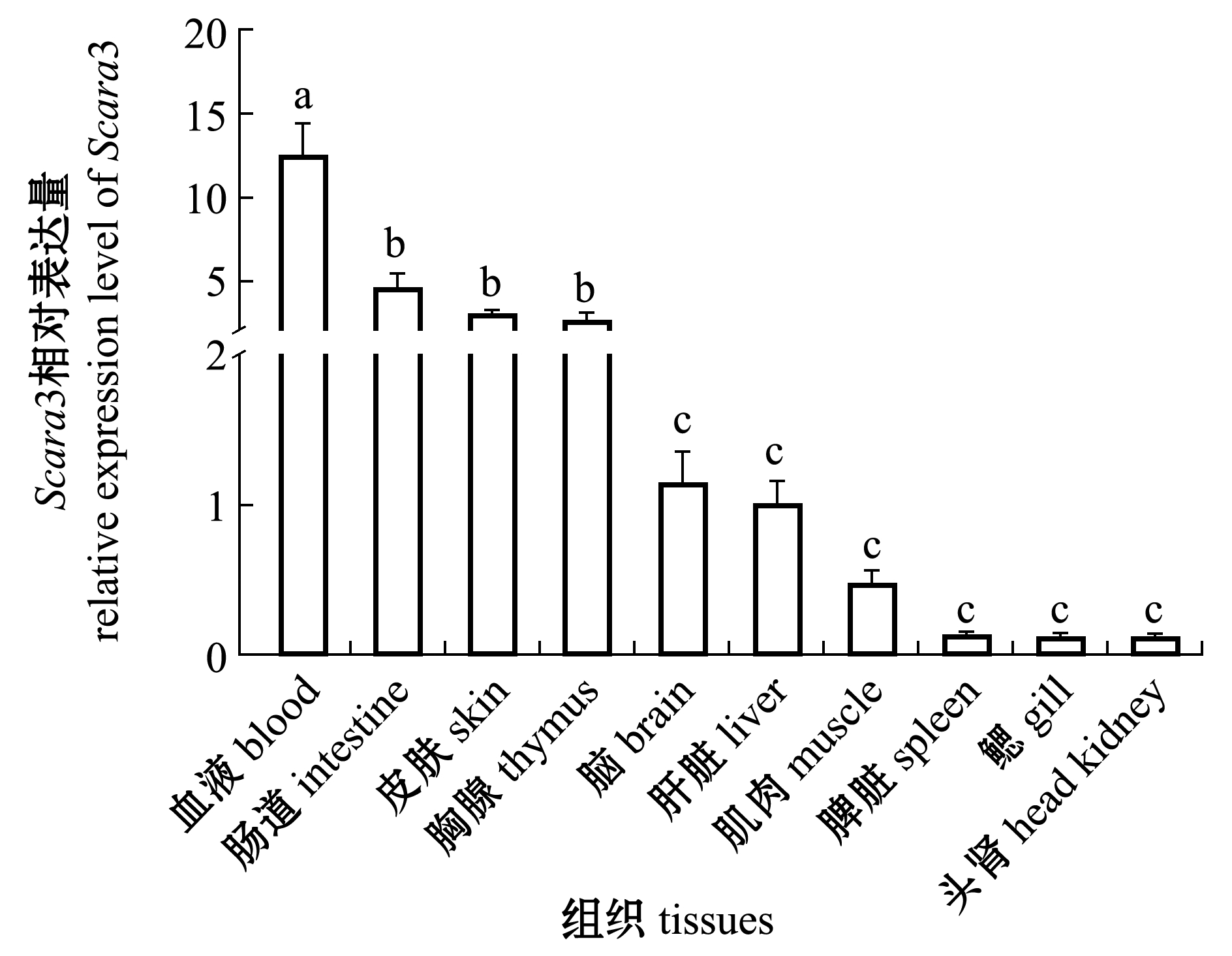
标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。The means with different letters are significantly different in the groups at the 0.05 probability level,and the means with the same letter are not significant differences.
图6 Scara3在不同组织中的表达量
Fig.6 Expression level of Scara3 in different tissues
2.4 不同刺激物刺激罗非鱼头肾白细胞后Scara3的表达模式
采用Percoll分离法分离获得罗非鱼头肾白细胞,采用 LPS、Poly I:C和灭活后的无乳链球菌孵育细胞,然后检测6、12、24、48 h时细胞中Scara3基因的表达情况。结果显示:LPS刺激罗非鱼头肾白细胞后,Scara3在6 h时极显著上调(P<0.01),12 h后逐渐下降;Poly I:C刺激头肾白细胞后Scara3表达量呈上升趋势,在48 h时达到高峰(P<0.01);灭活的无乳链球菌刺激头肾白细胞后Scara3表达量逐渐上调,在24 h时达到峰值(P<0.01),48 h后缓慢下降(图7)。

*表示与对照组有显著性差异(P<0.05);**表示与对照组有极显著性差异(P<0.01),下同。*means significant difference compared with the control (P<0.05);**means very significant difference compared with the control(P<0.01),et sequentia.
图7 不同刺激物孵育头肾白细胞后Scara3的时序表达
Fig.7 Temporal expression of Scara3 in head kidney leukocyte of immunized Nile tilapia Oreochromis niloticus
2.5 无乳链球菌和Poly I:C刺激后Scara3的组织表达模式
经灭活的无乳链球菌感染尼罗罗非鱼后,在头肾中,Scara3基因表达量6 h时出现表达高峰(P>0.05),而后逐渐降低;在脾脏中,表达量持续上升,在24 h时达到最大值(P<0.01),而后迅速恢复到正常水平;在肠道中,刚开始刺激时表达量上调,12 h时达到最大值(P<0.01),而后恢复到正常水平(图8(a))。
经Poly I:C病毒类似物感染尼罗罗非鱼后,肠道、头肾、脾脏组织中Scara3表达量在24 h时均有极显著上调(P<0.01)(图8(b))。
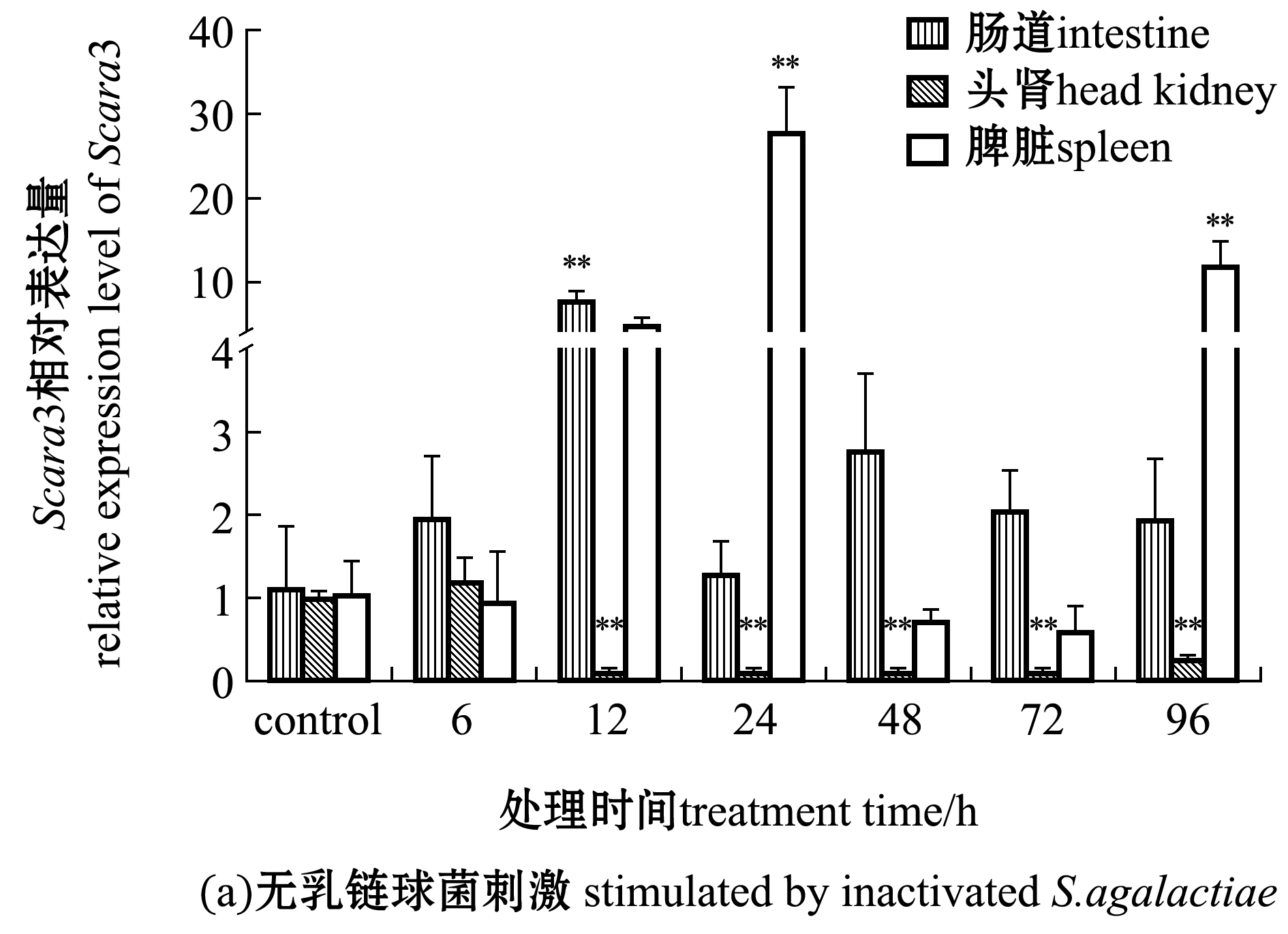
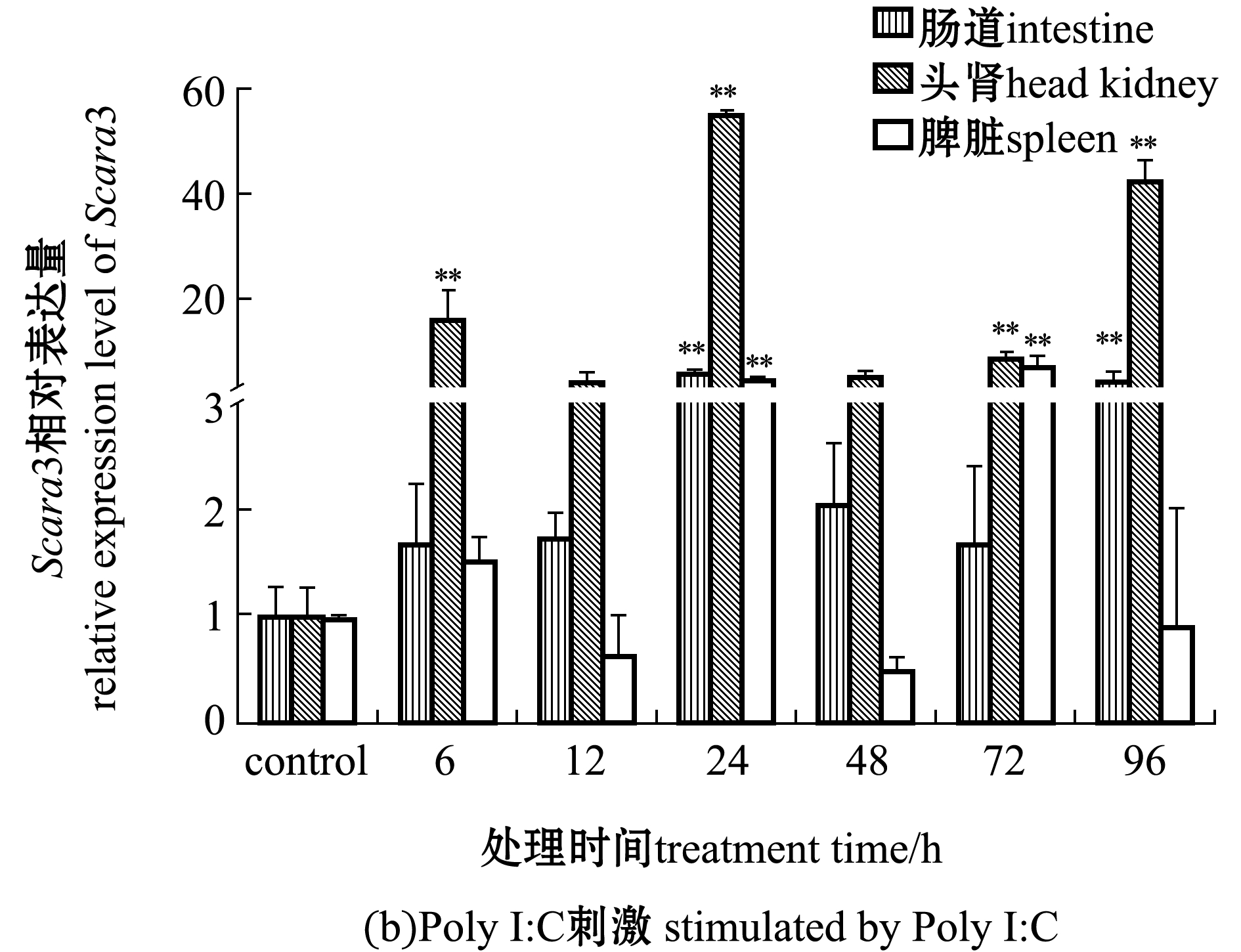
图8 灭活无乳链球菌和Poly I:C刺激后Scara3在3个组织中的时序表达
Fig.8 Temporal expression of Scara3 stimulated by inactivated Streptococcus agalactiae and Poly I:C in three tissues
3 讨论
3.1 尼罗罗非鱼Scara3基因的结构特征
在前期罗非鱼全长转录组测序结果基础上,本试验中克隆鉴定得到尼罗罗非鱼Scara3 cDNA全长序列3 889 bp,ORF区为1 827 bp,编码608个氨基酸。多序列比对结果显示,尼罗罗非鱼Scara3序列与河鲈的同源性较高(81.59%),在构建的系统进化树中,尼罗罗非鱼Scara3与奥利亚罗非鱼亲缘关系最近,聚为一支。此外,推导的Scara3氨基酸序列具有Collagen结构域(位于483 aa~547 aa),通过对比发现,Collagen结构域在不同物种中均较保守,只是长度、位置有所差异,但Collagen结构域均与受体三螺旋结构的形成相关[17]。Collagen结构域是重复的G-X-Y序列,多肽链形成一个三重螺旋,参与形成细胞外结构蛋白,可指引跨膜结构的形成[18]。重复序列中G位置总为甘氨酸,X、Y位置为任何残基,常见的三联体Gly-Pro-Hyp,胶原被脯氨酸羟化酶修饰形成羟基脯氨酸残基,羟基化缺陷可导致维生素C缺乏病[19-20]。Collagen结构域具有较高的序列及功能一致性,罗非鱼和智人的三维结构也表现出较高的相似性,说明Scara3在鱼类与哺乳类进化上高度保守。
3.2 尼罗罗非鱼Scara3基因的组织表达模式
本研究表明,Scara3在健康尼罗罗非鱼各组织及器官中均有表达,但其在血液、肠道、皮肤和肝脏等先天性免疫组织中的表达量较高,最低表达量出现在头肾中,这与对鲤[12]、虹鳟[14]的研究结果类似,但与大黄鱼[11]研究中的肾、肠表达量较高的结果相反。Scara3基因在罗非鱼、大黄鱼、鲤和虹鳟中的研究结果存在差异,可能是Scara3基因表达存在物种特异性,大菱鲆Scophthalmus maximus[21] 、大黄鱼[11]和虹鳟[14]中清道夫受体基因scara5中也存在这种现象。本研究中,Scara3在罗非鱼血液中表达量最高,可能是因为血液是鱼体的重要免疫场所,在鱼类免疫中发挥着重要的作用,血细胞是重要的免疫细胞,白细胞直接参与免疫应答及其相关的免疫应答过程[22-24],由此推测,Scara3基因参与罗非鱼的免疫应答过程。皮肤和肠道是罗非鱼重要的黏膜免疫组织,且均附着有淋巴组织,分布着大量的淋巴细胞、巨噬细胞和粒细胞等,皮肤还作为一个重要的屏障,可防止水分损失和外来分子及病原体的进入[25-26]。本研究中,Scara3基因在罗非鱼皮肤和肠道组织中的表达量较高,说明其在宿主抵御病原微生物侵袭的免疫应答过程中具有重要的作用。
3.3 3种不同刺激物刺激头肾白细胞后Scara3基因的表达特征
本研究中,通过 LPS、Poly I:C和灭活后的无乳链球菌孵育刺激尼罗罗非鱼头肾白细胞,发现LPS刺激后Scara3迅速显著上调并于6 h时达到表达高峰,无乳链球菌和Poly I:C刺激后Scara3也在发生上调,但是明显滞后,分别在24 h和48 h时才达到表达高峰,表明Scara3对LPS的刺激更为敏感[27]。LPS可以触发鱼白细胞产生炎症介质,对青斑、河豚Tetraodon nigroviridis的试验表明,清道夫受体基因SR参与清除鱼类宿主防御中的LPS识别分子[28]。无乳链球菌刺激后Scara3的表达上调,与小鼠试验中清道夫受体基因SR-A抵御金黄色葡萄球菌和其他革兰氏阳性菌结果一致[29]。对大鳞大马哈鱼试验证明,SR-A促进细胞和dsRNA的结合[13],dsRNA作为病毒的病原分子模式,Poly I:C与其结构类似[30]。综上,说明Scara3在对抗微生物感染中具有重要的免疫作用。
3.4 无乳链球菌和Poly I:C感染后Scara3基因的组织表达变化
肠道是主要的外周淋巴器官,具有免疫屏障的作用,细菌感染后通过产生抗菌蛋白来识别清除病原。本研究中,无乳链球菌感染后,罗非鱼肠道、头肾、脾脏组织中Scara3的表达量呈现先上调后下降趋势,肠道中该基因在12 h时表达量显著上升,脾脏中该基因在后一个时间段24 h时达峰值,这与鲤对嗜水气单胞菌感染表现类似[12]。以往研究也表明,无乳链球菌主要攻击的靶器官是脾脏和头肾,这主要是由于脾脏和头肾是鱼类主要的造血器官[31-32],因此,本研究中尼罗罗非鱼脾脏和头肾在无乳链球菌感染后Scara3表达量先上调后下降。这表明,Scara3可能参与罗非鱼多种免疫应答过程。本研究中,经Poly I:C病毒类似物刺激后,尼罗罗非鱼肠道、头肾和脾脏组织的Scara3表达量,在感染后的前6 h较对照组有增加,在24 h时显著上调,然后逐渐恢复正常,可能与鱼类先天抗病毒免疫有关[33],48 h下调后再次上调,可能是病原入侵后,激活细胞内抗病毒反应导致机体紊乱,Scara3的表达暂时被抑制。大鳞大马哈鱼Scara3起着dsRNA细胞表面受体的作用,结合dsRNA并介导IFN表达[13]。与大黄鱼CRFB13(IFN-y受体的亚型)头肾中表达相似[34],本研究中尼罗罗非鱼头肾Scara3表达量明显高于肠道和脾脏,头肾是免疫的关键器官,也是造血的主要部位,其中含有粒细胞、B细胞和T细胞,说明在感染病菌时,Scara3在头肾组织中表现出重要的免疫反应[35]。目前,针对Poly I:C刺激其他物种后Scara3的研究较少。本研究显示,Scara3可能在不同的免疫器官参与鱼类的抗病毒免疫应答过程,但对于病毒感染的转录意义有待进一步探索。细菌和病毒刺激后尼罗罗非鱼Scara3表达转录表现出不同的原因,反映了细菌和病毒感染所遇信号通路不同及各组织内Scara3活化的水平不同。这些结果表明,无乳链球菌感染和Poly I:C刺激均可引起Scara3基因在机体免疫反应中表达发生改变,表明Scara3参与了罗非鱼对细菌和病毒相关的免疫应答反应。
4 结论
1)本研究中成功克隆鉴定了尼罗罗非鱼Scara3的cDNA序列,预测的Scara3氨基酸序列具有与其他物种Scara3相似的结构特征。
2)Scara3在尼罗罗非鱼血液、肠道、皮肤和肝脏等先天性免疫组织中的表达量较高,表明该基因在免疫应答中起重要作用。
3)在无乳链球菌、LPS和Poly I:C刺激头肾细胞后Scara3表达量均上调。肠道、头肾和皮肤3个组织受到无乳链球菌感染和Poly I:C刺激后,表达量呈现显著变化,表明Scara3基因在罗非鱼抵御病原微生物入侵过程中有着重要作用。
[1] BROWN C O,SCHIBLER J,FITZGERALD M P,et al.Scavenger receptor class a member 3 (SCARA3)in disease progression and therapy resistance in multiple myeloma[J].Leukemia Research,2013,37(8):963-969.
[2] GUADAGNO N A,MORICONI C,LICURSI V,et al.Neuroserpin polymers cause oxidative stress in a neuronal model of the dementia FENIB[J].Neurobiology of Disease,2017,103:32-44.
[3] HAN H J,TOKINO T,NAKAMURA Y.CSR,a scavenger receptor-like protein with a protective role against cellular damage caused by UV irradiation and oxidative stress[J].Human Molecular Genetics,1998,7(6):1039-1046.
[4] BOCK A J,NYMOEN D A,BRENN E K,et al.SCARA3 mRNA is overexpressed in ovarian carcinoma compared with breast carcinoma effusions[J].Human Pathology,2012,43(5):669-674.
[5] MURPHY J E,TEDBURY P R,HOMER-VANNIASINKAM S,et al.Biochemistry and cell biology of mammalian scavenger receptors[J].Atherosclerosis,2005,182(1):1-15.
[6] TIAN Y,ZHOU K,HU J,et al.Scavenger receptor class a,member 3 is associated with severity of hand,foot,and mouth disease in a case-control study[J].Medicine,2019,98(40):e17471.
[7] PENG H,GUO Q,SU T,et al.Identification of SCARA3 with potential roles in metabolic disorders[J].Aging,2020,13(2):2149-2167.
[8] JIANG L,HU G,CHEN F F,et al.CSR1 suppresses tumor growth and metastasis of human hepatocellular carcinoma via inhibition of HPIP[J].European Review for Medical and Pharmacological Sciences,2017,21(17):3813-3820.
[9] ZHU Z H,YU Y P,SHI Y K,et al.CSR1 induces cell death through inactivation of CPSF3[J].Oncogene,2009,28(1):41-51.
[10] ZHENG Z L,TAN L Z,YU Y P,et al.Interaction of CSR1 with XIAP reverses inhibition of caspases and accelerates cell death[J].The American Journal of Pathology,2012,181(2):463-471.
[11] HE J Y,LIU H H,WU C W.Identification of SCARA3,SCARA5 and MARCO of class A scavenger receptor-like family in Pseudosciaena crocea[J].Fish &Shellfish Immunology,2014,41(2):238-249.
[12] FENG S S,JIANG Y L,ZHANG S H,et al.Genome wide identification of scavenger receptors class A in common carp (Cyprinus carpio)and their expression following Aeromonas hydrophila infection[J].Fish &Shellfish Immunology,2016,54:60-67.
[13] SEMPLE S L,VO N T K,POYNTER S J,et al.Extracellular dsRNA induces a type I interferon response mediated via class A scavenger receptors in a novel chinook salmon derived spleen cell line[J].Developmental &Comparative Immunology,2018,89:93-101.
[14] POYNTER S J,MONJO A L,DEWITTE-ORR S J.Identification of three class A scavenger receptors from rainbow trout (Oncorhynchus mykiss):SCARA3,SCARA4,and SCARA5[J].Fish &Shellfish Immunology,2018,76:121-125.
[15] 罗茵.《2020中国渔业统计年鉴》出版广东水产品总产量首次全国第一[J].海洋与渔业,2020(6):12-13.
LUO Y.“China Fishery Statistical Yearbook 2020”published that Guangdong’s total output of aquatic products ranked first in China for the first time[J].Ocean and Fishery,2020(6):12-13.(in Chinese)
[16] 苏友禄,刘婵,邓益琴,等.罗非鱼无乳链球菌病的研究进展[J].大连海洋大学学报,2019,34(5):757-766.
SU Y L,LIU C,DENG Y Q,et al.Research on Streptococcus agalactiae disease in tilapia:a review[J].Journal of Dalian Ocean University,2019,34(5):757-766.(in Chinese)
[17] HANSEN S,HOLMSKOV U.Structural aspects of collectins and receptors for collectins[J].Immunobiology,1998,199(2):165-189.
[18] BHATTACHARJEE A,BANSAL M.Collagen structure:the Madras triple helix and the current scenario[J].IUBMB Life,2005,57(3):161-172.
[19] MAYNE R,BREWTON R G.New members of the collagen superfamily[J].Current Opinion in Cell Biology,1993,5(5):883-890.
[20] WHATMOREA M.Streptococcus pyogenes sclB encodes a putative hypervariable surface protein with a collagen-like repetitive structure[J].Microbiology (Reading,England),2001,147(Pt 2):419-429.
[21] CHAO L,TIAN M Y,ZHANG L,et al.The characterization and initial immune functional analysis of SCARA5 in turbot (Scophthalmus maximus L.)[J].Fish &Shellfish Immunology,2018,81:242-249.
[22] COULIBALY I,GAHR S A,PALTI Y,et al.Genomic structure and expression of uncoupling protein 2 genes in rainbow trout (Oncorhynchus mykiss)[J].BMC Genomics,2006,7(1):203.
[23] CASTILLO A,RAZQUIN B,VILLENA A J,et al.Thymic barriers to antigen entry during the post-hatching development of the thymus of rainbow trout,Oncorhynchus mykiss[J].Fish &Shellfish Immunology,1998,8(3):157-170.
[24] DALMO R A,INGEBRIGTSEN K,BØGWALD J.Non-specific defence mechanisms in fish,with particular reference to the reticuloendothelial system (RES)[J].Journal of Fish Diseases,1997,20(4):241-273.
[25] PRESS C M,DANNEVIG B H,LANDSVER K T.Immune and enzyme histochemical phenotypes of lymphoid and nonlymphoid cells within the spleen and head kidney of Atlantic salmon (Salmo salar L.)[J].Fish &Shellfish Immunology,1994,4(2):79-93.
[26] ZWOLLO P,COLE S,BROMAGE E,et al.B cell heterogeneity in the teleost kidney:evidence for a maturation gradient from anterior to posterior kidney[J].The Journal of Immunology,2005,174(11):6608-6616.
[27] WANG Z W,XIE C X,LI Y,et al.Molecular characterization and expression of CD48 in Nile tilapia (Oreochromis niloticus)in response to different stimulus[J].Fish &Shellfish Immunology,2020,97:515-522.
[28] MENG Z,ZHANG X Y,GUO J,et al.Scavenger receptor in fish is a lipopolysaccharide recognition molecule involved in negative regulation of NF-κB activation by competing with TNF receptor-associated factor 2 recruitment into the TNF-α signaling pathway[J].The Journal of Immunology,2012,189(8):4024-4039.
[29] SUZUKI H,KURIHARA Y,TAKEYA M,et al.A role for macrophage scavenger receptors in atherosclerosis and susceptibility to infection[J].Nature,1997,386(6622):292-296.
[30] DEWITTE-ORR S J,MOSSMAN K L.dsRNA and the innate antiviral immune response[J].Future Virology,2010,5(3):325-341.
[31] LI Y W,LIU L,HUANG P R,et al.Chronic streptococcosis in Nile tilapia(Oreochromis niloticus)(L.),caused by Streptococcus agalactiae[J].Journal of Fish Diseases,2014,37(8):757-763.
[32] ZHU J J,LI C,AO Q W,et al.Trancriptomic profiling revealed the signatures of acute immune response in tilapia (Oreochromis niloticus)following Streptococcus iniae challenge[J].Fish &Shellfish Immunology,2015,46(2):346-353.
[33] MUGUE N,TEREKHANOVA N,AFANASYEV S,et al.Transcriptome sequencing of hybrid bester sturgeon:responses to poly (I:C)in the context of comparative immunogenomics[J].Fish &Shellfish Immunology,2019,93:888-894.
[34] 杨思司,母尹楠,丁连帅,等.大黄鱼(Larimichthys crocea)CRFB13基因的克隆与表达分析[J].应用海洋学学报,2018,37(1):60-67.
YANG S S,MU Y N,DING L S,et al.Cloning of large yellow croaker (Larimichthys crocea)CRFB13 gene and expression analysis[J].Journal of Applied Oceanography,2018,37(1):60-67.(in Chinese)
[35] GAN Z,WANG B,LU Y S,et al.Molecular characterization and expression of CD2BP2 in Nile tilapia (Oreochromis niloticus)in response to Streptococcus agalactiae stimulus[J].Gene,2014,548(1):126-133.