生长激素释放激素(growth hormone-releasing hormone,GHRH)由下丘脑合成分泌[1],广泛分布于中枢神经系统。生长激素释放激素不仅具有调节生长激素的释放和调节神经的作用,而且能促进动物的生长发育,参与免疫系统调节、细胞分化、细胞增殖等生物学过程[2-4]。在GHRH基因研究中,有关SNP位点与生长性状的关联分析在畜牧中报道较多。张存芳[5]发现,GHRH基因中有3个位于非编码区的SNP位点与南阳牛Nanyang cattle 的体质量、体斜长等生长性状相关。Cheong等[6]在韩国本土牛GHRH基因中发现了1个与胴体相关的SNPs位点。水产动物GHRH基因的SNP位点与生长性状的关联分析也有少量报道,如Guo等[7]在点带石斑鱼 Epinephelus coioides 的GHRH基因中筛选到2个与生长性状相关的SNP位点。
单核苷酸多态性(single nucleotide polymorphism,SNP)是指基因组中特定位置的单个核苷酸发生碱基突变引起的DNA序列的多态性[8-9]。SNP优势在于其一般只由两种碱基构成,因此,可快速、规模化地进行筛选[10-11]。目前,SNP在水产领域也有较多应用,如构建遗传图谱或关联分析。相关研究中,张建勇[12]根据对中国对虾 Fenneropenaeus chinensis 的高通量测序结果,设计了800组SNP引物,构建了中国对虾亲本遗传连锁图谱,其中,父本覆盖率为 51.94%,母本覆盖率为53.77%。樊佳佳等[13]在草鱼 Ctenopharyngodon idella柠檬酸合酶基因上筛选出2个SNPs位点,将其与草鱼的6个生长性状进行关联分析,结果显示,两位点不同基因型在6个性状的平均值上有差异但不显著,又将其组成7种双倍型,其中,D6属生长优势双倍型。
红鳍东方鲀 Takifugu rubripes在分类学上隶属于硬骨鱼纲Osteichthyes鲀形目Tetraodontiformes鲀亚目 Tetraodontoidei鲀总科Tetraodntoidea鲀科 Tetraodontidae东方鲀属Takifugu,在中国主要分布于东海、黄海,国外则主要分布于日本、韩国、朝鲜等沿海地区,属暖水性海洋底栖鱼类[14]。红鳍东方鲀味道鲜、营养高、肉质嫩,富含多种维生素,经济价值较高[15-16],是中国北方重要的经济鱼类之一。目前,国内养殖红鳍东方鲀一直源自日本红鳍东方鲀及其后代,长期繁育有可能导致该鱼生长缓慢、易患疾病,一定程度上影响了红鳍东方鲀养殖业的发展[17-19]。因此,开展红鳍东方鲀的选育研究,探索分子标记辅助育种技术势在必行。本研究中,挑选红鳍东方鲀与生长相关的GHRH基因作为候选基因,利用PCR直接测序技术筛选GHRH基因上的SNPs,分析SNPs与红鳍东方鲀生长性状的相关性,以期为红鳍东方鲀分子标记辅助育种提供科学参考。
1 材料与方法
1.1 材料
试验用红鳍东方鲀为本研究团队在河北省唐山市天正实业有限公司培育的红鳍东方鲀群体,从中随机挑选219尾个体,分别于该群体6月龄及12月龄时,参照王新安等[20]的方法测量其体长(BL)、体高(BD)、头长(HL)、尾柄长(CPL)、尾柄高(CPD)、尾柄宽(CPW)、眼间距(IS)、体宽(BW)、头长+躯干长(HL+TR)和体质量(BWH)共10个生长相关性状,并取尾鳍用于提取基因组DNA。
1.2 方法
1.2.1 引物设计及合成 参考红鳍东方鲀GHRH基因序列(GenBank登录号:101063685),利用Primer Premier 5.0软件,针对GHRH基因的6个外显子和5个内含子,共设计4对引物,覆盖了GHRH整段基因(表1)。引物由生工生物工程(上海)股份有限公司合成。
1.2.2 DNA提取及PCR扩增 按照天根海洋动物组织基因组DNA提取试剂盒说明书,以尾鳍为材料提取基因组DNA,用15 g/L琼脂糖凝胶电泳检测DNA质量。
PCR总反应体系(共25 μL):10×PCR buffer 2.5 μL,dNTP M Mixture 2 μL,上、下游引物各1 μL,DNA 0.5 μL,TaKaRa Taq 0.3 μL,用ddH2O补足至25 μL。
PCR反应程序为:94 ℃下预变性30 min;94 ℃下循环变性1 min,退火复性30 s,72 ℃下延伸1 min,共进行35个循环;最后在72 ℃下再延伸7 min,4 ℃下保存。退火温度见表1。从红鳍东方鲀样本中随机选取10尾鱼的基因组用于PCR扩增,扩增后进行PCR产物的分离纯化,然后经15 g/L琼脂糖凝胶电泳鉴定后,送生工生物工程(上海)股份有限公司进行测序。
表1 红鳍东方鲀GHRH基因的引物序列
Tab.1 Primers of GHRH gene in tiger puffer Takifugu rubripes

引物primer序列sequence(5′-3′)产物长度/bpproduct length退火温度/℃annealing temperatureG1-FCCCCTGTGTGAGTTGGAAGT50465G1-RTTGCGGTATGTGTGTCTGGAG2-FCGATGAGGAGGCTTGAGAAG59863G2-RGCCTCCATCATGTAGCACTGG3-FTGCTACATGATGGAGGCTTG52563G3-RAATCACCATCTACTCACAATCCAG4-FTGTGACGGAGGATGAGATGA123965G4-RCTGTGCTGTTGCTCAATGCT
1.2.3 SNP分型及统计分析 试验结果均以平均值±标准差(mean±S.D.)表示,利用直接测序法对序列进行分析,结合Chromas和DNAman 8软件查看测序结果峰图,查看已知位点的基因型,确定所需的SNP位点,并扩群测序、分型。利用SeqMan软件对全部基因型进行统计。用Excel 2019对数据进行整理。采用 SPSS 22 软件一般线性模型中的多元方差分析模块进行关联分析,因变量为形态性状,自变量为筛选到的SNPs位点的不同基因型,显著相关性水平设为0.05,极显著相关性水平设为0.01。
连锁不平衡是给定群体中不同位点等位基因的非随机关联现象[24],利用Hapioview 4.1软件,对筛选出的GHRH基因SNPs位点进行连锁不平衡分析,结果由 r2和D′表示,其中 r2表示重组和突变的程度,D′表示重组程度。当D′>0.8且r2>0.33时,两位点为完全连锁状态,此时两个位点将作为一个整体进行遗传,若只满足一个条件,则为连锁状态[25]。用Excel 2019的TTEST、AVERAGE和STDEV.P公式进行双倍型分析。
2 结果与分析
2.1 GHRH基因的PCR扩增结果
将设计好的4对引物均进行PCR试验,扩增目的片段,用15 g/L琼脂糖凝胶电泳进行检测,结果显示,PCR条带单一,特异性好,无杂带,将4对引物的PCR产物片段纯化回收后送生工生物工程(上海)股份有限公司进行测序。
2.2 GHRH基因序列的SNP位点获得和分析
通过PCR测序结果发现,在GHRH基因的4对引物中,除G2引物外其余3对引物中均检测出SNP位点。因此,将G1、G3和G4共3对引物分别进行PCR批量扩增,将PCR产物直接测序,对筛选出的SNP位点进行分型,采用Chromas软件查看结果,如图1所示,测序结果用DNAman 8软件进行比对,共发现7个SNPs位点。将起始密码子ATG的A碱基命名为1,7个SNPs位点分别为C81T、G315C(外显子)、C1083T(外显子)、A1524G(外显子)、G2107A、T2125C和G2256A。

图中黑色方框代表突变位置。The black box in the figure represents the location of the mutation.
图1 红鳍东方鲀GHRH基因7个SNPs位点基因型
Fig.1 Seven SNPs locus genotypes in GHRH gene of tiger puffer Takifugu rubripes
对3个处于外显子上的SNPs进行突变分析,发现只有C1083T和A1524G两位点突变引起氨基酸的变异,属于错义突变,其中,C1083T中由ATG(M)变为ACG(T),A1524G中GTC(V)变为ATC(I);而G315C位点无氨基酸的变化,属于同义突变(图2)。

图2 GHRH基因3个外显子SNPs氨基酸变化
Fig.2 Amino acid changes in SNPs of 3 exons of GHRH gene
2.3 GHRH基因SNPs位点与生长性状关联分析
各SNPs位点与生长性状相关性结果见表2、表3。从表2可见:在6月龄红鳍东方鲀群体中,C81T、G315C和A1524G位点与大多数生长性状的关联性均呈显著状态,而G2107A、T2125C和G2256A位点仅与头长和眼间距两个性状显著相关;在C81T位点上,TT基因型的体质量、头长+躯干长、眼间距和尾柄宽的平均值显著高于CC和CT基因型(P<0.05),TT基因型的体长平均值显著高于CT基因型(P<0.05);在G315C位点上,CC基因型的体质量、体高、头长、头长+躯干长、眼间距和体宽的平均值显著高于GC和GG基因型(P<0.05);在A1524G位点上,AA基因型的体质量、体长、尾柄高、体高和头长+躯干长的平均值显著高于GG基因型(P<0.05);在G2107A、T2125C和G2256A位点上,GG和GA基因型的头长和眼间距均显著高于CC基因型(P<0.05)。
表2 GHRH基因SNP位点基因型与6月龄红鳍东方鲀生长性状的关联分析
Tab.2 Correlation analysis between the SNP genotype of GHRH gene and the growth traits of 6-month-old tiger puffer Takifugu rubripes
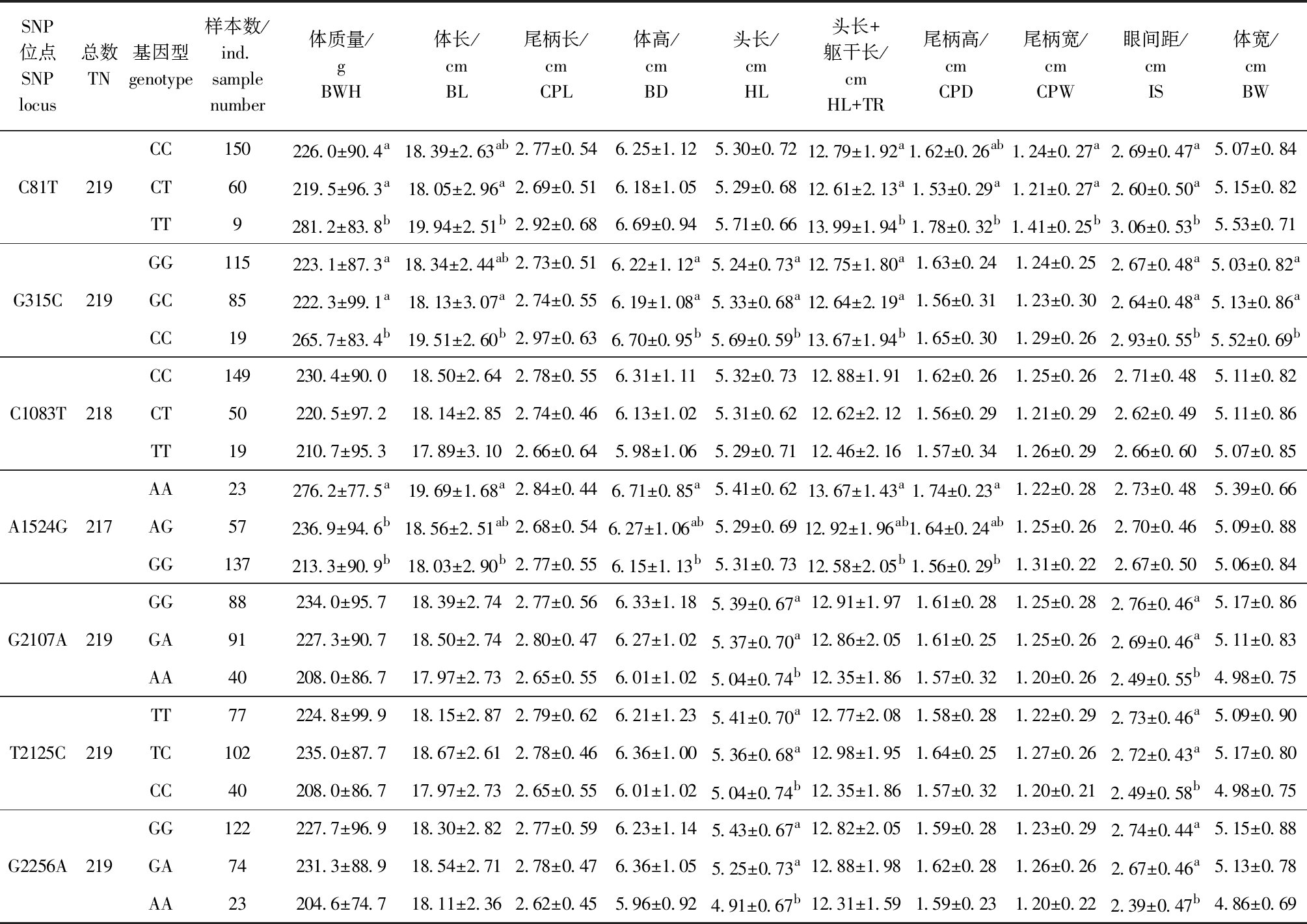
SNP位点SNP locus总数TN基因型genotype样本数/ind.sample number体质量/gBWH体长/cmBL尾柄长/cmCPL体高/cmBD头长/cmHL头长+躯干长/cmHL+TR尾柄高/cmCPD尾柄宽/cmCPW眼间距/cmIS体宽/cmBWCC150226.0±90.4a18.39±2.63ab2.77±0.546.25±1.125.30±0.7212.79±1.92a1.62±0.26ab1.24±0.27a2.69±0.47a5.07±0.84C81T219CT60219.5±96.3a18.05±2.96a2.69±0.516.18±1.055.29±0.6812.61±2.13a1.53±0.29a1.21±0.27a2.60±0.50a5.15±0.82TT9281.2±83.8b19.94±2.51b2.92±0.686.69±0.945.71±0.6613.99±1.94b1.78±0.32b1.41±0.25b3.06±0.53b5.53±0.71GG115223.1±87.3a18.34±2.44ab2.73±0.516.22±1.12a5.24±0.73a12.75±1.80a1.63±0.241.24±0.252.67±0.48a5.03±0.82aG315C219GC85222.3±99.1a18.13±3.07a2.74±0.556.19±1.08a5.33±0.68a12.64±2.19a1.56±0.311.23±0.302.64±0.48a5.13±0.86aCC19265.7±83.4b19.51±2.60b2.97±0.636.70±0.95b5.69±0.59b13.67±1.94b1.65±0.301.29±0.262.93±0.55b5.52±0.69bCC149230.4±90.018.50±2.642.78±0.556.31±1.115.32±0.7312.88±1.911.62±0.261.25±0.262.71±0.485.11±0.82C1083T218CT50220.5±97.218.14±2.852.74±0.466.13±1.025.31±0.6212.62±2.121.56±0.291.21±0.292.62±0.495.11±0.86TT19210.7±95.317.89±3.102.66±0.645.98±1.065.29±0.7112.46±2.161.57±0.341.26±0.292.66±0.605.07±0.85AA23276.2±77.5a19.69±1.68a2.84±0.446.71±0.85a5.41±0.6213.67±1.43a1.74±0.23a1.22±0.282.73±0.485.39±0.66A1524G217AG57236.9±94.6b18.56±2.51ab2.68±0.546.27±1.06ab5.29±0.6912.92±1.96ab1.64±0.24ab1.25±0.262.70±0.465.09±0.88GG137213.3±90.9b18.03±2.90b2.77±0.556.15±1.13b5.31±0.7312.58±2.05b1.56±0.29b1.31±0.222.67±0.505.06±0.84GG88234.0±95.718.39±2.742.77±0.566.33±1.185.39±0.67a12.91±1.971.61±0.281.25±0.282.76±0.46a5.17±0.86G2107A219GA91227.3±90.718.50±2.742.80±0.476.27±1.025.37±0.70a12.86±2.051.61±0.251.25±0.262.69±0.46a5.11±0.83AA40208.0±86.717.97±2.732.65±0.556.01±1.025.04±0.74b12.35±1.861.57±0.321.20±0.262.49±0.55b4.98±0.75TT77224.8±99.918.15±2.872.79±0.626.21±1.235.41±0.70a12.77±2.081.58±0.281.22±0.292.73±0.46a5.09±0.90T2125C219TC102235.0±87.718.67±2.612.78±0.466.36±1.005.36±0.68a12.98±1.951.64±0.251.27±0.262.72±0.43a5.17±0.80CC40208.0±86.717.97±2.732.65±0.556.01±1.025.04±0.74b12.35±1.861.57±0.321.20±0.212.49±0.58b4.98±0.75GG122227.7±96.918.30±2.822.77±0.596.23±1.145.43±0.67a12.82±2.051.59±0.281.23±0.292.74±0.44a5.15±0.88G2256A219GA74231.3±88.918.54±2.712.78±0.476.36±1.055.25±0.73a12.88±1.981.62±0.281.26±0.262.67±0.46a5.13±0.78AA23204.6±74.718.11±2.362.62±0.455.96±0.924.91±0.67b12.31±1.591.59±0.231.20±0.222.39±0.47b4.86±0.69
注:同一列中标有不同字母者表示同一位点不同基因型的生长性状间有显著性差异(P<0.05),标有相同字母者表示无显著性差异(P>0.05),下同。
Note:The means with different letters within the same column at the same SNP locus are significantly different at different genotypes at the 0.05 probability level,and the means with the same letter within the same column are not significant differences,et sequentia.
从表3可见:在12月龄红鳍东方鲀群体中,各突变位点与性状显著关联性与6月龄群体基本一致;在C81T位点上,TT基因型的体质量、头长和体宽平均值显著高于CC基因型(P<0.05);在G315C位点上,CC基因型的体质量和头长平均值显著高于GC和GG基因型(P<0.05);在A1524G位点上,AA基因型的头长平均值显著低于GG和AG基因型(P<0.05),而其体长、体宽和尾柄高平均值则显著高于GG基因型(P<0.05);在G2256A位点上,GG基因型的头长平均值显著高于AA基因型(P<0.05);在G2107A和T2125C位点上,各基因型生长性状均无显著性差异(P>0.05)。
另外,从表2和表3还可以看出,GHRH基因C1083T 位点的多态性与6月龄和12月龄红鳍东方鲀的生长性状均无显著关联(P>0.05)。
表3 GHRH基因SNP位点基因型与12月龄红鳍东方鲀生长性状的关联分析
Tab.3 Correlation analysis between the SNP genotype of GHRH gene and the growth traits of 12-month-old tiger puffer Takifugu rubripes

SNP位点SNP locus总数TN基因型genotype样本数/ind.sample number体质量/gBWH体长/cmBL尾柄长/cmCPL体高/cmBD头长/cmHL头长+躯干长/cmHL+TR尾柄高/cmCPD尾柄宽/cmCPW眼间距/cmIS体宽/cmBWCC150370.1±87.4a23.49±2.713.85±0.758.33±1.077.07±0.64a16.43±1.932.26±0.582.02±0.414.59±0.557.43±1.13aC81T219CT60383.2±106.9a22.91±2.743.68±0.748.12±0.967.01±0.60ab15.91±1.872.14±0.381.96±0.274.58±0.507.28±1.22abTT9474.1±108.3b23.44±3.673.83±0.948.37±2.016.62±0.93b16.07±2.682.25±0.112.00±0.354.49±0.658.13±1.68bGG115371.0±87.1a23.48±2.643.82±0.678.42±1.187.06±0.65a16.46±1.952.28±0.632.04±0.464.56±0.567.48±1.19abG315C219GC85373.6±100.2a23.14±2.893.83±0.858.14±0.977.07±0.61a16.09±1.932.13±0.381.96±0.244.57±0.537.23±1.15aCC19440.3±108.6b23.19±2.983.55±0.757.97±0.906.72±0.72b15.94±2.062.29±0.271.99±0.294.57±0.457.86±1.17bCC149373.5±89.323.57±2.773.88±0.768.36±1.147.09±0.6516.48±1.972.27±0.572.00±0.294.60±0.557.49±1.18C1083T218CT50381.8±105.422.65±2.513.56±0.657.99±0.946.87±0.6215.68±1.782.12±0.372.03±0.604.62±0.477.23±1.12TT19394.7±117.923.12±3.163.88±0.848.19±0.917.03±0.6616.15±2.022.11±0.411.96±0.284.39±0.567.25±1.37AA23394.6±85.824.22±2.34a3.97±0.528.45±1.146.76±0.65a16.62±2.052.43±0.24a2.00±0.304.60±0.468.14±0.79aA1524G217AG57387.9±99.723.76±2.78ab3.87±0.708.51±1.047.07±0.65b16.66±1.962.34±0.83ab2.01±0.264.61±0.587.51±1.27bGG137369.5±95.622.95±2.76b3.74±0.818.13±1.087.07±0.64b16.02±1.902.14±0.36b2.00±0.434.55±0.527.23±1.15bGG88369.3±86.923.39±2.763.86±0.828.31±1.147.11±0.6516.35±1.962.29±0.691.99±0.264.59±0.517.33±1.18G2107A219GA91382.1±98.523.33±2.843.75±0.738.12±1.007.01±0.6116.25±1.982.18±0.392.02±0.484.63±0.577.43±1.12AA40386.0±107.823.18±2.623.80±0.658.37±1.176.92±0.7116.17±1.902.19±0.342.01±0.324.49±0.527.56±1.35TT77369.2±90.323.34±2.923.84±0.868.42±1.207.17±0.6216.38±2.022.26±0.731.98±0.264.58±0.587.29±1.23T2125C219TC102380.7±94.923.34±2.703.77±0.728.12±0.946.98±0.6316.23±1.932.21±0.382.02±0.474.62±0.517.46±1.07CC40386.0±107.823.18±2.623.80±0.658.37±1.176.93±0.7116.17±1.902.19±0.342.01±0.324.49±0.527.56±1.35GG122378.8±97.523.28±2.873.79±0.858.29±1.157.11±0.65a16.25±1.992.22±0.621.98±0.274.58±0.537.37±1.22G2256A219GA74384.9±89.023.38±2.573.79±0.648.28±1.016.99±0.64ab16.33±1.882.21±0.392.02±0.534.61±0.577.42±1.06AA23347.2±102.923.40±2.813.90±0.548.17±1.026.80±0.61b16.24±2.072.32±0.322.07±0.294.53±0.517.64±1.37
2.4 连锁不平衡及双倍型分析
利用Hapioview 4.1软件对GHRH基因的7个SNPs位点进行连锁不平衡分析(图3)。从图3可见,G2256A与T2125C的D′值为0.81,两位点处于连锁不平衡状态,G2107A与C1083T的D′值为0.83,也处于连锁不平衡状态,然而二者的r2值分别为0.311和0.089,不能同时满足D′>0.8且r2>0.33的条件,这说明这些位点并非完全连锁;其他位点两两间既不能满足D′>0.8,也不能满足r2>0.33,说明位点间处于彼此独立的状态。
从上述6月龄和12月龄红鳍东方鲀的SNPs位点与生长性状关联分析中可知,7个SNPs位点中,C81T、G315C和A1524G位点与大多数生长性状的关联性均呈显著状态,因此,可用这3个位点进行双倍型与体质量的关联分析。在双倍型分析中,若一种双倍型组合的个体数小于10尾,则结果不具有参考价值,经排除,最终形成5种双倍型(理论双倍型数为8种),结果如表4所示。其中,双倍型D3出现频率最低(0.09),双倍型D5出现频率最高(0.48)。6月龄群体中,双倍型D3的体质量平均值最低且显著低于双倍型D4和D5(P<0.05),双倍型D4体质量平均值最高;12月龄群体中,双倍型D3的体质量平均值最低且显著低于双倍型D5(P<0.05)(表4)。推测6月龄时,双倍型D3对体质量有负相关影响,双倍型D4和D5对体质量有正相关影响,12月龄时,双倍型D4和D5对体质量有正相关影响。
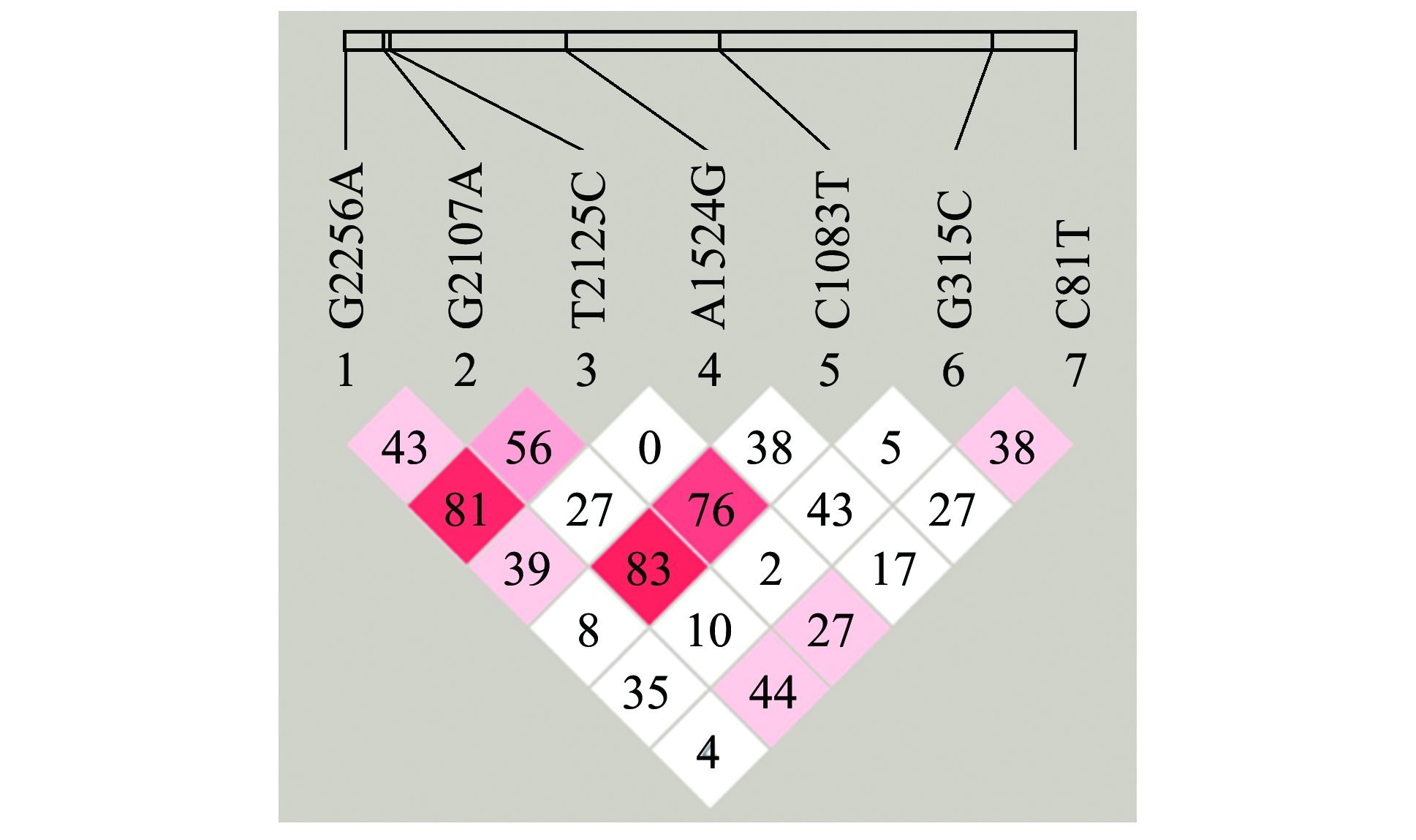
图中数值为D′值,单位为%。The numerical value in the figure is D′ value expressed as a percentage.
图3 GHRH基因SNPs连锁不平衡分析
Fig.3 Linkage disequilibrium analysis about the SNPs of GHRH gene
表4 GHRH基因双倍型与6月龄及12月龄红鳍东方鲀体质量的关联分析
Tab.4 Correlation analysis between GHRH gene diplotypes and body mass traits of 6-month-old and 12-month-old tiger puffer Takifugu rubripes

双倍型dipoltypeSNP位点locusC81TG315CA1524G样本数/ind.sample number频率frequency6月龄体质量/gBWH of 6-month-old12月龄体质量/gBWH of 12-month-oldD1TTCCGG120.10245.81±98.02ab350.90±70.79abD2TTGGGG180.15213.04±77.96ab362.12±73.58abD3CCCCGG110.09165.70±65.76a313.71±53.03aD4CCGGAA160.13251.25±101.01b402.39±109.20abD5CCGGGG570.48235.87±91.70b395.26±97.29b
注:同列中标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。
Note:The means with different letters within the same column are significantly different in the groups at the 0.05 probability level,and the means with the same letter within the same column are not significant differences.
3 讨论
GHRH是下丘脑分泌的肽类激素,其通过与垂体前叶生长激素受体结合来调节生长激素的合成和分泌。Farmer等[21]通过给不同年龄的猪注射GHRH,得出GHRH有促进个体生长效果的结论。张永亮等[22]通过直接注射将改良的生长激素释放因子导入兔和鼠的骨骼肌中,发现可增强其体内的GH浓度。在水产动物方面,Tao等[8]在北极红点鲑的 GHRH 基因中发现,特定 SNP 等位基因与早期生长之间存在显著关联。在红鳍东方鲀中,仅见王艳玲[23]利用PCR-SSCP方法和DNA测序方法研究GHRH基因突变的报道,但未发现SNPs。因此,本研究中首次发现了红鳍东方鲀GHRH基因突变与生长性状间的关联。
3.1 红鳍东方鲀GHRH基因的SNPs位点筛选及其与生长性状的关联分析
本试验中结果显示,红鳍东方鲀GHRH基因上共有7个SNP位点,分别为C81T、G315C、C1083T、A1524G、G2107A、T2125C及G2256A,其中,G315C、C1083T、A1524G存在于外显子上,C1083T位点所在的序列由ATG(M)变为ACG(T),A1524G中GTC(V)变为ATC(I),位点突变导致氨基酸种类的改变,属于错义突变。外显子上的突变如果导致氨基酸的改变,通常会导致蛋白质结构与活性的变化,甚至导致蛋白质翻译提前终止,从而发生一系列生物学效应并对动物机体产生影响。本研究中位于外显子上的3个SNP突变位点G315C、C1083T和A1524G,尤其是C1083T与A1524G 2个多态位点导致了氨基酸的改变,可能是红鳍东方鲀个体生长性状差异的主要原因。本研究结果显示,GHRH基因上只有3个突变位点G315C、A1524G和C81T与2个阶段的生长性状显著相关,且CC、AA、TT基因型分别为优势基因型,该研究结果也证实了GHRH基因是一个鱼类生长性状的功能候选基因,相似的研究结果在斑点交叉尾鮰 Ictalurus punetaus 中也有报道。张世勇等[24]对斑点交叉尾鮰采用DNA混池测序法筛选其GHRH基因的SNP位点,并进行连锁不平衡和单倍型分析,最终在GHRH基因中发现了4个与生长性状相关的SNPs标记位点。因此,笔者推测本试验中获得的3个与生长性状显著相关的位点可以作为红鳍东方鲀的分子标记辅助选择位点。
3.2 SNPs位点的连锁不平衡及双倍型分析
连锁不平衡是给定群体中不同位点等位基因的非随机关联现象。本研究中,G2256A与T2125C两位点及G207A与C083T两位点间都满足 D′>0.8,处于连锁不平衡状态,然而其他位点两两间既不能满足D′>0.8,也不能满足r2>0.33,说明位点间处于彼此独立的状态,这表明这7个SNPs位点更趋向于独立遗传。生物的生长性状并非受单一位点调控,而是多个位点或微效基因互相作用共同影响,因此,分析某一单个位点并不能准确得到相关性,应将多个位点组成双倍型组合共同分析[26-27]。本研究中对C81T、G315C和A1524G 3个SNPs位点进行双倍型与体质量的关联分析,去除个体数少于10的双倍型组合,共得到5种双倍型,将双倍型组合与红鳍东方鲀的体质量进行关联分析,结果表明,6月龄群体中双倍型D4、D5的平均值显著高于双倍型D3,12月龄群体中双倍型D5的平均值显著高于双倍型D3,推测6月龄时,双倍型D4、D5可能对红鳍东方鲀的体质量有正相关影响,12月龄时,双倍型D4、D5可能对红鳍东方鲀的体质量有正相关影响,双倍型D3可能有负相关影响。C81T的优势基因型为TT,G315C的优势基因型为CC,A1524G的优势基因型为AA。然而,TT、CC、AA的双倍型组合个体太少而无法进行分析,从双倍型分析结果上看,6月龄时双倍型D4、D5为优势基因型,12月龄时双倍型D5为优势基因型。
4 结论
1)本研究中根据红鳍东方鲀GHRH基因序列设计引物,用直接测序法在GHRH基因上共筛选到7个SNP位点,这表明红鳍东方鲀群体中GHRH基因存在较为丰富的突变。
2)C81T、G315C和A1524G 3个位点是对红鳍东方鲀生长有正向影响的优势位点,双倍型D4和D5为优势基因型,说明在后续红鳍东方鲀选育过程中可以优先选择这些位点及基因型,本研究结果为红鳍东方鲀的分子标记辅助选育提供了科学依据。
[1] BLOCH B,GAILLARD R C,BRAZEAU P,et al.Topographical and ontogenetic study of the neurons producing growth hormone-releasing factor in human hypothalamus[J].Regulatory Peptides,1984,8(1):21-31.
[2] AL-SAMERRIA S,ALOQAILY B,NEGRON A,et al.Interrupted IGF-1 feedback in GHRH neurons and somatotrophs results in impaired weight gain and increased energy expenditure[J].Journal of the Endocrine Society,2021,5(sup 1):A52.
[3] ICYUZ M,ZHANG F,FITCH M P,et al.Physiological and metabolic characteristics of novel double-mutant female mice with targeted disruption of both growth hormone-releasing hormone and growth hormone receptor[J].Aging Cell,2021,20(4):e13339.
[4] MU OZ-MORENO L,CARMENA M J,SCHALLY A V,et al.Stimulation of neuroendocrine differentiation in prostate cancer cells by GHRH and its blockade by GHRH antagonists[J].Investigational New Drugs,2020,38(3):746-754.
OZ-MORENO L,CARMENA M J,SCHALLY A V,et al.Stimulation of neuroendocrine differentiation in prostate cancer cells by GHRH and its blockade by GHRH antagonists[J].Investigational New Drugs,2020,38(3):746-754.
[5] 张存芳.黄牛PTHRP、GHRH和GHRHR基因SNP及其与生长性状关联分析[D].杨凌:西北农林科技大学,2008:74.
ZHANG C F.The analysis of PTHRP,GHRH and GHRHR genes SNP and their association with growth traits in cattle[D].Yangling:Northwest A &F University,2008:74.(in Chinese)
[6] CHEONG H S,YOON D H,KIM L H,et al.Growth hormone-releasing hormone (GHRH)polymorphisms associated with carcass traits of meat in Korean cattle[J].BMC Genetics,2006,7:35.
[7] GUO L,XIA J H,YANG S,et al.GHRH,PRP-PACAP and GHRHR target sequencing via an ion torrent personal genome machine reveals an association with growth in orange-spotted grouper (Epinephelus coioides)[J].International Journal of Molecular Sciences,2015,16(11):26137-26150.
[8] TAO W J,BOULDING E G.Associations between single nucleotide polymorphisms in candidate genes and growth rate in Arctic charr (Salvelinus alpinus L.)[J].Heredity,2003,91(1):60-69.
[9] LANDER E S.The new genomics:global views of biology[J].Science,1996,274(5287):536-539.
[10] 李艳杰,龙火生,李影,等.单核苷酸多态性(SNP)的研究进展和应用[J].畜牧兽医杂志,2003,22(4):16-18.
LI Y J,LONG H S,LI Y,et al.Research progress and application of single nucleotide polymorphism (SNP)[J].Journal of Animal Science and Veterinary Medicine,2003,22(4):16-18.(in Chinese)
[11] 卞光明,胡则辉,柴学军,等.SNP标记技术及其在水产动物遗传学中的应用[J].浙江海洋学院学报(自然科学版),2016,35(4):346-353.
BIAN G M,HU Z H,CHAI X J,et al.Single nucleotide polymorphism and its application in aquatic animal genetics[J].Journal of Zhejiang Ocean University (Natural Science Edition),2016,35(4):346-353.(in Chinese)
[12] 张建勇.中国对虾(Fenneropenaeus chinensis)基因组SNP标记的开发与应用[D].青岛:中国海洋大学,2011:127-133.
ZHANG J Y.Development and application of SNP markers in the genome of Fenneropenaeus chinensis[D].Qingdao:Ocean University of China,2011:127-133.(in Chinese)
[13] 樊佳佳,刘小献,白俊杰,等.草鱼柠檬酸合酶基因SNP筛选及与生长性状的关联分析[J].华中农业大学学报,2014,33(3):84-89.
FAN J J,LIU X X,BAI J J,et al.SNP screen of grass carp citrate synthase gene and correlation analysis with growth trait[J].Journal of Huazhong Agricultural University,2014,33(3):84-89.(in Chinese)
[14] 高露姣,黄艳青,夏连军,等.不同养殖模式下红鳍东方鲀的品质比较[J].水产学报,2011,35(11):1668-1676.
GAO L J,HUANG Y Q,XIA L J,et al.Quality comparison of Takifugu rubripes under different culture modes[J].Journal of Fisheries of China,2011,35(11):1668-1676.(in Chinese)
[15] 马爱军,陆丽君,陈超,等.东方鲀属主要经济鱼种繁育养殖、育种和基因研究现状[J].海洋科学,2011,35(11):128-133.
MA A J,LU L J,CHEN C,et al.Breeding,breeding and gene research status of the main economic species of the genus Takifugu[J].Marine Sciences,2011,35(11):128-133.(in Chinese)
[16] 黄玉林,王文利,刘源,等.我国养殖河鲀(业)发展现状及研究进展[J].中国食品学报,2018,18(1):217-224.
HUANG Y L,WANG W L,LIU Y,et al.The development and progress of cultured puffer fish in China[J].Journal of Chinese Institute of Food Science and Technology,2018,18(1):217-224.(in Chinese)
[17] 桂建芳,周莉,张晓娟.鱼类遗传育种发展现状与展望[J].中国科学院院刊,2018,33(9):932-939.
GUI J F,ZHOU L,ZHANG X J,et al.Research advances and prospects for fish genetic breeding[J].Bulletin of Chinese Academy of Sciences,2018,33(9):932-939.(in Chinese)
[18] 张迪,杨正勇,张英丽.中国河鲀养殖产业发展现状与发展对策[J].科学养鱼,2017(12):1-3.
ZHANG D,YANG Z Y,ZHANG Y L.Current situation and countermeasures of the development of the puffer fish aquaculture industry in China[J].Scientific Fish Farming,2017(12):1-3.(in Chinese)
[19] 苟盼盼,王秀利,窦冬雨,等.红鳍东方鲀不同家系群体的形态性状差异与相关性分析[J].大连海洋大学学报,2019,34(5):674-679.
GOU P P,WANG X L,DOU D Y,et al.Analysis of morphological differences and correlation in three families of tiger puffer Takifugu rubripes[J].Journal of Dalian Ocean University,2019,34(5):674-679.(in Chinese)
[20] 王新安,马爱军,庄志猛,等.红鳍东方鲀体型性状选育指标的综合判定[J].渔业科学进展,2012,33(6):10-15.
WANG X A,MA A J,ZHUANG Z M,et al.Comprehensive judgement on breeding indexes of shape characters of Takifugu rubripes[J].Progress in Fishery Sciences,2012,33(6):10-15.(in Chinese)
[21] FARMER C,POMMIER S A,BRAZEAU P.Validation of a culture system for porcine pituitary cells:effects of growth hormone-releasing factor and(or)somatostatin on growth hormone secretion[J].Journal of Animal Science,1993,71(4):923-929.
[22] 张永亮,欧阳红生,刘松财,等.生长激素释放因子(GRF)在动物肌肉组织的表达[J].中国兽医学报,2000,20(3):239-242.
ZHANG Y L,OUYANG H S,LIU S C,et al.Expression of growth hormone releasing factor (GRF)in animal muscle tissue[J].Chinese Journal of Veterinary Science,2000,20(3):239-242.(in Chinese)
[23] 王艳玲.红鳍东方鲀Myf5、GHRH基因的SNPs分析[D].大连海洋大学,2012.
WANG Y L.SNPs analysis of Myf5 and GHRH genes in Takifugu rubripes[D].Dalian:Dalian Ocean University,2012.(in Chinese)
[24] 张世勇,钟立强,秦钦,等.斑点叉尾鮰GHRH基因3个SNPs位点及其单倍型组合与生长性状的关联分析[J].水生生物学报,2016,40(5):886-893.
ZHANG S Y,ZHONG L Q,QIN Q,et al.Three SNPs polymorphism of growth hormone-releasing hormone gene (GHRH)and association analysis with growth traits in channel catfish[J].Acta Hydrobiologica Sinica,2016,40(5):886-893.(in Chinese)
[25] HOHENLOHE P A,BASSHAM S,CURREY M,et al.Extensive linkage disequilibrium and parallel adaptive divergence across threespine stickleback genomes[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2012,367(1587):395-408.
[26] LI S J,LIU H,BAI J J,et al.Transcriptome assembly and identification of genes and SNPs associated with growth traits in largemouth bass (Micropterus salmoides)[J].Genetica,2017,145(2):175-187.
[27] 薛倩,王金玉,张跟喜,等.黑素皮质素受体3基因(MC3R)多态性及其单倍型组合与京海黄鸡屠体性状的关联分析[J].农业生物技术学报,2015,23(3):344-351.
XUE Q,WANG J Y,ZHANG G X,et al.Association analysis of melanocortin 3 receptor (MC3R)polymorphism and its haplotype combination with carcass traits in jinghai yellow chicken[J].Journal of Agricultural Biotechnology,2015,23(3):344-351.(in Chinese)