三角鲂Megalobrama terminalis,因背鳍高、头尖尾长,从侧面观察形似三角形而得名,为鲂属鲤形目,主要分布在中国长江水系、黄河水系、黑龙江水系等淡水区域[1-2],其体大肉厚,肉质嫩滑,深受消费者的喜爱,是中国重要的经济鱼类之一。历史上,钱塘江渔业资源丰富,除盛产富春江鲥鱼Tenualosa reevesii、花鲈Lateolabrax japonicus和子陵鱼Rhinogobius giurinus等多个种[3]外,三角鲂的野生资源也很丰富[4]。近年来,钱塘江渔业资源衰退,三角鲂野生资源也日益衰退[5]。和其他鱼类一样,为了修复、补充野生资源,三角鲂也进行了大规模的增殖放流。据杭州市渔政渔港监督管理总站不完全统计,仅2018年钱塘江中下游就放流了三角鲂冬片5.4万kg。
目前,有关三角鲂的研究较多,早期主要集中在种群形态差异[6]、分类整理[7]、染色体核型与DNA含量[8]、胚胎发育[9]和脑垂体超微结构[10]等方面,近年来出现了一些有关线粒体基因组方面的研究报道。谢楠等[11]利用细胞色素b基因(cytb)研究了鲂属种间的分类情况;赖瑞芳等[12]和Hu等[13]对鲂属鱼类线粒体基因序列及种间亲缘关系进行了研究;另外,也有少量关于转录组的研究报道[14]。但有关三角鲂放流苗种的种质质量、放流群体的遗传多样性及遗传分化等研究尚未见报道。
种质是生物体亲代传递给子代的遗传物质,而遗传多样性是衡量种质优劣的重要指标。广义的遗传多样性是指生物种内和种间的遗传差异度[15],遗传多样性越高,物种或种群对环境的适应力越强。细胞色素C氧化酶亚基I(cytochrome C oxidase subunit Ⅰ, COⅠ)基因是线粒体基因的重要组成部分,具有结构简单、进化速率适中、多态性较高及易被通用引物扩增等特点。作为一种DNA条形码,COⅠ基因被广泛应用于不同生物类群的种类鉴定中[16-17],也被用于种群的遗传多样性分析[18-19]和系统进化分析中[20]。本研究中,以亲本群体、放流群体和钱塘江自然捕捞群体为研究对象,测定了钱塘江三角鲂6个群体的COⅠ基因序列,并对其进行遗传多样性和群体遗传分化研究,以期为评估放流苗种种质质量和指导其科学增殖放流提供理论依据和基础数据。
1 材料与方法
1.1 样本来源
试验群体为2个良种场亲本群体、3个放流群体和1个钱塘江自然捕捞群体,其中景山亲本群体(JS)和柏松亲本群体(BS)采集于各良种场,采集对象是当年参与繁殖的亲本;余杭(YH)、桐庐(TL)和建德(JD)放流群体采集于放流现场,采集对象是放流冬片鱼的苗种(体质量约20 g),利用渔业资源监测网点,在钱塘江中下游的桐庐、新桐乡、富阳、一桥和六桥5个捕捞位点采集自然捕捞群体(BL)亲本群体,尽量采集所有参加繁殖的个体,放流群体随机采集。所有样本取样部位均为尾鳍,所有样本保存于分析纯乙醇中,带回实验室备用。样本采集时间、采样信息见表1和图1。
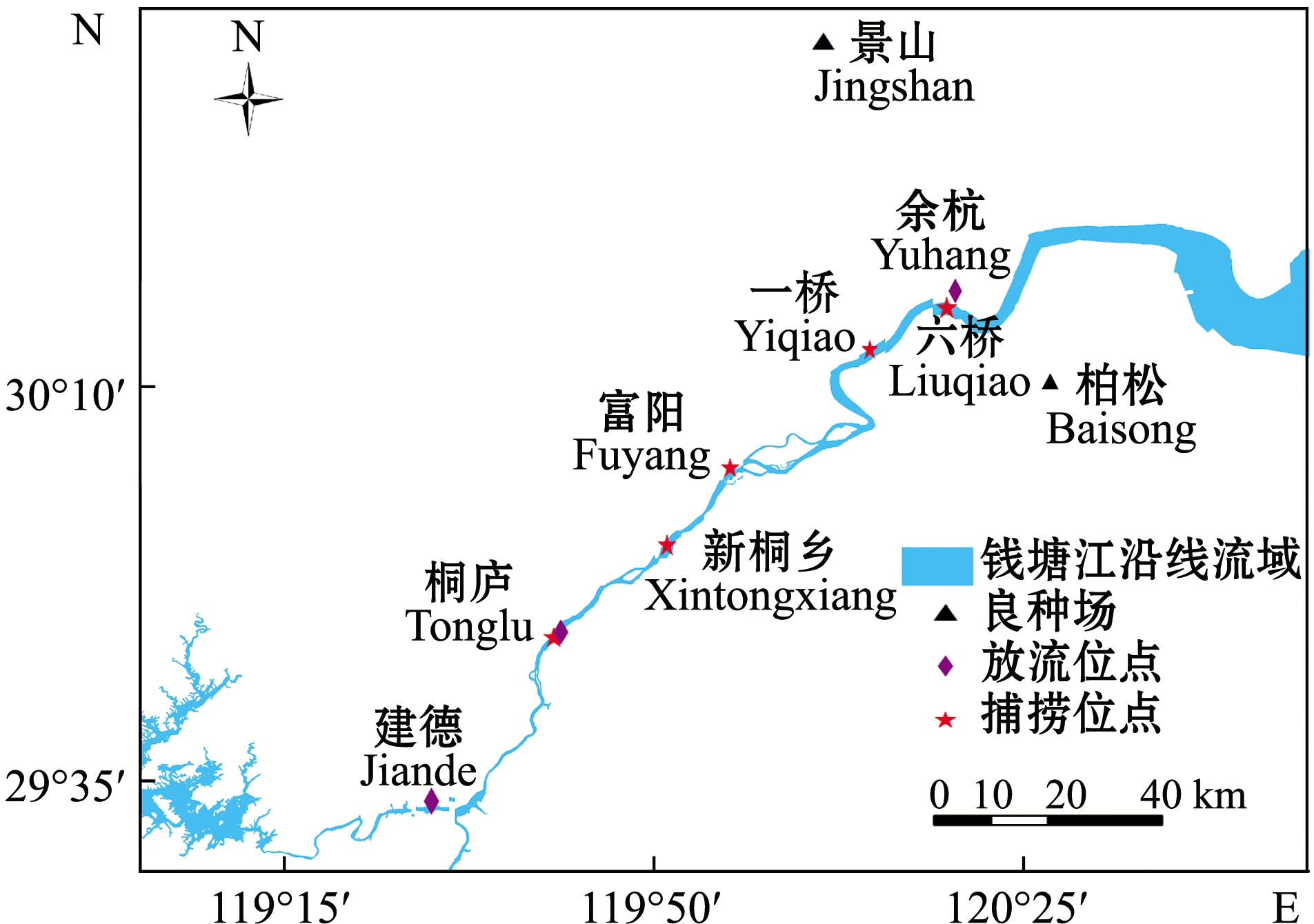
图1 三角鲂采样点分布
Fig.1 Sampling sites of black bream Magalobrama terminalis
表1 三角鲂样品采集信息
Tab.1 Sample collecting information on black bream Megalobrama terminalis

类型type群体population样品采集地collectingsite采集数量/ind.collectednumber分析样本量/ind.samplenumberanalyzed采集日期samplingdate亲本群体broodstockpopulation景山JS湖州东林镇景山良种场HuzhouDonglinJingshanHatchery196482018-04-22柏松BS杭州市萧山瓜沥镇柏松良种场HangzhouXiaoshanGualiBaisongHatchery75482018-05-09余杭YH余杭区钱塘江放流现场releasedsiteinYuhangQiantangRiver140322018-12-06放流群体releasedpopulation桐庐TL桐庐钱塘江放流现场releasedsiteinTongluQiantangRiver150322018-12-06建德JD建德钱塘江放流现场releasedsiteinJiandeQiantangRiver144322018-12-06自然捕捞群体naturalfishingpopulation捕捞BL钱塘江中下游middleandlowerQiantangRiver371482018年7—10月
1.2 DNA提取与PCR扩增测序
随机取各群体样品,采用试剂盒法(TIANamp Marine Animals DNA Kit)进行DNA抽提,用8 g/L琼脂糖凝胶(Nared染色)电泳检测DNA提取效果,将检测合格的DNA放入-20 ℃冰箱保存,用于PCR反应。
PCR扩增引物设计参照文献[21],其序列Fish F1:5′TCAACCAACCACAAAGACATTGGCAC 3′;Fish R1:5′TAGACTTCTGGGTGGCCAAAGAATCA 3′。引物由生工生物(上海)有限公司合成。PCR反应体系(共10 μL):Premix Taq 5 μL,双蒸水3.5 μL,正、反向引物各0.25 μL,DNA模板1 μL。PCR反应条件:94 ℃下预变性3 min;94 ℃下循环变性30 s,50 ℃下退火复性30 s,72 ℃下延伸40 s,共进行30个循环;最后再在72 ℃下延伸8 min,4 ℃条件下保存。PCR扩增产物经12 g/L琼脂糖凝胶电泳检验,用全自动凝胶成像分析系统(Syngene)观察目的条带的扩增效果。
将扩增效果好的产物送生工生物(上海)有限公司进行纯化、测序,测序引物与扩增引物相同。共成功测序224尾三角鲂样品,其中,景山亲本群体(JS)45尾,柏松亲本群体(BS)46尾,自然捕捞群体(BL)40尾,建德放流群体(JD)31尾,桐庐放流群体(TL)30尾,余杭放流群体(YH)32尾。
1.3 数据处理
将测定的序列运用ClustalX 1.81软件进行对位排列并人工校对,将对齐后的序列导入MEGA 3.1软件进行计算,得到碱基组成(nucleotide composition)、变异位点(variable sites)、简约信息位点(parsim-info sites)[22]。将得到的Meg文件导入DNASP 5.10软件包进行计算,得到群体的单倍型数(number of haplotypes)、单倍型多样性(haplotypes diversity,Hd)、核苷酸多样性(nucleotide diversity, Pi)、平均核苷酸差异数(nucleotide differences, K)[23],其中K代表每个群体内两个随机选取的mtDNA序列间平均每个位点的核苷酸数目[24]。利用Arlequin 3.11软件中的分子方差分析(analysis of molecular variance,AMOVA)估算遗传变异在群体内和群体间的分布及地理群体分化水平(F-statistics,Fst)[25],运用Nm=(1-Fst)/(4Fst)公式计算基因流(Nm)。利用 Network 4.6 软件构建单倍型的简约中介(reduced-median, medium-join(MJ))网络图。
2 结果与分析
2.1 序列多样性和单倍型分析
三角鲂样本经PCR扩增、电泳后得到清晰的COⅠ基因条带,长度约为700 bp(图2)。
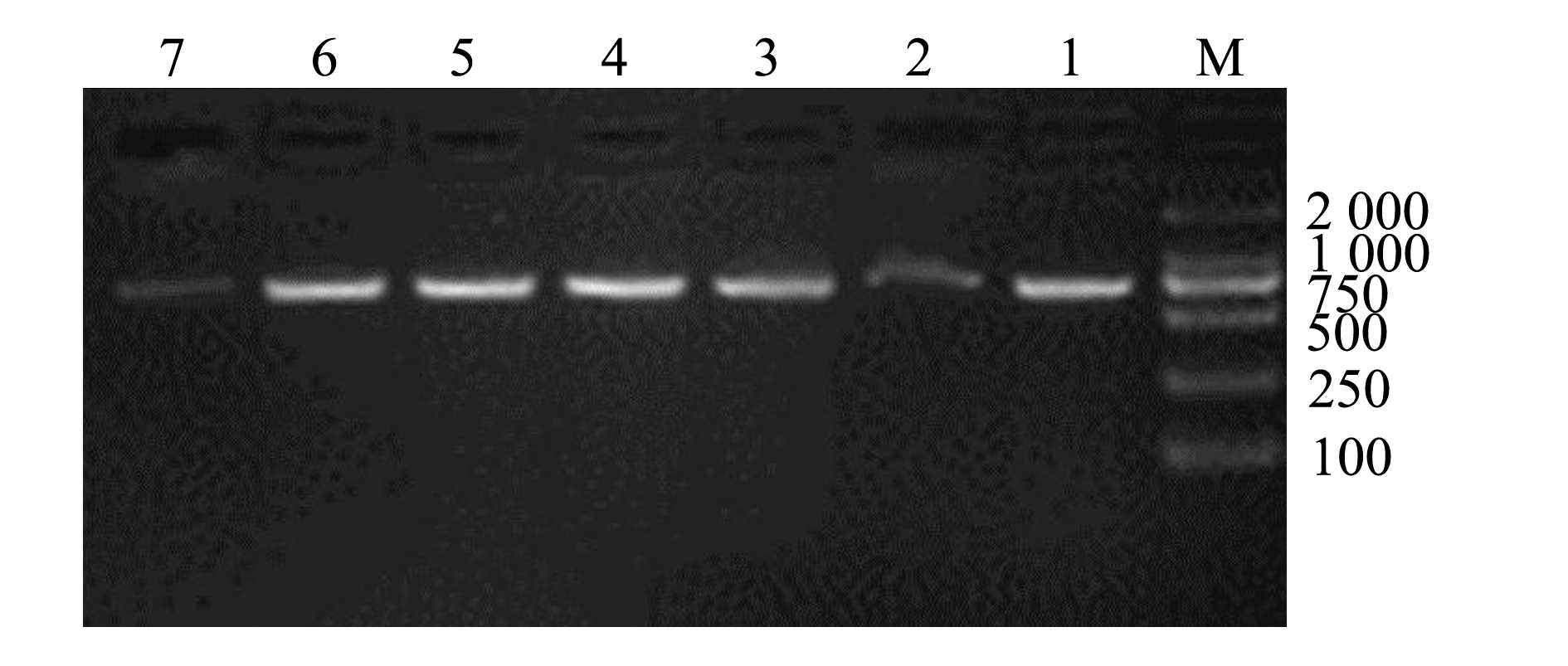
M—DNA maker;1~7—景山三角鲂样本。
M—DNA maker;1-7—JS populations of black bream Megalobrama terminalis.
图2 三角鲂mtDNA COⅠ基因PCR扩增电泳图
Fig.2 PCR amplification electrophoresis of mtDNA COⅠ gene of black bream Megalobrama terminalis
利用 ClustalX 软件对测序所获目的片段进行手工校对,获得长度为 661 bp 的同源序列, 经Blast同源性分析,与三角鲂线粒体DNA 全序列(GenBank:MN604 232.1)中COⅠ序列的一致性高达99.97%,确认所测序列为三角鲂COⅠ序列。在661 个位点中,共检测到 192个变异位点,占总位点数的29.05%,其中,简约信息位点 51个,单碱基变异位点 141 个。COⅠ 序列中碱基A、T、C、G 的平均含量分别为26.5%(25.8%~27.2%)、28.0%(27.0%~29.0%)、29.0%(28.1%~29.9%)和16.5%(15.4%~17.9%),其中,C 碱基含量最高,G 碱基含量最低,且 A+T 的含量(54.5%)高于 C+G 的含量(45.5%),具有一定的A.T 偏好性和C碱基偏好,表现出了碱基组成的偏向性,与赖瑞芳等[12]对鲂属种群研究结果相同。
224尾三角鲂样本共检出31种单倍型(表2),其中,6种为共享单倍型,25种为单个群体的特有单倍型。hap9是6个群体的共享单倍型,为第一优势单倍型,占样本总数的48.66%;hap2为5个群体的共享单倍型,占样本总数的22.32%;hap4和hap5为3个群体的共享单倍型,分别占样本总数的9.38%和4.46%;hap10和hap21为2个群体的共享单倍型,均占样本总数的0.89%。
表2 三角鲂6个群体线粒体COⅠ基因单倍型分布
Tab.2 Distribution of 31 haplotypes on mtDNA COⅠ gene of six populations of black bream Megalobrama terminalis

单倍型(个体分布数)numberanddistributionofhaplotype群体population景山JS柏松BS捕捞BL建德JD桐庐TL余杭YH每种单倍型占比/%haploidratiopertypeHap1(2)20.89Hap2(50)191791422.32Hap3(1)10.45Hap4(21)91119.38Hap5(10)6134.46Hap6(1)10.45Hap7(1)10.45Hap8(1)10.45Hap9(109)2427171881548.66Hap10(2)110.89Hap11(1)10.45Hap12(1)10.45Hap13(1)10.45Hap14(3)31.34Hap15(1)10.45Hap16(2)20.89Hap17(1)10.45Hap18(1)10.45Hap19(1)10.45Hap20(1)10.45Hap21(2)110.89Hap22(1)10.45Hap23(1)10.45Hap24(1)10.45Hap25(1)10.45Hap26(1)10.45Hap27(1)10.45Hap28(2)20.89Hap29(1)10.45Hap30(1)10.45Hap31(1)10.45单倍型数numberofhaplotype998686
仅单个群体特有的单倍型占样本总数 13.4%,柏松放流群体有6种特有单倍型,桐庐放流群体和自然捕捞群体各有5种特有单倍型;景山亲本群体、建德放流群体和余杭放流群体的特有单倍型数依次为4、3、2种。
2.2 群体遗传多样性及中性检验
遗传多样性是指生物群体间,以及群体内在分子、细胞和个体3个方面的遗传变异度。单倍型多样性(Hd)和核苷酸多样性(Pi)常用来度量遗传多样性,Hd和Pi值越高,群体的多态程度越高,遗传多样性越丰富。本研究中,三角鲂6个群体遗传多样性参数统计及中性检验(Tajima’s D检验)结果表明:6个群体的Hd为0.710~0.848(平均值为0.705),说明6个群体的单倍型多样性均较高;6个群体的Pi为0.003 07~0.008 40(平均值为0.005 78);6个群体的平均核苷酸差异数K
由高到低依次为景山亲本群体、桐庐放流群体、自然捕捞群体、建德放流群体、柏松亲本群体和余杭放流群体(表3)。这表明,6个群体整体遗传多样性水平较高,景山亲本群体的遗传多样性最高,余杭放流群体的遗传多样性最低。
中性检验表明,6个群体的Tajima’s D值为-2.658 38~-1.024 81,4个群体不同程度地显著偏离中性假说(P<0.05),柏松群体和余杭群体遵循中性假说。将所有样本作为一个整体进行分析,Tajima’s D值为负值偏离中性进化(表3),由此推测,6个三角鲂群体可能在进化过程中存在种群扩张事件。
表3 三角鲂6个群体的遗传多样性参数及Tajima’s D检验
Tab.3 Genetic diversity index and Tajima’s D tests of six populations of black bream Megalobrama terminalis
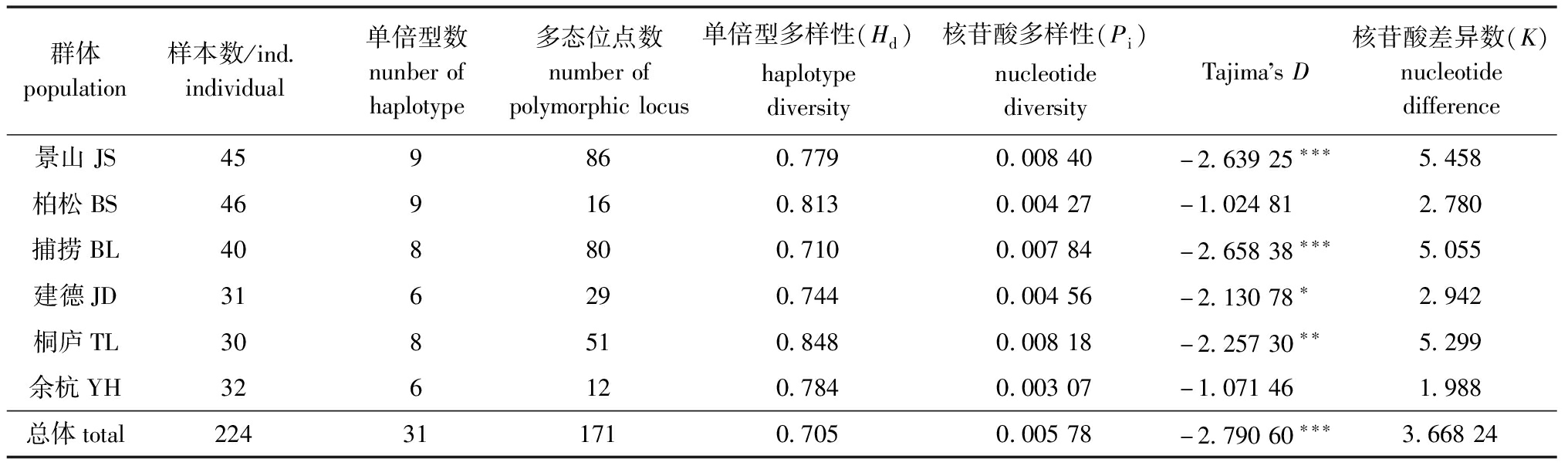
群体population样本数/ind.individual单倍型数nunberofhaplotype多态位点数numberofpolymorphiclocus单倍型多样性(Hd)haplotypediversity核苷酸多样性(Pi)nucleotidediversityTajima sD核苷酸差异数(K)nucleotidedifference景山JS459860.7790.00840-2.63925∗∗∗5.458柏松BS469160.8130.00427-1.024812.780捕捞BL408800.7100.00784-2.65838∗∗∗5.055建德JD316290.7440.00456-2.13078∗2.942桐庐TL308510.8480.00818-2.25730∗∗5.299余杭YH326120.7840.00307-1.071461.988总体total224311710.7050.00578-2.79060∗∗∗3.66824
注:* 表示达到显著性水平(P<0.05);**和***表示达到极显著性水平(P<0.01,P<0.001),下同。
Note:* means statistical significant(P<0.05); ** and *** denote very significant difference(P<0.01,P<0.001),et sequentia.
2.3 群体遗传分化及分子方差分析
采用双参数法(K2P)得到基于Nei’s的遗传距离,结果表明,不同群体间遗传距离为0.003~0.009,其中,建德放流群体和柏松亲本群体间的遗传距离最小(0.003),景山亲本群体与桐庐放流群体、捕捞群体间的遗传距离最大(均为0.009)(表4)。
表4 三角鲂群体间的K2P遗传距离
Tab.4 Pairwise K2P genetic distances among populations of black bream Megalobrama terminalis using COⅠ gene
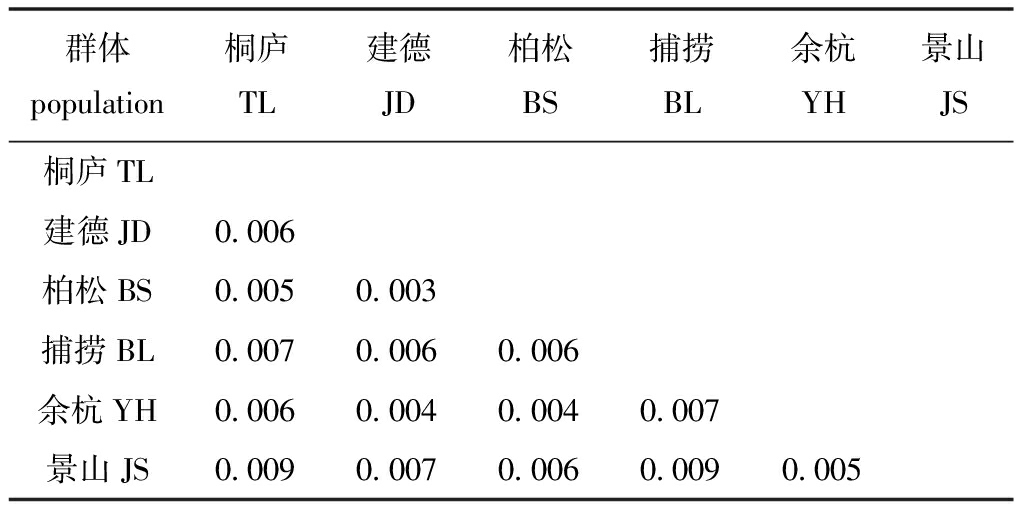
群体population桐庐TL建德JD柏松BS捕捞BL余杭YH景山JS桐庐TL建德JD0.006柏松BS0.0050.003捕捞BL0.0070.0060.006余杭YH0.0060.0040.0040.007景山JS0.0090.0070.0060.0090.005
基于COⅠ基因的6个群体间的遗传分化系数(Fst)为-0.009 34~0.193 55,大多在0.05~0.15,表明大部分群体间为中度遗传分化,且达到显著性水平(P<0.05);桐庐放流群体与柏松亲本群体间、桐庐放流群体与景山亲本群体间及柏松亲本群体与捕捞群体间的遗传分化系数均大于0.15,群体间为高度遗传分化,且达到极显著性水平(P<0.01);建德放流群体与余杭放流群体,柏松亲本群体与景山亲本群体,余杭群体、桐庐群体、建德群体与捕捞群体间的遗传分化系数均小于0.05,群体间为轻微的遗传分化,且未达到显著性水平(P>0.05)。从遗传分化指数看,大部分群体间具有一定程度遗传分化,但是群体间的基因流Nm大于1,部分群体间的基因流大于4(表5),说明群体间基因交流较频繁,制约种群间的遗传分化。
表5 基于 COⅠ 基因序列的三角鲂群体间遗传分化指数Fst(右上角)和基因流Nm(左下角)分析
Tab.5 Pairwise fixation indices(Fst, above diagonal)and gene flow(Nm, below diagonal)among populations of black bream Megalobrama terminalis based on COⅠgene

群体population桐庐TL建德JD柏松BS捕捞BL余杭YH景山JS桐庐TL0.07345∗∗0.19355∗∗0.003030.10880∗∗0.17383∗∗建德JD3.15370.13086∗∗0.01138-0.009340.07413∗∗柏松BS1.04171.66040.17366∗∗0.10614∗∗-0.00266捕捞BL82.258321.71841.18960.046050.13523∗∗余杭YH2.0478∞ 2.10545.17890.04969∗∗景山JS1.18823.1225∞1.59874.78120
将6个三角鲂群体不分组进行AMOVA分析,结果显示,群体间的遗传变异为9.91%,群体内的遗传变异为90.09%,遗传变异主要来自群体内;群体间遗传分化指数平均值为0.099 09,存在一定程度的遗传分化(P<0.01)(表6)。
将6个三角鲂群体分为两个组进行AMOVA分析,分为自然捕捞群体和养殖群体(包括2个亲本群体和3个放流群体),结果显示,组间遗传变异为-0.59%,组内群体间遗传变异为10.16%,群体内变异为90.44%,遗传变异主要来自群体内;组间遗传分化系数为-0.005 94,且未达到显著水平(P>0.05),这说明自然捕捞群体和养殖群体间不存在显著的遗传分化;群体间遗传分化指数平均值为0.095 64,存在一定程度的遗传分化(表6),这一结果与未分组的分析结果一致。
表6 三角鲂6个群体mtDNA COⅠ基因序列的分子方差分析(AMOVA)
Tab.6 Analysis of molecular variance(AMOVA)of mtDNA COⅠ gene in six populations of black bream Megalobrama terminalis
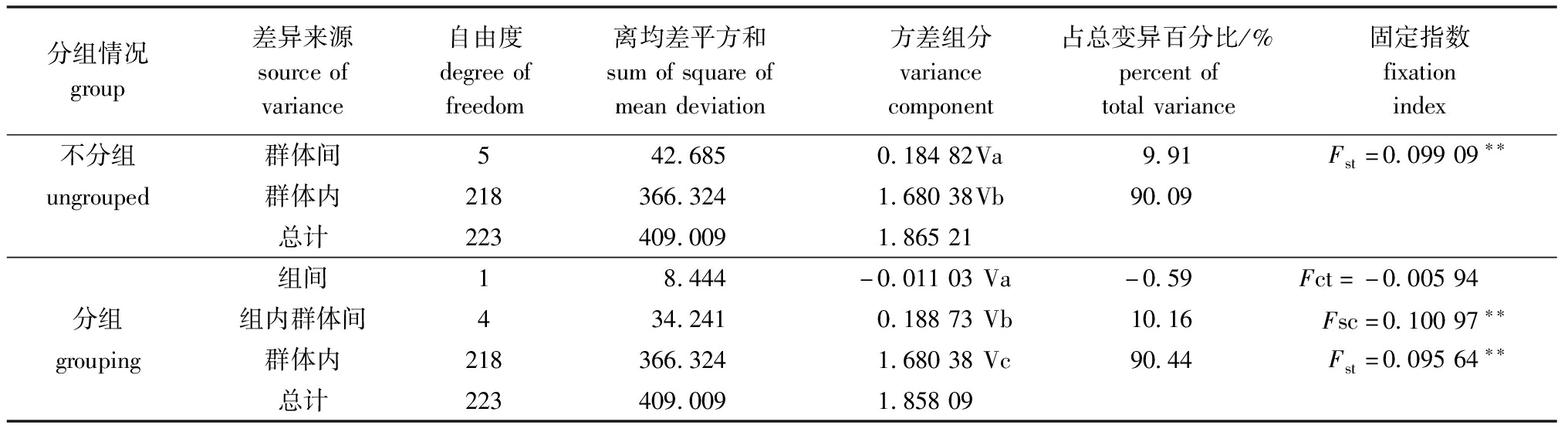
分组情况group差异来源sourceofvariance自由度degreeoffreedom离均差平方和sumofsquareofmeandeviation方差组分variancecomponent占总变异百分比/%percentoftotalvariance固定指数fixationindex不分组群体间542.6850.18482Va9.91Fst=0.09909∗∗ungrouped群体内218366.3241.68038Vb90.09总计223409.0091.86521组间18.444-0.01103Va-0.59Fct=-0.00594分组组内群体间434.2410.18873Vb10.16Fsc=0.10097∗∗grouping群体内218366.3241.68038Vc90.44Fst=0.09564∗∗总计223409.0091.85809
2.4 群体间系统进化分析
基于群体间Nei’s遗传距离(表4),采用UPGMA法构建系统进化树,结果显示,建德放流群体和柏松亲本群体最先聚为一支,然后依次与余杭放流群体、桐庐放流群体、自然捕捞群体聚在一起,最后与景山群体聚在一起(图3)。这表明,建德放流群体与柏松亲本群体亲缘关系最近,与景山亲本群体亲缘关系最远。
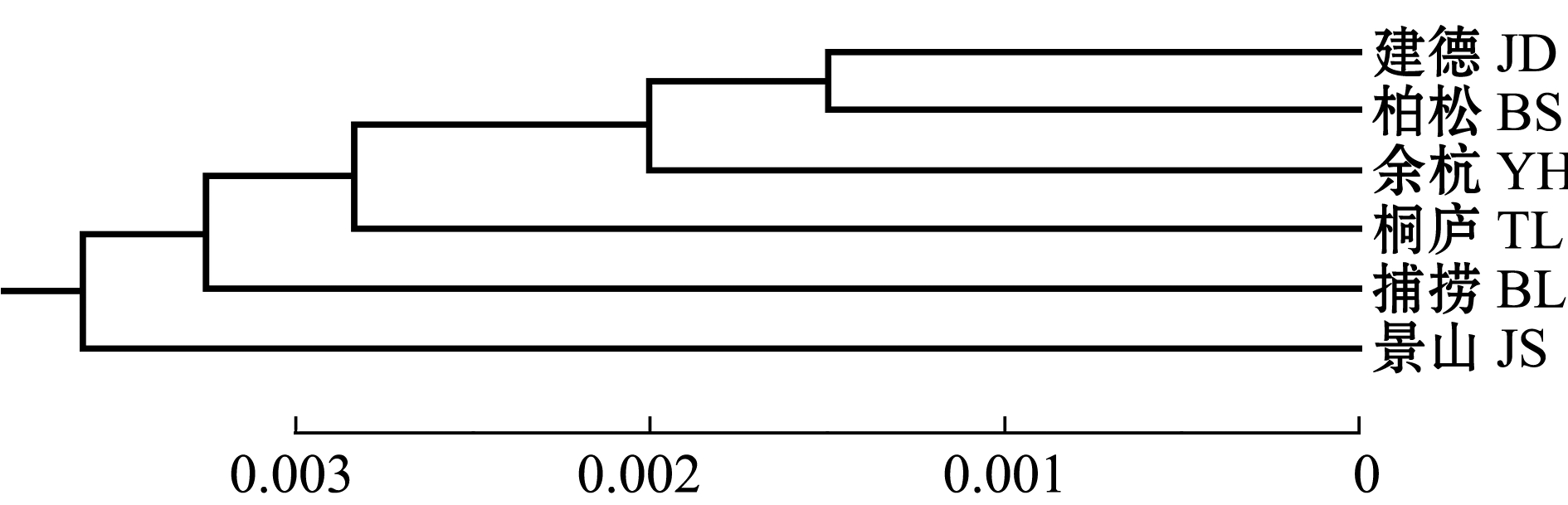
图3 基于遗传距离的三角鲂6个群体系统进化树
Fig.3 UPGMA molecular phylogenetic tree of six populations of black bream Megalobrama terminalis based on genetic distance
单倍型MJ网络图(图4)显示,hap9为第一优势单倍型,出现频率最高且位于网络图的中心,其他单倍型都是hap9经过一步或几步突变形成的,由此可以推测,hap9可能为最原始单倍型。每个群体都存在其特有的单倍型,也存在群体共享的单倍型,整体上并不存在明显的遗传分化。
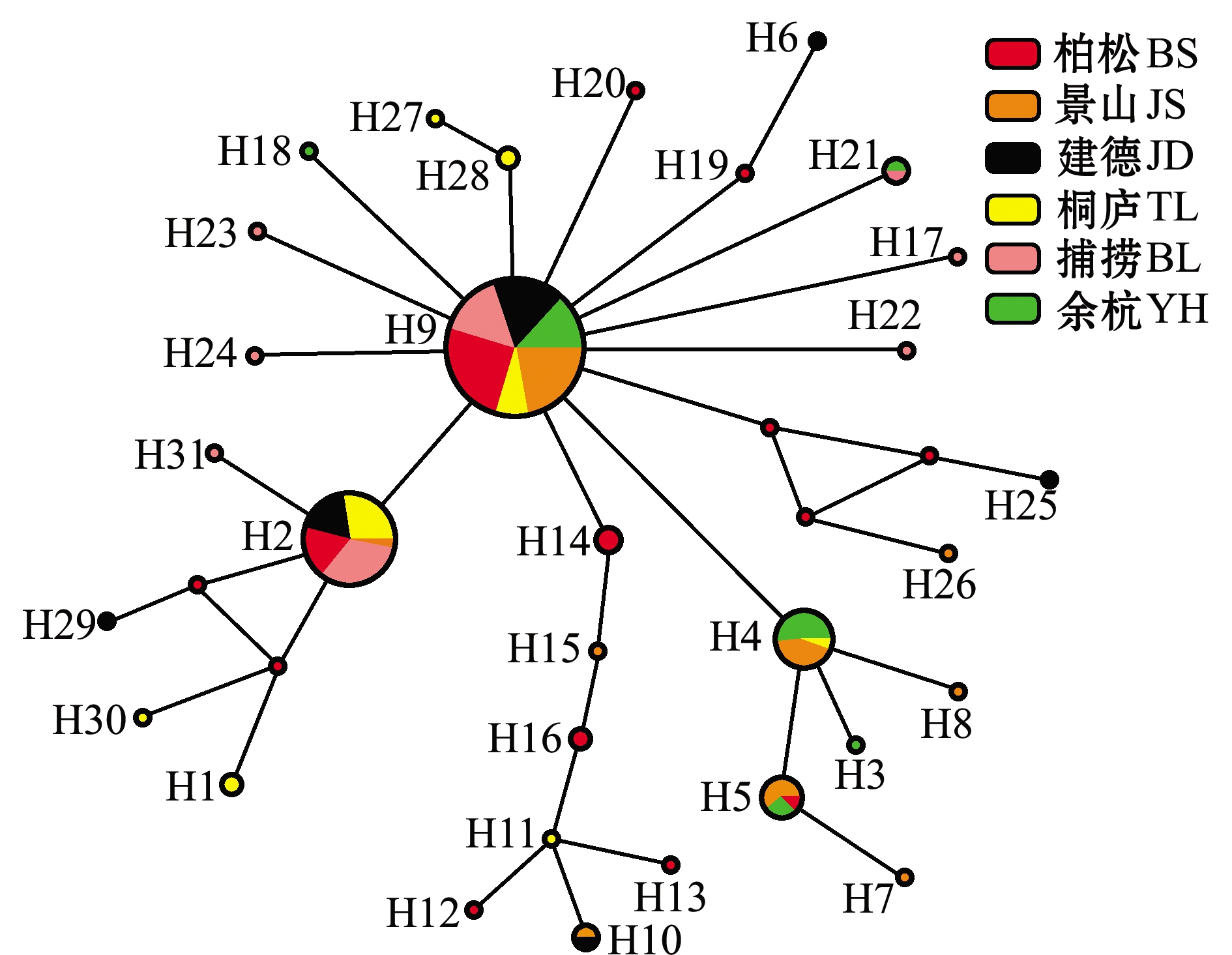
图4 三角鲂mtDNA COⅠ区31个单倍型的网络结构图
Fig.4 Network of mtDNA COⅠ region in 31 haplotypes of black bream Megalobrama terminalis
3 讨论
3.1 三角鲂6个群体的遗传多样性
遗传多样性是生物多样性的重要组成部分之一,是物种稳定和进化的基础。一个物种的遗传多样性或变异度越丰富,适应环境的能力就越强,具有的进化潜力就会越大[26]。本研究中,6个群体共检测到31个单倍型,单倍型种类较为丰富,且每个群体都含有其特有单倍型。其中,hap9是6个群体都含有的共享单倍型,这说明hap9单倍型有可能是十分稳定的,属于能够适应环境变化的原始单倍型,这与单倍型网络图显示的以hap9为网络中心的结果一致。遗传多样性判断的重要指标为单倍型多样性(Hd)和核苷酸多样性(Pi)。梁宏伟等[27]和潘贤辉等[28]均是利用单倍型多样性指数和核苷酸多样性指数判断群体遗传多样性的高低。Grant等[29]对海洋鱼类线粒体基因的研究表明,Hd>0.5,Pi>0.005时,说明单倍型多样性和核苷酸多样性高,这两个数值越大则表明遗传多样性越高。本研究中,6个三角鲂群体的Hd值范围为0.710~0.848(平均值为0.705),Pi值范围为0.003 07~0.008 40(平均值为0.005 78),因此,三角鲂6个群体作为一个种群来说,遗传多样性较高。其中,景山亲本群体、自然捕捞群体、桐庐放流群体的Hd值依次为0.779、0.710、0.848,Pi值依次为0.008 40、0.007 84、0.008 18,单倍型多样性均大于0.5且核苷酸多样性大于0.005,单倍型多样性和核酸多样性均较高,说明这3个群体的遗传多样性均较高。聂竹兰[14]指出,钱塘江野生三角鲂的遗传多样性较高,与本研究中自然捕捞群体的遗传多样性研究结果一致。本研究中建德放流群体、柏松亲本群体、余杭放流群体单倍型多样性较高(Hd>0.5),但核苷酸多样性较低(Pi<0.005),说明这3个群体的遗传多样性相对较低。
3.2 三角鲂群体的Tajima’s D检验及遗传分化
Tajima’s D值大于0代表群体观测杂合度高于预期杂合度,稀有等位基因频率降低(群体收缩或者平衡选择),小于0说明群体观测杂合位点少于预期值,稀有等位基因频率增加(群体扩张或者低频选择)[30]。本研究中,6个群体的D值为-2.658 38~-1.024 81,其中,3个群体的D值显著或极显著偏离0,6个群体作为一个总体时中性检验结果D值为-2.790 60,极显著偏离中性假说(P<0.001),这说明6个三角鲂群体内低频率基因(稀有基因)出现频率增大,群体在某种程度上经历了定向选择或群体个体数量的快速增长[31],这与本研究中三角鲂群体表现出的高单倍型多样性值和低核苷酸多样性值现象是吻合的,因为有研究表明,从一个较小的有效群体快速扩张而成的群体,极有可能积累了足够的单倍型多样性,并未积累足够的核苷酸序列多样性[32]。
本研究中,依据遗传距离可知,群体间遗传距离未达到亚种的分化水平[33]。本研究中基因流结果显示,大部分群体间的基因流Nm>1,其中,部分群体间的基因流Nm>4,显示6个群体间有着频繁的基因交流。近些年,在钱塘江中对三角鲂进行了大规模的增殖放流,两个亲本良种场群体(景山群体、柏松群体)提供三角鲂子代以供放流,3个放流群体(桐庐、余杭、建德群体)提供三角鲂冬片以供放流,因此,这5个养殖群体的三角鲂已经与钱塘江野生三角鲂频繁进行了基因交流,降低了这5个群体与天然捕捞群体间的遗传分化,这与分组AMOVA分析得出的养殖群体与天然捕捞群体间不存在显著的遗传分化结果是一致的(Fct=-0.005 94,P>0.05)。
本研究中,根据不分组群体间遗传分化指数为0.099 09可知,整体上群体间处于中等遗传分化水平,但并非所有群体间都为中等遗传分化[34],桐庐、余杭、建德3个放流群体与自然捕捞群体间遗传分化系数Fst<0.05,属轻度遗传分化,说明放流苗种遗传多样性与自然捕捞群体无显著性差异;而景山亲本群体、柏松亲本群体与自然捕捞群体间遗传分化程度相对较大(Fst为0.135 23和0.173 66),可能是放流群体直接与自然捕捞群体有基因交流,而亲本群体是通过繁育的子一代经过人工选择之后,再与自然捕捞群体进行基因交流。
3.3 三角鲂放流苗种的种质质量
本研究中,从单倍型多样性值和核苷酸多样性值来看,自然捕捞群体的遗传多样性低于景山群体和桐庐群体,高于余杭群体、柏松群体和建德群体。景山群体、桐庐群体与捕捞群体处于相近的遗传多样性水平,可认为不会对自然群体造成遗传学影响。捕捞群体的单倍型指数和核苷酸多样性指数均较高,三角鲂群体遗传多样性较为丰富,自然资源状况较好。分组AMOVA分析得出养殖群体(包括2个亲本群体和3个放流群体)与天然捕捞群体间不存在显著的遗传分化(表6,Fct=-0.005 94),说明放流的三角鲂群体进入钱塘江之后,可以较好地融入天然群体,进而补充钱塘江三角鲂资源量。增殖放流存在降低自然群体遗传多样性水平的可能性,因此,在增殖放流过程中选取良好的苗种是十分重要的。种质良好的苗种能保证放流群体和野生群体的遗传多样性处于同一水平,达到既补充自然资源又不改变自然资源的遗传结构的目的。
4 结论
1)两个亲本群体(景山和柏松群体)、3个放流群体(余杭、桐庐和建德群体)和捕捞群体遗传分化处于中等水平,基因交流较多。
2)自然捕捞群体的遗传多样性较高,钱塘江三角鲂种质资源较好。
3)自然捕捞群体与养殖群体间不存在显著的遗传分化,2个亲本群体和3个放流群体可作为良 好的增殖放流备选亲本群体和苗种群体。
[1] 成庆泰,郑葆珊.中国鱼类系统检索[M].北京:科学出版社,1987:135-136.
CHENG Q T,ZHENG Y S.China fish system search[M].Beijing:Science Press,1987:135-136.(in Chinese)(in Chinese)
[2] 陈宜瑜.中国动物志:硬骨鱼纲鲤形目(中卷)[M].北京:科学出版社,1998.
CHEN Y Y.Chinese animal records:the Cypriniformes of the bonefish(mei volume)[M].Beijing:Science Press,1998.(in Chinese)
[3] 陈马康.钱塘江鱼类资源[M].上海:上海科学技术文献出版社,1990:1-257.
CHEM M K.Qiantang River fish resources[M].Shanghai:Shanghai Science and Technology Literature Publishing House,1990:1-257.(in Chinese)
[4] 周少翌,曹富康,杜建明.钱塘江的渔业评价及合理利用渔业资源的初步设想[J].水利渔业,1989(6):21-23.
ZHOU S W,CAO F K,DU J M.The fishery evaluation of Qiantang River and the preliminary thoughts on rational utilization of fishery resources[J].Water Resources and Fisheries,1989(6):21-23.(in Chinese)
[5] 郝雅宾,刘金殿,张爱菊,等.钱塘江下游江段鱼类资源现状[J].浙江农业学报,2017,29(10):1620-1629.
HAO Y B,LIU J D,ZHANG A J,et al.The current status of fish resources in the lower reaches of Qiantang River[J].Journal of Zhejiang Agricultural Sciences,2017,29(10):1620-1629.(in Chinese)
[6] 宋文,王艺舟,祝东梅,等.三个地理种群鲂的形态差异与判别分析[J].水产学杂志,2013,26(4):1-7.
SONG W,WANG Y Z,ZHU D M,et al.Morphological differences and discriminant analysis of three geographical populations[J].Chinese Journal of Fisheries,2013,26(4):1-7.(in Chinese)
[7] 罗云林.鲂属鱼类的分类整理[J].水生生物学报,1990,14(2):160-165.
LUO Y L.A revision of fishes of the cyprinid genus Megalobeama[J].Acta Hydrobiologica Sinica,1990,14(2):160-165.(in Chinese)
[8] 吴成宾,李福贵,陈杰,等.鳊鲂鱼类的染色体核型及DNA含量分析[J].上海海洋大学学报,2015,24(6):801-809.
WU C B,LI F G,CHEN J,et al.Analysis of karyotype and DNA content of carp[J].Journal of Shanghai Ocean University,2015,24(6):801-809.(in Chinese)
[9] 万成炎,林永泰,黄道明.鲂胚胎的发育[J].湖泊科学,1999,11(1):70-74.
WAN C Y,LIN Y T,HUANG D M.Embryonic development of Megalobrama skolkovii[J].Journal of Lake Science,1999,11(1):70-74.(in Chinese)
[10] 林国辉,方展强,林爱薇.三角鲂(Megalobrama terminalis)脑垂体的超微结构[J].华南师范大学学报(自然科学版),2003(4):93-99.
LIN G H,FANG Z Q,LIN A W.Ultrastructure of the pituitary in Megalobrama terminalis[J].Journal of South China Normal University(Natural Science Edition),2003(4):93-99.(in Chinese)
[11] 谢楠,刘新轶,冯晓宇,等.鲂属鱼类细胞色素b片段序列分析[J].现代农业科技,2012(1):290-292.
XIE N,LIU X Y,FENG X Y,et al.Sequences analysis on mitochondrial cytochrome b gene fragment of Megalobrama spp.[J].Modern Agricultural Science,2012(1):290-292.(in Chinese)
[12] 赖瑞芳,张秀杰,李艳和,等.鲂属鱼类线粒体基因组的比较及其系统发育分析[J].水产学报,2014,38(1):1-14.
LAI R F,ZHANG X J,LI Y H,et al.Comparison of mitochondrial genomes of the genus Megalobrama and their phylogenetic analysis[J].Journal of Fisheries of China,2014,38(1):1-14.(in Chinese)
[13] HU X S,LUANP X,CAO C H,et al.Characterization of the mitochondrial genome of Megalobrama terminalis in the Heilong River and a clearer phylogeny of the genus Megalobrama[J].Scientific Reports,2019,9(1):8509.
[14] 聂竹兰.三角鲂转录组分析与不同地理种群遗传多样性研究[D].武汉:华中农业大学,2014.
NIE Z L.Triton transcriptome analysis and genetic diversity of different geographical populations[D].Wuhan:Huazhong Agricultural University,2014.(in Chinese)
[15] 施立明.遗传多样性及其保存[J].生物科学信息,1990,2(4):158-164.
SHI L M.Genetic diversity and conservation[J].Biological Science Information,1990,2(4):158-164.(in Chinese)
[16] 王亚楠,李秀启,客涵,等.基于线粒体COⅠ基因序列的南四湖外来须鳗虾虎鱼的种类鉴定[J].水产学杂志,2017,30(6):12-18.
WANG Y N,LI X Q,KE H,et al.Application of DNA barcoding gene COⅠ for indentifing Taenioides cirratus in Nansi Lake[J].Chinese Journal of Fisheries,2017,30(6):12-18.(in Chinese)
[17] 杨钰慧,谢佳燕,吴聪.基于线粒体COⅠ基因序列的武汉地区常见麦蚜蚜种鉴定[J].南方农业学报,2019,50(8):1742-1747.
YANG Y H,XIE J Y,WU C.Indentification of wheat aphid species in Wuhan based on mitochondrial coi gene sequence[J].Journal of Southern Agriculture,2019,50(8):1742-1747.
[18] 姜冰洁,傅建军,朱文彬,等.基于COⅠ基因分析7个罗非鱼群体的遗传变异[J].上海海洋大学学报,2019,28(6):827-834.
JIANG B J,FU J J,ZHU W B,et al.Genetic variation analysis of seven tilapia populations based on COⅠ gene[J].Journal of Shanghai Ocean University,2019,28(6):827-834.
[19] 汪曦,曾会玲,孟晓林,等.河南省小黄黝鱼群体遗传多样性[J].中国水产科学,2020,27(1):96-105.
WANG X,ZENG H L,MENG X L,et al.Genetic of wild Micropercops swinhonis populations in Henan province[J].Journal of Fishery Sciences of China,2020,27(1):96-105.(in Chinese)
[20] 徐岩,潘红平,阎冰,等.基于线粒体COⅠ和16S rRNA基因的中国沿海相手蟹系统发育研究[J].海洋学报,2019,41(8):63-71.
XU Y,PAN H P,YAN B,et al.Molecular phylogeny of the sesarmid crab based on the partial sequences of mitochondrial COⅠ and 16S rRNA genes from the coast of China[J].Acta Oceanologica Sinica,2019,41(8):63-71.(in Chinese)
[21] WARD R D,ZEMLAK T S,INNES B H,et al.DNA barcoding Australia’s fish species[J].Philosophical Transactions of the Royal Society of London.Series B:Biological Sciences,2005,360(1462):1847-1857.
[22] KRESS W J,WURDACK K J,ZIMMER E A,et al.Use of DNA barcodes to identify flowering plants[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102(23):8369-8374.
[23] SMITH M A,WOODLEY N E,JANZEN D H,et al.DNA barcodes reveal cryptic host-specificity within the presumed polyphagous members of a genus of parasitoid flies(Diptera:Tachinidae)[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(10):3657-3662.
[24] NEI M,LI W H.Mathematical model for studying genetic variation in terms of restriction endonucleases[J].Proceedings of the National Academy of Sciences of the United States of America,1979,76(10):5269-5273.
[25] LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
[26] 沈浩,刘登义.遗传多样性概述[J].生物学杂志,2001,18(3):5-7
SHEN H,LIU D Y.The role of metallothioneins and phytochelatins in heavy metal tolerance of plants[J].Journal of Biology,2001,18(3):5-7.(in Chinese)
[27] 梁宏伟,孟彦,罗相忠,等.基于线粒体COⅠ基因的6个黄鳝群体遗传多样性[J].中国水产科学,2018,25(4):837-846.
LIANG H W,MENG Y,LUO X Z,et al.Genetic diversity of six Monopterus albus populations based on COⅠ gene sequences[J].Journal of Fishery Sciences of China,2018,25(4):837-846.(in Chinese)
[28] 潘贤辉,周康奇,陈忠,等.基于线粒体D-loop区和COⅠ基因序列研究2个禾花鲤群体和野生鲤群体的遗传多样性与系统进化关系[J].淡水渔业,2019,49(6):33-40.
PAN X H,ZHOU K Q,CHEN Z,et al.Genetic diversity and phylogenetic relationship among two populations of Procypris merus and wild carp based on mtDNA D-loop region and COⅠ gene sequence[J].Freshwater Fisheries,2019,49(6):33-40.(in Chinese)
[29] GRANT W A S,BOWEN B W.Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J].Journal of heredity,1998,89(5):415-426.
[30] FU Y X.Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection[J].Genetics,1997,147(2):915-925.
[31] 孙鹏,彭士明,尹飞,等.南海海区鲻鱼(Mugil cephalus)COⅠ基因序列的遗传多样性分析[J].海洋与湖沼,2011,42(1):131-136.
SUN P,PENG S M,YIN F,et al.Genetic diversity of Mugil gephalus in the south China sea analyzed by mitochondrial COⅠ sequences[J].Oceanologia et Limnolgia Sinica,2011,42(1):131-136.(in Chinese)
[32] AVISE J C.Phylogegraphy:the history and formation of species[M].Cambridge,MA:Harvard University Press,2000.
[33] HEBERT P D N,RATNASINGHAM S,DE WARD J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceeding Biological Sciences,2003,270(sup 1):S96-S99.
[34] ALLENDORF F W.Isolation,gene flow,and genetic differentiation among populations[M]//CHONEWALD-COX C M,CHAMBERS S M,MACBRYDE B,et al.Genetics and conservation:a reference for managing wild animal and plant populations.Menlo Park:Benjamin Cummings Publishing,1983:51-65.