近年来,鱼类养殖业发展稳定,2019年中国水产品养殖产量达到5 079.07万t,鱼类占到2 708.61万t,其中,海水鱼类养殖产量160.58万t,淡水鱼类养殖产量2 548.03万t,养殖面积达711万hm2[1]。根据2018年联合国粮食及农业组织发布的报告,随着经济的增长,人们对优质蛋白质的选择趋势也增加,鱼产品作为蛋白质和必需微量营养素的重要来源之一,其发展的健康和稳定十分重要[2],并且鱼类产品消费的益处远大于危害[3]。然而,鱼类养殖生产活动易受到多种因素的影响,且鱼类生存对水质、水温等生存环境存在着严格的要求,近年来,为了满足水产品日益增长的需求,多数地区的水产养殖模式从粗放型转变为集约型,因此,极易导致养殖动物疾病的暴发[4],且不同的鱼类物种所面临的病害问题也不同,主要是细菌性和病毒性疾病。如由鳗弧菌Vibrio anguillarum引起的牙鲆Paralichthys olivaceus弧菌病[5],这对分布在中国、日本和韩国海域的海水养殖业造成了巨大的损失[6-7]。半滑舌鳎Cynoglossus semilaevis是分布在中国沿海地区具有极高经济价值的名贵鱼类,其生长快、肉质鲜美,但近年来随着其养殖规模的扩大,细菌病害威胁越来越严重,其中,哈维氏弧菌Vibrio harveyi是侵入半滑舌鳎的主要细菌性病原之一,能够引起烂鳍等疾病[8-9]。此外,哈维氏弧菌也可诱发大黄鱼Larimichthys crocea弧菌病[10],更为严重的是若误食感染哈维氏弧菌的食物或水,会引发人腹泻或败血症、中耳炎等疾病[11]。罗非鱼 Oreochromis spp.是联合国粮食与农业组织推荐的21世纪重要淡水养殖品种之一,是全球未来动物性蛋白质的主要来源之一[12],但罗非鱼集约化养殖模式的大规模推广及养殖环境的恶化,使得病害暴发越来越严重,特别是无乳链球菌Streptococcus agalactiae感染的传播速度和引起的死亡率逐渐增加。2009年以来,在以色列、厄瓜多尔、埃及、泰国和印度等多个国家和地区的罗非鱼中暴发了一种由罗非鱼湖病毒(Tilapia Lake virus, TiLV)引起的病毒性疾病,被世界动物卫生组织列为一种新生病毒,对罗非鱼养殖业造成了严重威胁[13-14]。除此之外,鲫疱疹病毒(Carassius auratus herpesvirus, CaHV)和草鱼Ctenopharyngodon idellus呼肠孤病毒(Grass carp reovirus, GCRV)等其他多种鱼类病害也频频暴发。因此,为保障鱼类养殖业的绿色健康发展,培育抗病力强的优良品种就显得十分重要。
1 鱼类抗病育种的起源
从病理学研究上来看,人们已经掌握了多种治疗或预防鱼类疾病的方法,如使用抗生素、化疗药物及接种疫苗等。但过度使用抗生素不仅会使细菌产生耐药性,抗生素进入到人体中也会对人造成伤害[11];化疗药物的使用易造成环境污染及副作用发生;而疫苗接种及益生菌和免疫刺激剂的使用等相关方法在养殖实践中难以大规模实施[15]。因此,国内外专家开始研究鱼类固有的抗病遗传因素,并进行选择育种从而建立抗病品系[16]。与其他物种相比,鱼类的选择育种研究起源较晚。
国外第一次有文献记载的鱼类选择育种是1920年在美洲红点鲑Salvelinus fontinalis中进行的抗病新品系培育[17]。国内科学系统的鱼类选择育种起源较晚,20世纪70年代出现相关研究[18]。抗病育种相关研究起源于吴维新等[19]将鲤和草鱼杂交一代与草鱼回交获得三倍体草鱼型杂种,并经多年从杂交一代异缘四倍体中选育出可育的二倍体83-2系抗病草鱼[20]。
近年来,随着鱼类遗传学的不断创新发展,以及对鱼类基因组的探索,研究人员建立了一系列的育种技术,包括家系与群体选育、分子标记辅助选育和全基因组选育等。
2 国内鱼类家系及群体选育研究
家系选育是应用较为广泛的育种方式,也为分子育种奠定了基础。其原理是通过逐代提高目的基因的纯合度,建立优良性状家系,获得具有优良性状且稳定的新品种,实际上就是对基因型的选择。近年来,国内外研究人员采用此方法在多种鱼类的抗病育种工作中取得了重要进展,如牙鲆、虹鳟Oncorhynchus mykiss和草鱼等。通过加性遗传方差分析证明,对虹鳟病毒性出血性败血病抗病力进行选育的方法是可行的[21]。通过自然选择和人工感染迟缓爱德华氏菌Edwardsiella tarda,对63个牙鲆家系的生长和抗病性能进行初步测定,从中鉴定得到3个抗病力强的家系[22];进一步以抗病牙鲆品种为亲本建立了57个家系,选择其中46个家系进行感染试验,从中鉴定出5个抗病家系[23];从28个优质抗病牙鲆家系中筛选得到7个高抗病力(攻毒感染存活率>66%)的家系[24];在建立的抗淋巴囊肿(Lymphocystis disease,LD)牙鲆品系中,通过在淋巴囊肿病高发养殖场进行天然攻毒试验,得到G2家系,以此为基础继续选育,有望得到抗LD牙鲆新品种[25]。另外,遗传评定模型分析表明,采用家系选育的方式适合对半滑舌鳎抗哈维氏弧菌性状进行选育[26]。在野生草鱼中已被证实抗GCRV能力较好的个体遗传力较高,为构建草鱼选择育种提供了基础[27]。
除此之外,采用上述类似的方法在其他鱼类选择育种中也取得了一定进展。许氏平鲉 Sebastes schlegelii 的遗传改良和优质新品系选育就是通过群体选育和家系选育相结合的方法进行的[28]。罗非鱼不同家系的抗链球菌能力存在显著性差异[29];通过对42个吉富罗非鱼 Oreochromis niloticus 家系人工腹腔注射感染无乳链球菌,评估筛选出抗病力强且生长速度快的7个家系[30];吉富罗非鱼经过2个世代的选育,抗病能力便得到了较大提高[31]。罗非鱼不同品种间的抗病力也不同[32-35],这为新品种的选育提供了基础和材料。此外,镜鲤Cyprinus carpio var.specularis抗锦鲤疱疹病家系选育已经进行到了F3代[36]。
以上研究结果均表明,家系选育是培育鱼类抗病优良品种的有效途径,且已在多种鱼类选育中取得成功,证明其是培育抗病新品系的重要手段。
3 鱼类抗病选育的分子标记辅助研究
常规的鱼类育种选育策略是利用细菌或病毒对种群内的个体感染以筛选出抗病能力较强的个体留种选育。但因鱼类的抗病力不仅受遗传影响,而且受环境影响,表型变异不能完全或真实地反映遗传变异[37],所以这种方法可信度不高,并且选育周期长。随着遗传学研究的发展,尤其是“数量遗传学理论”的提出,动物育种技术进入到快速发展时期。数量遗传学理论的观点是生物的若干性状(如抗病)是由许多微小的基因共同决定的,这些微小基因被称为数量性状位点(quantitative trait locus,QTL)。但若仅研究控制某一性状的多个微小基因,无法准确定位到每一个微小基因并准确估计其单个基因的效应[38]。分子标记技术是传统育种选育技术的辅助手段,即保留了家系选育的优点,也能够对于某些性状的QTL位点进行精确定位,筛选和培育优良品种。其基本原理是利用与目标基因紧密连锁或共分离的分子标记进行区域或全基因组筛选,从而减少连锁冗余,达到提高育种效率的目的。目前,在鱼类抗病育种中应用较多的分子标记辅助选育技术主要有微卫星标记和单核苷酸多态性(SNP)技术两种。
3.1 鱼类抗病相关微卫星标记的筛选及QTL定位
微卫星标记是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成,由于重复单位的重复次数在个体间呈高度变异性且数量丰富,故该技术应用广泛。采用微卫星标记技术对不同家系间的遗传结构和差异进行分析,构建高密度遗传连锁图谱并对抗病基因进行精准的QTL定位,可为后续的抗病选育奠定基础。
在中国,由微卫星标记构建的鱼类高密度遗传连锁图谱首先在鲤 Cyprinus carpio[39]、半滑舌鳎[40]、牙鲆[41]、鲢Hypophthalmichthys molitrix[42]和鳙Aristichthys nobilis[43] 等几种重要经济鱼类中完成(表1)。目前,将与 LD 抗性相关的遗传连锁图谱连锁群LG15上的主要基因座LD-R作为标记辅助育种的候选微卫星基因座,成功获得牙鲆Paralichthys olivaceus抗病品系[6, 44]。采用基因分型微卫星识别方法已成功对大西洋鲑Salmo salar 抗传染性鲑鱼贫血症进行QTL定位[45]。利用微卫星标记技术已成功对4种大菱鲆Scophthalmus maximus品系的杀鲑气单胞菌Aeromonas salmonicida抗性进行QTL定位[46]。利用微卫星标记技术在吉富品系尼罗罗非鱼中筛选出2个与抗病性状相关的位点[47]。采用混合分离子分析法(bulked segregant analysis,BSA)对半滑舌鳎中169个微卫星标记进行筛选,在感染试验存活率为52.22%的家系中得到了1个可能与抗哈维氏弧菌相关的微卫星标记,并成功对其进行了QTL定位,这为选育抗病新品种提供了基础和依据[9]。目前,在多种鱼类中完成了对抗病基因的精准QTL定位(表2)。
表1 部分鱼类微卫星遗传连锁图谱构建情况
Tab.1 Development of microsatellite genetic linkage map in some fishes
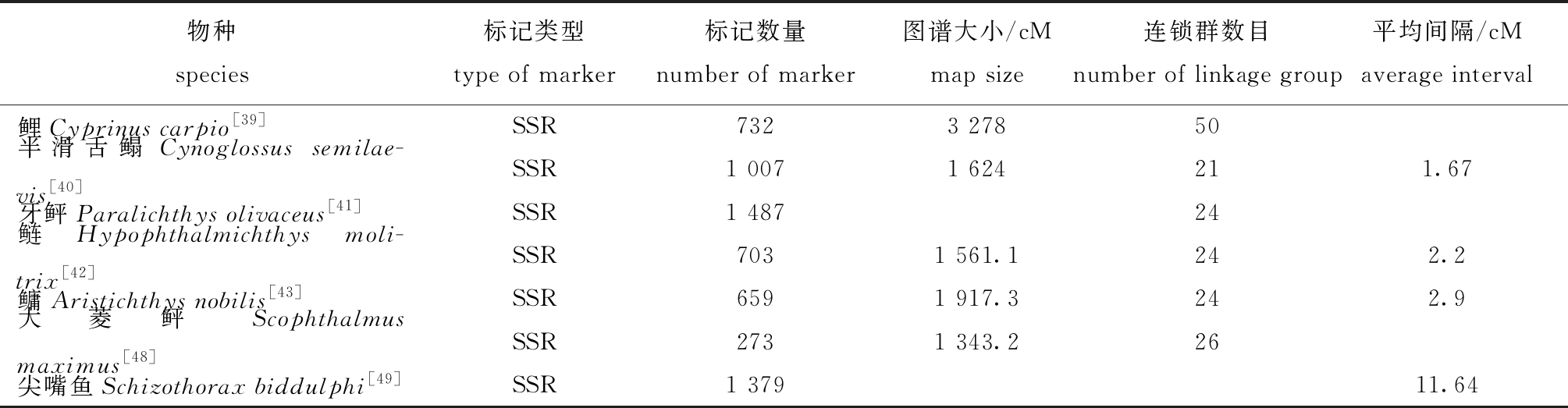
物种species标记类型type of marker 标记数量number of marker图谱大小/cMmap size连锁群数目number of linkage group平均间隔/cMaverage interval 鲤Cyprinus carpio[39]SSR7323 27850半滑舌鳎Cynoglossus semilae-vis[40]SSR1 0071 624211.67牙鲆Paralichthys olivaceus[41]SSR1 48724鲢 Hypophthalmichthys moli-trix[42]SSR7031 561.1242.2鳙 Aristichthys nobilis[43]SSR6591 917.3242.9大菱鲆 Scophthalmus maximus[48]SSR2731 343.226尖嘴鱼Schizothorax biddulphi[49]SSR1 37911.64
表2 部分鱼类抗病相关QTL定位
Tab.2 Mapping of QTL related to disease resistance in some fishes
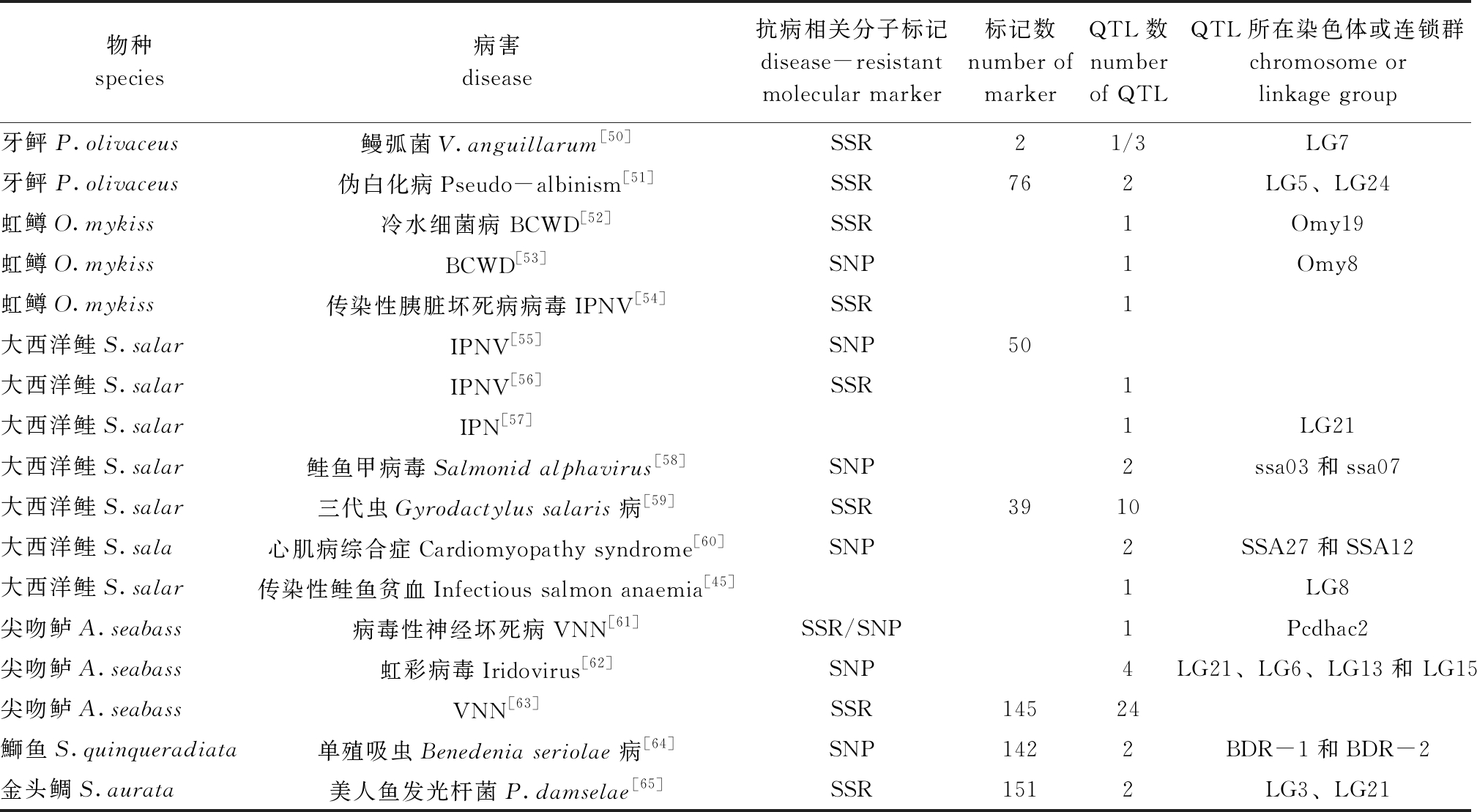
物种species病害disease抗病相关分子标记disease-resistant molecular marker标记数number of markerQTL数number of QTLQTL所在染色体或连锁群chromosome or linkage group牙鲆P.olivaceus鳗弧菌V.anguillarum[50]SSR21/3LG7牙鲆P.olivaceus伪白化病Pseudo-albinism[51]SSR762LG5、LG24虹鳟O.mykiss冷水细菌病 BCWD[52]SSR1Omy19虹鳟O.mykissBCWD[53]SNP1Omy8虹鳟O.mykiss传染性胰脏坏死病病毒IPNV[54]SSR1大西洋鲑S.salarIPNV[55]SNP50大西洋鲑S.salarIPNV[56]SSR1大西洋鲑S.salarIPN[57]1LG21大西洋鲑S.salar鲑鱼甲病毒Salmonid alphavirus[58]SNP2ssa03和ssa07大西洋鲑S.salar三代虫Gyrodactylus salaris病[59]SSR3910大西洋鲑S.sala心肌病综合症Cardiomyopathy syndrome[60]SNP2SSA27和SSA12大西洋鲑S.salar传染性鲑鱼贫血Infectious salmon anaemia[45]1LG8尖吻鲈A.seabass病毒性神经坏死病VNN[61]SSR/SNP1Pcdhac2尖吻鲈A.seabass虹彩病毒Iridovirus[62]SNP4LG21、LG6、LG13和 LG15尖吻鲈A.seabassVNN[63]SSR14524鰤鱼S.quinqueradiata单殖吸虫Benedenia seriolae病[64]SNP1422BDR-1和BDR-2金头鲷S.aurata美人鱼发光杆菌P.damselae[65]SSR1512LG3、LG21
3.2 基于候选基因的鱼类抗病相关SNP标记筛选
SNP是第三代分子标记技术,能够建立起更加密集的SNP图谱,遗传稳定性高、位点丰富且分布广泛、易于基因分型,从而定位出与目的性状更加紧密相关的标记或QTL区间[66]。近年来,SNP标记筛选技术在鱼类育种中得到广泛应用(表3)。
表3 部分鱼类抗病相关SNP标记的筛选研究进展
Tab.3 Progress of SNP markers related to disease resistance in some fishes
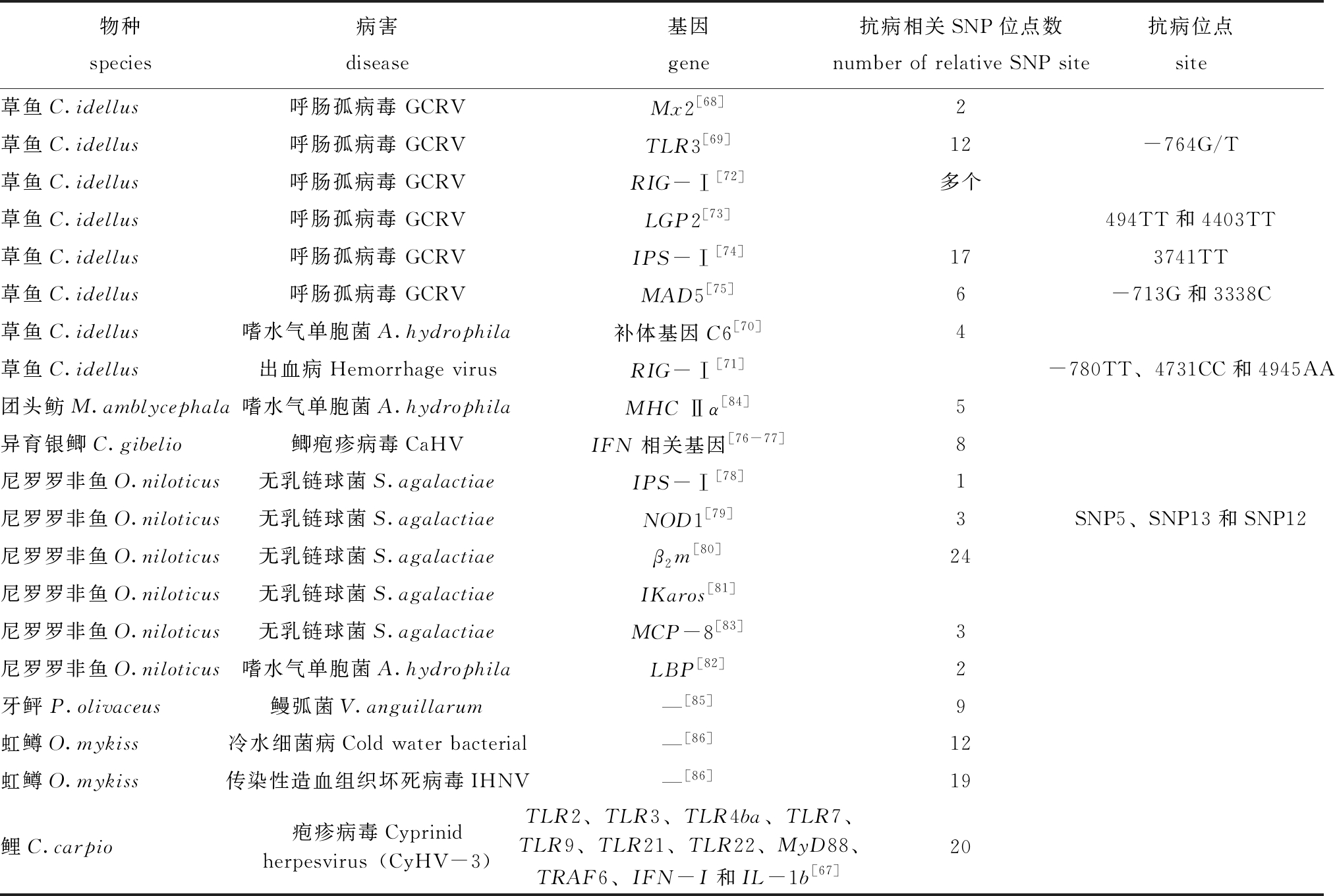
物种species病害disease基因gene抗病相关SNP位点数number of relative SNP site抗病位点site草鱼C.idellus呼肠孤病毒 GCRVMx2[68]2草鱼C.idellus呼肠孤病毒 GCRVTLR3[69]12-764G/T草鱼C.idellus呼肠孤病毒 GCRVRIG-Ⅰ[72]多个草鱼C.idellus呼肠孤病毒 GCRVLGP2[73]494TT和4403TT草鱼C.idellus呼肠孤病毒 GCRVIPS-Ⅰ[74]173741TT草鱼C.idellus呼肠孤病毒 GCRVMAD5[75]6-713G和3338C草鱼C.idellus嗜水气单胞菌A.hydrophila补体基因C6[70]4草鱼C.idellus出血病Hemorrhage virusRIG-Ⅰ[71]-780TT、4731CC和4945AA团头鲂M.amblycephala嗜水气单胞菌A.hydrophilaMHC Ⅱα[84]5异育银鲫C.gibelio鲫疱疹病毒CaHVIFN相关基因[76-77]8尼罗罗非鱼O.niloticus无乳链球菌S.agalactiaeIPS-Ⅰ[78]1尼罗罗非鱼O.niloticus无乳链球菌S.agalactiaeNOD1[79]3SNP5、SNP13和SNP12尼罗罗非鱼O.niloticus无乳链球菌S.agalactiaeβ2m[80]24尼罗罗非鱼O.niloticus无乳链球菌S.agalactiaeIKaros[81]尼罗罗非鱼O.niloticus无乳链球菌S.agalactiaeMCP-8[83]3尼罗罗非鱼O.niloticus嗜水气单胞菌A.hydrophilaLBP[82]2牙鲆P.olivaceus鳗弧菌V.anguillarum—[85]9虹鳟O.mykiss冷水细菌病Cold water bacterial—[86]12虹鳟O.mykiss传染性造血组织坏死病毒IHNV—[86]19鲤C.carpio疱疹病毒Cyprinid herpesvirus(CyHV-3)TLR2、TLR3、TLR4ba、TLR7、TLR9、TLR21、TLR22、MyD88、TRAF6、IFN-I和IL-1b[67]20
评估SNP位点的多态性并采用Snapshot法进行分型验证,在鲤的11个基因中检测到20个可用于连锁分析的标记[67]。草鱼作为中国主要的淡水养殖鱼类,病害频发严重,缺乏良种,但草鱼的优良性状选育已经取得了重要的进展。目前,利用测序筛查SNP位点,并采用Snapshot进行分型验证的方法,在草鱼抗呼肠孤病毒基因Mx2、TLR3(Toll样受体基因3)、补体C6、RIG-Ⅰ(视黄酸(维甲酸)诱导基因蛋白Ⅰ)、LGP2(RIG-Ⅰ样受体家族成员遗传生理学实验室2,laboratory of genetics and physiology 2)、MAD5(黑色素瘤分化相关基因5, melanoma differentiation-associated gene 5)和IPS-Ⅰ(β-干扰素启动子刺激因子Ⅰ, interferon-b promoter stimulator Ⅰ)中发现多个对草鱼GCRV抗性或易感性性状显著相关的SNP位点及单倍型。这些SNP位点和单倍型可以作为潜在的分子标记辅助筛选草鱼抗出血病品系[68-75]。通过人工感染筛选出异育银鲫Carassiusauratus gibelio 易感克隆系和抗性克隆系,进一步鉴定出8个干扰素(interferon, IFN)相关基因与抗CaHV的侵染有关,可为今后异育银鲫抗病育种提供科学依据[76-77]。利用测序筛查SNP位点并采用Snapshot进行分型验证的方法,在罗非鱼IPS-Ⅰ、NOD1(核苷酸结合和寡聚化结构域1,nucleotide binding and oligomerization domain 1)、β2m(β2-微球蛋白,β2-microglobulin)、IKaros(C2H2型锌指蛋白类转录因子)、LBP(脂多糖结合蛋白, lipopolysaccharide binding protein)和MCP-8(肥大细胞蛋白酶-8, mast cell protease-8)基因中筛选到与无乳链球菌抗性/易感相关单倍型或SNP位点[78-83]。这些SNP位点和单倍型可以作为潜在的分子标记辅助筛选罗非鱼抗链球菌病品系。
4 候选基因MHC多态性与抗病性的相关性
主要组织相容性复合体(major histocompatibility complex,MHC)是编码动物主要组织相容性抗原基因群的统称,分为MHCⅠ类和Ⅱ类分子,由其所编码的细胞表面转膜蛋白可以结合内外源抗原并呈递给T淋巴细胞,从而引发免疫反应。多基因性、高度多态性是与抗病力相关的MHC类基因的两个重要特性,目前公认其是研究与抗病/敏感表型相关联的多态性分子标记最合适的候选基因。Miretti等[87]2005年发表了涵盖人MHC基因区域4.46-Mb高分辨率(1.9 kb)的连锁不平衡图谱及第一代标签SNP,这使得基因分型效率增加了5倍,从而可用于未来与MHC相关的所有疾病研究中。由于鱼类MHC的高度多态性,其在抗病选育中备受关注,如在大西洋鲑、虹鳟和镜鲤等鱼类中发现,MHC位点具有高度多态性且与抗病力有关[88-89]。
4.1 鱼类MHCⅠ 类基因
1990年,研究者第一次扩增出鱼类的MHC ⅠA链基因[90],1993年,在大西洋鲑中测得第一条鱼类MHC ⅠA链 cDNA全序列[91],随后陆续报道了多种鱼类的MHC Ⅰ类基因cDNA序列。不同的硬骨鱼类中MHC基因的拷贝数不同,但其结构与哺乳动物相似。
4.2 鱼类MHC Ⅱ类基因
1993年,第一条鱼类MHC ⅡA基因cDNA序列首次在斑马鱼中被报道[92]。根据目前所得到的鱼类MHC ⅡA cDNA分子序列,推测其蛋白质结构与哺乳动物相似。MHC ⅡB基因是MHC基因中被研究最多的,其具有丰富的多态性,如牙鲆MHC ⅡB基因的多态性[93]。
4.3 MHC 基因多态性与鱼体抗病性的关系
1999年,Van Muiswinkel等[94]提出MHC与鱼类疾病抗性具有重要关系。之后,国内外对多种鱼体抗病性与MHC多态性的关系进行了研究(表4)。如急性病毒感染过程中,虹鳟MHC Ⅰ基因在病毒感染期间表达显著下调[89]。大西洋鲑MHC Ⅰ和MHC Ⅱ类基因的多态性也与抗贫血病毒和疖病有关[95]。
表4 MHC基因多态性与鱼体疾病抗性的关系
Tab.4 Relationship between MHC gene polymorphism and fish disease resistance
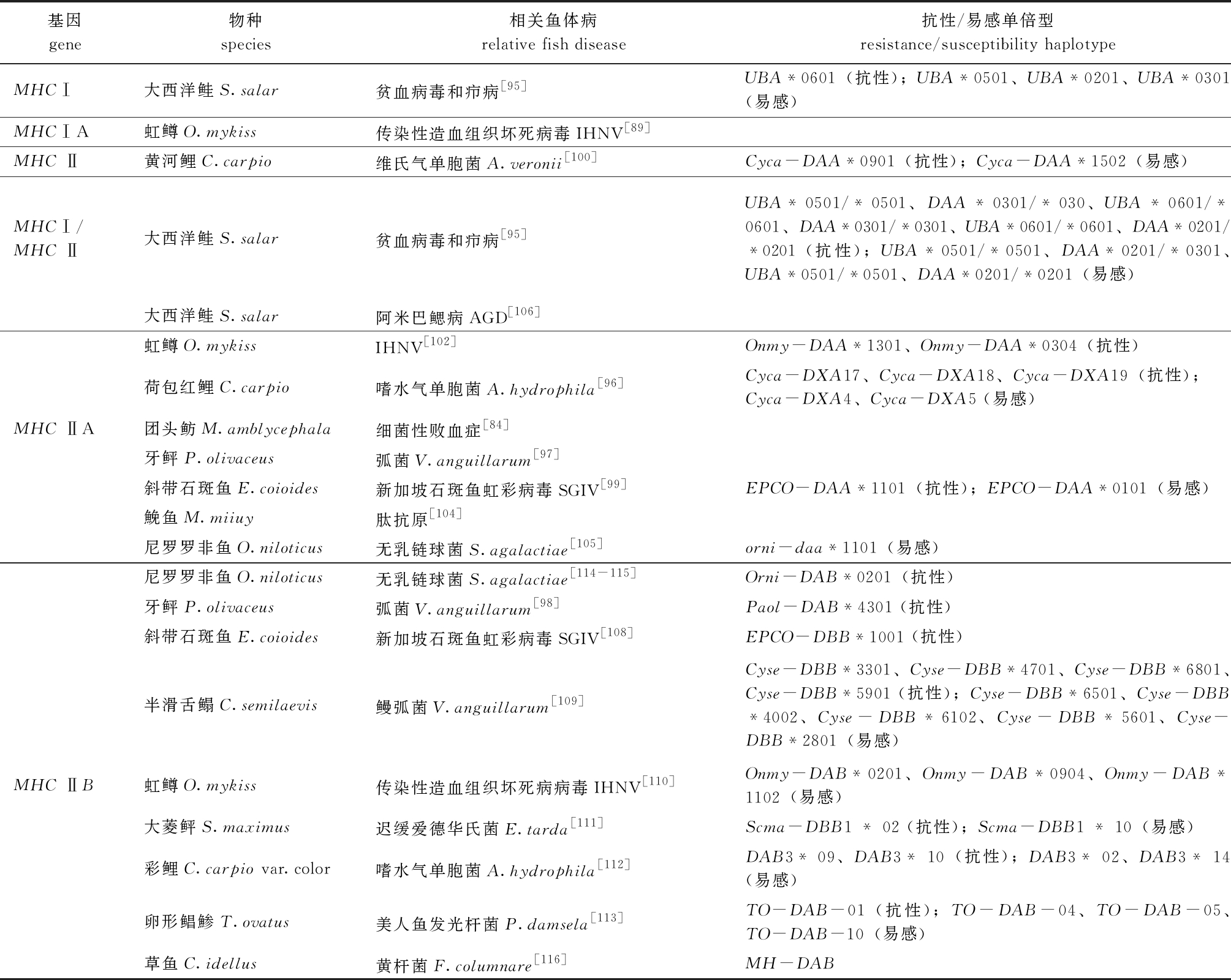
基因gene物种species相关鱼体病relative fish disease抗性/易感单倍型resistance/susceptibility haplotypeMHCⅠ大西洋鲑S.salar贫血病毒和疖病[95]UBA*0601(抗性);UBA*0501、UBA*0201、UBA*0301(易感)MHCⅠA虹鳟O.mykiss传染性造血组织坏死病毒IHNV[89]MHC Ⅱ黄河鲤C.carpio维氏气单胞菌A.veronii[100]Cyca-DAA*0901(抗性);Cyca-DAA*1502(易感)MHCⅠ/MHC Ⅱ 大西洋鲑S.salar贫血病毒和疖病[95]UBA*0501/*0501、DAA*0301/*030、 UBA*0601/*0601、DAA*0301/*0301、UBA*0601/*0601、DAA*0201/*0201(抗性);UBA*0501/*0501、DAA*0201/*0301、UBA*0501/*0501、DAA*0201/*0201(易感)大西洋鲑S.salar阿米巴鳃病AGD[106]虹鳟O.mykissIHNV[102]Onmy-DAA*1301、Onmy-DAA*0304(抗性)荷包红鲤C.carpio嗜水气单胞菌A.hydrophila [96]Cyca-DXA17、Cyca-DXA18、Cyca-DXA19(抗性);Cyca-DXA4、Cyca-DXA5 (易感)MHC ⅡA团头鲂M.amblycephala细菌性败血症[84]牙鲆P.olivaceus弧菌V.anguillarum[97]斜带石斑鱼E.coioides新加坡石斑鱼虹彩病毒SGIV[99]EPCO-DAA*1101(抗性);EPCO-DAA*0101(易感)鮸鱼M.miiuy肽抗原[104]尼罗罗非鱼O.niloticus无乳链球菌S.agalactiae[105]orni-daa*1101(易感)尼罗罗非鱼O.niloticus无乳链球菌S.agalactiae[114-115]Orni-DAB*0201(抗性)牙鲆P.olivaceus弧菌V.anguillarum[98]Paol-DAB*4301 (抗性)斜带石斑鱼E.coioides新加坡石斑鱼虹彩病毒SGIV[108]EPCO-DBB*1001 (抗性)半滑舌鳎C.semilaevis鳗弧菌V.anguillarum[109]Cyse-DBB*3301、Cyse-DBB*4701、Cyse-DBB*6801、Cyse-DBB*5901 (抗性);Cyse-DBB*6501、Cyse-DBB*4002、Cyse-DBB*6102、Cyse-DBB*5601、Cyse-DBB*2801(易感)MHC ⅡB虹鳟O.mykiss传染性造血组织坏死病病毒IHNV[110]Onmy-DAB*0201、Onmy-DAB*0904、Onmy-DAB*1102(易感)大菱鲆S.maximus迟缓爱德华氏菌E.tarda[111]Scma-DBB1 * 02 (抗性);Scma-DBB1 * 10(易感)彩鲤C.carpio var.color嗜水气单胞菌A.hydrophila[112]DAB3* 09、DAB3* 10(抗性);DAB3* 02、DAB3* 14(易感)卵形鲳鯵T.ovatus美人鱼发光杆菌P.damsela[113]TO-DAB-01(抗性);TO-DAB-04、TO-DAB-05、TO-DAB-10(易感)草鱼C.idellus黄杆菌F.columnare[116]MH-DAB
4.3.1 MHC ⅡA基因多态性与抗病性 荷包红鲤 Cyprinus carpio Linnaeus MHC ⅡA类基因具有较高的多态性,等位基因Cyca-DXA4和Cyca-DXA5与对嗜水链球菌的敏感性显著相关,而Cyca-DXA17、Cyca-DXA18和Cyca-DXA19与其对嗜水链球菌病抗性相关[96]。团头鲂MHC ⅡA基因的多态性与抗细菌性败血症性状也具有显著关联[84]。在牙鲆60个家系的MHC ⅡA和MHC ⅡB基因中,筛选到与弧菌病抗性密切相关的抗病/敏感家系MHC等位基因,其可作为分子标记进行抗弧菌病牙鲆品系的选育[97-98]。通过新加坡石斑鱼虹彩病毒(Singapore grouper iridovirus,SGIV)侵染试验研究了MHC ⅡA基因多态性与斜带石斑鱼Epinephelus coioides 抗病/敏感性的关系,结果得到了37个高多态性等位基因,经过验证分析发现,与SGIV敏感性和SGIV抗性相关的等位基因各一个[99]。在黄河鲤中发现多个维氏气单胞菌Aeromonas veronii易感性相关的等位基因,经验证MHC Ⅱ类基因在黄河鲤抵抗病原入侵的过程中有着不可或缺的重要作用[100]。大西洋鲑MHC Ⅱ基因多态性与对鲑疮痂鱼虱Lepeophtheirus salmonis的敏感性间具有相关性[101]。在虹鳟中采用聚合酶链反应-SSCP分析技术等方法检测MHC ⅡA类DAA基因(尤其是外显子2)的多态性,并研究其与对传染性造血器官坏死病病毒(IHNV)抵抗力的关系,结果发现与抗性有关的两个等位基因Onmy-DAA*1301和Onmy-DAA*0304[102]。从鮸鱼Miichthys miiuy中发现共有48个(Mimi-DAB)功能基因,并预测有14个MHC ⅡB类位点与抗原结合位点相关;还鉴定出40个MHC ⅡA类(Mimi-DAA)功能基因,预测有5个位点与肽抗原的结合有关[103-104]。尼罗罗非鱼个体中MHC ⅡA类基因多态性与无乳链球菌感染的易感程度间的关联分析表明,等位基因orni-daa*1101与无乳链球菌的易感性有关;而MHC ⅡA等位基因第2外显子可用于探索疾病易感性或抗性与MHC ⅡA的多样性间的联系[105]。Wynne等[106]利用MHC Ⅱ类基因作为分子标记成功地筛选出了抗阿米巴鳃病(Amoebic gill disease, AGD)的大西洋鲑品系。
4.3.2 MHC ⅡB基因多态性与抗病性 在关于MHC ⅡB基因的研究中,研究人员通过分析抗病和不抗病牙鲆的MHC ⅡB基因多态性,筛选得到与抗病相关联的MHC ⅡB等位基因[93]。采用RACE法成功克隆出赤点石斑鱼Epinephelus akaara MHC ⅡB基因的cDNA全序列,并利用单链构象多态性(SSCP)分析技术证明了赤点石斑鱼MHC ⅡB的表达多态性[107]。对斜带石斑鱼MHC ⅡB基因多态性也进行了研究,关联分析结果证明,等位基因EPCO-DBB*1001与SGIV抗性显著相关[108]。在对半滑舌鳎MHC ⅡB基因多态性与抗病相关性的研究中,得到9个与鳗弧菌病抗性/敏感性相关的位点[109]。在虹鳟中鉴定出45个不同的MHC ⅡB类等位基因,其中Onmy-DAB*0201、Onmy-DAB*0904和Onmy-DAB*1102与IHNV易感性相关[110]。在大菱鲆抗病性与MHC ⅡB基因多态性的关系研究中发现,与抗病/敏感相关的MHC ⅡB基因型各1个[111]。在“全红”体色瓯江彩鲤Cyprinus carpio var.color中也发现了与抗病/易感病群体相关的MHC ⅡB基因型[112]。卵形鲳鯵Trachinotus ovatus MHC ⅡB 基因第2外显子多态性与美人鱼发光杆菌Photobacterium damsela抗性/敏感性有关,关联分析结果表明,1个等位基因与其抗性有关,3个等位基因与其敏感性有关[113]。根据尼罗罗非鱼F1代基因型和F3代链球菌抗性/易感性状进行关联分析,结果表明,MHC ⅡB等位基因 Orni-DAB*0107、 Orni-DAB*0201和Orni-DAB*0302与链球菌抗性显著相关, 等位基因Orni-DAB*0701与链球菌易感显著相关[114]。在该研究之前,对尼罗罗非鱼的研究也表明, MHCⅡB等位基因Orni-DAB*0201与链球菌抗性显著相关[115]。因此,MHCⅡB等位基因Orni-DAB*0201可以作为尼罗罗非鱼抗链球菌病选育的分子标记,用于将来的抗病辅助育种。
5 全基因组抗病选择育种研究
2001年Meuwissen等[117]提出了全基因组选择的概念。用覆盖全基因组的分子标记进行辅助选育的手段,是对传统标记辅助选育的创新和改进,这已经成为动物遗传改良的研究热点及运用到育种生产中的重要手段。其主要方法是通过全基因组中大量的高密度遗传标记估计出不同染色体片段或单个标记效应值,然后将个体全基因组范围内片段或标记效应值累加,从而获得基因组估计育种值。目前,利用大黄鱼基因组育种值筛选抗性亲本取得了较好的效果[118]。所以全基因组测序及高密度遗传图谱的建立和遗传育种值的预测模型在全基因组选择技术中十分重要。
5.1 全基因组测序
国外对鱼类全基因组测序的研究起步较早,始于20世纪90年代中期。2002年研究者完成第一个全基因组测序的鱼类——红鳍东方鲀 Takifugu rubripes[119]。随着测序技术的发展,越来越多的鱼类全基因组被测序,如青鳉Oryzias latipes[120]、斑马鱼Danio rerio[121]、虹鳟[122]和大西洋鲑[123]等28种鱼类。
国内对鱼类全基因组测序的研究起步较晚,2014年,中国完成了半滑舌鳎的全基因组测序和精细图谱的绘制[124];同年,鲤的全基因组测序完成[125]。目前,研究人员已完成了包括4种滩涂鱼Scartelaos histophorus、 Boleophthalmus pectinirostris、 Periophthalmodon schlosseri、 Periophthalmus magnuspinnatus[126],以及大黄鱼[127]、草鱼[128]、牙鲆[129]、鞍带石斑鱼 Epinephelus lanceolatus[130]等共23种鱼类的全基因组测序[131]。
5.2 基于SNP标记的精细遗传图谱构建
目前,多种鱼类的高密度遗传连锁图谱已构建完成,如在牙鲆中构建了12 712个SNP标记平均间隔为0.47 cM的高密度遗传连锁图谱,并定位得到9个与鳗弧菌抗性相关的QTL位点[85]。除此之外,团头鲂Megalobrama amblycephala[132]、中华鲟Acipenser sinensis[133]、鳙[134]、鲤[135]等其他鱼类的高密度遗传连锁图谱也构建完成[131]。
5.3 全基因组抗病育种研究
5.3.1 基于高通量测序技术的全基因组SNP位点的分型 随着高通量SNP标记检测成本的不断降低,以及基因分型和测序技术的发展,全基因组选择技术开始被成功应用于育种实践当中。目前,基于2b-RAD高通量测序的基因型分型技术已建立并被广泛应用[136]。采用2b-RAD方法进行基于SNPs的基因分型,可以有效地捕获抗病力的遗传变异[137-138]。根据草鱼基因组SNP电子芯片对感染GCRV后的全同胞群体草鱼中存活个体和死亡个体的基因分型进行基因组重测序,从筛选得到的具有显著差异的1 822个基因型位点中获得9个抗病相关基因和10个显性等位基因位点,进一步采用高通量SNP分型技术筛选野生群体中携带抗病基因的草鱼个体,以此为亲本人工繁育出抗病群体[128]。降低基因分型成本的策略:1)选择极端表型(EP)或预选择SNP以构建候选基因的低密度标记,而且单标记分析是预选择SNP中节省候选基因分型成本的最可行方法;2)在基因组选择中,利用极端表型选择和单标记分析相结合的方法,显示出了较好的结果[139]。Wang等[140]也建立了一种高通量、低成本的基因型分析技术。
5.3.2 全基因组育种值的预测方法 在全基因组抗病育种技术研究过程中,研究人员开发了多种全基因组育种值预测方法(表5)。对虹鳟的研究发现,所有全基因组育种值的预测方法精度都高于系谱的BLUP(P-BLUP)方法,其中,贝叶斯C(BayesC)的精度相对提高最多[141-142]。在大黄鱼Larimichthys crocea育种中,采用基于测序的基因分型构建NGS测序文库,得到约30 000个SNPs,根据拟合预测精度曲线估计基因组预测中特定性状的算法达到理想预测精度所需的训练规模,并成功评估了预选择SNPs的可行性[143]。目前,Dong等[144]开发了两种新的大黄鱼基因组育种值的计算策略:一种是针对非基于马尔科夫链-蒙特卡洛算法(MCMC)的快速混合正态分布(FMixP)策略,另一种是针对基于MCMC的MixP(MMixP)策略。该研究中使用4种方法BayesA、BayesCπ、MMixP和FMixP来比较预测结果,结果表明,对于模拟数据,BayesCπ、MMixP和FMixP的性能优于BayesA,FMixP在计算上比基于MCMC的方法快得多,但是所有方法对于大黄鱼基因组育种值的预测能力都非常相似,该研究结果可能被应用于基因组育种值预测中;另外,Dong等[145]还提出了一种计算速度明显快于BayesC的快速贝叶斯C(fBayesC)方法,在基因组育种值预测上是可行的;该研究者还研究了对SNP基因型进行标准化与否条件下先验超参数的两种策略,并分别计算了7种预测模型(岭回归BLUP、BayesA、BayesB、BayesCπ、快速BayesB(fBayesB)、FMixP和MMixP)在两种策略下的基因组育种值,提出了先进行SNP基因型标准化,再设定先验分布参数值的建议[146]。“鲆优2号”牙鲆新品种培育过程中,采用BayesCπ和GBLUP两种算法进行独立个体育种值估算,从而建立了抗病牙鲆品系的基因组选择育种技术,成功培育出“鲆优2号”牙鲆新品种[147]。对半滑舌鳎良种选育研究表明,通过BayesCπ模型进行全基因组选择育种会取得较好效果,为通过芯片分型对半滑舌鳎全基因组选择育种奠定了基础[148]。
表5 全基因组选择计算方法
Tab.5 Calculation methods of whole genome selection
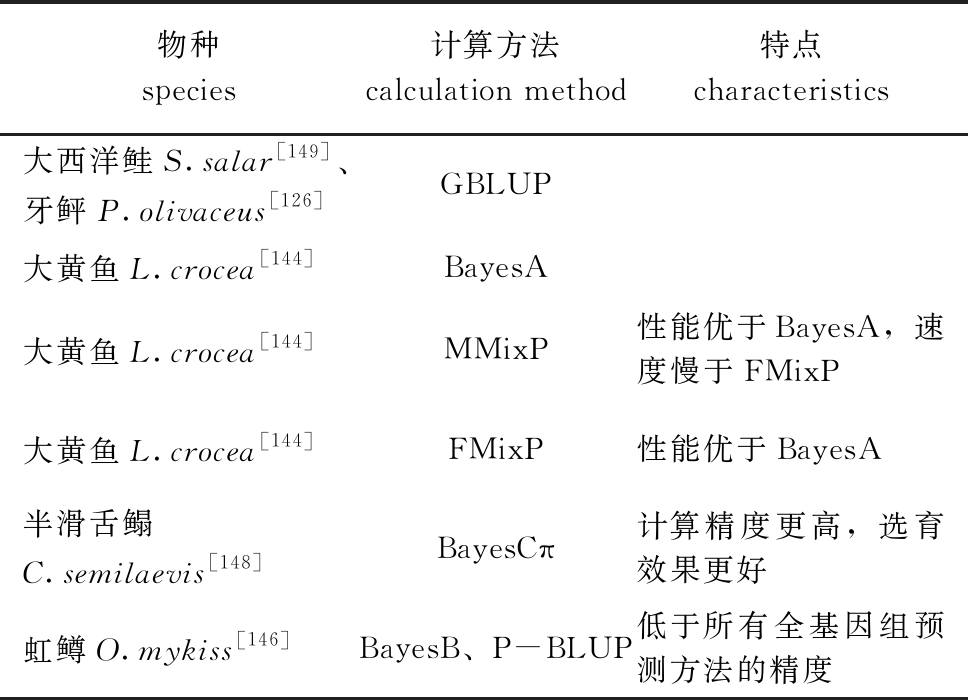
物种species计算方法calculation method特点characteristics大西洋鲑S.salar[149]、牙鲆P.olivaceus[126]GBLUP大黄鱼L.crocea[144]BayesA大黄鱼L.crocea[144]MMixP性能优于BayesA,速度慢于FMixP大黄鱼L.crocea[144]FMixP性能优于BayesA半滑舌鳎C.semilaevis[148]BayesCπ计算精度更高,选育效果更好虹鳟O.mykiss[146]BayesB、P-BLUP低于所有全基因组预测方法的精度
5.3.3 基于全基因组关联分析筛选抗病相关SNP位点 全基因组关联分析(GWAS)是筛选抗病相关SNP位点的重要手段。在鲈抗病研究中,采用一种简化的高通量测序技术——限制性位点相关DNA测序方法得到了病毒性神经坏死鲈的全基因组SNP数据,并对其进行GWAS,检测了个体SNP与VNN抗性间的关联[150]。对大西洋鲑的育种研究表明,基因组预测精度比传统预测模型获得的基因组预测精度高18%[151]。对大西洋鲑进行GWAS,获得5个与抗AGD显著相关的SNP位点[152]。在半滑舌鳎抗病性研究中,通过GWAS、Fst和核苷酸多样性筛选结合的方法识别对抗病性重要的位点,在基因组重测序中鉴定的 1 016 774 个SNPs位点中检测到 33 个与抗病性显著关联的SNPs位点[153]。
5.3.4 基于GWAS的QTL定位 采用GWAS法进行QTL定位,可以为鱼类抗病提供有价值的候选基因。如采用GWAS法发现了4个与斑点叉尾鮰Ictalurus punctatus的柱状病抗性相关的QTL[149],并发现了9个与斑点叉尾鮰爱德华氏菌Edwardsiella ictalurid免疫性相关的QTL[154]。在虹鳟中进行GWAS,检测到10个与抗病相关的QTL[155]。在感染锦鲤疱疹病毒(Koi herpesvirus,KHV)镜鲤的重要QTL或SNP周围发现了许多与免疫相关的基因,包括tnfa、hif1a、galectin-8、rootletin和palladin,并利用公开的KHV感染鲤的转录组数据,获得了候选基因的表达模式,这为鲤对KHV抗性的遗传结构研究提供了许多候选基因[114]。在鲤的LG 44上鉴定出一个与KHV抗性相关的QTL[156]。对虹鳟30 060个SNP进行GWAS,检测到几个QTL可能与抗冷水细菌病有关[157]。对大西洋鲑进行GWAS,在第二染色体的QTL区域发现3个可能与抗AGD有关的候选基因[152]。在大西洋鲑Ssa27和Ssa12染色体上发现两个与心肌炎病毒(piscine myocarditis virus,PMCV)有关的QTL,其中包括多个免疫相关基因,如MHC Ⅱ类基因等[158]。
6 存在问题及展望
在世界范围内,水产养殖业的发展速度快于所有其他食用动物生产部门,但却面临着巨大的疾病感染风险[4]。鱼类病害种类多样、病因复杂且传播性强,解决病害问题并减少病害带来的损失对整个水产养殖行业具有重要意义。近年来,随着育种技术的不断创新发展,育种水平不断提高,传统选育与分子标记辅助选育技术的结合,以及全基因组选择模型的不断优化,动植物育种工作都取得了很大的进展,同时在鱼类育种研究中也取得了一定进步。
6.1 抗病育种面临的问题
1)鱼类基因组资源开发发展迅速,但是后基因组时代,鱼类基因功能研究包括抗病相关基因功能研究相对滞后。影响鱼类基因功能研究的主要因素是相对于哺乳类,鱼类工具细胞系的建立相对滞后,目前建立的细胞系普遍存在转染效率低下的现象,严重影响了鱼类基因功能的研究、验证。另外,尚未建立以CRISPR/Cas9等为代表的基因组编辑技术和平台,严重影响了重要抗病性状关键基因的发掘及其在抗病育种中的应用。
2)鱼类抗病育种相关的基础研究及抗病分子育种的研究相对滞后。目前,对鱼类免疫系统的免疫机制研究还处于初级阶段。对鱼类免疫系统及其调控机制进行深入系统的研究,是推动鱼类抗病育种的基础,而抗病分子育种的研究是抗病育种的重要手段。
3)鱼类抗病育种的基因组选择研究尚处于初级阶段。价格低廉的分子标记筛选高通量分型平台尚未建立,鱼类抗病性状的界定手段还无统一的标准,全基因组育种值的统计方法还处于摸索阶段。
6.2 未来重点研究方向
1)鱼类免疫系统的解析。由于鱼类免疫知识的缺乏,限制了免疫系统进化研究、疫苗研发及抗病品种(系)的选育,因此,将来需要加强鱼类免疫系统的解析研究,包括鱼类先天免疫和获得性免疫系统的研究,同时也包括了免疫负调控因子的挖掘及其在先天免疫中作用机制的解析。
2)鱼类全基因结构解析及基因资源的深度挖掘。包括全基因组测序和精细图谱构建,鱼类基因组的拼接、组装和生物信息学分析技术,阐释不同鱼类基因组的结构特征、进化规律和功能,进行基因资源的深度挖掘,包括参考基因组序列及其注释(编码和非编码调节元件)、全基因组多态性标记、高效基因分型平台、高密度和高分辨率连锁图及转录组资源(包括非编码转录本)。
3)加强鱼类抗病性状的遗传解析及分子育种的基础研究。借助基因组编辑平台和鱼类工具细胞系培养技术,筛选抗病性状关键基因,研究和验证基因的具体功能和调控机制,阐明基因对抗病性状的决定机制,解析抗病性状基因的调控网络,为分子设计育种奠定理论基础;深入开展抗病性状的GWAS和基因组选择及分子设计的基础研究,包括全基因组育种值的统计方法研究、分子标记筛选的高通量分型平台技术的建立(如抗病育种芯片)、鱼类抗病性状界定方法标准的建立;拓展抗病性状相关分子标记和关键基因的筛选与鉴定研究,建立基因与抗病性状的关联性;定位抗病性状基因的QTL,明确基因型与抗病表型的关系。
4)确定抗病性状遗传和表观遗传调控。通过基因组甲基化分析、组蛋白修饰及小核糖核酸(RNA)分析等技术手段,解析环境因素介导的抗病性状形成的表观遗传调控机制;建立甲基化修饰因子在水产动物中的检测技术体系,筛选与抗病性状相关的表观遗传标记。
[1] 农业农村部渔业渔政管理局,全国水产技术推广总站,中国水产学会.2020中国渔业统计年鉴[M].北京:中国农业出版社,2020.
[2] 联合国粮食及农业组织.世界渔业和水产养殖状况-实现可持续发展目标[M].罗马:联合国粮食及农业组织,2018.
[3] FAO,WHO.Report of the joint FAO/WHO expert consultation on the risks and benefits of fish consumption[R].Rome:FAO,WHO,2011.
[4] INA-SALWANY M Y,AL-SAARI N,MOHAMAD A,et al.Vibriosis in fish:a review on disease development and prevention[J].Journal of Aquatic Animal Health,2019,31(1):3-22.
[5] 徐田军,陈松林,田永胜,等.牙鲆抗鳗弧菌病家系筛选及其分析[J].中国水产科学,17(1):59-68.
[6] FUJI K,KOBAYASHI K,HASEGAWA O,et al.Identification of a single major genetic locus controlling the resistance to lymphocystis disease in Japanese flounder(Paralichthys olivaceus)[J].Aquaculture,2006,254(1/2/3/4):203-210.
[7] LIU Y G,CHEN S L,LI B F.Assessing the genetic structure of three Japanese flounder(Paralichthys olivaceus)stocks by microsatellite markers[J].Aquaculture,2005,243(1/2/3/4):103-111.
[8] 徐晓丽,邵蓬,李贺密,等.半滑舌鳎体表溃疡病病原的分离鉴定[J].中国海洋大学学报,2015,45(11):29-35.
[9] 戴欢,刘洋,王文文,等.半滑舌鳎抗哈维氏弧菌病相关微卫星标记筛选及QTL定位[J].中国水产科学,2017,24(1):22-30.
[10] 吴立婷,廖金轩,庞茂达,等.大黄鱼中哈维氏弧菌毒力及耐药特性分析[J].食品安全质量检测学报,2019,10(8):2111-2119.
[11] 杨少丽,王印庚,董树刚.海水养殖鱼类弧菌病的研究进展[J].海洋水产研究,2005,26(4):75-83.
[12] 郭富强,张德锋,曹建萌,等.不同水温下无乳链球菌在尼罗罗非鱼体内的动态分布及其消除[J].渔业科学进展,2018,39(6):81-88.
[13] MUGIMBA K K,CHENGULA A A,WAMALA S,et al.Detection of tilapia lake virus(TiLV)infection by PCR in farmed and wild Nile tilapia(Oreochromis niloticus)from Lake Victoria[J].Journal of Fish Diseases,2018,41(8):1181-1189.
[14] 李嘉波,秦真东,赵丽娟,等.罗非鱼湖病毒对吉富罗非鱼和E-11细胞的感染[J].水产学报,2020,44(1):142-155.
[15] DAS S,SAHOO P K.Markers for selection of disease resistance in fish:a review[J].Aquaculture International,2014,22(6):1793-1812.
[16] 喻子牛,孔晓瑜,戴继勋.鱼类抗病遗传(上)[J].海洋湖沼通报,1995(2):63-69.
[17] EMBODY G C,HAYFORD C O.The advantage of rearing brook trout fingerlings from selected breeders[J].Transactions of the American Fisheries Society,2011,55(1):135-148.
[18] 桂建芳,周莉,张晓娟.鱼类遗传育种发展现状与展望[J].中国科学院院刊,2018,33(9):932-939.
[19] 吴维新,李传武,刘国安,等.鲤和草鱼杂交四倍体及其回交三倍体草鱼杂种的研究[J].水生生物学报,1988,12(4):355-363.
[20] 吴维新,李传武,曾国清.抗病草鱼(83-2系鱼)选育的研究[J].中国水产科学,1997,4(2):74-78.
[21] HENRYON M,JOKUMSEN A,BERG P,et al.Genetic variation for growth rate,feed conversion efficiency,and disease resistance exists within a farmed population of rainbow trout[J].Aquaculture,2002,209(1/2/3/4):59-76.
[22] 陈松林,田永胜,徐田军,等.牙鲆抗病群体和家系的建立及其生长和抗病性能初步测定[J].水产学报,2008,32(5):665-673.
[23] 郑卫卫,陈松林,李泽宇,等.牙鲆抗迟缓爱德华氏菌性状的遗传力和育种值分析[J].农业生物技术学报,2016,24(8):1181-1189.
[24] 李泽宇,陈松林,郭华,等.牙鲆抗迟缓爱德华氏菌病家系的继代选育与早期速生家系筛选[J].渔业科学进展,2018,39(3):36-43.
[25] 侯吉伦,王桂兴,张晓彦,等.牙鲆抗淋巴囊肿病家系选育及生长和抗病性能分析[J].中国水产科学,2017,24(4):727-737.
[26] 卢昇,李泽宇,李仰真,等.半滑舌鳎抗哈维氏弧菌病的遗传力和育种值分析[J].农业生物技术学报,2018,26(10):1770-1777.
[27] HUANG R,SUN J X,LUO Q,et al.Genetic variations of body weight and GCRV resistance in a random mating population of grass carp[J].Oncotarget,2015,6(34):35433-35442.
[28] 刘阳,韩慧宗,王腾腾,等.许氏平鲉(Sebastes schlegelii)速生选育群体的生长性能与遗传特征分析[J].海洋与湖沼,2018,49(2):432-443.
[29] 朱佳杰,李莉萍,唐瞻杨,等.吉富罗非鱼家系构建及抗病力检测[J].南方水产科学,2012,8(6):22-27.
[30] 沈夏霜,敖秋桅,甘西,等.吉富罗非鱼抗病品系F5代抗病性能和生长性能的评估[J].南方水产科学,2018,14(3):83-90.
[31] 朱佳杰,周宇,敖秋桅,等.吉富罗非鱼抗病选育品系的养殖效果评估[J].安徽农业科学,2015,43(16):157-158,207.
[32] 朱佳杰,周宇,谭芸,等.不同品系罗非鱼抗无乳链球菌病性能的评估[J].安徽农业科学,2015,43(10):168-170.
[33] 强俊,杨弘,马昕羽,等.吉富罗非鱼与奥利亚罗非鱼自繁与杂交F1遗传特性与抗病力分析[J].水产学报,2015,39(1):32-41.
[34] 祝璟琳,邹芝英,李大宇,等.四个罗非鱼选育品种抗链球菌病能力差异研究[J].水生生物学报,2017,41(6):1232-1241.
[35] 祝璟琳,邹芝英,李大宇,等.四种正交子一代奥尼罗非鱼耐链球菌病能力差异研究[J].农业生物技术学报,2019,27(1):127-138.
[36] 白姗姗,贾智英,郎延贺,等.CyHV-3感染镜鲤选育世代免疫基因表达及抗病能力比较[J].上海海洋大学学报,2017,26(5):651-657.
[37] 刘伟,苏胜彦,董在杰,等.3个鲤群体的微卫星标记与生长性状相关性分析[J].南方水产科学,2012,8(3):17-24.
[38] 张慧,王守志,李辉.畜禽全基因组选择[J].东北农业大学学报,2010,41(3):145-149.
[39] ZHANG X F,ZHANG Y,ZHENG X H,et al.A consensus linkage map provides insights on genome character and evolution in common carp(Cyprinus carpio L.)[J].Marine Biotechnology,2013,15(3):275-312.
[40] SONG W T,LI Y Z,ZHAO Y W,et al.Construction of a high-density microsatellite genetic linkage map and mapping of sexual and growth-related traits in half-smooth tongue sole(Cynoglossus semilaevis)[J].PLoS One,2012,7(12):e52097.
[41] SONG W T,PANG R Y,NIU Y Z,et al.Construction of high-density genetic linkage maps and mapping of growth-related quantitative trail loci in the Japanese flounder(Paralichthys olivaceus)[J].PLoS One,2012,7(11):e50404.
[42] GUO W J,TONG J G,YU X M,et al.A second generation genetic linkage map for silver carp(Hypophthalmichthys molitrix)using microsatellite markers[J].Aquaculture,2013,412/413:97-106.
[43] ZHU C,TONG J,YU X,et al.A second-generation genetic linkage map for bighead carp(Aristichthys nobilis)based on microsatellite markers[J].Animal Genetics,2014,45(5):699-708.
[44] FUJI K,HASEGAWA O,HONDA K,et al.Marker-assisted breeding of a lymphocystis disease-resistant Japanese flounder(Paralichthys olivaceus)[J].Aquaculture,2007,272(1/2/3/4):291-295.
[45] MOEN T,SONESSON A K,HAYES B,et al.Mapping of a quantitative trait locus for resistance against infectious salmon anaemia in Atlantic salmon(Salmo salar):comparing survival analysis with analysis on affected/resistant data[J].BMC Genetics,2007,8:53.
[46] RODR GUEZ-RAMILO S T,TORO M A,BOUZA C,et al.QTL detection for Aeromonas salmonicida resistance related traits in turbot(Scophthalmus maximus)[J].BMC Genomics,2011,12:541.
GUEZ-RAMILO S T,TORO M A,BOUZA C,et al.QTL detection for Aeromonas salmonicida resistance related traits in turbot(Scophthalmus maximus)[J].BMC Genomics,2011,12:541.
[47] 朱佳杰,周宇,沈夏霜,等.吉富品系尼罗罗非鱼抗病性状分子标记筛选及遗传多样性分析[J].华北农学报,2013,28(sup 1):16-21.
[48] BOUZA C,HERMIDA M,MILL N A,et al.Characterization of EST-derived microsatellites for gene mapping and evolutionary genomics in turbot[J].Animal Genetics,2008,39(6):666-670.
N A,et al.Characterization of EST-derived microsatellites for gene mapping and evolutionary genomics in turbot[J].Animal Genetics,2008,39(6):666-670.
[49] LUO W,NIE Z L,ZHAN F B,et al.Rapid development of microsatellite markers for the endangered fish Schizothorax biddulphi(Günther)using next generation sequencing and cross-species amplification[J].International Journal of Molecular Sciences,2012,13(11):14946-14955.
[50] WANG L,FAN C X,LIU Y,et al.A genome scan for quantitative trait loci associated with Vibrio anguillarum infection resistance in Japanese flounder(Paralichthys olivaceus)by bulked segregant analysis[J].Marine Biotechnology,2014,16(5):513-521.
[51] FUJI K,HASEGAWA O,SAKAMOTO T,et al.Quantitative trait loci associated with pseudo-albinism in Japanese flounder,Paralichthys olivaceus[J].Aquaculture,2007,272 (sup 1): S257.
[52] WIENS G D,VALLEJO R L,LEEDS T D,et al.Assessment of genetic correlation between bacterial cold water disease resistance and spleen index in a domesticated population of rainbow trout:identification of QTL on chromosome Omy19[J].PLoS One,2013,8(10):e75749.
[53] PALTI Y,VALLEJO R L,GAO G T,et al.Detection and validation of QTL affecting bacterial cold water disease resistance in rainbow trout using restriction-site associated DNA sequencing[J].PLoS One,2015,10(9):e0138435.
[54] OZAKI A,SAKAMOTO T,KHOO S,et al.Quantitative trait loci(QTLs)associated with resistance/susceptibility to infectious pancreatic necrosis virus(IPNV)in rainbow trout(Oncorhynchus mykiss)[J].Molecular Genetics and Genomics,2001,265(1):23-31.
[55] HOUSTON R D,DAVEY J W,BISHOP S C,et al.Characterisation of QTL-linked and genome-wide restriction site-associated DNA(RAD)markers in farmed Atlantic salmon[J].BMC Genomics,2012,13:244.
[56] MOEN T,BARANSKI M,SONESSON A K,et al.Confirmation and fine-mapping of a major QTL for resistance to infectious pancreatic necrosis in Atlantic salmon(Salmo salar):population-level associations between markers and trait[J].BMC Genomics,2009,10:368.
[57] GHEYAS A A,HOUSTON R D,MOTA-VELASCO J C,et al.Segregation of infectious pancreatic necrosis resistance QTL in the early life cycle of Atlantic salmon(Salmo salar)[J].Animal Genetics,2010,41(5):531-536.
[58] HILLESTAD B,MAKVANDI-NEJAD S,KRASNOV A,et al.Identification of genetic loci associated with higher resistance to pancreas disease(PD)in Atlantic salmon(Salmo salar L.)[J].BMC Genomics,2020,21:388.
[59] GILBEY J,VERSPOOR E,MO T A,et al.Identification of genetic markers associated with Gyrodactylus salaris resistance in Atlantic salmon Salmo salar[J].Diseases of Aquatic Organisms,2006,71(2):119-129.
[60] BOISON S,DING J W,LEDER E,et al.QTLs associated with resistance to Cardiomyopathy Syndrome in Atlantic salmon[J].Journal of Heredity,2019,110(6):727-737.
[61] LIU P,WANG L,WONG S M,et al.Fine mapping QTL for resistance to VNN disease using a high-density linkage map in Asian seabass[J].Scientific Reports,2016,6:32122.
[62] WANG L,BAI B,HUANG S Q,et al.QTL mapping for resistance to iridovirus in Asian seabass using genotyping-by-sequencing[J].Marine Biotechnology,2017,19(5):517-527.
[63] LIU P,WANG L,WAN Z Y,et al.Mapping QTL for resistance against viral nervous necrosis disease in Asian seabass[J].Marine Biotechnology,2016,18(1):107-116.
[64] OZAKI A,YOSHIDA K,FUJI K,et al.Quantitative trait loci(QTL)associated with resistance to a Monogenean parasite(Benedenia seriolae)in Yellowtail(Seriola quinqueradiata)through genome wide analysis[J].PLoS One,2013,8(6):e64987.
[65] MASSAULT C,FRANCH R,HALEY C,et al.Quantitative trait loci for resistance to fish pasteurellosis in gilthead sea bream(Sparus aurata)[J].Animal Genetics,2011,42(2):191-203.
[66] RISCH N,MERIKANGAS K.The future of genetic studies of complex human diseases[J].Science,1996,273(5281):1516-1517.
[67] KONGCHUM P,PALTI Y,HALLERMAN E M,et al.SNP discovery and development of genetic markers for mapping innate immune response genes in common carp(Cyprinus carpio)[J].Fish & Shellfish Immunology,2010,29(2):356-361.
[68] WANG L,SU J G,PENG L M,et al.Genomic structure of grass carp Mx2 and the association of its polymorphisms with susceptibility/resistance to grass carp reovirus[J].Molecular Immunology,2011,49(1/2):359-366.
[69] HENG J F,SU J G,HUANG T,et al.The polymorphism and haplotype of TLR3 gene in grass carp(Ctenopharyngodon idella)and their associations with susceptibility/resistance to grass carp reovirus[J].Fish & Shellfish Immunology,2011,30(1):45-50.
[70] SHEN Y B,ZHANG J B,XU X Y,et al.A new haplotype variability in complement C6 is marginally associated with resistance to Aeromonas hydrophila in grass carp[J].Fish & Shellfish Immunology,2013,34(5):1360-1365.
[71] WAN Q Y,SU J G,CHEN X H,et al.Genomic sequence comparison,promoter activity,SNP detection of RIG-I gene and association with resistance/susceptibility to grass carp reovirus in grass carp(Ctenopharyngodon idella)[J].Developmental & Comparative Immunology,2013,39(4):333-342.
[72] WAN Q Y,SU J G,WANG L,et al.Correlation between grass carp(Ctenopharyngodon idella)resistance to grass carp reovirus and the genetic insert-deletion polymorphisms in promoter and intron of RIG-I gene[J].Gene,2013,516(2):320-327.
[73] WAN Q Y,WANG L,SU J G,et al.Genetic structure,polymorphism identification of LGP2 gene and their relationship with the resistance/susceptibility to GCRV in grass carp,Ctenopharyngodon idella[J].Gene,2013,521(1):166-175.
[74] WAN Q Y,SU J G,CHEN X H,et al.Gene-based polymorphisms,genomic organization of interferon-β promoter stimulator1(IPS-1)gene and association study with the natural resistance to grass carp reovirus in grass carp Ctenopharyngodon idella[J].Developmental & Comparative Immunology,2013,41(4):756-765.
[75] WAN Q Y,SU J G,WANG L,et al.A 15 nucleotide deletion mutation in coding region of the RIG-I lowers grass carp(Ctenopharyngodon idella)resistance to grass carp reovirus[J].Fish & Shellfish Immunology,2012,33(2):442-447.
[76] GAO F X,WANG Y,ZHANG Q Y,et al.Distinct herpesvirus resistances and immune responses of three gynogenetic clones of gibel carp revealed by comprehensive transcriptomes[J].BMC Genomics,2017,18:561.
[77] MOU C Y,WANG Y,ZHANG Q Y,et al.Differential interferon system gene expression profiles in susceptible and resistant gynogenetic clones of gibel carp challenged with herpesvirus CaHV[J].Developmental & Comparative Immunology,2018,86:52-64.
[78] 高风英,卢迈新,曹建萌,等.尼罗罗非鱼IPS-1基因SNP位点的筛选及其与链球菌抗性的关联分析[J].农业生物技术学报,2018,26(1):20-33.
[79] 高风英,卢迈新,曹建萌,等.尼罗罗非鱼NOD1基因SNP位点和单倍型与抗无乳链球菌感染的关联分析[J].农业生物技术学报,2018,26(11):1949-1961.
[80] 张栋,高风英,卢迈新,等.尼罗罗非鱼β2m基因SNP位点和单倍型与无乳链球菌抗性的关联分析[J].水生生物学报,2018,42(5):903-912.
[81] 陈昆平.尼罗罗非鱼Ikaros基因克隆、组织表达及抗无乳链球菌相关SNP位点筛选[D].上海:上海海洋大学,2017.
[82] FU G H,LIU F,XIA J H,et al.The LBP gene and its association with resistance to Aeromonas hydrophila in tilapia[J].International Journal of Molecular Sciences,2014,15(12):22028-22041.
[83] FU G H,WAN Z Y,XIA J H,et al.The MCP-8 gene and its possible association with resistance to Streptococcus agalactiae in tilapia[J].Fish & Shellfish Immunology,2014,40(1):331-336.
[84] 柴欣.团头鲂MHC Ⅱα基因的SNP位点开发、鉴定及与抗病性状关联分析[D].武汉:华中农业大学,2017.
[85] SHAO C W,NIU Y C,RASTAS P,et al.Genome-wide SNP identification for the construction of a high-resolution genetic map of Japanese flounder(Paralichthys olivaceus):applications to QTL mapping of Vibrio anguillarum disease resistance and comparative genomic analysis[J].DNA Research,2015,22(2):161-170.
[86] CAMPBELL N R,LAPATRA S E,OVERTURF K,et al.Association mapping of disease resistance traits in rainbow trout using restriction site associated DNA sequencing[J].G3,2014,4(12):2473-2481.
[87] MIRETTI M M,WALSH E C,KE X Y,et al.A high-resolution linkage-disequilibrium map of the human major histocompatibility complex and first generation of tag single-nucleotide polymorphisms[J].American Journal of Human Genetics,2005,76(4):634-646.
[88] JIA Z Y,CHI X F,LI C T,et al.Development of MHC class I and Ⅱ B primers in common carp and its molecular characterization[J].Biochemical Genetics,2010,48(7/8):690-695.
[89] HANSEN J D,LA PATRA S.Induction of the rainbow trout MHC class I pathway during acute IHNV infection[J].Immunogenetics,2002,54(9):654-661.
[90] HASHIMOTO K,NAKANISHI T,KUROSAWA Y.Isolation of carp genes encoding major histocompatibility complex antigens[J].Proceedings of the National Academy of Sciences of the United States of America,1990,87(17):6863-6867.
[91] GRIMHOLT U,HORDVIK I,FOSSE V M,et al.Molecular cloning of major histocompatibility complex class I cDNAs from Atlantic salmon(Salmo salar)[J].Immunogenetics,1993,37(6):469-473.
[92] SÜLTMANN H,MAYER W E,FIGUEROA F,et al.Zebrafish MHC class Ⅱ alpha chain-encoding genes:polymorphism,expression,and function[J].Immunogenetics,1993,38(6):408-420.
[93] ZHANG Y X,CHEN S L,LIU Y G,et al.Major histocompatibility complex class ⅡB allele polymorphism and its association with resistance/susceptibility to Vibrio anguillarum in Japanese flounder(Paralichthys olivaceus)[J].Marine Biotechnology,2006,8(6):600-610.
[94] VAN MUISWINKEL W B,WIEGERTJES G F,STET R J M.The influence of environmental and genetic factors on the disease resistance of fish[J].Aquaculture,1999,172(1/2):103-110.
[95] KJØGLUM S,LARSEN S,BAKKE H G,et al.The effect of specific MHC class I and class Ⅱ combinations on resistance to furunculosis in Atlantic salmon(Salmo salar)[J].Scandinavian Journal of Immunology,2008,67(2):160-168.
[96] LIU J,LIU Z Z,ZHAO X J,et al.MHC class Ⅱα alleles associated with resistance to Aeromonas hydrophila in purse red common carp,Cyprinus carpio Linnaeus[J].Journal of Fish Diseases,2014,37(6):571-575.
[97] XU T J,CHEN S L,ZHANG Y X.MHC class Ⅱα gene polymorphism and its association with resistance/susceptibility to Vibrio anguillarum in Japanese flounder(Paralichthys olivaceus)[J].Developmental & Comparative Immunology,2010,34(10):1042-1050.
[98] XU T J,CHEN S L,JI X S,et al.MHC polymorphism and disease resistance to Vibrio anguillarum in 12 selective Japanese flounder(Paralichthys olivaceus)families[J].Fish & Shellfish Immunology,2008,25(3):213-221.
[99] YANG M,WEI J G,LI P F,et al.MHC class Ⅱα polymorphisms and their association with resistance/susceptibility to Singapore grouper iridovirus(SGIV)in orange-spotted grouper,Epinephelus coioides[J].Aquaculture,2016,462:10-16.
[100] 耿昕颖.基于维氏气单胞菌感染的黄河鲤MHCⅡ类基因多态性与抗病力关系研究[D].长春:吉林农业大学,2016.
[101] GLOVER K A,GRIMHOLT U,BAKKE H G,et al.Major histocompatibility complex(MHC)variation and susceptibility to the sea louse Lepeophtheirus salmonis in Atlantic salmon Salmo salar[J].Diseases of Aquatic Organisms,2007,76(1):57-65.
[102] LIU Z,HU D D,SHAO S J,et al.Polymorphisms in major histocompatibility complex class Ⅱα genes are associated with resistance to infectious hematopoietic necrosis in rainbow trout,Oncorhynchus mykiss(Walbaum,1792)[J].Journal of Applied Ichthyology,2013,29(6):1234-1240.
[103] LIU J,SUN Y Y,XU T J.Identification of 48 full-length MHC-DAB functional alleles in miiuy croaker and evidence for positive selection[J].Fish & Shellfish Immunology,2016,54:544-550.
[104] XU T J,LIU J,SUN Y Y,et al.Characterization of 40 full-length MHC class ⅡA functional alleles in miiuy croaker:polymorphism and positive selection[J].Developmental & Comparative Immunology,2016,55:138-143.
[105] GAO F Y,ZHANG D,LU M X,et al.MHC class ⅡA polymorphisms and their association with resistance-susceptibility to Streptococcus agalactiae in Nile tilapia,Oreochromis niloticus[J].Journal of Fish Biology,2018,93(6):1207-1215.
[106] WYNNE J W,COOK M T,NOWAK B F,et al.Major histocompatibility polymorphism associated with resistance towards amoebic gill disease in Atlantic salmon(Salmo salar L.)[J].Fish & Shellfish Immunology,2007,22(6):707-717.
[107] 丁少雄,张之文,杜佳莹,等.赤点石斑鱼(Epinephelus akaar)MHC ⅡB基因的克隆与表达多态性分析[J].海洋学报,2009,31(2):129-138.
[108] YANG M,WEI J G,LI P F,et al.MHC polymorphism and disease resistance to Singapore grouper iridovirus(SGIV)in the orange-spotted grouper,Epinephelus coioides[J].Science Bulletin,2016,61(9):693-699.
[109] DU M,CHEN S L,LIU Y H,et al.MHC polymorphism and disease resistance to Vibrio anguillarum in 8 families of half-smooth tongue sole(Cynoglossus semilaevis)[J].BMC Genetics,2011,12:78.
[110] YANG J,LIU Z,SHI H N,et al.Association between MHC Ⅱ beta chain gene polymorphisms and resistance to infectious haematopoietic necrosis virus in rainbow trout(Oncorhynchus mykiss,Walbaum,1792)[J].Aquaculture Research,2016,47(2):570-578.
[111] DU M,CHEN S L,LIU Y H,et al.MHC polymorphism and disease-resistance to Edwardsiella tarda in six turbot(Scophthalmus maximus)families[J].Chinese Science Bulletin,2012,57(25):3262-3269.
[112] 李雪松,刘至治,赵雪锦,等.“全红”体色瓯江彩鲤MHC-DAB基因多态性及其与鱼体抗病力关系的分析[J].水产学报,2011,35(9):1293-1301.
[113] ZHU K C,YU W B,GUO H Y,et al.The polymorphisms of MHC Ⅱß gene of Trachinotus ovatus and their association with resistance/susceptibility to Photobacterium damsela[J].Aquaculture,2018,485:160-165.
[114] GAO F Y,ZHANG D,LU M X,et al.MHC class ⅡB gene polymorphisms associated with resistance/susceptibility to Streptococcus agalactiae in Nile tilapia Oreochromis niloticus[J].Diseases of Aquatic Organisms,2019,133(3):253-261.
[115] 高风英,庞纪彩,卢迈新,等.尼罗罗非鱼MHC ⅡB基因多态性及其与链球菌病抗性的关系[J].中国农学通报,2014,30(2):76-83.
[116] YU H,TAN S W,ZHAO H J,et al.MH-DAB gene polymorphism and disease resistance to Flavobacterium columnare in grass carp(Ctenopharyngodon idellus)[J].Gene,2013,526(2):217-222.
[117] MEUWISSEN T H,HAYES B J,GODDARD M E.Prediction of total genetic value using genome-wide dense marker maps[J].Genetics,2001,157(4):1819-1829.
[118] ZHAO J,BAI H Q,KE Q Z,et al.Genomic selection for parasitic ciliate Cryptocaryon irritans resistance in large yellow croaker[J].Aquaculture,2021,531:735786.
[119] APARICIO S,CHAPMAN J,STUPKA E,et al.Whole-genome shotgun assembly and analysis of the genome of Fugu rubripes[J].Science,2002,297(5585):1301-1310.
[120] KASAHARA M,NARUSE K,SASAKI S,et al.The medaka draft genome and insights into vertebrate genome evolution[J].Nature,2007,447(7145):714-719.
[121] HOWE K,CLARK M D,TORROJA C F,et al.The zebrafish reference genome sequence and its relationship to the human genome[J].Nature,2013,496(7446):498-503.
[122] BERTHELOT C,BRUNET F,CHALOPIN D,et al.The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates[J].Nature Communications,2014,5:3657.
[123] LIEN S,KOOP B F,SANDVE S R,et al.The Atlantic salmon genome provides insights into rediploidization[J].Nature,2016,533(7602):200-205.
[124] CHEN S L,ZHANG G J,SHAO C W,et al.Whole-genome sequence of a flatfish provides insights into ZW sex chromosome evolution and adaptation to a benthic lifestyle[J].Nature Genetics,2014,46(3):253-260.
[125] XU P,ZHANG X F,WANG X M,et al.Genome sequence and genetic diversity of the common carp,Cyprinus carpio[J].Nature Genetics,2014,46(11):1212-1219.
[126] YOU X X,BIAN C,ZAN Q J,et al.Mudskipper genomes provide insights into the terrestrial adaptation of amphibious fishes[J].Nature Communications,2014,5:5594.
[127] WU C W,ZHANG D,KAN M Y,et al.The draft genome of the large yellow croaker reveals well-developed innate immunity[J].Nature Communications,2014,5:5227.
[128] WANG Y P,LU Y,ZHANG Y,et al.The draft genome of the grass carp(Ctenopharyngodon idellus)provides insights into its evolution and vegetarian adaptation[J].Nature Genetics,2015,47(6):625-631.
[129] SHAO C W,BAO B L,XIE Z Y,et al.The genome and transcriptome of Japanese flounder provide insights into flatfish asymmetry[J].Nature Genetics,2017,49(1):119-124.
[130] WANG D D,CHEN X Y,ZHANG X H,et al.Whole genome sequencing of the giant grouper(Epinephelus lanceolatus)and high-throughput screening of putative antimicrobial peptide genes[J].Marine Drugs,2019,17(9):503.
[131] 陈松林,徐文腾,刘洋.鱼类基因组研究十年回顾与展望[J].水产学报,2019,43(1):1-14.
[132] WAN S M,LIU H,ZHAO B W,et al.Construction of a high-density linkage map and fine mapping of QTLs for growth and gonad related traits in blunt snout bream[J].Scientific Reports,2017,7:46509.
[133] YUE H M,LI C J,DU H,et al.A first attempt for genetic linkage map construction and growth related QTL mapping in Acipenser sinensis using specific length amplified fragment sequencing(SLAF-seq)[J].Journal of Applied Ichthyology,2019,35(1):235-237.
[134] FU B D,LIU H Y,YU X M,et al.A high-density genetic map and growth related QTL mapping in bighead carp(Hypophthalmichthys nobilis)[J].Scientific Reports,2016,6:28679.
[135] PENG W Z,XU J,ZHANG Y,et al.An ultra-high density linkage map and QTL mapping for sex and growth-related traits of common carp(Cyprinus carpio)[J].Scientific Reports,2016,6:26693.
[136] WANG S,MEYER E,MCKAY J K,et al.2b-RAD:a simple and flexible method for genome-wide genotyping[J].Nature Methods,2012,9(8):808-810.
[137] ASLAM M L,CARRARO R,BESTIN A,et al.Genetics of resistance to photobacteriosis in gilthead sea bream(Sparus aurata)using 2b-RAD sequencing[J].BMC Genet,2018,19(1):43.
[138] BARR A A,CHRISTENSEN K A,YOSHIDA G M,et al.Genomic predictions and genome-wide association study of resistance against piscirickettsia salmonis in coho salmon (Oncorhynchus kisutch) using ddRAD sequencing[J].G3,2018,8(4):1183-1194.
A A,CHRISTENSEN K A,YOSHIDA G M,et al.Genomic predictions and genome-wide association study of resistance against piscirickettsia salmonis in coho salmon (Oncorhynchus kisutch) using ddRAD sequencing[J].G3,2018,8(4):1183-1194.
[139] DONG L S,XIAO S J,CHEN J W,et al.Genomic selection using extreme phenotypes and pre-selection of SNPs in large yellow croaker(Larimichthys crocea)[J].Marine Biotechnology,2016,18(5):575-583.
[140] WANG S,LIU P P,LV J,et al.Serial sequencing of isolength RAD tags for cost-efficient genome-wide profiling of genetic and epigenetic variations[J].Nature Protocols,2016,11(11):2189-2200.
[141] YOSHIDA G M,BANGERA R,CARVALHEIRO R,et al.Genomic prediction accuracy for resistance against Piscirickettsia salmonis in farmed rainbow trout[J].G3,2018,8(2):719-726.
[142] VALLEJO R L,LEEDS T D,GAO G T,et al.Genomic selection models double the accuracy of predicted breeding values for bacterial cold water disease resistance compared to a traditional pedigree-based model in rainbow trout aquaculture[J].Genetics Selection Evolution,2017,49(1):17.
[143] DONG L S,XIAO S J,WANG Q R,et al.Comparative analysis of the GBLUP,emBayesB,and GWAS algorithms to predict genetic values in large yellow croaker(Larimichthys crocea)[J].BMC Genomics,2016,17:460.
[144] DONG L S,FANG M,WANG Z Y.Prediction of genomic breeding values using new computing strategies for the implementation of MixP[J].Scientific Reports,2017,7(1):17200.
[145] DONG L S,WANG Z Y.Genomic prediction using an iterative conditional expectation algorithm for a fast BayesC-like model[J].Genetica,2018,146(4/5):361-368.
[146] 董林松,方铭,王志勇.基因组预测中先验分布的两种超参数设定策略比较[J].厦门大学学报(自然科学版),2018,57(4):539-545.
[147] LIU Y,LU S,LIU F,et al.Genomic selection using BayesCπ and GBLUP for resistance against Edwardsiella tarda in Japanese flounder(Paralichthys olivaceus)[J].Marine Biotechnology,2018,20(5):559-565.
[148] 刘峰.半滑舌鳎经济性状的遗传评估及基因组选择初步研究[D].上海:上海海洋大学,2015.
[149] GENG X,SHA J,LIU S K,et al.A genome-wide association study in catfish reveals the presence of functional hubs of related genes within QTLs for columnaris disease resistance[J].BMC Genomics,2015,16(1):196.
[150] PALAIOKOSTAS C,CARIOU S,BESTIN A,et al.Genome-wide association and genomic prediction of resistance to viral nervous necrosis in European sea bass(Dicentrarchus labrax) using RAD sequencing[J].Genetics Selection Evolution,2018,50(1):30.
[151] ROBLEDO D,MATIKA O,HAMILTON A,et al.Genome-wide association and genomic selection for resistance to amoebic gill disease in Atlantic salmon[J].G3,2018,8(4):1195-1203.
[152] ASLAM M L,BOISON S A,LILLEHAMMER M,et al.Genome-wide association mapping and accuracy of predictions for amoebic gill disease in Atlantic salmon(Salmo salar)[J].Scientific Reports,2020,10(1):6435.
[153] ZHOU Q,SU Z C,LI Y Z,et al.Genome-wide association mapping and gene expression analyses reveal genetic mechanisms of disease resistance variations in Cynoglossus semilaevis[J].Frontiers in Genetics,2019,10:1167.
[154] ZHOU T,LIU S K,GENG X,et al.GWAS analysis of QTL for enteric septicemia of catfish and their involved genes suggest evolutionary conservation of a molecular mechanism of disease resistance[J].Mol Genet Genomics,2017,292(1):231-242.
[155] VALLEJO R L,CHENG H,FRAGOMENI B O,et al.Genome-wide association analysis and accuracy of genome-enabled breeding value predictions for resistance to infectious hematopoietic necrosis virus in a commercial rainbow trout breeding population[J].Genetics Selection Evolution,2019,51:47.
[156] PALAIOKOSTAS C,ROBLEDO D,VESELY T,et al.Mapping and sequencing of a significant quantitative trait locus affecting resistance to koi herpesvirus in common carp[J].G3,2018,8(11):3507-3513.
[157] FRASLIN C,BRARD-FUDULEA S,D’AMBROSIO J,et al.Rainbow trout resistance to bacterial cold water disease:two new quantitative trait loci identified after a natural disease outbreak on a French farm[J].Animal Genetics,2019,50(3):293-297.
[158] HILLESTAD B,KRISTJ NSSON
NSSON  H,MAKVANDI-NEJAD S,et al.Genome-wide association study confirms previous findings of major loci affecting resistance to Piscine myocarditis virus in Atlantic salmon(Salmo salar L.)[J].Genes,2020,11(6):608.
H,MAKVANDI-NEJAD S,et al.Genome-wide association study confirms previous findings of major loci affecting resistance to Piscine myocarditis virus in Atlantic salmon(Salmo salar L.)[J].Genes,2020,11(6):608.