脊椎动物的性腺发育与成熟主要受下丘脑-垂体性腺轴(hypothalamic-pituitary-gonadal axis, HPG)为核心的内分泌系统调控[1]。促性腺激素释放激素(gonadotropin releasing hormone, GnRH)作为HPG轴重要的信号分子,在生殖活动中发挥了关键的调控作用。在哺乳动物中,GnRH由下丘脑神经内分泌细胞合成,以脉冲式释放,经下丘脑-垂体门脉系统到达垂体前叶,与促性腺释放激素受体(gonadotropin releasing hormone receptor, GnRHR)结合从而发挥作用[2]。大部分硬骨鱼类缺乏门脉系统,下丘脑合成的GnRH通过轴突末梢作用于垂体,与垂体中GnRHR结合,诱导促卵泡激素(follicle-stimulating hormone, FSH)和促黄体生成激素(luteinizing hormone, LH)的合成和释放。FSH和LH通过循环系统作用于性腺,调控类固醇激素的合成、配子发育和成熟[3]。此外,GnRH还可通过旁分泌或自分泌的方式,调整血液中局部促性腺激素的含量及固醇类性激素水平,从而调控动物的性行为[4]。
GnRHR隶属G蛋白偶联受体超家族,其由7个α跨膜螺旋构成,包括3个胞内环和3个胞外环。通常G蛋白偶联受体的胞内C末端,对于受体的脱敏和内化十分重要[5]。然而,GnRHR有包含和不包含胞内C末端两种蛋白变体。最早被鉴定的小鼠GnRHR蛋白缺乏一个典型的细胞内C末端[6]。在其他哺乳动物及某些低等脊椎动物和软骨鱼类中,也相继发现了缺少胞内C末端的GnRHR蛋白[7-8]。近年来,在哺乳动物和非哺乳动物中,还发现了具有典型胞内C末端的GnRHR蛋白。因此,依据是否存在细胞内C末端结构域,Roch等[7]将缺乏细胞内C末端的GnRHR归类为GnRHRⅠ,将具有C末端的GnRHR归类为GnRHRⅡA和GnRHRⅡB;Williams等[8]又将GnRHRⅡA进一步分为GnRHRⅡA-1、GnRHRⅡA-2和GnRHRⅡA-3。
硬骨鱼中存在GnRH-Ⅰ、GnRH-Ⅱ和GnRH-Ⅲ 3种GnRH旁系同源基因,其中,GnRH-Ⅱ存在于目前已经分析的所有硬骨鱼中[9],GnRH-Ⅰ存在于大多数的硬骨鱼中,其主要起刺激垂体合成和分泌促性腺激素的作用,而GnRH-Ⅱ和GnRH-Ⅲ主要起神经递质的作用[3, 9]。在鱼类生殖内分泌活动中,GnRH与GnRHR的结合是GnRH发挥作用的重要基础。因此,研究GnRHR的分子特征和表达模式对于理解GnRH-GnRHR信号通路的生理学功能具有重要意义。
黄颡鱼Pelteobagrus fulvidraco隶属于硬骨鱼纲Osteichthyes鲇形目Siluriformes鲿科Bagridae黄颡鱼属Pelteobagrus,其肉质鲜美、营养丰富、无肌间刺,是中国重要的淡水养殖品种。黄颡鱼精巢成熟比卵巢晚,雄性生长速率比雌性快,因而雄性个体偏大。在相同养殖条件下,第一年雄性黄颡鱼比同龄雌鱼的生长速度快30%左右,第二年雌雄生长差异甚至能接近3倍[10-11]。本实验室前期工作构建了黄颡鱼高密度遗传连锁图谱,鉴定出11个与性别相关的数量性状位点(quantitative trait locus,QTL),并从这些QTL附近的参考基因组区域鉴定出6个性别相关基因,其中包括黄颡鱼GnRHR基因[12],GnRHR可能参与黄颡鱼性别分化和性腺发育。本研究中,克隆了黄颡鱼GnRHR基因,分析其序列特征、系统进化关系和组织表达模式,并利用CRISPR/Cas9基因编辑技术构建和筛选出黄颡鱼GnRHR突变体,以期为深入研究GnRHR突变体在生殖发育过程中的作用提供材料,为进一步了解黄颡鱼生殖调控机制及探索黄颡鱼人工繁殖技术提供科学依据。
1 材料与方法
1.1 材料
试验用黄颡鱼取自珠海市斗门长源苗场,选取健康无病的黄颡鱼活体运回实验室。经过麻醉和放血后,取心、肝、脾、肾、端脑、中脑、小脑、下丘脑、垂体、精巢及卵巢等组织,在液氮中速冻后于超低温冰箱(-80 ℃)中保存备用。
用于人工繁殖的黄颡鱼雌雄亲本同样取自珠海市斗门长源苗场,在繁殖季节,选取发育状况良好的黄颡鱼雌雄亲本活体运回实验室,于28 ℃水温、自然光周期和持续充氧条件下暂养3 d,之后进行催熟和人工授精。
1.2 方法
1.2.1 总RNA的提取和cDNA的合成 按照50~100 mg组织样品与1 mL Trizol (Invitrogen)的比例,向各个样品管中加入适量Trizol,用均质破碎仪(托莫斯)匀浆40 s,其后按照操作手册提取总RNA。总RNA的质量和浓度通过15 g/L琼脂糖凝胶电泳分析和紫外分光光度仪(岛津)确定。根据PrimeScriptTM RT reagent Kit with gDNA Eraser (TaKaRa)操作手册,以1 μg总RNA作为模板反转录成cDNA。
1.2.2 黄颡鱼GnRHR基因克隆、序列分析及系统进化树构建 将黄颡鱼基因组中GnRHR片段在NCBI数据库进行同源检索,发现与已提交的一个黄颡鱼GnRHR基因序列(GenBank ID:113661459)一致性高达99%以上。因此,设计引物GnRHR-F1和GnRHR-R1,以脑和性腺混合cDNA文库为模板,扩增黄颡鱼GnRHR cDNA。PCR反应产物用10 g/L琼脂糖凝胶电泳进行检测,并回收目的产物。将纯化的PCR产物克隆到pMD-18T载体,转化至大肠杆菌DH5α感受态细胞中,挑取阳性克隆,送广州天一辉远公司进行测序。
将所得序列利用ORF Finder查找开放阅读框,并推导其相应的氨基酸序列。从GenBank中查询出其他物种GnRHR的氨基酸序列,利用Blast进行氨基酸同源性分析。利用MEGA6软件采用最大相似性法(Maximum-likelihood method),进行1 000次自展重复,构建基于GnRHR氨基酸序列的系统发生树。
1.2.3 基因的组织表达 采用RT-qPCR法检测GnRHR在黄颡鱼心、肝、脾、肾、端脑、中脑、小脑、下丘脑、垂体、精巢及卵巢组织中的表达情况。采用2ΔΔCT法计算GnRHR的相对表达量,以β-actin作为内参基因,以GnRHR-F2和GnRHR-R2作为特异引物进行RT-qPCR扩增(表1)。
表1 试验用引物及其序列
Tab.1 Sequences of the primers used for the PCR analysis
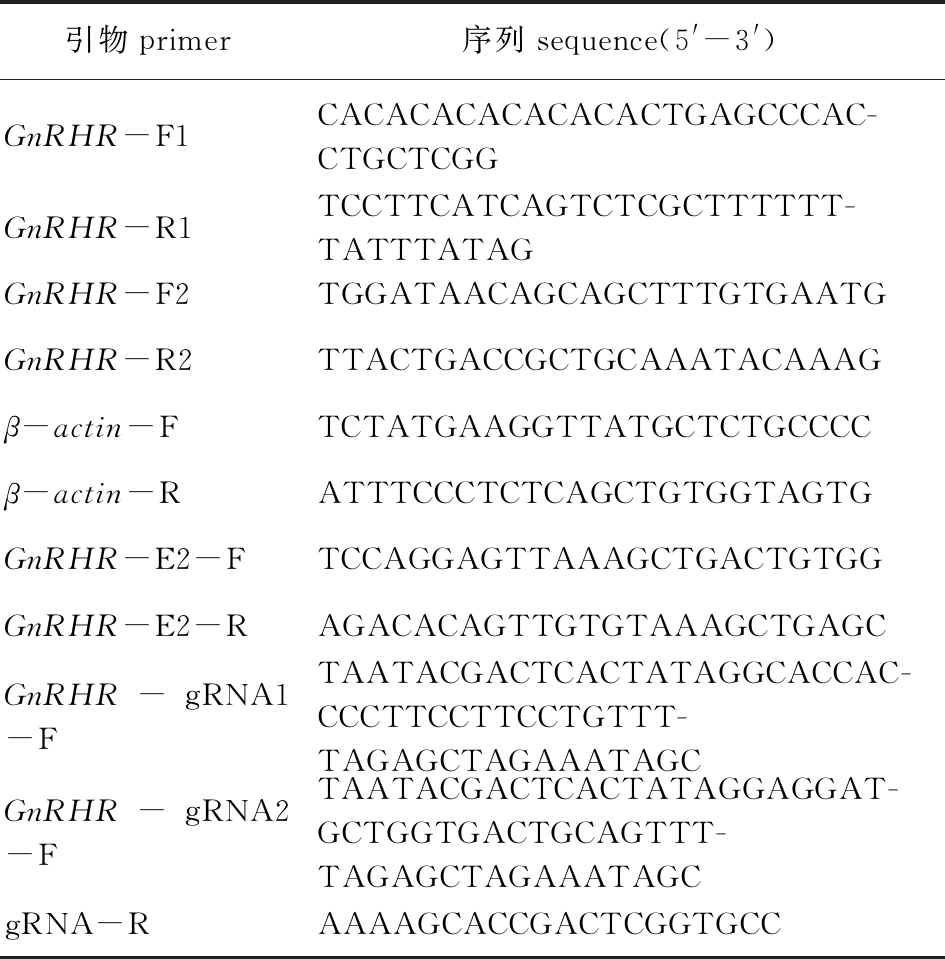
引物primer序列sequence(5'-3')GnRHR-F1CACACACACACACACTGAGCCCAC-CTGCTCGGGnRHR-R1TCCTTCATCAGTCTCGCTTTTTT-TATTTATAGGnRHR-F2TGGATAACAGCAGCTTTGTGAATGGnRHR-R2TTACTGACCGCTGCAAATACAAAGβ-actin-FTCTATGAAGGTTATGCTCTGCCCCβ-actin-RATTTCCCTCTCAGCTGTGGTAGTGGnRHR-E2-FTCCAGGAGTTAAAGCTGACTGTGGGnRHR-E2-RAGACACAGTTGTGTAAAGCTGAGCGnRHR-gRNA1-FTAATACGACTCACTATAGGCACCAC-CCCTTCCTTCCTGTTT-TAGAGCTAGAAATAGCGnRHR-gRNA2-FTAATACGACTCACTATAGGAGGAT-GCTGGTGACTGCAGTTT-TAGAGCTAGAAATAGCgRNA-RAAAAGCACCGACTCGGTGCC
1.2.4 黄颡鱼人工繁殖 黄颡鱼人工催熟和催产:雌鱼注射促黄体生成素释放激素类似物(LHRH-A2) 20 μg/kg;10~12 h后,雌鱼注射LHRH-A2 15 μg/kg、地欧酮(DOM)10 mg/kg和人绒毛膜促性腺激素(HCG)800 U/kg,雄鱼注射剂量为雌鱼的1/3~1/2,催产水温为28~29 ℃。黄颡鱼人工授精:解剖雄鱼取出精巢,用干净的剪刀剪碎,加2 mL生理盐水稀释,收集到干净的EP管中4 ℃下避光保存;轻轻挤压雌鱼腹部将鱼卵收集到无水的玻璃培养皿中,加入50 μL精液,充分混匀,在装有干净饱气水的玻璃皿中铺开受精。5~10 min后,用水轻轻清洗去除废液和杂质。
1.2.5 黄颡鱼GnRHR基因敲除突变体的构建与筛选 根据黄颡鱼GnRHR基因组序列,利用Cas9靶位点预测网站设计2个打靶位点(http://zifit.partners.org/ZiFiT/CSquare9Nuclease.aspx)。对试验所需的黄颡鱼雌雄亲本剪尾鳍,利用Tissue DNA Kit (Omega)提取基因组DNA,采用引物GnRHR-E2-F和GnRHR-E2-R PCR扩增靶位点附近序列,PCR产物送去测序,确认靶点序列不存在单核苷酸多态性。设计并合成特异的gRNA引物(表1),以pUC19-gRNA-scaffold质粒为模板,进行PCR扩增,产物采用Gel Extraction Kit(Omega)回收纯化。随后采用Transcript Aid T7 High Yield Transcription Kit(Thermo)体外转录gRNA。另外,pCS2-Cas9质粒经过限制性内切酶XbaⅠ线性化后回收并纯化,随后采用mMESSAGE mMACHINE T7 Kit(Invitrogen)体外转录Cas9 mRNA。
按照300、30、30 ng/μL的最终浓度混合Cas9 mRNA、GnRHR-gRNA1和GnRHR-gRNA2。将1 nL上述混合物注射到黄颡鱼的1细胞期胚胎中,于28 ℃下培养,受精72 h后收集40尾小鱼苗,提取基因组DNA。以上述基因组DNA为模板,用引物GnRHR-E2-F和GnRHR-E2-R进行PCR扩增,产物回收纯化后克隆到pMD-18T载体,转化至大肠杆菌DH5α感受态细胞中,挑选40个阳性克隆送去测序。
2 结果与分析
2.1 黄颡鱼GnRHR基因的克隆及序列分析
将黄颡鱼基因组中GnRHR片段在NCBI数据库进行同源检索,发现与已提交的一个黄颡鱼GnRHR基因序列(GenBank ID:113661459)一致性高达99%以上。
从图1可见,在脑和性腺混合cDNA文库中扩增得到的GnRHR片段符合预期大小,经克隆和测序证实与NBCI上提供的序列一致。本试验中扩增到的黄颡鱼GnRHR cDNA长为 2 894 bp,其中,包含5′非编码区239 bp、开放阅读框1 155 bp和3′非编码区1 500 bp,编码384个氨基酸。氨基酸序列分析发现,黄颡鱼GnRHR含有典型的7次跨膜结构域(transmembrane domain,TM),表明其属于G蛋白偶联受体家族成员(图2)。
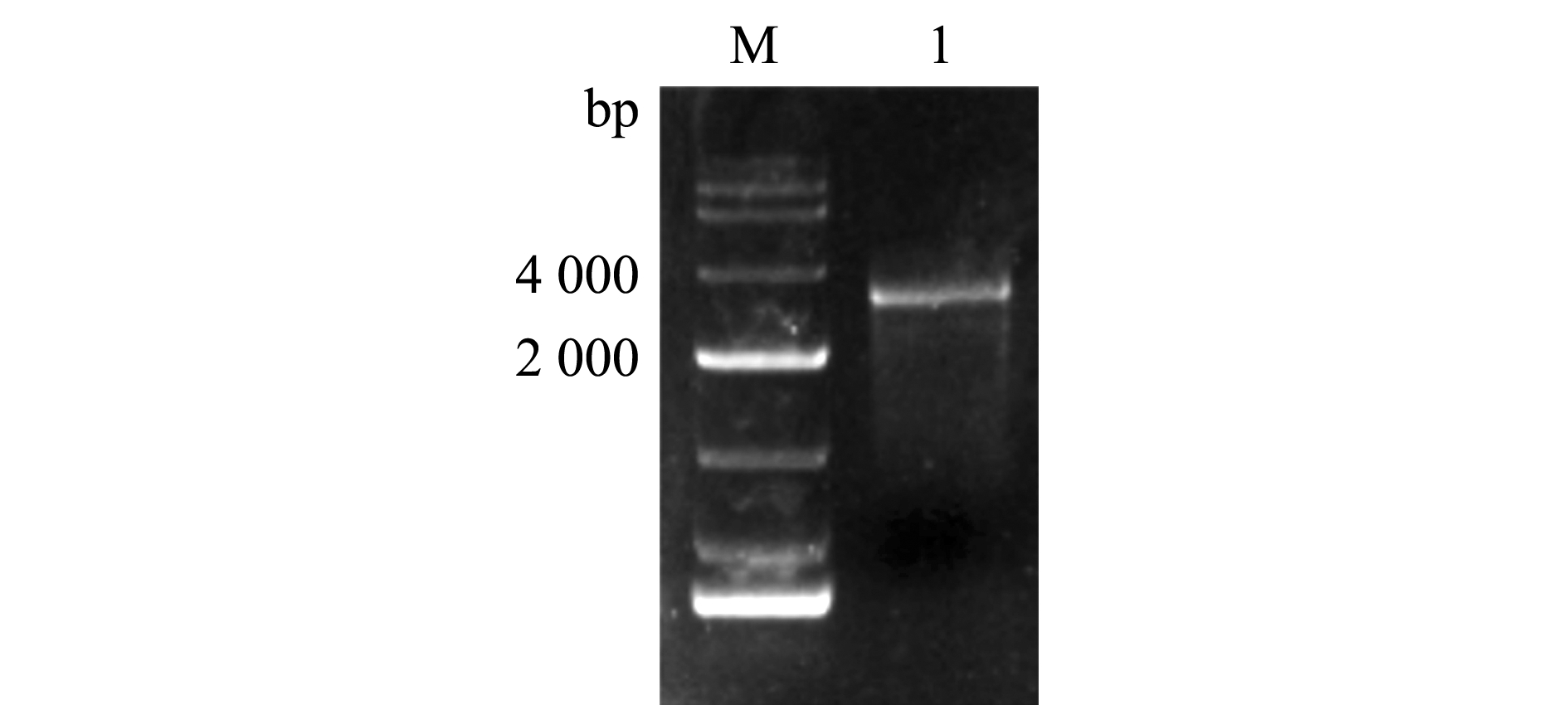
M—DL 10000 Maker; 1—GnRHR.
图1 黄颡鱼GnRHR cDNA扩增
Fig.1 Amplification products of GnRHR cDNA in yellow catfish

cDNA序列和氨基酸残基的编号位于左侧;终止密码子用*标出;7次跨膜结构域用方框标出。
Nucleotide and deduced amino acid residues are numbered on the left;the stop codon is indicated by *;the seven transmembrane (TM) domain regions are boxed.
图2 黄颡鱼GnRHR的cDNA序列及其推导的氨基酸序列
Fig.2 Nucleotide and deduced amino acid sequences of GnRHR cDNA in yellow catfish
将黄颡鱼GnRHR与其他物种GnRHR的氨基酸序列进行同源性分析(表2),结果表明:黄颡鱼GnRHR与鱼类GnRHR氨基酸序列的一致性较高(66%~96%),其中,与斑点叉尾鮰GnRHR的一致性最高(96%),与斑马鱼、鲤和银鲫等鲤科鱼类GnRHR的一致性超过80%;黄颡鱼GnRHR与其他非鱼类GnRHR的一致性较低(42%~55%),其中与爪蟾GnRHR的一致性为55%,与鸡GnRHR的一致性为48%,与人、小鼠、牛等哺乳类动物的GnRHR一致性低于45%。
表2 黄颡鱼与其他物种GnRHR氨基酸序列的同源性比较
Tab.2 Comparison of amino acid identities of GnRHR between yellow catfish and other species

物种species一致性identity/%GenBank accession No.斑点叉尾鮰 Ictalurus puncta-tus96XP_017309510.1斑马鱼 Danio rerio87XP_021323652.1鲤 Cyprinus carpio83XP_018942803.1银鲫Carassius auratus82XP_026087388.1虹鳟Oncorhynchus mykiss72NP_001117823.1银汉鱼 Odontesthes bonarien-sis68ABI75337.1青鳉 Oryzias latipes68NP_001098392.1罗非鱼 Oreochromis niloticus67NP_001266689.1朴丽鱼 Haplochromis burtoni66AAK29745.1爪蟾Xenopus tropicalis55NP_001107549.1鸡 Gallus gallus48NP_001012627.1小鼠 Mus musculus44NP_034453.1人 Homo sapiens43NP_000397.1狗 Canis lupus familiaris43NP_001003121.1牛 Bos taurus42NP_803480.1羊 Ovis aries42NP_001009397.1
2.2 GnRHR氨基酸序列的系统进化分析
通过最大相似性法构建脊椎动物GnRHR进化树进行系统进化分析(图3),结果显示,脊椎动物GnRHR形成3个分支,黄颡鱼与斑点叉尾鮰、鲤、银鲫、虹鳟等的GnRHR聚类到GnRHRⅡB分支,爪蟾、鸡及鱼类等其他一些GnRHR聚类到GnRHRⅡA分支,人、小鼠、牛等哺乳动物形成另一个独立的GnRHRⅠ分支。这表明,黄颡鱼GnRHR与斑点叉尾鮰GnRHR亲缘关系最近,这与氨基酸同源性分析结果一致。从3种类型的GnRHR中各选3个物种进行氨基酸序列比对,结果如图4所示,Ⅰ型GnRHR明显缺失了C末端,Ⅱ型GnRHR虽然都具有C端尾巴,但相比于GnRHRⅡA,GnRHRⅡB仍然缺失了两小段氨基酸序列。因此,系统进化树和氨基酸序列比对结果均证明,克隆得到的黄颡鱼GnRHR为ⅡB型GnRHR。
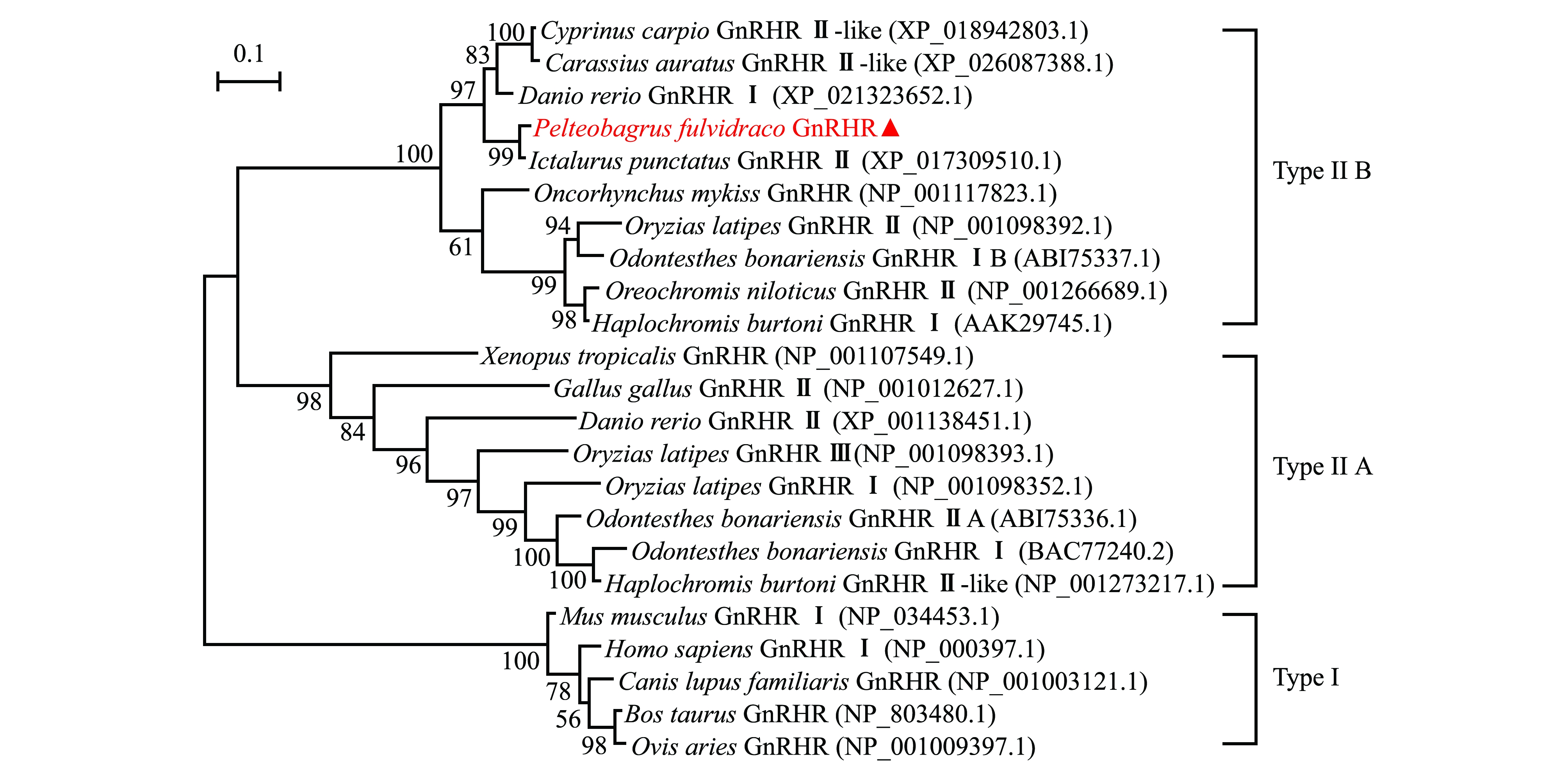
图3 黄颡鱼GnRHR氨基酸序列和其他物种同源氨基酸序列的系统进化树
Fig.3 Phylogenetic tree comparison of yellow catfish GnRHR and other vertebrate homologues amino acid sequence

图4 不同类型的GnRHR氨基酸序列比对
Fig.4 Amino acid alignment of different types of GnRHR
2.3 黄颡鱼GnRHR基因的组织分布
利用RT-qPCR检测黄颡鱼GnRHR mRNA的组织分布(图5),结果表明:黄颡鱼GnRHR基因主要在黄颡鱼端脑、中脑、下丘脑、垂体和精巢等组织中高表达,其中,在下丘脑和垂体中的表达量最高,而在心脏、肝脏、脾脏、肾脏、小脑和卵巢等组织中几乎检测不到;雌雄鱼在垂体中的相对表达量有显著性差异(P<0.05),在性腺中的相对表达量有极显著性差异(P<0.01),在其他组织中无显著性差异(P>0.05)。
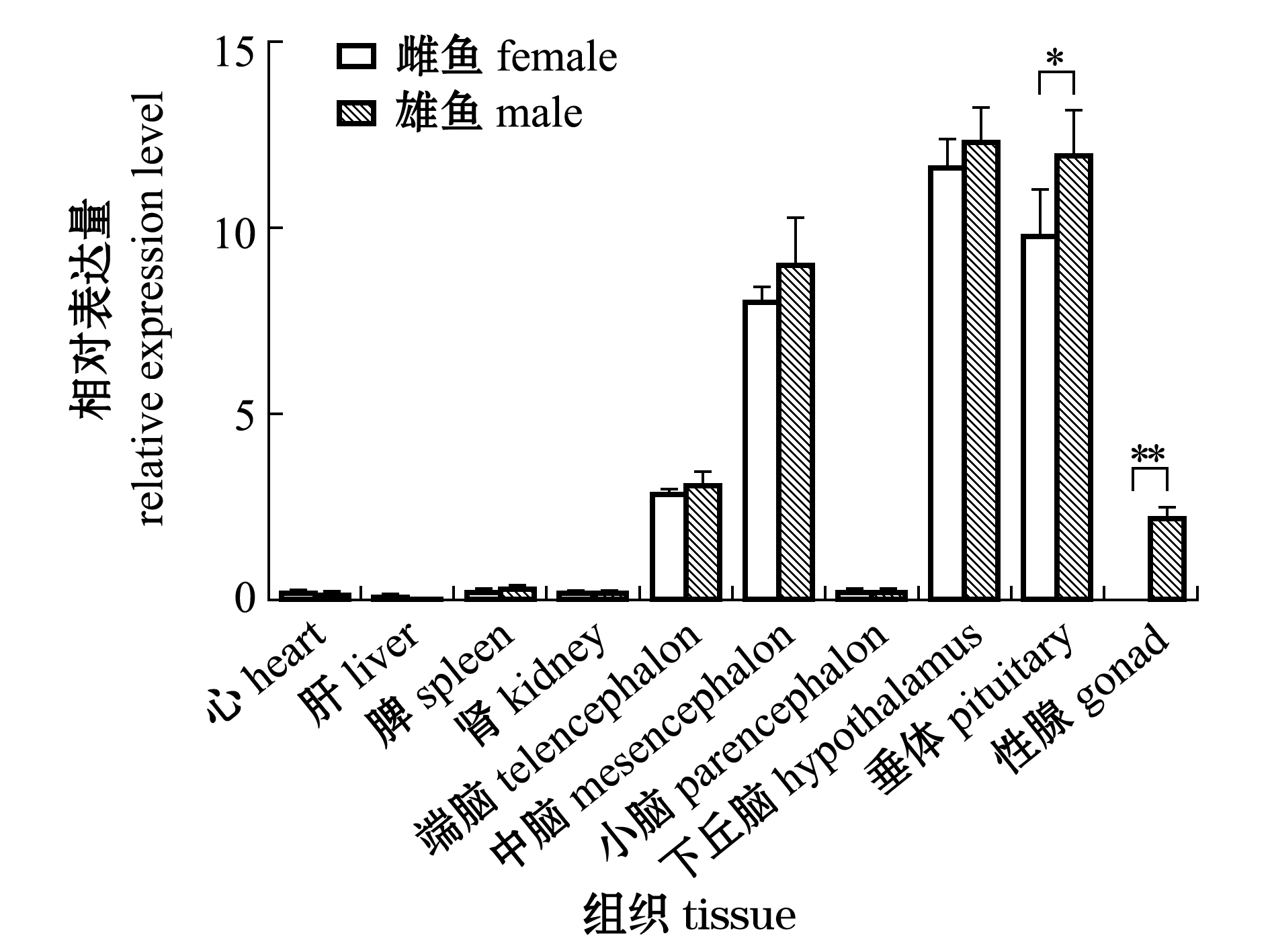
*表示组间有显著性差异(P<0.05);**表示组间有极显著性差异(P<0.01)。
*means significant difference in the groups (P<0.05);**means very significant difference in the groups(P<0.01).
图5 黄颡鱼GnRHR在不同组织中的表达模式
Fig.5 Relative expression of GnRHR in different tissues of yellow catfish
2.4 黄颡鱼GnRHR基因敲除突变体的构建与筛选
利用CRISPR/Cas9基因编辑技术构建黄颡鱼GnRHR基因敲除突变体,结果表明,黄颡鱼GnRHR基因全长9 797 bp,包含4个外显子和3个内含子,起始密码子位于第二外显子上。CRISPR/Cas9基因编辑靶位点的原则为:
1)靶位点包含20个碱基,其中,5′端应为GG,这是由于本研究中所用的gRNA体外转录采用的是T7启动子,T7启动子要求转录起始位点的前两位为GG,并且第三位最好为G或A;
2)紧邻靶位点3′端的3个碱基构成PAM区,要求序列为NGG(N为任意碱基);
3)靶位点尽量选在基因CDS的前2/3区域且在ATG之后,但不要在最后一个外显子上,最好能破坏重要的结构域;
4)靶位点也可选在外显子和内含子交界处,以破坏基因的剪接。
基于以上原则,本试验中选取黄颡鱼GnRHR第二外显子上的两个靶点,设计gRNA。将2个位点的gRNA和Cas9 mRNA共注射至1细胞期胚胎,以提高编辑效率(图6(a))。显微注射后的胚胎存活率为62.25%(381/612),而对照组的胚胎存活率为72.32%(473/654),可能是因为试验组中显微注射的机械损伤对胚胎的存活有一定的影响。如图6(b)、(d)所示,Sanger测序检测发现,F0代胚胎产生了各种突变类型,包括1~56 bp的缺失突变和1~17 bp的插入突变,其中,大部分突变会导致开放阅读框移码和终止突变。图6(c)、(f)显示了野生型和F0代突变体代表性的测序峰图。根据统计结果,靶点1的基因编辑效率为25%,靶点2的基因编辑效率为27.5%,总的编辑效率为45.6%。至此,本试验中成功构建了黄颡鱼F0代GnRHR基因敲除突变体,为后续的杂交筛选和基因功能研究提供了基础资料。

图6 CRISPR/Cas9构建黄颡鱼GnRHR突变体
Fig.6 Establishment of GnRHR mutant of yellow catfish by CRISPR/Cas9
3 讨论
3.1 GnRHR在进化上的保守与分歧
GnRHR主要位于促性腺激素细胞的细胞膜上,通过与来自下丘脑的十肽激素GnRH结合,激活腺苷酸环化酶-cAMP-蛋白激酶体系,促进垂体前叶合成和释放促性腺激素,从而调控性腺发育和生殖细胞的成熟[3]。自1992年从小鼠垂体细胞中克隆鉴定出首个GnRHR基因之后,研究人员陆续在包括哺乳动物、鸟类、爬行类、两栖类、鱼类等多个物种中克隆到GnRHR基因的全长序列或部分序列[13-16]。本研究中,以中国重要的淡水养殖鱼类黄颡鱼为研究材料,克隆了黄颡鱼GnRHR基因序列,通过氨基酸序列分析,证明黄颡鱼GnRHR与其他物种的GnRHR蛋白具有较高的保守性,均具有G蛋白偶联受体特有的7次跨膜结构域(图2)。氨基酸同源性分析表明,黄颡鱼GnRHR氨基酸序列与斑点叉尾鮰、斑马鱼、鲤、银鲫的一致性较高,分别为96%、87%、83%和82%(表2),表明黄颡鱼GnRHR在进化上比较保守。
在大多数脊椎动物中存在着3种GnRH,暗示其受体可能也以相似方式存在3种GnRHR。目前,通常将所有缺乏细胞内尾巴的GnRHR归类为GnRHRⅠ,而将具有典型胞内C末端尾巴的GnRHR归类为GnRHRⅡ[7-8]。在人中,仅保留了功能性的GnRHRⅠ,而GnRHRⅡ由于存在移码突变可能导致功能缺失[17]。在人促性腺激素细胞、促甲状腺激素细胞和促生长激素细胞中,均能检测到GnRHRⅠ的表达[18]。在猴和猪中,GnRHRⅠ和GnRHRⅡ同时存在,其中,GnRHRⅠ主要参与调控垂体促性腺激素的合成和分泌,而GnRHRⅡ的作用还不清楚,也有可能如同在人中一样功能缺失[19]。在一些非哺乳类脊椎动物中,如两栖类、爬行类、鸟类和硬骨鱼类中,GnRHRⅠ基因似乎已经缺失,取而代之的是发现了多个GnRHRⅡ亚型[3]。在许多硬骨鱼中已发现了多种类型的GnRHR,如青鳉 Oryzias latipes[20]、朴丽鱼 Haplochromis burtoni[21]、欧洲鳗鲡Anguilla anguilla[22]、河豚[23]。本试验中对黄颡鱼与其他物种的GnRHR氨基酸序列进行了系统进化分析(图3),从结果可以看出,黄颡鱼GnRHR与斑点叉尾鮰GnRHRⅡ、鲤GnRHRⅡ-like、银鲫GnRHRⅡ-like、虹鳟GnRHR聚类到GnRHRⅡB分支,斑马鱼GnRHRⅠ、青鳉GnRHRⅡ、银汉鱼GnRHⅠB、罗非鱼GnRHRⅡ及朴丽鱼GnRHRⅠ也聚类到GnRHRⅡB这一分支;同时,斑马鱼GnRHRⅡ、青鳉GnRHRⅠ与GnRHRⅢ、银汉鱼GnRHRⅠ与ⅡA、朴丽鱼GnRHRⅡ-like、鸡GnRHRⅡ和爪蟾GnRHR聚类到GnRHRⅡA分支;而人、小鼠、牛、羊、狗等哺乳动物的GnRHR形成另一个独立的分支。系统进化分析结果表明,有些物种中的GnRHR可能需要重新命名,例如斑马鱼中的两个GnRHR可能是按照发现时间先后顺序来命名的,但根据进化分析表明,斑马鱼GnRHR Ⅰ其实属于GnRHRⅡB类型。另外,斑马鱼、银汉鱼和青鳉等鱼类中存在多种GnRHR,聚类到不同的进化分支,可能是源于硬骨鱼中发生了额外的全基因组复制事件,且这些不同类型的GnRHR在随后的进化过程中有着不同的演化方向。在黄颡鱼基因组和转录组中进行检索显示:基因组中预测到的2个GnRHR序列,均对应为本研究中所述的序列(仅有3个核苷酸的差异);转录组中找到3个GnRHR片段,与本研究中所述的黄颡鱼GnRHR序列一致性为99%~100%。因此,黄颡鱼中只有一个GnRHR。序列比对和进化分析结果表明,黄颡鱼GnRHR应为ⅡB型GnRHR。
3.2 GnRH、GnRHR的表达及其功能多样性
GnRH基因的表达并不完全局限于HPG轴,在脑、胃、胰脏及交感神经节等组织中也发现有GnRH存在[24-26]。GnRH可以通过旁分泌调节生物体的免疫能力[27], GnRH还能够作为自分泌调节因子,在性腺中调控类固醇激素的合成和代谢[4]。这表明GnRH是一种多功能肽类,能够广泛参与神经、内分泌、生殖、消化及免疫系统的信号传导。GnRH通过形成GnRH-GnRHR复合物才能产生功能效应,因此,推测GnRHR同样在HPG轴以外的其他组织和器官中存在。研究表明,不同物种GnRHR基因的组织表达模式存在一定差异,如中华鲟Acipenser sinensis GnRHR主要表达于性腺、脑和肌肉[28],斑纹隐小鳉Kryptolebias marmoratus GnRHR主要表达于脑、垂体、性腺、肠和肝脏[29]。这表明,GnRHR可能在不同物种中发挥不同功能。GnRHR的组织表达定位可能与相应的GnRH分布密切相关,已有研究表明,GnRHⅠ主要在下丘脑表达,GnRHⅡ主要在中脑表达,GnRHⅢ主要在端脑表达,且GnRHⅠ主要作用是刺激垂体合成和分泌促性腺激素,而GnRHⅡ和GnRHⅢ主要作为神经递质发挥作用[30-32]。本研究表明,黄颡鱼GnRHR主要表达于下丘脑、垂体、中脑和端脑等中枢神经系统中,因此,GnRHR可能既参与促性腺激素的合成和分泌,同时也参与其他的神经内分泌调节作用。另外,黄颡鱼GnRHR的表达呈现显著的两性异形,GnRHR在精巢中有表达,但在卵巢中几乎检测不到。类似的结果在欧洲鳗鲡中也有报道,欧洲鳗鲡GnRHRⅠB也是在精巢中表达,而在卵巢中基本不表达[22]。这种表达模式可能暗示着黄颡鱼GnRHR对黄颡鱼精巢发育起着重要作用。
3.3 CRISPR/Cas9基因编辑技术在黄颡鱼及其他养殖鱼类中的成功应用
近年来,基因编辑技术的快速发展使得在经济养殖鱼类中进行基因功能性研究变得相对简单。CRISPR/Cas9系统是目前最常用的基因编辑技术之一,已在多种养殖鱼类中成功应用,如鲟、罗非鱼、大西洋鲑、黄颡鱼等[33-37]。Baloch等[33]在小体鲟Acipenser ruthenus中通过CRISPR/Cas9敲除原始生殖细胞(PGC)迁移关键基因dnd1,成功获得不育的小体鲟,可作为其他晚熟和大型鲟生殖细胞移植的不育受体。在罗非鱼Oreochromis mossambicus中,通过CRISPR/Cas9分别敲除性别相关基因gsdf和amhy来解析其基因功能[34-35],发现gsdf可能是罗非鱼dmrt1的下游基因,并通过抑制雌激素的分泌诱导精巢分化,而amhy敲除会导致罗非鱼由雄向雌性反转,因此,amhy可能是罗非鱼的性别决定基因。人工养殖的大西洋鲑Salmo salar外逃到自然环境中,可能会导致基因渗入,影响野生群体的遗传完整性。而培育出不育的大西洋鲑品系则可以解决这个问题,Wargelius等[36]利用CRISPR/Cas9敲除大西洋鲑dnd基因,成功获得了不育的大西洋鲑,可以有效阻止人工养殖家系和野生品系间的基因流。黄颡鱼是中国重要的经济养殖鱼类之一,具有明显的雌雄生长二态性,在相同养殖环境下,雄鱼生长显著快于雌鱼。因此,研究黄颡鱼性别发育相关基因的功能不仅能对鱼类性别发育分子机制提供新的见解,同时对黄颡鱼基于性别控制的遗传育种也具有重要的经济应用价值。然而目前黄颡鱼性别发育关键基因的功能研究还比较薄弱,Dan等[37]在黄颡鱼中通过CRISPR/Cas9敲除Y染色体上的pdz1基因,导致XY型雄鱼的精巢向卵巢分化,证明黄颡鱼pdz1基因是一个雄性决定和分化的重要基因。本研究中,通过CRISPR/Cas9基因编辑技术构建和筛选出了黄颡鱼GnRHR基因敲除F0代突变体,基因敲除的胚胎存活率为62.25%,2个靶向位点附近的突变率为45.6%,产生了多种多样的插入/缺失突变类型,由此可见,在黄颡鱼中可进行高效的基因编辑。基于此,作者在黄颡鱼中,还对其他生殖发育相关的基因如vasa、dmrt1、amh和cyp11b等也进行了基因编辑,获得了许多F0突变体(成果尚未发表)。因黄颡鱼性成熟周期为1年,杂合突变体和纯合突变体的筛选工作仍在进行,获得这些基因的纯合突变体也是研究这些基因的功能和作用机制必不可少的基础之一。
4 结论
1)本研究中克隆出黄颡鱼GnRHR基因,其cDNA长度为 2 894 bp,其中包含239 bp的5′非编码区、1 155 bp的开放阅读框和1 500 bp的3′非编码区,编码384个氨基酸。
2)系统进化分析和氨基酸序列分析结果显示,黄颡鱼GnRHR的进化类型为GnRHRⅡB型。
3)黄颡鱼GnRHR基因在黄颡鱼下丘脑和垂体中的表达量最高,在端脑、中脑和精巢等组织中表达量较高,而在心脏、肝脏、脾脏、肾脏、小脑和卵巢等组织中几乎不表达。
4)通过CRISPR/Cas9基因编辑技术,构建了黄颡鱼GnRHR基因缺失F0代突变体,F0代基因编辑效率为45.6%。
[1] KAPRARA A,HUHTANIEMI I T.The hypothalamus-pituitary-gonad axis:tales of mice and men[J].Metabolism,2018,86:3-17.
[2] BLISS S P,NAVRATIL A M,XIE J J,et al.GnRH signaling,the gonadotrope and endocrine control of fertility[J].Frontiers in Neuroendocrinology,2010,31(3):322-340.
[3] CHANG J P,PEMBERTON J G.Comparative aspects of GnRH-stimulated signal transduction in the vertebrate pituitary-contributions from teleost model systems[J].Molecular and Cellular Endocrinology,2018,463:142-167.
[4] LENTS C A,THORSON J F,DESAULNIERS A T,et al.RFamide-related peptide 3 and gonadotropin-releasing hormone-II are autocrine-paracrine regulators of testicular function in the boar[J].Molecular Reproduction and Development,2017,84(9):994-1003.
[5] VENKATAKRISHNAN A J,DEUPI X,LEBON G,et al.Molecular signatures of G-protein-coupled receptors[J].Nature,2013,494(7436):185-194.
[6] TSUTSUMI M,ZHOU W,MILLAR R P,et al.Cloning and functional expression of a mouse gonadotropin-releasing hormone receptor[J].Molecular Endocrinology,1992,6(7):1163-1169.
[7] ROCH G J,BUSBY E R,SHERWOOD N M.GnRH receptors and peptides:skating backward[J].General and Comparative Endocrinology,2014,209:118-134.
[8] WILLIAMS B L,AKAZOME Y,OKA Y,et al.Dynamic evolution of the GnRH receptor gene family in vertebrates[J].BMC Evolutionary Biology,2014,14:215.
[9] MU OZ-CUETO J A,ZMORA N,PAULLADA-SALMER
OZ-CUETO J A,ZMORA N,PAULLADA-SALMER N J A,et al.The gonadotropin-releasing hormones:lessons from fish[J].General and Comparative Endocrinology,2020,291:113422.
N J A,et al.The gonadotropin-releasing hormones:lessons from fish[J].General and Comparative Endocrinology,2020,291:113422.
[10] GONG G R,DAN C,XIAO S J,et al.Chromosomal-level assembly of yellow catfish genome using third-generation DNA sequencing and Hi-C analysis[J].Giga Science,2018,7(11):giy120.
[11] MEI J,GUI J F.Genetic basis and biotechnological manipulation of sexual dimorphism and sex determination in fish[J].Science China Life Sciences,2015,58(2):124-136.
[12] GAO D,ZHENG M,LIN G M,et al.Construction of high-density genetic map and mapping of sex-related loci in the yellow catfish (Pelteobagrus fulvidraco)[J].Marine Biotechnology,2020,22(1):31-40.
[13] LUMAYNO S D P,OHGA H,SELVARAJ S,et al.Molecular characterization and functional analysis of pituitary GnRH receptor in a commercial scombroid fish,chub mackerel (Scomber japonicus)[J].General and Comparative Endocrinology,2017,247:143-151.
[14] HILDAHL J,SANDVIK G K,EDVARDSEN R B,et al.Four gonadotropin releasing hormone receptor genes in Atlantic cod are differentially expressed in the brain and pituitary during puberty[J].General and Comparative Endocrinology,2011,173(2):333-345.
[15] IKEMOTO T,ENOMOTO M,PARK M K.Identification and characterization of a reptilian GnRH receptor from the leopard gecko[J].Molecular and Cellular Endocrinology,2004,214(1/2):137-147.
[16] WANG L,BOGERD J,CHOI H S,et al.Three distinct types of GnRH receptor characterized in the bullfrog[J].Proceedings of the National Academy of Sciences of the United States of America,2001,98(1):361-366.
[17] CHENG C K,LEUNG P C.Molecular biology of gonadotropin-releasing hormone (GnRH)-I,GnRH-II,and their receptors in humans[J].Endocrine Reviews,2005,26(2):283-306.
[18] LA ROSA S,CELATO N,UCCELLA S,et al.Detection of gonadotropin-releasing hormone receptor in normal human pituitary cells and pituitary adenomas using immunohistochemistry[J].Virchows Archiv,2000,437(3):264-269.
[19] DESAULNIERS A T,CEDERBERG R A,LENTS C A,et al.Expression and role of gonadotropin-releasing hormone 2 and its receptor in mammals[J].Frontiers in Endocrinology,2017,8:269.
[20] OKUBO K,ISHII S,ISHIDA J,et al.A novel third gonadotropin-releasing hormone receptor in the medaka Oryzias latipes:evolutionary and functional implications[J].Gene,2003,314:121-131.
[21] CHEN C C,FERNALD R D.Distributions of two gonadotropin-releasing hormone receptor types in a cichlid fish suggest functional specialization[J].The Journal of Comparative Neurology,2006,495(3):314-323.
[22] PE ARANDA D S,MAZZEO I,HILDAHL J,et al.Molecular characterization of three GnRH receptor paralogs in the European eel,Anguilla anguilla:tissue-distribution and changes in transcript abundance during artificially induced sexual development[J].Molecular and Cellular Endocrinology,2013,369(1/2):1-14.
ARANDA D S,MAZZEO I,HILDAHL J,et al.Molecular characterization of three GnRH receptor paralogs in the European eel,Anguilla anguilla:tissue-distribution and changes in transcript abundance during artificially induced sexual development[J].Molecular and Cellular Endocrinology,2013,369(1/2):1-14.
[23] IKEMOTO T,PARK M K.Identification and molecular characterization of three GnRH ligands and five GnRH receptors in the spotted green pufferfish[J].Molecular and Cellular Endocrinology,2005,242(1/2):67-79.
[24] SUWANSA-ARD S,ZHAO M,THONGBUAKAEW T,et al.Gonadotropin-releasing hormone and adipokinetic hormone/corazonin-related peptide in the female prawn[J].General and Comparative Endocrinology,2016, 236:70-82.
[25] 王立会,王莉,曹宏伟,等.不同月龄大鼠胰腺促性腺激素释放激素的表达变化及其与糖代谢的关系[J].中国糖尿病杂志,2010,18(11):863-865.
[26] UMATANI C,OKA Y.Multiple functions of non-hypophysiotropic gonadotropin releasing hormone neurons in vertebrates[J].Zoological Letters,2019,5:23.
[27] HUANG Q S,LI Q H,CHEN H M,et al.Neuroendocrine immune-regulatory of a neuropeptide ChGnRH from the hongkong oyster,Crassostrea hongkongensis[J].Fish & Shellfish Immunology,2019,93:911-916.
[28] 王桂苹,杜合军,冷小茜,等.中华鲟促性腺激素释放激素受体基因cDNA序列的克隆及表达分析[J].水生生物学报,2018,42(1):94-98.
[29] RHEE J S,SEO J S,RAISUDDIN S,et al.Gonadotropin-releasing hormone receptor (GnRHR) gene expression is differently modulated in gender types of the hermaphroditic fish Kryptolebias marmoratus by endocrine disrupting chemicals[J].Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology,2008,147(3):357-365.
[30] TOSTIVINT H.Evolution of the gonadotropin-releasing hormone (GnRH) gene family in relation to vertebrate tetraploidizations[J].General and Comparative Endocrinology,2011,170(3):575-581.
[31] WIRSIG-WIECHMANN C R.Function of gonadotropin-releasing hormone in olfaction[J].The Keio Journal of Medicine,2001,50(2):81-85.
[32] ABRAHAM E,PALEVITCH O,IJIRI S,et al.Early development of forebrain gonadotrophin-releasing hormone (GnRH) neurones and the role of GnRH as an autocrine migration factor[J].Journal of Neuroendocrinology,2008, 20(3):394-405.
[33] BALOCH A R,FRANEK R,TICHOP D T,et al.Dnd1 knockout in sturgeons by CRISPR/Cas9 generates germ cell free host for surrogate production[J].Animals,2019,9(4):174.
D T,et al.Dnd1 knockout in sturgeons by CRISPR/Cas9 generates germ cell free host for surrogate production[J].Animals,2019,9(4):174.
[34] JIANG D N,YANG H H,LI M H,et al.Gsdf is a downstream gene of dmrt1 that functions in the male sex determination pathway of the Nile tilapia[J].Molecular Reproduction and Development,2016,83(6):497-508.
[35] LI M H,SUN Y L,ZHAO J E,et al.A tandem duplicate of anti-Müllerian hormone with a missense SNP on the Y chromosome is essential for male sex determination in Nile tilapia,Oreochromis niloticus[J].PLoS Genetics,2015,11(11):e1005678.
[36] WARGELIUS A,LEININGER S,SKAFTNESMO K O,et al.Dnd knockout ablates germ cells and demonstrates germ cell independent sex differentiation in Atlantic salmon[J].Scientific Reports,2016,6:21284.
[37] DAN C,LIN Q H,GONG G H,et al.A novel PDZ domain-containing gene is essential for male sex differentiation and maintenance in yellow catfish (Pelteobagrus fulvidraco)[J].Science Bulletin,2018,63(21):1420-1430.