罗非鱼隶属于鲈形目Perciformes丽鱼科Cichlid罗非鱼属Oreochromis,为广盐性热带鱼类,最适生长水温为28~32 ℃,在世界范围内被广泛养殖[1-2]。罗非鱼与其他养殖鱼类相比,具有个体生长快、适应性强、耐高盐、病害少等特点,是目前中国重要的淡水养殖鱼类之一,年产量高达33万t,居中国第三位[3]。近年来,随着全球气候变暖及热污染不断加剧,罗非鱼养殖面临着热应激带来的不良影响。夏季经常会发生持续性高温现象,常导致罗非鱼的新陈代谢出现障碍,鱼体无法正常排出体内的排泄物,严重时甚至会出现死亡;此外,水温的升高加快了水体中有机物的分解速度,增加了微生物的繁殖,进而导致致病菌加速生长,使罗非鱼的发病率升高,给水产养殖产业带来重大的经济损失[4],严重威胁了罗非鱼产业的健康可持续发展。因此,应用分子生物学手段,开展罗非鱼在高温胁迫下相关功能基因的筛选及调控机理研究,对于揭示高温胁迫下罗非鱼的调控作用机制,开展罗非鱼的高温健康养殖至关重要。
RNA-seq是不依赖于基因组序列的信息,可以对互补DNA(cDNA)进行直接测序,是目前查找控制机体特异性状的关键基因和研究基因表达调控机制的有效方法[5]。应用转录组技术解析机体在非生物胁迫下的复杂分子机制,也已成为当前研究的热点。Bilyk等[6]通过对博氏南冰 Pagothenia borchgrevinki在4 ℃温度胁迫下的转录组进行分析,推测其在4 ℃温度胁迫下,相关基因的转录在一定程度上受到性别的影响。Quinn等[7]发现,北极鲑鱼 Salvelinus alpinus暴露在25 ℃温度胁迫下,机体内Hsp和泛素的表达增加,并报道了鱼类对高温的耐受性,具体表现为机体血红蛋白基因发生下调。
Pagothenia borchgrevinki在4 ℃温度胁迫下的转录组进行分析,推测其在4 ℃温度胁迫下,相关基因的转录在一定程度上受到性别的影响。Quinn等[7]发现,北极鲑鱼 Salvelinus alpinus暴露在25 ℃温度胁迫下,机体内Hsp和泛素的表达增加,并报道了鱼类对高温的耐受性,具体表现为机体血红蛋白基因发生下调。
目前,关于罗非鱼的研究主要集中在低温胁迫反应[8-9]、胚胎发育[10]、性别比例调控[11]和病菌敏感性[12-13]等方面,而关于罗非鱼在转录水平上进行的高温适应机理方面研究尚未见报道。本研究中,基于RNA-seq技术对尼罗罗非鱼Oreochromis niloticus 受到高温胁迫后的肝脏组织进行转录组分析,以揭示罗非鱼在高温胁迫下与常温对照组的转录差异,并从中挖掘关键基因及调控途径,以期为罗非鱼在持续性高温下的健康养殖管理,以及了解罗非鱼高温胁迫响应的调控机制提供科学依据。
1 材料与方法
1.1 材料
试验用尼罗罗非鱼亲鱼均来自广西南宁某罗非鱼良种场,平均体质量约为400 g,健康有活力且无疾病感染,于上海海洋大学罗非鱼养殖基地养殖。待雌雄鱼性腺发育成熟后,通过人工授精方式,收集尼罗罗非鱼同一家系的受精卵(约1 000粒)。正式试验前在温度为(28.0±0.2)℃、pH为8的环境中进行受精卵的人工孵化,于孵化12 d后开始试验。
1.2 方法
1.2.1 试验设计 将罗非鱼幼鱼随机分为试验组 (36 ℃) 和对照组 (28 ℃),每组约300尾。试验组初始水温为28 ℃,按每天升高1 ℃的速度升温,升高至36 ℃时停止升温,继续养殖70 d。分别在养殖30、50、70 d时,从各组随机取3尾麻醉后,统计其体长、体质量等指标,然后解剖采集高温组和对照组的肝脏组织样品,分别记为36 ℃-30 d、36 ℃-50 d、36 ℃-70 d、28 ℃-30 d、28 ℃-50 d、28 ℃-70 d,将各样品迅速转移至1.5 mL 离心管中并置于液氮中速冻后于超低温冰箱(-80 ℃)中保存。养殖试验期间,每天早晚投喂两次,每天换水一次,每次换水量为总量的1/3,换水前将新水预热至试验温度。为减少误差,各组取样的过程均在同一时间点进行,在各时间点从试验组与对照组随机取3尾。
1.2.2 总RNA的提取、文库构建及测序 利用Trizol法分别对高温组和对照组肝脏组织样品进行总RNA的提取,并使用DNaseⅠ(TaKaRa,中国)消化样品中残留的基因组DNA。采用10 g/L 琼脂糖凝胶电泳检测RNA降解及污染程度,采用紫外分光光度计检测提取的总RNA的纯度及浓度。根据Liu等[14]将组织样品进行混样的方法来消除个体间的差异,本试验中将各时间点每3尾鱼的肝脏组织样品进行等量混合,从每个时间点组织样品中取3 μg RNA用于文库构建,文库构建及测序工作由北京诺禾致源公司完成。
1.2.3 生物信息学分析 首先通过FastQC评估原始数据的质量,使用 Trimmomatic 对 raw data 进行处理,以去除带接头和低质量的碱基,得到高质量的clean data,且后续的转录组分析均基于高质量的clean data。TopHat2是一个来自RNA测序试验reads的高效比对系统[15],本研究中以罗非鱼的基因组作为参考进行序列比对及后续的分析,使用TopHat2比对系统将得到的clean reads与罗非鱼参考基因组进行序列比对(参考基因组下载地址:https://www.ncbi.nlm.nih.gov/genome)。
根据FPKM法[16]计算每个基因在不同样本中的表达量。使用DESeq软件包[17]对罗非鱼高温组和对照组进行差异表达分析,根据FPKM法对原始数据进行标准化处理,用Benjamini-Hochberg法控制错误发现率(false discovery rate, FDR)以调整所得的P值,将fold change≥2且FDR<0.05作为差异表达基因检测过程中的筛选标准。最后用 Cluster Profile 软件包对差异表达基因进行GO功能注释和KEGG通路富集分析,P<0.05时表示差异基因显著富集。
1.2.4 实时荧光定量PCR分析 根据差异基因对高温胁迫的响应程度,本研究中通过RT-qPCR试验对苏氨酸/丝氨酸激酶(mTOR)及其相关信号通路中的8个差异表达基因进行了验证,以此评价RNA-seq方法鉴定差异基因的可靠性。使用Primer 3.0软件对各差异基因进行引物设计(表1),并由生工生物工程(上海)股份有限公司合成引物。
表1 RT-qPCR引物设计
Tab.1 Primers for RT-qPCR

差异表达基因differentially expressed gene引物序列(5' -3')primer sequence(5' -3')socs2F:GAAGCAGTTTGACAGCGTGGR:CCTTTGGTTGATGGCGATGCccne2F:GGAGTGAACTTCCTGGAGCCR:AGTTATAGTCGAGCGCGGTGsocs1bF:GTTACCAAAGCGACGATGGCR:AGGGCACCGTCAGTTTACAGgrb10aF:GAGATGTGTGGAGGACCACGR:TGTGACGGAGGAATTGAGCCirs2aF:ACCTGAGGAAGCAGAAGCACR:GTTATAACCCGTTTCGCGGCprkczF:GCACGCAAGATTCTACGCTGR:AGCTTGATGTGTCCCTCGTGcdkn1aF:CGGTGGGGCTTTGACTTTAGR:CTCCACCCTTCCTCTCTTGGfoxo1aF:CACCATACAACAGCAGCCAGR:GCATCTGCATCTGGGTTTTCβ-actinF:CTGAACCCCAAAGCCAACAGR:GAACAGAAGGAAACCGGGGA
使用反转录试剂盒(TaKaRa,Dalian)将各样品的RNA反转录为cDNA,并以反转录产物为模板,以β-actin为内参基因,使用FastStart Universal SYBR® Green Master(ROX)试剂,采用CFX96 Real-Time PCR Detection System (Bio-Rad)进行实时荧光定量检测。每个样品设3个平行试验,采用2-△△CT方法计算出各候选基因的相对表达量。
2 结果与分析
2.1 尼罗罗非鱼体长、体质量指标分析
通过对持续性高温处理过程中(30、50、70 d)罗非鱼体长、体质量指标进行统计,结果显示,在高温阶段罗非鱼的生长受到水温的限制[18],与对照组(28 ℃)相比,随着高温(36 ℃)处理时间的不断延长,罗非鱼的体长、体质量增加速率逐渐变慢(图1、图2)。

图1 尼罗罗非鱼体长的变化趋势
Fig.1 Trend chart of body length in Nile tilapia Oreochromis niloticus

图2 尼罗罗非鱼体质量的变化趋势
Fig.2 Trend chart of body mass in Nile tilapia Oreochromis niloticus
2.2 转录组测序数据及比对分析
对罗非鱼高温组(36 ℃-30 d、36 ℃-50 d、36 ℃-70 d)和对照组(28 ℃-30 d、28 ℃-50 d、28 ℃-70 d)肝脏组织样品进行转录组测序,共获得39.23 Gb clean data(表2)。各样品的clean data数均达到6.18 Gb以上,Q30碱基百分比均在91.45%以上。将各样品的clean reads与尼罗罗非鱼基因组进行序列比对,比对效率均在82.37% 以上,这表明此次测序数据质量较高,可用于后续分析。
表2 测序数据质量和序列比对情况
Tab.2 Sequencing data quality and sequence alignment

样品sampleclean reads数目total readsclean data数目clean data碱基质量≥30所占的百分比/%Q30比对到参考基因组上的reads数(在clean reads中的占比/%)mapped reads(proportion)28 ℃-30 d42 480 4126.3791.4634 990 754(82.37)28 ℃-50 d47 232 4047.0892.0039 615 221(83.87)28 ℃-70 d43 598 5446.5492.1336 341 416(83.35)36 ℃-30 d41 577 6446.2491.4534 998 972(84.18)36 ℃-50 d41 219 2266.1891.5036 075 809(87.52)36 ℃-70 d45 476 3266.8292.0338 243 469(84.10)
2.3 差异表达基因分析
通过DEseq软件包进行样品组间的差异表达分析,在|log2(fold change)|≥1且FDR<0.05条件下,获得了高温胁迫不同时间点肝脏组织样品的差异基因表达情况。28 ℃-30 d与36 ℃-30 d文库比较中,检测到4 342个差异表达基因,其中,3 026个基因表达上调,1 316个基因表达下调;28 ℃-50 d与36 ℃-50 d文库比较中,检测到3 139个差异表达基因,其中,1 352个基因表达上调,1 787个基因表达下调;28 ℃-70 d与36 ℃-70 d文库比较中,检测到3 042个差异表达基因,其中,1 702个基因表达上调,1 340个基因表达下调(图3)。这表明,在持续性高温胁迫下,差异基因的表达发生了明显变化,且呈现出不同的高温应答模式。

图3 差异表达基因数目统计
Fig.3 Number of differentially expressed genes(DEG)
2.4 差异基因GO功能分析
因差异表达基因较多,为深入探究罗非鱼在持续性高温胁迫下的调控机制,对差异表达基因进行GO功能富集分析,将差异表达基因归类到GO 3大分支(生物学过程、细胞组分和分子功能)的26个子类别中(图4)。其中,28 ℃-30 d与36 ℃-30 d文库比较中发现,在细胞内信号转导、丝氨酸水解酶活性、细胞发育过程及细胞分化等调控过程中差异表达基因显著富集;28 ℃-50 d与36 ℃-50 d文库比较中发现,在细胞对外来生物的反应、细胞修饰氨基酸代谢过程、DNA复制、细胞周期等调控过程中存在差异表达基因富集现象;28 ℃-70 d与36 ℃-70 d文库比较中发现,脂类生物合成过程、类固醇代谢过程中存在差异表达基因富集现象。在高温胁迫过程中,应激反应、能量代谢等调控过程中差异基因富集较为显著,这表明在高温胁迫下,在罗非鱼肝脏组织中产生了大量参与调控代谢等相关的活动过程。

纵轴中色阶表示富集的P值,颜色越深,表示P值越小,白色单元格表示P>0.05,下同。
The color scale on the vertical axis indicates the enriched P value.The darker the color, the smaller the P value, and the white cell indicates P>0.05,et sequentia.
图4 差异表达基因GO富集分析
Fig.4 GO enrichment analysis of differentially expressed genes
2.5 KEGG富集分析
为了进一步探究在持续性高温胁迫下差异表达基因的生物学功能,对差异表达基因进行KEGG富集分析,以P<0.05为阈值,本试验中选取了前17条显著富集的代谢通路(图5)。在高温胁迫的不同时间下,差异表达基因在萜骨架生物合成通路、细胞周期、糖酵解/糖异生、精氨酸/脯氨酸的代谢通路、胰岛素信号通路、内质网的蛋白质加工通路及mTOR信号等通路中显著富集。其中,参与内质网蛋白质加工通路的差异基因数目最多,该通路涉及27个差异基因,且与高温胁迫最为相关的热应激蛋白基因Hsp70和Hsp90就属于这个通路。
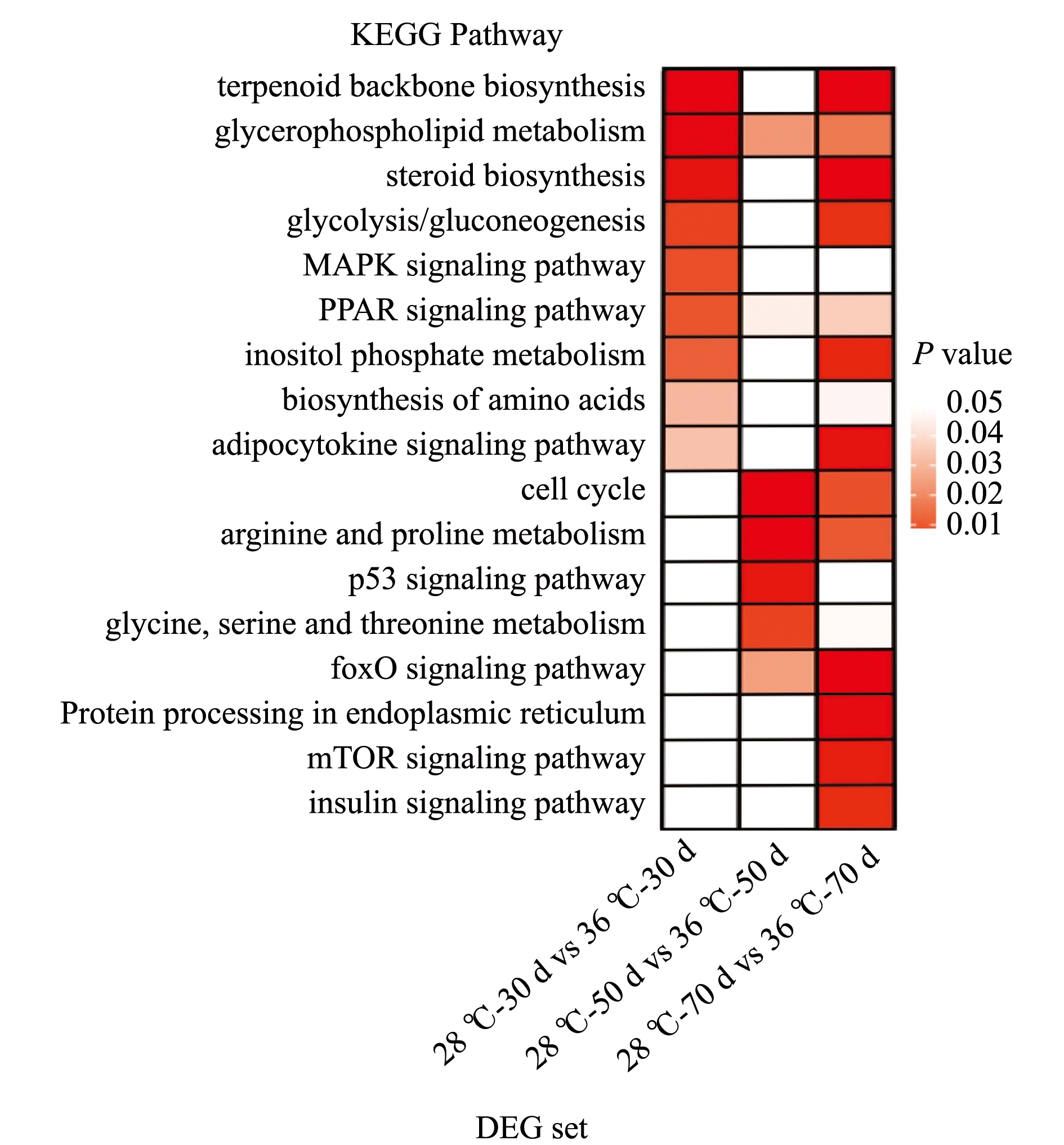
图5 差异表达基因KEGG富集分析
Fig.5 KEGG enrichment analysis of differentially expressed genes
2.6 实时荧光定量PCR验证分析
为了验证RNA-seq所得结果的准确性,本研究中通过RT-qPCR试验对mTOR及其相关信号通路中的8个差异表达基因进行验证。各基因的表达情况及各基因的转录组分析结果如图6所示,虽然表达量存在一定的偏差,但两者在整体上的表达趋势一致,由此证明本研究中转录组分析结果的可靠性。

图6 各差异表达基因的RT-qPCR验证
Fig.6 Validation of the differentially expressed genes by RT-qPCR
3 讨论
近年来,随着全球气温升高的情况日益加剧[19],罗非鱼的养殖面临着热应激带来的诸多不良影响。已有大量研究表明,转录组学技术逐渐在水产动物各方面的研究上发挥着重要的作用,比如应用转录组技术对水产动物在免疫应答反应、生长发育过程等方面进行的研究[20]。本研究中,通过对罗非鱼在持续性高温胁迫下的肝脏组织进行转录组分析,发现参与热应激相关的基因涉及生长、能量代谢及蛋白质折叠等多个生物学过程,对鱼类高温状态下的生存有着重要的作用。
3.1 高温胁迫下罗非鱼的生长调控分析
生长激素 (growth hormone,GH) -胰岛素样生长因子(insulin-like growth factor,IGF)体系,通常被认为是脊椎动物包括鱼类的生长内分泌调控轴的核心。已有研究表明,GH-IGF体系在鱼类的生长调控方面发挥着重要的作用,脑垂体分泌生长激素(GH)后,GH通过相应受体的介导以刺激肝脏和其他组织中合成并分泌IGF-1,且在其他组织中主要通过 IGF受体的介导机体发挥各种生物功能[21]。有研究发现,机体中IGF-1或IGF-2的缺失均将导致个体质量的损失[22-23];代谢通路中insulin/PI(3) K信号通路[24]在调控细胞生长方面发挥着重要的作用。本研究中,通过对罗非鱼高温处理组和常温对照组差异表达基因分析发现,在高温胁迫(30、50、70 d)过程中,肝脏组织中有关生长轴调控的重要因子IGF-1及相关受体基因的表达均呈现显著下调。由此推测,罗非鱼神经内分泌方面的调控在一定程度上抑制了罗非鱼的生长。同时,通过对差异表达基因进行KEGG富集分析发现,胰岛素信号通路被显著富集,主要参与罗非鱼的生长调控。当然,生长轴的调控还受到多种因子和调控模式的共同作用,有关罗非鱼神经内分泌调控机制的研究还有待后期深入探究。
3.2 高温胁迫下罗非鱼的代谢响应分析
在应激条件下,变温性鱼类的代谢活动高度依赖于环境温度。在多种鱼类如斑马鱼[25]、大黄鱼[26]等温度应激的研究中发现,细胞对环境温度升高得最快反应之一是机体代谢速率的改变。本研究中,通过对差异表达基因进行GO功能注释和KEGG富集分析后发现,这些差异基因也主要参与代谢相关的多个生物学过程;GO功能富集分析显示,差异表达基因富集较为显著的GO Term主要分布在脂质代谢过程、细胞修饰氨基酸代谢过程、类固醇代谢过程及蛋白质代谢过程等;KEGG富集分析也显示,代谢相关的通路被显著富集,如氨基酸的生物合成、萜类化合物、精氨酸/脯氨酸代谢及脂类代谢等通路。由此推测,罗非鱼在应对持续性高温胁迫的过程中,机体内的新陈代谢和能量消耗不断增强,差异基因可能更多地通过代谢通路相互作用的方式共同应答高温胁迫。
3.3 高温胁迫下罗非鱼的内质网蛋白质加工通路分析
早期研究表明,当机体处于环境胁迫的条件下,机体内许多酶和结构蛋白的功能和结构均会发生改变,机体此时则通过激发热应激蛋白的合成保护自身抵抗逆境。内质网不仅是细胞内Ca2+的贮存场所,也是细胞内重要的细胞器,其在蛋白质的合成转运及糖基化修饰等方面发挥着重要的作用,具有重要的生理功能。值得注意的是,内质网的蛋白质加工通路在持续性高温胁迫下,参与该通路的差异基因数目最多,且差异基因表达明显上调,其中,参与肝脏内质网相关性降解(ERAD)过程的Hsp70和Hsp90表达显著上调,Hsp90家族蛋白在维持细胞稳态及细胞应激响应方面发挥着重要的作用[27]。早期研究表明,在热应激状态下,机体中Hsp相关基因的表达可通过调节不同的细胞功能保护机体免于遭受热应激带来的伤害[28]。本研究中,热应激蛋白Hsp70和Hsp90发生了显著上调,推测热应激蛋白受高温胁迫时激发表达的特性,这些伴侣蛋白与变性蛋白质间相互作用,防止其发生聚集和错误折叠,可以使罗非鱼尽可能地避免高温带来的伤害。
4 结论
1)通过对尼罗罗非鱼高温处理组和常温对照组进行体长、体质量指标的统计分析,发现高温限制了罗非鱼的生长。
2)通过分析尼罗罗非鱼肝脏组织在高温胁迫下与常温对照组的转录差异,发现高温影响尼罗罗非鱼肝脏组织基因表达水平,差异基因主要富集在氨基酸代谢、内质网上的蛋白质加工及胰岛素信号转导相关的多个通路。
3)通过RT-qPCR试验对mTOR及其相关信号通路中的差异表达基因行验证,结果与转录组分析结果基本一致。
4)高温对罗非鱼的生长、能量代谢及蛋白质折叠有着显著的影响,本试验结果为解析罗非鱼高温胁迫响应的复杂分子机制提供了科学依据,为发展罗非鱼高温健康养殖管理技术和进一步研究和培育其他鱼类的耐高温性状提供了数据参考。
[1] 陈蓝荪.世界罗非鱼捕捞和养殖的动态特征研究[J].上海水产大学学报,2006,15(4):477-482.
[2] PAYNE A I,COLLINSON R I.A comparison of the biological characteristics of Sarotherodon niloticus (L.) with those of S.aureus (Steindachner) and other tilapia of the delta and lower Nile[J].Aquaculture,1983,30(1/2/3/4):335-351.
[3] 刘光琳,谢青夏,梁丽,等.罗非鱼年总产量全国第三位[J].农家之友,2018(12):30-31.
[4] 罗祖娟,林勇,杨慧赞,等.高温对罗非鱼生长发育及病害的影响[J].广西畜牧兽医,2013,29(1):48-51.
[5] LI E C,LI C.Use of RNA-seq in aquaculture research[J].Poultry,Fisheries & Wildlife Sciences,2014,2(2):1000e108.
[6] BILYK K T,CHENG C H C.RNA-seq analyses of cellular responses to elevated body temperature in the high Antarctic cryopelagic nototheniid fish Pagothenia borchgrevinki[J].Marine Genomics,2014,18:163-171.
[7] QUINN N L,MCGOWAN C R,COOPER G A,et al.Identification of genes associated with heat tolerance in Arctic charr exposed to acute thermal stress[J].Physiological Genomics,2011,43(11):685-696.
[8] 彭婷,胡庭俊,林勇,等.低温胁迫对罗非鱼血液生化、免疫及抗氧化指标的影响[J].水产科学,2012,31(5):259-263.
[9] 马旦梅,程光平,喻海燕.吉富罗非鱼对低温持续胁迫的死亡反应[J].广西农业科学,2010,41(7):726-728.
[10] 王令玲,仇潜如.尼罗罗非鱼胚胎及胚后发育的观察[J].动物学报,1981,27(4):327-335.
[11] 杨东,余来宁,张繁荣,等.筛选与尼罗罗非鱼性别相关的AFLP标记[J].水生生物学报,2007,31(6):901-904.
[12] NDONG D,CHEN Y Y,LIN Y H,et al.The immune response of tilapia Oreochromis mossambicus and its susceptibility to Streptococcus iniae under stress in low and high temperatures[J].Fish & Shellfish Immunology,2007,22(6):686-694.
[13] CHENG A C,CHENG S A,CHEN Y Y,et al.Effects of temperature change on the innate cellular and humoral immune responses of orange-spotted grouper Epinephelus coioides and its susceptibility to Vibrio alginolyticus[J].Fish & Shellfish Immunology,2009,26(5):768-772.
[14] LIU S K,WANG X L,SUN F Y,et al.RNA-Seq reveals expression signatures of genes involved in oxygen transport,protein synthesis,folding,and degradation in response to heat stress in catfish[J].Physiological Genomics,2013,45(12):462-476.
[15] KIM D,PERTEA G,TRAPNELL C,et al.TopHat2:accurate alignment of transcriptomes in the presence of insertions,deletions and gene fusions[J].Genome Biology,2013,14(4):R36.
[16] FLOREA L,SONG L I,SALZBERG S L.Thousands of exon skipping events differentiate among splicing patterns in sixteen human tissues[J].F1000 Research,2013,2:188.
[17] ANDERS S,HUBER W.Differential expression analysis for sequence count data[J].Genome Biology,2010,11(10):R106.
[18] 郭恩彦,郭忠宝,罗永巨.吉富罗非鱼最适生长水温研究[J].广东海洋大学学报,2011,31(1):88-93.
[19] Brander K M.Global fish production and climate change[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(50):19709-19714.
[20] 罗辉,叶华,肖世俊,等.转录组学技术在水产动物研究中的运用[J].水产学报,2015,39(4):598-607.
[21] BUTLER A A,LE ROITH D.Control of growth by the somatropic axis:growth hormone and the insulin-like growth factors have related and independent roles[J].Annual Review of Physiology,2001,63:141-164.
[22] BAKER J,LIU J P,ROBERTSON E J,et al.Role of insulin-like growth factors in embryonic and postnatal growth[J].Cell,1993,75(1):73-82.
[23] LIU J P,BAKER J,PERKINS A S,et al.Mice carrying null mutations of the genes encoding insulin-like growth factor I (Igf-1) and type 1 IGF receptor (Igf1r)[J].Cell,1993,75(1):59-72.
[24] RAMEH L E,CANTLEY L C.The role of phosphoinositide 3-kinase lipid products in cell function[J].Journal of Biological Chemistry,1999,274(13):8347-8350.
[25] VERGAUWEN L,BENOOT D,BLUST R,et al.Long-term warm or cold acclimation elicits a specific transcriptional response and affects energy metabolism in zebrafish[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2010,157(2):149-157.
[26] QIAN B Y,XUE L Y.Liver transcriptome sequencing and de novo annotation of the large yellow croaker (Larimichthy crocea) under heat and cold stress[J].Marine Genomics,2016,25:95-102.
[27] JAYASUNDARA N,GARDNER L D,BLOCK B A.Effects of temperature acclimation on Pacific bluefin tuna (Thunnus orientalis) cardiac transcriptome[J].American Journal of Physiology,2013,305(9):R1010-R1020.
[28] RYCKAERT J,PASMANS F,TOBBACK E,et al.Heat shock proteins protect platyfish (Xiphophorus maculatus) from Yersinia ruckeri induced mortality[J].Fish & Shellfish Immunology,2010,28(1):228-231.