精子缺乏基因(deleted in azoospermia,DAZ)是精子生成调控因子,DAZ基因家族成员均包含一个保守的RNA结合结构域,并在生殖细胞中特异表达。由DAZ基因家族编码的RNA结合蛋白在调控动物生殖细胞分化与发育过程中发挥重要作用[1-3]。目前,在DAZ基因家族中,研究较多的基因为boule、daz和dazl,其中,boule和dazl位于常染色体,daz位于Y染色体。boule是DAZ基因家族的最原始成员,在生殖细胞中特异表达,其分子结构和功能高度保守[4-5]。
boule基因在调控生殖细胞发育和分化的过程中发挥了重要作用,在不同物种中其表达模式也不同。在哺乳动物中,boule在精巢中特异表达,缺失boule会导致生精细胞分化为成熟精子的过程受阻,使人类患无精子症[6];在果蝇中,boule也在精巢中特异表达,缺失boule会使精子发生的进程一直停滞在初级精母细胞向次级精母细胞分化的阶段,从而使精子发生停止[7];在线虫中,boule仅在卵巢中表达[8],boule的缺失会使卵子发生的减数分裂过程受阻,从而造成卵子发生障碍[9];在淡水养殖动物中,青鳉和虹鳟卵巢和精巢中均能检测到boule的表达[10-11];在海水养殖动物中,栉孔扇贝boule在除了成熟精子外的一切雄性生殖细胞的细胞质中表达[12],紫海胆boule在其精巢中表达,但在卵巢中的表达较精巢微弱[13]。目前,针对boule在海水养殖动物尤其是棘皮动物中的研究较少,其表达情况及作用机理尚不明确。
光棘球海胆Mesocentrotus nudus隶属于棘皮动物门Echinodermata游在亚门Eleutherozea海胆纲Echinoidea正形目Camarodonta球海胆科Strongylocentrotidae,俗称大连紫海胆,是西北太平洋沿岸水域较常见的经济海胆类之一,也是中国北方沿海最主要的经济种类之一[14]。光棘球海胆体型属于大中型,性腺是其唯一可食用部分,成熟季节生殖腺呈淡黄色至橙黄色,质量好,富含不饱和脂肪酸[15],营养价值高,适合于加工冰鲜海胆。本研究中,以光棘球海胆为研究对象,通过克隆获得光棘球海胆boule cDNA序列全长,追踪了boule在光棘球海胆组织、各时期性腺及早期胚胎发育过程中的表达情况,并通过切片原位杂交技术对boule基因在性腺细胞中的定位情况进行了分析,旨在为海胆生殖细胞的深入研究提供科学参考。
1 材料与方法
1.1 材料
试验用光棘球海胆采自大连黄海海域(121°33′47″E,38°51′55″N),直径为(60.2±3.8)mm,湿体质量为(76.8±10.0)g。试验开始前将海胆暂养于大连海洋大学农业农村部北方海水增养殖重点实验室循环水槽,水温为(12.41±0.11)℃,pH 为7.96±0.02,每两天换水一次(换水量约30 L),每天投喂一次海带[16]。
试验用去离子甲酰胺购自AMRESCO(美国),DiethylPyrocarbonate(DEPC)购于Sigma-Aldrich®(上海),马来酸、Tween、20×SSC和PBS磷酸缓冲液粉末均购自索莱宝公司(北京)。
1.2 方法
1.2.1 组织总RNA的提取及cDNA的合成 解剖海胆,将所取组织样品迅速置于液氮中速冻后保存于超低温冰箱(-80 ℃)中备用。组织总RNA的提取按照SV Total RNA Isolation System(Promega Z3100)说明书进行,提取完毕后利用微量分光光度计Agilent 2100 Bioanalyzer (Aligent Technologies, Santa Clara, CA, USA)及琼脂糖凝胶电泳检测RNA的浓度及完整性。然后取1 μg总RNA,按照SMARTerTM RACE cDNA Amplification Kit(Clontech 634923)说明书操作建立cDNA文库。总RNA的反转录参照PrimerScriptTMRT reagent Kit(TaKaRa 634860)说明书进行。
1.2.2 cDNA全长克隆 在前期研究中,本实验室获得了光棘球海胆性腺转录组数据库[17],本研究中以人DAZL蛋白序列中保守的RRM结构域为查询序列,查询到了boule基因部分序列,设计5′和3′RACE引物(表1,设计网站:http://biotools. nubic.northwestern,edu/OligoCalc.html),并按照SMARTerTM RACE cDNA Amplification Kit(Clontech 634923)说明书克隆boule cDNA全长。PCR扩增使用的酶为LA Taq酶(TaKaRa RR02MA)。扩增程序为:94 ℃下预变性2 min;94 ℃下循环变性30 s,55 ℃下退火复性30 s,72 ℃下延伸1 min,共进行35个循环;最后在72 ℃下再延伸10 min。PCR产物经过琼脂糖凝胶电泳检测后,将其切胶回收并纯化,随后连接至pMD18-T 载体并转化入Trans5α感受态细胞中,摇床震荡1 h后将其均匀涂布于LB培养基平板上,37 ℃下倒置过夜。次日,用灭菌牙签挑取单菌落于盛有液体LB培养基的离心管中,摇床震荡,菌落PCR筛选阳性克隆并测序。
表1 PCR所用引物
Tab.1 Sequences of the primers used for PCR

引物 primer序列 sequence (5'-3')目的 purposeboule-F1CATTCGAGACGCAGAGCTCAGCAPCRboule-R1GCACCTCTGCAATGTCTTGTGTCPCRboule-5'F1CCATATCGTGGTGCAT5'RACEboule-5'F2TGGAGAGGGGGCTTGTGA5'RACEboule-5'F3CTCTCAAGGGTTGGCGAT5'RACEboule-3'F1AGCAGCAAGCAGCAGCCCCAGT3'RACEboule-3'F2ACCAACATCAACAGTTTGCCTACC3'RACEboule-ORF-F1GTTGATCACATCGCCATTORF全长boule-ORF-R1TACACTTCACATGACAGGCAORF全长qboule-F1GAGACGCCAGAGGAAGCACATAGART-qPCRqboule-R1CGTCCGAAGGATTGAAGTACGCGART-qPCRUbiquitin-FTGGTCGCACTCTCTCAGACTACAACRT-qPCRUbiquitin-RTGCCGTCTCTCAATTTGTCTATAGCRT-qPCRboule-control-FTAATACGACTCACTATAGGACGTACAGGCAGCTACACACGGTSISHboule-control-RGGTTCCATGATTGGCACCTCTGCASISHboule-probe-FACGTACAGGCAGCTACACACGGTSISHboule-probe-RTAATACGACTCACTATAGGGGTTCCATGATTGGCACCTCTGCASISH
注:粗体标记为T7启动子序列。
Note: The T7 promoter sequence is shown in bold font
1.2.3 boule序列及系统进化分析 利用NCBI中ORF-Finder预测boule基因的开放阅读框;使用SMART(http://smart.embl-heidelberg.de/)及ExPasy中的Protparam工具(https://web.expasy. org/ protparam/)预测Boule蛋白的结构域和基本理化性质;使用Predict(Proteinhttps://www. predictprotein.org/)及Swiss-model(https://swissmodel.expasy.org/)分析Boule蛋白的二级结构和三级结构;利用Clustal W进行多重序列比对,并用MEGA 7.0软件对Boule氨基酸序列进行系统进化分析。
1.2.4 荧光定量PCR检测基因表达 首先,取3只光棘球海胆,获得肠、管足、体腔液、性腺的组织样品。其次,考虑到boule基因在性腺中的表达量较高,本试验中通过持续取样,获得处于不同发育时期包括恢复期(stage Ⅰ)、生长期(stage Ⅱ)、成熟前期(stage Ⅲ)和成熟期(stage Ⅳ)的性腺样品。最后,通过催产并持续取样,获得光棘球海胆受精卵、4细胞期、8细胞期、16细胞期、囊胚、破膜期、棱柱幼虫、四腕幼虫和六腕幼虫的胚胎样品。
利用荧光定量PCR检测各样品的boule基因表达,每个样品孔进行3次技术重复,荧光定量PCR试验总体进行3次验证。以Ubiquitin为内参基因,采用2-ΔΔCt方法计算boule基因在各组织、各时期性腺组织、各胚胎发育时期的表达情况。荧光定量PCR仪器为LightCycler® 96(Roche)。PCR反应体系(共20 μL)包括:Fast Start Essential DNA Green Master 10 μL,上、下游引物各0.8 μL,cDNA 2 μL,DEPC水 6.4 μL。PCR反应程序为:95 ℃下预变性10 min;95 ℃下循环变性15 s,60 ℃下退火复性60 s,共进行40个循环;95 ℃下反应10 s,65 ℃下反应60 s,97 ℃下反应1 s。
1.2.5 RNA探针合成 在boule的cDNA全长序列中设计引物,PCR扩增后将特异的目的片段用切胶回收的方式进行DNA纯化,置于-20 ℃下备用。按照T7 RNA Polymerase(Roche,10881767001)和DIG RNA labeling mix(Roche,11277073910)说明书制备RNA探针,即在无酶微量离心管中配制PCR反应体系,PCR product or DNA 1 μg,DIG RNA Labeling Mix 1 μL,Transcription buffer 1 μL,T7酶 1 μL,RNAsein 0.5 μL,用DEPC水补足至总体积为10 μL,离心混匀后于37 ℃下反应2 h。完成后,加入1 μL DNA酶,37 ℃反应15 min,以消化剩余模板,反应结束后加入1 μL 0.2 mol/L EDTA终止消化,随后加入预冷后的2.5 μL 4 mol/L LiCl和75 μL无水乙醇,-20 ℃下沉淀过夜。次日,在4 ℃条件下,以16 170×g离心15 min,再用体积分数为75%的无水乙醇洗涤两次,最后加入25 μL DEPC水溶解RNA,并用微量分光光度计及琼脂糖凝胶电泳检测RNA探针的浓度及质量。
1.2.6 RNA原位杂交 参照本实验室以前的方法进行[18]。
1) 样品处理。以光棘球海胆卵巢及精巢各时期的性腺样品为试验组,以光棘球海胆精巢生长期性腺样品为对照组。将海胆性腺组织置于4%(体积分数)多聚甲醛中固定过夜;第2天,用DEPC-PBS摇洗3次,每次5 min,然后用300 g/L蔗糖-PBS溶液渗透过夜;第3天,使用冰冻包埋剂(OCT)进行包埋,包埋时注意组织方向;随后用LeicaCM1900-1-1进行冷冻切片,切片完毕后,将玻片于37 ℃烤箱中烘烤1 h。
2) 固定。烘烤后的玻片置于盛满DEPC-PBS的卧缸中复水,室温摇洗2次,每次5 min,用4%(体积分数)多聚甲醛固定30 min后,再将玻片置于盛满DEPC-PBS的卧缸中摇洗2次,每次5 min。
3) 杂交。在每个湿盒上平铺3张滤纸,用湿盒缓冲液(20×SSC 2.5 mL、DEPC水22.5 mL、去离子甲酰胺25 mL)充分浸透滤纸,探针预变性(70 ℃,5 min)后,每个玻片上滴加200 μL杂交液,盖上盖玻片,用封口膜封住湿盒边缘,置于60 ℃杂交箱中,杂交16~20 h。
4) 洗脱。配制洗脱液(20×SSC 12.5 mL、去离子甲酰胺125 mL、Tween 0.25 mL、无菌水112.5 mL)和0.2×SSC,预热至63 ℃。将玻片浸入盛有洗脱液的卧缸,轻柔摇掉盖玻片,63 ℃下摇洗2次,每次30 min;用0.2×SSC在63 ℃下摇洗2次,每次30 min;MABT室温摇洗2次,每次30 min;随后用封阻液(体积分数为10%的山羊血清+体积分数为90%的MABT)室温封阻1 h;抗体孵育(体积分数为2%的山羊血清+体积分数为98%MABT+Anti-dig-body,抗体的使用比例为 1∶5 000),4 ℃下过夜。
5) 显色及镜检。用MABT室温摇洗3次,每次5 min;用碱性磷酸酶显色缓冲液室温摇洗3次,每次5 min;按照说明书配制显色液(碧云天,C3206),避光显色,显色完成后, 用PBS室温摇洗2次,每次5 min;然后进行甲醇梯度脱色(25%、50%、75%、100%、75%、50%、25%,均为体积分数),每次5 min;再用PBS室温摇洗2次,每次5 min,完成后甘油封片,并使用显微镜(Leica DM4B)拍照。
1.3 数据处理
试验数据采用SPSS 23.0软件进行单因素方差分析(One-way ANOVA),采用Duncan法进行组间多重比较,显著性水平设为0.05。
2 结果与分析
2.1 boule cDNA全长序列分析
本研究中,克隆得到boule cDNA全长序列为1 788 bp,其中,5′UTR为146 bp,3′UTR为601 bp,开放阅读框(open reading frame,ORF)长度为1 041 bp,共编码346个氨基酸。结构域预测结果表明,Boule蛋白具有一个进化上高度保守的RRM结构域(图1),Boule蛋白理论相对分子质量为38 070,理论等电点为7.84。Boule蛋白二级结构中包含27.46%的α -螺旋(Alpha helix,H)、13.58%的延伸链(Extended strand,E)和58.96%的无规则卷曲(Random coli,C)。将Boule蛋白以人的Dazl蛋白三维结构为模板(模板号:2xs5.1.A)进行同源建模,发现Boule蛋白在第39个氨基酸(右框)和第112个氨基酸(左框)处存在弯曲和延伸的β1、β4链,这种构象在不同物种的蛋白序列中十分保守,对于RNA识别至关重要(图2)。Boule蛋白疏水残基106个,亲水残基232个,亲水残基数大于疏水残基数,为亲水性蛋白。

加粗部分为起始和终止密码子;画线部分为RRM。
Bold part indicates the start codon and the stop codon;RRM is indicated by underlined part.
图1 光棘球海胆boule基因cDNA序列及预测的氨基酸序列
Fig.1 Full-length cDNA and the deduced amino acid sequence of boule gene in the sea urchin Mesocentrotus nudus

黑色框架内代表的是以人Dazl蛋白为模板推测的Boule蛋白存在的β1、β4卷曲结构。
Black frame represents the β1 and β4 coil structure of Boule protein based on human Dazl protein as template.
图2 Boule蛋白结构域预测及其蛋白三级结构预测
Fig.2 Prediction of Boule protein domain and protein tertiary structure
2.2 多重序列比对和系统进化分析
用于多重序列比对物种及其Boule氨基酸序列在NCBI中的登录号见表2。氨基酸序列比对结果,光棘球海胆与紫海胆Strongylocentrotus purpuratus Boule氨基酸序列一致性最高,为66.48%,与长棘海星Acanthaster planci的一致性达到43.26%,与其他脊椎动物如黑腹果蝇Drosophila melanogaster、虹鳟Oncorhynchus mykiss、大西洋鲑Salmo salar、原鸡Gallus gallus和人Homo sapiens的一致性较低,分别为22.83%、20.62%、30.45%、27.43%、29.55%(图3(a))。光棘球海胆Boule蛋白具有一个进化上保守的RRM结构域,与紫海胆Boule氨基酸序列的一致性高达99.15%,与其他动物的一致性为41.53%~56.78%(图3(b))。
表2 多重序列比对和系统进化树所用的Boule信息
Tab.2 Information on Boule for multiple sequence alignment and phylogenetic analysis

物种 speciesNCBI登录号NCBI accession No.紫海胆 Strongylocentrotus purpuratusXP_011682290.1长棘海星 Acanthaster planciXP_022079994.1黑腹果蝇 Drosophila melanogasterNP_001286992.1虹鳟 Oncorhynchus mykissXP_ADW41783.1大西洋鲑 Salmo salarXP_014008057.1原鸡 Gallus gallusXP_015144981.1人 Homo sapiensXP_011509995.1

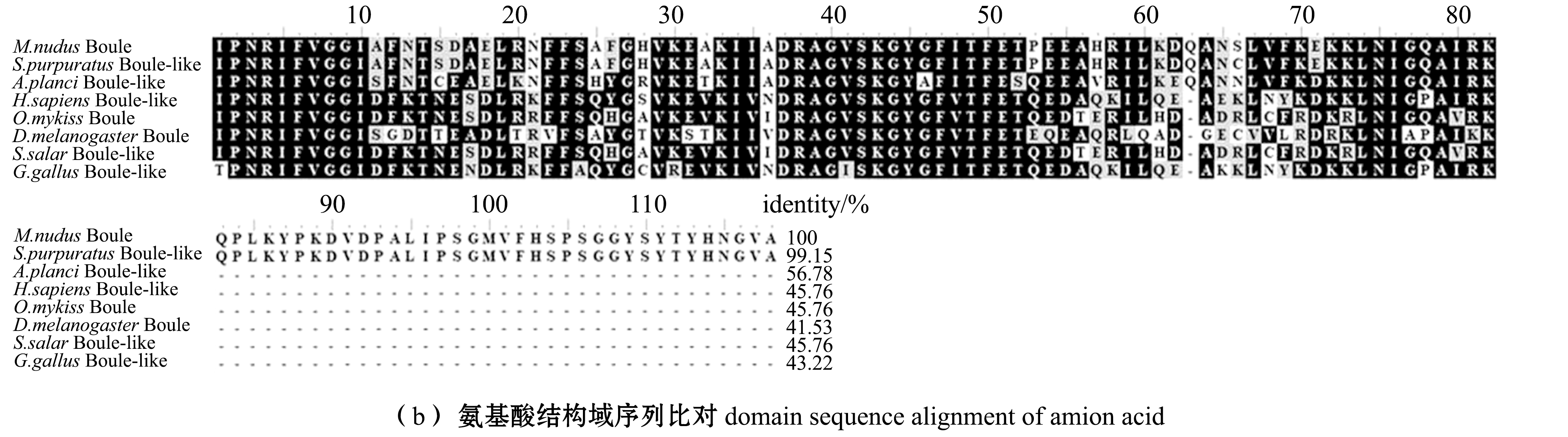
图3 boule基因编码的氨基酸多序列比对及结构域序列比对
Fig.3 Multiple sequence alignment and domain sequence alignment of amino acids encoded by the boule gene
基于以上试验结果,利用MEGA7构建了Neighbor-joining(NJ)系统进化树,结果显示,光棘球海胆、紫海胆和长棘海星的Boule氨基酸序列聚为一支,基本符合物种亲缘进化趋势(图4)。

图4 Boule氨基酸序列的系统进化树
Fig.4 Phylogenetic tree of Boule amino acid sequences
2.3 boule基因的组织表达分析
在光棘球海胆生长期(stageⅡ)的肠、管足、体腔细胞、卵巢及精巢中,均检测到了boule的表达,表达情况为精巢>卵巢>管足>肠>体腔液,其中,boule在精巢中表达量最高,且在精巢与卵巢中呈现差异表达,在精巢中的表达量约为卵巢中的25倍(图5)。
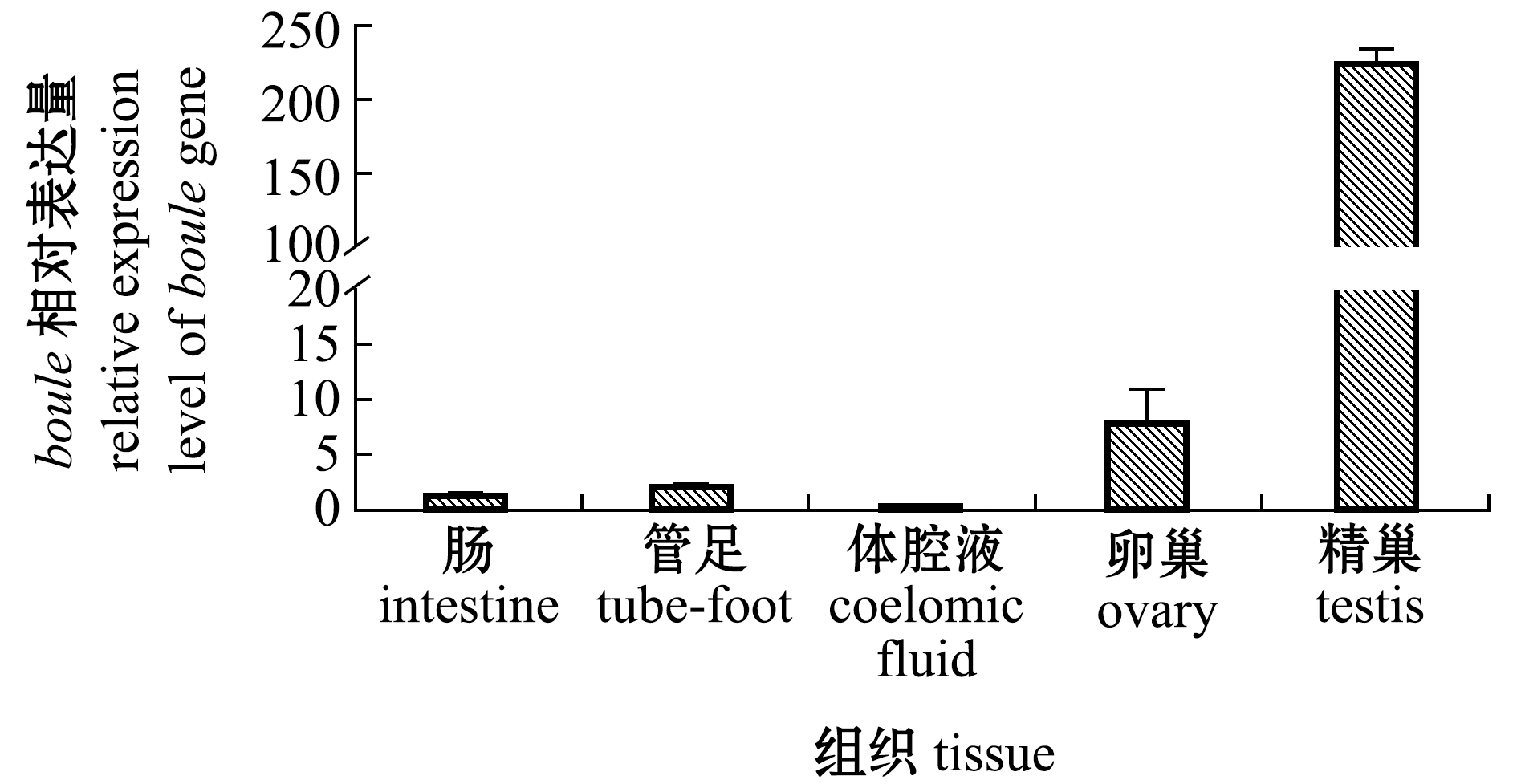
图5 boule基因的组织表达分析
Fig.5 Expression of boule gene in adult tissues
2.4 boule基因在不同时期性腺中的动态表达分析
荧光定量PCR结果表明,boule基因表达量在卵巢中总体呈现上升趋势,直至成熟期(stage Ⅳ)表达量最高;精巢中的boule表达量仅在生长期(stage Ⅱ)及成熟前期(stage Ⅲ)较高(图6)。
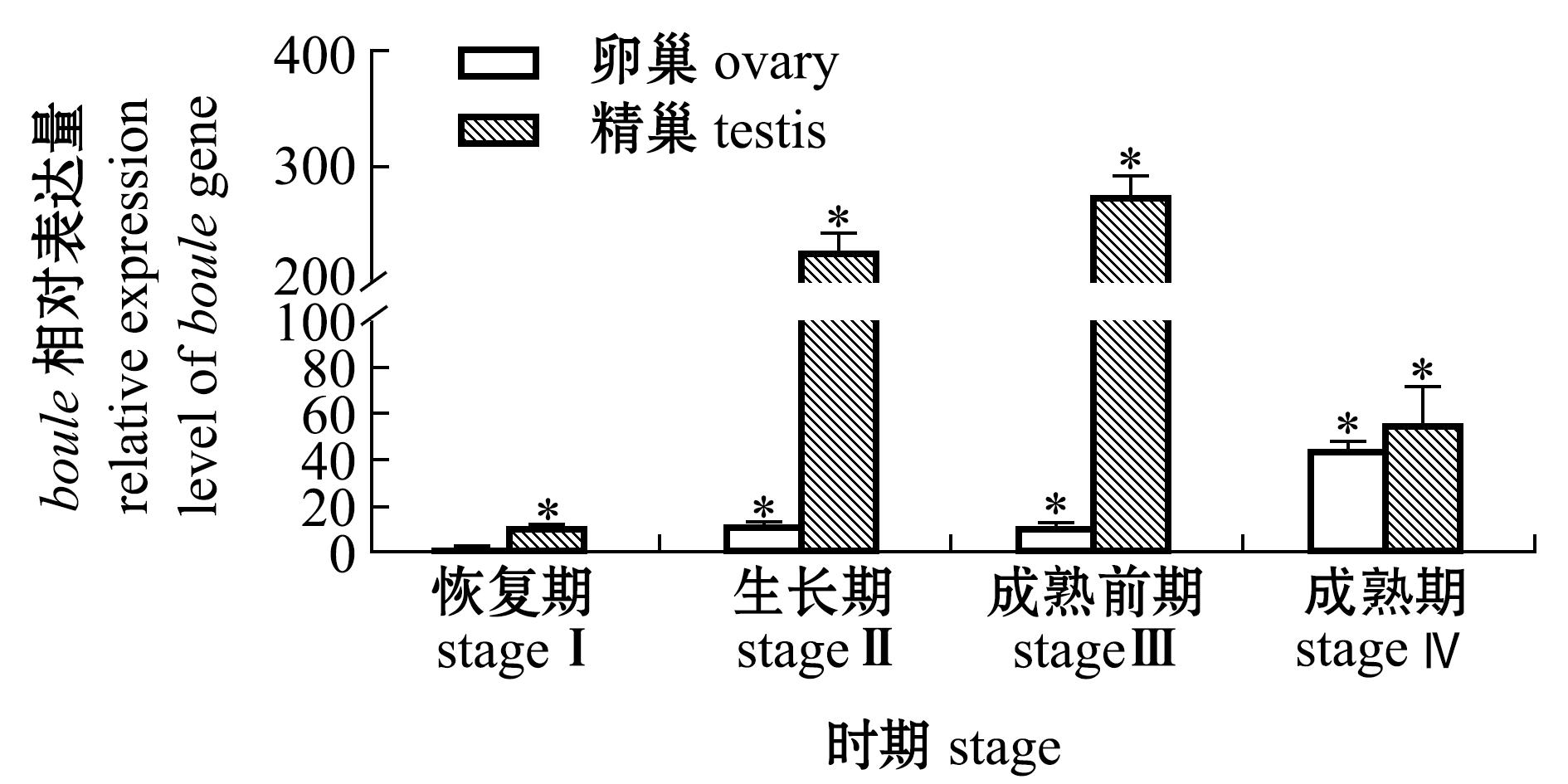
*表示与卵巢恢复期有显著性差异(P<0.05)。
* means significant difference compared with stage Ⅰ of the ovary(P<0.05).
图6 boule基因在不同性腺发育时期的动态表达分析
Fig.6 Dynamic expression analysis of boule gene in different gonadal development stages
通过RNA切片原位杂交技术检测boule基因在性腺细胞中的定位,结果显示,仅在生长期及成熟前期的精巢中检测到阳性细胞信号,其余时期未检测到信号(图7)。可能是由于表达量低、卵巢中的RNA不稳定及卵黄沉积等原因,并未在卵巢中检测到明显的阳性细胞信号。
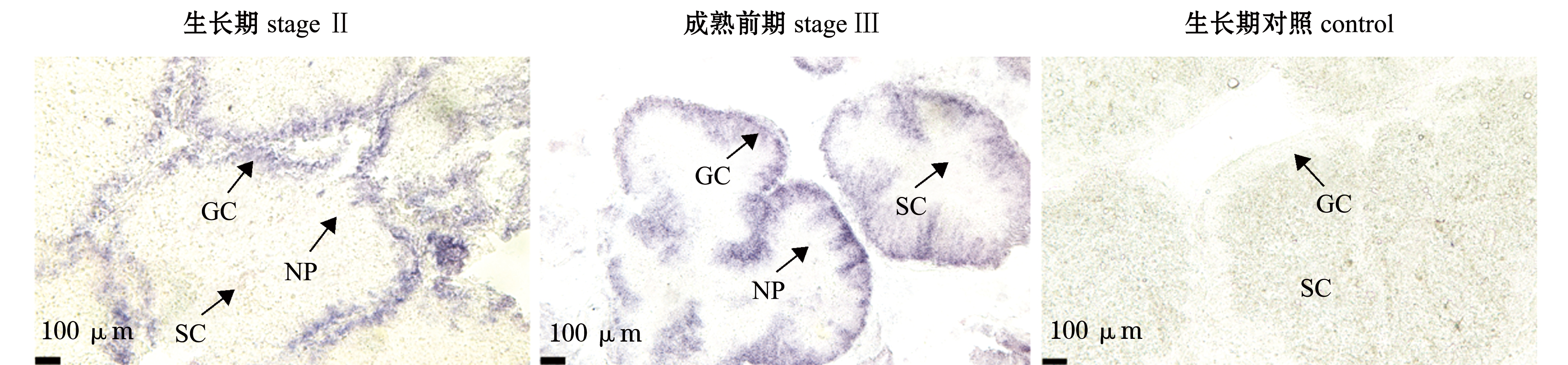
NP—营养吞噬细胞;SC—精细胞;GC—生殖细胞。
NP—nutritive phagocyte;SC—sperm cell;GC—germ cell.
图7 切片原位杂交分析boule基因在精巢组织中的表达情况
Fig.7 Expression of boule gene in testis by section in situ hybridization (SISH) analyses
2.5 boule基因在胚胎发育时期的表达特性分析
通过荧光定量PCR检测发现:光棘球海胆胚胎发育过程中,在受精卵内检测到大量boule转录本,由此说明boule为母源因子;随着细胞的增殖与分化和胚胎发育过程的进行,boule基因表达量逐渐降低,直到棱柱幼虫时期降到最低;从棱柱幼虫时期开始,由于幼虫开始变态及开始摄取饵料,boule基因表达量随着幼虫的发育,又呈现出逐渐升高的态势(图8)。
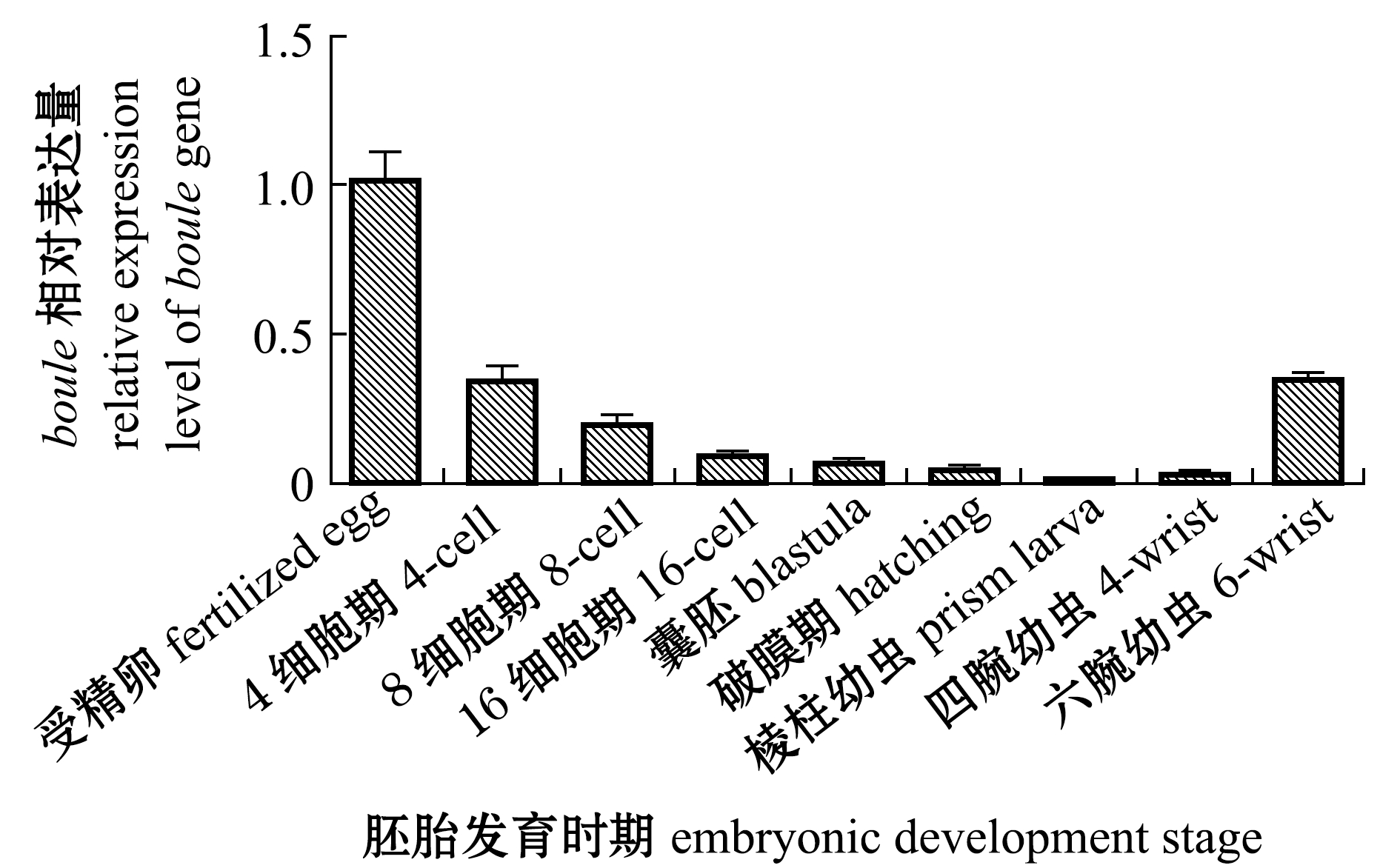
图8 boule基因在胚胎发育时期的表达分析
Fig.8 Expression of boule gene in embryonic development
3 讨论
3.1 光棘球海胆boule基因序列全长及RRM结构域分析
本研究中克隆了光棘球海胆boule cDNA全长序列,预测出Boule蛋白存在一个结构与功能十分保守的RRM结构域,RRM结构域隶属于RRM superfamily,包含两段保守的序列,即RNP1和RNP2 (RNP: Ribonucleoprotein,核糖核蛋白)。RRM结构域是由DAZ基因家族编码的RNA结合蛋白,在调控动物的生殖细胞分化与发育中发挥重要作用,为生殖细胞发育所必需。虽然不同物种间Boule氨基酸序列全长一致性较低(20.62%~66.48%),但Boule的RRM结构域氨基酸序列比对一致性较高,尤其与紫海胆的一致性高达99.15%,boule基因的进化趋势与光棘球海胆物种基本一致。
3.2 光棘球海胆boule基因在性腺中的表达情况
本研究中发现,不同于哺乳动物、果蝇等多种生物的boule仅在精巢或卵巢中特异表达,光棘球海胆中的boule在雌、雄性腺中均有表达,尤其是精巢中的表达量较高,这与栉孔扇贝中boule的表达情况类似。此外,boule在肠、管足、体腔液中也有少量的表达,意味着boule不仅在生殖细胞发育过程中发挥作用,还可能有未知的生物学功能。
本研究中,通过追踪boule在不同发育时期性腺中的表达情况,发现boule在精巢中的表达量高于卵巢,在生长期(stage Ⅱ)约为卵巢表达量的25倍,且在精巢生长期(stageⅡ)及成熟前期(stage Ⅲ)中的表达量最高。这种表达模式与紫海胆中的表达情况类似。在其他物种中,boule主要在精巢中表达,如boule基因特异地表达于果蝇的精巢,并参与调控果蝇的精子发生[7]。由此可以预测,boule可能参与调控光棘球海胆的精子发生过程。此外,本研究中还发现,boule基因的表达量随着雌性光棘球海胆卵母细胞的发育成熟而逐渐升高,由此推测,boule基因可能与光棘球海胆卵母细胞的成熟有关。
3.3 光棘球海胆boule基因在早期胚胎发育过程中的表达情况
在脊椎动物中,作为蝾螈母源因子的dazl基因在早期胚胎发育过程中持续表达[19],在硬骨鱼中,牙鲆dazl基因在起始阶段的表达量高于其他阶段[20];在无脊椎动物中,栉孔扇贝合子基因开始表达前,boule的表达量变化不大,但当胚胎发育至原肠胚期时其表达量明显增加[13]。这些均与本研究结果相似,因此,无论是无脊椎动物还是脊椎动物,boule在胚胎发育时期的表达模式均较相近,这表明boule在生殖细胞特化及形成中可能比较保守。
综上所述,本研究中既完善了无脊椎动物中棘皮动物门动物生殖腺的生长发育,又开拓了生殖细胞的分化在非模式生物中的研究空间。以上结果也预示光棘球海胆boule基因可作为生殖细胞标记基因,可为生殖细胞相关的研究提供参考。
4 结论
1) boule基因包含一个进化上高度保守的RRM结构域,其可能在调控动物生殖细胞发育分化过程中发挥重要作用。
2) boule基因只在光棘球海胆的生殖细胞中高表达,且在雄性生殖细胞中特异表达,是雄性生殖细胞的标记基因,这为研究其精巢形成和分化、精子发生等过程提供了数据资料。
3) 整个胚胎发育时期均检测到boule基因的持续表达,boule为母源因子,这为研究光棘球海胆生殖细胞特化及形成提供了数据支撑。
[1] GILL M E,HU Y C,LIN Y F,et al.Licensing of gametogenesis,dependent on RNA binding protein DAZL,as a gateway to sexual differentiation of fetal germ cells[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(18):7443-7448.
[2] SPRADLING A,FULLER M T,BRAUN R E,et al.Germline stem cells[J].Cold Spring Harbor Perspectives in Biology,2011,3(11):a002642.
[3] REIJO R A,DORFMAN D M,SLEE R,et al.DAZ family proteins exist throughout male germ cell development and transit from nucleus to cytoplasm at meiosis in humans and mice[J].Biology of Reproduction,2000,63(5):1490-1496.
[4] REIJO R,LEE T Y,SALO P,et al.Diverse spermatogenic defects in humans caused by Y chromosome deletions encompassing a novel RNA-binding protein gene[J].Nature Genetics,1995,10(4):383-393.
[5] SAXENA R,BROWN L G,HAWKINS T,et al.The DAZ gene cluster on the human Y chromosome arose from an autosomal gene that was transposed,repeatedly amplified and pruned[J].Nature Genetics,1996,14(3):292-299.
[6] XU E Y,MOORE F L,PERA R A R.A gene family required for human germ cell development evolved from an ancient meiotic gene conserved in metazoans[J].Proceedings of the National Academy of Sciences of the United States of America,2001,98(13):7414-7419.
[7] VAN GOMPEL M J W,XU E Y.The roles of the DAZ family in spermatogenesis[J].Spermatogenesis,2011,1(1):36-46.
[8] KARASHIMA T,SUGIMOTO A,YAMAMOTO M.Caenorhabditis elegans homologue of the human azoospermia factor DAZ is required for oogenesis but not for spermatogenesis[J].Development,2000,127(5):1069-1079.
[9] OTORI M,KARASHIMA T,YAMAMOTO M.The Caenorhabditis elegans homologue of deleted in azoospermia is involved in the sperm/oocyte switch[J].Molecular Biology of the Cell,2006,17(7):3147-3155.
[10] XU H Y,LI Z D,LI M Y,et al.Boule is present in fish and bisexually expressed in adult and embryonic germ cells of medaka[J].PLoS One,2009,4(6):e6097.
[11] LI M Y,SHEN Q,XU H Y,et al.Differential conservation and divergence of fertility genes boule and dazl in the rainbow trout[J].PLoS One,2011,6(1):e15910.
[12] 邵明瑜.栉孔扇贝生殖相关基因DEAD-box家族和boule的cDNA克隆及其发育表达图式[D].青岛:中国海洋大学,2007.
[13] JULIANO C E,VORONINA E,STACK C,et al.Germ line determinants are not localized early in sea urchin development,but do accumulate in the small micromere lineage[J].Developmental Biology,2006,300(1):406-415.
[14] 常亚青,丁君,宋坚,等.海参、海胆生物学研究与养殖[M].北京:海洋出版社,2004:199-220.
[15] 李磊,杨丹,亓守冰,等.中间球海胆繁殖前后性腺中脂类和脂肪酸含量变化[J].大连海洋大学学报,2018,33(4):423-429.
[16] 韩奋杰,张伟杰,王中,等.中间球海胆与马粪海胆杂交后代幼胆期耐热性研究[J].大连海洋大学学报,2019,34(6):811-816.
[17] SUN Z H,ZHANG J,ZHANG W J,et al.Gonadal transcriptomic analysis and identification of candidate sex-related genes in Mesocentrotus nudus[J].Gene,2019,698:72-81.
[18] SUN Z H,WANG Y,LU W J,et al.Divergent expression patterns and function implications of four nanos genes in a hermaphroditic fish,Epinephelus coioides[J].International Journal of Molecular Sciences,2017,18(4):685.
[19] JOHNSON A D,BACHVAROVA R F,DRUM M,et al.Expression of axolotl DAZL RNA,a marker of germ plasm:widespread maternal RNA and onset of expression in germ cells approaching the gonad[J].Developmental Biology,2001,234(2):402-415.
[20] 姜佳君.牙鲆(Paralichthys olivaceus)dazl基因的克隆、表达和功能初步研究[D].青岛:中国海洋大学,2015.