瘦素(Leptin)又称OB蛋白,是由肥胖基因OB编码的一类蛋白激素,OB基因最早由Zhang等[1]通过图位克隆技术从小鼠脂肪组织中分离、克隆得到。哺乳动物的Leptin主要在白色脂肪组织(WAT)中合成,其受体广泛表达于中枢和外周系统的组织中[2],Leptin与这些特定受体结合调控动物的摄食[3-4]、脂质代谢[3]、血管生成[5]、骨骼重塑[6]、免疫[7]和繁殖[8]等重要生命活动。
哺乳动物的Leptin属于长链螺旋细胞因子家族,其三维结构特征主要包括4个反向平行的α螺旋[9]。2005年,硬骨鱼的leptin基因首次从红鳍东方鲀Takifugu rubripes[10]中克隆得到,之后还从多种硬骨鱼中克隆和鉴定出了leptin基因。与哺乳动物不同,硬骨鱼的leptin基因主要在肝脏中表达[10-11],且哺乳动物的leptin只有一种,而已有研究表明,除鲀形目与部分鲈形目的种类只有一种leptin外,其他多数硬骨鱼含有2种甚至4种leptin亚型[12],不同亚型基因的空间表达有较大差异,表明这些亚型发挥的生理功能不同[13-15]。硬骨鱼Leptin氨基酸序列与哺乳动物相比相似性较低,但三级结构十分保守,均包含4个α螺旋[10, 13-16]。与哺乳动物相似,硬骨鱼的Leptin同样是影响多种生物学功能的调控因子。在同源Leptin注射试验[12, 16]及对鱼体不同摄食状态leptin基因表达量的检测[13-16]中均显示,leptin与抑制摄食有相关性。此外,leptin对硬骨鱼的脂质也有调节作用[17],但并不像在哺乳动物中一样作为主要的脂肪抑制因子。在leptin对硬骨鱼生殖影响的研究中发现leptin基因的表达与大西洋鲑Salmo salar及鲭Pneumatophorus japonicus的精巢发育有关[18-19];同源leptin能诱导雌性鲭鱼的促性腺激素分泌[20]。但在对缺乏功能性leptin受体斑马鱼的研究中发现,受体缺失的斑马鱼并不会像leptin受体缺乏的小鼠一样丧失生育能力,且未表现出食欲过高或肥胖[21],这表明leptin在硬骨鱼中的生理作用与哺乳动物相比有较大不同,还有待更进一步研究。
暗纹东方鲀Takifugu obscurus是中国特有的河鲀种类,属于硬骨鱼纲鲀形目鲀科东方鲀属,主要分布于中国东、黄海沿海,还可进入长江并栖息于太湖,属广盐性鱼类,是著名的“长江三鲜”之一,具有较高的营养和经济价值[22-24]。暗纹东方鲀和红鳍东方鲀是中国养殖及食用范围最广的两个河鲀品种[24],但前者和后者相比有着生长速度慢的缺点[25],这在一定程度上限制了暗纹东方鲀相关产业的发展。单核苷酸多态性 (single nucleotide polymorphism,SNP)是指生物不同个体 DNA 序列中单个核苷酸点突变产生的多态性[26]。SNP作为一种位点数量多、密度广、遗传多样性高的分子标记,已被广泛应用于与水产动物生长性状相关的研究中[27-29]。本研究中,克隆了暗纹东方鲀东方鲀leptin基因cDNA序列,并基于leptin在硬骨鱼生长发育过程中发挥的重要作用,通过直接测序筛选得到暗纹东方鲀leptin基因中分型稳定、多态性好的SNP位点,并与暗纹东方鲀生长性状进行关联分析,获得与生长性状显著相关的位点作为暗纹东方鲀辅助育种的候选分子标记,以提高选育效率。
1 材料与方法
1.1 材料
试验用暗纹东方鲀均来自江苏中洋集团,随机选择6尾健康的3月龄幼鱼作为基因克隆的试验样本;随机选择同期繁殖、同塘养殖的178 尾8月龄鱼,测量其体质量、体长、体全长指标,剪取背鳍风干保存,作为SNP与生长性状关联分析的试验样本。
1.2 方法
1.2.1 样品制备 取6尾3月龄暗纹东方鲀幼鱼,用 MS-222 麻醉后解剖,剪取肝脏组织经液氮低温处理后于超低温冰箱(-80 ℃)中保存备用。
1.2.2 总RNA提取与cDNA第一链的合成 使用Trizol试剂(TaKaRa)进行总RNA提取,用10 g/L琼脂糖凝胶电泳检测产物的完整性。使用Primer Script RT reagent Kit(TaKaRa)将总RNA反转录成cDNA,于超低温冰箱(-80 ℃)中保存备用。
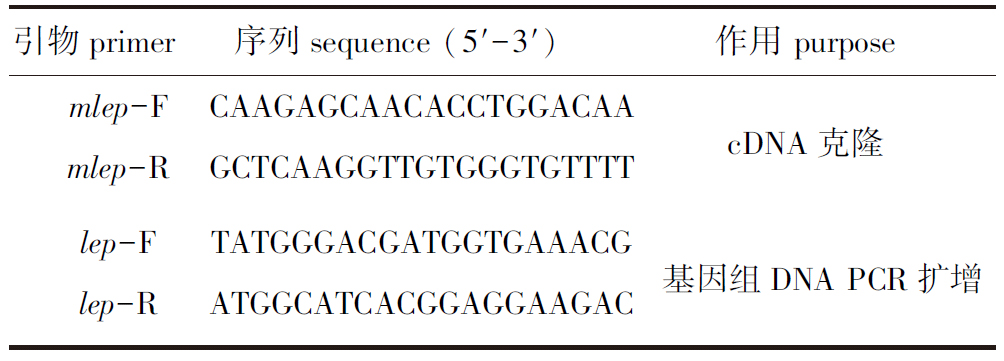
1.2.3 leptin cDNA克隆 参照NCBI数据库中登录的红鳍东方鲀leptin基因mRNA序列信息(GenBank登录号:NM_001032725.1),设计包含红鳍东方鲀蛋白编码区(CDS)的1对引物(表1),采用RT-PCR法克隆暗纹东方鲀的leptin基因。cDNA克隆的反应体系(25 μL)包含:10×PCR Buffer 2 μL,上、下游引物(mlep-F /mlep-R)各1 μL,Taq酶 0.5 μL,dNTP Mixture 2.5 μL,cDNA模板2 μL,ddH2O 16 μL。PCR反应程序为:94 ℃下预变性5 min;94 ℃下变性30 s,60 ℃下退火复性30 s,72 ℃下延伸30 s,共进行35个循环;最后在72 ℃下再延伸10 min,4 ℃下保存。PCR产物经10 g/L琼脂糖凝胶电泳检测后直接送北京六合华大基因科技有限公司测序。
表1 leptin基因克隆及基因组DNA扩增引物序列
Tab.1 Primers used for clone and genomic DNA amplification of the leptin gene
1.2.4 leptin基因序列分析 利用ORF Finder在线工具(https://www.bioinformatics.org/sms2/orf_find. html)预测基因的开放阅读框及其编码的氨基酸序列;以预测的氨基酸序列在NCBI数据库上进行同源比对,选取部分物种的Leptin氨基酸序列,利用MEGA Χ软件构建系统进化树;使用Expasy蛋白组服务器(https://www.expasy.org)中的Protparam分析Leptin蛋白的生理生化特征,用ProtScale预测其亲疏水性,用PROSITE预测氨基酸序列功能域;使用Signal P4.1 Server(http://www. cbs.dtu.dk/services/SignalP/)对Leptin蛋白信号肽进行分析;用SOPMA在线软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_ sopma.html)预测蛋白二级结构;用SWISS-MODEL(http: / /www.swissmodel.expasy. org)构建三维结构模型。
1.2.5 基因组DNA的提取与检测 使用TIANamp Marine Animals DNA Kit(天根生化科技(北京)有限公司)提取暗纹东方鲀基因组DNA,用10 g/L琼脂糖凝胶电泳检测提取的DNA质量并置于冰箱(-20 ℃)中保存待用。
1.2.6 leptin基因的PCR扩增及突变位点基因分型 根据NCBI数据库中红鳍东方鲀的基因组数据(GenBank登录号:NC_042293.1)设计1对引物(表1),从178个暗纹东方鲀DNA样本中随机挑选24个样本PCR扩增leptin基因的片段,通过比对测序结果筛选SNP位点。PCR反应体系(50 μL)包含:10×PCR Buffer 5 μL,上、下游引物(lep-F/lep-R )各2 μL,Taq酶 0.5 μL,dNTP Mixture 4 μL,暗纹东方鲀基因组DNA模板2 μL,ddH2O 34.5 μL。PCR反应程序为:94 ℃下预变性5 min;94 ℃下变性30 s,65 ℃下退火复性30 s,72 ℃下延伸30 s,共进行30个循环;最后在72 ℃下再延伸10 min,4 ℃下保存。PCR产物经10 g/L琼脂糖凝胶电泳检测后直接送北京六合华大基因科技有限公司测序。返回结果用seqMan软件进行拼接比对后得到多态性高、分型稳定的突变位点,并与克隆得到的暗纹东方鲀cDNA序列及红鳍东方鲀基因组数据(GenBank登录号:NC_042293.1)比对确定基因结构。通过直接测序对178尾暗纹东方鲀leptin基因的突变位点进行分型。
1.3 数据处理
采用Popgene 32软件计算有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He),并进行哈温平衡检验(HWE);采用PIC_CALC软件计算SNP位点的多态信息含量(PIC);采用Haploview软件对leptin基因的SNP位点进行连锁不平衡分析;采用SPSS 19.0软件对筛选到的SNP位点与暗纹东方鲀的生长性状进行关联分析。
2 结果与分析
2.1 暗纹东方鲀leptin基因cDNA序列及推测的氨基酸序列结构
克隆得到的暗纹东方鲀leptin cDNA序列长为661 bp,包括459 bp的开放阅读框,共编码152个氨基酸(图1),克隆序列已上传至GenBank数据库中(登录号:MT832308)。推测的Leptin蛋白相对分子质量为19 948.69,等电点为6.90,不稳定值为28.31,亲水性的总体平均值(GRAVY)为-0.089,结合ProtScale预测结果,推测该蛋白为亲水的稳定蛋白。Leptin氨基酸序列含19个氨基酸的信号肽序列,功能位点包含1个N-豆蔻酰化位点、3个蛋白激酶C磷酸化位点和1个N-糖基化位点(图1)。

方框表示起始密码子/终止密码子;双下划线表示信号肽序列;阴影部分表示N-豆蔻酰化位点;单下划线表示蛋白激酶C磷酸化位点;圆圈表示N-糖基化位点。
The box represents the start codon/stop codon; the double underline represents the signal peptide sequence; the shaded part represents the N-myristoylation site; the single underline represents the protein kinase C phosphorylation site; and the circle represents the N-glycosylation site.
图1 leptin cDNA序列及推测的氨基酸序列
Fig.1 Nucleotide sequence and the deduced amino acid sequence of leptin cDNA
推测的Leptin蛋白二级结构中α-螺旋(h)占 71.71%,β-折叠(e)占 2.63%,β-转角(t)占 1.32%,无规卷曲(c)占 24.34%。使用SWISS-MODEL在线工具获得Leptin蛋白的三级结构如图2所示。

图2 推测的Leptin蛋白三维结构模型
Fig.2 Predicted three-dimensional model of Leptin protein
2.2 Leptin氨基酸序列比对及系统进化分析
同源性分析结果显示:暗纹东方鲀Leptin氨基酸序列与红鳍东方鲀的一致性最高,为96.71%;其次为菊黄东方鲀,一致性为93.42%;与其他不同种属的鱼类一致性较低,其中与草鱼Leptin氨基酸序列的一致性仅为25.25%,而与其他脊椎动物的一致性则更低。基于鱼类和其他脊椎动物Leptin氨基酸序列的系统进化树显示,进化树可聚为鱼类、鸟类、两栖类和哺乳类4类,其中鱼类中,草鱼、鲤、鲢聚为一个分支,暗纹东方鲀、菊黄东方鲀、红鳍东方鲀聚为另一个分支(图3)。进化树分析与传统分类结果相一致。
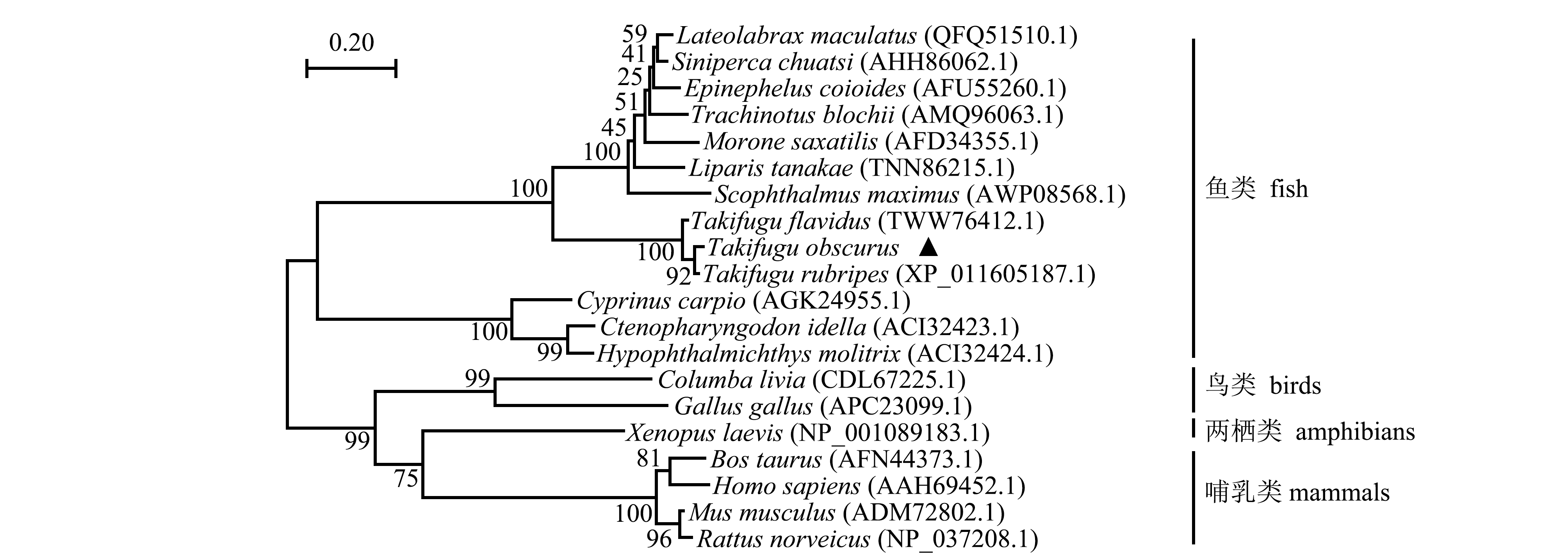
图3 基于Leptin氨基酸序列构建的NJ系统进化树
Fig.3 NJ phylogenetic tree based on Leptin amino acid sequence
2.3 leptin基因SNP位点的筛选
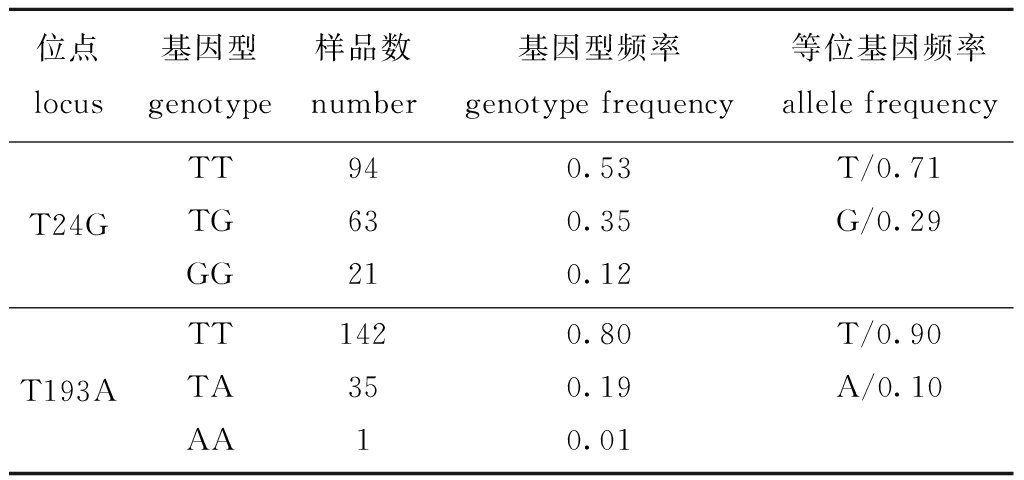
对24个暗纹东方鲀DNA样本进行PCR扩增及测序,获得了长度为691 bp的leptin基因片段,片段包括2个外显子(外显子2,外显子3)及1个内含子(内含子2)。通过序列比对,在暗纹东方鲀leptin基因中筛选得到2个分型稳定的SNP位点:一个位于外显子2(T24G),为同义突变未改变氨基酸的编码;另一个发生在内含子2(T193A)。本研究中,核酸位置以基因起始密码子的第一个碱基为参考值1,统计所有178个序列,得到2个SNP的等位基因频率和基因型频率如表2所示。
表2 leptin基因2个SNP等位基因及基因型频率
Tab.2 Two SNP alleles and genotype frequency of leptin gene
位点locus基因型genotype样品数number基因型频率genotype frequency等位基因频率allele frequency T24GTT940.53T/0.71TG630.35G/0.29GG210.12T193ATT1420.80T/0.90TA350.19A/0.10AA10.01
2.4 leptin基因SNP在暗纹东方鲀群体中的遗传结构分析
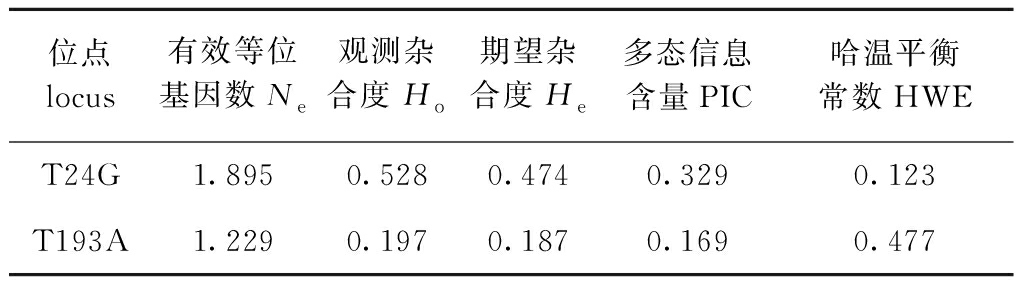
统计2个突变位点的遗传参数,结果如表3所示,其中,T24G中有效等位基因数Ne、观测杂合度Ho、期望杂合度He、多态信息含量PIC数值均高于T193A,T24G是中度多态性位点(0.25≤PIC<0.5),T193A位点是低度多态性位点(PIC<0.25)。经χ2检验,leptin基因的2个SNP在暗纹东方鲀群体中处于哈温平衡状态(P>0.05)。Haploview软件的连锁分析显示,2个SNP间的r2为0.005,表明两个位点彼此独立。
表3 leptin基因SNP位点的遗传参数
Tab.3 Genetic parameters of SNP loci in leptin gene
位点locus有效等位基因数Ne观测杂合度Ho期望杂合度He多态信息含量PIC哈温平衡常数HWET24G1.8950.5280.4740.3290.123T193A1.2290.1970.1870.1690.477
2.5 leptin基因SNP与生长性状的关联分析
采用SPSS 19.0软件分析不同基因型(频率低于2.5%的基因型排除)与生长性状的相关性,结果表明,T24G位点不同基因型个体间的体质量存在显著性差异(P<0.05),GG基因型个体体质量显著小于TG和TT基因型(P<0.05),为劣势基因型(表4)。
表4 leptin基因SNP位点与生长性状的关联分析
Tab.4 Correlation between SNP loci of leptin gene and growth traits

位点locus基因型(个体数)genotype(number)体质量/gbody mass体长/cmbody length体全长/cmtotal lengthT24GTT(94)244.42±53.94a18.79±1.5822.06±1.72TG(63)251.08±48.18a18.85±1.4322.19±1.56GG(21)221.88±54.17b18.42±1.5221.60±1.73T193ATT(142)243.73±49.8718.74±1.4721.99±1.61TA(35)251.49±58.7318.94±1.6222.38±1.78AA(1)———
注:同列中标有不同字母者表示同一位点不同基因型组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。
Note:The means with different letters within the same column are significantly different in the different genetype groups at the same locus at the 0.05 probability level, and the means with the same letter within the same column are not significant differences at the same locus.
将2个SNP位点的不同基因型进行组合,得到7种双倍型,去除频率低于2.5%的D3和D7,将其余5种与生长进行关联分析(表5),结果显示,双倍型D5的3个生长性状值最高,体质量显著高于D2、D4型个体(P<0.05),极显著高于D6型个体(P<0.01),体长和体全长均显著高于D6型个体(P<0.05)。
表5 leptin基因不同双倍型与生长性状的关联分析
Tab.5 Correlation analysis of different diplotypes of leptin gene and growth traits
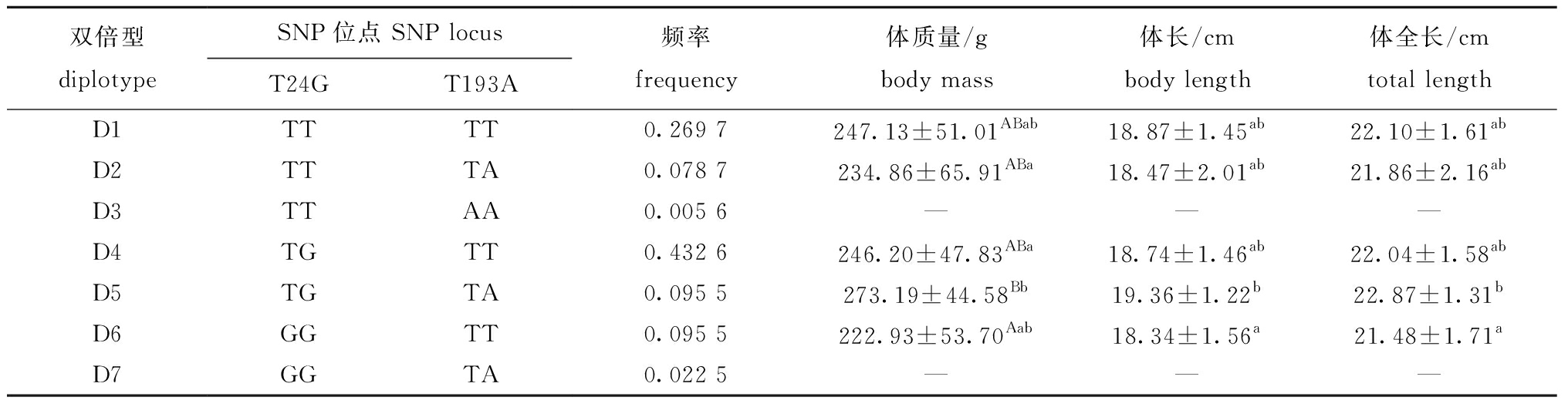
双倍型diplotypeSNP位点 SNP locusT24GT193A频率frequency体质量/gbody mass体长/cmbody length体全长/cmtotal lengthD1TTTT0.269 7247.13±51.01ABab18.87±1.45ab22.10±1.61abD2TTTA0.078 7234.86±65.91ABa18.47±2.01ab21.86±2.16abD3TTAA0.005 6———D4TGTT0.432 6246.20±47.83ABa18.74±1.46ab22.04±1.58abD5TGTA0.095 5273.19±44.58Bb19.36±1.22b22.87±1.31bD6GGTT0.095 5222.93±53.70Aab18.34±1.56a21.48±1.71aD7GGTA0.022 5———
注:同列中标有不同大写字母者表示组间有极显著性差异(P<0.01),标有不同小写字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)。
Note:The means with different capital letters within the same column are very significantly different among the groups at the 0.01 probability level,means with different letters being significantly different at the 0.05 probability level, and the means with the same letter within the same column are not significant differences.
3 讨论
3.1 暗纹东方鲀leptin基因编码的蛋白结构
本研究中,以近缘物种红鳍东方鲀leptin基因的mRNA序列为参照,设计引物并扩增得到了暗纹东方鲀的leptin cDNA。预测蛋白的三级结构模型显示,暗纹东方鲀Leptin符合Leptin的基本特征,蛋白结构包含4个α螺旋。多物种Leptin氨基酸序列比对结果表明,物种间Leptin蛋白的一级结构存在较大差异,这表明物种间Leptin的功能存在较大差异。已有研究显示,在哺乳动物中,Leptin是主要的脂肪抑制因子,但在硬骨鱼中Leptin对脂肪稳态的调节并不明显。Leptin受体缺失的斑马鱼并未表现出受体缺失小鼠食欲亢进、代谢提高和明显肥胖的状态。在硬骨鱼中Leptin主要在葡萄糖的稳态调控中发挥作用,且调节葡萄糖的稳态似乎是硬骨鱼和哺乳动物中的一种保守功能[21]。这可能与物种间Leptin相对稳定的三级结构有关。本研究中,同源性分析显示,暗纹东方鲀Leptin氨基酸序列与同属于东方鲀属的红鳍东方鲀和菊黄东方鲀序列的一致性较高,均在90%以上,这表明Leptin在东方鲀属物种中的功能相对保守,而与其他硬骨鱼、鸟类、两栖类及哺乳类Leptin序列的一致性较低,这表明Leptin在进化过程中出现了较大变化。
3.2 暗纹东方鲀leptin基因SNP与生长关联
本研究中,基于Leptin在硬骨鱼生长发育中的重要作用,以leptin基因作为暗纹东方鲀生长性状的候选基因,通过直接测序方法筛选到了2个分型稳定的SNP位点(T24G、T193A)。T24G位于编码区发生同义突变,未改变氨基酸的编码。但由于密码子的偏好性[30],突变的同义密码子可能不是相应生物的最佳密码子,甚至可能突变为低效密码子。这可能会影响氨基酸的编码效率,进而影响对应基因在生物中的功能发挥。本研究中关联分析发现,T24G位点不同基因型个体间的体质量性状存在显著性差异(P<0.05),GG基因型个体的平均体质量比TT基因型低9.22%,比TG基因型低11.63%,对暗纹东方鲀的生长不利,因此,可以通过筛除GG基因型提高暗纹东方鲀的生长性能。T193A位点发生于内含子,大多数SNP位点都发生在内含子,在人类基因组中内含子、外显子及毗连的非翻译区的SNP密度分别为8.21、5.288、7.51 SNP/10 kb[31]。内含子虽然不参与编码氨基酸,但可通过影响转录速率和转录稳定性增加转录水平,此外,内含子还可以提高mRNA的翻译效率[32]。已有的研究分别在大口黑鲈Micropterus salmoides[27]、红鳍东方鲀[28]、草鱼Ctenopharyngodon idella[29]等多种鱼类基因的内含子中筛选到了与生长相关的SNP位点。T193A位点未显现出明显的生长性状相关性,但单个SNP所含信息有限,不同的SNP位点间可能存在相互作用,将不同SNP位点组合成双倍型能显现更多基因频率信息[27, 33]。本研究中,将T24G和T193A的不同基因型进行组合得到7种双倍型,其中,D5型个体的体质量比D1、D2、D4、D6分别高出10.55%、16.32%、10.96%、22.55%,具有显著的生长优势。综上所述,可将T24G位点的TG和TT基因型及双倍型D5作为暗纹东方鲀辅助育种的候选分子标记。
4 结论
1) 使用反转录PCR技术克隆获得了暗纹东方鲀leptin cDNA序列长为691 bp。
2) 将暗纹东方鲀样本PCR产物测序并比对筛选得到2个分型稳定的SNP位点。
3) 通过SPSS分析,T24G位点的TG和TT基因型及双倍型D5是暗纹东方鲀优势生长的基因型,可作为辅助育种的候选分子标记。
[1] ZHANG Y Y,PROENCA R,MAFFEI M,et al.Positional cloning of the mouse obese gene and its human homologue[J].Nature,1994,372(6505):425-432.
[2] MARGETIC S,GAZZOLA C,PEGG G G,et al.Leptin:a review of its peripheral actions and interactions[J].International Journal of Obesity,2002,26(11):1407-1433.
[3] PELLEYMOUNTER M A,CULLEN M J,BAKER M B,et al.Effects of the obese gene product on body weight regulation in ob/ob mice[J].Science,1995,269(5223):540-543.
[4] CAMPFIELD L A,SMITH F J,GUISEZ Y,et al.Recombinant mouse OB protein:evidence for a peripheral signal linking adiposity and central neural networks[J].Science,1995,269(5223):546-549.
[5] SIERRA-HONIGMANN M R,NATH A K,MURAKAMI C,et al.Biological action of leptin as an angiogenic factor[J].Science,1998,281(5383):1683-1686.
[6] DUCY P,AMLING M,TAKEDA S,et al.Leptin inhibits bone formation through a hypothalamic relay[J].Cell,2000,100(2):197-207.
[7] LORD G M,MATARESE G,HOWARD J K,et al.Leptin modulates the T-cell immune response and reverses starvation-induced immunosuppression[J].Nature,1998,394(6696):897-901.
[8] BARASH I A,CHEUNG C C,WEIGLE D S,et al.Leptin is a metabolic signal to the reproductive system[J].Endocrinology,1996,137(7):3144-3147.
[9] ZHANG F M,BASINSKI M B,BEALS J M,et al.Crystal structure of the obese protein Ieptin-E100[J].Nature,1997,387(6629):206-209.
[10] KUROKAWA T,UJI S,SUZUKI T.Identification of cDNA coding for a homologue to mammalian leptin from pufferfish,Takifugu rubripes[J].Peptides,2005,26(5):745-750.
[11] 张沛,温海深,迟美丽,等.花鲈瘦素基因的克隆及低盐度条件下调控表达分析[J].大连海洋大学学报,2016,31(1):13-18.
[12] GORISSEN M,FLIK G.Leptin in teleostean fish,towards the origins of leptin physiology[J].Journal of Chemical Neuroanatomy,2014,61/62:200-206.
[13] HUISING M O,GEVEN E J W,KRUISWIJK C P,et al.Increased leptin expression in common carp (Cyprinus carpio) after food intake but not after fasting or feeding to satiation[J].Endocrinology,2006,147(12):5786-5797.
[14] RØNNESTAD I,NILSEN T O,MURASHITA K,et al.Leptin and leptin receptor genes in Atlantic salmon:cloning,phylogeny,tissue distribution and expression correlated to long-term feeding status[J].General and Comparative Endocrinology,2010,168(1):55-70.
[15] YUAN X C,LI A X,LIANG X F,et al.Leptin expression in mandarin fish Siniperca chuatsi (Basilewsky):regulation by postprandial and short-term fasting treatment[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2016,194:8-18.
[16] WON E T,BALTZEGAR D A,PICHA M E,et al.Cloning and characterization of leptin in a Perciform fish,the striped bass (Morone saxatilis):control of feeding and regulation by nutritional state[J].General and Comparative Endocrinology,2012,178(1):98-107.
[17] SALMER N C,JOHANSSON M,ASAAD M,et al.Roles of leptin and ghrelin in adipogenesis and lipid metabolism of rainbow trout adipocytes in vitro[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2015,188:40-48.
N C,JOHANSSON M,ASAAD M,et al.Roles of leptin and ghrelin in adipogenesis and lipid metabolism of rainbow trout adipocytes in vitro[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2015,188:40-48.
[18] TROMBLEY S,SCHMITZ M.Leptin in fish:possible role in sexual maturation in male Atlantic salmon[J].Fish Physiology and Biochemistry,2013,39(1):103-106.
[19] OHGA H,HIRATA D,MATSUMORI K,et al.Possible role of the leptin system in controlling puberty in the male chub mackerel,Scomber japonicus[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2017,203:159-166.
[20] OHGA H,ITO K,MATSUMORI K,et al.Leptin stimulates gonadotropin release and ovarian development in marine teleost chub mackerel[J].General and Comparative Endocrinology,2020,292:113442.
[21] MICHEL M,PAGE-MCCAW P S,CHEN W B,et al.Leptin signaling regulates glucose homeostasis,but not adipostasis,in the zebrafish[J].Proceedings of the National Academy of Sciences of the United States of America,2016,113(11):3084-3089.
[22] 成庆泰,王存信,田明诚,等.中国东方鲀属鱼类分类研究[J].动物学报,1975,21(4):359-378,398-399.
[23] KATO A,DOI H,NAKADA T,et al.Takifugu obscurus is a euryhaline fugu species very close to Takifugu rubripes and suitable for studying osmoregulation[J].BMC Physiology,2005,5:18.
[24] 张迪,张云霞,杨正勇.中国河豚鱼养殖产业发展现状研究[J].中国渔业经济,2019,37(4):87-96.
[25] YOO G Y,LEE T H,GIL H W,et al.Cytogenetic analysis of hybrids and hybrid triploids between the river puffer,Takifugu obscurus,and the tiger puffer,Takifugu rubripes[J].Aquaculture Research,2018,49(2):637-650.
[26] 卞光明,胡则辉,柴学军,等.SNP标记技术及其在水产动物遗传学中的应用[J].浙江海洋学院学报(自然科学版),2016,35(4):346-353.
[27] 李胜杰,樊佳佳,姜鹏,等.大口黑鲈HSC70-1基因多态性及其双倍型与生长性状的关联分析[J].南方水产科学,2018,14(6):74-80.
[28] WANG Y,WANG X L,MENG X Y,et al.Identification of two SNPs in myostatin (MSTN) gene of Takifugu rubripes and their association with growth traits[J].Molecular and Cellular Probes,2014,28(4):200-203.
[29] 陈静,何吉祥,樊佳佳,等.草鱼MyoD基因SNP和Indel标记的筛选及其与生长性状的关联分析[J].江苏农业学报,2018,34(3):612-616.
[30] ATHEY J,ALEXAKI A,OSIPOVA E,et al.A new and updated resource for codon usage tables[J].BMC Bioinformatics,2017,18:391.
[31] ZHAO Z M,FU Y X,HEWETT-EMMETT D,et al.Investigating single nucleotide polymorphism (SNP) density in the human genome and its implications for molecular evolution[J].Gene,2003,312:207-213.
[32] SHAUL O.How introns enhance gene expression[J].The International Journal of Biochemistry & Cell Biology,2017,91:145-155.
[33] 刘小献,白俊杰,于凌云,等.草鱼载脂蛋白A-I-1基因3′非编码区SNPs筛选及其与生长性状的关联分析[J].大连海洋大学学报,2012,27(1):12-17.