基因编辑(gene editing)技术是指在基因组水平进行基因的定点插入/缺失突变、敲除、多位点同时突变和小片段删除等精确操作技术。通过对基因编辑技术的研究,可以帮助人类探索生命本质,揭开疾病发生之谜,寻求疾病预防与治疗的有效途径[1]。基因编辑的最初技术手段为同源重组介导的基因打靶技术(gene targeting),该技术虽可以准确对特定基因进行修饰,但在实际操作中存在效率低、耗时长且可能导致基因突变等问题,影响了该技术的实际应用。近些年,基因编辑技术蓬勃发展,相继诞生了包括锌指核酸内切酶(zinc finger endonucleases, ZFN)、类转录激活因子效应物核酸酶(transcription activator-like effector nucleases, TALEN)和规律成簇短回文序列(clustered regularly interspaced short palindromic repeats, CRISPRs)等高效基因编辑工具。其中,CRISPR技术是当前应用最广泛的基因编辑技术之一。2012年至今,世界已发表数千篇关于CRISPR的文章,该技术还于2013年和2015年先后被《Science》杂志评为全球十大科技突破之一,于2020年获得诺贝尔化学奖。
海洋是人类不断探索的资源宝库,海洋中的许多生物如鱼、虾、贝、藻等经济种类更是人类餐桌上的美味佳肴,然而,随着过度捕捞、海洋环境污染等问题日益加剧,海洋生物数量不断减少,保护海洋生物的任务迫在眉睫,将传统的正向遗传学应用于海洋生物遗传育种研究更是困难重重。
作为时下生命科学领域最前沿的技术之一,基因编辑技术在生物基因功能研究、动植物病害防治及品种改良、遗传疾病基因治疗等诸多领域展现出广阔的应用前景。目前,该技术已经在许多模式生物、重要经济动植物乃至人类胚胎发育研究中取得一定突破[2-10]。2020年,基因编辑技术在淡水养殖鱼类中取得突破进展,成功培育出生长速度快、肉质质量高和规格大的新品系黄颡鱼Pelteobagrus fulvidraco等[11]。近年来,CRISPR等基因编辑技术应用于海洋生物的相关研究已有一些报道[12-13],已成功应用该技术进行遗传育种研究的物种有玻璃海鞘Ciona intestinalis[14-15]、海七鳃鳗Petromyzon marinus[16]、海葵Nematostella vectensis[17]、海胆Strongylocentrotus purpuratus[18]、三角褐指藻Phaeodactylum ricornutum[19]和大西洋鲑Salmo salar[20-21]等。本文主要综述了基因编辑技术的发展建立、作用机制及其应用现状,并结合海洋生物基因功能研究领域的研究进展,探讨了基因编辑技术在海洋生物资源保护与开发、遗传育种等领域的广泛应用及发展前景。
1 ZFN和TALEN技术
1.1 ZFN
基因编辑本质上是利用限制性核酸内切酶对生物遗传序列进行修饰,并产生稳定的突变型。这里的修饰包括插入/缺失突变、敲除、多位点同时突变和小片段删除等。然而,由于天然核酸内切酶的种类有限,科学家们迫切希望找到新的基因编辑方法。ZFN是由Kim等[22]成功构建的第一种人工核酸酶,目前,该技术已在多种模式生物中实现了靶基因的敲除或定点修饰[23-26],并于2005年实现了人类细胞的基因敲除[27]。由于ZFN对于其作用的靶点DNA序列无特异的对应性,该技术需要通过构建庞大的锌指表达文库来筛选出能够特异识别这些靶点的锌指蛋白。此外,由于ZFN序列识别的特殊性,目前仍无法设计出满足任意一段序列要求的ZFN,也无法确保每个基因都能够找到适合的ZFN作用靶点[28]。
1.2 TALEN
Moscou等[29]首次提出了一种类转录激活因子效应蛋白(TALE),该蛋白具有特异结合宿主基因的功能[30]。此后,以TALE为DNA结合域构建的TALENs技术在多个物种的基因修饰研究中均得到了成功应用,该技术被《Science》杂志评为2012年度十大科学进展之一[31]。TALENs技术的作用机制与ZFNs技术类似,其核心是识别特异DNA序列的TALEs蛋白和核酸内切酶FokI。TALEs蛋白是植物病原黄单胞杆菌Xanthomonas campestris进入植物细胞改变基因序列后转录翻译分泌的一类类似于真核生物转录因子的TALE蛋白家族,该家族首次发现的为AvrBs3[32],由12个或以上特异性识别DNA序列的串联“蛋白模块”和N′端、C′端两侧的序列组成。每个“蛋白模块”由34个氨基酸组成,其中,第12和第13位点是靶向识别的关键位置,即重复可变的双连氨基酸(repeat variant diresidue,RVD)位点。TALEs蛋白上的每个RVDs只能识别1个碱基(A、G、C、T),即NI识别A,NG识别T,NN识别G或者A,HD识别C[33-35] 。此外,在构建模块时,研究人员发现,NK识别G的特异性比NN要好,但由于NK活性较低,更倾向于使用NN[29,36]。由于不同物种基因组大小不同,选择的特异序列长度也不同,对于哺乳动物的基因修饰,识别靶点一般选取16~20 bp的DNA序列。
TALEs蛋白是识别靶基因,而FokI的作用则是切割DNA。FokI是一种核酸内切酶,必须形成二聚体才具有活性,所以大大减少了随机酶切的概率。TALEs蛋白和FokI连接成重组蛋白表达1个重组核酸酶,识别靶点核酸序列,发挥内切酶的活性,从而切断目的基因,实现基因敲除。然而这些方法在操作上技术难度大、耗时费力、费用昂贵且效率并不高[37-38]。
2 CRISPR编辑技术
CRISPR是细菌和古细菌自身的一种获得性免疫系统,通过特异性RNA介导相应的内源蛋白对侵入DNA进行特异性切割降解。1987年,日本学者Ishino等[39]在对大肠杆菌Escherichia coli K12克隆时发现了一系列相互间隔的短回文序列,并于2002年被正式命名为规律成簇短回文序列。CRISPR系统主要包括一系列Cas蛋白(Cas1、Cas2、Cas4和效应蛋白(如Cas9、Cpf1等)的编码基因和一段成簇的短回文序列组成,CRISPR系统因组分不同可以分成2类5型共16种亚型[40-41]。1类包括Ⅰ、Ⅲ和Ⅳ型,2类包括Ⅱ型和Ⅴ型。前3种发挥功能时需要利用多个效应蛋白复合物干扰靶基因,而另两种则只需单一的效应蛋白干扰靶基因即可发挥作用。
2.1 CRISPR/Cas
目前,研究最深入的CRISPR系统为CRISPR/Cas9(CRISPR associated gene 9)。CRISPR/Cas系统是一种RNA介导的适应性免疫系统,存在于大约48%的细菌和95%的古细菌中,可提供序列特异性保护以抵抗外来的DNA甚至RNA。2012年,Jinek等[42]将CRISPR/Cas9系统中的crRNA和tracrRNA改造成一条单导向RNA(single-guide RNA, sgRNA),它能够指导Cas9蛋白对指定DNA序列进行靶向断裂,这为CRISPR/Cas9系统的进一步应用奠定了基础。2013年,麻省理工学院Mail等[43]和哈佛大学Li等[44]首次实现了CRISPR技术在真核生物基因定点突变领域的突破,由此开始将CRISPR技术应用到基因编辑中。同年,来自不同地区的研究团队先后证实了CRISPR/Cas9系统在哺乳动物中的可行性和初步应用研究情况[44-45]。由于CRISPR/Cas9系统只需对sgRNA进行设计,因此,具有操作简单和节省时间等优点。
2.2 CRISPR-Cpf1
2015年,随着基因编辑技术的发展进步,华裔科学家张锋带领的小组发现了新一代基因编辑方法,这种新方法与CRISPR/Cas9较为相似,被称为CRISPR-Cpf1技术,这是一种更为高效的CRISPR系统[46]。与Cas9相比,Cpf1蛋白更加简洁且易于操作,这种蛋白仅需要一段crRNA即可构成切割复合体,进而完成对目的DNA片段的切割。这种新的蛋白切割后产生的末端为黏性末端,有利于目的基因以非同源重组的方式插入靶向位点,在克服了Cas9蛋白的缺陷后,CRISPR-Cpf1系统成为目前最有效的基因编辑方法。作为全新的基因编辑方法,CRISPR-Cpf1系统中的Cpf1蛋白主要有3个类别:氨基酸球菌 Acidaminococcus sp.BV3L6、土拉热弗朗西斯菌 Francisella novicida U112和毛螺科菌Lachnospiraceae bacterium ND2006,分别简写为 AsCpf1、FnCpf1和 LbCpf1。
CRISPR-Cpf1系统与CRISPR/Cas9系统的作用原理类似,Cas9系统集中在提高其特异性上,主要改造针对Cas9自身,诸如改变Cas9蛋白的结构,给其融合一个DNA结合域或者FokI核酶等;相比Cas9,Cpf1有许多超过Cas9的优点,如序列短,可用于包装腺病毒等,但对其研究相对较少,Moon等[47]提出,crRNA识别20个靶DNA且3′端加几个U可以提高Cpf1系统的在靶率,这为Cpf1提供了新的发展方向。
目前,随着基因编辑技术的不断发展,新的应用手段也在不断开发,如Co-CRISPR等技术的出现,为基因编辑技术的开发与应用展现了更广阔的前景。
3 基因编辑技术在海洋生物遗传育种中的应用
海洋生物能够为人类提供丰富的优质蛋白,开展海洋生物尤其是海水养殖品种的基因功能研究,通过揭示其遗传生长规律,以及培育高产、抗病和抗逆品种,不仅具有重要的科学意义,还可有效提高水产养殖业的经济效益。近年来, 随着全基因组测序工作成本的降低,水产生物后基因组时代到来,鱼、虾、蟹、贝、藻等诸多水产经济品种的基因组已被测序,为基因编辑在水产养殖物种中的应用提供了丰富的遗传信息资源。 CRISPR技术在海洋生物中的应用也从模式生物开始,该技术最早在斑马鱼Danio rerio、玻璃海鞘等模式动物中实现。与其他技术相比,CRISPR技术在模式生物中普及后开始迅速应用于非模式生物中。这从一方面反映了此项技术的普遍适用性和高效性,从另一方面也为海洋生物的研究带来了前所未有的机遇。新型基因编辑技术不仅可以敲除基因,还可以定向编辑目的基因,能够显著缩短育种时间,与传统的转基因技术相比也更加安全。CRISPR技术在不同水生生物类群中的研究进展参见表1。
3.1 鱼类
相对于其他物种,基因编辑技术在鱼类中的研究较为广泛, 该基因编辑技术迄今已在斑马鱼、青鳉Oryzias latipes等模式鱼类和其他水产经济鱼类遗传育种中建立了不少应用。 相对于小鼠等模式生物,斑马鱼的胚胎在显微镜下观察是透明的,可以看到各个器官和血管的发育。斑马鱼体外受精,繁殖周期短并与人类的器官高度相似,因此,该鱼已经成为生物医学研究的一个重要模型。
2013年,利用CRISPR技术实现了对斑马鱼fh基因的敲除并获得了成功[48]。研究证明,CRISPR/Cas9系统在斑马鱼体内可诱导胚胎中的靶向基因修饰,其效率与ZFN和TALEN的作用相似。2015年,研究者利用CRISPR技术成功实现了对海七鳃鳗中与人类同源的tyr基因的敲除,结果显示,海七鳃鳗完全适应CRISPR技术且提高注射量能够成倍地提高其突变量[16]。2017年,研究者利用CRISPR技术对斑马鱼的atplala.1、atplala.2、atplala.3、atplala.4、atplala.5、pkd2、aqp3a、bmper、s12a、tbx2a等10个前肾表达基因进行敲除,经过三代的筛选发现只有atplala.2和pkd2能够使斑马鱼胚胎发育缺陷[49]。同时该研究敲除了斑马鱼faf1基因,faf1基因参与早期胚胎的神经嵴细胞迁移分化,通过显微注射敲除该基因后斑马鱼的色素沉积延迟及尾部肌节部出现“结节样”变化[50]。
2019年,研究者成功通过CRISPR技术建立了青鳉foxl2基因缺失的突变体,foxl2突变体有遗传雌性、生理雄性的表型特征,结果表明,foxl2对维持青鳉性腺功能具有重要作用[51]。Nanog基因作为诱导多能性干细胞关键因子之一,对胚胎早期发育和维持胚胎干细胞全能性具有重要作用。CRISPR/Cas9系统可以高效率敲除青鳉tyr基因和nanog基因,且通过同源重组机制也可介导青鳉nanog基因敲入[52]。该技术还可以敲除青鳉的SLC45a2基因,产生的突变体显示出白化病表型[53]。
在研究大西洋鲑身体着色的关键基因slc45a2和tyr过程中,首次利用CRISPR技术对海洋冷水物种进行了敲除[20]。在随后的研究中,研究者利用Co-CRISPR技术同时靶定着色基因与一些不相干的基因,使得缺失着色基因的大西洋鲑的突变体更加容易被识别,而且第二个被靶定的基因拥有较高的突变率[21]。
除此之外,CRISPR技术在牙鲆Paralichthy olivaceus生长发育方面的应用也取得了一定进展,研究者采用CRISPR技术对牙鲆肌生成抑制素(MSTN)进行干扰,将靶向MSTN基因第一个外显子的Cas9 mRNA和sgRNAs共注射到牙鲆胚胎中,通过筛选,在F1代中得到了具有MSTN干扰的杂合双等位基因突变体,表现为身体增厚,肥满度增加[54]。
表1 CRISPR技术在海洋生物遗传育种中的应用
Tab.1 Application of CRISPR technology in genetic breeding of different marine organisms
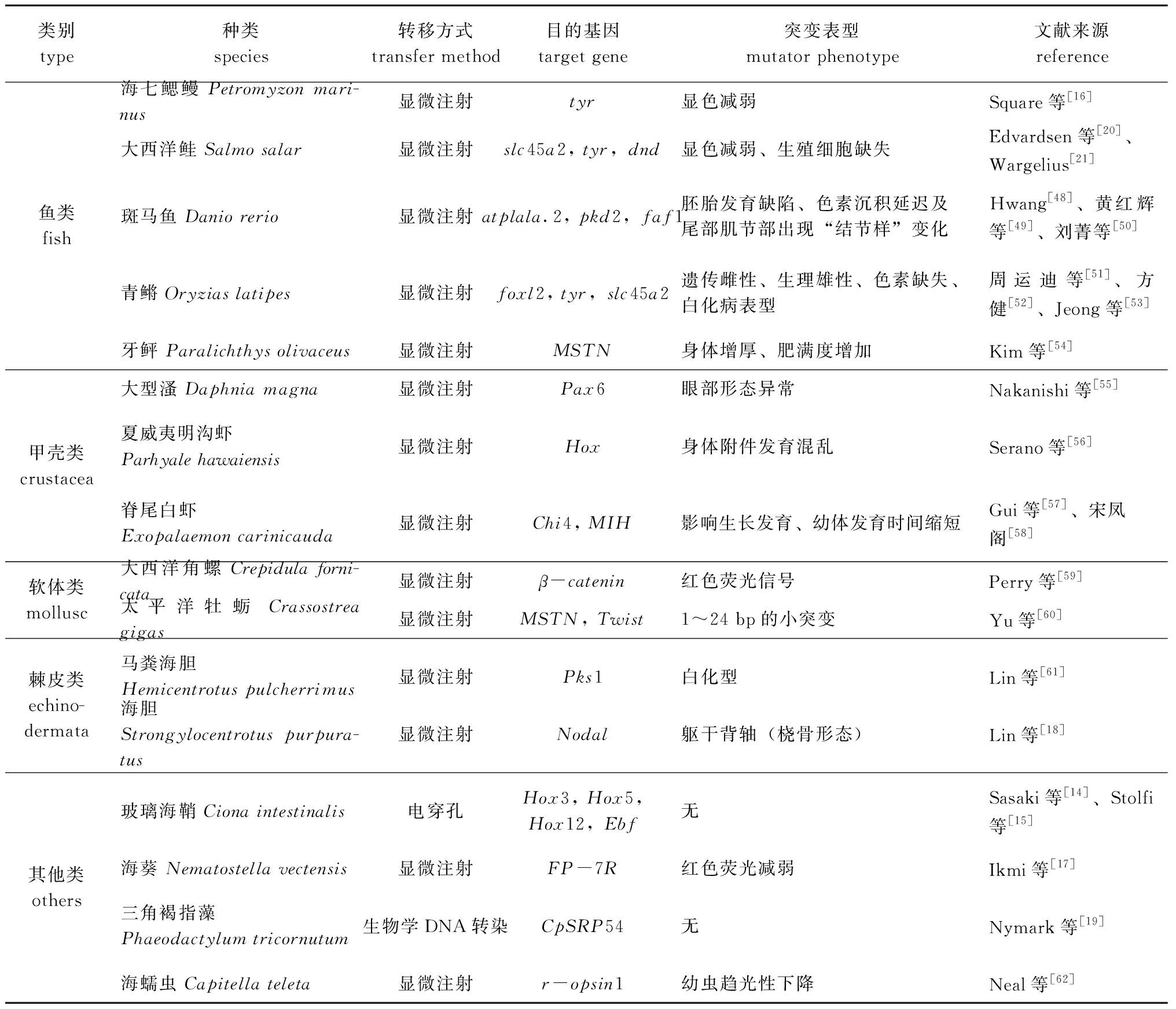
类别type种类species转移方式transfer method目的基因target gene突变表型mutator phenotype文献来源reference鱼类fish海七鳃鳗 Petromyzon mari-nus显微注射tyr显色减弱Square等[16]大西洋鲑 Salmo salar显微注射slc45a2, tyr,dnd显色减弱、生殖细胞缺失Edvardsen等[20]、Wargelius[21]斑马鱼 Danio rerio显微注射atplala.2, pkd2,faf1胚胎发育缺陷、色素沉积延迟及尾部肌节部出现“结节样”变化Hwang[48]、黄红辉等[49]、刘菁等[50]青鳉 Oryzias latipes显微注射foxl2, tyr,slc45a2遗传雌性、生理雄性、色素缺失、白化病表型周运迪等[51]、方健[52]、Jeong等[53]牙鲆 Paralichthys olivaceus显微注射MSTN身体增厚、肥满度增加Kim等[54]甲壳类crustacea大型溞 Daphnia magna显微注射Pax6眼部形态异常Nakanishi等[55]夏威夷明沟虾Parhyale hawaiensis显微注射Hox身体附件发育混乱Serano等[56]脊尾白虾Exopalaemon carinicauda显微注射Chi4, MIH影响生长发育、幼体发育时间缩短Gui等[57]、宋凤阁[58]软体类mollusc大西洋角螺 Crepidula forni-cata显微注射β-catenin红色荧光信号Perry等[59]太平洋牡蛎 Crassostrea gigas显微注射MSTN, Twist1~24 bp的小突变Yu等[60]棘皮类echino-dermata马粪海胆 Hemicentrotus pulcherrimus显微注射Pks1白化型Lin等[61]海胆 Strongylocentrotus purpura-tus显微注射Nodal躯干背轴(桡骨形态)Lin等[18]其他类others玻璃海鞘 Ciona intestinalis电穿孔Hox3, Hox5, Hox12,Ebf无Sasaki等[14]、Stolfi等[15]海葵 Nematostella vectensis显微注射FP-7R红色荧光减弱Ikmi等[17]三角褐指藻Phaeodactylum tricornutum生物学DNA转染CpSRP54无Nymark等[19]海蠕虫 Capitella teleta显微注射r-opsin1幼虫趋光性下降Neal等[62]
3.2 甲壳动物
目前,甲壳类动物中CRISPR技术的相关研究主要涵盖虾类和大型溞Daphnia magna。2014年,Nakanishi等[55]利用CRISPR技术将遗传突变引入到大型溞的内源盲基因中,这是该项技术在甲壳动物中的首次应用。研究者失活了大型溞的pax6基因,证明了该基因在眼发育中的关键作用。除此之外,研究者还将基因编辑后的个体继续培养至性成熟,并对获得的后代进行检测,发现这些编辑过的基因组能够顺利遗传给后代。
2016年,研究者利用CRISPR技术在夏威夷明沟虾Parhyale hawaiensis中成功实现了对Hox基因的定点敲除,结果表明,特定的Hox基因直接决定了甲壳动物头胸腹部不同形态的附肢形态[56]。这也使得CRISPR技术在经济甲壳动物(如虾蟹类)中的应用探索也有了突破。同年,研究者利用CRISPR技术成功实现了经济虾类脊尾白虾Exopalaemon carinicauda中几丁质酶基因Chi4的敲除,同时观察到Chi4敲除对脊尾白虾生长发育的影响,若对更多的基因进行改造,可能会获得更高产率的新品种[57]。在此基础上,通过共注射体外转录的Cas9mRNA和gRNA的方法敲除脊尾白虾的MIH基因,能够获得幼体发育时间缩短的个体[58]。
3.3 软体动物
CRISPR技术在软体动物中的研究相对较少。2015年,研究者利用CRISPR技术在大西洋角螺Crepidula fornicata幼虫中检测到了具有红色荧光信号的β-catenin蛋白[59]。2019年,一个基于核糖核酸蛋白复合物的太平洋牡蛎Crassostrea gigas基因编辑系统被开发出来[60]。在靶基因中,肌生成抑制蛋白(MSTN)和Twist被选为靶点,CRISPR诱导的突变主要是1~24 bp的小突变,这种方法为牡蛎和其他海洋双壳类的基因功能提供了强有力的工具,并有可能作为一种新的基因工程技术用于养殖并改善牡蛎的特性。
3.4 棘皮动物
在海洋棘皮动物的CRISPR技术相关研究方面,多个学者都针对海胆的基因进行了编辑。2016年,研究者利用CRISPR技术将靶向Nodal基因的sgRNA和编码Cas9蛋白的mRNA注射到海胆的受精卵中, 成功敲除了海胆的Nodal基因, 在全部6种gRNA中有5种出现了预期的敲除效果[18]。同年,研究者通过将目标对准一个非必需基因简化gRNA的构建步骤,该基因能快速评估胚胎中成功的CRISPR功能,从而能够简单目测出功能基因的破坏[63]。2019年,Lin等[61]详细介绍了CRISPR技术在海胆胚胎中的应用方法,包括gRNA设计、体外合成单导向RNA (sgRNA)、CRISPR技术在基因敲除和单核苷酸编辑中的应用,以及用于评价基因编辑效率的合成胚胎的基因分型方法。2019年,研究者将海胆中的Pks1基因成功敲除,结果表明,在海胆变态后的幼体中仍然保留了白化型,能够存活至少一年并发育为白化的成年海胆[64]。
3.5 其他
2014年,研究者开始使用CRISPR技术实现对玻璃海鞘基因的定点敲除[14],为了敲除玻璃海鞘中的Hox基因,研究人员选取了Hox3、Hox5、Hox12 3个等位基因共计8个靶点,结果发现,Hox3-sg3、Hox5-sg1能使受体目的基因产生突变,其余并未出现预期效果。进一步研究发现,导入的sgRNA表达框架质量必须大于1.5 pg,表达Cas9蛋白mRNA的质量必须大于15 pg,才能使受精卵发生突变,且突变率与使用量成正比。另一个研究选取了玻璃海鞘发育过程中非常重要的ebf基因作为靶向基因,并以其为基础构建了2个gRNA表达载体,将靶向ebf基因的gRNA与编码Cas9蛋白的mRNA以电穿孔的方式导入受精卵中,取得了较高的突变效率[15]。通过改造载体上的部分序列,试验还达到了帮助细胞中质粒转录的效果。
2014年,研究者分别利用CRISPR和TALEN技术对海葵Nematostella vectensis的一种内源的红色荧光蛋白(FP-7R)进行了敲除,被敲除基因的海葵不能发出红色荧光,该研究还证明了FP-7R并不是海葵发育所必需的[17]。2016年,CRISPR技术的应用领域拓展到了海洋藻类中,三角褐指藻Phaeodactylum tricornutum中的CpSRP54基因被成功敲除[19],该研究也证实,CRISPR技术可以在微藻中有效地产生稳定的靶向基因突变。2019年,研究者通过CRISPR/CAS9诱变基因编辑对海蠕虫Capitella teleta横纹肌视蛋白基因r-opsin1进行了破坏,虽然突变个体眼斑感光细胞和相关色素细胞正常形成并持续存在,但幼虫的趋光性显著下降[62]。
4 存在问题及展望
近年来,CRISPR系统和相关蛋白的研究不断地完善和优化,为整个生物技术领域提供了无限可能。相对于ZFN和TALEN这两种基因编辑技术,CRISPR/Cas作为第三代人工核酸酶成为目前研究的热点,在技术门槛、应用便捷程度、成本等关键方面具有显著的优势。然而,从目前的报道来看,CRISPR技术在研究与应用过程中仍存在诸多问题:
1) 应用受限:基因编辑技术主要是通过显微注射等方法对细胞系或胚胎进行操作,而海洋生物细胞系较少,一定程度上限制了基因编辑技术的应用,目前的研究只限于模式生物和少数海洋生物,广泛应用任重而道远。
2) 编辑效率和精确度不高:研究及应用中可能产生脱靶问题,引起基因组的重排或无法预测的突变,已有报道改良后的基因编辑技术在降低脱靶率和增强特异性等方面均取得了显著进展[65-66]。
3)公众认知度不高:多数公众对于CRISPR技术的发展、研究及应用的了解不多,对于基因编辑的水产品安全性存在一定的疑虑。
随着海洋生物基因组研究的快速发展,CRISPR技术也为海洋生物,特别是水产养殖领域的研究带来了前所未有的机遇。CRISPR技术通过高效、定向地编辑目的基因,进行基因的切除与导入,未来将对海洋生物的遗传育种产生深远影响,建议今后应在以下几方面开展更多研究工作:
1)扩大研究和应用范围:相对于其他物种,CRISPR技术在海洋生物领域的研究较少且多集中在鱼类,未来应在更多海洋经济品种中更深入和广泛地展开遗传育种的研究与应用。
2)提高基因编辑的效率和精确度:通过对基因更精准和高效的敲入/敲除或修饰,提升水产苗种的繁育效率和繁殖力、提高生长速度、增强抗逆性与抗病性,获得具有优良性状的新品种,实现经济与社会效益的双赢。
3)深入解析海洋生物基因功能:随着更多海洋生物基因组测序的完成,基因编辑技术将进一步解读不同基因在海洋生物遗传育种乃至生物进化发育中的作用。
4)提高透明度和公众认知度:一项技术的成功应用,推广也是其中重要一环,研究者应该让基因编辑技术的使用更透明,而管理者则应该不断完善政策和监管,从而减少公众的质疑,获得消费者的认可,使该技术理性健康发展,更好地服务于大众。
[1] CAPECCHI M R.Gene targeting in mice:functional analysis of the mammalian genome for the twenty-first century[J].Nature Reviews Genetics,2005,6(6):507-512.
[2] CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013, 339(6121):819-823.
[3] NAKAYAMA T,FISH M B,FISHER M,et al.Simple and efficient CRISPR/Cas9-mediated targeted mutagenesis in Xenopus tropicalis[J].Genesis,2013,51(12):835-843.
[4] 郭丹丹,郑国栋,陈杰,等.利用CRISPR/Cas9基因编辑技术探究团头鲂mdh基因功能[J/OL].水产学报,2020:1-9.(2020-04-03)[2020-12-29].http://kns.cnki.net/kcms/detail/31.1283.s.20200403.0925.002.html.
[5] 石红娟,陈立黎,蒋小龙,等.foxj1a和foxh1对尼罗罗非鱼生殖细胞发育功能的研究[C]//中国海洋湖沼学会、中国动物学会鱼类学分会第九届会员代表大会暨2014年学术研讨会论文集.天津:中国海洋湖沼学会,中国动物学会,2014.
[6] WOLTER F,PUCHTA H.The CRISPR/Cas revolution reaches the RNA world:Cas13,a new Swiss Army knife for plant biologists[J].The Plant Journal,2018,94(5):767-775.
[7] 傅俊豪,杨发誉,谢海华,等. 细菌中CRISPR/Cas系统的应用和优化[J]. 生物工程学报,2019,35(3):341-350.
[8] GERASHCHENKOV G A,ROZHNOVA N A,KULUEV B R,et al.Design of guide RNA for CRISPR/Cas plant genome editing[J].Molekuliarnaia Biologiia,2020,54(1):29-50.
[9] ARALDI R P,KHALIL C,GRIGNET P H,et al.Medical applications of Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR/Cas) tool:a comprehensive overview[J].Gene,2020,745:144636.
[10] WELTNER J,BALBOA D,KATAYAMA S,et al.Human pluripotent reprogramming with CRISPR activators[J].Nature Communications,2018,9(1):2643.
[11] 张晔,徐承旭.基因编辑技术“剪”出肉多个大的黄颡鱼[J]. 水产科技情报,2020,324(3):58-59.
[12] 李响,董波.CRISPR/Cas9技术及其在海洋生物中的应用现状与展望[J].水生生物学报,2017,41(1):244-256.
[13] 马杭柯,孙金秋,徐莞媛.CRISPR基因编辑技术研究进展及其在水生生物中的应用[J].海洋渔业,2018,40(5):632-640.
[14] SASAKI H,YOSHIDA K,HOZUMI A,et al.CRISPR/Cas9-mediated gene knockout in the ascidian Ciona intestinalis[J].Development,Growth & Differentiation,2014,56(7):499-510.
[15] STOLFI A,GANDHI S,SALEK F,et al.Tissue-specific genome editing in Ciona embryos by CRISPR/Cas9[J]. Development,2014,141(21):4115-4120.
[16] SQUARE T,ROM
 EK M,JANDZIK D,et al.CRISPR/Cas9-mediated mutagenesis in the sea lamprey Petromyzon marinus:a powerful tool for understanding ancestral gene functions in vertebrates[J].Development, 2015,142(23):4180-4187.
EK M,JANDZIK D,et al.CRISPR/Cas9-mediated mutagenesis in the sea lamprey Petromyzon marinus:a powerful tool for understanding ancestral gene functions in vertebrates[J].Development, 2015,142(23):4180-4187.
[17] IKMI A,MCKINNEY S A,DELVENTHAL K M,et al.TALEN and CRISPR/Cas9-mediated genome editing in the early-branching metazoan Nematostella vectensis[J].Nature Communications,2014,5:5486.
[18] LIN C Y,SU Y H.Genome editing in sea urchin embryos by using a CRISPR/Cas9 system[J].Developmental Biology,2016,409(2):420-428.
[19] NYMARK M,SHARMA A K,SPARSTAD T,et al.A CRISPR/Cas9 system adapted for gene editing in marine algae[J].Scientific Reports,2016,6(1):24951.
[20] EDVARDSEN R B,LEININGER S,KLEPPE L,et al.Targeted mutagenesis in Atlantic salmon (Salmo salar L.) using the CRISPR/Cas9 system induces complete knockout individuals in the F0 generation[J].PLoS One,2014,9(9):e108622.
[21] WARGELIUS A,LEININGER S,SKAFTNESMO K O,et al.Dnd knockout ablates germ cells and demonstrates germ cell independent sex differentiation in Atlantic salmon[J].Scientific Reports,2016,6(1):21284.
[22] KIM Y G,CHA J,CHANDRASEGARAN S.Hybrid restriction enzymes:zinc finger fusions to Fok I cleavage domain[J].Proceedings of the National Academy of Sciences of the United States of America,1996,93(3):1156-1160.
[23] BEUMER K J,TRAUTMAN J K,BOZAS A,et al.Efficient gene targeting in Drosophila by direct embryo injection with zinc-finger nucleases[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(50):19821-19826.
[24] MENG X D,NOYES M B,ZHU L J,et al.Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases[J].Nature Biotechnology,2008,26(6):695-701.
[25] DOYON Y,MCCAMMON J M,MILLER J C,et al.Heritable targeted gene disruption in zebrafish using designed zinc-finger nucleases[J].Nature Biotechnology,2008,26(6):702-708.
[26] CUI X X,JI D A,FISHER D A,et al.Targeted integration in rat and mouse embryos with zinc-finger nucleases[J].Nature Biotechnology,2011,29(1):64-67.
[27] URNOV F D,MILLER J C,LEE Y L,et al.Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J].Nature,2005,435(7042):646-651.
[28] DEFRANCESCO L.Erratum:move over ZFNs[J].Nature Biotechnology,2012,30(1):112.
[29] MOSCOU M J,BOGDANOVE A J.A simple cipher governs DNA recognition by TAL effectors[J].Science,2009,326(5959):1501.
[30] BOCH J,BONAS U.Xanthomonas AvrBs3 family-type III effectors:discovery and function[J].Annual Review of Phytopathology,2010,48:419-436.
[31] ALBERTS B.The breakthroughs of 2012[J].Science,2012,338(6114):1511.
[32] BONAS U,STALL R E,STASKAWICZ B.Genetic and structural characterization of the avirulence gene avrBs3 from Xanthomonas campestris pv. vesicatoria[J].Molecular and General Genetics MGG,1989,218(1):127-136.
[33] BOCH J,SCHOLZE H,SCHORNACK S,et al.Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors[J].Science,2009,326(5959):1509-1512.
[34] MILLER J C,TAN S Y,QIAO G J,et al.A TALE nuclease architecture for efficient genome editing[J].Nature Biotechnology,2011,29(2):143-148.
[35] CHRISTIAN M,CERMAK T,DOYLE E L,et al.Targeting DNA double-strand breaks with TAL effector nucleases[J].Genetics,2010,186(2):757-761.
[36] STREUBEL J,BLUCHER C,LANDGRAF A,et al.TAL effector RVD specificities and efficiencies[J].Nature Biotechnology,2012,30(7):593-595.
[37] CARROLL D.Genome engineering with targetable nucleases[J].Annual Review of Biochemistry,2014,83:409-439.
[38] YU Y J,BRADLEY A.Engineering chromosomal rearrangements in mice[J].Nature Reviews Genetics,2001,2(10):780-790.
[39] ISHINO Y,SHINAGAWA H,MAKINO K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion in Escherichia coli,and identification of the gene product[J].Journal of Bacteriology,1987,169(12):5429-5433.
[40] MAKAROVA K S,WOLF Y I,ALKHNBASHI O S,et al.An updated evolutionary classification of CRISPR-Cas systems[J].Nature Reviews Microbiology,2015,13(11):722-736.
[41] MAKAROVA K S,HAFT D H,BARRANGOU R,et al.Evolution and classification of the CRISPR-Cas systems[J].Nature Reviews Microbiology,2011,9(6):467-477.
[42] JINEK M,CHYLINSKI K,FONFARA I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science,2012,337(6096):816-821.
[43] MALI P,YANG LUHAN,ESVELT K M,et al.RNA-guided human genome engineering via Cas9[J].Science,2013,339(6121):823-826.
[44] CHO S W,KIM S,KIM J M,et al.Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease[J].Nat Biotechnol,2013,31(3):230-232.
[45] JINEK M,EAST A,CHENG A,et al.RNA-programmed genome editing in human cells[J].eLife,2013,2:e00471.
[46] ZETSCHE B,HEIDENREICH M,MOHANRAJU P,et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array[J].Nature Biotechnology,2016,35(1):31-34.
[47] MOON S B,LEE J M,KANG J G,et al.Highly efficient genome editing by CRISPR-Cpf1 using CRISPR RNA with a uridinylate-rich 3’-overhang[J].Nature Communications,2018,9(1):3651.
[48] HWANG W Y,FU Y F,REYON D,et al.Efficient genome editing in zebrafish using a CRISPR-Cas system[J].Nature Biotechnology,2013,31(3):227-229.
[49] 黄红辉,朱世诚,董国萍,等.运用CRISPR/Cas9技术对斑马鱼前肾表达基因的敲除研究[J].西南大学学报(自然科学版),2017,39(5):54-61.
[50] 刘菁,梁森,袁志,等.CRISPR/Cas9技术敲除faf1基因对斑马鱼软骨及肌节发育的影响[J].第三军医大学学报,2017,39(17):1709-1714.
[51] 周运迪,吴星星,赵海萍,等.青鳉foxl2基因敲除突变体的构建与表型分析[J].广东海洋大学学报,2019,39(2):20-30.
[52] 方健.利用CRISPR/Cas9技术编辑青鳉tyr和nanog基因[D].武汉:华中农业大学,2018.
[53] JEONG C B,KANG H M,HONG S A,et al.Generation of albino via SLC45a2 gene targeting by CRISPR/Cas9 in the marine medaka Oryzias melastigma[J].Marine Pollution Bulletin,2020,154:111038.
[54] KIM J,CHO J Y,KIM J W,et al.CRISPR/Cas9-mediated myostatin disruption enhances muscle mass in the olive flounder Paralichthys olivaceus[J].Aquaculture,2019,512:734336.
[55] NAKANISHI T,KATO Y,MATSUURA T,et al.CRISPR/Cas-mediated targeted mutagenesis in Daphnia magna[J].PLoS One,2014,9(5):e98363.
[56] SERANO J M,MARTIN A,LIUBICICH D M,et al.Comprehensive analysis of Hox gene expression in the amphipod crustacean Parhyale hawaiensis[J].Developmental Biology,2016,409(1):297-309.
[57] GUI T S,ZHANG J Q,SONG F G,et al.CRISPR/Cas9-mediated genome editing and mutagenesis of EcChi4 in Exopalaemon carinicauda[J].G3:Genes,Genomes,Genetics,2016,6(11):3757-3764.
[58] 宋凤阁.CRISPR/Cas9技术对脊尾白虾蜕皮抑制激素基因的靶向敲除研究[D].北京:中国科学院大学(中国科学院海洋研究所),2017.
[59] PERRY K J,HENRY J Q.CRISPR/Cas9-mediated genome modification in the mollusc,Crepidula fornicata[J].Genesis,2015,53(2):237-244.
[60] YU H,LI H J,LI Q,et al.Targeted gene disruption in Pacific oyster based on CRISPR/Cas9 ribonucleoprotein complexes[J].Marine Biotechnology,2019,21(3):301-309.
[61] LIN C Y,OULHEN N,WESSEL G M,et al.CRISPR/Cas9-mediated genome editing in sea urchins[J].Methods in Cell Biology,2019,151:305-321.
[62] NEAL S,DE JONG D M,SEAVER E C.CRISPR/CAS9 mutagenesis of a single r-opsin gene blocks phototaxis in a marine larva[J].Proceedings of the Royal Society B:Biological Sciences,2019,286(1904):20182491.
[63] OULHEN N,WESSEL G M.Albinism as a visual,in vivo guide for CRISPR/Cas9 functionality in the sea urchin embryo[J].Molecular Reproduction and Development,2016,83(12):1046-1047.
[64] LIU D M,AWAZU A,SAKUMA T,et al.Establishment of knockout adult sea urchins by using a CRISPR-Cas9 system[J].Development,Growth & Differentiation,2019,61(6):378-388.
[65] MANGHWAR H,LI B,DING X,et al.CRISPR/Cas systems in genome editing:methodologies and tools for sgRNA design,off-target evaluation,and strategies to mitigate off-target effects[J].Advanced Science,2020,7(6):1902312.
[66] DOMAN J L,RAGURAM A,NEWBY G A,et al.Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors[J].Nature Biotechnology,2020,38(5):620-628.