菲律宾蛤仔Ruditapes philippinarum隶属于软体动物门Mollusca双壳纲Veneroida帘蛤科Veneridae蛤仔属Ruditapes,俗称蛤蜊、蚬子、花蛤等[1],是中国四大养殖贝类之一,中国年产量约300万t,占世界总产量的90%以上,市场潜力巨大。目前,中国菲律宾蛤仔种质资源遗传基础不清、北黄海等滩涂土著地方种质丢失、人工培育的良种覆盖率低等问题制约了产业发展。理清中国菲律宾蛤仔野生群体及潜在可作修复用途的日本、朝鲜群体的种质遗传基础,开展蛤仔种质资源保护和可持续利用,可成为解决目前所存问题的主要途径。
动物线粒体DNA 由于具有分子量小、进化速度快、结构简单、母性遗传等特点[2],已经被广泛应用于水产动物的种质遗传研究中[3]。而16S rRNA 是线粒体基因组中既含有高度保守和中度保守的序列区域,又含有高度变化序列区域的一段序列,因此,被认为是科级以下水平物种间亲缘关系和系统演化研究的有效标记[4] 。目前,利用线粒体DNA基因序列进行菲律宾蛤仔遗传多样性的研究已有较多报道,胡利莎等[5]研究了10个菲律宾蛤仔野生群体的遗传多样性,结果表明,中国沿海的菲律宾蛤仔野生群体存在一定的遗传分化,辽宁大连群体和山东荣成群体聚为一支,其余8个群体聚为一支。刘相全等[6]对菲律宾蛤仔和杂色蛤仔Ruditapes variegata 的线粒体16S rRNA 基因序列进行了分析,结果表明,两种菲律宾蛤仔的亲缘关系比较接近, 且与同为帘蛤科的硬壳蛤Mercenaria mercenaria有明显的差异。在国外,Cordero等[7]结合线粒体细胞色素C氧化酶(COI)基因序列及SSR(简单重复序列)遗传标记对1936年由日本引种至北美西海岸的菲律宾蛤仔群体的种质遗传基础进行了评估。这些研究均为菲律宾蛤仔的种质资源保护提供了参考。
目前,关于中国菲律宾蛤仔野生苗种产区的广西北海、山东莱州、辽宁营口、天津群体,以及日本北海道、朝鲜新义州海域潜在可作为北黄海土著地方品种资源修复用途的群体的种质资源状况尚无报道。本研究中,利用16S rRNA序列多态性分析了中国4个野生苗种产区群体及日本北海道、朝鲜新义州群体的菲律宾蛤仔遗传多样性及遗传结构[8],以期为菲律宾蛤仔种质资源保护及可持续利用提供科学依据。
1 材料与方法
1.1 样本采集
菲律宾蛤仔样本(表1)分别采自中国辽宁营口(YK)、天津(TJ)、山东莱州(LZ)、广西北海(BH)、日本北海道(JH)和朝鲜新义州(NKS),每个地区收集10个个体,在实验室进行解剖,取其足和闭壳肌,使用体积分数为90%的乙醇固定,保存于冰箱(-20 ℃)中。
表1 菲律宾蛤仔6个群体的样本信息
Tab.1 Sample informations on 6 populations of Manila clam Ruditapes philippinarum
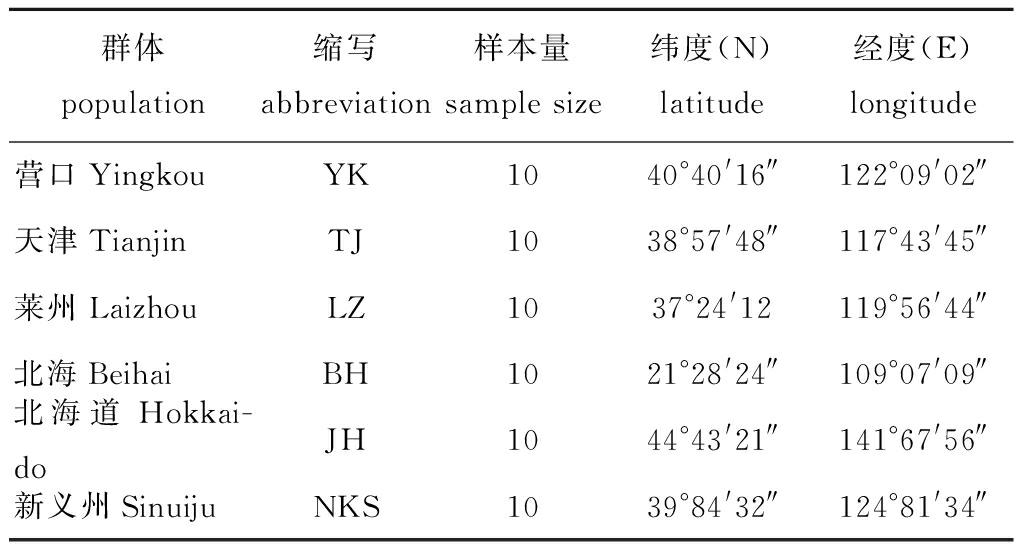
群体population缩写abbreviation样本量sample size纬度(N)latitude经度(E)longitude营口YingkouYK1040°40'16″122°09'02″天津TianjinTJ1038°57'48″117°43'45″莱州LaizhouLZ1037°24'12119°56'44″北海BeihaiBH1021°28'24″109°07'09″北海道Hokkai-doJH1044°43'21″141°67'56″新义州SinuijuNKS1039°84'32″124°81'34″
1.2 方法
1.2.1 DNA提取 采用海洋动物组织基因组DNA提取试剂盒(北京天根生物工程有限公司)提取菲律宾蛤仔基因组DNA,提取的DNA通过核酸定量仪检测浓度,并通过10 g/L琼脂糖凝胶电泳检测后,储存于-20 ℃用于后续试验。
1.2.2 PCR扩增 菲律宾蛤仔线粒体16S rRNA基因扩增所用的引物序列如下[9]:
正向16S rRNA:5′CGCCTGTTTAHYAAAAACAT 3′;
反向16S rRNA:5′CCGGTCTGAACTCAGMTCAYG 3′。
引物由生工生物工程(上海)股份有限公司合成。聚合酶链式反应(PCR)体系(50 μL):100 ng的DNA模板5 μL, Mix(宝生物工程(大连)有限公司)25 μL,正、反向引物各2 μL,用去离子水定容至50 μL。扩增反应条件为:94 ℃下预变性5 min;94 ℃下循环变性30 s,56 ℃下退火复性30 s,72 ℃下延伸1 min,共进行35个循环;最后在72 ℃下再延伸7 min;4 ℃下结束。每个PCR产物在10 g/L琼脂糖凝胶上进行检测,验证条带。所有经验证的PCR产物均采用TaKaRa MiniBEST 琼脂糖凝胶DNA提取试剂盒进行纯化,然后使用ABI 3730 xl DNA分析仪测序系统(生工生物工程(上海)股份有限公司)对PCR引物进行双向测序。
1.3 数据处理
测序完成后,采用DNAMAN软件手工检测和修剪DNA序列。采用DnaSP 5.10.01软件对菲律宾蛤仔进行遗传多样性分析[10]、中性测试及种群动态分析。采用MEGA 7软件[9]计算群体间的 K2P(Kimura-2-parameter) 遗传距离,并利用UPGMA法绘制单倍型遗传聚类树。采用Arlequin 3.0软件[11]计算核苷酸组成、群体间遗传分化指数(F-statistics,FST)。通过POPART[12-14] (http://www.http://POPART.otago.ac.nz/index.shtml)进行单倍型网络分析。设置菲律宾蛤仔16S rRNA基因序列的置信阈值为95%,用于不同地理群体单倍型网络图的绘制。采用DNASP软件将数据转换为Nexfile文件格式,用POPART构建菲律宾蛤仔Median-joining (MJ)[13]网络图,以检测不同地理群体菲律宾蛤仔间的遗传关系。
2 结果与分析
2.1 菲律宾蛤仔16S rRNA 基因片段碱基组成及单倍型分析
将测序序列剪切至相同长度的535 bp进行分析。6个地理群体16S rRNA 序列的平均碱基组成:T为33.46%,C为10.82%,A为33.79%,G为21.92%,A+T为67.26%。A+T含量依次为YK(67.36%)>LZ(67.28%)>TJ(67.25%)>JH(67.24%)>NPS(67.22%)>BH(67.18%),碱基偏倚显著,A+T含量显著高于G+C含量,且与线粒体碱基组成一致。
根据DnaSP 5.0 软件的分析结果,在菲律宾蛤仔6个地理群体中共获得16S rRNA 基因的单倍型20个,为Hap-1~Hap-20。其中,从单倍型来看,Hap-1、Hap-3、Hap-9和Hap-14 4个单倍型为共享单倍型,其余16个单倍型为群体的独享单倍型(表2)。
共享单倍型中,Hap-1 出现频率最高,为中国北方的营口(YK)、莱州(LZ)、天津(TJ),以及朝鲜新义州(NKS)和日本北海道(JH)群体所共有,占所有检测个体的46.67%;Hap-3为中国营口(YK)、莱州(LZ)、天津(TJ)、北海(BH)群体所共有;从不同地理群体来看,营口(YK)和天津(TJ)群体的单倍型数量最丰富,各自有7个单倍型,日本北海道(JH)群体的单倍型数量最少,只有2个单倍型,其余群体的单倍型数量为3~6个(表2)。
表2 菲律宾蛤仔16S rRNA 基因序列的单倍型序列比对及在不同群体中的分布
Tab.2 Haplotype sequence comparison of 16S rRNA gene sequence and distribution in different populations of Manila clam Ruditapes philippinarum

单倍型haplotype单体型序列比对haplotype sequence alignment营口(10)YK(10)莱州(10)LZ(10)北海(10)BH(10)天津(10)TJ(10)朝鲜(10)NKS(10)日本(10)JH(10)合计totalHap-1ATAATTGGCTGCGGCTCAATGATACGAAGGACA4528928Hap-2ATAATTGGCTGCGGCGTAACGATACGAAGGACA11Hap-3ATAATTGGCTGCGGCGTAATGATACGAAGGACA116311Hap-4ATAATTCGTTGCGGTGTAATGATACGAAGGACA11Hap-5ATATTTCGTTGCGGTGTAATGATACGAAGGACA11Hap-6ATATCCCGTGTTGATGTATTGATACAAAAAGCA11Hap-7ATATTTCGTTGCGGTTCAATGATACGAAGGACA11Hap-8ATAATTCGTTGCGGCGTAATGATACGAAAAACA11Hap-9ATAATTGGCTGCGGCTCAATGATACGAAGGACG1113Hap-10AATATTGGCTGCGGCGTAATGATACGAAGGACA11Hap-11ATAATTGGCTGCAGCGTAATGATACGAAGGACA11Hap-12ATAATTGACTGCGGCGTAATGATACGAAGAACA11Hap-13ATAATTCGCTGCGGCGTCATGACTTGCGGGATA11Hap-14TTAATTGGCTGCGGCGTAATGATACGAAGGACA112Hap-15ATAATTCGCTGCGGCGTAATGATACGAAGGACA11Hap-16ATAATTCGTTGCGGTTCAATGATACGAAGGACA11Hap-17ATAATTGGCTGCGGCGTAATAATACGAAGGACA11Hap-18ATAATTCGTTGCGGTTCAATGATACGAAAGACA11Hap-19ATAATTGGCTGCGGCTCAATGGTACGAAGGACA11Hap-20ATAATTCGCTGCGGCTCAATGATACGAAGGACA11
2.2 菲律宾蛤仔不同地理群体16S rRNA基因的遗传多样性分析
从表3可见:从各地理群体来看,基于16S rRNA基因片段,天津(TJ)、营口(YK)和莱州(LZ)群体的单倍型多样性(Hd)较高,分别为0.911、0.867和0.867,朝鲜新义州(NKS)和日本北海道(JH)群体单倍型多样性(Hd) 较低,均为0.378;营口(YK)群体的平均核苷酸多样性(Pi)和平均核苷酸差异数(K)在6个群体里最高,分别为0.010 60和5.578,日本北海道(JH)群体在6个群体里最低,分别为0.000 75和0.400。
2.3 菲律宾蛤仔单倍型发生关系
菲律宾蛤仔群体的单倍型网络中介图如图1(a)所示,6个群体单倍型呈分散状态,各个单倍型连接点间节点较多,单倍型分支呈现星状分布,其中,Hap-1和Hap-3分别处于2个集中分支的中心,且所占比例较大,由此可推测,这两个单倍型为菲律宾蛤仔的原始单倍型。
如图1(b)所示,单倍型在群体中分布网络图能更加直观地显示单倍型在不同地理群体中的分布和系统演化关系, 4个共享单倍型(Hap-1、Hap-3、Hap-9和Hap-14)分别分散在不同地理群体中。
表3 菲律宾蛤仔6个不同地理群体mtDNA 16S rRNA 基因的遗传多样性分析
Tab.3 Genetic diversity of mtDNA 16S rRNA gene of Manila clam Ruditapes philippinarum among 6 different geographic populations

群体population单倍型数number of haplotype单倍型(个体数)haplotype(number of individual)单倍型多样性haplotype diversity(Hd)核苷酸多样性nucleotide diversity(Pi)核苷酸平均差数average number of nucleotide difference (K)营口YK7Hap-1(4),Hap-2(1),Hap-3(1),Hap-4(1),Hap-5(1),Hap-6(1),Hap-7(1)0.867±0.1070.010 60±0.003 555.578±1.694莱州LZ6Hap-1(5),Hap-3(1),Hap-8(1),Hap-9(1),Hap-10(1),Hap-11(1)0.867±0.1070.005 41±0.001 382.867±1.168北海BH5Hap-3(6),Hap-9(1),Hap-12(1),Hap-13(1),Hap-14(1)0.667±0.1630.005 30±0.002 362.800±1.141天津TJ7Hap-1(2),Hap-3(3),Hap-9(1),Hap-14(1),Hap-15(1),Hap-16(1),Hap-17(1)0.911±0.0770.005 07±0.000 962.422±0.987新义州NKS3Hap-1(8),Hap-18(1),Hap-19(1)0.378±0.1810.001 89±0.001 171.000±0.407北海道JH2Hap-1(9),Hap-20(1)0.378±0.1810.000 75±0.000 390.400±0.163合计total20Hap-1~Hap-200.753±0.0530.005 48±0.001 062.870±0.989
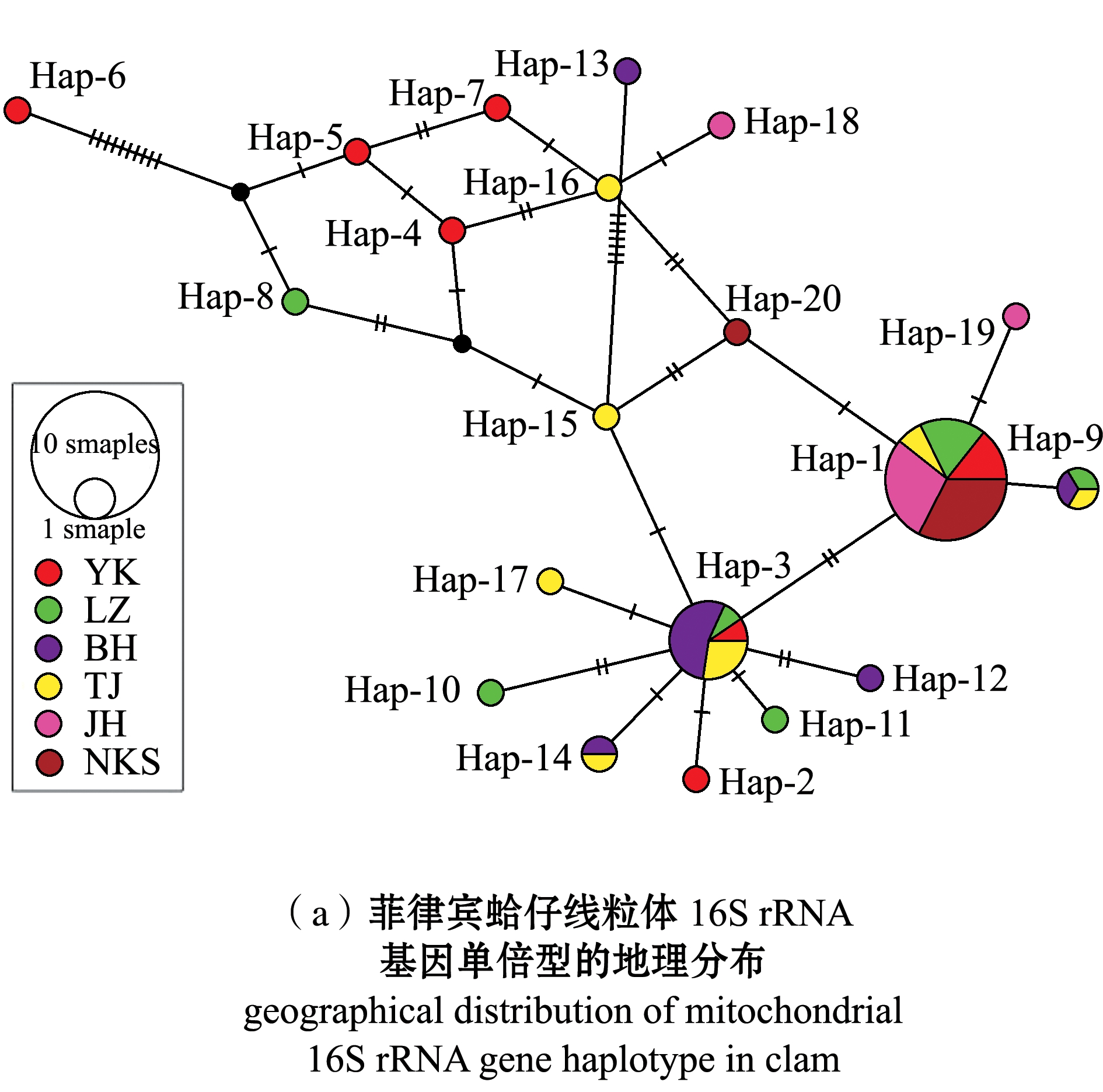
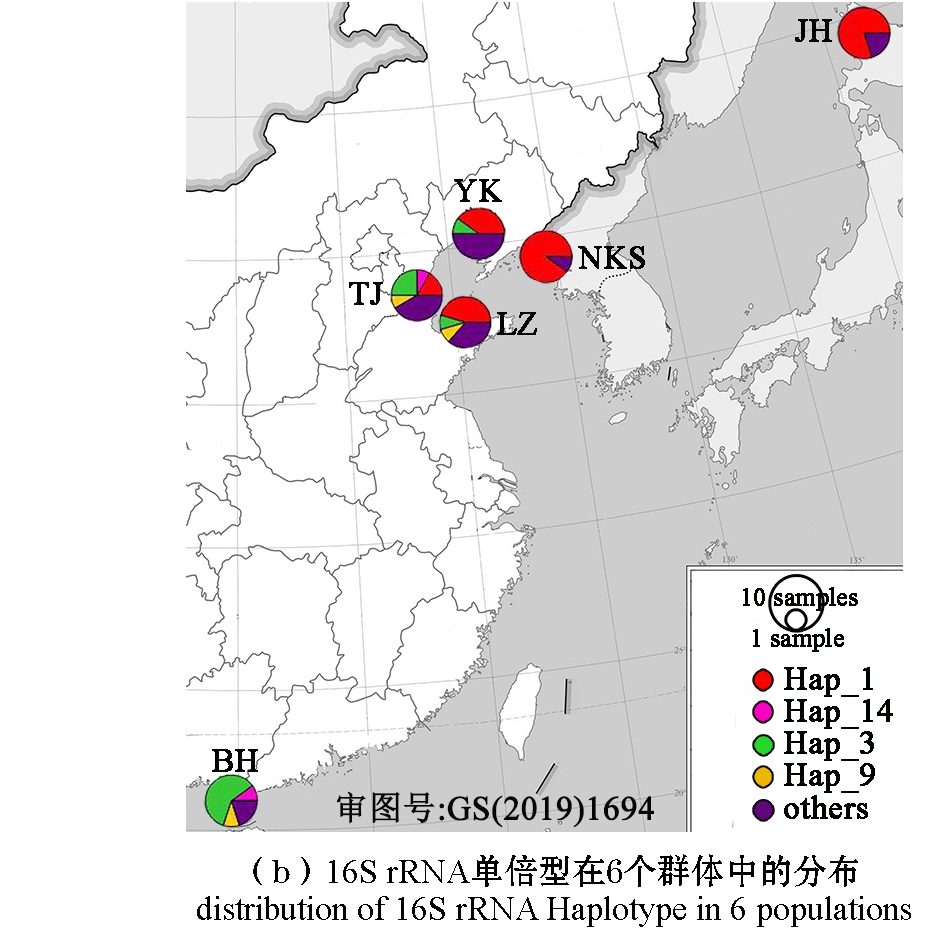
图(a)中每个圆的大小与总单倍型频率成正比;图(b)中扇区的颜色代表不同的单倍型,扇区的大小代表该地点出现此单倍型的频率。
The size of each circle is proportional to the total haplotype frequency in Fig.1(a); The color of the sector represents different haplotypes, and the size of the sector represents the frequency of occurrence of this haplotype at the location in Fig.1(b).
图1 菲律宾蛤仔群体的单倍型网络中介图
Fig.1 Haplotype web intermediary figure in populations of Manila clam Ruditapes philippinarum
根据菲律宾蛤仔6个群体的线粒体16S rRNA基因片段,构建20个单倍型Neighbor-joining (NJ)系统进化树(图2)。其中,共享单倍型Hap-1分布在YK、LZ、TJ、NKS和JH群体中,Hap-3分布在营口(YK)、莱州(LZ)、北海(BH)和天津(TJ)群体中,Hap-9分布在莱州(LZ)、北海(BH)和天津(TJ)群体中,Hap-14分布在北海(BH)和天津(TJ)群体中。在20个单倍型中存在2个优势单倍型(Hap-1和Hap-3),分别占个体数的46.67%和18.33%。
2.4 群体间遗传距离与遗传分化
从表4可见,6个群体间的K2P遗传距离均小于0.01,未达到种间分化水平。采用16S rRNA基因序列的遗传分化系数(FST)对群体间关系进行分析,结果表明:中国北方营口(YK)、莱州(LZ)、天津(TJ)3个群体间遗传分化不明显,南方北海群体(BH)与莱州(LZ)、营口(YK)群体间存在显著的遗传分化(P<0.05),但与天津(TJ)群体间遗传分化不显著(P>0.05);朝鲜新义州(NKS)与日本北海道(JH)群体间遗传分化不显著(P>0.05),两群体与北方营口(YK)和莱州(LZ)群体间遗传分化也不显著(P>0.05),但与天津(TJ)和北海(BH)群体间遗传分化显著(P<0.05)。
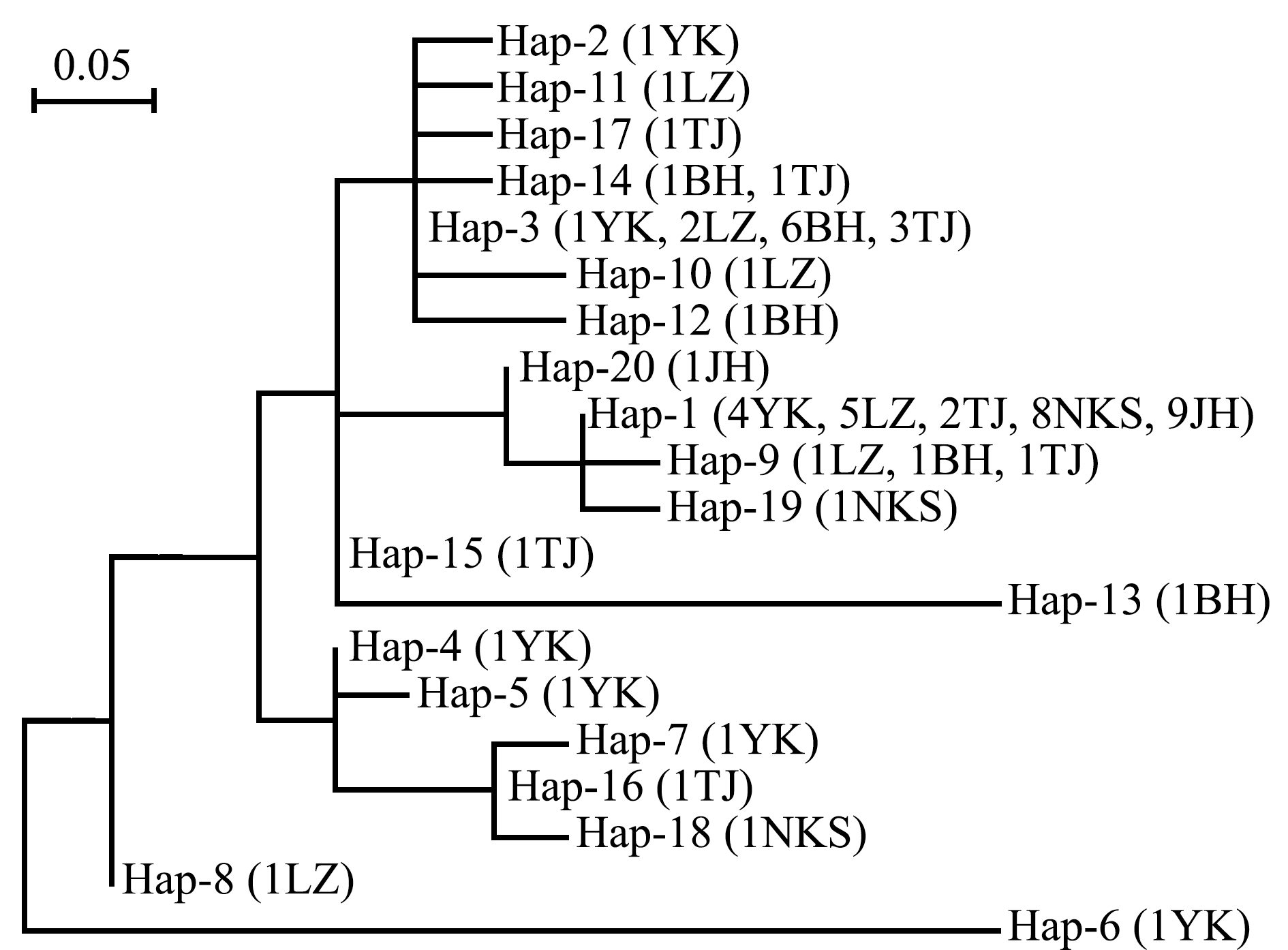
YK—营口群体;LZ—莱州群体;BH—北海群体;TJ—天津群体;NKS—新义州群体;JH—北海道群体。群体名前的数字代表群体中出现的个体数。
YK—Yingkou population; LZ—Laizhou population;BH—Beihai population;TJ—Tianjin population;NKS—Sinuiju population;JH—Hokkaido population.The number represents the number of individuals present in the populations.
图2 20种单倍型在6个群体中的分布及其系统发育树
Fig.2 Distribution of 20 haplotypes and NJ molecular phylogenetic tree in 6 populations
2.5 种群动态分析
通过线粒体16S rRNA基因对菲律宾蛤仔不同地理群体进行错配分析。错配分析(Mismatch distribution)曲线图可以对种群的历史动态进行分析,当种群为新形成或近期发生扩张时,其错配分析曲线呈单峰;当种群为一个长期稳定的群体时,错配分析曲线出现多个峰值。本研究结果显示(图3),6个群体均呈现双峰或多峰,6个地理群体在近期未发生快速扩张,群体处于相对稳定的状态。
表4 菲律宾蛤仔6个地理群体间K2P遗传距离(上三角)和遗传分化系数(FST) (下三角)
Tab.4 Genetic distance (upper triangle) and genetic differentiation coefficient (FST) (lower triangle) among 6 geographic populations of Manila clam Ruditapes philippinarum
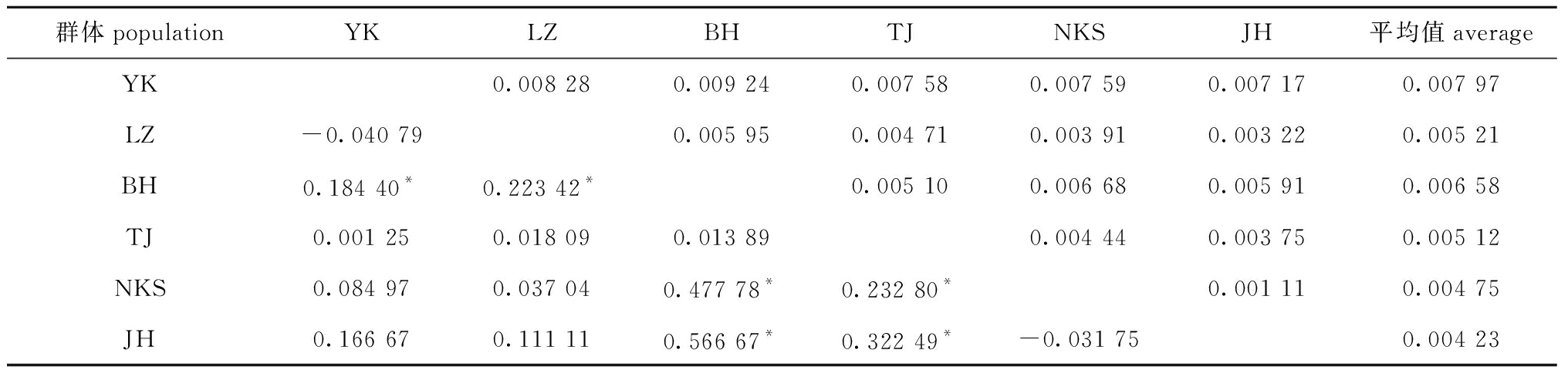
群体populationYKLZBHTJNKSJH平均值averageYK0.008 280.009 240.007 580.007 590.007 170.007 97LZ-0.040 790.005 950.004 710.003 910.003 220.005 21BH0.184 40*0.223 42*0.005 100.006 680.005 910.006 58TJ0.001 250.018 090.013 890.004 440.003 750.005 12NKS0.084 970.037 040.477 78*0.232 80*0.001 110.004 75JH0.166 670.111 110.566 67*0.322 49*-0.031 750.004 23
注: *表示显著相关(P<0.05)。
Note: * means significant correlations(P<0.05).
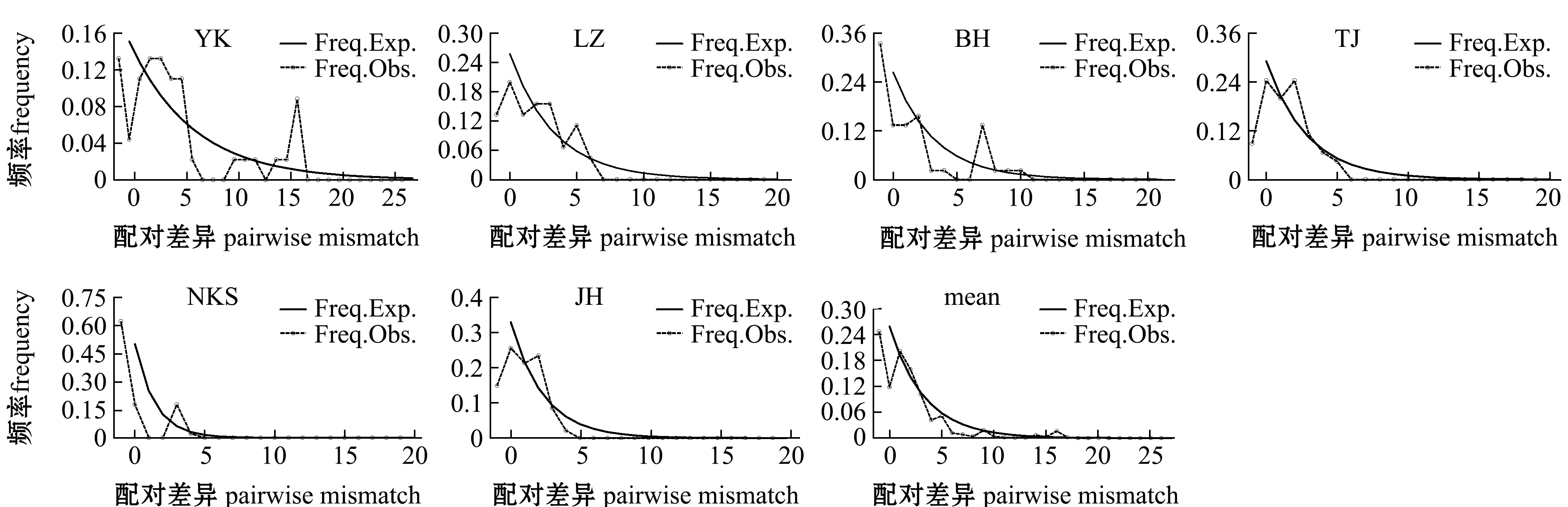
图3 基于16S rRNA 基因菲律宾蛤仔6个地理群体的错配分布分析
Fig.3 Mismatch distribution analysis of 6 populations of Manila clam Ruditapes philippinarum based on 16S rRNA gene
3 讨论
3.1 群体遗传多样性
本研究中对菲律宾蛤仔线粒体16S rRNA基因片段单倍型进行分析, 6个群体共检测到20个单倍型,包括4个共享单倍型和16个独享单倍型,这说明不同地理群体有部分基因交流的同时,也存在一定程度遗传分化。其中,共享单倍型Hap-1发生频率最高,在群体中分布最广,故认为该单倍型是最原始的单倍型,能够适应环境变化并在群体中保持稳定存在。
遗传多样性是指物种内部和物种间的遗传变异程度,是生物适应环境和物种进化的基础。单倍型多样性描述了样品间的等位基因差异,而核苷酸多样性代表了DNA序列间核苷酸差异的平均数量[15]。单倍型多样性指数和核苷酸多样性指数是衡量遗传多样性的两个重要参数[16]。一般来说,存在4种情况:
1) 单倍型多样性指数和核苷酸多样性指数的变异均较低(Hd<0.5,Pi<0.005)时,意味着最近发生过群体瓶颈效应或由单一、少数群体所产生的建立者效应。
2) 单倍型多样性指数较高而核苷酸多样性指数较低(Hd>0.5,Pi<0.005)时,预示着群体曾经历过瓶颈效应后,伴随了迅速的群体扩张与变异的积累。
3) 单倍型多样性指数较低而核苷酸多样性指数较高(Hd<0.5,Pi>0.005)时,提示可能是来自两个独立群体发生的二次接触所导致,或者是一个大而稳定的群体中发生过瓶颈效应。
4) 高单倍型多样性和高核苷酸多样性指数均较高(Hd>0.5,Pi>0.005)时,表明一个大而稳定的群体是经过长时间演化或是两个不同系群的群体二次接触所产生的[17]。
本研究中,检测到朝鲜NKS和日本JH群体遗传多样性指数均较低(Hd<0.5,Pi<0.005),其原因可能是群体建立者效应,2个群体在相对较短的时间内获得核苷酸变异的个体,但在短时间内核苷酸出现变异的数量不足,导致群体中核苷酸多样性降低,使其群体遗传多样性下降。另外,也可能是由于本研究中样本数量较少导致2个群体遗传多样性降低,在以后的研究中,应扩大群体样本数量,并结合其他分子标记和基因组测序方法,对这两个群体遗传多样性开展进一步分析。天津、营口、莱州和广西群体具有较高的遗传多样性,这反映了其具有较强的环境适应性和较大的遗传变异潜力。
物种的遗传多样性与其对环境的适应能力、物种进化潜力及生存能力密切相关。遗传多样性丰富的物种面对环境变化时具有较强的适应性,在物种进化方面表现出较好的潜力,而遗传多样性的匮乏对于种质资源保护和利用产生诸多不利影响。本研究中,根据单倍型遗传多样性、遗传及群体动态分析,在6个群体中,天津(TJ)、营口(YK)、莱州(LZ)群体的单倍型个数、单倍型多样性和核酸多样性较高,北海(BH)群体遗传多样性次之,朝鲜新义州(NKS)和日本北海道群体(JH)遗传多样性较低。菲律宾蛤仔在相邻海域内存在广泛的基因交流。中国人工异地移养菲律宾蛤仔活动对本地野生群体基因资源产生了一定影响。
3.2 群体遗传距离与遗传分化
K2P方法主要用来计算种间水平的遗传距离[18],一般种内K2P遗传距离小于0.01,只有个别物种的种内遗传距离大于0.02[19-22]。但K2P方法并不能准确反映群体间的遗传分化程度[18-19]。因此,本研究中首先使用 K2P方法检测不同群体间菲律宾蛤仔遗传距离,再使用FST评估群体间遗传分化程度。本研究表明,6个群体的遗传距离均小于0.01,并未达到种间分化水平。FST可以用于群体间遗传分化程度的评估,其值为-1~1,两个群体间FST值越大,表示两个群体的遗传分化程度越高[23]。一般认为,0<FST<0.05表示群体间无显著性分化,0.05<FST<0.15表示群体间分化程度中等,0.15<FST<0.25表示群体间分化程度较大,FST>0.25表示群体间分化程度极大。本研究中6个野生地理群体FST值为-0.040 79~0.566 67,中国南方北海群体与中国北方群体(除天津群体)、日本群体和朝鲜群体遗传分化显著,6个群体可划分为南、北2个谱系,即中国南方北海群体为南方谱系,其他5个群体为北方谱系。在前期的研究报道中,陈辰等[24]将毛蚶Scapharca subcrenata划分为南、北方2个群系,这可能是由于中国长江淡水形成的天然屏障阻断了海洋贝类南、北方自然种群的基因交流,同一贝类物种在不同的地理格局,经过上万年的隔离和群体演化,形成了具有明显特征的南、北方谱系。
菲律宾蛤仔幼虫浮游时间长(2~3周)、分布空间广、环境适应能力强的特点,提高了群体扩张和基因交流的效率。Cordero等[6]发现,菲律宾蛤仔日本群体自1936年被引入北美加拿大不列颠哥伦比亚海岸后,经过30余年的繁衍即扩张到美国南加利福尼亚州。Chiesa等[25]报道,1972年被引种到法国、英国、意大利、西班牙等欧洲沿岸的菲律宾蛤仔与本地土著品种发生了基因渐渗。本研究中,北海道群体和新义州群体的蛤仔遗传分化程度较低,可能与选择的分子标记有关,在今后的研究中可采用具有较高多态性的微卫星标记对群体遗传多样性进行分析;在明显的南北方谱系中,南方北海群体与北方天津群体遗传分化不显著,这两个群体均存在共享单倍型Hap-14,这可能与天津海域人工移养菲律宾蛤仔南方苗种有关[26],在此过程中,南方北海群体与北方群体得到了一定程度的基因交流;在北方谱系中,天津群体与日本、朝鲜群体均分化显著,天津与日本、朝鲜群体除了北方特有单倍型Hap-1外无其他共享单倍型,天津群体与北海群体有3个共享单倍型(Hap-3、Hap-9和Hap-14),说明天津群体由于人工移养南方苗种发生基因交流密切,由此与日本、朝鲜群体产生显著分化。中国北方营口、天津及莱州群体间遗传分化不显著,这可能与其在同一海域相互间广泛的基因交流有关。结合本研究的遗传分化和共享单倍型地理分布结果,分析中国北方3个群体、朝鲜新义州和日本北海道群体的基因流为距离隔离模式(Isolation-By-Distance Model),即菲律宾蛤仔属于活动范围有限,出生到死亡基本无较大移动范围的物种,各个群体在空间上连续分布,由于受个体间随机交配范围的限制,基因频率在相邻群体间差异小,不同群体在地理位置和遗传距离间存在联系。
3.3 种质资源的保护
根据本研究结果,今后的研究工作中可以将天津、营口、莱州和北海野生群体作为主要研究对象,利用线粒体COI基因序列及SSR(简单重复序列)等分子标记,以及SLAF(特异位点扩增片段)简化基因组技术对群体进行遗传多样性分析。日本朝鲜群体,种内检测到的遗传变异水平较低,基于该分子标记检测出蛤仔北海道和新义州群体遗传分化不显著,这可能与选择的分子标记有关,在今后的研究中可采用核基因标记(微卫星标记)进行分析。在进一步确定种质优良的蛤仔群体基础上,通过原良种场建设等方法,对种质资源进行保护,同时可以将原种场自然野生苗种进行有计划的采捕和换区移养,最终达到可持续利用目的。
4 结论
1) 菲律宾蛤仔天津、营口、莱州及北海4个群体具有较丰富的遗传多样性,朝鲜新义州和日本北海道群体遗传多样性较低。
2) 6个群体可划分为南、北方谱系,即北海群体为南方谱系,其他5个群体为北方谱系;中国北方3个群体(营口、天津和莱州)及日本、朝鲜群体间存在距离隔离模式基因流。
3) 中国人工异地移养菲律宾蛤仔活动对本地野生群体基因资源产生一定影响。
[1] 管华诗,王曙光.中华海洋本草[M].上海:上海科学技术出版社,2009.
[2] MORITZ C,DOWLING T E,BROWN W M.Evolution of animal mitochondrial DNA:relevance for population biology and systematics[J].Annual Review of Ecology and Systematics,1987,18:269-292.
[3] 徐敬明.蟹类线粒体DNA的研究与应用[J].中国海洋大学学报,2006,36(6):879-884.
[4] 胡利莎,张振,马培振,等.10个菲律宾蛤仔野生群体的遗传多样性研究[J].海洋与湖沼,2016,47(3):549-556.
[5] 刘相全,包振民,方建光,等.两种蛤仔线粒体16S rRNA基因和COⅠ基因的序列比较[J].海洋科学,2011,35(10):27-33.
[6] CORDERO D,DELGADO M,LIU B Z,et al.Population genetics of the Manila clam (Ruditapes philippinarum) introduced in North America and Europe[J].Scientific Reports,2017,7(1):39745.
[7] FOIGHIL D  ,GAFFNEY P M,HILBISH T J.Differences in mitochondrial 16S ribosomal gene sequences allow discrimination among American [Crassostrea virginica (Gmelin)] and Asian [C.gigas (Thunberg) C.ariakensis Wakiya] oyster species[J].Journal of Experimental Marine Biology and Ecology,1995,192(2):211-220.
,GAFFNEY P M,HILBISH T J.Differences in mitochondrial 16S ribosomal gene sequences allow discrimination among American [Crassostrea virginica (Gmelin)] and Asian [C.gigas (Thunberg) C.ariakensis Wakiya] oyster species[J].Journal of Experimental Marine Biology and Ecology,1995,192(2):211-220.
[8] AVISE J C,ARNOLD J,BALL R M,et al.Intraspecific phylogeography:the mitochondrial DNA bridge between population genetics and systematics[J].Annual Review of Ecology and Systematics,1987,18:489-522.
[9] KUMAR S,STECHER G,TAMURA K.MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1870-1874.
[10] ROZAS J,ROZAS R.DnaSP,DNA sequence polymorphism:an interactive program for estimating population genetics parameters from DNA sequence data[J].Bioinformatics,1995,11(6):621-625.
[11] EXCOFFIER L,LAVAL G,SCHNEIDER S.Arlequin (version 3.0):an integrated software package for population genetics data analysis[J].Evolutionary Bioinformatics Online,2007,1:47-50.
[12] HANSON K M.Performance optimized algebraic reconstruction technique[C]//Proceedings of SPIE 1001,Visual Communications and Image Processing’88:Third in a Series.Cambridge,MA,USA:SPIE,1988:94.
[13] BANDELT H J,FORSTER P,RÖHL A.Median-joining networks for inferring intraspecific phylogenies[J].Molecular Biology and Evolution,1999,16(1):37-48.
[14] CLEMENT M,SNELL Q,WALKE P,et al.TCS:estimating gene genealogies[C]//Proceedings 16th International Parallel and Distributed Processing Symposium.Ft.Lauderdale,FL,USA:IEEE,2002.
[15] FELSENSTEIN J.Molecular evolutionary genetics[M].New York:Columbia University Press,1987.
[16] 刘绍平,王珂,袁希平,等.怒江扎那纹胸鮡的遗传多样性和遗传分化[J].遗传,2010,32(3):254-263.
[17] GRANT W S,CLARK A M,BOWEN B W.Why restriction fragment length polymorphism analysis of mitochondrial DNA failed to resolve sardine (Sardinops) biogeography:insights from mitochondrial DNA cytochrome b sequences[J].Canadian Journal of Fisheries and Aquatic Sciences,1998,55(12):2539-2547.
[18] SRIVATHSAN A,MEIER R.On the inappropriate use of Kimura-2-parameter (K2P) divergences in the DNA-barcoding literature[J].Cladistics,2012,28(2):190-194.
[19] SHEN Y J,GUAN L H,WANG D Q,et al.DNA barcoding and evaluation of genetic diversity in Cyprinidae fish in the midstream of the Yangtze River[J].Ecology and Evolution,2016,6(9):2702-2713.
[20] HEBERT P D N,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B:Biological Sciences,2003,270(1512):313-321.
[21] HEBERT P D N,RATNASINGHAM S,DE WAARD J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J].Proceedings of the Royal Society B:Biological Sciences,2003,270(S1):S96-S99.
[22] WARD R D.FISH-BOL,a case study for DNA barcodes[M]//KRESS W J,ERICKSON D L.DNA Barcodes:Methods and Protocols.Totowa,NJ:Humana Press,2012.
[23] HUDSON R R,SLATKIN M,MADDISON W P.Estimation of levels of gene flow from DNA sequence data[J].Genetics,1992,132(2):583-589.
[24] 陈辰.毛蚶群体遗传学研究[D].青岛:中国海洋大学,2015:59-68.
[25] CHIESA S,LUCENTINI L,FREITAS R,et al.A history of invasion:COI phylogeny of Manila clam Ruditapes philippinarum in Europe[J].Fisheries Research,2017,186:25-35.
[26] 邢克智,高一力,郭永军,等.天津市贝类产业现状及发展展望[J].水产科学,2013,32(9):555-558.