罗非鱼是中国重要淡水养殖经济鱼类之一,2019年中国罗非鱼总产量达164.17万t,占世界罗非鱼养殖总产量的1/3以上[1]。培育具有生长速度快、抗病抗逆性强、雄性率高和体色均一等优良性状的罗非鱼新品种对罗非鱼产业的可持续发展具有重要意义。中国自1956年首次从国外引种莫桑比克罗非鱼以来,至今已成功培育了13个罗非鱼新品种(奥尼鱼、福寿鱼、尼罗罗非鱼、奥利亚罗非鱼、吉富品系尼罗罗非鱼、“新吉富”罗非鱼、“夏奥1号”奥利亚罗非鱼、“吉鲡”罗非鱼、尼罗罗非鱼“鹭雄1号”、吉富罗非鱼“中威1号”、吉奥罗非鱼、莫荷罗非鱼“广福1号”和罗非鱼“壮罗1号”),罗非鱼种质资源的不断发展壮大极大地促进了中国罗非鱼产量和品质的提升。
罗非鱼“粤闽 1号”为本研究团队近年来培育的新品种,是以经过多代选育的尼罗罗非鱼Oreochromis niloticus为母本,以超雄罗非鱼Oreochromis sp.为父本杂交获得的子一代,具有自然雄性率高、生长速度快和出肉率高等特点[2]。其母本是以台湾引进的尼罗罗非鱼为基础群体,以生长速度、体型和子代性别比例为主要指标连续多代选育获得,选育5代后其生长速度提高了18.99%,体长/头长比值提高了10.54%。但选育群体在选育过程中易受保种群体大小、人工选择和遗传漂变等因素的影响导致群体遗传多样性降低或近交衰退,从而降低其遗传潜力[3]。为了保证其优良性状的稳定遗传,有必要对其选育群体各世代的遗传多样性和遗传结构进行监测。在长江刀鲚Coilia ectenes[3]、大口黑鲈Micropterus salmoides[4]、新吉富罗非鱼Oreochromis niloticus NEW GIFT strian[5]、华南鲤Cyprinus carpio rubrofuscus[6]和草鱼Ctenopharyngodon idella[7]等鱼类中均发现,群体的遗传变异会随着选育世代的推进而不断下降。因此,对罗非鱼“粤闽 1号”母本选育过程中各世代的遗传多样性和遗传结构进行监测具有重要意义。同时,通过分析罗非鱼“粤闽 1号”母本选育群体与不同尼罗罗非鱼地理群体之间的遗传关系可揭示其遗传背景,也可为筛选外群个体用于提高选育群体遗传多样性提供理论依据。
微卫星标记具有数量多、分布广泛且均匀、多态性丰富、呈孟德尔共显性遗传、重复性好和可高通量检测等优点,广泛应用于鱼类选育过程中群体遗传多样性和遗传结构分析[3-8]、种质鉴定[9]、亲权分析[10]和遗传连锁图谱构建[11]等研究。为了揭示罗非鱼“粤闽1号”母本选育群体的遗传背景及人工选择对其遗传结构的影响,本研究中采用微卫星技术分析了罗非鱼“粤闽 1号”母本3个选育世代(F2、F4和F6世代)的遗传多样性和遗传分化,并进一步分析了其与4个尼罗罗非鱼地理群体(高要群体(GY)、惠州群体(HZ)、茂名群体(MM)和无锡群体(WX))之间的遗传关系,旨在为罗非鱼“粤闽1号”母本的进一步人工选育和遗传改良提供理论依据。
1 材料与方法
1.1 材料
罗非鱼“粤闽1号”母本(尼罗罗非鱼)3个选育世代群体(F2、F4和F6)的样本均采自福建省漳州市福建百汇盛源水产种业有限公司养殖基地罗非鱼繁育保种池塘。4个尼罗罗非鱼地理群体的样本分别采自肇庆高要、惠州、茂名和无锡的罗非鱼鱼苗繁殖池塘,并分别命名为GY群体、HZ群体、MM群体和WX群体。上述每个选育世代群体和地理群体均随机采集罗非鱼尾鳍样本20份,置于无水乙醇中-40 ℃下冷冻保存备用。
1.2 方法
1.2.1 基因组DNA的提取 采用动物组织基因组DNA提取试剂盒(TIANGEN)抽提上述尾鳍样本基因组DNA,提取步骤参照试剂盒说明书进行。采用10 g/L琼脂糖凝胶电泳检测提取的基因组DNA质量,采用超微量分光光度计(OSE-260, TIANGEN)检测DNA的浓度及纯度,并用无菌双蒸水将DNA稀释至质量浓度为50 ng/μL,-20 ℃下保存备用。
1.2.2 微卫星序列PCR扩增 从Lee等[11]报道的尼罗罗非鱼二代遗传连锁图谱的每个连锁群中随机选取2 ~ 4个微卫星位点,引物序列从GenBank数据库中获得,通过预试验从中筛选获得13个能够在尼罗罗非鱼中稳定扩增、多态性较好的微卫星位点,其分布于12个连锁群,相应位点的荧光标记引物由广州艾基生物技术有限公司合成,引物详细信息见表1。PCR扩增体系(总体积10 μL)包括:2×Taq PCR Master Mix (TaKaRa, Japan) 5.0 μL,上、下游引物(10 μmol/L) 各0.5 μL,DNA模板(50 ng/μL) 1.0 μL,无菌双蒸水3 μL。采用降落PCR进行扩增。反应程序为:95 ℃下预变性5 min;95 ℃下变性30 s,62~54 ℃下退火30 s,72 ℃下延伸30 s,共进行10个循环;95 ℃下变性30 s,52 ℃退火30 s,72 ℃延伸30 s,共进行22个循环;最后在72 ℃下延伸10 min。根据PCR产物大小不重叠的原则混合PCR产物后,利用ABI 3730 xl DNA测序仪进行多重毛细管电泳检测分型。
1.3 基因分型与数据分析
将毛细管电泳检测结果导入到GeneMarker软件中,分析每对引物扩增产物的大小,采用 Popgen 3.2软件统计各个微卫星位点上的等位基因数(allele number,Na)、有效等位基因数(effective allele number,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、Shannon 指数(Shannon index,I)、 遗传距离(genetic distance,Da)和遗传相似度(genetic similarity index)。 采用PIC-CALC软件计算多态性信息含量(polymorphism information content,PIC)。
采用 MEGA 4.1软件中非加权配对算术平均法(unweighted pair group method with arithmetic mean,UPGMA)构建系统进化树。利用Arlequin 3.0软件对群体遗传变异进行分子方差分析(AMOVA),并计算遗传分化系数(proportion of genetic differentiation,FST)。
表1 微卫星位点的退火温度及其荧光标记引物序列信息
Tab.1 Fluorescence labeled primer sequences and annealing temperature of microsatellite loci
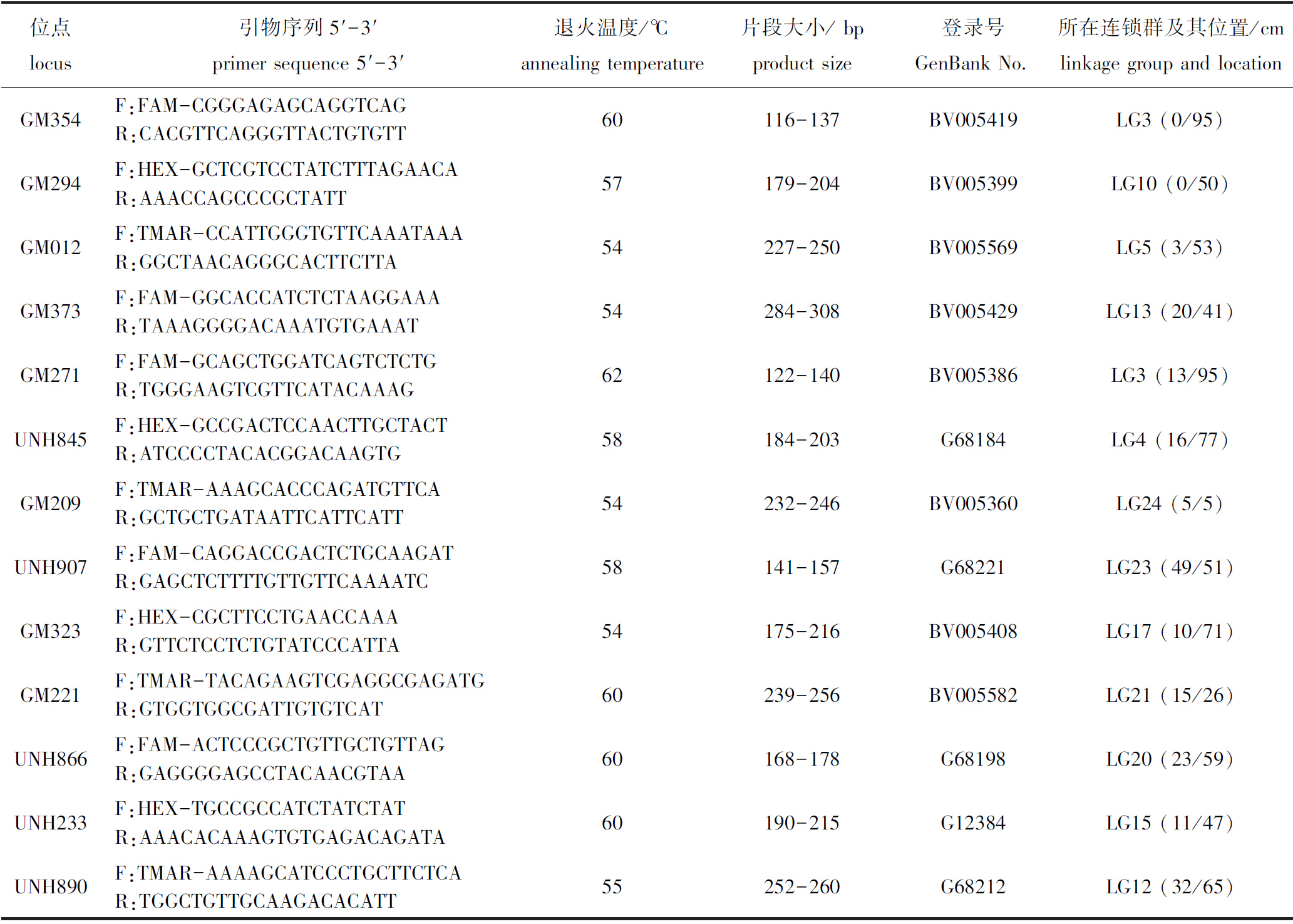
位点locus引物序列5'-3' primer sequence 5'-3'退火温度/℃annealing temperature片段大小/ bpproduct size 登录号GenBank No.所在连锁群及其位置/cmlinkage group and locationGM354F:FAM-CGGGAGAGCAGGTCAGR:CACGTTCAGGGTTACTGTGTT60116-137BV005419LG3 (0/95)GM294F:HEX-GCTCGTCCTATCTTTAGAA-CAR:AAACCAGCCCGCTATT57179-204BV005399LG10 (0/50)GM012F:TMAR-CCATTGGGTGT-TCAAATAAAR:GGCTAACAGGGCACTTCTTA54227-250BV005569LG5 (3/53)GM373F:FAM-GGCACCATCTCTAAGGAAAR:TAAAGGGGACAAATGTGAAAT54284-308BV005429LG13 (20/41)GM271F:FAM-GCAGCTGGATCAGTCTCTGR:TGGGAAGTCGTTCATACAAAG62122-140BV005386LG3 (13/95)UNH845F:HEX-GCCGACTCCAACTTGCTACTR:ATCCCCTACACGGACAAGTG58184-203G68184LG4 (16/77)GM209F:TMAR-AAAGCACCCAGATGTTCA R:GCTGCTGATAATTCATTCATT54232-246BV005360LG24 (5/5)UNH907F:FAM-CAGGACCGACTCTG-CAAGATR:GAGCTCTTTTGTTGTTCAAAATC58141-157G68221LG23 (49/51)GM323F:HEX-CGCTTCCTGAACCAAAR:GTTCTCCTCTGTATCCCATTA54175-216BV005408LG17 (10/71)GM221F:TMAR-TACAGAAGTCGAG-GCGAGATGR:GTGGTGGCGATTGTGTCAT60239-256BV005582LG21 (15/26)UNH866F:FAM-ACTCCCGCTGTTGCTGTTAGR:GAGGGGAGCCTACAACGTAA60168-178G68198LG20 (23/59)UNH233F:HEX-TGCCGCCATCTATCTATR:AAACACAAAGTGTGAGACAGATA60190-215G12384LG15 (11/47)UNH890F:TMAR-AAAAGCATCCCTGCTTCT-CAR:TGGCTGTTGCAAGACACATT55252-260G68212LG12 (32/65)
2 结果与分析
2.1 微卫星位点的多态性
13对微卫星引物在罗非鱼“粤闽1号”母本的3个选育世代群体(F2、F4和F6)和4个尼罗罗非鱼地理群体(高要群体(GY)、惠州群体(HZ)、茂名群体(MM)和无锡群体(WX))中均能稳定重复地扩增出特异性片段。13对微卫星引物在上述7个尼罗罗非鱼群体中共扩增出176个等位基因,平均等位基因数(Na)为13.538 5,平均有效等位基因数(Ne)、Shannon指数(I)、观测杂合度(Ho)、期望杂合度(He)、多态性信息含量(PIC)和近交系数(Fis)分别为6.220 7、2.027 9、0.648 0、0.810 9、0.789 9和0.101 5(表2)。
2.2 群体内遗传多样性
罗非鱼“粤闽1号”母本3个选育世代F2、F4和F6的平均等位基因数分别为7.307 7、5.538 5和3.923 1,呈现出随着选育世代增加而逐渐减少的趋势;此外,有效等位基因数、期望杂合度、Shannon指数和多态性信息含量等多态性参数也随选育进程逐渐下降,但3个选育世代仍表现为高度多态性(PIC>0.5)(表3)。
在4个尼罗罗非鱼地理群体中GY和MM群体的遗传多样性较高,其PIC分别为0.754 1和0.748 6,其次为HZ群体,WX群体的遗传多样性最低;GY、MM和HZ群体的遗传多样性显著高于罗非鱼“粤闽1号”母本的3个选育世代群体,而WX群体的遗传多样性与3个选育世代群体相当(表3)。
2.3 群体间遗传距离与亲缘关系
遗传距离和遗传相似度分析结果表明:在罗非鱼“粤闽1号”母本的3个选育世代中,F2和F4的遗传距离最小(0.155 6),遗传相似度为0.855 9;随着选育世代增加,世代间遗传距离增大,F2与F6之间的遗传距离为0.351 8,遗传相似度为0.703 5;在4个尼罗罗非鱼地理群体中,HZ群体与GY群体的遗传相似度最高,为0.795 2,遗传距离为0.229 2;MM群体与WX群体之间遗传相似度次之,为0.717 8,遗传距离为0.368 4;在7个尼罗罗非鱼群体中,F2、F4两个选育群体与F6群体的遗传距离要大于它们与HZ和GY群体的遗传距离(表4)。
表2 13个微卫星位点在7个尼罗罗非鱼群体中的遗传多样性参数
Tab.2 Genetic diversity parameters of the 13 microsatellite loci in 7 populations of Nile tilapia Oreochromis niloticus
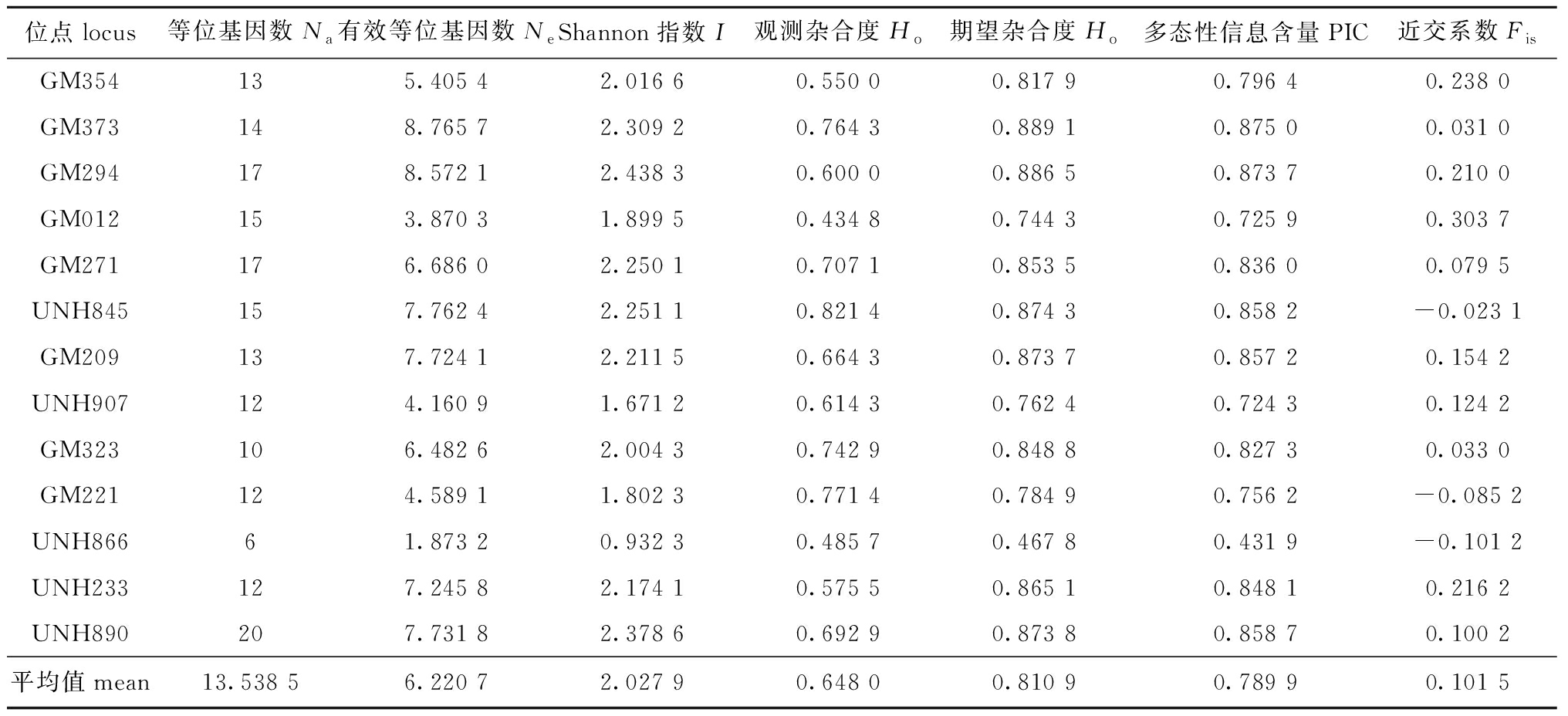
位点locus等位基因数Na有效等位基因数NeShannon指数I观测杂合度Ho期望杂合度Ho多态性信息含量PIC近交系数FisGM354135.405 42.016 60.550 00.817 90.796 40.238 0GM373148.765 72.309 20.764 30.889 10.875 00.031 0GM294178.572 12.438 30.600 00.886 50.873 70.210 0GM012153.870 31.899 50.434 80.744 30.725 90.303 7GM271176.686 02.250 10.707 10.853 50.836 00.079 5UNH845157.762 42.251 10.821 40.874 30.858 2-0.023 1GM209137.724 12.211 50.664 30.873 70.857 20.154 2UNH907124.160 91.671 20.614 30.762 40.724 30.124 2GM323106.482 62.004 30.742 90.848 80.827 30.033 0GM221124.589 11.802 30.771 40.784 90.756 2-0.085 2UNH86661.873 20.932 30.485 70.467 80.431 9-0.101 2UNH233127.245 82.174 10.575 50.865 10.848 10.216 2UNH890207.731 82.378 60.692 90.873 80.858 70.100 2平均值mean13.538 56.220 72.027 90.648 00.810 90.789 90.101 5
表3 罗非鱼“粤闽1号”母本的选育世代间和尼罗罗非鱼地理群体间的遗传参数
Tab.3 Average genetic parameters of three maternal selective breeding generations of tilapia “Yuemin No.1” and four geographical populations of Nile tilapia Oreochromis niloticus
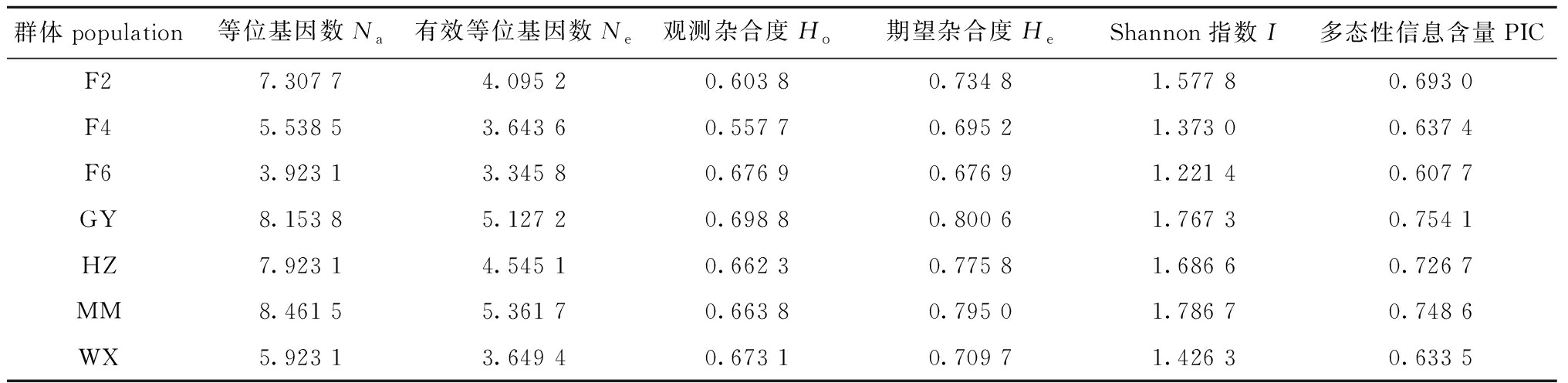
群体population等位基因数Na有效等位基因数Ne观测杂合度Ho期望杂合度HeShannon指数I多态性信息含量PICF27.307 74.095 20.603 80.734 81.577 80.693 0F45.538 53.643 60.557 70.695 21.373 00.637 4F63.923 13.345 80.676 90.676 91.221 40.607 7GY8.153 85.127 20.698 80.800 61.767 30.754 1HZ7.923 14.545 10.662 30.775 81.686 60.726 7MM8.461 55.361 70.663 80.795 01.786 70.748 6WX5.923 13.649 40.673 10.709 71.426 30.633 5
表4 罗非鱼“粤闽1号”母本的选育世代间和尼罗罗非鱼地理群体间的遗传距离(左下部分)和遗传相似度(右上部分)
Tab.4 Genetic distance (lower-left triangle) and the genetic similarity index (upper-right triangle) among three maternal selective breeding generations of tilapia “Yuemin No.1” and four geographical populations of Nile tilapia Oreochromis niloticus
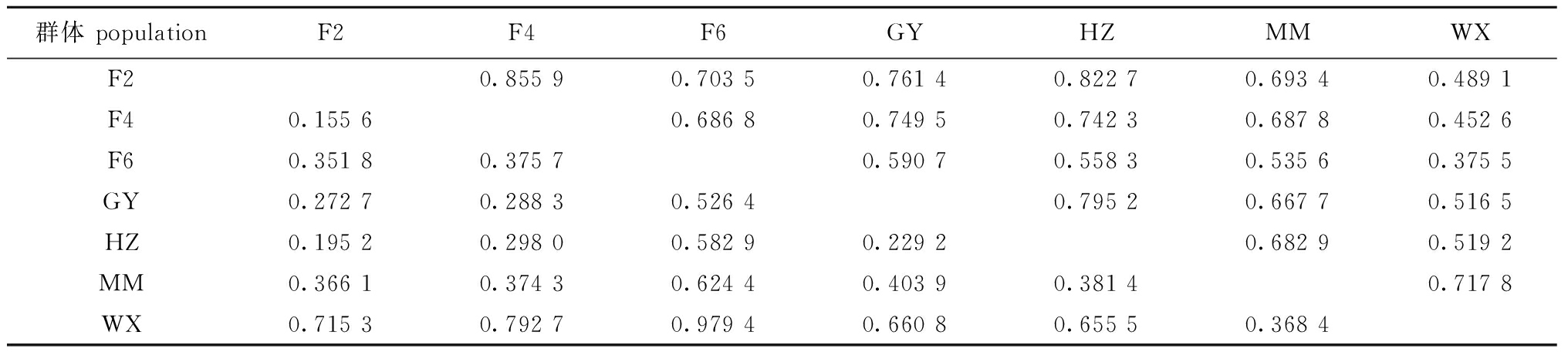
群体 populationF2F4F6GYHZMMWXF20.855 90.703 50.761 40.822 70.693 40.489 1F40.155 60.686 80.749 50.742 30.687 80.452 6F60.351 80.375 70.590 70.558 30.535 60.375 5GY0.272 70.288 30.526 40.795 20.667 70.516 5HZ0.195 20.298 00.582 90.229 20.682 90.519 2MM0.366 10.374 30.624 40.403 90.381 40.717 8WX0.715 30.792 70.979 40.660 80.655 50.368 4
基于群体间遗传距离的UPGMA系统树进化分析显示,MM群体与WX群体聚为一大支,罗非鱼“粤闽1号”母本3个选育世代群体与HZ、GY群体聚为另一大支,其中F6群体独立聚为一小支(图1)。
2.4 群体间遗传分化
根据Wright[12]的划分标准:当FST 为0~0.05时表示群体间遗传分化微弱;当FST 为0.05~0.15时表示群体遗传分化中等;当FST 为0.15~0.25时表示群体遗传分化较大;当FST 大于0.25时,表示遗传分化极大。罗非鱼“粤闽1号”母本的3个选育世代群体中,F2与F4遗传分化微弱,其FST 为 0.036 4;F2、F4二者与F6之间均为中等遗传分化,其FST 分别为0.095 5和 0.111 1,随着选育进程,世代间遗传分化不断加剧;4个尼罗罗非鱼地理群体中,HZ与GY群体间遗传分化微弱,但它们与WX和MM群体均具有中等遗传分化;罗非鱼“粤闽1号”母本的3个选育世代群体与4个地理群体中的GY、HZ和MM群体之间遗传分化中等,而与WX群体之间遗传分化较大(表5)。
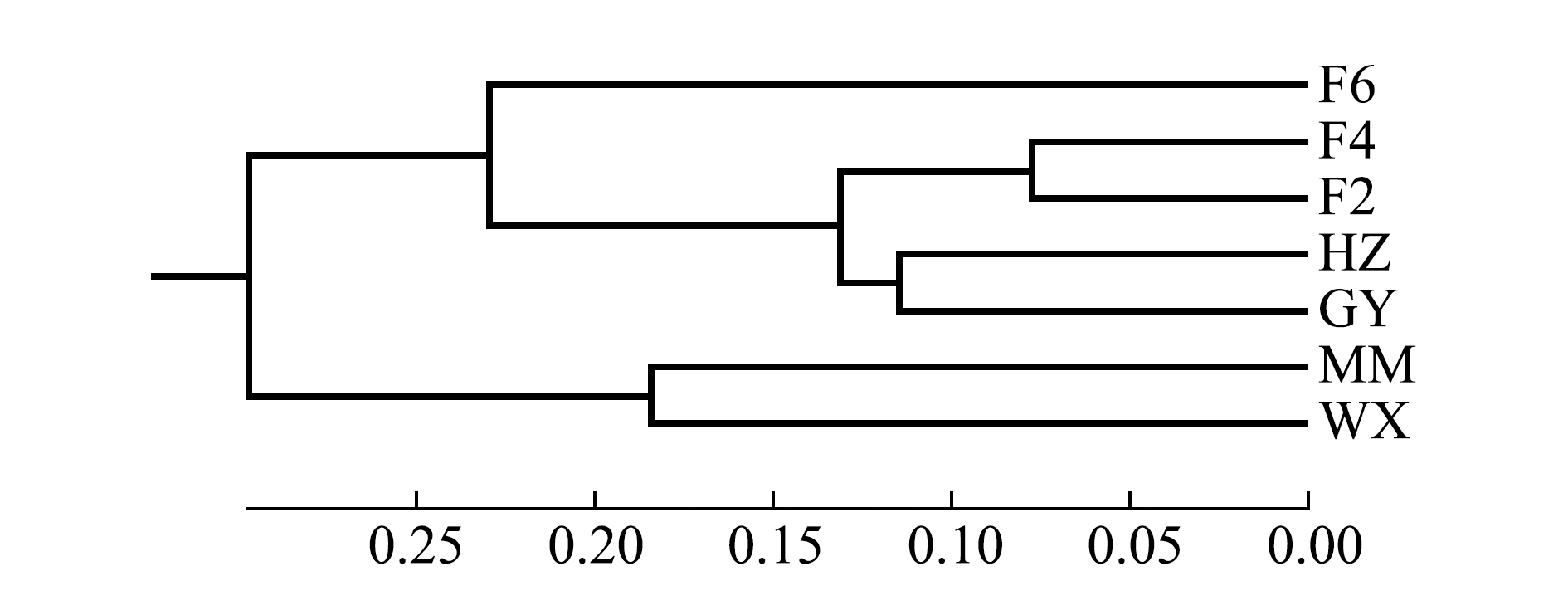
图1 基于遗传距离构建的7个尼罗罗非鱼群体的UPGMA系统进化树
Fig.1 Dendrogram of seven populations of Nile tilapia Oreochromis niloticus based on genetic distance using UPGMA method
AMOVA分析结果表明,罗非鱼“粤闽1号”母本的3个选育世代群体和4个尼罗罗非鱼地理群体的遗传分化有90.01%来自群体内个体间遗传差异,仅有9.99%来自群体间(表6)。
表5 罗非鱼“粤闽1号”母本的选育世代间和尼罗罗非鱼地理群体间的遗传分化系数(FST)
Tab.5 FST values of pairwise comparison among three maternal selective breeding generations of tilapia “Yuemin No.1” and four geographical populations of Nile tilapia Oreochromis niloticus
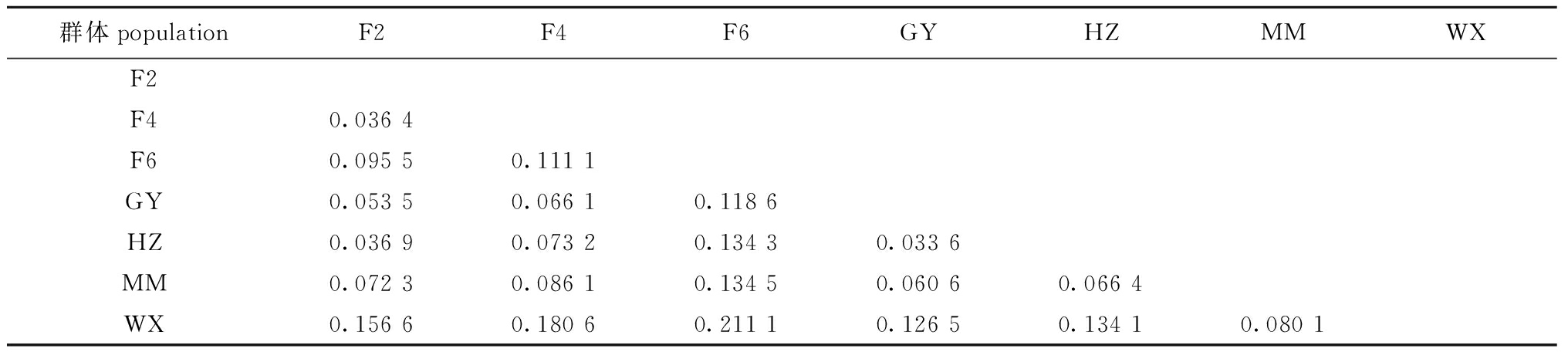
群体populationF2F4F6GYHZMMWXF2F40.036 4F60.095 50.111 1GY0.053 50.066 10.118 6HZ0.036 90.073 20.134 30.033 6MM0.072 30.086 10.134 50.060 60.066 4WX0.156 60.180 60.211 10.126 50.134 10.080 1
表6 尼罗罗非鱼7个群体间的分子方差分析
Tab.6 AMOVA analysis of three maternal selective breeding generations of tilapia “Yuemin No.1” and four geographical populations of Nile tilapia Oreochromis niloticus
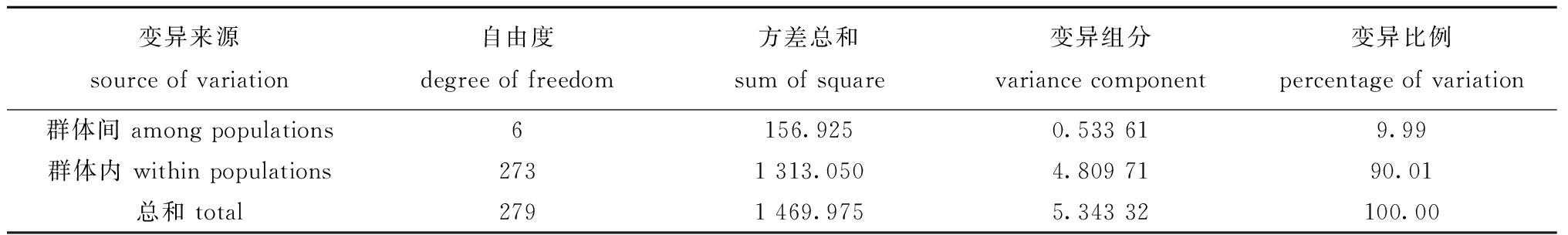
变异来源 source of variation自由度 degree of freedom方差总和 sum of square变异组分 variance component变异比例 percentage of variation群体间 among populations 6156.9250.533 619.99群体内 within populations2731 313.0504.809 7190.01总和 total2791 469.9755.343 32100.00
3 讨论
3.1 不同选育世代的遗传多样性分析
本研究中采用13对荧光标记微卫星引物和多重毛细管电泳方法对罗非鱼“粤闽 1号”母本3个选育世代和4个尼罗罗非鱼地理群体进行遗传多样性和遗传结构分析,与传统的聚丙烯酰胺凝胶电泳微卫星基因分型方法相比,荧光标记多重毛细管电泳具有所需样品量低、分辨率高、重复性好、准确度高、通量高等优点,可大大提高等位基因分型效率[13]。此外,Barker[14]认为具有4个或4个以上等位基因的微卫星位点才能较好地用于物种遗传多样性分析。本研究中采用的13对微卫星引物在7个尼罗罗非鱼群体中共扩增出176个等位基因,平均等位基因数为13.538 5,平均有效等位基因数为6.220 7,平均多态信息含量为0.789 9,这说明13个微卫星位点的多态性较高,可用于尼罗罗非鱼群体的遗传参数分析。本研究结果还表明,罗非鱼“粤闽 1号”母本F2、F4和F6 3个选育世代的等位基因数、有效等位基因数、期望杂合度、Shannon多样性指数和多态性信息含量等遗传多样性参数均呈现出随着选育世代增加而降低的趋势,这说明人工选育导致该选育群体的遗传变异有所下降,逐步趋向纯化,符合选育预期。在“新吉富”罗非鱼[5]、罗氏沼虾Macrobrachium rosenbergii[15]和翘嘴鳜Siniperca chuatsi[16]等多种鱼类选育群体中也有类似报道,选育群体遗传多样性下降可能与人工选育过程中基因逐渐趋于纯合,以及低频等位基因的丢失有关。Botstein等[17]认为,当PIC>0.5时为高度多态性,当PIC为0.25~0.5时为中度多态性,当PIC<0.25时为低度多态性,由此可知,虽然本研究中选育群体(F2、F4和F6)的遗传变异随着选育世代的进行有所下降,但仍为高度多态性(PIC>0.5),具有进一步选育的潜力。为了避免选育过程中近亲繁殖和“瓶颈”效应导致遗传多样性进一步下降,需要保种足够数量的有效亲本,也可通过引进遗传多样性较高的外群尼罗罗非鱼个体进行杂交而得到改善。本研究中对4个尼罗罗非鱼地理群体的遗传多样性分析结果表明,GY、MM和HZ群体的遗传多样性显著高于罗非鱼“粤闽 1号”母本的选育群体,可以作为潜在的候选引种群体。
3.2 不同选育世代的遗传分化
遗传距离和遗传相似度是衡量群体间遗传分化程度的重要指标。根据Thorpe[18]提出的理论,同物种群体间的遗传距离在0.03~0.2之间,遗传相似度在0.8~0.97之间。本研究结果表明,罗非鱼“粤闽 1号”选育世代群体F2与F4之间遗传距离为0.155 6,遗传相似度为0.855 9,符合该理论;但F2与F6之间的遗传距离增大至0.351 8,遗传相似度为0.703 5,F4与F6之间的遗传距离为0.375 7,遗传相似度为0.686 8,均超出了该理论界定的同物种群体间遗传距离和遗传相似度范围。这说明人工选育对选育群体的遗传结构产生了较大影响,随着选育世代的进行,世代间遗传距离逐渐增大,F2与F4亲缘关系较近,为同种群体,而F6已经演变成一个独立的亚种群体。根据 Wright[12]对遗传分化系数的界定,本研究中F2与F4之间存在微弱遗传分化(FST <0.05),而F2和F4二者与F6之间均存在中等遗传分化(0.05<FST <0.15),这与对遗传距离的分析结果相吻合。在UPGMA系统进化树中,MM群体与WX群体聚为一大支,F2、F4和F6 3个选育世代与HZ群体、GY群体聚为另一大支,这说明罗非鱼“粤闽 1号”母本的3个选育世代群体与HZ群体和GY群体遗传背景相近,可能具有共同的遗传来源。
3.3 选育群体遗传基础的拓宽
选择育种往往需要连续多代选育才能获得可稳定遗传的优良性状,但随着选育代数增加,群体遗传多样性往往会不断下降,影响进一步选育的效果。适当拓宽选育群体的遗传基础可为其选育工作的持续进行提供有力保障。Melchinger等[19]研究表明,在遗传距离小于0.54的范围内,亲本间的遗传距离越大,其杂交子代的基因型杂合度越高,杂种优势越强。本研究结果表明,GY、HZ、MM和WX 4个尼罗罗非鱼地理群体与选育世代群体F6的遗传距离分别为0.526 4、0.582 9、0.624 4和0.979 4,只有GY群体与F6的遗传距离小于0.54,而且该地理群体的遗传多样性最高,因此,其最适合作为潜在的引种群体与罗非鱼“粤闽1号”母本选育群体进行杂交,从而拓宽其遗传基础,提高选育群体的遗传多样性和选育潜能。
4 结论
1) 罗非鱼“粤闽1号”母本的选育群体在选育过程中遗传多样性随选育世代进行不断下降,遗传分化不断加剧,F6世代与F2、F4之间存在中等遗传分化,选育群体仍然保持着较高的遗传多样性,具有进一步选育的潜力。
2) 4个尼罗罗非鱼地理群体中GY群体的遗传多样性最高,与F6群体的遗传距离适中,可作为潜在的引种群体与罗非鱼“粤闽1号”母本选育群体进行杂交,用于提高选育群体的遗传多样性。
[1] 农业农村部渔业渔政管理局,全国水产技术推广总站,中国水产学会.2020中国渔业统计年鉴[M].北京:中国农业出版社,2020:21-36.
[2] 刘志刚,卢迈新,曹建萌,等.罗非鱼“粤闽1号”及其繁育群体的遗传多样性和遗传关系分析[J].渔业科学进展,2018,39(6):31-41.
[3] 于爱清,施永海,邓平平.长江刀鲚选育和野生群体遗传多样性的微卫星分析[J].水产科技情报,2019,46(3):121-125.
[4] 樊佳佳,白俊杰,李胜杰,等.驯食配合饲料的大口黑鲈3个选育世代的遗传多样性分析[J].渔业科学进展,2019,40(4):57-64.
[5] 唐首杰,李思发,赵金良.“新吉富”罗非鱼选育后期世代遗传潜力的微卫星分析[J].海洋湖沼通报,2018(3):120-131.
[6] 马冬梅,苏换换,朱华平,等.华南鲤选育群体不同世代遗传多样性与遗传结构的微卫星分析[J].水生生物学报,2018,42(5):887-895.
[7] 王沈同,沈玉帮,孟新展,等.草鱼野生与选育群体遗传变异微卫星分析[J].水产学报,2018,42(8):1273-1284.
[8] 刘永新,刘奕,周勤,等.利用微卫星标记指导红鳍东方鲀亲本选配[J].大连海洋大学学报,2015,30(2):113-119.
[9] TIAN C,GUO W J,LIANG X F,et al.Identification of species-specific microsatellite markers in three Siniperca species by RNA-Seq[J].Biochemical Systematics and Ecology,2017,70:126-131.
[10] HERN NDEZ S,DUFFY C,FRANCIS M P,et al.Evidence for multiple paternity in the school shark Galeorhinus galeus found in New Zealand waters[J].Journal of Fish Biology,2014,85(5):1739-1745.
NDEZ S,DUFFY C,FRANCIS M P,et al.Evidence for multiple paternity in the school shark Galeorhinus galeus found in New Zealand waters[J].Journal of Fish Biology,2014,85(5):1739-1745.
[11] LEE B Y,LEE W J,STREELMAN J T,et al.A second-generation genetic linkage map of tilapia (Oreochromis spp.)[J].Genetics,2005,170(1):237-244.
[12] WRIGHT S.Variability within and Among natural populations[M].Chicago:University of Chicago Press,1978.
[13] CAWKWELL L,BELL S M,LEWIS F A,et al.Rapid detection of allele loss in colorectal tumours using microsatellites and fluorescent DNA technology[J].British Journal of Cancer,1993,67(6):1262-1267.
[14] BARKER J S F.A global protocol for determining genetic distances among domestic livestock breeds[C]//Proceedings of the 5th world congress on genetics applied to livestock production.Guelph:University of Guelph,1994,21:501-508.
[15] 董丁健,戴习林.罗氏沼虾不同群体世代遗传多样性的SSR分析[J].南方农业学报,2020,51(2):421-428.
[16] 郑荷子,易提林,梁旭方,等.翘嘴鳜连续4代选育群体遗传多样性及遗传结构分析[J].淡水渔业,2013,43(6):8-12.
[17] BOTSTEIN D,WHITE R L,SKOLNICK M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[18] THORPE J P.The molecular clock hypothesis:biochemical evolution,genetic differentiation and systematics[J].Annual Review of Ecology and Systematics,1982,13(1):139-168.
[19] MELCHINGER A E,LEE M,LAMKEY K R,et al.Genetic diversity for restriction fragment length polymorphisms:relation to estimated genetic effects in maize inbreds[J].Crop Science,1990,30(5):1033-1040.