鲤春病毒血症(Spring Virernia of Carp,SVC)是一种高致病性、高传染性的鲤科鱼类病毒性疾病。在自然条件下,可感染锦鲤Cyprinus carpio、草鱼Ctenopharyngodm idellus、银鱼Hypophthalmichthys molitrix、胖头鱼Aristichthys obilis、鲫Carassius auratus、金鱼C.auratus、拟鲤Rutilus rutilus、圆腹雅罗鱼Leuciscus idus、丁鲷Tinca tinca和六须鲇Silurus glanis[1-2]。该病最早流行于中世纪欧洲大部分地区。1998年英国环境、渔业和水产养殖科学研究中心从中国对英国出口的锦鲤中检测出SVCV之前,中国无该病毒流行的报道。中国内地对于该病的首次报道是在2003年Liu等[3]从天津某观赏鱼养殖场分离鉴定出SVCV。
SVCV是一种有类脂囊膜的弹状病毒,长为90~180 nm,宽为60~90 nm,隶属单股负链RNA病毒目弹状病毒科Rhabdoriridae水泡性病毒属Vesiculorius[1]。SVCV的基因组全长约为11 000 bp,共包含5个主要的开放阅读框(Open Reading Frame, ORFs),分别编码5种结构蛋白,即核蛋白(Nuclear protein,N)、磷蛋白(Phosphoprotein,P)、基质蛋白(Matrix protein,M)、糖蛋白(Glycoprotein,G)和RNA聚合酶(RNA polymerase,L),各结构蛋白之间的连接顺序为3′-N-P-M-G-L-5′[4]。目前GenBank中共收录16株SVCV的全基因组序列,其中11株来自中国。Stone等[5]于2003年对来自不同国家的15株SVCV分离株部分G蛋白的基因序列(550 bp)进行了系统发育分析,将世界范围内的SVCV分成Ⅰa、Ⅰb、Ⅰc和Ⅰd 4个基因型。此后,编码G蛋白的基因序列成为划分SVCV基因型的主要参考基因序列。目前,国内有3株SVCV分离株(SVCV-A1、SVCV-265、BJ0505-2)完成了全基因组序列生物学分析工作[6-8]。
本课题组在水生动物疫病监测期间,获得一株中国北方地区现行毒株SVCV-shlj4[9]。为了更好地了解该毒株的分子特征,本研究中通过分段克隆、测序、拼接,获得了该毒株的全基因组序列,结合生物信息学研究方法对该基因组序列及其编码M蛋白、G蛋白、P蛋白的基因序列进行了系统发育和基因型分析,旨在明确中国北方地区SVCV分离株与国内乃至世界各地SVCV毒株间的进化关系,为揭示SVCV分子进化机制提供参考。
1 材料与方法
1.1 材料
鲤上皮瘤细胞(Epithelioma papulosum cyprini,EPC)由中国水产科学研究院长江水产研究所曾令兵研究员惠赠;SVCV-shlj4毒株由本课题组在2016年黑龙江地区养殖鲤流行病调查中分离保存。
大肠杆菌(E.coli)DH5α、pMD18-T克隆载体、PCR纯化试剂盒、RACE试剂盒购自宝生物工程(大连)有限公司;Easy-A High Fidelity Cloning Enzyme购自Stratagene公司;MEM(Minimum essential medium)细胞培养基、胰酶购自Hyclone生物公司;胎牛血清(Fetal bovine serum, FBS)购自Gibco生物公司。
1.2 方法
1.2.1 引物设计 根据GenBank公布的SVCV-A2基因组序列(NCBI登录号:DQ491000.1),利用Primer Premier 5.1软件设计特异性引物。将SVCV-shlj4毒株全基因组编码区分成3个部分,分别命名为1st、2nd、3rd,为保证扩增的准确性,相邻的扩增片段之间含有部分重叠区域。3对重叠引物分别为
1st F:5′ATGAGTGTCATTCGGATCAAAACAA 3′,
1st R:5′TTCAGCTGCATGGCAGATCCATCCA 3′;
2nd F:5′TACACGGAAATCTACCTAACACA 3′,
2nd R:5′TCATCATTGTTGATGATTAATCCTAG 3′;
3rd F:5′ACACAGTTGTAAAAGTGTTGGCT 3′,
3rd R:5′CTATTCTACCCATGTCCCAGAGT 3′。
根据已获得SVCV-shlj4毒株的CDS序列信息,利用RACE试剂盒,设计合成用于扩增5′末端和3′末端非编码区的上、下游引物,分别为
5′RACE:GTTAGCCGGAAGCACGGCAGCAACT,
3′RACE:TCGGTACCAACAGTGTCTTCCGCTC。
以上引物由长春库美生物公司合成。
1.2.2 PCR产物的克隆及测序 采用Trizol法对SVCV-shlj4毒株进行总RNA的提取[10-11]。以提取的总RNA作为模板,利用Easy-A高保真DNA聚合酶,对SVCV株 3段基因序列进行RT-PCR的扩增及3′末端加A处理。利用PCR纯化试剂盒对扩增获得的PCR产物回收。将回收后的产物与pMD18-T载体连接,转化至E.coli DH5α中,在含有氨苄青霉素抗性的LB平板中培养,直至长出单菌落。每个连接选择3个阳性克隆并送至库美公司测序,以得到SVCV-shlj4毒株的编码区基因序列。利用RACE试剂盒,结合已获得的编码区基因序列,继续扩增SVCV-shlj4分离株5′末端和3′末端非编码区,对其进行回收、连接、转化、测序,操作方法同上。
1.2.3 SVCV-shlj4毒株系统发育分析 利用DNAsis软件对该毒株的3段基因序列及5′末端和3′末端非编码区的测序结果进行拼接,结合Lasergene(DNAStar,Inc.,USA)、Clustal W及MEGA 5.1生物信息学软件,对SVCV-shlj4毒株基因组及GenBank中检索得到的其他16株SVCV基因组序列进行同源性分析并绘制系统发生树。同时,利用MEGA 5.1软件和Lasergene对SVCV-shlj4分离株及其他SVCV参考毒株编码的M蛋白、P蛋白、G蛋白基因序列进行多重对比,采用Neighbor-Joining法(1000 runs)构建系统进化树。
2 结果与分析
2.1 SVCV-shlj4毒株基因组特征
SVCV-shlj4毒株基因组全长为11 029 bp,已提交至GenBank( NCBI登录号:MT675953)。该基因组共由5个开放阅读框构成,从3′端到5′端共编码5 个结构蛋白,各结构蛋白之间的连接顺序为 3′-N-P-M-G-L-5′(表1)。N基因全长为1335 bp,开放阅读框长度为1257 bp,编码蛋白418 aa,蛋白相对分子量约为47 000;P基因全长为967 bp,开放阅读框长度为930 bp,编码蛋白309 aa,蛋白相对分子量约为36 000;M基因全长为716 bp,开放阅读框长度为672 bp,编码蛋白223 aa,蛋白相对分子量约为26 000;G基因全长为1598 bp,开放阅读框长度为1530 bp,编码蛋白约509 aa,蛋白相对分子量约为57 000;L基因全长为6325 bp,开放阅读框长度为6288 bp,编码蛋白2095 aa,蛋白相对分子量约为121 000。
表1 开放阅读框信息
Tab.1 Information on the open reading frame
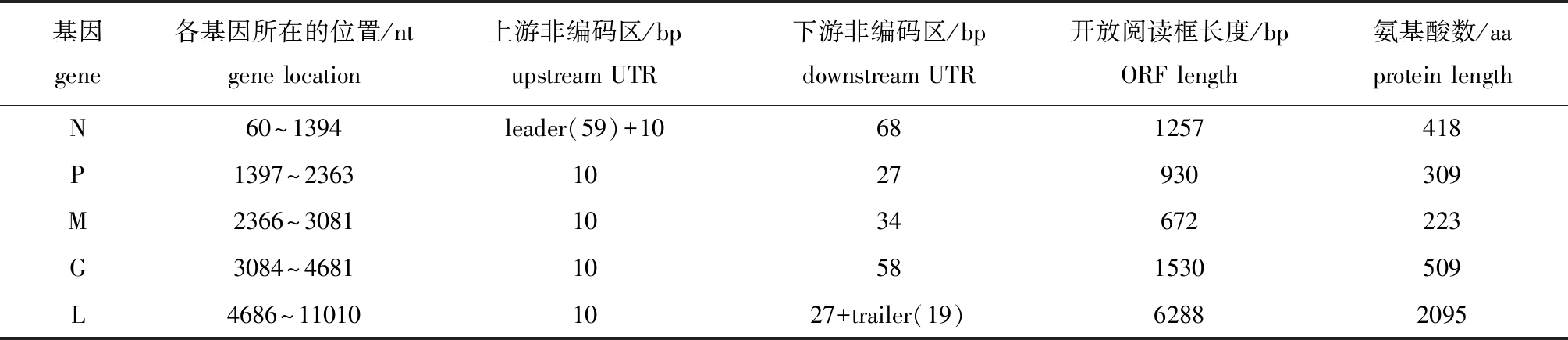
基因gene各基因所在的位置/ntgene location上游非编码区/bpupstream UTR下游非编码区/bpdownstream UTR开放阅读框长度/bpORF length 氨基酸数/aaprotein lengthN60~1394leader(59)+10681257418P1397~23631027930309M2366~30811034672223G3084~468110581530509L4686~110101027+trailer(19)62882095
SVCV-shlj4毒株的N基因、M基因、G基因、L基因均由AACAGACATC(ATG)作为转录起始信号,而P基因的转录起始信号序列为AACAGAGATC(ATG),有一个碱基发生突变,各基因序列均以TATG(A)7作为转录终止信号。此外,毒株各基因的连接区高度保守,其中N基因、P基因、M基因及G基因之间的连接区均为CT,G基因和L基因之间的连接区为CTAT。SVCV-shlj4毒株基因组的3′ 端非编码区(1 nt~8 nt)与5′ 端非编码区(11029 nt~11022 nt)存在8对反向互补序列(ACGAAGAC)(图1)。

图1 SVCV-shlj4毒株基因组特征
Fig.1 Features of SVCV-shlj4 strain genome
2.2 SVCV基因组序列的系统发育分析
使用MEGA 5.1软件,采用邻位相连法(Neighbor-Joining)通过自举分析(Bootstrap 1000 runs)构建包括SVCV-shlj4在内的17株SVCV全基因组的系统发育进化树,各参考毒株具体的信息如表2所示。进化分析结果显示:SVCV-shlj4毒株与中国内地株(10株)、中国香港株(1株)及韩国株(3株)聚为一簇,同属亚洲分支,一致性在97.1%~99.0%,与SVCV-A2毒株一致性最高(99.0%),与SVCV-202238毒株一致性较低(97.1%);Fijan株和VR-1390株属于欧洲分支,SVCV-shlj4株与二者在进化上有明显差异,一致性最低(93.0%)(图2)。
表2 本研究所用SVCV基因组序列
Tab.2 The SVCV genome sequence used in this study

编号No.分离株isolateGenBank登录号GenBank accession No.宿主host来源country一致性/%identity全长/bpnucleotide1A1DQ097384.2—China98.0111002A2DQ491000.1—China99.0109903BJ0505-2EU177782.1—China98.7110474SVCV-265KJ513477.1carpChina98.3110295SH140501KR012465.1goldfishChina97.4110296SH140502KR012466.1goldfishChina97.9110297SH140503KR012467.1goldfishChina97.8110298SH140522KR012468.1common carpChina97.6110299SH150514KT321307.1goldfishChina97.71102910SH160901KY475636.1grass carpChina97.61102911Fijan reference strainAJ318079.1carpYugoslavia93.01101912ATCCVR-1390NC_002803.1(U18101.2)Cyprinus carpioYugoslavia93.01101913202238KU230365.1Cynops orientalis (Chinese fire belly newt)Hong Kong97.11099114ADC-SVC2016-1MG663514.1Cyprinus carpio (common carp)South Korea97.61103415ADC-SVC2016-3MG663513.1Micropterus salmoides (largemouth bass)South Korea97.61103416ADC-SVC2016-5MG663512.1Cyprinus carpio nudus (leather carp)South Korea98.011029

图2 SVCV分离株基因组序列系统发育分析
Fig.2 Phylogenetic analysis of complete genomic sequence of SVCV isolates
2.3 编码M、G、P蛋白的基因系统发育分析
利用BLAST检索(https://blast.ncbi.nlm. nih.gov/Blast.cgi)获得其他SVCV参考株编码M、G、P蛋白的基因序列,进行系统发育进化分析。结果显示:根据编码M蛋白的核苷酸序列可将SVCV分为两大分支,亚洲分支和欧洲分支,其中亚洲分支包括中国所有毒株、韩国株ADC-SVC2016-1和美国株(207914、266921、316715、212364及322383)(图3);根据编码G蛋白的核苷酸序列可将SVCV分为4个基因型,分别为Ⅰa、Ⅰb、Ⅰc和Ⅰd,其中,SVCV-shlj4毒株的基因型为Ⅰa,与中国株SVCV-265可能来源于同一祖先,聚为一簇(图4);根据编码P蛋白的核苷酸序列可进一步将Ⅰa基因型划分成Ⅰai和Ⅰaii两个基因亚型,SVCV-shlj4株与中国株BJ0505-2、SVCV-A1、SVCV-A2及美国株OM-24聚为一簇,均为Ⅰaii基因亚型,其中与SVCV-A2株的一致性最高(99.0%)(图5)。
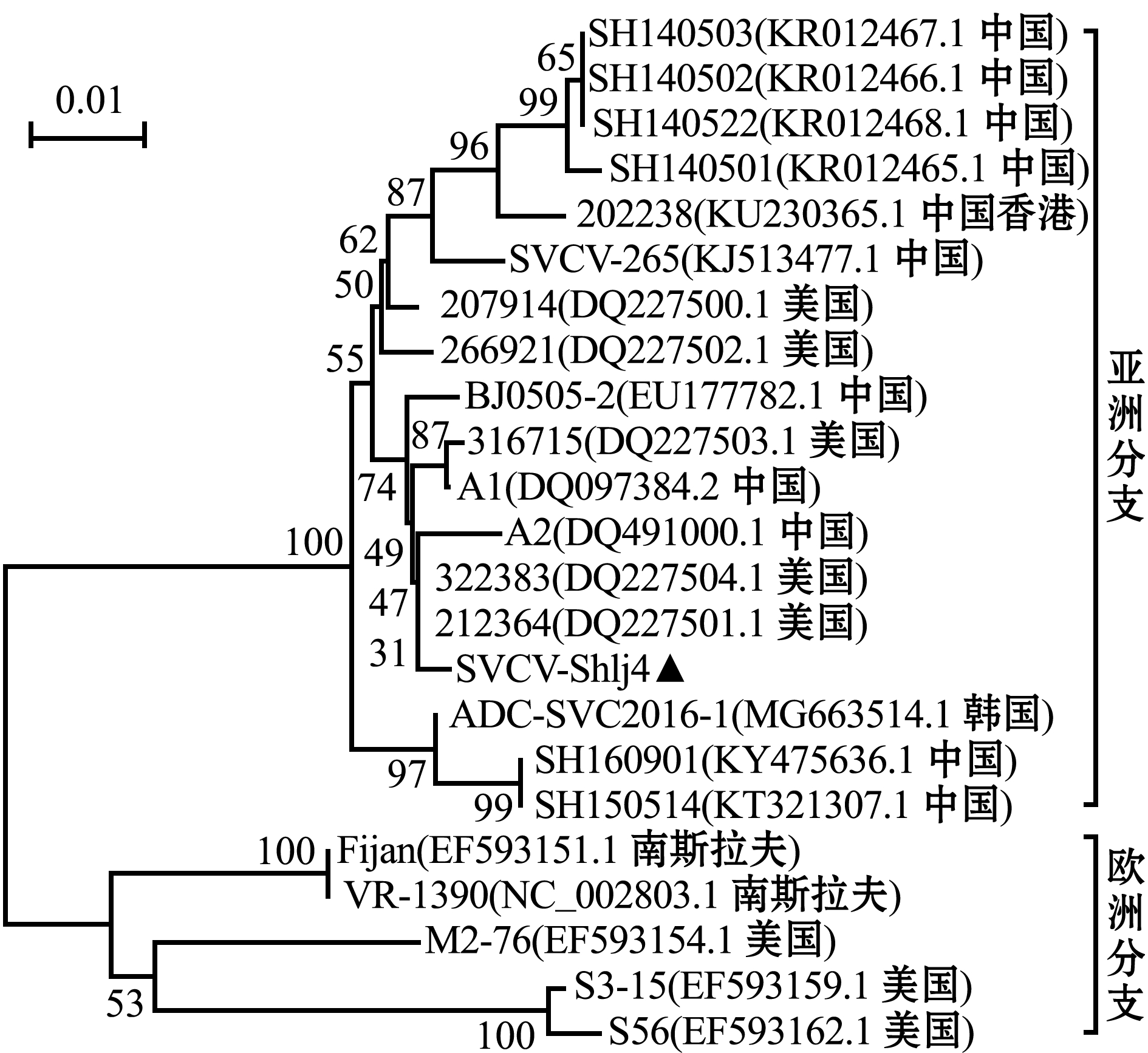
图3 编码M蛋白的SVCV基因系统发育树
Fig.3 Phylogenetic tree of SVCV gene encoding M protein

图4 编码G蛋白的SVCV基因系统发育树
Fig.4 Phylogenetic tree of SVCV gene encoding G protein
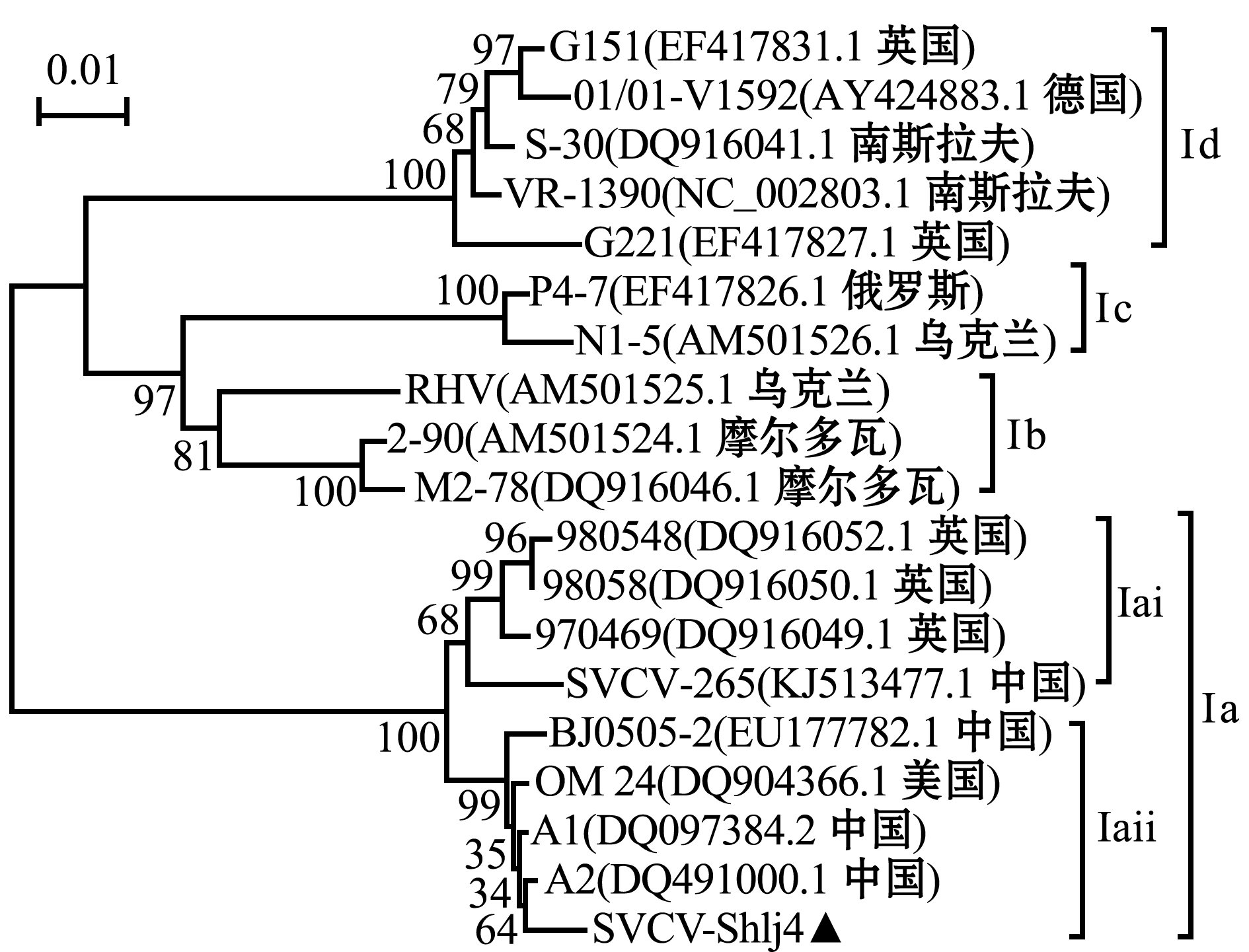
图5 编码P蛋白的SVCV基因系统发育树
Fig.5 Phylogenetic tree of SVCV gene encoding P protein
2.4 各基因序列变异位点分析
SVCV-shlj4毒株各基因序列变异位点的分析结果显示(图6):与中国株SVCV-A1相比,SVCV-shlj4毒株在N基因编码区第1357、1364和1376 nt分别缺失1个碱基,在G基因3′端非编码区4630 nt处和4639 nt处缺失两段基因序列(分别为44 bp和31 bp);与欧洲株VR-1390相比,SVCV-shlj4毒株在G基因3′端非编码区(4637 nt位点)插入一段10 bp的基因片段;与中国株BJ0505-2相比,SVCV-shlj4毒株在L基因编码区(5822 nt位点)缺失一段18 bp的基因片段,该基因缺失导致L基因编码区缺失6个氨基酸(序列为KSLANA)(图7)。
3 讨论
SVC首次被报道是在20世纪70年代,Fijan[12]从患病的鱼体中分离得到SVCV。该病毒严重威胁到欧洲鲤鱼养殖业的发展,并对其造成了巨大的经济损失。中国对于SVCV的报道是在2002—2004年间,刘荭等[3,13]在对全国范围内SVCV监控过程中,首次分离报道了该病毒。近年来,该病在中国虽然没有大规模的暴发,但在多个地区均有关于SVCV分离鉴定的报道,这应当引起国内鲤科鱼类养殖业的高度重视。

注:以SVCV-Sh1j4毒株基因序列作为参比
Note:The gene sequence of the SVCV-shlj4 is used as a reference
图6 各基因序列变异位点分析
Fig.6 Analysis of variation in open reading frame
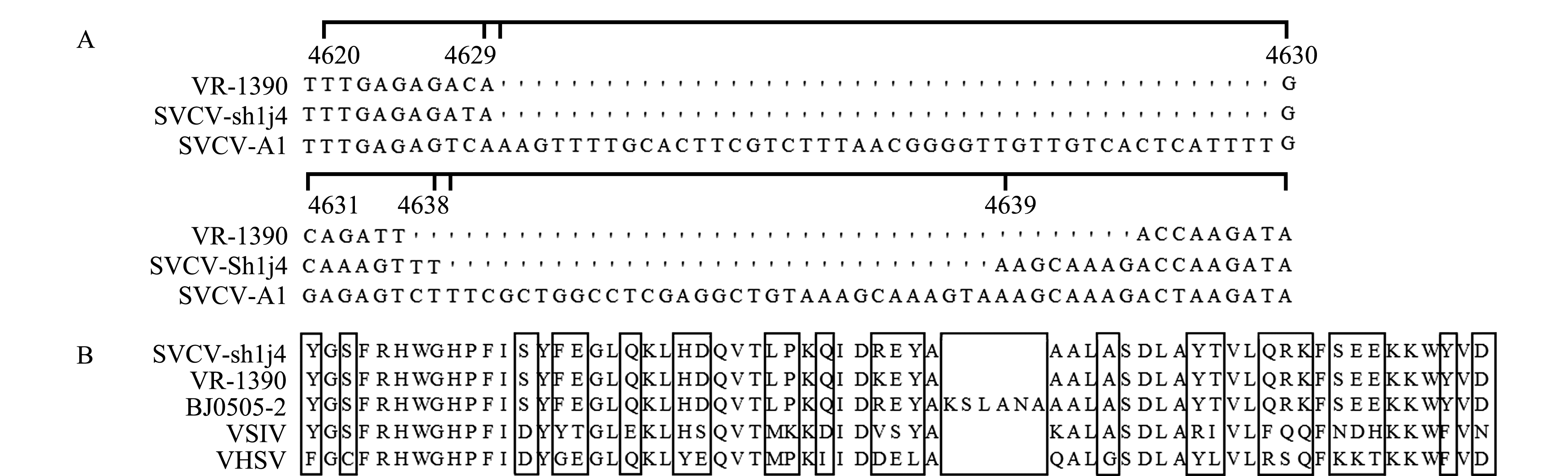
注:A为G蛋白基因序列变异位点;B为L蛋白氨基酸序列变异位点
Note: A, the variation sites of nucleic acid in glycoprotein gene sequence in G protein; B, the variation sites of amino acid sequences in L protein
图7 SVCV-shlj4毒株的部分基因序列及其编码的氨基酸序列变异位点
Fig.7 Variation sites of nucleic acid and amino acid sequences among partial gene sequences in SVCV-shlj4 strain
3.1 参考毒株的选择及基因组结构分析
由GenBank SVCV分离株的收录情况可知,迄今为止,国际上共完成了16株SVCV毒株基因组序列的测定工作,其中11株为中国株。本课题组在水生动物疫病监测期间,分离获得了一株中国北方地区现行毒株SVCV-shlj4。根据OIE检测标准对该病毒进行鉴定,RT-PCR产物测序结果对比显示,SVCV-shlj4与中国株SVCV-A2的核酸一致性最高。因此,本研究中选用SVCV-A2作为参考毒株设计引物,以获得SVCV-shlj4全基因组序列。测序结果显示,该毒株的基因组大小为11 029 bp,共编码5种结构蛋白,各基因之间的连接区高度保守,N基因、P基因、M基因及G基因连接区均为CT,G基因与L基因连接区为CTAT,这些基因间连接区与其他水泡病毒属成员一致,区别于弹状病毒科的其他属成员[14]。同时,SVCV-shlj4毒株基因组的3′ UTR与5′ UTR存在8对反向互补序列,这是弹状病毒基因组和不分段负链RNA病毒的共同特征[15]。
3.2 全基因组及编码M、G、P蛋白的基因系统发育分析
在得到SVCV-shlj4毒株全基因组序列后,将其与其他16株SVCV参考株全基因组序列进行系统发育分析,结果显示,SVCV基因组序列相对稳定,但不同地区的毒株存在一定的差异,一致性为93.0%~99.0%。通过对比SVCV参考株编码M、G、P蛋白的基因序列,构建系统发育进化树,可以发现,编码M蛋白的核苷酸序列可将SVCV分为两大分支,亚洲或亚洲原始分支及欧洲分支[16]。亚洲SVCV分离株(包括韩国株和中国株)均处于同一分支,各毒株间核苷酸一致性较高,亚洲原始株包括美国株207914、266921、316715、212364、322383[16],其中SVCV-shlj4株与美国株322383的一致性最高(99.7%),说明二者可能来源于同一祖先。根据编码G蛋白的部分核苷酸序列可将SVCV分为4种基因型,分别为Ⅰa、Ⅰb、Ⅰc、Ⅰd,其中,Ⅰa基因型包括亚洲株和亚洲原始株,Ⅰb和Ⅰc基因型包括来自摩尔多瓦、乌克兰和俄罗斯等国家的分离株,Ⅰd基因型包含来自英国和其他欧洲国家的分离株。目前,中国SVCV分离株均为Ⅰa基因型[3]。由于编码P蛋白的基因系统发育树与编码G蛋白的基因拓扑结构相似[6],可将Ⅰa基因型进一步分为Ⅰai和Ⅰaii两个基因亚型,SVCV-shlj4与中国株A1、A2、BJ0505-2及美国株OM-24聚为一簇,均为Ⅰaii基因亚型,印证了之前关于美国毒株由亚洲传入的假说[8,17]。
3.3 各基因变异位点分析
在对比分析各个参考毒株和SVCV-shlj4毒株各基因核苷酸变异位点时发现,SVCV-shlj4毒株与中国株SVCV-A1、BJ0505-2、欧洲株VR-1390在N基因、G基因及L基因存在明显差异。其中,SVCV-shlj4毒株与中国株SVCV-A1差异性主要存在于N蛋白的编码区和G蛋白的3′端非编码区;与中国株BJ0505-2的差异性主要存在于L蛋白的编码区;与欧洲株VR-1390株的差异性主要存在于G蛋白的3′端非编码区。这些差异性可能是导致各个地区毒株毒力强弱的主要原因之一。
综上所述,SVCV-shlj4毒株在进化上属于亚洲分支,属Ⅰa基因型,Ⅰaii基因亚型。虽然当前国内SVCV基因型均为Ⅰa,但来源于不同地区SVCV分离株的核酸序列依然存在一定差异,SVCV毒株在中国不同地区不同环境中正在不断地进化。本研究结果可为SVCV分子进化研究提供数据支撑。
[1] Fijan N.Vaccination of fish in European pond culture:prospects and constraints[J].Symposia Biologica Hungarica,1984,23:233-241.
[2] Shchelkunov I S,Shchelkunova T I.Rhabdovirus carpio in herpivorous fishes:isolation,pathology,and comparative susceptibility of fishes[M]//Ahne W,Kurstak E.Viruses of Lower Vertebrates.Berlin,Heidelberg:Springer,1989:333-348.
[3] Liu H,Gao L,Shi X,et al.Isolation of spring viraemia of carp virus (SVCV) from cultured koi (Cyprinus carpio Koi) and common carp (C.carpio carpio) in P.R.China[J].Bulletin of the European Association of Fish Pathologists,2004,24(4):194-202.
[4] 徐耀先,周晓峰,刘立德.分子病毒学[M].武汉:湖北科学技术出版社,2000.
[5] Stone D M,Ahne W,Denham K L,et al.Nucleotide sequence analysis of the glycoprotein gene of putative spring viraemia of carp virus and pike fry rhabdovirus isolates reveals four genogroups[J].Diseases of Aquatic Organisms,2003,53(3):203-210.
[6] Xiao Yu,Shao Ling,Zhang Chongwen,et al.Genomic evidence of homologous recombination in spring viremia of carp virus:a negatively single stranded RNA virus[J].Virus Research,2014,189:271-279.
[7] Teng Y,Liu H,Lv J Q,et al.Characterization of complete genome sequence of the spring viremia of carp virus isolated from common carp (Cyprinus carpio) in China[J].Archives of Virology,2007,152(8):1457-1465.
[8] Zhang Nianzhi,Zhang Lifeng,Jiang Yinan,et al.Molecular analysis of spring viraemia of carp virus in China:a fatal aquatic viral disease that might spread in East Asian[J].PLoS One,2009,4(7):e6337.
[9] 纪锋,赵景壮,刘淼,等.黑龙江地区鲤春病毒血症病毒的分离与基因型分析[J].中国水产科学,2017,24(5):1141-1148.
[10] 徐黎明,刘红柏,尹家胜,等.传染性造血器官坏死病毒糖蛋白原核表达及免疫原性分析[J].病毒学报,2013,29(5):529-534.
[11] 纪锋,徐黎明,赵景壮,等.鲤春病毒血症病毒Shlj1株糖蛋白空间结构及其B细胞抗原表位的预测[J].大连海洋大学学报,2017,32(4):440-446.
[12] Fijan N.Spring viraemia of carp and other viral diseases and agents of warm-water fish[M]//Woo P T K,Druno D W.Fish Diseases and Disorders.Oxon:CAB International,1999:177-244.
[13] 刘荭,付峰,黄倢,等.鲤春血症病毒中国分离株糖蛋白基因和氨基酸序列的初步解析[J].中国病毒学,2005,20(6):647-651.
[14] Björklund H V,Higman K H,Kurath G.The glycoprotein genes and gene junctions of the fish rhabdoviruses spring viremia of carp virus and hirame rhabdovirus:analysis of relationships with other rhabdoviruses[J].Virus Research,1996,42(1-2):65-80.
[15] Hoffmann B,Schütze H,Mettenleiter T C.Determination of the complete genomic sequence and analysis of the gene products of the virus of spring viremia of carp,a fish rhabdovirus[J].Virus Research,2002,84(1-2):89-100.
[16] Warg J V,Dikkeboom A L,Goodwin A E,et al.Comparison of multiple genes of spring viremia of carp viruses isolated in the United States[J].Virus Genes,2007,35(1):87-95.
[17] Miller O,Fuller F J,Gebreyes W A,et al.Phylogenetic analysis of spring virema of carp virus reveals distinct subgroups with common origins for recent isolates in North America and the UK[J].Diseases of Aquatic Organisms,2007,76(3):193-204.